摘要
背景
纯合子植株的选择是新转基因植株评价的一个关键环节。在这里,我们描述了一种高效和高度灵活的基于实时pcr的方法,用于在具有复杂(多个)基因组和/或相对较长生成时间(>3个月)的植物模型中开发纯合子系,使用直接拷贝数测定。
结果
一种现有的DNA提取方法被转化为一种高通量的植物叶片DNA提取程序,得到适合实时PCR分析的DNA。针对面包小麦内参基因(Epsilon Cyclase)和用于谷物转化的基因盒中的标准序列元件(内含子桥和诺帕林合成酶终止子)开发了高度特异性和高效的引物对。实时聚合酶链反应(real-time PCR)技术可以可靠地将具有单一基因拷贝的小麦植株与具有多个基因拷贝的小麦植株以及缺乏该基因的小麦植株区分开来。为了尽可能有效地获得携带唯一插入事件的纯合子系,T0选择单拷贝的转基因植株(由转化的愈伤组织培养的植株),并筛选其后代为纯合子植株。最后,将该方法应用于水稻。
结论
在我们的案例研究中,实时聚合酶链反应(real-time PCR)检测技术提供了快速、容易和准确量化构建拷贝数的能力,极大地提高了纯合转基因植物选择的效率和可靠性。我们能够选择早期世代的纯合子植株,避免了大量分析后代分离模式和/或Southern blotting等耗时的方法。此外,专门开发携带独特插入事件的纯合子系的能力对于避免因共抑制而导致的基因沉默很重要,如果需要,还可以帮助选择适合未来解除调控的系。相同的引物对可以用来量化许多不同的小麦转基因事件,因为结构特异性引物对针对谷物基因盒的标准序列元素,使该方法广泛适用于小麦转基因研究。此外,由于这里描述的所有步骤都是标准化的,该方法可以很容易地适用于缺乏这里使用的目标区域的向量和/或其他植物模型。
背景
基因修饰,即在宿主基因组中插入构建的DNA,是提高作物作物生产力和/或产品质量的一种新兴的强大工具[1].然而,在转化过程中产生半合子植株(插入没有等位基因对应物的转基因),通过孟德尔分离,该结构可能在后代中丢失。因此,大多数遗传转化的下游应用都要求转基因在宿主基因组中是纯合的。对于模型,例如拟南芥具有简单的筛选选项,如标记选择[2]、较短的生成时间(<1个月)以及单拷贝数到低拷贝数的转换方法(通过根癌土壤杆菌),通过几代孟德尔分离研究,纯合子植物的选择可以在相当长的时间内实现[3.].然而,这种方法对于小麦、大麦、水稻和玉米等农业模型来说非常缓慢和繁琐。
迄今为止,大多数小麦转化都是通过粒子轰击实现的[4在许多情况下,该结构的多个副本被插入,要么在整个基因组中随机插入,要么在单个插入位点中插入(链接副本)。多个未连接的拷贝会导致复杂的分离模式,可能需要多代筛选来建立一个系的合子性。链接副本将作为一个单独的副本分离,因此在孟德尔分离研究中不会被注意到。然而,多个插入拷贝(链接或未链接)与转基因沉默问题有关[5],不适合考虑放宽管制的行,表明需要识别链接副本。最近,在效率方面取得了进展农包括小麦在内的谷物的介导转化(Sood等.[4),这可能会限制插入基因组的拷贝数量,从而减少(但不能避免)这些并发症。尽管如此,多拷贝问题以及大多数农业模型的生成时间较长(>3个月)强调了对孟德尔分离研究更有效的替代方法的需要,例如直接确定拷贝数。
直接量化DNA片段(基因、结构等)拷贝数的两种最流行的方法是Southern blot和real-time PCR(由Bubner和Baldwin审阅[2])。Southern blotting是最早的方法,通常用于确定DNA片段的拷贝数。它包括对酶消化的基因组DNA进行印迹,然后对与感兴趣的序列相对应的特定标记DNA探针进行杂交[6].尽管该技术功能强大且可靠,但这种有些繁琐的技术显示出一些重要的局限性。由于DNA改变和/或酶切位点缺失,Southern blot可能无法准确评估副本的确切数量,或无法区分位于紧密位置的副本(特别是链接副本)[2].此外,Southern印迹通常不会给出合子性信息,而这是在T中选择纯合子植株所需要的1的一代。使用这种技术,合子性的信息必须在T中收集2通过分析感兴趣的性状或额外的南方印迹的分离比率来生成。此外,Southern blotting要求每个结构都有一个特定的标记探针,这使得它不太适合筛选包含多个结构的样本集合。
实时PCR是基于检测PCR扩增过程中产生的荧光。这种荧光可以由与双链DNA结合时发出荧光的插层染料(如SYBR绿)产生,也可以由同时含有荧光团和引物之间结合的淬灭剂(如Taqman)的探针产生。在引物延伸过程中,探针被分解,从淬灭器中释放荧光团。反应的结果用C表示T值,表示荧光超过预先设置的阈值的周期号。比较CT方法被认为是最鲁棒的副本数确定方法[2].这种技术需要比较CT从感兴趣基因(GOI)到CT内源性内参基因的价值。这种方法的好处是不需要标准线,并且允许DNA浓度在样品之间有一定的变化,但关键是目标反应和参考反应都具有几乎相同的扩增效率。理论上,实时PCR分析将检测插入的转基因基因,无论它位于基因组的哪个位置。
Real-time PCR已用于测定小麦等植物的转基因拷贝数[7,8)、玉米(9)、大米(10,番茄11和甘蔗[12].然而,也有人担心实时PCR不够准确,无法可靠地确定合子性[7,13],特别是当由于需要筛选的植物数量众多而限制了技术复制的数量时。然而,我们认为,以前的结果发现实时PCR不可靠的拷贝数分析和/或合子度测定可能是由于次优优化程序可以克服。在这种情况下,real-time PCR在协助建立转基因纯合子系方面的能力可能没有得到充分利用,特别是在世代时间较长的植物中。
在这篇论文中,我们描述了一种基于实时PCR的方法,该方法具有所需的准确性和精密度,能够区分只有一个转基因拷贝的植物与纯合子植物和/或有多个插入的植物。我们展示了这些信息如何能最好地用于开发具有独特插入事件的转基因纯合子系。该方法经过专门设计,具有高度的通用性,所有程序都经过标准化和充分详细的描述,以促进其适应新的生物和/或载体。
方法
转基因植物
在第一个实验中,我们使用了来自自受精植物后代的DNA样本,这些自受精植物来自一个葡聚糖-水二激酶(GWD) RNAi转化的单插入纯合子小麦系之间的杂交(基于通过端点PCR观察到的5代孟德尔分离历史,如Ral所述)et al。[14)和野生型植物。该试验的目的是确定是否可以使用相对定量方法来区分单拷贝和纯合子植物(见方法-实时PCR拷贝数测定)。
在第二个实验中,我们跟踪了四种小麦植株,这些植株用RNAi盒式磁带进行了生物上的转化,时间长达四代(T0T3.),研究我们的方法在选择纯合子转基因株系中的有效性。
DNA隔离
DNA是从树叶样本中分离出来的,使用的是从Ellis那里获得的一种改进的方法et al。[15].约1厘米的幼叶样本被放入96x1 mL的母块(Greiner Bio-one)中,同时放入一个5毫米的玻璃珠(Sigma-Aldrich)。用密封垫(Thermo Scientific)密封块,并在−80°C下冷冻至少1小时。接下来,将冷冻块放入Retch MM300球磨机中,以最大频率(30/s)粉碎组织1分钟。每孔加入300 μL萃取缓冲液(0.1 M Tris-HCl pH8, 0.05 M EDTA, 1.25% SDS,预温至60℃),更换密封垫。将块放入定制的金属夹中以防漏出,然后在60°C的水浴中培养1-2小时,倒置进行定期混合。冰水冷却,加入150 μL 6 M醋酸铵,用PCR膜封片。将样品倒置6-8次混合,在冰水中孵育10分钟。然后在4000 g(4℃)离心20分钟后,将250 μL上清转移到新的母块中。加入冰镇2-丙醇150 μL, PCR膜封块,倒置搅拌6-8次,冰水中孵育10 min, 4000 g(4℃)离心20 min,倒上清,在薄纸上晾干。加入150 μL的70%乙醇,重复离心步骤,再次倒出上清,室温干燥5min。 Finally, the precipitated DNA was re-dissolved in 400 μL 0.05 M Tris pH9 overnight at 4°C. This method routinely yields 10–30 ng/μL genomic DNA for wheat.
端点PCR筛选
引物对GWDcontrol_for x GWDcontrol_rev和JP_bx17pro5 ' x IB_GWD3rev(表1)用于GWD RNAi转化植株的端点PCR筛选。PCR反应采用MyTaq HS (Bioline),包括:0.5 μL FP (10 μM), 0.5 μL RP (10 μM), 2 μL 5 × buffer, 5.9 μL H2O、0.07 μL MyTaq HS和1 μL DNA模板(约15 ng/μL)。协议遵循制造商的建议,在58°C退火。GWDcontrol引物对扩增内源性GWD基因,产生约980 bp (DNA质量控制)的PCR产物。当引物对存在时,尽管内源产物在大多数情况下仍然可见,但它优先从感觉序列和反感觉序列扩增568 bp的产物。第二个引物对是结构特异性的,针对Bx17启动子/GWD-sense过渡。
实时PCR引物开发
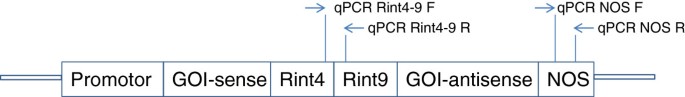
为了产生对我们课题组使用的广泛谷物结构有用的引物,我们选择了水稻内含子4和9(来自淀粉分支酶1基因,存在于我们所有谷物RNAi结构中)和诺paline合成酶(NOS)终止子(存在于我们所有谷物沉默和过表达结构中)之间的边界进行引物开发(图)1).对6对引物进行特异性和效率试验(每个区域3对)。必须采取标准措施以避免污染(例如,专门的PCR罩和单独的移液管)。
Epsilon Cyclase (EC),参与类胡萝卜素生物合成途径的基因[16]被选为小麦的参考基因,因为众所周知,它在每个基因组中只存在一次,而且基因组特异性序列可用。利用小麦的六倍性,验证了引物设计和筛选程序的有效性。开发和试验了两组3对引物,针对三个基因组之间相同的保守区域(EC ABD)和基因组a的特定区域(EC a)(图)2).这两个引物对之间1:3的比例表明我们的引物设计和选择过程产生了匹配效率的引物对。
所有引物均使用Primer3软件生成(http://frodo.wi.mit.edu/primer3/)使用默认值的以下修改:Product size = 50 - 150 bp;引物Tm = 59 - 65°C;产品Tm = 80 - 88°C;二价阳离子浓度= 3 mM, dNTPs浓度= 0.2 mM;热力学参数表= SantaLucia 1998;盐校正公式= 1998年桑塔卢西亚。
引物对的试验分为三步。首先,通过使用两个阳性对照和两个阴性对照(所有引物对使用相同的样本)确定引物对的特异性,选择良好的熔点曲线(预期温度附近的单峰),而无模板对照用于选择没有或只有低引物二聚体形成的引物对。如果有几个引物对合格,则通过选择最低C进行效率预选T值。用单拷贝GWD RNAi细胞系的DNA (40 ng DNA/μL - 0.1 ng DNA/μL,分3步稀释)对引物进行样品稀释。为了建立动态范围,当稀释系列的最高DNA浓度显示高于预期的CT值,一个被抑制反应的指标。当DNA浓度最低的DNA在重复之间出现较大的变异时,将被删除,这是实时PCR技术检测下限较低的一个指标。在必要时,丢弃两端反应后剩下的DNA浓度范围,构成动态范围,用于回归分析,计算R2和效率值。最后,采集检测样品进行测定,以确定测定的准确性和精密度。
实时PCR拷贝数检测
反应在MyIQ实时PCR仪(BioRAD)中进行。典型反应为:10 μl含荧光素的Sensimix SYBR绿,5 μl引物混合物(前后引物均为1.4 μM), 5 μl DNA模板(1 - 6ng /uL)。Sensimix采用标准的2步方案:95°C酶激活10分钟,然后95°C循环40次,持续15秒,60°C持续45秒。每次运行后,从60°C开始,每增加1°C生成一条熔融曲线,以检查引物二聚体的形成。在所有运行中,荧光阈值设置为150相对荧光单位(RFU)。反应进行了三次重复。如果三个重复的标准偏差高于0.4个CT,用肉眼筛选出明显的异常值,去除异常值后得到一个标准差≤0.4的重复。C的样本T在进一步的分析中,去除标准差大于0.4且无异常值的情况很少发生。所有拷贝号都是用ΔC计算的T方法使用参考基因和校准样本(本质上与ΔΔC相同T方法(2但计算顺序不同),用自制的电子表格进行计算。简而言之,“原始”拷贝数的计算公式为:原始拷贝数= 2(CT(参考)- CT(感兴趣的基因))×(参考基因拷贝数)。接下来,原始值除以校准器的原始值(所有运行中都包含一个已知的单拷贝样本),得到(校正后的)拷贝数。这个变体相对于标准ΔΔC的优点T方法是(i)在我们的实验过程中,我们发现这种方法计算起来更直观,(ii)原始拷贝数对引物对的匹配效率提供了额外的验证(原始值与校正值越接近,匹配越好),以及(iii)当在一次运行中识别单个拷贝样本时,与仅使用校准器的原始值相比,用原始值除以单个拷贝样本(包括校准器)的原始值的平均值可以提高拷贝数确定的精度。
结果
引物对选择和样品稀释
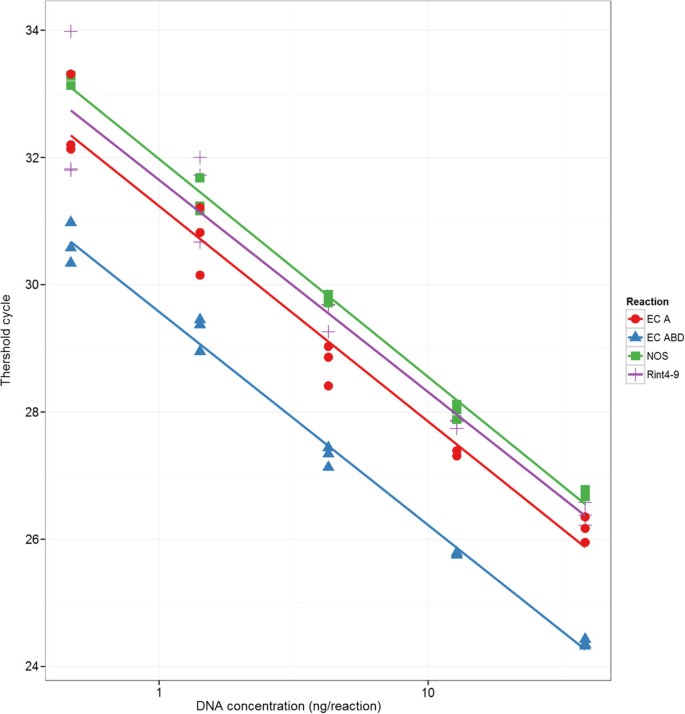
在特异性测试(干净的熔体曲线和琼脂糖凝胶上的单条带)和缺乏或低水平引物二聚体(无模板对照中无或极低信号)后,选择了4对引物:Rint4-9、NOS、EC A和EC ABD(表1).随后的样品稀释级数和回归分析表明,它们的效率非常匹配,接近100%(图3.).
在样品稀释过程中,我们注意到当DNA浓度为bb0 - 13 ng/μL时,样品中有CT数值高于预期。同样,浓度< 0.3 ng/μL的样品在技术重复之间的变异也有所增加。因此,在0.3 - 13 ng/μL范围之外的稀释系列样品不包括在回归分析中。正因为如此,实验样品被稀释到1-6 ng/ μL范围内。
EC A与EC ABD的比值为1:3.3。这接近于预期的1:3比率,在没有参考样本(即原始拷贝数)的情况下比较两对不同引物时,也完全在实时PCR的误差范围内。这一结果进一步表明,我们的引物设计和选择过程产生了非常匹配的引物对。
实验1
为了测试该方法是否能够区分纯合子和半合子植物,使用上述4对引物筛选了8个样品。结果汇总见表2.对于所有可用引物对组合,该试验明确地识别了只有一个拷贝的4个植物,显示了约30%的拷贝数估计误差(从0.7到1.2)。对于纯合子植物,估计的范围在1.6到2.6之间,表明正确的鉴定具有相似的30%的误差。校正值(校准器和单拷贝样本的平均值)在运行之间有所不同,表明不同运行之间的效率有一些轻微的变化,但始终在0.6至1.8的范围内。
实验2
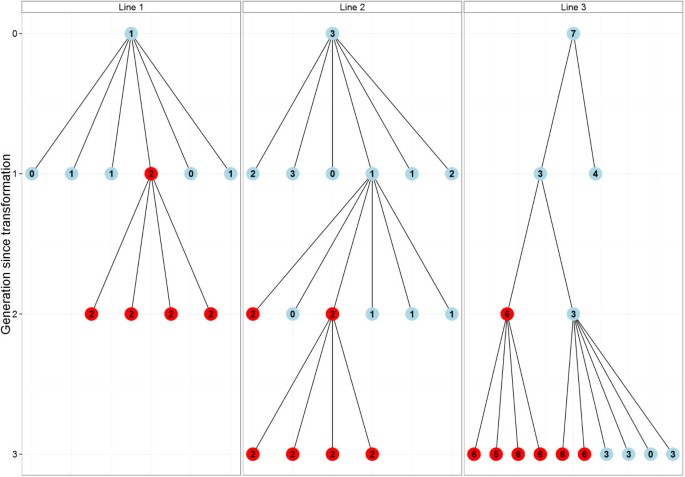
我们对3株转化植株进行了三代的跟踪研究,以研究实时PCR技术在纯合子转化系选择中的有效性(图4).从单株拷贝植株开始,筛选T基因中的纯合子植株,这是获得纯合子系的最快途径1.当T0是一个低拷贝数(2-3)的植株,单个的拷贝植株往往可以在T1先代,然后在T2(第2行)。但是,当从较高的拷贝号T开始时0植物,它可能不可能选择一个单一的复制植物,因为几个副本可能被链接,有效地表现为一个单一的副本。以株系3为例,表明该株系可被鉴定为高拷贝纯合子株系。
实验2的real-time PCR结果通过两种方法进行验证。首先,我们用末端PCR法筛选了一个半合子植株和一个纯合子植株的24个后代。单拷贝植株的7个后代为阴性,而纯合子植株的所有后代均为阳性,证实了实时PCR的结果。其次,我们用real-time PCR对3个纯合子植株的4个后代进行了筛选,确认它们都具有相同的偶数个转基因拷贝数。
大米
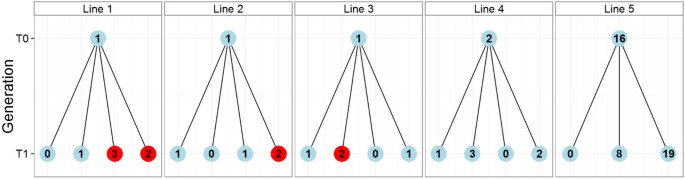
我们成功地使我们的分析方法适用于水稻。由于在转化过程中使用的水稻表达盒包含与小麦相同的标准序列元素,因此可以使用相同的结构特异性引物对(在本例中选择了Rint4-9引物对)。然而,需要一个水稻参考基因。我们选择蔗糖磷酸合酶(SPS),因为它是一个被证实的单拷贝基因,可以获得良好的序列信息[17].一旦产生了引物对,并选择了最好的一个(如方法-实时PCR引物开发中所述),该检测对水稻和对小麦一样有效(表1和图5).5株植物在T0和多达4个后代(T1)进行了筛选。携带唯一插入的纯合子植株在植株1、2和3中被鉴定出来,而在植株4和5中多次插入使纯合子系无法被鉴定出来1的一代。
纯合子水稻系的发育。水稻(T0和T1使用Rint4-9和SPS引物对(表1).红色表示鉴定的纯合子植株。注意,多次插入阻止了T中第4行和第5行纯合子系的阳性识别1经鉴定的纯合子系的稳定性尚未在下一代(T2)。
讨论
该方法为纯合子转基因株系的开发提供了有力的工具。理想情况下,该分析被用来选择一个单拷贝的转基因T0植物作为检测方法在检测单个拷贝时是最精确的,在这些系中不存在由于多拷贝插入而导致基因沉默的可能性。筛选6株下一代植株,一般足以鉴定出一株纯合子植株(预期频率为0.25),可作为转基因纯合子系的建立者。低拷贝数T0植物可以在下一代中筛选一个单拷贝分离体,然后它可以用作携带单插入纯合子系的建立植物。在T0具有多个副本的植物,可能需要反向交叉来实现单副本生成(未测试),尽管如果植物携带链接副本,这将不起作用。具有假定的连锁拷贝的植物可以在一定程度上被制成纯合子,但是在这些分析中,关于精确拷贝数的不确定性仍然存在,这对于单插入纯合子系是不存在的(下面讨论)。
所要求的高精度是通过以下几个简单的规则可靠地实现的:(i)选择(接近)100%效率的引物(通过在标准化PCR条件下设计和测试每个位点的数对引物,并选择(接近)100%效率的那一个);(ii)使用高质量的商业qPCR主混合物和(自制的)引物预混合物,通过限制移液步骤的数量来最小化运行之间的差异;(iii)使用正确浓度(在动态范围内)的优质DNA。这种方法一直导致CT标准偏差≤0.4的值,用于拷贝数和合子度测定[13].
在这些分析中确定的约30%误差的一个重要方面是,对于单个拷贝植物的确切拷贝数几乎没有不确定性,但这种不确定性随着拷贝数的增加而逐渐增加。例如,6个副本的结果应该被解释为有4到8个副本。这个误差可以通过运行更多的技术复制(未测试)来减少;然而,在我们的分析中,我们认为这是没有必要的,因为主要目的是尽可能有效地选择具有独特插入的植物。为了反映这个误差范围,拷贝数结果应该以整数形式报告。此外,为了尽量减少假阳性的可能性,一个鉴定的纯合子植物的几个后代应该被筛选以验证拷贝数的稳定性。
布伯纳和鲍德温[2]表明之前的大多数研究都使用Taqman探针进行拷贝数分析(一个例外是[12]),这表明这是因为这些探针更优越,因为它们不受引物二聚体形成的影响。然而,我们认为SYBR绿色在拷贝数分析方面比Taqman探针具有重要的优势,对Taqman探针的关注可能是以前的研究难以达到可靠合子筛选所需精度的原因之一。根据我们的经验,使用SYBR绿色有助于选择能以接近100%的效率放大的引物对。每个引物对的低得多的成本使得每个靶点订购几个引物对更加可行。此外,使用SYBR绿色为引物找到合适的DNA序列的机会更高(在小区域内工作时尤其重要),因为产品可以在60到200 bp之间的任何地方,不需要适合探针。不是优化PCR对引物的反应,而是对PCR条件进行了标准化,只选择在标准条件下以接近100%的效率扩增且不产生干扰引物二聚体的引物对。
在拷贝数确定方面,Taqman探针相对于SYBR的一个优点是它们提供了多路复用的能力,这是高通量筛选的一个强大优势。然而,我们的经验是,选择纯合子系的过程本身并不是一个高通量的过程,而是受益于筛选许多不同结构的灵活性和对新靶点的简单适应过程,正如我们的试验的情况一样。任何含有两个标准序列元素之一的小麦结构都可以使用所提供的引物进行筛选,水稻的例子表明,该方法可以很容易地适用于其他植物。
由于面包小麦是六倍体,我们能够为参考基因制造基因组非特异性和基因组特异性引物对。任何一对都可以作为参考,但重要的是要知道参考引物对扩增了多少拷贝,因为这将影响对原始转基因拷贝数的估计。其他生物,如水稻和大麦,没有这种复杂性,因此引物设计更容易。
菲茨杰拉德et al。[18]已经在小麦中使用基因组特异性实时聚合酶链反应(real-time PCR)在大样本收集中检测Null-plants(在三个基因组中有一个缺失感兴趣的基因的植物)。我们提出,我们的方法可以帮助选择双(和三)零线通过这些单零线植物的交叉。用我们的方法,应该可以选择一个基因组为零的植物,另一个基因组为半合子的植物,这可能是非常有益的,特别是在双零难以捉摸的情况下(例如,由于负选择压力)。当将多个结构物交叉到单个植物系(使用结构物特异性引物对)时,也可以获得类似的优势。最后,我们的分析可以用于研究低拷贝数基因的内源基因拷贝数变化。
结论
在这里,我们提出了一种基于实时PCR的方法,以快速跟踪转基因纯合子系的发展,在商业上重要的植物品种,相对较长的世代,如小麦和水稻。DNA分离、引物设计、引物选择和实时聚合酶链反应(real-time PCR)的标准化程序形成了一种能够可靠地从低拷贝数到高拷贝数植物中区分单个拷贝的方法,这可以在T0的一代。合子性测定可以在其后代上进行,以选择携带单插入的纯合子系,避免转基因沉默的潜在问题和与多次插入的植物相关的复杂的解除管制程序的商业应用。我们专门设计了这种方法,使其相对快速、经济和多功能,因此它可以用于许多不同的谷物结构,并可以很容易地适应不同的载体和植物种类。SYBR绿色被认为在拷贝数分析方面比Taqman探针有几个优点,包括引物设计、灵活性和成本效益。该方法大大有助于纯合子系的发展,并有望大大节省时间和收集管理工作。
参考文献
- 1.
雷默·PG:转基因植物与食品:科学家的问题分析(上).植物学报,2008,59:771-812。
- 2.
IT:利用实时荧光定量PCR技术测定转基因植物的拷贝数和合子度。植物细胞学报,2004,23(5):263-271。
- 3.
Butaye KMJ、Goderis IJWM、Wouters PFJ、Pues JMTG、Delaure SL、Broekaert WF、Depicker A、Cammue BPA、De Bolle MFC:利用基因沉默突变体和基质附着区在拟南芥中稳定表达转基因。植物学报,2004,39(3):440-449。
- 4.
Sood P, Bhattacharya A, Sood A:单子叶转化的问题和可能性。植物学报,2011,55(1):1-15。
- 5.
唐伟,陈晓燕,陈晓燕,等:一株东方白松转基因基因的克隆及基因沉默。中国机械工程学报,2007,58(3):545-554。
- 6.
转基因植物的分子和遗传分析:考虑和方法。植物生态学报,2004,25(4):369 - 369。
- 7.
Gadaleta A, Giancaspro A, Cardone MF, Blanco A:用于硬粒小麦转基因拷贝数精确检测的实时PCR。细胞分子生物学杂志,2011,16(4):652-668。
- 8.
李志伟,Hansen JL,刘宇,Zemetra RS, Berger PH:用实时荧光定量PCR法测定小麦转基因拷贝数。植物生物学杂志,2004,22(2):179-188。
- 9.
寿红霞,Frame BR, Whitham SA,王凯:粒子轰击或农杆菌介导的转基因玉米事件的评价。分子育种,2004,13(2):201-208。
- 10.
杨丽彤,丁建勇,张春明,贾金伟,翁海红,刘文祥,张东宝:利用实时定量PCR技术估算转化水稻转基因拷贝数。植物学报,2005,23(10-11):759-763。
- 11.
德国MA, Kandel-Kfir M, Swarzberg D, Matsevitz T, Granot D:一种转基因植物合子性的快速分析方法。植物科学学报,2003,24(2):366 - 366。
- 12.
Casu RE, Selivanova A, Perroux JM:利用实时定量PCR技术对甘蔗转基因拷贝数进行高通量评价。植物细胞学报,2012,31(1):167-177。
- 13.
Bubner B, Gase K, Baldwin IT: real-time PCR检测植物中转基因拷贝数的检出限为双重差异。生物技术,2004,4:14。
- 14.
Ral JP、Bowerman AF、Li Z、Sirault X、Furbank R、Pritchard JR、Bloemsma M、Cavanagh CR、Howitt CA、Morell MK:降低小麦胚乳中葡聚糖、水-二激酶活性可提高营养物质生物量和产量。植物生态学报,2012,10(7):871- 871。
- 15.
Ellis MH, Rebetzke GJ, Azanza F, Richards RA, Spielmeyer W:面包小麦中赤霉素反应矮化基因的分子定位。中国科学(d辑),2005,29(3):369 - 369。
- 16.
Cunningham FX, Gantt E:植物类胡萝卜素生物合成的基因和酶。植物生态学报,1998,29(4):557-583。
- 17.
蒋丽霞,杨丽彤,张红红,郭春春,Mazzara M, Van den Eede G,张东斌:用于转基因水稻定性和定量分析的内源参比基因蔗糖磷酸合酶(SPS)的国际合作研究。农业化学学报,2009,57(9):3525-3532。
- 18.
Fitzgerald TL, Kazan K, Li ZY, Morell MK, Manners JM:一种检测六倍体小麦同源基因缺失的高通量方法。植物生物学杂志,2010,10:264。
确认
我们要感谢玛丽安·布洛姆斯玛在抄本数字分析和校对手稿方面的帮助,感谢亚历克斯·万在数据准备方面的帮助。我们还要感谢Andrew Bowerman对real-time PCR的讨论,感谢三位审稿人对手稿的有益评论。我们感谢澳大利亚谷物研究和发展公司的资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者声明他们没有竞争利益。
作者的贡献
JM构思了这项研究,参与了设计,进行了分子研究并起草了手稿。JR和CH参与了研究的设计,并帮助起草了手稿。所有作者阅读并批准了最终稿件。
权利和权限
本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(http://creativecommons.org/licenses/by/2.0),它允许在任何媒体上不受限制地使用、分发和复制,只要原著被恰当地引用。
关于这篇文章
引用这篇文章
miieog, j.c., Howitt, C.A. & Ral, JP。利用简单而灵活的实时荧光定量PCR技术快速开发纯合子转基因谷物品系。BMC植物杂志13,71(2013)。https://doi.org/10.1186/1471-2229-13-71
收到了:
接受:
发表:
关键字
- 实时聚合酶链反应
- qPCR
- 麦片
- 小麦
- 转基因
- 拷贝数
- 纯合子的线