抽象的
背景
细胞内配置的电池状态的一个重要特征。在显微成像技术的最新进展使我们可以容易地获得大量细胞内结构的显微图像。在这种情况下,自动显微图像识别技术的极端重要性,以未来型组学/可见的筛选方法。然而,有一种用于在特定植物细胞类型的细胞内细胞器没有基准显微镜图像数据集。我们以前建立的植物气孔(LIPS)数据库,植物保卫细胞的各种细胞内结构的光学截面图像的可公开获得的采集的实时图像,如环境信号感知和传导的模型系统。在这里,我们报告的最近更新嘴唇数据库,并建立一个数据库表,LIPService的。
描述
我们更新了LIPS数据集,并建立了新的命名LIPService接口,以促进细胞内的结构配置的有效检验。细胞核,微管,微丝,线粒体,叶绿体,内质网,过氧化物酶体,内体,高尔基体和液泡可使用探针名称或形态学参数如气孔孔径进行过滤。除了原始LIPS数据库的串行光学截面图像,用于三维细胞内结构的容易的网页浏览新体绘制数据已被释放,以允许与小区状态及其配置或关系的简易检查/形态。我们还展示了从图像聚类分析另一种植物细胞图像数据库的自动识别细胞器图像的新LIPS图像数据库的效用。
结论
更新后的LIPS数据库提供了代表性的细胞内结构的基准图像数据集拟南芥保卫细胞。新发布的LIPService允许用户检查器官三维构型与形态参数之间的关系。
背景
在影像设备和探测器的最新进展,使得它能够获得bioimages的巨大数字。一位研究人员可以很容易地检查几百图像。然而,当图像增大到数千,数百万甚至数十亿的数量,任何个人都不可能单独处理它们。细胞成像技术在特定的产率的大规模系统的图像数据表征基因和蛋白本地化[1].在这些情况下,计算机辅助成为图像检测越来越重要。要制定和图像检查评估的计算机程序,基准图像数据集是至关重要的。为了这个目的,所述基准数据集不应该是一个杂图像集合,但系统地拍摄图像设计成具有精确的目标。例如,MitoCheck财团集中于鉴定参与有丝分裂进程和收集67个平均HeLa细胞的时间推移图像两天表达的组蛋白-GFP与化学合成的短干扰RNA(siRNA)敲低系统21000蛋白编码人类基因基因[2].这次延时图像数据集可自由使用(http://www.mitocheck.org/).此外,卡内基梅隆大学Murphy博士的实验室获得了HeLa细胞的荧光标记细胞核、核仁、内质网(ER)、高尔基体、溶酶体、线粒体、质膜、核内体、肌动蛋白微丝和微管的图像,以及针对HeLa图像数据集提出的细胞器识别算法[3.].每10个标记的80-91个Hela细胞的基准图像在墨菲的实验室网站上免费提供(http://murphylab.web.cmu.edu/data/2dhela_images.html.).这些图像数据集的公开发布,将有助于计算图像分析的未来发展。
在植物科学,几个荧光显微镜图像数据库都集中在细胞内结构,如植物细胞成像(http://deepgreen.stanford.edu/)[4.],被照射的植物细胞(http://www.illuminatedcell.com.)[5.],以及植物细胞器数据库(http://podb.nibb.ac.jp/Organellome/)[6.].这些数据库提供了植物细胞结构和蜂窝动态的美丽图片,并且具有潜在的价值作为模型分析的资源[7.].但是,这些图像数据库包含不同植物细胞类型的图像,复杂分析。
植物气孔保卫细胞已经成为表征从环境感知的信号转导机制,膨压运动的模型系统[8.].保卫细胞的生物以前的研究已经证明,都需要健康的气孔运动细胞内结构[9.-11.].为了全面观察气孔运动过程中保卫细胞的动态变化,我们已经发布了50-60对保卫细胞的显微图像拟南芥植物气孔(嘴唇)数据库的实时图像中用18种细胞器标记荧光标记的防护细胞[12.].然而,与第一版本的数据库,可视化细胞内三维构型和/或挖掘生物学上有意义的信息,如细胞内的结构和气孔孔径之间的关系是不方便的。
我们已经用额外的数据集更新了LIPS数据库。原系列光学切片数据集仍可用为LIPS数据集I,体绘制数据集和对齐图像数据集分别新发布为LIPS数据集II和III。此外,还新建立了一个名为LIPService的数据库表,方便地检查细胞内配置和细胞状态/形态之间的关系。在本文中,我们描述了LIPService的更新内容和功能。该数据库将作为一个图像数据挖掘工具,一个基于网络的教育资源,以及一个植物保卫细胞内植物细胞内结构的基准数据集。
建筑和内容
拟南芥蒂利亚纳植物在23.5℃生长于土壤 - 蛭石在生长室,在60%相对湿度,并用一个12/12小时明/暗循环使用100微摩尔米-2S.-1白光。将4-5周龄植物的完全扩展玫瑰花叶片的表皮条放入荧光显微镜(IX70;奥林巴斯,日本)的倒置平台上,配备了浸没物镜(Uplanapo 100×/ 1.35油; Olympus),旋转圆盘共焦单元(CSU10; Yokogawa,Tokyo,Japan)和冷却的CCD摄像头(Coolsnap HQ; Photometrics,Tucson,AZ,USA)。GFP / YFP成像使用了488个NM励磁激光器(HPU-50101-PFS2; FFUKAWA,TOKYO,JAPAN)和524-546 NM发射过滤器(FF01-535 / 22-25; SEMROCK,ROCHESTER,NY,USA)。对于RFP成像,使用561个NM励磁激光器(85-YCA-025-040; CVI MELLES GRIT,Albuquerque,NM,USA)和524-546 NM发射过滤器(FF01-590 / 20-25; Semrock)。串行光学部分(0.5μm间隔)采用Metamorph 7.0软件(分子装置; Sunnyvale,CA,USA)。所有原始图像为500×500像素(32×32μm)。基于先前植物细胞生物学研究的可靠性和有效性选择这些荧光标记物。嘴唇数据库中使用的所有转基因植物具有健康的生长和昼夜周期的气孔运动[12.].由于样品制备中的机械损坏,我们没有用伪影捕获明显的异常防护细胞。要制作卷载数据,我们使用了imagej插件3d卷查看器(在imagej菜单插件-3d-3d查看器中;http://3dviewer.neurofly.de/)和转换后的数据为动画GIF用ImageJ的(http://rsbweb.nih.gov/ij/).对于对准的图像,我们用我们原来的KBI ImageJ的插件。有关详细过程,请参考我们的协议[13.].图像和HTML文件都位于我们的内部Web服务器上。搜索功能与谷歌自定义搜索实现(http://www.google.com/cse/).该LIPService使用谷歌的Fusion Tables(http://support.google.com/fusiontables/).
效用与讨论
原始嘴唇图像数据集的详细内容先前已报告[12.].在这里,我们重点介绍如何使用LIPS数据库网站和新的附加内容。LIPS数据库网站访问者可以通过在首页搜索框中输入查询词(如微管)来查找图像和/或文献数据。要获得LIPS图像数据集,用户应访问“图像”页面(http://hasezawa.ib.k.u-tokyo.ac.jp/lips/images.html;数字1一种)。在那里,用户将看到链接到三种类型的图像数据集,如下所述。
数据集I.原始串行光学切片
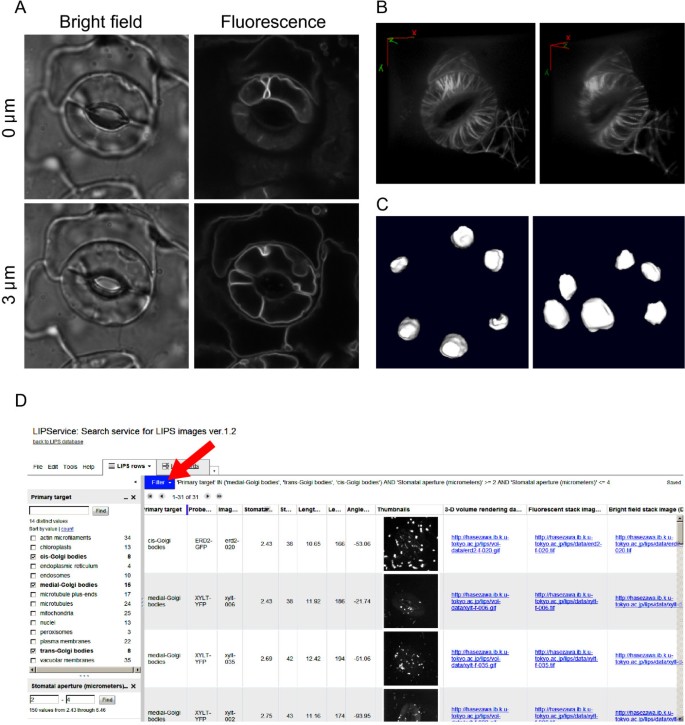
数据集I是18种荧光标记物的原始光学切片的集合,在我们之前的报告中列出了[12.].用户可以选择使用平铺缩略图细胞器(图1b)并访问用户选择页面(图1C).在这个页面上,用户可以通过点击缩略图下载原始序列光学部分16位TIFF图像。要浏览下载的图像,我们建议使用图像可视化工具,如ImageJ,它是免费的。关于图像可视化工具的概述,请参阅Walter等人的综述[1].嘴唇图像的代表性示例如图所示2答:此数据集包含可以使用适当的图像分析工具进行处理和分析原始图像[1].例如,三维模型可以从与ImageJ的插件3D查看器(ImageJ中菜单插件-3D-3D查看器的原始的连续切片被重建;图2b,c)。
LIPS数据库图像的实施例的应用程序。(一种)从数据集中LIPS I.明场和荧光图像的样本图像拟南芥在不同焦平面(0和3μm)处表达真空膜标记GFP-VAM3的防护细胞。所有图像尺寸为500×500像素(32×32μm)。(B.) gfp微管蛋白标记微管的体积效果图。左边和右边的图像显示了不同的视角。360°旋转动画在LIPS数据集II中可用。(C)用CT-GFP标记的叶绿体的表面图像。左边和右边的图像显示了不同的视角。(D.) LIPService接口。点击“筛选”下拉列表(箭头)允许用户选择“筛选”项目(例如,主要目标)。指定滤镜条件后,用户将看到一个带有下载图像数据集I和II链接的列表(蓝色部分)。
数据集II。体绘制数据
数据集I是原始串行光学截面数据集可用于与适当的图像处理软件来重建三维模型。然而,这样的三维重建需要技巧和劳动。为了克服这个问题,数据集II可以方便地查看目标细胞内结构的三维组织。体绘制数据(图2B)来自所有930双保卫细胞是可用的。原始图像是在10帧/秒的播放180帧。用户可以轻松地通过简单地点击在网络浏览器的链接,没有具体的图像可视化工具检查360个旋转GIF动画。我们相信,此功能将是受教育的宝贵援助。
为了更有效地搜索和浏览LIPS数据集I和II中的图像,我们准备了一个名为LIPService的数据库接口(图2d)。用户可以从“图像”页面访问Lipservice网站(http://hasezawa.ib.k.u-tokyo.ac.jp/lips/images.html).在此页面上,用户可以从下拉列表中选择“过滤器”诸如“主目标”和“气孔孔径”(图2D、 箭头)。当用户指定过滤条件时,将显示匹配的缩略图和下载图像数据集I和II的链接(图1)2d)。我们认为Lipservice对图像数据挖掘有用,尤其是在研究细胞内构造和形态计量参数之间的关系,例如气孔孔宽度(孔径)或长度。
数据集III。对准的图像本地化分析
DataSet III是荧光串行光学部分的对齐最大强度投影的集合。在我们最近的协议纸上描述了使用用于制作数据集III的ImageJ的逐步图像处理[13.].所有保护区区域与平均尺寸的304×119像素(19.5×7.6μm)对齐。荧光强度也被标准化为平均值0,标准偏差为1.在“图像”页面上,用户可以将DataSet III下载为包含1,860个32位TIFF文件的ZIP文件(52 MIB)(100-120示例 × 18 probes) with probe-name tags in the file names. Part of dataset III is presented as a tile in Figure1D.
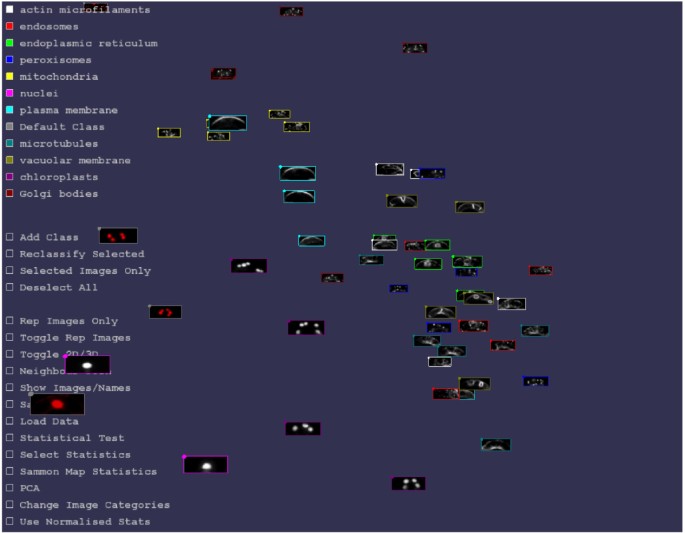
使用DataSet III,用户可以检查自己的保护单元的荧光图像。要演示此实用程序,我们执行了从另一个数据库中获得的图像的图像聚类分析,植物细胞器数据库(http://podb.nibb.ac.jp/Organellome/)[6.],使用GFP融合核定位序列标记的细胞核保卫细胞图像(NLS-GFP;额外的文件1:图S1A),由DSRED融合标记的线粒体,具有线粒体F1-ATPase的三角形亚基的预序(F1-ATPase-Δ-dsred;附加文件1:图S1b),由GFP融合用CAS,囊体膜 - 局部化蛋白(Cas-GFP)标记的叶绿体[14.)(附加文件1:图S1C)。经过预处理(附加文件1:图S1D-H),使用免费的图像聚类软件iCluster (http://icluster.imb.uq.edu.au/)[15.] (数字3.,附加文件2:Movie S1) and the 55 selected LIPS images (5 examples × 11 probes) that users can download as a ZIP file (860 KiB). iCluster gathered images of the same organelles (Figure3.),包括核(NLS-GFPvs。组蛋蛋蛋蛋蛋蛋白酶H2B-RFP;额外的文件1:图S2A),线粒体(F1 ATP酶-δ-DsRedvs。Mt-GFP;额外的文件1:图S2B)和叶绿体(CAS-GFPvs。自体荧光;额外的文件1:图S2C)。这些数据显示了该数据集的潜在有用性,用于用户按需本地化分析。
可用性和要求
LIPS数据库是免费提供的http://hasezawa.ib.k.u-tokyo.ac.jp/lips/index.html.。最新的信息不断从Twitter帐户的LIPS(发布https://twitter.com/lipsDb).
缩写
- ER:
-
内质网
- GFP:
-
绿色荧光蛋白
- 嘴唇:
-
植物气孔的现场图片
- RFP:
-
红色荧光蛋白。
参考
- 1。
Walter T,Shattuck DW,Baldock R,Bastin Me,Carpenter Ae,Duce S,Ellenberg J,Fraser A,汉密尔顿N,Pieper S,Ragan Ma,Schneider Je,Tomancak P,HérichéJK:从细胞到生物的图像数据的可视化。NAT方法。2010,7:S26-S41。10.1038 / nmeth.1431。
- 2。
诺伊曼B,沃尔特T,HérichéJK,BulkescherĴ,Erfle H,康拉德C,罗杰斯P,波塞尔我举行男,Liebel U,切廷C,Sieckmann女,保罗G,Kabbe R,WünscheA,Satagopam V,施米茨MH那Chapuis C, Gerlich DW, Schneider R, Eils R, Huber W, Peters JM, Hyman AA, Durbin R, Pepperkok R, Ellenberg J: Phenotypic profiling of the human genome by time-lapse microscopy reveals cell division genes. Nature. 2010, 464: 721-727. 10.1038/nature08869.
- 3.
Boland的MV,墨菲RF:一个神经网络分类器能够识别在HeLa细胞中的荧光显微镜图像的所有主要的亚细胞结构的图案的。生物信息学。2001,17:1213年至1223年。10.1093 /生物信息学/ 1213年12月17日。
- 4.
Cutler SR,EHRHARDT DW,GRIFFITTS JS,Somerville CR:随机GFP:cDNA融合使得CDNA融合能够以高频在拟南芥细胞中可视化亚细胞结构。Proc Natl Acad Sci USA。2000,97:3718-3723。10.1073 / pnas.97.7.3718。
- 5。
马瑟Y:该照明植物细胞。趋势植物SCI。2007年,12:506-513。10.1016 / j.tplants.2007.08.017。
- 6。
马诺S,三轮T,西川S,三村T,西村玛:植物细胞器数据库(PODB):可视化植物细胞器和协议植物细胞器研究的集合。核酸RES。2008年,36:D929-D937。
- 7.
Mano S,Miwa T,Nishikawa S,Mimura T,Nishimura M:眼见为实:关于使用图像数据库可视化探索植物细胞器动态。植物细胞生理学。2009, 50: 2000-2014. 10.1093/pcp/pcp128。
- 8.
SCHROEDER JI,ALLEN GJ,HUGOUVIEUX V,KWAK JM,WANER D:保护单元信号转导。Annu Rev植物植物植物Mol Biol。2001,52:627-658。10.1146 / annurev.arplant.52.1.627。
- 9.
福田男,Hasezawa S,浅井N,中岛N,近藤N:在保卫细胞微管的动态组织蚕豆根尖L.与昼夜周期。植物细胞生理。1998年,39:80-86。10.1093 / oxfordjournals.pcp.a029293。
- 10。
Sutter JU, Sieben C, Hartel A, Eisennach C, Thiel G, Blatt MR:脱落酸触发拟南芥KAT1 K的内吞作用+信道和其再循环至质膜。Curr Biol。2007年,21:1496至02年。
- 11.
桧垣T,久津N,佐野T,近藤N,Hasezawa S:量化与在植物细胞中肌动蛋白细胞骨架结构聚类分析:肌动蛋白时在拟南芥保卫细胞昼夜交替在气孔运动捆绑的作用。植物杂志2010,61:156-165。10.1111 / j.1365-313X.2009.04032.x。
- 12.
Higaki T,Kutsuna N,Hosokawa Y,Akita K,Ebine K,Ueda T,Kondo N,Hasezawa S:使用图像数据库嘴唇的统计细胞器解剖拟南芥防护细胞。SCI批准。2012,2:405。
- 13。
桧垣T,久津N,Hasezawa S:概率映射和用于在拟南芥保卫细胞荧光蛋白本地化的定量评估图像聚类。Protoc EXCH。2012年,10.1038 / protex.2012.013。
- 14。
Nomura H,Komori T,Kobori M,Nakahira Y,Shiina T:外部CA叶绿体控制的证据2+- 诱导细胞溶溶胶CA.2+瞬变和气孔闭合。工厂J. 2008,53:988-998。
- 15.
汉密尔顿NA,TEASDALE RD:可视化和聚类高吞吐量子蜂窝定位成像。BMC Bioinforma。2008,9:81.101186 / 1471-2105-9-81。
- 16。
基于迭代聚类的主动学习框架。中国社会科学。2012,3:1032。
致谢
这项工作是由高级测量和分析赠款JST到SH支持,文部科学省KAKENHI授权号24114007和24114704到SH,JSPS KAKENHI批准号24770038向NK和JSP KAKENHI授权号码25711017,以TH。
作者信息
隶属关系
通讯作者
附加信息
利益争夺
两位作者宣称他们没有相互竞争的利益。
作者的贡献
该计划数据库,进行了所有微观成像,准备了数据库网站,执行了映像聚类分析,并写了稿件。NK开发了KBI Imagej插件,并帮助了数据库设计。SH支持该项目。所有作者阅读并认可的终稿。
电子辅料
A-C.
附加文件1:图S1:使用嘴唇图像进行荧光保护单元图像的聚类预处理。()在本研究中使用的图像。这些图像从植物细胞器数据库下载(http://podb.nibb.ac.jp/Organellome/)[6.].GFP融合物与核定位序列(NLS-GFP)标记的核(一种);与线粒体标记F1-ATP酶(F1-ATP酶的DsRed)线粒体的δ-素亚基的前序列的DsRed融合体(B.); GFP与标记叶绿体的类囊体膜定位蛋白CAS融合[14.(C). (d - h)预处理图像的聚类的工作流程。图片。(A)作为一个例子。首先,图像被旋转(D.)用边界盒裁剪(E.那F).对于彩色图像,只选择目标通道得到灰度图像。然后将裁剪后的图像插值到与数据集III相同的大小,其强度归一化为平均强度0,标准差1 (G).在类似处理之后,单面防护电池旋转180°(倒置)(H).图S2。放大的图像群集的结果。注意相同的细胞器成像,包括细胞核(一种),线粒体(B.)和叶绿体(C),被聚集在三维萨姆米地图中。另请参阅附加文件2:电影S1。(PDF 2 MB)
12870_2013_1286_MOESM2_ESM.mov
附加文件2:电影S1。图3中显示的iCluster聚类结果的三维三文鱼映射结果如图3所示(MOV 2 MB)
作者的原始提交的图像文件
权利和权限
本文在BioMed Central Ltd.的许可下发布了这是一个开放的访问文章,根据Creative Commons归因许可(http://creativeCommons.org/licenses/by/2.0.)提供任何介质中的不受限制使用,分发和再现,所以提供了正确的工作。
关于这篇文章
引用这篇文章
Higaki,T.,Kutsuna,N.&Hasezawa,S. Lips Database with Lipservice:一种微观图像数据库拟南芥保卫细胞。BMC植物BIOL.13,81(2013)。https://doi.org/10.1186/1471-2229-13-81
已收到:
公认:
发表:
关键词
- 三维结构
- 拟南芥蒂利亚纳
- 荧光蛋白
- 微观图像
- 细胞器
- 气孔