摘要
背景
植物共表达数据库(PLANEX)是一种新型的基于网络的植物基因分析数据库。在内(http://planex.plantbioinformatics.org)包含从国家生物技术信息中心(NCBI)的基因表达综合(GEO)获得的公开的基因芯片数据。PLANEX是一个全基因组共表达数据库,它允许从各种各样的实验设计中鉴定基因的功能。它可以用于基因的特征,用于功能鉴定和分析一个基因与其他基因之间的依赖性。已经为其他物种开发了基因共表达数据库,但植物的基因共表达信息目前有限。
描述
我们将PLANEX构建为共表达基因和功能注释的列表拟南芥,大豆,大麦,水稻,番茄茄,小麦,葡萄而且玉蜀黍。PLANEX报告Pearson相关系数(PCCs;r-values)从感兴趣的基因分布到对应于特定生物体的给定微阵列平台集。为了支持PCCs, PLANEX对基因本体术语和Cohen’s Kappa值进行了富集测试,以比较共表达数据库中所有基因的功能相似性。PLANEX绘制了一个具有共表达基因的聚类网络,该网络使用k意思是方法。为了构建PLANEX,各种数据集由超级计算中心的IBM超级计算机Advanced Interactive eXecutive (AIX)解释。
结论
PLANEX提供了一个相关数据库、聚类网络和8种植物富集试验结果的解释。一个典型的共表达基因会生成一系列的共表达数据,其中包含数百个富集分析感兴趣的基因。此外,通过使用“共表达基因比较”功能,可以根据比较基因组学对共表达基因进行识别和编目。这种类型的分析将有助于解释实验数据,并确定感兴趣的基因之间是否有一个共同的术语。
背景
基因组学、蛋白质组学和生物信息学领域的方法组合为研究生物过程提供了强大的方法。基因的生物学功能通常是由蛋白质或基因产物的相互作用决定的,在生物过程中基因的表达经常是相关的。因此,共表达基因可能与生物途径有关,并可能为理解复杂的生物系统提供关键信息[1,2].许多技术方法已经被用于全基因组实验,同时测量数千个基因的调节的能力已经彻底改变了分析生物过程的方式。为了理解生物系统,共表达数据已用于各种各样的实验设计,包括基因靶向、调控研究和蛋白质-蛋白质相互作用中潜在伙伴的识别[3.].
需要大量这样的表达数据来估计共表达基因的依赖性。不幸的是,这些实验既昂贵又耗时。然而,大量的基因表达数据集最近已经可以用于一些植物物种。最流行的公共微阵列数据库是ArrayExpress [4],基因表达综合系统(GEO) [5],纳斯卡雷[6]和genevarcheator [7].然而,如果没有生物信息学背景,生物研究人员很难管理如此大量的基因表达数据。为此,生物信息学领域加速了生物过程的共表达分析。此外,完成了模式植物的基因组序列拟南芥[8),大豆[9),栽培稻[10),茄属植物lycopersicum[11],葡萄[12),玉米[13有先进的基因组和基因表达分析。对于其他基因表达数据分辨率较低的物种,例如大麦芽而且小麦,随着达纳·费伯癌症研究所(DFCI)的基因指数计划,基因组资源正在改善[14].注释基因组序列刺激了许多功能基因组方法的发展。这些材料对于基因组尺度微阵列中的基因表达很有价值。
在共表达数据集构建过程中,采用总结方法对基因表达数据进行归一化,包括RMA [15], gcrma [16]及MAS5 [17].鉴定共表达基因集的一种方法是通过估计基因表达相似性。估计基因表达相似性最方便的方法是使用皮尔逊相关系数(PCCs) [1,18].如果相似性是由相关度量(例如PCCs)确定的,则会生成一个表示表达式相似性的全面的相关值成对矩阵。
在共表达数据集分析的基础上,我们重点改进了基因网络的构建。主成分分析(PCA)是一种常用的技术,用于查找多元数据集的主成分。在DNA微阵列分析中,它被用来寻找在几个实验中合作改变表达的基因群[19],在基因空间中进行主成分分析。然后,k结合-mean聚类算法来揭示贡献较大的样本。
植物共表达数据库之前已经建立拟南芥,栽培稻而且大麦芽。拟南芥共表达工具包(ACT) [20.],星网2 [21], RiceArrayNet [22], atted-ii [23],共表达生物过程(CoP)数据库[24]和PlaNet [25],用于搜索共表达式关系和合并函数数据。考虑到最近高性能计算机的快速发展,能够执行快速计算,使用大规模基因表达数据构建共表达数据库是可能的。
在本报告中,我们描述了PLAnt共表达数据库(PLANEX;额外的文件1:表S1),并讨论用户查询产生的输出。PLANEX已经计算出了8种植物的基因对相关性。与PLANEX,我们提供拟南芥,甘草,大麦,水稻,番茄茄,小麦,葡萄而且玉米带有用户友好的web界面的共表达数据集,用于检索感兴趣的共表达基因列表和功能丰富数据。构建PLANEX的核心动机是在进行额外昂贵的湿式实验室实验之前,利用大量的微阵列数据资源进行生物相互作用、表达多样性和发现假定的基因调控关系。该数据库提供了可能有助于理解输入基因的表达相似性和功能丰富的细节。
结构与内容
表达数据
原始微阵列数据从国家生物技术信息中心(NCBI)的GEO获得,截至2011年4月。我们从拟南芥,甘草,大麦,水稻,番茄茄,小麦,葡萄而且玉米Affymetrix GeneChip基因组阵列,这是最常用和最公开的植物平台之一(表1).
所有原始数据(CEL文件格式)都是通过编程访问GEO (http://www.ncbi.nlm.nih.gov/geo/info/geo_paccess.html).我们终止了GEO系列(GSEs),其中包括截断的GEO样本(GSM)。跨平台GSMs也被终止,包括GSE13641 (Rorippa两栖类表达式配置文件拟南芥Affymetrix基因芯片平台;GPL198)。我们还收集了原始数据,排除亚种表达数据,包括甘氨酸大豆在大豆平台(GPL4592;例如GSE20323)和拟南芥亚属。佩特拉-而且拟南芥halleri在拟南芥Affymetrix基因芯片平台(GPL198;例如GSE5738)。
CEL文件用于汇总探针集,这是对芯片像素值进行强度计算的结果。所有表达水平均采用背景减法、归一化和汇总探针集进行分析。我们估计分位数归一化使用RMA算法检测背景信息。所有微阵列都使用Affymetrix Power Tools计算探针集,总结了8个物种中的每一个[26].
实现
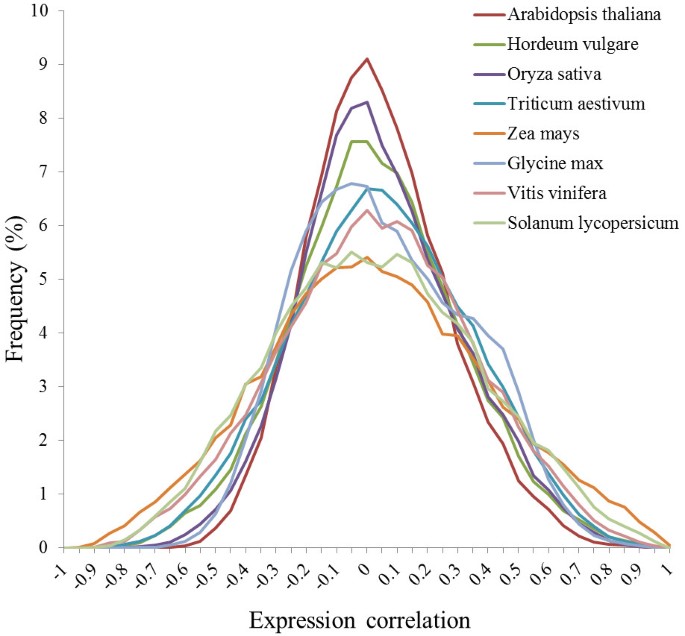
基因共表达数据预先输入PLANEX系统。数据采用表达式探测集汇总数据实现。我们提供了PCCs来评估基因共表达的程度,我们开发了新的c++代码来生成共表达数据。成对共表达式计算不需要大量的CPU,但大量的CPU有助于减少计算时间。我们使用了韩国科学技术信息研究所超级计算中心的GAIA系统,[27]包含1536个CPU内核。GAIA系统基于IBM的AIX (Advanced Interactive eXecutive)系统,支持MPI (Message Passing Interface)接口[28].我们独特的c++代码支持MPI和共表达式数据由512个CPU核估计。为了检索共表达式数据,我们为共表达式值设置阈值。为了明确共表达基因集的阳性(前1%的PCCs)和阴性(后1%的PCCs)值,随机基因对的分布通过PCCs进行评估(图1).随机基因对的数量对应于阵列上探针的数量(表2).
聚类
对于聚类,使用基因表达值进行分析。我们应用了k对表达式数据进行-mean聚类,将每个点分配到中心最近的聚类[29].我们使用PCA来确定聚类的数量k.主成分分析是使用CLUSTER进行的,这样聚类是有序的,选择聚类来最大限度地解释数据向量中的剩余方差[1].因此,k-平均聚类分析各物种的聚类数量。大量的表达式数据需要较长的聚类时间。因此,我们编译了Parallel K-mean Data Clustering代码[30.],在带有MPI的AIX超级计算机系统上执行。的k-mean算法提供了PLANEX中共表达网络的节点。
将基因标识符映射到探测集id上
基因组序列和注释计划Phytozome最近完成并发布[31].我们通过下载所有Affymetrix GeneChip探针序列来澄清该物种的注释和序列[26],我们通过探针将它们映射到六种已测序植物的基因组核苷酸上:拟南芥,甘草,水稻,葡萄,茄属而且玉米(Phytozome V9.0)。相比之下,其他基因组序列仍未完成的物种,如大麦芽而且小麦与DFCI的暂定一致序列进行了映射。探测匹配是使用我们独特的Perl脚本进行的。该脚本针对任何给定物种的单个基因芯片探针处理字符串匹配的核苷酸序列(包括反向补体),并返回与每个物种序列对应的探针集亲和力列表。具体地说,玉米每个探针有15对序列对,所有其他植物物种每个探针有11对序列对。
基因本体术语赋值
由于基因本体(GO)术语的层次树和术语的冗余性,我们将GO术语映射到代表性的基因功能。DFCI提供了GO映射注释。Phytozome序列注释不支持GO映射注释,但提供Pfam id;我们将代表性的Pfam id与GO项进行映射。我们将外部分类系统映射为GO [32].GO- termfinder用于估计GO术语的富集程度[33].GO- termfinder通过web界面集成到PLANEX中,该界面评估了GO主要类别的丰富程度,包括细胞成分、生物过程和分子功能的超几何分布以及Benjamini和Hochberg描述的错误发现率(FDR)。
共表达基因组的比较分析
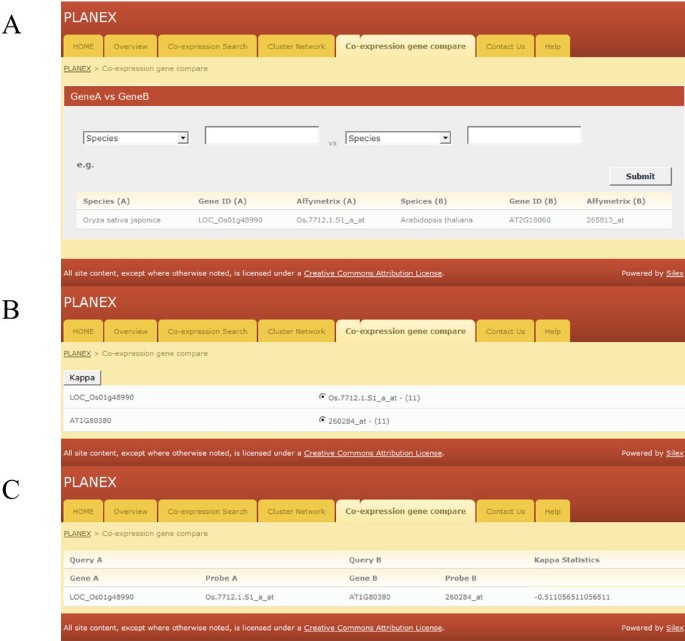
Cohen’s Kappa统计数据用于比较物种间的共表达数据[34].类似于在线DAVID工具的内部模块35,36]使用Kappa统计数据来评估共同表达的相似性,这些统计数据通过web界面集成。蛋白质序列被用来从物种中选择两个基因拟南芥,大豆,栽培稻,葡萄而且玉米.提交两个查询基因后,模块比较每个查询基因的共表达数据集,转换为Pfam ID [37].Kappa测量了表的主对角线上的数据值的百分比,然后调整这些值,以确定由于偶然性而可能产生的协议数量。
系统开发
PLANEX的web应用程序是用Dancer (Perl web应用程序框架)开发的[38]为服务器端和JQuery (Javascript框架)[39]为客户端。共表达式数据库与MongoDB(面向文档的数据库)相结合[40]和TokyoCabinet(数据库管理)[41].MongoDB将共表达式数据存储为文档文件,使得成对共表达式应用程序中的数据集成更容易、更快。TokyoCabinet通过单键存储基因ID数据,并使用哈希技术快速检索查询基因的共表达数据。这种组合显著提高了搜索的处理和访问速度。我们使用了Cytoscape Web [42],在浏览器上显示网络。Cytoscape Web不需要安装插件,可以快速运行于各种浏览器。PLANEX在Ubuntu 10.04上运行[43配备2.66GHz双CPU和8GB内存的服务器。
实用和讨论
Web界面
PLANEX可通过用户友好的网页界面(http://planex.plantbioinformatics.org/,见可用性需求部分),提供三个搜索菜单:“共同表达搜索”,“集群网络”和“共同表达基因比较”(图2).“共表达搜索”可用于共表达的基因集和PCC值。搜索数据库时,使用Affymetrix GeneChip ID或代表性基因ID进行“按ID搜索”或使用粘贴序列进行“用BLAST搜索”[44];两个或多个具有代表性的基因id用于“检索带有基因列表的PCC”(图3.A).如图所示3.A, PLANEX依赖于物种、目标、截止、BLAST程序和e-value等选项的选择。随机基因的分布被确定为每个物种的截止值。
将查询提交到“按id搜索”或“使用BLAST搜索”后,将显示探测匹配结果页面。探测匹配页面表示与查询匹配的探测数量占探测总数的比例,以及它们的亲缘关系,如' match '所示(图3.B).这个探针匹配页面将帮助丢弃多余的基因探针。PLANEX发现许多共表达基因在临界值内(图3.C).复制的Affymetrix id显示在结果页面的“复制”部分(图3.D).可通过GO-TermFinder下载CSV格式的共表达基因集进行分析。GO- termfinder提供了三个GO术语丰富分析与超几何p-value在FDR≤10-6时< 0.05(图3.E).向“Retrieve PCCs with gene list”提交查询后,基因列表将以成对格式显示相关性(图3.F). PLANEX不提供探针匹配页面,但是,相反,它为一个基因列表提供了所有潜在匹配的探针集,这表明PCCs和亲缘性。数据由GO-TermFinder支持,这与其他搜索相似。
PLANEX允许在浏览器中显示共表达式网络数据。“集群网络”是基于k-mean聚类分析和PCCs,支持“按id搜索”和“使用BLAST搜索”功能(图4A).网络由实验的结果组成k-均值聚类分析,表示节点,节点大小,表示边缘个数,边缘由pcc表示(图4B)。
PLANEX中的Kappa统计分析工具可用于与其他物种的共表达基因进行比较,使用“共表达基因比较”功能(图5).它只接受拟南芥,甘氨酸,水稻,葡萄而且玉米作为蛋白质注释植物基因id。任何两个物种都可以与Phytozome中的代表性基因ID进行比较。简单的Kappa统计系数显示了两个共表达基因集之间的一致性,这是在二进制尺度上测量的。这种分析在比较基因组学中是有用的,可以确定共表达基因集的相似性或家族基因的功能相似性。这种方法与医学文献中常见报道的测量值进行了比较分析。
讨论
PLANEX是一个新的数据库,帮助研究人员研究复杂的生物过程,通过共表达的基因集覆盖在一个k意味着集群。ATTED-II、STARNET 2、RiceArrayNet和CoP提供了共表达关系,但它们只包含一到三组共表达数据。因此,PLANEX的一个优势是它结合了来自8个不同物种的共表达数据集。此外,它聚类和比较共表达基因的成员。据我们所知,PLANEX是唯一一个将集群和PCCs数据结合在一起的系统。
PLANEX的另一个优点是探针通过字符串匹配而不是BLAST来匹配代表性基因。我们的探针匹配脚本如果探针序列中的每个碱基与代表性基因序列完全匹配,没有任何缺口,就会得到阳性结果。
PLANEX的一个潜在应用是GO-TermFinder。我们生成了一个酿酒每个物种的基因组数据库(SGD)文件格式。模式物种,比如拟南芥而且栽培稻拥有大量功能注释基因,由各种实验衍生的证据代码支持GO术语。相比之下,其他生物只有通过电子注释推断出的注释(例如,葡萄而且玉米)或完全缺乏功能注释。由于我们最初缺乏功能性GO数据,我们将Pfam转换为GO id,并构建了一个SGD文件用于功能丰富分析。但是,这个映射只能用作指导。
我们之前的报告栽培稻基因组复制[45]证明了阳性(前1%的PCCs)值为0.545,但我们使用0.646作为阳性PCCs的阈值栽培稻本期报道。我们建立了这个不同的标准,因为我们包含了超过给定数量的微阵列,因为我们相信更多的微阵列对表达研究产生更大的意义。此外,Aoki等人。[46]指定了共表达基因检索的最小PCCs值(0.55-0.66),以减少错误的基因功能关系。我们为每个物种提供了一个特定的阈值来检索正态分布的共表达基因(图1).
PLANEX菜单上的“共表达基因比较”选项卡提供了比较基因组学的数据。的拟南芥基因组被认为包含与水稻基因组相似的基因数量,并且两者都经历了全基因组复制事件[47,48].Kappa统计系数的使用应与数据的表达差异程度相一致。之前,我们报道了水稻基因家族使用水稻公共微阵列在成员之间证明了类似的高度表达多样性[45].共表达基因的比较可能支持对基因家族成员在进化过程中复杂生物过程方向的专门化的理解[49].
结论
比较共表达基因的小而重要的功能可能为直系同源基因之间的分子功能守恒或多样性提供线索禾本科家族的基因。PLANEX可用于解释共表达基因的结果,也可用于比较基因组学中的精细分析。PLANEX补充了现有的数据库和工具,如ATTED-II、CoP和STARNET 2。
可用性和需求
项目名称:在内
操作系统:平台独立(在Windows、i386 Linux和Mac上测试)
编程语言:Perl
其他要求:Web浏览器(在Chrome, Safari和Explorer上测试)
许可:创作共用署名许可协议
缩写
- 行为:
-
拟南芥共表达工具包
- AIX:
-
高级交互执行
- DFCI:
-
Dana faber癌症研究所
- 地理:
-
基因表达综合
- 走:
-
基因本体论
- KEGG:
-
京都基因和基因组百科全书
- 主成分分析:
-
主成分分析
- PCC:
-
皮尔逊相关系数
- 在内:
-
PLAnt共表达数据库
- 质子泵抑制剂:
-
蛋白质相互作用
- SGD:
-
酿酒基因组数据库
- TAIR:
-
拟南芥信息资源。
参考文献
- 1.
Eisen MB, Spellman PT, Brown PO, Botstein D:全基因组表达模式的聚类分析和显示。中国科学:自然科学,1998,29(2):344 - 344。10.1073 / pnas.95.25.14863。
- 2.
李洪亮,徐佳佳,秦娟,潘丽萍,王晓明,王晓明。基于微阵列的人类基因共表达分析。基因组学报,2004,14:1085-1094。10.1101 / gr.1910904。
- 3.
杨晓明,杨晓明,杨晓明:植物基因共表达网络中实用信息的提取方法。中国生物医学工程学报,2007,29(4):366 - 366。10.1093 /卡式肺囊虫肺炎/ pcm013。
- 4.
张志刚,张志刚,张志刚,张志刚,等:微阵列基因表达库的构建与应用研究。核化学学报,2003,31:68-71。10.1093 / nar / gkg091。
- 5.
Barrett T, Troup DB, Wilhite SE, Ledoux P, Rudnev D, Evangelista C, Kim IF, Soboleva A, Tomashevsky M, Edgar R: NCBI GEO:挖掘数千万个表达配置文件-数据库和工具更新。核科学通报,2007,35:D760-D765。10.1093 / nar / gkl887。
- 6.
克根·DJ,詹姆斯·N,奥克耶尔,希金斯,乔瑟姆·J,梅:NASCArrays:由NASC转录组服务生成的微阵列数据存储库。核科学通报,2004,32:D575-D577。10.1093 / nar / gkh133。
- 7.
Zimmermann P, Hirsch-Hoffmann M, Hennig L, Gruissem W:基因研究者。拟南芥微阵列数据库和分析工具箱。中国生物医学工程学报,2004,36(3):344 - 344。10.1104 /页。104.046367.
- 8.
开花植物拟南芥基因组序列分析。自然科学,2000,30(4):344 - 344。10.1038 / 35048692。
- 9.
Schmutz J, Cannon SB, Schlueter J, Ma J, Mitros T, Nelson W, Hyten DL, Song Q, Thelen JJ,程杰,等:古多倍体大豆基因组序列分析。自然科学进展,2010,38(4):344 - 344。10.1038 / nature08670。
- 10.
Ouyang S, Zhu W, Hamilton J, Lin H, Campbell M, Childs K, Thibaud-Nissen F, Malek RL, Lee Y, Zheng L,等:TIGR水稻基因组注释资源的改进与新特征。核化学学报,2007,35:D883-D887。10.1093 / nar / gkl976。
- 11.
番茄基因组联盟:番茄基因组序列为肉质水果的进化提供了见解。自然科学学报,2012,34(4):344 - 344。10.1038 / nature11119。
- 12.
Jaillon O, Aury J-M, Noel B, Policriti A, Clepet C, Casagrande A, Choisne N, Aubourg S, Vitulo N, Jubin C, Vezzi A, Legeai F, Hugueney P, Dasilva C, Horner D, Mica E, Jublot D, Poulain J, Bruyère C, Billault A, Segurens B, Gouyvenoux M, Ugarte E, Cattonaro F, Anthouard V, Vico V, Del Fabbro C, Alaux M, Di Gaspero G, Dumas V,等。自然科学学报,2007,29(3):344 - 344。10.1038 / nature06148。
- 13.
Schnable PS, Ware D, Fulton RS, Stein JC, Wei F, Pasternak S, Liang C, Zhang J, Fulton L, Graves TA:玉米B73基因组的复杂性、多样性和动态。科学通报,2009,29(4):344 - 344。10.1126 / science.1178534。
- 14.
杨晓东,王晓明,王晓明,等:基于基因索引的生物发现研究。清华大学学报(自然科学版),2010,27(1):1 - 3。第一章:Unit1.6.1-36
- 15.
Irizarry RA, Hobbs B, Collin F, Beazer-Barclay YD, Antonellis KJ, Scherf U, Speed TP:高密度寡核苷酸阵列探针水平数据的探索、归一化和总结。生物科学学报,2003,4:449 - 464。10.1093 /生物统计学/ 4.2.249。
- 16.
吴震,伊瑞扎里,mr R, Martinez-Murillo F, Spencer F:基于模型的寡核苷酸表达阵列的背景调整。中国统计杂志,2004,29(1):1 - 4。10.1198 / 016214504000000683。
- 17.
刘文敏,梅锐:表达分析的鲁棒估计。生物信息学,2002,18:1585-1592。10.1093 /生物信息学/ 18.12.1585。
- 18.
Soper HE, Young AE, Cave BM, Lee A, Pearson K:小样本中相关系数的分布。“学生”和r. a. Fisher论文的附录二。合作研究。生物计量学,1917,11:328-413。10.1093 / biomet / 11.4.328。
- 19.
Brunet JP, Tamayo P, Golub TR, Mesirov JP:利用矩阵分解发现元基因和分子模式。美国国家科学研究院。2004, 30(4): 344 - 344。10.1073 / pnas.0308531101。
- 20.
Manfield IW, Jen CH, Pinney JW, Michalopoulos I, Bradford JR, Gilmartin PM, Westhead DR:拟南芥共表达工具(ACT):基于微阵列的基因表达分析的web服务器工具。核化学学报,2006,34:W504-W509。10.1093 / nar / gkl204。
- 21.
Jupiter D, Chen H, Van Buren V: STARNET 2:使用微阵列共表达数据加速发现基因调控网络的基于web的工具。中国生物医学工程学报,2009,10:332。10.1186 / 1471-2105-10-332。
- 22.
Lee TH, Kim YK, Pham TTM, Song SI, Kim JK, Kang KY, An G, Jung KH, Galbraith DW, Kim M,等:RiceArrayNet:水稻转录组相关基因表达数据库及其在共表达基因分析中的应用。植物科学进展,2009,29(3):344 - 344。10.1104 /页。109.139030.
- 23.
Obayashi T, Hayashi S, Saeki M, Ohta H, Kinoshita K: ATTED-II提供拟南芥的共表达基因网络。中国生物医学工程学报,2009,37:D987-D991。10.1093 / nar / gkn807。
- 24.
杨春华,杨春华,杨春华,杨春华。植物共表达基因模块的研究进展。生物信息学,2010,26:1267-1268。10.1093 /生物信息学/ btq121。
- 25.
Mutwil M, Klie S, Tohge T, Giorgi FM, Wilkins O, Campbell MM, Fernie AR, Usadel B, Nikoloski Z, Persson S: PlaNet:来自七个物种的植物网络的组合序列和表达比较。植物学报,2011,23:895-910。10.1105 / tpc.111.083667。
- 26.
Affymetrix。http://www.affymetrix.com/estore/.
- 27.
KISTI超级计算中心。http://www.ksc.re.kr/.
- 28.
工作人员:使用带有消息传递接口的可移植mpi并行编程。科学通报,1996,27(5):379 - 379。
- 29.
Hartigan JA, Wong MA:算法AS 136:一种k均值聚类算法。J皇家统计Soc系列C(应用统计学)。1979, 28: 100-108。
- 30.
并行k均值数据聚类。http://users.eecs.northwestern.edu/~wkliao/Kmeans/.
- 31.
Phytozome。http://www.phytozome.net.
- 32.
Hunter S, Apweiler R, Attwood TK, Bairoch A, Bateman A, Binns D, Bork P, Das U, Daugherty L, duquene L, Finn RD, Gough J, Haft D, Hulo N, Kahn D, Kelly E, Laugraud A, Letunic I, Lonsdale D, Lopez R, Madera M, Maslen J, McAnulla C, McDowall J, Mistry J, Mitchell A, Mulder N, Natale D, Orengo C, Quinn AF,等:InterPro:整合蛋白特征数据库。中国生物医学工程学报,2009,37:D211-D215。10.1093 / nar / gkn785。
- 33.
Boyle EI,翁s, Gollub J, Jin H, Botstein D, Cherry JM, Sherlock G: GO: termfinder -用于访问基因本体信息并发现与基因列表相关的显著丰富的基因本体术语的开源软件。生物信息学,2004,20:3710-3715。10.1093 /生物信息学/ bth456。
- 34.
科恩J:名义比例尺的一致系数。《心理教育》,2000,20:37-46。10.1177 / 001316446002000104。
- 35.
黄DW, Sherman BT, Lempicki RA:利用DAVID生物信息学资源对大型基因列表进行系统和综合分析。Nat协议,2009,4:44-57。
- 36.
Huang DW, Sherman BT, Lempicki RA:生物信息学富集工具:通往大型基因列表综合功能分析的路径。中国生物医学工程学报,2009,37:1-13。10.1093 / nar / gkn923。
- 37.
Bateman A, Coin L, Durbin R, Finn RD, Hollich V, griffith - jones S, Khanna A, Marshall M, Moxon S, Sonnhammer ELL, Studholme DJ, Yeats C, Eddy SR: Pfam蛋白家族数据库。中国生物医学工程学报,2004,32:D138-D141。10.1093 / nar / gkh121。
- 38.
- 39.
Jquery。http://www.jquery.com.
- 40.
MongoDB。http://www.mongodb.com.
- 41.
Tokyocabinet。http://fallabs.com/tokyocabinet.
- 42.
Lopes CT, Franz M, Kazi F, Donaldson SL, Morris Q, Bader GD: Cytoscape Web:基于Web的交互式网络浏览器。生物信息学,2010,26:2347-2348。10.1093 /生物信息学/ btq430。
- 43.
Ubuntu。http://www.ubuntu.com.
- 44.
Johnson M, Zaretskaya I, Raytselis Y, Merezhuk Y, McGinnis S, Madden TL: NCBI BLAST:一个更好的web界面。核酸工程,2008,36:W5-W9。10.1093 / nar / gkn201。
- 45.
林文伟,李保民,张春春:水稻重复基因的表达多样性和进化动态。中国生物医学工程学报,2009,27(1):483-493。10.1007 / s00438 - 009 - 0425 - y。
- 46.
杨晓明,杨晓明,杨晓明:植物基因共表达网络中实用信息的提取方法。中国生物医学工程学报,2007,29(4):366 - 366。10.1093 /卡式肺囊虫肺炎/ pcm013。
- 47.
Simillion C, Vandepoele K, Van Montagu MCE, Zabeau M, Vande Peer Y:拟南芥隐藏复制历史。美国国家科学研究院。2002, 99: 13627-13632。10.1073 / pnas.212522399。
- 48.
杨晓明,王晓明,杨晓明,王晓明,等:基于染色体复制事件的被子植物基因组进化研究。自然科学,2003,29(4):433-438。10.1038 / nature01521。
- 49.
张春春,林卫西,文建昌,郑俊华,李tg,林士德,赵帅,李kk, Kim W,徐耀文,李保明:Poaceae家族非特异性脂质转移蛋白(nsLTP)基因的进化:复制与多样性。中国生物医学工程学报,2008,29(4):489 -497。10.1007 / s00438 - 008 - 0327 - 4。
确认
作者要感谢Kunho Kim对PLANEX项目的贡献,从而使生物信息学研究成为可能。我们感谢Silex和Jongjin Lee建立了这个网页界面。我们也要感谢Hojung Yun对perl和这篇手稿的改进所做的贡献。
本研究由韩国教育科学技术部资助的国家研究基金(NRF)基础科学研究计划(NRF-2011-0011643)支持。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突.
作者的贡献
WCY、YY和CSJ设计并实现了数据库。WCY和YY建立了网站页面和在线工具。WCY, CSJ, KS和BML负责数据收集。WCY和KS参与了数据库模式的设计。WCY和BML构思了这项研究。WCY, CSJ, KS和BML起草了手稿。所有作者都阅读并批准了手稿。
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
严炜聪,余勇,宋凯。et al。PLANEX:植物共表达数据库。BMC植物生物学13日,83(2013)。https://doi.org/10.1186/1471-2229-13-83
收到了:
接受:
发表:
关键字
- Co-expression
- 数据库
- 皮尔逊相关系数
- 聚类