抽象的
背景
Orobanchaceae是唯一具有成员的植物家庭,代表全系列的寄生虫生活和自由生活谱系姐妹到所有寄生谱系,林恩伯加斯.这个家庭的一般成员,以及重要的寄生植物模型,TripHysaria Versicolor.定期喂食各种宿主植物。在这里,我们比较德诺维组装从主寄生虫界面处的激光微扰动组织产生的转录om,以揭示寄生植物和其宿主之间的大量不交易化相互作用的细节。
结果
互动Triphysaria与远距离相关的主机玉米和Medicago truncatula揭示了宿主特异性基因表达模式。相对于地上组织,基因家族不成比例地出现在界面上,包括转录因子富集和功能未知的基因。实时荧光定量PCR检测t .杂色的β-expansin在单子叶寄主应答中表现出强烈的差异上调(120x)z梅斯;结果与我们的读数估计很合一协调一致。病因相关的蛋白质,其他细胞壁修饰酶,以及具有未知功能的基因的直肠(如序列植物基因组的注释)是高度表达的寄生虫基因中t .杂色的在寄生-宿主界面。
结论
激光捕获显微解剖使在寄生虫宿主相互作用中心的细胞小区域取样成为可能。我们的分析结果表明t .杂色的一般主义的策略涉及依赖重叠但不同的基因套装,这取决于它寄生化的宿主植物。巨大上调的一个t .杂色的β-扩张蛋白提示了寄生在草上的成功机制。在这个界面转录组的初步研究中,我们已经证明t .杂色的,以及一般的Orobanchaceae,为鉴定功能未知的植物基因提供了极好的机会。
背景
大约1%的现存被子植物是寄生的,它们通过被称为吸器的特殊喂养结构从宿主植物中获得全部或部分的水分和营养。在含有寄生虫的科中,只有Orobanchaceae含有代表从潜在的自由生活兼性形式到非光合的专性寄生虫的全部寄生光谱的物种[1].林恩伯加斯是Orobanchaceae的一个非寄生系,是所有寄生物种的姐妹种[2];因此,该家庭代表了研究寄生派的演变的理想比较框架。非洲生长和地中海的寄生羚羊都包括毁灭性的农业害虫巫婆(斯特)和飞天扫帚(奥诺坎普和菲律宾人),分别。的斯特侵染占12350万英亩,导致年产损失大于70亿美元[3.,4].巨大威胁临近4000万英亩,尽管由于频繁放弃侵扰领域和关于产量损失的不可靠数据而难以评估难以评估[5].斯特是撒哈拉以南非洲农业的主要生物制约因素之一,受影响地区的规模正在扩大[6].杂草,寄生羚羊都威胁到亚洲,欧洲和北美的部分[7].
受一些寄生Orobanchaceae所带来的农艺威胁的驱使,TripHysaria Versicolor.已成为家庭的模型寄生厂。作为一个可转化的[8和易驯服的兼性多面手寄生虫,t .杂色的是研究寄生、吸器发育、植物-植物交流、宿主-寄生虫相互作用和寄生虫生物学许多其他方面的一个极好的物种[9,10.].为了发现在寄生植物生物学中重要的过程,我们将我们的分析重点分析了寄生植物的统一解剖学特征。这种改进的根结构适于使寄生物饲喂并且是寄生植物的独特,因此它是寄生虫和寄主之间相互作用的焦点[11.].
Heide-Jorgensen和Kuijt [12.,13.的吸器t .杂色的包含许多特殊的细胞,包括吸器毛,宿主和寄生虫之间的木质部桥梁,以及宿主-寄生虫界面毗邻导管元素的转移样细胞。尽管组织学证据表明木质部之间的吸器t .杂色的它的宿主有很好的记录[12.,13.,则没有韧皮部连通性的证据。然而,有证据表明,韧皮部可移动的病毒颗粒在全寄生虫中在宿主和寄生虫之间移动菲律宾人(syn。奥诺坎普)拉莫斯[14.],韧皮部连续性Orobanche crenata[15.].寄主韧皮部和寄生物韧皮部之间的运输机制可能不同于直接韧皮部连接的寄生虫[15.]通过采食性途径运输。在宿主和寄生虫之间的小RNA的双向运动已记录在其中T.versicolor攻击转基因生菜[16.].出血界面细胞的解剖学和对宿主寄生虫接口的双向传输的经验证据作为寄生虫对话的震中。
亲密的Symbioss倾向于专业化(例如寄生派)[17.].一个真正的通用策略,其中寄生虫常规地喂食许多远距离相关的宿主物种,在寄生生物中相对罕见[18.].从表面上看,这是令人惊讶的,因为广泛的宿主范围提供了更多的进食机会。例如,大多数寄生植物的幼苗在萌发后不久必须接触和寄生一个合适的寄主植物[10.]并进入更广泛的潜在宿主工厂增加生存的可能性,无论在附近生长的特定植物19.].虽然不像寄主植物专家那么常见,但许多寄生植物科确实包含通才,包括Orobanchaceae、Lauraceae、Convolvulaceae、Krameriaceae的部分或全部寄生成员,以及santtalales(檀香木、槲寄生及其亲属[20.])。
如果在特定主机资源可预测的情况下增加专业喂养策略的突变会增加频率[21.,那么与维持通才能力有关的特质可能会减少。如果一种通才策略涉及到一套通用基因的进化,这套基因对于成功寄生于广泛的宿主是必要的和足够的,那么这种趋势可能导致一种长期稳定的通才策略。或者,如果通才维持了针对不同宿主的不同的基因集,那么攻击不同宿主的基因集的长期维持可能不太可能,除非有来自不同宿主的频繁强化。
Triphysaria(orobanchaceae)是一种通用寄生虫,其在自然界中的高度多样化的Anviaperms收集,包括至少30种单码头和eudicot宿主植物中的17个家庭[22.].我们推断从吸器的转录组测序t .杂色的在远处相关宿主上生长将最大限度地识别识别基因表达的共享和宿主特异性模式的可能性。转录组数据集t .杂色的提供了利用寄生植物基因组计划(PPGP)新建立的基因组资源的独特机会。[23.),并有完善的功能协议,包括寄生虫-宿主共培养[9,24.,吸器诱导试验[25.,以及寄生虫转化[8,16.,26.].通过表征主寄生虫相互作用的分子签名,我们应该进入洞察过程中的过程中的过程,促进广泛的主机范围,并了解能够促进通用寄生虫策略的分子机制。
在表征的转录组时出现了两个实质性的障碍t .杂色的Haustoria。首先,在收获甚至收获微小的ukastorium(1-2毫米直径)时,稀释的稀释性细胞的基因表达谱变化杂色的。Heide-Jorgensen和Kuijt的优秀组织学和电子显微镜工作[12.,13.[揭示留在具有转移细胞形态的宿主寄生虫界面的细胞。这些专用细胞的解剖性包括致密细胞质,许多小型液泡,高度血管的细胞膜和迷宫细胞壁(用于评论,参见[27.])。我们假设,小的界面细胞集合,包括那些具有转移细胞样形态的细胞,促进了宿主和寄生虫之间难以捉摸的分子相互作用,使它们成为转录组分析的最佳候选细胞。第二个障碍是,如果没有测序和注释良好的基因组,就很难在全基因组范围内发现基因和随后的基因表达分析,而这正是目前所缺乏的t .杂色的.下一代测序(NGS)技术已经成为探索新基因组的强大工具,因为其每碱基的成本大大低于传统的染料终止子甚至焦磷酸测序(454)方法[28.].随着NGS革命的到来,一些数据分析工具应运而生。29.]),包括高性能德诺维转录组合体等三位一体[30.[已举起以促进在非特征模型系统中的转录组分析。
为了克服少数难以收获细胞的参考独立转录组分析的局限性,我们开发了用于样本寄生虫 - 宿主界面细胞的方法t .杂色的生长在远亲和序列模型宿主上玉米(b73)(单子叶)和Medicago truncatula(A17) (euicot)通过激光压力弹射显微解剖(LPCM)。我们提取了非常小的RNA样本,通过基于t7的线性扩增,然后对每个扩增的寄生虫-宿主界面转录组进行深度测序。我们收集了数百万对端Illumina读取新创,通过读取映射向每个组件进行估计的基因表达水平德诺维转录组组装。通过这种方法,我们识别出了属于宿主特异性反应的基因以及属于共同反应的基因t .杂色的给不同的主人。我们还验证了两者的宿主特异性差异表达模式Triphysaria扩展基因。扩张素是在Haustoria差异调节的少数基因中[31.,32.].扩展素基因的分析使我们验证界面序列数据中存在的差异基因表达模式,并证明了β-扩展素高度上调的第一种证据t .杂色的在成长时z梅斯主持人。我们的结果表明,维护一般喂养策略Triphysaria涉及推广和专门的基因反应,帮助我们了解Triphysaria的一般喂养能力。
结果
寄生虫宿主共同培养和微放置t .杂色的吸根
t .杂色的宿主在单独的培养板中萌发并长期生长。要开始共同文化,主持人被转移到新的盘子上t .杂色的加入并置于近距离(〜1mm)置于主管根部。附件率t .杂色的对寄主根的诱导率约为90%m . truncatula〜50%z梅斯。这种差异可能是由于更快的增长率z梅斯(相比m . truncatula)与共同培养的培养皿的限制尺寸相结合而不是差分寄生虫宿主相容性。在共同培养的早期阶段,宿主根部在琼脂生长培养基上保持或多或少地静止,附着率t .杂色的高(> 90%)和等同于之间z梅斯和m . truncatula。
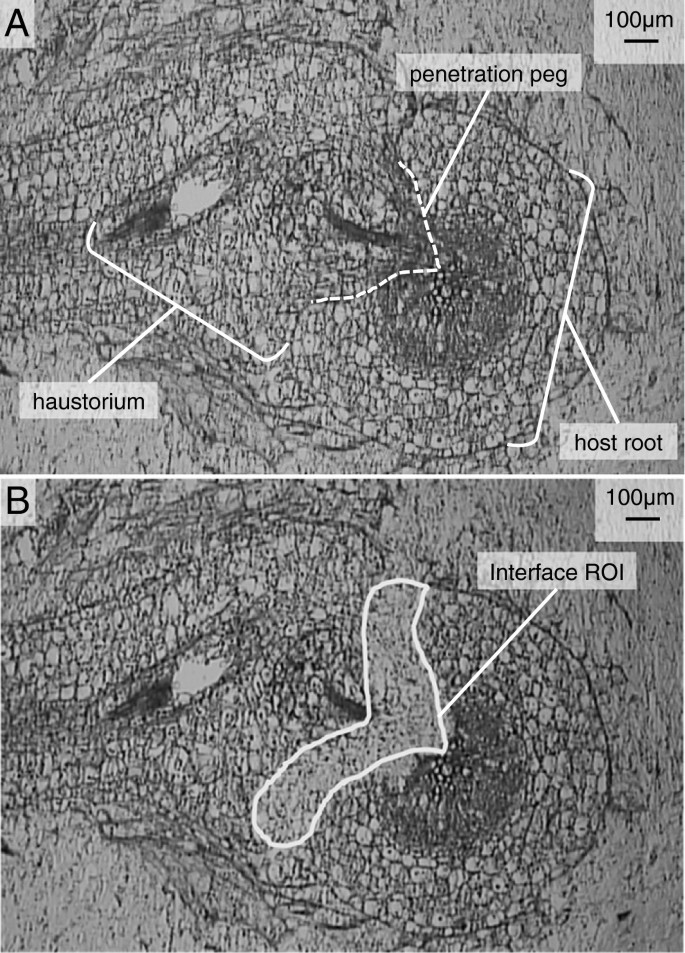
LPCM样品制备的第一步是在每份宿主上形成的Haustoria的分离和低温。通过从1μm截面厚度间隔从18-30μm切割的部分从切片的微观切割的样品统一地确定最佳部分厚度。为了t .杂色的吸器:我们确定25 μm冷冻切片是最理想的,可以有效地从涂有粘合剂的StarFrost™LPCM玻片中释放组织,并获得最大的组织收获量。在界面上相互接触的寄生虫和宿主细胞很难分离,所以为了确保捕获整个寄生虫界面细胞群,我们故意包含了少量宿主组织,知道宿主转录本可以被识别并被信息移除。图1显示(a)之前和(b)Lpcm之前的典型的横截面。
为了生成有代表性的界面细胞样本,我们从生物复制体(>8吸器)中收集了约110个感兴趣的界面区域(ROIs)。平均投资回报率t .杂色的接口转录组样品生长在内m . truncatula是54,910微米2总面积为610万μm2产生144 ng总RNA。该汇总样品具有7.6的RNA完整性数(rin),a a260./一种280.1.58和一个a260./一种230.0.76。平均投资回报率t .杂色的接口转录组样品生长在内z梅斯是56079μm2总面积为640万米2总RNA为160 ng, RIN为6.9,a a260./一种280.1.68和a a260./一种230.0.11。
激光显微解剖组织的线性mRNA扩增
基于T7的扩增的第一步是用寡聚-DT / T7 RNA引物/启动子合成cDNA合成。重要的是,该步骤非常有效,以最小化扩增样本中的较短片段长度的偏差[33.].我们经常在寄主寄生虫的界面上观察到一种黄褐色的物质,这可能导致界面RNA样品的初始纯度较低,即低a260./一种280.和一个260./一种230.比率。因此,我们用Zymo™RNA清洁和浓缩器试剂盒清洁界面总RNA。随后,我们观察到清洁界面总RNA的技术和生物学复制之间的一致扩增性能以及与阳性对照样品一致的性能A. Thaliana.高质量和纯度的幼叶RNA (28s/18s比:1.9;RIN: 8;一个260./一种280.: 2.0,260./一种230.: 2.1)。
扩增~100 ng总RNA,两轮扩增后常规扩增RNA (aRNA)为50-100 ug,与阳性对照(拟南芥young leaf total RNA), Message Amp™II aRNA试剂盒的预期性能,以及以前的报告[34.].aRNA产量单轮放大后100 ng,这是足够的图书馆建筑的Illumina公司测序,但我们选择放大样品对两轮自可取的额外aRNA基因表达谱进一步分析包括存在验证。通过Bioanalyzer™确定的片段长度谱从第一轮扩增减少到第二轮扩增,这与之前的报告一致[33.].
排序和装配统计
扩增的界面RNA样本在Illumina 's Genome Analyzer IIx上以83 × 83 bp的配对端循环方案进行测序。测序数据可在http://ppgp.huck.psu.edu[23.].的t .杂色的接口转录组数据集1)包含1790万读对z梅斯并且有1910万对阅读m . truncatula.从每个接口转录组数据集的主机读数被映射到其各自的主机基因组,导致移除150万m . truncatula阅读和0.4百万z梅斯从每个相应的转录组数据集读取。读取质量修剪和过滤(参见方法),留下> 2600万读取(孤儿和配对对),每个样品使用九百文分别组装(三位一体,[30.])和后处理以去除精确的重复或不可转换的序列。接口转录组合组件t .杂色的成长为z梅斯所得序列为12.77 Mbp,由28126个unigenes组成,N50为525 bp1).接口转录组合组件t .杂色的成长为m . truncatula所得序列为12.25 Mbp,由26709个unigenes组成,N50为536 bp1).所有类别的测序和装配统计数据相似(表1)表示两个数据集具有等同的质量。
Unigene注释
Unigenes使用PlantTribes 2.0数据库中已知植物基因的客观分类进行注释[35.,36.]如Wikeett等人所述。[37.].我们将基因分配成基因簇的层次,其包括近似基因家族(部落),以及潜在较窄的谱系(Orthogroups),其寻求代表参考植物物种的收集中的单一祖先基因的后代[35.- - - - - -37.].我们还将Unigenes从我们的实验中分类为使用BLAST到查询序列数据库(表 1).为了鉴定混合物种转录om中的宿主衍生的未成体,我们通过查询集合来建立95%的成对核苷酸同一性阈值z梅斯est序列为t .杂色的成长为m . truncatula和反之亦然(附加文件1:图S1)。的情节t .杂色的与互酷主机数据库查询相比,每个主机数据库的Unigene身份在95%下显然发散。要验证事件的高身份不是由于样本之间的交叉污染,我们还将主机EST数据库查询德诺维转录组的组装Lindenbergia Philippensis,orobanchaceae的非寄生成员[23.].爆炸身份图林恩伯加斯转录组显示出类似的趋势Triphysaria接口转录OMES针对相应的非主机数据库查询(附加文件1:图S1)。
为了除去在预装配读取映射期间可能已经过脱落检测的宿主基因,我们进一步基于与cDNA(植物血红型,[38.])和EST数据库(PlantGDB,[39.])。该筛选从其转录组中移除4967个unigenest .杂色的成长为z梅斯来自转录组的7,785个ungenenest .杂色的成长为m . truncatula(桌子 1).用于筛选RAW读取数据的相同参考转录物也用于筛选组件。大量推定主机派生的Unigenes表示,单独用Mosaik读取读取筛选不足以去除所有宿主污染。主机屏幕后,将筛选剩余的未成年粒t .杂色的基于序列相似性与其他PPGP文库中检测到的基因TripHysaria Versicolor.[23.].我们鉴定了17887个unigenest .杂色的成长为z梅斯14352个unigenes来自于t .杂色的成长为m . truncatula在核苷酸水平上具有> 95%的同一性t .杂色的来自其他PPGP库的基因。除了高相似性的unigenes之后t .杂色的unigenes来自PPGP数据库中其他组装的unigenes,余下的5272个unigenes在PPGP的界面转录组中t .杂色的成长为z梅斯和4572个unigenes在介面转录组t .杂色的成长为m . truncatula用于查询NCBI非冗余蛋白序列数据库(NR) [40使用BLASTx设置阈值e为1e-10。在每个界面转录组中,大约有一半剩余的单基因对包括模式物种在内的植物具有最佳的命中拟南芥,杨树,血管,其他Orobanchaceae,或> 30种其他植物物种(“其他植物命中”表 1).
每个t .杂色的接口转录组〜2300个未成熟,在任何上述外部数据库中没有明显对齐序列(表 1).我们采取了几个额外的步骤来试图识别这些未知序列。虽然这些unigenes未按来源进行分类,但我们通过Planttribes 2.0数据库的查询识别潜在的植物基因直脑〜20-25%的剩余unigenes。然后我们查询广泛的迭代(IPS)[41.]及OrthoMCL DB [42.]数据库具有剩余的剩余序列的翻译序列。这些unigenes中的大多数(每个转录组中> 75%)对orthomcl数据库中的基因缺乏显着的相似性,也没有含有IPS肽图案的基因(附加文件2:图S2);因此,它们被称为“无命中”unigenes(表1).通过对OrthoMCL数据库的扫描,鉴定出100个单株基因和7个非单株基因Zea.种植t .杂色的和85株植物和16名非植物unigenesMedicago种植t .杂色的(桌子 1).另外,370Zea.种植t .杂色的和310年Medicago种植t .杂色的unigenes含有IPS主题。大约一半的orthomcl db命中缺乏描述或具有最小的(例如一个字)描述(附加文件3.).在IPS搜索结果中存在类似的模式,其中大约一半的使用IPS图案包含仅推定的分泌信号和/或跨膜域(附加文件3.).总的来说,我们对每个组装中的unigenes进行了分类,结果发现了82%的潜在同源序列Zea.种植t .杂色的界面unigenes和88%的Medicago种植t .杂色的unigenes。我们能够确定>的假定起源,两个转录组中90%的unigenes和每个转录组中只有5%的unigenes未分类。其中,493个ungenes来自t .杂色的成长为Medicago装配和536个unigenest .杂色的成长为Zea.组装长于300 BP,并具有读取支持。
接口转录组t .杂色的成长为z梅斯和m . truncatula共包含127个和329个unigenes,其中对非植物物种的影响最大(表1)1).每个接口转录组的非工厂组件包括两个接口库共享的最佳点击率为16个分类群。这些包括在内埃斯克里希亚洲,曲霉菌,clavispora.,洋葱, 和别的。在这套中,洋葱在两个界面转录om的非植物组分中,是最高度代表的分类群(> 20倍)在任何其他物种中的增加。这些界面洋葱在参考转录组中未检测到序列(TRVEBC1,序列同一性截止值> 90%)。
剩余的“无击中”序列,尤其是具有读取支持的300bp,可以代表未经发布的宿主或寄生虫基因,无表称相关的共生或偶然污染。所有剩余的未成年人未分配给NR数据库中的主机工厂或非工厂源(23,032t .杂色的在z梅斯和18595年t .杂色的在m . truncatula)被统称为“推定”t .杂色的衍生的unigenes。“
比较界面转录组简档t .杂色的
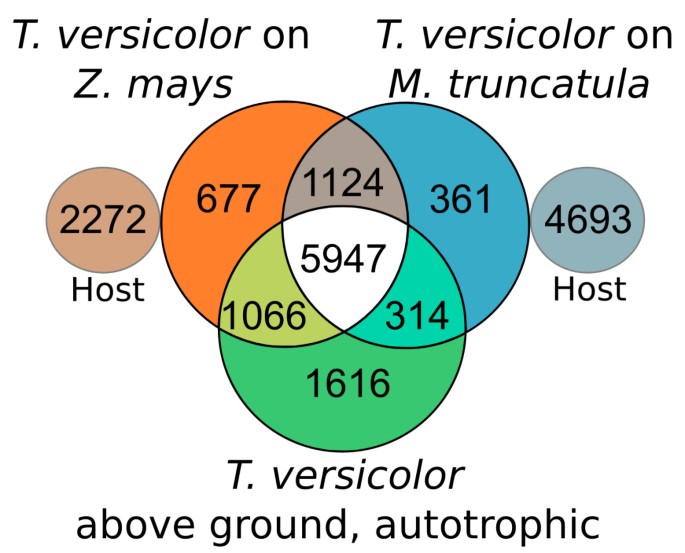
检查的概况t .杂色的界面转录组,我们使用来自自养生长的地上组织的注释转录组t .杂色的作为参考([23.]Triphysaria装配trvesbc1)。我们通过(PlantTribes 2.0)OrthoGroups对unigenes进行了分类,以确定接口转录组的主机特定和共享组件t .杂色的成长为z梅斯和m . truncatula(图2).正如预期的那样,最大数量的正交群(5947,或在占所述总量的53.6%)t .杂色的)在所有三种转录组之间进行共享,并且可能代表参与各种细胞类型共同的方法的基因的表达。在接口转录组之间共享大量的正交组(1124)t .杂色的与两个主机进行交互。这些基因可能包括一种寄生核寄生虫基因,其无论宿主植物种类如何都有活性。许多额外的OrthoGroups都是界面和特定于主机的(677 forz梅斯和361 for.m . truncatula)或与成长的地面阶段分享(1066z梅斯和314 for.m . truncatula).
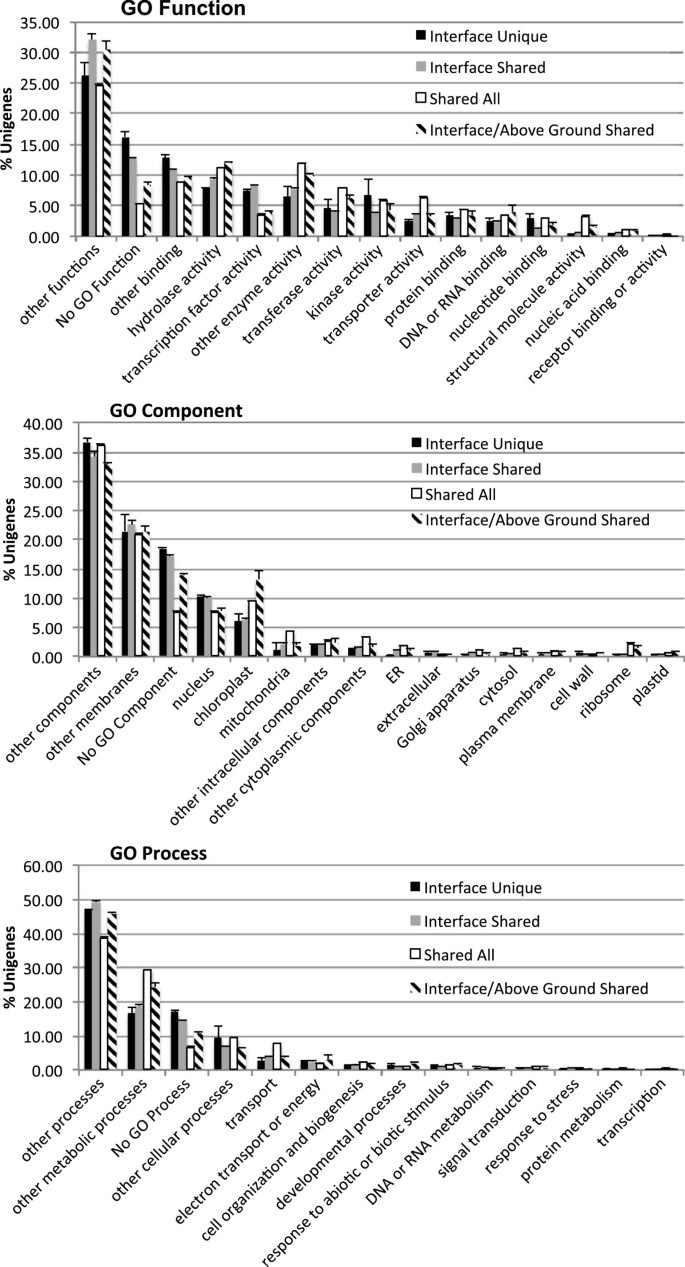
我们的注释策略包括从Planttribes 2.0中获得最佳爆炸的Go Slim类别术语分配。Go Slim类别是最广泛的Go标准,可用于转录组的比较。特定于主体的组成部分t .杂色的界面转录组可能含有与宿主生物学的独特方面相互作用的基因,而分享的那些可能含有寄生寄生基因的基因。为了确定注释轮廓在重叠和独特的转录组件之间类似,我们绘制了未经成根类的比例(图 3.),由图中唯一或重叠的正群表示2.Go Slim类别概况在每个接口转录组的等效组件之间的简档通常彼此相似,但通常与每个接口转录组的非等效组件的简档不同(图 3.).例如,在两个界面转录组之间,界面唯一的Orthogroup谱是相似的,但不同于地面上的两个界面转录组的Orthogroup谱。
go slim类别摘要。在接口转录om中GO SLIM类别的未成语条款t .杂色的和地面上的参考组件杂色的。每个系列显示等效转录组件中的平均未成语数量,具有给定的GO Slim术语。例如,“界面唯一”表示两个来自界面唯一组件的平均unigenes的数目Medicago和Zea.种植t .杂色的转录组。误差棒是标准的平均误差。“Interface Unique”=来自宿主和界面特异性正交组的unigenes,“Interface Shared”=来自界面特异性正交组的unigenes,并在界面转录组之间共享,“Shared All”=来自交互转录组和上面的转录组之间共享的unigenes,“界面/地面上共享”=来自地面上、自食性转录组和宿主特异性界面转录组之间共享的正交群的unigenes。
每个界面转录组内重叠的和独特的正交组中的Slim类别概况通过卡方检验进行比例性检验(附加文件)4:图S3A-F)。6项试验结果均显示出歧化且极显著(P< 0.0001)。每个GO Slim类别中具有强残差值(强正或强负,因此不成比例)的unigenes的数量在附加文件中显示4:图S3A-F。该分析的结果与Go Slim类别曲线的曲线相辅调(图 3.).最引人注目的结果是缺少界面唯一的Orthogroups中缺乏Go Slim类别的Unigenes的始终如一的强大过度表示(附加文件4:图S3A-C:列A和B,图S3D-F:列E和F)与缺少共享Orthogroups中缺少Go Slim类别的unigenes的一致弱表示相比(附加文件4: S3A-C:柱D,图S3D-F:柱H)t .杂色的两个主机都是通过“转录因子活动”的超级级别的超薄函数术语(附加文件4:图S3A:B栏和3D:第F列和“传输”Go Slim Process术语(附加文件)4:图3.:B栏和3F:第F)。这表明存在在上述地基参考转录组中不表达的寄生虫宿主界面处有活性的转录因子基因。相反,在界面表达的转运蛋白基因包括在上述地面参考转录组中表达。
在宿主寄生虫接口处高表达基因
使用a的优点德诺维组装的RNA-Seq转录组是转录本检测的内在阈值。如果文字记录被充分读取德诺维组装,存在目标德诺维RNA-SEQ是表征的证据。参考组件TRVEBC2 [23.包括吸器的数据t .杂色的成长为m . truncatula并用作对来自每个接口转录组的地图读取的引用。我们将归一化读取(读取/千级读数/百万读数/百万拍卖读取(RPKM))从属于接口转录OMES和上述地面参考转录组之间共享的Orthogroups,Trvesbc1(附加文件5:图S4)。对于在两个界面转录组中检测到的unigenes,相关性很高(Pearson’s R= 0.81),这表明界面转录组之间的技术和生物变异性很低。
要确定每个UNIGENE的表达式,我们还将映射到每个相应的接口读取德诺维组装。在两个转录组的每组共享和独特的正交群中,20个最高表达的ungenes列于附加文件中6:数字S5A-C。我们使用Blastx(E-Value阈值:1E-10)和17个注释的植物基因组来查询这组120个未通过NR数据库使用Blastn和Blastx。使用BLAST的数据库查询的结果在附加文件中呈现6:数字S5A-C。来自NR中搜索的最佳点击描述与使用Planttribes 2.0分配的注释协调(请参阅爆炸导致其他文件7和8).我们还使用Transocan来预测额外文件中列出的未在附加文件中列出的unigenes的信号肽和跨膜域6:图5A-C(附加文件9和10.).motif预测工具经常在注释为转运蛋白的unigenes中识别假定跨膜结构域,在注释为分泌蛋白的unigenes中识别分泌信号。列出的120个unigenes(附加文件6(图S5A-C), 9个unigene在NR查询时没有命中,其余unigene对植物物种的命中最好。在这120个单基因中,约有30%在NR中没有BLAST命中,或与预测的、假设的或其他未特征的序列相匹配。这一结果与我们的发现一致,即缺少GO Slim类别赋值的unigenes丰富了界面(因此是功能性注释)。
在界面样本的共享正交组中最高表型的基因中t .杂色的在两者中生长z梅斯和m . truncatula(附加文件6图S5A)是一个β-扩展蛋白基因(见下文),几个其他细胞壁修饰酶的基因,和一个编码推测ap2-erf域转录因子的基因。在共享界面正交群中一个显著的模式是10个unigenes(包括来自t .杂色的成长为Medicago)在其他edicot物种中具有序列同一性与带注释的发病机制相关蛋白质。一个单一的m . truncatulaunigene在公共界面组分(ID 5537)中通过去除宿主植物过程;这也与致病相关蛋白具有高序列一致性。
在附加文件中列出的unigenes6:图S5A-C,42来自接口转录组t .杂色的成长为z梅斯34个来自于t .杂色的成长为m . truncatula对分发性基因组有最强的爆炸点击(包括Mimulus guttatus).当我们询问17个植物基因组数据库时,豆类中有稍微更好地击中Medicago种植Triphysaria数据集,可能是因为双二核苷酸之间的序列差异较小Triphysaria和Medicago而不是在远亲之间Triphysaria和玉蜀黍属。这导致宿主和寄生虫之间的略微更广泛的模糊序列同一性。尽管过滤了严格的过滤,但一个占有的z梅斯文字记录和三个假设m . truncatula在附加文件的高表达基因列表中,转录本保持不变(用粗体表示)6:图S5。
一种新的β-扩展蛋白在寄主-寄主界面上差异表达
在介面转录组中观察到的高表达unigenest .杂色的成长为z梅斯是推定的β-expansin(附加文件6:图S5A)。读取映射数据的手动策展表明它在成长时高度表达z梅斯,以及来自的几乎相同的Unigenem . truncatula -种植Triphysaria界面略低表示。这显然是宿主特异性的基因表达模式感兴趣,因为膨胀蛋白是细胞壁松动蛋白(如审查,见[43.])这一点涉及寄生植物与其宿主之间的相互作用[31.,32.].虽然在两个样品中表达β-扩展蛋白基因,但读取映射证据表明该基因(Unigene 772)高度差异表达。作为比较的点,我们研究了一个推定的α-扩展蛋白,Unigene 11,其在界面转录组中显示出高表达的互易型模式t .杂色的成长为m . truncatula。我们通过从界面ARNA扩增的PCR产物的染料 - 终止子测序验证了未终原772和11的核苷酸序列。
β-expansin unigene 772的系统发育分析显示,它嵌套在双子叶β-expansin序列支持的分支中(附加文件)11.图S6A)表明unigene 772是双子叶β-扩展素,而不是az梅斯衍生序列。通过Interprocan注释支持772的扩展Identity(附加文件9)并显示推定的5'信号肽(附加文件6图S5A),与膨化蛋白在质外体中的作用一致。此外,所有BLAST搜索的结果表明unigene 772是at .杂色的衍生序列。UNIGENE 11的系统发育证据不会产生α-expansionins的良好分辨的树(附加文件11.:图S6B),但BLAST结果表明,已知或假定(如est)的成对核苷酸身份m . truncatula基因<70%,而Unigene 11具有高同型(195%的成对核苷酸同一性)Triphysaria在其他PPGP组件中的未成年体。
定量实时PCR核查宿主特异性扩展蛋白表达
我们试图通过QRT-PCR验证这两种扩展蛋白的互核表达模式。unigenes 772和11分别分配正式名称TVEXPB1和TVEXPA4。我们通过熔化曲线分析验证了引物特异于其靶标。为了进一步验证TVEXPB1,TVEXPA4和TVACTIN引物对寄生虫转录物特异,我们收获了立即邻近成熟的主体根部的部分t .杂色的并通过qRT-PCR对其进行检测。在这些宿主根样本中,我们能够检测到ZmActinz梅斯根样本和mtactinm . truncatula样品,而寄生虫引物产生与背景一致的信号。
我们询问了生物复制t .杂色的寄主-寄主界面细胞在两者上生长z梅斯和m . truncatula通过qRT-PCR检测内参基因TvActin、TvEXPA4、TvEXPB1的表达(图1)4).TvEXPB1上调>120倍(P=0.024)t .杂色的调整的界面界面细胞z梅斯关系到t .杂色的成长为m . truncatula。TVEXPA4显示较弱的互易模式(P = 0.17)。tvexpa4和tvexpb1的表达式模式与我们的协调德诺维RNA-Seq结果。此外,当我们检测整个吸器样本中TvEXPB1的表达时,信号与背景信号难以区分,这表明TvEXBP1的大量上调是特定于少数界面细胞的。
讨论
使用允许我们取样、排序和德诺维在宿主-寄生虫界面组装细胞转录组,我们已经证明了这一点t .杂色的以宿主特有的方式表达基因。对寄生植物-寄主植物界面基因表达的初步研究表明,通用性寄生的基础至少部分是由寄主特定的基因表达模式构成的。总的来说,这项工作显示了发现基因的潜力德诺维并以高度组织特异性的方式研究基因表达的基因组表达模式,其在缺乏测序基因组的生物体中。
激光微生物(LM)是植物转录组织的强大工具
全面了解任何生物系统的能力在于理解复杂器官和组织中进行的无数过程。揭示这一图像的一个主要障碍是识别、分离、捕获和分析感兴趣的组织和细胞的能力。一些作者强调了组织和细胞特异性基因表达的高分辨率、高通量研究的重要性,以及以全球方式调查基因表达的必要性,以及为什么LM(包括LPCM)正在成为基因组学的强大工具[44.- - - - - -47.].
LM从一些模型系统中生成的样本已经使用微阵列进行了检测[48.- - - - - -53.]允许调查人员分析特定组织和细胞类型中的全球基因表达模式。最近,LM越来越多地与NGS汇集以序列各种组织的转录组z梅斯[54.- - - - - -56.] 和S. lycopersicum.[34.].出现德诺维转录组组件现在使非测量生物的特定组织和细胞中基因表达的全局调查成为逻辑下一步。检查寄生虫 - 主机接口转录组t .杂色的,我们将LPCM与基于鲁棒T7的线性RNA扩增组合[33.,57.- - - - - -60.[音乐会与illumina mrna-seq,高性能德诺维转录组组件[30.]以及各种装配后处理工具.
对于储质的t .杂色的我们的取样策略主要基于详细的组织学和透射电子显微镜对野外采集标本的研究[12.,13.].这一关键的背景信息使我们能够识别吸器内感兴趣的细胞,然后在冷冻切片中识别吸器内包含这些细胞的区域。植物组织必须在LPCM之前被包埋,准备步骤可能会影响RNA制备的数量和质量[61.].然而,必须通过下游可用性来识别感兴趣组织的能力。该部分的组织学质量是可以确定样品制备方法的重要考虑因素。石蜡嵌入部分通常以牺牲RNA质量和产量为代价提供高的组织学质量[62.,63.].为了在更细的空间分辨率下取样,组织学质量随着更薄的切片而增加,压力弹射效率随着更薄的切片而增加,然而Kerk等[64.报告称,较厚的切片增加了RNA产量。我们发现,最佳的切片厚度,以捕获界面细胞t .杂色的吸器直径20 ~ 25 μm。这是基于我们对组织和细胞的组织学识别能力与LPCM压力弹射阶段有效组织释放的平衡而确定的。易受瞬时冷冻损伤的植物组织的完整性可以通过注入冷冻保护剂来保存[65.].使用Cryojane™(冷冻转移系统)冷冻呼吸允许我们容易地捕获连续冷冻冻干t .杂色的从精心挑选的样品常规地产生高质量的RNA的Hautroium。
通过设计,我们的采样策略最小化了从时间或空间采样伪影产生基因表达的差异的可能性。共同文化t .杂色的不是高度同步,所以我们的Haustoria样本代表了一个大约8-10天的连接的广泛的连接窗口。我们收集了〜110界面ROI,减少了空间采样伪影的可能性。此外,在整个样本处理和数据分析中具有高度相似的统计数据,包括共享Orthogroups中未成语的读取计数的高相关(附加文件5:图S4),并在额外的生物复制实验中验证TvEXPB1的表达模式(图4),表示从生物或技术来源的低变异。
寄生虫-宿主界面转录组有助于识别寄生虫的核心基因
orobanchaceae包括有害杂草斯特,奥诺坎普和菲律宾人以及模型寄生虫Triphysaria。我们推理通过发现可能包括这些响应核心的基因子集t .杂色的为了远方相关的主机,我们将更接近识别在Weedy Orobanchaceae中运行的寄生基因的核心集。寄生植物的分子对话与其宿主在很大程度上[66.].在这里,我们报道了在宿主-寄生虫界面表达的基因子集的鉴定t .杂色的对两个远亲主人的反应z梅斯和m . truncatula.此外,我们还鉴定了宿主特异的基因表达模式,表明通才寄生植物t .杂色的维持基因套件,以供不同的主机使用。
基因t .杂色的在主机 - 寄生虫接口上表达,响应两者z梅斯和m . truncatula包括一组在其他植物物种中被注释为致病机理(或病原体反应)相关蛋白的大量基因。这包括6个高表达基因Triphysaria界面长大m . truncatula和三个在成长时z梅斯但是,许多额外的unigenes也是在病原体侵袭期间上调的基因的诱发同源物(脱脂,酸性的内奇素酶,疾病抵抗蛋白等)。虽然预期这些课程的基因的上调将作为对病原体的防御反应,但该观察结果表明,寄生虫在宿主入侵过程中也接通了植物保护的途径。已经提出,寄生植物可能征收来自异质信号转导期间使用的等等化学解毒的途径[67.].无论Triphysaria在具有病原体抵抗(PR)基因的入侵过程中捍卫自己,或者招募了PR途径,因为令人反感的“武器”尚不清楚,但它似乎一部分核心基因设置Triphysaria寄生宿主衍生自植物用于防御病原体的途径。
对于为什么真正的通用寄生虫发生或持续存在,甚至不经常,尽管有证据表明专业化趋势([18.]并参考其中)。我们假设成功的一般喂养策略可能严重依赖于部署任何宿主供饲喂的通用基因。相反,我们看到界面转录组的大组件仅在喂食其中一个测试的主机植物时被检测到。在仅在界面转录om中检测到的2162 rOthogroups,在两个宿主物种上表达约一半(52%),但只有在玉米种植(31%)或on的生长时,才检测到大部分界面正交组。Medicago(17%)。广泛的遗传机械的长期维护,朝向可能的宿主子集的寄生机会将需要维持以宿主特异性方式部署的基因上的选择性压力。Unless the parasite routinely encounters and parasitizes a wide range of host plants, selective pressure on some host-specific genes will be relaxed and the gene functions eventually lost, limiting the plant’s potential to parasitize some hosts, despite the potential advantages of a large host range. The evidence for host specific gene expression suggests thatt .杂色的定期寄生在广泛的寄主范围内,对以寄主特定方式使用的基因保持选择性压力。事实上,在宿主种群在空间和时间上具有适度可变的情况下,广泛的宿主范围的优势已经被探索过[19.].本研究作为对推定和潜在主人进行更广泛的调查的起点Triphysaria以及首先看看寄生植物Haustoria中高度专业化组织的基因表达谱。
有趣的是,与上面的转录组相比,宿主特异性和共享界面基因家族(Orthogroups)中没有GO Slim类别术语分配的unigenes过多。因为正群是根据已测序和注释的植物基因组分类定义的[35.- - - - - -37.,这一观察结果突出了这样一个事实,即吸器中表达的基因包括许多来自于基因子集的基因,而这些基因的功能在任何植物中都尚不清楚。此外,来自每个界面转录组的大约1300个unigenes与任何已知序列缺乏强同源性,尽管25%的unigenes是来自每个宿主的界面数据集的高同源互反最佳匹配。此外,由于这种模式在介面转录组中是可复制的t .杂色的当在不同的宿主植物上生长时,这些数据表明未知功能的基因以特定于宿主的方式在宿主中以宿主表达。我们的数据还表明,地下阶段的增长t .杂色的用于未知功能的基因。
在那些具有GO Slim类别分配的unigenes中,GO Slim Function类别“转录因子”的共享接口特定的Orthogroups代表过多,而GO Slim Process类别“运输”的代表不足。这表明,界面上存在独特的转录因子正交群(相对于地面上的参考转录组),但参与运输过程的正交群在所有三个转录组中都是活跃的。关于转运的后一种观察并不排除本研究中在所有三个转录组中表达的特定基因的差异表达。
TvEXPB1编码一个t .杂色的β-扩展蛋白是特定于宿主的反应的一部分
扩展蛋白基因家族包括4个主要群体:α、β、扩展蛋白样A和扩展蛋白样B [43.].通过允许细胞壁聚合物的滑动来思考扩展蛋白旨在松开植物细胞壁[43.].它们的活性是非酶的,但它们与糖苷水解酶家族蛋白有很大的关系[43.].扩张蛋白与细胞生长有关[68.],并与植物病原菌的相互作用有关[69.,70,植物寄生线虫[71.,72.]和寄生植物[31.,32.]与他们的植物主持人。在每种情况下,都建议扩展签到宿主入侵中的作用。
在单码本的细胞壁中已经测定了扩展素活动[73.- - - - - -75.]和dicots [76.,77.].β-扩展蛋白具有特异于草的细胞壁的活性,但不是单萝卜或大多数其他单子叶细胞壁[78.].α-expansin的作用模式相反,这表明α-和β-expansin蛋白都具有底物特异性[43.,79.].通常,在低浓度下发现扩展蛋白[43.除草花粉除草量分泌大量的β-膨胀蛋白[76.在花粉管生长过程中可能用来松弛花柱组织的物质[80,81.].虽然确切的动作机制仍然是未知的,但是草细胞壁具有相对少量的木葡聚糖和果胶;这些被β-(1→3),(1→4)-D-葡聚糖和葡萄糖醛酸诺氧基氧基替换。这两种草细胞壁组分都是其壁宽松活性中β-膨胀蛋白的潜在靶标[82.].在十多年的研究中,认为β-膨胀蛋白的积累被认为是仅对草花粉的特异性[82.].
在这项研究中,我们已经表明,β-扩展蛋白TVEXPB1的转录物水平是界面转录组中最高表达的基因之一t .杂色的成长为z梅斯.相对于的界面转录组t .杂色的成长为m . truncatulaTvEXPB1在寄主-寄主界面组织中表达显著上调(>120倍)t .杂色的成长为z梅斯。TvEXPB1的大量和宿主特异性表达提示了双子叶寄生虫与草宿主相互作用的作用。此外,TvEXPB1在全吸器样品中的信号t .杂色的成长为z梅斯表明TvEXPB1对宿主-寄生虫界面具有高度特异性。
携带的β-膨胀素的草细胞壁特异性活性的证据以及寄生铁矿 - 宿主界面在寄生虫β-扩展蛋白的大规模上调表明t .杂色的与单子台主机交互时表达TVEXPB1z梅斯努力操纵宿主墙壁。海德·贾格森和奎德[12.,13.]观察到寄主的皮层和表皮细胞在吸器内的寄主界面处似乎被压碎和移位杂色的。宿主组织位移的机制可包括细胞壁改性蛋白质,如具有宿主细胞壁的膨胀菌素的膨胀蛋白。这种机制将允许寄生虫软化或分离宿主壁,而不会影响其在穿透式胰岛素中的其自身细胞壁的完整性。扩展蛋白在宿主寄生虫相互作用中发挥的具体作用只会通过详细的功能分析来揭示。这包括重点的基因表达分析,靶向沉默t .杂色的扩展基因和生物化学表征,以确定这些基因编码的扩展蛋白的底物特异性。
是一个普遍存在的土壤细菌属的共生t .杂色的吗?
非植物转录组组分中最好代表的属(〜60%的非植物击中t .杂色的成长为z梅斯约45%的非植物进入t .杂色的成长为M. Truncatula)曾是伯克德利亚,一种常见的土壤细菌属。这种细菌的高频率令人惊讶,原因有三:(1)其他非植物属的相对频率要低得多,(2)t .杂色的在官方共同培养之前,种子在阳极培养之前积极灭菌,(3)伯克德利亚 -在地面参考装配中未检测到衍生的unigenes.其他属(包括植物病原真菌、人类和其他细菌)感染的低频率可以解释为来自实验室环境的偶然污染,然而洋葱仅在界面样本中击中表明存在属于共培养系统中该属的生物体。unigenes通常是<1kbp(表明转录尺寸的未成根)和RNA样品是DNA酶处理的,降低这些unigenes源自基因组DNA污染的可能性。
属洋葱在过去二十年中,由于多样化的宿主相互作用,从人病原体到植物生长的不同目录,促进了根际动物的植物生长83.].这里特别有趣的是它们作为有益植物内共生体的潜在作用[83.].Attsat [84.]注意到在出血细胞中存在丝状细菌的结构,并且灌溉霉素的应用显着降低了海芥末形成Orthocarpus purpurascens(syn。Castilleja exserta),一种半寄生虫,是t .杂色的[85.].存在洋葱在轴静脉共同培养系统中,指向有趣的可能性:一种物种洋葱通过与种子的表面灭菌进行杂色的。提示原核内共生体在寄生植物生物学中作用的证据[84.与存在一种有益的土壤细菌的证据相结合,暗示了两者之间的共生关系洋葱和Triphysaria。存在和可能的角色洋葱在寄主-寄主界面t .杂色的目前正在调查中。
方法
植物材料的生长
TripHysaria Versicolor.
种子是从生长在加州纳帕实验室(加州大学戴维斯分校,美国)沿海草原的数百种开放授粉的植物中收集的,这些植物与我们之前的转录组测序研究相同来源的种群[37.,87.].将种子在70%乙醇中灭菌10分钟。轻轻摇动,然后用无菌蒸馏水洗涤3倍2种子用50%漂白剂+ 0.01% Triton X-100 (Sigma)溶液进一步消毒并划痕30分钟,同时轻轻摇晃。种子用无菌蒸馏水洗涤10次2o并置于含有共培养基的培养皿(1 / 4X Hoagland的基础盐和营养混合物,7.5g / L蔗糖,6g / L植物组织培养级琼脂糖,pH为6.1)并用Parafilm™包裹。在黑暗中,在4℃下将种子分层4天,然后在12小时的光线状态下转移到16℃的生长室,光强度为30μmols/ m2/秒。t .杂色的幼苗生长14-17天,然后转移到与寄主共培养的新鲜平板上。
Medicago Truncatula(A17)
种子是由曾玉王(俄罗斯俄克拉荷马州,美国俄罗斯州贵族基金会)慷慨提供。通过在18mh H中偶尔孵育来孵育种子2所以48分钟后,用无菌蒸馏水冲洗5次2种子表面用50%漂白剂+ 0.01% Triton X-100 (Sigma)洗涤3分钟,同时轻轻摇晃。种子用无菌蒸馏水洗涤10次2O,置于50ml锥形底管中,250ml无菌蒸馏H2o在25°C,在黑暗中,在轻微摇晃的同时。将第二天幼苗转移到无菌滤纸(Whatman#5)中,用无菌蒸馏H湿润2o,并放在培养皿中。幼苗在25℃下置于25℃,在黑暗中,过夜。将幼苗转移到共培养基中,然后用Parafilm TM包裹,然后在25℃下置于生长室,在16小时的光线状态下,光强度为100μmols/ m2/秒培养5-7天,然后转移到新鲜的寄生虫共培养板上。
Zea Mays(B73)
种子由David Braun(密苏里大学,哥伦比亚,美国)慷慨提供。种子表面用20%漂白剂+0.08% Triton X-100 (Sigma)洗涤10分钟,同时轻轻摇晃。种子用无菌蒸馏水冲洗3x2种子进一步用70%乙醇清洗表面消毒5分钟,同时轻轻摇晃。将乙醇倒出,种子在层流罩中晾干。干燥的种子放置在含有共培养介质的培养皿中,并用parafiltm包好,然后放置在25°C、16小时光照条件下的生长室中,光照强度为100 μmol光子/m2/秒培养5-7天,然后转移到新鲜的寄生虫共培养板上。
寄生虫和宿主共同文化
玉米或Medicago truncatula在上述时间转移到新鲜的共培养基中。主持人根本定向,以允许放置t .杂色的(如上所述生长)离寄主根很近,寄生根尖离寄主根0.5-1毫米。Co-culture plates were sealed with Micropore™(3M) surgical tape and placed in a growth chamber at 25°C under a 16-hour light regime with a light intensity of 100 μmoles photons/m2/秒8-10天。
组织处理和测序的样品制备
组织收获
8-10天共同培养,Haustoriat .杂色的通过在立体显微镜下与宿主根系和寄生虫根部〜1-2mm的切割进行切割,并嵌入在10 mm x 10 mm x 5 mm cryomold™中的Shandon Cryomatrix™(Thermo Scientific)中嵌入组织tek)。解剖组织在垂直轴上用宿主根系迅速定向,并且与模具底部的所有Haustoria等距离。在干冰上迅速冷冻样品并储存在-80℃。
低温和脱水
用Cryotome SME (ThermoFisher Scientific)在20-25 μm处对埋入的吸器进行切片。冷冻切片使用Cryo-Jane™磁带传输系统(徕卡微系统)安装到载玻片上。安装的切片在一系列有机溶剂浴中脱水(RT 70%乙醇(10分钟),4℃70%乙醇(2分钟),4℃95%乙醇(2分钟),4℃100%乙醇(2分钟),4℃100%二甲苯(2分钟),4℃100%二甲苯(2分钟),最后RT 100%二甲苯(2分钟))。载玻片在通风柜中干燥30分钟。
激光压力弹射显微解剖(LPCM)
P.A.L.M.微束™系统(蔡司)用于收集感兴趣的细胞。仅使用压力弹射器功能,最大限度地减少了压力弹射器功能的激光能量设定。激光聚焦是固定的,但在LPCM过程中需要手动调整客观聚焦以确保有效去除感兴趣的组织。解剖组织用不透明的胶帽0.5 mL管捕获(蔡司,第415101-4400-250部分)并在-80°C保存。
RNA提取和RNA清理
使用Picopure™RNA分离试剂盒(Arcturus)从LPCM收获材料中提取总RNA(Arcturus),部分,部分,从伴随不透明的粘合剂帽0.5ml管(Zeiss,零件#415101-4400-250)。变化包括在Arcturus方案中详述的RNA提取步骤的改性(步骤1),如下:我们取下了粘合剂帽并将其放入了0.5ml EPPendorf Safe-Lock™管(部分#022363719的管体)为了实现更好的管帽密封。将50μl缓冲液Xb加入管中,涡旋倒置30秒。然后将管置于温度平衡(42℃)定制夹紧装置(未示出)中以防止裂解步骤中的泄漏,其中管被反转并在42℃下温育30分钟。在空气孵化器中。在10分钟。间隔含有管的整个夹紧装置被涡旋倒置30秒。在适应裂解步骤之后,我们在步骤2,RNA分离开始时遵循Picopure™RNA分离试剂盒协议。根据Picopure™RNA分离试剂盒协议的附录A,RNA是DNA酶的柱子处理。使用RNA 6000纳米试剂盒(Agilent)在植物总RNA测定中对Agilent生物分析仪进行评估总RNA,具有特异性关注RNA完整性数(rin,1(降解)至10(完整))[88.].我们需要对总RNA prep进行额外的清理,以去除我们怀疑是干扰下游酶处理的多酚和次级代谢物。RNA Clean and Concentrator™-5试剂盒(Zymo Research)用于清洁和浓缩从LPCM收集的样品中提取的总RNA,遵循协议版本2.0.6中的一般程序,RNA用10 μL RNase-free水洗脱。
基于T7的mRNA的RNA扩增
消息AMP™II ARNA套件(AMALION)用于扩增总RNA样品中含有的多RNA,得到扩增的RNA(ARNA)。输入量约为100ng总RNA。制造商的说明随后和第一轮在体外允许转录(IVT)进行14小时。将整个ArNA样品在速度 - VAC(保密)中浓缩至<10μl然后进入第二轮cDNA合成。第二轮IVT被允许进展4小时。使用具有mRNA测定的RNA 6000纳米试剂盒(Agilent)在沉肠生物分析仪上评估总RNA。第一轮扩增后的典型产量高达100ng arna,并且在第二轮扩增后的产量范围为50-100μg。优质拟南芥幼叶RNA用作阳性对照和RT-PCR级水(Ambion,包括ampii试剂盒包含的ambion)作为扩增的阴性对照。
Illumina成对的终端图书馆建设
在Illumina的mRNA Seq文库准备协议(Rev D)中使用aRNA作为输入。我们省略了poly-A选择步骤,直接移动到“片段RNA”。“100ng的aRNA被片段化,其余的库制备均遵循制造商的说明,但有以下例外:1)我们在“纯化cDNA模板”步骤中选择了300 bp而不是200 bp的适配器连接文库片段。2)我们进行了第二个大小选择/纯化步骤,以“纯化cDNA模板”步骤中描述的类似方式运行凝胶,并在大约325 bp处切除包含文库富集产物的条带。第二次大小选择是为了纯化文库,并进一步限制Illumina推荐的配对端mRNA Seq片段的分布。Illumina RNA Seq文库在安捷伦生物分析仪上使用DNA 1000试剂盒(安捷伦)进行评估。
测序、生物信息学和系统发育学
测序
在VA USA夏洛斯维尔的弗吉尼亚大学基因组学核心设施对Illumina基因组分析仪进行配对(83×83bp)测序。每个库在一条车道中测序。
柱上测序数据处理和注释
通过与注释的编码DNA序列比对,将污染序列从预组装的配对端读取Medicago truncatula[38.] 和玉米[38.使用版本1.1.0014的Mosaik Assembler [89.]推荐参数(HS = 15,mm = 12,和ACT = 35)。然后修剪未对准的读取以使用CLC组装单元3.2的质量调整程序从结束中移除低质量的基础(
后注释组件过滤
为了确定适当的成对身份(在核苷酸水平)以参考宿主序列,用于确定未来成对频率的频率对参考宿主EST(玉米和Medicago truncatula, mRNA est [39.])。界面转录omt .杂色的成长为z梅斯和m . truncatula被爆炸到两者中z梅斯和m . truncatulaEST数据库。对非宿主数据库的BLAST是为了确定不应该出现在汇编中的一组宿主参考的单基因的事件核苷酸成对身份。为了确定对宿主互反序列具有>95%同源性的ungene是否由于交叉污染,我们对整个植物标准化装配(Orobanchaceae的一个非寄生成员,Lindenbergia philippensis(PPGP组装Liphgnb1,[23.])。建立了95%的同一性阈值,以最小化宿主污染,同时将寄生虫转录物与寄生参考序列的偶然高的身份保持较高。
通过BLASTN过滤最终组件,以除去unigenes与> 95%同一性对宿主衍生序列(玉米和Medicago truncatula,来自[38.[39.])。然后通过BLASTN过滤剩余的unigenes至可用的PPGP转录组组件t .杂色的[23.[筛选>95%同一性的单基因Triphysaria转录物。ungenes持有少于95%的宿主或t .杂色的在非冗余蛋白序列数据库中查询序列[40]使用BLASTX(1E-10)。序列被最佳击中物种分类为非植物和植物类别。基于Estscan ORF预测,翻译在上述广泛努力之后保持未经发布和未分类的序列,并提交给Interporan [41.通过blast2go [92.]使用默认参数。这些unigenes也提交给了orthomcl db [42.数据库使用默认参数。
在unique and overlap Orthogroups中RPKM最大的ungenes,不包括属于所有三个转录组共享的Orthogroups和上述转录组中唯一的那些ungenes,用于查询NCBI的非冗余蛋白序列数据库(NR) [40]使用Blast2Go的Blastx(1E-10)[92.].使用BLASTn和BLASTx对17个注释的植物基因组进行了查询,以确定NR中不一定存在的注释植物序列的最佳匹配。查询的17个基因组如下:拟南芥,番木瓜,Fragaria vesca, Glycine max, Medicago truncatula, Mimulus guttatus, Oryza sativa, Phoenix dactylifera, Populus trichocarpa, Selaginella moellendorffii, Solanum lycopersicum, Solanum tuberosum, Sorghum bicolor, Thellungiella parvula,可可树,葡萄,和。此外,这些unigenes与interxocan注释[41.通过blast2go [92.]有默认设置。
瘦身类目分析
一个带注释的t .杂色的PPGP转录组没有界面转录om的组织重叠(TRVEBC1,[23.])纳入正交组分析,作为界面转录组分析的比较点。假定的t .杂色的基于OrthoGroup分配使用Venny对unigenes(不包括宿主和非植物unigenes)进行排序[93.].从orthogroups中存在的unigenes的苗条注释是使用r [94.].alpha设定为0.05。
TvEXPB1和TvEXPA4的系统发育分析
TVEXPB1的同源物拟南芥,栽培稻,Mimulus guttatus,Selaginella Moellendorfii.提取自Phytozome [38.基因家族#28891348 (β-expansins)。分别从PlantTribes 2.0中提取TvEXPA4的α expansin同源物[36.Orthogroup 1292年。将这些序列与PPGP项目中BLAST效果最好的一组可翻译序列进行组合,该序列分别与PlantTribes v2.0 Orthogroup 6163 (β-expansins)和1292 (α-expansins)序列组合。使用MUSCLE对每个数据集的推断氨基酸序列进行比对[95.然后将编码的DNA序列强制进行比对,以进行系统发育分析。RAxML [96.]版本7.2.8用于估计GTR+gamma分子进化模型下的最大似然树,并使用100个快速bootstrapping重复估计bootstrapping支持值。
验证扩展素转录序列和相对转录性丰度
互补脱氧核糖核酸的合成
从用于测序的相同扩增样品中合成cDNA,加上各种生物重复,用作使用该符号的QRT-PCR的模板TM值RT-QPCR(BiORAD)的逆转录超纤维。所有RNA样品都随着该试剂盒利用寡核苷酸和随机喷射而进行制造商的说明。
从宿主根中分离RNA
RNA从宿主根中分离,如前所述,与杂色的。在邻近Haustoria和液体液体中收获的主体根部在邻近的液体中收获2.金布尔大约50毫克的组织大力浸渍在追逐玻璃组织研磨机(部分# kt885450 - 0020)在450μL缓冲RLT +β-mercapto-ethanol(试剂盒)3分钟。然后将溶解产物转移到QiaShredder列和RNeasy工厂迷你包(试剂盒)指令被跟随的一个(附录D: RNeasy迷你手册4TH.版本)直至洗脱,在30μlRNase游离水中进行。使用具有植物总RNA测定的RNA 6000微微试剂盒(Agilent)在藿香生物分析仪上评估总RNA。
底漆设计和序列验证
引物采用Geneious Pro (v5.5.4 [97.])基于由由此产生的未成熟的unigenes扩增来自Arna的TVEXPA4和TVEXPB1德诺维Illumina配对端mRNA Seq数据的组装和后处理。
底漆序列:
tvexpa_f5:gcttttgcctacgaccaacttg.
TvEXPA_R3: GACAGTTTTGCCATCGCTTGTAG
tvexpb_f1:gccatagttcaacccgggac.
TvEXPB_R2: GGCTTCTTCCTGCTCTCCTTACTTG
利用这些引物,推测的α-和β-expansins转录本被扩增并提交Sanger测序。通过肉眼检查电铁图,手工调整测序序列的质量。使用MUSCLE将Sanger reads与unigene进行比对,以确定unigene序列。其余的空白均根据第一轮PCR产物验证的Sanger序列,采用相同的引物进行测序。
实时定量聚合酶链反应
(QRT-PCR)验证TvEXPB1和TvEXPA4的宿主特异性表达模式德诺维读取结果,使用QRT-PCR估计TVEXPB1,TVEXPA4,TVACTIN,Zmactin和MTACTIN的转录物水平。
底漆序列:
tvexpa4:
为:5'-TGGGAGGTGCTTGTGGGTAT-3'; Rev: 5’-CCGCAGGATAACCCATTGTT-3’
TvEXPB1:
为:5'-gatggcctgactgaagttgca-3'; Rev: 5’-GCGGCAAATTCACCCTAAAA-3’
TvActin:
: 5“-ACCCGATCCTTCTCACTGA-3”;牧师:5“-CATGACAATACCAGTCGTACG-3”
ZmActinB:
: 5“-CAATGGCACTGGAATGGT-3”;牧师:5“-ATCTTCAGGCGAAACACG-3”(98.]
MtActin:
为:5'-ATGTGCTATTCAGGCCG-3'; Rev: 5’-GCTCATAGTCAAGGGCAAT-3’ [99.]
初始特异性的验证
为了确定引物特异于其预期靶标,对每个PCR产物进行熔融曲线分析。通过提交从共培养的主体根提取的宿主RNA与寄生虫基因引物提取宿主RNA来验证寄生虫靶基因的引物特异性。在两种情况下,检测寄主肌动蛋白转录物,但特异于寄生虫基因的引物对产生与背景一致的信号。所有无模板控件(NTC)显示与背景信号一致的信号和所有反转转录负(RTN)控制显示与背景一致的信号。
QRT-PCR测定条件
使用Kapa TM Sybr Fast QPCR套件(Kapa Biosystems,KK4602)制备的QRT-PCR反应在制造商的说明之后。通过以下计划在Biorad Myiq(170-9770)上运行反应:
95°C 8分钟。(初始熔体)
95°C 0.5 min(循环熔化)
60℃0.5分钟。(循环退火/延伸)
重复40个周期
熔融曲线:0.5°C增量从95°C - 25°C。
存在数据分析
每3个技术重复的交叉点(Ct)值计算平均Ct值。使用2^(-ddCt)方法计算每个样本相对于对照的表达的fold change [100.].单尾,两个样本t- 使用Rest使用r [94.];alpha设定为0.05。
缩写
- (门店):
-
新一代测序
- (LPCM):
-
激光压力弹射显微解剖
- (ROI):
-
兴趣区
- (RIN):
-
RNA完整性
- (arna):
-
放大RNA
- (t .杂色的):
-
TripHysaria Versicolor.
- (Z.Mays):
-
玉米
- (m . truncatula):
-
Medicago truncatula
- (rpkm):
-
读取/千碱基/百万可映射读取
- (LM):
-
激光显微解剖
- (PR):
-
病原体抵抗力
- (存在):
-
定量实时PCR
- (NCBI):
-
国家生物技术信息中心
- (NR):
-
非冗余蛋白序列数据库。
参考文献
- 1。
植物寄生性的研究进展。植物科学进展,2010,15(4):227-235。10.1016 / j.tplants.2010.01.004。
- 2。
Olmstead Rg,Depamphilis Cw,Wolfe Ad,Young Nd,Elisons WJ,Reeves PA:盲味的疏症。我是J机器人。2001,88(2):348-361。10.2307 / 2657024。
- 3.
ejeta g:斯特非洲的祸害:日益严重的流行病。整合Striga控制的新技术:走向终结猎巫。编辑:Gressel J. Singapore: World Scientific Publishing Co;2007: 3-16。
- 4.
Scholes JD,按MC:斯特谷物作物的侵犯 - 资源有限公司农业中的未解决问题。CurrOp植物BIOL。2008,11(2):180-186。10.1016 / J.PBI.2008.02.004。
- 5。
Parker C:对目前状况的观察奥诺坎普和斯特全球问题。植物病虫害学报,2009,65(5):453-459。10.1002 / ps.1713。
- 6。
Vurro M, Bonciani B, Vannacci G:发展中国家作物植物新出现的传染病:对农业的影响和社会经济后果。粮食安全。2010,2(2):113-132。10.1007 / s12571 - 010 - 0062 - 7。
- 7。
Press MC, Graves JD:寄生植物。伦敦:查普曼和霍尔;1995.
- 8。
Tomilov A,Tomilova N,Yoder Ji:农杆菌肿瘤术和根出杆菌根草杆菌寄生植物转化后的根TripHysaria Versicolor.保留寄生能力。植物学报。2007,25(5):1059-1071。10.1007 / s00425 - 006 - 0415 - 9。
- 9。
yoder ji:一种特异性识别系统指导寄生植物中的海芥发Triphysaria(玄参科)。Planta。1997,202(4):407-413。10.1007 / S004250050144。
- 10.
Westwood Jh,De Pamphilis CW,Das M,Fernández-Aparicio M,Honaas La,Timko Mp,Wafula Ek,Wickett NJ,Yoder Ji:寄生植物基因组项目:理解生物学的新工具奥诺坎普和斯特.植物学报,2012,60(2):295-306。10.1614 / ws - d - 11 - 00113.1。
- 11.
寄生开花植物的生物学研究。伯克利:加州大学出版社;1969.
- 12.
陈志强,陈志强:木质部分子和转移细胞的表皮衍生物:对两种植物寄主-寄主界面的研究Triphysaria(玄参科)。原生质。1993,174(3):173-183。10.1007 / BF01379049
- 13。
heidej ørgensen HS, Kuijt J:根寄生虫的吸器Triphysaria(玄参科),尤指木质部桥状超微结构。中国生物医学工程学报,2012,31(6):591 - 598。10.2307 / 2445619。
- 14。
黄志强,王志强,王志强,等:马铃薯纺锤形块茎类病毒在番茄和番茄之间的传播Orobanche ramosa.病毒学。2010,399(2):187-193。10.1016 / J.Virol.2009.12.022。
- 15.
植物的韧皮部运动物质从寄主植物转移到全寄生植物cuscuta.acta physica sinica, 2010, 32(4): 489 - 494。10.1093 / jxb / erj076。
- 16。
Tomilov AA,Tomilova NB,Wrablewski T,Michelmore R,Yoder Ji:宿主和寄生植物之间的跨特异性基因沉默。工厂J. 2008,56(3):389-397。10.1111 / J.1365-313x.2008.03613.x。
- 17。
Thompson JN:共施加过程。芝加哥:芝加哥大学出版社;1994年。
- 18。
Agosta SJ,Janz N,Brooks博士:专家如何成为通情:解决“寄生虫悖论”并对新兴传染病的影响。动物毒素(库里奇巴)。2010年,27:151-162。
- 19。
草地一年生半寄生虫的种群生物学研究。主机环境。地球科学进展。1970,24(2):278-291。10.2307 / 2406804。
- 20.
寄生植物的联系。http://www.parasiticplants.siu.edu/.
- 21。
雪neeweiss GM:非光合寄生开花植物生活史和寄主范围的相关进化奥诺坎普和菲律宾人(Orobanchaceae)。J EVOL BIOL。2007,20(2):471-478。10.1111 / J.1420-9101.2006.01273.x。
- 22。
Thurman Ld:畸变研究在畸变研究亚属植物。加州大学,伯克利:博士论文。伯克利;1966年。
- 23。
寄生植物基因组计划。http://ppgp.huck.psu.edu/.
- 24。
王志强,王志强,王志强,等:植物与植物间的通讯:被子植物与寄主间的根际信号。植物生理学。1998,116(1):1-7。10.1104 / pp.116.1.1。
- 25。
Jamison DS,Yoder Ji:醌诱导的寄生植物中醌诱导的海芥子发育的可遗传变异Triphysaria.植物理性。2001,125(4):1870-1879。10.1104 / pp.125.4.1870。
- 26。
Bandaranayake PCG, Filappova T, Tomilov A, Tomilova NB, jamson - mcclung D, Ngo Q, Inoue K, Yoder JI:单电子还原醌氧化还原酶是诱导根寄生植物吸器发育所必需的Triphysaria.植物学报。2010,22(4):1404-1419。10.1105 / tpc.110.074831。
- 27。
Offler CE, McCurdy DW, Patrick JW, Talbot MJ:转移细胞:专门用于特殊目的的细胞。植物生态学报,2003,19(4):431- 434。10.1146 / annurev.arplant.54.031902.134812。
- 28。
墙壁PK,Leebens-Mack J,Chanderbali As,Barakat A,Wolcott E,Liang H,Landerr L,Tomsho LP,Hu Y,Carlson Je:下一代测序技术的比较转录组特征。BMC基因组。2009,10(1):347 .101186 / 1471-2164-10-347。
- 29。
STRICKLER SR,BOMBALLY A,MUELLER LA:设计用于非典范植物物种的转录组下一代测序项目。我是J机器人。2012,99(2):257-266。10.3732 / AJB.1100292。
- 30.
Grabherr MG, Haas BJ, Yassour M, Levin JZ, Thompson DA, Amit I, Adiconis X, Fan L, Raychowdhury R, Zeng Q:不含参考基因组的RNA-Seq数据的全长转录组组装。生物技术。2011,29(7):644-652。10.1038 / nbt.1883。
- 31.
寄生性被子植物Expansin的信息调控:在发育中的标记时间。植物学报。2000,12(8):1455-1465。
- 32.
Wrabel RL,yoder ji:寄生植物植物中α-扩展蛋白基因家族成员的差异RNA表达TripHysaria Versicolor.(玄参科)。基因工程学报。2001,26(2):85-93。
- 33.
Kenzelmann M, Klaren R, Hergenhahn M, Bonrouhi M, Grone HJ, Schmid W, Schutz G:纳米级总RNA数量的高精度扩增用于基因图谱。基因组学。2004,83(4):550-558。10.1016 / j.ygeno.2003.09.026。
- 34.
Matas AJ, Yeats TH, Buda GJ, Zheng Y, Chatterjee S, Tohge T, Ponnala L, Adato A, Aharoni A, Stark R:扩展番茄果实的组织和细胞类型特异性转录组分析提供了代谢和调控专门化和角质层形成的深入了解。植物学报。2011,23(11):3893-3910。10.1105 / tpc.111.091173。
- 35。
PlantTribes:植物比较基因组学的基因和家族资源。核酸学报2008,36(4):593 - 598。
- 36。
花卉基因组计划。http://fgp.bio.psu.edu..
- 37。
Wickett NJ,Honaas La,Wafula Ek,Das M,Huang K,Wu B,Landerherr L,Timko MP,Yoder J,Westwood JH:寄生植物家庭Orobanchaceae的转录组揭示了杂草合成的令人惊讶的保护。Curr Biol。2011,21:2098-2104。10.1016 / J.CUB.2011.11.011。
- 38。
Phytozome。http://www.phytozome.net.
- 39。
PlantGBD。http://www.plantgdb.org.
- 40.
国家生物技术信息中心。http://www.ncbi.nlm.nih.gov.
- 41。
欧洲生物信息学院。http://www.ebi.ac.uk/.
- 42。
orthomcl db。http://www.orthomcl.org/.
- 43.
桑佩德罗·J,科斯格罗夫DJ:不断扩大的超级大家庭。中国生物医学工程学报。2005,6(12):242。10.1186 / gb - 2005 - 6 - 12 - 242。
- 44.
Brandt SP:微基因组学:在组织特异性和单细胞水平上的基因表达分析。植物营养与肥料学报。2005,32(4):427 - 434。10.1093 / jxb / eri066。
- 45。
日RC, Grossniklaus U, Macknight RC:具体点!激光辅助植物细胞显微解剖。植物科学进展,2005,10(8):397-406。10.1016 / j.tplants.2005.06.006。
- 46。
Nelson T, Gandotra N, Tausta SL:植物细胞类型:新技术的报告和采样。植物生态学报,2008,11(5):567-573。10.1016 / j.pbi.2008.06.006。
- 47。
激光显微解剖及其在分析丛枝菌根共生中基因表达的应用。植物病虫害学报,2009,65(5):504-511。10.1002 / ps.1715。
- 48。
Woll K:新型玉米横向和精细根引发突变体Rum1的分离,表征和特异性细胞特异性转录组分析。植物理性。2005,139(3):1255-1267。10.1104 / pp.105.067330。
- 49。
黄志强,王志强,王志强,等:大豆包囊线虫侵染过程中寄主和病原菌的全基因组表达谱分析。MPMI。[j] .环境科学学报,2007,20(3):293-305。10.1094 / mpmi - 20 - 3 - 0293。
- 50.
Gomez Sk,Javot H,Deewatthanawong P,Torres-Jerez I,Tang Y,Blancaflor EB,Udvardi Mk,Harrison MJ:Medicago truncatula和Glomus mortraradice.丛枝菌根共生中含有丛枝的皮层细胞的基因表达。华北农学报。2009,9(1):10。10.1186 / 1471-2229-9-10。
- 51.
Klink VP,Hosseini P,Matsye P,Alkharouf NW,Matthews BF:Syncytia激光从根系中的基因表达分析甘氨酸最大(大豆)基因型PI 548402(北京)感染后抗性反应Heterodera甘氨酸(大豆囊肿线虫)。植物mol biol。2009,71(6):525-567。10.1007 / s11103-009-9539-1。
- 52.
Matas AJ,AgustíJ,TadeoFr,TalónM,玫瑰JKC:使用激光捕获微量碎片的柑橘类果皮和骨膜细胞的组织特异性转录组谱。J Exp Bot。2010,61(12):3321-3330。10.1093 / JXB / ERQ153。
- 53.
Pillitteri LJ,Peterson Km,Horst RJ,Torii Ku:气孔夹层的分子剖面揭示了拟南芥干细胞种群的非对称细胞分裂和常见的新组分。植物细胞。2011,23(9):3260-3275。10.1105 / TPC.111.088583。
- 54.
Emrich SJ,Barbazuk WB,Li L,Schnable PS:基因发现和使用LCM-454转录组测序的注释。Genome Res。2007,17(1):69-73。
- 55.
ohtsu k,史密斯Mb,emrich sj,borsuk la,zhou rl,chen tl,张xl,timmermans mcp,beck j,buckner b:玉米枝顶部分析的全局基因表达分析(Zea Mays L.).工厂J. 2007,52(3):391-404。10.1111 / J.1365-313x.2007.03244.x。
- 56.
Li PH, Ponnala L, Gandotra N, Wang L, Si YQ, Tausta SL, Kebrom TH, Provart N, Patel R, Myers CR:玉米叶片转录组的发育动态。acta photonica sinica, 2010, 42(12): 1060-1067。10.1038 / ng.703。
- 57.
Feldman Al,Costouros Ng,Wang E,Qian M,Marincola FM,Alexander HR,Libutti SK:微阵列分析mRNA扩增的优点。生物技术。2002,33(4):906。
- 58.
Polacek DC,Passerini AG,Shi C,Francesco NM,Manduchi E,Grant Gr,Powell S,Bischof H,Winkler H,Stoeckert CJ:纳米中纳米内皮mRNA线性扩增后鉴别转录谱的富有度和增强敏感性。physiol基因组学。2003,13(2):147-156。
- 59。
李啊,李t,刘sz,邱我,汉泽,姜zl,李丽,y k,谢y,毛ym:使用cDNA微阵列进行ARNA,mRNA和T-RNA对基因表达分析的保真度的系统比较.j biotech。2004,107(1):19-28。10.1016 / J.JBBIOTEC.2003.09.008。
- 60。
King C, Guo N, Frampton GM, Gerry NP, Lenburg ME, Rosenberg CL:利用激光显微解剖的乳腺组织寡核苷酸阵列扩增RNA测量基因表达的可靠性和重现性。中华医学杂志。2005,7(1):57。10.1016 / s1525 - 1578(10) 60009 - 8。
- 61。
日RC,McNoe La,Macknight RC:激光微量植物细胞的转录分析。physiol platararum。2007,129(2):267-282。
- 62.
固定对激光捕获显微解剖组织RNA提取和扩增的影响。acta physica sinica, 1999, 25(2): 86-91。10.1002 / (SICI) 1098 - 2744 (199906) 25:2 < 86:: AID-MC2 > 3.0.CO; 2 - 4。
- 63.
Gillespie JW, Best CJM, Bichsel VE, Cole KA, Greenhut SF, Hewitt SM, Ahram M, Gathright YB, Merino MJ, Strausberg RL:非福尔马林组织固定在分子谱研究中的评价。王志强,王志强,王志强,等。10.1016 / s0002 - 9440 (10) 64864 - x。
- 64.
激光捕获植物组织细胞的显微解剖。植物生理学报。2003,132(1):27-35。10.1104 / pp.102.018127。
- 65。
Nakazono M:激光捕获微小,一种用于特定植物细胞类型中基因表达的全局分析的工具:鉴定表达表达表达表达表达的基因或玉米血管组织。植物细胞。2003,15(3):583-596。10.1105 / TPC.008102。
- 66。
Rispail N,Dita Ma,Gonzalez-Verdejo C,Perez-De-Luque A,Castillejo Ma,Prats E,Roman B,Jorrin J,Rubiales D:植物抵抗寄生植物:古老敌人的分子方法。新植物。2007,173(4):703-711。10.1111 / J.1469-8137.2007.01980.x。
- 67.
引用本文:Tomilov A, Tomilova N, Shin DH, Jamison D, Torres J, Reagan R, Horning T, Truong R, Nava A, Nava A:植物间的化学信号转导:植物毒性化感作用与寄主识别的机制相似性。化学通讯:从基因到生态系统。编辑:Dicke M. Wageningen: Frontis;2006: 55 - 69。
- 68.
Cosgrove DJ:植物细胞壁的生长。中国生物医学工程学报。2005,6(11):850-861。10.1038 / nrm1746。
- 69。
关键词:基因表达素类(L.)在用细菌病原体感染后osbeckCandidatus libibacter在佛罗里达造成黄龙病的亚洲人。植物科学学报,2008,35(3):441 - 446。10.1016 / j.plantsci.2008.05.001。
- 70。
丁Xh,Cao Yl,Huang Ll,Zhao J,Xu Cg,Li Xh,王SP:吲哚-3-乙酸 - amido合成酶GH3-8的激活抑制扩张素表达,促进水杨酸盐和淫苗依赖性基础免疫力白饭。植物细胞。2008,20(1):228-240。10.1105 / TPC.107.055657。
- 71。
一种植物扩张素的表达参与了番茄根结线虫寄生的建立。植物学报。2006,34(1):155-162。10.1007 / s00425 - 005 - 0204 - x。
- 72。
Kikuchi T,Aikawa T,Kosaka H,Pritchard L,Ogura N,Jones JT:表达序列标签(EST)分析松木线虫Bursaphelenchus Xylophilus.和B-mucronatus.Mol Biochem寄生醇。2007,155(1):9-17。10.1016 / J.Molbiopara.2007.05.002。
- 73。
Li ZC, Durachko DM, Cosgrove DJ:一种燕麦胚芽鞘壁蛋白,在体外诱导壁延伸,它与黄瓜下胚轴的一种类似蛋白具有抗原相关性。植物学报。1993,191(3):349-356。
- 74.
深水水稻节间膨化素的研究。植物营养与肥料学报。1997,13(4):593 - 598。10.1104 / pp.113.4.1137。
- 75.
科斯格罗夫DJ, Bedinger P, Durachko DM:作为细胞壁松弛剂的草花粉I组过敏原。美国国立科学院。[j] .地理科学进展,2017,36(12):1259 - 1263。10.1073 / pnas.94.12.6559。
- 76.
McQueenmason SJ,Cosgrove DJ:诱导壁延伸的蛋白质植物细胞壁聚合物之间的氢键合。美国国立科学院。1994,91:6574-6578。10.1073 / pnas.91.14.6574。
- 77.
细胞壁上的膨胀素作用模式-细胞壁水解、应力松弛和结合的分析。植物营养与肥料学报。1995,27(1):87-100。
- 78.
李玉玲,李玉玲,李玉玲:细胞扩张素与细胞生长的关系。植物学报,2003,6(6):603-610。10.1016 / j.pbi.2003.09.003。
- 79.
Cosgrove DJ:通过扩展蛋白松开植物细胞壁。自然。2000,407(6802):321-326。10.1038 / 35030000。
- 80.
KAPU NUS,Cosgrove DJ:授粉后玉米丝绸的增长和细胞壁延长性的变化。J Exp Bot。2010,61(14):4097-4107。10.1093 / JXB / ERQ225。
- 81.
Tabuchi A,Li Lc,Cosgrove DJ:由玉米花粉的β-扩展蛋白(1型过敏原)削弱的基质溶解和细胞壁。工厂J. 2011,68(3):546-559。10.1111 / J.1365-313x.2011.04705.x。
- 82.
Yennawar NH,Li L-C,Dudzinski DM,Tabuchi A,Cosgrove DJ:ExpB1(Zea M 1),α,β-扩展素和来自玉米的1组花粉过敏原的晶体结构和活动。美国国立科学院。2006,103(40):14664-14671。10.1073 / pnas.0605979103。
- 83。
小瓶L, Chapalain A,格罗罗MC, Deziel E:不同的生活方式伯克不过复杂物种:适应的贡献。环境微生物学报,2011,13(1):1-12。10.1111 / j.1462-2920.2010.02343.x。
- 84。
Atsatt Pr:寄生开花植物:他们是如何发展的?我是nat。1973,107(956):502-510。10.1086 / 282853。
- 85。
Chuang Ti,Heckard LR:子系统Castillejinae的通用重新调整和概要(Scrophulariaceae-Tribulareae)。SYST BOT。1991,16(4):644-666。10.2307 / 2418868。
- 86.
王志强,王志强,王志强,等:全寄生植物的转化和再生Phelipanche aegyptiaca.植物学报。2011,7(1):36。10.1186 / 1746-4811-7-36。
- 87.
Torres MJ, Tomilov AA, Tomilova N, Reagan RL, Yoder JI: Pscroph,一个丰富寄生虫相关转录本的寄生植物EST数据库。植物学报。2005,5:24。10.1186 / 1471-2229-5-24。
- 88.
SCHROEDER A,MUELLER O,Stocker S,Salowsky R,Leiger M,Gassmann M,Lightfoot S,Menzel W,Granzow M,Ragg T:Rin:RNA完整性编号,用于将完整性值分配给RNA测量。BMC MOL BIOL。2006,7:3. 10.1186 / 1471-2199-7-3。
- 89.
- 90。
ESTScan:一种用于检测、评估和重建EST序列中潜在编码区域的程序。计算机科学与技术。1999,138-148。
- 91。
Edgar RC:搜索和聚类比爆炸速度快。生物信息学。2010,26(19):2460-2461。10.1093 / Bioinformatics / BTQ461。
- 92。
blast2go。http://www.blast2go.com/.
- 93。
- 94。
统计计算的R项目。http://www.r-project.org/.
- 95。
Edgar RC:肌肉:具有高精度和高吞吐量的多个序列对齐。核酸RES。2004,32:1792-1797。10.1093 / nar / gkh340。
- 96。
RAxML-VI-HPC:基于数千个分类群和混合模型的最大可能性系统发育分析。生物信息学。2006,22:2688-2690。10.1093 /生物信息学/ btl446。
- 97。
Geneious。http://www.geneious.com/.
- 98.
邢盖夫,郭金望,姚益,彭人力,孙QX,氮效:玉米幼苗根系新型杂交上调长期非蛋白质编码RNA的鉴定与表征。植物SCI。2010,179(4):356-363。10.1016 / J.PlantsCi.2010.06.008。
- 99.
Jones Km,Sharopova N,Lohar DP,张JQ,Vandenbosch Ka,Walker GC:植物Medicago Truncatula的差分响应为其SymbiontSinorhizobium meliloti或者是胞外多糖缺陷突变体。美国国立科学院。[j] .环境科学学报,2008,29(2):429 - 434。10.1073 / pnas.0709338105。
- One hundred.
Pfaffl MW:实时rt - pcr中相对定量的一个新的数学模型。中国生物医学工程学报2001,29 (9):e45。10.1093 / nar / 29.9.e45。
致谢
作者通过NSF Plant Genome奖DBI-0701748对该项目进行了对该项目的部分资金,DBI-0701748到Westwood JH,Depamphilis CW,Timko MP和Yoder Ji,NSF奖IOS-0821954和IOS-1104334至Taylor CG,McIntyre L和Koch,K和宾夕法尼亚州立大学植物生物学和生物学系中的阶级研究生课程。 We thank Paula Ralph for lab assistance and Yongde Bao (University of Virginia) for Illumina sequencing. We also thank Daniel J. Cosgrove for helpful discussion and encouragement throughout the course of this research. We thank dePamphilis lab members past and present for their assistance with various aspects of this work, and two anonymous reviewers for helpful comments.
作者信息
从属关系
相应的作者
额外的信息
利益争夺
提交人声明他们没有竞争利益。
作者的贡献
PPGP转录组(JHW, CWD, MPT, JIY)和界面转录组研究(LAH, CWD)的构思与设计;种子和Triphysaria成长和分析:jiy;植物栽培和组织:LAH;激光捕获方法和抽样:LAH,CGT;RNA,扩增和测序图书馆:LAH;QRT-PCR:LAH,ZY;数据分析和演示:LAH,JPD,EKW,NJW,ZY,NSA,CWD;写了稿件:LAH和CWD与所有作者的贡献。所有作者阅读并认可的终稿。
电子补充材料
单基因配对核苷酸同源图。
附加文件1:图S1:本研究中考虑的未经成果之间的序列标识和参考EST集合(PlantGDB公共Ests,http://www.plantgdb.org/)给主人z梅斯和m . truncatula。Triphysariaunigenes与宿主参考对齐以鉴定宿主污染物并与往复式非宿主参考组对齐以鉴定偶然核苷酸成对相同特性。整个植物标准化的转录组合组件Lindenbergia philippensis(Orobanchaceae的非寄生成分)用于确定对每个宿主的非寄生虫的成对标识的分布,并控制来自潜在的交叉污染的宿主的高未植物标识。选择了95%的阈值,以通过保留排除宿主转录物Triphysariaunigenes与宿主est具有高度同源性。(PDF 72 KB)
OrthoMCL DB和InterProScan (IPS)结果的VENN图总结。
附加文件2:图S2:从整个接口转录组中的eStscan ORF预测,在基于广泛的基于炸的数据库搜索之后仍未入围,并将其提交给Orthomcl DB和Internoscan。从每个转录物组的unigenes之间的图案相似,指示当量的未植入分类t .杂色的在两个主人上生长。鉴定出直牙或肽基序的未植物的数量相对较小,表明使用PlantTribes 2.0和外部数据库查询的Unigene分类是强大的。分享大约25%的从每个转录组中鉴定的已知的正常表然。分享到每种转录组的大多数unigenes仍然未知,其中包括> 300个核苷酸Bp的许多(〜500〜500),并具有读取的载体。(PDF 220 KB)
12870 _2012_1267_moesm3_esm.xls
附加文件3:筛选PPGP数据库、宿主cDNA和EST序列、NCBI非冗余蛋白序列数据库后剩余unigenes的OrthoMCL DB和InterProScan注释摘要电子表格。(XLS 252 KB)
瘦身分类分析。
图S3:在Venn的指示区域中,GO Slim术语的卡方检验(P< 0.0001)。表中列出了每个GO类别中指示区域的unigenes的数量。残值为强阳性(>4)的细胞表示为大胆的+强负残差值(<-4)表示为大胆的,.GO瘦身功能(一个),组件(B)和过程(C)的界面转录组的分类分析t .杂色的成长为z梅斯.GO瘦身功能(D),组件(E)和过程(F)的界面转录组的分类分析t .杂色的成长为m . truncatula.(PDF 125 KB)
在接口转录om和参考组装TrvestBC1(PPGP.HUCK.PSU.EDU)之间共享的正常读数(RPKM)对unigenes的正常读数(RPKM)的相关性。
附加文件5:图S4:从每个接口读取的读数被映射到包括整个Hautroium数据的参考组件(Trvesbc2,ppgp.huck.psu.edu)t .杂色的成长为m . truncatula.在界面转录组中更高度表达了未成年的子集t .杂色的成长为m . truncatula;没有类似的模式t .杂色的成长为z梅斯.这是由于对Medicago种植Triphysaria参考数据集TRVEBC2中的Unigenes,其由读取构建Medicago种植Triphysaria.对于共享正群中的unigenes,界面转录组之间的RPKM值高度相关(Pearson’s R = 0.81),表明技术和生物学变异较低。(PDF 205 KB)
高度表达的界面unigenes。
附加文件6:图S5:每个转录组维恩图中20个最高表达(RPKM)的unigenes (ID)显示了相互作用的转录组维恩图t .杂色的对每个宿主物种。NR BLASTx -描述,种类和%id。:TH.e description, species of origin, and percent pairwise identify, respectively, of the best unigene alignment (<1e-10) resulting from the NR database query, 17 genomes BLAST and %id.: best hit species in a BLAST database of 17 annotated plant genomes with the percent pairwise identity in the nucleotide BLAST (N) or translated nucleotide BLAST (P).TIPS跨膜预测,年代XXX = IPS分泌信号预测。(PDF 2 MB)
BLAST的文本文件(BLAST默认输出格式)来源于对NCBI的非冗余蛋白序列数据库的查询
额外的文件7:z梅斯种植t .杂色的unigenes列于附加文件6。(TXT 341 KB)
BLAST的文本文件(BLAST默认输出格式)来源于对NCBI的非冗余蛋白序列数据库的查询
附加文件8:m . truncatula种植t .杂色的unigenes列于附加文件6。(TXT 347 KB)
InterProScan分析结果
附加文件9:z梅斯种植t .杂色的unigenes列于附加文件6。(XLS 48 KB)
InterProScan分析结果
附加文件10:m . truncatula种植t .杂色的unigenes列于附加文件6。(XLS 73 KB)
raxml分析A:
附加文件11:图S6:TriphysariaB: α expansin基因TvEXPB1 (TrVeIntZeamaGB1_772,绿色文本)和B: α expansin基因TvEXPA4 (TrVeIntMedtrGB1_11,绿色文本)。在每个节点上给出了自举比例。A的分类简写:拟南芥(在),栽培稻(操作系统),Mimulus guttatus(mg),TripHysaria Versicolor.(龙),Striga hermonthica(sthe),菲律宾人(=奥诺坎普)aegyptiaca(orae),卷柏moellendorffii(Smoellendorffii)。分类单元B的缩写:栽培稻(操作系统),高粱双色(某人),Striga hermonthica(sthe),菲律宾人(=奥诺坎普)aegyptiaca(orae),TripHysaria Versicolor.(龙),卡里卡番木瓜(卡帕),Populus Trichocarpa.(Poptr),Medicago truncatula(MEDTR),vitis Vinifera(Vitvi),拟南芥(在),卷柏moellendorffii(Selmo),Physcomitrella patens.(phypa)。(PDF 87 KB)
作者的原始提交的图像文件
权利和权限
开放访问本文在“生物资源”中央有限公司的许可下公布了这是一个开放式访问条款,分配根据创意公约归因许可证的条款(https://creativecommons.org/licenses/by/2.0)提供任何介质中的不受限制使用,分发和再现,所以提供了正确的工作。
关于这篇文章
引用这篇文章
霍纳斯,洛杉矶,瓦夫拉,e.k.,杨,Z。等等。通才寄生植物的功能基因组学:激光显微解剖寄主-寄主界面揭示寄主特异性寄主基因表达模式。BMC植物BIOL.13,9(2013)。https://doi.org/10.1186/1471-2229-13-9
收到了:
接受:
发表:
关键词
- 寄生植物
- RNA-SEQ.
- illumina
- 德诺维集会
- 转录组
- 激光微生物
- expans
- 通才寄生虫
- 列当科
- Triphysaria
- 玉米
- Medicago
- 洋葱