- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
早期玉米减数分裂的转录组格局gydF4y2Ba
植物生物学gydF4y2Ba体积gydF4y2Ba14gydF4y2Ba,文章编号:gydF4y2Ba118gydF4y2Ba(gydF4y2Ba2014gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
高等植物生命周期的一个重要步骤是决定离开有丝分裂细胞周期,开始减数分裂细胞周期的进程,从而形成配子。调控这一转变和早期减数分裂的分子机制在很大程度上仍不清楚。为了深入了解减数分裂重组启动过程中的基因表达特征,我们对玉米早期前期I型减数分裂细胞进行了分析gydF4y2Ba(玉米)gydF4y2Ba利用毛细管收集分离减数细胞,然后进行RNA-seq。gydF4y2Ba
结果gydF4y2Ba
我们检测到约2000个基因在减数分裂早期优先表达,其中大多数没有特征。功能分析揭示了几个细胞过程在早期减数分裂中的重要性。分离的减数细胞中显著富集的过程包括蛋白质水解、蛋白质靶向、染色质修饰和氧化还原稳态调节。减数细胞中上调最显著的是碳水化合物代谢过程。与此一致的是,许多线粒体基因在减数细胞中上调,包括核和线粒体编码基因。用real-time PCR和PCR方法对数据进行验证gydF4y2Ba原位gydF4y2Ba杂交,并用于从拟南芥中生成已知减数分裂基因的候选玉米同源列表。gydF4y2Ba
结论gydF4y2Ba
综上所述,我们对一种重要作物减数分裂早期的转录组进行了高分辨率分析,为选择基因进行重组启动和早期减数分裂调控的详细描述提供了支持。我们的数据还揭示了减数分裂过程和能量生产变化/增加之间的重要联系。gydF4y2Ba
背景gydF4y2Ba
减数分裂是高等植物生命周期中的一个关键过程,在此过程中发生重组,导致亲本等位基因的新组合。许多减数分裂基因的特征是迄今为止直接参与减数分裂重组机制和识别整套减数分裂基因是一个持续的过程。在经过充分研究的双子叶植物模型中,gydF4y2Ba拟南芥,gydF4y2Ba大约70个参与减数分裂的基因已被功能鉴定[gydF4y2Ba1gydF4y2Ba- - - - - -gydF4y2Ba6gydF4y2Ba].在农作物中,很少有特征良好的减数分裂基因,但已经尝试在玉米、水稻、小麦和大麦中生成与其他生物的特征良好的同源基因相对应的完整减数分裂基因图谱[gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba].一些使用完整花药的转录组研究已经在拟南芥等物种中进行。gydF4y2Ba9gydF4y2Ba,矮牵牛花[gydF4y2Ba10gydF4y2Ba,大米[gydF4y2Ba11gydF4y2Ba- - - - - -gydF4y2Ba13gydF4y2Ba,六倍体小麦[gydF4y2Ba14gydF4y2Ba和玉米[gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba].对花药发育过程中多个阶段的研究在转录组动力学和阶段特异性转录本方面获得了有价值的数据[gydF4y2Ba10gydF4y2Ba,gydF4y2Ba13gydF4y2Ba- - - - - -gydF4y2Ba15gydF4y2Ba].此外,一些研究通过比较减数分裂突变花药与野生型花药,有助于阐明减数分裂的转录组[gydF4y2Ba9gydF4y2Ba,gydF4y2Ba16gydF4y2Ba- - - - - -gydF4y2Ba19gydF4y2Ba].gydF4y2Ba
然而,这些研究检查了整个花药的转录组,虽然在技术上比分离减数细胞(发生减数分裂的细胞)难度小得多,但没有区分减数细胞基因表达和花药其他各种组织中的基因表达。与减数分裂突变花药进行比较可以改善这一点,但突变会导致组织组成和基因表达的扭曲。为了深入了解减数细胞的转录组,最近的工作涉及到分离纯减数细胞的技术。利用CCM (gydF4y2BaCgydF4y2BaapillarygydF4y2BaCgydF4y2Baollection的gydF4y2Ba米gydF4y2Baeiocyte),其中减数细胞用微毛细血管收集[gydF4y2Ba20.gydF4y2Ba].在这些研究中,使用表达微阵列分析了分离的拟南芥减数细胞在前期I到四分体阶段的mRNA [gydF4y2Ba21gydF4y2Ba或下一代测序[gydF4y2Ba5gydF4y2Ba,gydF4y2Ba22gydF4y2Ba].这些研究发现,大量的拟南芥基因在减数分裂过程中表达[gydF4y2Ba5gydF4y2Ba,gydF4y2Ba22gydF4y2Ba].有趣的是,减数细胞也大量表达转座因子[gydF4y2Ba5gydF4y2Ba,gydF4y2Ba22gydF4y2Ba]以及高水平的转录本映射到核基因组中的线粒体基因组插入(MGI) [gydF4y2Ba5gydF4y2Ba].虽然这些研究是基于所有减数分裂阶段的细胞集合,但之前没有研究专门检查分离的减数分裂细胞中的早期减数分裂转录组。减数分裂前期I中减数分裂的早期步骤是孢子母细胞离开有丝分裂细胞周期进入减数分裂细胞周期,染色体开始配对重组的阶段[gydF4y2Ba1gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba23gydF4y2Ba].这些过程对减数分裂的成功至关重要。识别特定富集的基因和过程对于理解早期减数分裂、重组启动和配子形成的调控分子机制非常重要。gydF4y2Ba
在本研究中,我们利用了玉米雄花序(流苏)中存在的发育同步性。gydF4y2Ba玉米gydF4y2Ba我们对分离的减数分裂细胞的早期减数分裂转录组进行了更仔细的研究,主要有两个目的:首先,我们想用玉米减数分裂候选基因列表来补充之前的研究,报告它们在分离的减数分裂细胞和花药中的表达水平。我们的第二个目标是采用更普遍的方法,揭示在减数分裂早期的重要过程,而不仅仅是那些直接参与重组保守过程的过程。gydF4y2Ba
结果gydF4y2Ba
分离的玉米雄性减数细胞基因表达谱gydF4y2Ba
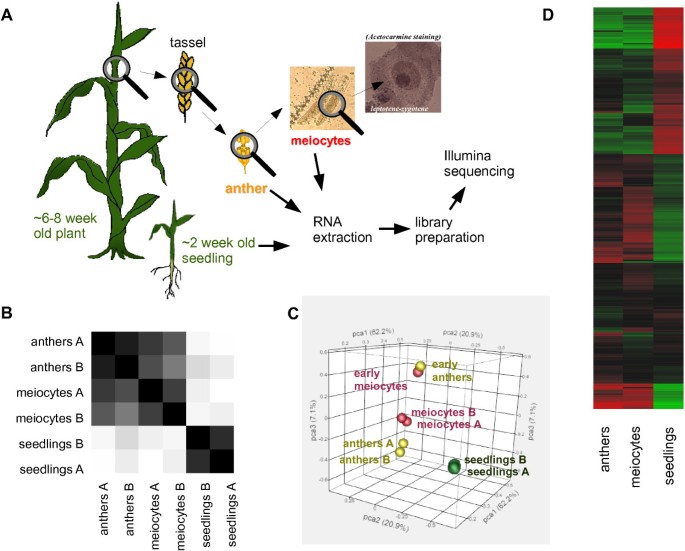
我们使用CCM (gydF4y2BaCgydF4y2BaapillarygydF4y2BaCgydF4y2Baollection的gydF4y2Ba米gydF4y2Ba),然后进行RNA提取和Illumina测序,生成分离的I期瘦线期和接合线期减数细胞的转录组谱,包含相同阶段减数细胞的完整花药,以及两周大的玉米幼苗(图gydF4y2Ba1gydF4y2BaA).每个转录组在两个生物重复中生成,重复之间的相关系数分别为0.9174(减数细胞)、0.9419(花药)和0.7990(幼苗;额外的文件gydF4y2Ba1gydF4y2Ba:表S1)。RNA产量在2.3-6.7 μg之间,总测序reads从36,126,210-77,551,649,其中至少18,535,914 reads是唯一对齐的gydF4y2Ba1gydF4y2Ba:表S1)。JMP Genomics使用Ward方法通过分层聚类生成的相关树状图显示成对的生物复制彼此之间的相关性最好(图1)gydF4y2Ba1gydF4y2BaB).花药和减数细胞之间也有很高的相关性(图gydF4y2Ba1gydF4y2BaB-D,附加文件gydF4y2Ba1gydF4y2Ba:表S1),当将早期前期的减数分裂细胞和花药样本与额外的减数分裂前的减数分裂细胞和花药样本进行比较时,我们的生物复制的相关性尤其明显(图1)gydF4y2Ba1gydF4y2BaC)。gydF4y2Ba
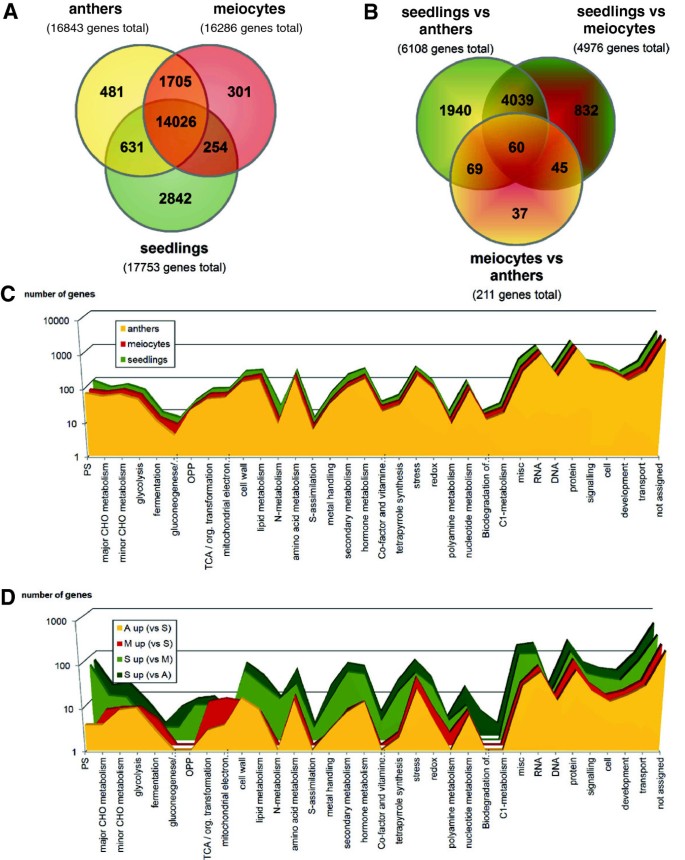
超过5个reads / million的阈值,在减数细胞中表达了16286个基因,在花药中表达了16843个基因,在幼苗中表达了17753个基因,其中79% -86%的基因是所有样本共有的(图gydF4y2Ba2gydF4y2Ba一个);在2或10 RPM情况下的基因数量,并与等效的比较gydF4y2Ba拟南芥gydF4y2Ba数据见[gydF4y2Ba24gydF4y2Ba].很少有基因在一个样本中唯一表达,即2%的减数细胞基因,3%的花药基因和16%的幼苗基因(图gydF4y2Ba2gydF4y2BaA).注意花药样品中含有减数细胞,这可能导致花药和减数细胞之间差异表达的基因数量较少(包括上调和下调);在花药或减数细胞与幼苗之间发现了更多差异表达的基因(图gydF4y2Ba2gydF4y2BaB、附加文件gydF4y2Ba2gydF4y2Ba:图S1A-C;使用DEseq包生成的用于R统计分析的上调和下调基因列表,使用的阈值显著性gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba≤0.01,附加文件gydF4y2Ba3.gydF4y2Ba:表S2)。表达热图中花药和减数细胞的高度一致性也清楚地将它们与幼苗区分开来(图gydF4y2Ba1gydF4y2BaD、附加文件gydF4y2Ba2gydF4y2Ba:图S1D)。gydF4y2Ba
玉米B73自交系减数细胞、花药和幼苗的基因表达谱。(一)gydF4y2Ba每个样本至少有5 RPM的所有基因的维恩图。gydF4y2Ba(B)gydF4y2Ba样本间差异表达基因的维恩图(gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba≤0.01)。gydF4y2Ba(C)gydF4y2BaGO基因分布如图所示gydF4y2Ba(一)gydF4y2Ba.gydF4y2Ba(D)gydF4y2Ba差异表达基因的GO分布gydF4y2Ba(B)gydF4y2Ba.PS =光合作用,CHO =碳水化合物,OPP =氧化磷酸戊糖途径,TCA =三羧酸循环,misc =杂项gydF4y2Ba
由于近85%的玉米基因组由转座因子(TEs)组成[gydF4y2Ba25gydF4y2Ba,我们分析了TE在减数细胞、花药和幼苗中的差异表达。与相对基因组丰度相比,标注的TEs仅对全局表达贡献约12%,而标注的基因贡献约80%,在所有数据集中平均;来自参考文献中不存在的基因组区域的读取,或质量太差以致于无法对齐的读取gydF4y2Ba4gydF4y2Ba:图S2A)。我们对减数细胞中TE的表达进行了彻底的分析gydF4y2Ba4gydF4y2Ba:图S2B-E),并检测到对[RLX]未知TE超家族的LTRs的偏好,以及在减数分裂期间表达较高水平的TEs的染色体起源的强烈偏见(附加文件gydF4y2Ba4gydF4y2Ba图S2D + E):大多数减数分裂特异性TEs起源于染色体6,与非减数分裂特异性TEs相比,从线粒体基因组中检测到的TEs要多得多gydF4y2Ba4gydF4y2Ba:图S2E)。[RLX]未知TE超家族以及6号染色体衍生的TEs的过度表达是由于“Ipiki”家族TEs(数据库ID AC212468_11834),其中许多在减数分裂特异性TEs中上调,一些高达约200倍。255个Ipiki基因中有231个位于6号染色体上,位于短臂的远端,核仁组织者区远端。此外,我们注意到,以前vicent等人报道的在减数分裂或有丝分裂组织中高度表达的TE家族的发生率增加。[gydF4y2Ba26gydF4y2Ba在我们的减数分裂特异性TEs中,特别是Giepum, Cinful和Flip(附加文件gydF4y2Ba4gydF4y2Ba:图S2F)。gydF4y2Ba
使用MapMan对每个样本表达的所有基因进行功能注释[gydF4y2Ba27gydF4y2Ba]表明样本之间功能类别分布的差异是最小的(图gydF4y2Ba2gydF4y2BaC).唯一的区别是与光合作用和次生代谢相关的基因,这些基因在幼苗中富集(图1)gydF4y2Ba2gydF4y2BaC).在这种方法中,花药和减数细胞之间没有明显的区别。gydF4y2Ba
将我们样本中上调的基因用MapMan软件进行功能术语概述(图1)gydF4y2Ba2gydF4y2BaD)则表现出明显的差异。大多数在花药中富集的功能类别也只在减数细胞中富集,这与它们相似的表达谱一致(附加文件gydF4y2Ba2gydF4y2Ba:图S1D)。与幼苗相比,整个花药中上调的基因表明参与染色质包装和组织、转录、RNA生物合成过程以及调控过程的基因富集(图gydF4y2Ba2gydF4y2BaD)。此外,花药和减数细胞的转录组普遍存在与能量生产相关的基因,如糖酵解、发酵、TCA(三羧酸循环)和线粒体电子运输(图)gydF4y2Ba2gydF4y2BaD).幼苗中上调的基因富集于参与光合作用、OPP(氧化戊糖磷酸途径)、细胞壁和脂质代谢、次生代谢、氮和硫代谢的基因(图)gydF4y2Ba2gydF4y2BaD)。gydF4y2Ba
减数细胞中上调基因的详细GO分析gydF4y2Ba
为了更深入地了解减数分裂早期的过程,我们使用AgriGO ([gydF4y2Ba28gydF4y2Ba),gydF4y2Bahttp://bioinfo.cau.edu.cn/agriGO/gydF4y2Ba).对上调或下调基因的分析并没有产生显著的GO(基因本体论)术语来比较减数细胞和花药。所有其他比较返回多个重要的GO术语(附加文件gydF4y2Ba5gydF4y2Ba表S3)。gydF4y2Ba
在减数细胞和幼苗中上调的基因在一些重要的氧化石墨烯术语中被富集(表gydF4y2Ba1gydF4y2Ba,附加文件gydF4y2Ba5gydF4y2Ba表S3),包括与能量和线粒体相关的过程和各种调节机制,如氧化还原稳态和染色质修饰。在减数细胞与幼苗比较中,氧化石墨烯富集最显著的术语是“细胞碳水化合物代谢过程”。在减数细胞和幼苗中富集的其他氧化石墨烯类别是“定位”(包含编码线粒体或ER中的跨膜蛋白和受体的基因,以及RasGTPases,见附加文件gydF4y2Ba6gydF4y2Ba:图S3A)、“信号转导”(包括RasGTPase基因)、“DNA修复”(特别是编码错配或切除修复蛋白的基因)、“蛋白质水解”(包括蛋白酶体亚基和细胞周期进展蛋白SKP1的基因)和“糖基化”(包括核蛋白和半乳糖凝集素的基因)。除了这些高度富集的氧化石墨烯类别外,“染色质”(含有大部分组蛋白和组蛋白修饰物的基因)、“RNA”(含有转录因子的基因,见附加文件gydF4y2Ba6gydF4y2Ba:图S3B,核糖体蛋白和组蛋白)和“内稳态”(主要由硫氧还蛋白和戊氧还蛋白基因组成)在减数细胞中与幼苗相比也显著富集,但富集程度较低。gydF4y2Ba
我们使用Chen等人先前定义的以下标准将2223个基因指定为减数细胞基因。[gydF4y2Ba5gydF4y2Ba]:基因在减数细胞中的表达量至少是花药中的2倍(M/A≥2),或者,基因在减数细胞和花药中的表达量至少是幼苗的2倍,花药中的表达量低于减数细胞的4倍(如果M/S和A/S均≥2,则A/M < 4)。总的来说,457个基因符合第一标准,2187个基因符合第二标准。第一组的大部分(429个)基因也出现在第二组中,在组合列表中有2223个基因。利用AgriGO对这些基因进行氧化石墨烯分析[gydF4y2Ba28gydF4y2Ba]发现了与“核小体组装”和“DNA包装”相关的术语更加富集,以及与“碳水化合物代谢”和“定位”相关的几个额外富集的氧化石墨烯术语(附加文件gydF4y2Ba5gydF4y2Ba表S3)。gydF4y2Ba
减数分裂早期线粒体转录物丰富gydF4y2Ba
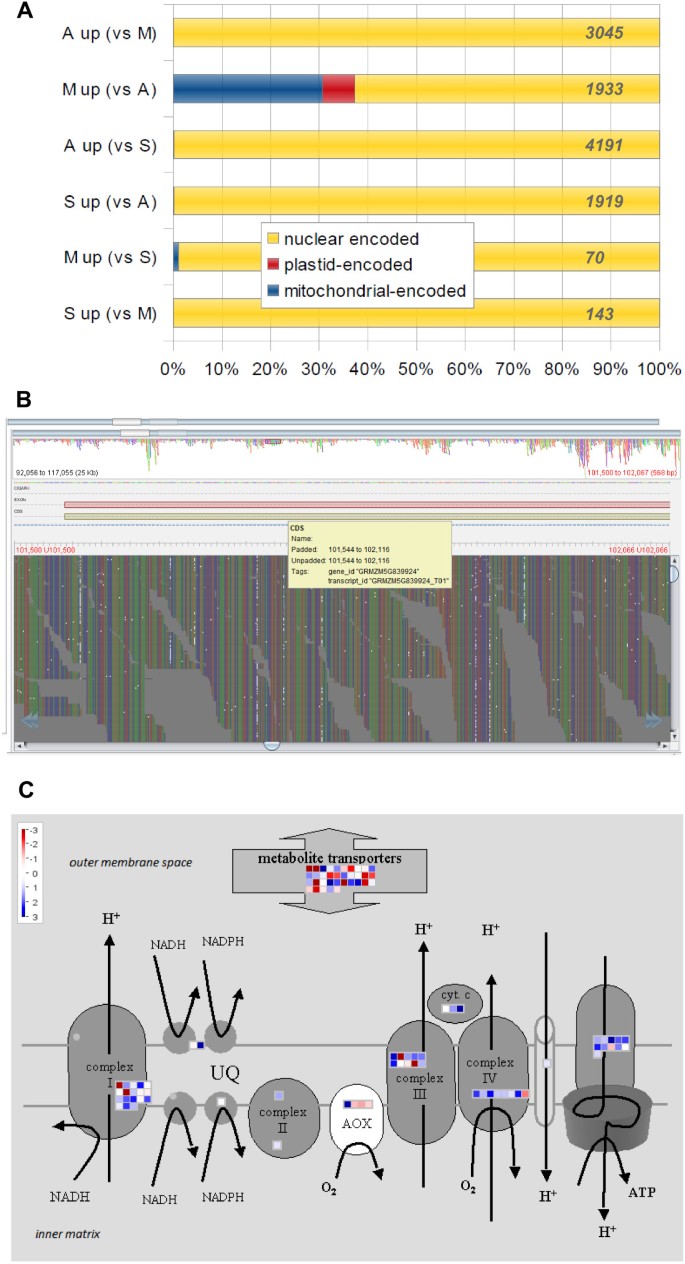
与花药和幼苗相比,我们在分离的减数细胞中检测到大量线粒体功能基因的高表达(图gydF4y2Ba3.gydF4y2BaA、附加文件gydF4y2Ba4gydF4y2Ba:图S2B和附加文件gydF4y2Ba7gydF4y2Ba:图S4)。这些转录本来源于核基因组和线粒体基因组中的基因。在减数细胞与花药中上调的69个基因中,有24个是线粒体编码的,考虑到玉米线粒体中只有58个被识别的基因,这是一个相当大的比例[gydF4y2Ba29gydF4y2Ba为支持这些转录本的线粒体起源,在我们的数据集中对线粒体转录本的仔细检查发现C→U RNA编辑(图2)gydF4y2Ba3.gydF4y2BaB).通过对线粒体染色体的综合SNP分析,我们检测到G→a和C→T转变,它们都转化为C→U RNA编辑,只是链的起源不同(前向和互补)。我们采用了一种精细化的方法,只针对注释基因(123个基因,包括58个描述基因加上新的orf和1个假基因)中的C→U转换,发现多达2%的C被编辑(附加文件)gydF4y2Ba8gydF4y2Ba:表S4)。gydF4y2Ba
与幼苗相比,在减数细胞中,线粒体电子链组成部分的59个核和线粒体编码基因中的35个上调,而只有9个下调,其中3个编码替代氧化酶(图gydF4y2Ba3.gydF4y2BaC,也参见附加文件gydF4y2Ba9gydF4y2Ba:图S5);编码代谢物转运蛋白的基因在减数细胞和幼苗中没有表现出表达偏倚。gydF4y2Ba
减数分裂候选基因的表达水平gydF4y2Ba
我们生成了一个玉米减数分裂候选基因的列表gydF4y2Ba拟南芥gydF4y2Ba已知参与减数分裂的基因编译自[gydF4y2Ba5gydF4y2Ba]和[gydF4y2Ba22gydF4y2Ba].为了在玉米中找到这些基因的同源性,拟南芥的基因被提交到一个使用Phytozome (gydF4y2Bahttp://www.phytozome.netgydF4y2Ba[gydF4y2Ba30.gydF4y2Ba])。根据相似结构域的存在选择假定的玉米同源物,并进一步检查其在玉米RNA-seq数据集中的表达水平。在81个假定的玉米减数分裂基因候选基因中,一些但不是全部被发现在减数细胞中高度上调:24个基因在减数细胞中的表达比在幼苗中至少高5倍,但只有4个基因在减数细胞与花药中表达水平为2倍或更高(附加文件gydF4y2Ba10gydF4y2Ba:表S5,表中举例gydF4y2Ba2gydF4y2Ba).一般来说,在我们的玉米数据集中,减数分裂基因候选基因的良好指标是:1)减数分裂细胞中的表达量至少是幼苗中的5倍(M/S≥5),或如拟南芥[gydF4y2Ba5gydF4y2Ba]gydF4y2Ba,gydF4y2Ba2)减数细胞/花药表达量比幼苗高2倍以上,而花药表达量低于减数细胞的4倍(当M/S和A/S均≥2时,A/M < 4;减数分裂基因表中81个候选基因中有44个为真,与第一标准M/S≥5有很大重叠)。gydF4y2Ba
Mus81gydF4y2Ba是将这些标准用于减数分裂功能候选基因以支持选择减数分裂功能的最佳候选基因的一个例子:gydF4y2BaGRMZM2G361501gydF4y2Ba在减数细胞样本中表达最低,而gydF4y2BaGRMZM5G822970gydF4y2Ba在减数细胞与幼苗的比例几乎是10倍,在减数细胞与花药的比例几乎是2倍。其他例子,比如gydF4y2BaRad51DgydF4y2Ba与幼苗相比,减数细胞表达明显增加gydF4y2BaRad51A1gydF4y2Ba而且gydF4y2BaRad51A2gydF4y2Ba[gydF4y2Ba31gydF4y2Ba]在减数细胞和幼苗中也有高表达,但表达程度较低。检测gydF4y2BaRad51A1gydF4y2Ba而且gydF4y2BaRad51A2gydF4y2Ba证实了使用Phytozome方法的可行性,尽管并不是所有的东西都可以被检测到,例如对于拟南芥gydF4y2BaSWI1 /双gydF4y2Ba基因的搜索发现了玉米类似性gydF4y2BaAm1gydF4y2Ba但不是gydF4y2BaAC194609.2_FG029gydF4y2Ba尽管它们起源于复制事件[gydF4y2Ba32gydF4y2Ba].gydF4y2Ba
通过RNA原位杂交、实时RT PCR和硅片分析验证基因表达及其在减数细胞中的重要性gydF4y2Ba
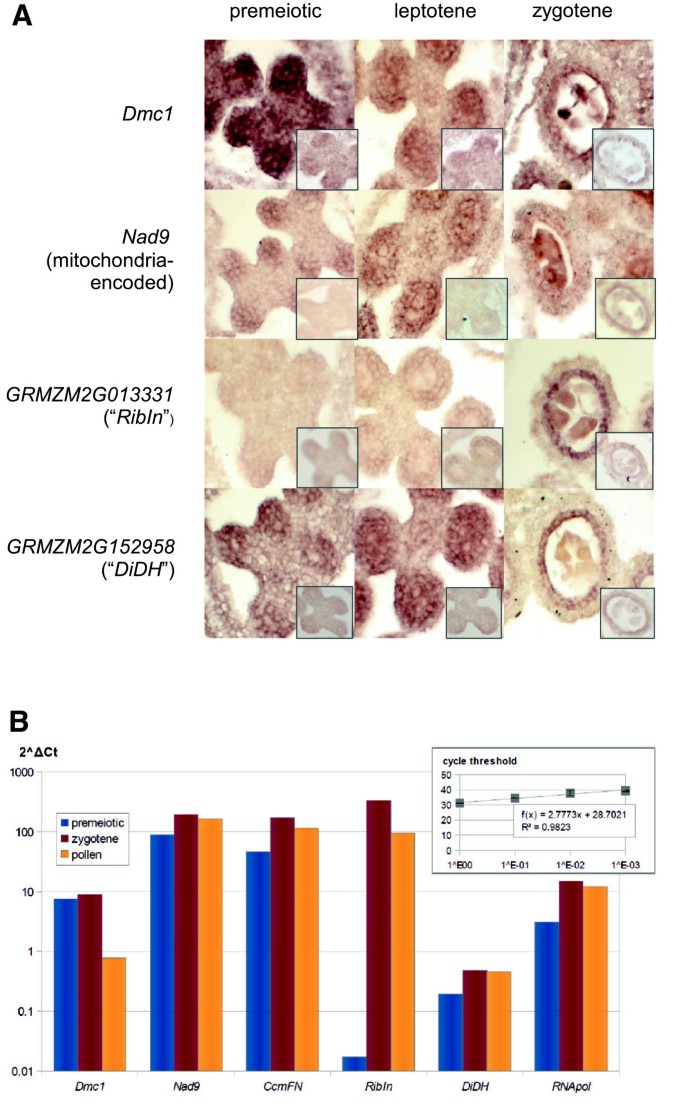
之前在拟南芥中类似的方法也发现了对减数分裂很重要的基因[gydF4y2Ba5gydF4y2Ba],随后进行启动子研究,以证明候选基因的减数细胞特异性表达[gydF4y2Ba33gydF4y2Ba].在这里,我们选择多种方法来验证RNA-seq数据中检测到的基因表达模式。为了将分析扩展到组织特异性的检测,我们选择了几个基因进行进一步的RNA分析gydF4y2Ba原位gydF4y2Ba杂交和实时RT-PCR。我们选择的示例基因包括一个著名的减数分裂基因作为阳性对照,线粒体编码基因在本研究中被发现感兴趣,以及表达水平低的基因,以确定其表达模式。选择RNA的基因gydF4y2Ba原位gydF4y2Ba包括杂交gydF4y2Ba一代gydF4y2Ba(已知对减数分裂重组至关重要[gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,在减数细胞中高度表达),gydF4y2BaNad9gydF4y2Ba(线粒体编码,线粒体电子传递链中复合体I的组成部分,在减数细胞中高度表达),gydF4y2BaGRMZM2G013331gydF4y2Ba(编码一种无特征核糖体失活蛋白,低水平表达)和gydF4y2BaGRMZM2G152958gydF4y2Ba(编码一种未鉴定的二氢脂酰脱氢酶,低水平表达)。核糖核酸gydF4y2Ba原位gydF4y2Ba杂交结果gydF4y2Ba一代gydF4y2Ba而且gydF4y2BaNad9gydF4y2Ba确实表明这两种基因在花药裂片中都有很强的表达gydF4y2Ba4gydF4y2BaA、附加文件gydF4y2Ba11gydF4y2Ba:图S6)。这种信号在减数分裂前期和细端花药中特别强烈,并且集中在减数分裂细胞发育的区域,而不在花药叶之间的结缔组织中。的发生gydF4y2Ba一代gydF4y2Ba减数分裂开始前的表达以前也有报道,例如在小麦中[gydF4y2Ba14gydF4y2Ba].在接合线中,信号更多地局限于减数细胞。gydF4y2Ba一代gydF4y2Ba在绒毡层中也有表达(图gydF4y2Ba4gydF4y2Ba一个)。gydF4y2Ba
核糖核酸gydF4y2Ba原位gydF4y2Ba杂交和实时RT - PCR。(一)gydF4y2Ba核糖核酸gydF4y2Ba原位gydF4y2Ba不同花药发育阶段的花药横截面杂交显示了RNA转录本的位置。信号范围从非常强(gydF4y2Ba一代gydF4y2Ba早减数分裂到不存在(gydF4y2BaGRMZM2G013331gydF4y2Ba减数分裂前的)。面板上较小的插入显示控件(感知探针)。gydF4y2Ba(B)gydF4y2Ba实时RT-PCR分析减数分裂前期、合子线和花粉期整个花药的RNA。表达水平归一化与参考基因HMG (gydF4y2BaGRMZM5G834758gydF4y2Ba),描述为2gydF4y2BaΔCtgydF4y2Ba.gydF4y2Ba一代gydF4y2Ba=减数分裂重组酶DMC1编码;gydF4y2BaNad9gydF4y2Ba= nadh脱氢酶亚基编码,线粒体编码;gydF4y2BaRibIngydF4y2Ba=推定核糖体失活蛋白的编码;gydF4y2BaDiDHgydF4y2Ba=已知的二氢脂酰脱氢酶编码;gydF4y2BaCcmFNgydF4y2Ba=编码细胞色素C生物发生成分,线粒体编码;gydF4y2BaRNApolgydF4y2Ba=编码推定的RNA聚合酶,线粒体编码。gydF4y2Ba
核糖核酸gydF4y2Ba原位gydF4y2Ba杂交与gydF4y2BaGRMZM2G013331gydF4y2Ba而且gydF4y2BaGRMZM2G152958gydF4y2Ba在接合线花药的绒毡层细胞中有表达。此外,gydF4y2BaGRMZM2G152958gydF4y2Ba在减数分裂前期和细端花药的整个花药裂片中表达,其强度与gydF4y2Ba一代gydF4y2Ba而且gydF4y2BaNad9gydF4y2Ba(图gydF4y2Ba4gydF4y2Ba然而,A)。gydF4y2Ba原位gydF4y2Ba杂交作为比较样本中组织的相对定量方法更好,但可能不是比较不同基因表达强度的选择工具,原因是杂交强度差异(这也是微阵列实验的一个缺点)。Real-time PCR和RNA-seq数据通常很一致,可以帮助决定是唯一计数还是分配计数更好地反映实际表达水平(见附加文件)gydF4y2Ba12gydF4y2Ba:表S6)。gydF4y2Ba
为了验证所选基因的表达水平,我们还对整个花药在减数分裂前期、合子线期和花粉期的cdna进行了实时PCR(图gydF4y2Ba4gydF4y2BaB).样品包括四个基因检测gydF4y2Ba原位gydF4y2Ba杂交和两个额外的线粒体编码基因,gydF4y2BaCcmFNgydF4y2Ba(gydF4y2BaGRMZM5G867512gydF4y2Ba编码细胞色素C生物发生途径的一个组成部分)和gydF4y2BaRNApolgydF4y2Ba(gydF4y2BaGRMZM5G827309gydF4y2Ba编码一种推测依赖于dna的RNA聚合酶)。结果与从gydF4y2Ba原位gydF4y2Ba杂化,包括强gydF4y2Ba一代gydF4y2Ba和几乎无法检测到的水平gydF4y2BaGRMZM2G013331gydF4y2Ba在减数分裂前期花药中(另见附加文件)gydF4y2Ba11gydF4y2Ba:图S6,附加文件gydF4y2Ba12gydF4y2Ba:表S6)。gydF4y2Ba
为了验证在减数细胞中优先表达的基因的表达,以及它们的重要性,我们分析了生成的基因表中命名玉米基因的存在,亲缘间描述和减数分裂候选基因(附加文件gydF4y2Ba3.gydF4y2Ba:表S2)。最值得注意的是,我们的减数分裂候选基因中包含了20个gydF4y2BaAm1gydF4y2Ba而且gydF4y2BaPhs1gydF4y2Ba这种基因已经在玉米中被证明参与了减数分裂重组,其缺失导致雄性不育[gydF4y2Ba16gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba36gydF4y2Ba].gydF4y2Ba
讨论gydF4y2Ba
之前的研究已经解决了植物减数分裂转录组的重要问题:基于微阵列的方法在玉米、矮牵牛、小麦和水稻[gydF4y2Ba10gydF4y2Ba,gydF4y2Ba13gydF4y2Ba- - - - - -gydF4y2Ba15gydF4y2Ba]研究了花药的不同发育阶段,并提供了有关特定阶段的转录组动态和基因的宝贵信息。这些和其他研究有助于识别拟南芥、玉米、大麦和水稻的减数分裂基因。在这里,我们通过提供玉米减数分裂候选基因的综合图谱,以及分离的早期减数分裂细胞的表达水平,来补充这些努力。我们目前研究的另一个目标是利用我们从分离的减数细胞中获得的数据,通过转录丰度来检测在早期减数分裂中至关重要的过程。通过对分离的玉米减数分裂细胞细线和接合线的转录组进行测序,我们得到了植物早期减数分裂前期I的表达谱。gydF4y2Ba
分离的减数细胞和完整的花药有非常相似的表达谱,这并不奇怪,因为完整的花药包含减数细胞。之前的一项研究表明,玉米减数细胞RNA贡献了整个花药RNA的20% [gydF4y2Ba16gydF4y2Ba].他们的计算是基于水稻的数据。gydF4y2Ba8gydF4y2Ba]和拟南芥[gydF4y2Ba5gydF4y2Ba],估计水稻PMCs和拟南芥减数细胞的RNA分别是典型二倍体细胞的25倍和100倍[gydF4y2Ba16gydF4y2Ba].在玉米中,pmc只占总花药细胞的不到1%,但因为它们很大(约占花药体积的10%)[gydF4y2Ba16gydF4y2Ba],使用整个花药来获取减数分裂转录组的信息是合理的。我们的数据显示,至少在玉米中,减数细胞对整个花药转录组格局的贡献可能远远大于之前的假设。考虑到这一点,可以重新分析之前的转录组花药数据,未来针对减数分裂转录组的RNA水平研究可以合理地使用整个花药而不是孤立的减数细胞。然而,使用分离的减数细胞产生了前所未有的分解和对特定表达模式的新见解。此外,在基于dna的实验中,减数细胞对整个花药的贡献要低得多,因此强烈建议使用减数细胞。gydF4y2Ba
减数细胞中线粒体转录物的丰富gydF4y2Ba
与拟南芥不同,拟南芥在减数细胞和花药之间发现了显著的基因表达图谱差异[gydF4y2Ba5gydF4y2Ba分离的玉米减数细胞和完整的花药具有非常相似的表达谱。我们在早期前期I减数细胞中检测到线粒体转录本显著增加,这在整个花药数据中并不明显。细胞器转录本以前在其他转录组研究中也遇到过[gydF4y2Ba37gydF4y2Ba,但通常不作进一步解释就不予理会,或被视为人工制品[gydF4y2Ba27gydF4y2Ba].根据经典的观点,在文库准备过程中polyA的选择确实应该去除大多数线粒体mrna。然而,仅在细胞核转录本上稳定3'polyA和在细菌和细胞器转录本上瞬时降解靶向内外polyA的经典观点最近受到了各种研究的挑战[gydF4y2Ba37gydF4y2Ba- - - - - -gydF4y2Ba39gydF4y2Ba].在最近的拟南芥研究中,发现了与增强耐热性相关的线粒体基因聚腺苷酸转录本的强烈增加[gydF4y2Ba37gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba]以及一些关于植物发育的研究发现特定线粒体基因的转录水平升高[gydF4y2Ba42gydF4y2Ba- - - - - -gydF4y2Ba46gydF4y2Ba].解释有基因剂量效应、转录激活和更高的稳定性等,但其分子基础和意义尚未阐明。gydF4y2Ba
在植物中,有一个持续的线粒体基因组片段洗牌到细胞核,导致numt (gydF4y2BaνgydF4y2Ba清晰的编码gydF4y2Ba米gydF4y2Ba我gydF4y2BatgydF4y2Ba线粒体DNA),可通过插入整个或部分线粒体基因组(DNA)或经过处理的转录本(mRNA)而发生[gydF4y2Ba47gydF4y2Ba- - - - - -gydF4y2Ba50gydF4y2Ba].gydF4y2Ba
必须提出的问题是,在我们的RNA-seq中检测到的线粒体基因转录物是否源于numt(也称为MGI),gydF4y2Ba米gydF4y2BaitochondrialgydF4y2BaggydF4y2BaenomegydF4y2Ba我gydF4y2BaNsertion)或直接来自线粒体。我们在RNA-seq数据中检测到高比例的编辑线粒体转录本,这指向线粒体起源。大约三分之一在减数细胞和花药中高表达的基因编码在线粒体中,但在线粒体中具有功能的核编码基因也在减数细胞和幼苗中上调。我们假设,这是前期I早期旺盛的染色体运动发生时的高能量需求的一个迹象[gydF4y2Ba51gydF4y2Ba].因此,我们的数据表明,编码糖酵解步骤和线粒体电子传递链的蛋白质的基因在减数细胞中表达水平显著增加。来自其他生物的一些研究也指出了线粒体过程对减数分裂配对过程的重要性gydF4y2Ba秀丽隐杆线虫gydF4y2Ba而且gydF4y2Ba粟酒裂殖酵母gydF4y2Ba染色体配对或重组依赖于线粒体呼吸[gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba].线粒体基因对减数分裂或减数分裂后过程的功能要求在细胞质雄性不育现象中得到强调,这是由于线粒体基因突变,已在玉米和其他植物中报道[gydF4y2Ba54gydF4y2Ba- - - - - -gydF4y2Ba59gydF4y2Ba].尽管这些线索表明了线粒体定位过程对减数分裂的重要性,但之前的两项拟南芥减数细胞转录组研究都没有指出这一点。没有明确提到任何线粒体基因上调在减数细胞,杨gydF4y2Ba等gydF4y2Ba.[gydF4y2Ba22gydF4y2Ba]仍然列出了18个最丰富的PFAM家族,包括mitto - carr(线粒体载体)和TPR_1和TPR_2(四肽重复序列),它们可以在NADPH氧化酶亚基中找到,作为线粒体输入蛋白的受体。陈gydF4y2Baet al。gydF4y2Ba[gydF4y2Ba5gydF4y2Ba]注意到线粒体来源转录本的增加,但将其归因于线粒体基因组插入(MGI),因为读取映射到该区域。一个新的分析,比较gydF4y2Ba拟南芥gydF4y2Ba我们实验室最近生成的玉米数据显示,大量的转录本gydF4y2Ba拟南芥gydF4y2Ba也直接来源于线粒体基因组[gydF4y2Ba24gydF4y2Ba].gydF4y2Ba
减数分裂早期氧化还原稳态和染色质修饰gydF4y2Ba
在减数细胞中高度表达的两组基因也值得密切关注。一个是“内稳态”,包括编码硫氧还蛋白和戊氧还蛋白的基因。氧化还原状态最近被认为是减数分裂前花药发育中细胞命运的决定因素[gydF4y2Ba60gydF4y2Ba].建立生殖细胞起始的一个关键因素是gydF4y2BaMsca1 (gydF4y2Ba米gydF4y2Ba啤酒gydF4y2Ba年代gydF4y2BaterilegydF4y2BacgydF4y2BaonvertedgydF4y2Ba一个gydF4y2BangydF4y2Ba1gydF4y2Ba)gydF4y2Ba[gydF4y2Ba60gydF4y2Ba它编码一种glutaredoxin,也在我们的“内稳态”基因列表中,其表达在早期减数分裂中上调,此外还有另外两种假定的glutaredoxin和5种硫oredoxin(附加文件gydF4y2Ba5gydF4y2Ba表S3)。硫氧还蛋白是普遍存在的二硫调节蛋白,似乎将氧化还原状态与多细胞生物发育过程中的细胞命运和生长联系在一起[gydF4y2Ba61gydF4y2Ba,gydF4y2Ba62gydF4y2Ba],在拟南芥中,硫氧还蛋白Trx H9甚至似乎能够进行细胞-细胞迁移和通信[gydF4y2Ba63gydF4y2Ba].检测到的氧化还原调控因子候选人可能不仅需要建立生殖细胞,而且还需要维持,推进,特别是同步以后的减数分裂过程。gydF4y2Ba
另一个富集的过程组是“染色质”,表明染色质相关基因的表达可能代表减数分裂重组的一个环节。例如,组蛋白3赖氨酸4三甲基化(H3K4me3)标志着小鼠双链断裂热点[gydF4y2Ba64gydF4y2Ba],组蛋白乙酰化通常与重组活性区域相联系[gydF4y2Ba65gydF4y2Ba- - - - - -gydF4y2Ba67gydF4y2Ba].我们的数据提供了可能影响重组的减数分裂特异性组蛋白修饰物的候选列表,包括两个SET结构域蛋白和两个来自组蛋白去乙酰化酶超家族的蛋白质gydF4y2Ba5gydF4y2Ba表S3)。gydF4y2Ba
蛋白质定位和降解gydF4y2Ba
我们还发现减数细胞中与分子靶向、定位和蛋白质水解相关的过程显著丰富。虽然一些参与定位过程的基因可能是由于丰富的线粒体转录本,但也与蛋白质水解相关的成分有关。泛素化主要与蛋白质水解有关,在植物中具有多种作用,对调节生长和发育很重要,综述在[gydF4y2Ba68gydF4y2Ba,gydF4y2Ba69gydF4y2Ba],具有调节细胞定位和活性的非降解功能,在[gydF4y2Ba70gydF4y2Ba].单泛素化作用与膜转运和转录过程的调控有关[gydF4y2Ba71gydF4y2Ba],使参与这些过程的减数细胞中的基因上调也成为进一步研究的有趣候选对象。gydF4y2Ba
结论gydF4y2Ba
生成的玉米早期减数分裂特异性转录组数据集是了解减数分裂过程的宝贵资源。使用它,我们能够揭示早期减数分裂的新和未被认识的方面,如高能生产,er连接过程和RNA调控。除了识别新的减数分裂基因候选基因外,该数据集还有助于区分减数分裂花药中减数细胞和其他组织中功能的基因,从而深入了解减数分裂花药中减数细胞和体细胞之间的相互作用。综上所述,未来的研究可能旨在研究本文中检测到的过程是如何相互联系的,以及它们在早期减数分裂过程中发挥何种调节作用。gydF4y2Ba
方法gydF4y2Ba
玉米雄性减数细胞的植物材料与分离gydF4y2Ba
玉米的植物gydF4y2Ba(玉米)gydF4y2BaB73自交系在温室内,光照16 h (~450 μmol × m)gydF4y2Ba-2gydF4y2Ba×交会gydF4y2Ba-1gydF4y2Ba),在24°C和22°C的黑暗8小时,在表层土壤和SunGro LC8(2:1)的混合物中,施肥约30 g缓释肥料(渗透压14-14-14)和每两周添加约1-2 g Peterson’s 20:20:20溶于水。为了确定减数分裂阶段,我们使用了Sheehan和Pawlowski(2012)中描述的乙酰红胺染色,并使用氧化铁强化。我们只收集减数细胞和早期前期I的花药(细线和接合线),将它们储存在RNA中gydF4y2Ba晚些时候gydF4y2Ba.用于实验的花药来自于用于减数细胞收集的同一株植物(每次重复约10-15株)。植物减数细胞的分离遵循CCM (gydF4y2BaCgydF4y2BaapillarygydF4y2BaCgydF4y2Baollection的gydF4y2Ba米gydF4y2Ba拟南芥的描述[gydF4y2Ba5gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba22gydF4y2Ba和玉米[gydF4y2Ba72gydF4y2Ba],在将分离的细胞浸入RNA之前,处理刚摘下的小穗不超过20-30分钟gydF4y2Ba晚些时候gydF4y2Ba.以玉米为例,有早期减数分裂阶段的花药(细线和接合线)被解剖并用扁平的移液管尖端压扁。平均每小时收集约800个细胞,大约2 -3万个细胞用于RNA提取(RNA产量见附加文件)gydF4y2Ba1gydF4y2Ba:表S1)。对于每个幼苗复制,使用三个大约两周大的有三片叶子的幼苗。所有生物重复均在温室内独立时间取样。gydF4y2Ba
RNA提取和测序gydF4y2Ba
使用Ambion RNAqueous Micro Kit (Ambion)从减数细胞、整个花药和幼苗中提取总RNA。使用量子比特®RNA BR检测试剂盒和量子比特®荧光计(Invitrogen)测量RNA量。gydF4y2Ba
RNA文库的制备采用标准的Illumina TruSeq RNA文库试剂盒,该试剂盒选择polyA RNA。文库在Illumina HiSeq 2000仪器上测序,产生单端50 nt长reads。序列用标准的Illumina管道进行过滤,附加的过滤步骤用于识别适配器(附加文件gydF4y2Ba13gydF4y2Ba:表S7)。gydF4y2Ba
转录组分析gydF4y2Ba
用GSNAP v 2011_03_28将每个样本的Reads与玉米B73基因组参考文献(RefGenv2,注释版本5b.60)对齐[gydF4y2Ba73gydF4y2Ba除了max-mismatches = 2, indl -penalty = 2, novelsplicing = 1, localsplicedist = 1000, disttsplicpunishment = 1000, terminal-penalty = 1000, npaths = 10,已知的拼接位点被输入到对齐中。基因读取计数使用NCGR开发的管道生成[gydF4y2Ba74gydF4y2Ba].只考虑给定读取的最佳匹配。如果一个基因映射到该基因的坐标上,那么该基因就会被指定为Reads,包括重叠但不完全包含在该基因内的Reads。不同的剪接异构体没有单独考虑。对于用于基因分析的唯一读取计数,只考虑单个最佳命中的读取。对于TE分析以及比较基因和TEs的分析,使用相同的参数重新运行GSNAP校准,除了最多返回100条路径(npaths = 100)。与以前一样,只考虑最佳对齐。我们不是只考虑唯一对齐的读取,而是通过将它们按比例分配到所有可能的基因,来解释映射不明确的读取(读取可以同样很好地映射到多个基因)。以分级的方式将Reads分配给基因,从只有一个匹配的Reads开始,然后是两个匹配的Reads,以此类推。考虑对齐模糊性对TEs很重要,因为它们本质上是重复的。 Indeed, there was nearly a three-fold increase in the number of TEs showing expression with these criteria compared to using only unique hits. Gene-based analyses changed minimally with the two criteria sets (< 4% increase in the number of genes expressed in each replicate with an average of 1.6%). Therefore, to be able to compare directly between genes and TEs in analyses that involved both genes and TEs the apportioned read data were used.
对于基于基因的差异表达分析,使用唯一计数数据。去除所有零表达的基因后,只考虑归一化数据的前70%进行大多数分析,包括检测差异表达基因,类似于[gydF4y2Ba75gydF4y2Ba].当考虑所有唯一计数而不是修剪集(花药:0.9419修剪/0.9438全部;减数细胞:修剪0.9174 /全部0.9187;幼苗:0.7990修剪/0.8031全部)。使用JMP®Genomics(版本6.0,64位版),用Ward方法测试生物重复之间相关性的层次聚类。用R[的DEseq包进行差异表达基因的检测分析gydF4y2Ba76gydF4y2Ba作为Bioconductor R工具套件的一部分公开提供。使用DEseq包与裁剪后的数据进行MA比值计算和绘图,并生成样本间表达差异显著的基因列表,多次检测经Benjamini-Hochberg校正后调整P值≤0.01 [gydF4y2Ba77gydF4y2Ba].当使用带有调整的差异表达基因列表时,得到了类似的结果gydF4y2BaPgydF4y2Ba值≤0.05。使用JMP Genomics计算和描述Venn图,使用完整的数据归一化阅读每百万读数(RPM)或r生成的差异表达基因列表,除了使用r生成的没有修改的列表,进一步的减数分裂基因定义(M/A > 2,或A/M < 4,如果M/S和A/S都≥2)应用于花药或减数细胞与幼苗中上调的差异表达基因的组合列表。gydF4y2Ba
为了进行GO(基因本体)分析,将差异表达基因列表提交到AgriGO GO工具[gydF4y2Ba28gydF4y2Ba),gydF4y2Bahttp://bioinfo.cau.edu.cn/agriGO/gydF4y2Ba对于奇异富集分析,默认设置为Fisher精确测试,调整为多次测试与Benjamini-Yekutieli方法和MapMan [gydF4y2Ba27gydF4y2Ba].gydF4y2Ba
对于TE分析,文件ZmB73_5a_MTEC_repeats. zip的修改版本。gff (maizesequence.org) where duplicates had been removed was used to generate read counts for the annotated ~1.7 million TE locations. TEs with less than or equal to five reads total in our three tissue sources were excluded. This equals a 23.5% upper cut instead of the 70% used with the gene data set. Downstream analyses were performed with lists of differentially expressed TEs generated with DEseq, and with a list of TEs designated as meiosis-specific. For the latter one, we used only highly expressed TEs (row sum > 1000), and applied the same definition criteria as for genes (M/A > 2, or A/M < 4, if M/S and A/S both ≥ 2), and used the remaining TEs for comparison.
使用Tablet Viewer Version 1.12.08.29对基因组的对齐读取进行可视化[gydF4y2Ba78gydF4y2Ba],并装载参考基因特征进行比较。Tablet Viewer还用于线粒体基因RNA编辑的检测和验证。gydF4y2Ba
RNA原位杂交gydF4y2Ba
设计引物,将完整CDS克隆到pGEM-Teasy载体上,使用DIG RNA标记试剂盒(Roche)与RNA聚合酶T7和SP6进行探针合成。RNAgydF4y2Ba原位gydF4y2Ba杂交过程严格遵循Yong等人(2003)描述的方案,并进行了少量修改。全株玉米穗在真空下冰上固定1小时,然后在4℃的固定液中保存一夜。用组织清液代替二甲苯进行清除,检测反应前用低严格度缓冲液(2× SSC)对载玻片进行RNAse处理和洗涤。所有探头在黑暗中显影3小时,并给出足够的信号。gydF4y2Ba
实时RT - PCRgydF4y2Ba
将B73玉米花药进行分期、解剖并保存在RNA中gydF4y2Ba晚些时候gydF4y2Ba.处理前,花药立即在1xPBS中洗涤,然后使用Qiagen RNeasy®Mini Kit进行RNA提取。dna酶消化使用Optizyme rdnasi (Fisher Scientific)进行。使用Qubit®RNA BR检测试剂盒(Invitrogen)测定的每个样品约2.5 μg总RNA用于cDNA合成,并使用包含寡聚dT引物的SuperScript®III第一链合成系统(Invitrogen)进行RT-PCR cDNA合成。在实时PCR中,使用iQ™SYBR®Green Supermix (Bio-Rad),每次反应含有约125 ng总RNA和25 μl末端体积的cDNA,在3个副本中进行。使用的引物列在附加文件中gydF4y2Ba12gydF4y2Ba:表S6。用Real-time PCR Miner计算引物效率[gydF4y2Ba79gydF4y2Ba),gydF4y2Bahttp://www.miner.ewindup.info/gydF4y2Ba每个基因在90-110%的范围内。Bio-Rad real-time PCR系统(C100TM Thermal Cycler,带有CFX96™实时系统)与Bio-Rad CFX Manager™软件2.0版本一起使用。gydF4y2Ba
支持数据的可用性gydF4y2Ba
原始的Illumina读取已经被存入NCBI的SRA(序列读取档案),研究标题为“mRNA-seq ofgydF4y2Ba玉米gydF4y2BaB73早期前期减数细胞、花药和幼苗控制”,注册号SRX218264-218270。gydF4y2Ba
参考文献gydF4y2Ba
- 1.gydF4y2Ba
王晓燕,王晓燕,王晓燕:植物减数分裂前期I的遗传学研究。植物生态学报,2006,29(4):366 - 366。10.1146 / annurev.arplant.57.032905.105255。gydF4y2Ba
- 2.gydF4y2Ba
Ma H:分子肖像gydF4y2Ba拟南芥gydF4y2Ba减数分裂。阿拉伯的书。编辑:Somerville CR, Meyerowitz EM. 2006, Rockville, MD:美国植物生物学家协会,1-39。gydF4y2Ba
- 3.gydF4y2Ba
植物减数分裂:十年的基因发现。中国生物医学工程学报,2008,29(4):359 - 359。10.1159 / 000121077。gydF4y2Ba
- 4.gydF4y2Ba
de Muyt A, Pereira L, Vezon D, Chelysheva L, Gendrot G, Chambon A, Lainé-Choinard S, Pelletier G, Mercier R, Nogué F, Grelon M:高通量基因筛选发现拟南芥早期减数分裂重组功能。科学通报,2009,5:e1000654-10.1371/journal.pgen.1000654。gydF4y2Ba
- 5.gydF4y2Ba
Chen C, Farmer AD, Langley RJ, Mudge J, Crow JA, May GD, Huntley J, Smith AG, Retzel EF:植物减数分裂特异性基因的发现:RNA-Seq应用于分离的拟南芥雄性减数细胞。植物生物学杂志,2010,10:280-10.1186/1471-2229-10-280。gydF4y2Ba
- 6.gydF4y2Ba
王晓燕,王晓燕,王晓燕,王晓燕。拟南芥减数分裂重组途径的研究。中国植物学报,2011,29(5):529 -544。10.1111 / j.1469-8137.2011.03665.x。gydF4y2Ba
- 7.gydF4y2Ba
Bovill WD, Deveshwar P, Kapoor S, Able JA:全基因组方法识别谷物早期减数分裂基因候选基因。功能整合基因组学,2009,9:219-229。10.1007 / s10142 - 008 - 0097 - 4。gydF4y2Ba
- 8.gydF4y2Ba
唐旭,张志勇,张文杰,赵晓明,李旭,张东,刘启强,唐文华:激光捕获的水稻花粉母细胞整体基因谱分析揭示了水稻减数分裂的分子通路和基因亚家族。植物生理学报,2010,32(4):359 - 364。10.1104 / pp.110.161661。gydF4y2Ba
- 9.gydF4y2Ba
Wijeratne AJ,张伟,孙颖,刘伟,Albert R,郑铮,Oppenheimer DG,赵丹,马红:拟南芥野生型和突变型花药的差异基因表达:对花药细胞分化和调控网络的认识。植物学报,2007,32(4):447 - 447。10.1111 / j.1365 - 313 x.2007.03217.x。gydF4y2Ba
- 10.gydF4y2Ba
Cnudde F, Hedatale V, de Jong H, Pierson ES, Rainey DY, Zabeau M, Weterings K, Gerats T, Peters JL:矮矮矮植物雄性减数分裂过程中基因表达的变化。染色体研究,2006,14:919-932。10.1007 / s10577 - 006 - 1099 - 5。gydF4y2Ba
- 11.gydF4y2Ba
王铮,梁勇,李超,徐勇,兰兰,赵东,陈超,徐铮,薛勇,冲凯:水稻花药发育相关基因表达的微阵列分析。植物生物学杂志,2004,24(3):378 - 378。10.1007 / s11103 - 005 - 8267 - 4。gydF4y2Ba
- 12.gydF4y2Ba
黄明东,魏凤娟,吴春春,邢永成,黄ahc:对水稻高级花药转录组的分析揭示了整体绒毡层的分泌功能和脂质外膜形成的潜在蛋白。植物生理学报,2009,29(4):369 - 371。gydF4y2Ba
- 13.gydF4y2Ba
Deveshwar P, Bovill WD, Sharma R, Able JA, Kapoor S:分析水稻花药转录组,识别参与减数分裂和雄性配子体发育的基因。植物生物学杂志,2011,11:78-10.1186/1471-2229-11-78。gydF4y2Ba
- 14.gydF4y2Ba
Crismani W, Baumann U, Sutton T, Shirley N, Webster T, Spangenberg G, Langridge P, Able JA:六倍体面包小麦减数分裂和小孢子发生的微阵列表达分析。中国生物医学工程学报,2006,26(3):366 - 366。gydF4y2Ba
- 15.gydF4y2Ba
马杰,Skibbe DS, Fernandes J, Walbot V:玉米雄蜂生殖发育:花药和花粉个体发生的基因表达谱。中国生物技术学报,2008,9:R181-10.1186/gb-2008-9-12-r181。gydF4y2Ba
- 16.gydF4y2Ba
南G-L, Ronceret A, Wang RC -1, Fernandes JF, Cande WZ, Walbot V:玉米花药中两个无穗数分裂等位基因的转录组分析。植物生物学杂志,2011,11:120-10.1186/1471-2229-11-120。gydF4y2Ba
- 17.gydF4y2Ba
Alves-Ferreira M, Wellmer F, Banhara A, Kumar V, Riechmann JL, Meyerowitz EM:全局表达谱在拟南芥雄蕊发育分析中的应用。植物生理学报,2007,29(4):347 - 347。10.1104 / pp.107.104422。gydF4y2Ba
- 18.gydF4y2Ba
Ma J, Duncan D, Morrow DJ, Fernandes J, Walbot V:利用遗传消融分析玉米花药减数分裂前和绒毡层细胞类型的转录组分析。植物学报,2007,30(4):369 - 371。10.1111 / j.1365 - 313 x.2007.03074.x。gydF4y2Ba
- 19.gydF4y2Ba
Wang D, Oses-Prieto JA, Li KH, Fernandes JF, Burlingame AL, Walbot V:玉米雄性不育8突变扰乱了转录组的时间进程,导致代谢功能的调控失调。植物学报,2010,32(4):339 - 351。10.1111 / j.1365 - 313 x.2010.04294.x。gydF4y2Ba
- 20.gydF4y2Ba
陈超,rezel EF:利用分离的减数分裂细胞进行减数分裂转录组分析gydF4y2Ba拟南芥gydF4y2Ba.植物减数分裂方法。编辑:Pawlowski W, Grelon M, Armstrong SS. 2013,纽约:Humana出版社,203-213。gydF4y2Ba
- 21.gydF4y2Ba
Libeau P, Durandet M, Granier F, Marquis C, Berthomé R, Renou JP, taconnatat - soubirou L, Horlow C:拟南芥减数细胞基因表达谱分析。植物生物学(Stuttg)。2011, 13: 784-793。10.1111 / j.1438-8677.2010.00435.x。gydF4y2Ba
- 22.gydF4y2Ba
杨红,陆萍,王莹,马红:拟南芥雄性减数分裂细胞转录组图谱的高通量测序:减数分裂过程的复杂性和进化。植物学报,2011,29(5):563 - 566。10.1111 / j.1365 - 313 x.2010.04439.x。gydF4y2Ba
- 23.gydF4y2Ba
李晓燕,李晓燕。减数分裂过程中的染色体动力学。细胞分裂控制植物。编辑:Verma DPS, Hong Z. 2007,海德堡:施普林格,103-124。gydF4y2Ba
- 24.gydF4y2Ba
Dukowic-Schulze S, Harris A, Li J, Sundarajan A, Mudge J, Retzel E, Pawlowski W, Chen C:拟南芥和玉米早期减数分裂的转录组学比较。中国基因工程学报,2014,41:139-152。10.1016 / j.jgg.2013.11.007。gydF4y2Ba
- 25.gydF4y2Ba
Schnable PS、Ware D、Fulton RS、Stein JC、Wei F、Pasternak S、Liang C、Zhang J、Fulton L、Graves TA、Minx P、Reily AD、Courtney L、Kruchowski SS、Tomlinson C、Strong C、Delehaunty K、Fronick C、Courtney B、Rock SM、Belter E、Du F、Kim K、Abbott RM、Cotton M、Levy A、Marchetto P、Ochoa K、Jackson SM、Gillam B等:玉米B73基因组:复杂性、多样性和动态。科学通报,2009,36(5):527 - 527。10.1126 / science.1178534。gydF4y2Ba
- 26.gydF4y2Ba
玉米转座因子的转录活性。中国生物医学工程学报,2010,26(6):694 - 694。gydF4y2Ba
- 27.gydF4y2Ba
Thimm O, Bläsing O, Gibon Y, Nagel A, Meyer S, Krüger P, Selbig J, Müller LA, Rhee SY, Stitt M: MAPMAN:一种用户驱动的工具,将基因组数据集显示在代谢途径和其他生物过程的图表上。植物学报,2004,37:914-939。10.1111 / j.1365 - 313 x.2004.02016.x。gydF4y2Ba
- 28.gydF4y2Ba
张凌Du Z,周X, Y, Z,苏Z: agriGO:去分析工具箱农业社区。核酸学报,2010,38:W64-W70。10.1093 / nar / gkq310。gydF4y2Ba
- 29.gydF4y2Ba
Clifton SW、Minx P、Fauron CM-R、Gibson M、Allen JO、Sun H、Thompson M、Barbazuk WB、Kanuganti S、Tayloe C、Meyer L、Wilson RK、Newton KJ:玉米NB线粒体基因组序列与比较分析。植物生理学报,2004,24(3):366 - 366。10.1104 / pp.104.044602。gydF4y2Ba
- 30.gydF4y2Ba
Goodstein DM, Shu S, Howson R, Neupane R, Hayes RD, Fazo J, Mitros T, Dirks W, Hellsten U, Putnam N, Rokhsar DS: Phytozome:绿色植物基因组学比较平台。核酸学报,2012,40:D1178-D8116。10.1093 / nar / gkr944。gydF4y2Ba
- 31.gydF4y2Ba
Li J, Harper LC, Golubovskaya I, Wang CR, Weber D, Meeley RB, McElver J, Bowen B, Cande WZ, Schnable PS:玉米RAD51在减数分裂和双链断裂修复中的功能分析。遗传学杂志,2007,176:1469-1482。10.1534 / genetics.106.062604。gydF4y2Ba
- 32.gydF4y2Ba
Pawlowski WP, Wang C-JR, Golubovskaya IN, Szymaniak JM, Shi L, Hamant O, Zhu T, Harper L, Sheridan WF, Cande WZ:玉米AMEIOTIC1在多个早期减数分裂过程中是必不可少的,可能是开始减数分裂的必要条件。中国生物工程学报,2009,29(3):369 - 369。10.1073 / pnas.0810115106。gydF4y2Ba
- 33.gydF4y2Ba
李娟,Farmer AD, Lindquist IE, Dukowic-Schulze S, Mudge J,李婷,Retzel EF,陈c:一组拟南芥减数分裂活性启动子的研究。植物工程学报,2012,29(3):366 - 366。gydF4y2Ba
- 34.gydF4y2Ba
Couteau F, Belzile F, Horlow C, Grandjean O, Vezon D, Doutriaux MP:拟南芥dmc1突变体雄性和雌性减数分裂细胞中无减数分裂停止的随机染色体分离。植物学报,1999,11:1623-1634。10.1105 / tpc.11.9.1623。gydF4y2Ba
- 35.gydF4y2Ba
Kurzbauer M-T, Uanschou C, Chen D, Schlögelhofer P:拟南芥减数分裂过程中重组酶DMC1和RAD51在功能和空间上分离。植物学报,2012,24:2058-2070。10.1105 / tpc.112.098459。gydF4y2Ba
- 36.gydF4y2Ba
Ronceret A, Doutriaux M-P, Golubovskaya IN, Pawlowski WP: PHS1通过控制RAD50到细胞核的运输来调节减数分裂重组和同源染色体配对。美国国家科学研究院。2009, 106: 20121-20126。10.1073 / pnas.0906273106。gydF4y2Ba
- 37.gydF4y2Ba
Adamo A, Pinney JW, Kunova A, Westhead DR, Meyer P:热胁迫增强拟南芥多聚腺苷化线粒体转录本的积累。科学通报,2008,3:e2889-10.1371/journal.pone.0002889。gydF4y2Ba
- 38.gydF4y2Ba
LaCava J, Houseley J, Saveanu C, Petfalski E, Thompson E, Jacquier A, Tollervey D:外泌体对RNA的降解是由核聚腺苷化复合体促进的。细胞科学,2005,21:713-724。10.1016 / j.cell.2005.04.029。gydF4y2Ba
- 39.gydF4y2Ba
Slomovic S, Laufer D, Geiger D, Schuster G:人类线粒体RNA的聚腺苷酸化和降解:原核生物的过去留下了它的印记。中国生物医学工程学报,2005,29(4):447 - 447。10.1128 / mcb.25.15.6427 - 6435.2005。gydF4y2Ba
- 40.gydF4y2Ba
Shedge V, Davila J, Arrieta-Montiel MP, Mohammed S, Mackenzie SA:拟南芥线粒体基因组的广泛重排引发了细胞的耐热性条件。植物生理学报,2010,32(4):366 - 366。10.1104 / pp.109.152827。gydF4y2Ba
- 41.gydF4y2Ba
Kim M, Lee U, Small I, des Francs-Small CC, Vierling E:在缺少主要分子伴侣HSP101的情况下,拟南芥线粒体转录终止因子相关蛋白的突变增强了耐热性。植物学报,2012,24:3349-3365。10.1105 / tpc.112.101006。gydF4y2Ba
- 42.gydF4y2Ba
小麦叶片发育过程中线粒体基因的表达。植物学报,1990,32(4):369 - 371。10.1007 / BF02411391。gydF4y2Ba
- 43.gydF4y2Ba
Monéger F, Mandaron P, Niogret MF, Freyssinet G, Mache R:玉米小孢子发生过程中叶绿体和线粒体基因的表达。植物生理学报,1999,19(4):369 - 371。10.1104 / pp.99.2.396。gydF4y2Ba
- 44.gydF4y2Ba
聪明CJ, Monéger F, Leaver CJ:向日葵花药发育过程中线粒体基因表达的细胞特异性调控。植物生态学报,1998,16(6):557 - 557。10.1105 / tpc.6.6.811。gydF4y2Ba
- 45.gydF4y2Ba
Logan DC, Millar AH, Sweetlove LJ, Hill SA, Leaver CJ:玉米胚萌发过程中的线粒体生物发生。植物生理学报,2001,29(3):366 - 366。10.1104 / pp.125.2.662。gydF4y2Ba
- 46.gydF4y2Ba
杨建军,张建军,张建军,等:水稻萌发过程中线粒体的有序组装始于富含蛋白质输入体成分的前线粒体结构。植物生物学杂志,2006,30(4):371 - 371。10.1007 / s11103 - 005 - 3688 - 7。gydF4y2Ba
- 47.gydF4y2Ba
Adams KL, Daley DO, Qiu YL, Whelan J, Palmer JD:开花植物线粒体基因向细胞核的重复、近期和多样化转移。自然学报,2000,408:354-357。10.1038 / 35042567。gydF4y2Ba
- 48.gydF4y2Ba
帕默JD,亚当斯KL,曹Y,帕金森CL,秋黄,K歌:动态植物线粒体基因组的进化:移动基因和内含子和高度变量变异率。中国生物医学工程学报,2000,29(3):366 - 366。10.1073 / pnas.97.13.6960。gydF4y2Ba
- 49.gydF4y2Ba
Henze K, Martin W:线粒体基因是如何进入细胞核的?研究进展,2001,17:383-387。10.1016 / s0168 - 9525(01) 02312 - 5。gydF4y2Ba
- 50.gydF4y2Ba
Lough AN, Roark LM, Kato A, Ream TS, Lamb JC, Birchler JA, Newton KJ:玉米线粒体DNA转移到细胞核产生广泛的插入位点变异。遗传学报,2008,38(4):347 - 355。10.1534 / genetics.107.079624。gydF4y2Ba
- 51.gydF4y2Ba
WP:植物减数分裂过程中的染色体动态成像。方法酶学杂志,2012,505:125-143。gydF4y2Ba
- 52.gydF4y2Ba
Jambhekar A, Amon A:通过呼吸控制减数分裂。中华微生物学杂志,2008,18:969-975。10.1016 / j.cub.2008.05.047。gydF4y2Ba
- 53.gydF4y2Ba
Labrador L, Barroso C, Lightfoot J, Muller-Reichert, Flibotte S, Taylor J, Moerman DG, Villeneuve AM, Martinez-Perez E:同源性搜索需要线粒体蛋白SPD-3促进的染色体运动gydF4y2Ba秀丽隐杆线虫gydF4y2Ba减数分裂。科学通报,2013,9:e1003497-10.1371/journal.pgen.1003497。gydF4y2Ba
- 54.gydF4y2Ba
玉米花粉不育基因。遗传学杂志,1931,17:413-431。gydF4y2Ba
- 55.gydF4y2Ba
Young EG, Hanson MR:一个与细胞质雄性不育相关的融合线粒体基因受到发育调控。细胞科学,1987,50:41-49。10.1016 / 0092 - 8674 (87) 90660 - x。gydF4y2Ba
- 56.gydF4y2Ba
de Paepe R, Chétrit P, Vitart V, Ambard-Bretteville F, Prat D, Vedel F:烟草原无性系中控制雄性不育和线粒体蛋白合成的几个核基因。中华分子生物学杂志,1999,21(4):366 - 366。10.1007 / BF00633819。gydF4y2Ba
- 57.gydF4y2Ba
Goldberg RB, Beals TP, Sanders PM:花药发展:基本原理和实际应用。植物科学学报,1998,19(5):369 - 369。10.1105 / tpc.5.10.1217。gydF4y2Ba
- 58.gydF4y2Ba
Hernould M, Suharsono, Zabaleta E, Carde JP, Litvak S, Araya A, Mouras A:烟草工程雄性不育株绒毡层和线粒体的损伤。植物生物学杂志,1998,26(3):369 - 371。10.1023 /: 1005946104983。gydF4y2Ba
- 59.gydF4y2Ba
细胞质雄性不育与生育恢复的分子基础。植物科学进展,1998,3:175-180。10.1016 / s1360 - 1385(98) 01235 - 7。gydF4y2Ba
- 60.gydF4y2Ba
Kelliher T, Walbot V:低氧引发玉米减数分裂命运获取。科学通报,2012,36(3):354 - 354。10.1126 / science.1220080。gydF4y2Ba
- 61.gydF4y2Ba
氧化还原稳态和抗氧化信号:应激感知和生理反应之间的代谢界面。植物学报,2005,17:1866-1875。10.1105 / tpc.105.033589。gydF4y2Ba
- 62.gydF4y2Ba
Fujino G, Noguchi T, Takeda K, Ichijo H:氧化还原信号通路中的硫氧还蛋白和蛋白激酶。中华癌症杂志,2006,16:427-435。10.1016 / j.semcancer.2006.09.003。gydF4y2Ba
- 63.gydF4y2Ba
黄志华,王志华,Feldman LJ, Lemaux PG, Buchanan BB:植物生长所需的膜相关硫氧还蛋白在细胞间的传递,提示其在细胞间通信中的作用。中国生物工程学报,2010,29(3):359 - 359。10.1073 / pnas.0913759107。gydF4y2Ba
- 64.gydF4y2Ba
Buard J, Barthès P, Grey C, de Massy B:不同组蛋白修饰定义小鼠减数分裂重组的启动和修复。中华医学杂志,2009,28:2616-2624。10.1038 / emboj.2009.207。gydF4y2Ba
- 65.gydF4y2Ba
山田T,水野K,广田K, Kon N, Wahls WP, Hartsuiker E, Murofushi H,柴田T, Ohta K:组蛋白乙酰化和染色质重塑因子在减数分裂重组热点中的作用。中华医学杂志,2004,23:1792-1803。10.1038 / sj.emboj.7600138。gydF4y2Ba
- 66.gydF4y2Ba
Seo H, Masuoka M, Murofushi H, Takeda S, Shibata T, Ohta K:通过增强同源重组快速生成特异性抗体。中国生物工程学报,2005,23:731-735。10.1038 / nbt1092。gydF4y2Ba
- 67.gydF4y2Ba
Merker JD、Dominska M、Greenwell PW、Rinella E、Bouck DC、Shibata Y、Strahl BD、Mieczkowski P、Petes TD:酿酒酵母中组蛋白甲基化酶Set2p和组蛋白去乙酰化酶Rpd3p在HIS4减数分裂重组热点抑制减数分裂重组。DNA修复(Amst)。2008, 7: 1298-1308。10.1016 / j.dnarep.2008.04.009。gydF4y2Ba
- 68.gydF4y2Ba
Sullivan JA, Shirasu K, Deng XW:泛素和26S蛋白酶体在植物生命中的不同作用。科学通报,2003,4:948-958。gydF4y2Ba
- 69.gydF4y2Ba
王晓燕,王晓燕,王晓燕。泛素-蛋白酶体途径与植物发育的关系。植物细胞学报,2004,16:3181-3195。10.1105 / tpc.104.161220。gydF4y2Ba
- 70.gydF4y2Ba
泛素和泛素结合蛋白的非传统功能。中国生物医学工程学报,2003,29(5):557 - 561。10.1074 / jbc.R300018200。gydF4y2Ba
- 71.gydF4y2Ba
Hicke L:单泛素调节蛋白质。细胞生物学杂志,2001,2:195-201。10.1038 / 35056583。gydF4y2Ba
- 72.gydF4y2Ba
Dukowic-Schulze S, Chen C:基于测序的大规模基因组学方法与少量分离的玉米减数细胞。前沿植物学报,2014,5:57-doi:10.3389/fpls.2014.00057gydF4y2Ba
- 73.gydF4y2Ba
Wu TD, Nacu S:短读中复杂变异和剪接的快速和snp耐受检测。生物信息学,2010,26:873-881。10.1093 /生物信息学/ btq057。gydF4y2Ba
- 74.gydF4y2Ba
Miller NA, Kingsmore SF, Farmer A, Langley RJ, Mudge J, Crow JA, Gonzalez AJ, Schilkey FD, Kim RJ, van Velkinburgh J, May GD, Black CF, Myers MK, Utsey JP, Frost NS, Sugarbaker DJ, Bueno R, Gullans SR, Baxter SM, Day SW, Retzel EF:高通量DNA测序项目的管理:Alpheus。中国生物医学工程学报。2008,1:132-10.4172/jcsb.1000013。gydF4y2Ba
- 75.gydF4y2Ba
Bullard JH, Purdom E, Hansen KD, Dudoit S:评估mRNA-Seq实验中归一化和差异表达的统计方法。中国生物医学工程学报,2010,26(5):591 - 591。gydF4y2Ba
- 76.gydF4y2Ba
序列计数数据的差分表达式分析。中国生物技术学报,2010,11:R106-10.1186/gb-2010-11-10-r106。gydF4y2Ba
- 77.gydF4y2Ba
Hochberg Y, Benjamini Y:多重显著性检验的更强大的程序。中华医学杂志1990,9:811-818。10.1002 / sim.4780090710。gydF4y2Ba
- 78.gydF4y2Ba
Milne I, Bayer M, Cardle L, Shaw P, Stephen G, Wright F, Marshall D:平板-下一代序列装配可视化。生物信息学,2010,26:401-402。10.1093 /生物信息学/ btp666。gydF4y2Ba
- 79.gydF4y2Ba
赵硕,Fernald RD:定量实时聚合酶链反应的综合算法。中国生物医学工程学报,2005,12:1047-1064。10.1089 / cmb.2005.12.1047。gydF4y2Ba
确认gydF4y2Ba
我们感谢A. Harris, J. Jensen, R. Meissner对植物的照顾。科恩(J. Cohen)和海格曼(A. Hegeman)的讨论和技术支持。本研究得到了美国国家科学基金会(IOS: 1025881)对W.P, S.K, J.P, J.M, E.R.和C.C.的支持gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
相应的作者gydF4y2Ba
额外的信息gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明他们没有竞争利益。gydF4y2Ba
作者的贡献gydF4y2Ba
SD进行研究,分析数据并撰写稿件,AS, JM, TR, AF分析数据并编辑稿件,MW, QS, JP协助数据分析和稿件编辑,SK, ER, WP, CC设计研究并编辑稿件。所有的作者都阅读并批准了最终的手稿,除了欧内斯特·雷策尔,他不幸去世了。gydF4y2Ba
电子补充材料gydF4y2Ba
12870 _2013_1532_moesm1_esm.xlsxgydF4y2Ba
附加文件1:表S1: RNA产量,序列读取的对齐分数,和所有RNA-seq之间的Pearson相关系数。(xlsx12kb)gydF4y2Ba
12870 _2013_1532_moesm2_esm.pdfgydF4y2Ba
附加文件2:图S1:差异表达基因。差异表达基因的减数细胞,花药和幼苗gydF4y2Ba玉米gydF4y2BaB73。(A)-(C)减数细胞与花药(A)、花药与幼苗(B)、减数细胞与幼苗(C)中DE基因的MA图。(D)花药或减数细胞与幼苗中组合的DE基因的热图。(pdf 264kb)gydF4y2Ba
12870 _2013_1532_moesm4_esm.pdfgydF4y2Ba
附加文件4:图S2:可转置元素。(A)全局表达式的平均比例(分配读,最多100个同样好的匹配)。(B)每个样本的每个特征的分配读取百分比。y轴刻度为对数。(C-F) TEs亚群中不同模式的分布分析:转座子顺序(C)、转座子超家族(D)、转座子的染色体位置(E)、先前显示与减数分裂或有丝分裂组织有联系的特殊家族(F)。差异表达的所有基因亚群见(C),定义为减数分裂特异性的亚群与(C-F)中非减数分裂特异性的亚群进行比较。(pdf 206kb)gydF4y2Ba
12870 _2013_1532_moesm6_esm.pdfgydF4y2Ba
附加文件6:图S3:定位和转录因子。MapMan分析。刻度显示对数gydF4y2Ba2gydF4y2Ba样本间的折叠变化,蓝色=高的减数细胞,红色=低的减数细胞。(A)减数细胞和幼苗之间分子靶向机制的基因。(B)减数细胞与幼苗之间转录因子的基因。(pdf 176kb)gydF4y2Ba
12870 _2013_1532_moesm7_esm.pdfgydF4y2Ba
附加文件7:图S4:在减数细胞和花药中富集的细胞成分。(A)减数细胞与幼苗细胞组分显著上调的曲线图(PgydF4y2Ba邻接的gydF4y2Ba≤0.01)。(B)花药与幼苗细胞成分显著上调图(PgydF4y2Ba邻接的gydF4y2Ba≤0.01)。(pdf 360kb)gydF4y2Ba
12870 _2013_1532_moesm9_esm.pdfgydF4y2Ba
附加文件9:图S5: TCA循环和电子传递链的细节。(A)减数细胞与幼苗TCA循环的差异。(B)在被定义为减数分裂基因的基因中,线粒体电子传递链的详细差异。刻度显示对数gydF4y2Ba2gydF4y2Ba样本间的折叠变化,蓝色=高的减数细胞,红色=低的减数细胞。用MapMan进行分析。(pdf 132kb)gydF4y2Ba
12870 _2013_1532_moesm11_esm.pdfgydF4y2Ba
附加文件11:图S6: RNA原位杂交所有阶段的原始图像,以及相应的RNA-seq计数。(A)用于原位杂交的基因及其RPM (reads per million)计数表。(B)原始原位杂交图像。所有的图片都使用相同的显微镜和相机设置,没有对裁剪的图片进行编辑。(pdf 2mb)gydF4y2Ba
作者提交的图片原始文件gydF4y2Ba
下面是作者提交的原始图片文件的链接。gydF4y2Ba
权利与权限gydF4y2Ba
开放获取gydF4y2Ba本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(gydF4y2Bahttps://creativecommons.org/licenses/by/2.0gydF4y2Ba),它允许在任何媒体上不受限制地使用、分发和复制,只要原始作品的名称正确。创作共用公共领域奉献放弃书(gydF4y2Bahttps://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)除另有说明外,适用于本条提供的资料。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
杜科维克-舒尔茨,S., Sundararajan, A., Mudge, J.。gydF4y2Baet al。gydF4y2Ba早期玉米减数分裂的转录组格局。gydF4y2Ba植物生物学gydF4y2Ba14,gydF4y2Ba118(2014)。https://doi.org/10.1186/1471-2229-14-118gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/1471-2229-14-118gydF4y2Ba
关键字gydF4y2Ba
- 玉米gydF4y2Ba
- 减数分裂gydF4y2Ba
- 性母细胞gydF4y2Ba
- 线粒体gydF4y2Ba
- RNA-seqgydF4y2Ba
- 转录组gydF4y2Ba