摘要
背景
可利用的役用作物植物基因组可以预测编码NB-LRR抗性基因同源物的全部基因,从而实现更有针对性的抗病育种。最近,我们开发了RenSeq方法来重新注释土豆中的完整NB-LRR基因补体,并识别自动基因预测软件未提取的新序列。在此,我们在番茄参考基因组上建立了RenSeq (茄属植物lycopersicum) Heinz 1706,在更新的茄科RenSeq诱饵文库中使用260个先前鉴定的NB-LRR基因。
结果
我们对Heinz 1706的基因组DNA进行RenSeq后的250 bp MiSeq reads,鉴定出105条新的NB-LRR序列。重注释包括基因模型的分裂、部分基因组合成较长的序列以及组装间隙的闭合。在草案中美国pimpinellifolium在LA1589基因组中,RenSeq使355个NB-LRR基因得以注释。然而,其中大多数是碎片化的,5 ' -端和3 ' -端位于独立的contigs边缘。系统发育分析表明,在Heinz 1706、LA1589和马铃薯无性系DM之间,所有NB-LRR类都具有高度保守性,这表明所有亚科都已经存在于最后的共同祖先中。系统发育的比较拟南芥NB-LRR补体验证了更古老CC的高度保守性RPW8类型NB-LRRs。利用RenSeq对未受感染和晚疫病感染的番茄叶片的cDNA进行分析,可以避免对非表达的副产物进行序列分析。
结论
RenSeq是一种很有前途的植物抗性基因补体分析方法。本文所提供的重新注释的番茄NB-LRR补体、系统发育关系和染色体位置将为育种家和科学家识别新的抗病性状提供有用的工具。cDNA RenSeq首次实现了针对这个非常低表达基因家族的下一代测序方法,而不需要标准化。
背景
为了控制病原体,植物会激活防御机制,最终导致受感染细胞和邻近细胞的超敏反应(HR) [1].防御激活需要通过两种已知的不同识别机制之一来检测病原体,病原体可以发生在植物细胞外或细胞内[2- - - - - -4].第一道检测线位于细胞表面,包括通过细胞表面跨膜受体识别病原体相关分子模式(PAMPs)。适应性病原体已经进化出克服pamp触发免疫(PTI)的机制,通过使用“效应分子”抑制免疫信号传递[4].反过来,植物拥有第二道防线,这道防线由检测特定效应分子或其对宿主细胞成分的影响的蛋白质所代表。这种机制被称为“效应者触发免疫”(ETI)。这些细胞内免疫受体,称为R(抗性)基因,编码类似哺乳动物nod样受体的蛋白质,通常携带核苷酸结合和富含亮氨酸的重复结构域(NB-LRR)。
植物NB- lrr蛋白(也称为NLR、NBS-LRR或NB- arc - lrr蛋白)通常被分为TIR类或非TIR类,这是基于NB结构域前的序列的一致性以及该结构域内的基序[5].植物NB-LRR蛋白(TNLs)的TIR类在氨基端含有一个Toll,白细胞介素1受体,R蛋白同源(TIR)蛋白-蛋白相互作用域。非tir类(CNLs)定义不太清楚,但该类的一些成员在其氨基端结构域中包含螺旋状线圈状(CC)序列[1].根据与含有EDVID氨基酸基序的典型CNLs的序列相似性,以及其n端与拟南芥RPW8蛋白的卷曲卷曲结构相似的RPW8-like蛋白的序列相似性,该类先前被分为亚类[6].
西红柿是世界上第二大重要的蔬菜作物(faostat.org),抗病育种是一个主要目标。几种NB-LRR型R从番茄、土豆和辣椒中克隆出的基因已被用于目前的育种工作。第一个番茄基因组组装草图揭示了NB-LRR基因家族的庞大规模,从而揭示了其潜力R基因库[7].第一个番茄R基因注释[7]是基于番茄基因组联盟现有的自动化基因和蛋白质预测[8].
最近,我们能够证明,利用抗性基因富集和测序(RenSeq)方法,对马铃薯参考序列的自动化基因和蛋白质预测未能揭示马铃薯中300多个潜在的NB-LRR基因[9].RenSeq方法利用基于Solanaceous NB-LRR序列设计的自定义生物素化120 mer RNA探针与已连接到Illumina适配器的感兴趣植物基因组DNA片段之间的退火。在洗去非结合部分后,捕获的包含约50% NB-LRR序列的文库可以在任何下一代测序平台上进行扩增和测序,这有助于在多基因家族中存在的许多NB-LRR基因上获得足够的序列深度[9].然而,即使使用RenSeq数据来绘制对特定位点的抗性,在一个多基因家族中定义每个旁位的序列仍然具有挑战性。
在本研究中,我们采用了RenSeq方法的改进版本[7,9,10]结合Illumina MiSeq 250 bp基因组DNA (gDNA)和两个已测序番茄基因组的cDNA对端测序美国pimpinellifoliumLA1589和美国lycopersicum1706年亨氏。gDNA上的RenSeq使我们能够纠正大约25%的先前描述的番茄NB-LRR基因,并从以前未注释的区域识别出105个新基因。我们进一步报道了第一个全面研究个体NB-LRR基因之间的系统发育关系美国pimpinellifoliumLA1589,美国lycopersicum亨氏1706和芸苔科拟南芥.RenSeq未来应用的一个重要结果是通过从cDNA中富集NB-LRR基因来降低序列数据的复杂性,从而避免了对非表达副序的序列分析。
结果与讨论
番茄和马铃薯RenSeq饵库的设计与应用
为了重新标注已测序番茄基因组的NB-LRR基因补体茄属植物lycopersicum亨氏1706和美国pimpinellifoliumLA1589(因此分别被称为Heinz 1706和LA1589),我们为NB-LRR基因靶向序列富集设计了定制RenSeq诱饵文库的更新版本[9].该版本的诱饵库包括28,787个独特的120 mer诱饵,这些诱饵是根据先前从番茄和土豆基因组中描述的260和438个nb - lrr类序列设计的(Jupe et al. (2013), [9])1) [7,10].RenSeq实验在基因组DNA上进行,以促进完整NB-LRR补体的重新注释,此外还在双链cDNA上进行,以测试仅对表达基因进行测序是否可以进一步降低该多基因家族测序数据的复杂性。在一次SureSelect NB-LRR捕获反应中,多达5个条形码样本被组合在一起,在测序前进一步合并到多达12个单个样本。
所得到的平均插入大小为700 bp的RenSeq库在MiSeq平台上测序(读取250 bp)。对于Heinz 1706,从gDNA中产生了9,395,874个reads。其中,50%(4,867,603)可以分别映射到12个(加ch00)参考番茄染色体上1).同样,对于LA1589,从MiSeq测试中获得了4,980,032个reads, 34%(1,680,734)映射到超级支架。对未映射的gDNA的分析揭示了一些来自线粒体和叶绿体DNA的序列污染,如早些时候报道的[9].
RenSeq数据可以在Heinz 1706和LA1589中实现NB-LRR基因的重注释
为了定位所有潜在的NB-LRR编码区域,将gDNA RenSeq reads映射到相应的参考基因组。从Heinz 1706和LA1589中分别提取了至少45个核苷酸的读覆盖大于20×的序列,共获得7290个和6465个基因组片段,两端延伸500 bp。重叠序列被连接并用于MAST搜索,以识别与NB-LRR基因相似的氨基酸基序组成[9,10].结果分别从Heinz 1706和LA1589中获得326和355个潜在的NB-LRR序列2,其他文件2,3.而且4).所有鉴定的序列被提交给植物抗性基因(http://prgdb.crg.eu/wiki/Main_Page),可从那里下载或用于BLAST搜索。
利用现有的MAST基序,可以将基因分为TNL或CNL,基序的存在/缺失可以对所识别的基因是部分基因还是全长基因做出结论。与以往的努力相比[7,11], RenSeq方法在Heinz 1706和LA1589基因组中分别建立了105和126个额外的nb - lrr。大约70%(221)的亨氏1706 NB-LRR基因是潜在的全长基因美国pimpinellifoliumLA1589仅占NB-LRR补体总数的37% (124)1而且2)编码全长基因所必需的最小结构域(NB-ARC和LRR)。这不太可能反映真实的结构,可能是由于LA1589基因组的碎片性,因为大约35%(124)的部分基因是在contigs边界发现的片段,其缺失的对应体预计位于其他contigs上。与n端结构域或NB-ARC开头相关联的基序的位置信息进一步用于预测推定的开始密码子,最后一个LRR特定基序和阅读帧信息用于为潜在的全长序列建立停止密码子(表2)2和附加文件2).
Heinz 1706中NB-LRR基因模型的修正
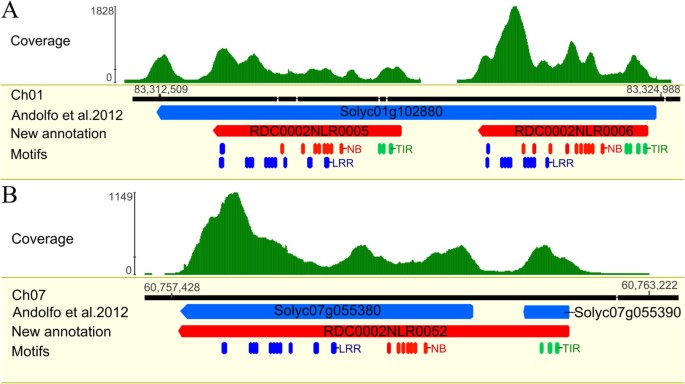
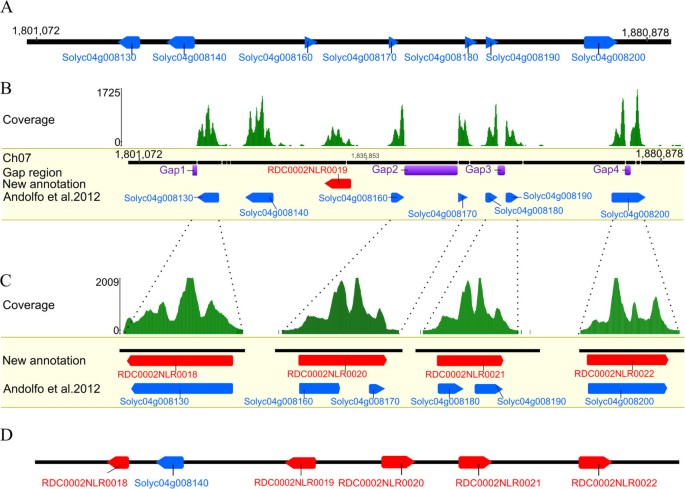
与之前的研究相比,我们的结果确定了72个错误注释的NB-LRR序列[7],其中使用了自动注释(表1).自动化基因预测软件并不能正确地注释所有的基因模型,基因组测序联盟的工作通常不包括对单个基因和基因家族的详细验证[7].为了完全重新注释NB-LRR补体,我们手动分析了所有已识别的位点,以纠正错误的开始和停止密码子,缺失或额外的外显子,以及错误的融合或分裂基因(附加文件)5).在图1一个和1我们展示了两个使用RenSeq数据进行校正的基因示例。尽管番茄基因组质量很高,但它仍然包含许多未知序列内容的区域,在注释的NB-LRR基因中,我们发现了8个长度不同的N延伸(在97到7851 bp之间)。这一数字明显小于马铃薯NB-LRR序列中发现的39个缺口[9].通过使用长250 bp的RenSeq读取和相应的配对结束信息,从两侧创建序列读取的拱形来填补这些空白。如图所示2,其中鉴定出4个序列间隙(Gap1-Gap 4,图2B为紫色)位于4号染色体上的基因簇中,最初由3个部分和4个全长NB-LRR基因组成[7].Solyc04g008130 (CC-NB-LRR)在预期的停止密码子位置有一个缺口,然后更正了这个缺口。在四个部分NB-LRR基因Solyc04g008160、Solyc04g008170、Solyc04g008180和Solyc04g008190之间发现了两个缺口,这些缺口的关闭使得部分基因重新注释为两个完整大小的CC-NB-LRR基因(RDC0002NLR0020和RCD0002NLR0021)。Solyc04g008200在序列中间有784 nt的预测缺口,校正为503个核苷酸。RenSeq数据进一步在这个集群中发现了一个新的NB-LRR (RDC0002NLR0019,图2B为红色),最终的基因模型如图所示2C.与Jupe等人相比。9]他们依赖于76 bp的配对读取数据,更长的读取允许非常快速地关闭高置信度的间隙,使用最少数量的重复映射轮。
两个错误融合/分裂的NB-LRR基因的重新注释。(一)RenSeq测序在Solyc01g102880中发现了两个不同的模式,表明两个基因融合(蓝框);(B)相比之下,Solyc07g055380和Solyc07g055390是预测的单个基因(红框),然而无间隙的RenSeq读覆盖模式表明它们都是一个更长的序列的一部分。修正后的注释在使用NB-LRR特定MEME motif的MAST分析中得到确认(TIR、NB和LRR motif分别显示在绿色、红色和蓝色框中[10]),并以方框箭头(绿色)表示新的全长TIR-NB-LRR基因RDC0002NLR0005、RDC0002NLR0006和RDC0002NLR0052。
4号染色体1.81 ~ 1.87 Mb位置的NB-LRR簇的详细分析(一)海因茨1706区域与来自Andolfo等人的注释[7].NB-LRR基因用蓝框表示。(B)Illumina miseq平台的RenSeq读覆盖显示为绿色峰值,并识别出一个尚未注释的NB-LRR RDC0002NLR0019(红框)。紫色框表示N的延伸为未知的基因组序列(紫色为Gap1到Gap4)。(C)被分析的位点的特写,其中的间隙被关闭了。图中显示了先前的基因模型(蓝框)、新模型(红框)和RenSeq读覆盖(绿色峰值)。(D)重注释NB-LRR基因簇的表达。
番茄和马铃薯NB-LRR分布的守恒性
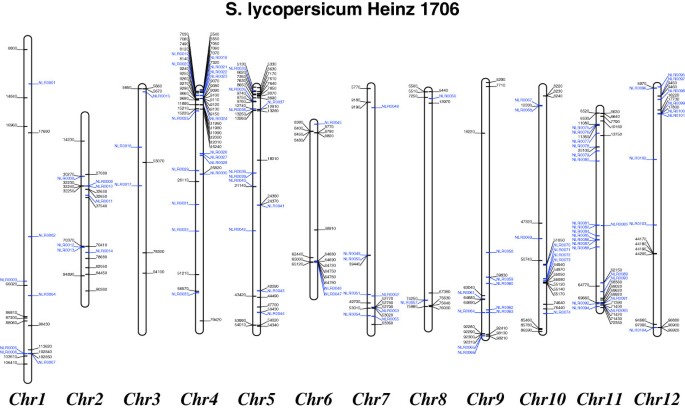
基于染色体大小,NB-LRR基因的全基因组分布明显是非随机的(χ2= 96, P <0.001)(图3.).在第4、5和11号染色体上发现的NB-LRR基因数量最多(约占已定位基因的45%),在第3号染色体上发现的NB-LRR基因数量最少(9个),这与马铃薯等茄科其他品种一致[9].TNL和CNL基因的基因组分布存在明显差异,TNL在1号染色体上最多(43%),而在3、6和10号染色体上不存在TNL。然而,cnl存在于所有染色体上。番茄中大多数(约66%)NB-LRR基因呈簇状组织(一个区域在200 kb以内包含4个或更多基因;[7]),包括串联阵列。我们发现20个基因簇共携带107个NB-LRR基因,平均5个,最多14个NB-LRR编码基因。最大的簇位于第4染色体的短臂(Solyc04g009070 ~ Solyc04g009290),分布在约110 kb宽的区域内。
Heinz 1706 NB-LRR基因的染色体分布。先前标注的NB-LRR基因[7]是黑色的,而在这项研究中发现的是蓝色的。染色体左边的基因在正链上,右边的基因在反链上。
有趣的是,与双单倍体参考马铃薯相比,番茄的NB-LRR基因数量不到一半。然而,这些存在于两个物种之间的同步染色体簇中。总的来说,这种差异不是由于基因亚家族的缺失,而是由于番茄中这些集群中的单基因数量明显较少。全基因组复制事件对马铃薯的扩增没有贡献[8].
番茄NB-LRR基因间的系统发育关系
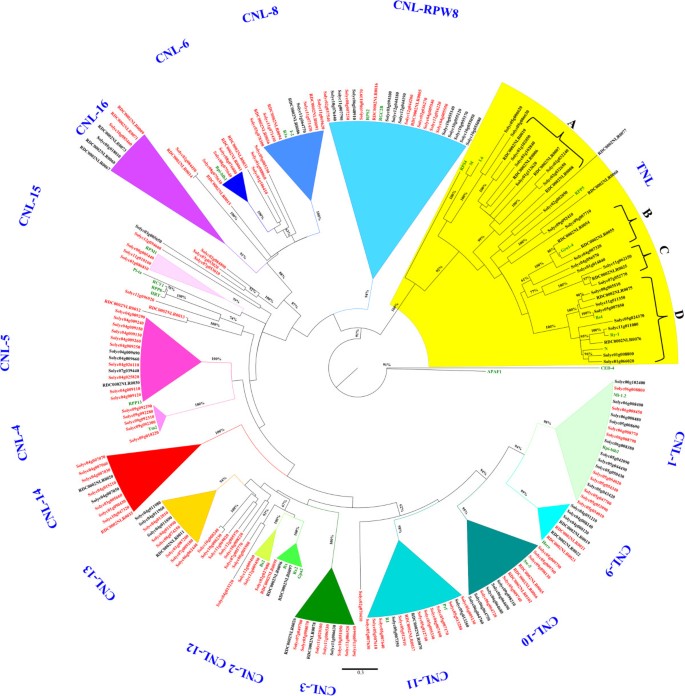
NB-LRR基因的NB-ARC结构域已被证明是分析系统发育关系最可靠的蛋白质结构域。因此,从每个具有完整NB-ARC结构域的NB-LRR基因中提取该结构域的氨基酸序列,分别用于对Heinz 1706和LA1589进行系统发育分析(图4和附加文件6).为了比较,我们纳入了30篇特征良好的克隆文献R来自11种不同植物物种的基因和两个具有核苷酸结合域的外群基因,即人类基因Apaf1.1和线虫Ced-4,分别(附加文件7,绿色的数字4而且5).Heinz 1706和LA1589的NB-ARC结构域分别排列240个和222个。这些序列被分为自举值≥75%支持的稳健支,并允许定义Heinz 1706中具有高度序列相似性的17和16支支(图4)和LA1589(附加文件6),分别。
系统发育树显示TNL, CNL之间有明显的区别RPW8和补偿中子测井EDVID(CNL-1至CNL-18)基因(图4和附加文件6),正如之前报道的马铃薯,我们也发现这种区别在拟南芥中非常明显(附加文件8) [5,6,10].有趣的是,尽管这种区别是非常保守的,并指向最后的共同祖先,包括茄科参考R基因之间没有任何相似之处答:芥NB-LRR,反之亦然(图4和附加文件8).此外,茄科CNL基因比TNL基因表现出更大的多样性和簇扩展,这与拟南芥和其他芸苔科形成对比。在TNL组中,确定了两个被分析物种之间共同的三个主要亚分支(A, B和D)。TNL-B和TNL-D的成员在功能特征上具有同源性R基因;线虫抗性基因Gro1.4(茄属植物tuberosum),Bs4,Ry1而且N,分别。在Heinz 1706中有四个成员的子分支TNL-C在LA1589中缺席。
不同于规范CNLEDVID基因是那些具有类似RPW8的cc结构域的基因,这些基因被认为具有保守的功能,并且可以在整个植物王国中找到[6].番茄、马铃薯和拟南芥在系统发育树中的古老位置,以及其他报告表明,这一类群在单子叶/双子叶分裂之前就存在了[6].这个分支的典型成员是n必需基因1(NRG1)n benthamiana和拟南芥激活抗病1(ADR1)基因。
CNL内部EDVID在Heinz 1706中定义了15个分支,在LA1589中定义了14个分支(图4和附加文件6;两个分析种与马铃薯的分支id相对应[10])。支CNL-1由Mi1.2,Rpi-blb25号和6号染色体上也有相似的序列。值得注意的是,演化支CNL-1与演化支CNL-9和CNL-10(由93%的引导索引支持)具有共同的祖先,这两个演化支由CNL-9和CNL-10组成英雄家族编码在4号染色体上Sw-5分别是9号染色体上的家族。在LA1589系统发育树中,前三个相似的分支(CNL-1, CNL-17和CNL-10)定义不太清楚英雄只有两个相似的序列(RDC0003NLR0189和RDC0003NLR0120)不被认为是进化支。像这样的差异可能是由于LA1589基因组组装的质量较差,以及由此注释的基因的碎片性。CNL-11在两个系统发育树中都与R1而且脉冲重复频率,所有序列均位于第5染色体上。在Heinz 1706和LA1589中存在的两个小分支是CNL-2和CNL-12,它们与特征基因具有相似性处方,Rx2而且Gpa2,Bs2,分别。5个独立的大分支(CNL-3, CNL-13, CNL-14, CNL-16和CNL-18)在任何功能上都没有相似性R基因,因此可能是新的耐药性的潜在来源。支CNL-4包含参考蛋白Tm-2在这两个物种的9号染色体上编码的序列高度相似。14和10个基因相似A. thaliana RPP13聚集在Heinz 1706和LA1589,分别存在于演化支CNL-5中。番茄特有的是CNL-15,它包括类似于RPM1.CNL-16分别含有7个和8个来自Heinz 1706和LA1589的基因。小分支CNL-6包括同源Rpi-blb1在两个系统发生树中具有高度同源性。9和13个非常相似的番茄的同源物I2和土豆R3a基因分别在Heinz 1706和LA1589的CNL-8分支中发现.进化支CNL- rpw8位于TNL和CNL基因之间的祖先位置,并含有特征基因RPS2而且RGC2B[12,13].
cDNA RenSeq显著降低了NB-LRR基因补体的复杂性
RenSeq被建立为一种工具,用于对NB-LRR基因补体进行靶向测序,以确定隔离人群中耐药和易感个体之间与疾病抗性相关的多态性[9].然而,对于一些NB-LRR亚家族,在染色体簇和系统发育支中定义许多副同源的NB-LRR基因,并确定共分离SNP源自的个体副同源仍然具有挑战性。NB-LRR基因不高表达,可能是为了防止自身免疫,因此RNA-seq方法不太可能恢复足够的序列深度。我们测试了使用RenSeq富集NB-LRR序列500-1000×的能力是否能够提供足够的阅读深度来测序这些低表达基因的cDNA。对未处理和晚疫病混合RNA样本的双链cDNA进行了RenSeq实验(5种)感染了海因茨1706和LA1589的叶子
从富集了Heinz 1706 cDNA的NB-LRR中共获得2,882,986对250 bp的MiSeq序列;其中65% (1,863,598 reads)映射到12条参考染色体。未映射到染色体的Reads被鉴定为源于核糖体RNA。高严格的Bowtie作图,省略了可以映射到多个序列的reads(参见方法),分别将214,050和235,656 reads放置在167个Heinz 1706和154个LA1589 NB-LRR基因上。平均每个NB-LRR序列映射1281和1560个读。一些序列具有非常少的映射读取数量(最少2个;附加文件2而且3.),并可能映射人工制品,但仍被考虑。总体而言,在Heinz 1706中NB-LRR补体的复杂性降低了51%(图4)和43%的LA1589(附加文件6),因此,任何候选人的对话录的数量R需要分析的基因减半。更重要的是,这种减少甚至出现在所有的进化支中。然而,这些数据并不能得出任何关于读取数和表达水平之间相关性的结论,因为不能排除来自诱饵库的某些偏差(尽管在gDNA上进行RenSeq后没有发现)。在表达的基因中,90%为全长基因,10%为部分基因。表达的部分基因数量高于其他植物物种,这可能表明其在NB-LRR基因调控中发挥作用[14].
整合遗传学和基因组学,定位最佳NB-LRR抗性基因候选
植物抗病育种是基于抗性等位基因的遗传定位。本文提出的结果通过使用可用的标记数据以及注释NB-LRR基因的位置和序列信息,为基因组学和遗传学的集成构建了一个框架。下面的案例将展示一个最近映射但尚未克隆的实例R基因,和另一个在高度进化压力下的位点R番茄中的基因已经被鉴定出来。
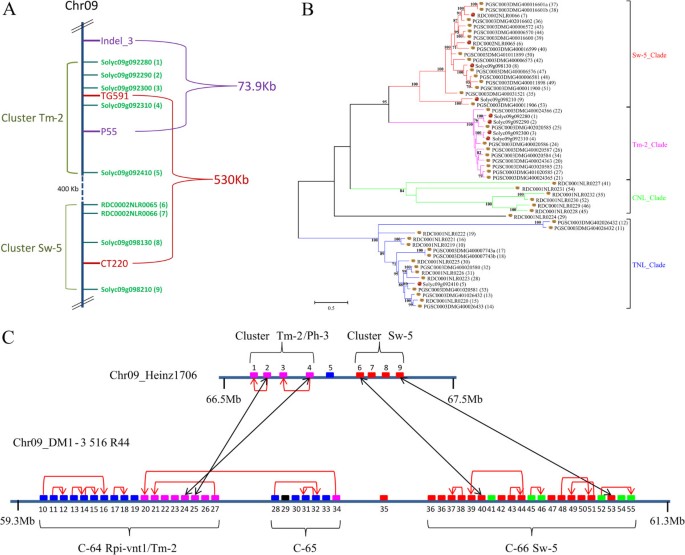
最近的两份出版物分别提出了一套四个侧翼标记R基因Ph-3这给某些人带来了阻力p . 5隔离在美国lycopersicum[15,16].将这些标记序列(Indel_3, CT220, TG591和P55)定位到参考染色体上,在9号染色体的短臂上识别出一个600kb的区域5A).该基因组区域包含与番茄高度相似的序列R基因Tm-2而且Sw-5分别对番茄花叶病毒(ToMV)和番茄斑点枯萎病毒(TSWV)具有抗性。的Tm-2Heinz 1706的聚类由4个CC-NB-LRR基因组成,具有90%以上的成对同源性。的Sw-5该簇由3个完整的CC-NB- lrr基因和1个部分CC-NB基因组成。有趣的是,这两个独立鉴定的标记对的共同区域只有30kb,其中只有一个NB-LRR基因位于TG591和P55之间。CNL Solyc09g092310是Heinz 1706中最接近的同源物,因此是潜在的候选Ph3在抗性番茄系中[15- - - - - -17].该CNL与Rpi-vnt1.1和Tm-2的氨基酸同源性分别为77.4%和73%。数字5C表示的是同步守恒R周围的基因簇Ph-3番茄与马铃薯的候选基因[9].在这一共线区域发现的马铃薯和番茄序列的系统发育分析并没有导致来自两个物种的序列的明确区分,这表明这些簇已经存在于最后的共同祖先中(图5B). 4个高度相似的基因对,同源性在82 - 89%之间(图5C;黑色箭头)被鉴定为可能是最古老的。
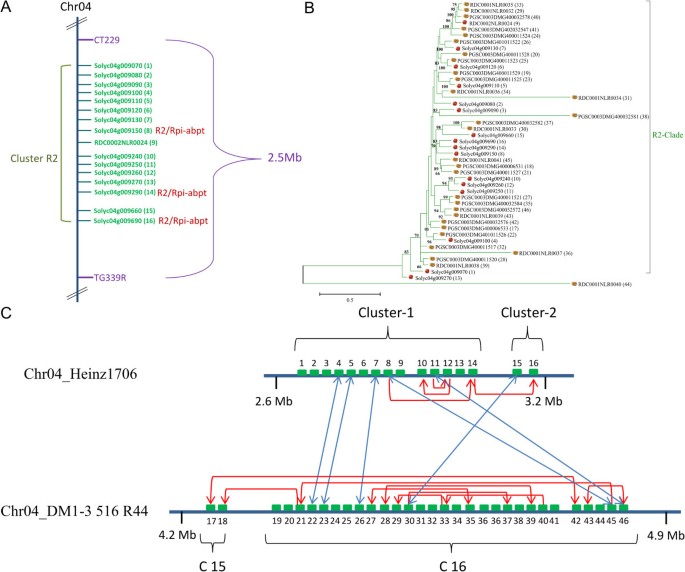
Heinz 1706的4号染色体拥有最大的NB-LRR基因簇,共有14个成员(均位于CNL-11)(图6A).该聚类的所有成员彼此之间具有高度的序列相似性,并衍生出野生马铃薯R基因R2,Rpi-blb3而且Rpi-abpt它们位于马铃薯4号染色体的同线区[18,19].Synteny也通过标记CT229和TG339R显示,两者都与之相关Rpi-blb3[17].对来自番茄和土豆的这些簇的成员编码的蛋白质的详细系统发育分析表明,所有基因都属于一个独特的分支,其平均身份为80%,自举值为83%(图2)6B). Solyc04g009290具有较高的序列同一性R2(88%;数字6A).系统发育树进一步确定了马铃薯中的9个重复事件,这些事件必须发生在马铃薯和番茄的分化之后(图6C).微共线分析在两个物种中鉴定出6个NB-LRR基因,序列相似性在78%至85%之间(蓝色箭头;数字6C).没有功能R从这个快速进化的集群中,基因尚未在番茄中被鉴定出来,但可以推测,该位点的一些等位基因可能编码有价值的抗病性。
结论
RenSeq有助于植物NB-LRR基因完整补体的深度测序和鉴定。Illumina MiSeq平台具有250 bp读数,便于无错误地关闭装配中的间隙。我们预计,在其他组装的植物基因组上进行RenSeq将增加标注NB-LRR序列的数量,并将实现更有针对性和特异性的抗性育种策略。虽然在散装抗性和散装易感植物上的RenSeq允许使用“快速”定位或基因型特异性定位识别与抗性表型共隔离的NB-LRR基因等位基因,但候选基因的列表可以通过cDNA RenSeq进一步减少,限制了数量R候选基因只分析那些已表达的基因。这些方法的结合将大大加快作物改良中自然抗性基因多样性的招募。
方法
植物材料和RenSeq库的制备
完全展开的叶子美国lycopersicum亨氏1706和美国pimpinellifoliumLA1589从3周龄的温室植株中分离。每叶接种2滴20 μl的水,或接种悬液接种3片叶片p . 5分离2006_3928A(50,000个游动孢子/ml)。接种24小时后,每叶取1个接种点,形成直径为10mm的叶盘,冷冻于液氮中。在6 dpi时观察剩余斑点,以成功定殖5页。按照制造商的建议,将两种处理的叶片混合,使用tri试剂(Sigma-Aldrich)和Directzol RNA Mini-prep (Zymo Research)提取RNA。使用oligo-dT和随机六聚体引物和First-strand上标II (Sigma-Aldrich)制备第一链cDNA。第二股是按照[20.].
gDNA是从相同植物的幼叶组织中提取的,使用DNeasy Plant Mini试剂盒(Qiagen),遵循制造商的建议。
Illumina MiSeq文库使用NEBNext Ultra DNA文库准备试剂盒(NEB),使用2 ~ 3 μg起始材料制备。使用NEBNext Multiplex Oligos for Illumina (Index Primers Set I)对库进行多路复用。如jpe等人所述,多达三个库被池化,并捕获NB-LRR类序列。[9]使用安捷伦SureSelect套件,更新的诱饵库包括28,787个独特的120 mer oligos(附加文件1).富集文库扩增至1 μg,送到基因组分析中心(TGAC, Norwich Research Park, UK)进行MiSeq 250 bp配对端测序。
中NB-LRR基因的鉴定与注释茄属植物spp
所有Illumina MiSeq数据分析都是使用Sainsbury实验室的Galaxy项目实例进行的,如果没有其他说明的话[21].鉴定和注释番茄基因组中的NB-LRR位点[8],使用BWA version 0.5.9(默认参数)(tgc_sl2 .40 _pseudomoleculs .fasta)将富集NB-LRR的配对端Illumina MiSeq reads映射到12条染色体上。将作图信息(bam格式)导入geneous 6.0,并将每条染色体可视化(http://www.geneious.com/).Illumina对先前确定的nb - lrr的读取覆盖率确定如Jupe等人所述。[9].如Jupe等人所述,使用MAST输出确定潜在的全长序列。[10],并进一步用于识别每个基因的开始和停止位置。按照Jupe等人描述的方法关闭组件中的间隙。[9].新基因的id按照Jupe等人的定义。[9]为R基因发现联盟(RDC),包括物种代码RDC0002 (Heinz 1706)和RDC0003 (LA1589)。
cDNA RenSeq文库分析
在严格的条件下,使用Bowtie 0.12.7版本将原始的高质量MiSeq reads映射到重新注释的NB-LRR基因补体中(省略多次映射的reads)。对所得到的sam文件进行筛选以获得映射的reads,并计算每个NB-LRR基因的数量。没有对映射读取的数量应用截止。
系统发育和基因重复分析
为鉴定NB结构域序列用于系统发育分析,对NB结构域的氨基酸序列进行了参考R基因(在附加文件中报道7),在BLASTx分析中进行搜索,期望值<1e−3.全长NB-ARC结构域小于50%的序列(Pfam数据库ID: PF00931)被排除。进化分析使用MEGA5进行[22].分别推断映射的NB-LRR基因的系统发育关系(如:美国lycopersicum亨氏1706和美国pimpinellifoliumLA1589组)采用基于WAG模型的最大似然方法[23].162拟南芥从TAIR数据库中提取NB-LRR基因序列(http://www.arabidopsis.org/).对于核苷酸序列,使用一般时间可逆模型。采用从100个重复中推断出的自举一致性树来表示所分析序列的进化历史[24].所有序列使用ClustalW 1.74进行比对[25].
支持数据的可用性
支持本文结果的数据集可以作为附加文件找到,序列读取可在欧洲核苷酸档案,http://www.ebi.ac.uk/ena/data/view/ERP002644.所有NB-LRR序列都可以在附加文件中找到4并被上传到植物抗性基因(http://prgdb.crg.eu/wiki/Main_Page),并可透过搜寻RDC0002NLR或RDC0003NLR编号进入。
参考文献
- 1.
Heath MC:与过敏反应相关的死亡。植物化学学报,2000,44:321-334。10.1023 /: 1026592509060。
- 2.
丹格尔,琼斯:植物病原体与综合防御反应。自然科学,2001,411:826-833。10.1038 / 35081161。
- 3.
王志强,王志强,王志强,等。植物免疫应答机制的研究进展。电子学报,2006,34(4):344 - 344。10.1016 / j.cell.2006.02.008。
- 4.
Jones JDG, Dangl JL:植物免疫系统。自然科学,2006,44(4):323-329。10.1038 / nature05286。
- 5.
王晓燕,王晓燕,王晓燕,等。抗病基因的进化。中国植物学报,2005,28(4):529 - 529。10.1016 / j.pbi.2005.01.002。
- 6.
Collier SM, Hamel LP, Moffett P:一种独特且高度保守的NB-LRR蛋白n端结构域介导的细胞死亡。植物与微生物相互作用,2011,24:918-931。10.1094 / mpmi - 03 - 11 - 0050。
- 7.
Andolfo G, Sanseverino W, Rombauts S, Van der Peer J, braden JM, Carputo D, Frusciante L, Ercolano MR:番茄(Solanum lycopersicum)候选病原体识别基因概述揭示了重要的Solanum R位点动态。中国生物工程学报,2013,27(3):344 - 344。10.1111 / j.1469-8137.2012.04380.x。
- 8.
番茄基因组联盟:番茄基因组序列为肉质水果的进化提供了见解。自然科学学报,2012,34(4):344 - 344。10.1038 / nature11119。
- 9.
Jupe F, Witek K, Verweij W, Sliwka J, Leighton P, Etherington GJ, Maclean D, Cock PJ, Leggett RM, Bryan GJ, Milne L, Hein I, Jones JDG:抗性基因富集测序(RenSeq)可以从已测序的植物基因组中重新注释NB-LRR基因家族,并在分离群体中快速绘制抗性位点。植物学报,2013,37(3):344 - 344。10.1111 / tpj.12307。
- 10.
Jupe F, Pritchard L, Etherington GJ, Mackenzie K, Cock PJ, Wright F, Kumar Sharma S, Bolser D, Bryan GJ, Jones JD, Hein I:马铃薯基因组中NB-LRR基因家族的鉴定和定位。中国生物医学工程学报,2012,13:75-10.1186/1471-2164-13-75。
- 11.
肖勋N,京晶Y,思龙S,文才Y:番茄基因组中抗性样基因的鉴定与分析。中国植物学报,2013,29(3):344 - 344。
- 12.
Bent AF, Kunkel BN, Dahlbeck D, Brown KL, Schmidt RL, Giraudat J, Leung JL, Staskawicz BJ:拟南芥RPS2:一个富含亮氨酸的重复类植物抗病基因。科学通报,1994,29(4):344 - 344。10.1126 / science.8091210。
- 13.
沈佳,Chin DB, Arroyo-Garcia R, Ochoa OE, Lavelle DO, Wroblewski T, Meyers BC, Michelmore RW: Dm3是核苷酸结合位点富含亮氨酸重复编码基因组成性表达家族的成员之一。植物与微生物的相互作用,2002,15:251-261。10.1094 / MPMI.2002.15.3.251。
- 14.
Marone D, Russo MA, Laido G, De Leonardis AM, Mastrangelo AM:植物核苷酸结合位点富含亮氨酸重复序列(NBS-LRR)基因:宿主防御反应中的积极守护者。中华分子生物学杂志,2013,14:7302-7326。10.3390 / ijms14047302。
- 15.
杨晓东,杨晓东,杨晓明,陈晓明,等。番茄抗病候选基因的筛选与分析。摩尔繁殖。2013, 9928: 7-
- 16.
张超,刘玲,郑志,孙艳,周玲,杨艳,程峰,张震,王霞,黄松,谢波,杜艳,白艳,李娟:番茄晚疫病抗性Ph - 3基因的精细定位。应用理论与实践,2013,26(1):1 - 4。10.1007 / s00122 - 013 - 2162 - 1。
- 17.
杨春旺,刘春旺,王晓明,王春旺,王晓明,等:L. pimpinellifolia L3708晚疫病病原Ph-3基因的定位。《番茄》,1998,48:13-14。
- 18.
李X, Van Eck HJ, Van der Voort JNAM R, Huigen DJ, Stam P, Jacobsen E:马铃薯抗疫霉基因在4号染色体上的定位。应用理论,1998,26(1):1- 4。10.1007 / s001220050847。
- 19.
Park T、Gros J、Sikkema、Vleeshouwers VGAA、Muskens M、Allefs S、Jacobsen E、Visser RGF、van der Vossen EAG:球栗茄(Solanum bulbocastanum)的晚疫病抗性位点Rpi-blb3属于马铃薯4号染色体上一个主要的晚疫病R基因簇。植物与微生物相互作用,2005,18:722-729。10.1094 / mpmi - 18 - 0722。
- 20.
Okayama H, Berg P: cDNA的高效克隆。中国生物医学工程学报,2002,23(2):366 - 366。
- 21.
Maclean D, Kamoun S:小地方的大数据。生物技术,2012,30:33-34。10.1038 / nbt.2079。
- 22.
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S: MEGA5:利用最大似然,进化距离和最大简约方法进行分子进化遗传学分析。分子生物学杂志,2011,28:2731-2739。10.1093 / molbev / msr121。
- 23.
惠兰S,戈德曼N:一个一般的经验模型的蛋白质进化源于多个蛋白质家族使用最大似然方法。中华生物医学杂志,2001,18:691-699。10.1093 / oxfordjournals.molbev.a003851。
- 24.
Felsenstein J:系统发生的置信限制:一种使用自举法的方法。《科学进展》,1985,39:783-791。10.2307 / 2408678。
- 25.
Thompson JD, Higgins DG, Gibson TJ: Clustal w:通过序列加权、位置特定间隙惩罚和权重矩阵选择提高渐进式多序列对齐的灵敏度。中国生物医学工程学报,1994,22(4):344 - 344。10.1093 / nar / 22.22.4673。
致谢
我们要感谢莎拉·珀金斯在温室里维护番茄生产线。我们非常感谢Mark McMullan博士和Agathe Jouet博士在系统发育和基因进化分析方面的宝贵讨论。本作品由BBSRC基金BB/H019820/1和盖茨比基金会资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
AG, FJ和KW设计了这项研究。AG进行了注释和系统发育分析。KW和FJ计划并实施了RenSeq实验。FJ分析了cDNA RenSeq数据。GJE对生物信息学分析做出了贡献。AG和FJ撰写了手稿。KW, ME和JJ严格审查了手稿。所有作者都阅读并批准了最终的手稿。
电子辅助材料
定制的
附加文件1:R基因富集和测序(RenSeq)诱饵文库。在马铃薯和番茄的NB-LRR基因上设计了28,787个120 mer探针。(txt 4mb)
详细资料
附加文件2:茄属植物lycopersicum亨氏1706 NB-LRR。该表包含326个注释的Heinz 1706 NB-LRR基因的信息,包括链信息,基因是否表达,染色体上的起始和结束位置以及蛋白质类。(xlsx60kb)
详细资料
附加文件3:茄属植物pimpinellifoliumLA1589 NB-LRR。该表包含355个注释的LA1589 NB-LRR基因的信息,包括链信息、基因是否表达、在contig/支架上的起始和结束位置以及蛋白质类别。(xlsx71 kb)
亨氏1706和
附加文件4:茄属植物lycopersicum亨氏1706和美国pimpinellifoliumFASTA格式标注NB-LRR基因。所有已确定的番茄NB-LRR基因家族成员都包括在这个文件中。(zip 456 kb)
12870 _2013_1536_moesm5_esm.xlsx
附加文件5:重新注释的亨氏1706 NB-LRR基因列表。此列表详细介绍了原亨氏1706 solyc - id,包括RenSeq分析后所做的更改和新的基因id。(xlsx43 kb)
的系统发育树
附加文件6:茄属植物pimpinellifoliumLA1589 NB-ARC结构域。进化分析在Heinz 1706中进行,基于222个已命名的nb - lrr的NB-ARC结构域。红色标签显示表达的NB-LRR基因的基因id;黑色代表未表达基因。(png 908 kb)
12870 _2013_1536_moesm7_esm.xlsx
附加文件7:系统发育研究的参考NB-LRR基因。30个先前克隆和鉴定的植物NB-LRR基因和两个外群基因的列表,这些基因被用于系统发育研究作为参考基因。(xlsx45kb)
的系统发育树
附加文件8:拟南芥NB-LRR基因。进化分析进行了茄属植物。194个NB-ARC结构域。(png 6mb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是原创作品的名称要注明出处。创作共用公共领域奉献弃权书(https://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
安道尔福,G.,朱佩,F.,维泰克,K.。et al。利用基因组和cDNA RenSeq确定番茄NB-LRR抗性基因的完整库。BMC植物生物学14日,120(2014)。https://doi.org/10.1186/1471-2229-14-120
收到了:
接受:
发表:
关键字
- RenSeq
- NB-LRR
- 互补脱氧核糖核酸
- 基因模型
- 抗病性
- Paralogous
- 植物育种
- 茄属植物lycopersicum
- 茄属植物pimpinellifolium
- 拟南芥