摘要
背景
SPLs是植物特有的转录因子家族,通过调节多种生理生化过程在植物生长发育中发挥重要作用。虽然杨树trichocarpa是一棵模范森林的树吗PtSPL基因家族尚未得到系统的研究。
结果
在这里我们报告了28个全长的鉴定PtSPLs,分布在14p . trichocarpa染色体。基于植物中SPLs的系统发育关系p . trichocarpa而且拟南芥,植物SPLs可分为6类。每个组至少包含一个PtSPL和一个AtSPL。第6组SPLs SBP结构域的n端锌指1 (Zn1)含有4个半胱氨酸残基(cccc型),其他组SPLs的Zn1主要含有3个半胱氨酸残基和1个组氨酸残基(c2hc型)。对PtSPLs和AtSPLs的基因结构、保守基序和表达模式的比较分析表明,植物SPLs在一个群体内是保守的,而在群体间则是保守的p . trichocarpa而且拟南芥SPLs有显著差异。在PtSPLs中发现了多种保守基序,而在AtSPLs中没有发现,这表明植物SPLs具有多样性。共11对染色体内重复PtSPLs被确认,这表明基因复制在SPL基因扩增p . trichocarpa.此外,28个PtSPLs中有18个属于G1、G2和G5,被发现是miR156的靶点。一致地,这些组中的所有AtSPLs都受到miR156的调控。这表明mir156介导的植物转录后调控具有保护性。
结论
从其全基因组序列中鉴定出28个全长SPLsp . trichocarpa.通过对基因结构、系统发育关系、染色体位置、保守基序、表达模式和mir156介导的转录后调控的综合分析,研究人员发现PtSPL对基因家族进行了鉴定。我们的研究结果为植物SPLs的进化和生物学功能提供了有用的信息。
背景
SPL蛋白是一个多种转录因子家族,在植物生长发育中起着至关重要的作用。SPLs是植物特有的,具有高度保守的SBP (SQUAMOSA PROMOTER BINDING PROTEIN, SQUAMOSA PROMOTER BINDING PROTEIN)结构域,约有78个氨基酸残基。该结构域包含三个功能上重要的基序,包括锌指1 (Zn1)、锌指2 (Zn2)和核定位信号(NLS) [1,2].在SBP1和SBP2中首次鉴定出编码SPLs的基因金鱼草majus[3.].最近,它在各种绿色植物中被发现,包括单细胞绿藻、苔藓、裸子植物和被子植物。结果显示:作为一个大的基因家族存在于植物中。例如,SPL基因家族拟南芥、大米、Physcomitrella金属盘,玉米和番茄分别含有16、19、13、31和15个成员[4- - - - - -9].
16拟南芥:被称为AtSPL1来AtSPL16[2],其中AtSPL1,AtSPL7,AtSPL12,AtSPL14而且AtSPL16是相对较大的构成型表达,而其他的相对较小,在花中高度表达[4,10].16个中的10个AtSPLs,包括AtSPL2- - - - - -AtSPL6,AtSPL9- - - - - -AtSPL11,AtSPL13而且AtSPL15的miRNAs调控MIR156家庭(11- - - - - -17].AtSPL3,AtSPL4而且AtSPL53 ' UTR中含有miR156互补序列,均促进营养相变和开花[10,14,18].AtSPL2,AtSPL10而且AtSPL11调节茎生叶和花的形态性状[19].miR156b的过表达降低了细胞的积累AtSPL2,AtSPL10而且AtSPL11信使rna (12,14,20.].AtSPL9而且AtSPL15在控制青苗到成虫的生长阶段过渡和起叶率方面起了冗余作用拟南芥[21].六个AtSPLs,包括AtSPL1,AtSPL7,AtSPL8,AtSPL12,AtSPL14而且AtSPL16,都不是miR156的靶标拟南芥.其中,AtSPL7可以直接结合到含有GTAC核心序列的Cu响应元件(CuRE)上,是铜稳态稳态的调节因子拟南芥[22].AtSPL8调节花粉囊发育[23]、男性生育能力[24], GA生物合成及信号传导[25].AtSPL14在植物发育和对伏马菌素B1的敏感性中起重要作用[26].19种大米中:,一半主要在各种年轻器官中表达[27].miR156靶向的OsSPLs参与水稻花的发育。OsSPL14由miR156调控,也控制营养期芽的分枝[8,28,29].在玉米、liguleless1包含SBP结构域调节舌叶和耳廓的形成[30.,31].
杨树trichocarpa是一种全基因组序列可用的模式植物[32].总共鉴定出352种miRNA前体,包括12种miR156前体[33- - - - - -39].然而,miR156的调控在trichocarpa PtSPLs没有分析过。在我们之前的研究中[40],从中鉴定出17个PtSPLs,这些PtSPLs似乎是全长或部分序列,至少含有300个氨基酸杨树基因组组装v1.1 (http://genome.jgi-psf.org/Poptr1_1/Poptr1_1.home.html).它们分别被命名为PtSPL1-PtSPL17,其中PtSPL3和PtSPL4与参与Cu稳态的AtSPL7相似度最高[40].为了描述整体SPL基因家族p . trichocarpa,我们搜索了杨树基因组组装v1.1、v2.2和v3.0 [32].结果鉴定出28个全长PtSPLs.基因结构、染色体位置、系统发育关系、保守蛋白基序和表达模式全部鉴定PtSPLs系统分析。mir156介导的转录后调控PtSPL研究基因。这些结果为阐明植物的生物学功能提供了有用的信息:在p . trichocarpa.
结果
28的鉴定SPL基因在p . trichocarpa基因组
分析杨树v1.1、v2.2和v3.0基因组组装显示存在28个全长SPL基因p . trichocarpa基因组(表1).推导出的PtSPL蛋白均含有保守的SBP结构域。理论p我推断出的PtSPL蛋白在5.87 ~ 9.49之间。长度在148到1044个氨基酸之间变化。分子量(Mw)从16.2到116.1 kDa(附加文件)不等1).p的分布我类似于AtSPLs(附加文件2);但ptsps的长度和Mw均大于atsps。
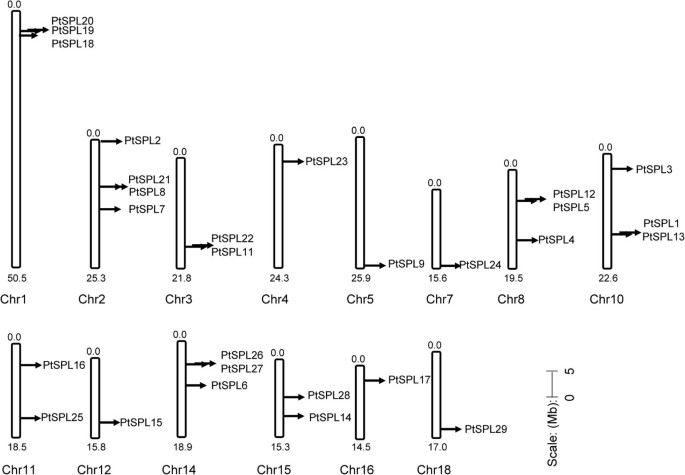
映射PtSPLs到p . trhichocarpa基因组显示PtSPLs染色体不均匀分布在14条染色体上,其中Chr2染色体4条,Chr1、Chr8、Chr10和Chr14染色体各3条,Chr3、Chr11和Chr15染色体各2条,Chr4、Chr5、Chr7、Chr12、Chr16和Chr18染色体各1条(图1).相对高密度的PtSPLs主要分布在Chr8、Chr10、Chr11和Chr14的顶部和底部,Chr1、Chr4和Chr16的顶部,Chr3、Chr5、Chr7、Chr12和Chr18的底部。很少在染色体的中心区域。此外,11对PtSPLs(Ks < 1.0)是由染色体内复制进化而来的2),说明基因复制对PtSPL基因的扩张。
植物sps的系统发育分析p . trichocarpa而且拟南芥
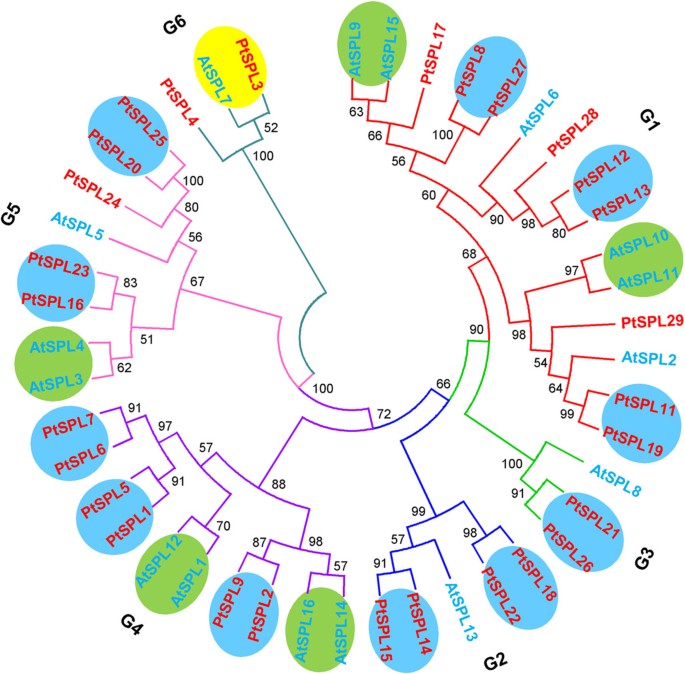
为了研究它们之间的进化关系p . trichocarpa而且答:芥利用MEGA5.1构建了28个PtSPLs和16个AtSPLs的邻居连接(NJ)系统发育树。采用1000次自举重复的自举重采样方法评估分支的可靠性。仅使用引导值>50%支持的节点进行进一步分析。结果显示,44个SPL蛋白聚成6组(命名为G1-G6),每组至少含有1个AtSPL和1个PtSPL(图2).结果与SmSPLs在丹参[41].为了进一步确认有6组SPLs,我们还构建了28个PtSPLs、16个AtSPLs、18个水稻OsSPLs和15个SmSPLs的系统发育树。如附加文件所示3., 77个SPLs也聚成6组。所构建的两个树之间的差异(图2,附加文件3.)为图中属于G1的PtSPL12、PtSPL13、PtSPL28和AtSPL62是否包含在附加文件中的G2中3..在所有的SBP结构域编码区都发现了一个内含子SPL基因p . trichocarpa而且拟南芥(图3.);序列特征分析显示,G6中SPLs的SBP结构域(AtSPL7、PtSPL3和PtSPL4)与其他组存在差异。G6 SPLs的n端锌指在SBP结构域有4个半胱氨酸残基,而其他组SPLs主要含有3个半胱氨酸和1个组氨酸,表明植物SPL进化的多样性。另一方面,:一组内具有相似的内含子数量、外显子-内含子结构和编码序列长度。长度、Mw和理论p我在一个组内,所推导出的SPL蛋白也相似,尽管它们在组间是不同的。这表明了植物群落中SPLs的守恒性。系统发育分析表明,PtSPL3和PtSPL4与AtSPL7具有较高的同源性拟南芥SPL具有在MIR398启动子在体外参与了对铜缺乏的反应拟南芥[22].这与我们之前对PtSPLs的结果一致[40].根据系统进化树,PtSPL3和AtSPL7极有可能是同源蛋白(图2).此外,5对AtSPLs和11对PtSPLs似乎是副同源蛋白(图2).包括G1的AtSPL9/15、AtSPL10/11、PtSPL8/27、PtSPL12/13和PtSPL11/19, G2的PtSPL18/22和PtSPL14/15, G3的PtSPL21/26, G4的AtSPL14/16、AtSPL1/12、PtSPL2/9、PtSPL1/5和PtSPL6/7, G5的AtSPL3/4、PtSPL16/23和PtSPL20/25聚类。16个AtSPLs中的62.5%和28个PtSPLs中的78.5%是副同源对。它暗示了扩张SPL副同源基因分离后产生基因。从副同源对鉴定的结果是一致的部分重复在p . trichocarpa基因组(http://chibba.agtec.uga.edu/duplication/) [32],暗示了parogous的起源PtSPLs从分段复制。使用同义替换(Ks)值对串列复制事件的潜在年龄进行预测PtSPLs出现在9-21 mya(表2).这与年代是一致的p . trichocarpa基因组复制事件[32].
比较分析PtSPL而且AtSPL基因结构
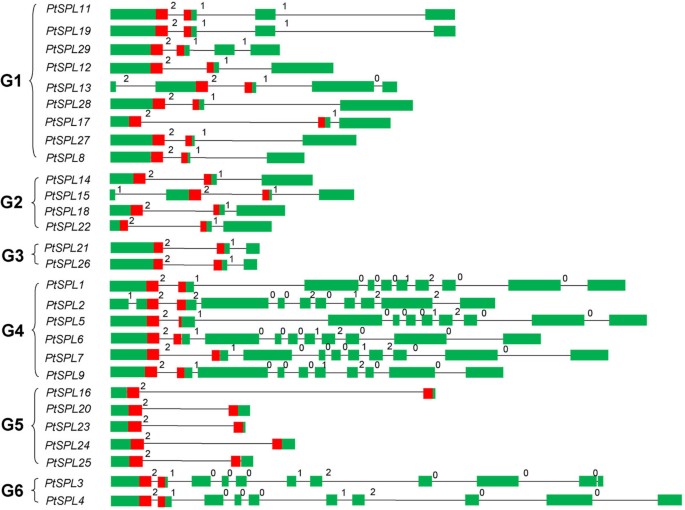
基因结构分析表明,编码区内含子数为28个PtSPL基因从1到10不等。的数量PtSPLs含1、2、3、4、9和10个内含子分别为5、10、4、1、6和2个(图3.,附加文件1).同样,内含子数量为16AtSPLs变化在1到9之间(附加文件2).内含子的分布模式PtSPLs很类似于AtSPLs以2和9内含子为主,其次是1和3内含子(图3.,其他文件1而且2) [41].此外,内含子在SBP结构域的位置高度保守。它位于SBP结构域第48个氨基酸的密码子(附加文件)4).这些结果表明外显子-内含子之间的结构守恒PtSPLs而且AtSPLs.
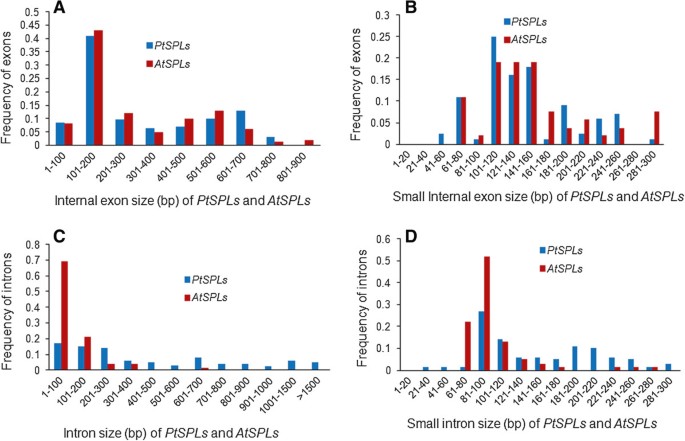
内含子的长度在不同物种间差异显著PtSPL如G1、G2和G5中的基因(图2)3.).我们分析了其内部的外显子和内含子PtSPLs而且AtSPLs.结果表明PtSPLs大小为43 - 884 bp,平均长度为314 bp,略大于平均长度为293 bp的AtSPL外显子。大约59%PtSPL外显子和63%的AtSPL外显子的大小在300 bp以下,71%和70%的外显子在60 - 160 bp之间PtSPLs而且AtSPLs,分别为(图4).虽然大小分布PtSPLs外显子是相似的AtSPL外显子、内含子大小分布变化较大,从30 bp到3.0 kb不等。有6个PtSPL内含子(5%)大小为>1.5 kb;然而,不存在这样的内含子AtSPLs.大约55%PtSPLs内含子大小在300 bp以下,56%的内含子在60 - 160 bp之间;然而,大多数的AtSPLs(94%)的尺寸小于300 bp。的平均大小PtSPL内含子的长度为476 bp,远高于内含子的120 bpAtSPLs.这些结果提示了外显子和内含子大小分布的差异PtSPLs而且AtSPLs.
鉴定25个保守基序
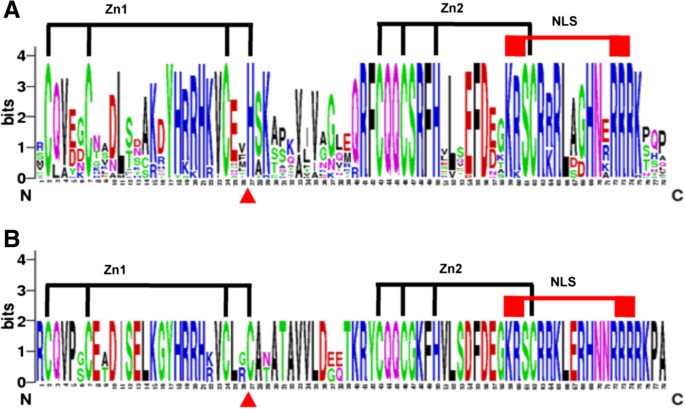
PtSPLs的保守结构域用Pfam (http://pfam.sanger.ac.uk),并通过BLAST分析蛋白质序列与保守域数据库(CDD,http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi).结果显示,28个PtSPLs和16个AtSPLs均含有一个长度约78个氨基酸残基的SBP结构域(图5).这并不奇怪,因为SBP域被用于PtSPL识别。SBP结构域序列分析显示,在PtSPLs的SBP结构域中也存在保守的锌结合位点Zn1和Zn2(图2)5).Zn1在G1-G5 SPLs中是cys3his型(CCCH-type)(图5一个);然而,Zn1中的His残基被G6中的Cys残基所取代,导致G6 SPLs的签名序列为CCCC(图5B).与Zn1不同,Zn2的特征序列(C2HC)在所有分析的SPLs中都是高度保守的。除了Zn1和Zn2外,SBP结构域的c端还包含一个保守的核定位信号(NLS)(图5).SBP结构域在PtSPLs和AtSPLs之间的保守性表明在古代植物中已经建立了结构域组织。此外,6个属于G4的PtSPLs (PtSPL1, PtSPL2, PtSPL5, PtSPL6, PtSPL7和PtSPL9)包含一个ANK或ANK -2域,具有3个或4个锚蛋白重复序列(附加文件)5),它们参与蛋白质与蛋白质的相互作用[42].这与AtSPLs和SmSPLs先前的结果一致[41].
除了保守结构域,其他保守基序也可能对SPLs的功能起重要作用[27,43].我们使用MEME搜索保守motif,并应用e-value cut off 1e−10感谢大家的认可。结果鉴定出28个PtSPLs的25个motif(图6、表3.).大多数确定的基序在PtSPLs和AtSPLs之间是保守的[41],而包括基序11、19和23在内的3个基序是特定于PtSPLs的。这表明了ptsps和atsps的保护性和多样性。每个SPL中的图案数量从1到16不等(图6).Motif 1实际上是SBP结构域。一致地,它存在于所有分析的SPLs中。G1和G2 SPLs中存在的Motif 14包含miR156的靶基因序列,表明miR156对G1和G2 SPLs的转录后调控。除了motif 1和14之外,还有一些motif广泛存在于两个SPL组中,例如motif 12存在于G1和G2中,motif 2、4、5、6、15和16存在于G4和G6中6),表明这些母题的重要性。我们还发现一些motif是群体唯一的,例如motif 24专门存在于G6 SPLs中,motif 7、9、10和18专门存在于G4中(图4)6).这些群体特有的母题对于群体中SPLs的特定角色可能很重要。此外,PtSPLs和AtSPLs [41]在一个群体中具有相似的基序,这表明它们可能在植物生长发育中发挥类似的作用。
表达模式:在p . trichocarpa
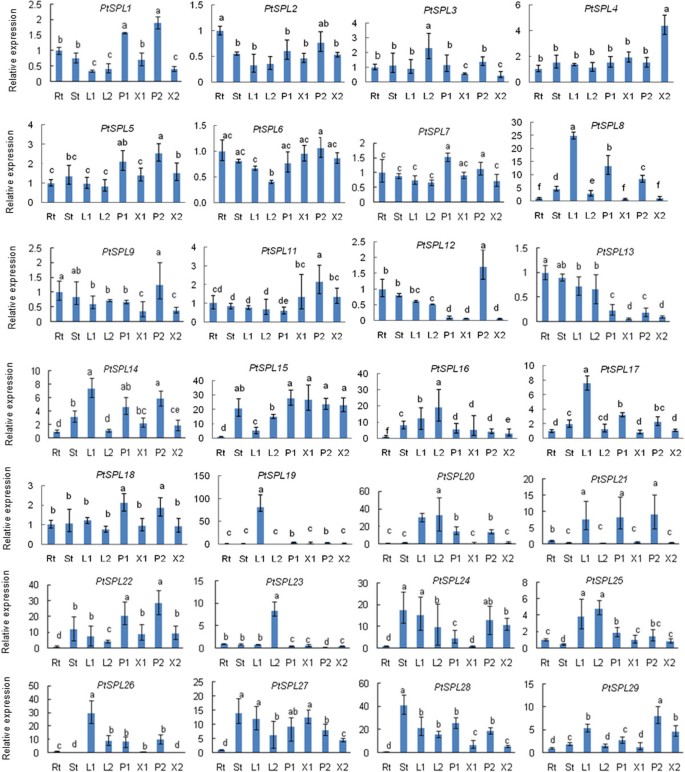
基因的表达模式往往与其功能相关。为了初步阐明的作用PtSPLs在p . trichocarpa在开发过程中,我们首先从微阵列分析中搜索PopGenIE的基因表达数据[44].除了PtSPL17,表达水平为27PtSPLs分别在根、茎、幼叶和成熟叶中进行了测定(图7).接下来,我们检测了28的相对表达水平PtSPLs1年生的幼叶、成熟叶、幼茎、幼根和发育中的次生木质部和韧皮部组织,分别为4 ~ 6节和12 ~ 25节p . trichocarpa采用实时定量RT-PCR方法对植物进行定量检测(图8).结果表明,qRT-PCR数据与基因芯片数据基本一致PtSPLs在根、茎、幼叶和成熟叶中(图7而且8).尽管所有的PtSPLs在至少一个被检查的组织中表达,观察到差异表达。许多假定的副同源基因,如PtSPL18/22在G2,PtSPL21/26在G3,PtSPL2/9,PtSPL1/5而且PtSPL6/7在G4和PtSPL16/23属于G5的,表现出相似的表达模式,说明它们的作用是冗余的PtSPL基因对。然而,很少有基因对的表达模式,包括PtSPL12/13在G1,和PtSPL14/15属于G2是不同的。它表示这些PtSPLs可能扮演不同的角色p . trichocarpa发育,尽管它们是副同源基因。
mir156介导的转录后调控PtSPLs
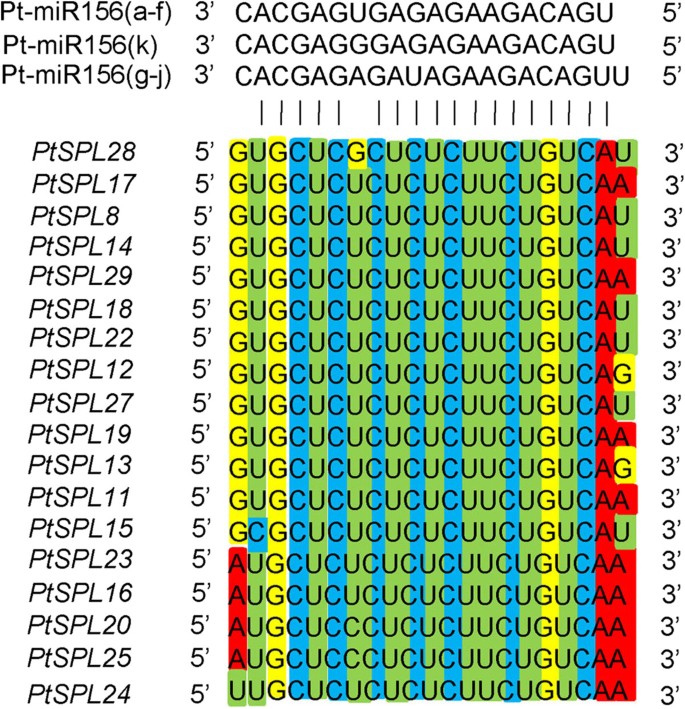
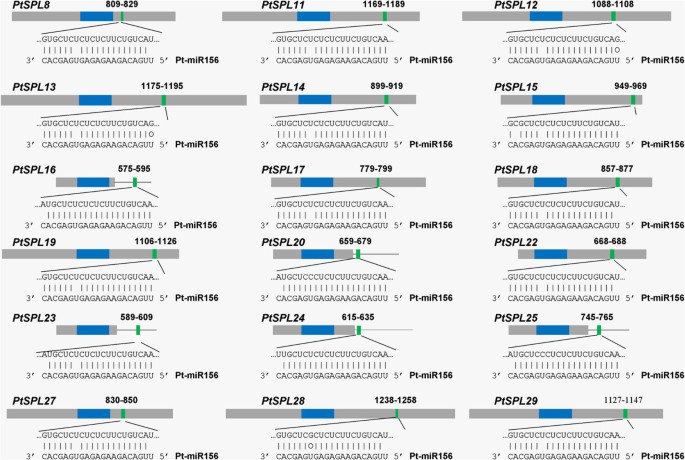
已证明10AtSPLs都受到miR156的调控[11].miR156的互补位点位于基因的编码区或3 ' UTRsAtSPLs.从而了解mir156给药后的转录后调控PtSPLs,我们搜索了所有编码区域和3 ' utrPtSPLs针对p . trichocarpamiR156a-miR156j在psRNATarget服务器上使用默认参数[45].结果表明,18PtSPLs是miR156的潜在靶点(图9而且10).13 . mir156靶向位点PtSPLsG1和G2位于最后一个外显子,编码保守肽ALSLLS。目标站点为其他5个PtSPLs属于G5的位于靠近停止密码子的3 ' utr(图10).一致地,AtPSLs在G1, G2和G5中聚集是miR156的靶点拟南芥.这表明mir156介导的SPLs转录后调控是保守的p . trichocarpa而且拟南芥.
讨论
SPLs是植物特异性转录因子,包含高度保守的SBP (SQUAMOSA启动子结合蛋白)结构域。它能特异性结合花分生组织鉴定基因的启动子SQUAMOSA及其同源基因,在植物生长发育中起着重要的调控作用[46- - - - - -49].基因编码:已从各种植物物种中鉴定出来,如拟南芥[2,10,23,26],玉米[30.),金鱼草majus[3.],大米[50]、白桦[51),而美国miltiorrhiza[41].SPL基因在植物中作为一个大的基因家族存在。的数量:在拟南芥、大米、p .金属盘,玉米和番茄分别为16、19、13、31和15 [4- - - - - -9].全基因组序列的可用性使我们能够进行全基因组的鉴定:在p . trichocarpa.分析了三个版本的注释p . trichocarpa基因组显示存在28个全长PtSPLs,分布在14条染色体上。这是第一次尝试分析PtSPL基因家族。研究结果为阐明其功能提供了依据:在p . trichocarpa一棵模型森林树。
的数量SPL基因在p . trichocarpa比那大得多吗拟南芥、大米、p .金属盘还有番茄,虽然和玉米的数量差不多:[4- - - - - -9].序列同源分析表明,基因重复在发病中起重要作用SPL基因扩增p . trichocarpa.共11对染色体内重复PtSPLs在本研究中被确定。在系统发育树中,它们都聚集在一起(图2).这与先前在其他生物(如小鼠、人类和动物)中基因家族的生成和维持的发现是一致的拟南芥[52,53].事实上,许多植物转录因子基因家族都有基因复制的报道,例如MYB,AP2,麦斯等等[54- - - - - -56]和重复的SPL基因对已在拟南芥(AtSPL10/11,AtSPL4/5而且AtSPL1/12)和米(OsSPL2/19,OsSPL3/12,OsSPL4/11,OsSPL5/10而且OsSPL16/18) [57- - - - - -61].然而,同源的数目PtSPL基因对明显大于在拟南芥而水稻,说明在水稻中发生了更多的节段复制事件杨树和最SPL基因在拟南芥而且杨树以特定物种的方式扩展[62- - - - - -64].
比较分析trichocarpa PtSPLs而且拟南芥AtSPLs揭示了许多保守序列特征。例如,所有推导出的蛋白质都含有高度保守的SBP结构域,约有78个氨基酸残基。在sbp结构域编码区内含子的位置和内含子相位也是保守的SPL基因在p . trichocarpa而且拟南芥,表示植物SPL基因起源于共同的祖先。基于MEGA 5.1构建的邻居连接(NJ)系统发育树。, 44个SPL蛋白p . trichocarpa而且拟南芥聚类为6组。每组至少包括一个PtSPL和一个AtSPL。同一组内PtSPLs和AtSPLs的内含子数量和内含子相位相似。结果表明,之间的守恒trichocarpa PtSPLs而且拟南芥AtSPLs.
已经证明AtSPLs在各种发育过程中起着重要的调节作用拟南芥.例如,茎生叶和花的形态性状是由AtSPL2,AtSPL10而且AtSPL11[19].幼体到成体的生长阶段转变和起叶率是由叶片的冗余作用控制的AtSPL9而且AtSPL15[21].花粉囊发育、雄性育性和GA生物合成及信号通路受其调控AtSPL8,是G3的成员[23- - - - - -25].铜稳态拟南芥由第6组成员控制,AtSPL7[22].在本研究中,我们发现许多基序是独一无二的或主要存在于一组SPLs中。这与AtSPLs在一个组中的冗余角色相一致,表明同一组中的PtSPLs成员可能扮演与他们相似的角色拟南芥同行。不同群体中SPLs的功能可能不同。另一方面,发现了3个PtSPLs特异性基序,包括基序11、19和23,这表明一些PtSPLs可能发挥了物种特异性作用。一致地,大部分都是谬误的PtSPL同一组中的基因对表现出相似的表达模式,而少数基因对表现出差异模式。结果表明,在同一植物种内和不同物种之间,SPLs存在亚功能化和新功能化。
mir156给药的转录后调节是重要的功能:[11,41,65].目标预测显示全部PtSPLs组1、组2、组5均受miR156调控。miR156的互补位点位于G1和G2的编码区:,而位于G5的3 ' UTR:.结果与拟南芥:并提示mir156介导的植物转录后调控的保守性。
结论
在本研究中,从植物的全基因组序列中共鉴定出28个全长SPLsp . trichocarpa.通过对基因结构、系统发育关系、染色体位置、保守基序、表达模式和mir156介导的转录后调控的综合分析,研究人员发现PtSPL对基因家族进行了特征分析,并与:在拟南芥.结果表明,28个PtSPLs和16个AtSPLs聚为6组。组内许多PtSPLs和AtSPLs在序列特征、基因结构、基序、表达模式和转录后调控等方面高度保守,表明植物组内SPLs具有一定的保守性。然而,观察到显著差异:组间。此外,在PtSPLs中鉴定出了各种基序,但在AtSPLs中没有。这表明植物SPLs的多样性。研究结果为阐明蛋白的功能提供了有用的信息:在p . trichocarpa.
方法
的识别PtSPL基因
已知的核苷酸序列和推导的氨基酸序列有16个SPL基因在拟南芥[2,4]从TAIR数据库(http://www.arabidopsis.org)(附加文件2).的SBP域AtSPLs使用Pfam (http://pfam.sanger.ac.uk).BLAST搜索PtSPLs反对杨树trichocarpaV1.1、v2.2和v3.0采用AtSPLSBP为查询序列[32) (http://genome.jgi-psf.org/Poptr1_1/Poptr1_1.home.html,http://www.phytozome.net/poplar.php#B).从1e截去的e值−5被应用于识别。我们还使用网络服务器上的关键字搜索工具搜索数据库中的SBP。提取的蛋白质序列杨树trichocarpa基于序列恒等式对V1.1、v2.2、v3.0进行对齐组合。
染色体定位与序列特征分析
染色体位置PtSPL基因通过BLAST分析确定PtSPLs反对杨树trichocarpav3.0 (http://www.phytozome.net/poplar.php#B).植物基因组复制数据库(PGDD)服务器(http://chibba.agtec.uga.edu/duplication/index/locus),显示范围为100kb。复制事件的大致日期是用T = Ks/2λ来计算的,假设类似时钟的速率(λ)在杨树适用于9.0 × 10−9[32,57,66].利用DnaSP计算副同源基因对的同义替换(Ks)值[67].理论等电点(p我)和分子量(Mw)使用ExPASy服务器上的Compute pI/Mw工具进行预测(http://web.expasy.org/compute_pi/) [68].的内含子/外显子结构SPL基因结构显示服务器(http://gsds.cbi.pku.edu.cn/chinese.php) [69].
系统发育构建和母序分析
在MEGA5.1中使用邻居连接(NJ)方法构建系统发育树。分支可靠性通过使用1000个自举重复的自举重采样方法进行评估。只分析引导值大于50%支持的节点。PtSPLs的保守结构域用Pfam (http://pfam.sanger.ac.uk),并通过BLAST分析蛋白质序列与保守域数据库(CDD,http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi),预期e值阈值为1.0,最大命中量为500个氨基酸[70].用clustalW对SBP结构域的78个氨基酸进行了比对。序列logo是使用weblogo平台生成的(http://weblogo.berkeley.edu/).利用MEME包预测潜在的蛋白基序(http://meme.sdsc.edu/meme/),并应用以下参数。它包括motif的分布:每序列0个和1个,motif的最大寻找数量:25个,motif的最小宽度:8个,motif的最大宽度:150个。从1e截去的e值−10被应用于识别。
定量实时逆转录pcr (qRT-PCR)
p . trichocarpa植物在人工气候室中生长了大约一年。幼叶(顶部第2 ~ 3位)、成熟叶(顶部第12位)、幼茎(顶部第1 ~ 3位)、幼根、发育次生木质部和韧皮部组织(顶部第4 ~ 6位和第12 ~ 25位节间)p . trichocarpa收集植物。进行了三次生物重复。总RNA采用植物总RNA提取试剂盒(Aidlab, China)提取。基因组DNA污染通过用RNase-free DNase预处理总RNA来消除(Promega,美国)。RNA完整性在1.2%琼脂糖凝胶上进行分析,其数量使用NanoDrop 2000C分光光度计(Thermo Scientific, USA)测定。总RNA用Superscript III逆转录酶(Invitrogen, USA)进行逆转录。使用基因特异性引物对每个组织样本进行三份qrt - pcr(附加文件)6),如上文所述[71].qRT-PCR的程序如下:95℃预变性30s, 95℃5 s扩增40次,60℃18 s, 72℃15 s。扩增子长度在80 ~ 250 bp之间。肌动蛋白作为前文所述的内参基因[72].分离曲线用于评估扩增特异性。比较Ct法分析转录物的相对丰度[73].使用算术公式2-ΔΔCq来获得相对量化的结果。Cq表示阈值周期。对来自三个生物重复的基因表达数据进行标准化,如所述[74].统计分析采用SPSS (Version 19.0, IBM, USA)计算方差分析(ANOVA)。P < 0.05为差异有统计学意义。
微阵列数据分析
微阵列数据PtSPLs由PopGenIE的eplant组织表达工具获得(http://www.popgenie.org/).数据进行基因归一化,然后使用Cluster 3.0中的平均连锁聚类技术进行分析[75].
的预测PtSPLsmiR156靶向
的序列p . trichocarpamiR156a-miR156j是从miRBase [36) (http://www.mirbase.org/).PtSPLs通过对所有基因的编码区和3 ' utr进行搜索,预测miR156靶向的基因PtSPLs的互补序列p . trichocarpamiR156a-miR156j在psRNATarget服务器上使用默认参数[45) (http://plantgrn.noble.org/psRNATarget/?function=3).
支持数据的可用性
支持本文结果的数据集包含在本文及其附加文件中。
缩写
- 治疗:
-
Cu-response元素
- 兆瓦:
-
分子量
- NJ:
-
Neighbor-joining
- NLS:
-
核定位信号
- pI:
-
等电点
- 存在:
-
定量实时逆转录pcr
- SBP:
-
鳞状突起启动子结合蛋白
- Zn1:
-
锌指1
- Zn2:
-
锌指
参考文献
- 1.
Yamasaki K, Kigawa T, Inoue M, Tateno M, Yamasaki T, Yabuki T, Aoki M, Seki E, Matsuda T, Nunokawa E, Ishizuka Y, Terada T, Shirouzu M, Osanai T, Tanaka A, Seki M, Shinozaki K, Yokoyama S: dna结合域溶液结构揭示的一种新的锌结合基序拟南芥sbp家族转录因子。中华分子生物学杂志,2004,37:49-63。10.1016 / j.jmb.2004.01.015。
- 2.
Birkenbihl RP, Jach G, Saedler H, Huijser P:植物特异性sbp结构域的功能解剖:dna结合和核定位结构域的重叠。中国生物医学工程学报,2005,36(4):585-596。10.1016 / j.jmb.2005.07.013。
- 3.
李志强,李志强,李志强,等:DNA结合蛋白家族的研究进展金鱼草majus花分生组织鉴定基因。中国生物医学工程学报,1996,25(3):344 - 344。
- 4.
王志刚,王志刚,王志刚,王志刚,王志刚,王志刚拟南芥SBP-box基因。基因工程学报,1999,37(3):344 - 344。10.1016 / s0378 - 1119 (99) 00308 - x。
- 5.
王晓明,王晓明,王晓明,王晓明,王晓明。苔藓微rna的克隆与鉴定。植物学报,2005,29(3):344 - 344。10.1111 / j.1365 - 313 x.2005.02499.x。
- 6.
胡尔特奎斯特JF,多韦勒JE:女性化的玉米流苏mop1而且壹空间突变体表现出miR156和特定SBP-box基因水平的改变。植物学报,2008,29(4):344 - 344。10.1007 / s00425 - 008 - 0813 - 2。
- 7.
李志刚,李志刚,李志刚,李志刚。苔藓中sbp结构域转录因子对隐色素介导的蓝光信号通路的影响Physcomitrella金属盘.植物学报,2008,27(3):344 - 344。
- 8.
Miura K, Ikeda M, Matsubara A, Song X, Ito M,浅野K, Matsuoka M,北野H, Ashikari M: OsSPL14促进水稻穗部分枝,提高籽粒产量。王志强,2010,30(4):549 -549。10.1038 / ng.592。
- 9.
Salinas M, Xing S, Höhmann S, Berndtgen R, Huijser P:番茄SBP-box转录因子家族的基因组组织系统发育比较及差异表达。植物学报,2012,35(3):344 - 344。10.1007 / s00425 - 011 - 1565 - y。
- 10.
王志强,王志强,王志强,王志强拟南芥SBP-box基因SPL3:参与花转化的新基因。植物学报,1997,12:367-377。10.1046 / j.1365 - 313 x.1997.12020367.x。
- 11.
Rhoades MW, Reinhart BJ, Lim LP, Burge CB, Bartel B, Bartel DP:植物microRNA靶点的预测。光子学报,2002,29(4):531 - 532。10.1016 / s0092 - 8674(02) 00863 - 2。
- 12.
谢泽,谢志强,李志强,李志强,李志强,李志强,李志强。RNA沉默病毒抑制因子P1/HC-Pro的研究进展拟南芥发育和miRNA功能。生物工程学报,2003,30(4):344 - 344。10.1016 / s1534 - 5807 (03) 00025 - x。
- 13.
陈娟,李文霞,谢东,彭建荣,丁文文:病毒毒力蛋白抑制RNA沉默介导的防御,但上调微RNA在宿主基因表达中的作用。中国生物医学工程学报,2004,26(3):344 - 344。10.1105 / tpc.018986。
- 14.
Schwab R, Palatnik JF, Riester M, Schommer C, Schmid M, Weigel D: microrna对植物转录组的特异性影响。生物工程学报,2005,8:517-527。10.1016 / j.devcel.2005.01.018。
- 15.
吴刚,李志刚,李志刚:植物茎部发育的时间调控拟南芥miR156和它的靶标SPL3.发展,2006,133:3539-3547。10.1242 / dev.02521。
- 16.
王继伟,王俊伟,捷克B, Weigel D: mir156调控的SPL转录因子在植物内源开花通路中的作用拟南芥.细胞学报,2009,38(3):344 - 344。10.1016 / j.cell.2009.06.014。
- 17.
于楠,蔡文军,王珊珊,单cm,王丽娟,陈晓霞:microrna156靶向SPL基因对毛状体分布的时间调控拟南芥.中国生物医学工程学报,2010,29(3):344 - 344。10.1105 / tpc.109.072579。
- 18.
Gandikota M, Birkenbihl RP, Höhmann S, Cardon GH, Saedler H, Huijser P: miRNA156/157的3'UTR识别元件拟南芥SBP box基因SPL3通过翻译抑制抑制幼苗早花。植物学报,2007,29(3):344 - 344。10.1111 / j.1365 - 313 x.2006.02983.x。
- 19.
四方M,小山T,三田N, Ohme-Takagi M:拟南芥SBP-box基因SPL10,SPL11而且SPL2在繁殖阶段控制与芽成熟相关的形态变化。植物生理学报,2009,30(4):344 - 344。10.1093 /卡式肺囊虫肺炎/ pcp148。
- 20.
Vazquez F, Gasciolli V, Crété P, Vaucheret H:核dsRNA结合蛋白HYL1是microRNA积累和植物发育所必需的,而不是转录后转基因沉默。中国生物医学工程学报,2004,14:346-351。
- 21.
Schwarz S, Grande AV, Bujdoso N, Saedler H, Huijser P: microRNA调控SBP-box基因SPL9而且SPL15控制嫩枝成熟拟南芥.植物化学学报,2008,29(3):344 - 344。10.1007 / s11103 - 008 - 9310 - z。
- 22.
Yamasaki H, Hayashi M, Fukazawa M, Kobayashi Y, Shikanai T: SQUAMOSA启动子结合蛋白样7是铜稳态的中心调节因子拟南芥.植物学报,2009,21:347-361。10.1105 / tpc.108.060137。
- 23.
Unte US, Sorensen AM, Pesaresi P, Gandikota M, Leister D, Saedler H, Huijser P:影响花粉囊发育的SBP-Box基因SPL8拟南芥.植物学报,2003,29(3):344 - 344。10.1105 / tpc.010678。
- 24.
邢星,Salinas M, Höhmann S, Berndtgen R, Huijser P: mir156靶向和非靶向SBP-box转录因子协同作用的男性生育能力拟南芥.中国生物医学工程学报,2010,26(3):357 - 357。10.1105 / tpc.110.079343。
- 25.
张艳,王晓明,张晓明,王晓明,等。赤霉素介导的植物生长发育过程中SPL8基因的表达拟南芥.中国生物医学工程学报,2007,29(4):429-439。10.1007 / s11103 - 006 - 9099 - 6。
- 26.
Stone JM, Liang X, Nekl ER, Stiers JJ:拟南芥AtSPL14植物特异性sbp结构域转录因子参与植物发育和对伏马菌素B1的敏感性。植物学报,2005,41:744-754。10.1111 / j.1365 - 313 x.2005.02334.x。
- 27.
谢坤,吴超,熊玲:水稻SQUAMOSA启动子结合样转录因子与微rna156的基因组组织、差异表达及相互作用。植物科学进展,2006,29(3):344 - 344。10.1104 / pp.106.084475。
- 28.
焦艳,王颖,薛东,王娟,闫敏,刘刚,董刚,曾东,刘震,朱霞,钱倩,李娟:OsmiR156对水稻OsSPL14基因调控的研究进展。王志强,2010,30(4):541-544。10.1038 / ng.591。
- 29.
王松,吴凯,袁强,刘旭,刘震,林旭,曾锐,朱辉,董刚,钱倩,张刚,付旭:用颗粒尺寸形状和质量控制OsSPL16在大米。王文杰,2012,30(4):357 - 357。10.1038 / ng.2327。
- 30.
克拉夫特PW,邦加德-皮尔斯DK,西尔维斯特AW, Poethig RS, Freeling M: Theliguleless-1基因在玉米叶片发育中起组织特异性作用。中国生物医学工程学报,1998,29(4):344 - 344。10.1016 / 0012 - 1606(90) 90117 - 2。
- 31.
莫雷诺MA,哈珀LC,克鲁格RW,德拉波塔SL,弗里林M:liguleless1编码一种在玉米叶片器官发生过程中诱导叶舌和耳廓所需的核定位蛋白。基因工程学报,1997,11:616-628。10.1101 / gad.11.5.616。
- 32.
Tuskan GA, DiFazio S, Jansson S, Bohlmann J, grigoriiev I, Hellsten U, Putnam N, Ralph S, Rombauts S, Salamov A, Schein J, Sterck L, Aerts A, Bhalerao RR, Bhalerao RP, Blaudez D, Boerjan W, Brun A, Brunner A, Busov V, Campbell M, Carlson J, Chalot M, Chapman J, Chen GL, Cooper D, Coutinho PM, Couturier J, Covert S, Cronk Q,等:黑杨木基因组杨树trichocarpa(托。&灰色)。科学通报,2006,29(3):344 - 344。10.1126 / science.1128691。
- 33.
吕松,孙永华,史锐,陈志强,李玲,常维林:白杨植物中缺失的机械胁迫响应microrna拟南芥.中国生物医学工程学报,2005,27(3):366 - 366。10.1105 / tpc.105.033456。
- 34.
Tuskan GA, DiFazio SP, Teichmann T:杨树基因组学越来越流行:杨树基因组计划对树木研究的影响。植物学报,2004,6:2-4。
- 35.
吕松,孙永华,蒋维林:胁迫响应microrna杨树.植物学报,2008,29(3):344 - 344。10.1111 / j.1365 - 313 x.2008.03497.x。
- 36.
Kozomara A, griffith - jones S: miRBase:集成microRNA注释和深度测序数据。生物工程学报,2011,39:D152-D157。10.1093 / nar / gkq1027。
- 37.
普西JR,卡格A,阿克塞特尔M,克雷默EM:深度注释杨树trichocarpa来自不同组织的microrna。公共科学学报,2012,7:e33034-10.1371/journal.pone.0033034。
- 38.
陆松,李强,魏华,张俊梅,Tunlaya-Anukit S, Kim H,刘杰,宋杰,孙玉华,袁林,叶tf, Peszlen I, Ralph J, Sederoff RR, Chiang VL: Ptr-miR397a是影响木质素含量的漆酶基因负调控因子杨树trichocarpa.美国国家科学研究院。2013, 30(4): 457 - 457。10.1073 / pnas.1308936110。
- 39.
潘帅,梁东,张震,殷伟,夏霞:干旱响应性的新鉴定杨树trichocarpa通过高通量测序microRNAs及其降解体分析的靶标。中国生物医学工程学报,2013,30(4):344 - 344。
- 40.
吕松,杨超,蒋维林:microrna相关铜调控网络的多样性与保护杨树trichocarpa.中国生物医学工程学报,2011,29(3):349 - 349。10.1111 / j.1744-7909.2011.01080.x。
- 41.
张磊,吴斌,赵东,李超,邵峰,陆松:植物全基因组分析与分子解剖SPL基因家族丹参.中国生物医学工程学报,2013,29(3):344 - 344。
- 42.
王志刚,彭志刚,王志刚:锚蛋白重复基序的结构决定因素分析。Proc Natl Aca science美国。2002, 29(3): 344 - 344。10.1073 / pnas.252537899。
- 43.
郭爱英,朱庆华,顾霞,葛霞,杨娟,罗娟:植物特异性SBP-box转录因子家族的全基因组鉴定与进化分析。基因学报,2008,418:1-8。10.1016 / j.gene.2008.03.016。
- 44.
Sjödin A, Street NR, Sandberg G, Gustafsson P, Jansson S: The Populus Genome Integrative Explorer (PopGenIE):探索Populus基因组的新资源。新植醇。2009, 42(1): 1 - 4。10.1111 / j.1469-8137.2009.02807.x。
- 45.
戴旭,赵px: psRNATarget:植物小RNA靶标分析服务器。中国生物工程学报,2011,39:W155-W159。10.1093 / nar / gkr319。
- 46.
Huijser P, Klein J, Lonnig WE, Meijer H, Saedler H, Sommer H:苞片狂花(Bracteomania)是一种花序异常,其原因是小麦中MADS-box基因SQUAMOSA功能缺失金鱼草majus.中国农业科学,2002,23(4):349 - 349。
- 47.
Saedler H, Becker A, Winter KU, Kirchner C, Theissen G: MADS-box基因参与花序发育和进化。生物化学学报,2001,48:351-358。
- 48.
Fornara F, Parenicova L, Falasca G, Pelucchi N, Masiero S, Ciannamea S, Lopez-Dee Z, Altamura MM, Colombo L, Kater MM: MADS box基因AP1/SQUA亚家族成员OsMADS18的功能特征植物科学学报,2004,27(3):337 - 344。10.1104 / pp.104.045039。
- 49.
Robles P, Pelaz S:花卉和果实发育拟南芥.中国生物医学工程学报,2005,29(3):344 - 344。10.1387 / ijdb.052020pr。
- 50.
Shao CX, Takeda Y, Hatano S, Matsuoka M, Hirano HY:水稻编码SBP结构域蛋白的基因,SBP结构域蛋白是一种控制植物发育的新型转录因子。农学通报,1999,16:414 -
- 51.
Lännenpää M, Jänönen I, Hölttä-Vuori M, Gardemeister M, Porali I, Sopanen T:一个新的SBP-box基因BpSPL1在白桦树上(桦木属翻车机).中国生物医学工程学报,2004,29(4):349 - 349。10.1111 / j.0031-9317.2004.00254.x。
- 52.
Kent WJ, Baertsch R, Hinrichs A, Miller W, Haussler D:进化的大锅:小鼠和人类基因组的重复、删除和重排。美国国家科学研究院。2003, 30(4): 344 - 344。10.1073 / pnas.1932072100。
- 53.
卡农SB, Mitra A, Baumgarten A, Young ND, May G:片段和串联基因复制在大基因家族进化中的作用拟南芥.中国生物医学工程学报,2004,4:10-10.1186/1471-2229-4。
- 54.
杜华,杨世升,梁震,冯斌斌,刘玲,黄一斌,唐玉玉:大豆MYB转录因子超家族的全基因组分析。中国生物医学工程学报,2012,29(4):366 - 366。
- 55.
Zahn LM, Kong H, Leebens-Mack JH, Kim S, Soltis PS, Landherr LL, Soltis DE, Depamphilis CW, Ma H: MADS-box基因SEPALLATA亚家族的进化:被子植物历史上的多个重复的前被子植物起源。中国生物医学工程学报,2005,29(4):529 - 529。10.1534 / genetics.104.037770。
- 56.
沈洋,李志刚,李志刚,李志刚:AP2亚家族的分子进化。基因工程学报,2006,36(3):366 - 366。10.1016 / j.gene.2005.08.009。
- 57.
Blanc G, Wolfe KH:从复制基因的年龄分布推断模式植物中广泛的古多倍体。植物科学学报,2004,16:1667-1678。10.1105 / tpc.021345。
- 58.
杨晓明,王晓明,杨晓明,王晓明,等:基于染色体复制事件的被子植物基因组进化研究。自然科学,2003,29(4):433-438。10.1038 / nature01521。
- 59.
Paterson AH, Bowers JE, Chapman BA:在谷物分化之前的古代多倍体化及其对比较基因组学的影响。美国国家科学研究院。2004, 101: 9903-9908。10.1073 / pnas.0307901101。
- 60.
王霞,石霞,郝波,葛松,罗娟:水稻基因组复制与DNA片段丢失:二倍体化的意义。环境科学学报,2005,29(3):337 - 344。10.1111 / j.1469-8137.2004.01293.x。
- 61.
杨震,王霞,顾松,胡震,徐宏,徐成:水稻SBP-box基因家族的比较研究拟南芥和米饭。基因学报,2008,407:1-11。10.1016 / j.gene.2007.02.034。
- 62.
白j, Pennill LA, Ning J, Lee SW, Ramalingam J, Webb CA,赵b, Sun Q, Nelson JC, Leach JE, Hulbert SH:谷物中核苷酸结合位点富含亮氨酸重复基因的多样性。中国生物工程学报,2002,29(4):344 - 344。10.1101 / gr.454902。
- 63.
张松,陈超,李玲,孟l, Singh J,姜宁,邓晓伟,何志华,Lemaux PG:水稻壁相关激酶基因家族的进化扩展基因结构与表达。植物科学进展,2005,29(4):457 - 457。10.1104 / pp.105.069005。
- 64.
杨晓明,王晓明,王晓明,等:水稻早期生长素响应基因SAUR家族的基因表达及基因扩增(栽培稻).基因组学,2006,88:360-371。10.1016 / j.ygeno.2006.04.008。
- 65.
吴刚,Park MY, Conway SR, Wang JW, Weigel D, Poethig RS: miR156和miR172的序列作用调控了植物的发育时间拟南芥.细胞学报,2009,38(3):344 - 344。10.1016 / j.cell.2009.06.031。
- 66.
邵绍辉,潘瑞,曾玉华,梅凯芳,李文华:拟南芥和水稻受体样激酶家族的比较分析。植物学报,2004,16:1220-1234。10.1105 / tpc.020834。
- 67.
Librado P, Rozas J: DnaSP v5: DNA多态性数据综合分析软件。生物信息学,2009,25:1451-1452。10.1093 /生物信息学/ btp187。
- 68.
Bjellqvist B, Basse B, Olsen E, Celis JE:来自不同人类细胞类型的蛋白质的二维图的比较参考点,定义在pH值尺度,其中等电点与多肽组成相关。电泳,1994,15:529-539。10.1002 / elps.1150150171。
- 69.
郭爱英,朱庆华,陈晓霞,罗景昌:GSDS:一种基因结构显示服务器。易川,2007,29:1023-1026。10.1360 / yc - 007 - 1023。
- 70.
Marchler- bauer A, Lu S, Anderson JB, Chitsaz F, Derbyshire MK, dewese - scott C, Fong JH, Geer LY, Geer RC, Gonzales NR, Gwadz M, Hurwitz DI, Jackson JD, Ke Z, Lanczycki CJ, Lu F, Marchler GH, Mullokandov M, Omelchenko MV, Robertson CL, Song JS, Thanki N, Yamashita RA, Zhang D, Zhang N, Zheng C, Bryant SH: CDD:用于蛋白质功能注释的保守域数据库。生物工程学报,2011,39:D225-D229。10.1093 / nar / gkq1189。
- 71.
马勇,袁磊,吴波,李霞,陈松,陆松:萜类生物合成新基因的全基因组鉴定与鉴定丹参.应用物理学报,2012,29(3):379 - 379。10.1093 / jxb / err466。
- 72.
苏旭,范斌,袁琳,崔霞,陆松:RT-PCR定量分析基因表达内参基因的选择与验证杨树trichocarpa.植物学报,2013,48:507-518。
- 73.
Livak KJ, Schmittgen TD:使用实时定量PCR和2(−Delta Delta C(T))方法分析相关基因表达数据。方法,2001,25:402-408。10.1006 / meth.2001.1262。
- 74.
王丽娟,王丽娟,王丽娟,等。实时PCR基因表达数据的标准化研究。中国生物医学工程学报,2008,29(3):329 - 329。10.1016 / j.ab.2008.04.036。
- 75.
Eisen MB, Spellman PT, Brown PO, Botstein D:全基因组表达模式的聚类分析和显示。美国国家科学研究院。1998, 95: 14863-14868。10.1073 / pnas.95.25.14863。
确认
本工作得到国家重点基础研究计划(973计划)(2012CB114502给予S.L)和中国医学科学院北京协和医学院协和学者计划(SL)的资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
CL参与了生物信息学和qRT-PCR分析,并参与了手稿的撰写。SL设计实验,进行生物信息学分析并撰写稿件。两位作者都已阅读并认可了手稿的版本。
电子辅助材料
中PtSPLs的序列特征
附加文件1:p . trichocarpa。蛋白质长度,内含子数,p我中SPLs的分子量p . trichocarpa所示。(xls 33 kb)
的atsps序列特征
附加文件2:答:芥。基因id,蛋白质长度,内含子数,p我中SPLs的分子量答:芥所示。(xls 22 kb)
构建了77个SPLs的邻居连接(NJ)系统发育树
附加文件3:p . trichocarpa拟南芥,大米和美国miltiorrhiza。所鉴定的同源基因组和自举值显示。采用1000次自举重复的自举重采样方法评估分支的可靠性。引导值显示在节点下面。(doc 394 kb)
内含子在SBP结构域上的分布
附加文件4:杨树而且拟南芥。内含子在SBP结构域上的分布杨树而且拟南芥所示。(xls 17kb)
qRT-PCR分析所用引物
附加文件6:PtSPL基因。qRT-PCR所用引物全套。(xls 14kb)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/4.0),允许在任何媒介上不受限制地使用、传播和复制,前提是原创作品的名称要注明出处。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
李春华,陆胜,李春华SPL基因家族杨树trichocarpa.BMC植物生物学14日,131(2014)。https://doi.org/10.1186/1471-2229-14-131
收到了:
接受:
发表:
关键字
- 转录后的调控
- 基因内区相
- 副同源基因对
- SQUAMOSA启动子结合蛋白
- 25日节间