摘要
背景
温带地区用于粮食生产的高粱基因型光周期不敏感,开花早,在繁殖期避免不利环境。相比之下,能量高粱杂交种对光周期高度敏感,营养阶段在长时间内延长,从而增加了生物量积累。SbPRR37和SbGHD7通过抑制高粱的光周期敏感性SbEHD1而且英国《金融时报》,类似基因,因此在较长的白天推迟开花,而在较短的白天影响最小(PNAS_108:16469-16474, 2011;植物基因组_in press, 2014)。GIGANTEA (GI)-CONSTANS (CO)-开花位点T (FT)通路调节拟南芥和禾草的开花时间(J Exp Bot_62:2453-2463, 2011)。在长日开花植物,如拟南芥和大麦,CONSTANS激活英国《金融时报》在漫长的日子里表达和开花。在水稻中,短日开花植物Hd1,与CONSTANS同源,激活短日开花,抑制长日开花。
结果
通过对BTx642和Tx7000的重组自交系(RILs)在长日、短日和田间条件下进行筛选,发现了影响高粱开花时间的QTL。分析SBI-10上的开花时间QTL,发现BTx642编码一个隐性基因君士坦斯在B-box 2中含有His106Tyr替换的等位基因,已知可以使CONSTANS失活拟南芥.遗传分析表明,高粱CONSTANS是一种花激活剂,通过诱导表达促进开花提前启航日期1(SbEHD1)和玉米FT基因的高粱同源基因ZCN8(SbCN8),ZCN12(SbCN12).花抑制因子伪反应调节蛋白37(PRR37)对高粱CONSTANS活性和长日开花有抑制作用。
结论
高粱CONSTANS是一种开花激活因子,在长时间内被花抑制因子PRR37转录后抑制,有助于光周期敏感的开花高粱二色的,一种短日照植物。
背景
花转变时机的优化调控对作物的繁殖成功和产量至关重要。C4草高粱二色的广泛适应,在北纬40度0到>的地区作为一年生作物种植。根据生长地点的不同,高粱作物有不同的开花时间,并可作为粮食、糖、饲料或生物量的来源[1- - - - - -3.].一般选择花期早(60-80天)的高粱,通过在繁殖阶段避免干旱、不利温度和昆虫压力来提高粮食产量的稳定性。相比之下,为了延迟开花和延长营养生长的时间,能量高粱杂交品种设计了高光周期敏感性,从而使生物量产量增加了2倍以上[3.,4].综合植物发育阶段、光周期、温度、赤霉素等因子信号调控高粱花期[5].
光周期响应开花时间调控通路的遗传结构已在许多植物中被表征[6- - - - - -18].在拟南芥中,通过光信号和生物钟输出的巧合,在长日(LD)中促进开花,从而使植物能够感知光周期的季节性变化并作出反应。开花途径的时钟输出部分由GIGANTEA(胃肠道).GI是由中央时钟振荡器组成的驾驶室表达时机(TOC1),生物钟相关1(CCA1),晚细长的下胚轴(LHY).在漫长的白天,胃肠道活动君士坦斯(有限公司)表达结合黄素结合KELCH结构域F盒蛋白1(FKF1)通过诱导CDF1阻遏子的降解君士坦斯转录。由于隐色素(CRY1/2)、光敏色素A (PHYA)和抑制素介导的稳定作用,CO在LD中积累巴- 105(SPA1),以对抗光敏色素B (PHYB)介导的CO降解:本构PHOTOMORPHOGENIC 1(COP1) [6,9].长时间内CO蛋白水平的增加会激活开花轨迹T(英国《金融时报》)的表达和产生,从叶片移动到茎尖分生组织(SAM),在那里它与FD结合并诱导花的转变。
GI-CO-FT调控通路在长日(LD)植物拟南芥中被发现,在短日(SD)植物水稻中也存在[10].当水稻暴露在感应SD下时,标题DATE1(即),对应的有限公司,激活的表达英国《金融时报》例如基因Hd3a这是水稻中致花素的两个来源之一。在无感性LD中,Hd1作为一种阻遏剂Hd3a和开花19].因此,水稻的光周期敏感性部分取决于CO (Hd1)在长日和短日活动的差异。在水稻中发现了禾本科植物特有的两种花期调节剂:提前启航日期1(EHD1) [20.),粒数、株高、抽穗期7(GHD7) [21,22].EHD1激活的表达Hd3a而且水稻开花位点t1(RFT1),在漫长的白天,它是致花素的来源。GHD7通过下调EHD1而且Hd3aLD在水稻中的含量[23),SbEHD1而且SbCN8在高粱2].
光周期对开花时间的影响在草种之间和草种内部差异很大。大麦和小麦是LD植物,水稻和高粱是SD植物。大多数栽培玉米是光周期不敏感的,因此植株在一定的度数天后开花;然而,热带玉米是一种对光周期敏感的短日照植物[11].高粱是一种短日照植物,尽管谷物高粱通常对光周期不敏感,饲料高粱和能量高粱基因型表现出不同程度的光周期敏感性[3.].高粱花期QTL已鉴定出40多个[24].的Ma1-Ma4位点是在美国育种早花光周期不敏感谷物高粱时发现的(1920-1960)[25].Ma1对应于伪反应调节蛋白37(SbPRR37),是LD开花的抑制因子[1].马编码光敏色素B(PhyB),一种红光感受器,在光周期感应和抑制开花中发挥重要作用[26- - - - - -28].Ma6编码SbGhd7,一种SbEHD1在漫长的日子里表达和开花[2].Ma2, Ma4,Ma5高粱的开花时间位点是否增强了光周期敏感性[25,29].
CONSTANS (CO)最初被认为是一种转录激活因子英国《金融时报》和拟南芥的开花[30.].CO属于植物特有的转录因子家族,包含一个或两个n端锌指B-box结构域和一个c端CCT结构域。Zn指结构域中两个保守的半胱氨酸和组氨酸氨基酸对CO活性至关重要[6].在这些位置有氨基酸取代的拟南芥突变体具有晚开花表型。在该基因家族中发生了大量的基因复制事件,导致拟南芥中有~17个CO家族成员,水稻中有~16个,大麦中有~9个[31,32].水稻中CONSTANS的同源基因Hd1通过激活SD中的开花和抑制LD中的开花,在开花的光周期调控中起关键作用[19].等位基因的即约占栽培水稻开花时间变异的44% [33].即LD和SD中Hd1的转录物和蛋白水平相似,这与PHYB在转录后调节Hd1活性的发现一致[34]和PRR37 [35,36].
结果
花期QTL的鉴定
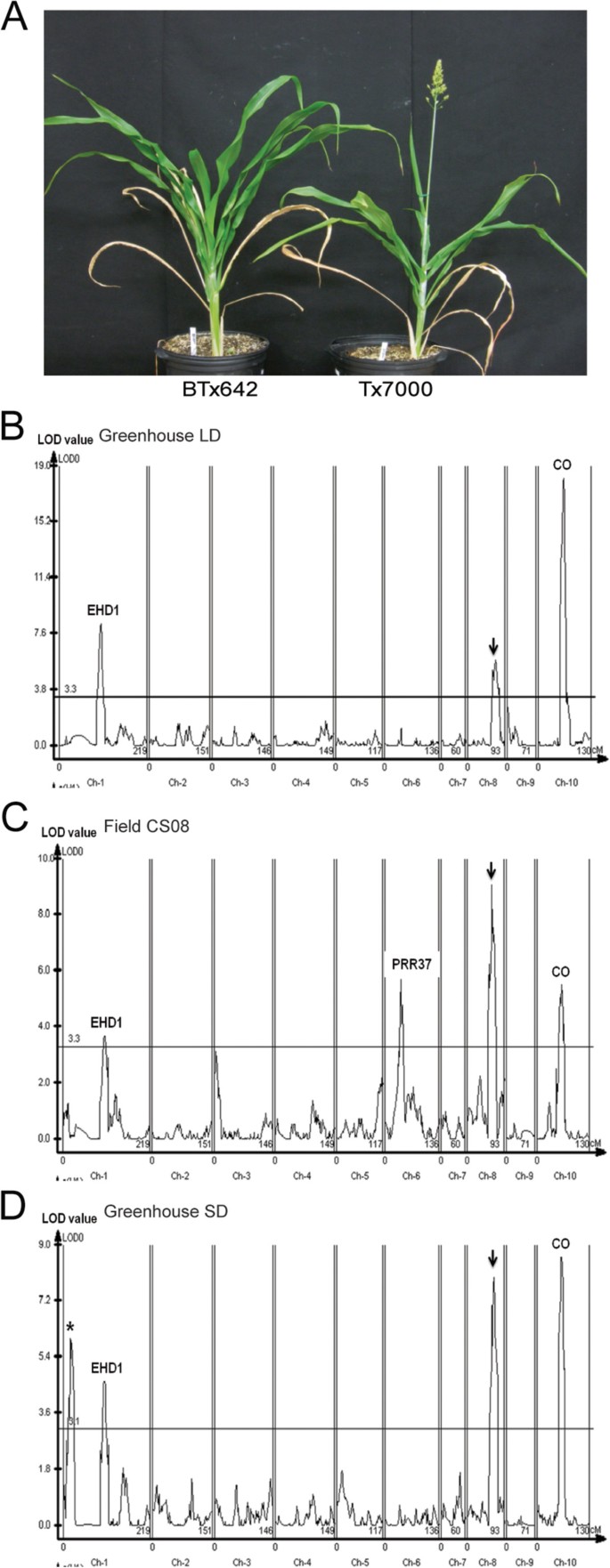
开花时间QTL在美国高粱育种项目中用作耐旱性来源的基因型BTx642和Tx7000的杂交所衍生的RIL群体中进行了定位[37].此前,从这些基因型衍生的RIL群体(n = 90)利用基于RFLP标记的遗传图谱来定位花期QTL和保持绿色耐旱性性状[38].最近对BTx642和Tx7000的基因组进行了测序,并分析了区分这些基因型的DNA多态性变异[39].利用数字基因分型创建了与该RIL群体基因组序列相匹配的高分辨率遗传图谱[39,40].数字基因分型鉴定出在RIL群体中分离的1462个SNP标记,并利用重组频率数据创建了跨越10条高粱染色体的1139 cM遗传图谱[39].开花时间QTL在该群体中进行了定位,通过对RIL群体在长14小时(LD)、短10小时(SD)和4月中旬植株出苗后昼长从12.6小时增加到7月14.3小时的田间条件下,在温室中到一半花粉脱落的天数进行表型分析。Tx7000花了73天,BTx642花了大约4天,在德克萨斯州的大学站的田间条件下。当夏季在温室中以恒定的14小时日长(LD)种植时,Tx7000在84天内开花,BTx642在19天后开花1A).当Tx7000和BTx642在冬季10 h日长(SD)条件下在温室中种植时,Tx7000在54天内开花,而BTx642在约11天后开花。
2008-2010年在田间条件下,2009年和2010年在LD温室中,2011年冬季在SD温室中,对BTx642/Tx7000 RIL种群进行了开花前数天的培养和测定。WinQTL Cartographer使用从每个地点/年收集的开花时间数据和Evans等人生成的遗传地图来识别开花时间QTL。[39].在每个环境中观察到3个花期QTL,仅在一个环境中发现2个额外的QTL(表2)1).
在LD温室条件下筛选RILs,确定了3个开花期QTL(图1B). SBI-01上的QTL (19.2 ~ 22.0 Mbp)解释了该环境下开花时间表型变异的12.3%。SbEHD1在跨越该QTL的一个LOD区间内发现了一个位于SBI-01上的草开花激活因子(Sb01g019980, 21921315-21925396)。SbEHD1根据与水稻的序列相似性,在高粱中被鉴定为花的激活因子EHD1观察到SbEHD1相对于SD的表达,与此函数一致[1].Tx7000和BTx623的SbEhd1蛋白序列之间没有氨基酸差异。BTx623是一种谷物高粱,广泛用于育种、遗传和基因组研究[40].然而,比较BTx642和Tx7000中的SbEhd1发现了两个氨基酸替换,Asp144Asn和Thr157Ile1:表S1)。Ehd1蛋白序列的差异发生在高度保守的GARP结构域OsEHD1,SbEHD1而且拟南芥响应调控因子1/2(ARR1/2).的SbEHD1BTx642等位基因(暂定为Sbehd1-2)相对于Tx7000,延迟LD和SD的开花(SbEHD1-1),与氨基酸变化的假设一致Sbehd1-2降低了该花激活因子的活性,这解释了为什么在SBI-01的这个区域检测到开花时间QTL。
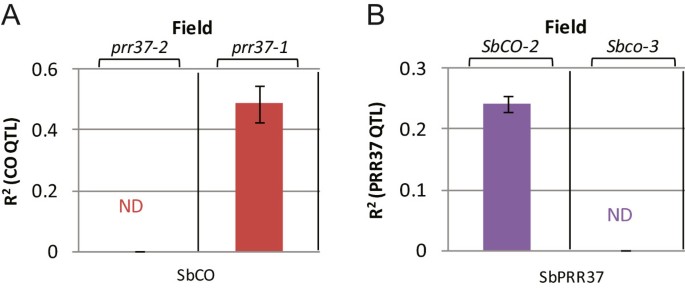
开花时间QTL位于SBI-10上(10.1-13.7 Mbp),在所有环境中均可观察到君士坦斯而且即(Sb10g010050, 12275128-12276617),分别是拟南芥和水稻花期的重要调控因子1罪犯)。QTL跨越高粱同系物君士坦斯在LD温室中解释了约40%的花期方差,而在田间或SD温室中解释了16-17%的花期方差(表2)1).花期QTL SBI-08在LD、SD和田间条件下均为48.1 ~ 50.3 Mbp。该QTL解释了田间环境中8-14%的表型变异和18-22%的表型变异。需要进一步的分析来确定与这个开花时间QTL相对应的基因。只有在SD温室中培养BTx642/Tx7000 RIL种群时,才观察到位于SBI-01末端的一个QTL (~7.2 Mbp)1D).当rl在田间种植时,在SBI-06上发现了一个QTL(约40.2 Mbp),解释了大约15%的开花时间方差(图1C)。SbPRR37 (Ma1)是LD开花的抑制因子,位于SBI-06的开花时间QTL中。序列分析显示BTx642编码Sbprr37-1和PRR37的删减版本,以及Tx7000所包含的Sbprr37-2,编码PRR37的全长版本,其中包含伪响应调控域中Lys162Asn位点的变化[1].基因型包含Sbprr37-2开花时间晚于基因型编码Sbprr37-1(null)在现场条件下,表明Sbprr37-2是活跃但弱的等位基因SbPRR37.这一结论与在杂交基因型里约热内卢和BTx623的RIL群体中发现的与PRR37相匹配的开花时间QTL的分析一致[41].序列的分析SbPRR37等位基因显示里约热内卢编码Sbprr37-2和BTx623包含Sbprr37-3,一个空等位基因[1].的Ma1来自里约热内卢(Sbprr37-2)在大学院站的田间条件下,相对于BTx623的延迟开花,其方式类似于Tx7000的相同等位基因相对于BTx642的延迟开花,而BTx642编码了空等位基因Sbprr37-1.
高粱品种的鉴定
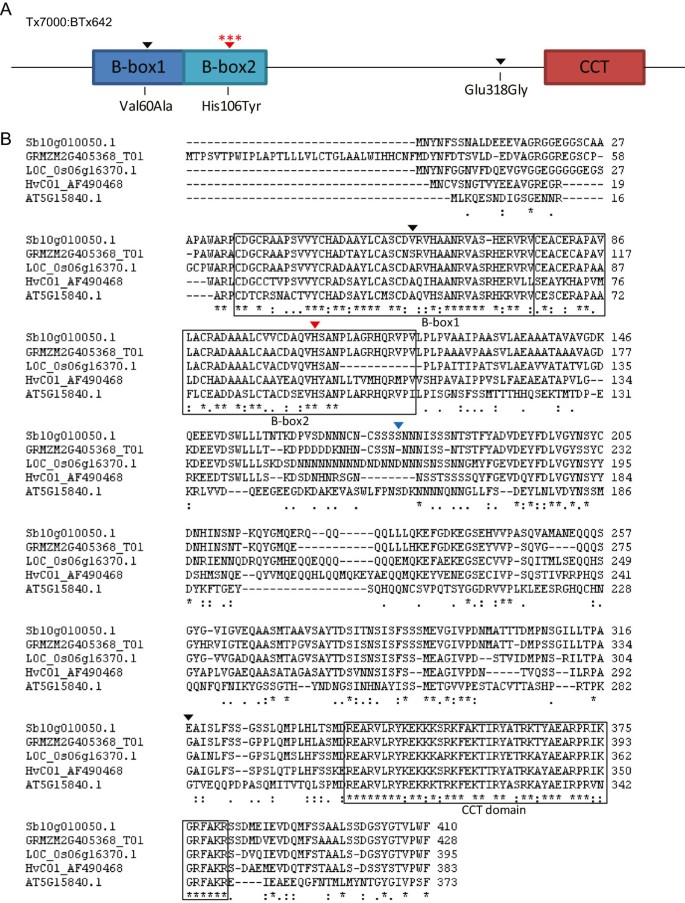
SBI-10上的开花时间QTL是由的等位基因引起的假说君士坦斯/ Hd1通过基因序列比对和共线性分析进一步研究。利用Phytozome v9.1的数据,利用水稻Hd1的氨基酸序列来鉴定高粱、玉米、大麦和拟南芥的同源性[42].Sb10g010050(评分= 71.9)、GRMZM2G405368_T01(评分= 80.7)、AF490468(评分= 63.2)和AT5G15850(评分= 40.5)与Hd1的相似性最高。GRMZM2G405368_T01和AF490468之前被鉴定为玉米君士坦斯例如基因,conz1[43)和大麦君士坦斯例如基因,HvCO1[36], AT5G15850编码有限公司在拟南芥30.].CO同源物的多重序列比对显示,Sb10g010050具有在constans样基因家族中发现的所有特征蛋白结构域(图2),包括n端B-box1(残基35-76)、B-box2(残基77-120)和c端CCT结构域(残基339-381)。
CONSTANS同系物的多重对准分析。一个.SbCO的蛋白质结构显示出constans样基因家族的结构域特征:B-box1、B-box2和CCT结构域为框状结构域。His106Tyr突变上方的红色星号表明该功能突变也在水稻和拟南芥中被发现。B.高粱(Sb10g010050, SbCO)、玉米(GRMZM2G405368_T01, conz1)、水稻(Os06g16370, OsHd1)、大麦(AF490468, HvCO1)和拟南芥(AT5G15850, AtCO) CO同源物的多重序列比对。用于比对的高粱序列来源于BTx623 (SbCO-1).所有5个物种的蛋白质残留都用星号标出。用结肠标记的氨基酸残基表明具有很强的保守性,而用句号标记的氨基酸残基表明具有较弱的相似性。一个氨基酸取代使BTx623 (SbCO-1)及Tx7000 (SbCO-2)(用蓝色箭头标记)。区分BTx623和BTx642的独特氨基酸取代(Sbco-3)分别用黑色箭头(容忍)和红色箭头(不容忍)标记。
高粱的同系物君士坦斯(Sb10g010050)位于SBI-10和水稻上即(Os06g16370)位于水稻同源染色体6上,提示这些基因可能是同源的。将这些基因的序列与每条染色体上的相邻序列进行比对,以确定是否SbCO而且OsHd1处于基因共线的区域。高粱Sb10g010050侧翼序列从Phytozome下载,与水稻6号染色体侧翼序列进行比对即使用各种[44].三个基因和即在两条染色体的100 kbp区域内排列一致,相对顺序相同,这与Sb10g010050作为水稻的同源物的鉴定一致即(附加文件2:图S1)。因此,基于序列相似性和共线性,Sb10g010050被指定为水稻的同源基因即可能与拟南芥同源有限公司,被称为“SbCO”。
SBI-10上的开花时间QTL是由不同的等位基因引起的假说SbCO通过比较BTx642和Tx7000的含量SbCO这些基因型的序列。比较发现内含子序列有1个差异,编码区有4个差异,其中3个差异导致氨基酸序列发生变化2).氨基酸Val60Ala的变化发生在B-box1中2,黑色箭头),是一种基于SIFT分析预期可容忍的氨基酸序列保守变化[45].Glu318Gly的氨基酸变化发生在b -box和cct结构域之外2,黑色箭头),并基于SIFT分析预测其耐受性。虽然蛋白质序列的Val60Ala和Glu318Gly的变化可能不会破坏CO的功能,但有可能CO的其他方面会被这些差异所修饰。位于B-box2的BTx642 CO蛋白序列中His106Tyr的变化(图2(红色箭头)被预测会破坏CO的功能。在野生型版中君士坦斯,His106是锌配合和蛋白质活性所必需的[6].的BTx642等位基因君士坦斯被指定为Sbco-3因为拟南芥的等位基因co-3具有相同的His106Try替换,破坏功能[30.].的野生型等位基因有限公司在BTx623和Tx7000中,除了在Tx7000中有一个Ser177Asn替换外,其余CO蛋白序列相同(图2B,蓝色箭头),一种不影响B盒或CCT域的修饰,并且被SIFT预测对CO函数的影响最小。在此分析的基础上君士坦斯BTx623和Tx7000的等位基因被指定为SbCO-1而且SbCO-2, BTx642中的等位基因为Sbco-3.BTx642 (Sbco-3)花晚于Tx7000 (SbCO-2),不论白昼长短。
SbCO等位基因调节开花时间通路中的基因表达
的影响SbCO对花期调控通路中其他基因表达的等位基因进行分析,进一步了解其作用机理SbCO影响开花时间。RIL105和RIL112的等位基因存在差异SbCO但在其他影响花期的主要基因座上没有。在SBI-01上的开花时间QTL中,RIL105和RIL112是BTx642等位基因的纯合子Sbehd1-2), SBI-06(跨越Sbprr37-1), SBI-08。BTx642编码的空等位基因Ma1(Sbprr37-1),一个有助于光周期敏感性的基因。Tx7000含有一个弱等位基因Ma1(Sbprr37-2),根据QTL分析,它编码了一个抑制开花的全长蛋白质[1,41].因此,选择RIL105和RIL112进行表达研究,因为它们都包含SBI-06上0-42 Mbp的BTx642 DNA,确保这些基因型为空Ma1(Sbprr37-1).此外,两个RILs都编码空等位基因Ma6(Sbghd7-1)位于SBI-06的近端[2].因此,比较RIL105和RIL112中基因表达的差异所引起的SbCO等位基因不会受到Ma1或Ma6高粱光周期敏感性的主要决定因素[1,2].
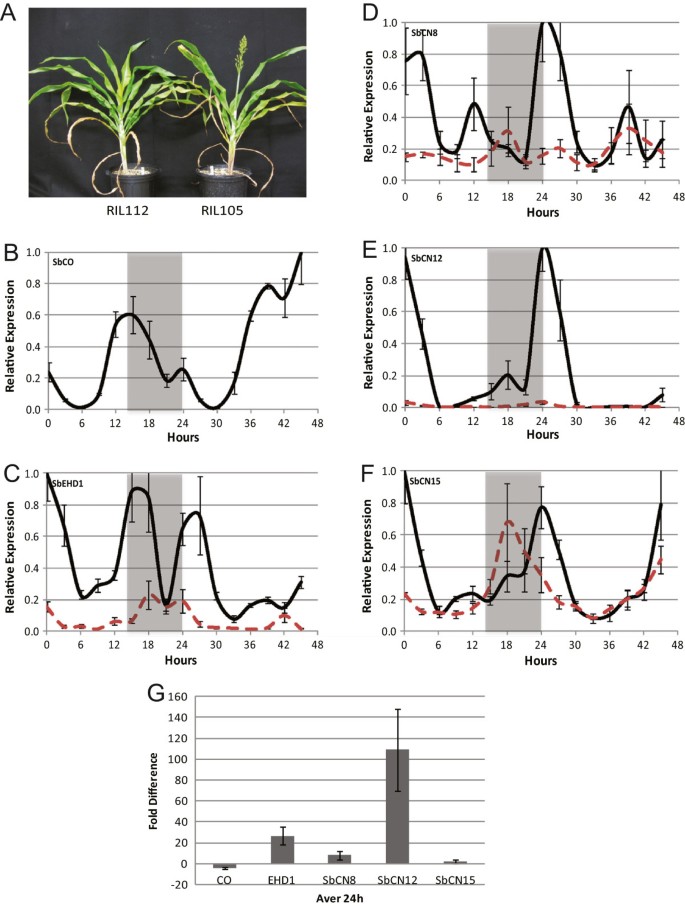
在LD温室种植时,RIL105 (SbCO-2)约75天开花,而RIL112 (Sbco-3)在大约113天内开花,这与SbCO作为开花激活因子的假设相一致(图3.一个)。SbCORIL105表达(SbCO-2)采用qRT-PCR方法进行分析,在24 h LD循环后,连续光照和温度24 h (LL)。SbCO表达在黎明下降,并在一天的大部分时间里保持在低水平,然后在傍晚增加到一个峰值,大约在黎明后15小时,随后下降和第二个较小的峰值在黎明(图3.B)SbCO以前在高粱中观察到傍晚和接近黎明的表达[1),对conz1在玉米43].的增加SbCO表达也发生在持续光(LL)下,这与之前的研究一致,表明光和生物钟调节这个峰值有限公司表达式。的模式SbCORIL112中的表达(Sbco-3)与RIL105 (SbCO-2),尽管表达水平略高(<2倍)(数据未显示)。
SbCO通过诱导促进开花SbEHD1而且英国《金融时报》样基因在LD(14小时光照/10小时黑暗)。一个.RIL112和RIL105的开花时间表型。(RIL112和RIL105的开花天数分别为113和75。)B-F.RIL105(黑色实线)和RIL112(红色虚线)开花时间基因的相对表达量。灰色阴影表示每个24小时周期的黑夜部分。前24小时包括一个明暗循环,随后24小时连续光照和温度(LL)。B.SbCO。C.SbEHD1。D.SbCN8。E.SbCN12。F.SbCN15。G.绘制出RIL105和RIL112中每个基因mRNA水平在前24小时(明暗周期)的平均折叠差异。阳性表示RIL105中检测到高表达。每个表达数据点对应三个生物重复中的三个技术重复。
RIL105 (SbCO-2)及RIL112 (Sbco-3)用来分析的等位基因是如何君士坦斯影响高粱花期调控通路中其他基因的表达。时钟基因的表达TOC1, LHY而且胃肠道在RIL105和RIL112中相似,表明这些基因不受SbCO上游基因的预期等位基因SbCO(附加文件3.:图S2)。在拟南芥中,CO通过诱导基因表达激活开花英国《金融时报》在水稻中Hd1被激活Hd3a / RFT1编码PEBP(磷脂酰乙醇胺结合)结构域蛋白“成花因子”的基因从叶片移动到茎尖分生组织,在那里它们与FD相互作用并诱导花的转变。在水稻中,PEBP基因家族的两个成员,Hd3a而且RFT1被鉴定为编码花原素[10].在玉米、ZCN8, PEBP基因家族的另一个成员,被确定为成花原的来源[46,47].在高粱,SbCN8而且SbCN12两种基因的表达都受光周期的调控,是否都是成花原的潜在来源Ma1等位基因,表达的诱导与花的起始同时发生[1].高粱与玉米的同源物ZCN8(SbCN8),ZCN12(SbCN12)和大米Hd3a(SbCN15)进行鉴定,并设计针对每个基因的qRT-PCR引物,以进行基因表达分析(附加文件4:表S2)。没有直接同源RFT1存在于高粱基因组中。
在RIL105 (SbCO-2)生长在LD,SbEHD1表达在黎明时高,然后在白天下降,在黎明后约15小时的晚上上升(图3.C,黑色实线),图案类似SbEHD1以100 M表示(Ma1)在短的日子里[1].在24小时的LD循环中,SbEHD1RIL105中RNA水平较高(SbCO-2)与RIL112 (Sbco-3)表明CO激活的表达SbEHD1(图3.C, RIL112 =红色虚线)。平均差异SbEHD1在24小时的LD周期内,两种RILs中的RNA水平是20倍(图3.G). RIL105 (SbCO-2)生长在LD,SbCN8而且SbCN12mRNA水平在黎明时最高,然后在白天下降,在黎明后约12-18小时出现第二个较小的表达峰值(图3.D和E,黑色实线)在RIL112 (Sbco-3),观察到相同的表达模式;然而,SbCN8而且SbCN12mRNA水平要低得多(图3.D和E,红色虚线)。的表达SbCN8RIL105组高约10倍(SbCO-2)与第112条相比(Sbco-3)(图3.C)和表达SbCN12RIL105基因高约100倍(SbCO-2)与RIL112 (Sbco-3)(图3.D)超过24小时的LD循环SbCO-2背景(图3.G).相比之下,SbCN15(Hd3a)在两种基因型中相似(图3.G),虽然RIL105基因的表达峰值在黎明(SbCO-2)和RIL112在18 h (Sbco-3)(图3.F).这些结果与SbCO通过诱导表达促进开花的假说一致SbEHD1,SbCN8,而且SbCN12,SbCN12显示出最大的co介导的LD表达增加。
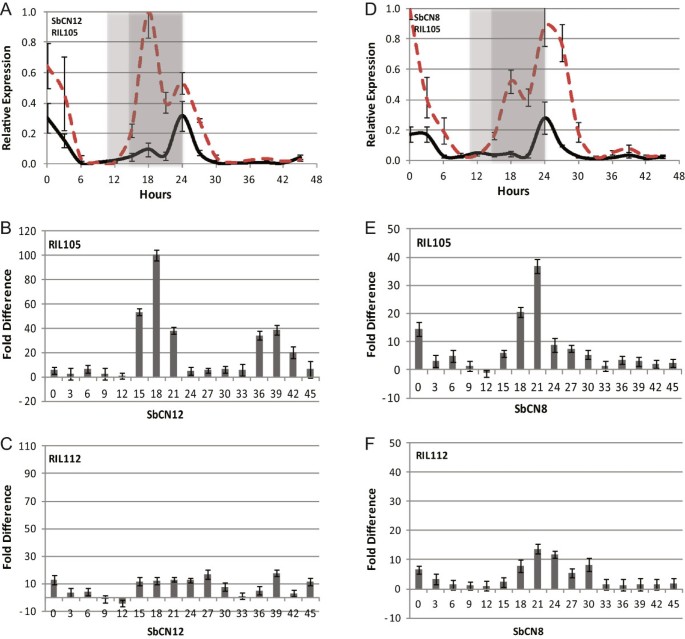
SbCO对SD和LD促花活性的调控
RIL105开花时间与开花途径基因表达的比较ma1 ma6,有限公司)及RIL112 (ma1、ma6 co-3)表明SbCO会激活SbCN8/12下一个问题是光周期是否改变了高粱SbCO的活性。在SD温室种植时,RIL105 (SbCO-2)约55天开花,而RIL112 (Sbco-3)在约72天内开花,这与SbCO作为高粱短天开花激活因子的假说相一致。的相对表达式的比较SbCO在SD和LD中表明SbCO表达对日长的响应没有显著改变(附加文件5:图S3)。然而,SbCO激活表达的相对能力的差异SbCN12而且SbCN8RIL105 (ma1 ma6,有限公司)及RIL112 (ma1、ma6 co-3)(图4).在RIL105,SbCN12而且SbCN8与LD相比,SD的表达更高,特别是在夜晚,两种基因的表达都最高(图4A和D;SD =红色虚线,LD =实线)。之间的区别SbCN12SD和LD的mRNA水平随时间的变化而变化,最大的差异发生在夜间,在18小时时达到峰值(图)4B)。相似的模式被观察到SbCN8其中SD在夜间的表达高出20-40倍,在18-21小时之间达到峰值(图4E).当比较SbCN8/12在SD/LD中表达RIL112 (Sbco-3),在夜间观察到SD和LD的表达差异约10倍(图4C和F)。总的来说,这些结果表明,与LD相比,CO在SD中有更大的活性,导致高达10倍的表达SbCN8/12在夜里,基因背景中包含无等位基因Ma1而且Ma6.
PRR37 (Ma1)对SbCO活性的转录后抑制
在高粱,Ma1(SbPRR37)通过抑制的表达增加光周期敏感性SbEHD1而且SbCN8/12,导致LD的开花延迟,但SD的影响很小[1].SbPRR37的抑制表达能力SbCN8而且SbCN12可能是由于抑制SbEHD1或SbCO,激活SbCN8而且SbCN12表达式,和/或通过SbCN8和SbCN12的直接抑制。花期QTL与之吻合Ma1在2008年、2009年和2010年野外条件下生长的BTx642/Tx7000 RIL种群中被发现(例如图1C)以及德克萨斯州的Lubbock(数据未显示)。正如预期的那样,在SD条件下没有观察到这个QTL,因为SbPRR37在这些条件下对开花的影响最小。如上所述,BTx642编码一个空等位基因Sbprr37-1然而,Ma1在Tx7000等位基因,Sbprr37-2在田间条件下,编码一个具有一个氨基酸取代的全长蛋白Lys62Asn,具有足够的活性来延迟开花时间。
如果SbPRR37通过抑制SbCO来延迟开花,SbCO会增加SbEhd1而且SbCN8/12,那么在RIL群体中可能检测到SbPRR37与SbCO的上位互作。SbPRR37而且SbCO等位基因相互作用是通过首先将RILs分类到包含Sbprr37-1(空)或Sbprr37-2,然后分析影响SbCO而且SbEHD1各背景中开花时间的等位基因。的零版本在种群中所占的部分Sbprr37-1,对应的QTL为SbCO/Sbco-3(LOD = 13)解释了田间开花时间表型变异的48%(图5A).相反,在RIL群体中含有活性等位基因的部分Ma1(Sbprr37-2),无QTL对应SbCO被观察到。在这部分的RIL人群中(Sbprr37-2),对应的QTL为SbEhd1-1/Sbehd1-2解释了约20%的表型变异(日期未显示)。这一结果表明,Sbprr37-2抑制sbco介导的开花诱导。如果这个假设是正确的,那么能力Sbprr37-2抑制开花可能依赖于一个活跃的等位基因SbCO.为了验证这一假设,RIL种群被分为包含SbCO-2和包含Sbco-3,和开花时间QTL在各背景中鉴定(图5B)这一分析表明SbPRR37等位基因影响花期SbCO-2背景,但不包含遗传背景Sbco-3表明SbPRR37抑制开花的能力依赖于SbCO。
讨论
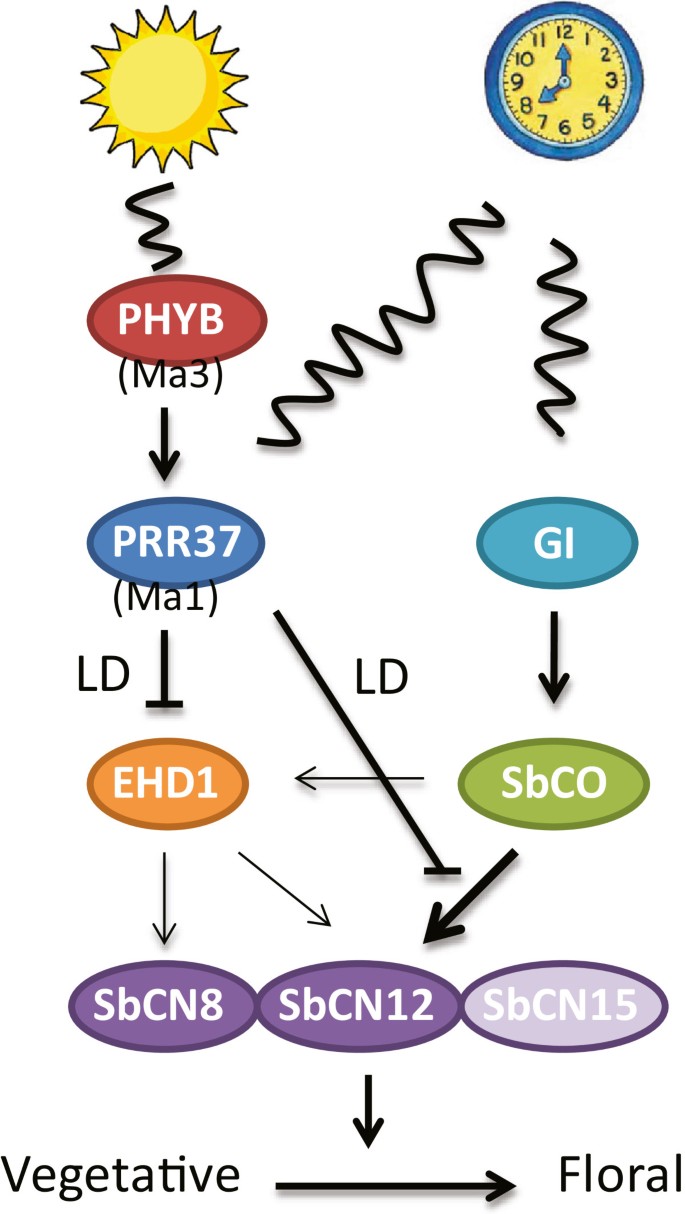
高粱品种在长时间种植时(例如,在德克萨斯州大学城的田间条件下,48d到bb0 175d)开花时间范围很广[2].这种变异很大程度上是由由编码的花阻遏子介导的光周期敏感性的差异引起的Ma1而且Ma6抑制长时间开花[1,29].关于高粱中的花激活因子,我们所知甚少。草特异性花激活剂SbEHD1之前是基于基因序列与水稻的相似性来识别的吗EHD1和激活的SbEHD1与花起始相一致的表达[2].在本研究中,我们鉴定并表征了高粱开花的第二个激活因子SbCO,是花激活剂的同系物君士坦斯在拟南芥中的同源即在大米。编码的等位基因君士坦斯通过对SBI-10上一个花期QTL的分析进行鉴定。结果表明,SbCO在高粱基因型的LD和SD中起激活作用Sbprr37-1而且Sbghd7-1。的Sbco-3BTx642中的等位基因是显著的,因为它包含His106Tyr氨基酸替代,也失活有限公司在拟南芥中的作用[30.].高粱和拟南芥基因型含有不活跃的His106Tyrco-3在高粱中,等位基因开花时间在白天较晚,开花时间在白天较晚,表明CONSTANS在两种高粱中都是开花的激活因子。SbCO与TOC1、PRR37、Ghd7和血红素激活蛋白(HAP或NF-Y蛋白)共享一个保守的CCT (CO, CO-like, TOC1)结构域。酵母双杂交筛选显示,CO可以通过其cct结构域与HAP3和HAP5亚基相互作用,形成与ccaat结合的cbf复合体,与FT启动子结合并激活转录[48,49].在高粱中,SbCO被发现激活转录SbEHD1,SbCN8而且SbCN12,与其作为开花激活剂的作用一致,可能是通过cbf复合物的形成,但也可能是通过直接与DNA结合[50].
的能力SbCO在含有无等位基因的RIL遗传背景中检测诱导开花途径基因表达和开花的等位基因Ma1(Sbprr37-1),Ma6(Sbghd7)以消除这些LD花抑制因子的影响。在这种缺失遗传背景下,SbCO促进了LD和SD的早开花,增加了表达SbEHD1(~ 25倍),SbCN8(~ 10倍),SbCN12(~ 100倍)SbCN15(~5倍)相对于携带非活性基因的基因表达Sbco-3等位基因。这些信息在图中显示的开花时间调节模型中得到了总结6.该模型包括pebp基因家族的三个成员,它们可能是高粱中致花原的来源,SbCN8,SbCN12而且SbCN15。SbCN8是玉米的同类吗ZCN8的基因表达模式与已证实的作用相一致ZCN8作为玉米发花素的来源[47].SbCN12PRR37抑制其表达,SD诱导其在叶片中表达SbCO(本研究),表明该基因也是高粱成花原的一个可能来源。在大米、Hd3a而且RFT1已确定为发花原的来源;因此表达SbCN15,直接同源的Hd3a,进行了分析。SbCN15显示SbPRR37和SbCO的基因表达响应光周期和突变的相对较小的变化。高粱基因组不编码与之对应的RFT1,是劳工处水稻中的花素来源。
结果表明,在补语方面发生了显著的变化英国《金融时报》类基因是高粱发花素的主要来源(SbCN8,SbCN12)和大米(Hd3a=SbCN15;RFT1因此,开花时间的调节也可能不同,即使这两种草都是短日照植物。SbCO激活的表达SbCN8而且SbCN12,虽然SbCN12被诱导的程度明显更大。SbCO也增加了SbEHD1的激活剂Hd3a表达大米。SbEHD1在LD中生长的光敏高粱转移到SD时,SbPRR37和SbGhd7抑制了其表达,并诱导其表达[1,2].增加SbEHD1表达的发生与基因的增加是平行的SbCN8而且SbCN12说明SbEhd1可以诱导这些基因的表达,如图所示6.然而,SbEhd1的这种活性的程度和特异性还需要在SbCO对这些基因表达影响最小的背景下进一步分析(Sbco-3背景)。本研究结果表明,SbCO增加了SbEHD1推测SbEhd1可以激活SbCN8而且SbCN12和开花。相比之下,水稻Hd1没有增加表达EHD1[20.,33].有趣的是,发现SbCO激活了SbCN12在很大程度上比SbEHD1。因此我们得出结论,SbCO直接增加SbCN12这可能是SbCO激活开花最重要的方式。
本研究发现SbCO能激活LD和SD基因型中缺失的高粱开花Ma1而且Ma6提出了这样一个问题:在这种短日照植物中,这种基因的活性是如何受光周期的调节的。SbCO在白天表达量低,然后在傍晚增加,在黎明后约15小时达到峰值,随后下降,在黎明出现第二个峰值(图3.B).在拟南芥中,类似的增加有限公司由于GI和蓝光激活的FKF1的相互作用,导致抑制的cdf因子的降解有限公司表达式[9].这一机制也可以解释晚峰SbCO在高粱表达式。的第二个不太突出的峰SbCO黎明的表达是由等位基因调节的SbPRR37并在LD中增强[1].函数的峰值SbCO尽管在这个时候产生SbCO有助于激活,但目前还不清楚黎明时的表达SbEHD1早上的表情。
功能等位基因SbCO增加了SbCN8而且SbCN12与LD相比,SD的影响更大(图4).SbCO的表达水平在晚上增加,这有助于解释其中的原因SbCN8/12表情在晚上增加。自的表达SbCO没有被光周期显著改变(附加文件5图S3), SD中SbCO活性的增加最有可能是由于蛋白质水平或活性的增加。在长日照植物拟南芥中,由于蛋白质COP1-SPA1-Cry2的稳定,LD中的CO水平较高[6].这种稳定模块在高粱中可能缺失或减弱。相对于LD,减少PhyB/ c介导的SD中SbCO的降解可能导致SbCO介导的SD中SbCN8/12的更大激活。在高粱基因型中含有活性等位基因SbPRR37的晚高峰SbCO表达量没有改变,表达峰值在黎明时增加,但SbCO活性强烈减弱。的表达SbPRR37在LD生长的植物中,夜间的含量较高,而在SD生长的植物中则较低。因此,更高的水平SbPRR37在这些条件下,SbPRR37对SbCO活性的抑制,可以阻止SbCO在LD中激活开花。
SbPRR37是一种cct结构域蛋白,已被证明与HAP3相互作用,与SbCO相互作用的cbf亚基相同[51].因此,SbPRR37可能是SbCO与HAP复合物结合的竞争性抑制剂。SbPRR37也可能以类似于TOC1和其他prr蛋白的方式直接与DNA结合[52].TOC1与LHY/CCA1启动子中的同源基序结合是由其cct结构域介导的,导致prr结构域介导的转录抑制。如果PRR37绑定到SbCN12它可以直接抑制转录,阻断SbCO与HAP复合体的结合,并/或与CO或其他蛋白相互作用从而抑制SbCN12转录。最近的研究结果表明,PRR37在水稻Hd2中具有直接的抑制作用Hd3a转录(35].需要进一步的遗传和生化分析来区分这些可能性。
在大麦这种长日照植物中,HvCO1在LD中激活开花,而激活依赖于Ppd-H1, SbPRR37的同源物。过度的HvCO1但光周期敏感介导的Ppd-H1仍在此背景下观察到[36].Ppd-H1不直接影响HvCO1但增强了HvCO1的激活能力HvFT1有趣的是,Ppd-H1在LD中增加HvCO1的活性,而SbPRR37在LD中抑制SbCO的促花活性,SbPRR37和Ppd-H1在LD中的表达和活性增加,都影响CO调节ft -基因表达的能力,但方式相反,这与大麦是长日植物,高粱是短日植物一致。PRR37活性的差异可能是由于PRR37直接结合到的启动子的差异SbCN12而且HvFT1(同族体Hd3a,SbCN15),或通过PRR37/Ppd-H1与激活子、阻遏子、HAP亚基、HvCO1和SbCO的相互作用间接实现。在水稻中,有研究表明,PRR37被Hd6磷酸化可能导致PRR37与Hd1一起成为一种抑制因子Hd3aLD的表达[35].PRR37可与CO形成共抑制复合物的可能性与高粱和水稻的结果一致。然而,在缺乏PRR37的情况下,CO作为一种激活剂英国《金融时报》在高粱和水稻中的表达。虽然PRR37活性变化的生化基础还有待阐明,但综合来看,结果表明,PRR37和CO在特定成花素相关pebp基因启动子上的相互作用导致了LD和SD草在光周期敏感开花方面的根本差异。
图中开花时间模型6表明SbEhd1和SbCO可通过激活独立诱导开花SbCN8和/或SbCN12。EHD1和Hd1已被证明独立激活Hd3a (FT)和水稻开花[20.].在高粱中,我们发现这些通路之间存在交叉信号,因为SbPRR37激活了SbCO而SbCO在LD中有诱导作用SbEHD1在SD表达式。水稻EHD1是一种b型响应调控因子,由多个上游调控因子控制,包括抑制因子GHD7、粒数、株高和抽穗期8 (GHD8)、OsLEC1和FUSCA-LIKE1 (OsLFL1)、OsMADS56和激活因子GI、早抽穗期2 (EHD2)和OsMADS50 [8,53].两条平行途径的存在可以激活高粱开花,提供了对不同环境因素的广泛响应,有助于高粱广泛的地理适应。高粱作物育种家利用这些平行途径中关键基因的不同等位基因,培育出早开花的谷物高粱杂交种和晚开花的能量高粱杂交种。
结论
等位基因的高粱君士坦斯(SbCO)通过分析从BTx642和Tx7000衍生的RIL群体中发现的10号染色体上的花期QTL进行鉴定。遗传分析和基因表达研究表明,SbCO在缺乏活性SbPRR37和GHD7等位基因的基因型中是长日开花和短日开花的激活因子。PRR37被发现在长时间内阻断co介导的花的激活。
方法
植物材料
BTx642/Tx7000 RIL群体(n = 90)和亲本系于2008年、2009年和2010年在德克萨斯州大学城(College Station)附近的德州农工大学研究农场(Texas A&M Research Farm)采用重复随机区组设计在田间条件下种植,种植时间为4月1日至14日。花中期(花粉脱落)的天数被确定为开花时间的测量。田间日长由4月的~12.6 h增加到7月的14.3 h,日平均最高气温为31.7℃,日平均最低气温为20.0℃。每个RIL和亲本系的10株植株在温室中生长10 h日长(SD, 2011)或14 h日长(LD, 2009和2010),花期表型与田间生长的种群相似。RIL105和RIL112在原BTx642/Tx7000 RIL群体中对应4_6和12_14 [38].
基因分型测序和QTL分析
利用数字基因分型(DG)进行测序基因分型[40]研究从BTx642和Tx7000衍生的90个RILs [39].利用Mapmaker/EXP ver构建1462个多态DG标记的遗传连锁图谱。3.0b,其中使用Kosambi映射函数计算重组频率。在WinQTL Cartographer v2.5中使用复合区间映射(CIM)检测qtl [54].基于α = 0.05时1000次重复的实验特异性排列确定QTL检测的显著LOD阈值[55].在基于qtl的上位性分析中,根据的等位基因将90个RILs划分为亚群体SbPRR37或等位基因SbCO分别。的每个等位基因纯合的亚居群SbPRR37的每个等位基因SbCO进行QTL分析。
系统发育和共线性分析
利用水稻Hd1(Os06g16370)的氨基酸序列搜索Phytozome v9.1 [42]用于水稻、玉米、大麦和高粱中CONSTANS的同源物。使用ClustalW2进行多序列比对、比对评分和系统分析[56]使用Sb10g010050(高粱CO)、GRMZM2G405368_T01(玉米conz1)、Os06g16370(水稻Hd1)、AF490468(大麦HvCO1)和AT5G15850(拟南芥CO)的蛋白质序列。水稻和高粱基因组序列(Phytozome v9.1, 100 kbp)跨越同系物有限公司用于共线分析。共线性由GEvo确定[44],一个来自CoGe(加速比较基因组学)工具包的基因组区高分辨率序列分析工具[57].进行了类似的系统发育/共线性分析EHD1.
等位基因特征
通过比较BTx623、BTx642和Tx7000的DNA序列,确定候选基因中的SNPs。的BTx623Sbi1装配和Sbi1.4基因注释作为参考基因组序列(Phytozome)。用于分析的BTx642和Tx7000基因组序列组合先前已获得[39].使用CLC Genomics Workbench 4.9的SNP Detection功能调用SNP。变量调用的最小覆盖率设置为5,最大覆盖率设置为150。等位基因类型是根据snp来指定的。SIFT算法(从容差中筛选出不容差)[45]被用来预测氨基酸取代是否影响蛋白质功能,这是基于来自密切相关基因序列的序列比对中氨基酸残基的保守程度。从BTx642/Tx7000 RIL群体中选择RIL105和RIL112,利用每个QTL峰值两侧的DG标记,跨越每个候选基因。RIL112包含横跨所有开花时间的BTx642单倍型QTL,包括SBI-10上的QTL (Sbco-3).RIL105包含跨越SBI-01、SBI-06、SBI-08上QTL的BTx642单倍型和跨越SBI-10上QTL的Tx7000单倍型(SbCO-2)(额外的文件6:表S3)。
LD、SD和昼夜节律实验
在昼夜节律试验中,RIL105和RIL112在LD (14 h光照)温室中生长32 d。对于夹带,在收集组织进行表达分析之前,将植物转移到设置为LD (14 h光照/10 h暗)或SD (10 h光照/14 h暗)处理一周的生长室中。生长室内白天温度为30℃,光照强度为~300 μmol·s−1·米−2夜间(暗)温度23℃,相对湿度约60%。在第39天,从三种不同植物的不同基因型和光周期中,每3小时采集顶部三片叶片的充分展开部分,经过24小时的光-暗循环,然后24小时的连续光(连续30°C) (LL)。采用TRI试剂(MRC)从叶片组织中提取RNA,提取方法为多糖含量高的组织。使用RNeasy Mini Kits (QIAGEN)进行RNA清理,包括通过列上DNA酶I消化去除DNA。在1% MOPS凝胶上检测RNA完整性。第一链cDNA合成使用SuperScript®III第一链合成系统(Invitrogen)与寡聚dT和随机六聚体引物混合。合成第一链cDNA后,将反应稀释至初始总RNA的最终浓度为10 ng/μl。基因特异性qRT-PCR使用Power SYBR Green PCR Master Mix (Applied Biosystems)进行。选择18S rRNA作为内对照,使用TaqMan通用PCR Master Mix (Applied Biosystems)、rRNA探针(VIC®探针)和rRNA正向/反向引物进行检测。所有反应均在7900HT PCR系统上进行,使用SDS v2.3软件(Applied Biosystems)。 The specificity of each qRT-PCR primer set was validated using melting temperature curve analysis. Amplification efficiency of each primer set was determined by the series dilution method [58],它可以通过由每个Ct值和稀释因子组成的曲线的斜率来计算(附加文件7:表S4)。使用比较周期阈值(ΔΔCt)方法确定相对表达量,并使用RNA水平最高的样本进行校准。采用引物效率调整数据,按照效率修正法进行相对定量[59].每个表达数据点来自三个生物重复中的三个技术重复的分析。
支持数据的可用性
所有支持数据都包含在附加文件中。用于分析的BTx642和Tx7000全基因组序列集合此前发表在Evans J, McCormick RF, Morishige D, Olson SN, Weers B, et al.(2013)高粱基因组DNA多态性密度和分布的广泛变异。科学通报8(11):e79192。doi: 10.1371 / journal.pone.0079192。基因组序列保存在NCBI SRA中,标识如下:BioProjectPRJNA189453, Accession SRP019171 (http://www.ncbi.nlm.nih.gov/sra/?term=PRJNA189453);SAMN01942195包含BTx642的序列信息,SAMN01942194包含Tx7000的序列信息。
参考文献
Murphy RL、Klein RR、Morishige DT、Brady JA、Rooney WL、Miller FR、Dugas DV、Klein PE、Mullet JE:伪反应调节蛋白37 (PRR37)同步光和时钟调控控制高粱光周期开花美国国家科学研究院。2011, 38(4): 359 - 359。10.1073 / pnas.1106212108。
Murphy RL、Morishige DT、Brady JA、Rooney WL、Yang S、Klein PE、Mullet JE: Ghd7 (Ma6)抑制长日开花:能量高粱杂交种的一个关键性状。植物基因组学报,2014,doi:10.3835/plantgenome2013.11.0040
Rooney WL, Blumenthal J, Bean B, Mullet JE:设计高粱作为专用的生物能源原料。生物燃料生物制品生物炼制。2007,1(2):147-157。10.1002 / bbb.15。
Olson SN, Ritter K, Rooney W, Kemanian A, McCarl BA, Zhang Y, Hall S, Packer D, Mullet J:高生物量产能高粱:C4草类生物能作物遗传模型的建立。生物燃料与生物炼制。2012,6(6):640-655。
Morgan PW, Finlayson SA:成熟和身高的生理和遗传学。高粱:起源、历史、技术和生产。编辑:Smith CW, Frederiksen RA。2000,纽约:作物科学的威利级数,240-242。
康斯坦斯和开花光周期时间的进化起源。实验机械学报,2011,62(8):2453-2463。10.1093 / jxb / erq449。
Tsuji H, Taoka K, Shimamoto K:水稻中的成花素:成花素转录、激活复合体和多重功能的复杂基因网络。植物生物学杂志,2013,16(2):228-235。10.1016 / j.pbi.2013.01.005。
长日条件下水稻成花原基因表达和花过渡的临界日长识别的符合性。植物学报,2013,6(3):635-649。10.1093 / mp / sst022。
Turck F、Fornara F、Coupland G:成花原的调控与鉴定:开花位点T转移到中心阶段。植物学报,2008,29(5):573-594。10.1146 / annurev.arplant.59.032607.092755。
Tsuji H, Taoka K, Shimamoto K:水稻开花调控:两个成花原基因,一个复杂的基因网络和自然变异。植物学报,2011,14(1):45-52。10.1016 / j.pbi.2010.08.016。
科拉桑提,科涅娃V:草类花诱导机制:有借来的,有新的。植物生理学报,2009,29(1):369 - 369。10.1104 / pp.108.130500。
Greenup A, Peacock WJ, Dennis ES, Trevaskis B:拟南芥和谷类植物季节性开花响应的分子生物学研究。中国生物工程学报,2009,32(4):366 - 366。10.1093 / aob / mcp063。
植物对光周期的响应。植物生态学报,2009,32(3):517-531。10.1111 / j.1469-8137.2008.02681.x。
安德烈斯F,库普兰G:开花对季节线索反应的遗传基础。科学通报,2012,13(9):627-639。10.1038 / nrg3291。
Imaizumi T, Kay SA:开花的光周期控制:不仅仅是巧合。植物动态,2006,11(11):550-558。10.1016 / j.tplants.2006.09.004。
Coles ND, McMullen MD, balintt - kurti PJ, Pratt RC, Holland JB:联合多群体分析揭示玉米光周期敏感性的遗传控制。中国生物医学工程学报,2010,38(3):369 - 371。10.1534 / genetics.109.110304。
Buckler ES、Holland JB、Bradbury PJ、Acharya CB、Brown PJ、Browne C、Ersoz E、Flint-Garcia S、Garcia A、Glaubitz JC、Goodman MM、Harjes C、Guill K、Kroon DE、Larsson S、Lepak NK、Li H、Mitchell SE、Pressoir G、Peiffer JA、Rosas MO、Rocheford TR、Romay MC、Romero S、Salvo S、Sanchez Villeda H、da Silva HS、Sun Q、Tian F、Upadyayula N等:玉米花期的遗传结构科学通报,2009,29(5):514 - 514。10.1126 / science.1174276。
Chardon F, Virlon B, Moreau L, Falque M, Joets J, Decousset L, Murigneux A, Charcosset A:玉米花期遗传结构的定量性状基因座元分析及与水稻基因组的同源性保守中国生物医学工程学报,2004,38(4):369 - 369。10.1534 / genetics.104.032375。
Yano M、Katayose Y、Ashikari M、Yamanouchi U、Monna L、Fuse T、Baba T、Yamamoto K、Umehara Y、Nagamura Y、Sasaki T: Hd1是水稻光周期敏感性的主要数量性状位点,与拟南芥花期基因CONSTANS密切相关。植物生态学报,2000,12(12):2473-2483。10.1105 / tpc.12.12.2473。
Doi K, Izawa T, Fuse T, Yamanouchi U, Kubo T, Shimatani Z, Yano M, Yoshimura A: Ehd1是一种水稻b型响应调控因子,能在短时间内促进开花,独立于Hd1控制ft样基因表达。基因学报,2004,18(8):926-936。10.1101 / gad.1189604。
赵翁雪W,邢Y, X, Y,唐W,王L,周H, S,徐C,李X,张问:自然变异Ghd7基因是一个重要的监管机构的标题日期和在水稻产量潜力。植物学报,2008,40(6):761-767。10.1038 / ng.143。
Yan L, Loukoianov A, Blechl A, Tranquilli G, Ramakrishna W, SanMiguel P, Bennetzen JL, Echenique V, Dubcovsky J:小麦VRN2基因是受春化作用下调的开花抑制因子。科学通报,2004,32(4):339 - 339。10.1126 / science.1094305。
伊藤H, Nonoue Y, Yano M, Izawa T:一对花调节剂设定了水稻Hd3a成花原表达的临界日长。植物学报,2010,42(7):635-638。10.1038 / ng.606。
Mace ES, Hunt CH, Jordan DR:超模:高粱和玉米提供了对开花时间遗传学的相互洞见。中国生物工程学报,2013,29(5):369 - 369。10.1007 / s00122 - 013 - 2059 - z。
昆比:高粱改良和生长遗传学。大学站:德州农工大学出版社1974。
杨文华,杨文华,杨文华,杨文华。高粱光敏色素基因Ma3的克隆与表达分析。植物生理学报,2004,22(2):366 - 366。10.1104 / pp.113.2.611。
Izawa T, Oikawa T, Sugiyama N, Tanisaka T, Yano M, Shimamoto K:光敏色素介导外光信号抑制水稻光周期开花中的FT同源分子。基因学报,2002,16(15):2006-2020。10.1101 / gad.999202。
杨文华,张文华,张文华,张文华,等:大麦光敏色素系的光敏感性研究进展[j] .植物生理学报,2001,22(3):366 - 366。10.1104 / pp.119.3.1033。
杨志强,王文华,李文华,等:高粱光敏反应的遗传调控Moench。作物科学,1999,39(2):397-400。10.2135 / cropsci1999.0011183X0039000200016x。
Robson F, Costa MM, Hepworth SR, Vizir I, Pineiro M, Reeves PH, Putterill J, Coupland G:花期基因CONSTANS保守结构域的功能重要性(突变等位基因和转基因植株分析)。植物学报,2001,28(6):619-631。
Griffiths S, Dunford RP, Coupland G, Laurie DA:大麦、水稻和拟南芥中constans样基因家族的进化。植物生理学报,2003,31(4):369 - 369。10.1104 / pp.102.016188。
Ballerini ES, Kramer EM:从进化的角度:重新评价CO-FT调控的保守性及其在开花时间光周期调控中的作用。植物科学进展,2011,2:81-
高桥Y, Teshima KM, Yokoi S, Innan H, Shimamoto K: Hd1蛋白、Hd3a启动子和Ehd1表达水平的变化影响了栽培水稻开花时间的多样性。美国国家科学研究院。科学通报,2009,36(6):559 - 561。10.1073 / pnas.0812092106。
石川R,青木M,黑谷K,横井S,信村T,高野M,岛本K:光敏色素B调节抽穗期1 (Hd1)介导的水稻成花素Hd3a的表达和水稻临界日长。中国生物医学工程学报,2011,29(6):563 - 566。10.1007 / s00438 - 011 - 0621 - 4。
Koo BH, Yoo SC, Park JW, Kwon CT, Lee BD, An G, Zhang Z, Li J, Li Z, Paek NC: OsPRR37基因的自然变异调节了抽穗期,并对宽纬度地区水稻种植有贡献。植物学报,2013,6(6):1877-1888。10.1093 / mp / sst088。
Campoli C, Drosse B, Searle I, Coupland G, von Korff M:与大麦(Hordeum vulgare)开花时间同源的HvCO1的功能特征。植物学报,2012,29(5):366 - 366。10.1111 / j.1365 - 313 x.2011.04839.x。
Rosenow DT, Clark LE:优质高粱作物的抗旱和抗倒伏能力。第50届玉米和高粱产业研究年会论文集:1995年12月6日至7日,芝加哥,伊利诺斯州:美国种子贸易协会,
徐伟,Subudhi PK, Crasta OR, Rosenow DT, Mullet JE, Nguyen HT:籽粒高粱(sorghum bicolor L. Moench)保持绿色qtl的分子定位。中国生物医学工程学报,2000,43(3):461-469。10.1139 / gen - 43 - 3 - 461。
Evans J, McCormick RF, Morishige D, Olson SN, Weers B, Hilley J, Klein P, Rooney W, Mullet J:高粱基因组DNA多态性密度和分布的广泛变异。科学通报,2013,8 (11):e79192-10.1371/journal.pone.0079192。
Morishige DT, Klein PE, Hilley JL, sahraian SM, Sharma A, Mullet JE:高粱的数字基因分型——高粱是一种具有大量重复序列丰富基因组的多样化植物物种。中国生物医学工程学报,2013,31(1):448-10.1186/1471-2164-14-448。
Murray SC, Sharma A, Rooney WL, Klein PE, Mullet JE, Mitchell SE, Kresovich S:高粱作为生物燃料原料的遗传改良:I.蔗糖和谷物非结构性碳水化合物的QTL。作物科学,2008,48(6):2165-2179。10.2135 / cropsci2008.01.0016。
Phytozome v9.1。[http://www.phytozome.net/]
Miller TA, Muslin EH, Dorweiler JE:玉米constans样基因conz1在不同光周期下表现出不同的日表达模式。植物生态学报,2008,27(6):1377-1388。10.1007 / s00425 - 008 - 0709 - 1。
基因组进化分析。[http://genomevolution.org/CoGe/GEvo.pl]
Kumar P, Henikoff S, Ng PC:使用SIFT算法预测编码非同义变体对蛋白质功能的影响。通信学报,2009,4(7):1073-1081。
Danilevskaya ON,孟X, Hou Z, Ananiev EV, Simmons CR:玉米PEBP基因家族扩展的基因组和表达谱。植物生理学报,2008,26(1):441 - 441。
fft -like ZCN8基因在玉米中是一种花激活因子,与光周期敏感性有关。植物细胞学报,2011,23(3):942-960。10.1105 / tpc.110.081406。
Wenkel S、Turck F、Singer K、Gissot L、Le Gourrierec J、Samach A、Coupland G: CONSTANS与CCAAT box结合复合体共享一个重要功能域,相互作用调节拟南芥开花。植物学报,2006,18(11):2971-2984。10.1105 / tpc.106.043299。
Ben-Naim O, Eshed R, Parnis A, Teper-Bamnolker P, Shalit A, Coupland G, Samach A, Lifschitz E: CCAAT结合因子可以介导constans样蛋白和DNA之间的相互作用。植物学报,2006,26(3):366 - 366。10.1111 / j.1365 - 313 x.2006.02706.x。
Tiwari SB, Shen Y, Chang HC, Hou Y, Harris A, Ma SF, McPartland M, Hymus GJ, Adam L, Marion C, Belachew A, Repetti PP, Reuber TL, Ratcliffe OJ:开花时间调节剂CONSTANS通过一个独特的顺式元素被引入到开花位点T启动子中。植物生态学报,2010,32(1):447 - 447。10.1111 / j.1469-8137.2010.03251.x。
李超,狄斯菲尔德,科米斯,杜bcovsky J:小麦开花抑制因子VRN2和启动子CO2与核因子y配合物的相互作用。植物生态学报,2011,37(5):369 - 369。10.1111 / j.1365 - 313 x.2011.04630.x。
Gendron JM, Pruneda-Paz JL, Doherty CJ, Gross AM, Kang SE, Kay SA:拟南芥生物钟蛋白TOC1是一种dna结合转录因子。中国生物医学工程学报,2012,29(4):366 - 366。10.1073 / pnas.1200355109。
宾利AR, Jensen EF, Mackay IJ, Hönicka H, Fladung M, Hori K,矢野M, Mullet JE, Armstead IP, Hayes C, Thorogood D, Lovatt A, Morris R, Pullen N, Mutasa-Göttgen E, Cockram J:开花时间。气候适应性作物的基因组学与育种。编辑:科尔C. 2013,柏林出版社:施普林格,2:1-66。
王硕,Basten CJ,曾志斌:Windows QTL制图师2.5。2012年,北卡罗来纳州罗利:北卡罗莱纳州立大学
Churchill GA, Doerge RW:定量性状映射的经验阈值。中国生物医学工程学报,1994,38(3):369 - 371。10.1186 / 1478-1336-1-1。
ClustalW2。[http://www.ebi.ac.uk/Tools/msa/clustalw2/]
加速比较基因组学。[http://genomevolution.org/CoGe/]
Bookout AL, Mangelsdorf DJ:用于核受体信号通路分析的定量实时PCR协议。核接收信号。2003,1:1-7。
Pfaffl MW:实时RT-PCR中相对定量的新数学模型。核酸学报,2001,29(9):2002-2007。
确认
作者感谢William Rooney在大学院站的田间地块中增加BTx642*Tx7000 RIL种群。这项研究的资金由农业生物学(JEM)的Perry Adkisson主席、Ceres公司和Pioneer Hi-Bred国际公司提供。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者声明他们没有相互竞争的利益。
作者的贡献
SY参与了实验的设计,数据的收集和分析,主要负责图形的构建和论文的撰写。JM构思并获得了研究资金,帮助设计和解释实验结果,并编辑了文本。BW与SY合作在多个地点对RIL群体的开花时间进行表型分析,并进行QTL分析。DM有助于识别CO等位基因和其他开花时间基因等位基因的性质,并有助于实验设计、用于分析开花基因表达的组织收集和文本编辑。所有作者阅读并批准了最终稿件。
电子辅料
12870 _2014_1543_moesm2_esm.pdf
附加文件2:图S1:水稻Hd1和高粱CONSTANS的共线性。参考基因组序列包括高粱SbCO(上图)和米饭OsHd1(下面板)对对齐的序列进行分析(红框)。对齐区域内的共线基因由红线连接。a-d代表水稻和高粱中的4个共线基因(Sb10g010020- Sb10g010050),包括SbCO(Sb10g010050, d). (PDF 406kb)
12870 _2014_1543_moesm3_esm.pdf
附加文件3:图S2:在LD(14小时光照/10小时黑暗)或SD(10小时光照/14小时黑暗)条件下RIL105(黑色实线)和RIL112(红色虚线)中生物钟基因和GI的表达水平(ΔCt)。灰色阴影区域代表黑暗时期。第一个24小时包括一个明暗循环,然后是24小时的连续光照。答:TOC1。LHY。胃肠道。每个表达数据点对应三个生物重复中的三个技术重复。(PDF 468 KB)
12870 _2014_1543_moesm5_esm.pdf
附加文件5:图S3:相对表达水平SbCO在LD (14 h光照/10 h暗)或SD (10 h光照/14 h暗)条件下生长。黑色实线表示LD的相对表达,红色虚线表示SD的相对表达,然后是LL的24小时。(PDF 317 KB)
权利和权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。
本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。
欲查看此许可证的副本,请访问https://creativecommons.org/licenses/by/4.0/.
创作共用公共领域奉献放弃书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
杨,S,韦尔斯,b.d.,莫里什奇,D.T.et al。君士坦斯是一种光周期调节的高粱开花激活剂。BMC植物杂志14,148(2014)。https://doi.org/10.1186/1471-2229-14-148
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-14-148
关键字
- 光周期
- 高粱
- 开花的时间
- QTL
- 君士坦斯
- PRR37