摘要
背景
栽培水稻由两种重要的生态型组成,旱稻和灌溉稻,它们分别适应旱地或灌溉栽培。旱稻因其需水量少而在旱作地区广泛采用,它含有大量未开发的遗传资源,如干旱适应基因。随着水资源短缺的加剧和人口的增长,选育抗旱作物品种的需求日益迫切。然而,过去在旱稻研究中的一个疏忽,对旱稻适应的遗传机制知之甚少,这极大地阻碍了利用其遗传资源进行育种和栽培的进展。
结果
在本研究中,我们选择了来自世界各地的84个旱地和82个灌溉生态类型,分别在灌溉和旱地环境下进行表型分析,利用全基因组变异数据研究了旱地生态类型的系统发育关系和种群结构。进一步的比较分析得出了一组可能解释旱稻和灌溉水稻之间表型和生理差异的分化基因。
结论
该研究首次对旱稻大样本基因组进行了研究,为了解旱稻的适应能力,特别是干旱相关的适应能力及其在现代农业中的应用提供了有价值的基因列表。
背景
亚洲栽培水稻(栽培稻籼型和栽培稻.型粳稻)具有表型上的分化生态型[1].灌溉水稻和旱作水稻分别适合在水田和旱地生长,是两种主要的生态类型,通常被认为表现出明显的表型分化。旱地品种较旱地品种耐旱性强,植株较高,分蘖潜力较低,抗稻瘟病能力较强,根系较灌水品种长而粗[2,3.].因此,旱地品种在旱地地区被广泛采用,以优化作物产量,因为它们更有效地利用水分和适应旱地。虽然旱稻的产量相对低于灌溉水稻,但它为近亿人提供了日常主食。4].由于我们现在面临着全球人口膨胀和水资源日益短缺的问题,旱稻,特别是关于旱地适应遗传机制的知识变得更加重要,因为它们使将高产灌溉水稻品种转化或改良为旱稻品种成为可能。
抗旱性是水稻适应大多数旱作旱地的一个重要组成部分,因为在这种生态系统中,水无法很好地保存,每年可能发生频繁或零星的干旱事件[5].之前的研究提出了三种不同的植物抗旱性机制:逃避干旱、避免脱水和耐脱水[6].通过干旱逃逸,在每年干旱时间可预测的情况下,调节植物生命周期的时间以逃逸干旱时间。脱水避免是指植物通过较发达的根系或较厚的角质层来避免水分的过度流失[6].脱水耐受性指的是渗透调节等诱导机制,它帮助植物在发生过量失水时防止损伤并存活下来[6].针对旱地干旱胁迫频繁且难以预测的情况,旱地作物应采取防脱水和耐脱水措施。事实上,旱稻在适应过程中似乎更多地依赖于本构性脱水规避,而非诱导性脱水耐受[7- - - - - -9].到目前为止,一些基因已被报道与抗旱性有关[10],如脱落酸(ABA)合成途径中的基因(如9-独联体-环氧类胡萝卜素双加氧酶基因)[11,12],转录因子与顺式作用脱水反应元件(DRE)相互作用[13],合成渗透保护剂的基因[14,15]和丝裂原激活蛋白激酶(MAPK)基因[16].然而,这些研究集中在单基因的影响,而适应旱地可能涉及基因组中的许多基因。此外,旱稻中有价值的遗传资源也值得加强利用,而这些资源在以往的研究中被忽视了。
在自然选择和人工选择的作用下,旱作水稻和灌溉水稻各自进化出各自的特征性状,这导致了对各自环境的表型适应和生态型间的遗传分化。在种群分化过程中,一些用于识别特定区域的统计工具已经建立起来。固定指数(F圣)是一种常用的基于遗传多态性数据来衡量群体分化的统计方法。F圣与等位基因频率差异正相关。跨种群复合似然比检验(XP-CLR)是一种新发明的方法,它不仅包含遗传分化,还包含等位基因频谱信息[17].通过对多位点等位基因频率分化和中性选择下的频谱进行联合建模,证明了XP-CLR在群体分化过程中具有较高的选择性扫描检测能力。此外,Nielsen的CLR也是一种基于复合似然比的参数检验,它利用站点频谱的空间分布来检测选择性扫瞄[18].该方法只使用一个种群的SNP数据,因此在单个种群中检测选择性扫瞄而不考虑种群分化。
为了研究旱地适应的遗传机制,并识别其重要组成部分“抗旱性”的基因,我们使用了大量资源的全基因组重测序数据,包括我们之前工作报告的84个旱地资源和82个灌溉资源[19],这些数据被用来测试含有脱落酸合成关键酶基因的低多样性区域是由于人类的选择。考虑到旱稻在世界上的广泛分布,我们的样本来自亚洲、非洲和南美洲,覆盖了大多数主要的旱稻分布区。探讨了旱作与灌溉生态型之间的系统发育关系和当前旱稻种群结构。更重要的是,通过检测选择性扫描区域使用F圣和XP-CLR,我们编制了一个候选基因列表,这些基因可能负责旱地适应或与旱地和灌溉生态类型之间的表型分化有关,为旱地作物育种提供了有价值的见解。
结果
旱地品种的表型与灌溉品种不同
关于84个全球取样和具有代表性的旱稻品种(见附加文件)1而且2),我们想确认它们是否具有报道的表型,从而将它们与灌溉水稻区分开来[2,3.].因此我们种植了这些品种来进行表型分析(参见附加文件)3.而且4).我们的研究集中在旱稻和灌溉水稻之间的4种非产量表型:根重、最大主根长、分蘖数和株高。此外,我们还研究了千粒重、生产穗数、每穗实粒数和每穗空粒数4个产量性状。在水源充足和旱地(雨养旱地)环境下,旱地材料均显著表现出较高的植株结构、较少的分蘖、较长的主根和较重的根重,这支持了之前报道的表型差异(表1)1,请参阅附加文件5表型差异)。就产量性状而言,在两种环境下,旱地与灌溉品种相比,表现出更大的千粒重和更少的高产穗1).灌水条件下,两种生态型玉米穗实粒数和空粒数无显著差异。而旱作条件下,旱作表现较好,籽粒充实程度显著提高(Student 's t检验,P= 0.016)和较少的空粒(学生t检验,P= 5.3e-05)。
利用SPSS 17.0统计软件,计算了旱地条件下4个非产量性状与各产量性状之间的pearson相关系数(ρ)6).株高与千粒重显著相关(ρ = 0.263,P= 0.002),每穗空粒数(ρ =−0.226,P= 0.009)和生产穗数(ρ =−0.2,P= 0.022)。分蘖数与生产穗数显著相关(ρ≈1,P< 0.001)和千粒重(ρ =−0.169,P= 0.05)。根重与每穗实粒数呈极显著相关(ρ = 0.295,P= 0.001)。最大根长与千粒重显著相关(ρ = 0.443,P< 0.001)和每穗空粒数(ρ =−0.232,P= 0.008)。因此,旱作条件下4个非产量性状均与产量性状显著相关。从ρ值可以推测,在旱地条件下,旱地水稻更大的根重和更大的根长将有助于净产量的增加,这可能是因为更发达的根系有助于更好地避免脱水。然而,株高和分蘖数对产量的影响是复杂的。较高的株高可能导致高产穗数减少,但可增加千粒重,减少空粒数。旱地分蘖数越少,生产穗数越少,但千粒重越高。
在比较两种栽培条件下的表型时,我们发现旱地环境对旱地和灌溉生态类型的株高和千粒重略有降低,但对分蘖数、根重、生产穗数和每穗实粒数均有显著影响。但主根长度和每穗空粒数对不同环境的响应不同。我们观察到,与灌溉条件下相比,灌溉水稻的主根长度轻微减少(- 2.5%),但旱地旱地水稻的根长度显著增加(+9.9%)(表2)1学生的学习任务,P= 0.002,附加文件7),显示旱稻的适应性特征。具体来说,61%的旱地材料在旱地条件下根系长度增加,而只有大约38%的灌溉材料在旱地条件下根系长度增加。此外,就每穗空粒数而言,我们观察到在旱地条件下,灌溉区的空粒数有轻微且不显著的减少(学生t检验,P= 0.29,附加文件7),但在高地地区则显著减少(学生t检验,P= 5.9e-07,附加文件7),表明旱稻的另一种适应性特征。基于旱地和灌区之间的表型差异以及旱地条件下非产量表型与产量表型之间的相关性,本研究将“旱地适应”定义为旱地水稻的特征非产量表型,这些表型与旱地条件下的产量性能相关,如株高、分蘖数、根重和最大根长,这些表型与抗旱性直接相关。
snp调用和注释的166个加入
对于82个灌溉和84个高地,我们获得了402,052,644对端reads通过了reads Pass Filter,总计约8 G碱基对(NCBI登录代码SRA066116)。84个旱地和82个灌区的重测序数据在我们以前的工作中报道过[19].在此,我们对这些重测序数据进行综合分析,以确定可能导致两种生态型之间的旱地适应和表型分化的遗传成分,这是水稻研究中长期未解决的问题。
使用SOAP-2.20软件,大约87%的reads可以与参考基因组(IRGSP/RAP build 5 Nipponbare基因组)对齐[20.].籼型和粳型reads的平均映射率分别为81%和92%。平均而言,每次加入的参考基因组覆盖29.54%(范围从18.65%到46.58%)。每一次登陆的平均深度约为0.50 ×(范围从0.27 ×到0.96 ×)(见附加文件)8).旱地种群和灌溉种群的基因组覆盖度分别达到41 ×和43 ×。经过SNP调用和一系列的质量控制过滤(见方法),共识别出3,571,104个高质量SNP9包括所有snp),其中3,563,681个是非单例snp。通过随机选择56个SNP并通过Sanger测序进行基因分型,我们能够验证我们的SNP调用准确率为96.4%。测序数据,虽然相对较低的深度,足以很好地满足我们的分析,我们将在下面讨论。
在接下来的分析中,仅使用了3,029,822个snp,其基因型数据覆盖了两个群体中超过10个个体。我们根据基因组背景将这些snp分类为不同的类别(参见附加文件)10).snp的非同义与同义之比为1.32,与先前报道的水稻的1.29之比一致[21].引起激进突变的snp,如过早停止密码子或帧中断,也被识别出来10).
旱稻的系统发育和群体遗传分析
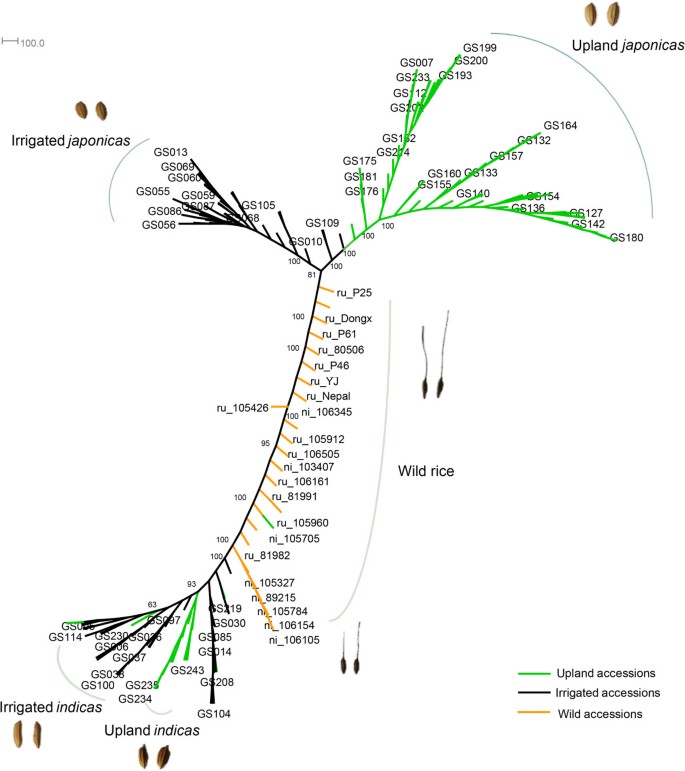
利用SNP基因型对166份资料进行系统发育分析。此外,从我们之前的数据中,有25个狂野的加入(10o . nivara和15由额外的文件11)被添加,以帮助解决高地资源的系统发育问题[21].我们使用带有100个自举的邻居连接方法构建了这些材料的系统发育树1).与以前的一些报告一致[21],我们的研究结果支持通常被认为是两个亚种的籼稻和粳稻是非常不同的类群,因为它们大部分聚集在各自的类群中,并且都有密切的野生亲缘关系(图1,也参见附加文件12).结果还表明,可能目前大部分的高地材料属于粳稻类型(在我们的样本中76%的高地材料是粳稻)(图)1额外的文件12),与采集中心基于传统表型特征的分类和此前旱稻主要属于粳稻的报道一致[22].系统发育树显示,所有的高地粳稻材料都聚集在一起,表明高地粳稻可能有一个单一的起源。而对于类型籼稻,旱地籼稻的分散模式表明旱地籼稻可能是用不同灌溉水稻作为基本遗传材料进行多次单独繁殖的。
旱稻群体结构与旱稻杂交种
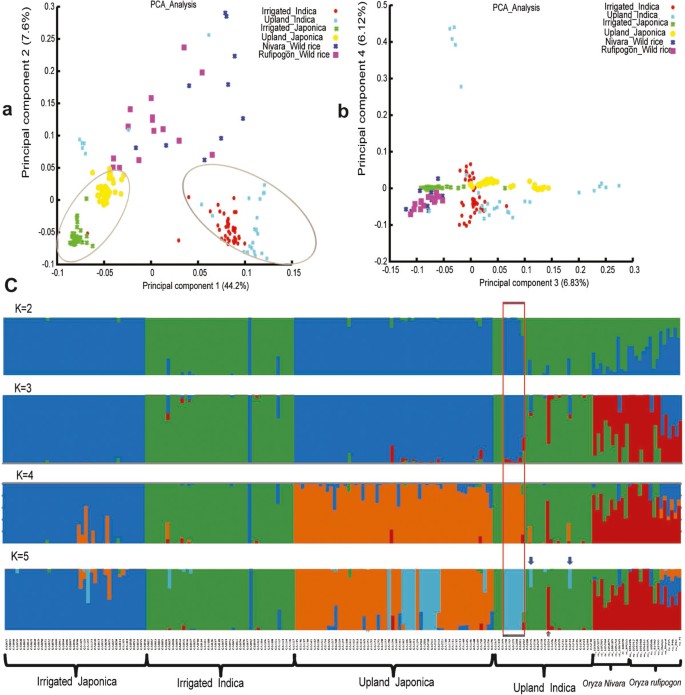
我们使用24,890个SNPs和超过100个资料的基因型数据来分析我们样本中的群体结构(见方法)。主成分分析(PCA)总结了整个种群的种群结构(图2a, b).前四个主要成分解释了大约65%的遗传变异。籼稻与粳稻在主成分1上的巨大差异。相比之下,旱地和灌溉生态型之间的遗传差异似乎要低得多。的F圣基于全基因组SNPs计算的两种生态型的值仅为0.06。即使在粳稻型中,高地亲本几乎是单系亲本,生态型分化非常明显(图1,图2A)旱地和灌水生态型仍存在一定的遗传分化(F圣= 0.13),远低于报道的籼型和粳型的分化(F圣= 0.55) (23].
人口结构的评估。PCA分析的前四个成分,如(一)而且(b).这四种成分加在一起解释了65%的遗传变异。(一)主成分1将粳稻型与籼稻型区分开来。这两种类型包括两种生态型(旱地型和灌溉型)。粳稻的生态型分化明显。(b),主成分3和4。(c),即FRAPPE报告的人口结构。结果显示K = 2到K = 5。当K = 2时,只区分籼稻和粳稻。当K = 3时,分离野稻。当K = 4时,粳稻出现生态型分化,即旱地粳稻开始与灌溉粳稻不同。结果表明,旱灌生态型分化在遗传上低于籼粳生态型分化。红色矩形表示6个旱地品种,它们以前被分类为旱地籼稻品种,但在遗传上更接近旱地粳稻群体。K = 5图中蓝色箭头表示两个陆地籼稻GS215和GS233,它们的基因组比例接近于陆地粳稻。红色箭头表示旱稻GS224,与野生稻相近。
我们还利用FRAPPE对种群结构进行了研究,以估计每个系的祖先和群体混合[24].将K(种群数)从2增加到5,旱地和灌区的种群结构如图所示2c).当K = 2时,我们看到籼稻型和粳稻型的分化,这种分化也存在于选用高产稻而且选用nivara在野生种群中。当K = 3时,野生稻与栽培稻发生分离。当K = 4时,灌水粳稻与旱地粳稻的分化出现,而灌水粳稻与旱地粳稻的结构仍然相同,说明旱地粳稻内部的生态型分化高于旱地粳稻内部。当K = 5时,高地粳稻中出现了一个新的亚群,这表明高地粳稻中的一些亲本形成了一个独特的亚群,与其他亚群有所区别。
从结构分析中,我们发现一些高地籼稻品种虽然主要按照表型划分为籼稻型,但其基因组组成却相当复杂。例如,6个接入(GS190、GS192、GS199、GS200、GS201、GS202)(在图中红色矩形中包含)2c),以前根据其表型(例如,种子形状,见附加文件)被分类为高地籼稻13),结果更接近高地粳稻(图2C),说明对于山地生态型,种子形状表型可能会误导籼型和粳型的区分。除了这6个系外,GS215和GS233也有很大的基因组比例接近高地粳稻(图中蓝色箭头所示)2c),这使得它们在系统发育树中与粳稻聚类(图1额外的文件12).而旱稻GS224 (Dular),用红色箭头表示,其野生稻血统比例较大,在系统发育树中与野生稻成簇(图1额外的文件12).这些结果表明,目前的高地籼稻类群是一个外加剂,包含了不同起源的亲本。它们被归为籼稻,可能是因为它们的某些性状更接近籼稻型而不是粳稻型。此外,在该树的籼稻类群中保留的19份旱地籼稻品种也具有多系性,表明它们可能是多次起源的,其旱地适应的遗传机制可能是异质性的。相反,形成单系系的高地粳稻品种更有可能具有类似的高地适应机制。考虑到众所周知的籼粳杂交,很可能旱稻和部分旱稻有相似的适应基因,而其余旱稻可能有不同的适应基因。因此,在接下来的生态型分化基因研究中,我们首先使用大样本量的方法,通过比较合并后的旱地和灌地群体,来寻找表现出最强信号的基因,并且这些基因可能在旱地粳稻和部分旱地籼稻之间共享。然后以粳稻类型为研究对象,寻找粳稻特有的高地适应基因。需要更多的数据和实验来全面研究复杂籼稻群体的适应机制。
基因组中的生态型分化基因组区(edr)
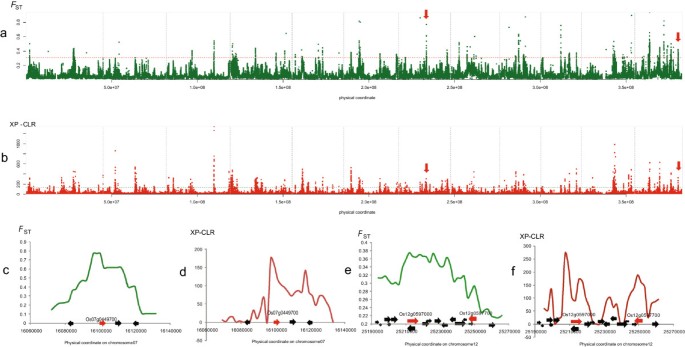
edr可能包含了解释适应性分化的基因。采用滑动窗口的方法来筛选地区与两个前5‰F圣和跨群体复合似然比检验(XP-CLR)评分(方法),用选择性信号对edr进行识别,以缩小旱稻和灌溉水稻之间表型分化的潜在基因(图)3.).在合并邻近的edr后,我们最终在基因组中识别出74个edr,中位长度为22.7 kb,在旱稻和灌溉水稻之间进行了区分14).在这些EDR区域内,共有8980个snp(附加文件15).与我们分析所用的全基因组snp相比,edr中的snp在基因区显著富集(卡方检验,P< 2.2e-16)(参见附加文件16).在EDR snp中,2623个生态型间的等位基因频率差异大于60%,我们随后将其标记为“生态型分化snp”(EDSs)(见附加文件)17).
EDRs扫描。(一),F圣根据基因组的物理坐标绘制值;红色水平虚线指的是最高5‰的门槛。(b),根据基因组坐标绘制XP-CLR评分;绿色横虚线为5‰的门槛。通过取两种方法给出的edr的交集得到edr。垂直虚线(一)而且(b)表示两条染色体之间的边界;垂直的红色箭头表示在(c - f)。(c)而且(d)分别显示了F圣和XP-CLR值Os07g0449700,一个有趣的基因注释为a型反应调节因子(用红色箭头表示)。(e)而且(f)分别显示F圣和XP-CLROs12g0597700,类似于WRKY2和Os12g0597000编码一种sos3样钙结合蛋白1(用红色箭头表示)。所有这些有趣的基因都有很强的F圣和XP-CLR值,在生物学上与水稻的旱地适应有关。
edr中的生态型分化基因(EDGs)
在edr中有154个基因,我们称之为edg(见附加文件)18).在这些EDGs中,74个基因在基因区有EDSs,另外53个基因在上游或下游1 kb内有EDSs。通过基因富集分析,我们发现EDGs在几个类中富集(表2额外的文件19),例如含有GDSL域类的脂肪酶(P= 7.49e-05)、过氧化物酶(P= 1.80e-03)、谷胱甘肽相关类(P= 5.44e-08),生长素信号通路(P= 3.80 e-06)。
据报道,含有GDSL结构域的脂肪酶参与了对非生物胁迫的响应,如干旱[25- - - - - -27],说明这些基因在旱稻中的遗传分化可能与它们对旱地的适应有关。过氧化物酶和谷胱甘肽相关蛋白具有抗氧化活性,这是植物适应类似干旱环境的另一个关键因素[6,7].此外,生长素,一种著名的生长促进激素,在决定植物的结构方面起着重要作用,并已被证明可以控制不定根的形成[8,28- - - - - -30.].因此,旱稻和灌溉水稻生长素信号的差异可能是其不同的结构和根系的潜在原因。此外,我们还发现了几个感兴趣的个体edg,它们可能在旱地适应或两种生态类型之间的表型分化中也很关键。Os07g0449700是一种a型响应调节器,具有特殊的意义(图3.c, d). a型反应调控因子属于细胞分裂素诱导的ARR基因家族,在细胞分裂素信号转导中起重要作用[31].不少证据表明,ARR基因影响根和芽的发育,a型ARR之间存在明显的功能重叠[31- - - - - -34].此外,a型ARR基因与旱稻和灌溉水稻根和茎表型的相关性也得到了实验支持;在转基因水稻中,a型反应调控因子的过表达已被证明会导致矮化和根系发育不良[32].因此这种a型ARR基因的生态型分化,Os07g0449700,可以解释旱稻和灌溉水稻根高表型的某些差异。在旱稻和灌溉水稻之间进一步的互惠转基因实验将最终解决水稻的贡献Os07g0449700在适应旱稻方面。
其他edg也被直接或间接的证据暗示与山地适应有关。Os12g0597700(图3.e, f)具有WRKY dna结合结构域,WRKY基因已被证实是耐旱性的重要元素[35- - - - - -38].Os12g0597000编码一种sos3样钙结合蛋白1及其在拟南芥与对缺水的反应有关[39].Os11g0474600编码一种萜类合成酶,萜类植物激素已被证明能抑制水稻分蘖[40].因此,这个基因可以解释旱稻和灌溉水稻分蘖数的差异。此外,几个五肽重复蛋白编码基因(Os01g0646000,Os06g0184866,Os06g0185800)和两个丝氨酸/苏氨酸蛋白激酶基因(Os04g0616400,Os04g0616600)也可能与干旱反应有关,因为这类基因已被报道参与对干旱/渗透胁迫的反应和根系发育[38,41].这些结果说明这154个EDGs可能是研究这两种生态类型的表型分化和旱稻适应性的一个有价值的候选基因表。
在个体群体中选择edg
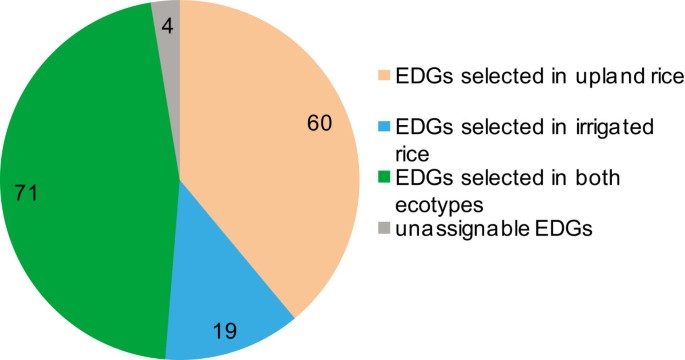
研究的EDGs表明了两种生态型分化过程中的选择。为了进一步确定选择发生在哪个种群中,我们使用尼尔森的CLR检验来确定单个种群中可能的选择性扫描(参见方法)。我们在旱地种群中确定了661个选择性扫描区域,中位数长度为27 kb,在灌溉种群中确定了351个选择性扫描区域,中位数长度为41 kb20.).在选定的旱地和灌地群体中,分别有2542和1285个基因。因此,旱稻的基因组区域/基因选择数量是灌溉水稻的两倍,这表明旱稻品种可能在适应或育种中承受了更多的选择压力。通过将154个edg与这些潜在选择的基因重叠,我们发现几乎所有这些选择的edg(150/154)都可以使用Nielsen的CLR方法再次确认为选择的基因。其中,60个可能在旱稻群体中特异选择,19个可能在灌溉群体中特异选择,而71个可能在两个群体中特异选择(图4额外的文件18).
japonica特异性和indica特异性EDGs
水稻复杂性状的遗传结构通常既丰富又复杂,因为它们在亚种之间通常是异质的[42].据此,我们还对籼型和粳型的生态型分化基因进行了研究。如图1说明,旱地籼稻有多个起源,因此在旱地适应方面可能是遗传异质性的。在研究特定类型的edr时,我们相应地主要关注于japonica类型。
我们采用了类似的管道来筛选japonica类型的edr(附加文件)21),找到了52份中位数长度为24 kb的edr(见附加文件)22).在这些edr中,识别出169个edg(见附加文件)23),其中47个EDG与先前的总种群EDG列表重叠,其余122个为日本特有EDG列表。对于这些日本特有的edg,显著富集的基因家族包括丝裂原激活蛋白激酶(P= 1.37e-14), WRKY基因(P= 0.03)和硫氧还蛋白编码基因(P= 0.04)(表2).先前的研究表明丝裂原激活蛋白激酶在生物胁迫响应和根发育中发挥重要作用[11,43];这类粳稻特有的EDG与稻瘟病诱导的激酶非常相似,这可能解释了为什么旱稻通常比灌溉水稻更耐稻瘟病。此外,如前所述,WRKY基因和硫氧还蛋白可能对干旱反应至关重要。有趣的是,在我们的列表中,我们发现Os09g0410500与著名的玉米驯化基因teosinter -branched 1(tb1),表明该基因也可能与旱地型和灌水型分蘖能力的不同有关。
在籼型中,由于混合了旱地籼,只有19个仍聚在籼稻组中的旱地籼可用于分析。但是,F圣旱地和灌地籼稻的差异(0.06)比粳稻的差异(0.13)低得多,这意味着需要更大的样本来检测籼稻的生态型分化。然而,由于世界上的旱地籼稻群体规模小,迄今为止难以收集到足够的样品。仅使用19个高地籼稻供试品系进行分析,得到了通过交叉鉴定的56个基因列表F圣和XP-CLR顶部信号(附加文件24额外的文件25).因此,考虑到籼稻的样本量小,且可能存在异质性的适应机制,56个基因与先前的EDG列表在总居群中没有重叠也就不足为奇了。然而,我们注意到,来自籼稻XP-CLR顶部信号的9个和4个基因分别与合并居群的edg和粳稻居群的edg重叠26额外的文件27),表明一些与陆地生态型相关的基因可能在一些籼稻和粳稻之间共享,但由于样本数量有限且可能是异质性的,因此较难检测。需要更多的实验和数据来全面研究籼型特异高地适应基因。
讨论
很大程度上由于灌溉水稻的普及和繁殖力,对区分旱稻的表型研究有限。Chang等人在这方面做了一些工作,他们调查了11个旱地品种的叶片产量、分蘖产量、株高、根系和抗旱性作为不同表型的解释因素[3.].同样,小野也指出,日本的山地品种分蘖高、分蘖低,叶宽,穗长,抗旱、抗爆,肥料利用效率较低[44].尽管发现了有趣的结果,但这些研究只是基于少数几种旱稻品种。据我们所知,本研究中旱稻和灌溉水稻表型比较的样本量是迄今为止旱稻品种的一个非常大的样本,样本覆盖了世界上大多数旱稻分布地区,为Chang等人的观察提供了坚实的证据[3.].我们的表型分析还发现了一些新的特征,如旱地栽培增加了旱稻的主根长度而减少了灌溉水稻的主根长度,这表明旱稻在适应旱地环境方面具有显著的优势。
除了我们彻底的表型分析外,据我们所知,该研究还提供了关于当前旱稻系统发育和种群结构的第一个基因组视角。与我们基于表型特征的分类和Glaszmann的报告一致,该报告指出,大部分山地品种属于粳稻型[22],系统发育分析表明,大部分高地品种属于粳稻型,形成一个单系系。群体结构推断表明,两个生态型之间的遗传分化远远小于籼粳型之间的遗传分化,这可能是由于生态型之间的基因流动比籼粳型之间频繁得多,也可能是由于旱稻的起源较近。
虽然我们研究中使用的每个加入的测序深度相对较低,但数据集本身的信息量足够解决我们想要解决的关于旱稻适应的问题。首先,即使考虑到较低的覆盖率,识别出的snp仍具有较高的准确性(96.4%)。其次,在旱地或灌溉群体中,每个位点的平均测序深度大于40×,为等位基因频率估计提供了足够的样本量。同样地,每个群体中只有在超过10个个体中确定基因型的SNP位点被用于计算等位基因频率等分析,只有不少于100个个体的基因型的SNP位点被用于估计群体结构。第三,虽然每个加入的基因组覆盖率不高,但对于每一个成对比较,都有足够的信息位点来精确计算任意两个加入之间的距离(见附加文件)28).因此,正如之前的一些研究所表明的那样,我们提出了一个有趣的例子,证实了低覆盖率人群基因组数据的遗传分析的可行性[45,46].
除了一些QTL定位工作外,很少有报道涉及到旱稻适应性表型的遗传机制[47,48].在这项研究中,我们发现了一些有趣的候选基因或基因家族,它们可能有助于旱地和灌溉生态型之间的旱地适应性性状或表型分化,这不仅为利用旱地水稻的遗传适应性提供了令人兴奋的新证据,而且证明了我们的方法的有效性。虽然还需要更多的实验证据来验证所有已识别的等位基因的精确表型效应,但数量有限的基因(约150个)为功能研究和旱地育种提供了一个高度可行的列表。最重要的是,我们想强调的是,我们的方法很可能捕获了大多数解释生态型分化和旱稻适应的主要基因组区域,包括粳稻型的特定区域,但确定一些籼稻加入的特定适应基因仍需要进一步的工作和更多的数据。
结论
总之,我们调查了旱地和灌溉条件下的大量旱地水稻的表型,并将它们与类似条件下的灌溉水稻进行比较,我们能够证明两种生态类型之间的一些显著表型差异。通过对大量水稻材料的高通量重测序,我们首次明确了旱稻粳稻的单一起源模式,而旱稻籼稻的多重起源模式。通过群体遗传分析,确定了旱稻与灌水生态型分化过程中的生态型分化区域和重要的选择基因,为旱稻适应和两生态型表型分化提供了重要的理论依据。这些发现对育种中旱稻品种的推广具有重要意义。例如,可能与抗旱性有关的基因可以用来从现有的高产灌溉品种中培育出高抗旱性的品种;同时,可能与分蘖能力有关的基因可以用来增强旱稻的分蘖能力,这是限制其产量和随后广泛使用的主要因素。
方法
调查高地资源的表型
为了评估报告的旱稻和灌溉水稻的表型,我们种植了这两个品种。由于一些资源的种子数量有限,只有131个资源被表型化(附加文件3.).表型分析在灌溉和旱地条件下进行(即,防止浸入水中以模拟旱地环境)。灌水条件下,种子发芽,秧苗移栽到浸水的水田中,整个生长发育阶段。对于旱地条件,我们采用了直播。为充分模拟雨养条件,整个生育期均不灌水。下雨时,我们放水防止被淹没。根据我们的记录,在高地条件下发生了几次中度干旱事件,并造成了一些单株植物死亡。每增栽12株,两行栽,每行6株,行距30厘米,株行距20厘米。抽穗期前,统计分蘖数。在植株成熟后,对其根重、主根长、株高及产量性状进行了测定。 For each accession, about five individuals were randomly selected and phenotyped (Additional file4).由于旱作条件下分蘖结穗率接近100%,本研究旱作条件下分蘖数和生产穗数结果一致。
重排序的数据
84个旱地和82个灌区的重测序数据在我们以前的工作中报道过[19].在我们之前的研究中,这些数据只是用来测试所选择的含有ABA合成关键酶基因的区域不是由于人口统计学原因。它们既未用于研究旱稻群体结构,也未用于研究两生态型间的群体分化区域/基因。在此,我们利用这些重测序数据进一步挖掘两种生态类型之间的山地适应和表型分化的遗传成分。84份旱地和82份灌溉资料来自中国云南省农业科学院。生态型信息由遗传资源中心提供。籼稻和粳稻都被选为灌溉组或旱地组2).在灌水生态型中,我们选择了42个籼稻品种和40个粳稻品种。对于旱地生态型,我们选择了28个籼稻和56个粳稻品种,因为在我们的遗传资源中心,大多数旱地水稻品种属于粳稻类型2).这些藏品的起源覆盖了广泛的地理范围(附加文件1),包括大部分主要的旱稻分布地区(南亚、东南亚、南美洲、西非等)。所有种子都是在灌溉条件下播种的。当幼苗发育到三叶草期时,用CTAB法从叶片中提取基因组DNA [49].
读取映射
我们先前获得的这些高地和灌溉地区的重测序数据保存在NCBI Short Read Archive中,登录号为SRA066116 [19].野生水稻的原始解读来自我们之前的工作(NCBI短解读档案登录号为SRA023116)[21].IRGSP 5.0 Nipponbare基因组从RAP-DB数据库下载(http://rapdblegacy.dna.affrc.go.jp/download/latest/IRGSPb5.fa.masked.gz).我们分别使用SOAP将短reads映射到Nipponbare基因组[20.].
调用SNP和SNP注释
在映射读取之后,我们使用SOAPsnp -v1.02调用灌溉和旱地接入的整个种群的snp [50].为了保证snp的质量,进行了一系列的筛选。例如,删除质量值小于15或拷贝号接近> 1.5的snp。此外,我们还排除了深度小于6或大于300的snp位点,因为深度过低的位点容易出现测序错误,而深度过高的位点很可能位于重复或重复区域[51].为了确认SNP呼叫的准确性,我们随机选取了60个SNP,用PCR扩增其周围的序列,并对其进行Sanger测序。在我们的实验中,60对引物中有56对起作用。随后,我们使用基因组注释将这些snp分配到与转录和翻译相关的不同类别中,包括基因区域(CDS、内含子和utr)、启动子区域和基因间区域,使用我们内部的PERL脚本。导致停止密码子过早、开始/停止密码子中断或剪接供体/受体位点的snp被定义为自由基snp。
确定每个加入的SNP基因型
对于每一个加入,我们从SOAPsnp的输出中提取SNP基因型信息。再次,我们进行了一系列的质量控制程序,以确保基因型的可靠性。共识文件中包含了各站点的最佳基础基因型及其平均质量分数、次优基础基因型及其平均质量分数、共识基因型及其质量分数等信息,可作为过滤器[50].我们使用非常严格的标准,以确保只考虑质量评分> =20(错误率不超过1%)的基因型。更明确地说,如果最佳碱基的平均质量分> =20,而次最佳碱基的平均质量分< 20,则该基因型为最佳碱基。如果最佳碱基和次最佳碱基的平均质量分数都为> =20,则该基因型为杂合型。如果最佳和次最佳碱基的平均质量分数都< 20,但一致基因型的质量分数为> =20,则使用一致基因型。由于我们的重测序数据覆盖率相对较低,平均每次添加的SNP基因型缺失67%。然而,这些缺失的数据可能不会影响我们的分析,因为系统发育分析是基于许多位点的成对平均距离,而群体遗传学分析是基于等位基因频率。因此,这些数据足以估计成对距离和等位基因频率。除对166份材料重新测序外,对野生材料的snp基因型也采用相同的方法进行了测定。
构建系统发育树
结合野生稻品种的基因型信息,我们使用每个品种的SNP基因型,利用PHYLIP [52].我们使用默认参数来计算距离,即使用F84模型,转换/横向比为2.0,没有跨站点的Gamma分布率。我们假设整个基因组的突变率是一致的,因此对不同的snp给予相同的权重。对全基因组SNP数据集进行100次重复的引导。在计算了100个重复数据集的遗传距离后,利用基于遗传距离的Neighbor-Joining (NJ)方法构建了系统发生树[52].然后使用PHYLIP内置的“共识”程序建立共识树,参数包括“严格多数原则”和“被视为无根树”。
人口结构推理
我们首先使用报道的程序进行了PCA分析[53].共使用24890个snp,基因型数据覆盖100多个资料。此外,FRAPPE软件[24],该方法使用频率论方法来估计个体混合,也被用于检验种群结构。EM运行的最大迭代次数设置为10000。
计算F圣
根据材料的基因型确定SNP等位基因频率。在计算特定群体的等位基因频率时,一个群体中小于10个个体基因型信息的snp被丢弃。根据等位基因频率,我们计算出F圣'使用Nei [54].全基因组扫描采用20kb滑动窗口和2kb滑动步长。通过平均F圣每个窗口中SNP位点的值,F圣获取了Windows的值。
使用复合似然方法的扫描选择性扫描
交叉种群复合似然比检验(lp -CLR)和尼尔森复合似然比检验(CLR)是利用SNP数据集识别选择性扫描信号的两种方法[17,18].XP-CLR结合两个群体间的等位基因频率分化模式和多位点频谱,识别群体分化中发生的选择性扫瞄。尼尔森的CLR方法使用特定人群中等位基因频谱的分布模式来搜索该人群中的选择性扫描信号。在我们的分析中,我们分别使用XP-CLR和SweepFinder软件将XP-CLR和尼尔森的CLR结合起来。在XP-CLR分析中,我们使用了旱地和灌溉群体的等位基因频率,计算XP-CLR得分,并选取了排名前5‰的地区。在计算XP-CLR评分时,我们使用0.1 cM的窗口大小、2 kb的网格大小和每个窗口的最大SNP数150。将这些XP-CLR得分最高的区域与前5‰的区域重叠F圣,得到了旱稻和灌水水稻生态型分化区域(edr)的保守集,以及这些区域内的生态型分化基因(EDGs)。在Nielsen的CLR分析中,我们使用单一种群(高地或灌溉种群)的等位基因频率,发现基因组中最高似然比(LR)值的前5%网格点。这些网格点10 kb内的基因被认为是两个水稻群体的潜在选择目标[55].在计算LR值时使用了细尺度网格点(每1000个核苷酸一个网格点)。
EDGs的基因富集分析
基因富集分析采用卡方检验。对于特定类别的基因,我们利用水稻基因注释信息计算整个基因组内该类别的基因数量[56].例如,对于基因类别“含脂肪酶GDSL域”,我们统计了水稻基因注释数据库中带有该域注释的脂肪酶基因的数量[56].在edg中也计算了这类基因的数量。然后根据edg中感兴趣的基因数、edg中总基因数、全基因组中感兴趣的基因数、全基因组中总基因数构建列联表。然后进行卡方检验,得到P值。
加入代码
166个品种的原始序列数据已存入NCBI Short Read Archive (SRA,http://www.ncbi.nlm.nih.gov/sra/),注册号为SRA066116。
缩写
- XP-CLR:
-
跨种群复合似然比
- CLR:
-
复合似然比
- 功能:
-
生态型分化区域
- EDG:
-
基因功能
- EDS:
-
生态型分化snp。
参考文献
- 1.
水稻的起源、传播、栽培和变异。植物生物学杂志,1997,35(1-2):25-34。
- 2.
Chang TT VS:旱稻品种多样性与形态农艺特征。1975年,菲律宾拜奥斯:旱稻的主要研究,国际水稻研究所,72-90。
- 3.
Chang TT LC, Tagumpay O:旱稻和低地水稻品种的农艺和生长特性。1972年,菲律宾拜奥斯:水稻育种,国际水稻研究所,645-661。
- 4.
旱稻研究综述。1982年Bouake. 1984, LOS BAÑOS,拉古纳,菲律宾:象牙海岸高地水稻研讨会,国际水稻研究所(IRRI), 1-500。
- 5.
IRR研究所:旱稻的主要研究。1975年,菲律宾拜奥斯:国际水稻研究所(IRRI)
- 6.
Bernier J, Atlin GN, Serraj R, Kumar A, Spaner D:回顾:旱稻抗旱性育种。2008
- 7.
Price AH, Steele KA, Moore BJ, Barraclough PB, Clark LJ:旱稻(Oryza sativa L.)根系渗透能力qtl的RFLP和AFLP组合连锁图谱。应用理论与实践,2000,20(1):49-56。
- 8.
Cairns JE, Audebert A, Mullins CE, Price AH:旱稻(Oryza sativa L.)暴露于不同土壤性质的土壤水分亏缺的土壤中,与根系生长相关的数量性状位点的绘制。农学通报,2009,34(1):1 - 4。
- 9.
Price AH, Steele KA, Moore BJ, Jones RGW:在充满土壤的室内种植的旱稻和暴露于不同的缺水条件II。定位根系形态和分布的数量性状位点。田间作物学报2002,76(1):25-43。
- 10.
朱建军:植物盐和干旱胁迫信号转导。植物生态学报,2003,24(3):347 - 347。
- 11.
Thompson AJ, Jackson AC, Symonds RC, Mulholland BJ, Dadswell AR, Blake PS, Burbidge A, Taylor IB:番茄9-顺式环氧类胡萝卜素双加氧酶基因异位表达导致脱落酸过量产生。植物学报,2000,23(3):363-374。
- 12.
Iuchi S, Kobayashi M, Taji T, Naramoto M, Seki M, Kato T, Tabata S, Kakubari Y, Yamaguchi-Shinozaki K, Shinozaki K:拟南芥脱落酸生物合成关键酶9-顺式-环氧类胡萝卜素双加氧酶基因调控的抗旱性。植物学报,2001,27(4):325-333。
- 13.
夏佳佳,刘强,三浦S,山口-筱崎K,筱崎K:单胁迫诱导转录因子基因转移提高植物抗旱性、耐盐性和抗冻性。中国生物工程学报,1999,17(3):287-291。
- 14.
Garg AK, Kim JK, Owens TG, Ranwala AP, Choi YD, Kochian LV, Wu RJ:海藻糖在水稻植株中的积累使水稻对不同非生物胁迫具有较高的耐受性。中国生物工程学报,2004,25(3):369 - 369。
- 15.
Jang IC, Oh SJ, Seo JS, Choi WB, Song SI, Kim CH, Kim YS, Seo HS, Choi YD, Nahm BH, Kim JK:在转基因水稻植株中表达海藻糖-6-磷酸盐合成酶和海藻糖-6-磷酸盐磷酸酶的大肠杆菌基因双功能融合增加了海藻糖的积累和非生物胁迫的耐受性,而不阻碍生长。植物生理学报,2003,31(2):516-524。
- 16.
熊林,杨燕:水稻的抗病性和抗非生物胁迫能力是由脱落酸诱导的有丝分裂原激活蛋白激酶反向调节的。植物科学学报,2003,15(3):745-759。
- 17.
Chen H, Patterson N, Reich D:种群分化作为选择性扫瞄的检验。中国生物医学工程学报,2010,20(3):393-402。
- 18.
Nielsen R, Williamson S, Kim Y, Hubisz MJ, Clark AG, Bustamante C:使用SNP数据进行选择性扫描的基因组扫描。中国生物医学工程学报,2005,15(11):1566-1575。
- 19.
吕军、张胜、董颖、何伟、张俊、邓鑫、张颖、李鑫、李斌、黄伟、万伟、余勇、李强、李俊、刘鑫、王斌、陶东、张刚、王俊、徐旭、胡峰、王伟:旱稻优良品种标签snp分析揭示了一个重要的等位基因。科学通报,2013,4:2138-
- 20.
李锐,李勇,Kristiansen K,王娟:SOAP:短寡核苷酸对齐程序。生物信息学,2008,24(5):713-714。
- 21.
徐鑫、刘鑫、葛森、Jensen JD、胡峰、李鑫、董勇、Gutenkunst RN、方磊、黄磊、李俊、何伟、张刚、郑鑫、张峰、李勇、于超、Kristiansen K、张鑫、王俊、Wright M、McCouch S、Nielsen R、王伟:对50份栽培稻和野生稻产量标记进行重测序,鉴定农艺重要基因。中国生物工程学报,2012,30(1):105-111。
- 22.
亚洲水稻品种的同工酶与分类。应用科学学报,1987,74(1):21-30。
- 23.
魏黄X, Kurata N, X,王ZX,王,赵问,赵Y,刘K,卢H,李W,郭Y, Y,周C, D,粉丝翁Q,朱C,黄T,张L,王Y,冯L, Furuumi H,久保T, Miyabayashi T,元X,徐问董G,詹Q,李C,富士山,丰田章男,et al:水稻基因组变异的地图揭示了种植水稻的起源。自然学报,2012,490(7421):497-501。
- 24.
唐宏,彭军,王萍,Risch NJ:单掺合料的估算:分析和研究设计的考虑因素。中华流行病学杂志,2005,28(4):289-301。
- 25.
胡海红,尤娟,方玉军,朱晓燕,齐志勇,熊丽珍:水稻耐寒耐盐转录因子基因SNAC2的鉴定。植物生物学杂志,2008,29(1-2):169-181。
- 26.
洪俊杰,崔华伟,黄is, Kim DS, Kim NH, du Choi S, Kim YJ, Hwang BK:新型gsll型辣椒脂肪酶基因CaGLIP1在病害易感性和非生物胁迫耐受性中的作用。植物学报,2008,27(3):539-558。
- 27.
Gupta S、Bharalee R、Bhorali P、Bandyopadhyay T、Gohain B、Agarwal N、Ahmed P、Saikia H、Borchetia S、Kalita MC、Handique AK、Das S:基因表达分析鉴定茶叶抗旱子代。功能整合基因组学,2012,12(3):543-563。
- 28.
吴强,林杰,刘建忠,王鑫,林伟,Oh M, Park J, Rajashekar CB, Whitham SA, Cheng NH, Hirschi KD, Park S:拟南芥glutaredoxin AtGRXS17异位表达增强番茄耐热性。植物生态学报,2012,10(8):945-955。
- 29.
Price AH, Steele KA, Gorham J, Bridges JM, Moore BJ, Evans JL, Richardson P, Jones RGW:在充满土壤的室内生长并暴露于不同缺水条件下的旱稻I.根系分布、水分利用和植株水分状况田间作物学报2002,76(1):11-24。
- 30.
刘德东,董清良,方美琴,陈克勤,郝玉军:异位表达苹果无融合相关基因MhFIE诱导共抑制,导致番茄营养和生殖发育异常。植物生理学报,2012,29(6):673 - 673。
- 31.
对JP、Haberer G、Ferreira FJ、Deruere J、Mason MG、Schaller GE、Alonso JM、Ecker JR、Kieber JJ: a型拟南芥反应调控因子是细胞分裂素信号的部分冗余负调控因子。植物学报,2004,16(3):658-671。
- 32.
广濑N,牧田N,小岛M, Kamada-Nobusada T,坂原H: a型反应调控因子过表达改变水稻形态和细胞分裂素代谢。植物生理学报,2007,48(3):523-539。
- 33.
Argyros RD、Mathews DE、Chiang YH、Palmer CM、Thibault DM、Etheridge N、Argyros DA、Mason MG、Kieber JJ、Schaller GE:拟南芥B型反应调控因子在细胞分裂素信号转导和植物发育中发挥关键作用。植物学报,2008,20(8):2102-2116。
- 34.
Hill K, Mathews DE, Kim HJ, Street IH, Wildes SL, Chiang YH, Mason MG, Alonso JM, Ecker JR, Kieber JJ, Schaller GE:拟南芥细胞分裂素反应中b型反应调控因子的功能表征。植物生理学报,2013,32(1):369 - 369。
- 35.
Ulker B, Somssich IE: WRKY转录因子:从DNA结合到生物功能。植物生态学报,2004,7(5):491-498。
- 36.
张颖,王磊:WRKY转录因子超家族:在真核生物中的起源和在植物中的扩展。中国生物医学工程学报2005,5:1-
- 37.
Eulgem T, Rushton PJ, Robatzek S, Somssich IE:植物转录因子WRKY超家族。植物科学进展,2000,5(5):199-206。
- 38.
雷贝洛,陈建军,陈建军,陈建军,陈建军。旱稻根系抗旱基因的克隆与鉴定。华北农学报,2008,29 (2):137 - 137
- 39.
Cheong YH, Pandey GK, Grant JJ, Batistic O, Li L, Kim BG, Lee SC, Kudla J, Luan S:两个钙调磷酸酶b样钙传感器与蛋白激酶CIPK23相互作用,调控拟南芥叶片蒸腾和根系钾吸收。植物学报,2007,32(3):366 - 366。
- 40.
梅原M,花田A,吉田S,秋山K, Arite T,武田-神谷N, Magome H,神谷Y,白苏K, Yoneyama K,京祖卡J,山口S:新型萜类植物激素对枝条分枝的抑制作用。自然学报,2008,455(7210):195-200。
- 41.
王辉,黄铮,陈强,张铮,张辉,吴勇,黄东,黄锐:番茄JERF3在烟草中异位过表达激活下游基因表达,增强耐盐性。植物生理学报,2004,25(2):366 - 366。
- 42.
Zhao K, Tung CW, Eizenga GC, Wright MH, Ali ML, Price AH, Norton GJ, Islam MR, Reynolds A, Mezey J, McClung AM, Bustamante CD, McCouch SR:全基因组关联图谱揭示了水稻复杂性状的丰富遗传结构。科学通报,2011,2:467-
- 43.
邢涛,王新军,Malik K, Miki BL:一种拟南芥钙调素样结构域蛋白激酶异位表达增强番茄原生质体NADPH氧化酶活性和氧化爆裂。植物生态学报,2001,14(10):1261-1264。
- 44.
小野S:日本的旱稻育种。1971,日本农业研究季刊:东京
- 45.
Turner TL, Bourne EC, Von Wettberg EJ, Hu TT, Nuzhdin SV:种群重测序揭示拟南芥对蛇纹石土壤的局部适应性。植物学报,2010,42(3):260-263。
- 46.
赵魏黄X, X,唱T, Q,冯Q,赵Y,李C,朱C,陆T,张Z,李米,风扇D,郭Y,小王,小王L,邓L,李W,陆Y,翁Q,刘K,黄T,周T, Jing Y, Z,盾牌,钱Q,张QF,李J,汉族B: 14个农艺性状的全基因组关联研究大米一起。植物学报,2010,42(11):961-967。
- 47.
黄宁,Courtois B, Khush GS,林红霞,王桂良,吴萍,郑克林:水稻株高数量性状位点与矮化基因的关联。遗传。1996,77:130-137。
- 48.
穆鹏,李振春,李春平,张海林,吴春春,李超,王晓康:水稻与旱稻杂交DH系根系性状的QTL定位及其与抗旱性的相关分析。科学通报,2003,48(24):2718-2724。
- 49.
Richards E, Reichardt M, Rogers S:从植物组织中制备基因组DNA。3 .中国生物医学杂志,2001,第2章:第2单元
- 50.
李锐,李勇,方旭,杨辉,王杰,Kristiansen K: SNP检测在大规模并行全基因组重测序中的应用。基因组学报,2009,19(6):1124-1132。
- 51.
林敏敏,徐旭,刘旭,陈伟,杨刚,王福林,李明伟,何伟,秦宁,王斌,李娟,简明,王杰,邵刚,孙世生,张刚:31个野生大豆和栽培大豆基因组的重测序鉴定遗传多样性和选择模式。植物学报,2010,42(12):1053-1059。
- 52.
Felsenstein J: PHYLIP——系统发育推断包(版本3.2)。支序分类学。1989年,164 - 166。5
- 53.
Patterson N, Price AL, Reich D:种群结构与特征分析。科学通报,2006,2 (12):e190-
- 54.
分子进化遗传学。1987,纽约:哥伦比亚大学出版社
- 55.
Williamson SH, Hubisz MJ, Clark AG, Payseur BA, Bustamante CD, Nielsen R:在人类基因组中定位最近的适应性进化。科学通报,2007,3 (6):e90-
- 56.
田中T, Antonio BA,菊池S,松本T, Nagamura Y, Numa H, Sakai H, Wu J, Itoh T, Sasaki T, Aono R,藤井Y,叶原T,原田E,菅野M,川原Y,川岛H,库布卡H,松屋A,中冈H,赛一N,三本松R,佐藤Y, Shinso Y,铃木M,武田J,谷野M, Todokoro F, Yamaquchi K,水稻注释项目,等:水稻注释项目数据库(rapa - db): 2008更新。核酸学报,2008,36(数据库问题):D1028-D1033。
致谢
计算资源由中国科学院西南超算中心提供。感谢哈佛大学公共卫生学院的华晨博士就数据分析提出的建设性建议。并感谢惠翔博士帮助编辑稿件。云南省科技厅项目(2011BB015, 2013HA004, 2013GA004),中国973项目(2013CB835200, 2013CB835201)资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者声明他们没有竞争利益。
作者的贡献
JL, WW, FH设计了该项目。FH和DT提供了大米材料。HL, JL, LM进行测序,JL, ZG, WH和XL分析数据。BL做了表型分析。JL、SZ、JZ、WH和BL栽培稻。JL和WW写了手稿。所有作者阅读并批准了最终稿件。
电子辅料
12870 _2014_1574_moesm1_esm.docx
附加文件1:入学名额的地理分布。蓝色的圆点表示这些资源的来源,而圆点的大小大致对应于来自某些地区的资源的数量。(多克斯1 MB)
12870 _2014_1574_moesm4_esm.xlsx
附加资料4:旱地条件(a)和灌溉条件(b)下的品种表型
12870 _2014_1574_moesm5_esm.docx
附加文件5:五个旱地和五个灌溉地的表型比较。左边5个品种为灌水稻(从左到右依次为:红油4号、滇屯502、云辉290、河西42、台中65)。右边的5个品种是旱稻(从左到右依次为IRAT104、CNA4140、Sanlicun、Arias Halus、Dourado)。这幅图表明,高地类型通常有较高的结构,较发达的根,和较少的分蘖。(多克斯17 MB)
12870 _2014_1574_moesm6_esm.xlsx
附加资料6:旱地条件下4个性状(根重、最大根长、分蘖数和株高)与产量性状(千粒重、每穗实粒数、每穗空粒数和生产穗数)的相关性(XLSX 14 KB)
12870 _2014_1574_moesm7_esm.xlsx
附加文件7:灌溉和旱作条件下的最大主根长度(a)和空粒数(b)及统计检验(XLSX 21 KB)
12870 _2014_1574_moesm10_esm.docx
附加文件10:166个条目的SNP注释。a,利用基因组注释信息将snp分为基因间snp和基因区snp,并进一步将其分为内含子或UTR区snp、非同义snp和同义snp。b, snp导致激进突变,包括中断/启动密码子,导致过早停止密码子或中断剪接位点。(多克斯14 KB)
12870 _2014_1574_moesm12_esm.docx
附加文件12:高地资源的系统发育分析。绿枝、黑枝、橙枝分别为旱地、灌溉地和野生地。叶节点用访问的样本号标记。这棵树表明旱稻稻型似乎有多次起源,而旱稻稻型可能源于单一起源。多克斯(660 KB)
12870 _2014_1574_moesm13_esm.docx
附加文件13:六份错误分类资料中的五份。(a),典型的籼稻种子;(b),典型的粳稻种子;(c) GS190(TOS2300)、GS192(WAB56-125)、GS199(CNA4140)、GS200(瓜拉尼)、GS201(Dourado)、GS202(TGR78),根据其表型(即种子形状等)以前被归类为旱地籼稻。根据我们的全基因组系统发育分析,它们更接近高地粳稻。由于没有种子,GS200没有被拍摄到。多克斯(432 KB)
12870 _2014_1574_moesm15_esm.docx
附加文件15:话单中snp的注释。在edr中,共有8980个snp,其中2409个位于基因区域内。在基因区域中,同义snp和非同义snp分别为241个和266个。(多克斯12 KB)
12870 _2014_1574_moesm20_esm.docx
附加文件20:在高地(a)和灌溉(b)种群中选择性清扫区域的长度分布。中位长度分别为27002 bp(a)和41001 bp(b)。(多克斯14 KB)
日本型EDR扫描。
补充文件21:(a)F圣高地粳稻和灌溉粳稻之间的值根据基因组的物理坐标绘制;红色水平虚线指的是最高5‰的门槛。(b),根据基因组坐标绘制XP-CLR评分;绿色横虚线为5‰的门槛。通过取两种方法给出的edr的交集得到edr。(a)和(b)中的垂直虚线表示两条染色体之间的边界。多克斯(278 KB)
EDR扫描indica型。
补充文件25:(a)F圣根据基因组的物理坐标绘制了旱地和灌溉籼稻之间的值;红色水平虚线指的是最高5‰的门槛。(b),根据基因组坐标绘制XP-CLR评分;绿色横虚线为5‰的门槛。通过取两种方法给出的edr的交集得到edr。(a)和(b)中的垂直虚线表示两条染色体之间的边界。多克斯(222 KB)
12870 _2014_1574_moesm26_esm.docx
附加文件26:来自籼稻XP-CLR顶端信号的9个基因与合并群体先前的edg重叠。(多克斯12 KB)
12870 _2014_1574_moesm27_esm.docx
附加文件27:来自籼稻XP-CLR顶部信号的4个基因与粳稻种群先前的edg重叠。(多克斯11 KB)
12870 _2014_1574_moesm28_esm.xls
附加文件28:用于计算任意两个访问之间的成对距离的信息性站点的数量。(XLS 254 KB)
权利和权限
本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(http://creativecommons.org/licenses/by/4.0),它允许在任何媒体上不受限制地使用、分发和复制,只要原始作品的名称正确。创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条提供的资料。
关于这篇文章
引用这篇文章
吕杰、李斌、何伟、李晓燕、李晓燕、李晓燕。et al。水稻旱地适应重要遗传机制的基因组分析。BMC植物杂志14日,160(2014)。https://doi.org/10.1186/1471-2229-14-160
收到了:
接受:
发表:
关键字
- 旱稻
- 高地适应
- 遗传机制
- 系统发生学
- 人口结构
- 人工选择