摘要
背景
大豆是最重要的作物之一,提供了大量的膳食蛋白质和食用油,也是研究重复基因进化的极好模型。然而,相对于模型植物拟南芥在水稻方面,目前关于大豆转录组的知识非常有限。
结果
在本研究中,我们利用RNA-seq技术研究了大豆11个组织的转录组,以期在全基因组范围内发现真正表达的基因、新的和替代的转录本,并分析了重复基因的保守性和差异性及其功能意义。共检测到54132个高可信表达基因,并鉴定出6,718个新转录区,平均长度为372 bp。我们还为15.9%的具有两个或两个以上外显子的基因提供了选择性剪接(AS)事件的有力证据。其中,1834个基因表现为阶段依赖性AS, 202个基因存在组织偏向的外显子跳跃事件。我们进一步从近期的全基因组复制(WGDs)中定义了重复基因对之间表达模式的保守性和差异性;在茎尖分生组织和花发育过程中发现了差异表达基因、组织优先表达基因、转录因子和特异基因家族成员。
结论
本研究结果显著提高了大豆基因注释功能,为大豆WGDs重复基因的功能基因组学和进化研究提供了有价值的资源。
背景
豆科植物是三个最大的开花植物家族之一,从大约5000万年前的一个共同祖先(mya)分化而来,是生物固氮的主要参与者,对农业系统有重要贡献[1].大豆(大豆(l)稳定。]is the most important crop among legumes, providing ~70% dietary proteins and ~30% edible oil [2].大豆有20对染色体,预测基因组大小为1115 mb [3.是一个具有两个谱系特异性全基因组复制(WGD)的古多倍体。大豆史上最近的一次WGD发生在大约1300万年前(mya) [4,比模型工厂历史上的那些更近拟南芥和米饭。最近测序的大豆基因组中有950兆酶(Mb)(约占估计总序列的85%)的组装序列显示,由于WGD,最近出现了数千个类似序列[4],使其成为研究重复基因进化的一个极好的模型。
基因组序列可以注释6.6万个以上的基因,其中4.6430万个被指定为高置信度基因,2万个被生物信息预测为低置信度基因[4].最近的转录组数据提供了证据,证明大豆总共有55,616个转录本[5].相对近期的WGD和串联重复(TD)已经产生了一个大约75%的基因是多基因家族成员的基因组[4,6,7].特别是在46430个高置信度基因中,有15632组2-6个相近的类似基因,包括串联重复基因,而其他15166个基因为单拷贝[4].最近的一项研究将重复基因更新到17547对/组,其中8910对来自最新的WGD [8].此外,大豆基因组有38581个重复元件,占基因组的59%,这涵盖了该植物大部分类型的转座子[9].然而,大豆基因组中的基因注释还不完整,可以利用全基因组的基因表达信息进行进一步的改进,包括检测新的转录区域和选择性剪接事件。
近年来高通量RNA测序(RNA-seq)技术的发展极大地提高了转录组学的敏感性,使得无需先验基因模型就能检测转录本[10- - - - - -12].RNA-seq已被广泛应用,并成功地探索全基因组表达模式,识别新的转录本,检测替代剪接事件和反剪接RNA,从酵母到人类[13- - - - - -16].转录组学也在模型植物中广泛应用拟南芥对于水稻,在特定组织甚至单细胞类型的水平上,以及对新转录区域和剪接模式的识别[17- - - - - -22].它也越来越多地应用于其他植物物种,如玉蜀黍属米投赞成票者(23)、小麦(24),草莓属vesca[25,以及大豆[5,8,26,27].然而,目前对大豆转录组的认识还不完全。例如,大豆基因组中许多预测基因尚未得到表达信息的支持;此外,对大豆可变剪接事件的模式所知相对较少。在本研究中,我们对11个大豆组织进行了RNA-seq测序,获得了大量的数据集,用于发现新的转录区域和剪接转录本、组织优先表达或差异表达基因、转录因子、复制基因对之间表达模式的保守和差异,以及比较转录组分析的功能意义。
结果与讨论
RNA-seq揭示了大豆中约54000个转录活性基因
分析大豆(g·马克斯)转录组,就像我们之前所做的那样拟南芥而且斑马鱼[21,28,29],我们收集了大豆的11个组织,包括根尖、下胚轴、子叶、愈伤组织、第6、17和38天的茎尖分生组织(为方便称为SAM6D、SAM17D和SAM38D),以及腋生分生组织(简称AM)、减数分裂前后的花序(简称IBM和IAM,类似于拟南芥分别在第1-9和9-12阶段的花序)和开放的花朵(简称OF),每个样本获得了111 - 3.26亿条~50 bp的reads,比之前的大豆RNA-seq研究多了~30-50倍[5,30.].其中52.3% ~ 71.6%的reads被映射到g·马克斯参考基因组(4], ~90%的reads与标注的大豆基因相匹配(见附加文件1:图S1a和附加文件2:表S1)。此外,reads的基因分布显示,75%的reads与外显子对应,而其余的reads分布在内含子(10%)、基因间区(7%)和剪接区(8%)(见附加文件)1:图S1b和附加文件2:表S2)。因此,我们的RNA-seq为进一步研究大豆转录组提供了高质量的数据。
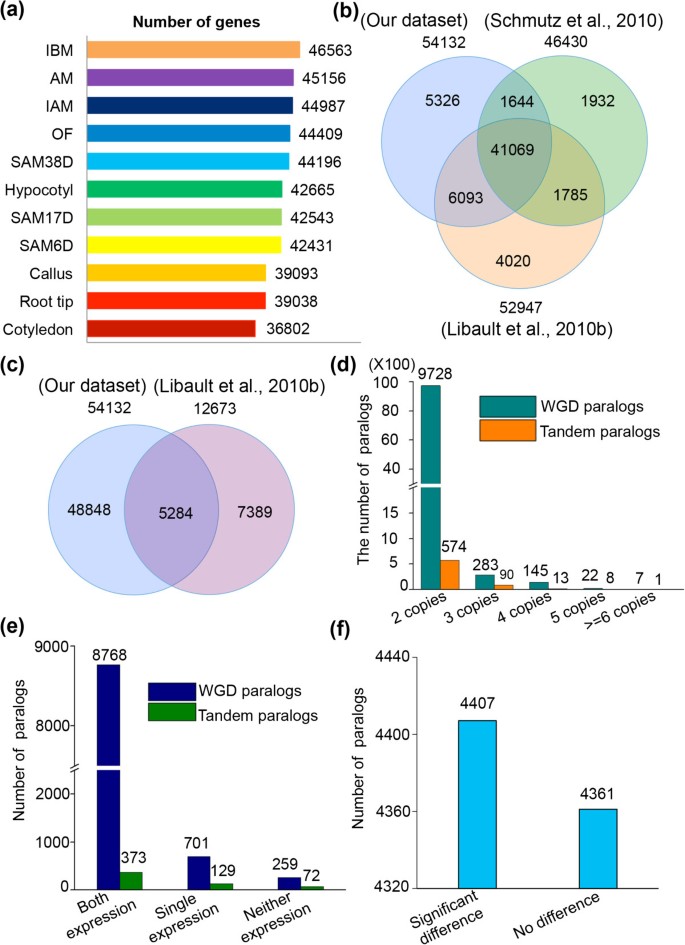
为了估计在被检测的大豆组织中表达的基因数量,我们首先使用RPKM方法的变异对基因表达值进行归一化(R欧洲航空防务与航天公司P呃Kilo-base mRNA长度per米百万次映射读取)[13,31,32],通过比较基因表达水平和基因间区域的表达水平,从实验的背景噪声中区分出可靠的基因表达信号(见附加文件1:图S2,参见Methods)。我们在11个样本中至少检测到54132个表达基因(log2 (RPKM)≥-2),占66210个标注大豆基因的81.8% [4].在不同组织中检测到的基因数量变化很大,在36,802到46,563之间(图1A),在活性组织中检测到更多的基因,这与水稻的结果一致[18].与最近在大豆中检测到的52,947个表达基因相比[5],其中47162个基因在我们的数据集中被识别,5805个基因未被纳入(图1b),而我们的数据检测到另外6970个表达基因,在52,947个基因中不存在(图1b)在先前定义的46,430个高置信度基因中[4]时,42713个(92%)基因在我们的数据集中具有转录活性,而3717个(8%)基因未检测到(图2)1b).相反,我们的数据还检测到额外的11419个先前被定义为低置信基因的基因[4],包括最近被指定为假基因的12673个基因中的5284个[5)(图1C),表明与多个组织耦合的超高通量测序能够识别更多的低水平或组织优先表达基因。综合本研究和前人数据,共转录了61849个基因(接近大豆基因组中注释的基因),显著提高了大豆转录组注释水平。
11个组织中检测到的基因及重复基因的分析。(一)11个组织中各检测到的基因数。SAM6D、SAM17D和SAM38D分别为萌发后6、17和38 d的茎尖分生组织;IBM和IAM为减数分裂前后的花序;:开放的花朵;问:腋分生组织。(b)54132个检测基因与46430个高信度基因的比较(Schmutz等人[4])和此前报道的大豆中52,947个转录本(Libault等[30.])。(c)将54132个检测到的基因与先前指定的12673个大豆假基因进行比较(Libault等人[30.])。(d)基于66210个大豆基因组预测的WGD和TD位点分布在2-6个拷贝。(e)54132个基因中有9728个WGD基因同源性和574个TD基因同源性成对分布。(f)T-检验分析8768个检测到的对照语中两个对照语之间表达水平的显著性差异。
最新WGD引起的重复基因分析
基因复制是理解进化新颖性的重要机制之一,而复制基因表达的差异与其功能差异高度相关[33].近年来,由于大豆发生了两起萎蔫病,大豆已成为研究这方面的一种极具吸引力的模式植物。基于大豆基因组中标注的基因,我们鉴定出TD和WGD引起的重复基因分别为2713和37746个(2 ~ 6拷贝)。这些基因按拷贝数2 (9728/WGD和574/TD)、3 (283/WGD和90/TD)、4 (145/WGD和13/TD)、5 (22/WGD和8/TD)和6或更多(7/WGD和1/TD)进一步分为5种类型(图1)1d).我们的11个样本经WGD和TD检测出重复基因35569个(94.23%)和2139个(78.84%),表明WGD现有的重复基因绝大多数都得到了表达。为了进一步研究重复基因间的表达差异,我们对来自WGD的9728对同源基因进行了分析。我们的结果显示,8768对在两个拷贝中都有表达,701对在一个拷贝中表达,而259对在两个拷贝中都没有表达1e).在8768个双拷贝表达基因中(除非另有说明,下文中提到的配对指的是对),t-检验统计分析显示,有4407个(50.26%)在两组间表达差异显著(p < 0.05)(图1f和在附加文件中2:表S3),表明监管子功能化和/或新功能化,而其他4361个组(49.74%)彼此之间没有显著差异(p < 0.05)(图3)1f,在附加文件中2:表S3),建议功能守恒和两个论证之间可能存在的冗余。此外,701对单拷贝表达的一个拷贝缺乏表达,表明它们可能是调控非功能化的候选对象,尽管其中一些可能是亚/新功能化的额外例子,因为它们可能在这里没有取样的其他组织或在不同的生长条件下表达。574个TD基因也发现了类似的趋势1e)。
转录组分析鉴定出大豆中约6718个高信度NTRs
RNA-Seq已被广泛应用于鉴定NTRs酿酒酵母而且美国非洲酒[13,34),拟南芥[35)、大米(19,22,鼠标36和human [37].我们的转录组数据显示,大量的reads被映射到注释的基因间区域,这表明它们是潜在的NTRs。因此,我们组装了映射读取,获得了19752个NTRs。通过严格要求>的大小为150bp,读取数为>10,并在至少两个样本中检测到,我们总共获得了6718个高可信的NTRs,平均长度为~372 bp,其中2265个之前报道过[5].
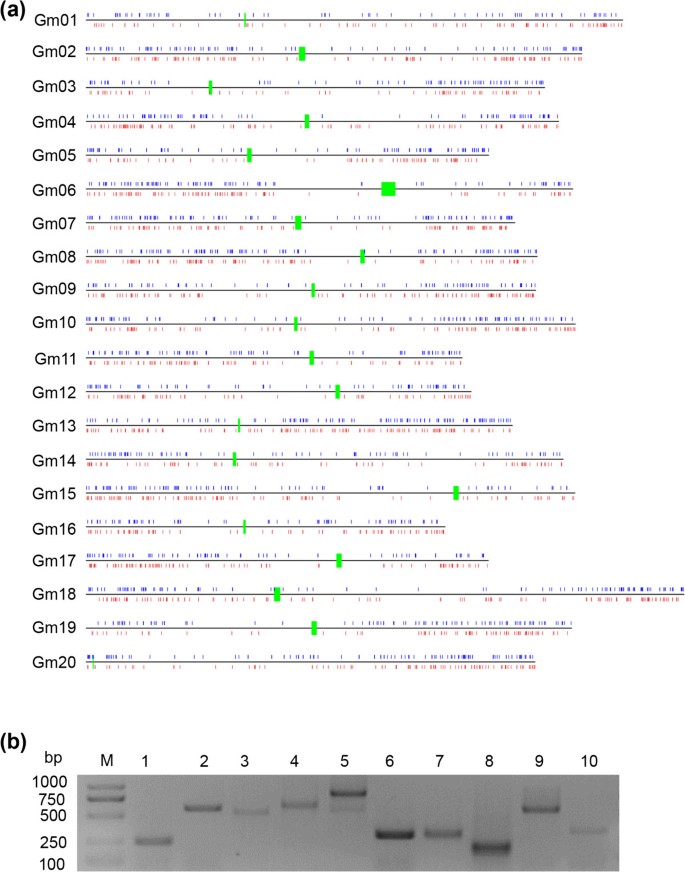
据报道,NTRs可能是新的基因或代表附近注释转录本的延伸,可能构成新的外显子。为了验证第二种可能性,我们在距离假设的NTRs相同转录方向的近距离(405 bp)内寻找注释基因,发现6718个NTRs中有1509个被检测到通过内部脚本扩展注释基因的5'UTR(见附加文件)2:表S4)。对这些新的和扩展的utr的进一步分析将有助于识别额外的调控元素。除了1509个扩展基因外,其他5209个NTRs被组装成4949个新的转录单位(nTUs),平均分布在20条染色体上,但富集于染色体臂(图1)2a和在附加文件中2:表S5)。此外,随机选择的10个NTRs通过RT-PCR验证为真实转录本(图2b),进一步支持已识别的NTRs的可靠性。
在4949个ntu中,有2326个(47%)得到了国家生物技术信息中心(NCBI)标注的1532条大豆est(附加文件)的支持2:表S6),但目前没有在g·马克斯基因组。在其他2623个ntu中,有698个(53%)被发现与其他物种有同源性(见附加文件)2:表S7),提示它们可能是保守基因。只有47个ntu位于转座元件(TE)区域,表明TE活性(在附加文件中)2:表S8)。为了从2623个ntu中识别潜在的非编码RNA,我们使用ntu与Rfam进行BLASTN比对,发现40个ntu的非编码RNA注释为tRNA、rRNA、snoRNA或miRNA(见附加文件)2:表S9)。例如,XLOC_015015被标注为miR159,这表明一些新型的ntu具有非编码rna的功能。其余国立大学的性质需要进一步调查。
然后,我们分析了4949个ntu在11个组织中的时空分布(见附加文件)1:图S3),发现其中1393个表达为构成型表达,3556个表达为组织优先表达。有趣的是,目前的大豆基因组只注释了一个CLAVATA1A(CLV1A的同源基因拟南芥CLV1基因调节分生组织大小[38,而识别XLOC_047893南洋理工大学是类似于CLV1A在大豆。这两个基因在SAM17D和SAM38D中均有特异性表达,表明存在冗余功能CLV1A而且XLOC_047893用于调控大豆中的SAM。
选择性剪接转录本及其差异表达
AS是高等生物产生蛋白质组学和功能复杂性的主要贡献者之一[16],但目前对大豆中的AS事件知之甚少。在之前注释的66210个大豆基因中,52460个基因至少有两个外显子[4].我们在11个样本中共鉴定了12,810个AS事件,覆盖了7,084个基因(包括504个类似位点)(在附加文件中)2: Table S10),表明~15.9%的多外显子基因具有AS模式。这一比例明显低于两项研究中观察到的48%拟南芥或大米(19,20.,22].一个可能的原因是大豆最近经历了两次基因组复制,这导致了许多保留的重复基因,这也是蛋白质组学和功能复杂性的主要来源[39].
我们还总结了大豆中可能存在的11种AS类型,包括内含子保留型(32.2%)、ES型(26.3%)、A3SS型(20.8%)、A5SS型(11.2%)4种常见类型(Table)1).不同于动物体内主要的ES类型[15,16],内含子保留是大豆中AS的主要类型,这与在拟南芥、水稻、玉米及大豆[19,20.,23,40].本研究和其他研究结果表明,植物中IR的调控机制是保守的。大豆中ES事件的比例较高(26.3%),这也与水稻和玉米中相同[19,23],但明显高于拟南芥.有报道称ES参与调节组织特异性功能[16].为了研究AS的组织特异性表达,我们进行了MISO程序分析[41]来识别202个组织偏向外显子跳过事件,包括2个类似事件(在附加文件中2:表S11)。它们大多编码蛋白质降解、RNA调控、信号转导和转运的酶和转录因子。我们还发现,一些外显子在一个组织中主要是外显子,在另一个组织中主要是内含子。例如,如图所示3.A,第7外显子Gm15g15960,结果显示Ψ,根尖占88%,子叶占6%(见附加文件)2:表S11),表明根尖和子叶的功能不同。此外,在SAM和花的发展过程中,1834个AS事件发生了很大的变化(图3.b和在附加文件2:表S12), GO分析表明,许多编码蛋白质的基因参与了生殖发育过程。除了已知的开花基因表现出AS变化外,许多未被表征的基因也被观察到有显著的AS变化,例如Gm05g28120,该基因有三组表达模式相互排斥的外显子(Figure3.c)。
组织转录组的比较显示了保护和分化
为了研究这11种组织的相似性,我们比较了它们的转录组,并根据任意两种转录组之间的Pearson相关系数绘制了热图(图2)4).根尖与of之间的系数最低,为0.62,AM与SAM38D之间的系数最高,为0.922:表向)。我们进一步用层次聚类(HCL)将11个样品分为4组:(I)根尖和愈伤组织;(II)子叶和下胚轴;(III) SAM6D、SAM17D、SAM38D和AM;(IV) IBM和IAM;(图(V)4).第I组的根尖与愈伤组织的相似性与之前的发现一致拟南芥愈伤组织,即使是来自于气生器官,在特定的基因表达谱方面类似于根顶端分生组织的发展[42].最近的一项研究进一步证实了这种相似性,因为叶子到愈伤组织的转变过程涉及到根偏好基因表达的表观激活[43].在大豆中观察到相似的转录组,表明在植物中决定愈伤组织形成的细胞命运的分子机制是保守的。子叶和下胚轴聚在一起,表明与其他组织相比,它们更相似4).与上述4种组织相比,其他7个样本被归为一个进化支,这得到了相近的Pearson相关系数值的支持,尤其是AM与SAM38D或IBM的相关系数值。对三种组织进行额外的比较,发现了1884个重叠基因(见附加文件1:图S4),主要参与花器官的确定、雄蕊发育等生殖细胞过程(见附加文件2:表S14),表明AM在这个发育状态下具有茎部AM和花分生组织之间的一些共同特征。综上所述,这些结果表明,器官识别和细胞命运决定高度受基因的时间和空间表达的调节。
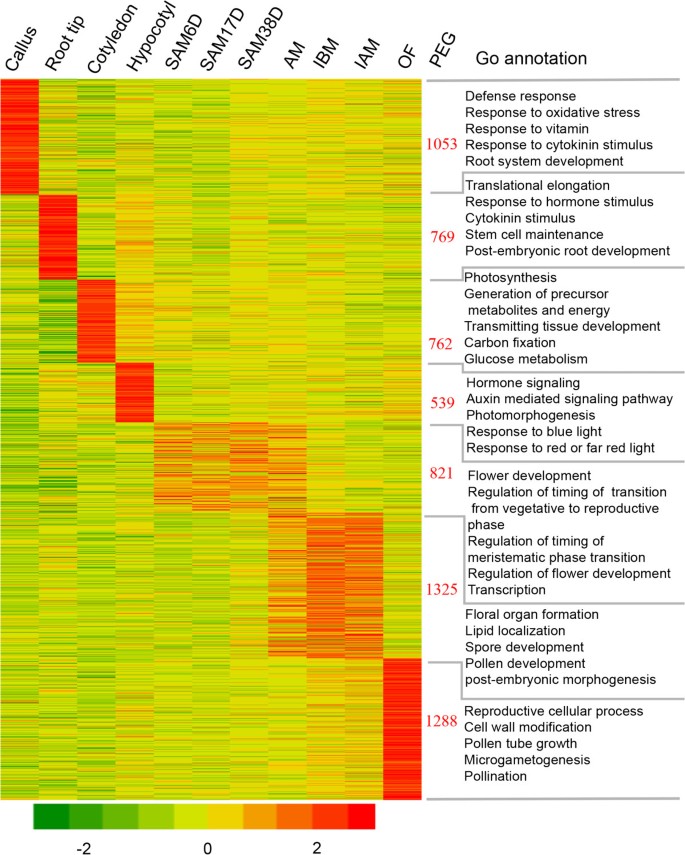
组织优先表达基因的鉴定
组织或细胞特异性基因的特性为揭示其分子机制提供了基础。以往对多种植物的研究表明,每个器官或组织都有其特定的转录本[18,21,44],包括大豆[5].这些基因在一个组织(或一组密切相关的组织)中表达高于所有其他组织,被称为优先表达基因(PEGs)。为了研究组织peg,我们首先比较了11个组织的转录组,发现了6557个组织peg(图5).在这些基因中,根尖有769个peg,包括65个parogh;GO注释显示,它们与翻译延伸、激素信号转导、细胞分裂素刺激、干细胞维持和胚胎后根发育相关(图4)5,在附加文件中2:表S15和S16)。在拟南芥,PIN2是根发育过程中生长素运输所必需的[45].两个类似的类比PIN2在大豆中发现,在根尖上也有类似的特异表达,表明它们在根系发育中可能与PIN2在拟南芥.相比之下,在愈伤组织中发现的1053个peg,包括102个转录因子和48个转录因子,主要参与生物和非生物反应,如防御、氧化应激、维生素、无定物和细胞分裂素刺激(图5,在附加文件中2:表S17和S18)。762个子叶peg(49个)在光合作用、能量、传输组织发育和糖代谢等方面富集(图4)5,在附加文件中2:表S19和S20)。生长素是子叶发育的重要调节剂[46].我们检测到其他几个生长素相关基因,包括一对Gm09g38700而且Glyma18g47630的同系物拟南芥pin - form5这是生长素平衡和配子体发育所必需的[47,48].然而,这两种基因都在子叶中表达量最高,但在生殖组织中几乎检测不到,这表明平快在大豆中可能有不同的作用。相比之下,539个下胚轴peg(27个近端)富集于生长素介导的信号通路和/或照片形态发生(图5,在附加文件中2:表S21和S22),包括拟南芥非嗜光下胚轴3基因(49].
如图所示4, AM与SAM38D和IBM高度相似,两两比较可能会漏掉许多活跃在分生组织中的基因。为了识别这些分生组织中的peg(但不是专门在其中一个分生组织中),我们将相似的分生组织分组在一起,检测到821个基因(20个类似基因)。GO注释表明,最丰富的种类与花的发育和调控、花从营养阶段到生殖阶段的转变,或分生组织阶段的转变和转录调控有关(图5,在附加文件中2:表S23和S24),这与之前关于大豆的报告一致[50,51].例如,peg中包含几个同源的营养期短(高级副总裁),确定生殖器官的身份和控制开花时间拟南芥和大米(52,53和基因参与WUSCHEL(本人)软件系统维修所必需的规管网络[54].我们还发现了同源基因(Gm14g15820而且Gm7g30920)的生长素合成和反应基因,如YUCCA4,根据这个事实拟南芥YUCCA4表达仅限于SAM和花分生组织或幼花原基[55],以及20个与调节SAM发育的生长素应答基因相关的基因[56].我们的GO富集结果与分生组织中已知功能之间的良好一致性表明,所收集的样品用于SAM的可靠性和控制SAM之间的分子机制的保守性拟南芥和大豆。
相应地,AM、IBM和IAM总共有1325个peg(60个paralogs)5,在附加文件中2:表S25),主要参与花器官的确定和发育、雄蕊发育、绒毡层发育、花粉发育等生殖过程(Figure S25)5).例如,除了从ABC模型中鉴定几个花器官的身份基因外(在附加文件中)2: Table S26),我们还发现了减数分裂的特异性基因,如5级(Gm08g47070而且Gm18g38060),MMD1(Gm14g39310而且Gm02g41020)[57].不同于拟南芥5级而且MMD1在减数细胞中受限的基因中,大豆同源基因在AM中高表达,这表明其在大豆中的功能可能未知。有趣的是,拟南芥DREB1B是冷反应的关键调控因子之一,也被广泛表达[58],而大豆同源物(Gm11g19340而且Gm12g09130)在AM、IBM和IAM中有特殊的表达,而在其他营养组织中没有,这表明它可能在大豆生殖发育中获得了新的功能。另外,还有一个同源的DREB1A(Gm17g14110)也被确认,这与最近的一项新发现一致拟南芥DREB1A基因对花的发育很重要,尤其是在不利条件下[59].
最终,开放花有1,288个peg(78个),富集于生殖细胞过程、细胞壁修饰、花粉管生长、授粉和信号转导(图1)5,在附加文件中2:表S27及S28)。特别是,至少有50个基因(大多数在两个拷贝中)编码花粉和子房相互作用的信号转导蛋白,如124年提前受体,富亮氨酸重复蛋白激酶,Rop结合蛋白激酶,钙依赖性蛋白激酶[60,61].
大豆SAM转录组的动态重编程
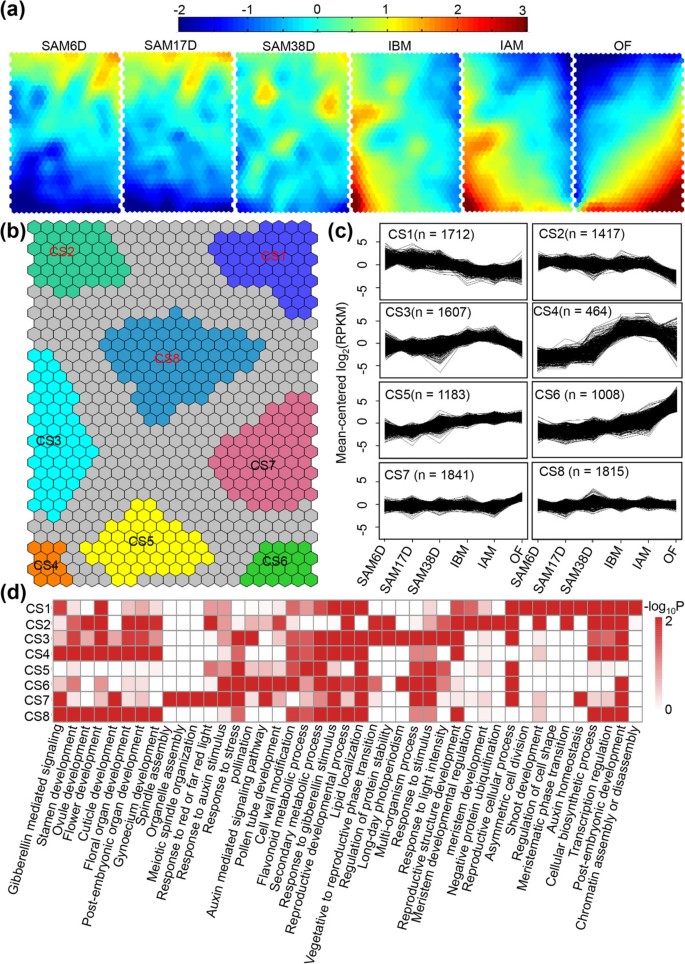
大豆与拟南芥为SAM发育过程中关键基因的保存提供了线索。为了获得更清晰的SAM发育过程中的转录组变化,我们主要关注大豆SAM和花发育过程中的22,571个DEGs(在附加文件中)2:表S29)。通过qRT-PCR对随机选取的20个基因在SAM中的表达进行验证,结果显示二者具有较高的相关性(R2= 0.93)与RNA-seq(在附加文件2:表S30),支持我们数据集的可靠性。然后我们应用了自组织地图(SOMs) [62],以寻求与发育阶段相关的deg的共同模式(图6a),随后根据类似的共享模式确定了8个区域(CS1-CS8)(图6b和在附加文件2:表S31)。其中,CS1基因在SAMs早期的表达水平高于平均水平,而在IBM、IAM和of后期的表达水平低于平均水平(图1)6c),表明它们对SAM的早期发育很重要,而不是之后。GO富集分析显示,这些基因主要参与染色质组装和拆卸、转录调控、分生组织相变时间调控、细胞不对称分裂和生长素稳态(图1)6d和在附加文件中2:表S32),提示转录调控在SAM早期发育中起重要作用。CS2和CS7的基因在除OF外的5个早期生殖组织中均有稳定表达,但在OF中分别出现大幅表达下降和表达增加(图7)6c).这说明CS2基因在花的早期发育中起作用,但在花的后期发育中作用不明显。该表达基因主要参与分生组织发育、生殖结构发育和转录调控,也参与蛋白质泛素化的负调控(图6d和在附加文件中2:表S32)。相反,CS7基因在花发育后期更加活跃。这些基因参与了对刺激的反应、生长素信号、脂质定位和纺锤体组织(图6d和在附加文件中2:表S32)。CS3和CS4的基因表现出相似的表达模式,从SAM38D到IAM增加,然后OF下降,但CS3的增加水平远高于CS4(图4)6C),表明这些基因可能对减数分裂后的花发育很重要。的确,GO分析丰富了基因在生殖发育中的作用(图6d和在附加文件中2表S32),包括ABC模型和花药或花粉发育所需的基因。CS5和CS6基因在SAM组织(SAM6D、SAM17D、SAM38D)和生殖组织(IBM、IAM、of)中表达量随发育阶段逐渐增加,CS6基因在of中的表达量高于CS5基因(图3)6c). GO富集分析表明,CS6中OF表达上调的基因在花粉管发育和授粉等生殖发育后期过程中更加活跃(图6)6d和在附加文件中2:表S32)。CS8的基因在6种组织中呈结构性表达,部分基因在SAM38D中呈波动性表达(图6c).这些基因不仅对细胞的基本发育很重要,而且对分生组织和花的发育也很重要(图6d和在附加文件中2:表S32)。因此,对不同簇的基因进行进一步的功能研究有助于更好地理解它们在大豆SAM和花发育过程中的生物学意义。
转录因子在SAM中表达明显
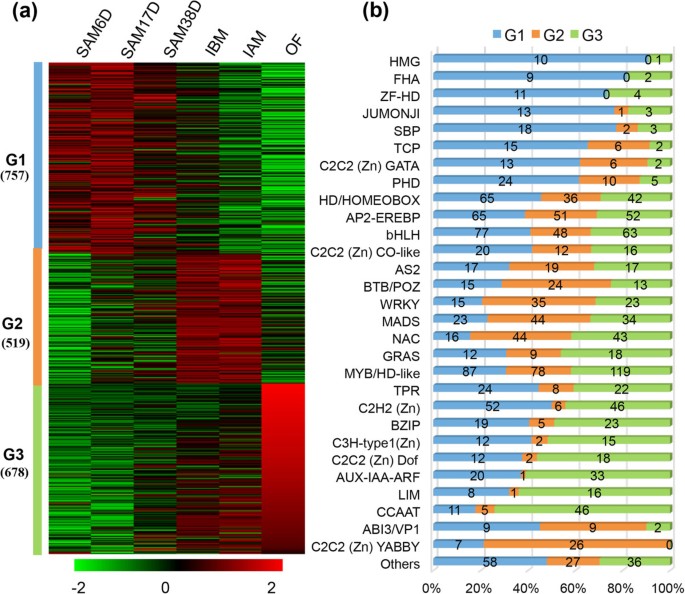
确定大豆SAM和花发育过程中动态积累的TFs是了解其潜在调控网络的第一步。目前大豆基因组中注释了5671个TF基因,分别属于63个不同的家族[4].我们在六个样本(SAM6D, SAM17D, SAM38D, IBM, IAM, of)中至少一个中检测到4806个TF基因(642个类似位点)表达。其中有1954个(141个组)差异表达(GFOLD >1或GFOLD < -1;RPKM >(图1)7a和在附加文件中2:表S33),揭示了几乎所有的家庭。然后我们根据不同的表达模式将1954个TF基因分为三个簇(图7a).这些TF基因在sam(指定为G1)、IBM和IAM(指定为G2)或of(指定为G3)中分别表达量最高的分别为39.8%、29.6%和30.6%(图3)7b)。
进一步的家族优先表达分类显示,G1主要包括HMG、FHA、ZF-HD、SBP、TCP、C2C2 (Zn) GATA和PHD家族(图2)7b),表明SAM的早期发育在很大程度上需要这些转录因子家族。例如,鳞状细胞启动子结合蛋白样蛋白质是一个具有保守的SBP (SQUAMOSA启动子结合蛋白)结构域,在植物生长发育中发挥多种作用[63].16 - 48:被发现在拟南芥和大豆,分别[64],并被分为八个进化枝(在附加文件中1:图S5和附加文件2:表S34-S35)。23:7个分枝在大豆SAM和花发育过程中均有差异表达。可用的数据拟南芥,水稻和番茄支持了这样一种观点,即来自一些不同支系的基因功能可能仍然保持不变,但来自其他支系的基因可能已经分化了[63- - - - - -65].例如,16个中的10个拟南芥:(SPL2-6、SPL9-11 SPL13,而且SPL15)来自5个进化枝miR156/157目标(66,并在相变中起类似的作用[65],而包含进化支I-、II-和iii的基因则缺乏miR156而且miR157绑定的网站。只包含进化枝ISPL7在调节铜的体内平衡中具有广泛的表达和独特的功能[67].一致,两个大豆SPL7类似的模式也被广泛表达,表明它在大豆中的保守作用(图8).演化支II有四个成员SPL1、SPL12 SPL14而且SPL16广泛表达于拟南芥(图8),但只AtSPL14已经被证明参与了从营养到生殖的转变[63].这个分支包括大豆中的八个成员。其中7人的表达模式与对照组相似拟南芥(图8在附加文件中2:表S36),但仅限Gm17g04400在SAM中有差异表达,表明其功能不同于其对应物。进化支III只有AtSPL8在根生长和小孢子发生中起作用[68].在这支分支中发现了4个大豆基因,其中2个没有表达,可能是非功能化的。另外两个基因在SAM和生殖组织中特异表达。但是,与AtSPL8与番茄相似,大豆的同源物在根中不表达,但在根中具有功能SPL8同源染色体(64].进化支IV包含AtSPL6具有本构表达式和未知函数.然而,一个Physcomitrella金属盘据报道同源物会抑制生殖发育[69,某种程度上类似于AtSPL14[70].该分枝具有6个大豆同源性;除了无法察觉的表情Gm06g22450,其余5个基因在SAM和生殖组织中高表达。与其他7个支系相比,大豆支系中来自大豆的基因较多拟南芥在拟南芥中,进化支V有3个基因,但只有2个同源基因来自大豆(图8).因此,研究导致该支系大豆基因丢失的可能原因将是一项有趣的研究。
大豆热图分析SPL转录因子家族存在于11种组织中。在附加文件中对I-VIII支进行了系统发育分析1:图S8。只有拟南芥列出各进化支的基因作为参考。用相同颜色标记的基因名称是一对配对。
最广泛特征的功能:是促进从营养生长过渡到生殖生长,特别是为SPL3-5在进化枝VI中拟南芥[71].值得注意的是,这个分支包含15个:其中14个在SAM中高表达(图8),在其他组织中几乎检测不到,这表明分子机制在调节从营养生长到生殖生长之间的转变中是保守的拟南芥和大豆。第七和第八的最后两个分支包括AtSPL13而且AtSPL9/15,分别在附加文件中1:图S5)。AtSPL13与叶片发育有关,而AtSPL9而且AtSPL15在相变中起部分冗余作用[72,73].七和四SPL分枝VII和分枝VIII的基因在SAM和花组织中的表达模式非常相似,与大豆的功能一致拟南芥同源染色体。总共有7对陪练被包括在内SPL家庭(图8).表达模式的比较表明,一对同源基因可能经历了亚功能化,进一步支持了亚功能化可能是大豆WGD后重复基因的主要事件的观点。
与G1不同,G2主要包含MADS、AS、BTB/POZ、WRKY、C2C2 (Zn) YABBY(图1)7b).有报道称MADS-box基因家族不仅是开花转变的关键抑制因子或激活因子,而且是生殖器官特性的主要调控因子[74].我们的数据在花发育过程中检测到101个MADS-box基因7b),等Gm01g08150、Gm04g42420 Gm08g12730而且Gm08g27670的同系物AP1,π,AG)而且SEP2,这与它们在花器官特性中的已知功能一致。因此,MADS-box基因家族对花发育的调控功能在大豆和大豆之间可能是保守的拟南芥.相比之下,BZIP、C3H-type1 (Zn)、C2H2 (Zn) Dof、AUX-IAA-ARF、LIM和CCAAT基因家族在OF中优先表达(图7b).许多研究表明,生长素依赖的转录调控需要TFs的生长素/吲哚-3-乙酸(Aux/IAA)和生长素反应因子(ARF)家族[75和Aux/IAA-ARFs异质二聚体的形成抑制生长素信号传导[75].除了已知的生长素在拟南芥花粉发育、授粉和受精似乎也需要增加生长素水平[76].事实上,我们在OF中检测到33个差异表达成员,这表明Aux/IAA-ARF调控通路在后期生殖发育中也是保守的。然而,of中其他富集的TFs的功能仍在很大程度上未知。
结论
大豆基因组的古多倍体性和快速分化使其成为一种理想的进化分析基因组。然而,目前的大豆基因组注释和基因表达信息不完整。这项研究展示了大豆的整体转录格局,并提供了大量证据,表明大豆的转录调控比之前预期的要复杂得多。该数据显著提高了基因组预测大豆基因的注释,为研究最新WGD与大豆功能基因组之间的重复基因表达水平提供了必要的数据来源。
方法
植物材料及生长状况
本研究使用的大豆(Glycine max)植物材料均来自HX3品种。萌发后3天,在石英砂培养基上,在28°C(光照)/25°C(黑暗)条件下,在70%相对湿度下,在14 h/10 h光照/黑暗条件下培养出较老的幼苗,用于获得0.2-0.3 cm长的根尖。同样,用制备好的4天幼苗收集子叶和下胚轴。用镊子和解剖针分别采集萌发后6、17和38天的土壤生长植株的茎尖分生组织(sam)。在萌发38天后,在土壤生长的植株茎尖第2或第3节间收集腋生分生组织。每个分生组织RNA-seq样本包含大约1000株植物的材料。对于减数分裂前后的花序,我们通过四分体和染色体分布来确定合适的花序大小,然后在显微镜下解剖土壤生长45天的植株的花序,并分离出开放的花和未开放的芽。愈伤组织诱导采用子叶-节点法,如前所述[77略作修改[78].所有样品均在室温25°C下采集,并迅速置于液氮中。
RNA分离,RNA序列文库制备和测序,实时RT-PCR
采用前述方案进行RNA分离、RNA-seq文库制备和测序[21,28,29].RT-PCR按原程序进行[21,29].本研究中使用的引物列在附加文件中2:表S30。通过将每个发育阶段的Ct值与内源性对照(Gmβ-actin:Gm15g05570)使用2-ΔΔCt方法(79].
读取图谱绘制和基因表达水平计算
通过固态测序获得的Reads与大豆基因组装配版本9 (Glyma1.1;http://www.phytozome.net/),使用Lifescope软件包。Lifescope使用一种种子和扩展方法来根据参考进行映射读取。通过GFOLD V1.0.7软件计算归一化的基因表达水平为:Reads Per Kilo-base of mRNA length Per Millions of mapped Reads (RPKM) [80].比较基因和基因间区域的表达水平,以找到在上述背景下可检测表达的阈值。0.25 RPKM的值是将注释基因分为两个大簇的阈值,并定义为“表达”和“未表达”之间的阈值。然后,用GFOLD差异程序(GFOLD >1或GFOLD < -1;Log2 (fold change) >2 or Log2 (fold change) < -2)。与所有其他组织相比,特定组织的优先表达基因是通过在相关组织中至少满足GFOLD >1和RPKM > 4来确定的。
鉴定假定的相似性和差异表达分析
我们使用MCScanx软件[81]以识别潜在的类似集群。采用默认参数检测WGD基因和TD基因。根据11个样本和的log2归一化RPKM值,分析各组的微分表达t-检验评估统计学意义。
相关分析
采用R软件制作相关矩阵,Pearson相关系数作为统计度量,比较11份样本54132个基因的全转录组值。使用RNA-seq数据集的log2归一化RPKM值创建相关矩阵,然后使用R脚本分析样本之间的相关性。相关系数值转换为距离来定义树状图的高度尺度。相关性的热图由pheatmap包中的pheatmap (Pretty Heatmaps)函数实现(R版本,2.15,pheatmap版本,0.6.1;R核心团队,维也纳,奥地利)。
NTRs的发现和RT-PCR验证
我们使用Cufflinks软件[82使用Lifescope提供的高质量的图谱读取(无错配)进行装配转录本的比对,并根据类代码“u”比较标注的大豆基因组(http://www.plantgdb.org/GmGDB/),使用以下标准:(1)大于150bp的大小,(2)读取编号> 10和(3)在至少两个组织样本中检测支持。基于这些标准,我们得到了约6718个高置信度的NTRs。利用inGap软件对大豆基因组上的RNA-seq reads进行可视化[83].随机选取10个NTRs,使用本研究设计的特异性引物进行RT-PCR验证(见附加文件)2:表S37)。此外,BLAST还用于识别与Rfam相对应的ntu [84,85].
可变剪接分析
我们使用ASTALAVISTA软件[82]来根据Cufflinks软件收集的转录文本来量化AS事件的类型。味噌(41]和MISO管道分别用于评估11个样本的表达转录本及其差异表达。首先,我们需要生成两个文件库:可选拼接事件的注释文件和索引对齐文件。对于AS事件文件,我们使用MISO通过贝叶斯推断来度量差分表达式。在比对文件中,利用大豆基因组特征文件,将高质量过滤的不同样品reads与大豆基因组进行比对,以提高对拼接连接的检测。通过测试不同的截止和滤波器的组合产生MISO输出,最终使用贝叶斯系数0.7作为截止值来检测差分as事件。使用RPKM的刺身作图工具在大豆基因组上可视化RNA-seq读取。
自组织映射
我们使用SOM(自组织地图)方法[86]用于聚类和可视化SAM和花发育过程中DEGs的模式。使用赫尔辛基理工大学信息与计算机科学实验室开发的SOM MATLAB工具箱(http://www.cis.hut.fi/projects/somtoolbox/).一个SOM拟合了来自特定发育阶段/组织数据的平均归一化log2转化(RPKM值)基因表达估计。随后,通过对SOM单元进行k-means聚类(发育数据集k = 8),确定了SOM中与特征和连贯表达模式相对应的区域。更相干的SOM单元的上半部分是通过剪影系数确定的揭示集群。最后,我们可视化了每个SOM区域的原型基因表达模式。基因在这些区域中绘制出最匹配的SOM单元。
去富集分析
利用在线工具AgriGO (http://bioinfo.cau.edu.cn/agriGO/analysis.php)与Fisher精确检验和错误发现率(FDR)校正[87].从大豆基因组注释中下载转录因子(TF)家族注释,包含63个家族的5671个转录因子大豆[4].表达的tf的热图由热图生成。2function in the gplots package (R version, 2.15, R Core Team, Vienna, Austria). In addition, all gene functional descriptions were from modified MapMan annotations [88].
支持数据的可用性
支持本文结果的数据集可以在NCBI GenBank存储库中找到[http://www.ncbi.nlm.nih.gov/bioproject/?term=PRJNA241144]和NCBI SRA库中[http://www.ncbi.nlm.nih.gov/sra/?term=SRP040057].
缩写
- RPKM:
-
读取/ Kb /百万
- 走:
-
基因本体论
- RNA-Seq:
-
RNA序列
- 山姆:
-
技条顶端分生组织
- 聚合酶链反应:
-
聚合酶链反应
- 存在:
-
定量逆转录聚合酶链反应
- RNA:
-
核糖核酸
- TF:
-
转录因子
- 耶鲁大学管理学院:
-
自组织映射
- 为:
-
可变剪接
- 正常:关系
-
小说转录区域。
参考文献
王志强,刘志刚:大豆和其他豆类植物的古多倍体和基因复制。植物学报,2006,9(2):104-109。
林鸿茂,徐旭,刘旭,陈伟,杨光,王福林,李伟,何伟,秦宁,王波,李晶,建明,王晶,邵光,王晶,孙世盛,张光:31个野生和栽培大豆基因组的遗传多样性和选择模式的重测序分析。中国生物医学工程学报,2010,42(12):1053-1059。
陈晓明,张建军,张志强,陈志强,张志强,陈志强。荧光原位杂交技术在大豆染色体核型鉴定中的应用。中国生物医学工程学报,2010,30(3):327 - 327。
Schmutz J, Cannon SB, Schlueter J, Ma J, Mitros T, Nelson W, Hyten DL, Song Q, Thelen JJ, Cheng J, Xu D, Hellsten U, May GD, Yu Y, Sakurai T, Umezawa T, Bhattacharyya MK, Sandhu D, Valliyodan B, Lindquist E, Peto M, Grant D, Shu S, Goodstein D, Barry K, Futrell-Griggs M, Abernathy B, Du J, Tian Z, Zhu L,等:古多倍体大豆基因组序列。地球科学进展,2010,29(3):349 - 356。
李宝强,徐东,李志强,李志强,李志强,李志强,何军,徐东,李志强:水稻作物模型的转录组整合图谱大豆,以及在植物比较分析中的应用。植物学报,2010,63(1):86-99。
刘志强,刘志强,刘志强,刘志强,刘志强,刘志强,刘志强,刘志强。大豆基因复制和古多倍性及其对全基因组测序的影响。中国生物医学工程学报。2007,8:330-
张志强,李志强,张志强,张志强,张志强:大豆异源多倍体的分子遗传学证据。植物生理学报,2009,15(3):397 - 397。
刘琳,李宝强,李宝强,李宝强,刘志刚:多倍体植物基因组中重复基因的命运。植物学报,2012,33(1):1 - 5。
杜军,田智,Hans CS, Laten HM, Cannon SB, Jackson SA, Shoemaker RC,马军:开花植物ltr -逆转录转座子的进化保护、多样性和特异性:基于全基因组分析和多特异性比较的研究。植物学报,2010,30(4):584-598。
Sultan M, Schulz MH, Richard H, Magen A, Klingenhoff A, Scherf M, Seifert M, Borodina T, Soldatov A, Parkhomchuk D, Schmidt D, O’keeffe S, Haas S, Vingron M, Lehrach H, Yaspo ML:通过对人类转录组的深度测序对基因活性和选择性剪接的全局观点。科学。2008,321(5891):956-960。
Wang Z, Gerstein M, Snyder M: RNA-Seq:转录组学的革命性工具。热内。2009,10(1):57-63。
Martin JA, Wang Z:下一代转录组组装。中国科学:地球科学,2011,29(3):371 - 382。
Nagalakshmi U, Wang Z, Waern K, Shou C, Raha D, Gerstein M, Snyder M: RNA测序确定的酵母基因组转录格局。科学通报,2008,32(4):344- 349。
Aanes H, Winata CL, Lin CH, Chen JP, Srinivasan KG, Lee SG, Lim AY, Hajan HS, Collas P, Bourque G, Gong Z, Korzh V, Alestrom P, Mathavan S:斑马鱼mRNA测序破译了母体向合子转变过程中转录组动力学的新变化。中国生物医学工程学报,2011,21(8):1328-1338。
Graveley BR、Brooks AN、Carlson JW、Duff MO、Landolin JM、Yang L、Artieri CG、van Baren MJ、Boley N、Booth BW、Brown JB、Cherbas L、Davis CA、Dobin A、Li R、Lin W、Malone JH、Mattiuzzo NR、Miller D、Sturgill D、Tuch BB、Zaleski C、Zhang D、Blanchette M、Dudoit S、Eads B、Green RE、Hammonds A、Jiang L、Kapranov P等:发育转录组黑腹果蝇.自然,2011,471(7339):473-479。
Wang ET, Sandberg R, Luo S, Khrebtukova I, Zhang L, Mayr C, Kingsmore SF, Schroth GP, Burge CB:人类组织转录组中另类亚型调控。自然学报,2008,456(7221):470-476。
Filichkin SA, Priest HD, Givan SA, Shen R, Bryant DW, Fox SE, Wong WK, Mockler TC:选择性剪接的全基因组定位拟南芥.中国生物医学工程学报,2010,20(1):45-58。
焦艳,陶士达,刘涛,孙宁,刘涛,Clay NK, Ceserani T,陈明,马玲,Holford M,张海燕,赵慧,邓晓伟,Nelson T:水稻细胞类型转录组图谱揭示了细胞、功能和发育层次。热纳特。2009,41(2):258-263。
卢涛,卢国光,范丹,朱超,李伟,赵强,冯强,赵颖,郭勇,李伟,黄晓明,韩波:RNA-seq对水稻转录组单核苷酸解析的功能注释。中国生物医学工程学报,2010,20(9):1238-1249。
Marquez Y, Brown JWS, Simpson C, Barta A, Kalyna M:转录组调查显示拟南芥中选择性剪接环境的复杂性增加。中国生物医学工程学报,2012,22(6):1184-1195。
杨慧,卢萍,王勇,马慧:拟南芥雄性减数分裂细胞的高通量测序转录组图谱:减数分裂过程的复杂性和进化。植物学报,2011,65(4):503-516。
张国杰,郭国伟,胡晓东,张勇,李启元,李荣强,庄荣荣,陆兆坤,何志强,方晓东,陈丽,田伟,陶勇,Kristiansen K,张晓强,李国刚,杨鸿茂,王俊,王俊:水稻转录组的高复杂性和单碱基对分辨率的深度RNA测序。中国生物医学工程学报,2010,20(5):646-654。
李萍,刘倩,孙倩,王玲,杨思,陶斯达,凯brom TH, proart N, Patel R, Myers CR, Reidel EJ, Turgeon R,刘萍,孙倩,Nelson T, Brutnell TP:玉米叶片转录组发育动态。热纳特。2010,42(12):1060-1067。
刘志强,刘志强,刘志强,刘志强,刘志强,刘志强。四倍体小麦转录组同源性的研究进展。中国生物医学工程学报,2013,14 (6):R66-
刘志刚,刘志刚,刘志刚,刘志刚:森林草莓早期果实发育的基因组转录组研究草莓属vesca.植物细胞。2013,25(6):1960-1978。
刘丽萍,刘文斌,张晓燕:大豆花启动过程中组蛋白修饰基因和RNA沉默基因的RNA-seq转录组分析。科学通报,2013,8 (10):e77502-
Severin AJ, Woody JL, Bolon YT, Joseph B, Diers BW, Farmer AD, Muehlbauer GJ, Nelson RT, Grant D, Specht JE, Graham MA, Cannon SB, May GD, Vance CP, Shoemaker RC: RNA-Seq Atlas of大豆:大豆转录组指南。中国生物医学工程学报,2010,29 (3):416 - 416
杨慧,周勇,顾军,谢松,徐勇,朱刚,王磊,黄军,马慧,姚军:利用深度mRNA测序技术捕获斑马鱼胚胎和幼鱼转录组图谱。科学通报,2013,8 (5):e64058-
王勇,肖瑞,王慧,程智,李伟,朱刚,马华:The拟南芥RAD51假字RAD51B, RAD51D而且XRCC2在体细胞DNA修复和基因调控中发挥部分冗余作用。叶绿醇,2014,201(1):292-304。
李宝特,马农A,布莱辛马切尔L, Drnevich J,兰利RJ, Bilgin DD, Radwan O, Neece DJ, Clough SJ, May GD, Stacey G:大豆根毛细胞的完全转录组,一个单细胞模型及其对大豆根毛细胞的响应Bradyrhizobium日本血吸虫感染。植物生理学报,2010,32(2):353 - 356。
Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B: RNA-Seq定位和定量哺乳动物转录组。Nat方法。2008,5(7):621-628。
Marioni JC, Mason CE, Mane SM, Stephens M, Gilad Y: RNA-seq:技术可重复性的评估和与基因表达阵列的比较。中国生物医学工程学报,2008,18(9):1509-1517。
Taylor JS, Raes J:复制和分化:新基因和旧思想的进化。王志军。2004,38:615-643。
Wilhelm BT, margaret S, Watt S, Schubert F, Wood V, Goodhead I, Penkett CJ, Rogers J, Bahler J:单核苷酸分辨率下真核转录组的动态保留库。自然。2008,453(7199):1239-1243。
Lister R, O’malley RC, Tonti-Filippini J, Gregory BD, Berry CC, Millar AH, Ecker JR:高度集成的表观基因组单基分辨率图拟南芥.细胞。2008,133(3):523-536。
Cloonan N, Forrest ARR, Kolle G, Gardiner BBA, Faulkner GJ, Brown MK, Taylor DF, Steptoe AL, Wani S, Bethel G, Robertson AJ, Perkins AC, Bruce SJ, Lee CC, Ranade SS, Peckham HE, Manning JM, McKernan KJ, Grimmond SM:通过大规模mRNA测序的干细胞转录组分析。Nat方法。2008,5(7):613-619。
Morin R, Bainbridge M, Fejes A, Hirst M, Krzywinski M, Pugh T, McDonald H, Varhol R, Jones S, Marra M:使用随机引物cDNA和大规模并行短读测序分析HeLa S3转录组。生物技术。2008,45(1):81-94。
的CLAVATA信号通路受体拟南芥调节果实器官形成和分生组织中的细胞增殖。遗传,2011,189(1):177-194。
苏智,王军,于军,黄旭,顾昕:基因复制后选择性剪接的进化。中国生物医学工程学报,2006,16(2):182-189。
沈勇,周智,王智,李伟,方超,吴敏,马勇,刘涛,孔丽娜,彭丽丽:古多倍体大豆选择性剪接的全局解剖。植物细胞。2014,26(3):996-1008。
Katz Y, Wang ET, Airoldi EM, Burge CB:用于识别亚型调控的RNA测序实验分析和设计。Nat方法。2010,7(12):1009-1015。
Sugimoto K, Jiao Y, Meyerowitz EM:拟南芥多个组织的再生通过根的发育途径发生。开发学报,2010,18(3):463-471。
何晨,陈欣,黄辉,徐玲:H3K27me3基因的重编程对体外培养获得多能性至关重要拟南芥组织。中国科学:地球科学,2012,31 (5):691 - 698
Rogers ED, Jackson T, Moussaieff A, Aharoni A, Benfey PN:细胞类型特异性转录谱分析:代谢物谱分析的意义。植物学报,2012,70(1):5-17。
a、Hove CA、Hogeweg P、Maree AF、Scheres B:从细胞形态到生长素运输的根系结构。中国生物医学工程学报,2008,6 (12):e307-
赵燕:生长素的生物合成及其在植物发育中的作用。植物学报,2010,35(3):349 - 356。
Mravec J, Skupa P, Bailly A, Hoyerova K, Krecek P, Bielach A, Petrasek J, Zhang J, Gaykova V, Stierhof YD, Dobrev PI, Schwarzerova K, Rolcik J, Seifertova D, Luschnig C, Benkova E, Zazimalova E, Geisler M, Friml J:植物激素生长素的亚细胞内稳态是由er定位的PIN5转运蛋白介导的。自然。2009,459(7250):1136-1140。
丁智,王波,Moreno I, Duplakova N, Simon S, Carraro N, Reemmer J, Pencik A, Chen X, Tejos R, Skupa P, Pollmann S, Mravec J, Petrasek J, Zazimalova E, Honys D, Rolcik J, Murphy A, Orellana A, Geisler M, Friml J: er定位的生长素转运蛋白PIN8调控生长素稳态和雄性配子体发育拟南芥.通讯。2012,3:941-
Motchoulski A, Liscum E:拟南芥NPH3:一种NPH1光感受器相互作用蛋白,对趋光性至关重要。科学通报。1999,28(4):391 - 396。
黄志强,黄志强,王晓东,张晓明,王志强:大豆茎尖分生组织中基因表达的全基因组分析。植物生理学报,2009,35(6):711-727。
郑振华,黄志强,辛格明,黄志强,黄志强:大豆开花基因的比较基因组分析。科学通报,2012,7 (6):e38250-
Dreni L, Pilatone A, Yun D, Erreni S, Pajoro A, Caporali E, Zhang D, Kater MM: AGAMOUS亚家族成员在水稻生殖器官鉴定和分生组织确定中的功能分析。植物细胞。2011,23(8):2850-2863。
Dorca-Fornell C, Gregis V, Grandi V, Coupland G, Colombo L, Kater MM: The拟南芥SOC1例如基因AGL42,AGL71而且AGL72在茎尖和腋生分生组织中促进开花。植物学报,2011,67(6):1006-1017。
刘玉玲,刘玉玲。激素在茎尖分生组织中的作用。植物学报,2006,9(5):484-489。
程燕,戴霞,赵燕:丝兰黄素单加氧酶生物合成生长素调控植物花器官和维管组织的形成拟南芥.中国生物医学工程学报,2006,20(13):1790-1799。
植物生长素:植物发育变化的一个触发因子。Cell. 2009, 136(6): 1005-1016。
杨欣,马卡洛夫,马洪:拟南芥男性性母细胞DEATH1基因编码一种雄性减数分裂所需的PHD-finger蛋白。植物细胞。2003,15(6):1281-1295。
Novillo F, Alonso JM, Ecker JR, Salinas J:CBF2 / DREB1C负调节器是CBF1 / DREB1B而且CBF3 / DREB1A表达和抗压能力起着核心作用拟南芥.中国生物医学工程学报,2004,24(4):391 - 396。
苏智,马晓燕,郭辉,苏基兰,郭斌,苏志明,马慧:干旱胁迫下花的发育:植物的形态和转录组学分析揭示了干旱胁迫的急性响应和长期适应拟南芥.植物细胞。2013,25(10):3785-3807。
刘志强,刘志强,刘志强:开花植物花粉壁发育的研究。植物化学学报,2007,34(3):483-498。
杨维武,史德强,陈玉华:开花植物雌配子体发育。植物学报,2010,35(3):349 - 356。
Spencer WC, Zeller G, Watson JD, Henz SR, Watkins KL, McWhirter RD, Petersen S, Sreedharan VT, Widmer C, Jo J, Reinke V, Petrella L, Strome S, Von Stetina SE, Katz M, Shaham S, Ratsch G, Miller DM:一个时空图秀丽隐杆线虫基因的表达。中国生物医学工程学报,2011,21(2):325-341。
杨智,王旭,顾胜,胡智,徐慧,徐晨:《中国文化》的比较研究SBP-box基因家族在拟南芥和米饭。基因工程。2008,407(1):1-11。
张志强,邢淑萍,张志强,张志强,张志强,张志强,张志强,张志强SBP-box家族的转录因子在番茄。植物学报,2012,235(6):1171-1184。
Preston JC, Hileman LC:工厂的功能进化SQUAMOSA-PROMOTER绑定蛋白质像(SPL)基因家族。植物学报。2013,4:80-
Addo-Quaye C, Eshoo TW, Bartel DP, Axtell MJ:通过测序确定内源性siRNA和miRNA靶点拟南芥degradome。中国生物医学工程学报,2008,18(10):758-762。
山崎H,林M,深泽M,小林Y, Shikanai T:SQUAMOSA启动子结合蛋白样7是拟南芥中铜稳态的核心调节因子。植物细胞。2009,21(1):347-361。
Unte US、Sorensen A-M、Pesaresi P、Gandikota M、Leister D、Saedler H、Huijser P:SPL8在拟南芥中,SBP-box基因影响花粉囊的发育。植物细胞。2003,15(4):1009-1019。
Cho SH, Coruh C, Axtell MJ: miR156和miR390调节tasiRNA的积累和发育时间Physcomitrella金属盘.植物细胞。2012,24(12):4837-4849。
Stone JM, Liang X, Nekl ER, Stiers JJ:拟南芥AtSPL14,一个植物SBP-结构域转录因子,参与植物发育和对伏马菌素B1的敏感性。植物学报,2005,41(5):744-754。
Bäurle I, Dean C:植物发育转变的时间。Cell. 2006, 125(4): 655-664。
刘p - p, Kristof JR, Coppersmith JL, Pluskota WE, Bassel GW, Goloviznina NA, Nguyen TT, Martínez-Andújar C:微rna靶向对萌发后子叶-营养叶阶段过渡的调控Squamosa promoter nnbinding protein like13在拟南芥.种子科学,2010,20(02):89-96。
Schwarz S, Grande AV, Bujdoso N, Saedler H, Huijser P: microRNA被调控SBP基因盒SPL9而且SPL15控制嫩枝成熟度拟南芥.植物生理学报,2008,30(1):353 - 356。
Alvarez-Buylla ER, Benítez M, Corvera-Poiré A, Cador ÁC, De Folter S, Gamboa De Buen A, Garay-Arroyo A, García-Ponce B, jaims - miranda F, V R, Pérez-Ruiz RV, Piñeyro-Nelson A, Sánchez-Corralesa YE:花的发展。拟南芥,2010,8:e0127-
辅助/IAA蛋白的作用和活性拟南芥.植物学报,2001,6(9):420-425。
生长素在生殖发育过程中具有独特而动态的活性。冷泉港湾展望生物。2009,1 (6):a001628-
张智,张智,王凯,王凯,王凯:农杆菌介导的大豆子叶节外植体转化的影响因素分析。植物学报,2004,136(2):167-179。
王艳,索慧,郑艳,刘凯,庄昌,Kahle KT,马慧,闫欣:大豆根系特异蛋白激酶GmWNK1对根系构型ABA信号的调控作用。植物学报,2010,64(2):230-242。
Livak KJ, Schmittgen TD:利用实时定量PCR和2-ΔΔCT方法。方法。2001,25(4):402-408。
冯军,Meyer CA,王倩,刘建军,刘小霞,张勇:GFOLD:一种基于RNA-seq数据的差异表达基因排序的广义折叠变化。生物信息学。2012,28(21):2782-2788。
王勇,唐慧,德barry JD,谭欣,李军,王欣,李天辉,金辉,马勒B,郭辉:MCScanX:一种检测和分析基因共线性和共线性的工具。中国生物医学工程学报,2012,40 (7):e49-e49。
Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley DR, Pimentel H, Salzberg SL, Rinn JL, Pachter L: TopHat和Cufflinks RNA-seq实验的差异基因和转录本表达分析。网络学报,2012,7(3):562-578。
齐军,赵峰:inGAP-sv:一种基于成对端映射数据的结构变异识别与可视化方法。中国生物医学工程学报,2011,39:W567-W575。
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ:基本的局部对齐搜索工具。分子生物学杂志,1990,15(3):403-410。
griffith - jones S, Moxon S, Marshall M, Khanna A, Eddy SR, Bateman A: Rfam:在完整基因组中注释非编码rna。中国生物医学工程学报,2005,33:D121-D124。
Kohonen T:自组织形成的拓扑正确的特征地图。生物学报,1982,43(1):59-69。
杜智,周欣,凌宇,张智,苏中:agriGO:面向农业社区的GO分析工具。中国生物医学工程学报,2010,30(5):632 - 632。
Thimm O, Bläsing O, Gibon Y, Nagel A, Meyer S, Krüger P, Selbig J, Müller LA, Rhee SY, Stitt M: Mapman:一个用户驱动的工具,将基因组学数据集显示到代谢途径和其他生物过程的图表上。植物学报,2004,37(6):914-939。
确认
感谢国家转基因生物研究重点项目(2014ZX0800921B-001)、复旦大学卓学计划、上海市科委2013启名星项目(13QA1400200)和中国农业研究系统(H.N.)的资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者声明他们之间没有利益冲突。
作者的贡献
LW、YXW、HN、HM设计实验。YXW、ZHC、QBM、QYZ收集组织。LW和GFZ制备了用于固体测序的mrna和cDNA文库。LW进行了RT-PCR和qRT-PCR实验。LW、CLC、HFW和JQ进行生物信息学分析。LW, HM和YXW撰写了这篇论文。所有作者阅读并批准最终稿。
王磊、曹晨龙对这项工作的贡献相当。
电子辅料
12870 _2014_1570_moesm1_esm.pdf
附加文件1:图S1:样品中被映射的reads总数及在大豆注释基因组中的分布。图S2。RPKM值在11个样本中的分布。(a)比较11个大豆组织中基因(蓝色)和基因间背景区(红色)的表达水平。我们放大了在-15到15个经log2转换的RPKM值之间的表达式的效果。(b)经过log2转换的RPKM值在11个样本中的分布情况。垂直虚线表示阈值,高于该阈值的基因被确定为表达。将每个样本经log2转换后的基因RPKM值装箱,间隔大小为0.1。图S3。4949个ntu的表达谱。图S4。SAM38D、AM和IBM的表达基因比较。图S5。基于SBP结构域AA序列的SBP-box家族基因未根系统发育树。(PDF 417 KB)
12870 _2014_1570_moesm2_esm.xlsx
额外的文件2:S1 ~ S37的表内容。表S1。映射读取的摘要。表S2。基因组中reads的分布。表S3。的t- 8768个模拟测试。表S4。基因5'UTR上游1509个nTRs列表。表S5。4949 ntu在染色体上的分布。表S6。2326个ntu及NCBI注释基因。表S7。698 ntu的Go注释。表S8。TE中的47个ntu。表S9。40名ntu。表S10。AS事件列表。表S11。描述了202个跳过外显子基因。表S12。1834年的Psi值。表向。整个数据集的相关矩阵。表S14系列。AM与IBM重叠基因的GO注释。表S15。根尖peg列表。表S16。根尖peg的GO注释。表S17、S18。愈伤组织peg列表及GO标注。表S19、S20。子叶peg列表及GO注释。表S21和S22。下胚轴peg列表及GO注释。表S23。多个分生组织中peg的列表。表S24。多个分生组织中peg的GO注释。表S25。AM、IBM和IAM中的peg列表。表S26。AM、IBM、IAM中peg的GO注释。表S27。of peg列表。表S28。of peg的GO注释。表S29。6份SAM样品中聚乙二醇的含量清单。表S30。mRNA-seq与qRT-PCR的相关性。表S31。8个集群的RPKM值和功能描述。表S32。各GO项的P值。表S33。TF家族在G1、G2、G3中的分布。表S34。系统发育分析SBP-box家庭。表S35。sbp结构域序列和植物的登录号SBP年代。表S36。48个大豆的表达SBP年代。表S37。RT-PCR引物列表。(XLSX 7 MB)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇根据知识共享署名许可条款发布的开放获取文章(http://creativecommons.org/licenses/by/4.0),允许不受限制地在任何媒体上使用、分发和复制,前提是正确地注明原创作品。知识共享公共领域转让豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
王璐,曹超,马倩,王璐。et al。大豆多个分生组织的RNA-seq分析:新转录本和替代转录本、进化和功能意义。BMC植物杂志14,169(2014)。https://doi.org/10.1186/1471-2229-14-169
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-14-169
关键字
- 大豆
- RNA-seq
- 转录组
- 小说转录区域
- 可变剪接
- 分生组织
- 转录因子