摘要
背景
内源性小(sm) rna(主要是si-和mirna)很重要反式/ cis -参与多种细胞功能的代理调节器。在植物中,依赖RNA的RNA聚合酶(RDRs)是smRNA生物发生所必需的。目前已经证实,RDR2参与了24 nt sirna依赖的rna定向DNA甲基化(RdDM)通路。最近的研究表明,RDR1参与了第二个RdDM通路,该通路主要依赖于21个nt smrna,其功能是沉默通常对正常RdDM通路不敏感的基因组位点子集拟南芥.的同源是否以及在何种程度上RDR1可能在其他植物中也有类似的功能,目前尚不清楚。
结果
我们鉴定了一个功能丧失突变体(Osrdr1)的OsRDR1水稻中的基因(栽培稻l)源自一个反转录转座子Tos17插入。微阵列分析鉴定出1175个差异表达基因(占水稻茎尖组织中所有表达基因的5.2%)Osrdr1和WT,其中896个基因上调,279个基因下调Osrdr1.smRNA测序揭示了水稻基因组中smRNA簇的区域性改变。一些smRNA簇改变的区域与DNA甲基化的变化相关。此外,检测到几种mirna的表达改变Osrdr1,其中至少有一些与预测miRNA靶基因表达的改变有关。尽管有这些变化,在Osrdr1正常情况下相对于WT;然而,在一些非生物胁迫条件下,表型波动是短暂的。
结论
我们的研究结果表明OsRDR1通过smrna介导的DNA甲基化途径调控水稻大量具有不同功能的内源基因,参与非生物胁迫反应。
背景
RNA沉默是真核生物中由20-25 nt非编码小RNA介导的一种进化保守的基因调控机制。这些smRNAs由双链(ds)或发夹RNA分子经Dicer-like (DCL)蛋白加工而成,并基于序列互补引导RNA诱导的沉默复合物同源单链RNA,导致目标RNA降解[1- - - - - -3.].在植物中,有几种不同类型的smRNAs,包括由DCL1处理的20-24 nt微rna (miRNAs),由DCL4和DCL2处理的21-22 nt小干扰rna (siRNAs),以及由DCL3处理的24 nt异染色质相关siRNAs。miRNAs通过指导转录因子编码等调控基因的转录后基因沉默(PTGS)在植物发育中发挥重要作用。类似地,21-22 nt sirna指导病毒rna和一些内源性mrna的降解,对植物防御病毒和植物发育的某些方面很重要[4- - - - - -6].与这些ptgs相关的smrna不同,24 nt siRNAs与rna定向的DNA甲基化(RdDM)相关,这是一种植物特异性新创转座元件和其他DNA重复序列转录沉默以维持基因组稳定所需的DNA甲基化途径[7- - - - - -10].
植物中siRNAs的生物发生需要RNA依赖的RNA聚合酶(RDR)的活性,它将单链RNA转化为siRNAs的dsRNA前体。双柯模型植物拟南芥有6个RDR基因,也就是说,RDR1,RDR2,RDR3a,RDR3b,RDR3c而且RDR6[11],其中三份纪录册(RDR1,RDR2而且RDR6)在RNA沉默通路中发挥作用。RDR2是24 nt siRNA生物发生所必需的,因此参与了典型的RdDM途径[7- - - - - -9].RDR6是否参与内源性2nt的产生反式-作用sirna,也是转基因诱导的PTGS所必需的[12,13].这两个RDR6而且RDR2也参与了病毒siRNA在感染拟南芥植物[14- - - - - -16].的功能RDR1在RNA沉默中的作用尚不清楚,但最近的研究表明,它与RNA病毒亚群的siRNA生物生成有关[17- - - - - -19].此外,rdr1突变体的拟南芥与野生型相比,在一个基因组位点子集中DNA甲基化缺失拟南芥植物(20.,21],表明RDR1在最近发现的非规范的21 nt sirna导向的RdDM通路中发挥作用[20.,21].然而,的功能RDR1从全基因组的角度来看,基因调控还没有在任何植物中进行过研究。
相比之下拟南芥有6个RDR基因,水稻的RDR家族(栽培稻L.)是单子叶植物的模式植物,仅包含三个成员,即OsRDR1,OsRDR2而且OsRDR6[11,22,23].之前的一项研究表明OsRDR1具有与同类产品相似的功能拟南芥而烟草(烟草)在以ptgs为基础的某些RNA病毒的沉默中,如溴病毒[6,19,24].调查是否OsRDR1在水稻内源基因调控中起作用,我们鉴定了一个功能丧失突变体OsRDR1源自破坏性LTR逆转录转座子(Tos17)插入到基因的第2外显子。我们研究了这种水稻在正常和几种非生物胁迫条件下基因表达和smRNA谱的全基因组变化、DNA甲基化的局部变化以及表型rdr1突变体。
结果
描述大米RDR1突变体(Osrdr1)
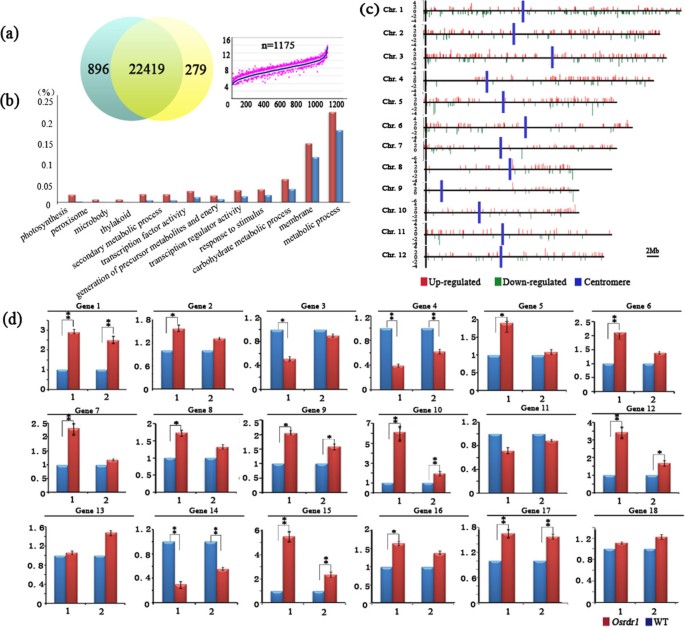
我们获得了一个LTR逆转录转座子Tos17[25插入线OsRDR1(登记编号H0643)Tos17水稻cv插入突变文库。Hitomebore(http://www.cns.fr/spip/Oryza-sativa-retrotransposon-Tos17.html).分子特征鉴定H0643为aTos17的第二外显子插入OsRDR1(图1a).获得纯合突变体(OsRDR1−−或Osrdr1)和它的兄弟野生型(WT)植物,通过杂合植物(OsRDR1+/−)连续五代。在每一代中,根据基因座特异性PCR扩增选择WT、杂合子和纯合子突变体三种基因型(图1a).杂合子和纯合子植物OsRDR1在正常的田间条件下,在整个生长和发育时期,在多代中没有明显的表型改变(图1b).半定量和实时定量(q)RT-PCR分析证实纯合子OsRDR1突变体(Osrdr1)完全失去了OsRDR1在茎尖组织中表达,该基因在WT植物中高度表达(图1c).这表明Tos17敲掉的表情OsRDR1,因此,废除了它的功能。
基因表达的全基因组变化Osrdr1
我们对茎尖组织的转录组进行了分析Osrdr1和它的兄弟WT植物使用Affymetrix基因芯片水稻®基因组阵列(Affymetrix, Inc.)。Santa Clara, CA, USA)。对微阵列数据进行归一化处理后,在水稻茎尖组织中检测到57381个表达基因。之间有22419个基因表达量保守Osrdr1而896个基因和279个基因表达显著上调和下调Osrdr1,分别为(图2a).对这1175个差异表达基因进行基因本体论(GO)类别分析,结果显示它们在多种GO类别中富集(图2b).然而,这1175个差异表达基因被发现非随机分布在12条水稻染色体上(P = 2.2E-16,基于卡方检验)。例如,染色体1、2和3比其他染色体包含更多的分布(图2c).从数据中还可以清楚地看出,在给定的染色体内,分布也是非随机的,例如,相对于各自的短臂,分布几乎完全局限于染色体4、8和9的长臂(图2c)。
的零突变效应OsRDR1水稻全局基因表达研究。(一)微阵列分析的摘要显示了检测到的基因总数和WT和差异表达基因的数量Osrdr1.(b)基因本体论(GO)对1175个差异表达基因进行分类分析Osrdr1.的y-axis是由GO类别项映射的基因的百分比:百分比由的数量计算Osrdr1vs. WT差异表达基因除以映射到特定GO类别的基因总数。的x-轴为GO类项,按相对丰度排序(水稻茎尖组织中表达的基因总数为45,078个)。蓝色条表示所有注释基因的每个类别的百分比,红色条表示所有表达基因的GO类别的百分比。(c)分布1175个Osrdr1与WT相比,12条水稻染色体中的每条都有差异表达的基因(水平线)。上面的红色柱状图和下面的绿色柱状图分别表示染色体中上调和下调的基因Osrdr1和WT,分别。的y-axis表示之间基因表达的折叠变化Osrdr1垂直的蓝条表示着丝粒区域。(d)通过qRT-PCR验证微阵列结果,其中蓝色和红色列代表WT和Osrdr1,分别。每个基因下方的数字1和2分别代表microarray和qRT-PCR结果的数据。有统计学意义P< 0.05和P<0.01水平用一个或两个星号标记。
在三个生物重复中高度可重复的微阵列配置文件Osrdr1和它的WT兄弟植物证明了数据和分析的可靠性。所有微阵列数据已提交到GEO存储库,登录号为GSE58007。为了进一步验证微阵列数据和分析的质量,我们分析了18个表达上调和下调的基因Osrdr1 vs。利用qRT-PCR技术对相同的cdna进行基因芯片检测,检测两系间的表达相等。qRT-PCR结果与几乎所有18个被测基因在表达水平或至少在表达变化趋势上都与微阵列数据高度一致(图2D),确认微阵列分析的可靠性。
smRNA簇的改变Osrdr1
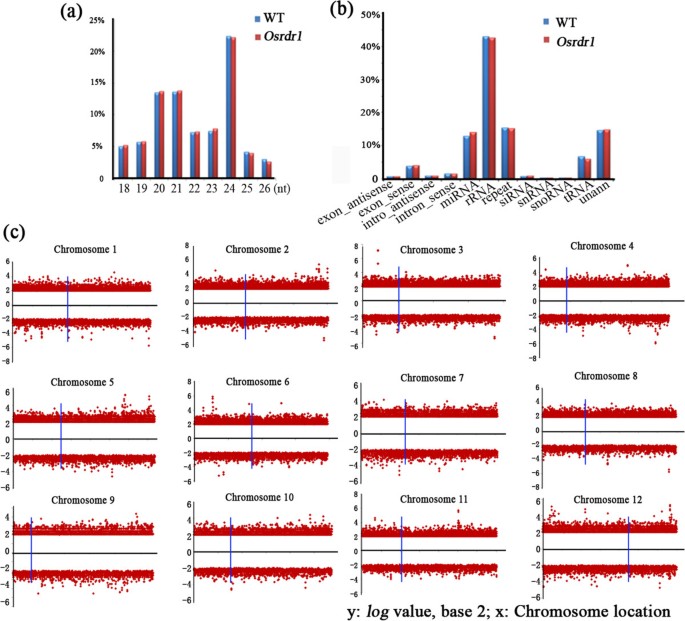
之前的研究已经证实了这一点RDR1功能是生物发生和/或某些类型的RNA病毒相关smRNA积累的扩增所必需的拟南芥[26]及烟草(烟草) [27].这些发现促使我们测试是否丧失功能OsRDR1可能对水稻中“正常”的smRNA丰度有普遍影响,我们通过高通量smRNA测序研究了这一问题。比较10398,592个清洁smRNA的读取Osrdr1有9,339,435条来自其兄弟姐妹WT (看到方法)显示两者之间的smrna的大小分布和序列类别都高度相似Osrdr1和WT(图3.a, b),表明总体smRNA丰度一般不受功能丧失的影响OsRDR1.
smRNA丰度的全基因组整体相似性并不一定意味着局部smRNA簇中没有波动,因为上调和下调的smRNA积累可以通过相互补偿来掩盖。因此,我们研究了之间的局部smRNA积累Osrdr1通过将清洗后的smRNA reads映射到12条水稻染色体上的100 bp滑动窗口(反映为smRNA簇),将reads归一化为每百万reads (RPM),然后比较两者之间的RPM smRNA簇Osrdr1使用4倍差异作为临界值,我们识别出了许多之间丰度差异的smRNA簇Osrdr1和它的兄弟姐妹WT,它们均匀分布在每条染色体的整个长度(图3.c).接下来,我们提取之间的差异表达的smrnaOsrdr1和WT(同样基于4倍差异的截止阈值)在20-24 nt的大小范围内,应该简约地包含所有sirna,并将它们映射到每条染色体上相同的100 bp窗口。我们发现这个差异smRNA簇的子集也分布在每条染色体的两个臂上(附加文件)1),但由于它们的数量较少,我们不能排除在特定染色体内出现“热点”分布的可能性。综上所述,smRNA测序数据表明,功能缺失突变在OsRDR1在每条染色体和整个基因组中引起了smRNA簇的广泛改变,但它没有导致总体smRNA谱的显著波动,这可能是由于相互抵消的smRNA簇的丰度或多或少相等地增加或减少。
miRNAs及其靶基因表达的改变Osrdr1
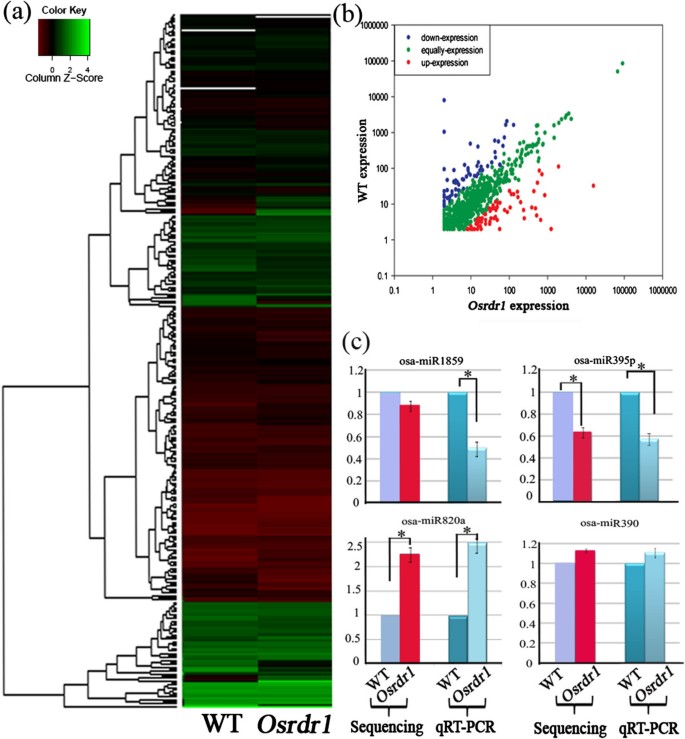
考虑到mirna所起的各种重要作用,我们研究了它们的积累是否可能在乳腺癌中受到影响Osrdr1突变体.之前的计算和克隆研究已经证实ca。水稻86个miRNA家族中的300个miRNA [28,29].基于这些信息,我们首先分析了水稻中已知miRNAs (Osa-miRNAs) (miRBase14.0中列出)的丰度Osrdr1和WT.此分析(图4a、b;额外的文件2表明:(1)大多数(90.7%)已知的oso - mirnas在两者之间表达相等或接近Osrdr1和WT;(2)部分mirna(5%)的表达量增加了2倍Osrdr1相对于WT,最高表达比达到9.0:1 (t-test, P < 0.05);(3)部分mirna(4.3%)的表达量下降2倍Osrdr1相对于WT, miR167j在突变体与WT之间的最低表达比为1:6.1 (t-test, P < 0.05);(4) Osa-miR395p和osa- mir395属于同一个miRNA家族,表达变化方向相反,突变体和WT中Osa-miR395p的表达比为1:6 6,而osa- mir395的表达比为4.2:1 (t-test, P < 0.05)。为了验证基于smRNA测序数据的表达差异,我们对突变体和WT中的4个miRNAs进行了半巢式qRT-PCR分析。qRT-PCR结果与smRNA测序数据一致(图4c),确认之间miRNA表达的变化Osrdr1和WT。
除了已知的mirna,我们根据水稻基因组中围绕smRNA序列的序列中预mirna样茎环结构的预测,从突变体和WT植物的smRNA数据中鉴定出总共10个假定的新的oso - mirna(附加文件)3.).其中三个新miRNA (Osr-miRNA-N5.1,−N5.2和-N5.3)具有相同的成熟序列,但对应于三个独立的基因组位点,从而形成了一个新的miRNA家族。对于假定的miRNA Osr-miRNA-N7,从预测的茎环结构的5 ' (5p)和3 ' (3p)一半中检测到smRNA reads,但5p smRNA显示出比3p smRNA更高的丰度(附加文件)3.),表明5p smRNA是引导链(miRNA),而3p smRNA是客链(miRNA*) [30.].与已知的mirna一样,这些新的mirna也表现出表达差异Osrdr1WT,其中3只在in中表达Osrdr1和5只在WT植物中表达(附加文件3.).综上所述,我们的结果表明OsRDR1可能与水稻miRNA的积累有关。
研究miRNA的积累是否发生改变Osrdr1相对于WT与miRNA靶基因表达的变化相关,我们比较了来自smRNA测序数据的miRNA表达谱(丰度)与基于微阵列数据的靶基因表达水平。我们没有发现miRNA丰度和靶基因表达水平之间的普遍关系(附加文件)4a).相反,在mirna子集及其预测目标中识别出四种类型的关系(附加文件4b),其中包括:(1)miRNA丰度降低与靶基因表达上调相关Osrdr1相对于WT(附加文件4我);(2) miRNA丰度增加与靶基因表达下调相关Osrdr1相对于WT(附加文件4b-ii);(3)中miRNAs及其靶基因均上调Osrdr1相对于WT(附加文件4b-iii);(4)中miRNAs及其靶基因均下调Osrdr1相对于WT(附加文件4b-iv)。前两种类型的关系支持了mirna在下调其预测靶基因表达中的作用。后两种类型的关系可能是由另一种更上游的调控因子引起的miRNAs及其靶基因的协调转录调控的结果,该调控因子的表达或活性由于其功能的丧失而被修改OsRDR1。所有小RNA数据已提交到GenBank,登录号为SRP042238。
DNA甲基化的位点特异性改变Osrdr1
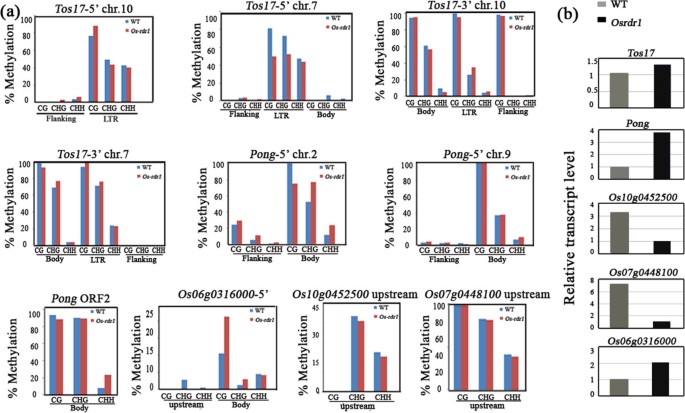
随着拟南芥RDR1已被证明在非规范的、依赖于nerd的RdDM通路中发挥作用[20.,21,我们很想知道OsRDR1可能在大米中也有类似的作用。因此,我们检测了10个选择的基因组位点的胞嘧啶甲基化和基因表达水平Osrdr1亚硫酸盐测序和qRT-PCR分析。这些位点与两个转座元件(TEs)和三个蛋白质编码基因重叠,之所以选择这三个基因作为代表,是因为它们都表现出smRNA簇的改变Osrdr1相对于WT(附加文件5).两个te的亚硫酸根测序区(反转录转座子Tos17和DNA转座子发出难闻的气味)包括:(1)5 ' -和3 ' - ltr的部分及其相邻的两个侧翼区域Tos17副本(分别位于染色体10和7上)(附加文件5a、b);(2) 5 '末端及其相邻两侧的两个区域发出难闻的气味副本(分别位于染色体2和9)(附加文件5C, d),和;(3)转座酶编码ORF的体区发出难闻的气味(附加文件5E)由元素的所有保守副本共享。三个蛋白质编码基因的亚硫酸根序列区域都在其5 '上游区域内(附加文件)5F, g, h)。
亚硫酸根测序结果表明:(1)其中五种Tos17区域分析,只有5 ' LTR区域为Tos17位于7号染色体上的拷贝明显减少ca。30%)的CG和CHG甲基化,而CHH甲基化在Osrdr1相对于WT(图5a和附加文件5b);(2)为三发出难闻的气味区域分析,只有位于染色体2上的拷贝的5 '区域显示出明显的甲基化变化:CG甲基化减少20%,CHG和CHH甲基化分别增加约30%和50%Osrdr1相对于WT(图5a和附加文件5c);在分析的三个基因位点中,只有一个(操作系统06g0316000)显示CG甲基化增加ca。50%Osrdr1相对于WT,而其他两个区域的甲基化没有变化(图5a和附加文件5f)。
两种te均明显上调Osrdr1相对于WT(图5b),与每个te的一个副本的5 '区域甲基化减少一致Osrdr1(图5a).值得注意的是,所分析的三个基因均未显示其5 ' -调控区DNA甲基化状态与表达水平之间的预期关系。具体来说,一个基因(操作系统06g0316000)显示CG甲基化增加Osrdr1表达上调(图5b);其余2个基因表达下调Osrdr1相对于WT,尽管亚硫酸根序列区域没有甲基化变化(图5b)。
接下来,我们研究了smRNA积累和DNA甲基化之间的可能关系。我们发现几乎所有CHH甲基化的改变都与smRNA簇的改变有关。例如,CHH甲基化的增加发出难闻的气味位于染色体2上的拷贝与smRNA积累的中度增加有关,而CHH甲基化在侧翼区域略有减少,而CHH甲基化在基因体区域增加发出难闻的气味位于9号染色体的拷贝分别与smRNA积累的中度减少和增加相关(附加文件5c). smRNA积累与CHH甲基化的正相关表明OsRDR1起作用新创CHH甲基化在水稻基因组位点的一个子集中,可能是通过影响RdDM所需smRNAs的产生/积累,如图所示拟南芥[20.,21].甲基化改变的位点特异性或两种分析的TEs表明,它们的甲基化模式是由侧翼序列和局部染色质环境决定的,这一问题值得进一步研究。
表型的Osrdr1在正常和非生物胁迫条件下
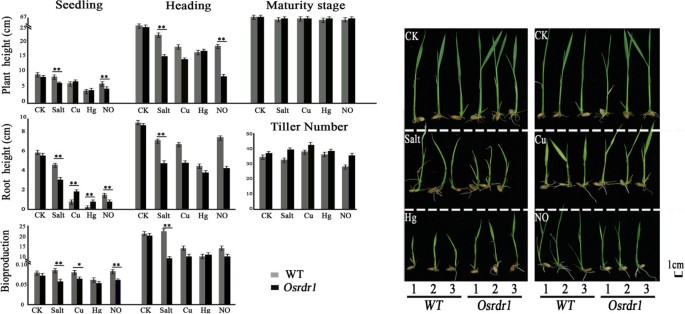
众所周知,各种应激条件可能对基因组稳定性产生持久影响,导致基因组结构的跨代变化,这种变化被认为是由表观遗传机制启动的[31- - - - - -37].因此我们很想知道OsRDR1可能在水稻的应激反应中起作用。我们量化了之间的表型Osrdr1和野生型植物在正常和几种短期非生物胁迫条件下(看到材料和方法),包括用盐、重金属铜处理2 +和汞2 +,以及过量的一氧化氮(NO)。结果表明,间无显型差异Osrdr1但在某些不同的胁迫条件下,两种基因型之间出现了显著的短暂表型差异(图6).具体来说,(1)幼苗O年代rdr1对盐和过量NO处理的敏感性高于WT,表现为苗期株高、根长和生物量的降低,去除胁迫后株高和根长差异持续到抽穗期;(2)Osrdr1对重金属Cu的耐受性增强2 +/ Hg2 +随着根系长度的增加,在抽穗期差异逐渐减小,并完全消失;(3)突变体和WT的非胁迫和瞬态胁迫植株生长至成熟时,株高、分蘖数、穗数和籽粒性状在两种基因型间均无差异。总的来说,我们的结果表明OsRDR1在水稻的胁迫反应中具有潜在的作用,但当胁迫存在时,其影响是偶然的,而当应力消除时,其影响不会持久。
讨论
RNA沉默途径在双子叶模型植物中已被很好地表征拟南芥但对许多主要作物所属的单子叶植物等其他植物的研究仍然很少。利用转座子Tos17插入突变体OsRDR1在水稻中,我们进行了全基因组分析,以揭示基因的功能OsRDR1中显示的组件拟南芥参与非典型的,21 nt sirna导向的RdDM通路[20.,21],对内源性基因表达的影响。通过对smRNAs的深度测序和基因表达的微阵列分析Osrdr1在突变体及其兄弟体WT中,我们发现> 1000个基因的表达发生了显著变化Osrdr1相对于WT,这表明OsRDR1在水稻全基因组基因调控中起作用。此外,Osrdr1该突变体在整个水稻基因组中显示了smRNA积累和/或滴定的区域性改变,其中至少有一些与DNA甲基化的位点特异性改变有关。
在差异积累的smrna中,许多是本研究中已知和新发现的mirna。许多这些mirna的表达变化与其靶基因表达的变化相关,这可能部分原因是在乳腺癌中观察到的基因表达变化Osrdr1的突变机制OsRDR1引起水稻miRNA表达变化的原因尚不清楚。它是在拟南芥没有一种rdr在miRNA的生物发生中起直接作用[2,38].然而,RDRs可以通过TGS或PTGS途径影响miRNA前体基因的表达,间接影响miRNA的积累[1,2,38]或生成dsRNAs,竞争miRNA生物发生所需的DCL1功能[1,2].
我们的研究结果表明OsRDR1可能在维持内在基因座特异性DNA甲基化模式中发挥作用,因为它的突变导致了我们分析的一些基因座的甲基化改变。特别是CHH甲基化的变化,这表明新创通过RdDM途径甲基化,显示与smRNA积累的变化相关。在CHH甲基化减少和随之而来的smRNA积累减少方面,OsRDR1可能在功能上类似于拟南芥RDR1并在21 nt sirna依赖的非规范RdDM通路中发挥作用[20.,21].然而,一些分析的位点显示CHH甲基化增加,这与smRNA积累增加有关Osrdr1.我们应该注意的是,由于我们只分析了10个位点,观测到的结果可能无法外推到全球范围。在拟南芥的突变RDR1导致在4949个CHH低甲基化DMRs(差异甲基化区域)之间的所有三个序列上下文(CG, CHG和CHH)的甲基化胞嘧啶几乎完全丢失数字版权管理1/2和WT [20.].因此,全基因组甲基化分析(甲基化组)Osrdr1需要变种人来确认是否OsRDR1在全球水稻中起着类似的作用。
以前的研究拟南芥而且烟草已经确定了RDR1在植物病毒反应中的既定作用[24,26,27].我们在这里展示了Osrdr1在正常生长条件下,其表型与同类野生型植株无差异,但在几种非生物胁迫条件下,其表型出现短暂波动。这一观察结果,连同丰富的GO类包括那些参与代谢过程的差异表达基因之间Osrdr1和WT(图2B),认为改变基因表达的影响是由于OsRDR1突变在正常条件下大部分已被疏导,但在某些非生物胁迫条件下可被释放[39,这个问题值得进一步调查。无论如何,我们的结果表明,除了其在受感染植物中外源病毒来源的siRNAs (vsiRNAs)的产生和扩增中的既定作用外,[26,27,大米RDR1同族体(OsRDR1)也可能在某些非生物应激反应中发挥作用,但这可能不涉及这方面的稳定表观遗传变化。在这方面,应该再次强调的是,smRNA谱和基因表达的全基因组分析Osrdr1是在正常条件下生长的植物上进行的。因此,需要进一步研究突变体和WT在短期和长期胁迫条件下的植物。分析压力处理的后代也会很有趣Osrdr1植物调查OsRDR1参与压力诱导的表观遗传变化的跨代遗传,如果它们发生。
结论
如何RDR1影响全局基因表达和smRNA谱的研究尚未在任何植物物种中进行。通过分析水稻的零突变体RDR1基因(OsRDR1),我们发现该突变体中有1000多个不同基因本体(GO)类别的内源性基因表达发生了显著改变,表明其在基因本体(GO)中的功能作用OsRDR1调控水稻内源基因表达。通过smRNA深度测序,我们发现突变体中12条水稻染色体中的每一条都发生了广泛的smRNA簇的改变,这表明了基因在水稻中的作用OsRDR1水稻中smRNA的生物发生和/或滴定。我们还发现,至少部分基因表达变化与miRNAs的差异相关。我们进一步表明,smrna的改变可能与CHH环境中胞嘧啶甲基化的位点特异性改变相伴而生,因此相互关联OsRDR1到水稻中的DNA甲基化最后,我们发现,虽然没有明显的表型异常与功能丧失相关OsRDR1的结果,各种短期非生物胁迫条件可产生短暂的表型波动OsRDR1突变,暗示一种作用OsRDR1在植物非生物胁迫反应中。
方法
植物材料
基于水稻逆转录转座子的信息Tos17插入线(http://tos.nias.affrc.go.jp/),得到了水稻品种Hitomebore的一个品系(#RDR704)Tos17的第二外显子插入的OsRDR1处于杂合状态(登记号H0643)。根据BlastN在NCBI网站(http://blast.ncbi.nlm.nih.gov/Blast.cgi),我们发现大米含有一份插入基因(OsRDR1).这三个OsRDR1基因型,WT (RDR1 / RDR1),杂合(RDR1 / RDR1)和纯合突变体(rdr1 / rdr1)被两对特异性引物识别(图1a).具体地说,WT由一对固定在OsRDR1吉恩却夹击了Tos17插入网站;通过一对引物鉴定了纯合突变体OsRDR1吉恩和另一个目标是Tos17;通过两种引物的组合鉴定杂合子。的杂合子OsRDR1(+/−)连续自育5代,在前4代(S1-S4)中,根据PCR鉴定只选择杂合个体。在最后一代(S5),新分离的纯合突变体,即OsRDR1−/−(指定为Osrdr1),以及它的兄弟野生型,即OsRDR1+/+,被选择并繁殖另一代,以获得本研究所需的足够种子。这样,突变体和WT在基因上应该是相同的,除了相关的位点,即OsRDR1.用蒸馏水彻底清洗两种基因型的种子,然后在含有蒸馏水的培养皿中28°C在黑暗中发芽。将发芽种子移栽到26℃、光照/暗光照16 h/8 h的温室中,进行0.15 mMol/L NaCl(盐)、0.25 mMol/L CuSO四种非生物胁迫处理4(重金属铜2 +), 0.25 mMol/L HgCl2(重金属汞2 +)、1 mMol/L硝基铁氰化钠(III)在霍格兰德营养液中脱水(SNP, for NO胁迫)7天。模拟对照(CK)平行生长。育苗移栽到正常水田。在适当的生长发育阶段、苗期、抽穗期和成熟期对植物进行调查。
从植物幼苗中分离出基因组DNAOsrdr1用改良的CTAB法和WT在同一发育阶段。根据制造商的说明,用Trizol试剂(Invitrogen)从同一幼苗中分离总RNA。然后用无RNase- DNase I (Invitrogen)处理RNA,以消除可能的基因组DNA污染,然后用SuperScript RNase H-逆转录酶(Invitrogen)进行逆转录。
SmRNA文库构建与测序
根据Illumina Sample Preparation Protocol制备总RNA进行smRNA测序。这些样本被定量和均一化,以便等量的RNA从Osrdr1和WT进行分析。简而言之,在15% tbe -尿素变性PAGE凝胶上通过电泳分离纯化总RNA,并切除和回收标记通道中15-30个核苷酸条带对应的smRNA区域。15-30 nt smrna分别用T4 RNA连接酶连接5 '和3 ' RNA接头,每一步都用尿素PAGE凝胶电泳分离进行验证和纯化。随后,通过Super- Script II逆转录酶(Invitrogen)将连接适配器的smRNA转录成cDNA,并使用退火到适配器末端的引物进行PCR扩增。对扩增后的cDNA也进行了纯化和回收。使用安捷伦技术2100生物分析仪对样品的大小、纯度和浓度进行了验证,从而确保了文库的最终质量。构建的两个cDNA文库随后进行Solexa/Illumina专有的流细胞簇生成和桥接扩增。
smRNA簇分析
沿着水稻基因组,每滑动100 bp的窗口内统计18-26 nt大小的SmRNA reads。读数被标准化为RPM(每百万读数),然后进行比较Osrdr1和WT植物使用中位RPM值,表示为XOsrdr1Y表示WT植物。折叠值由公式log计算2X-log2Y = log2(X / Y)。
Affymetrix genchip®水稻基因组阵列
微阵列转录分析由Gene Company Ltd.(中国上海)的Affymetrix, Inc.使用《GeneChip®表达分析技术手册》中描述的程序进行。
实时定量(q)逆转录酶(RT) pcr分析
qRT-PCR实验采用SYBR预混料进行前女友Taq (TOYOBO)根据制造商在Roche LightCycler480仪器(Roche Inc.)上的说明。扩增18个研究基因的引物使用Primer 5软件设计,并在附加文件中列出6.qRT-PCR分析转座酶基因的引物已在先前的报道中描述[40].大米的表达β肌动蛋白的基因,eEF基因和UBQ5内控基因为5 ' -atgccattctccgtctt和5 ' -gctcctgctcgtagtc引物对;5 ' -tttcactcttggtgtgaagcagat和5 ' -gacttccttcacgatttcatcgtaa;5 ' -accacttcgaccgccactact和5 ' - acccctaagcctgctggtt。RT-qPCR条件报道[40].
半巢式RT-PCR检测miRNA表达
根据之前的报道,设计了四种mirna特异性寡核苷酸引物[41,42]对特异性miRNAs进行逆转录,并设计了与RT引物相对应的四对特异性引物来扩增cDNA(附加文件6).RT-PCR方法如前所述[41,42].
亚硫酸氢盐测序
使用EZ DNA甲基化金试剂盒对每株植物的DNA (~1 μg)进行亚硫酸盐处理,并使用特定引物进行扩增(附加文件)6).将亚硫酸氢盐PCR产物克隆到载体NTI中,送阳性克隆进行测序。利用网站上的分析软件对结果进行分析http://katahdin.mssm.edu/kismeth根据先前的报告[43].
参考文献
- 1.
Baulcombe D:植物中的RNA沉默。自然科学进展,2004,29(3):349 - 349。
- 2.
Eamens A, Wang MB, Smith NA, Waterhouse PM:植物RNA沉默:昨天,今天和明天。植物学报,2008,37(2):357 - 357。
- 3.
Zamore PD: RNA干扰:听沉默之声。中国生物工程学报,2001,8:746-750。
- 4.
Tang G: siRNA和miRNA:对RISCs的洞察。生物化学,2005,30(2):106-114。
- 5.
Taylor RS, Tarver JE, Hiscock SJ, Donoghue PC:植物microrna的进化史。植物科学进展,2014,19(3):175-182。
- 6.
王博明,马苏田C,史密斯NA,志村H: RNA沉默与植物病毒性疾病。植物与微生物相互作用,2012,25(10):1275-1285。
- 7.
Haag JR, Pikaard CS:多亚基RNA聚合酶IV和V:植物基因沉默的非编码RNA的提供者。中国生物医学工程学报,2011,12:483-492。
- 8.
王晓燕,王晓燕,王晓燕,王晓燕,王晓燕。植物rna介导染色质蛋白沉默的研究进展。中国生物医学工程学报,2009,21(3):367-376。
- 9.
张宏,朱建科:rna定向的DNA甲基化。植物学报,2011,14:142-147。
- 10.
孙峰,郭伟,杜娟,倪震,孙强,姚勇:发育中的小麦te相关sirna分布广泛、丰富且多样性。基因学报,2013,522(1):1-7。
- 11.
王志刚,王志刚,王志刚:RNA定向聚合酶的命名与功能研究。植物科学进展,2006,11(3):142-151。
- 12.
宋旭,王东,马磊,陈震,李鹏,崔霞,刘超,曹松,褚超,陶勇,曹旭:水稻RNA依赖RNA聚合酶6在小RNA生物发生和小穗发育中的作用。植物学报,2012,31(3):388 - 388。
- 13.
Vazquez F, Vaucheret H, Rajagopalan R, Lepers C, Gasciolli V, Mallory AC, Hilbert JL, Bartel DP, Crete P:内源性反式作用sirna调节拟南芥mrna的积累。生物化学学报,2004,16(1):69-79。
- 14.
Shivaprasad PV, Rajeswaran R, Blevins T, Schoelz J, Meins F, Hohn T, Pooggin MM: CaMV transactivator/ virus plasmin干扰拟南芥rdr6依赖反式作用和次级siRNA通路。中国生物医学工程学报,2008,36(18):5896-5909。
- 15.
李志强,李志强,李志强,等。rdr6基因缺失对植物基因沉默的影响。植物学报,2009,29(3):379 - 379。
- 16.
朱庆华,王晓梅,王文杰,王文杰:拟南芥对尖孢镰刀菌侵染反应的长链非编码rna。植物学报,2014,201(2):574-584。
- 17.
丁文文,李华,卢瑞,李峰,李文霞:RNA沉默:一种保守的动植物抗病毒免疫机制。病毒决议2004,102(1):109-115。
- 18.
廖玉文,孙志辉,周永华,史凯,李霞,张国强,夏新军,陈志新,于建强:过氧化氢和一氧化氮在烟草花叶病毒基础防御中诱导植物编码RNA依赖RNA聚合酶1的作用。公共科学学报,2013,8 (9):e76090-
- 19.
Rakhshandehroo F, Takeshita M, Squires J, Palukaitis P: RNA依赖性RNA聚合酶1对马铃薯Y病毒感染及其他抗病毒反应基因的影响。植物微生物相互作用,2009,22(10):1312-1318。
- 20.
Stroud H, Greenberg MV, Feng S, Bernatavichute YV, Jacobsen SE:沉默突变体的综合分析揭示了拟南芥甲基化组的复杂调控。中国生物医学工程学报,2013,32(3):344 - 344。
- 21.
Pontier D, Picart C, Roudier F, Garcia D, Lahmy S, Azevedo J, Alart E, Laudie M, Karlowski WM, Cooke R, Colot V, Voinnet O, Lagrange T: NERD,植物特异性GW蛋白,在拟南芥中定义了一个额外的rnai依赖的染色质通路。生物化学学报,2012,48(1):121-132。
- 22.
Chen H, Kobayashi K, Miyao A, Hirochika H, Yamaoka N, Nishiguchi M:在水稻dna损伤反应中,OsRecQ1和OsRDR1都是产生小RNA的必要条件。公共科学学报,2013,8 (1):e55252-
- 23.
陈华,田井A, Mori M, Ugaki M, Tanaka Y, Samadder P, Miyao A, Hirochika H, Yamaoka N, Nishiguchi M:病毒介导RNA沉默后水稻RNA依赖RNA聚合酶1 (OsRDR1)的分析。中国植物学报,2010,36(3):344 - 344。
- 24.
应小斌,董磊,朱红,段广昌,杜秋生,吕德强,方YY, Garcia JA,方RX,郭海生:烟草RNA依赖RNA聚合酶1抑制RNA沉默并增强本色烟病毒感染。植物细胞学报,2010,22(4):1358-1372。
- 25.
Hirochika H, Sugimoto K, Otsuki Y, Tsugawa H, Kanda M:水稻组织培养诱导突变的逆转录转座子。中国科学:自然科学,2006,26(3):349 - 349。
- 26.
Donaire L, Barajas D, Martinez-Garcia B, Martinez-Priego L, Pagan I, Llave C:烟草拨浪鼓病毒衍生的小干扰rna生物发生的结构和遗传要求。中国病毒学杂志,2008,32(4):567 - 567。
- 27.
王晓波,吴强,Ito T, Cillo F,李文霞,陈霞,余建林,丁文文:rnai介导的病毒免疫需要病毒源sirna的扩增拟南芥.中国科学:自然科学,2010,27(3):489 -489。
- 28.
孙嘉荣,周晓霞,郑勇,张伟,朱建军:利用高通量测序鉴定水稻新mirna和候选mirna。中国生物医学工程学报,2008,8:25-
- 29.
朱qh, Matthew L, Fan L, Kennedy G, Gubler F, Helliwell C:发育中的水稻籽粒中多种microrna和microrna样小rna。基因组学报,2008,18(9):1456-1465。
- 30.
Lu C, Jeong DH, Kulkarni K, Pillay M, Nobuta K, German R, Thatcher SR, Maher C, Zhang L, Ware D, Liu B, Cao X, Meryers B, Green P:水稻microRNAs的全基因组分析揭示了天然反义microRNAs (nat-miRNAs)。中国科学:自然科学,2008,29(3):349 - 349。
- 31.
朱建科,朱建科:植物胁迫响应的表观遗传调控。植物学报,2009,12(2):133-139。
- 32.
Karan R, DeLeon T, Biradar H, Subudhi PK:盐胁迫诱导不同基因型水稻DNA甲基化模式变异及其对基因表达的影响。公共科学学报,2012,7 (6):e40203-
- 33.
金建敏,杜志明,石田J,松井A,木ura H, Seki M:拟南芥干旱胁迫恢复过程中染色质状态的转变。植物生理学报,2012,29(5):344 - 344。
- 34.
柯惠萍,李勇,宋晓霞,欧晓峰,邢少生,马军,冯·Wettstein D,刘波:水稻DNA甲基化在氮素胁迫下的遗传改变与子代抗逆性的增强。中国生物医学工程学报,2011,29(3):344 - 344。
- 35.
辛敏,王勇,姚勇,谢超,彭华,倪震,孙强:不同microrna对小麦白粉病感染和热胁迫的响应。中国生物医学工程学报,2010,30 (3):344 - 344
- 36.
欧旭,张勇,徐超,林霞,臧青,庄涛,姜玲,冯wettstein D,刘波:重金属胁迫诱导水稻DNA甲基化模式改良和耐受性的跨代遗传。公共科学学报,2012,7:e41143-
- 37.
朱勇,董安,沈文华:植物非生物胁迫响应中的组蛋白变异和染色质组装。生物化学学报,2012,19(3-4):343-348。
- 38.
巴特尔DP: MicroRNAs:基因组学,生物发生,机制和功能。光子学报,2004,29(2):344 - 344。
- 39.
王晓明,王晓明,王晓明。隐性变异与遗传鲁棒性的群体遗传理论。中国生物医学工程学报,2004,18(4):379 - 379。
- 40.
王宁,王辉,张东,吴艳,欧旭,刘松,董智,刘波:紫莓渐进杂交水稻Dart转座子家族的转座子再活化。中国生物医学工程学报,2010,30 (4):344 - 344
- 41.
陈c, Ridzon DA, Broomer AJ, Zhou Z, Lee DH, Nguyen JT, Barbisin M, Xu NL, Mahuvakar VR, Andersen MR, Lao K, Livak K, Guegler K:利用茎环RT-PCR实时定量microrna。中国生物医学工程学报,2005,33 (20):e179-
- 42.
Wan G, Lim QE, Too HP:利用脱氧尿苷结合寡核苷酸和半嵌套引物实时RT-PCR高效定量成熟microrna。RNA。2010, 16(7): 1436-1445。
- 43.
徐超,王慧,姜玲,庞娟,刘波:水稻mPing转座活性与胞嘧啶甲基化的关系。中国生物医学工程学报,2009,29 (3):344 - 344
确认
国家自然科学基金项目(30990243,31200198)、国家重点基础研究发展计划项目(2013CBA01404)、吉林省基础研究基金项目(201201097,201205048)、吉林农业大学青年科学家专项基金(201201)资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
NW和DZ进行了实验的主要部分,分析了数据并起草了手稿。ZHW、HWX、JM、HW、WH、YL、XYL、CYZ、OXF和NL参与了所有实验。BL和MBW设计了作品并最终定稿。所有作者都阅读并批准了最终的手稿。
王宁宁、张迪对这项工作做出了同样的贡献。
电子辅助材料
12870 _2014_1583_moesm1_esm.tiff
附加文件1:图S1:差异smRNA簇之间的染色体分布Osrdr1其中x轴为染色体的长度(每100 bp窗口),y轴为不同rpm的值(对数值,以2为基底)。蓝色纵线表示每条染色体的着丝点区域。(tiff 202 kb)
12870 _2014_1583_moesm3_esm.tiff
附加文件3:图S2:从Osrdr1和广式。(a)新miRNAs的序列、表达状态(包括读数)和基因组位置。5p,成熟的miRNA序列位于预测的茎环结构的5 '一半;3p,成熟的miRNA序列位于预测的茎环结构的3 '一半。Mfe,最小自由能。(b)预测的新型miRNAs前体RNA的茎环结构。茎环内成熟的miRNA序列用红线表示,miRNA的5 '到3 '方向用箭头表示。(tiff格式651 kb)
12870 _2014_1583_moesm4_esm.tiff
附加文件4:图S3:(a)已知miRNAs的表达与其靶标的相关性。(b)已知mirna的表达水平与其靶标的两两比较。黄色和蓝色列分别表示目标和miRNA的表达水平。Y-axis表示log的值2褶皱的变化。(tiff 854 kb)
12870 _2014_1583_moesm5_esm.tiff
附加文件5:图S4:两个转座元件(TEs)的10个基因组位点的smRNA簇和DNA甲基化之间的区域关联Tos17(四个地区)和发出难闻的气味(三个区域)和三个基因(各一个区域)。红色、蓝色和绿色圆圈分别表示CG、CHG和CHH序列上下文,其中填充的是甲基化的,空的是未甲基化的。(tiff格式1mb)
12870 _2014_1583_moesm6_esm.xlsx
附加文件6:表S2: 18个基因的qRT-PCR检测引物,10个位点的亚硫酸氢盐测序和4个miRNAs的半嵌套qRT-PCR分析。(xlsx10kb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是原创作品的名称要注明出处。创作共用公共领域奉献弃权书(https://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
王宁,张东,王珍。et al。基因突变RDR1基因导致水稻基因表达的全基因组变化、小RNA簇的区域变异和DNA甲基化的局部改变。BMC植物生物学14,177(2014)。https://doi.org/10.1186/1471-2229-14-177
收到了:
接受:
发表:
关键字
- 基因表达
- 表观遗传学
- 小核糖核酸
- DNA甲基化
- RDR1
- 栽培稻l