摘要
背景
Theobroma可可L.是一种热带果树,它的种子被用来制作巧克力。在体外可可体细胞胚胎发生(SE)是一种繁殖系统,可用于快速批量繁殖,加速育种计划,并直接为农民提供植物。可可SE的两个主要局限性仍然存在:胚胎产生的效率高度依赖于基因型,缺乏完整的子叶发育导致胚胎到植物的低转化率。为了更好地了解SE的发育并提高SE转化的效率,我们使用全基因组芯片检测了合子胚胎和体细胞胚胎之间的基因表达差异。
结果
在合子胚胎发生(ZE)的4个发育时间点和可可体细胞胚胎发生(SE)的2个发育时间点测定了28752个基因的表达量。在ZE时间过程中,10288个差异表达基因被富集,用于与对非生物和生物刺激的反应、代谢和细胞过程相关的功能。比较ZE和SE的表达谱,鉴定出10175个差异表达基因。许多可能参与乙烯代谢和反应的TF基因在SEs中比ZEs表达更强。脂肪酸代谢基因、类黄酮生物合成基因和种子贮藏蛋白基因在两种胚中的表达水平也存在差异。
结论
在可可籽粒ZE和SE发育的不同阶段,大量基因受到差异调控。乙烯和类黄酮相关基因在SE中相对较高的表达表明,发育中的组织可能在SE成熟过程中经历了高水平的应激在体外环境。尽管缺乏脂质和代谢物的积累,但与生长素、多不饱和脂肪酸和次生代谢物合成相关的基因表达在SEs中高于ZEs。这些与种子发育过程中关键过程相关的基因转录水平的差异与体细胞胚胎没有完全发育出合子胚胎中发现的大储存子叶的事实是一致的。这些结果为可可体细胞胚胎发生的改进方案的设计提供了参考。
背景
Theobroma可可L.(可可)是一种二倍体树木,生长在50多个热带国家,是一种主要的经济作物,为数百万小农户提供收入。[1].经过发酵和干燥的可可种子为这些地区的经济贸易和社会发展提供了价值数十亿美元的经济作物的基础。可可种子主要由胚胎子叶组成,形成一个高度内陷的组织,富含油、萜烯、蛋白质、淀粉和类黄酮。
植物胚胎的发育始于雄性和雌性配子的双重受精,随后是合子胚胎发生(ZE)过程,在此过程中,胚胎的形成通过横向、径向和纵向生长的复杂程序进行[2,3.].胚胎发育可分为球状发育、心脏发育、鱼雷发育和成熟发育[4]而这些发育阶段的适当进展是由复杂的调节网络控制的[2,3.,5,6].
尽管胚胎发生过程复杂,但可以从许多植物物种的体细胞组织中人工诱导,包括可可[5,7],这一过程被称为体细胞胚胎发生(SE),于1958年首次在胡萝卜中报道[8].在SE成熟过程中,细胞完全在母体环境外发育成体细胞胚胎,并通过“转化”发育成完整的形态完整的植物[9].在一些植物中,这一复杂的过程可以通过简单的激素操作来启动,而在另一些植物中,其效率取决于基因型和外植体组织类型,并且需要对激素和环境条件进行精确控制。广泛的研究集中于发现控制高度复杂的调控机制的基因,这些机制导致胚胎发生的起始和调控[5,10- - - - - -13].体细胞胚胎的开始是先进行体细胞的去分化,而不是像有性生殖那样通过减数分裂形成单倍体配子,并且是在没有胚乳和母体卵巢组织的情况下发生的,尽管如此,体细胞胚胎和合子胚胎的发育总体上遵循相同的模式[11,14].在植物种子中,胚乳在胚胎发育和萌发过程中为胚胎提供营养物质方面起着重要作用[3.,15,16]并已被证明在整合种子发育的不同成分和遗传程序方面发挥作用[17].可可SEs的主轴在形态上与合子的主轴相似,具有双极结构和典型的胚胎器官。然而,在合子种子发育过程中,胚胎子叶扩张并积累大量的脂类、蛋白质、碳水化合物和萜烯,而体胚则产生不发育的小子叶。
可可的SE方案包括4个主要步骤:初级体细胞胚胎发生、次级体细胞胚胎发生、体细胞胚胎转化为幼苗和植物驯化[18,19].可可的se已由许多不同的基因型产生,se植物已在田间条件下生长,并已证明生长类似于种子繁殖的植物[18- - - - - -23].尽管该系统足够高效,可用于商业用途,但它包括几个高度依赖基因型的步骤。体细胞胚胎转化是该过程中效率最低的步骤之一,它涉及将成熟的体细胞胚胎转化为整株植物[24].转换率取决于许多因素,包括基因型,成功率从7%到75%不等(作者未发表的数据)。
为了研究可可ZE和SE成熟过程中涉及的调控和基因表达网络,我们使用全基因组微阵列对基因表达谱进行了表征。4个合子胚和2个体细胞胚发育阶段的表达谱显示,合子胚和体细胞胚成熟过程中转录因子、类黄酮生物合成基因和脂类生物合成基因的表达存在显著差异,说明体细胞胚子叶发育不完全的分子基础。了解调控SE发育级联的遗传成分,可为调控体胚成熟过程,通过控制化学和物理培养环境,提高植株生产效率和质量提供指导。此外,我们的研究结果为可可合子胚胎发育过程中的基因表达程序提供了新的认识,这为一种重要的热带树木作物种子发育的基本生物学提供了见解。
结果
概述和微阵列分析
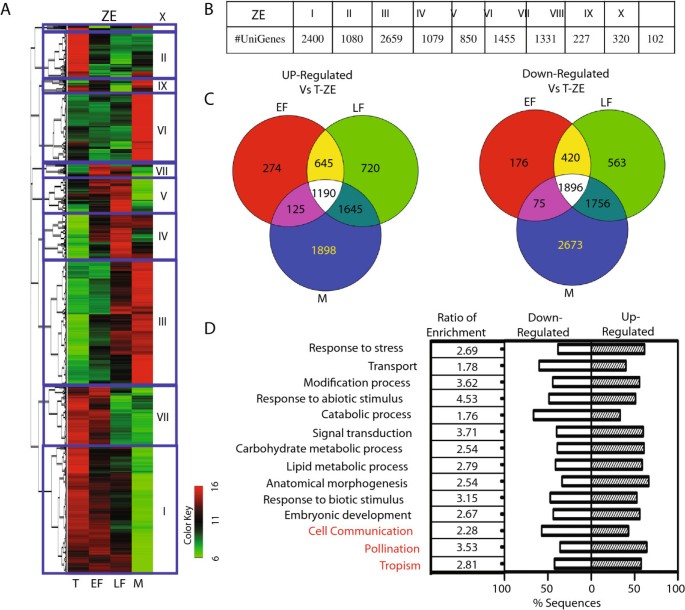
利用已发表的可可克里奥罗基因组汇编V1.0 [1],制造了一个代表28752个基因的全基因组12 plex Nimblegen表达微阵列,其中只有46个预测的可可基因没有被至少一个探针表示。为了研究可可胚胎发育,从6种不同组织类型和胚胎发育阶段的4个生物重复中分离出总RNA:合子胚胎组织-鱼雷(T-ZE)、早满(EF-ZE)、晚满(LF-ZE)和成熟(M-ZE)胚胎;体胚-鱼雷晚期(LT-SE)和成熟(M-SE)阶段。胚胎在这些阶段的代表性图像如图所示1a . 24个RNA样本同时用双色荧光标记杂交到单个芯片上。在背景减法和归一化之后,生成了一个热图来表示合子胚胎和体细胞胚胎成熟过程中基因表达的整体动态(图1B).数据集的聚类分析表明,SE的两个发育阶段与ZE成熟发育阶段的剖面聚类最接近。
合子胚胎发育过程中基因表达的动态
为了识别ZE成熟过程中的差异表达基因,配对t检验分析(p < 0.01,校正Bonferroni校正),截断p < 0.01,≥2倍变化。在这个数据集中鉴定出10288个基因被上调或下调(附加文件)1).该列表包括与T-ZE表达值相比,在至少一个阶段差异表达的所有重要基因。k -均值聚类建立了10个代表不同表达模式的聚类,包含102到2659个显著调控基因(图2A和B)。相对于T-ZE,大组基因在每个阶段都有独特的调控,并且还观察到所有阶段之间的基因组重叠。差异表达基因的总数(相对于T-ZE)在胚胎发生过程中依次增加(图2C)。
为了进一步探索ZE成熟过程中涉及的生物学途径,我们将基因本体(Gene Ontology, GO)术语分配给显著调控的基因,发现5700个基因具有与各种生物学过程相关的注释(图2D).我们探索了氧化石墨烯类别在受显著调控的基因集中的富集情况,并将其在整个基因组中的比例进行了比较。14种不同的氧化石墨烯种类显著富集,其中对非生物和生物刺激的反应以及对压力的反应富集程度最高(图2D、附加文件2).
体细胞胚成熟过程中基因表达的动态
利用体细胞胚发育晚期(LT-SE和M-SE)的基因表达谱进行了比较p< 0.01和≥2倍变化过滤器,检测基因表达水平显著差异。显著基因(4420个)分布在4个主要的k -均值簇(附加文件)3.而且4).GO富集分析表明,“转运”、“分解代谢过程”、“信号转导”和“应激反应”相关基因在从LT-SE阶段向M-SE阶段过渡过程中富集(附加文件)4B).除了三个GO类别外,其他所有GO类别也在ZE发展中得到了丰富(图2D)。综上所述,我们的结果表明,可可合子胚和体细胞胚的成熟在很大程度上具有相似的转录组谱,但在ZE中存在一些独特的差异,包括参与细胞通信、授粉和向性的基因(图2D),在细胞周期,次生代谢,细胞稳态在SE(附加文件4).
合子胚胎和体细胞胚胎成熟的比较
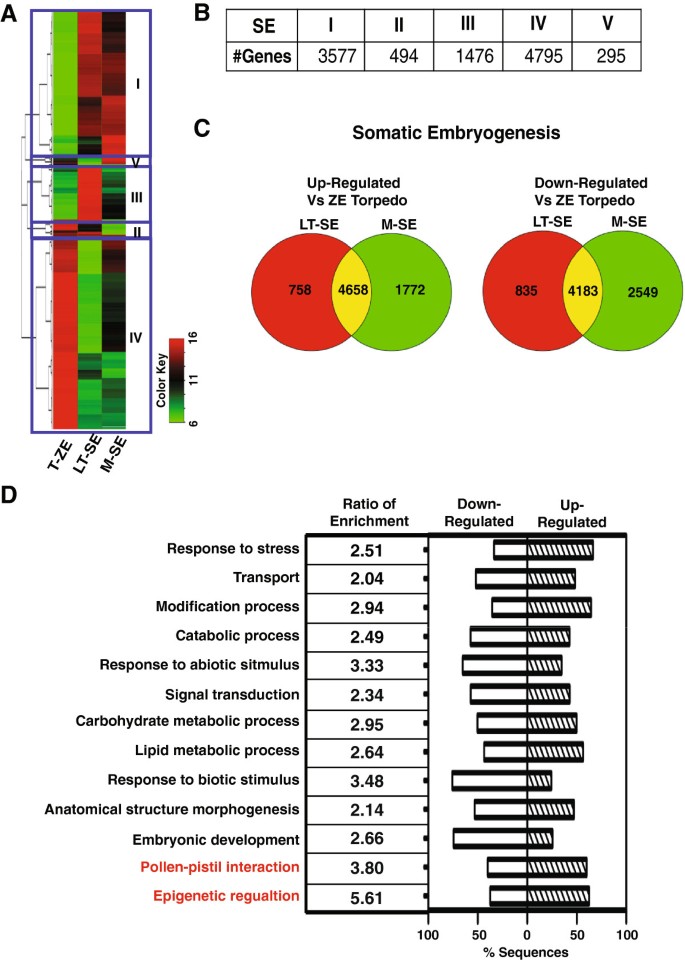
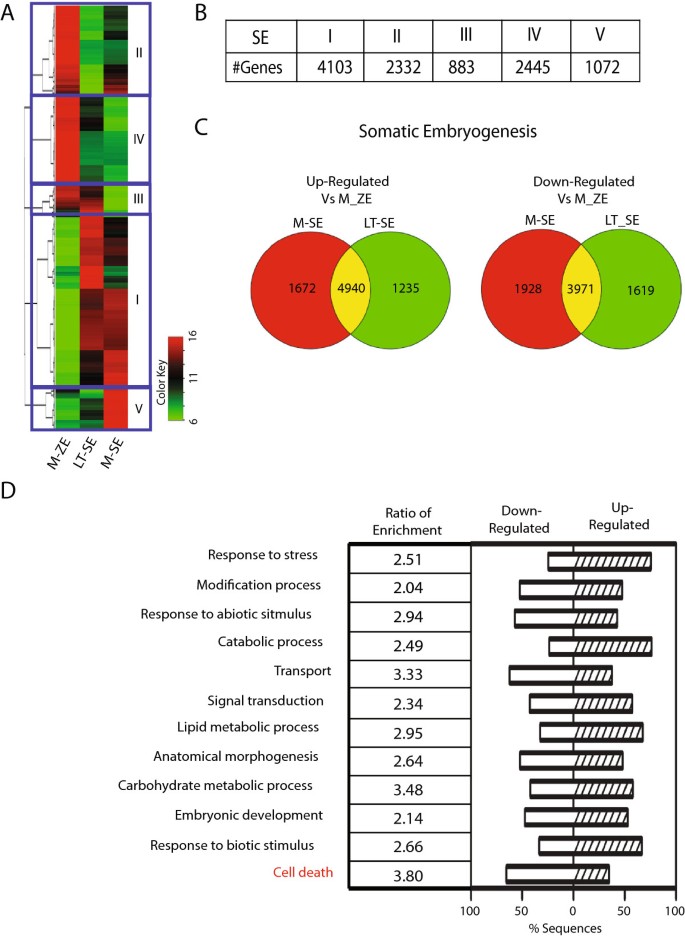
为了进行比较分析,我们进行了两种不同的聚类分析,将两个体胚期与T-ZE或M-ZE合子期进行对比(图3.而且4).在SE/T-ZE比较中检测到大量差异调控基因,这些基因被分为五个簇(图3.B和附加文件5).T-SE和T-ZE之间差异表达基因的数量(10,434)与从T-ZE到M-ZE发育转变中鉴定的差异表达基因的数量相当(11,258个基因,图2C),这说明了鱼雷期体细胞胚胎和合子胚胎之间的巨大差异。
SE/M-ZE相比,基因表达差异较小(4708上调,4506下调,图4和附加文件6).其中,1949个基因被分配到上调和下调的组,因为它们的表达根据SE分期或高或低。与M-ZE在两个SE阶段的初始聚类一致,与T-ZE相比,差异表达基因的数量减少了30%。通过GO注释富集分析,探讨合子胚胎和体细胞胚胎基因表达谱的差异(图3.D和4D).对于这两个比较,最广泛代表的富集氧化石墨烯术语与对非生物和生物刺激的反应、代谢和细胞过程相关。我们还仅在T-ZE特异比较中检测到“花粉-雌蕊相互作用”和“表观遗传调控”基因的富集(图3.D).在成熟的合子胚胎比较中,“Cell death”被独特地富集,这表明在不同的胚胎成熟模式中,凋亡的调控存在差异(图4D)。
合子胚胎和体细胞胚胎成熟中差异表达基因的功能分类
与K-means聚类方法鉴定的10个具有相似基因表达谱的基因簇相比(图2A和附加文件7)在比较ZE分期时,将两个SE发育阶段与T-ZE比较时,鉴定出5个基因表达簇(图3.而且4和附加文件8).为了获得不同聚类中所涉及的生物过程的定量评估,进行了氧化石墨烯富集分析(附加文件)7而且8).每个聚类中占主导地位的功能GO术语注释与“代谢和细胞过程”相关。ZE组中下调的I簇在信号和生物调控方面的基因数量比例较大(5%)。在SE中,下调的簇IV包含更多的“细胞过程”基因,而在上调的簇中获得更多的“代谢过程”基因(附加文件)8).这些结果表明,代谢过程的上调可能在体细胞胚成熟中起重要作用。这使我们进一步研究了与种子发育相关的特定基因类别,即种子主要代谢途径的转录因子和主要酶。
转录因子基因和主要种子代谢途径相关基因的表达谱
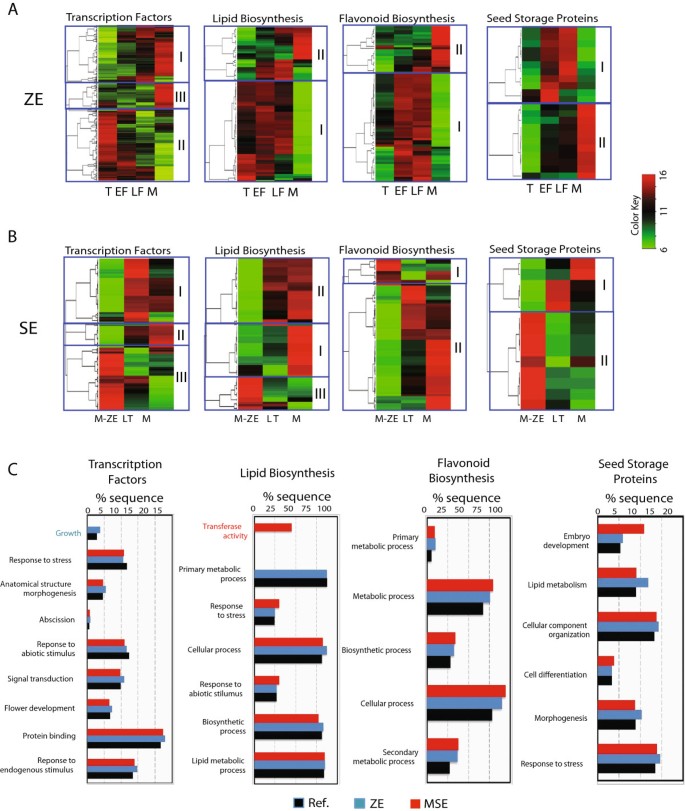
基于模型植物系统积累的知识,我们选择了转录因子、脂质生物合成、类黄酮生物合成和种子贮藏蛋白4个功能基因组进行分析(图5我们研究了这些类别中在合子胚胎成熟过程中差异表达的所有基因,并基于两个SE发育阶段最接近M-ZE的观察,我们选择了相对于成熟合子胚胎的体胚成熟阶段进行比较。
合子胚和体细胞胚成熟过程中转录因子基因和主要种子代谢途径相关基因的表达谱。(一)合子胚成熟过程中所有差异表达的转录因子、脂质生物合成、类黄酮生物合成和种子贮藏蛋白基因的热图:鱼雷(T)、早期饱满(EF)、晚期饱满(LF)、成熟(M)。用K-means算法将差异表达的基因分组为不同的簇;(B)体胚成熟过程中所有差异表达的转录因子、脂质生物合成、类黄酮生物合成和种子储存蛋白基因的热图:与成熟合子胚胎阶段(M-ZE)相比,鱼雷晚期(LT-SE)和成熟(M-SE)。使用K-means算法将差异表达的基因分组到不同的簇中。(C)GO富集分析了ZE和SE中所有差异表达的转录因子、脂质生物合成、类黄酮生物合成和种子贮藏蛋白基因的结果。使用blast2go软件进行比较。蓝色的GO类别仅代表ZE组富集的基因,红色仅代表SE组富集的基因。
转录因子基因
根据Mitsuda和Ohme-Takagu的一篇文章[25]生成了一个植物转录因子列表,用于在可可基因组中执行tBlastX同源性搜索。同源性分析鉴定出736个可能编码转录因子(Tc-TF)的可可基因,其中463个和417个分别在ZE和SE成熟过程中差异表达(附加文件)9而且10).其中54%(303)在ZE和SE成熟过程中均有差异表达。在至少一个阶段,差异表达的Tc-TF基因的K-means聚类在SE和ZE发育过程中都产生了3个亚簇(簇I和簇III上调,簇II下调,图5A和B)。
氧化石墨烯富集分析表明,转录因子的主要功能类别的表达只有微小的差异(图5C).使用MapMan软件对重要Tc-TF基因进行功能分类[4](附加文件11).MapMan分析显示,在“乙烯代谢和乙烯反应”类别中有26个转录因子,其中19个在SE中表达水平显著高于ZE。这些可可基因大多与乙烯快速诱导的拟南芥基因有关,属于ERF/AP2家族,控制介导乙烯生理和发育反应的蛋白质的合成[26].这可能表明乙烯,一种已知的应激反应激素,正在我们的培养系统中积累,这可能引发下游乙烯介导的不利于胚胎发育的应激反应。
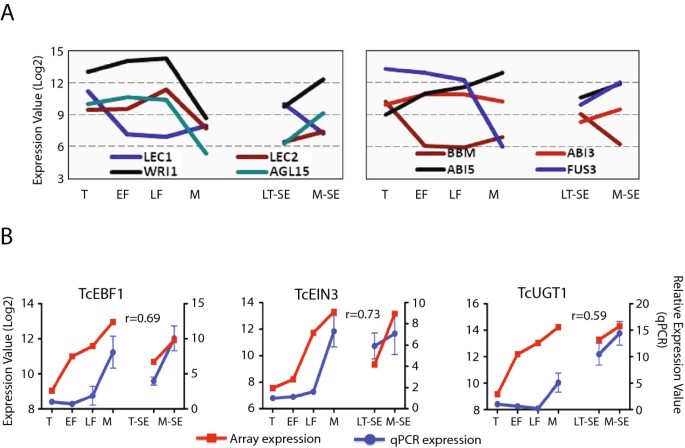
可可TF基因的一个目标子集,类似于其他物种的基因,以前被认为在胚胎发生中起作用[5,13,27- - - - - -30.]进行了检查,包括:TcLec1(Tc07g001180),TcWRI1(Tc10g012790),TcLec2(Tc06g015590),TcAGL15(Tc01g040120),TcBBM(Tc05g019690),TcABI5(Tc05g008870),TcABI3(Tc01g024700),TcFUS3(Tc04g004970)(图6A).与其他ZE期相比,M-ZE期表达值显著降低的基因包括TcLec1,TcLec2,TcWRI1,TcAGL15,TcFUS3.令人惊讶的是,除了TcLec1,这些基因在M-SE样本中的表达高于不成熟的LT-SE样本。TcBBM而且TcABI5,从LF-ZE到M-ZE表达略有下降。TcBBM然而,在SE阶段表现出相反的行为(图6A,右边面板)。这些结果揭示了合子胚胎和体细胞胚胎之间与胚胎发生相关的转录因子表达的巨大差异,这可能导致了这些结构在形态外观上的巨大差异(图1一个)。
可可合子胚和体细胞胚成熟过程中脂肪酸生物合成途径的差异调控
可可种子中含有约50%的可可脂,可可脂是工业用途中最有价值的脂肪之一,而子叶中脂肪的积累与种子发育成熟密切相关[31].我们研究了84个可可基因(Tc-FA)的表达变化,这些基因之前被鉴定和注释为可能参与脂肪酸生物合成[1].在M-SE和M-ZE的比较中,40个差异表达基因中有32个在M-SE中上调(附加文件)12).重要Tc-FA基因的功能特征表明,与M-ZE相比,M-SE中大量的质体脂肪酸生物合成基因上调,这表明成熟SE组织中脂肪酸代谢途径上调(附加文件)12).例如,基因参与脂肪酸生物合成的起始步骤,如乙酰辅酶a羧化酶(Tc08g009450;EC 6.4.1.2,3.6;折上调),乙酰辅酶a羧化酶生物素羧基载体蛋白2(Tc04g010240;BCCP;EC 6.4.1.2;6.2倍上调),生物素羧化酶(Tc00g000210;Cac2, ec 6.3.4.14;10.3倍上调)和几个亚型3-ketoacyl-CoA合酶(Tc00g015810而且Tc04g024470;;;与M-ZE相比,M-SE上调47倍),均控制碳通量导致新创脂肪酸生物合成。此外,与M-ZE相比,M-SE中涉及脂肪酸谱测定的基因也上调,包括stearoyl-ACP desaturase(Tc08g012550;FAB2,EC 1.14.19.1),oleoyl-ACP thioesterase(Tc01g022130;法达;EC 3.1.2.14),质体β-酮酰基- acp合成酶II(Tc09g006480;KASII;EC 2.3.1.179),Plastidial omega - 3去饱和酶(Tc05g002310;FAD7/8;EC 1.14.19).与合子种子组织相比,这些关键酶表达水平的差异可能导致成熟体细胞胚胎中观察到的脂肪酸不饱和程度更高(Y. Zhang,未发表数据)。值得注意的是,脂质积累的动态可能有助于观察到的FA转录动态:在所选的成熟合子胚胎阶段之前已经发生了大量的脂质积累,但在“成熟”体细胞胚胎阶段没有发生。
可可合子胚和体细胞胚成熟过程中类黄酮代谢基因的差异调控
类黄酮在植物王国中普遍存在,具有多种功能,包括防御、紫外线防护和生长素运输抑制[11,32- - - - - -34].94个可可基因的表达(Tc-FB, [1]),与已知的类黄酮生物合成基因同源,其中49个和52个分别在ZE成熟和SE成熟过程中差异表达(附加文件)13而且14).使用K-mean分析来生成两组上调和下调的基因簇(图5在SE中调控的52个基因中,有39个在ZE中也被调控。
分析还表明,在SE/M-ZE比较的52个差异表达基因中,有45个基因在M-SE中表达显著高于M-ZE,表明M-SE中次级代谢更活跃。MapMan分配的功能类别支持了Tc-FB基因确实参与次生代谢产物的合成,包括类黄酮、苯丙素和木质素,但MapMan还将其中6个基因分配到“乙烯合成和降解”类别(附加文件)15),表明黄酮类化合物和乙烯生物合成途径可能共同调控。这些基因包括:柚皮素,2-氧戊二酸3-双加氧酶(Tc01g033570、Tc03g022510、Tc01g001700),Leucoanthocyanidin加双氧酶(Tc03g026420)、黄酮醇合酶基因(Tc08g010270)、黄酮醇合酶/黄酮3-羟化酶(Tc05g019560).这些基因中的五个(不包括Tc01g033570)在M-SE中表达高于M-ZE。
可可合子胚和体细胞胚成熟过程中种子贮藏蛋白基因的差异调控
在大多数高等植物中,胚胎成熟与大量储存蛋白质的积累有关,这是由激素、遗传和代谢控制共同调节的[35,36].根据Higashi [37种子中贮存蛋白质的清单拟南芥生成查询,在可可基因组内执行tBlastX同源性搜索。在我们的分析中,我们选择了37个编码潜在种子储存蛋白(Tc-SSP)的可可基因。LIMMA测试分别鉴定了其中13个和9个在ZE和SE成熟过程中差异表达的基因(图5A和B,附加文件16).K-mean分析为ZE组产生了上调(聚类II)和下调的聚类I,而SE组则产生了“低”和“高”表达的基因聚类(分别为聚类I和聚类II)。13个差异表达基因包括1个vicilin基因,1个推测为甜蛋白mabinlin基因,3个编码21-kDa胰蛋白酶抑制剂蛋白基因,7个编码为胚胎发生晚期蛋白质丰富(LEA)(附加文件16).与M-ZE相比,在SE成熟过程中鉴定出的重要基因包括2个相同的21-kDa胰蛋白酶抑制剂基因,其在M-SE中的表达量高于M-ZE(11.77倍和2.08倍)。编码LEA蛋白的7个ZE显著基因中的6个在SE中也有差异表达。在ZE成熟过程中,9个差异表达中的6个表达LEA基因随发育而增加,在M-ZE末期达到最高表达值;剩下的3个LEA基因表现出相反的模式,M-ZE表达减少。相比之下,SE期间6个差异调控LEA蛋白的表达值明显低于M-ZE(附加文件)16).尽管从LT-SE到M-SE阶段,所有6个LEA基因的表达都有所增加,但这些结果表明,与M-ZE相比,两个SE阶段可能发展得更早。
一个有趣的观察是基因的表达Tc01g005060M-SE显著低于M-ZE。这个基因与拟南芥几乎完全相同ABA1该基因编码一种玉米黄质环氧化酶,对合成脱落酸(ABA)的第一步至关重要。由于已知ABA对诱导参与储存化合物合成和胚胎成熟的基因很重要,这也表明与ZE样品相比,SE样品处于更早的发育阶段。
通过RT-qPCR验证芯片数据
为了验证微阵列结果,使用随机选择的10个显著调控基因进行RT-qPCR分析。M-ZE和LT-SE样品的基因表达比较使用相同的RNA提取用于微阵列杂交。这10个基因在微阵列和RT-qPCR分析中表现出相似的表达模式(附加文件17).尽管r20.54的值表明,在定量绝对值上只有适度的相关性,在检测的10个基因中,除1个外,ZE和SE样品之间的表达水平的相对差异在微阵列和RT-qPCR数据中是相同的。因此,我们认为微阵列数据可以充分测量相对基因表达的变化,而RT-qPCR数据可以更准确地量化精确的定量差异。
考虑到观察到大量参与乙烯信号通路的TF基因似乎在ZE和SE发育过程中都受到调控,我们还额外验证了三个选择的涉及乙烯信号通路的基因的表达模式:tcein3结合F-box蛋白1, (TcEBF1, Tc09g011440),乙烯不敏感3(TcEIN3, Tc09g033150),TcIndole-3-acetate beta-glucosyltransferase2 (TcUGT1, Tc02g020270).从微阵列和RT-qPCR数据中绘制出每个发育阶段的平均表达值。在所有三个基因的两种分析的表达值之间观察到明显的相关性(图6B)。
讨论
植物的发育过程非常复杂,需要许多相互关联的分子过程的相互作用,这些分子过程的活动需要精细控制和协调。我们的全基因组表达分析揭示了这种复杂性,因为它与可可胚胎成熟的机制有关。在近29,000个基因中,约36%的基因在合子胚胎成熟过程中表达差异,上调和下调基因的数量大致相等。显著基因的数量随着成熟而增加,在成熟的合子胚胎中达到11258个。这些结果表明,可可合子发育的特征是基因表达的显著变化。这与拟南芥的研究一致,其中54.9%(约14,000个独特mrna,总数为25,498)在不同胚胎发育阶段差异表达[1,3.].在目前的研究中,在合子胚胎发生和体细胞胚胎发生中,我们观察到大量编码转录因子(TFs)的差异调控基因。这与Gliwicka et al., 2013探索拟南芥体细胞胚胎发生过程中基因表达变化的研究结果一致[38].他们观察了729个在SE期间表达发生变化的tf。与我们的结果一致,他们报道了许多TF基因在合子胚胎发育后期下调,但在体胚发育后期继续高表达。与在可可中一样,许多tf被注释为与植物发育、植物激素和应激反应相关的功能。
SEs和ZEs之间的一个主要区别是子叶的成熟,在ZE中,子叶成熟导致含有类黄酮、脂类和种子储存蛋白质的大型折叠组织,而在SE中,只导致小而不发达的组织。虽然SE的基因表达谱与成熟的ZE表达谱的聚集性最强,但也鉴定了一组在SE中强烈表达的基因,这表明特定的代谢途径可能与SE特异性相关,并有助于解释子叶发育的差异。
对导致种子发育的特定基因和途径的进一步分析表明,虽然许多种子发育的特定基因在两种类型的胚胎中都有表达,但许多与生长素和乙烯反应以及胁迫反应相关的基因在SEs中表达上调。这些基因中的许多在种子发育的早期阶段表达水平较高,在SEs中没有像在正常种子发育中那样下调。基于这些结果,我们推测体胚子叶发育不全是由于培养过程中应激反应信号的积累导致基因表达模式改变的结果。这可能是由次优介质组成或环境条件引起的压力引起的。
激素和类黄酮生物合成基因的调控
超过150个生长素或乙烯代谢相关基因和352个生物和非生物胁迫相关基因在SEs中有差异表达。这表明在在体外条件,乙烯信号在可可SEs中上调,导致乙烯介导的应激反应基因表达增加。在白云杉和黑云杉体细胞胚胎发生中也报道了类似的观察结果,乙烯信号的增加与异常胚胎发育有关[39,40].在SEs中,增加的应激反应基因表达限制了子叶的发育,这是子叶储备资源重新分配的结果。
乙烯也参与黄酮醇生物合成的上调Arabidposis[41,42],这与我们观察到的可可体胚相对于合子胚中参与类黄酮生物合成的基因表达升高一致。类黄酮通常与植物应激反应有关,它们似乎通过调节生长素运输在响应生长素和乙烯信号的生长发育控制中发挥作用[42,43],因此对调节种子发育至关重要。综上所述,体胚中大量参与乙烯功能的基因上调与乙烯介导的应激反应在可可体胚异常发育中起重要作用的假设是一致的。
体胚脂肪酸生物合成的调控
我们还观察到体细胞胚胎中控制脂代谢的关键酶编码基因的高水平错表达。我们观察到表达增加FAB2[44,45),KASII[46),法达[47都与油酸的合成有关,油酸是多不饱和脂肪酸生物合成的底物。此外,ω - 3和ω - 6去饱和酶基因表达上调FAD2[45),FAD7/8[48]也与我们最近的观察相一致,与可可种子相比,成熟的可可SEs含有更高比例的多不饱和脂肪酸,包括亚油酸和α-亚麻酸,但硬脂酸和油酸的比例较低(未发表的数据,Yufan Zhang),这是未成熟合子种子和可可叶的特征[31].
结论
在可可合子胚胎成熟过程中,基因组的很大一部分受到差异调节,这与其他物种的报道相似。综上所述,合子胚胎发育成熟阶段的主要基因表达程序仅在体胚成熟阶段部分表达。考虑到所涉及的环境和发展途径的显著不同,这并不奇怪。本研究结果表明,生长素、乙烯和黄酮类化合物的生物合成和调控基因在SEs中异常表达,这些通路可能是未来体细胞胚胎发生过程优化的重要靶点。
本研究提供了一份全面的差异表达基因列表及其表达谱,可进一步作为ZE和SE成熟的标记物。这些数据代表了与这两种胚胎发育过程相关的代谢过程的有用的新知识t .可可,并为功能基因组学研究提供资源在体外优化培养基和环境条件,以改善可可SE体系。这些发现将为进一步分析这些因素提供假设和工具,并导致改进的可可体细胞胚方案。
方法
植物材料
合子胚胎(ZE)
可可豆荚是在宾夕法尼亚州立大学温室用基因型西非Amelonado植物的花粉对基因型Scavina6 (Sca6)植物进行人工授粉获得的。果实成熟期为授粉后14周(WAP),分别为鱼雷期(T-ZE), 16 WAP(早满,EF-ZE), 18 WAP(晚满,LF-ZE)至20 WAP(成熟,M-ZE),此时豆荚完全成熟。从每个荚果中提取合子胚胎,切除子叶组织,在液氮中冷冻,并保存在- 80°C下用于RNA提取。对于每个发育阶段,冷冻四组生物重复。
躯体胚胎(SE)
临时浸泡系统生物反应器中产生次级SEs [49,50]源自基因型Sca6的花部分,如前所述[18],并进行了如下微小的修改:在生物反应器中使用直径为2毫米的玻璃珠来支撑胚胎。体细胞胚胎阶段的详细描述先前已报道过[18,19,51,52].选择处于鱼雷早期发育阶段的SEs,转移到胚胎发育(ED)培养基(每个生物反应器20个胚胎)。在ED培养基上培养2周后,在以下两个发育阶段收获组织:1)30 g/L蔗糖培养的整个晚期鱼雷SEs (LT-SE); 2) 60 g/L蔗糖培养的成熟SEs (M-SE)子叶组织(见图)1).将每个生物复制的1克组织冷冻在液氮中,并保存在−80°C下用于RNA提取。每个发育阶段收集4个重复的生物,并立即在液氮中冷冻。
总RNA提取
总RNA的提取方法由[53,54].简单地说,冷冻样品在液氮中研磨,粉末悬浮在含有4 M异硫氰酸胍、0.24 M乙酸钠、0.03 M n -月桂酰肌氨酸钠盐、22.5 mM PVP-40 (MW 40,000)和14 mM β-巯基乙醇的萃取缓冲液中。4℃14000 g离心30 min后,提取氯仿rna,用异丙醇沉淀,用70%乙醇洗涤,然后在无rnase的无菌水中重新悬浮。然后用DNase l (Invitrogen)处理RNA,并使用RNA 6000纳米测定生物分析仪(Aglient)评估完整性,使用RNA完整性编号为7或以上的RNA。
微阵列设计与制造
根据基因组序列Theobroma可可(混血儿)[1],使用28798个预测基因模型提取所有预测编码序列(CDS),对于那些预测3' untranslation region (UTR)序列的基因,我们提取22489个额外的3' UTR序列。3' utr被纳入,允许基因特异性探针解析多基因家族的表达特异性。所得到的51,287个序列用于设计独特的60 mer寡核苷酸探针(NimbleGen Chip design Service)。最终选择134357个60 mer寡核苷酸序列(84,650个CDS探针和49707个3'UTR探针),平均每个基因序列4.67个探针(每CDS 2.93个探针和每3'UTR 2.21个探针),其中4473个目标序列要么太短,要么复杂度有限,无法设计探针。在原始的28798个基因模型中,只有46个基因没有出现在该阵列中。
单个探针元件是在NimbleGen 12-plex微阵列上合成的,每张幻灯片上有12个子阵列,每个子阵列上有135,000个探针位置。随机对照序列被用来填充数组上的任何剩余空间。
探针标记,杂交和检测
杂交由PSU的基因组学核心设施根据公布的NimbleGen微阵列设施协议进行[55].在标记和杂交前,使用mRNA扩增试剂盒(Amino Allyl MessageAmp II™,Ambion, Austin, TX, AM1753)扩增1 μg总RNA (RNA完整性数(RIN)为7或以上)。将aRNA与Cy3或Cy5 (GE Health Care #RPN5661)偶联染色,然后根据Ambion Kit说明书进行纯化。对24个样品进行配对,对于一个12 plex阵列,每个Cy3标记的样品(1.5 μg)与Cy5标记的样品(1.5 μg)结合,并根据制造商的说明使用RNA裂解试剂(Ambion AM8740)进行裂解。破碎后,样品在快速真空中完全干燥。重悬的样品对随后在42°C下杂交到单个12 plex阵列18小时。根据制造商说明书(Roche NimbleGen)清洗阵列以去除非特异性结合目标,并使用相关软件使用Axon 4000A扫描仪进行扫描。
数据分析
R编程环境[56]和Bioconductor软件进行分析[57].探针级数据的图像图允许对微阵列数据异常进行评估。日志224个阵列的图像图显示“正常”高质量阵列数据,使用控制探针进行了背景和质量检查。
数据经过质量预处理后,得到每个基因的最终表达值。我们使用LIMMA包进行背景调整、归一化和汇总[58].采用RMA程序,在多阵列模型拟合的基础上,使用中值抛光算法进行卷积背景校正、分位数归一化和汇总,获得平均Log2每个基因的表达值(所有探针的平均值)在2到16之间。背景噪声计算为控制探头的平均强度水平,值为6 (Log2)作为截止值,以消除任何由噪声引起的信号。统计学意义是使用Bioconductor的“LIMMA”包中实现的调节t统计量。它基于实证贝叶斯方法,详细资料见[59].计算调节后的t统计量及其对应的p值进行统计分析。在本研究中,我们以截点p < 0.01和≥2倍变化作为统计分析的常规截点进行分析。数据保存在NCBI的基因表达Omnibus (GEO) [60, GEO项目GSE55476http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE55476).
聚类分析
将SE组和ZE组归一化和过滤后的数据用r中的LIMMA包进行分析。根据6个样本的差异表达基因的表达模式,采用k-means无监督聚类技术将差异表达基因聚类到不同的组。简而言之,该算法任意地将基因(“载体”)分为不同的组。每组的质心是通过平均每个基因的坐标来计算的。在一次迭代中,每个克隆被重新分配到它最接近的质心,并重新计算质心的坐标。
GO注释使用Blast2GO
可可基因组数据库中的所有预测基因[1]使用Blast2GO进行功能注释[61]使用默认参数对非冗余(nr)蛋白质序列数据库。同样,使用Blast2GO软件v2.5.0从检索到的数据库匹配中,通过GO映射函数获取基因本体(Gene ontology, GO)信息。最后,使用“ANNEX (Annotation Argumentation)”函数来细化注释。GOslim goslim_plant。obo '用于生成特定的GO术语。输出数据导出为注释格式文件(annot),选择不同聚类中的基因id生成饼图。GO的层次表示是根据不同的级别进行结构的,从最高(一级)父术语对应3个主要的GO类别(细胞成分,生物进展,分子功能)到最低,更专业的子术语(二级,三级,四级等)。在我们的研究中,GO注释的数据集表示在第2级。
去充实
用Blast2GO软件进行GO富集分析(Fisher’s Exact Test),鉴定差异表达基因的全基因组GO类别。在此分析中,使用可可注释文件(如上所述)被用作执行分析的参考。GO术语假定值小于0.01被认为在我们的差异表达基因集中有显著差异和丰富。
MapMan代谢途径分析
地图人[62包含所有预测基因的映射文件Theobroma可可基因组(Criollo)数据库[1通过MapMan网站使用墨卡托管道自动序列注释生成。简而言之,我们将28802个肽序列FASTA文件上传到Mercator工具(分配默认参数加上保守参数和InterProScan),以便与包含已知蛋白质序列的参考数据库进行比较,以获得功能注释。生成的映射文件包含Tc基因id,并指定了MapMan bin(基因功能类别)。该文件用于MapMan分析,其中包括重要基因进入代谢途径的位置和可视化。从先前的微阵列分析中富集的差异表达基因被分组在各自的功能类别中。将M-SE和M-ZE基因表达的倍数差异纳入每个基因。蓝色所示的正值表示M-SE与M-ZE相比基因表达增加。红色所示的负值表示M-SE与M-ZE相比基因表达的减少。
实时荧光定量PCR分析
基因特异性引物(从微阵列中随机选择的10个基因和2个对照管家基因在宾夕法尼亚州立核酸设施用MerMade12自动DNA合成器合成(Bioautomation, Plano TX)(附加文件)18).基因特异性荧光探针由Biosearch Technologies (Novato, CA)合成。在可可基因探针的5'端使用的荧光标记是6-羧基荧光素(6-FAM),基因探针3'端的猝灭剂是BHQ1 (Biosearch)。25 μl PCR反应包括:5 μl cDNA (~12.5 ng), 12.5 μl 2X TaqMan®Universal Master Mix (#4304437, Applied Biosystems, Foster City, CA),每个引物400 nmol,探针200 nmol。PCR反应在应用生物系统公司7300 Q-PCR系统(福斯特城,CA)的96孔薄壁PCR板上进行,反应条件如下:在50°C下2分钟。,10min at 95°C, followed by 40 cycles of 15 sec at 95°C and 1 min at 60°C. Each sample was amplified in duplicate and the results were averaged.
两种可可管家控制基因肌动蛋白和泛素的平均表达量(Tc05g027250而且Tc09g021610)对数据进行归一化。所有靶基因和内参基因的扩增效率由每个样品稀释曲线的斜率计算(E = 10)(−1 /坡)1) (63].然后计算每个基因的平均效率,并用于效率数据校正。采用REST软件计算数据归一化、效率校正、统计随机化检验和相对转基因/对照非转基因表达比[64].比(折差)与假定值小于0.05为显著性。
参考文献
Argout X, Salse J, Aury JM, Guiltinan MJ, Droc G, Gouzy J, Allegre M, Chaparro C, Legavre T, Maximova S, Abrouk M, Murat F, Fouet O, Poulain J, Ruiz M, Roguet Y, rodieer - goud M, Barbosa-Neto J, Sabot F, Kudrna D, Ammiraju J, Schuster S, Carlson J, Sallet E, Schiex T, Dievart A, Kramer M, Gelley L, Shi Z, Bérard A: Theobroma coco的基因组。自然遗传学,2011,43(2):101-108。
张志刚,刘志刚,刘志刚,刘志刚:拟南芥胚胎模式的研究进展。细胞发育生物学杂志,2007,23:207-236。
Linkies A, Graeber K, Knight C, Leubner-Metzger G:种子的进化。中国生态学报,2010,36(4):344 - 344。
以扫K:胚胎和幼苗。种子植物解剖,第二版。纽约:John Wiley & Sons出版社,1977:475-500。
京晶:LECs在胚胎发育过程中变得疯狂。植物科学进展,2008,13(12):624-630。
Spencer MW, Casson SA, Lindsey K:拟南芥胚的转录谱分析。植物科学进展,2007,26(2):344 - 344。
组织培养介导的生物技术对石榴的干预:综述。植物细胞学报,2011,30(5):707-721。
Steward FC, Pollard JK, Patchett AA, Witkop B:氮化合物对植物组织培养生长的影响。生物化学与生物物理学报,1958,28(2):308-317。
体细胞胚胎发生:高等植物早期发育的一个模型。中国生物医学工程学报,1993,5(10):1411-1423。
santo - mendoza M, Dubreucq B, Baud S, Parcy F, Caboche M, Lepiniec L:拟南芥种子发育和成熟的基因调控网络的破译。植物学报,2008,44(4):366 - 366。
Broun P:类黄酮生物合成的转录控制:一个涉及拟南芥分化多个方面的保守调节的复杂网络。植物学报,2005,8(3):272-279。
李华昌,庄k, Henderson JT, Rider SD,白杨,张华,Fountain M, Gerber J, Ogas J: PICKLE在萌发过程中抑制胚性状的表达。植物学报,2005,44(6):1010-1022。
Cernac A, Benning C: wrinkle - 1编码一个AP2/EREB结构域蛋白,参与控制拟南芥贮藏化合物的生物合成。植物学报,2004,40(4):575-585。
Goldberg RB, de Paiva G, Yadegari R:植物胚胎发生:合子到种子。科学通报,1994,26(4):528 - 528。
Lopes MA, Larkins BA:胚乳的起源,发育和功能。中国生物医学工程学报,1993,5(10):1383-1399。
Sabelli PA, Larkins BA:草类胚乳的发育。植物生理学杂志,2009,149(1):14-26。
Berger F, Grini PE, Schnittger A:胚乳:种子生长发育的整合器。植物学报,2006,9(6):664-670。
王晓明,王晓明,王晓明,王晓明,王晓明,王晓明:植物体细胞胚发生的基因型变异及其与细胞起源的关系。植物营养学报,2002,29(4):357 - 357。
李智,赵晓明,王晓明,王晓明,王晓明:可可花外植体的体细胞胚胎发生与植株再生(可可树。)使用噻地脲。中国生物医学工程学报,1998,29(3):344 - 344。
李志刚,李志刚,李志刚:可可花胚的体细胞胚发生。林学学报,1996,30(4):344 - 344。
李志刚,李志刚,李志刚。可可树合子胚胎发生与花外植体体细胞胚胎发生的比较。植物学报,1997,33(3):163-172。
Lopez-Baez O, Bollon H, Eskes A, Petiard V: Embryogenèse somatique de cocooyer Theobroma cocoa L. á partir de pièces florales。中国科学院学报(自然科学版),1993,29(3):366 - 366。
马志刚,杨永杰,李志刚,李志刚:可可树体胚发生繁殖的田间表现。植物学报,2008,44(6):487-493。
博士论文。可可树的体细胞胚胎发生,胚转化,微繁殖和影响遗传转化的因素,2000,大学公园:宾夕法尼亚州立大学
杨晓明,杨晓明,杨晓明:拟南芥转录因子的功能分析。植物生理学报,2009,30(7):344 - 344。
Weber H, Hellmann H:拟南芥BTB/ POZ-MATH蛋白与ERF/AP2转录因子家族成员相互作用。FEBS杂志。2009, 276(22): 6624-6635。
郑强,郑勇,Perry SE: agamus - like15在拟南芥和大豆中通过调控乙烯生物合成和反应促进体细胞胚胎发生。中国生物医学工程学报,2013,26(4):344 - 344。
Boutilier K, Offringa R, Sharma VK, Kieft H, Ouellet T, Zhang L, Hattori J, Liu CM, van Lammeren AA, Miki BL, Custers JB, van Lookeren Campagne MM: BABY BOOM的异位表达引发了从营养生长到胚胎生长的转换。植物细胞学报,2002,14(8):1737-1749。
Nambara E, Keith K, Mccourt P, Naito S: ABI3基因在胚胎成熟建立中的调节作用拟南芥.发展。1995,121(3):629-636。
杨晓明,张晓明,张晓明,等。拟南芥脱落酸应答基因ABI5编码一种碱性亮氨酸拉链转录因子。植物学报,2000,12(4):599-609。
王志刚,王志刚,王志刚。硬脂酰酰基载体蛋白去饱和酶表达与脂肪脂肪酸组成的关系Theobroma-cacaoL胚胎。植物学报,1994,19(1):1 -8。
李平,李志刚,李志刚,李志刚,黄酮类化合物的遗传与生物化学研究。植物学报,2006,29(4):357 - 357。
Brown DE, Rashotte AM, Murphy AS, Normanly J, Tague BW, Peer WA, Taiz L, Muday GK:拟南芥体内黄酮类化合物对生长素转运的负调节作用。植物营养学报,2001,26(2):344 - 344。
Lazar G, Goodman HM: MAX1是类黄酮通路的调节因子,控制拟南芥营养性腋芽的生长。美国国家科学院院刊。中国农业科学,2006,34(2):472-476。
Gutierrez L, Van Wuytswinkel O, Castelain M, Bellini C:调控种子成熟的联合网络。植物科学进展,2007,12(7):294-300。
Itoh Y, Kitamura Y, Arahira M, Fukazawa C:大豆种子贮藏11S球蛋白基因顺式作用调控区及其与种胚因子的相互作用。植物分子生物学,1993,21(6):973-984。
Higashi Y, Hirai MY, Fujiwara T, Naito S, Noji M, Saito K:拟南芥种子的蛋白质组学和转录组学分析:种子蛋白质连续加工的分子证据及其在硫营养胁迫响应中的意义。植物学报,2006,48(4):557-571。
李志刚,李志刚,李志刚,李志刚。拟南芥体细胞胚胎发生过程中转录因子转录组的广泛调控。PLoS ONE。2013, 8(7): 1-20。
孔玲,杨娥:乙烯和乙烯抑制剂对白云杉体胚成熟的影响。中国植物科学,1994,26(1):1-8。
El Meskaoui A, Tremblay FM:乙烯参与不同成熟能力的黑云杉胚性细胞系的成熟。实验植物学杂志,2001,52(3):369 - 369。
杨晓明,王晓明,王晓明,等。乙烯对拟南芥根类黄酮积累和向地性的影响。中国生物医学工程学报,2006,40(4):344 - 344。
Lewis DR, Ramirez MV, Miller ND, Vallabhaneni P, Ray WK, Helm RF, Winkel BS, Muday GK:生长素和乙烯通过不同的转录网络诱导黄酮醇积累。中国生物医学工程学报,2011,26(1):344 - 344。
Grunewald W, De Smet I, Lewis DR, Lofke C, Jansen L, Goeminne G, Vanden Bossche R, Karimi M, De Rybel B, Vanholme B, Teichmann T, Boerjan W, Van Montagu MCE, Gheysenc G, Mudaye GK, Friml J, Beeckman T:转录因子WRKY23通过局部控制黄酮醇生物合成,协助生长素在拟南芥根发育过程中的分布模式。中国科学:自然科学,2012,29(3):357 - 357。
杨晓明,王晓明,王晓明,等。拟南芥硬脂酸突变体的研究进展。中国植物学报,2004,26(4):344 - 344。
Okuley J, Lightner J, Feldmann K, Yadav N, Lark E, Browse J:拟南芥FAD2基因编码多不饱和脂质合成所必需的酶。植物生理学报,2004,26(1):457 - 457。
王晓明,王晓明,王晓明,王晓明,王晓明。种子β-酮酰基-酰基载体蛋白合成酶的研究进展[J]:通过调控种子β-酮酰基-酰基载体蛋白合成酶的水平,将温带种子油的成分转化为热带棕榈油。中国科学:自然科学,2007,26(4):344 - 344。
DeVeaux LC, Cronan JE, Smith TL:允许反式不饱和脂肪酸取代大肠杆菌必需的顺式不饱和脂肪酸的突变(fatA)的遗传和生化特征。中国生物医学工程学报,2004,27(3):344 - 344。
Collados R, Andreu V, Picorel R, Alfonso M:大豆光合细胞悬浮液中omega3脂肪酸去饱和酶(FAD3, FAD7和FAD8)转录和转录稳定性的光敏感机制。清华大学学报(自然科学版),2006,37(4):344 - 344。
聂梅娜,李志刚,李志刚,李志刚,李志刚,李志刚:利用临时浸泡生物反应器对可可胚体胚的再生及不同组织游离氨基酸的分析。植物细胞学报,2008,27(4):667-676。
肖S:荣誉论文。植物组织培养繁殖临时浸泡生物反应器的改进设计。宾夕法尼亚州立大学:大学公园2012。
陈志伟,陈志伟,陈志伟。碳源和外植体类型对4种基因型可可体细胞胚胎发生的影响。HortScience。2006,41(3): 753-758。
Maximova SN, Young A, Pishak S, Miller C, Traore A, Guiltinan MJ:传播的集成系统Theobroma可可木本植物体细胞胚胎发生规程。编辑:Jain SM, Gupta PK. 2005,多德雷赫特,荷兰:施普林格,209-229。
李文杰,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚可可树.植物学报,2008,27(4):853-866。
Rodriguez Lopez CM, Wetten AC, Wilkinson MJ:愈伤组织衍生可可的遗传和表观遗传变异的渐进侵蚀(Theobroma可可)植物。植物保护学报,2010,36(4):344 - 344。
NimbleGen 4 plex和12 plex微阵列协议。http://www.huck.psu.edu/facilities/genomics-up/protocols/nimblegen-protocols,
统计计算R项目。http://www.r-project.org/,
Gentleman RC, Carey VJ, Bates DM, Bolstad B, Dettling M, Dudoit S, Ellis B, Gautier L, Ge Y, Gentry J, Hornik K, Hothorn T, Huber W, Iacus S, Irizarry R, Leisch F, Li C, Maechler M, Rossini AJ, Sawitzki G, Smith C, Smyth G, Tierney L, Yang JY Zhang J: Bioconductor:计算生物学和生物信息学的开放软件开发。中国生物医学工程学报,2004,5 (10):R80-
Ritchie ME, Silver J, Oshlack A, Holmes M, Diyagama D, Holloway A, Smyth GK:双色微阵列背景校正方法的比较。生物信息学,2007,23(20):2700-2707。
线性模型和经验贝叶斯方法评估微阵列实验中的差异表达。中华分子生物学杂志,2004,3:第3篇
Edgar R, Domrachev M, Lash AE:基因表达Omnibus: NCBI基因表达和杂交阵列数据库。中国生物医学工程学报,2002,30(1):207-210。
Conesa A, Gotz S, Garcia-Gomez JM, Terol J, Talon M, Robles M: Blast2GO:功能基因组学研究中注释、可视化和分析的通用工具。生物信息学,2005,21(18):3674-3676。
Thimm O, Bläsing O, Gibon Y, Nagel A, Meyer S, Krüger P, Selbig J, Müller LA, Rhee SY, Stitt M: Mapman:一种用户驱动的工具,可以将基因组学数据集显示在代谢途径和其他生物过程的图表上。植物学报,2004,37(6):914-939。
Bustin SA:定量PCR的A-Z。加州拉霍亚:国际大学线2004。
Pfaffl MW, Horgan GW, dempple L:相对表达软件工具(REST),用于实时PCR中相对表达结果的分组比较和统计分析。中国生物医学工程学报,2002,30 (9):e36-
确认
作者要感谢Kabin Xie, Muya Shu, Luis Mejia在生物信息学技术方面的帮助,以及Jeff Larsen在生物反应器安装方面的帮助。这项工作得到了NSF CBET拨款#1035072给WC, SM和MJG的支持。本材料中表达的任何观点、发现、结论或建议都是作者的观点,并不一定反映美国国家科学基金会的观点。这项工作的额外支持来自宾夕法尼亚州立大学可可分子生物学的美国可可研究基金会。Nicolas Niemenak获得了富布赖特奖学金。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
SM参与了整个项目的协调、实验设计、数据分析和解释;SF进行MapMan数据分析,转录因子基因数据分析和解释,微阵列数据形成并提交GEO数据库;XS进行微阵列数据分析;利用神经网络对树木进行授粉、合子胚样本采集、体胚组织培养及样本采集、RNA提取、qRT-PCR分析;YZ进行脂质生物合成基因数据分析和解释;WC和MG为实验设计和数据解释做出了贡献;所有作者均参与了稿件的准备和编辑工作。所有作者都阅读并批准了最终的手稿。
电子辅助材料
12870 _2014_1596_moesm1_esm.xls
附加文件1:合子胚胎成熟过程中差异表达的基因。相对基因表达值(log2)中所有基因在合子胚胎成熟期间相对于鱼雷期合子胚胎(T-ZE)上调或下调。列表包括基因上调或下调至少在一个发育阶段的比较和T-ZE(截止p< 0.01和≥2倍变化)。表示合子胚胎成熟期归一化表达值相对于鱼雷胚胎表达值的倍差(fold difference M-ZE/T-ZE)。基因ID号和预测功能(描述)是基于criollo cacao基因组浏览器V1.0注释[1].(xls2mb)
12870 _2014_1596_moesm3_esm.xlsx
附加文件3:鱼雷晚期与体细胞胚胎发育成熟阶段的差异基因表达。相对基因表达值(log2)中所有基因的上调或下调比较晚期鱼雷(LT-SE)和成熟(M-SE)阶段的体细胞胚胎发生(切断p< 0.01和≥2倍变化)。表示成熟期与鱼雷期体胚归一化表达值的倍差(fold difference M-SE/LT-SE)。基因ID号和预测功能(描述)是基于criollo cacao基因组浏览器V1.0注释[1].(xlsx412kb)
12870 _2014_1596_moesm4_esm.pdf
附加文件4:体细胞胚胎发育晚期和成熟发育阶段差异表达基因的聚类分析。(一)体细胞胚胎发生(SE)晚期鱼雷(LT-SE)和成熟(M-SE)发育阶段所有差异表达基因的热图。利用转化数据的K-means聚类对两个SE阶段进行比较,鉴定出2213个上调基因和2207个下调基因分布在4个簇中。(B)对LT-SE和M-SE所有差异表达基因进行GO富集分析(FDR < 0.01, Material and Methods)。红色标签中的GO类别表示在SE发育阶段富集的基因,但在ZE发育阶段不富集。(pdf 581 kb)
12870 _2014_1596_moesm5_esm.xlsx
附加文件5:体胚成熟过程中差异表达的基因与合子胚胎发生的鱼雷阶段有关。相对基因表达值(log2),以鱼雷合子胚胎(T-ZE)为参照,比较了晚期鱼雷胚(LT-SE)和成熟体胚(M-SE)。的微分表达式p< 0.01和≥2倍变化过滤器,我们鉴定出10637个差异表达基因。表示成熟期体胚和鱼雷合子胚归一化表达值的倍差(fold difference M-SE/T-ZE)。基因ID号和预测功能(描述)是基于criollo cacao基因组浏览器V1.0注释[1].(xlsx996 kb)
12870 _2014_1596_moesm6_esm.xlsx
附加文件6:体胚成熟过程中差异表达的基因与合子胚胎发生的成熟阶段有关。相对基因表达值(log2以成熟的合子胚胎(M-ZE)为参照,比较晚期鱼雷体胚(LT-SE)和成熟体胚(M-SE)的上调或下调基因p< 0.01和≥2倍变化)。表示成熟期体胚与成熟合子胚归一化表达值的倍差(fold difference M-SE/M-ZE)。基因ID号和预测功能(描述)是基于criollo cacao基因组浏览器V1.0注释[1].(xlsx1mb)
12870 _2014_1596_moesm7_esm.pdf
附加文件7:合子胚胎成熟过程中的基因表达模式。利用K-means聚类算法将合子胚胎成熟4个发育阶段的差异表达基因分为鱼雷(T)、早满(EF)、晚满(LF)、成熟(M) 10个聚类。确定的10个ZE聚类包括:上调基因(聚类III、IV和VI)、下调基因(聚类I、II和VII)、上调下调基因(聚类V和VIII)和下调表达基因(聚类IX和X)2)相对于T-ZE期为y轴,发育阶段为x轴。红线表示每个聚类中表达式值的平均值。给出了生物过程(P)的饼图GO分类。本分析中包括的基因是那些相关的2级生物过程功能GO术语注释:ZE组中有5700个基因(10288个中的55.4%),SE组中有5754个基因(10210个中的56.3%)。饼图中省略了未分类的基因;分类基因的数量显示在图表下面的括号中。给出了每个聚类中富集GO项的百分比。(pdf 2mb)
12870 _2014_1596_moesm8_esm.pdf
附加文件8:体胚成熟过程中的基因表达模式与合子胚胎发生的鱼雷阶段有关。以T-ZE为参照,采用K-means聚类算法将SE 2个发育阶段的差异表达基因分为5个聚类。将表达模式分为三大类:SE高表达基因(聚类I和III)、T-SE下调表达基因(聚类IV)和T-SE上调表达基因(聚类II和V)2)表示y轴,胚胎发育阶段表示x轴。红线描述了每个聚类中的平均表达式值。生物过程的饼图分类(P)在聚类下表示。本分析中包括的基因是那些相关的2级生物过程功能GO术语注释:ZE组中有5700个基因(10288个中的55.4%),SE组中有5754个基因(10210个中的56.3%)。饼图中未分类的基因被省略,分类基因的数量显示在图下的括号中。在每个整个子簇中富集GO项的百分比表示。(pdf 1mb)
12870 _2014_1596_moesm9_esm.xls
附加文件9:转录因子基因在可可合子胚成熟过程中受到显著调控。相对基因表达值(log2转录因子基因(TF)基因在合子胚胎(ZE)成熟阶段与合子胚胎(T-ZE)鱼雷期相比上调或下调。的微分表达式截止点p< 0.01和≥2倍变化滤波器。将成熟合子胚与鱼雷合子胚归一化表达值的倍差(fold difference M-ZE/T-ZE)表示出来。基因ID号和预测功能(描述)是基于criollo cacao基因组浏览器V1.0注释[1].(xl158kb)
12870 _2014_1596_moesm10_esm.xls
附加文件10:转录因子基因在可可体胚成熟过程中显著调控,参照成熟合子胚。相对基因表达值(log2late鱼雷- se (LT-SE)和maturity - se (M-SE)中TF基因的上调或下调参照了maturity - ze (M-ZE)。的微分表达式截止点p< 0.01和≥2倍变化滤波器。用归一化表达值在成熟期合子胚胎和鱼雷合子胚胎之间的倍差(fold difference M-SE/M-ZE)表示。基因ID号和预测功能(描述)是基于criollo cacao基因组浏览器V1.0注释[1].(xl142kb)
12870 _2014_1596_moesm11_esm.jpeg
附加文件11:比较成熟体细胞和成熟合子胚胎阶段差异表达转录因子基因的功能概述。使用MapMan软件生成的图表Theobroma可可28k基因本体映射文件。彩色条表示体细胞胚胎与合子胚胎中基因表达的比例(蓝色=合子胚胎中高表达的基因,红色=体细胞胚胎中高表达的基因)。使用路径工具(未显示)将基因分组到以下箱子中:RNA调控(290个基因),发育(32个基因),蛋白质合成/降解(34个基因),激素代谢(18个基因),DNA合成(6个),信号传导(3个),细胞组织(9个基因),杂项(3个基因),卡尔文周期(1个基因),非生物应激热(1个基因),未分配(31个基因)。这项研究特别感兴趣的两个类别是激素调节和细胞发育,分别占35和32个基因。(jpeg76 kb)
12870 _2014_1596_moesm12_esm.xls
附加文件12:可可体胚成熟过程中脂质生物合成基因的差异调控参考成熟合子胚。相对基因表达值(log2鱼体胚(LT-SE)和成熟体胚(M-SE)参照成熟合子胚(M-ZE)中脂质生物合成基因的上调或下调。的微分表达式截止点p< 0.01和≥2倍变化滤波器。用归一化表达值在成熟期合子胚胎和鱼雷合子胚胎之间的倍差(fold difference M-SE/M-ZE)表示。基因ID号和预测功能(描述)是基于criollo cacao基因组浏览器V1.0注释[1].(xl880kb)
12870 _2014_1596_moesm13_esm.xls
附加文件13:可可合子胚成熟过程中黄酮类生物合成基因的差异调控参考了鱼雷发育阶段。相对基因表达值(log2)在晚期鱼雷体胚(LT-SE)和参照鱼雷合子胚(T-ZE)的成熟体胚(M-SE)中类黄酮生物合成基因的上调或下调。的微分表达式截止点p< 0.01和≥2倍变化滤波器。用归一化表达值在成熟期合子胚胎和鱼雷合子胚胎之间的倍差(fold difference M-SE/M-ZE)表示。基因ID号和预测功能(描述)是基于criollo cacao基因组浏览器V1.0注释[1].(xls44kb)
12870 _2014_1596_moesm14_esm.xls
附加文件14:可可体胚成熟过程中黄酮类生物合成基因的差异调控参考成熟合子胚。相对基因表达值(log2在鱼雷晚期体胚(LT-SE)和成熟体胚(M-SE)参照成熟合子胚(M-ZE)中类黄酮生物合成基因的上调或下调。的微分表达式截止点p< 0.01和≥2倍变化滤波器。用归一化表达值在成熟期合子胚胎和鱼雷合子胚胎之间的倍差(fold difference M-SE/M-ZE)表示。基因ID号和预测功能(描述)是基于criollo cacao基因组浏览器V1.0注释[1].(xls 37 kb)
12870 _2014_1596_moesm15_esm.jpeg
附加文件15:不同次生代谢产物生物合成途径中编码酶的差异表达基因的功能概述。MapMan图是使用Theobroma可可28k基因本体图谱文件突出了黄酮类化合物、苯丙类化合物和木质素三个类群的富集。蓝色表示成熟体细胞胚胎(M-SE)中表达量高于成熟合子胚胎(M-ZE)的基因,红色表示表达量下降。(jpeg39kb)
12870 _2014_1596_moesm16_esm.xls
附加文件16:种子贮藏蛋白基因在可可合子胚和体胚成熟过程中显著调控。A)以鱼雷合子胚胎(T-ZE)为参考,在合子胚胎成熟过程中显著调控种子储存蛋白基因。B)以成熟合子胚胎(M-ZE)为参照,体胚成熟过程中种子贮藏蛋白基因显著调控。相对基因表达值(log2),截点设为p< 0.01和≥2倍变化。指出了成熟期合子胚与鱼雷合子胚归一化表达值的倍数差异。基因ID号和预测功能(描述)是基于criollo cacao基因组浏览器V1.0注释[1].(xl26kb)
12870 _2014_1596_moesm17_esm.pdf
附加文件17:用qRT-PCR验证10个差异表达基因的微阵列结果。通过qRT-PCR(红条)和芯片分析(蓝条)得到的10个基因的表达值(M-ZE/LT-SE)之比。(pdf 575 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。
本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。
如欲查看本牌照的副本,请浏览https://creativecommons.org/licenses/by/4.0/.
创作共用公共领域奉献弃权书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
马克西莫娃,s.n.,弗洛雷兹,s.s,沈,X。et al。全基因组分析揭示了不同的基因表达模式在合子胚胎和体细胞胚胎成熟Theobroma可可L,巧克力树。BMC植物生物学14日,185(2014)。https://doi.org/10.1186/1471-2229-14-185
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-14-185
关键字
- Theobroma可可
- 体细胞胚胎发生
- 合子的胚胎发生
- 胚胎发生
- 微阵列
- 基因表达