- 研究文章
- 开放获取
- 发表:
根寄生植物Orobanche aegyptiaca和寄生植物五角南极光获得Brassicaceae-specificstrictosidine synthase-like基因通过水平基因转移
BMC植物生物学体积14,文章号:19(2014)
摘要
背景
除了基因复制新创水平基因转移(HGT)是获得新基因的另一种重要途径。HGT可能赋予受体新的表型性状,这对物种进化和适应新的生态位很重要。由于它们长期的物理接触,寄生系统预计允许HGT以相对较高的频率发生。在植物中,寄生虫细胞器与寄主之间的HGT事件已被报道,但寄主和寄主核基因组之间的HGT事件很少被发现。
结果
一项彻底的转录组筛选显示Astrictosidine synthase-like(SSL)基因在根寄生植物Orobanche aegyptiaca而茎寄生植物五角南极光它们与芸苔科的基因序列相似度远高于与近缘种的基因相似度,表明从芸苔科到这些寄生虫的基因水平转移是独立的。这些发现得到了系统发育分析及其相同的独特氨基酸残基和缺失的有力支持。有趣的是,核定位SSL芸苔科属一个新成员SSL基因家族,源于基因复制。内含子的存在表明,在两种寄生虫中,这种转移是通过DNA整合直接发生的。此外,在国外检测到阳性选择SSL基因o . aegyptiaca但不是在c .南极光.外国人的表达SSL这两种寄生植物的基因在多个发育阶段和组织中均有检测到,且外源植物也有SSL基因经损伤处理后诱导c .南极光茎。这些数据表明,外源基因在受体物种中仍可能保留某些功能。
结论
我们的研究有力地支持了寄生植物可以通过HGT从远亲寄主中获得新的核基因,并且外源基因可能在新的寄主中发挥一定的功能。
背景
新的基因为生物提供了新的特性,从而有助于物种适应新的生态位[1,2].生物获得新基因的机制已被深入研究,迅速积累的基因组数据进一步支持了大多数基因起源于复制事件的观点[3.,4].除了基因复制,反转,外显子洗牌,反式剪接,还有新创基因进化中,水平基因转移(HGT)是通过直接向远亲受体物种引入新基因来获得新基因的另一个关键来源[1].
在原核生物中已发现大量HGT事件[2]及单细胞真核生物[5- - - - - -7].虽然在多细胞动物中,生殖系细胞从躯体分离预计会导致低HGT频率,但在包括蛭形轮虫在内的多种动物中发现了原核、真菌或植物起源的基因[8,9],被囊[10]、海蜇[11],小海葵[12]、线虫[13]、蚜虫[14,15]和其他昆虫[16,17].HGT的进化意义已经在一些动物身上得到了很好的说明。例如,从真菌中获取两个类胡萝卜素生物合成基因使豌豆蚜虫具有红色的身体颜色,在此基础上,捕食者和寄生虫进一步驱动了绿色蚜虫和红色蚜虫之间的变化[18].
HGT还参与植物的适应和基因组进化[19,20.].在非维管植物中,Yue等人在苔藓核基因组中鉴定出57个原核、真菌和病毒起源的基因家族[21].这些基因中的大多数很可能转移到绿色植物的祖先,并可能在植物从水生向陆生过渡的过程中发挥了重要作用[21].高等植物细胞器基因组之间广泛发生HGT [19,22- - - - - -24].然而,植物核基因组间的基因转移却鲜有报道[19,22].但也有例外,高等植物中的一种Mu-like element (MULE)被发现在水稻和水稻之间转移Setaria核基因组[25],同样,在Panicoideae植物中,C4光合作用途径中的4个独立基因从C4植物反复转移到C3植物,这种HGT事件可能解释了C4植物在不同物种中的起源[26].这些HGT都发生在核基因组之间,目前尚不清楚这些HGT事件是如何发生的,因为供体和受体之间没有直接的物理相互作用。
理论上,供体和受体之间的身体接触有助于HGT的发生,因为它增加了遗传物质转移的机会[7,27].寄生虫与寄主可形成长期亲密的身体接触,因此寄生可导致相对较高的HGT频率[5,7].与此相一致的是,迄今为止发现的高等植物HGT事件大多来自植物寄生系统。在植物中,据报道有数百种寄主细胞器基因从寄主转移到寄主[23,28,29],或反之[30.- - - - - -32],其中大多数靶向受体线粒体。相比之下,寄生植物与寄主核基因组间HGT的报道较少[19,22].在涉及核基因组的寄生系统中,第一个被证实的HGT是一个功能未知的基因从单子叶宿主转移(可能高粱)到寄生植物Striga hermonthica[33],通过分析表达的序列标签美国hermonthica.随着新一代测序技术的快速发展,越来越多的转录组数据被获取。大花草属cantleyi通过对该寄生生物及其专性寄主的转录组数据的筛选,发现该寄生生物是一种专性全寄生植物Tetrastigma rafflesiae,34].同样,Zhang等人发现,编码种子储存和昆虫毒素蛋白的白蛋白基因从豆科植物转移到根寄生植物Orobanche aegyptiaca和寄生植物五角pentagona可能在HGT后仍保持相同的功能[35].
属植物Orobanche是根寄生植物,是全寄生植物中最大的一类。大多数Orobanche物种寄主范围较窄,而o . aegyptiaca是一个例外,因为它寄生在许多植物中,包括芸苔科、豆科、茄科、蜂科、菊科和葫芦科[35,36].o . aegyptiaca主要分布在地中海地区和西亚,在非洲、澳大利亚、美洲和东亚也有零星分布[37].五角(旋花科)植物是寄生植物,是专性寄生植物的另一类群,该属大多数成员寄主范围广泛,与所感染的植物科有很大的重叠o . aegyptiaca[38],并且在地理上分布广泛,与o . aegyptiaca[37].
物种树和基因树之间的不一致是HGT发生的重要标志。基因组数据Mimulus guttatus而且茄属植物(美国tuberosum而且美国lycopersicum的序数亲戚Orobanche而且五角分别已发布[39].m . guttatus(Phrymaceae)和o . aegyptiaca属于拉米亚莱斯骑士团茄属植物属(茄科)和c .南极光(旋花科)属龙葵目。可用的基因组信息m . guttatus而且茄属植物为鉴定HGT提供了极好的机会Orobanche而且五角.在这里,我们报道strictosidine合酶-就像(SSL)来自芸苔科的基因被两种寄生植物所吸收,o . aegyptiaca而且c .南极光.此外,这些SSL基因属于一个新的成员SSL基因家族,源于十字花科特有的基因复制。内含子在异体中的存在ssl寄生植物的DNA,而不是信使rna,被直接整合o . aegyptiaca而且c .南极光分别。此外,表达水平SSL基因在o . aegyptiaca而且c .南极光在不同的发育阶段和器官和SSL基因c .南极光伤后诱发。这些结果表明,在寄生过程中,核基因可以从寄主转移到寄生植物中,外源基因可能为新寄主提供新的性状,这可能有利于适应。
结果
外国人的识别SSL基因在Orobanche aegyptiaca而且五角南极光
o . aegyptiaca是一种寄生于许多植物物种的根全寄生植物。最近,一大批转录组数据被公布(寄生植物基因组计划;[40])。利用这些已发表的数据库,我们下载了组装的转录组o . aegyptiaca并筛选外源基因(见附加文件)1对于程序)。最初获得的2100多个序列被AlienG预测为HGT候选序列[41].其中,一段691 bp的转录片段在核苷酸水平和氨基酸水平上分别具有88%和89%的同源性strictosidine synthase-like(SSL)基因拟南芥(以下称拟南芥)(AT2G41300),而与中同源体高度分化Orobanche相对Mimulus guttatus(氨基酸水平49%的同源性,核苷酸水平无显著相似性)。获得完整的SSL转录序列,我们重新组装的RNA-seq数据集o . aegyptiaca使用Trinity [42],获得了全长1597 bp、编码369个氨基酸的完整开放阅读框(ORF) cDNA序列OaSSL, NCBI登录号:KF817594)。
考虑到植物的茎部寄生c .南极光也寄生于芸苔科植物,我们调查了是否c .南极光也获得了SSL基因。一个自制的转录组组装c .南极光使用OaSSL随着查询,发现了2个高度相似的转录本,它们很可能是通过替代剪接从一个基因中衍生出来的(见下文)。这C. australis SSL(以下CaSSL)也显示出与同一拟南芥的高度相似性SSL在核苷酸和氨基酸水平上(分别为84%和88%),但与Solanales同源物的相似性要低得多美国tuberosum而且美国lycopersicum(氨基酸水平49%和50%,核苷酸水平无显著相似性)。这些数据有力地表明,这两种寄生植物获得了一种SSL来自拟南芥或某些其他芸苔属植物的基因。
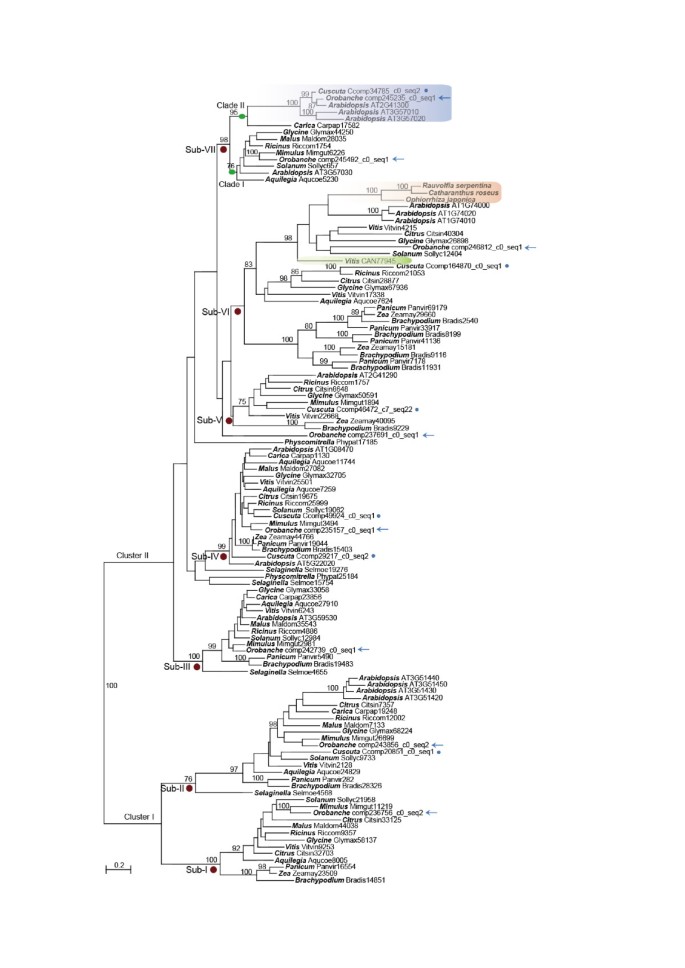
来深入了解进化过程SSL基因,排除了这种可能性OaSSL而且CaSSL都起源于非芸苔科植物,SSL利用15种代表性植物基因组和2种寄生植物的序列构建基因树(图1).系统发育分析SSL拟南芥中的成员和来自其他物种的代表性同源序列(在系统发生树中聚集为姐妹分支的序列被删除)揭示了SSL基因家族包含两个高度分散的簇,簇I和簇II,以及多个亚家族。类群I包括2个亚科,每个亚科都有来自陆生植物主要分支的同源物,包括真花科和单花科;聚类II包含5个亚科,其中亚科VII (subvii)仅由小叶子科同源组成,其余4个亚科包括小叶子科和单叶子科同源。转录组分析显示,两种寄生植物均含有多种SSL复印,7份o . aegyptiaca属于6个亚科和5个拷贝c .南极光来自5个亚科。
植物中伸缩糖苷合成酶样(SSL)蛋白家族的分子系统发育。为了减小树的大小,除了的所有SSLs之外,每个植物中的多个高度相似副本中只使用了一个序列拟南芥被使用。属名和序列id显示在节点中。分支上方的数字表示引导支持值大于75%时的最大可能性。分支下面的红色圆圈表示亚族I至VII (Sub-I至Sub-VII)。绿色圆圈表示支系I和(芸苔类特定基因)的分支。寄生植物的序列o . aegyptiaca而且c .南极光分别由箭头和圆点表示。感兴趣的SSL基因在o . aegyptiaca而且c .南极光和芸苔科供体用蓝色表示。三个序列来自Rauvolfia serpenti,Catharanthus roseus也叫,Ophiorrhiza粳稻与生化证据,活性的strictosidine合酶表示在橙色。SSL在葡萄,没有检测到strictosidine合成酶活性,用绿色表示。
亚vii包含两个枝:枝一和枝二。虽然枝I包含了来自不同种类的长尾科植物的同源物,但枝II只包含了SSL芸苔属植物和2种寄生植物的序列(图1),以及OaSSL而且CaSSL与3个拟南芥成簇SSL基因100%自举支持。因此,分枝II可能是在芸苔属植物分化之前的分枝I的基因复制而起源于芸苔属SSL进化枝I中的基因因此被称为SSLClade II中的基因)和OaSSL而且CaSSLHGT很可能来自芸苔科。
为了更深入地了解潜在的基因捐赠者SSL基因在Orobanche而且五角,我们收集了所有SSL从具有可用基因组序列的物种和3种寄生植物的转录组组合中获得的序列,五角pentagona(旋花科植物),Cassytha filiformis(樟科),Pilostyles thurberi(蒲公英科)(http://www.onekp.com/project.html),以及2种根寄生植物,Triphysaria杂色的而且Striga hermonthica寄生植物基因组计划[40].系统发育分析表明,只有某些十字花科SSL基因紧密聚集在一起OaSSL而且CaSSL.所有这些序列都被用来推断外来ssl的具体来源。该树显示了3组,组I到III,和SSL来自两种寄生植物的基因被分配到II组,并获得100%的自举支持(图2).重要的是,这两个OaSSL而且CaSSL它们共享一个氨基酸缺失和多个唯一的氨基酸,这些氨基酸在II组SSL蛋白中是唯一保守的(图2).这些结果表明OaSSL而且CaSSL起源于芸苔科,虽然我们不能确定确切的供体种,因为OaSSL而且CaSSL显示出与第二组其他物种的模糊关系(图2).
芸苔科特有的结构SSL基因,OaSSL在O。aegyptiaca,而且CaSSL在c .南极光
23的比较SSL5种芸苔科植物的mRNA序列与基因组DNA序列的比较表明,芸苔科植物的基因组DNA序列与芸苔科植物的基因组DNA序列一致SSL基因最多含有3个内含子,其中18个基因缺少第一个内含子。内含子2和内含子3的位置很保守,但内含子长度在种内和种间存在差异(附加文件)2).
只有一份OaSSL转录本在转录组中被识别o . aegyptiaca.我们以基因组DNA为模板,克隆了其基因组序列(NCBI登录号:KF817597):包含2个内含子,其位置与该基因的内含子2和3相同SSL芸苔科基因(图3.).分析c .南极光转录组组装显示2种亚型CaSSL分别命名为Seq1 (NCBI登录号:KF817596)和Seq2 (NCBI登录号:KF817595)。Seq1与Seq2共享100%的身份,除了Seq1有一个21 bp的插入(附加文件3.).在芸苔科特异性SSL基因中,Seq1的插入位置与内含子2相同,因此Seq1很可能源于内含子2的部分保留。假定的Seq1和Seq2蛋白序列与来自芸苔科的SSLs序列一致,没有任何帧移位,表明Seq1和Seq2编码功能蛋白(附加文件)3.).利用基因组PCR扩增基因序列CaSSL基因,但跨越内含子区域的各种引物未能产生任何产物。我们推测内含子CaSSL可能有复杂的二级结构,也可能很长。内含子3明显缺失CaSSL(NCBI接入号:KF817598,图3.).
通过HGT, DNA可以转移到植物的核基因组、质体基因组或线粒体基因组中。目前已知的芸苔科特有SSL基因由核基因组编码。细胞核或线粒体基因组Orobanche或五角属,但BLAST搜索的多个叶绿体基因组五角物种(43](叶绿体基因组c .南极光)表明没有一个质体基因组含有同源的CaSSL基因。我们推测CaSSL基因可能存在于核基因组中c .南极光,但不能完全排除存在于叶绿体或线粒体基因组中的可能性。这对OaSSL在o . aegyptiaca,目前质体基因组从Orobanche不拥有SSL基因(44].
表达分析AtSSL1在A. thaliana, OaSSL在o . aegyptiaca而且CaSSL在c .南极光
因为还没有关于芸苔科特异功能的研究报道SSL基因,我们选择了AtSSL1拟南芥基因(AT2G41300, in Clade II,图1),两者的相似度最高OaSSL在o . aegyptiaca而且CaSSL在c .南极光,并研究其在不同发育阶段的表达谱,以及在不同应激刺激和处理后使用genevestikator [45].AtSSL1在所有发育阶段的表达水平相当相似(附加文件4).强烈上调AtSSL1在添加1-萘乙酸的花序茎和接种线虫的根中观察到表达异皮线虫属schachtii;在低氧处理后发现根中有较大的下调(附加文件)5).因此,我们推测AtSSL1可能参与植物发育和胁迫响应。
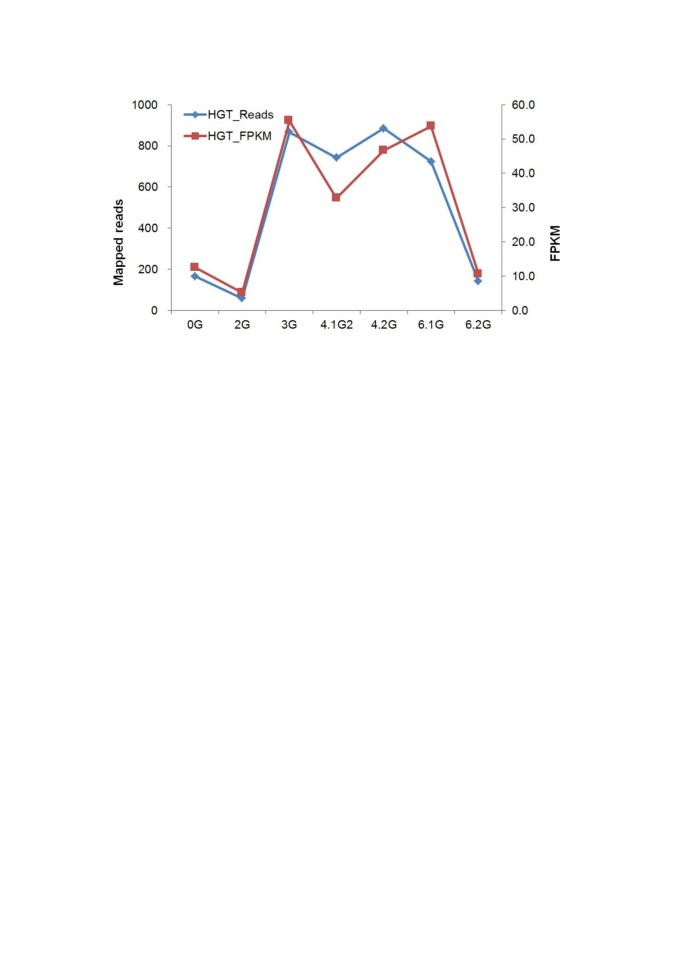
的潜在功能OaSSL基因o . aegyptiaca的表达谱OaSSL使用PPGP中来自不同发育阶段的配对端RNA-seq数据集o . aegyptiaca[40)(图4).OaSSL显示在吸器形成和蔬菜生长阶段转录物丰度增加了3- 5倍(图4),暗示OaSSL基因可能在吸器的形成和蔬菜的生长中起作用。
表达水平OaSSL不同发育阶段的基因。在左y轴上显示了映射到cDNA上的reads的数量。标准化表达水平由Illumina RNA-seq数据集中来自不同发育阶段(PPGP)的每千克碱基外显子每百万片段映射的片段数(FPKM,一个片段指来自配对端数据的一对reads)估计。0G,种子吸收,提前萌发;2G,幼苗暴露吸器诱导因子后;3G,吸器附着于寄主根;早期渗透阶段,血管前连接;4.1G,早期形成的寄生虫;寄生虫血管连接后的营养生长;4.2G,蜘蛛期;5.1G,土芽预出; 5.2G, pre-emergence from soil - roots; 6.1G, post emergence from soil - vegetative structures, leaves/stems; 6.2G, post emergence from soil - reproductive structures, floral buds.
鉴于目前还没有公开的转录组数据库五角采用定量实时荧光定量pcr (qPCR)技术检测各基因的表达水平CaSSL在c .南极光在不同的发育阶段和伤口治疗后。由于选择合适的内参基因对qPCR检测很重要,四种常用的内参基因肌动蛋白,EF-1α,EF-TU,PP2A,用geNorm评估[46, DeltaCT [47]、BestKeeper [48], Normfinder [49],和RefFinder [50].结果表明EF-1α在所有条件下都稳定表达(附加文件6).因此,EF-1α作为内参基因。CaSSL在所有9个不同发育阶段或器官中均有表达(图5A),成熟茎中表达量最高,茎尖中表达量最低(差异为19.7倍)(图5A).此外,转录水平CaSSL分别测定伤前、伤后1、3、9、24 h。的表达级别CaSSL基因在治疗1 h后上调1.3倍,并随时间增加(治疗24 h后上调3.2倍)(图5B).这些结果暗示CaSSL可能与发育和非生物应激有关,如损伤。
功能分化与选择分析
与原始基因拷贝相比(Clade I,图1)、芸苔科特有基因(Clade II,图1)在系统发育树中呈现相对较长的分支,这表明两个亚家族之间的功能分歧可能是在基因复制事件发生后发生的。为了评估这种情况,使用DIVERGE 2进行后验分析,估计这两个亚家族之间的两种类型的功能分歧(I型和II型)[51].I型功能差异(功能约束改变)表示一个分支中氨基酸位点保守,而另一个分支中高度可变;II型功能分化检测到两支进化枝中保守的氨基酸位点,但生化性质发生变化[52].I型功能散度系数显著大于0 (θ1=0.21040±0.0343,LRT = 37.5692, p < 0.01), 7个氨基酸位点的后验概率大于0.7,表明这7个位点的功能约束在两个进化支之间发生了转移。基因复制后,两个支间未发生ii型相关的根本变化。这些结果表明,I型功能差异是分枝I和分枝II的显性模式,改变的位点特异性选择约束可能导致了它们之间的群体特异性功能变化。
我们进一步研究了强选择是否参与了SSL进化枝I和II的基因(图1而且2)使用Yang等人开发的似然比测试。[53].我们首先利用站点模型检测了芸苔科特有的某些站点是否发生了正向选择SSL基因,它们的原始副本,还有外来的SSL两种寄生植物的基因。在这些基因中未检测到阳性选择(附加文件7).值得注意的是,M0模型下的最大似然估计值在大数据集(芸苔科特异性基因及其原始拷贝)接近0,在小数据集(芸苔科特异性基因拷贝和两个外源基因拷贝)接近0.1903SSL寄生植物中的基因)。模型M0和M3之间的对数似然差对于两个大数据集(LRT = 630.293,p< 0.01)小数据集(LRT = 387.605,p< 0.01)(附加文件7).这表明,放松的纯化选择是这些基因进化的主要驱动力。
在分支点模型下,通过设置芸苔科植物特异性来检验零假设和备择假设SSL各基因和两个寄生基因分别作为前景。没有证据表明正选择作用于芸苔科SSL基因和CaSSL基因c .南极光(附加文件8).然而,两个密码子的站点OaSSL基因的o . aegyptiaca可能经历了正向选择(LRT = 5.754,p< 0.05),后验概率大于75%(附加文件8).密码子181编码了芸苔科特有的甘氨酸(G)或精氨酸(R)SSL基因,而它编码一个天冬酰胺(N)在OaSSL基因的o . aegyptiaca;位点267是一个丝氨酸(S)OaSSL该基因在芸苔科特定基因中编码了天冬酰胺(N)或谷氨酸(E),显示出0.975的正选择后视概率(附加文件)9).
讨论
的Brassicaceae-specificSSL基因和外星起源SSL寄生植物的基因o . aegyptiaca而且c .南极光
的SSL植物的基因构成了一个庞大的基因家族,不同亚家族之间的差异很大。其中亚vii相对特殊,因为它包含一个枝(枝I)包含SSL而另一个分枝(分枝II)仅存在于芸苔科和本研究的两种寄生植物中。很有可能SSL分枝II的基因仅在芸苔属中通过一个祖先的基因复制形成SSL第I枝基因。
有趣的是,在这项研究中,我们发现两个SSL采用水平基因转移的方法,将十字花科植物的基因引入到两种寄生植物中。以下证据有力地支持了这一点:1)多序列比对中的高同一性值,2)共享的indels和氨基酸,3)系统发育分析中的稳健的bootstrap值。从这些证据来看,趋同进化的可能性可以被排除。因为o . aegyptiaca而且c .南极光属于不同类群的两种远亲寄生植物中导致两个外源基因的基因转移事件应该是独立发生的。
可能的转移机制
内含子的存在OaSSL而且CaSSL基因序列表明,这两种基因转移事件都不是发生在mRNA水平,而是发生在DNA水平。同样,有报道称外源白蛋白基因在o . aegyptiaca也含有内含子,因此应该以DNA的形式转移[35].已知寄生植物会从寄主那里运输数千种mrna [54,55];然而,从一个植物转移到另一个植物的DNA,而不是mRNA,很可能是植物中HGT的主要形式。这可能是因为mRNA在整合到植物基因组之前必须逆转录为DNA,而这种逆转录的活性非常低。
植物DNA水平的任何HGT事件的发生都需要多个步骤:1)穿过细胞壁和细胞膜后进入细胞质,2)通过核膜进入细胞核,3)整合到基因组中,4)固定并传递给下一代,5)在群体内传播。目前已提出的植物HGT机制包括非法授粉、媒介介导和植物-植物接触假说[5,19].非授粉假说认为,基因供体的花粉粒可以在生殖隔离种的柱头上萌发,授粉后,重组可能会使一些外源基因整合到另一组染色体中,同一物种的杂交和自然选择可能会导致群体内保留含有一些外源基因的目标染色体组。这种HGT机制被认为发生在密切相关的物种之间,如之间的HGT案例Setaria而且选用[25之间),《行动纲领》而且羊茅属[56],或在C4和C3物种之间Alloteropsis[26].因为两个o . aegyptiaca而且c .南极光与芸苔科有远亲关系,非传粉假说是非常不可能的。
载体介导转移假说假设病毒、病原体、内生植物或其他介质作为载体将外源DNA转移到受体中。由于大多数载体的寄主范围有限,从十字花科(或从豆科,在这种情况下白蛋白1[35])和旋花科似乎不太可能。只有宿主范围广的载体,如苹果潜伏球形病毒[57],可能能够调解这些转移事件。但是,目前还没有发现携带外源基因的植物病毒。这可能是由于病毒复制速度快,在扩增过程中外源基因丢失所致。需要病毒携带外源基因的证据来证实载体介导的HGT是一种合理的机制。
植物-植物接触假说认为外源DNA是通过植物之间的直接接触进入受体的,如寄生系统中长期的亲密接触。寄生在寄主和寄生植物之间建立了一种功能性的共生途径,允许营养物质、rna的运输[54,58]、蛋白质[59- - - - - -61]、病毒[62]和植原体[63]从宿主到寄生虫。目前,没有直接证据表明寄主DNA分子可以被转运到寄生植物中,但在同一物种之间的移植物区域观察到大的DNA片段甚至整个叶绿体的移动[64]或两性不相容的物种之间[65].寄生植物与寄主之间的连接与人工嫁接的连接高度相似;因此,无论DNA的转移是局限于细胞间的短范围内,还是可以在相对较长的距离内发生,宿主和寄生虫血管系统的融合为外源DNA传播到受体提供了可能性。因为输入大分子的浓度也沿着茎降低[55,61],外来遗传物质整合的概率随着与吸器距离的增加而降低。我们假设芸苔科SSL基因首先被整合到o . aegyptiaca而且c .南极光吸器或附近的组织,以及某些细胞以某种方式发展成花分生组织,使宿主基因传递给下一代。
外文的功能意涵SSL基因在o . aegyptiaca而且c .南极光
a功能的最初生化证据SSL吉恩来自Rauvolfia Serpentina,检测一种催化色胺与色胶素立体缩合形成strictosidine (Pictet-Spengler反应)的strictosidine合酶的活性[66],后来又克隆出编码基因[67].由于strictosidine是吲哚类生物碱合成的关键中间体,是几种临床上有用的抗癌生物碱的前体,STR基因先后在Catharanthus roseus也叫[68),萝芙木属verticillata[69),而Ophiorrhiza粳稻[70].然而,与STRs的催化特性和三维结构中的活性位点信息相比[71,72], Hickes等人认为绝大多数标注为STR的序列并不催化Pictet-Spengler反应[73].
在我们的综合系统发育分析中,我们添加了所有编码具有证实STR活性的蛋白质的序列,它们聚为一个高度支持的分支(图1).其中一个序列葡萄(CAN77945)与我们系统发育树中已验证STR活性的基因相关。无STR活性,但有水解活性SSL基因的诉酿酒用葡萄[73].鉴于演化支II的基因与真正的STRs,芸苔科特有和外来STRs的序列相似性低,关系遥远SSL寄生植物中的基因可能不催化Pictet-Spengler反应。我们发现SSL拟南芥和寄生植物的基因在多个发育阶段和不同处理后都有积极转录,但其生物学功能有待进一步研究。
寄生植物至少面临两个挑战。首先,它们需要克服宿主的防御,同时从宿主那里吸收营养;其次,它们必须抵抗食草动物、细菌、真菌或非生物环境的压力,如伤害。以前的研究表明,从微生物转移到线虫体内的基因可能使寄生虫能够调节宿主的防御系统[74].寄生卵菌可能从真菌中获得某些分泌蛋白,真菌与卵菌生活在同一生态位,因此能够抑制某些宿主防御反应[75].在公共微阵列数据库中进行数据挖掘,发现与拟南芥最相关AtSSL1也对激素,非生物和生物压力的治疗有反应。OaSSL在o . aegyptiaca在不同发育阶段表现出不同的表达水平,并经历了正向选择。同样,在c .南极光,CaSSL还表现出发育阶段和器官特异性表达水平;此外,损伤在转录上被激活CaSSL.因此,国外SSL两种寄生植物的基因可能参与植物发育和次生代谢,而次生代谢通常与草食性和病原抗性有关[76,77]或其他环境压力[78].
结论
迄今为止,从寄主核基因组到寄生植物的HGT很少报道,在这里我们提供了确凿的证据,表明两个核编码SSL来自芸苔科物种的基因被两种远亲的全寄生植物(根寄生植物)增选o . aegyptiaca和寄生植物c .南极光.因此,与寄主植物的物理联系以及寄主营养物质和大分子的运输等寄生虫生活方式可能为寄生虫提供了通过HGT获取寄主基因的机会,并且寄主和寄生植物核基因组之间的HGT可能并不罕见。我们的分析也提示了水平转移基因在进化和适应寄生生活方式或环境中的潜在作用。
方法
数据源
所有的12个Illumina数据集o . aegyptiaca原始转录组组装OrAeBC4从根寄生植物基因组计划(PPGP)网站(http://ppgp.huck.psu.edu/).来自PPGP的其他寄生植物的转录组组合,Striga hermonthica(StHeBC2),Triphysaria杂色的(TrPuRnBC1),Triphysaria pusilla(TrVeBC2),也下载了。AlienG所需的蛋白质数据库41]用于BLAST搜索的数据包括NCBI非冗余(nr)数据库(2012年9月)和Phytozome(9.0版本)上提供的22种植物基因组的所有预测蛋白[39但是在nr数据库中不存在。核苷酸数据库包括NCBI核苷酸集合(nt)和来自Phytozome的22个植物基因组的所有预测转录本。1 KP项目中的数据库(http://www.onekp.com/project.html;包括转录组集合五角pentagona,Cassytha filiformis,Pilostyles thurberi), PlantGDB (http://www.plantgdb.org),以及SOL基因组网络(http://solgenomics.net)。
水平转移基因的转录组筛选o . aegyptiaca
由于寄生植物从寄主那里运输了数千种mrna,因此用于原始转录组组装的数据集包括附着在寄主上的样本,因此组装包含来自寄主mrna的污染。为了去除被污染的序列,从两个未附着在宿主上的样品,种子萌发(OrAe0G)和发芽种子,胚根出现和吸器前生长(OrAe1G)中读取的reads使用RSEM映射到唯一的序列[79],并保留带有映射读的组装序列。对于长度不小于300 nt的组装序列,利用OrfPredictor对潜在的orf进行局部预测[80],长度不小于100的氨基酸序列使用AlienG [41].如果第一次非Lamiales命中与第一次Lamiales命中的得分之比大于1.2,则可以预测基因的外星起源。得到的候选人进一步筛选,以排除那些具有m . guttatus由于发现一些非编码基因被严格翻译成蛋白质,因此BLAST搜索发现它们在相应的cDNA序列中存在关联。
总RNA提取,cDNA文库构建及转录组测序c .南极光
的种子c .南极光用硫酸处理20分钟,使种皮松动,然后用水大量冲洗除去硫酸。种子在25°C的湿滤纸上保存3-4天,直到幼苗长约4厘米。幼苗被轻轻固定在幼大豆上(大豆)植株(~ 20-25厘米高;在26±2℃,光照12 h条件下培养),用棉线和水喷2 - 3次/天,防止菟丝子幼苗变干,直至寄生建立。提取总RNAc .南极光按照制造商的说明,使用RNAeasy Plant Mini Kit (Qiagen)。通过DNase处理(Qiagen)去除潜在的DNA污染。使用Truseq RNA and DNA Sample Prep Kit构建cDNA文库,但将连接接头后的插入片段设置为300 bp (cDNA插入长度约180 bp)。配对的短读数(2 × 101 bp)在Illumina Hiseq™2Clade I仪器上生成。采用Illumina GA 1.6管道进行图像反褶积和质量计算。
的识别ssl在两种寄生植物的转录组中
潜在的适配器标签及其后续序列从Illumina数据集中的所有原始读取中删除o . aegyptiaca而且c .南极光使用cutadapt [81].通过使用Btrim进一步过滤低质量值(低于25)的碱基来获得清洁读数[82].新创RNA-seq数据的组装使用Trinity软件包[42],片段对之间期望的最大长度设置为300。外国SSLcDNA片段的组装o . aegyptiaca在PPGP中对该物种的自制Trinity组合进行了搜索。得到的假定全长cDNA被用于对转录组组装进行搜索c .南极光以及其他寄生植物,包括Striga hermonthica,Triphysaria杂色的,Triphysaria pusilla,五角pentagona,Cassytha filiformis,Pilostyles thurberi.
表达水平估计OaSSL通过转录组分析,RNA-seq Reads映射到组装序列的可视化,以及微阵列表达分析拟南芥AtSSL1基因
外文的表达水平SSL基因o . aegyptiaca用RSEM估计不同组织和发育阶段的差异[79]通过将上面得到的所有干净的读数映射到我们的三位一体集会o . aegyptiaca.为了检查可能的装配错误,将所有这些数据集的所有干净读取映射到外部SSL用平板电脑显示cdna [83].基于web的表达分析A. thaliana AtSSL1基因(AT2G41300)在不同组织、不同发育阶段和不同处理下的差异https://www.genevestigator.com/gv/, (45].
克隆SSL寄生植物的基因
获得外来者的基因组序列SSL基因,从3mg干种子中提取基因组DNAo . aegyptiaca和0.1克茎c .南极光采用改良的十六烷基三甲基溴化铵(CTAB)方法[84].引物对oaeF2/R1370 (5 ' - ggaacgatagtgtgaagctt -3 ' /5 ' - catctctttcttgaaccctac -3 ')和cusF52/ R1470 (5 ' - aggaacgaagggagtatttt -3 ' /5 ' -TATTCATGAACTTCCGATATGG-3 ')根据研究结果设计SSLcDNA序列o . aegyptiaca而且c .南极光,分别。使用AxyPrep DNA Gel Extraction Kit (Axygen Bioscienes)提取预期大小的扩增产物,克隆到pMD18-T载体(Takara)中,并进行测序。
的qRT-PCRCaSSL基因c .南极光
新鲜组织采集于9个不同发育阶段:干燥种子、下胚轴2毫米和3厘米的萌发种子、吸器前(附着在伪寄主、竹棒上2 d后)、成熟茎(距离茎尖至少4厘米)、茎尖、花蕾、花和蒴果。在距茎尖4 cm处,用铁丝纱布将长2 cm的菟丝子茎用4排直径0.1 mm的细丝击伤。分别于处理后0、1、3、9、24 h采集样品。每个时间点取5个生物重复。按照制造商的说明,使用Fruit-mate™和RNAiso Plus (TaKaRa)从每个样品中提取总RNA。RNA浓度定量,每个RNA样本500ng用oligo_(dT)反转录。18RevertAid™H Minus逆转录酶(Fermentas),总体积为10 μL。将得到的cDNA样品稀释至25 μL。根据两种mRNA亚型的共享区域设计特异性引物RT-cus34-F2/RT-cus34-R1 (5 ' -CTGCGACGGTTACCTTGGAA-3 ' /5 ' -CCACCATGTCCACCACTTTCT-3 ')。用q-PCR法检测外源蛋白的表达SSL基因c .南极光根据制造商的说明,使用iTaq™Universal SYBR Green Supermix (BIO-RAD),在CFX Connect™实时系统(BIO-RAD)上切割处理过的茎干。从四个候选基因中选择了cDNA浓度变化正常化的内参基因,肌动蛋白,伸长因子1α(EF-1α),延伸系数Tu(EF-Tu),丝氨酸/苏氨酸蛋白磷酸酶2A(PP2A).使用相同数量的总RNA记录所有样品的Ct值。通过使用在线服务器refinder (http://www.leonxie.com/referencegene.php).外源表达水平的变化SSL采用比较CT法对基因进行检测。用Student 's法测定损伤处理诱导的SSL表达水平的差异t测试。
系统发育分析
利用BLAST搜索技术从Phytozome的代表性植物中提取蛋白质序列OaSSL基因o . aegyptiaca作为查询。因为OaSSL同源基因在每个物种中都有多个拷贝数,我们利用ClustalX2构建了自举NJ树[85]并且只保留了每个分支中每个物种的代表性序列。的同系物o . aegyptiaca而且c .南极光通过tblastn搜索得到的RNA-seq组装在o . aegyptiaca作为查询。
所有收集到的蛋白质序列都使用ClustalX2进行比对[86].我们目视检查了校准并进行了手工改进。缺口和不明确的位置被从对齐中移除。通过ModelGenerator (v_851)确定蛋白质替代矩阵和速率异质性的最优模型[87].采用PHYML 3.0最大似然法重建系统发育树[88].Bootstrap分析使用100个伪重复。使用NJplot对树的拓扑结构进行查看和编辑[89].
功能分化分析
利用DIVERGE 2.0估算了甘蓝类特异基因与原始拷贝之间的I型和II型功能分化系数[51].第一类功能分化(θ1)改变基因复制后部分位点的功能限制,而II型功能分化(θ11)会导致两个复制副本之间的氨基酸性质发生根本变化。采用1自由度(DF)分布进行似然比检验。
阳性选择检测
对Phytozome的所有转录本和寄生植物的转录组组合进行BLAST搜索,得到相应的cdna。选择压力作用于SSL通过计算程序的非同义替换与同义替换(或)的比率来分析编码区域codeml在PAML v4.7中实现[53].我们创建了大数据集和小数据集。大数据集包括芸苔类特有基因副本和原始基因副本;小数据集包括芸苔类特异性基因拷贝和外源基因拷贝SSL两种寄生植物的基因。联机服务器PAL2NAL [90]将蛋白质序列比对转化为相应的密码子比对,作为的输入文件codeml在PAML。使用ClustalX 2.1生成具有分支长度的无根NJ树[85基于蛋白质排列,并被喂食codeml在PAML。
我们分别用似然比检验(LRT)对大型和小型数据集上的站点模型和分支站点模型进行了分析。对于站点模型,采用M0-M3比较的LRT检验DF设置为3时站点间的变量。采用M1a-M2a和M7-M8比较的lrt进行阳性选择,DF设为2。对于分支位点模型,零假设与备择假设进行了比较,以检验正选择是否作用于甘蓝类特异性ssl而外国人ssl在两种寄生植物中,以其为前景进行分析。我们选择了贝叶斯实证贝叶斯(BEB)分析codeml计算感兴趣谱系中正选择位点的后验概率。
参考文献
- 1.
丁勇,周强,王伟:新基因的起源及其新功能的进化。环境科学学报,2012,43(3):344 - 344。10.1146 / annurev - ecolsys - 110411 - 160513。
- 2.
高加腾,杜立德,李志刚:基因转移对原核生物进化的影响。分子生物学杂志,2002,19(12):2226-2238。10.1093 / oxfordjournals.molbev.a004046。
- 3.
李文杰,李文杰,李文杰:植物基因复制与进化新颖性研究。植物保护学报,2009,36(3):557-564。10.1111 / j.1469-8137.2009.02923.x。
- 4.
Kondrashov FA:基因复制是基因组适应环境变化的机制。中国生物医学工程学报,2012,29(4):457 - 457。10.1098 / rspb.2012.1108。
- 5.
王志刚,王志刚:水平基因转移在真核生物进化中的应用。植物学报,2008,9(8):605-618。10.1038 / nrg2386。
- 6.
水平基因转移的进化意义。科学通报,2012,46:341-358。10.1146 / annurev -麝猫- 110711 - 155529。
- 7.
Wijayawardena BK, Minchella DJ, Dewoody JA:宿主,寄生虫和水平基因转移。寄生虫学杂志,2013,29(7):329-338。10.1016 / j.pt.2013.05.001。
- 8.
Boschetti C, Carr A, Crisp A, Eyres I, Wang-Koh Y, Lubzens E, Barraclough TG, Micklem G, Tunnacliffe A:蛭形轮虫外源基因表达的生化多样化。公共科学学报,2012,8 (11):e1003035-10.1371/journal.pgen.1003035。
- 9.
孙晓明,王晓明,王晓明。蛭形轮虫横向基因转移研究进展。科学通报,2008,32(5):528 - 528。10.1126 / science.1156407。
- 10.
倪涛,岳娟,孙刚,邹勇,文娟,黄娟:藻类向动物的基因转移机制及其进化意义。中国生物医学工程学报,2012,12(1):83-10.1186/1471-2148-12-83。
- 11.
Denker E, Bapteste, Le Guyader H, Manuel M, Rabet N:水平基因转移与刺胞细胞进化。动物学报,2008,18 (18):R858-R859。10.1016 / j.cub.2008.07.031。
- 12.
Starcevic A, Akthar S, Dunlap WC, Shick JM, Hranueli D, Cullum J, Long PF:基底后生动物维氏线虫(Nematostella vectensis)基因组编码莽草酸途径酶具有微生物来源。中国科学:自然科学,2008,29(3):344 - 344。10.1073 / pnas.0707388105。
- 13.
Danchin EGJ, Rosso MN:横向基因转移已经打磨了动物基因组:线虫的经验教训。中国生物医学工程学报,2012,27(2):329。
- 14.
Moran NA, Jarvik T:真菌的基因横向转移是蚜虫产生类胡萝卜素的基础。科学通报,2010,38(5):528 - 528。10.1126 / science.1187113。
- 15.
Nikoh N, Nakabachi A:蚜虫通过横向基因转移获得共生基因。中国生物医学工程学报,2009,7:12-10.1186/1741-7007-7-12。
- 16.
李世杰,叶芝,王志刚,王志刚,等:咖啡侵染性害虫基因水平转移的研究进展。美国国家科学研究院。2012, 39(4): 357 - 357。10.1073 / pnas.1121190109。
- 17.
Keeling CI, Yuen MM, Liao NY, Roderick Docking T, Chan SK, Taylor GA, Palmquist DL, Jackman SD, Nguyen A, Li M,等:山松甲虫(Dendroctonus ponderosae Hopkins)基因组草图。中国生物医学工程学报,2013,14 (3):R27-10.1186/gb-2013-14-3-r27。
- 18.
Fukatsu T:一种真菌过去的昆虫颜色。科学通报,2010,38(4):344 - 344。10.1126 / science.1190417。
- 19.
Bock R: DNA的交换:植物中的水平基因转移。植物科学进展,2010,15(1):11-22。10.1016 / j.tplants.2009.10.001。
- 20.
岳娟,胡晓霞,黄娟:水平基因转移在陆生植物创新适应中的作用。植物信号行为学学报,2013,8 (5):e24130-10.4161/psb.24130。
- 21.
岳健,胡晓霞,孙辉,杨勇,黄娟:水平基因转移对植物在土地上定殖的广泛影响。环境科学学报,2012,30(3):344 - 344。
- 22.
理查森·奥,帕尔默·JD:植物水平基因转移。实验学报,2007,58(1):1-9。
- 23.
公共科学学报,2013,9 (2):e1003265-10.1371/journal.pgen.1003265。
- 24.
Renner SS, Bellot S:叶绿体和线粒体基因组学,真核生物水平基因转移:真菌到植物和植物到植物转移的ganellar DNA。荷兰:施普林格;2012:223 - 235。
- 25.
刁晓霞,李丽玲,李丽霞,等。植物转座子水平转移的研究。公共科学图书馆,2006,4 (1):e5-10.1371/journal.pbio.0040005。
- 26.
Christin PA, Edwards EJ, Besnard G, Boxall SF, Gregory R, Kellogg EA, Hartwell J, Osborne CP: C-4光合作用通过周期性横向基因转移的适应性进化。动物学报,2012,22(5):445-449。10.1016 / j.cub.2012.01.054。
- 27.
黄娟:真核生物水平基因转移:弱链模型。生物学报,2013,35(10):868-875。
- 28.
杨晓明,杨晓明,杨晓明:开花植物寄主-寄主基因转移的研究进展。科学通报,2004,35(4):344 - 344。10.1126 / science.1100671。
- 29.
李霞,张天成,乔强,任志,赵娟,Yonezawa T, Hasegawa M, Crabbe MJ,李娟,钟勇:全寄生生物肉苁蓉(orobanchaceae)叶绿体全基因组序列显示宿主梭梭(Haloxylon ammodendron, chenopodiaceae)的基因缺失和水平基因转移。公共科学学报,2013,8 (3):e58747-10.1371/journal.pone.0058747。
- 30.
杨建军,杨建军,杨建军:植物遗传学研究:寄主植物的基因转移。自然科学进展,2004,27(3):344 - 344。
- 31.
杨文杰,杨文杰,杨文杰:开花植物向蕨类植物的基因转移。中国生物医学工程学报,2005,27(3):357 - 357。10.1098 / rspb.2005.3226。
- 32.
莫文平,李志刚,王志刚,王志刚,王志刚,王志刚,王志刚:寄生植物线粒体基因的横向获取及其与寄主线粒体基因的转化。中国生物医学工程学报,2010,30(4):344 - 344。
- 33.
吉田,马山,野崎,白苏K:寄生植物异月直蝽的水平基因转移。科学通报,2010,29(4):344 - 344 / 344。
- 34.
中国生物医学工程学报,2012,13:227-10.1186/1471-2164-13-227。
- 35.
张Y, Fernandez-Aparicio M,奥库穆埃克,Das M,焦Y, Wickett新泽西,Honaas洛杉矶,拉尔夫PE、Wojciechowski MF,子弹擦过蒂姆科MP, et al:进化水平获得了豆类的基因,白蛋白1,寄生植物Phelipanche aegyptiaca和相关的物种。中国生物医学工程学报,2013,29(4):344 - 344。
- 36.
非光合寄生开花植物金合欢(Orobanche)和金合欢(Phelipanche)的生活史和寄主范围的相关进化。中华流行病学杂志,2007,20(2):471-478。10.1111 / j.1420-9101.2006.01273.x。
- 37.
寄生杂草:一个世界性的挑战。杂草学报,2012,60(2):269-276。10.1614 / ws - d - 11 - 00068.1。
- 38.
唐文杰,李志刚,王志刚,王志刚,王志刚,王志刚。植物科学进展,1994,6:366 - 366。
- 39.
Goodstein DM, Shu S, Howson R, Neupane R, Hayes RD, Fazo J, Mitros T, Dirks W, Hellsten U, Putnam N,等:Phytozome:绿色植物基因组学比较平台。核酸研究,2012,40(数据库issue): D1178-D1186。
- 40.
Westwood JH, dePamphilis CW, Das M, fernando - aparicio M, Honaas LA, Timko MP, Wafula EK, Wickett NJ, Yoder JI:寄生植物基因组计划:了解Orobanche和Striga生物学的新工具。杂草学报,2012,60(2):295-306。10.1614 / ws - d - 11 - 00113.1。
- 41.
田娟,孙刚,丁强,黄娟,谢波:第三届生物信息学与计算生物学国际会议(BICoB): 2011年3月23 - 25日。路易斯安那州新奥尔良:第三届生物信息学和计算生物学国际会议(BICoB);2011.
- 42.
王丽娟,王丽娟,王丽娟,王丽娟,等:基于RNA-Seq数据的转录组组装研究。中国生物工程学报,2011,29 (7):644-10.1038/nbt.1883。U130。
- 43.
McNeal JR, Kuehl JV, Boore JL, de Pamphilis CW:完整的质体基因组序列表明寄生植物菊科植物中光合基因的保留存在强烈的选择作用。中国生物医学工程学报,2007,27(3):377 - 388。
- 44.
Wicke S, Muller KF, de Pamphilis CW, Quandt D, Wickett NJ, Zhang Y, Renner SS, Schneeweiss GM:飞天菊科光合和非光合寄生植物功能和物理基因组减少机制。植物学报,2013,25(10):3711-3725。10.1105 / tpc.113.113373。
- 45.
Hruz T, Laule O, Szabo G, Wessendorp F, Bleuler S, Oertle L, Widmayer P, Gruissem W, Zimmermann P:用于转录组元分析的参考表达数据库。中国生物工程学报。2008,2008:420747。
- 46.
Vandesompele J, De Preter K, Pattyn F, Poppe B, Van Roy N, De Paepe A, Speleman F:多内控基因几何平均实时定量RT-PCR数据的精确归一化。中国生物医学工程学报,2002,3 (7):RESEARCH0034。
- 47.
银N,贝斯特S,姜娟,登林林:利用实时荧光定量PCR技术研究人网织细胞基因表达。生物化学学报,2006,7:33-10.1186/1471-2199-7-33。
- 48.
Pfaffl MW, Tichopad A, Prgomet C, Neuvians TP:稳定管家基因、差异调控靶基因和样本完整性的测定:使用成对相关性的bestkeeper - excel工具。生物技术通报,2004,26(6):509-515。
- 49.
Andersen CL, Jensen JL, Orntoft TF:实时定量逆转录pcr数据的归一化:一种基于模型的方差估计方法,用于识别适合归一化的基因,应用于膀胱癌和结肠癌数据集。癌症研究,2004,64(15):5245-5250。0008 - 5472. - 10.1158 / - 04 - 0496。
- 50.
谢峰,肖鹏,陈东,徐玲,张波:miRDeepFinder:一种用于植物小rna深度测序的miRNA分析工具。植物分子生物学,2012,80(1):75-84。10.1007 / s11103 - 012 - 9885 - 2。
- 51.
顾旭,范德尔·维尔登·K:一个蛋白质家族功能结构差异的系统发育分析。生物信息学,2002,18(3):500-501。10.1093 /生物信息学/ 18.3.500。
- 52.
顾旭,邹勇,苏智,黄伟,周泽,Arendsee Z,曾勇:用于蛋白质家族功能差异分析的DIVERGE软件的更新。分子生物学杂志,2013,30(7):1713-1719。10.1093 / molbev / mst069。
- 53.
杨忠:PAML 4:最大似然系统发育分析。分子生物学杂志,2007,24(8):1586-1591。10.1093 / molbev / msm088。
- 54.
王丽娟,王丽娟,王丽娟,王丽娟:寄生植物与寄主间RNA易位的研究进展。昆虫学报,2009,65(5):533-539。10.1002 / ps.1727。
- 55.
勒布朗M,金正日G, Patel B, Stromberg V,韦斯特伍德J:量化的番茄和拟南芥移动rna贩运到寄生植物五角pentagona。植物学报,2013,200(4):1225-1233。10.1111 / nph.12439。
- 56.
杨晓明,王晓明,王晓明,王晓明,等。绵羊PgiC基因水平转移的研究进展。分子系统进化学报,2008,46(3):890-896。10.1016 / j.ympev.2007.11.031。
- 57.
Igarashi A, Yamagata K, Sugai T, Takahashi Y, Sugawara E, Tamura A, Yaegashi H, Yamagishi N, Takahashi T, Isogai M,等:苹果潜伏球形病毒载体在包括烟草、番茄、拟南芥、葫芦和豆类在内的广泛植物中可靠有效的病毒诱导基因沉默。中华病毒学杂志,2009,36(2):344 - 344。10.1016 / j.virol.2009.01.039。
- 58.
李志强,李志强,李志强:植物寄生系统中RNA的转运。前沿植物学报,2012,3:203。
- 59.
Aly R, Hamamouch N, Abu-Nassar J, Wolf S, Joel DM, Eizenberg H, Kaisler E, Cramer C, Gal-On A, Westwood JH:寄主植物与寄生杂草Phelipanche aegyptiaca Pers之间蛋白质和大分子的运动。植物细胞学报,2011,30(12):2233-2241。10.1007 / s00299 - 011 - 1128 - 5。
- 60.
胡晓峰,王晓峰,王晓峰,王晓峰:大分子分子间的迁移烟草还有全息寄生虫五角reflexa.应用物理学报,2001,29(3):344 - 344。10.1093 / jexbot / 52.354.173。
- 61.
姜丽,曲峰,李志,杜汉D:菟丝子种间蛋白质转运使其具有宿主特异性的耐除草剂特性。植物学报,2013,198(4):1017-1022。10.1111 / nph.12269。
- 62.
王志刚,王志强,王志强,等。植物韧皮部物质对全寄生植物的迁移特性研究。植物营养学报,2006,29(4):457 - 457。10.1093 / jxb / erj076。
- 63.
Pribylova J, Spak J:植物原质体的Dodder传播。方法Mol生物学,2013,938:41-46。10.1007 / 978 - 1 - 62703 - 089 - 2 - _4。
- 64.
Stegemann S, Bock R:植物组织移植物细胞间遗传物质交换。科学通报,2009,29(4):344 - 344。10.1126 / science.1170397。
- 65.
李志刚,李志刚,李志刚。植物叶绿体基因组水平转移的研究进展。美国国家科学研究院。2012, 39(7): 457 - 457。10.1073 / pnas.1114076109。
- 66.
潘建平,张志刚,张志刚,等:蛇尾螺细胞悬浮培养的均相strictosidine合酶。植物化学,1988,27(12):3811-3815。10.1016 / 0031 - 9422(88) 83023 - 1。
- 67.
Kutchan TM, Hampp N, Lottspeich F, Beyreuther K, Zenk MH:蛇纹蛇尾螺(Rauvolfia serpentina) strictosidine合成酶cDNA克隆。DNA序列测定及在大肠杆菌中的表达。中国生物医学工程学报,1998,37(1):1 -4。
- 68.
McKnight TD, Roessner CA, Devagupta R, Scott AI, Nessler CL:大红花空泡蛋白狭糖苷合成酶cDNA的核苷酸序列。核酸研究,1990,18 (16):4939-10.1093/nar/18.16.4939。
- 69.
陈锐,廖志华,陈敏,王强,杨彩霞,杨玉娟:轮孢罗纹蝽Strictosidine合成酶基因的克隆与鉴定。植物物理学报,2008,55(5):670-675。10.1134 / S1021443708050117。
- 70.
陆勇,王辉,王伟,钱忠,李玲,王娟,周刚,凯刚:蛇根狭甘醇合成酶cDNA的分子特征及表达分析。Mol生物学报,2009,36(7):1845-1852。10.1007 / s11033 - 008 - 9389 - y。
- 71.
Ma X, Panjikar S, Koepke J:蛇纹蛇尾螺(Rauvolfia serpentina) strictosidine合成酶的结构是一种新型的六叶片β -螺旋桨折叠结构。植物细胞学报,2006,18(4):907-920。10.1105 / tpc.105.038018。
- 72.
张志刚,张志刚,张志刚,张志刚。单萜类吲哚类生物碱合成的关键酶——狭甘醇合成酶的结构与功能。植物理化,2008,46(3):340-355。10.1016 / j.plaphy.2007.12.011。
- 73.
Hicks MA, Barber AE, Giddings LA, Caldwell J, O 'Connor SE, Babbitt PC: strictosidine合成酶样蛋白功能的进化。中国生物医学工程学报,2011,39(4):344 - 344。10.1002 / prot.23135。
- 74.
杨晓明,杨晓明,杨晓明等:线虫水平基因转移对植物寄生的催化作用。植物微生物相互作用,2011,24(8):879-887。10.1094 / mpmi - 03 - 11 - 0055。
- 75.
Richards TA, Soanes DM, Jones MD, Vasieva O, Leonard G, Paszkiewicz K, Foster PG, Hall N, Talbot NJ:水平基因转移促进了卵菌中植物寄生机制的进化。美国国家科学研究院。2011, 38(4): 457 - 457。10.1073 / pnas.1105100108。
- 76.
Mithofer A, Boland W:植物防御食草动物:化学方面。植物学报,2012,29(3):344 - 344。10.1146 / annurev - arplant - 042110 - 103854。
- 77.
凯斯勒A,鲍德温IT:植物对昆虫食草性的反应:新兴的分子分析。植物学报,2002,29(3):344 - 344。10.1146 / annurev.arplant.53.100301.135207。
- 78.
Ramakrishna A, Ravishankar GA:非生物胁迫信号对植物次生代谢产物的影响。植物信号行为学学报,2011,6(11):1720-1731。10.4161 / psb.6.11.17613。
- 79.
李波,杜威CN: RSEM:基于参考基因组的RNA-Seq数据的精确转录物定量。中国生物医学工程学报,2011,29(2):323-10.1186/1471-2105-12-323。
- 80.
闵晓娟,Butler G, Storms R, Tsang A: OrfPredictor预测EST-derived序列中蛋白质编码区域的方法。Nucleic Acids Res. 2005, 33 (Web Server issue): W677-W680。
- 81.
Martin M: Cutadapt从高通量测序读取中移除适配器序列。工程机械学报,2011,17:10-12。
- 82.
Kong Y: Btrim:用于下一代测序技术的快速、轻量级适配器和高质量微调程序。中国生物医学工程学报,2011,32(2):339 - 339。10.1016 / j.ygeno.2011.05.009。
- 83.
Milne I, Bayer M, Cardle L, Shaw P, Stephen G, Wright F, Marshall D:平板下一代序列装配可视化。生物信息学,2010,26(3):401-402。10.1093 /生物信息学/ btp666。
- 84.
奥苏贝尔F, Brent R, Kingston R, Moore D, Seidman J, Smith J, Struhl K:分子生物学的当前协议。纽约:威利;1987.
- 85.
Larkin MA, Blackshields G, Brown NP, Chenna R, McGettigan PA, McWilliam H, Valentin F, Wallace IM, Wilm A, Lopez R,等:Clustal W和Clustal X 2.0版本。生物信息学,2007,23(21):2947-2948。10.1093 /生物信息学/ btm404。
- 86.
Thompson JD, Gibson TJ, Plewniak F, Jeanmougin F, Higgins DG: CLUSTAL_X窗口界面:由质量分析工具辅助的多序列比对的灵活策略。中国生物医学工程学报,1997,25(24):476 - 482。10.1093 / nar / 25.24.4876。
- 87.
Keane TM, Creevey CJ, Pentony MM, Naughton TJ, McLnerney JO:对氨基酸基质选择方法及其在经验数据上的应用的评估表明,对基质选择的临时假设是不合理的。中国生物医学工程学报,2006,6:29-10.1186/1471-2148-6-29。
- 88.
Guindon S, Gascuel O:通过最大似然估计大系统发育的简单,快速和准确的算法。中国生物医学工程学报,2003,52(5):696-704。10.1080 / 10635150390235520。
- 89.
杨晓明,王晓明,王晓明,等。生物序列库的在线检索系统。生物化学学报,1996,28(5):364-369。10.1016 / 0300 - 9084(96) 84768 - 7。
- 90.
Suyama M, Torrents D, Bork P: PAL2NAL:蛋白质序列对齐到相应密码子对齐的鲁棒转换。核酸决议2006,34 (Web服务器问题):W609-W612。
致谢
感谢马永清教授(西北农林科技大学资源与环境学院,陕西省杨凌)提供的信息o . aegyptiaca种子,PPGP项目(弗吉尼亚理工大学,宾夕法尼亚州立大学,弗吉尼亚大学和加州大学戴维斯分校)用于转录组数据的生成Orobanche,Triphysaria,斯特1 KP项目(Gane Ka-Shu Wong,阿尔伯塔大学)用于生成转录组数据五角,Cassytha,Pilostyles.大部分计算工作在中国科学院昆明植物研究所高性能计算中心完成。支持这项工作的资助自然科学基金(31301037)、云南省自然科学基金的资助,中国(2013号fb068)赞助的一个项目为中华民国公司、SEM、创业资助从昆明植物研究所、中科院的资助云南招聘专家科学项目(2012号ha016),数千青年人才的资助计划,一个开放的基础遗传资源与进化国家重点实验室,中科院,以及马克斯·普朗克合作小组的拨款。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
构思和设计研究:GS和JQW。生成数据并进行分析:GS DZ JQ JY TS ZY JSW LW。贡献试剂/材料/分析工具:JLH SL JFW。撰写论文:GS JQW。所有作者都阅读并批准了最终的手稿。
Dale Zhang, Jinfeng Qi对这项工作也有同样的贡献。
电子辅助材料
中水平转移基因鉴定的概要图
附加文件1:o . aegyptiaca转录组。(pdf 249kb)
芸苔科特有的基因位点和结构
附加文件2:SSL基因。(pdf 110kb)
基因结构及其两种mRNA异构体
附加文件3:CaSSL.(pdf 88kb)
的
附加文件4:AtSSL1表达水平答:芥在不同的发育阶段计算。(pdf 229 kb)
的
附加文件5:AtSSL1表达变化答:芥根据genevestikator计算的不同处理。(pdf 3mb)
12870 _2013_1442_moesm6_esm.pdf
附加文件6:4个候选内参基因的引物序列及其refinder表表达排序。(pdf 60kb)
场地经过了积极的选择
附加文件9:OaSSL基因的o . aegyptiaca。(pdf 171kb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是原创作品的名称要注明出处。创作共用公共领域奉献弃权书(https://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
张,D,齐,J,岳,J。et al。根寄生植物Orobanche aegyptiaca和寄生植物五角南极光获得Brassicaceae-specificstrictosidine synthase-like基因通过水平基因转移。BMC植物生物学14,19(2014)。https://doi.org/10.1186/1471-2229-14-19
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-14-19
关键字
- 五角
- 水平基因转移
- 新基因
- Orobanche
- 寄生植物
- 类Strictosidine合成酶基因