摘要
背景
小麦种子萌发直接影响小麦产量和品质。虽然在一些作物植物中已经报道了种子萌发过程中的转录组和蛋白质组分析,但小麦种子萌发过程中的动态转录组特征尚未进行。利用Affymetrix小麦基因组阵列首次对面包小麦优良品种冀麦20种子萌发不同阶段进行了全面的动态转录组分析。
结果
在吉麦20号五个种子萌发阶段共鉴定出61703个探针集,代表51411个转录本,其中采用方差分析和随机方差模型鉴定出2825个差异表达探针集,对应2646个功能不同的转录本。种子萌发过程包括快速的初始吸收阶段(吸胀后0-12小时[HAI])、平台期(12-24 HAI)和进一步的吸水阶段(24-48 HAI),对应于小分子蔗糖降解到三大营养物质代谢和光合作用的转换。分层聚类分析和MapMan分析揭示了种子萌发过程中几种重要代谢途径及相关功能基团的变化。KEGG构建的信号通路网络显示了编码磷酸果糖激酶家族蛋白的三个重要基因,其中果糖- 1,6 -二磷酸酶和utp -葡萄糖-1-磷酸脲基转移酶位于中心,表明它们分别在糖酵解途径、糖异生和糖发生中起关键作用。我们选择了几种重要的途径,根据它们的程度值建立了代谢途径网络,从而找到了对种子萌发至关重要的途径。利用基因共表达网络和k-core算法分析了涉及转运、信号通路、发育、脂质代谢、防御反应、氮代谢和转录调控的51个基因,确定了哪些基因在萌发过程中起关键作用。发现23个有意义基因,定量RT-PCR分析验证了12个有意义基因的表达模式。
结论
小麦种子萌发包括三个不同的阶段,包括复杂的调控网络,涉及大量的基因。这些基因属于许多功能群,它们的共同调控保证了正常的萌发。我们的研究结果为种子萌发过程中的代谢变化和一些重要基因之间的相互作用提供了新的见解。
背景
小麦(小麦L., 2n = 6x = 42, AABBDD)是一种异六倍体品种,是世界上最重要和最广泛栽培的作物之一。小麦具有广泛的农艺适应性,可在斯堪的纳维亚半岛和俄罗斯北纬67°至阿根廷北纬45°的地区种植,包括热带和亚热带地区[1,2].这种作物主要用于人类食品和牲畜饲料。
小麦种子的化学成分包括蛋白质、碳水化合物、脂类、核酸、色素、维生素、酶和无机物质。成熟的小麦种子主要由淀粉(高达70%)和蛋白质(12-15%)组成。碳水化合物主要以淀粉的形式存在,占整个种子重量的55-70%,在种子发芽时被激活提供能量。种子蛋白包括白蛋白、球蛋白、麦胶蛋白和谷蛋白[3.,4],其中麦胶蛋白和谷蛋白是决定面团粘弹性的主要贮藏蛋白[5].它们的动员还在种子萌发过程中提供能量和其他中间产物。白蛋白和球蛋白在人体营养中很重要,因为它们含有丰富的必需氨基酸,如赖氨酸、色氨酸和蛋氨酸[5].
种子萌发始于吸胀,即静止的干燥种子吸收水分,并随着胚轴的伸长而终止[6].随后的储备调动与幼苗生长有关。种子经历了三个阶段的生理和形态变化过程。第一阶段(第一阶段)是快速的初始吸收,种子开始膨胀,种皮变软。同时,淀粉、蛋白质、脂类等贮藏物质的物理状态逐渐发生变化。然后,发芽的种子进入平台期(第二阶段),水分吸收增加(第三阶段)。第三阶段发生在发芽完成后胚轴拉长的时候[7].呼吸作用和能量产生在整个种子萌发过程中起着关键作用。开始时,种子萌发的能量主要由无氧呼吸提供,在吸胀过程中,随着吸氧和二氧化碳释放的加快,呼吸活动增加。氧摄取与细胞色素氧化酶的氧化磷酸化有关[8].
由于种子萌发与小麦幼苗成活率及后续营养生长密切相关,直接影响小麦产量和品质。种子萌发的初步研究主要集中在种子的生理和生物化学方面[6,7,9,10],让我们对种子的萌发过程有了基本的了解。近年来,蛋白质组和转录组方法已被用于研究植物种子萌发的生化机制。大量的蛋白质组学研究已经在许多植物的种子萌发,如拟南芥[11],水芹[12]、甜菜[13),Medicago truncatula[14],大麦[15],玉米[16]和米[17];然而,蛋白质组方法检测到的蛋白质数量是有限的,因此不允许进行全基因组比较。干燥成熟的种子含有大量的mRNA种类。棉花是第一种被发现在成熟的干燥种子中储存RNA的植物[18];20世纪90年代后,人们发现植物成熟的干燥种子中普遍存在储存的RNA [19- - - - - -21].种子中储存的mRNA反映了种子萌发过程中的基因表达模式[22].Affymetrix阵列可以提供种子萌发过程中全转录组水平的全面描述和实时变化,已被用于研究许多植物的种子萌发生物学过程,如拟南芥[22],大麦[23,24],大米[25]和玉米[26].研究小麦籽粒发育主要采用转录组方法[27- - - - - -29].在转录水平上对小麦种子萌发的研究仍然局限于一个特定的萌发阶段[30.];小麦种子萌发过程中转录组的全面动态表征尚未完成。
具有A、B和D基因组的现代异源六倍体小麦具有巨大而复杂的基因组(高达17000 Mb)。小麦基因组测序最近取得进展[31].A基因组计划u基因组小麦属植物urartu和Dt基因组山羊草属tauschii,六倍体小麦A和B基因组的祖细胞已分别完成[32,33],促进了小麦种子萌发的蛋白质组和转录组的进一步研究。本研究选用高产、适应性广、品质优良的我国面包小麦优良品种冀麦20号[34],使用GeneChip®小麦基因组阵列(Affymetrix, Santa Clara, CA)在五个萌发阶段进行首次动态转录粒微阵列分析。本研究结果为研究小麦种子萌发的分子机制提供了新的思路。
结果
种子萌发过程的形态和扫描电镜观察
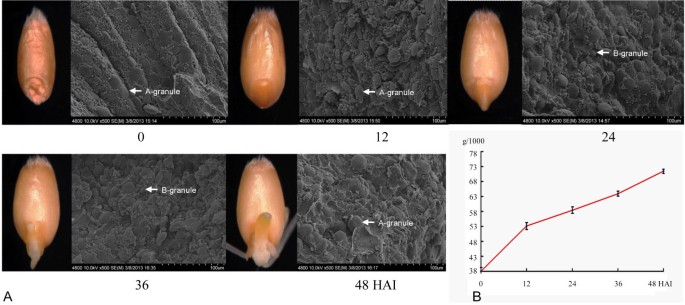
自吸后,小麦种子逐渐膨胀,但自吸后12小时(HAI)胚外观基本不变,如图所示1A.在24 HAI时,胚芽周围的结构被自由基穿透,在36 HAI时自由基和芽都出现了。在48hai时胚芽呈微绿色,自由基伸长。
在萌发过程中,种子重量随着吸胀而增加(图1B)发芽初期吸水快;结果表明,种子重在12 HAI时迅速增加,在24 ~ 48 HAI时继续稳定增加。丰富的早期吸胀为水溶性代谢产物的动员奠定了基础,为种子萌发和随后的幼苗生长提供了持续的能量。
小麦胚乳中含有三种淀粉粒:a型(直径大于10 μm)、b型(直径5 ~ 10 μm)和c型(直径小于5 μm)。这些淀粉粒为种子萌发提供了重要的能量供应。通过扫描电镜观察胚乳,a型淀粉颗粒的直径随着种子的吸吸而增大(图1A和附加文件1:图S1)。胚乳吸水后空间稀疏,淀粉粒几乎全部膨胀,说明贮藏淀粉逐渐降解,为萌发提供能量。
种子萌发过程中转录组表达谱及功能分类
为了研究种子萌发过程中的转录组表达谱,我们使用GeneChip®小麦基因组阵列进行了基因芯片分析,其中包含61,127个探针集,代表55,052个转录本[35].在我们的实验中,共有61,703个探针集(61,115个实际探针集和其他588个重复探针集的总和)被识别出代表51,411个转录本(附加文件2:表S1)。61,703套探针可分为35个bin;在19782个不同功能的探针组中,34个bin被自动标记为代谢途径和大酶家族。另有41,921个探测集被分类为未知或未分配(附加文件2:表S2)。与蛋白质、RNA、酶家族、信号和转运蛋白相关的探针集在具有功能的探针集中所占比例较大,说明它们在种子萌发过程中起着重要作用。
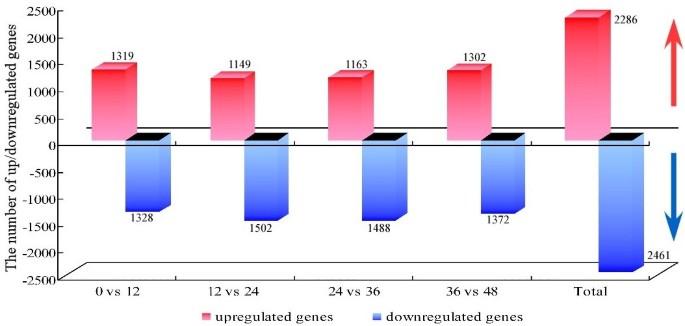
全基因组转录谱分析表明,在萌发过程中出现了广泛的基因表达。共有2825个探针组对应2646个转录本被宣布在五个种子萌发阶段差异表达(附加文件2:表S3)。将连续时间点进行比较,得到两个连续时间点之间上调或下调基因的数量(图2).结果表明,在种子萌发过程中,2286个基因表达上调,2461个基因表达下调。
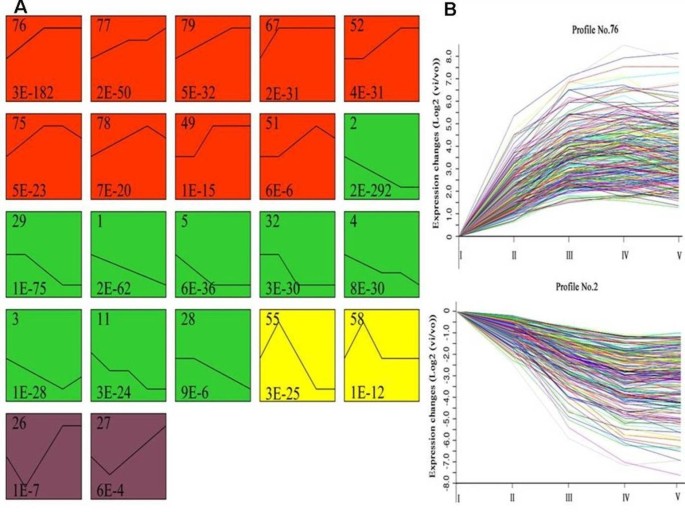
对2646份转录本的进一步分析表明,有22个表达谱高度显著,并将这些表达谱分为4组。如图所示3., I组上调基因和II组下调基因占总差异表达基因的87.7%。III组基因在0 ~ 12 HAI之间表达上调,但在萌发后期表达急剧下调。IV组基因起初下调,12 HAI后逐渐上调。在22个表达式配置文件中,最重要的两个模式(配置文件2和76)根据他们的P-值差异表达基因数量最多,分别为239个和245个(图3.).
用模型谱分析了2646份转录本的主要表达模式和两个显著表达模式(谱2号和76号)。一个,每个方框代表一个模型表达式概要。配置文件框中上面的数字为型号配置文件编号,下面的数字为p-value用于总结不同的基因表达模式。共有22个基因表达模式显著p值(p< 0.05)。相同的颜色代表同一组。用红色标记的模型概要文件(组I)表示上调的表达模式,而绿色概要文件框(组II)表示下调的表达模式。第55和58号剖面(III组)属于先上调后下调的模式,其余剖面箱(IV组)属于与III组相反的模式。B在种子萌发过程中,2号基因表达量增加,76号基因表达量减少。横轴为萌发期,纵轴为Log2归一化后基因表达量的时间序列。
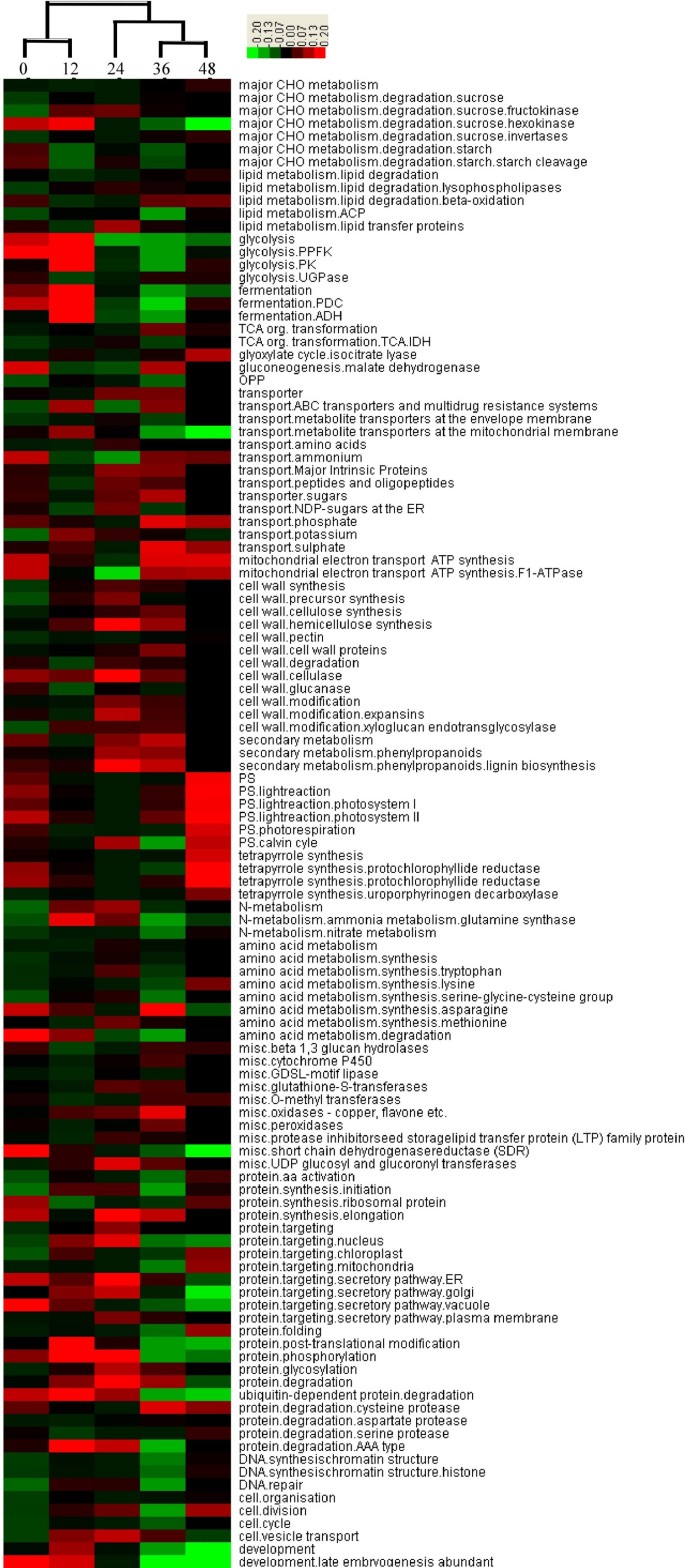
利用层次聚类分析(HCA)揭示了种子萌发各阶段被激活的一些重要基因功能类别的坐标变化(图4).根据聚类可以划分出3个不同的相,对应于12和24 HAI的两个明确开关点。第一个快速摄取的阶段是0-12 HAI,此时参与小分子蔗糖降解的基因被激活。第二阶段为12 - 24hai,与三大营养物质(碳水化合物、蛋白质和脂类)相关的代谢基因和细胞壁代谢开始激活。24 ~ 36 HAI,种子萌发进入第三阶段,这是一个明显的过渡阶段,代谢基因持续大量表达。在萌发末期(48hai),与光合作用相关的基因被激活,胚轴明显拉长。因此,种子萌发过程经历了从小分子蔗糖的降解到三种主要营养物质的代谢再到光合作用的两次主要转换。因此,能量储备在萌发过程中呈下降趋势,尤其是在24和36 HAI。
从储备动员中提供能源
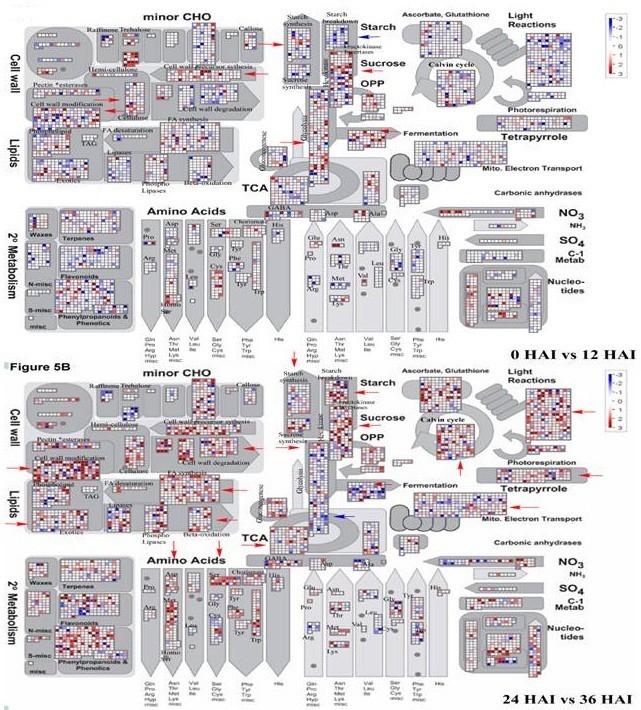
MapMan已被证明是绘制转录组数据、定义功能类别和进行时间过程分析以识别显著过度代表的功能基团的有效工具,并已应用于拟南芥、大麦和水稻的转录组分析[24,25,36].本研究通过MapMan对不同种子萌发阶段被激活的重要基因功能群进行分析,结果如图所示5和附加文件2:表S2。
胚乳中的储备积累开始于种子发育早期,然后结束于种子成熟。在成熟过程中,小麦种子积累储备物质,包括淀粉、蔗糖、脂类和储存蛋白质。种子内的代谢活动在休眠期间急剧下调,然后在吸胀、萌发和幼苗生长期间重新激活。当种子开始吸水时,休眠就被打破,静止的胚就能发芽。除了储备的调动外,种子萌发过程也是茎尖分生组织(SAM)和根尖分生组织(RAM)发育的重要阶段。在24 HAI位点发现了几个发育相关基因的显著上调,如与WUSCHEL-CLAVATA通路相关的CK202188和BJ272365,与SAM和RAM相关的CD869453和CK2018931).此外,一些编码信号受体激酶的分生组织相关基因,如BJ266952和BJ307684,在种子萌发过程中也表现出上调的表达模式。
根据基因表达数据,沉积淀粉、蔗糖和储存蛋白的动员在种子萌发早期就开始了(图5A和附加文件3.:图S2A)。例如,糖酵解在吸胀开始时随着积累的蔗糖的动员而被激活(图)4而且5).在此过程中,蔗糖合成酶(SUSY)、己糖激酶(hexokinase)、磷酸果糖激酶(PPFK)、丙酮酸激酶(PK)等促进糖酵解的关键酶的编码基因均表达上调(图)4而且5A).例如,从0 - 12 HAI,作为糖酵解的关键基因,编码PPFK的基因CD866884上调了7倍,而编码PK的基因BJ252827和CK207050分别上调了14倍和15倍(表2)1);然而,编码淀粉合成关键酶udp -葡萄糖焦磷酸化酶(UGPase)的基因(CD491538)在糖酵解过程中被下调(表2)1和图4).相应的,由于丙酮酸不断合成,丙酮酸转化为酒精进入发酵途径,参与发酵过程的基因随着糖酵解而上调。例如,发酵中两种重要的催化酶,丙酮酸脱羧酶(PDC, CD934753)和乙醇脱氢酶(ADH, CD868238)被显著上调(图4和表1).糖酵解和发酵途径相关基因在12 HAI后均下调,可能是由于储存的蔗糖的动员结束(图)4而且5一个)。
种子萌发进入静止期。淀粉动员发生在蔗糖降解后。当种子胚乳吸收了一定的水分后,在12 ~ 24 HAI之间胚乳中的淀粉基因被激活,24 HAI后达到一个非常繁荣的时期,36 HAI后由于淀粉的持续消耗,相关基因逐渐下调(图5B和附加文件3.:图S2)。
淀粉酶是小麦淀粉裂解的主要酶。在我们的研究中,大多数与α -淀粉酶相关的基因,如CA719001、AL820663和BF293263,在种子萌发过程中从12 HAI开始上调,在37 HAI达到最大值,而β -淀粉酶在种子萌发过程中表达下调(表2)1).这一结果表明-淀粉酶是水解淀粉以提供葡萄糖的主要酶。随着淀粉降解基因的转录激活,编码果糖激酶转化酶和己糖激酶、蔗糖合成和糖酵解的基因转录物增加(图5B和附加文件3.:图S2B),这可能是由于已糖供应和动员淀粉储备合成蔗糖[24].
种子胚乳或子叶中储存的有机物质在利用过程中需要分解成铵、磷酸盐、氨基酸、多肽、寡肽、糖以及主要的固有蛋白等小分子化合物并运输到胚根和胚中,因此运输基因在储备动员后被激活并上调(图)4).我们的结果显示,编码线粒体膜上代谢物转运蛋白的转录本在0到12 HAI期间积累最快,而编码包膜上代谢物转运蛋白的转录本在12到24 HAI期间上调最快(图4).
脂质动员在吸胀后不久开始,随后是一个迟滞期。脂肪酶是脂类水解的关键酶。因此,脂肪酸(FA)不断合成(图5A和附加文件3.:图S2A)。24 HAI后,FA合成转录物高度上调,脂质动员结束时,其表达逐渐下调(图5B和附加文件3.:图S2B)。β-氧化途径(FA降解的主要途径)和磷脂酶相关基因也在24 ~ 36 HAI之间显著上调(图5B). β-氧化参与脂质降解,最终导致过氧化物酶体释放能量,为蔗糖的产生提供碳供应[24].
贮藏蛋白主要位于胚乳和糊粉层。根据我们的基因表达数据,沉积的存储蛋白的动员开始于种子萌发后期(图5B),其代谢产物氨基酸是幼苗发芽后早期生长的重要营养资源。在此期间及之后的时间(从约36 HAI开始),用于存储蛋白降解的半胱氨酸蛋白酶、天冬氨酸蛋白酶和丝氨酸蛋白酶等转录物显著增加(图4).Asp、Met、Ser、Cys、Phe、Trp等氨基酸代谢基因同时被激活(图5B和附加文件3.:图S2B)。从12 - 24 HAI,磷酸化和糖基化等蛋白修饰相关的基因表达增加(图4).这表明在种子萌发阶段,需要合成一些碎屑酶来调动贮藏蛋白。
根据我们的实验,在种子萌发过程中,光合作用基因在储备动员开始后被轻度动员。如图所示1A, 48hai时子叶逐渐变绿,说明光合作用准备基本完成。光合作用相关基因,包括光反应、光呼吸和光周期相关基因被激活,并在36hai结束时表达高度上调(图)4而且5B)。
种子在萌发初期因吸水而迅速膨胀。与细胞壁主要成分(纤维素、果胶、半纤维素、膨胀素)相关的基因在0 ~ 12 HAI时被激活(图4而且5A).由于吸胀后的停滞,细胞壁合成酶和降解相关基因几乎下调。直到胚根冲破种皮,种子才再次吸水,这时这些基因逐渐上调(图5B和附加文件3.:图S2A)。24 HAI后细胞壁大量降解(图5B),它激活一些与降解有关的重要酶,如纤维素酶和果胶酸裂解酶(附加文件2:表S1)。与此同时,细胞壁合成和修饰基因大幅上调(图4而且5B).这说明在种子萌发过程中,大量断裂的细胞壁被降解成小分子,为细胞壁合成提供原料。
信号通路网络分析
在京都基因与基因组百科全书(Kyoto Encyclopedia of genes and Genomes, KEGG)数据库中,利用Java语言根据信号基因的功能建立信号通路网络(图6).在该网络中,三个基因处于信号通路网络的中心:Os01g0191700(磷酸果糖激酶家族蛋白),Os01g0866400(果糖- 1,6 -二磷酸酶)和Os09g0553200 (utp -葡萄糖-1-磷酸脲基转移酶)(附加文件)4:表S4和表S5)。磷酸果糖激酶是糖酵解途径中的一种限速酶,它从ATP中提供一个磷酸基,以促进各种各样的生物过程。PPFK家族蛋白的表达模式属于第55号谱线,在12 HAI后先上调后急剧下调。果糖- 1,6 -二磷酸酶是糖异生中将果糖- 1,6 -二磷酸转化为果糖-6-磷酸的酶。其转录水平位于27号染色体,趋势与PPFK家族蛋白相反。糖异生中果糖- 1,6 -二磷酸酶的活化在氨基酸代谢中起着重要作用。UTP-葡萄糖-1-磷酸uridylyltransferase,又称UTP-葡萄糖焦磷酸化酶(UGPase),与糖发生有关,由葡萄糖-1-磷酸和UTP合成udp -葡萄糖,并在整个萌发过程中上调,在36 HAI时达到峰值。
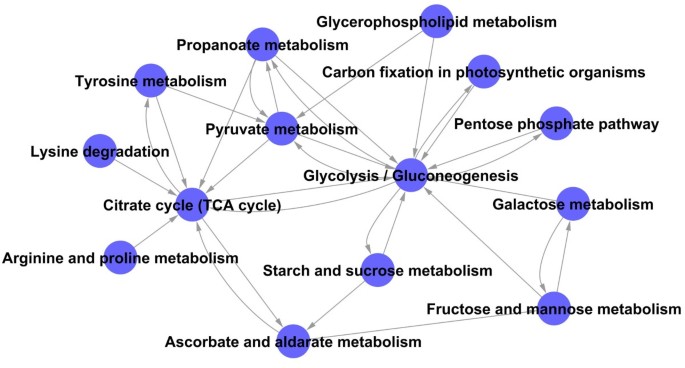
代谢途径网络分析
代谢通路网络建立如图所示7.根据度值(附加文件5:表S6),糖酵解和糖异生代谢处于通路网的中心位置,提示糖酵解和糖异生相关基因如编码PPFK家族蛋白、PK和果糖-1的基因、6-双磷酸酶的基因在为种子萌发提供能量方面起着关键作用。柠檬酸循环(TCA循环)在途径网络中度值第二高,主要为种子的后期萌发提供能量。作为TCA循环的关键酶,4个酶编码基因(异柠檬酸脱氢酶家族蛋白、丙酮酸脱氢酶E1组分β亚基、苹果酸脱氢酶和2-羟戊二酸脱氢酶复合物二氢脂酰赖氨酸残基琥珀酰转移酶组分)在萌发过程中逐渐上调。这些与能量代谢相关的基因在这一过程的早期启动,表明线粒体呼吸活动的激活和ATP的释放。据此,至少有10个与液泡ATP酶和线粒体ATP合成酶复合物以及线粒体载体蛋白相关的基因在种子萌发过程中优先上调(附加文件)2:表S1)。发酵途径中的醇脱氢酶(ADH)基因也大幅上调,为种子萌发提供能量。丙酮酸是一种重要的中间产物,起着介导作用,丙酮酸代谢是另一种重要途径。丙酮酸代谢通过两种方式将丙酮酸转化为乙酰辅酶a,用于脂质生物合成和TCA循环[17,37].一种方法是由丙酮酸脱氢酶(PDH)催化,它催化丙酮酸转化为乙酰辅酶a。另一种方法是由丙酮酸脱羧酶(PDC)和乙醛脱氢酶(ALDH)催化丙酮酸不可逆地转化为乙醛和CO2,所以乙醛可以转化为乙酸,产生额外的乙酰辅酶a。
从代谢途径网络来看,赖氨酸、酪氨酸、精氨酸和果仁氨酸等多种氨基酸被触发,为氮代谢提供主要氮源,为TCA循环提供中间产物。赖氨酸降解涉及三种酶:乙醛脱氢酶(ALDH)家族蛋白,含有SET结构域的蛋白和2-羟戊二酸脱氢酶复合物的二氢脂酰赖氨酸残基琥珀酰转移酶组分。富马酰乙酰乙酰酶、天门冬氨酸转氨酶和乙醇脱氢酶2参与酪氨酸代谢。在精氨酸和脯氨酸代谢中,与干种子相比,4个酶基因(乙醛脱氢酶、天门冬氨酸转氨酶、δ -1-吡罗-5-羧酸合成酶和精氨酸琥珀酸裂解酶)的表达上调,而2个酶基因(乙醛脱氢酶家族蛋白和谷氨酰胺合成酶根同工酶2)在萌发过程中表达逐渐下调(附加文件)2:表S1)。
基于k核算法的基因共表达网络
为了确定在小麦种子萌发过程中起关键作用的功能基因,我们利用k-core算法的基因共表达网络进一步分析了一些重要通路的基因(图2)8).网络中最中心的基因具有最高的度值。根据k-core值,我们的结果中k-core水平较高的31个基因被认为具有核心状态(附加文件6:表S7)。大多数基因与转运、信号通路、发育、脂质代谢、应激与防御反应、氮代谢和转录进程有关。编码胚胎丰度蛋白1的CD490513、编码CAX3(阳离子交换器3)的CD452864和编码CYP78A8的CK208119三个最重要的核心基因位于k核水平最高的网络中心。他们直接调节24个邻近基因根据他们的程度(附加文件6:表S7)。这些基因在萌发过程中显著降低,CD490513基因降幅最大(表490513)1).在该网络中,乙烯形成酶CK216168(乙烯形成酶)是上调基因之一(表216168)1),位于网络中心,k核分别为14度和19度,表明其参与多种功能,包括防御反应、信号转导、脂质代谢过程、碳水化合物代谢过程和乙烯生物合成过程。
在该网络中,BJ291458、BJ282439和CD453856三个基因参与蛋白质代谢。BJ291458 (UBQ3) k核分别为12度和16度,BJ282439 (Ubiquitin E3) k核分别为10度和13度,主要参与调控蛋白质降解(表2)1).由于蛋白质降解产物需要从胚乳转运到胚胎,蛋白质降解相关基因与转运相关基因(CD452864和CD373830)之间的共调节是一致的,表明这些基因在网络中正调控,说明蛋白质降解基因的下调/上调会导致转运基因的下调/上调。CD453856 (Chaperonin;折叠)是调控蛋白质折叠,然后通过调控另一个参与翻译后修饰的基因CA649522来催化蛋白质的修饰(图8和表1).由于CD453856和CA649522的功能都是反向蛋白降解的过程,我们可以推断CD453856和CD452864的共同调节是负调控,这与图中结果一致8.
qRT-PCR检测基因表达谱
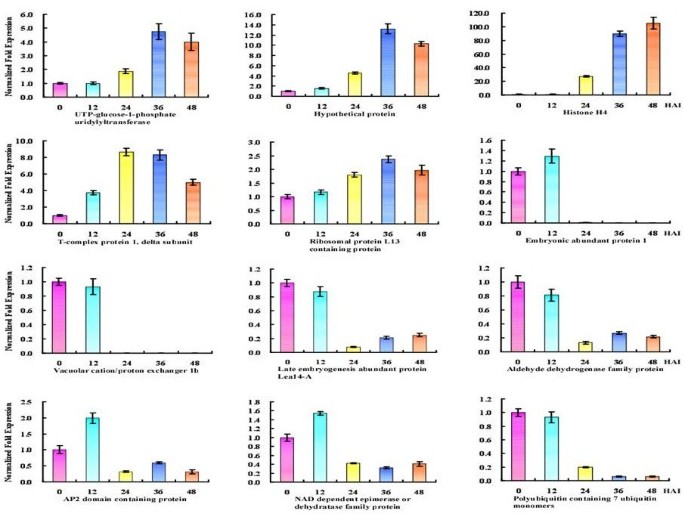
实时定量逆转录PCR (Real-time quantitative reverse transcriptpcr, qRT-PCR)检测了12个代表性基因的表达,其中5个基因表达上调,7个基因表达下调7:表S8)。优化后的实验结果表明,12个基因的扩增效率和特异性较高8:图S3)。如图所示9,这12个基因的表达模式与其转录表达模型基本一致。
讨论
种子萌发所需能量
植物在种子萌发过程中需要大量的能量进行生理活动。由于缺乏矿物吸收系统和光合作用装置,发芽的种子依靠储备动员,主要是淀粉、蛋白质和脂类,为生长提供必需的能量,直到幼苗成为光自养的[7,38- - - - - -40].小麦是单子叶作物,其能量储备主要储存在胚乳中,胚乳在萌发过程中因胚生长而降解。由于含氧量有限,在种子吸胀的早期,能量由糖酵解和酒精发酵补充。速率限制酶PPFK和PK参与糖酵解,而PDC和ADH参与乙醇发酵。谷物种子萌发时的能量需求似乎主要是通过糖酵解来满足的[17].厌氧呼吸提供的能量不能满足种子发芽的需要;此时,TCA循环在富氧条件下提供了大量的能量。TCA循环中的限速酶之一IDH在种子萌发和萌发后积累。淀粉动员、脂质动员和蛋白质动员的最终目的是提供能量。能量储备的降解产物总是进入糖酵解,然后通过TCA循环和线粒体电子传递合成ATP。因此,随着淀粉、脂质和蛋白质的降解,能量代谢相关基因(糖酵解、TCA循环、氧化磷酸化和线粒体电子传递)从36 - 48 HAI开始启动,表明线粒体呼吸活动的激活和ATP的释放(图)5B和附加文件3.:图S2B)。上述三种呼吸途径对多种细胞功能提供能量至关重要[5,41],如本著作所述。
淀粉和蔗糖代谢
贮藏的胚乳储备主要是淀粉,淀粉的降解是由淀粉酶催化的。本研究发现了一些对淀粉降解至关重要的基因,如编码α -淀粉酶的BF293263和AL820663(表2)1).淀粉产物的降解可以通过两种方式进入糖酵解。首先,淀粉酶可以将淀粉转化为麦芽糖,麦芽糖再分解成葡萄糖,参与糖酵解;第二,淀粉磷酸化酶可以将淀粉转化为葡萄糖-1-磷酸,葡萄糖-1-磷酸进入糖酵解。在本研究中,α -淀粉酶和淀粉磷酸化酶相关基因在12 HAI后大多上调(图5和附加文件2:表S1)。这一结果表明,淀粉在胚根突出前12 HAI左右发生了大规模的动员,与大麦的结果相似[24].Pritchard等人认为,大部分蔗糖来自于贮藏时的脂质降解,而不是来自拟南芥种子内的其他可溶性糖[40];然而,我们的研究结果显示,最初的蔗糖来源于可溶性糖,而大部分蔗糖来自于储存淀粉脂质的动员以及一些氨基酸(图5) -类似于大麦种子的发芽[24].蔗糖可被蔗糖合成酶(SUSY)分解为葡萄糖和果糖,然后进入糖酵解。我们发现了一个与AT3G43190高度相似的基因(M26672.1),该基因编码SUS41和附加文件2:表S1)。SUS4是SUSY的一种酶,SUSY是一种高度调控的酶,可可逆地将蔗糖和核苷二磷酸转化为相应的核苷二磷酸葡萄糖和果糖;这一过程可发生在大多数植物中,包括谷类作物[42- - - - - -49].
蛋白质动员
除了淀粉之外,种子中其他最重要的能量储备是蛋白质。在萌发早期,许多与蛋白质合成、修饰和降解相关的基因明显上调(图4).我们假设这些蛋白质是水解酶和与激素信号有关的蛋白质,它为当前和随后的代谢做准备。根据前面的分析,我们还可以推测,在干种子中,许多基因编码了一些前期代谢酶。当种子吸收水分时,酶直接激活并催化重要的代谢过程,如糖酵解。蛋白质降解发生在种子萌发后期(约48 HAI);但在这一过程中,编码半胱氨酸蛋白酶、天冬氨酸蛋白酶、丝氨酸蛋白酶等重要酶的基因表达被激活的时间稍早(图4).已知种子萌发部分受到赤霉素(GAs)的控制,其作用是刺激种子萌发[40],因为植物激素GA在萌发过程中对触发上述主要蛋白酶有重要作用[24,50,51].
发育基因的激活
茎分生组织和根分生组织作为植物的主要分生组织,在植物胚的相反极性开始发育。SAM含有一种自我更新的未分化多能干细胞群,为所有地上结构的发育提供细胞[52,53],而RAM则产生主根和侧根系统[54].在本研究中,sam相关基因CD869453属于RNA功能类,参与转录调控,并随着种子吸吸而被激活。该基因在种子萌发过程中表达上调,在36 HAI时达到最高表达水平。ram相关基因CK201893在种子吸吸后迅速激活,并在种子萌发过程中呈上调表达模式,属于氧化还原功能类,参与胁迫响应(表2)1).以前的报道推测被子植物的RAM是从SAM进化而来的,这可能是由于植物对不断变化的环境要求的适应[55,56].
在拟南芥, SAM中的干细胞稳态受WUSCHEL-VLAVATA (WUS-CLV)负反馈环路控制[52,57,58].在本研究中也发现了这种负反馈。BJ272365 (WUS)和CK202188 (CLV1)在种子萌发过程中表达谱相似;即,它们从0上调至24 HAI,从24下调至36 HAI, \然后在48 HAI再次上调(表1).这一结果表明,在小麦种子萌发过程中,有类似的发育途径拟南芥.
光合作用基因的激活
我们的研究结果表明,在48hai左右,光合装置逐渐整合到种子中,表明随后(48hai后)的生长不仅依赖于能量储备的动员,还依赖于光合作用。与光合作用相关的基因,包括光反应、光呼吸和卡尔文循环,在48hai开始大量激活。四吡咯已被证明对光合作用很重要[59].根据大麦的报告[24],在36 ~ 48 HAI期间,四吡咯合成基因和光合作用基因均被激活(图4而且5).在此期间,与四吡咯合成相关的两个编码原叶绿素还原酶和尿卟啉原脱羧酶的基因表达显著上调(图4).光合作用利用叶绿素将光转化为化学能,原叶绿素还原酶和尿卟啉原脱羧酶是合成叶绿素的关键酶;由此可以推断,四吡咯是叶绿素合成的中间产物,四吡咯可以通过调节叶绿素来调节光合作用。
防御基因的激活
种子的发育和成熟伴随着耐旱性的增强[60].当置于水中时,由于外部环境的变化,小麦种子在萌发过程中能够激活一系列机制来应对许多生物和非生物胁迫(附加文件)9:图S4)。细胞识别外部压力并诱导许多信号蛋白,包括信号受体激酶,光相关蛋白,钙依赖蛋白激酶,g蛋白和14-3-3蛋白(附加文件)10:图S5)。研究发现,对水、盐、缺氧、冷应激和创伤或病原体反应的耐受性需要基因表达的改变[61].结果表明,这些信号蛋白的转录本在萌发过程中积累,多数基因的表达在萌发早期上调,说明胁迫反应主要由基因调控。呼吸活动的恢复是吸胀后的最初变化之一,可在几分钟内检测到。7].据报道,在48 HAI条件下,大麦种子萌发过程中参与清除活性氧(ROS)的谷胱甘肽过氧化物酶、氧化还原代谢和超氧化物歧化酶基因被激活。24].ROS在生物和非生物胁迫中作为基因表达的触发器发挥关键作用[49].在我们的研究结果中,氧化还原状态、过氧化物酶和谷胱甘肽- s转移酶基因的转录激活是由呼吸爆发引起的。它们的表达主要出现在24 HAI左右,略早于大麦。
植物激素调节植物生长,调节植物对生物和非生物胁迫的反应[62].在激素信号通路中,除了脱落酸和赤霉素这两种主要激素参与控制萌发事件外,其他激素还参与一个复杂的相互作用网络,调节小麦种子萌发[24].研究表明,脱落酸(ABA)是一种积极的休眠调节因子,是一种消极的萌发调节因子,赤霉素(GA)、乙烯和油菜素内酯(BA)促进种子萌发并抵消ABA的作用[63].我们的结果表明,大多数ABA生物合成基因由于休眠的打破,从0到12 HAI略有上调,然后在萌发后期急剧下调(附加文件)10:图S5)。而GA、BA和乙烯生物合成基因在小麦种子萌发初期至萌发后期均有显著上调。这些结果表明,双子叶种子和单子叶种子对植物激素的调控是相似的。此外,泛素-蛋白酶体途径已被证明在激素信号传导中起着令人惊讶的重要作用[62].我们的结果表明,依赖于泛素的蛋白质降解开始于萌发的最开始,紧跟着吸胀(图4).这些蛋白质的降解明显早于贮藏蛋白质和淀粉的降解,这可能是种子萌发的前提条件,如在水稻中发现的[17].
丝裂原活化蛋白激酶(MAPK)是丝氨酸/苏氨酸特异性蛋白激酶,可被多种应激刺激激活,包括渗透压、干旱、温度、盐度、病原体感染、损伤和ROS,并调节细胞活动,如增殖、基因表达、分化、有丝分裂、细胞存活和细胞死亡[64,65].有研究报道,应激激活MAPKs的底物大多是动物、酵母细胞和植物中的转录因子[66,67].在小麦种子萌发过程中,许多与生物和非生物胁迫相关的转录因子(tf)的表达发生了显著变化,如乙烯响应元件结合蛋白家族(ERF TFs)、bZIP转录因子家族(bZIP TFs)、WRKY结构域转录因子家族(WRKY结构域TFs)和MYB转录因子家族(MYB TFs)。见于大麦中[24],这些转录因子均在种子萌发早期上调,说明它们的作用至关重要。相关研究表明MAPK的激活可以导致转录因子的磷酸化,进而激活基因的表达[64].在烟草中,MAPKs可诱导防御基因的表达[68].MAPKs接收激素等信号,通过MAPK级联反应介导转录因子,然后这些转录因子调节编码应激相关蛋白的防御基因对外界生物和非生物胁迫的反应。在目前的研究中,许多与防御基因相关的蛋白质和途径,如细胞壁合成、β葡聚糖酶pr蛋白、蛋白水解和次生代谢产物的表达在发芽早期逐渐上调,这可以保护发芽种子免受生物和非生物胁迫的损害(附加文件)9:图S4)。
结论
本研究采用转录组方法研究了中国面包小麦优良品种吉麦20种子萌发5个阶段的基因表达变化。微阵列分析使我们能够检测到大量与种子萌发相关的基因。小麦种子萌发过程包括三个不同的阶段,大量参与种子萌发的基因被划分为不同的功能群。MapMan和层次聚类分析显示了种子萌发过程中基因表达谱的变化。这些分析揭示了许多与种子萌发有关的重要代谢(如能量储备动员、TCA循环、氧化磷酸化、线粒体电子传递、光合作用和细胞壁代谢)和官能团(如细胞过程、激素和信号传递和运输)。相关基因的共同调控保证了种子的正常萌发。除了种子萌发过程中明显的整体变化外,KEGG和共表达网分析揭示了许多过滤后的显著差异表达基因的相互关系,从而发现了一些关键基因及其在种子萌发中的作用。qRT-PCR分析进一步验证了部分重要基因的表达模式。我们的转录组水平的结果为种子萌发的彻底代谢变化以及一些重要基因之间的关系提供了新的见解。
方法
植物材料和种子萌发
优质中华面包小麦品种(小麦l .)吉麦20,面筋品质优良,产量性能高34],在本研究中使用。选取大小和重量相近的种子作为试验材料。种子用蒸馏水冲洗三次,在黑暗中用蒸馏水吸收。12 HAI后室温孵育。在萌发过程中,研究了5个阶段的萌发,包括I组(种子不吸水)、II组(种子吸水12h)、III组(胚根穿透外胚根约24h)、IV组(胚根和胚根明显,约36h)和V组(胚根绿色,胚根可见,约48h)。在五个萌发阶段测试了1000粒种子重量的变化。称量前,用纸吸干种子10秒。采集样品进行形貌和扫描电子显微镜(SEM)观察,并固定在不同的固定液体中。收集的种子在RNA提取前保存在-80°C。采用微阵列杂交技术,对5个萌发期进行3个生物重复。
形貌和扫描电镜观察
固定样品用立体显微镜进行形态观察。用于扫描电镜观察,从FAA固定液(50%乙醇89 ml +冰醋酸6 ml +甲烷5 ml)中收集的种子与100 ml固定24小时以上。然后用一系列脱水溶液(95%乙醇、100%乙醇、75%乙醇+ 25%乙酸异戊酯、50%乙醇+ 50%乙酸异戊酯、25%乙醇+ 75%乙酸异戊酯、100%乙酸异戊酯,各处理10-20 min)处理固定材料。脱水后,在乙醇和乙酸异戊酯比例为3:1、1:1和1:3的混合物中分步处理15 min,最后在乙酸异戊酯中浸泡80 min。临界点干燥后,用扫描电镜S-4800 FESEM(日立,日本)观察。
RNA分离和微阵列杂交
总RNA采用预冷冻Trizol试剂(Invitrogen, Carlsbad, CA)根据制造商的说明进行了一些修改。RNA进一步用DNase处理以去除潜在的基因组DNA污染,并使用Agilent 2100生物分析仪(Agilent Technologies, Palo Alto, CA)评估完整性和浓度,260/280吸光比约为2.0。总rna用Oligo dT/T7引物孵育,反转录为双链cDNA。利用Affymetrix的IVT标记试剂盒对扩增的rna进行纯化和生物素标记。将生物素化的cdna片段化并与Affymetrix GeneChip®小麦基因组阵列(Affymetrix, Inc., Santa Clara, CA)杂交16小时。清洗染色后扫描记录结果。
数据处理和显著差异基因分析
使用microarray套件5.0版本(Affymetrix Inc.)分析微阵列成像数据,随后使用Spotfire (Spotfire, Somerville, MA)进行分析。原始数据(CEL文件)使用统计算法(MAS 5.0算法)在转录水平进行标准化。MAS 5.0算法使用Tukey的双权估计器来提供一个鲁棒的平均信号值,Wilcoxon的秩检验来计算一个显著性或p值,并对每个探测集进行检测调用。背景估计由最低2%的特征强度的加权平均值提供。错配探针用于调节完全匹配(PM)强度。使用修剪后的平均值,对特征级强度值进行线性缩放,是默认的,以使所有分析数组的平均值相等。MAS 5.0算法(也称为统计算法)独立分析每个阵列;因此,不能考虑单个探针特定的亲和性,与RMA或PLIER相比,检测实验和控制样本之间微小变化的能力降低。MAS 5.0算法的主要用途是获得关于阵列性能的快速报告,并在将最终阵列提交给多芯片分析方法之一(RMA, PLIER)之前识别任何明显的问题。
基因表达采用连通环设计,在两个时间点间进行比较分析。每个时间点进行三次微阵列,结果显示每个时间点重复之间的相关性大于0.98。为了准确估计每组基因的表达值,不同萌发阶段的差异表达探针集采用方差分析(ANOVA)过滤,并采用随机方差模型(RVM)进行校正[69- - - - - -71].基因表达值为每个时间点3个样本的RMA归一化基因信号的几何平均值。通过重复比较检验和置换检验1000次,估计错误发现率(FDR)来确定某些探针集是否确实显著;FDR旨在评估随机发生的特定样本的意义。差异表达的探针集由两个参数选择(p-value < 0.05, FDR < 0.05)。
STC (Series Test of Cluster)完全用Java实现。聚类算法首先选取了一组不同且具有代表性的时态表达谱。这些模型概要文件的选择与数据无关。然后,聚类算法将每个通过过滤条件的基因分配给由相关系数确定的与基因表达谱最接近的模型谱。由于选择的模型配置文件与数据无关,因此算法可以确定哪些配置文件具有统计上显著高于使用置换测试分配的基因数量。该测试确定了使用大量时间点排列的模型配置文件的基因分配。然后,它使用标准假设检验来确定哪些模型配置文件在时间点的真实顺序下具有明显更多的基因分配,与排列运行中分配给模型配置文件的平均数量相比。重要模型概要可以独立分析,也可以根据相似度分组,形成重要概要的集群[72- - - - - -74].
采用聚类3.0进行分层聚类分析。MapMan已被证明是绘制转录组数据、定义功能类别和执行时间过程分析以识别显著过度代表的功能组的有效工具。MapMan分析主要基于大麦参考文献[24].MapMan BIN是基于自动注释和手动注释推断的。
本研究中使用的微阵列数据已存入NCBI GEO数据库,可通过GEO系列登录号GSE49821 (http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE49821).
Signal-net分析
利用Java语言在KEGG数据库中构建信号网络图。如果有确凿的证据表明两个基因相互作用,则在它们之间分配一条相互作用边[75- - - - - -77].在网络中,节点主要是基因(蛋白质、化合物等)和表示节点之间关系类型的边;例如,活化或磷酸化。度定义为与其他网络基因的连接强度之和:.对于网络中的一个基因,一个基因的源基因数称为该基因的进度,一个基因的目标基因数称为其出度。对每个基因按照程度由高到低进行排序后,网络焦点代表程度值最高的基因。这些基因对整个网络的结构和其他基因之间的关系影响最大。
路径和路径网分析
同样,利用KEGG、Biocarta和Reatome数据库进行通路分析,找出差异表达基因的显著通路。尽管如此,双面费雪的精确检验x2采用检验对显著性途径进行分类,显著性阈值定义为p-value < 0.05, FDR < 0.05。FDR值用于纠正p值(78- - - - - -80].
Path-Net是差异表达基因显著通路之间的相互作用网络,是根据KEGG数据库中各通路之间的相互作用建立的,目的是直接系统地发现差异表达基因显著通路之间的相互作用。它可以总结差异表达基因的通路相互作用,并确定某一通路被激活的原因[79].
共表达网络的构造与拓扑属性
在生物过程中,基因共表达网络可以从功能基因关联构建,以识别基因相互作用。对于每一对基因,我们计算Pearson相关,并选择显著相关对来构建网络[81].在网络中,循环节点代表基因,两个节点之间的边代表基因之间的相互作用。网络结构分析的目的是找到连接最多相邻基因且k-core值或度值最高的核心调控因子(基因)。网络的k核是一个子网络,其中所有节点都连接到该子网络中至少k个其他基因。因此,k-核值的秩描述了基因关联关系的复杂程度。最大核阶被称为图的主核或最高k核[82].相同颜色的循环属于同一子图[83,84];此外,为了研究网络的各种特性,度中心性也是构建的网络中基因中心性的重要衡量标准,以确定相对重要性。度中心性定义为一个节点到另一个节点的链路编号。
实时定量PCR (qRT-PCR)
差异表达基因经qRT-PCR验证。RNA分离后,在含有0.5 μl AMV逆转录酶(Promega)、0.5 μl RNase inhibitor (Promega)、1 μl oligo dT引物、2 μl dNTP混合物、4 μl MgCl2 (25 mM)、2 μl 10×逆转录酶缓冲液和4 μl RNA样品的20 μl体积中合成第一链cDNA。反应混合物在42°C下孵育60分钟。
采用双标准曲线法检测基因表达水平。ADP-ribosylation factor作为内控,被鉴定为表达最稳定的基因之一[85].使用Primer 5.0设计基因特异性引物,通过RT-PCR产物熔融曲线检测其特异性。每个qRT-PCR反应在20 μl体积中进行,每次qRT-PCR反应含有10 μl 2 × SYBR Premix Ex Taq (TaKaRa)、2 μl 50倍稀释cDNA、每个基因特异性引物0.4 μl和7.2 μl ddH2O。PCR条件如下:95°C持续3 min, 95°C持续15秒,57°C持续15秒,72°C持续20秒,45个循环。每个样品重复3次。反应在CFX96实时PCR检测系统(Bio-Rad)中进行。所有数据均使用CFX管理软件(Bio-Rad)进行分析。
缩写
- 阿坝:
-
脱落酸
- 抗利尿激素:
-
乙醇脱氢酶
- ALDH:
-
醛脱氢酶
- 芭:
-
Brassinosteroids
- 费尔南多-阿隆索:
-
脂肪酸
- 罗斯福:
-
错误发现率
- 遗传算法:
-
Gibberelin
- 海:
-
吸胀后一小时
- HCA:
-
层次聚类分析
- IDH:
-
异柠檬酸脱氢酶
- KEGG:
-
京都基因与基因组百科全书
- MAPK:
-
丝裂原激活蛋白激酶
- PDC:
-
丙酮酸脱羧酶
- PDH:
-
丙酮酸脱氢酶
- PPFK:
-
磷酸果糖激酶
- PK:
-
丙酮酸激酶
- 存在:
-
实时定量逆转录PCR
- RMA:
-
鲁棒多芯片平均
- 数位视讯
-
随机方差模型
- 内存:
-
根顶端分生组织
- ROS:
-
活性氧
- 扫描电镜:
-
扫描电子显微镜
- 山姆:
-
茎顶分生组织
- 苏西:
-
蔗糖合酶
- STC):
-
聚类的系列检验
- TF:
-
转录因子
- UGPase:
-
UDP-glucose焦磷酸化酶。
参考文献
- 1.
Shewry PR:小麦。应用物理学报,2009,30(3):344 - 344。10.1093 / jxb / erp058。
- 2.
费尔德曼M:作物植物的进化。哈洛,英国:朗曼科技;1995:185 - 192。
- 3.
Bietz JA, Wall JS:小麦面筋亚基:脱癸基硫酸钠-聚丙烯酰胺凝胶电泳测定分子量。中国食品科学,1997,49:416-430。
- 4.
Singh NK, Donovan GR, Batey IL, MacRitchie F:超声和阻垢高效液相色谱在小麦粉蛋白质研究中的应用。I.在没有还原剂的情况下总蛋白质的溶解。中国食品科学,2000,27(3):357 - 357。
- 5.
郭刚,吕东,闫旭,Subburaj S,葛萍,李霞,胡艳,闫艳:面包小麦品种发育籽粒蛋白质组特征分析(小麦l .)。中国生物医学工程学报,2012,29(3):344 - 344。
- 6.
Bewley JD, Black M:种子:发育和萌发的生理学。纽约:充气出版社;1994.
- 7.
Bewley JD:种子萌发和休眠。植物科学学报,1997,26(3):357 - 357。10.1105 / tpc.9.7.1055。
- 8.
Botha FC, Potgieter GP, Botha AM:种子萌发过程中的呼吸代谢与基因表达。植物学报,2001,30(4):344 - 344。10.1007 / BF00024560。
- 9.
Bewley JD:种子萌发和幼苗生长过程中蛋白质和核酸的合成。14日版。柏林:斯普林格出版社;1982:559 - 591。
- 10.
王志刚,王志刚,王志刚:种子萌发过程中的生理生化研究。第二版。柏林:斯普林格出版社;1982.
- 11.
Gallardo K, Job C, Groot SPC, Puype M, Demol H, Vandekerckhove J, Job D:拟南芥种子萌发和启动的蛋白质组学分析。中国农业科学,2001,26(3):457 - 457。10.1104 / pp.126.2.835。
- 12.
Müller K, Job C, Belghazi M, Job D, Leubner-Metzger G:蛋白质组学揭示了水芹(Lepidium sativum L.)胚乳帽蛋白质组的组织特异性特征及其在种子萌发过程中激素诱导的变化。蛋白质组学,2010,10:406-416。10.1002 / pmic.200900548。
- 13.
Catusse J, Strub JM, Job C, Van Dorsselaer A, Job D:甜菜种子活力的蛋白质组特征及其组织特异性表达。自然科学进展,2008,29(4):344 - 344。10.1073 / pnas.0800585105。
- 14.
Boudet J, Buitink J, Hoekstra FA, Rogniaux H, Larré C, Satour P, Leprince O:植物胚根热稳定蛋白质组的比较分析Medicago truncatula萌发期的种子鉴定出胚胎发生后期丰富的与耐干燥性相关的蛋白质。植物科学进展,2006,30(4):344 - 344。10.1104 / pp.105.074039。
- 15.
Finnie C, Maeda K, Østergaard O, baker - jensen KS, Larsen J, Svensson B:大麦种子发育和萌发过程中蛋白质组的研究。生物化学学报,2004,32:517-519。10.1042 / BST0320517。
- 16.
付忠,金鑫,丁东,李勇,付忠,唐娟:玉米种子萌发过程杂种优势的蛋白质组学分析。蛋白质组学,2011,11:1462-1472。10.1002 / pmic.201000481。
- 17.
杨鹏,李霞,王霞,陈辉,陈峰,沈松:水稻蛋白质组学分析(栽培稻)种子萌发时。蛋白质组学,2007,7:3358-3368。10.1002 / pmic.200700207。
- 18.
Dure L, Waters L:长寿信使RNA:来自棉花种子萌发的证据。科学通报,2005,27(3):344 - 344。10.1126 / science.147.3656.410。
- 19.
Almonguera C, Jordano J:向日葵干籽贮藏低分子热休克蛋白和Lea mrna的发育与环境同步表达。植物化学学报,1997,19:781-792。10.1007 / BF00027074。
- 20.
石bashi N, Yamauchi D, Minamikawa T:大豆子叶中贮存的mRNA豇豆属unguiculata种子:为储存的mRNA克隆cDNA的核苷酸序列,并通过早熟萌发诱导其合成。植物化学学报,2000,19(4):457 - 457。10.1007 / BF00017724。
- 21.
杨晓明,李志刚,张晓明,等:蕨类植物早期胚胎中mRNA的表达Marsilea vestita:父系和母系血统。中国生物医学工程学报,2001,30:27-33。10.1002 / mrd.1080300104。
- 22.
Nakabayashi K,冈本M, Koshiba T, Kamiya Y, Nambara E:存储mRNA的全基因组分析拟南芥种子萌发:种子转录的表观遗传和遗传调控。植物学报,2005,41:697-709。10.1111 / j.1365 - 313 x.2005.02337.x。
- 23.
Potokina E, Sreenivasulu N, Altschmied L, Michalek W, Graner A:大麦种子萌发过程中的差异基因表达(大麦芽l .)。中国生物医学工程学报,2002,2:28-39。10.1007 / s10142 - 002 - 0050 - x。
- 24.
Sreenivasulu N, Usadel B, Winter A, Radchuk V, Scholz U, Stein U, Weschke W, Strickert M, Close TJ, Stitt M, Graner A, Wobus U:大麦籽粒成熟和发芽:新的MapMan/PageMan分析工具突出的代谢途径和调控网络的共性和差异。植物科学学报,2008,29(3):344 - 344。10.1104 / pp.107.111781。
- 25.
Howell KA, Narsai R, Carroll A, Ivanova A, Lohse M, Usadel B, Millar AH, Whelan J:水稻萌发过程中代谢和转录时间开关的定位突出了特定转录因子和RNA不稳定性在萌发过程中的作用。植物科学学报,2009,29(3):344 - 344。
- 26.
Jiménez-López S, Mancera-Martínez E, Donayre-Torres A, Rangel C, Uribe L, March S, Jiménez-Sánchez G, Sánchez de Jiménez E:玉米表达谱(玉米L.)萌发期胚胎轴:核糖体蛋白mrna的翻译调节。中国生物医学工程学报,2011,29(3):344 - 344。10.1093 /卡式肺囊虫肺炎/ pcr114。
- 27.
万云,Poole RL, Huttly AK, Toscano-Underwood C, Feeney K, Welham S, Gooding MJ, Mills C, Edwards KJ, Shewry PR, Mitchell RAC:六倍体小麦籽粒发育的转录组分析。中国生物医学工程学报,2008,29(4):344 - 344。
- 28.
陈志伟,陈志伟,陈志伟,等。小麦糊粉粉和淀粉胚乳的基因表达。中国生物工程学报,2012,30(4):366 - 366。10.1111 / j.1467-7652.2012.00705.x。
- 29.
Laudencia-Chingcuanco DL, Stamova BS, You FM, Lazo GR, Beckles DM, Anderson OD:基于cDNA微阵列的小麦颖果发育转录分析。中国生物医学工程学报,2007,29(3):344 - 344。10.1007 / s11103 - 006 - 9114 - y。
- 30.
Wilson ID, Barker GLA, Lu C, Coghill JA, Beswick WB, Lenton JR, Edwards KJ:六倍体冬小麦胚转录组的改变(小麦简历。麦花)在成熟和发芽期间。中国生物医学工程学报,2005,30(4):344 - 344。10.1007 / s10142 - 005 - 0137 - 2。
- 31.
Brenchley R, Spannagl M, Pfeifer M, Barker GLA, D 'Amore R, Allen AM, McKenzie N, Kramer M, Kerhornou A, Bolser D, Kay S, Waite D, Trick M, Bancroft I, Gu Y, Huo N, Luo MC, Sehgal S, Gill B, Kianian S, Anderson O, Kersey P, Dvorak J, McCombie WR, Hall A, Mayer KFX, Edwards KJ, Bevan MW, Hall N:利用全基因组散弹测序分析面包小麦基因组。自然科学进展,2012,29(4):344 - 344。10.1038 / nature11650。
- 32.
凌海海,赵升,刘东,王杰,孙辉,张超,范宏,李东,董林,陶勇,高超,吴红,李勇,崔勇,郭鑫,郑松,王波,于凯,梁强,杨伟,楼霞,陈杰,冯明,简杰,张欣,罗刚,姜艳,刘杰,王铮,沙勇,等:小麦a基因组祖基因草图小麦属植物urartu.自然科学进展,2013,37(3):344 - 344。10.1038 / nature11997。
- 33.
李贾J,赵年代,香港的X, Y,赵G,他W, appel R, Pfeifer M,道Y,张X, Jing R,张C,马Y,高,高C, Spannagl M,梅耶尔KFX,李D,锅年代,郑F,胡问,夏X,李J,梁问陈J,柳条T,郭台铭C,旷H, G,罗Y, B·凯勒,夏Q,陆P,王J,邹H,张R,徐J,高J,米德尔顿C,关丽珍Z,刘G,王J,杨H,刘X, Z,毛L,王J J,国际小麦基因组测序联盟:山羊草属tauschii基因组序列草图揭示了小麦适应的基因库。自然科学进展,2013,37(3):344 - 344。10.1038 / nature12028。
- 34.
罗娟,焦娟,任东,刘娟:高产优质小麦新品种吉麦20号。麦类作物,2006,26:159。
- 35.
Crismani W, Baumann U, Sutton T, Shirley N, Webster T, Spangenberg G, Langridge G, Able JA:六倍体面包小麦减数分裂和小孢子发生的芯片表达分析。BMC基因组学杂志,2006,7:267-10.1186/1471-2164-7-267。
- 36.
Usadel B, Nagel A, Thimm O, Redestig H, Blaesing OE, palacoos - rojas N, Selbig J, Hannemann J, Piques MC, Steinhauser D, Scheible WR, Gibon Y, Morcuende R, Weicht D, Meyer S, Stitt M:可视化工具MapMan的扩展,允许对数组进行统计分析,显示相应的基因,并与已知反应进行比较。中国生物医学工程学报,2005,29(4):344 - 344。10.1104 / pp.105.060459。
- 37.
Gass N, Glagotskaia T, Mellema S, Stuurman J, Barone M, Mandel T, Roessner-Tunali U, Kuhlemeier C:丙酮酸脱羧酶使矮牵牛花粉管生长具有竞争优势。中国生物医学工程学报,2005,27(3):344 - 344。10.1105 / tpc.105.033290。
- 38.
柯恩尼夫,李志强,李志强。种子休眠与萌发。植物学报,2002,5:33-36。
- 39.
谢晓兰,李志刚,李志刚,李志刚:番茄胚和胚乳的蛋白质组学分析。蛋白质组学,2005,5:3752-3764。10.1002 / pmic.200401209。
- 40.
Pritchard SL, Charlton WL, Baker A, Graham IA:拟南芥的萌发和贮藏储备动员是独立调控的。植物学报,2002,31(5):639-647。10.1046 / j.1365 - 313 x.2002.01376.x。
- 41.
李志刚,李志刚,李志刚。呼吸代谢:糖酵解、TCA循环与线粒体电子传递。植物学报,2004,7:254-261。
- 42.
Li J, Baroja-Fernández E, Bahaji A, Muñoz FJ, Ovecka M, Montero M, Sesma MT, Alonso-Casajús N, Almagro G, Sánchez-López AM, Hidalgo M, Zamarbide M, Pozueta-Romero J:玉米蔗糖合成酶活性增强导致淀粉和adp -葡萄糖水平升高(玉米l)种子胚乳。中国生物医学工程学报,2013,34(2):344 - 344。10.1093 /卡式肺囊虫肺炎/ pcs180。
- 43.
潘·D,斯特劳洛·李,纳尔逊·OE:许多玉米自交系缺乏胚乳细胞质磷酸葡萄糖转化酶。中国农业科学,2004,27(3):457 - 457。10.1104 / pp.93.4.1650。
- 44.
Nakai T, Konishi T, Zhang XQ, Chollet R, Tonouchi N, Tsuchida T, Yoshinaga F, Mori H, Sakai F, Hayashi T:绿豆蔗糖合酶对蔗糖表观亲和力的增加是由Ser的体外磷酸化或定向诱变引起的。植物生理学报,1998,29(3):344 - 344。10.1093 / oxfordjournals.pcp.a029339。
- 45.
Purcell PC, Smith AM, Halford NG:马铃薯中一种蔗糖非发酵1相关蛋白激酶序列的反义表达导致块茎中蔗糖合酶表达降低,叶片中蔗糖合酶转录物的蔗糖诱导性丧失。植物学报,1998,14(2):195-202。10.1046 / j.1365 - 313 x.1998.00108.x。
- 46.
Asano T, Kunieda N, Omura Y, Ibe H, Kawasaki T, Takano M, Sato M, Furuhashi H, Mujin T, Takaiwa F, Wu C, Tada Y, Satozawa T, Sakamoto M, Shimada H:水稻SPK,一种钙调蛋白样结构域蛋白激酶,在种子发育过程中贮藏产物的积累是必需的:蔗糖合酶的磷酸化是一个可能的因素。植物学报,2002,14:619-628。10.1105 / tpc.010454。
- 47.
Ciereszko I, Kleczkowski LA:葡萄糖和甘露糖通过己糖激酶依赖机制调节拟南芥中一种主要蔗糖合酶基因的表达。植物化学学报,2002,30(4):357 - 357。10.1016 / s0981 - 9428(02) 01452 - 3。
- 48.
Hardin SC, Winter H, Huber SC:玉米蔗糖合酶氨基端磷酸化与膜结合和酶活性的关系。中国农业科学,2004,27(4):344 - 344。10.1104 / pp.103.036780。
- 49.
Pucciariello C, paranti S, Banti V, Novi G, Perata P:缺氧条件下拟南芥活性氧物种驱动转录。中国生物医学工程学报,2012,29(4):344 - 344。10.1104 / pp.111.191122。
- 50.
陈凯,安玉春:大麦糊粉对赤霉素和脱落酸的转录应答。中国生物工程学报,2006,29(4):531 - 531。10.1111 / j.1744-7909.2006.00270.x。
- 51.
黄毅,朱涛,朱涛,李丽丽:玉米糊粉蛋白基因表达模式的研究矮人1突变的大米。植物科学学报,2006,30(4):344 - 344。10.1104 / pp.105.074435。
- 52.
谢敏,Tataw M, Reddy GV:对芽分生组织干细胞生态位中细胞生长动态的功能理解。中国生物医学工程学报,2009,20:1126-1133。10.1016 / j.semcdb.2009.09.014。
- 53.
金春春,刘玲,金建勇:干细胞维持和功能的信号网络拟南芥茎顶分生组织。植物学报,2007,50(3):274-281。10.1007 / BF03030655。
- 54.
Hanur VS:令人兴奋的植物干细胞研究进展。动物科学学报,2007,29(2):344 - 344。
- 55.
施晓峰,李志刚,李志刚:植物初级分生组织的功能与调控机制。植物学报,2010,13:53-58。10.1016 / j.pbi.2009.09.008。
- 56.
姜凯,Feldman LJ:根尖分生组织发育的调控。中国生物医学工程学报,2005,29(4):349 - 349。10.1146 / annurev.cellbio.21.122303.114753。
- 57.
黄志刚,李志刚,王志刚:被子植物茎干细胞生态位的表达模式本人水稻和玉米的同源基因暗示了单偶进化过程中的重大变化。分子生物学杂志,2006,23(12):2492-2504。10.1093 / molbev / msl125。
- 58.
Schulze S, Schäfer BN, Parizotto EA, Voinnet O,’S K: LOST分生组织基因调控拟南芥芽分生组织中央带子代细胞分化植物学报,2010,29(3):344 - 344。10.1111 / j.1365 - 313 x.2010.04359.x。
- 59.
Watanabe S, Hanaoka M, Ohba Y, Ono T, Ohnuma M, Yoshikawa H, Taketani S, Tanaka K:红藻铁螯合酶线粒体定位Cyanidioschyzon merolae.植物生理学报,2013,doi: 10.1093/pcp/pct077。
- 60.
杨晓明,杨晓明,杨晓明,杨晓明。大麦籽粒灌浆和籽粒成熟的蛋白质组学分析。植物科学学报,2002,29(3):366 - 366。10.1104 / pp.003681。
- 61.
芬克尔斯坦RR,甘帕拉SSL,岩石CD:脱落酸信号在种子和幼苗。植物学报,2002,14:S15-S45。
- 62.
Santner A, Calderon-Villalobos LIA, Estelle M:植物激素是植物生长的多种化学调节剂。中国生物医学工程学报。2009,5:301-307。10.1038 / nchembio.165。
- 63.
李文杰,李志刚,李志刚:植物激素在种子休眠释放和萌发过程中的作用。种子科学,2005,15:281-307。10.1079 / SSR2005218。
- 64.
Zhang S, Klessig DF: MAPK在植物防御信号中的级联作用。植物科学进展,2001,6(11):520-527。10.1016 / s1360 - 1385(01) 02103 - 3。
- 65.
张志刚,张志刚,张志刚,张志刚:植物丝裂原活化蛋白激酶信号级联。植物学报,2001,4:392-400。10.1016 / s1369 - 5266(00) 00191 - 6。
- 66.
Widmann C, Gibson S, Jarpe MB, Johnson GL:丝裂原活化蛋白激酶:从酵母到人的三激酶模块的守恒。自然科学进展,1999,29(3):344 - 344。
- 67.
Davis RJ: JNK组MAP激酶的信号转导。2000年细胞103:239 - 252。
- 68.
杨凯凯,刘颖,张松:丝裂原活化蛋白激酶通路的激活与烟草抗病性有关。自然科学进展,2001,29(4):344 - 344。10.1073 / pnas.98.2.741。
- 69.
Wright GW, Simon RM:在小微阵列实验中检测差异基因表达的随机方差模型。生物信息学,2003,19:2448-2455。10.1093 /生物信息学/ btg345。
- 70.
Yang H, Crawford N, Lukes L, Finney R, Lancaster M, Hunter KW:正常组织中存在转移预测特征。中华肿瘤杂志,2005,22:593-603。10.1007 / s10585 - 005 - 6244 - 6。
- 71.
陈峰,朱海辉,周丽芳,李娟,赵丽丽,吴世生,王娟,刘伟,陈震:cona诱导的暴发性肝炎早期晚期的Geseeanrche asrt循环:基于基因芯片的小鼠模型研究。中国生物医学工程学报,2010,30(4):344 - 344。
- 72.
赵伟,王玲,陈萍,赵娟,齐颖,迟松,孙娟,朱玲,程燕:HCV HVR1 7-aa肽模拟物通过降低关键促炎因子表达对大鼠肝损伤的保护作用。炎症过敏药物靶点,2010,9:135-145。10.2174 / 187152810791292845。
- 73.
李安,宋涛,王峰,刘东,范震,张超,何娟,王松:微型猪牙胚发育的microrname及其表达谱。公共科学学报,2012,7:e52256-10.1371/journal.pone.0052256。
- 74.
霍瑞麟,梁小斌,李斌,梁俊涛,何勇,贾玉军,贾军,龚小玲,余峰,王小明:100 Hz电针治疗6-羟多巴胺诱导帕金森病模型皮层和纹状体基因表达谱。基于Evid的Compl Alt, 2012, 2012: 14。
- 75.
李超,李华:基因组数据分析的网络约束正则化和变量选择。生物信息学,2008,24:1175-1182。10.1093 /生物信息学/ btn081。
- 76.
魏智,李华:基于网络的基因组数据分析的马尔可夫随机场模型。生物信息学,2007,23:1537-1544。10.1093 /生物信息学/ btm129。
- 77.
张建东,魏曼,张建东:KEGG图:R和生物导体中KEGG通路的一种图形方法。生物信息学,2009,25:1470-1471。10.1093 /生物信息学/ btp167。
- 78.
Kanehisa M, Goto S, Kawashima S, Okuno Y, Hattori M:破译基因组的KEGG资源。中国生物医学工程学报,2004,32:D277-D280。10.1093 / nar / gkh063。
- 79.
Yi M, Horton JD, Cohen JC, Hobbs HH, Stephens RM: WholePathwayScope:用于高通量数据的基于路径的综合分析工具。中国生物医学工程学报,2006,30(4):344 - 344。
- 80.
张志刚,张志刚,张志刚,张志刚。基于神经通路水平分析的系统生物学方法。基因组学报,2007,17:1537-1545。10.1101 / gr.6202607。
- 81.
Vermeirssen V, Barrasa MI, Hidalgo CA, Babon JAB, Sequerra R, doucte - stamm L, Barabási AL, Walhout AJM:一个以基因为中心的秀丽隐杆线虫核心神经元蛋白- dna相互作用网络中的转录因子模块化。基因组学报,2007,17:1061-1071。10.1101 / gr.6148107。
- 82.
Huber W, Carey VJ, Long L, Falcon S, Gentleman R:分子生物学中的图。中国生物医学工程学报,2007,28(增刊6):S8-10.1186/1471-2105-8-S6-S8。
- 83.
王晓峰,王晓峰,王晓峰,王晓峰,Barabási AL:代谢网络模块化的分层组织。科学通报,2002,29 (3):377 - 377 / ei。
- 84.
Barabási AL, oltai ZN:网络生物学:了解细胞的功能组织。中国科学院学报,2004,(5):344 - 344。10.1038 / nrg1272。
- 85.
Paolacci AR, Tanzarella OA, Porceddu E, Ciaffi M:小麦定量RT-PCR归一化内参基因的鉴定与验证。生物化学学报,2009,10:11-10.1186/1471-2199-10-11。
确认
作者非常感谢堪萨斯州立大学研究与推广学院通讯与农业教育系的Sarah Hancock女士对本文进行的语言编辑。本研究得到国家自然科学基金(31271703,31101145)、科技部中澳合作项目(2013DFG30530)、中国转基因作物国家重点项目(2011ZX08009-003-004)的资助。这是14-088-J号来自堪萨斯农业实验站。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
这份手稿没有经济或非经济上的竞争利益。
作者的贡献
YY和GG进行了所有实验和数据分析。LD和HY分别进行RNA、cDNA制备、qRT-PCR和生物信息学分析。JL帮助英文写作和校对手稿。LX和YY构思了这项研究,计划了实验,并帮助起草了手稿。所有作者都阅读并批准了最终的手稿。
于永龙、郭广芳、吕东文对这项工作有同样的贡献。
电子辅助材料
12870 _2013_1756_moesm1_esm.jpeg
补充资料1:图S1: a型淀粉粒在种子萌发5个阶段的直径变化横轴为种子萌发期,纵轴为种子直径变化。(jpeg32 kb)
12870 _2013_1756_moesm2_esm.zip
附加文件2:表S1:从三个基于独立生长的植物材料的实验系列中获得的归一化表达值的完整列表。表S2。Affymetrix GeneChip®小麦基因组阵列的MapMan功能类(bin和subbin)概述表S3。从归一化表达值的整个列表中获得的差异表达基因的完整列表p-value和FDR。(zip 19mb)
12870 _2013_1756_moesm3_esm.tiff
附加文件3:图S2: MapMan代谢概览图显示了种子萌发期间转录水平的差异(12与24 HAI和36与48 HAI)(更多详细信息,参见图中的图例)5).(tiff格式4mb)
12870 _2013_1756_moesm4_esm.xls
附加文件4:表S4:构建信号网络的基因列表。表S5。以小麦的差异基因或EST序列为基础,建立稻瘟病后水稻基因序列的完整列表。(xls1mb)
12870 _2013_1756_moesm9_esm.jpeg
附加文件9:图S4: Mapman显示了经过一些修饰后的12 HAI和24 HAI之间种子萌发过程中外部环境调控和响应相关基因的表达。(jpeg101kb)
12870 _2013_1756_moesm10_esm.pdf
附加文件10:图S5: Mapman显示了种子萌发过程中0和48 HAI转录水平差异的调控概述图。在对数色标度上,蓝色表示转录本下调,红色表示转录本上调。(pdf 1mb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是原创作品的名称要注明出处。创作共用公共领域奉献弃权书(https://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
于洋,郭国强,吕,D。et al。面包小麦优良品种吉麦20种子萌发过程转录组分析。BMC植物生物学14,20(2014)。https://doi.org/10.1186/1471-2229-14-20
收到了:
接受:
发表:
关键字
- 面包小麦
- 种子发芽
- 转录组
- 中存在