摘要
背景
植物是一种无根的生物,它们以良好的调节方式处理它们的——有时是不利的——环境。涉及SWI/ snf2型atp酶的染色质重塑被认为是调控不同发育程序中基因表达的重要表观遗传机制,并将这些程序与对环境信号的反应结合起来。在这项研究中,我们报道了染色质重塑在拟南芥中与生长和基因表达变异性有关的环境条件的作用。
结果
已经适度(2倍)的过度表达AtCHR23与野生型相比,拟南芥中的atp酶基因导致总体生长降低。详细分析表明,在根中,生长的减少是由于细胞伸长的减少。生长减少表型需要充足的光照,并通过施加非生物(盐、渗透)胁迫而放大。相反,基因敲除突变AtCHR23不会导致这种可见的表型效应。此外,我们还展示了AtCHR23增加基因相同植物种群生长的可变性。这些数据表明AtCHR23有助于增长的稳定性或稳健性。详细的RNAseq分析表明AtCHR23过度表达和基因表达的变异在与环境压力相关的基因子集中增加。通过独立的qRT-PCR分析,证实了单株中基因表达的较大变异。
结论
表达的AtCHR23使拟南芥具有明显不同于生长停滞表型的表型AtCHR12的平行AtCHR23,以应对非生物应激。这证明了拟南芥中高度相似的atp酶的功能亚特化。表达的AtCHR23增加基因相同个体之间的生长变异性,其方式与与环境压力相关的特定基因子集的表达变异性增加相一致。我们认为atchr23介导的染色质重塑是植物缓冲系统的一个潜在组成部分,可以保护植物免受环境诱导的表型和转录变异。
背景
植物已经进化出精细的协调机制来调节其生长,以响应环境,作为其无根生活方式的一个程序化部分。这些机制帮助它们在生存的任何时期应对(可能不利的)环境。值得注意的是,发育中的幼苗易受短期不利环境的影响[1,2]。因此,植物表现出很大的生长变异性,这种现象也被称为生长可塑性[3.]。这种可塑性使植物能够根据当时的环境条件优化其生长和发育,确保以最佳策略完成其生命周期和繁殖。生长可塑性在不利环境下影响产量和质量,对农艺应用具有潜在的重要意义。随着生长,性状的可塑性很大程度上是在分子水平上组织起来的,其中表观遗传机制起着关键作用[3.]。染色质重塑是表观遗传机制的一部分,仅次于DNA甲基化、组蛋白修饰和基于小rna的机制[4],这是整个植物发育的一个组成部分,与植物对生物[5]和非生物胁迫[6]。
我们之前已经证明,由AtCHR12是否参与了生长的调控拟南芥当感知到非生物压力,如干旱或高温时[7]。拟南芥植株过表达AtCHR12在胁迫下,正常活跃的初芽生长停滞,初茎生长减少。在没有压力的情况下,它们与野生型无法区分。生长停滞反应取决于施加的压力的严重程度。另一种SWI/ snf2型atp酶,伸展开的(悉德),被证明是抵抗坏死性病原体所必需的葡萄孢菌[5],然而击倒AtDRD1ATPase基因对真菌病原体的易感性增加Plectosphaerella cucumerina[8]。SWI/ snf2型atp酶被认为介导染色质重塑和参与DNA和组蛋白修饰的酶之间的复杂相互作用。这强调了atp依赖性染色质重塑在植物对环境胁迫反应中的重要性。
此外,这种染色质修饰在发育过程中起调节作用[9建立具有在时间和空间上受到严格调控的表达模式的表观遗传状态。在动物中,这种表观遗传状态是在发育早期确定的,而在植物中,表观遗传机制也在胚胎发育后起作用[10]。几个染色质重塑atp酶基因在植物发育中起作用。chd3亚家族atp酶泡菜(PKL)在胚胎发生、种子萌发和根系发育过程中选择性地调控一系列基因[11- - - - - -13]。近年来,该基因被鉴定为光形态发生的负调控因子[14]。拟南芥atp酶SWI/ snf2亚家族的四个基因中[15],悉德和BRM参与各种部分重叠的发育过程,如根和花的发育或种子的成熟[16- - - - - -18]。这个亚家族的另外两个成员,AtCHR12和AtCHR23,在胚胎和胚乳发育中起作用。几乎致命的atchr12/atchr23含有弱等位基因的双突变体表现出多种严重的多效性形态缺陷,包括茎和根分生组织维持不良[19]。这种atp酶介导的染色质修饰建立了一种水平的基因调控,可能将发育程序与对环境信号的反应结合起来。
据认为,表观遗传修饰有助于建立对环境扰动的缓冲[20.这导致了有机体的表型稳健性。无论是在果蝇[21]和酵母[22- - - - - -24]染色质调节基因的缺失显著增加了所研究表型的可变性,表明适当的染色质修饰可以抵消遗传、环境和/或随机扰动[25,26]。
我们在此报告过表达的显著影响AtCHR23基因在拟南芥生长、逆境反应和全基因组表达水平方面对表型的影响。AtCHR23是平行的吗AtCHR12[27]其中过表达的影响在前面已经介绍过了[7]。表达的AtCHR23结果与野生型拟南芥相比生长降低,但表型细节之间存在差异AtCHR12和AtCHR23过度表达有明显的不同,表现出这两种类比的次专门化。的影响AtCHR23在生长表型和基因表达方面,过表达都是显著的定量。过度表达AtCHR23增加相同植物群体中基因亚群的生长变异和表达变异。强调了染色质修饰在植物基因表达调控中的重要作用。基于这些结果,我们提出了精确和有控制地表达AtCHR23是稳定或稳健增长所必需的。我们提出atchr23介导的染色质重塑可能是植物缓冲系统的一部分,可以保护植物免受环境诱导的表型和转录变异[20.]。
结果
构建拟南芥突变体AtCHR23表达式
产生过表达该基因的转基因拟南芥系AtCHR23基因是一个包含35S CaMV启动子的结构体AtCHR23(包括5 ' -UTR)从加入Columbia(附加文件)1:图S1)用于野生型拟南芥(Col-0)的转化。对2个单拷贝转基因株系进行了鉴定和分析:AtCHR234 ov和AtCHR235机汇。此外,转基因系过表达的cDNA拷贝AtCHR23在框架内融合到绿色荧光蛋白前面35S CaMV启动子下的基因(附加文件)1:图S1)生成。鉴定并分析了两个独立的单拷贝转基因系:G_AtCHR231 ov和G_AtCHR233机汇。用cDNA拷贝转化获得了第三种过表达转基因株系AtCHR23包括5 ' -UTR融合在框架绿色荧光蛋白由本地驱动AtCHR23-promoter(附加文件1:图S1)。作为比较,两条功能缺失的T-DNA插入线影响AtCHR23从拟南芥砧木中心获得表达。两个敲除系均未显示全长表达AtCHR23成绩单。本文提供的数据来自SALK_057856,在本文的其余部分将其指定为atchr23。另一条插入线给出了类似的结果(数据未显示)。
表达的AtCHR23减少根的生长,增加表型变异
基因敲除菌幼苗的生长动态atchr23)和过度表达AtCHR23在使用垂直琼脂板的根伸长试验的帮助下进行了分析[7]。野生型和突变型植物的分层种子萌发时间和频率大致相同。在不同的环境条件下,反复测量其发育过程中主根和下胚轴的长度以及其他表型特征。防止上一代所经历的环境可能造成的混杂影响[28],所有的比较都是使用在同一时间和同一环境中生长的亲本植物(包括野生型和突变型)的种子进行的。每个条件至少有40个根,最多16个根(8个突变体;每个琼脂板8个野生型),每个实验5个琼脂板。
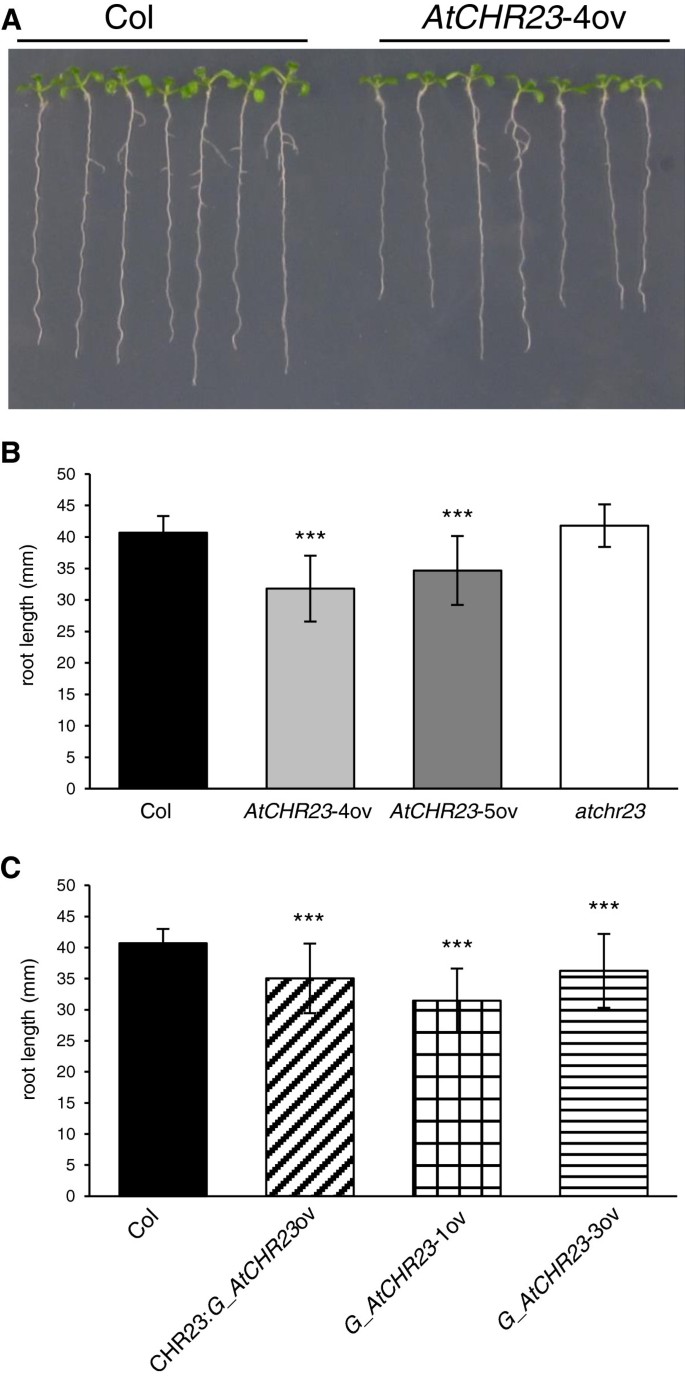
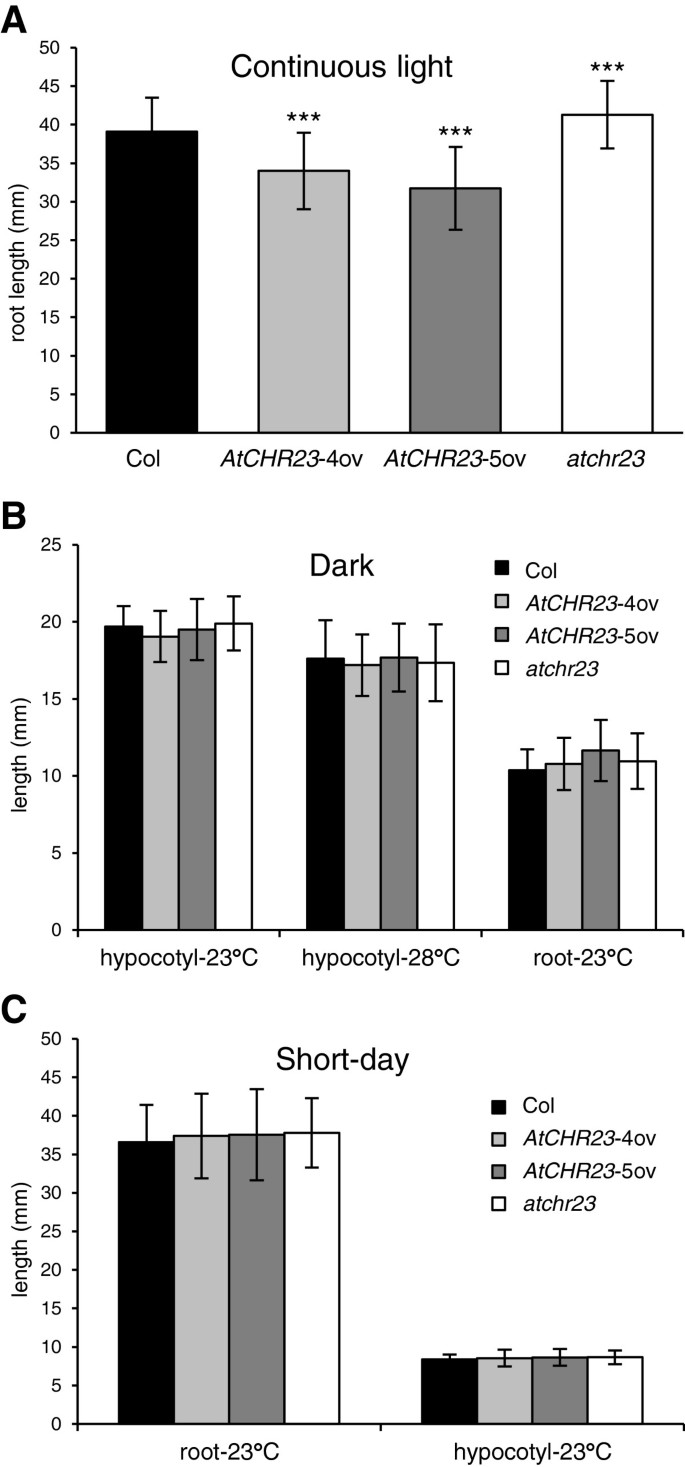
观察到不同线条之间明显的差异,特别是在根的长度方面(图2)1A)根长度的差异取决于所施加的环境条件。当在23°C的长时间条件下生长时,这两种植物的根AtCHR23-ov突变体比哥伦比亚野生型短得多(图2)1A和B)。数据汇总见表1。野生型8日龄幼苗的平均根长为40.7 mm,野生型幼苗的平均根长为40.7 mmAtCHR23- 40v苗期为31.9 mm(减少21.6%)AtCHR23- 5v 34.6 mm(减少14.9%)。同时也上调了AtCHR23带有基因的cDNA拷贝(两个)G_AtCHR23-ov系)的幼苗根系比野生型短14%,比野生型短22.7%,而具有本地启动子的转基因系的根系比野生型短11%(图2)1C;表格1)。在这些试验中,根长变化相当大,过表达系的变异系数(CV)范围为0.161至0.164,而野生型的变异系数为0.052(表2)1)。过表达突变体的变异显著高于野生型(Levene’s test;表格1)。这些数据表明,当过表达时AtCHR23,根不仅明显变短,而且更加多变和不均匀。相反,基因敲除突变体atchr23其根只比野生型稍长(图1)1B).在40株幼苗的种群中,这种差异无统计学意义。这些根的生长差异各不相同AtCHR23突变体和野生型在不同生长条件下,温室或生长室中生产的几种种子中一致观察到。此外,在18°C和25°C生长的幼苗中观察到类似的根长差异和变异模式(数据未显示)。
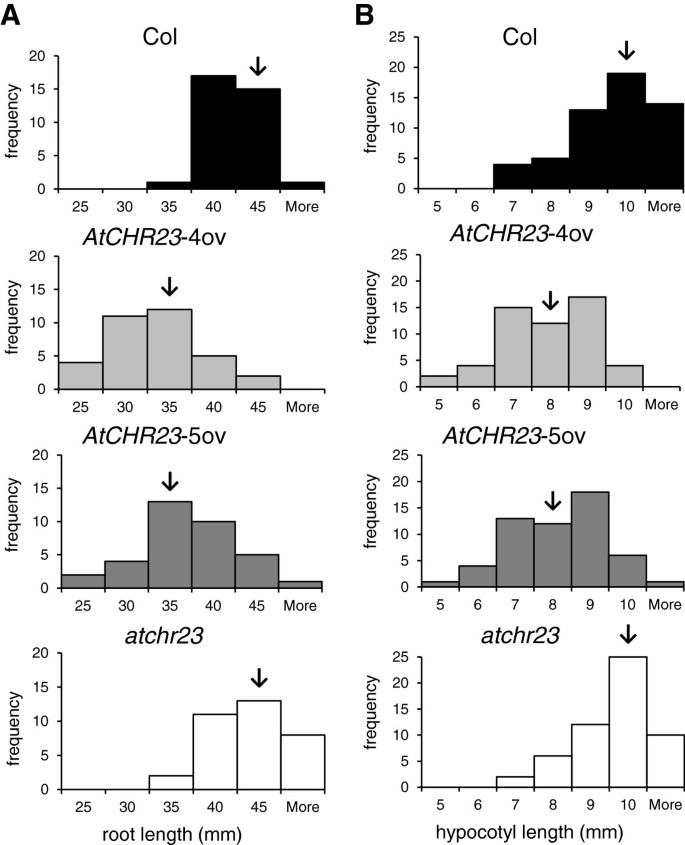
通过分析长度数据的频率分布,更详细地评估了表型分析中的变异性(图2)2)。根长度的频率分布表明,根长度的分布向更短的根偏移AtCHR23过度表达(图2A),但仍有相当多的个别幼苗的根与野生型一样长(图2)2A,中间两个面板)。对于下胚轴长度的分布,过表达幼苗群体的差异也大于野生型(图2)2B,中间两个面板)。鉴于所有在表型分析中标准化环境的实验努力,我们认为过表达系个体之间的差异可能具有分子和/或功能基础。
将生长停滞表型与AtCHR23mRNA的量AtCHR23利用qRT-PCR技术对8株幼苗的mRNA进行检测。定量结果总结于表中1。增加了两倍AtCHR23在CHR23中观察到mRNA(与野生型相比):G_AtCHR23机汇。这显然足以使生长停滞表型被检测到。较高水平的mRNA倾向于使表型更加明显,然而,上调水平与根的长度之间没有明确的相关性。这种关联表明稳态mRNA水平与根长度表型外显率之间存在复杂的相互作用。根长与土壤养分水平之间缺乏相关性AtCHR23在野生型和突变型的单个幼苗(各10个幼苗)中也证实了表达(数据未显示)。
根生长的减少是由于细胞伸长的减少
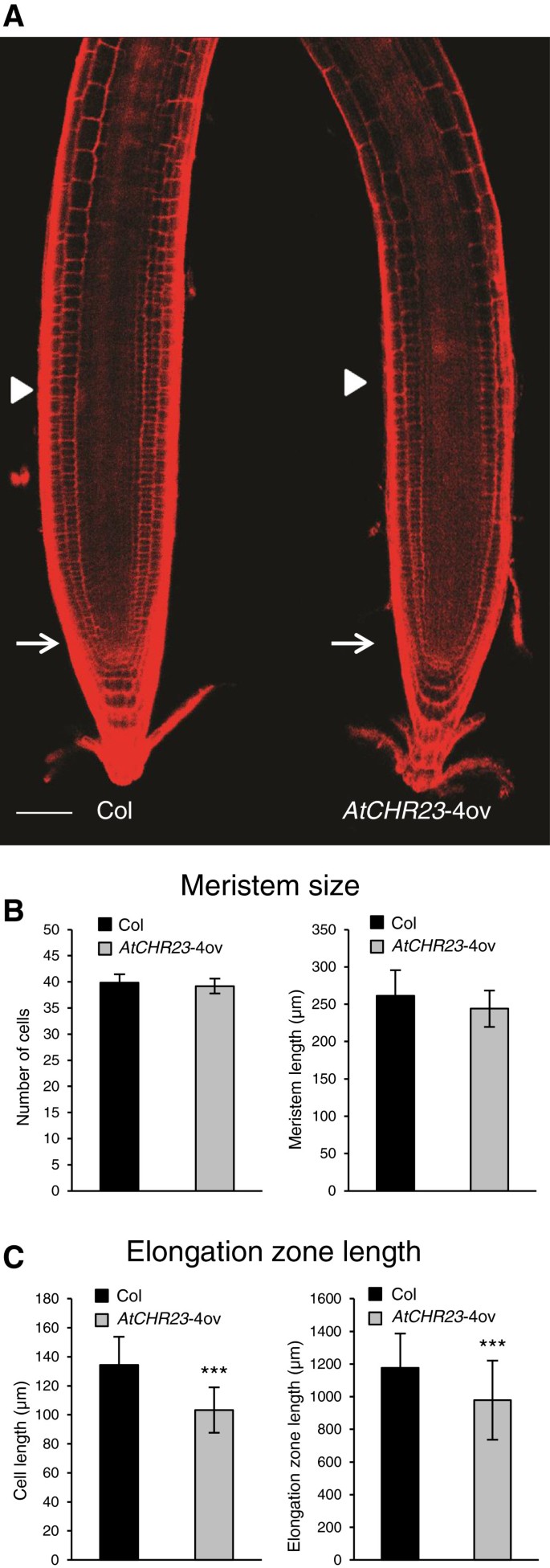
为了确定根长减少是由于细胞分裂减少还是细胞伸长减少,我们分析了6 d龄幼苗的分生组织和伸长区大小。AtCHR23与野生型相比,- 40v的根表现出正常的细胞模式(图2)3.A).对于分生组织,我们测量了分生组织带的长度和分生组织皮层细胞的数量。在野生型和突变根之间没有任何差异(图2)3.B).为了进一步评估细胞分裂的作用,我们还使用了细胞G2-M期周期标记pCYCB1;1:CYCB1;1- gus [29]。图案上没有明显的区别(附加文件)1:图S2),在野生型和过表达突变体之间观察到gus阳性细胞的数量(数据未显示)。这与野生型和突变型的分生组织大小一致(图2)3.B)。另一方面,与野生型相比,突变体的伸长区明显缩短(16.8%),完全伸长的细胞长度缩短(23.1%)(图2)3.综上所述,这些结果表明……的主要影响是AtCHR23根部的上调是细胞伸长的减少。
表达的AtCHR23导致幼苗和植株更小
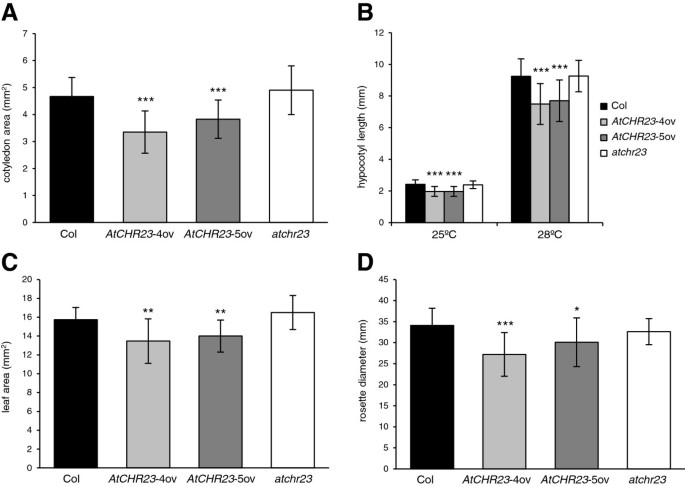
二的分析AtCHR23-ov行演示了AtCHR23也导致幼苗和植株生长总体下降(图2)4)。过表达系子叶生长减少(图2)4A)和下胚轴(图4B).子叶平均面积由4.67 mm减少2在野生型为3.35毫米2在AtCHR23- 4v(减少28.3%)至3.83 mm2在AtCHR23- 50伏(减少18%)下胚轴的长度由25°C或28°C生长的幼苗测定。后一种温度可诱导下胚轴伸长[30.]。过表达系25°c生长8 d苗的平均下胚轴长度比野生型的2.42 mm减少了1.97 mm(约减少20%),而敲除系的下胚轴长度与野生型的差异不显著。这种差异在28℃时更为明显(图1)4B)AtCHR23导致幼苗生长的总体显著减少。突变体子叶和下胚轴的生长变异性增加不显著(Levene检验;额外的文件2:表1)。
以确定是否和如何影响植物的大小由于AtCHR23过表达在发育过程中产生表型变化,测定了土生植物营养生长的两个参数:叶面积和莲座直径。这两个参数都是根据15种土壤种植植物的数字图像确定的。野生型第一片莲座叶的平均表面积为15.7 mm2。这被减少到13.5毫米2在AtCHR23- 40毫米和14.0毫米2在AtCHR23- 5v,因此过表达系的叶片比野生型小15%(图2)4C)。敲除系的叶片略大(5%),但在选择的实验设置中,这种差异再次没有统计学意义。在第三个莲座叶上也观察到类似的生长差异(数据未显示)。与野生型相比,过表达突变体的叶片也表现出显著增加的生长变异性(Levene’s test;额外的文件2:表1)。此外,4周龄过表达突变体的平均莲座直径减小(图2)4D)。野生型莲座直径为34.1 mm,而野生型莲座直径为27.2 mmAtCHR23- 40v和30.1 mm inAtCHR235机汇。与野生型相比,突变体的莲座大小分别减少了20%和12%。这也表明植物在营养发育过程中过度表达AtCHR23往往比野生型要小。
光照条件决定过表达系的生长特性
由于光是影响植物生长的重要环境因素[31],我们评估了各种的生长动态AtCHR23不同光照条件下的表达变异。在连续的光中,一切AtCHR23突变体证实了上述长日照条件下根系长度的模式。相对于野生型,过表达系的根长度明显减少,而敲除系的根往往(在这种情况下确实明显)更长(图2)5A)。然而,在黑暗中,所有品系的根长与野生型没有显著差异(图2)5B).在黑暗中,根的生长明显减少[32,33],而下胚轴比光照下更长(黄化)[34]。因此,在这种环境下进一步减少根长度是不可靠的。然而,在23°C或28°C的黑暗中生长的幼苗的下胚轴长度(图2)5B)与野生型没有什么不同。同样在短日照条件下(在23°C下,10天8小时光照/16小时黑暗;数字5C),突变体的根和下胚轴的长度都无法与野生型区分。在黑暗和白天较短的环境中缺乏这种表型的一个可能原因可能是基因的不稳定性AtCHR23信使rna表达。的定量表达分析AtCHR23在黑暗和短日照条件下,与野生型相比,生长的幼苗证实了与长日照条件下相同的上调水平(数据未显示)。因此,在黑暗和短日照生长突变体中缺乏表型不能用降低水平来解释AtCHR23过表达。结果表明,光对改性的效果有明显的影响AtCHR23表达对拟南芥幼苗生长动态的影响:需要足够(量)的光来建立AtCHR23介导的生长表型。
非生物的压力放大的影响AtCHR23过表达
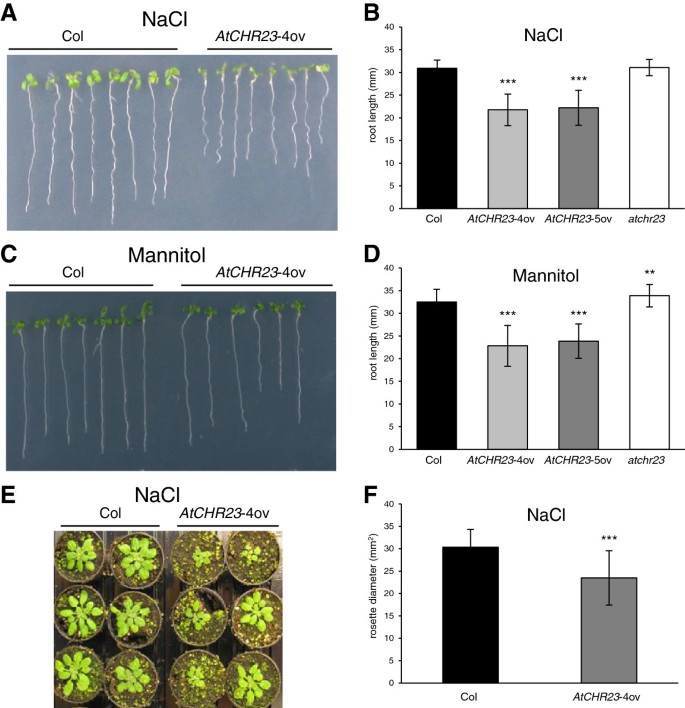
修改后的影响AtCHR23这种表达在环境应激中也很明显。在非生物胁迫条件下,在含75 mM NaCl(盐胁迫;数字6A)或200mm甘露醇(渗透胁迫;数字6C).正如预期的那样,两种胁迫对根的生长都有明显的负面影响。盐胁迫环境下野生型幼苗根的平均长度为30.92 mm(图2)6B),在渗透胁迫下32.51 mm(图6D),而在没有这种应力的情况下,长度为40.7 mm(见表1和图1)。结果表明,盐胁迫使野生型根长减少24%,渗透胁迫使野生型根长减少20%。过度表达的突变体AtCHR234 ov和AtCHR23- 5v对盐的响应分别使根长减少32%和36%(图2)6B)。在渗透胁迫下,这种减少分别为29%和31%(图2)6D).过表达的细胞系也得到了类似的结果AtCHR23cDNA拷贝(附加文件1:图S3)。相比之下,淘汰线atchr23比野生型的根略长,但仅在渗透胁迫下(平均长度33.9 mm;数字6D,这些数据表明AtCHR23过表达系对胁迫条件的反应是根长比野生型更强的生长阻滞。非参数因子分析显示,基因型和胁迫处理对根长均有极显著(P < 0.001)的影响,除基因敲除系外,所有突变系中基因型X处理互作对根长均有极显著(P < 0.01)的影响2表2)。在进一步的营养发育中也观察到同样的情况。在3天内用100 mM NaCl对2周龄植株进行2次盐胁迫后,土生植株的莲座直径(图26E)被测量。无胁迫野生型莲座直径为34.1 mm2应力后为30.34 mm2,即降价11%。的AtCHR23- 40v植株对盐胁迫的反应是,莲座直径减少了两倍(22%),从30.1毫米减少2至23.49毫米2(图46 D, f)。非参数因子分析显示基因型和处理对莲座直径的影响均极显著(P < 0.001),而基因型X处理交互作用的影响不显著(附加文件)2表2)。结果表明,非生物胁迫放大了AtCHR23过表达对幼苗生长有影响,且影响延伸到苗期以后。
非生物胁迫强调在…情况下生长的减少AtCHR23过表达。(一)8日龄幼苗在23℃长日条件下在添加75 mM NaCl的培养基上生长的照片。(B)75 mM NaCl处理下8日龄幼苗主根的平均(±SD)长度。(C)8日龄幼苗在23°C长日条件下在添加200 mM甘露醇的培养基上生长的照片。(D)在200 mM甘露醇上生长的8日龄幼苗主根的平均(±SD)长度。(E)4周大的野生型和AtCHR23在施用盐胁迫两周后- 40株。(F)施用盐胁迫2周后4周龄植株蔷薇丛直径平均值(±SD)。在每个试验和品系中,测量了40株幼苗或15株植株。星号表示与野生型的显著差异:**, p < 0.01;***, p < 0.001。
全基因组RNAseq分析显示基因表达变异性增加AtCHR23过表达
的生长表型AtCHR23通过RNA测序评估过表达。2个8日龄鱼苗的生物重复AtCHR23 -在23°C条件下生长的长日(生长表型减少)和短日(没有生长表型减少)光周期对4ov和野生型(哥伦比亚)进行了评价。对于这8个样本中的每一个,都产生了超过6000万的读取。考虑到实验设置,相对于所有其他样品,在长日照条件下的过表达系之间,与生长表型降低相关的表达差异是预期的。
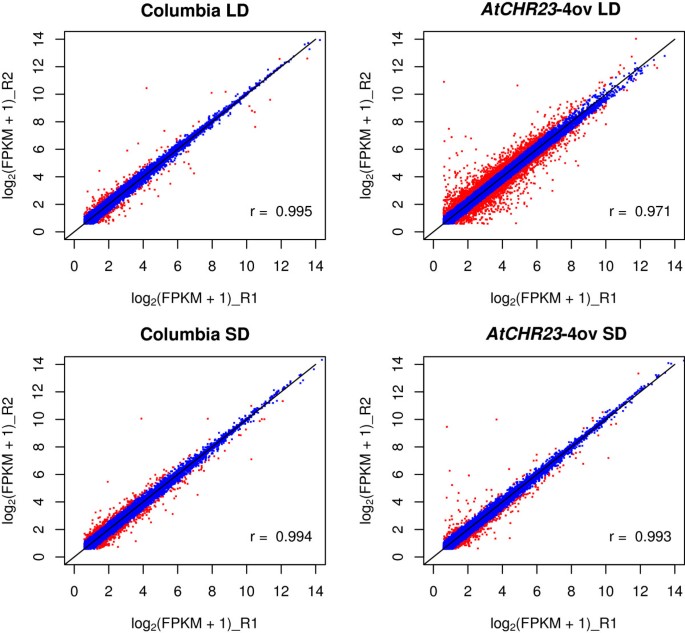
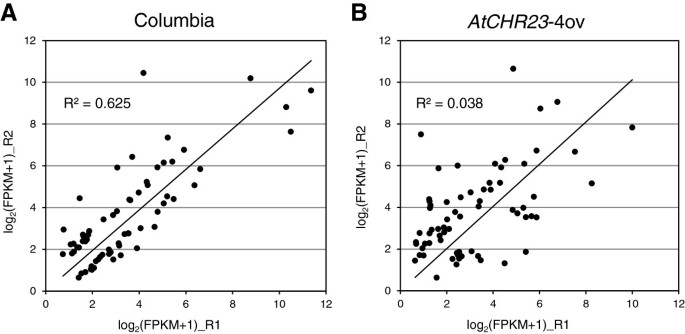
应用DESeq进行差异表达分析[35]或cuffdiff [36]生成了潜在差异表达(DE)基因列表。然而,在其他的生物复制中,其中许多不能得到证实。从96个经DESeq鉴定为长日突变体潜在DE的基因中(附加文件)3.),通过qRT-PCR分析了24个基因,其中7个基因被确认为差异表达(占检测基因的33.3%)。我们的结论是,已鉴定的DE基因无法进行生物学验证。因此,进一步的分析集中在基因表达的明显变化上。将每个样本以重复R1和R2的每百万映射读数(FPKM)的转录本(外显子模型)每千碱基的片段数总和表示的表达值进行比较,Pearson相关系数大于0.99(图2)7),除了存在生长表型的唯一样本:AtCHR23在长时间工作条件下过度表达。在这种情况下,数据离最佳拟合线更加分散,Pearson相关系数刚刚超过0.97(图1)7)。为了评估长日突变体中更大的重复间表达变异性,我们计算了所有基因的对数之间的绝对差异2(FPKM + 1)在两个重复中的表达水平。在野生型(195个基因)中,前1%的基因表现出较大的表达差异作为变异性的截止值,并用于选择变异性高于指定截止值的所有其他样本中基因的数量(和身份)。这一阈值相当于正常尺度上约1.5倍的表达差异。在全基因组基因表达的散点图中,这些基因用红色表示(图2)7)。
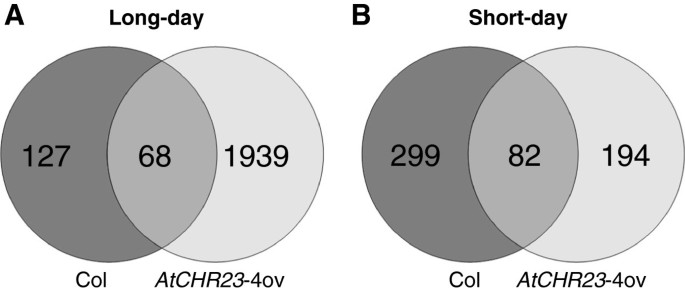
在长时间工作的情况下AtCHR23过表达突变体有不少于2007个变异较大的基因(图2)8其中,68个基因在野生型中也是可变的(图2)8;额外的文件4)。这表明AtCHR23与野生型相比,过度表达增加了相当一部分基因的表达变异性。相比之下,在短日照条件下,在野生型中鉴定出381个基因是可变的,而在突变系中鉴定出276个基因,其中82个基因是共享的(图2)8B;额外的文件4)。因此,较大的可变基因亚群与较高的过表达有关AtCHR23在长日照条件下观察。这可能指向两者之间的因果关系AtCHR23基因表达的过度表达和变异性增加。野生型和突变型之间共有的68个长日可变基因在两个重复之间相关性较小AtCHR23过表达突变体(R2= 0.038)相对于野生型(R2= 0.625)(图9)。这表明在自然条件下已经嘈杂的基因(野生型)的表达在自然条件下变得更加嘈杂AtCHR23是过度的。
评价基因表达变异较大的基因的功能AtCHR23是过度,进行基因本体(GO)分析。为此,选择了298个基因子集(来自2007年),这些基因在两个生物重复之间具有至少3倍的表达差异。使用Classification SuperViewer对基因进行分类[37代表人数过多或不足。主要结果总结在附加文件中1:图S4。除了其他生物过程外,过度代表的生物过程子类别还包括对压力的反应,压力刺激和发育过程。这与上述表型观察结果很好地吻合。
AtCHR23过度表达扩大了特定基因亚群在个体间的基因表达差异
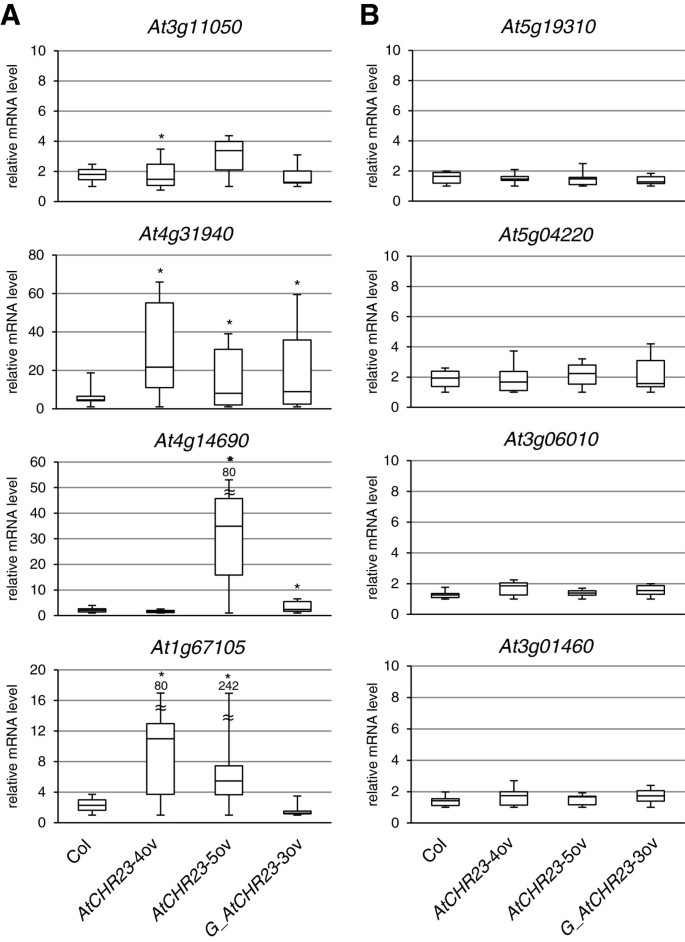
为了解决变异对单个幼苗基因表达的影响,我们选择了8个基因进行额外的分析。从基因列表中随机选择4个基因AtCHR23 -在长日照条件下,有40个可变基因。此外,随机选择4个被确定为非变量的基因(包括AtCHR23/At5g19310)。这些基因的细节在附加文件中给出2:表S3。通过定量RT-PCR分析了这8个基因在过表达突变体的6株幼苗和长日照条件下生长的野生型幼苗中的表达情况。汇总这些数据的箱形图显示,与野生型相比,不同突变体在个体幼苗中的表达差异要大得多10A)。相比之下,在任何被测试的品系中,没有一个选择为缺乏变异的基因在单个幼苗之间表现出如此大的表达差异(图2)10B).与图中分析的幼苗不同的个体幼苗10附加三个变量(At1g04220,At3g22640,At3g12580)和三个不变基因(At5g02490,At5g10140,At2g01422)进行分析。对所有人来说,除了At3g12580,证实了RNAseq检测到的变异性。虽然基于有限数量的个体,但这些数据表明,不同基因子集的基因表达变异在个体幼苗中也很明显。这强调了研究单株中表达模式的重要性。直接的生物学证据证明增加的表达变异AtCHR23过度表达突变的基因应该进行测试,最好是通过大量的个体幼苗的RNAseq分析。
讨论
在此之前,我们已经详细分析了AtCHR12[7]。AtCHR12是平行的吗AtCHR23:这两个基因在(蛋白质)序列上非常相似,被认为是拟南芥最近基因复制的结果[27]。然而,它们的表达模式在空间和数量上是不同的[38],研究结果表明,这两个基因的过表达对拟南芥的生长参数有不同的影响。表达的AtCHR12这里的表型效应不是有记录的吗AtCHR23过表达。在正常的环境条件下,表型AtCHR12突变体与野生型无法区分。然而,暴露AtCHR12在轻度胁迫条件下过表达突变体导致正常活跃的初芽生长停滞,初茎生长减少。研究表明,在向生殖发育过渡后,染色质重塑相关的生长停滞为植物在实际压力下的生长停滞做了准备[7]。
AtCHR23过表达影响幼苗和营养莲座的生长。由于这两种atp酶在不同的发育阶段以不同的方式影响植物生长,这两种基因在拟南芥中是一个非常相似的基因在基因复制后亚功能化的清晰而不常见的例子[38]。在马铃薯和番茄等作物的基因组中,只有一个基因而不是两个类似基因。27]。这个单一的正交应该结合两者的功能AtCHR12和AtCHR23在这类作物的环境生长反应。如果在未来的实验中得到证实,这将为改善农业相关作物的环境生长反应开辟新的可能性。
观察生长表型需要过度表达而不是功能丧失
的atchr23功能丧失突变体没有表现出任何可见的表型,同等突变体也没有atchr12丧失的突变。的atchr12/atchr23双敲除不能启动根和茎的分生组织,是胚胎致死的(自己的观察;[19])。一个含有弱敲除等位基因的勉强存活的双突变体在干细胞的维持方面存在严重缺陷,发育极度延迟,外观浓密,花器官畸变,根明显缩短[19]。这种双敲除的短根显著减少了细胞分裂和分生组织的其他缺陷[19]。这表明在胚胎和胚乳发育过程中,这两个同源基因似乎是多余的[19]。与弱双敲除的表型影响相反,AtCHR23过表达与根分生组织的形态学影响无关(图2)3.A).过表达与根伸长区缩短以及下胚轴和子叶生长减少有关。这些器官的胚胎后生长主要是受控制的细胞伸长的结果,不涉及显著的细胞分裂[34,39]。因此,AtCHR23过表达对细胞扩增的影响大于对细胞分裂的影响。弱双敲除的表型伴随着几个分生组织标记基因的表达变化(WOX5,月,可控硅)和几个细胞周期基因[19]。在RNAseq分析中,这些基因均未被鉴定为差异表达和/或可变AtCHR23本文介绍过表达表型。
原生或更强的CaMV 35S启动子的过表达导致相同的生长表型。一个相对较小的折叠向上调节约2(表)1;CHR23行:G_AtCHR23v)显然是充分的,折叠上调与根长度减少之间没有明显的相关性(表1)1)。这种缺乏剂量依赖性的上调表明可能涉及其他限制因素。SWI/SNF2 atp酶在蛋白质复合物中的作用[40而这种情结的参与者之一可能会变得受限。大量的ATCHR23可能与这种因子竞争,或者从这种复合物中排出。如果是这样,过表达表型将代表一个开/关的情况,并且允许对ATCHR23在生长调节中的详细作用进行比敲除分析更细致的分析[41]。这种微小的上调皱褶所产生的严重影响很难研究,但特别是在营养发育这样的复杂表型中,这种微妙的影响可能更多的是规律而不是例外[42]。已知的其他几个例子中,相对较小的折叠过表达导致明显的表型效应[43,44]。
ATCHR23功能需要长日照条件
的生长表型AtCHR23过度表达只有在(充足的)光线下才明显。因此,它的函数或(其中一个)直接伴星的函数必须与光周期相关。光周期是植物与环境之间最重要、最复杂的相互作用之一。它是植物用来感知季节的主要刺激物。45]。众所周知,对光周期的响应是开花,但它也影响种子萌发、叶片形成速率、叶片大小和干物质产量[46]。在黑暗和短日照条件下缺乏明确的表型可能是由于黑暗诱导ATCHR23或ATCHR23靶点的蛋白水解降解所致。这种调控的一个例子是促进植物开花的转录调控因子CONSTANS (CO)拟南芥在漫长的夏日里,而不是在短暂的冬日里[47]。需要对ATCHR23进行更详细的分析,以评估蛋白质稳定性是否在调节和/或通过AtCHR23。相反,ATCHR23的假定靶基因可能受到轻度调控,ATCHR23重塑复合体有助于这种调控的微调。鉴于各种植物系对环境胁迫的反应(图2)6),轻微或轻微的持续时间可能会被视为压力。
表达的AtCHR23增加生长和基因表达的可变性
幼苗群体中根(或下胚轴)的长度表现为典型的数量性状:在这种情况下,它在遗传相同的植物的群体平均值附近具有频率分布。表达的AtCHR23将分布转移到较短根的平均值(图1)2A)和下胚轴(图2)2B).这种连续分布表明是一种多基因性状,而不是由单一基因座控制的表型和/或环境的参与,表观遗传过程被认为在其中起重要作用[48,49]。频率分布倾向于重叠,当从个体的角度考虑时,一些过表达的幼苗是与单个野生型幼苗生长相当的个体(图2)1一个,图2)。在个别情况下,过度表达AtCHR23可能不一定会导致增长下降。这表明过表达对生长没有抑制作用本身。过表达后,突变苗比野生型表现出更广泛的生长参数分布(图2)2)。因此,AtCHR23过表达增加了种群内生长的变异性,最好用变异系数(CV;表格1)的增长。
在长时间工作的情况下,AtCHR23过度表达与生物复制之间基因表达的变异性增加有关。人们很容易假定这两种相关现象之间存在因果关系。虽然较大的差异可能来自环境因素和/或使用的生物材料(种子批次),但我们非常小心地排除了这些实验因素的干扰。对光照强度、温度、湿度进行了严格控制和监测;野生型和突变型幼苗无一例外地在同一时间、相同条件下生长;同时分离RNA样品,并尽可能同步RNA处理程序。此外,同一过表达系在短日照条件下的变异性与野生型相似。
的明显效果AtCHR23过表达增加表达变异性在其他生物系统中并非未知。在许多生物体中,包括人类、小鼠、鱼类和酵母,都观察到基因表达的个体间差异[50- - - - - -52]。同样在植物中,在相同环境中基因相同的个体之间,基因表达也会发生相当大的差异。在基因相同的细胞之间,基因表达的幅度、频率和时间似乎是随机的。这种基因表达的随机性本质上是非遗传的或表观遗传的,被认为是基因表达本身的内在特性:随机噪声[53]。在野生型和过表达突变体中被鉴定为可变的68个基因可能代表了在所检查的组织和条件中内在可变或嘈杂的基因。因为AtCHR23过度表达使内在可变的基因更加可变,ATCHR23重塑复合体可能参与调节可变基因的噪声水平。随机噪声可能是有益的,如。为了在波动的压力环境中生存[54,55],但一般认为它会降低健康或干扰发育[53,56]。
染色质相关事件被认为是基因表达随机噪声调控的一个组成部分。例如,在酵母中,染色质重塑复合体的单个组分(如SWI/SNF)的缺失显著增加了PHO5启动子的表达波动[55]和可变基因明显受到染色质修饰因子的调控[57]。在当今已知的大多数生物系统中,表达的内在可变性必须得到控制或缓冲[58以确保最佳的发展和增长。缓冲由分子噪声或环境波动产生的变化的能力称为鲁棒性[20.]。研究表明,在发育或分化中起重要作用的基因的表达是高度稳健的[59],而应激反应基因的表达往往在细胞和个体之间变化更大[53,60]。后一种建议与GO分析中对应激和应激刺激反应基因的显著富集一致,这些基因在两个重复之间高度可变AtCHR23在长时间条件下生长的过度表达行(附加文件1:图S4)。
假定的合用风险
RNAseq分析的最佳方法仍在讨论中[61,62]并可能取决于生物学和统计学问题,如抽样、汇集、汇集设计、(生物或环境)变异的分布等[63,64]。考虑基因表达的生物学变异对于可靠和生物学相关的差异表达分析非常重要[65]。池中单个幼苗之间的基因子集表达差异较大(图2)10)可能导致来自不同数据池的数据之间再现性差。
我们在这里提出了一项RNAseq数据分析,重点关注基因表达的可变性作为感兴趣的主题。通过qRT-PCR的独立验证显示了所开发方法的有效性,尽管更高级的统计可能会从本文提供的RNAseq数据中提取更多的理解。这些数据是基于基因相同的幼苗池,然而这些幼苗可能表现出高度可变的基因表达。这种基因表达的个体间差异尚未引起太多关注,但新测序技术的深度可以提供绕过这一限制的方法[66]。事实上,对单个幼苗的分析也涉及到不同发育阶段的各种组织,这些组织可能在基因表达上存在差异。在未来,大规模的单细胞转录组学可能会解决这些复杂问题[67,68]。
结论
我们已经证明,在转基因拟南芥中,SWI/ snf2型atp酶的过表达AtCHR23在遗传相同的植物群体中,增加生长的可变性和不同基因子集的表达可变性。这些结果提示AtCHR23有助于更稳定或健壮的基因表达,导致更均匀的生长表型。基于表型和表达数据,我们提出ATCHR23重塑复合体可能是植物基因表达缓冲系统的一个组成部分。如果这个缓冲系统被过度表达破坏AtCHR23,下游基因变得更加可变,并以导致此处记录的生长表型降低的方式损害其他基因的表达。表型稳健性影响植物生长、产量和品质的所有重要参数。这里提出的研究结果将有助于更好地理解和利用染色质重塑基因作为表型和转录变异的潜在缓冲指数,特别是在不断变化的环境条件下。
方法
构建用于转化的T-DNA质粒
产生过度表达的植物AtCHR23从拟南芥Col-0野生型中获得基因序列的基因组拷贝(包括全部11个内含子)。三组引物与Phusion™DNA聚合酶一起使用(见附加文件)2:详情见表S4)。将三个PCR片段克隆到pJET (Fermentas)中,并进行测序验证。基因组拷贝AtCHR23将三个PCR序列的合适限制性片段连接到pENTR4 (Invitrogen)中组装。由此产生的质粒携带整个基因,包括127个5'UTR核苷酸。在LR Gateway (Invitrogen)与pB2GW7 (http://gateway.psb.ugent.be/)。得到的二进制矢量35S::AtCHR23(附加文件1:图S1)被引入根癌土壤杆菌C58C1 (pMP9),用于拟南芥转化。根据PPT抗性(5 μg ml)筛选转基因株系1phosphinothricin-DL),筛选转基因表达水平。这些线路被指定为AtCHR23机汇。此外,两个gfp标记的构建体携带cDNA拷贝AtCHR23分别由CaMV 35S或内源性启动子驱动(附加文件)1:图S1)。的cDNA拷贝AtCHR23用上标®采用寡核苷酸(dT)的第一链合成体系20.引物(Invitrogen)和PCR扩增使用CHR23_F4和CHR23_R4引物(附加文件2表S3)。用Gateway BP克隆酶将全长cDNA序列重组到pDONR221进入载体(Invitrogen)中。将得到的质粒在LR Gateway反应中重组到目的载体pK7FWG2 (http://gateway.psb.ugent.be/)。得到的二进制矢量35S::绿色荧光蛋白-AtCHR23用于拟南芥转化。根据卡那霉素抗性(50 μg ml)筛选转基因株系1)和转基因表达水平。这些线路被指定为G_AtCHR23机汇。的启动子序列(918个碱基)AtCHR23利用引物pCHR23_F和pCHR23_R(附加文件)进行PCR分离2(表S4)并克隆到pENTR4入口载体中。用合适的限制性内切酶选择所需的启动子序列,克隆到携带该基因的pENTR4衍生物中绿色荧光蛋白基因。由此产生的克隆是,连同AtCHR23上述cDNA进入克隆,通过多步LR Gateway反应组装到修饰的目的载体pBGW (http://gateway.psb.ugent.be/)。得到的二进制向量pCHR23::绿色荧光蛋白-AtCHR23转化为拟南芥。根据PPT抗性(5 μg ml)筛选转基因植株1phosphinothricin-DL),用于进一步分析的品系命名为CHR23;G_AtCHR23机汇。
植物材料和生长条件
所有转基因拟南芥植株均过表达AtCHR23是由野生型转化产生的吗拟南芥colo -0使用花浸法[69]。分析采用纯合子F3植株。的丧失功能突变系AtCHR23(At5g19310) SALK_057856和SALK_139883来自拟南芥库存中心(Salk实验室,基因组学分析研究所,美国;由J.R. Ecker [70])。SALK_057856在第一个外显子携带T-DNA插入,SALK_139883在第5个外显子携带T-DNA插入AtCHR23。在30 μg ml浓度下测定两株SALK的合合性1卡那霉素盘子。两种基因敲除均未检测到全长cDNA产物(数据未显示)。标记系pCYCB1;1:CYCB1;1- gus [29]在Col-0中的表达来源于M. Koornneef (Cologne/Wageningen)。在所有情况下,在播种或分析前,种子在黑暗中4°C分层3天以同步发芽。幼苗垂直生长在完全受控的生长室内,由Philips TD 32w /84HF灯照明,温度分别为23°C、25°C或28°C。16小时光照/8小时黑暗)或短昼(SD;8 h光照/16 h暗)光周期。根据实验设置调整光照条件。植物在标准盆栽土壤中生长,在21±2°C下,在16小时光照/8小时黑暗(长白天条件)下,在由Philips- master 36w /830灯照明的生长室内或在受控温室中生长,必要时由四个Son-T (Philips Greenpower, 400 W)灯提供补充光。
生长参数分析
根伸长测定方法如下所述[7在0.5 × MS琼脂板上垂直生长的幼苗。在盐胁迫和渗透胁迫下,幼苗分别生长在添加75 mM NaCl和200 mM甘露醇的培养皿上。幼苗生长8-10天后拍照(佳能SX120),用ImageJ (http://rsb.info.nih.gov/ij)。离体的下胚轴、子叶和叶片在双面胶带上被压平,并拍照供ImageJ分析。为了分析营养莲座的生长,植物在萌发后4周,即开花前拍摄了照片。利用ImageJ将整个花环围成一个矩形选区后,估计花环的直径。在0.5 × MS琼脂板上垂直培养6 d的幼苗,测定分生组织的大小和伸长区。分生区是指从静止中心到过渡区之间的长度,以及从静止中心到过渡区第一个细胞之间皮层中细胞的数量。在伸长区分析了从过渡区到分化区开始的距离长度和完全伸长细胞的大小。使用徕卡显微镜(Leica Microsystems)获得图像,并在ImageJ中用于长度测定。对于大多数生长参数,至少进行了两到三次重复。测量用15-20根。 GUS patterns were observed as described previously [7用尼康Optiphot-2显微镜观察。
统计分析
在统计分析方面,采用Shapiro-Wilk检验评价数据的正态性[71],方差齐性采用Levene检验[72] (http://www.stat.ufl.edu/~winner/sta6166.html)。采用非参数Mann-Whitney U检验计算相同生长条件下野生型与突变体均数差异的显著性。为了比较不同生长条件,采用非参数调整秩变换检验[73]被使用。在图表和表格中,星号*,**,***,分别在0.05、0.01和0.001的置信水平上表示显著性。
qRT-PCR和RNAseq分析基因表达
幼苗在琼脂板上生长,在相同的设置和条件下用于生长测量(见上文)。使用e.zn.a.™植物RNA迷你试剂盒(Omega Bio-Tek, Inc., USA)从8个完整的8日龄幼苗池中分离总RNA,然后进行柱上dna酶处理(Qiagen, RNase-free DNase Set)。1微克RNA使用iScript™cDNA合成试剂盒(Bio-Rad Laboratories, Inc., USA)进行cDNA合成。使用iQ™SYBR进行10倍稀释的cDNA定量RT-PCR®Green Supermix (Bio-Rad Laboratories, Inc., USA)在iCycler热循环器中。反应进行了三次。的哥伦比亚大学基因(At5g25760)作为参考[74]。所使用的引物序列在附加文件中给出2:表S3。
对于RNA测序,从8个8日龄突变体(AtCHR23- 4v)或野生型,在23°C下生长,长日照(具有减少的生长表型)或短日照(没有减少的生长表型)光周期。在所有试验中,选取1年龄(生物重复1)和半年龄(生物重复2)两种不同种源的2个生物重复。采用4种不同条件,每个条件下设置2个生物重复,共8个样品进行分析。所有8个RNAseq文库制备均按照制造商建议(Illumina Truseq RNA样品制备低通量协议)进行。8个样本在一个Hiseq 2000 lane (WUR测序设备)中进行多路处理,并在100个碱基配对的末端读取中测序,插入长度约为300个碱基。在解复用之后,对于8个样本中的每一个,都产生了超过6000万个读取。
RNASeq生物信息学
使用FastQC(从http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)。使用CLCbio Genomics工作台(v. 5.5.1)使用默认设置执行适配器和质量修剪。读取被映射到拟南芥使用TopHat (v. 2.0.5;[75]),默认参数设置:——no-mixed,——no-discordant, -M, -g 1,——min-intron-length 50,——max-intron-length 11000。采用DEseq (v1.10.1;[35])和袖扣包装中的袖扣(v. 2.0.2;[36])使用设置选项-u和-b,没有质量修剪。通过计算FPKM(每百万映射读取的转录物(外显子模型)的每千碱基片段)值来确定基因表达水平。为了分析表达变化,我们比较了每个样品和条件在两个重复中FPKM > 0.5的所有基因的表达水平。对数的绝对差2转换后的FPKM值[log .2(fpkm + 1);计算了大约相当于正常尺度上的倍变化],并使用在长日照条件下生长的野生型植物的前1%基因来定义所有其他条件下的截止值,以确定差异基因的数量(和身份)(即。变化)大于这个截止值。使用Classification SuperViewer工具进行基因本体(GO)分析[37]来自Bio-Array资源(http://bbc.botany.utoronto.ca/ntools/cgi-bin/ntools_classification_superviewer.cgi)。基因根据氧化石墨烯的三个主要类别进行功能分类:生物过程、分子功能和细胞成分。
参考文献
李建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军。植物学报,2012,32(3):557 - 563。10.1146 / annurev - arplant - 042811 - 105550。
Lianes A, Reinoso H, Luna V:不同盐溶液中豆豆幼苗的萌发和早期生长。世界农业科学学报,2005,1(2):120-128。
Nicotra AB, Atkin OK, Bonser SP, Davidson AM, Finnegan EJ, Mathesius U, Poot P, puruganan MD, Richards CL, Valladares F, van Kleuven M:气候变化下植物表型可塑性。植物科学进展,2010,15(12):684-692。10.1016 / j.tplants.2010.09.008。
王晓明,王晓明,王晓明,等。植物逆境适应的表观遗传机制。植物学报,2011,14(3):267-274。10.1016 / j.pbi.2011.03.004。
Walley JW, Rowe HC, Xiao Y, Chehab EW, Kliebenstein DJ, Wagner D, Dehesh K:染色质重塑因子SPLAYED调控胁迫信号通路。中华疾病杂志,2008,4 (12):e1000237-10.1371/journal.ppat.1000237。
金建明,陶国明,王志刚,王志明:植物非生物胁迫响应中染色质调控的研究进展。植物细胞环境,2010,33(4):604-611。10.1111 / j.1365-3040.2009.02076.x。
李春华,李春华,李春华,等。SWI/SNF染色质重塑基因AtCHR12调控拟南芥在环境胁迫下的生长。植物学报,2007,31(5):874-885。10.1111 / j.1365 - 313 x.2007.03185.x。
赵建军,刘建军,刘建军,等。RNA沉默酶RNA聚合酶在植物免疫中的作用。科学通报,2011,7 (12):e1002434-10.1371/journal.pgen.1002434。
冯绍平,李建平,李建平,等。表观遗传重编程在植物和动物发育中的应用。科学通报,2010,33(6):622-627。10.1126 / science.1190614。
王晓明,王晓明,王晓明,等。植物生长发育过程中染色质重构的研究进展。中国生物医学工程学报,2009,35(5):559 - 561。
Ogas J, Kaufmann S, Henderson J, Somerville C: PICKLE是调控拟南芥胚胎向营养发育过渡的CHD3染色质重塑因子。[j] .中国科学:自然科学,1999,35(4):839- 844。10.1073 / pnas.96.24.13839。
李春华,李春华,李春华,等。拟南芥种子萌发过程中染色质重塑因子PKL在渗透胁迫响应中的作用。植物学报,2007,32(5):927-936。10.1111 / j.1365 - 313 x.2007.03288.x。
Aichinger E, Villar CB, Di Mambro R, Sabatini S, Kohler C: CHD3染色质改造因子PICKLE和polycomb group蛋白拮抗拟南芥根分生组织活性。植物学报,2011,23(3):1047-1060。10.1105 / tpc.111.083352。
景艳,张东,王鑫,唐伟,王伟,怀军,徐刚,陈东,李勇,林荣:拟南芥染色质重塑因子PICKLE与转录因子HY5互作调控下胚轴细胞伸长。植物学报,2013,25(1):242-256。10.1105 / tpc.112.105742。
李建军,李建军,李建军,李建军,等。基因序列分析与基因序列分析。核酸学报,2006,34(10):2887-2905。10.1093 / nar / gkl295。
Farrona S, Hurtado L, Bowman JL, Reyes JC:拟南芥SNF2同源AtBRM调控茎部发育和开花。发展,2004,131(20):4965-4975。10.1242 / dev.01363。
张春华,张春华,张春华,张春华,张春华。拟南芥SWI/SNF染色质重构ATPases BRAHMA和SPLAYED的研究进展。植物学报,2007,19(2):403-416。10.1105 / tpc.106.048272。
唐新,侯安,Babu M, Nguyen V, Hurtado L, Lu Q, Reyes JC, Wang A, Keller WA, Harada JJ, Tsang EW,崔勇:拟南芥叶片中染色质重塑ATPase参与种子成熟基因的抑制。植物生理学。中国机械工程,2008(3):1143-1157。10.1104 / pp.108.121996。
桑颖,吴胜,吴明义,吴明义,吴明义,Pfluger J, Stewart Gillmor C, Gallagher KL, Wagner D:两个非标准拟南芥SWI2/SNF2染色质重塑atp酶突变导致胚胎发生和干细胞维持缺陷。植物科学学报,2012,32(2):1010 - 1010。
李建军,李建军,李建军,等。植物抗逆性的分子机制。植物学报,2013,16(1):62-69。10.1016 / j.pbi.2012.12.002。
Sollars V, Lu X, Xiao L, Wang X, Garfinkel MD, Ruden DM: Hsp90作为形态进化的催化剂的表观遗传机制证据。植物学报,2003,33(1):70-74。
Lehner B, Crombie C, Tischler J, Fortunato A, Fraser AG:秀丽隐杆线虫遗传相互作用的系统定位及其不同信号通路的共同修饰因子。生物医学工程学报,2006,38(8):896-903。10.1038 / ng1844。
李世峰,李志明,李志明:网络枢纽缓冲环境变化的酿酒酵母。生物医学进展,2008,6 (11):e264-10.1371/journal.pbio.0060264。
李建军,李建军,李建军,刘建军,等。基因表达调控因子的研究进展。中国生物医学工程学报,2010,32 (6):444 - 444
Landry CR, Rifkin SA:染色质调控因子塑造基因型表型图谱。中国生物医学工程学报,2010,26 (6):444 - 444
在酵母中,基因对环境、随机和遗传扰动具有相似的稳健性。科学通报,2010,5 (2):e9035-10.1371/journal.pone.0009035。
Bargsten JW, Folta A, Mlynarova L, Nap JPH:高等植物基因组中Snf2基因的计算分析:独立进化和DRD1扩增的证据。公共科学图书馆。2013, 8: e81147-10.1371/journal.pone.0081147。
Elwell AL, Gronwall DS, Miller ND, Spalding EP, Brooks TL:亲本环境和种子大小对拟南芥下一代生长发育的影响。植物细胞环境,2011,34(2):291-301。10.1111 / j.1365-3040.2010.02243.x。
刘建军,刘建军,李建军,等。细胞周期蛋白- gus融合蛋白的克隆及其在细胞分裂中的应用。植物学报,1999,20(4):503-508。10.1046 / j.1365 - 313 x.1999.00620.x。
王晓明,王晓明,王晓明,等。高温对拟南芥下胚轴伸长的影响。[j] .中国科学:自然科学,1998,35(12):7197-7202。10.1073 / pnas.95.12.7197。
焦艳,刘玉生,邓小文:高等植物光调控转录网络。水土保持学报,2007,8(3):217-230。
王晓明,王晓明,王晓明,等。拟南芥根系生长动力学的时空分辨率分析。中国生物医学工程学报,2010,35(5):783-791。10.1093 / aob / mcq048。
Kircher S, Schopfer P:光合蔗糖作为子叶源远程信号调控拟南芥幼苗发育早期根系生长。美国科学进展,2012,109(28):11217-11221。10.1073 / pnas.1203746109。
王晓明,王晓明,王晓明,等。拟南芥下胚轴生长的细胞机制研究。植物生理学。中国生物医学工程学报,2009,31(1):394 - 394。10.1104 / pp.114.1.295。
Anders S, Huber W:序列计数数据的差异表达分析。基因组生物学,2010,11 (10):R106-10.1186/gb-2010-11-10-r106。
trappnell C, Williams BA, Pertea G, Mortazavi A, Kwan G, van Baren MJ, Salzberg SL, Wold BJ, Pachter L: RNA-Seq转录本组装和定量分析揭示细胞分化过程中无注释转录本和异构体转换。生物工程学报,2010,28(5):511-515。10.1038 / nbt.1621。
朱涛。基于浏览器的拟南芥基因组功能分类SuperViewer。中国生物医学工程学报,2003,27(2):444 - 444。
Duarte JM,崔玲,Wall PK,张强,张晓明,Leebens-Mack J,马宏,Altman N, de Pamphilis CW .拟南芥调控基因的亚功能化和新功能化表达模式变化。生物医学进展,2006,23(2):469-478。
李春华,黄春华,黄春华,等。拟南芥子叶细胞分裂和细胞增殖的研究进展。植物学报,2004,32(2):471-479。10.1111 / j.1469-8137.2004.01031.x。
Mohrmann L, Verrijzer CP: SWI2/SNF2类染色质重塑复合物的组成和功能特异性。生物化学学报,2005,31(2):59-73。
基因过表达:用途、机制和解释。遗传学报,2012,31(3):841-854。10.1534 / genetics.111.136911。
Birchler JA, Veitia RA:基因平衡假说:跨生物学科的剂量敏感性连接问题。中国科学:自然科学,2012,37(3):14746-14753。10.1073 / pnas.1207726109。
陈慧聪,Stone SJ,周鹏,Buhman KK, Farese RV:过度表达酰基辅酶a:二酰基甘油酰基转移酶1的小鼠肥胖解离和葡萄糖处理障碍。糖尿病杂志,2002,51(11):3189-3195。10.2337 / diabetes.51.11.3189。
Delgado MD, Leon J: Myc在造血和白血病中的作用。癌症基因,2010,1(6):605-616。10.1177 / 1947601910377495。
戴维斯SJ:光周期主义:对季节的巧合感知。中国生物医学工程学报,2002,12(4):444 - 444。10.1016 / s0960 - 9822(02) 01348 - 9。
[3]王晓明,王晓明。植物生长与光周期关系的研究进展。园艺科学与生物技术杂志。[j] .中国科学:地球科学。
光周期开花时间的进化起源。实验学报,2011,62(8):2453-2463。10.1093 / jxb / erq449。
richard EJ:定量表观遗传学:DNA序列变异不需要应用。基因发育,2009,23(14):1601-1605。10.1101 / gad.1824909。
李建军,李建军,李建军,李建军,等。拟南芥重组自交系新群体生长相关性状的基因座分析。植物生理学。中国生物医学工程学报,2004,31(1):444-458。10.1104 / pp.103.036822。
李建军,张建军,张建军,等。基因表达在自然种群中的变化。植物学报,2002,32(2):261-266。10.1038 / ng983。
Fay JC, McCullough HL, Sniegowski PD, Eisen MB:酿酒酵母基因表达的群体遗传变异与表型变异相关。中国生物医学工程学报,2004,5 (4):526 - 526 /gb。
李军,刘勇,金涛,闵锐,张志:人群内和人群间基因表达变异及其对疾病易感性的影响。科学通报,2010,6 (8):e1000910-10.1371/journal.pcbi.1000910。
《自然、培养或机遇:随机基因表达及其后果》。细胞学报,2008,35(2):216-226。10.1016 / j.cell.2008.09.050。
陈晓明,陈晓明,陈晓明,陈晓明,陈晓明。基因表达噪声与调控网络结构的相互作用。趋势与进展,2012,28(5):221-232。10.1016 / j.tig.2012.01.006。
Raser JM, O 'Shea EK:真核生物基因表达的随机性控制。科学通报,2004,34(6):1811-1814。10.1126 / science.1098641。
王铮,张杰:基因表达噪声对生物适应度和自然选择效能的影响。美国科学进展,2011,108 (16):E67-E76。10.1073 / pnas.1100059108。
崔建军,金玉军:表观遗传调控与基因表达变异性。植物学报,2008,40(2):141-147。10.1038 / ng.2007.58。
李建军,李建军,李建军,等。基因表达的不完全外显性。自然科学,2010,36(7):913-918。10.1038 / nature08781。
李建军,李建军,李建军,等。基因调控网络在基因表达控制中的作用。遗传学报,2011,21(5):645-657。10.1101 / gr.097378.109。
Blake WJ, Balazsi G, Kohanski MA, Isaacs FJ, Murphy KF, Kuang Y, Cantor CR, Walt DR, Collins JJ:启动子介导的转录噪声的表型后果。生物质化学工程,2006,24(6):853-865。10.1016 / j.molcel.2006.11.003。
McCarthy DJ, Chen Y, Smyth GK:生物变异的多因子RNA-Seq实验差异表达分析。核酸学报,2012,40(10):4288-4297。10.1093 / nar / gks042。
Trapnell C, Hendrickson DG, Sauvageau M, Goff L, Rinn JL, Pachter L:基于RNA-seq的基因调控差异分析。生物工程学报,2013,31(1):46-53。
endziorski C, Irizarry RA, Chen KS, Haag JD, Gould MN:微阵列实验中生物样本池的应用。科学通报,2005,32(3):452 - 457。10.1073 / pnas.0500607102。
Kliebenstein DJ:生物学中的模型误解:表型,统计,网络和推理。植物科学前缘,2012,3:13-
Oshlack A, Robinson MD, Young MD:从RNA-seq读取到差异表达结果。基因组生物学,2010,11 (12):220-10.1186/gb-2010-11-12-220。
王志强,李志强。基因表达在物种内部和物种之间的变异:进化的原始材料。中国生物医学工程学报,2006,15(5):1197-1211。10.1111 / j.1365 - 294 x.2006.02868.x。
唐峰,老K, Surani MA:单细胞转录组分析的发展与应用。自然科学学报,2011,8(4增刊):6-11。
李建军,李建军,李建军。单细胞分析在生物技术、系统生物学和生物催化中的应用。化学与生物工程,2012,3(3):129-155。10.1146 / annurev - chembioeng - 062011 - 081056。
郭文杰,李文杰,李文杰,等。农杆菌介导的拟南芥转化方法的研究进展。植物学报,1998,16(6):735-743。10.1046 / j.1365 - 313 x.1998.00343.x。
Alonso JM, Stepanova AN, Leisse TJ, Kim CJ, Chen H, Shinn P, Stevenson DK, Zimmerman J, Barajas P, Cheuk R, Gadrinab C, Heller C, Jeske A, Koesema E, Meyers CC, Parker H, Prednis L, Ansari Y, Choy N, Deen H, Geralt M, Hazari N, home, Karnes M, Mulholland C, Ndubaku R, Schmidt I, Guzman P, Aguilar-Henonin L, Schmid M,等:拟南芥全基因组插入突变。科学通报,2003,31(6):653-657。10.1126 / science.1086391。
亨德森AR:检验单变量正态性的实验数据。临床医学学报,2006,366(1-2):112-129。
林天生,陆文勇:方差相等检验的比较。计算机统计数据,1996,22(3):287-301。10.1016 / 0167 - 9473(95) 00054 - 2。
leysc, Schumann S:一种非参数交互分析方法:调整秩变换检验。[J]心理学报,2010,46(4):684-688。10.1016 / j.jesp.2010.02.007。
赵建军,刘建军,刘建军,等。拟南芥转录调控内参基因的克隆与鉴定。植物生理学。中国机械工程,2005,(1):5-17。10.1104 / pp.105.063743。
trappnell C, Pachter L, Salzberg SL: TopHat:利用RNA-Seq发现剪接连接。生物信息学,2009,25(9):1105-1111。10.1093 /生物信息学/ btp120。
致谢
我们感谢2012年生物系统基因组学中心(CBSG2012)作为荷兰基因组学倡议(NGI)的一部分提供的财政支持。AF由荷兰科学研究组织(NWO)通过其ALW/TTi绿色遗传学计划提供支持。JPN和HvdG还得到了由格罗宁根汉泽应用科学大学协调的RaakPRO BioCOMP项目的支持。我们感谢Elio Schijlen博士和Ing博士。拥有Lintel Hekkert (WUR测序设施)进行RNA测序和讨论,以及Joachim Bargsten理学硕士(植物育种和应用生物信息学,WUR)进行系统发育分析和输入。Ton Bisseling教授博士(分子生物学,WUR)提供了广泛的支持和鼓励。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称不存在利益竞争。
作者的贡献
AF(进行实验,分析数据,撰写稿件);EIS(进行基因表达分析,撰写部分稿件);JK和HG(协助进行表达分析);JV(生成的构造);JPN(协助解读数据并撰写稿件);LM(设计实验,分析数据,撰写论文)。所有作者都阅读并批准了最终的手稿。
电子补充资料
12870 _2014_1488_moesm1_esm.pdf
附加文件1:图S1:用于产生过表达转基因拟南芥的质粒的T-DNA区域示意图AtCHR23基因。35S, CaMV 35S启动子;pCHR23,AtCHR23启动子;绿色荧光蛋白,绿色荧光蛋白基因;Kan,卡那霉素耐药基因;BAR,麻秆酶抗除草剂基因;RB, LB,左右T-DNA边界。灰色阴影框表示存在5'UTR。图S2。CYCB1;1:CYCB1;1- gus野生型(左)和CYCB1AtCHR23两种转基因均为-4ov纯合。AtCHR23-4ov与转基因系pCYCB1;1:CYCB1;1- gus杂交格斯报告基因与有丝分裂破坏序列(D-box)和细胞周期蛋白融合CYCB1; 1启动子。在记者这一行,格斯在进入细胞周期G2期时通过CYCB1; 1启动子及其蛋白产物在中期通过D-box退出后被降解。bar: 20 μm。图S3。的负面影响AtCHR23盐胁迫增强了cDNA过表达对生长的影响。在75 mM NaCl条件下生长的10日龄野生型(Col)和突变型幼苗主根的平均(±SD)长度。每个品系测量40株幼苗。星号表示与野生型的显著差异:***(P < 0.001)。图S4。基因本体(GO)分析的基因显示高变异性在两个重复之间的表达AtCHR23过表达突变体在长日照条件下生长。用Classification SuperViewer对298个重复表达差异大于3倍的基因进行分类。给出了GO分类归一化频率±bootstrap SD。归一化频率大于1的类别代表过度,低于1的类别代表不足。深灰色和粗体显示的类别的代表性过高或不足在P<0.01具有统计学意义;p值在SD旁边表示。(pdf 104kb)
12870 _2014_1488_moesm2_esm.docx
附加文件2:表S1:修改后的效果AtCHR23生长性状变异的表达。表S2。非参数调整秩变换检验结果。表S3。定量RT-PCR检测基因定义如图10所示。表S4。研究中使用的引物。(docx 26kb)
权利和权限
开放获取本文由BioMed Central Ltd.授权发表。这是一篇开放获取的文章,在知识共享署名许可(https://creativecommons.org/licenses/by/2.0),允许在任何媒体上不受限制地使用、分发和复制,前提是原创作品要有适当的署名。创作共用公共领域免责声明(https://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有说明。
关于本文
引用本文
Folta, A, Severing, e ., Krauskopf, J。et al。拟南芥的过表达AtCHR23染色质重塑三磷酸腺苷酶导致生长和基因表达的变异性增加。BMC Plant Biol14日,76(2014)。https://doi.org/10.1186/1471-2229-14-76
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-14-76
关键字
- 拟南芥
- 染色质重塑
- 增长
- 基因表达
- 可变性
- 鲁棒性