摘要
背景
扩张素是植物细胞壁松动蛋白,参与细胞扩大和多种其他发育过程。扩展素超家族包含四个亚家族;即α-膨胀素(EXPA)、β-膨胀素(EXPB)、类膨胀素A (EXLA)和类膨胀素B (EXLB)。虽然大豆的基因组测序已经完成,但我们对大豆扩张素基因的扩张模式和进化史的了解仍然有限。
结果
在大豆基因组中共鉴定出75个扩增素基因,并根据其系统发育关系将其分为4个亚家族。结构分析表明,扩增素基因在各亚科内保守,但在各亚科间存在差异。此外,在大豆和拟南芥,扩增素基因家族主要通过串联复制和节段复制进行扩增;然而,在水稻中,节段复制似乎是产生这个超家族的主要过程。转录组图谱显示,在正常生长条件下,无论是转录丰度还是表达模式都存在显著差异。这一发现与差异分布相一致独联体该超家族在启动子区有明显的功能分化。此外,还检测到一些有助于功能分化和正向选择的关键氨基酸。最后,正选择的位点模型和分支-位点模型分析表明,大豆扩张素基因超家族处于强正选择状态,四个亚家族的进化可能受到不同选择约束的影响。
结论
本研究表明大豆扩张素基因超家族通过串联复制和分段复制实现了扩张。差异表达表明该超家族存在广泛的功能分化。此外,正选择分析表明,不同的选择约束可能影响了四个亚科的进化。综上所述,本研究结果为大豆扩张素基因超家族的分子进化提供了新的详细信息。
背景
扩张素由一个多基因家族编码,由一个植物细胞壁松动蛋白超家族组成,这些蛋白以一种特有的、独特的方式诱导ph依赖的细胞壁延伸和应激松弛[1].扩张素在植物细胞壁扩大机制的研究中首次被发现,并从黄瓜下胚轴中分离出来[2].最近,在包括燕麦在内的其他植物物种中发现了越来越多的扩张素[3.],西红柿[4]和玉米[5].根据Kende等人提出的命名法[6],根据系统发育序列分析,植物中的扩张素超家族可分为4个亚家族;这些亚家族被命名为α-膨胀素(EXPA)、β-膨胀素(EXPB)、类膨胀素A (EXLA)和类膨胀素B (EXLB)。α-扩张蛋白和β-扩张蛋白具有细胞壁松动活性,参与细胞扩张和其他发育过程;然而,类膨胀蛋白A和类膨胀蛋白B只能从它们的基因序列中得知[7],目前还没有关于它们在细胞壁上活动的实验证据发表[8].
功能研究表明膨胀素参与了许多发育过程,如果实软化[9],木质部形成[10]、脱落(叶脱落)[11],种子萌发[12],以及花粉管的穿透[13,14].植物细胞壁由纤维素微原纤维组成,与各种聚糖结合,包括木葡聚糖和木聚糖。细胞壁的延伸涉及到纤维素微原纤维的运动和分离,通过分子爬行的过程。α-膨胀素被认为通过诱导木葡聚糖的局部解离和滑移来促进这种运动,而β-膨胀素则被认为以类似的方式作用于另一种聚糖,也许是木聚糖[7].然而,没有实验证明膨胀素具有水解活性或任何其他酶活性[15- - - - - -17].
膨胀蛋白通常有250-275个氨基酸长,包含两个结构域,前面是一个长度为20-30个氨基酸的信号肽[7].结构域I与糖苷水解酶家族家族-45 (GH45)蛋白具有显著但遥远的同源性,包括一系列保守的半胱氨酸和组成家族-45内切葡聚糖酶催化位点的his - phi - asp (HFD)基序[9,18].域II与第2类草花粉过敏原有远亲关系[9].结构域II被推测为基于蛋白质表面保守的芳香族残基和极性残基的多糖结合结构域[18].只有一种细菌膨胀蛋白的晶体结构[19]和玉米中的Zea m 1 [20.都被解决了。
大豆基因组测序完成[21]为我们提供了一个机会来提高我们对这种植物物种中扩张素超家族的进化和其他特征的理解。在本研究中,我们鉴定了大豆基因组中的扩增素基因,并将其分为4个亚家族。此外,还研究了扩增素基因家族的扩增模式拟南芥,大米和大豆被检查。结果表明,大豆扩增素基因是通过串联复制和分段复制的方式产生的。正常条件下大豆扩张素基因在不同组织中的转录组图谱分析表明,亚家族间表达存在显著差异。这一发现表明在这个超家族中存在广泛的功能分歧。发现了导致功能分化的关键氨基酸。此外,负责功能分化和/或阳性选择的氨基酸位点的位置表明I结构域和C端是保守的。本研究的结果有望促进对该基因家族的进一步研究,并为扩展蛋白的进化史提供新的见解。
结果
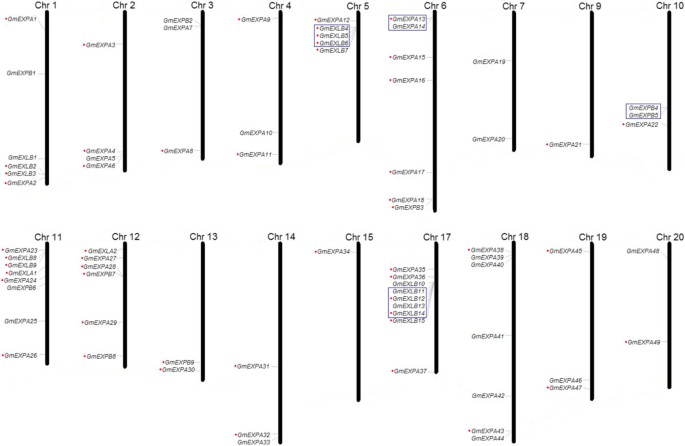
大豆扩张素基因超家族的全基因组鉴定
通过大豆基因组blast和在线软件鉴定,共鉴定出75个大豆扩增素基因(附加文件)1)根据扩展命名法[6].基于Pfam和SMART测试,所有75个成员都包含两个域(PF03330和PF01357)。只有其中一个结构域或没有完整的开放阅读框的蛋白质被排除在外。蛋白质序列(附加文件2),编码序列(CDS)(附加文件3.),基因组序列(附加文件4),以及翻译起始密码子上游的核苷酸序列的1500 bp(附加文件5)都是从Phytozome数据库(http://www.phytozome.com).此外,还从Phytozome数据库中获得了扩增素基因的物理位置,并将其映射到相应的染色体上(图1).结果表明,除8号和16号染色体外,扩增素基因可定位于1 ~ 20号染色体上。17号染色体的扩增素基因密度最高,有9个成员,而7号、9号、13号、15号和20号染色体含有不超过2个扩增素基因。为了明确这些扩增素基因属于哪个亚家族(EXPA、EXPB、EXLA或EXLB),我们利用MEGA v5.0软件,采用邻居连接(NJ)方法构建了无根系统发育树。拟南芥和大米(附加文件6).由于基因的膨胀拟南芥水稻已经被分类,我们可以根据系统进化树上显示的聚类对大豆扩增素基因进行分类。据此将大豆膨胀素基因分为4个已知亚家族:α-膨胀素(EXPA)、β-膨胀素(EXPB)、膨胀素样A (EXLA)和膨胀素样B (EXLB)。根据Kende等人提出的命名规则[6],根据基因座及其所属亚家族,对75个大豆扩增素基因进行了命名。所有大豆膨胀蛋白的基本信息(包括基因名称、基因座、蛋白长度、信号肽长度、内含子数、pI值、分子量)均在附加文件中提供1.大豆中的75个膨胀蛋白长218 ~ 309个氨基酸,分子量在23.5 ~ 33.8kD之间。除10个成员缺乏信号肽外,其余75个扩展蛋白均含有长度为16 - 31个氨基酸的信号肽。大豆膨胀素超家族的pI值在4.5 ~ 9.8之间,EXLB与其他亚家族存在差异。EXPA、EXPB和EXLA亚家族中几乎所有成员的pI值都在7.0以上,而EXLB中大多数成员的pI值都在7.0以下。
为了获得更多关于4个扩张素亚家族的大小特征的信息,我们比较了5种植物的扩张素基因(拟南芥大米、大豆和其他两种豆类,Medicago truncatula而且菜豆).中四个亚科大小的数据拟南芥大米是从一篇综述[7].此外,我们对扩增素基因超家族进行了全基因组鉴定Medicago truncatula而且菜豆(附加文件7),按照与鉴定大豆膨胀素基因超家族相同的方法。扩增素基因分别为36和18个菜豆而且Medicago truncatula,分别。然后,我们根据系统发育树将这些扩展蛋白基因分为四个亚家族(附加文件)7).5个种间亚科大小比较结果见表1.扩增素基因在4个亚家族中的分布不均匀。在5个种中,EXLA亚科大小最小,EXPA亚科大小最大1).这两种豆科植物,大豆和菜豆在大豆中有15个成员,在大豆中有5个成员菜豆),而两者中只有一个成员拟南芥和米饭。相比之下,豆类Medicago truncatula只有一个EXLB成员。此外,水稻中的EXPB亚家族比其他四种双子叶虫要大得多。
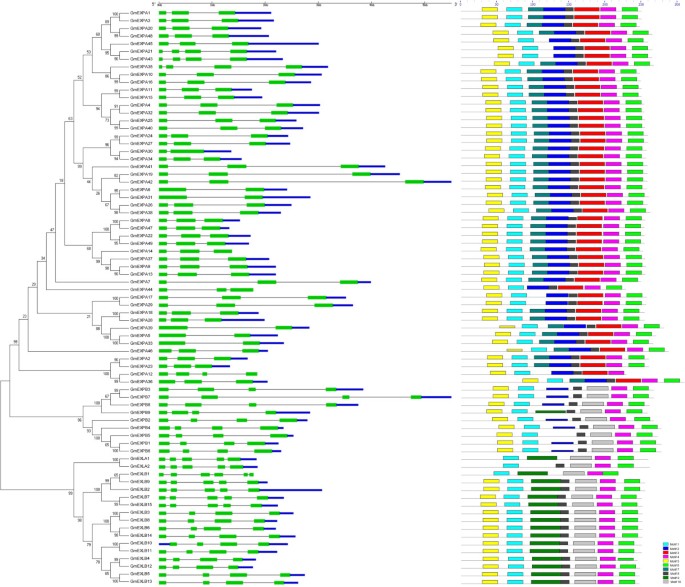
大豆扩增素基因的系统发育及结构分析
我们执行了多个序列比对(附加文件8),根据推测的氨基酸序列构建了75个大豆膨胀素基因的系统发育树(图2).来自同一亚家族的膨胀蛋白聚集在一起。四个亚家族的系统发育分类与基序位置和外显子-内含子组织一致。
如图所示2, 10种类型的主题(附加文件9)。同一亚家族蛋白的基序、类型和数量基本相同,但与其他亚家族蛋白存在差异。在EXPA亚家族中,49个成员中有42个(85.7%)的成员具有相同顺序的8个motif (motif 1 ~ 8),这与其他3个亚家族中缺少motif 3和7的成员有显著差异。此外,除EXPA外,所有亚家族基因均存在motif 10。因此,EXPA亚科的motif分布与其他3个亚科的motif分布存在显著差异,导致EXPB、EXLA和EXLB亚科具有更紧密的进化和系统发育关系。然而,大多数扩张素(77.8%;EXPB亚家族9个中有7个含有基序2,该基序在EXPA亚家族的所有扩展子中均存在,但在EXLA和EXLB亚家族中不存在。这一发现表明EXPA和EXPB具有更紧密的进化和系统发育关系,而EXPA与EXLA/EXLB亚家族的关系更为密切。因此,这表明属于同一亚科的膨胀蛋白的基序位置是保守的,而来自四个亚科的膨胀蛋白之间存在差异。
利用在线软件GSDS (http://gsds.cbi.pku.edu.cn/),以进一步了解它们可能的基因结构演化。因为一个ATG序列位于的第一个起始密码子附近GmEXLB10时,GSDS软件将后续ATG识别为起始密码子。因此,该基因的外显子-内含子组织之前有一个短的5 ' -UTR,而在其他基因中则没有(图2).我们的结果表明,同一家族的基因一般具有相似的外显子-内含子结构,外显子数量相同。例如,来自EXPB和EXLA亚家族的所有基因都包含4个外显子,来自EXPA亚家族的大多数基因包含3个外显子,而来自EXLB家族的基因包含5个外显子。反过来,这一发现支持了大豆中膨胀素基因的分类。此外,这一结果反映了四个亚家族在基因结构上的差异。此外,来自EXPA和EXLB亚家族的基因的外显子-内含子结构存在差异,几个基因含有不同数量的外显子。EXPA亚家族中的大部分扩增素基因含有3个外显子,其余的则含有2个或4个外显子。这种变异可能是由于在漫长的进化过程中外显子的丧失或获得造成的。此外,4个亚家族基因的外显子-内含子结构比较表明,EXPB和EXLA亚家族比EXPA和EXLB亚家族更为保守。
系统发育和结构分析结果表明,4个亚科均具有一定的保守性,亚科间也存在较大的多样性。大豆膨胀素超家族的基因序列高度一致,外显子-内含子结构相似,表明大豆膨胀素超家族在进化过程中经历了基因复制。因此,扩增素基因家族包含多个拷贝,这些拷贝在功能上可能部分或完全重叠,本研究对大豆基因扩增和表达模式的分析支持了这一假设。
扩增素基因扩增模式分析
基因复制被认为是基因组和遗传系统进化的主要驱动力之一[22].复制的基因为新基因的生成提供了原料,而新基因又促进了新功能的生成。节段复制、串联复制和转位事件,如转位和复制转位[23],被认为代表了三种主要的进化模式。在这些模式中,节段复制和串联复制被认为是植物基因家族扩张的两个主要原因[24].分段复制通过多倍体产生多个基因,然后染色体重排[25].它最常发生在植物中,因为大多数植物都是二倍体多倍体,在它们的基因组中保留了大量重复的染色体块[24].串联复制的特征是一个家族的多个成员出现在同一个基因间区或相邻的基因间区[26].在本研究中,我们将串联复制基因定义为单个染色体上相邻的同源基因,中间不超过一个基因。在这个分析中,我们主要关注分段复制和串联复制事件。为了更深入地了解大豆扩增素基因在这个庞大基因家族中的扩增模式,我们根据基因位点确定了串联复制簇,并搜索了植物基因组复制数据库[27]来定位分段复制的对。我们在共享区和邻近区寻找相邻的扩张蛋白基因。我们发现该家族75个基因中有11个(14.7%)为大豆串联重复序列(图1),表明串联复制促进了这个家族的扩张。我们还验证了区段复制事件在大豆膨胀蛋白超家族的进化中起重要作用的假设。我们搜索了PGDD中每个大豆膨胀素基因(http://chibba.agtec.uga.edu/duplication/),并发现68%(75个中的51个)的基因参与片段复制(图1).有趣的是,当我们将我们研究中发现的51个节段复制基因与Du等人的结果进行比较时。[28,29], 40 (78.4%;51个扩增素基因中有40个来自全基因组复制(WGDs),其余11个(21.6%;51个扩增素基因中有11个是单基因(GmEXPA2,GmEXPA8,GmEXPA17,GmEXPA21,GmEXPA22,GmEXPA23,GmEXPA29,GmEXPA43,GmEXPA45,GmEXPA47,GmEXPA49).这一发现表明,其余11个节段复制的扩张蛋白基因可能来源于独立的复制事件。因此,大豆在WGDs后保留了部分扩增素基因。先前的研究表明,WGD事件后以重复对的形式保留的基因往往属于特定的类别,如转录因子和大型多蛋白复合物的成员[30.- - - - - -32],这支持了本研究的结果。
同时,我们计算了这些串联重复基因对的4DTv(表2)使用PAML v4.4。4DTv值从0(最近复制的多肽)到0.5(具有古老进化历史的平行多肽)不等。结果表明,所有的4DTv值都在0.2左右,远远大于0。因此,我们推断串联复制的基因对可能有一个古老的进化历史。基因图谱显示,EXLB家族的两个大串联复制基因簇出现在5号染色体和17号染色体上;因此,17号染色体是大豆中扩增素基因密度最高的染色体。显然,复制事件,特别是串联复制,在一定程度上可能导致扩增素基因在染色体上的分布不均匀。此外,我们用Ks作为时间的代表,用保守的侧翼蛋白编码基因来估计片段复制事件的日期。表中列出了与扩增素基因相对应的所有片段复制事件的平均Ks值和估计日期3..除了独立的复制事件外,大豆的片段复制事件似乎发生在10-25 mya和40-65 mya这两个相对较近的关键时期。这些推论与大约发生在5900万年前和1300万年前的大豆基因组复制事件的年龄一致[21].这与我们的结果相一致,40 (78.4%;根据Du等人的数据,51个扩增素基因中有40个起源于WGDs。[28,29].因此,我们的研究结果表明,大多数参与片段复制的基因是全基因组复制事件的结果,而其余的可能是单独的片段复制事件的结果。
综上所述,这些结果表明扩增素基因超家族通过节段复制和串联复制,特别是节段复制进行了扩增。此外,大部分参与片段复制的基因在WGDs后被保留。
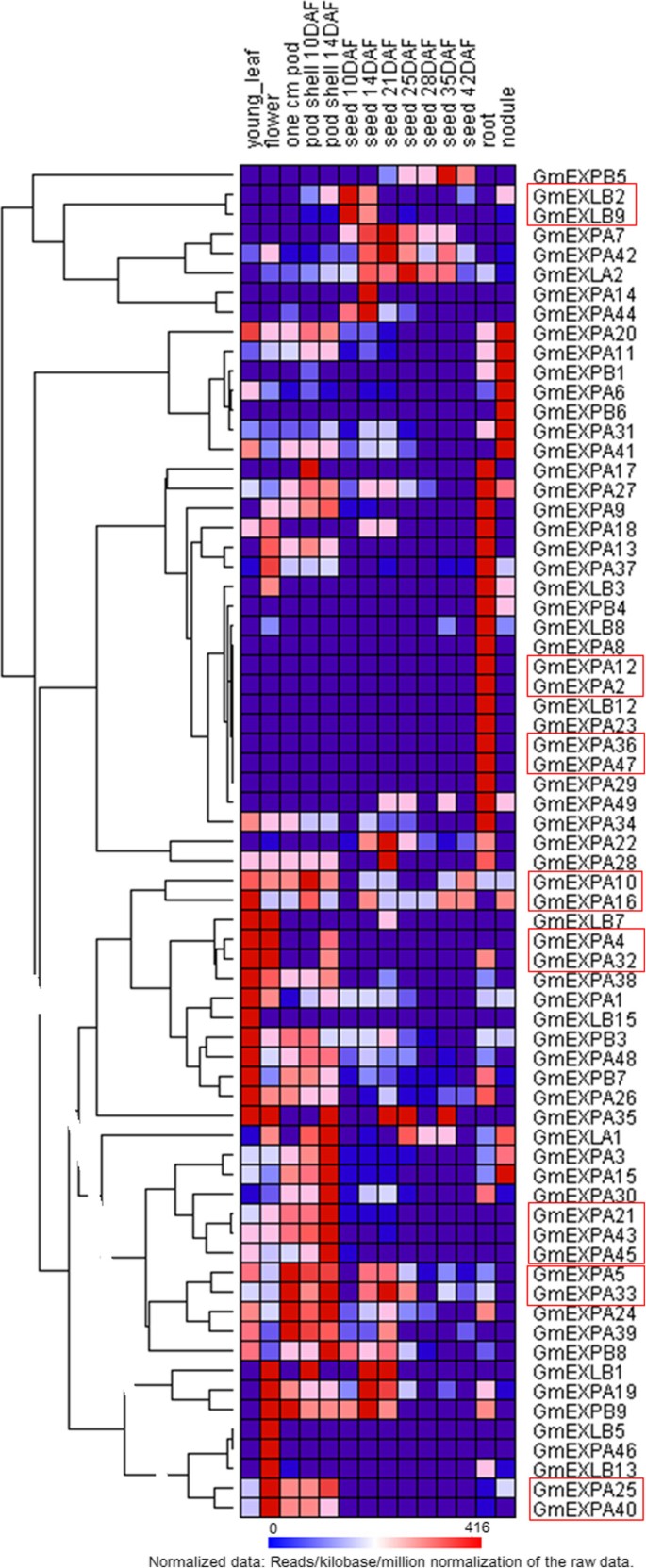
大豆膨胀素基因超家族的表达分析
最近开发的RNA-Seq网络工具,包括跨多个组织和器官的基因表达数据,允许对大豆的基因转录组图谱进行表征和比较。因此,在大豆扩增素基因的RNA-Seq图谱数据集中,可以很容易地识别出不同的转录丰度模式。大豆膨胀素基因的RNA-Seq图谱数据(附加文件)10)已从Soybase (http://soybase.org/soyseq/).然而,六种扩张素基因(GmEXPB2,GmEXLB4,GmEXLB6,GmEXLB14,GmEXLB10,GmEXLB11)缺乏RNA-Seq图谱数据,这可能表明这些基因是假基因,或者只在特定发育阶段或特殊条件下表达。RNA-Seq图谱分析表明,许多大豆扩增素基因的转录丰度较低。我们观察到扩张素基因转录物的积累与不同的组织相关,并且每个扩张素基因成员的表达模式不同(图3.).在大豆中,31%(75个中的23个)所分析的扩张蛋白在所有7种被检测的组织类型中都有组成性表达。这一发现表明,膨胀素在大豆发育过程中参与了多个过程。相比之下,大多数大豆膨胀蛋白表现出优先表达。RNA-Seq图谱数据显示,大多数(72%;75个大豆扩张蛋白中的54个)仅在一种组织类型中表现出明显的转录丰度峰值。这一结果表明,这些膨胀蛋白具有细胞壁松动蛋白的功能,并且局限于离散的细胞或器官。大约25%(总n =75)、20%、13%、11%、9%和7%的大豆膨胀素分别在根组织、种子组织、豆荚壳组织、叶组织、结节组织和花组织中表现出最高的转录物积累水平。首次报道大豆根特异性扩张蛋白基因[33]在大豆根中具有较高的表达水平,在大豆根中起重要作用。根据基因位点,它只对应GmEXPA37(Glyma17g37990)。如图所示3.,GmEXPA37仅在根组织的转录本丰度谱上有一个明显的峰值,这与以往的研究一致[33].根据Libault地图集[34](附加文件11),GmEXPA37倾向于在根毛中表达;因此,它可能有助于根毛的发育。这些基因的广泛表达说明大豆中的扩张素基因在正常情况下参与了所有器官和组织的发育。虽然在某些情况下,扩增素基因可能具有一般的重叠表达,但在其他情况下,表达可能是高度特异性的,并且仅限于单个器官或细胞类型。一些扩张素仅在单个组织中表达:7个基因(GmEXPA12,GmEXPA8,GmEXPA2,GmEXLB12,GmEXPA23,GmEXPA29,GmEXPA36,GmEXPA47)只在根中表达;三个基因(GmEXPA7,GmEXPA14,而且GmEXPB5)只在种子中表达;两个基因(GmEXLB5而且GmEXPA46)只在花中表达;一个基因(GmEXPB6)仅在结节中表达;一个基因(GmEXLB15)只在叶片中表达。我们的分析表明这些基因可能是组织特异性的,或者至少是优先表达的。有趣的是,这些结果表明,扩张素基因超家族的更多基因可能在根中特异性或优先表达。另一个热图(附加文件11)提供了更多的根中优先表达的基因信息。Libault图谱集中于地下组织,提供了更多地下组织中高表达基因的信息,特别是根、根毛、根尖。
此外,在热图中聚集在分支的扩增素基因表现出相似的转录丰度谱。然而,这些基因中的大多数在系统发育树中并不是聚集在一起的,并且具有相对的系统发育差异。只有几个小的系统发育演化支具有大致相似的转录物丰度谱,并在热图上用红色框线标出(图3.).大豆扩展蛋白具有较高的序列相似性和共享表达谱,是评价大豆基因功能的良好候选基因。因此,红色框中的基因可能在相同的组织中具有相似的功能。例如,GmEXPA2而且GmEXPA12,在系统发育树中聚在一起,序列相似性较高,仅在根组织中表达,这表明这两个基因在根组织中可能具有相同的功能。
转录组图谱显示,大豆扩张素超家族的4个亚家族均存在差异表达,这可能与扩张素基因启动子区域的差异有关。基因上游区域的启动子在基因表达的发育和/或环境调控中发挥关键作用[35].因此,独联体-作用元件可能提供有关基因表达调控机制的有用信息。计算工具PlantCARE [36,用来识别独联体大豆扩增素基因翻译起始密码子上游1500 bp DNA序列中的-作用元件。四种类型独联体-作用元件在大豆膨胀素基因超家族的启动子区显著丰富(附加文件12).第一种独联体启动子区富集的-作用元件是光响应元件,包括G-box [37,38],第4栏[39]和盒子I [40].在大豆膨胀蛋白基因中,G-box的丰度最高,平均为1.386个拷贝,而在EXLB中,G-box的丰度较低,平均为0.8000个拷贝。另一类顺式扩增素基因启动子区富集的作用元件是植物激素反应元件,包括tca元件[41], TGA-element [42]和GARE-motif [43].水杨酸反应性tca元素似乎是最丰富的激素相关元素独联体说明水杨酸调控了部分大豆膨胀素基因的表达。大豆膨胀素基因中tga元件和GARE-motif的丰度表明生长素和赤霉素也在调节大豆膨胀素基因的表达中发挥作用。其他元素也与生长素或赤霉素反应性有关,如AuxRR-core [44], TGA-box [45], P-box [46],及TATC-box [47].这些结果与之前的研究一致,即一些扩张素受到生长素的调控[48,49]和赤霉素[50,51].第三丰富的独联体-作用元件类包含响应外部环境压力的元件。我们观察到大多数大豆膨胀素基因似乎含有ARE [52], MBS [53], hse [54],以及富tc元素[52].ARE是参与厌氧诱导的元素;因此,我们推测扩张蛋白表达的厌氧调控可能与组织或发育阶段有关。干旱响应元件MBS在启动子区也很丰富。除了少数例外,扩增素基因包含该元素的至少一个副本(附加文件12).这些结果与膨胀素活性受到包括干旱在内的各种非生物应激因素影响的事实是一致的[55,56]及水浸[57- - - - - -61].昼夜节律元素,涉及昼夜节律控制[62的第四类独联体在大豆膨胀素基因的启动子区大量发现的一种作用元件。PlantCARE分析显示大豆膨胀素基因含有昼夜因子,这可能表明大豆膨胀素具有明显的昼夜表达模式[63].启动子分析显示存在多样性独联体大豆膨胀素基因超家族上游区域的作用元件。这一发现进一步支持了扩张蛋白在与细胞壁修饰相关的广泛发育过程中的各种功能作用。
上述结果表明,大豆75个扩增素基因在4个亚家族中的表达存在差异,无论是在转录物的丰度上还是在正常生长条件下的表达模式上。
大豆膨胀蛋白的功能分化分析
使用DIVERGE v2.0程序进行后验分析,推断大豆膨胀素超家族亚家族之间的功能分化。计算每个位点发散的后验概率(Qk)来预测某些关键氨基酸位点(CAASs)的位置[64]与功能分化高度相关。在我们的研究中,估计了两种类型的功能分化。i型功能分化是指基因复制后导致进化速率发生位点特异性转移的进化过程,ii型功能分化是指位点特异性氨基酸理化性质的转移。这些方法由于对同义位点的饱和不敏感,已被广泛应用于各种基因家族的研究[64- - - - - -66].这一估计是基于由75个大豆膨胀蛋白基因的所有蛋白质序列构建的邻居连接树。相比之下,只有2个成员的EXLA亚家族被排除在外,因为该方法无法分析少于4个序列的组。将其余三个亚家族的副同源扩增素基因进行两两比较,并估计每个序列位置的氨基酸进化速率。我们的研究结果(表4)表明,3个膨胀蛋白亚家族之间的i型功能分歧系数(θI)具有很强的统计学意义(p < 0.01), θI值在0.498 ~ 0.783之间。因此,显著的位点特异性变化改变了超家族成员扩张的选择限制,导致了多样化后亚群特异性功能进化。亚家族(EXPA/EXLB)之间的ii型功能差异(θII)很明显,θII值为0.136 (p < 0.05),这表明氨基酸性质发生了根本性的变化。EXPA/EXPB和EXPB/EXLB之间的II型函数散度θ系数不明显,θII值小于0,但标准误差较大。因此,对于大豆膨胀素超家族亚家族的功能分化而言,Type-I和Type-II的相对重要性似乎有所不同。
此外,我们预测一些关键的氨基酸残基是导致功能分化的原因,从每个比较的Qk中推导出合适的临界值。考虑到使用经验Qk值0.8作为截断值时,DIVERGE2识别出了太多与功能差异相关的残基(数据未显示),我们使用Qk > 0.95来预测CAASs,以排除其他位点进行进一步分析。结果表明,通过type-I功能分歧分析,共预测了19个CAASs,而type-II功能分歧分析则识别了63个概率较高的氨基酸位点(Qk > 0.95),这在一定程度上反映了进化速度和氨基酸性质的根本性转变。此外,有12个氨基酸对i型和ii型的功能分化都至关重要,这表明在这些氨基酸位点上,进化速度的改变和氨基酸理化性质的改变是共同发生的。因此,在进化过程中,这些位点可能在功能分化中发挥了重要作用。此外,我们还注意到预测位点的数量(表4)在每一对中,i型和ii型功能分化存在差异;即,在每个亚科对中,更多caas被鉴定为ii型功能分化。因此,两组基因之间的功能差异主要是由于氨基酸理化性质的快速变化,其次是进化速度的变化。
此外,与EXPA/EXPB和EXPB/EXLB相比,EXPA/EXLB具有较大的功能分歧系数(θI和θII),与功能分歧相关的位点也更多。因此,与EXPA/EXPB和EXPB/EXLB相比,EXPA和EXLB之间存在的功能差异更为显著,尽管EXLB的任何成员尚未确定其生物学或生化功能[8].此外,根据功能分化系数和鉴定出的caas数量,我们还推断EXPA/EXPB和EXPB/EXLB之间的功能分化程度较小。因此,与EXPA和EXLB相比,EXPB和EXLB具有更紧密的系统发育关系,motif分析也表明了这一点。基序分析表明,EXPA亚家族的基序组织与其他两个亚家族明显不同,而EXPB和EXLB亚家族的基序类型和数量相似。
正选择分析
为了验证大豆膨胀素基因正选择的假设,我们使用PAML v4.4软件包的CODEML程序中的位点模型和分支位点模型[67].计算非同义(dN或Ka)突变与同义(dS或Ks)突变的替代率(dN/dS或ω)。中性选择的基因Ka/Ks比值为1,负选择的基因Ka/Ks比值<1,正选择的基因Ka/Ks比值为>1 [68].在位点模型中,实现密码子位点模型M0、M3、M7和M8,使用似然比检验在氨基酸位点上是否存在可变ω (dN/dS)比值。M0是单比模型,在所有地点假设一个ω比。在离散模型(M3)中,将每个位点的概率(p0, p1, p2)分别提交给净化、中性和阳性选择,并从数据中推断出它们对应的ω比值(ω0, ω1, ω2)。Beta模型(M7)是正选择的零检验,假设Beta分布ω在0和1之间。最后,Beta & ω模型(M8)添加一个相同比例的额外类ω1 [69].在我们的研究中,我们选择了两对模型(M0/M3和M7/M8)进行比较(表2)5).首先对模型M0和M3进行比较,采用dN/dS比值值的密码子位点异质性检验,两倍对数似然差2Δℓ= 560表明结果具有较强的统计学意义(p < 0.01),说明大豆膨胀素超家族存在较大的选择压力;即大豆膨胀素经历了强烈的正向选择。M3和M0的比较表明,没有一个密码子位点受到正选择的影响(ω > 1)。相比之下,M7 (beta)和M8 (beta + ω > 1)的比较,被认为是最严格的正选择检验[70],表明~0.001%的密码子落在2.02644的估计ω值范围内(这表明存在正选择)。基于贝叶斯后验概率,从M8模型中筛选出14个密码子候选位点(42G、43 T、123H、146S、153R、166S、172 L、184G、186A、190S、195 M、196P、198S和203Q)进行正选择。其中8个阳性选择位点处于0.01显著水平,其余位点处于0.05显著水平。在位点模型中鉴定的四个氨基酸残基(172l、184G、190S和196P)也导致了功能分化;即172l和184G是i型功能分化的主要原因,190S和196P是ii型功能分化的主要原因。
在分支位点模型中,ω允许在蛋白质的不同位点和树上的不同分支之间变化,目的是检测只影响特定谱系中少数位点的正选择[71].正在进行积极选择测试的分支被称为前景分支,而树上剩余的分支被称为背景分支。BEB方法用于计算位点类的后验概率(Qks),如果LRT表明前景分支上存在正选择下的密码子[67].选取各大豆膨胀素亚科作为前景分支,进行阳性选择试验。研究结果(表6)表明,4个亚科间均存在分化的阳性选择。当选择EXPB、EXLA或EXLB作为前景分支时,前景ω值相当大,几乎所有密码子候选位点都被识别出来;但所有密码子的后验概率均不高于0.95,除192 T选择EXLB作为前景分支后验概率为0.984外。当选择EXPB或EXLA亚家族作为前景分支时,未发现后验概率大于0.95的位点。然而,积极选择通常只在少数几个位点上发生,且发生在较短的进化时间内;因此,信号可能会被广泛的负选择所淹没[72].而当选择EXPA作为前支时,前景ω值(1.32036)要低得多,共识别出10个后验概率大于0.95的位点(56Q, 133 L, 166S, 169 N, 186A, 172 L, 174 T, 190S, 198S, 203Q)。
这些结果表明四个亚科存在不同的选择限制。与EXPA和EXLB亚家族相比,EXPB和EXLA亚家族更为保守。此外,EXPA亚家族可能是四个亚家族中最强的阳性选择,因为在这个亚家族中检测到最显著的阳性位点。
讨论
大豆膨胀素基因超家族的起源
最近的研究认为70% ~ 80%的被子植物都经历过复制事件[73- - - - - -76].例如,90%和62%拟南芥而且栽培稻基因座经历了重复事件[22].作为一种古老的多倍体,大豆的基因组高度复制,近75%的基因出现在多个拷贝中[21].目前的研究揭示了大豆扩张素基因家族的复制模式。11个基因被鉴定为串联重复序列,表明串联重复也促进了大豆扩张素基因超家族的扩增。此外,还发现了51个基因是由片段复制进化而来的,这表明片段复制可能在大豆基因组中的扩增素基因扩增中发挥了关键作用。基因组测序结果显示,大豆全基因组复制(WGD)大约发生在5900万年前和1300万年前(MYA),这与本研究结果一致。我们推测扩增素基因家族的扩增与WGD事件一起发生,并且这些基因在进化过程中被保留。先前的研究表明,快速的功能分化和重复基因的偏倚表达似乎是促进它们在基因组中保留的主要因素[77- - - - - -81].在我们的研究中,四个亚家族之间存在明显的功能差异,重复的基因表现出不同的表达。例如,在一个重复的基因对中,GmEXPA30&GmEXPA34,两个基因在基因组复制事件后被保留,仅与GmEXPA30在叶片中表达,表示偏表达。类似的情况也在其他片段复制的基因对中观察到,如GmEXPA4&GmEXPA32,GmEXPA6&GmEXPA31,GmEXPA18&GmEXPA28.这些结果进一步验证了我们的假设,即大部分区段复制的大豆膨胀素基因从基因组复制事件中保留下来。扩增素基因超家族的扩增模式分析表明,大豆基因组发生了大规模复制。节段复制和串联复制都是扩增素基因超家族扩增的重要因素。
我们还分析了中膨胀素超家族的膨胀模式拟南芥(附加文件13)和大米(附加文件14).本研究结果显示,50%(18 / 36)的基因参与了片段复制,27.8%(10 / 36)的基因参与了串联复制拟南芥.相比之下,27.6%(16 / 58)的基因参与了片段复制,55.2%(32 / 58)的基因参与了串联复制。在大豆中,68%(51 / 75)的基因参与片段复制,14.7%(11 / 75)的基因参与串联复制。因此,我们观察到片段复制和串联复制在大豆扩展蛋白超家族的扩展中都发挥了重要作用。拟南芥和大米。以往的研究表明,编码转录因子和核糖体成分的基因在四倍体后明显过度保留[82].然而,影响应激反应的基因在串联复制后有较高的保留概率[83].扩增素基因与细胞壁增大有关。然而,虽然这些基因不是转录因子、核糖体成分或影响应激反应的基因,但它们通过串联复制和节段复制进行了扩展,而不仅仅是一种形式的复制。更有趣的是,我们还注意到这三个物种都表现出了物种特有的扩张模式。例如,节段复制似乎是两个双子代的扩张素基因超家族的主要扩张形式,拟南芥和大豆。单子叶水稻扩增素基因家族的扩增方式以串联复制为主。
水稻中exb家族规模较大,大豆中EXLB家族规模较大
先前的研究表明,β-扩张素基因在草类中数量特别多,表达量特别丰富,但在双子叶中也发现数量较少[84].我们的研究结果比较了大豆中扩张素基因家族的大小,拟南芥与之前的研究结果一致。水稻中的EXPB家族比大豆和大豆中的EXPB家族大得多拟南芥.我们还发现大豆中的EXLB家族比大豆大得多拟南芥和米饭。然而,在所有三个物种中,EXPA家族的体型最大。以往的研究表明,家族大小的主要变化和大多数基因家族的分布受到串联复制和节段复制的影响[24].因此,我们比较了三个物种中四个亚科的复制事件(表7).亚科和种间变异较大。水稻中EXPB家族的规模大得多可能是由于这个家族与其他两个物种相比以不同的速度扩张造成的。水稻EXPB家族基因均参与片段复制或串联复制;而在大豆和大豆中,EXPB家族只有部分基因参与片段复制和串联复制拟南芥都参与了重复事件。另一方面,从适应性角度看,水稻的EXPB亚家族在重复事件后可能保留较多,而大豆和大豆的EXPB亚家族在重复事件后可能保留较多拟南芥容易发生大规模的基因丢失,导致保留的EXPB基因减少。因此,水稻中EXPB家族的扩大和保留程度越高,它的个头就越大。同样,大豆中EXLB亚家族的扩展和保留程度高也可能解释了其规模大得多的原因。结果表明,串联复制是大豆EXLA亚家族扩展的主要原因,EXLB中有74%的基因参与串联复制,并可能在较长的进化周期中被保留。然而,EXLB亚家族的基因拟南芥而水稻不参与节段或串联复制事件。因此,片段复制和串联复制事件都促进了EXLB亚家族在大豆中的不断扩大。
近年来的研究表明,玉米中的Zea m1 (EXPB1)和其他禾本科植物中的同源1族花粉过敏原在花粉中含量很高。这些基因可能只在草的细胞壁上诱导延伸,但对双叶菊的细胞壁无效,通过软化母细胞壁来帮助花粉管穿透柱头和花柱[9,84].此外,β-expansin基因在草类中表达尤其丰富[84].在本研究中,我们推断出水稻EXPB亚家族的大小在漫长的进化过程中不断增加,以适应特定的功能需求。或者,水稻EXPB亚家族的更多基因可能在水稻发育中具有更高程度的重复后保留,以发挥重要功能。相比之下,其他两个物种的EXPB亚家族的基因可能在进化过程中经历了大规模的基因丢失。大豆中EXLB亚家族更大的尺寸也可能反映了对某些功能或环境的适应。因此,EXLB成员在大豆发育过程中可能具有特殊的作用;然而,实验证据尚未证实它们在细胞壁中的活性[8].
功能分化与正向选择分析
基因复制被认为是基因组和遗传系统进化的主要驱动力之一[22].通常情况下,一个氨基酸残基在一个重复基因中高度保守,但在另一个重复基因中高度可变[85].氨基酸位点突变频繁,突变的积累可能导致重复基因的功能分化[30.,80,86,87].通过功能分化分析,关键氨基酸位点(表4)。这些位点是四个大豆亚科之间功能分化的主要原因。快速的功能分化和重复基因的偏倚表达有望促进两个同源基因或来自WGD的同源基因的保留[77- - - - - -81].在我们的研究中,expansin基因超家族经历了大规模的基因复制,许多基因在WGD事件后被保留。重复基因的突变,以及随后对它们的选择限制,预计将导致功能分化。在分子水平上,导致适应度降低的氨基酸变化通过负选择被去除,而增加适应度的变化通过正选择被保留[88].通过正选择分析,经过强正选择的氨基酸位点(表5,6)。最后,我们鉴定出7个同时负责功能分化和正向选择的位点(56Q、133 L、172 L、184G、190S、192 T和196P),表明这些位点在大豆扩张素基因超家族的进化历史中具有重要意义。
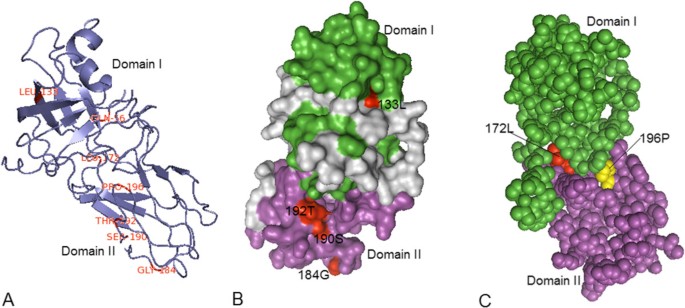
我们使用了瑞士模式[89- - - - - -91的三维结构模型GmEXPA1通过同源建模,并在其上标记了7个关键氨基酸位点。三维结构显示,172l和196P位于两个畴接触的表面(图4).与Zea m1的晶体结构比较20.],通过对两个序列的Clustal比较,我们推断172 L可能参与了膨胀蛋白两个结构域的接触,因为它对应于Zea m1的164 L,而Zea m1位于与两个结构域接触相关的疏水区域。因此,我们推断非极性残基196P可能也参与了两个畴的接触。基于蛋白质表面存在保守的芳香残基和极性残基,推测结构域II是一个多糖结合结构域[18].有趣的是,蛋白质表面有4个关键氨基酸位点(133 L, 184G, 190S, 192t)4);因此190S和192t可能参与多糖结合。
基于大豆膨胀蛋白(GmEXPA1)与Zea m1 (protein Data Bank [PDB] code: 2HCZ)相似性的三维结构模型构建。在不同程度上显示了7个负责功能分化和积极选择的关键氨基酸位点(一),(B),(C).该图形是使用瑞士模型和pyMOL程序生成的。(一)7个关键氨基酸位点的三维结构整体视图。这七个地点用红色标注。(B)gmexp1表面关键氨基酸位点的观察。位于分子表面的四个负责功能分化和积极选择的氨基酸位点被标记为红色。在这些氨基酸位点中,190S和192t可能是多糖结合结构域II的关键位点。灰色区域代表N端和C端。(C)与两个结构域接触相关的关键氨基酸位点。只有两个结构域较好地表现出172l和196P两个残基,这可能与两个结构域的接触有关。
虽然我们在本研究中没有绘制其他只负责功能分化和积极选择的位点,但我们基于的三维结构分析了这些位点的位置GmEXPA1(表8).结构域II(假定的多糖结合结构域)中负责功能分化或正向选择的氨基酸位点数量明显大于结构域I,表明结构域I相对于结构域II具有保守性。这种差异可能与功能适应性有关。前人研究表明,花粉expb(第1类过敏原)对禾本科植物细胞壁有明显的松动作用,而对双子叶植物细胞壁无明显松动作用;然而,expa的情况正好相反。因此,这两种形式的膨胀蛋白似乎针对细胞壁的不同组分[13,92].因此,假定的多糖结合结构域(结构域II)可能已经进化到适应细胞壁的不同成分,从而促进功能分化和更快的进化。
在C端未发现负责功能分化和阳性选择的位点,表明C端是严格保守的。相反,在N端发现了6个负责功能分化的氨基酸位点和3个负责正选择的氨基酸位点,说明该末端有助于功能分化。此外,N端扩张素的变化可能促进了扩张素对不同功能需求的适应性。玉米EXPB1基因的n端延伸包含一个基序(VPPG-PNITT),该基序在第1组草花粉过敏原中始终存在,只有很小的变异,但在其他expb中没有[20.].虽然这种n端延伸的功能尚不清楚,但它可能有助于蛋白质识别、运输、包装和花粉分泌装置的加工[20.].
结论
先前的研究表明,扩张素基因家族的成员在细胞扩大和各种其他发育过程中发挥重要作用。本研究结果表明,串联复制和节段复制都有助于扩张素基因家族在大豆中的扩展。通过比较植物中扩增素基因家族的扩增模式,鉴定了种间的特异性扩增特征拟南芥大豆和大米。节段复制似乎是两种双子代扩张素基因超家族的主要扩张形式,拟南芥和大豆。相比之下,串联复制似乎是单子叶水稻扩增素基因家族的主要扩增形式。此外,正选择可能是复制基因功能分化的主要驱动力,这可能是促进植物在其进化历史中对各种应激源做出反应的关键。此外,不同的选择约束可能影响了四个亚科的进化。本研究结果将进一步加深对大豆膨胀素基因进化过程的认识,并有助于加强对大豆膨胀素这一重要模型系统的功能基因组研究。
方法
大豆膨胀蛋白超家族基因的鉴定
扩增素超家族的35个基因序列拟南芥收集于扩展新中心(http://www.personal.psu.edu/fsl/ExpCentral/),并单独用于攻击Phytozome v9.1中的大豆基因组数据库(http://www.phytozome.net/soybean).E值≤1e-10的序列作为候选蛋白。最后,Pfam (http://www.sanger.ac.uk/Software/Pfam/)和简单模块化体系结构研究工具(SMART;http://smart.embl-heidelberg.de/smart/batch.pl),证实每个预测的扩张蛋白序列都是一个扩张蛋白超家族成员,共享结构域I (PF03330)和结构域II (PF01357)。人工去除冗余基因(只有两个结构域之一的基因,或ORF未整合的基因)。每个查询序列都找到了位于不同染色体上的假定基因。一个包含目标基因所有信息的数据文件(包括染色体上的位置、基因组序列、完整的CDS序列、蛋白质序列和翻译起始密码子上游的1500 bp核苷酸序列)从Phytozome网站(http://www.phytozome.net).使用SignalP 4.1服务器(http://www.cbs.dtu.dk/services/SignalP/).利用ExPASy Compute pI/Mw工具计算理论pI(等电点)和Mw(分子量)值[93- - - - - -95].
系统发育遗传树构建及结构分析
无根邻接的构造[96]利用分子进化遗传学分析(MEGA) 5.0程序进行系统发育树和bootstrap分析[97].使用默认设置的MEME统计识别副同源扩张蛋白的基序;然而,要找到的图案的最大数量被设置为10个。利用在线软件GSDS (http://gsds.cbi.pku.edu.cn/).
扩增素基因扩增模式分析
大豆扩张蛋白基因在染色体上呈分散分布。此外,根据它们的位点,几个基因明显相邻。因此,我们重点研究了分段复制和串联复制的过程。根据Schauser等人[98],一种检测片段复制事件的有效方法是在每个家族成员的邻近区域识别额外的副同源蛋白对。因此,我们在植物基因组复制数据库(Plant Genome Duplication Database)中搜索每个扩展蛋白成员的同构块[27].在植物基因组复制数据库中检索每个扩增蛋白成员,以确定其是否参与分段复制。扩增素基因在大豆基因组中的串联复制是通过检查它们在单个染色体上的物理位置来确定的。串联复制基因被定义为在单个染色体上相邻的同源基因,中间插入的基因不超过一个。例如,Glyma17g15670/Glyma17g15680/Glyma17g15690/Glyma17g15710经鉴定为串联重复基因簇。
确定复制事件的日期
植物基因组复制数据库直接为Ka和Ks提供了相应的复制基因对。当确定分段复制事件的日期时,所有Ks值在0到1之间的可用锚点都被用来计算平均Ks。然而,少于3个锚点的重复基因对被删除。重复事件的大致日期是用T = Ks/2λ [99],其中Fabaceae的同义替代率(λ)为6.1 × 109[One hundred.].对于串联复制事件,基因对的蛋白质序列在Clustal X 1.83和PAL2NAL [101]被用来指导最终的编码序列(CDS)比对。Ks是每个位点同义取代的数量,在Codeml程序系统发育分析中使用最大似然(PAML) 4.4 [67]在所有对准间隙被消除后。同时,PAML也计算了4倍同义密码子位置的转换率4DTv。
RNA-Seq图谱和启动子分析
引入RNA-Seq数据进一步分析扩增素基因的表达,数据来源于Soybase (http://soybase.org/soyseq/) [102].的独联体调控基因表达的-作用元件分布在编码区上游300-3000 bp处,PlantCARE也考虑了序列限制[36].从Phytozome下载了每个大豆膨胀素基因编码区上游共计1500 bp的核苷酸序列,并提交给PlantCARE进行分析insilico分析。
功能分歧估计
采用DIVERGE2软件检测大豆膨胀素亚家族成员之间的功能分化[103].计算了大豆膨胀素亚家族之间的i型和ii型功能分歧系数θI和θII。如果θI或θII显著大于0,则意味着基因复制和/或物种形成后发生了位点特异性的选择性约束改变或氨基酸理化性质的根本转变[103].此外,使用特定位点的后验分析来预测对功能分化至关重要的氨基酸残基。在该分析中,大后验概率(Qk)表明两个聚类之间一个位点的功能约束(或进化速率)和/或氨基酸性质的根本变化很可能是不同的[103].
积极选择测验
正向选择采用PAML 4.4中的Codeml程序的最大似然方法进行研究[67],在站点模型和分支站点模型下。首先,通过PAL2NAL获得了大豆扩张蛋白的精确核苷酸序列和相关的多蛋白序列比对[101].由此得到的密码子比对和NJ树随后被用于PAML包中的Codeml程序中,以计算每个位点的dN/dS(或ω)比值,并测试不同的进化模型。
在站点模型中,选择PAML中的两对站点模型,使用似然比检验(LRT)检验正选择,并使用朴素经验贝叶斯(NEB)和贝叶斯经验贝叶斯(BEB)估计方法在正交组中识别正选择的站点。首先,对模型M0(一个比率)和M3(离散)进行比较,使用dN/dS比率值ω的密码子位点之间的异质性检验。第二次比较是M7 (beta) vs M8 (beta + ω >1);这种比较是积极选择的最严格的检验。70].当LRT表明正选择时,采用BEB方法计算每个密码子来自模型M3和M8下正选择位点类的后验概率[72].
分支位点模型假设密码子位点之间的ω比值不同,序列中有4个位点类。第一类位点在所有世系中都高度保守,ω比ω0很小。第二类包括中性或弱约束位点,其中ω = ω1, ω1接近或小于1。在第三和第四类中,背景分支显示ω0或ω1,而前景分支显示ω2,其值可能大于1。在构建LRTs时,零假设修正了ω2 = 1,允许站点在背景谱系的负选择下从约束中释放出来,并在前景谱系上中性地进化。备择假设约束ω2≥1 [72,104].使用Yang等人描述的BEB方法计算与特定密码子落在受正选择影响的位点类相关的后后概率。[105].
支持数据的可用性
支持本文的数据集包括:
附加文件2。大豆扩增素基因超家族的蛋白质序列数据,拟南芥和米饭。
附加文件3。大豆膨胀素基因超家族的编码序列数据。
附加文件4。大豆扩张素基因超家族的基因组序列研究。
附加文件5。大豆扩张蛋白基因超家族翻译起始密码子上游1500 bp的核苷酸序列。
附加文件8。大豆膨胀蛋白的多序列比对。
附加文件10。扩增素基因的RNA-Seq图谱数据。
参考文献
- 1.
科斯格罗夫DJ,李LC,赵HT,霍夫曼-本宁S,摩尔RC,布莱克D:膨胀子的成长世界。植物生理学报,2002,43(12):1436-1444。10.1093 /卡式肺囊虫肺炎/ pcf180。
- 2.
McQueen-Mason S, Durachko DM, Cosgrove DJ:诱导植物细胞壁延伸的两种内源性蛋白质。《植物细胞学报》,1992,4(11):1425-1433。10.1105 / tpc.4.11.1425。
- 3.
李zc, Durachko DM, Cosgrove DJ:一种诱导体外壁延伸的燕麦胚轴壁蛋白,与黄瓜下胚轴的一种类似蛋白在抗原上相关。植物学报,1993,19(3):349-356。
- 4.
Keller E, Cosgrove DJ:生长番茄叶片中的膨胀素。植物学报,2004,18(6):366 - 366。10.1046 / j.1365 - 313 x.1995.8060795.x。
- 5.
吴勇,Sharp RE, Durachko DM, Cosgrove DJ:低水势下玉米初生根的生长维持与细胞壁延伸特性、膨胀素活性和细胞壁对膨胀素的敏感性有关。植物科学进展,2004,27(3):344 - 344。
- 6.
Kende H, Bradford K, Brummell D, Cho HT, Cosgrove DJ, Fleming AJ, Gehring C, Lee Y, McQueen-Mason S, Rose JKC, Voesenek LACJ:基因和蛋白质膨胀蛋白超家族成员的命名法。植物分子生物学,2004,55(3):311-314。10.1007 / s11103 - 004 - 0158 - 6。
- 7.
Sampedro J, Cosgrove DJ:超级家族的扩张。中国生物医学工程学报,2005,6:12-
- 8.
Sampedro J, Carey RE, Cosgrove DJ:基因组历史阐明了扩张素超家族的进化:来自杨树基因组和松树ESTs的新见解。植物保护学报,2006,29(1):1- 6。10.1007 / s10265 - 005 - 0253 - z。
- 9.
Cosgrove DJ:植物细胞壁因膨胀素而松动。自然科学学报,2000,27(2):332 - 332。10.1038 / 35030000。
- 10.
灰色- mitsumune M, Mellerowicz EJ, Abe H, Schrader J, Winzéll A, Sterky F, Blomqvist K, McQueen-Mason S, Teeri TT, Sundberg B:次生木质部中丰富的扩张素属于α-扩张素基因家族的A亚群。植物科学进展,2004,29(3):344 - 344。10.1104 / pp.104.039321。
- 11.
张晓明,李志刚,张晓明,杨晓明,等。乙烯促进植物叶片脱落过程中扩增素活性和基因表达的变化Sambucusnigra.应用物理学报,2005,26(3):344 - 344。10.1093 / jxb / eri076。
- 12.
陈芳,陈志军,陈志军:番茄种子萌发过程中一种扩张蛋白的表达与胚乳弱化有关。植物营养学报,2000,27(3):344 - 344。10.1104 / pp.124.3.1265。
- 13.
科斯格罗夫DJ, Bedinger P, Durachko DM:草花粉作为细胞壁松动剂的I类过敏原。自然科学进展,2004,26(3):349 - 349。10.1073 / pnas.94.12.6559。
- 14.
Pezzotti M, Feron R, Mariani C:授粉调节雌蕊特异性β-膨胀素PPAL基因的表达。植物分子生物学,2002,49(2):187-197。10.1023 /: 1014962923278。
- 15.
Li LC, Cosgrove DJ:草I类花粉过敏原(β-膨胀素)缺乏蛋白酶活性,不会通过蛋白水解引起壁松动。中国生物医学工程学报,2001,26(3):344 - 344。10.1046 / j.1432-1327.2001.02336.x。
- 16.
McQueen-Mason SJ, Cosgrove DJ:扩张蛋白对细胞壁的作用模式(分析细胞壁水解、应力松弛和结合)。植物营养学报,2004,27(1):1 - 8。
- 17.
McQueen-Mason SJ, Fry SC, Durachko DM, Cosgrove DJ:木葡聚糖内转糖基酶与黄瓜下胚轴细胞壁延伸的关系。植物学报,1993,19(3):327-331。
- 18.
科斯格罗夫DJ:高压环境下的放松:可扩展细胞壁和细胞扩大的分子基础。植物细胞学报,1997,9 (7):1031-10.1105/tpc.9.7.1031。
- 19.
Kerff F, Amoroso A, Herman R, Sauvagea E, Petrellab S, Filéea P, Charliera P, Jorisa B, Tabuchic A, Nikolaidisc N, Cosgrovec DJ:晶体结构和活性枯草芽孢杆菌YoaJ (EXLX1),一种细菌扩张素,促进根定殖。自然科学进展,2008,29(4):344 - 344。10.1073 / pnas.0809382105。
- 20.
Yennawar NH, Li LC, Dudzinski DM, Tabuchi A, Cosgrove DJ:玉米花粉变应原EXPB1 (Zea m1)的晶体结构和活性。自然科学进展,2006,39(4):344 - 344。10.1073 / pnas.0605979103。
- 21.
Schmutz J, Cannon SB, Schlueter J, Ma J, Mitros T, Nelson W, Hyten DL, Song Q, Thelen JJ, Cheng J, Xu D, Hellsten U, May GD, Yu Y, Sakurai T, Umezawa T, Bhattacharyya MK, Sandhu D, Valliyodan B, Lindquist E, Peto M, Grant D, Shu S, Goodstein D, Barry K, Futrell-Griggs M, Abernathy B, Du J, Tian Z, Zhu L:古多倍体大豆基因组序列。自然科学学报,2010,38(4):344 - 344。10.1038 / nature08670。
- 22.
Moore RC, Purugganan MD:复制基因进化的早期阶段。自然科学进展,2003,30(4):344 - 344。10.1073 / pnas.2535513100。
- 23.
孔海华,李志强,马海华,李志强,李志强,李志强:植物基因复制模式的研究SKP1被子植物中的基因家族:基因快速诞生的多种机制的证据。植物学报,2007,30(5):873-885。10.1111 / j.1365 - 313 x.2007.03097.x。
- 24.
卡农SB, Mitra A, Baumgarten A, Young ND, May G:片段和串联基因复制在大基因家族进化中的作用拟南芥.中国生物医学工程学报,2004,4(1):10-10.1186/1471-2229- 10。
- 25.
王于J, J,林W,李,李H,周J, Mi P,董W,胡年代,曾C,张J,张Y,李R,徐Z,李,李X,郑H, Cong L林L,阴J,耿J,李G, J,刘J, Lv H,李J,王J,邓Y, L, Shi X:的基因组栽培稻重复的历史。公共科学图书馆,2005,3 (2):e38-10.1371/journal.pbio.0030038。
- 26.
Ramamoorthy R, Jiang SY, Kumar N, Venkatesh PN, Ramachandran S:不同非生物和植物激素处理下水稻WRKY基因家族的综合转录谱分析。植物生理学报,2008,49(6):865-879。10.1093 /卡式肺囊虫肺炎/ pcn061。
- 27.
唐华,王霞,王晓明,王晓明,王晓明,王晓明。被子植物六倍体基因图谱分析。基因组学报,2008,18(12):1944-1954。10.1101 / gr.080978.108。
- 28.
杜娟,田忠,隋勇,赵敏,宋强,陈建平,马娟:古多倍体大豆复制基因的分离、保留和表达模式与周围熵效应的关系。植物细胞学报,2012,24(1):21-32。10.1105 / tpc.111.092759。
- 29.
尹刚,徐辉,肖松,秦勇,李勇,闫勇,胡勇:大大豆(大豆WRKY TF家族通过分段复制事件和随后在亚组间的分化选择而扩展。中国生物医学工程学报,2013,13(1):148-10.1186/1471-2229-13-148。
- 30.
Blanc G, Wolfe KH:从复制基因的年龄分布推断模式植物中广泛的古多倍体。植物细胞学报,2004,16(7):1667-1678。10.1105 / tpc.021345。
- 31.
Seoighe C, Gehring C:基因组复制导致高度选择性的扩增拟南芥蛋白质组。植物学报,2004,20(10):461-464。10.1016 / j.tig.2004.07.008。
- 32.
Maere S, De Bodt S, Raes J, Casneuf T, Van Montagu M, Kuiper M, Van De Peer Y:真核生物基因和基因组复制的建模。中国科学:自然科学,2005,26(3):344 - 344。10.1073 / pnas.0501102102。
- 33.
李德昌,安建华,宋雪峰,杜才勇,李建军:大豆根伸长与扩张素基因表达的关系。植物学报,2003,31(3):985-997。10.1104 / pp.009902。
- 34.
李波M,张志刚,张志刚,高桥K,李志刚,张志刚,何俊,徐东,李志刚,张志刚:作物模型Glycine max转录组综合图谱及其在植物中比较分析的应用。植物学报,2010,30(1):344 - 344。
- 35.
薛涛,王东,张松,Ehlting J,倪峰,Jakab S,郑成,钟艳:水稻和拟南芥蛋白质磷酸酶2C全基因组及表达分析。生物医学工程学报,2008,9(1):550-10.1186/1471-2164-9-550。
- 36.
Lescot M, Déhais P, Thijs G, Marchal K, Moreau Y, Van de Peer Y, Rouzé P, Rombauts S: PlantCARE,植物数据库独联体-代理监管要素和门户工具在网上启动子序列分析。中国生物医学工程学报,2002,30(1):325-327。10.1093 / nar / 30.1.325。
- 37.
Sommer H, Saedler H:查尔酮合成酶基因的结构金鱼草majus.Mol Gen Genet MGG。1986, 202(3): 429-434。10.1007 / BF00333273。
- 38.
Menkens AE, Schindler U, Cashmore AR: G-box:植物中普遍存在的由GBF家族bZIP蛋白结合的调控DNA元件。生物化学,1995,20(12):506-510。10.1016 / s0968 - 0004(00) 89118 - 5。
- 39.
李文杰,李志刚,李志刚,李志刚:香芹苯丙氨酸解氨酶基因的结构、调控及光响应基因的鉴定独联体表演元素。中国生物医学工程学报,1998,8 (6):1641-
- 40.
Arguello-Astorga GR, Herrera-Estrella LR:光响应植物启动子中的祖先多部单元具有与特定光转导途径相关的结构特征。植物营养学报,2004,27(3):344 - 344。10.1104 / pp.112.3.1151。
- 41.
pasuglia M, Roby D, Dumas C, Cock JM: S基因家族受体样激酶基因在甘蓝中的损伤和细菌感染快速诱导。植物细胞学报,1997,9(1):49-60。10.1105 / tpc.9.1.49。
- 42.
刘志博,李艳,刘志博,刘志强,陈志强,陈志强,陈志强,陈志强:生长素调控转录。植物学报,1993,20(5):489-502。
- 43.
小川M,花田A,山内Y,库原A,神谷Y,山口S:拟南芥种子萌发过程中赤霉素的生物合成及其响应。植物细胞工程学报,2003,15(7):1591-1604。10.1105 / tpc.011650。
- 44.
王丽娟,王丽娟,王晓明,王晓明,王晓明,王晓明,等。早期吲哚乙酸诱导基因ps - ia4 /5的表达与生长素反应结构域的相互作用。自然科学进展,2004,22(3):389 - 397。10.1073 / pnas.92.8.3483。
- 45.
帕斯库齐P汉密尔顿D身体K Arias J:生长素诱发的应激增强反式-由保守的植物碱性/亮氨酸拉链因子激活。中国生物医学工程学报,1998,27(4):344 - 344。10.1074 / jbc.273.41.26631。
- 46.
金景江,曹娟,吴瑞:水稻高pl α-淀粉酶基因近端启动子区多种蛋白因子的调控及其相互作用。Mol Gen Genet MGG。1992, 32(3): 383-393。
- 47.
Jacobsen JV, Gu B: Pp 246-271bler F, Chandler PM。发芽谷物中的赤霉素和脱落酸。植物激素:生理学、生物化学和分子生物学。编辑:Davies PJ1995年,荷兰多德雷赫特:Kluwer学术
- 48.
Catalá C, Rose JKC, Bennett AB:编码细胞壁修饰蛋白的生长素调控基因在番茄果实早期生长过程中表达。植物营养学报,2000,22(2):344 - 344。10.1104 / pp.122.2.527。
- 49.
Hutchison KW, Singer PB, McInnis S, diazs - sala C, Greenwood MS:扩张素在针叶树中保守,并在下胚轴表达,以响应外源生长素。植物营养学报,2004,27(3):344 - 344。10.1104 / pp.120.3.827。
- 50.
Cho HT, Kende H:扩张素基因表达与深水水稻生长发育相关。《植物细胞学报》,1997,9(9):1661-1671。10.1105 / tpc.9.9.1661。
- 51.
李勇,kade H: β-扩张素表达与深水水稻节间伸长相关。植物学报,2001,27(2):344 - 344。10.1104 / pp.010345。
- 52.
Klotz KL, Lagrimini LM:植物激素对烟草阴离子过氧化物酶启动子的控制。植物分子生物学,1996,31(3):565-573。10.1007 / BF00042229。
- 53.
山口-信崎K,信崎K:拟南芥DNA编码两个干燥反应理查德·道金斯29岁的基因。植物营养学报,2003,26(3):339 - 339。
- 54.
弗雷塔斯FZ,贝托里尼MC:基因组组织Neurosporacrassagsn基因:热休克时可能参与了STRE和HSE元素的转录调节。中国生物医学工程学报,2004,27(5):357 - 357。10.1007 / s00438 - 004 - 1086 - 5。
- 55.
吴勇,Meeley RB, Cosgrove DJ:玉米α-膨胀素和β-膨胀素基因家族的分析与表达。中国农业科学,2001,26(1):1 -2。10.1104 / pp.126.1.222。
- 56.
Jones L, McQueen-Mason S:膨胀蛋白在复活植物脱水和再水化中的作用Craterostigmaplantagineum.中国生物医学工程学报,2004,29(1):344 - 344。
- 57.
Cho HT, Kende H:深海水稻节间膨化蛋白。中国农业科学,2004,27(4):457 - 457。10.1104 / pp.113.4.1137。
- 58.
黄娟,Takano T, Akita S: α-扩张素基因在水稻幼苗中的表达(栽培稻l .)。植物学报,2000,21(4):467-473。10.1007 / s004250000311。
- 59.
金俊华,赵晓东,刘志刚:半水生蕨类植物α-膨胀素的研究进展Marsileaquadrifolia而且Regnellidiumdiphyllum:轴伸长的进化方面和生理作用。植物学报,2000,29(1):1 - 5。10.1007 / s004250000367。
- 60.
王志刚,王志刚,王志刚,王志刚,王志刚:植物抗洪能力的研究进展Rumexpalustris而不是不耐洪水r . acetosa.植物学报,2000,21(6):956-963。10.1007 / s004250050703。
- 61.
Colmer TD, Peeters AJM, Wagemaker CAM, Vriezen WH, Ammerlaan A, Voesenek LACJ: α-扩张素基因在根系适应缺氧过程中的表达Rumexpalustris.植物分子生物学,2004,56(3):423-437。10.1007 / s11103 - 004 - 3844 - 5。
- 62.
Pichersky E, Bernatzky R, Tanksley SD, Breidenbach RB, Kausch AP, Cashmore AR:叶绿素编码基因簇的分子特征和遗传定位a / b-结合蛋白Lycopersiconesculentum(西红柿)。基因学报,1985,40(2):247-258。
- 63.
Yamaji N, Ma JF:水稻硅转运体Lsi1的时空分布。中国农业科学,2007,27(3):344 - 344。10.1104 / pp.106.093005。
- 64.
刘强,王辉,张志,吴娟,冯勇,朱忠:nod26样本征蛋白在植物中的功能和表达差异。生物医学工程学报,2009,10(1):313-10.1186/1471-2164-10-313。
- 65.
刘强,朱华:植物的分子进化枣疯病基因家族栽培稻以及它们的函数散度。基因学报,2008,409(1):1-10。
- 66.
王敏,王强,赵宏,张晓,潘勇:叉头域进化选择压力与功能分化。基因学报,2009,432(1):19-25。
- 67.
杨忠:PAML 4:最大似然系统发育分析。分子生物学杂志,2007,24(8):1586-1591。10.1093 / molbev / msm088。
- 68.
杨喆,尼尔森R:在现实进化模型下估计同义和非同义替代率。分子生物学杂志,2000,17(1):32-43。10.1093 / oxfordjournals.molbev.a026236。
- 69.
杨振:PAML:最大似然系统发育分析3.14版。2004年,伦敦:伦敦大学学院
- 70.
杨智,杨志平,杨志平:似然比检验在自适应分子进化检测中的准确性和有效性。分子生物学杂志,2001,18(8):1585-1592。10.1093 / oxfordjournals.molbev.a003945。
- 71.
杨志:PAML:最大似然系统发育分析程序包。计算应用生物科学,1997,13(5):555-556。
- 72.
张军,杨震,尼尔森R:一种改进的分支位点似然方法在分子水平上检测正选择。分子生物学杂志,2005,22(12):2472-2479。10.1093 / molbev / msi237。
- 73.
李志刚,李志刚,李志刚。多倍体的发生与进化。科学通报,2000,34(1):401-437。10.1146 / annurev.genet.34.1.401。
- 74.
Blanc G, Hokamp K, Wolfe KH:一种新的多倍体叠加在古老的大规模重复拟南芥基因组。基因组学报,2003,13(2):137-144。10.1101 / gr.751803。
- 75.
杨晓明,王晓明,杨晓明,王晓明,等:基于染色体复制事件的被子植物基因组进化研究。自然科学学报,2003,26(3):344 - 344。10.1038 / nature01521。
- 76.
Paterson AH, Bowers JE, Chapman BA:在谷物分化之前的古代多倍体化,及其对比较基因组学的影响。中国科学:自然科学,2004,26(3):389 - 397。10.1073 / pnas.0307901101。
- 77.
Force A, Lynch M, Pickett FB, Amores A, Yan YL, Postlethwait J:利用互补退行性突变保存重复基因。中国生物医学工程学报,1999,31(4):344 - 344。
- 78.
Lynch M,力A:亚功能化重复基因保存的概率。中国生物医学工程学报,2000,54(1):459-473。
- 79.
何霞,张娟:基因复杂性和基因可复制性。动物学报,2005,15(11):1016-1021。10.1016 / j.cub.2005.04.035。
- 80.
Sémon M, Wolfe KH:基因组复制的后果。中国生物医学工程学报,2007,17(6):505-512。10.1016 / j.gde.2007.09.007。
- 81.
Schnable JC,施普林格NM, Freeling M:玉米亚基因组通过基因组优势和古代和持续基因损失的分化。自然科学进展,2011,29(3):379 - 379。10.1073 / pnas.1101368108。
- 82.
Freeling M:亚功能化的进化位置,降级。2008
- 83.
韩田,邹超,李志刚,李志刚,李志刚:植物串联复制体在植物对环境刺激的适应性反应中的作用。中国生物医学工程学报,2008,29(2):344 - 344。10.1104 / pp.108.122457。
- 84.
Cosgrove DJ:扩张蛋白的新基因和新生物学作用。植物学报,2000,3(1):73-79。10.1016 / s1369 - 5266(99) 00039 - 4。
- 85.
郑勇,徐东,顾旭:基因复制后的功能分化与序列结构关系——以g蛋白α亚基为例。中国生物医学工程学报,2007,30(1):489 - 497。
- 86.
顾晓,张卓,黄伟:酵母基因复制后表达和调控差异的快速进化。中国科学:自然科学,2005,26(3):344 - 344。10.1073 / pnas.0409186102。
- 87.
河敏,金东德,陈志军:重复基因增加近缘种和异源多倍体的表达多样性。自然科学进展,2009,26(3):344 - 344。10.1073 / pnas.0807350106。
- 88.
张志刚,张志刚,张志刚,张志刚:5个叉头亚科进化选择压力的分子水平分析。中国生物医学工程学报,2008,8 (1):261-
- 89.
Arnold K, Bordoli L, Kopp J, Schwede T: SWISS-MODEL工作空间:基于网络的蛋白质结构同源建模环境。生物信息学,2006,22(2):195-201。10.1093 /生物信息学/ bti770。
- 90.
基弗F,阿诺德K, Künzli M, Bordoli L, Schwede T:瑞士模型资源库和相关资源。核酸研究,2009,37(增刊1):D387-D392。
- 91.
Peitsch M:通过电子邮件建立蛋白质模型。生物技术学报,1995,13:658- 658。10.1038 / nbt0795 - 658。
- 92.
Li LC, Bedinger PA, Volk C, Jones AD, Cosgrove DJ:从玉米花粉中分离纯化4种β-扩张蛋白(Zea m1亚型)。植物科学进展,2003,27(4):344 - 344。10.1104 / pp.103.020024。
- 93.
Bjellqvist B, Hughes GJ, Pasquali C, Paquet N, Ravier F, Sanchez JC, Frutiger S, Hochstrasser D:多肽在固定化pH梯度中的聚焦位置可以从其氨基酸序列预测。电泳,1993,14(1):1023-1031。10.1002 / elps.11501401163。
- 94.
Bjellqvist B, Basse B, Olsen E, Celis JE:来自不同人类细胞类型的蛋白质的二维图的比较参考点,定义在pH值尺度,其中等电点与多肽组成相关。电泳,1994,15(1):529-539。10.1002 / elps.1150150171。
- 95.
王志强,王志强,王志强,等。蛋白质组学技术在蛋白质组学分析中的应用[M]//2005,美国:Humana出版社
- 96.
齐藤,倪明:邻居连接法:一种重建系统发育树的新方法。分子生物学杂志,1987,4(4):406-425。
- 97.
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S: MEGA5:利用最大似然,进化距离和最大简约方法进行分子进化遗传学分析。分子生物学杂志,2011,28(10):2731-2739。10.1093 / molbev / msr121。
- 98.
李志强,李志强,李志强,等:植物NIN-like蛋白的进化拟南芥,大米,和Lotus对虾.中华生物医学杂志,2005,60(2):229-237。10.1007 / s00239 - 004 - 0144 - 2。
- 99.
李志强,李志强,李志强。分子进化与系统发育。2000年,牛津:牛津大学出版社
- One hundred.
Lynch M, Conery JS:重复基因的进化命运和后果。科学通报,2000,29(4):344 - 344。10.1126 / science.290.5494.1151。
- 101.
Suyama M, Torrents D, Bork P: PAL2NAL:蛋白质序列对齐到相应密码子对齐的鲁棒转换。核酸研究,2006,34(增编2):W609-W612。
- 102.
Severin AJ, Woody JL, Bolon YT, Joseph B, Diers BW, Farmer AD, Muehlbauer GJ, Nelson RT, Grant D, Specht JE, Graham MA, Cannon SB, May GD, Vance CP, Shoemaker RC: RNA-Seq Atlas of大豆:大豆转录组指南。中国生物医学工程学报,2010,10(1):160-10.1186/1471-2229-10-160。
- 103.
Gu X:估计蛋白质序列ii型(聚类特异性)功能分歧的简单统计方法。分子生物学杂志,2006,23(10):1937-1945。10.1093 / molbev / msl056。
- 104.
杨震,Nielsen R:基于密码子替代的分子适应性检测方法。分子生物学杂志,2002,19(6):908-917。10.1093 / oxfordjournals.molbev.a004148。
- 105.
杨忠,王卫伟,尼尔森R:正选择下氨基酸位点的Bayes经验Bayes推断。分子生物学杂志,2005,22(4):1107-1118。10.1093 / molbev / msi097。
确认
感谢国家自然科学基金(30971783)和北京市自然科学基金(5132005)的资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
YZ进行了生物信息学分析并起草了手稿。YH设计了这项研究,并为整个研究提供指导。NW, WS和GY参与了研究并帮助起草了手稿。YQ、YY协调研究,对稿件进行阐述。所有作者都阅读并批准了最终的手稿。
电子辅助材料
大豆扩增素基因超家族的蛋白质序列数据,
附加文件2:拟南芥和米饭。(txt 47kb)
12870 _2013_1512_moesm5_esm.txt
附加文件5:大豆扩张蛋白基因超家族翻译起始密码子上游的核苷酸序列数据1500 bp。(txt 151kb)
大豆中所有膨胀蛋白的邻居连接系统发育树,
附加文件6:拟南芥和大米。(png 924 kb)
膨胀素基因超家族
附加文件7:Medicago truncatula而且菜豆.大豆所有膨胀蛋白的邻居连接系统发育树,Medicago truncatula而且菜豆提供了。蓝色分支支系为EXPA亚科;黄色分支支系为EXPB亚科;绿色分支支系为EXLA亚科;红枝支系属于EXLB亚科。扩增素基因超家族的各个亚家族的基因Medicago truncatula而且菜豆上市。(tiff 874 kb)
12870 _2013_1512_moesm9_esm.docx
附加文件9:大豆膨胀蛋白基序示意图。原理图来源于MEME。图中膨胀蛋白基序的排序由MEME根据分数自动生成。(docx28kb)
12870 _2013_1512_moesm11_esm.tiff
附加文件11:基于Libault Atlas的表达式模式分析。分层集群颜色代码:最大值显示为最红(热),最小值显示为最蓝(冷),中间值是较浅的蓝色或红色。采用Pearson相关聚类对发育调控基因进行分组。(tiff 2mb)
大豆膨胀素基因家族启动子分析。
附加文件12:轨迹名称,独联体的元素名称,以及不同类型的元素的平均值独联体列出了-元素副本。(xlsx55kb)
扩增素基因超家族的扩增模式
附加文件13:拟南芥.(docx17 kb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是原创作品的名称要注明出处。创作共用公共领域奉献弃权书(https://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
朱勇,吴宁,宋伟。et al。大豆(大豆)扩增素基因超家族起源:分段和串联复制事件,随后亚家族间的分化选择。BMC植物生物学14,93(2014)。https://doi.org/10.1186/1471-2229-14-93
收到了:
接受:
发表:
关键字
- 串联重复
- 节段重复
- 棒曲霉素
- 氨基酸位点
- 棒曲霉素的基因