抽象的
背景
揭示植物和动物发育背后复杂的转录调控网络(TRNs)仍然是一个挑战。然而,来自公共微阵列实验的大量数据是可用的,这些数据可以通过推理算法来恢复可靠的TRN架构。
结果
在本研究中,我们提出了一种简单的生物信息学方法,使用公开的、精心策划的微阵列数据和互信息算法ARACNe,以获得转录相互作用的数据库。我们使用了来自拟南芥根样本的转录调控网络成功地恢复了之前鉴定的根转录模块,并提出了新的转录因子短的根/稻草人和过多途径。我们进一步表明,这些网络是一种强大的工具,可以集成和分析高通量表达数据,如我们对短根诱导时间课程微阵列数据集的分析,并且是预测新型根基因功能的可靠源。特别是,我们使用我们的数据库预测涉及根次级细胞壁合成的新型基因,并确定了疯狂箱TFXAL1/AGL12作为这个过程中的意外参与者。
结论
这项研究表明,使用精心策划的微阵列数据进行网络推断可以产生可靠的TRN架构。与以往获得根trn的努力相比,我们的根转录相互作用提供了目前的转录途径的概述拟南芥并可能产生大量有待实验验证的新假设。
背景
转录因子在基因表达的调控中起着重要的作用。的拟南芥(拟南芥)TF数据库Agris [1,2],Rartf [3.,4]或DATF [5,6]包含大约1900个条目,相当于TAIR10基因组释放的27416个蛋白编码基因的6.9%。正向和反向遗传学、诱导表达系统以及近年来的大规模方法,如染色质免疫沉淀、阵列杂交或大规模平行测序,为许多拟南芥转录因子的靶基因和功能提供了大量信息。然而,获得一个特定生物体或发育过程的转录相互作用的完整概述仍然是一项具有挑战性和昂贵的任务。Brady等人利用富集石柱的TFs、这些TFs的启动子以及来自几个miRNA编码基因的启动子,获得了一个包含蛋白质- dna和蛋白质-蛋白质相互作用的富含石柱的根TRN,该TRN通过Y1H和Y2H测定确定[7].然而,至少有一个TF结合的TF启动子的比例很低,并且TF和它们的靶蛋白之间的表达富集有少量重叠,这表明在这个网络中可能缺失了几个可能是stele TRN重要组成部分的基因。
基因表达微阵列可以快速量化给定生物样本中大量基因的表达水平。目前应用最多的拟南芥基因表达芯片是Affymetrix ATH1-121501 (ATH1)基因芯片。截至2010年10月,使用EBI ArrayExpress数据库中列出的ATH1芯片进行的实验共686例[8].这些实验提供了在多种实验条件下拟南芥组织中基因表达的定量分析,为拟南芥转录调控网络(TRN)推断提供了合适的数据来源。尽管genevinvestigative [9,10], ATTED-II [11,12],或者BAR表达垂钓者[13,14]有用于分析拟南芥微阵列数据的工具,它们要么使用有限的微阵列实验,AtgenExpress系列[15] (ATTED-II和BAR表达式垂钓者),或其质量控制、策划、注释和标准化数据不公开(genevinvestigator)。
鉴于这些限制,我们决定创建自己的策略和注释的rabidopsis微阵列数据库,并使用此数据来推断TRNS。在ArrayExpress数据库中索引的686微阵列实验包含超过9000个单独的芯片杂交或CEL文件。我们实验室中完成的初步工作表明,来自不同组织中获得的样品的网络推断,例如全植物,根和叶,产生次优效果,并且推断网络难以在生物学环境中解释。因此,我们决定使用从单个器官获得的微阵列数据,即根。拟南芥根系具有几种特征,使其成为我们目的的合适器官:根部解剖学是相对简单,发育改变可以容易观察到,有大量的文献关于根部发育和根本表达的TFS,最后,有从根样本获得的相当大量的高质量ATH1微阵列数据。为了鉴定在根组织中发生的转录相互作用,我们使用微阵列数据作为输入的算法ARACNe [16].
Aracne是一种用于识别基因产品之间的转录相互作用的信息方法,使用微阵列表达式数据数据,能够在变量之间恢复非线性统计依赖性,并且先前已用于TRN重建[17- - - - - -20.].在这项工作中,我们表明,我们的数据库,以及从它衍生出来的trn,已经能够恢复先前描述的转录因子的功能和靶基因。我们进一步证明,推断出的trn可以准确预测新的TF函数,例如MADS-box TF的预测作用XAL1/AGL12(AT1G71692)在次级细胞壁的形成及其与功能缺失突变根表型的确认为该基因。
结果与讨论
为了推断拟南芥根系发育和生理过程的trn,我们使用了两个精心整理的数据集,这些数据集来自56个ATH1微阵列实验的656份根系特异性CEL文件(附加文件)1).第一个数据集,即我们所说的只TFS-数据集,是一个656列由2088行表对应于我们的2088个TF探针列表。我们称之为数据集的第二个数据集是656到22810表,其中包含ATH1芯片中存在的所有22810个探针。我们将两个数据集用作Aracne软件的输入[21].ARACNe输出是通过一个互信息值及其关联的p值排列的交互探针集对的列表。ARACNe的理论背景和实际应用的细节可参阅[16]和[21但是,简单地说,基因A和基因B之间的相互作用意味着基因A在所有656个实验中的表达谱可以解释基因B在同样的656个实验中的表达谱,反之亦然,因为相互作用不是定向的。在生物环境下,基因a和B之间的交互将意味着基因a和B参与相同的生理过程,进一步,如果基因是一个特遣部队和B是一个non-TF,交互(基因解释基因B)将表明,基因是基因的转录监管机构B。
网络推理以ATH1芯片中存在的2088 TF探头为中心,在三个数据处理不等式(DPI)值0.0、0.1和0.2下获得。DPI是一种已知的信息理论属性,在[21].简单地说,在DPI 0.0时,当存在三个节点的小团(三角形)时,具有最低互信息的交互将被删除,因为这种交互被认为是一种间接交互。在DPI值非0.0时,允许有三个基因循环,在DPI 1.0时,不去除相互作用。DPI值为0.2(如果交互的互信息值的差值为20%或更少,则会保留三角形)增加了真实积极交互的恢复,同时仍然最小化了假阳性的恢复[16].将ARACNe输出邻接文件转换为Cytoscape兼容表后,我们得到了相应的TFs-only (TFsNet;额外的文件2)并完成(FullNet;附加文件3.)数据库。如表所示1时,边数从DPI 0.0急剧增加到DPI 0.1到DPI 0.2。为清晰起见,本文中网络的所有图形表示都是在DPI 0.0处得到的。
参与推断交互的tf用根表示
关于我们的网络,一个重要的问题是确定参与推断交互的tf是否实际上在根组织中表达。来自affy R包的mas5calls功能,用于标记微阵列表达值为Present, Absent或Marginal,是一个不可靠的工具来确定一个基因是否被表达[22],特别是涉及拟南芥转录因子[23].因此,为了确定TFs出现在我们的网络表达在根组织内,我们从TFsNet和FullNet获得0.0 DPI TFs,参与互动,我们比较两个实验确定root-expressed基因的列表,列表(见方法)。结果如表所示2和附加文件4.实验证明,两种网络中92%以上的恢复转录因子在根中表达。因此,我们相信,在我们的数据集中出现的tf确实是根tf,我们恢复的交互代表了真实的在足底转录的交互。
参与相同过程的tf被归类到TFsNet中
TFsNet来自一个只包含TFs的数据集,该数据集排除了所有非tf基因,构成了拟南芥根TFs推断相互作用的概述(图)1).参与相同过程的TFS预计将在不同的集群或模块中分组。已经识别和实验表征了这些功能模块中的一些并用作推断网络的可靠性的探讨。
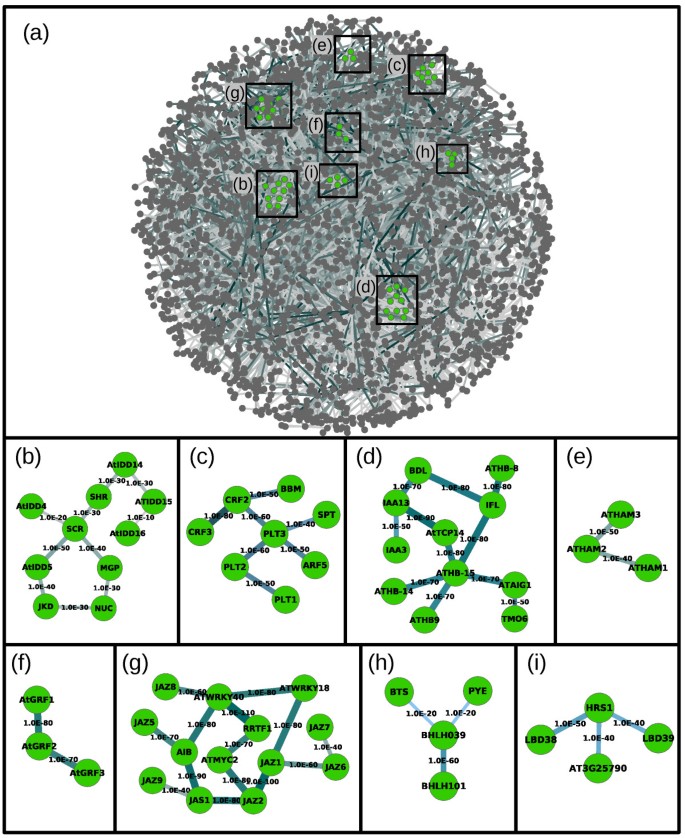
TFSNet。(一)在DPI 0.0时获得的TFsNet概述。基因被表示为节点,推断的交互作用被表示为边。节点是灰色的,除了在文本中提到的基因是绿色的。边宽和颜色强度与交互的互信息(Mutual Information, MI)值成正比,MI值越大,边缘越厚越深。为了清晰起见,省略了基因名称。对特定TF组的放大将在后面的面板中显示。(我)在shrr - scr组中存在tf的子网络(b)PLT小组(c)血管开发群体(d), AtGRF组(e)AtHAM集团(f)中,茉莉酮酸酯反应组(g)缺铁性组(h)还有硝酸盐反应组(我).边被标记为交互作用的p值。为了清晰起见,从组到网络其余部分的边缘被省略了。
已经确定了控制干细胞生态位模式的两个转录途径[24- - - - - -28].第一个途径由GRAS系列组成短的根(月;AT4G37650)和稻草人(可控硅;AT3G54220)和C2H2家族,不确定域名(IDD)喜鹊(MGP;AT1G03840)和寒鸦(jkd.;AT5G03150)。如图所示1b,这四个tf与IDD一起分组胡桃夹子(n;AT5G44160)的SSXT域转录共激活因子ANGUSTIFOLIA 3(AN3;AT5G28640)和GRAS家族衣衫褴褛的3放到(SCL-3;AT1G50420)。n和SCL-3是SHR的直接转录靶点[29- - - - - -31].注意,由于在DPI 0.0处获得的网络不能包含三角形,例如在中间没有边月和n,并不意味着这两个基因之间缺乏相互作用,而仅仅意味着这两个基因有其他相互作用,从而获得更好的MI评分。此外,该模块中基因间的相互作用具有较低的MI值,对应于1e-30和1e-40的p值(相对于数据集中最低的p值1e-140)。这可能并不奇怪,因为这种途径有一个复杂的分子相互作用模式[32这将阻碍ARACNe算法从具有较高p值的微阵列数据中恢复其交互作用的能力[21].额外IDD基因,AtIDD4(AT2G02080),AtIDD5(AT2G02070),AtIDD14(AT1G68130),AtIDD15(AT2G01940)和AtIDD16(AT1G25250),存在于此模块中。据报道了SCL-3-NUC,MGP-SCR,MGP-SHR,MGP-JKD,SCR-JKD和SHR-JKD的蛋白质 - 蛋白质相互作用[33].另一方面,IDD蛋白JKD和MGP通过转录和蛋白质 - 蛋白质相互作用通过转录和蛋白质 - 蛋白质相互作用调节REC和SCR表达和运动横跨根组织[27,34].最后,通过在其vhiid基序中取代单个苏氨酸残基来消除了ShR蛋白的运动,这提出了介导SHR的蛋白质 - 蛋白质相互作用[35]及其核心定位[34].因此可以推测这一点是有趣的AtIDD4,AtIDD5,AtIDD14,AtIDD15和AtIDD16也可能通过与SHR和SCR的转录调控或蛋白-蛋白相互作用参与根的发育和模式形成。
第二种途径涉及生长素信号通过激活尚未识别的生长素反应因子(ARFs)和过多(PLT)转录因子,属于AP2-EREBP家族。PLT的基因,PLT1(AT3G20840),PLT2(AT1G51190),PLT3/AIL6.(AT5G10510)和婴儿潮(BBM;AT5G17430),具有重叠的表情轮廓,并以冗余的方式行事[26].在TFSNet中,四个PLT基因是同一组的一部分,也包括bHLH抹刀(AT4G36930),ARF5/MONOPTEROS(AT1G19850)和erf家族细胞分裂素反应因子CRF2/TMO3(AT4G23750)和CRF3(AT5G53290;数字1c).值得注意的是,有四个过多蛋白质(26),ARF5[36]均表现在苗根石碑首字母上。根的血管模式已被证明依赖于生长素-细胞分裂素的相互作用[37和一些基因的相互交流的参与,例如SHY2[38],BRX[39] 或者AHP6[40,41已经得到证明。然而,一个转录网络连接过多通路和细胞分裂素应答的转录因子仍然缺失。本模块中出现的两个CRF tf为这一方向提供了新的线索。
BODENLOS(BDL;Aux / Iaa家族的成员AT1G04550是一种转录抑制剂ARF5其表达由胚胎中的ARF5控制[42].奇怪的是,BDL,以及另外两个Monopteros的目标(TMO)基因,ATAIG1/TMO5(AT3G25710)和TMO6(AT5G60200),不要群体ARF5TFsNet。相反,它们是一组涉及血管发育的转录因子的一部分,包括基因IAA13(AT2G33310),IAA3/SHY2(AT1G04240) [39],ATHB-14/PHABULOSA(AT2G34710),ATHB-15/电晕(AT1G52150) [43],IFL/罗沃拉(AT5G60690)[44],ATHB-8(AT4G32880) [45],ATHB9 / PHAVOLUTA(AT1G30490)和AtTCP14(AT3G47620)[46)(图1d)。pBDL:绿色荧光蛋白在4-5日龄幼苗的根中观察到表达(见图S6 [42]),从而指向这些与血管发育中这些与这些生长素相关基因的新典型。
其他涉及器官发育的转录因子也被归在转录因子网中。例如,密切相关的ATHAM1(AT2G45160),ATHAM2(AT3G60630)和ATHAM3(AT4G00150)的基因,属于GRAS家族,是参与维持分生组织的不确定性,并且在功能上是冗余的[47,48].这三个tf也在我们推断的TFsNet中的同一个模块中分组(图1e).另一个例子涉及AtGRFGRF家族的基因,在发育中的组织中表达,如茎尖、花蕾和根。单一突变体Atgrf1.(AT2G22840),atgrf2.(AT4G37740)或atgrf3(AT2G36400)基因没有表现型,双突变体具有较小的表现型[49,这表明这三个基因有多余的作用。Atgrf1.,atgrf2.和atgrf3组一起在TFsNet提出这里(图1有趣的是,我们的网络推断也恢复了相互作用atgrf3-AN3(p值1E-70)和AN3-可控硅(P值1E-40),建议AtGRF模块和月-可控硅在根开发期间的模块。
所述TFsNet还恢复已知参与比发展其他根生理过程基因之间的转录相互作用。参与茉莉响应的第一个例子涉及基因(图1g).这组包括TIFY域基因JAZ1(AT1G19180),JAZ2(AT1G74950),JAZ5(AT1G17380),JAZ6(AT1G72450),JAZ7(AT2G34600),JAZ8(AT1G30135),JAZ9(AT1G70700),JAS1/JAZ10(AT5G13220),参与病原体响应2个WRKY基因,WRKY18(AT4G31800)和WRKY40(AT1G80840) [50), bHLH-family爱尔兰联合银行(AT2G46510)[51),MYC2(AT1G32640) [52]和AP2/ERFRRTF1(AT4G34410)。有趣的是,染色质免疫沉淀实验表明WRKY40可以结合JAZ8和RRTF1监管区域(53),而MYC2最近被证明参与茉莉酸盐依赖的根发育抑制[54].
第二个示例包括bHLH TFBHLH038(AT3G56970),BHLH039(AT3G56980),BHLH100(AT2G41240),BHLH101(AT5G04150),大力水手(py;AT3G47640)和dna结合蛋白编码哈库斯(BTS;AT3G18290),参与缺铁应激调节[55,56].BHLH039,BHLH101,py和BTS在TFsNet中分组(图1h;BHLH038和BHLH100不在ATH1芯片中)。
第三个例子涉及硝酸盐反应TFs [57].应响应硝酸盐刺激表达的最早TFS是HRS1(AT1G13300),LBD37(AT5G67420),LDB38(AT3G49940),LBD39(AT4G37540)和AT3G25790.(在集群157])。这五个助教中的四个,HRS1,LDB38,LBD39和AT3G25790.在TFsNet中分组(图1h).请注意,EBI数据库中Long等人(E-GEOD-21443)和Krouk等人(E-GEOD-20044)的微阵列数据是在我们的微阵列实验下载后几天发布的,并不是我们用于分析的数据的一部分。
使用FullNet集成和分析高通量功能基因组数据
FullNet的数据包括ATH1芯片中所有22810个探针集,并以2088 TF探针集列表为中心(附加文件)3.).在这个网络中,tf将是中心节点,它们的交互者,无论是tf还是非tf,都是相邻节点。参与相同过程的基因应该再次被归类到一起。例如,TFsNet中确定的TF组仍然存在于FullNet中的相同组中。必须记住,在这个网络中,存在非tf节点。当一个非tf与两个tf相互作用,且这些相互作用的MI得分高于TF-TF相互作用时,在DPI 0.0时,后者的相互作用将被认为是间接相互作用,因此不会出现在网络中。然而,这并不意味着TF-TF交互不存在,只是它被一个中间的非tf节点“屏蔽”了。当TF-TF MI值不是三角形中的最低值时,它在DPI 0.0 FullNet中可见。这就是相互作用的情况PLT1,PLT2和PLT3/AIL6.,在p值为1e-50处可控硅-月相互作用的p值为1e-30,表明早期的硝酸盐响应TF的相互作用HRS1与LBD38,LDB39和AT3G25790.在p值1e-40及以下,以及相互作用BHLH039与BHLH101p值为1e-60pyp值为1e-20。之间的相互作用AGL71和AGL72,在TFsNet中以p值1e-20存在,现在以p值1e-50恢复。这两个MADS-box基因最近被证明在顶端和腋生分生组织中有冗余作用[58].
在FullNet中,TF节点的交互子是该TF的潜在靶基因。如果是这样的话,人们可以预期大量实验确定的TF靶基因会出现在相应的ARACNe相互作用者列表中。一个由aracne推断的相互作用被实验证实的TF例子对应于VND7/ANAC030 (AT1G71930)。VND7是一种参与次级细胞壁合成的na -family TF,目前已有几种推测的靶基因列表[59- - - - - -62].我们比较了这些实验确定的列表VND7我们的目标基因列表VND7在DPI 0.0, 0.1和0.2时,来自完整数据集的交互作用子3.和附加文件5).在DPI 0.0,在DPI 0.1和24中24个中的16个基因中的14个,其中107个在DPI 0.2中为107个。VND7邻居列表是出于对实验设置至少一个差异表达。几乎所有的差异表达的基因在高MI值发现,对应于1E-50和下部的p值。最后,三山口等人确定的四个差异表达的转录因子。[62],jlo.(AT4G00220),MYB46(AT5G12870)和MYB103(AT1G63910),是VND7TFSNet中的群集,在1E-50和更低的P值下。好奇地,排名第一VND7我们数据集中的互动器,Pinoreinol还原酶ATPRR1(AT1G32100),不存在于任何实验中VND7目标基因列表。ATPRR1在DPI 0.0,TF互动器,比MI值更高VND7这表明,它可以由一个或多个高分助教来管理。或者,VND7-ATPRR1转录相互作用可能是年龄特异性的,在上述任何实验设置中都无法检测到。
也有一些转录因子的例子,在aracne推断的相互作用子列表和实验的靶基因列表之间几乎没有重叠。两个例子是SHR和SCR tf。月和可控硅是根发育的重要基因,目前已有几个拟转录靶基因列表[29- - - - - -31,63].Sozzani等人[30.],通过微阵列数据分析,获得了SCR或SHR诱导时间过程中差异表达基因的综合列表,Cui等[31]通过染色质免疫沉淀(ChIP)鉴定SHR靶基因。将Sozzani等人的目标基因列表(我们将其称为Sozzani-SCR和Sozzani-SHR列表)与我们在DPI 0.0、0.1和0.2条件下获得的推断SCR或SHR交互子ARACNe列表进行直接比较,结果发现总体重叠率较低:在DPI 0.2位点有732个ARACNe-SCR和719个ARACNe-SHR相互作用体,其中68个(9.2%)和159个(22%)存在于相应的SCR-或sr - sozzani列表中。特别是,我们希望在ARACNe和Sozzani lists中发现已知参与shrr - scr转录调控途径的基因,即jkd.,MGP,n和CYCD6; 1(AT4G03270)。前三个基因是tf,它们可以在同一个模块中找到月和可控硅TFsNet。CYCD6; 1,一个非tf,在SCR-Sozzani和sr - sozzani列表中都存在,但不是一个aracne推断的交互因素月,可控硅,jkd.,MGP也不n.在DPI 0.0时,它唯一的交互TF是AGL92(AT1G31640),这是不接近月-可控硅TFSNet或FullNet中的模块。虽然令人失望,但这结果可能并不令人惊讶:Cycd6; 1以非常特殊的野生型根细胞类型表达,皮质/内胚层初始干细胞和侧根原始内胚层细胞[30.,64].此外,Cycd6; 1参与涉及蛋白质 - 蛋白质相互作用,蛋白质磷酸化和蛋白质降解的复杂调节机制[64].这些机制很可能在整个根样本中翻译成相应基因的转录水平较差,这是ARACNe的输入数据。
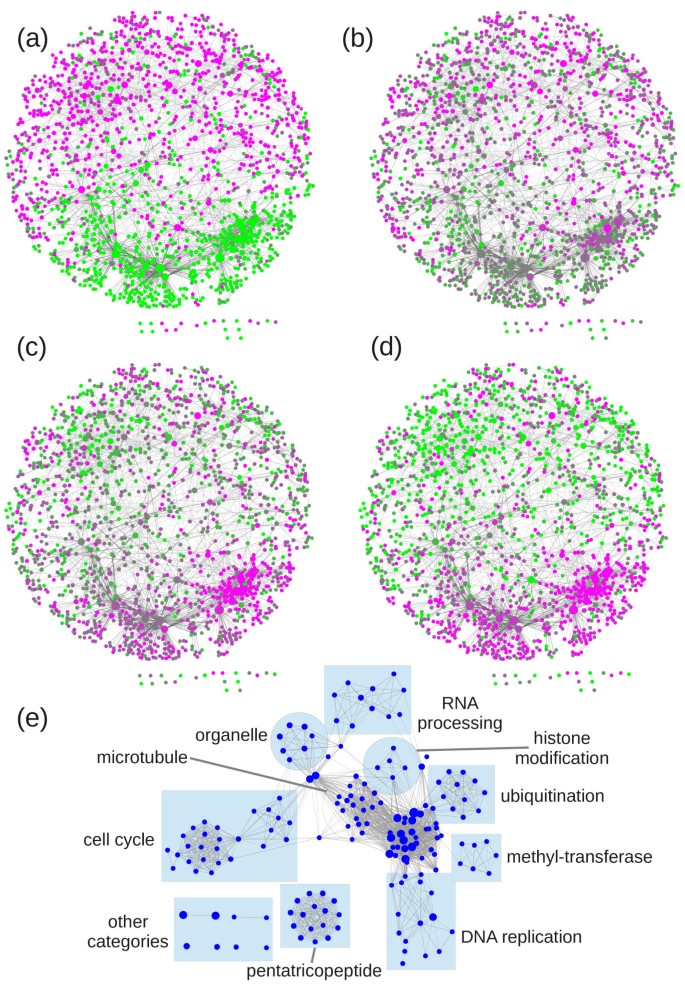
ARACNe恢复实验确定的TF靶基因的能力很可能反映了TF参与的调控相互作用的数量和复杂性。VND7是一种只参与次级细胞壁合成(SCWS)的TF [65,66].因此,我们预计VND7参与非常具体的转录模块,并准确地回收其实验鉴定的靶基因。另一方面,REC和SCR最有可能参与许多转录途径,因为这些基因的突变体在根瘤发育中受强烈影响[67,68]在SCR或SCR诱导的差异表达基因的列表中可以发现超过200TFS,其分析了特定的根细胞型,即地面组织[30.].如此重要数量的差异表达转录因子(约占拟南芥转录因子总数的10%)进一步表明,这些实验确定的靶基因中有相当一部分是间接靶基因。此外,SCR和SHR对根发育的调控涉及特定细胞类型的表达、细胞类型间的运输、核-细胞质易位、蛋白-蛋白相互作用和蛋白磷酸化[27,30.,34,35,64].在这种情况下,我们预计可以通过在他们参与的网络的背景下可视化实验识别的目标基因来获得更好的结果。因此,我们决定从DPI 0.0获得的Fullnet数据集和1E-30的截止值P值,所有相互作用都存在于来自Sozzani的SHR诱导动力学中的2481差异表达基因的列表中。collaborator’s study [30.],我们补充说月(AT4G37650)。结果数据集现在包含1668个基因(原始列表的67%),相应的网络由Cytoscape绘制[69].1647个节点(66%),包括月的分组在一个单一的子网络(图2广告)。我们注意到,这个子网中的两个部分分得很清楚,对应的基因,如诱导动力学时间的推移,交换机从低表达的一种过度表达状态,反之亦然。该子网的分析,现在可以帮助识别相关节点,应在SHR的转录通路中发挥重要作用。例如,三个主节点,从不足到过度表达切换是PRMT3(AT3G12270),kyp.(AT5G13960)和HD2A(AT3G44750),是编码染色质修饰(组蛋白甲基转移酶和组蛋白去乙酰化酶)蛋白的基因。当使用David时,所有基因从低表达转换为过度表达的分析[70,71]及富集图[72揭示了该模块在细胞周期、微管、rna加工和推测的染色质修饰蛋白编码基因中富集(图)2e)。
Aracne推断的网络允许预测根表达的TFS的新型遗传相互作用:可能的作用抹刀在里面过多通路
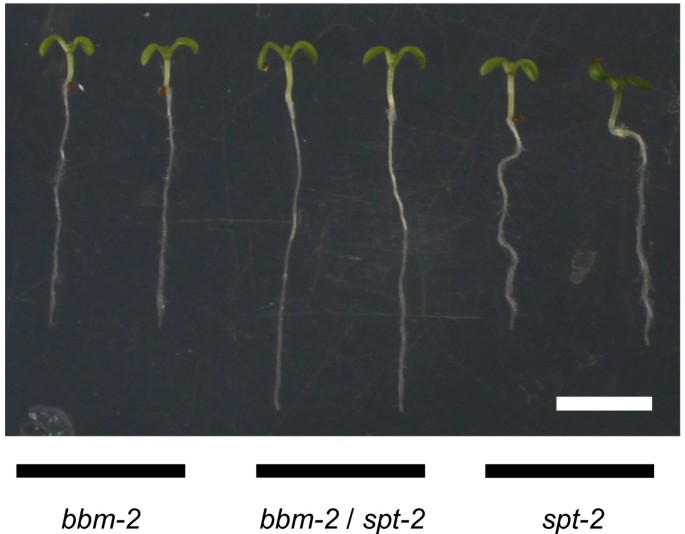
TFsNet的数据来源于我们列出的2088个TF探针集(见方法)。在这个网络中,参与同一生物过程的转录因子应该被分组在一起。因此,我们期望同一模块中基因的高阶突变植株表现出在单个突变植株中所没有的根表型。我们用存在于同一模块中的基因来验证这一假设,这些基因1)属于不同的TF家族,2)在TFsNet中不是直接邻居,3)突变体具有不同的根表型。的基因婴儿潮(BBM),抹刀(SPT)符合这些标准。两个基因都存在于同一个模块中(图)1c)和突变体BBMap2结构域TF基因的根略短[26的突变体SPTbHLH TF基因的根比野生型植物略长[73].当生长在垂直的盘子上时,BBM-2/SPT-2双突变体表现出比任何一种更长的根SPT-2或BBM-2单个突变体幼苗(图3.).之前的报告表明PIN4和DR5 :: GUS.根分生组织中的表达改变spt-11突变体苗(73].总之,这些结果指出了可能的转录相互作用过多途径和抹刀在拟南芥根分生组织中生长素运输和/或反应的调控。
ARACNe推断的网络允许的新颖功能根表达的转录因子的预测:的情况下XAL1/AGL12, MADS-box TF参与次级细胞壁合成
由于我们的Aracne推断网络能够恢复已知的基因关联,因此我们希望它们还能够预测新的TF功能。作为我们数据库的预测能力的示例,我们决定寻找可以参与二次细胞壁合成(SCWS)的新TFS。为此目的,我们的策略组成,在选择几个基因中,既是涉及SCW的TF和非TF,恢复他们与FullNet的交互,并绘制所得到的网络以识别新的SCWS TFS。已知几个TFS参与SCWS,其中包括我们选择VND6/ANAC101(AT5G62380),VND7/ANAC030(AT1G71930)[74],SND2/ANAC073(AT4G28500) [75],MYB46[76),IXR11(AT1G62990) [77].作为SCWS非TF基因我们选择了纤维素合酶CESA4(AT5G44030),CESA7.(AT5G17420)和CESA8.(AT4G18780) [78],漆包酶LAC4(AT2G38080)和LAC17(AT5G60020) [79]中,所述半胱氨酸肽酶XCP1(AT4G35350)和XCP2(AT1G20850) [80), chitinase-likeATCTL2(AT3G16920) [81.,即DUF6域WAT1(AT1G75500) [82.],TED6(AT1G43790) [83.],duf231域TBL3(AT5G01360) [84.和8糖基转移酶家族GAUT12/IRX8(AT5G54690) [85.].然后,我们从FullNet中检索所有涉及这些基因在DPI 0.0和p值截断1e-30的相互作用,并使用Cytoscape [69来可视化相应的网络(附加文件6).它立即似乎这些基因确实是SCWS基因网络的一部分,包括我们的输入基因加上其他其他已知或推定的SCWS基因,包括MYB83(AT3G08500) [86.],ANAC007/VND4(AT1G12260) [65] 或者ATPRR1[87.]但还有血管开发TFS喜欢ATHB-15[43],ATHB-16(AT4G40060)88.),jlo.(AT4G00220) VND7的靶标[62].
在SCWS网络的一个高度连接的部分,22个不属于我们输入基因列表的tf现在存在(图4).我们从FullNet中检索所有涉及这些TFs的交互作用,DPI为0.0,0.1和0.2,p值截止值为1e-30。利用David对三个新确定的助教的助教名单进行了浓缩分析,XAL1/AGL12(一个疯子盒),BEE2和AT1G68810(两个bHLH)显示它们在SCWS基因中特别富集(数据未显示);每个TF的高MI值交互者列表显示在附加文件中7.由于这三个tf存在于SCWS网络的高度连接部分,因此发现它们共享几个相互作用者并不令人惊讶。AGL12/XAL1是一种MADS-box转录因子,在韧皮部组织中表达,参与调控根的发育和花期[89.].BEE2被首批认定为油菜素内酯响应TF [90.].油菜素内酯促进根系生长[91.,是拟南芥茎维管系统发育所必需的[92.增强拟南芥悬浮培养的木质部导管转分化[74].AT1G688101)我们发现TF是TFsNet中血管发育簇的一部分,2)与ATAIG1/TMO5,这也是TFS-ock血管开发群的一部分,以及3)寂寞高速公路的蛋白质 - 蛋白蛋白互联网,其参与血管发育的转录活化剂[93.].这些结果预测XAL1,BEE2和AT1G68810是SCWS的重要tf。
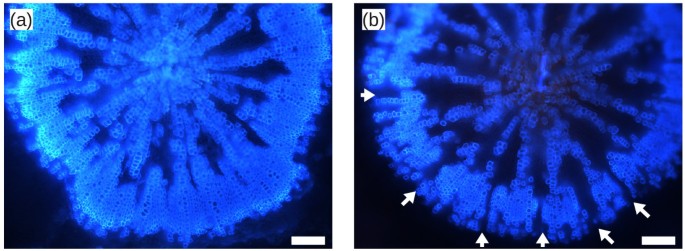
由于MADS-box tf通常与SCWS不相关,我们决定寻找SCW沉积在xal1-2失去功能的突变根[89.].自xal1-2相同年龄植株的根可能揭示了发育阶段相关的SCWS差异,而不是直接的SCWS表型。因此,col0和xal1-2当主干长度为29-32cm时,收集根部,其可谓对应于同一发育阶段的植物。正如我们推断的网络所预测的那样,xal1-2成年根实际上具有改变的次级细胞壁图案,具有次级木质和纤维环中的间隙(n = 10/10),在相同尺寸的野生型植物中很少观察到表型(n = 1/10;图5).Sibout等人在一篇有趣的论文中指出,下胚轴和根的木质部扩张与开花时间有关[94.].巧合的是,xal1-2植物推迟开花[89.和根SCWS的改变,强烈提示XAL1可以是连接两个进程的TRN的一部分。
SCWS改变的确认xal1-2根组织表明,我们推断trn的生物信息学方法是准确预测根表达的转录因子新功能的成功方法。这一结果进一步加强了我们的网络可能会提供有关根系发育的功能模块的新假设。另外一个例子,DUF6蛋白WAT1 [82.]在DPI 0.0和p值截止值1e-50时,具有TF交互作用ATHB-15/中央社,AT1G68810,AT4G29100,STH2.,ATHB-16和AT2G28510,这些都是TFsNet血管发育簇的一部分(图)1d).这表明,首先,这些tf中的一个或多个是转录调节因子WAT1其次,这些TFs中的一个或多个,至少部分地通过直接的转录调控来控制维管的发育WAT1.最后,DUF6结构域蛋白编码基因AT1G43650,AT1G01070,AT3G45870,AT3G18200和AT4G30420是否已知tf的相互作用者参与SCWS,表明他们可能具有与SCWS相似的作用WAT1在根标准铜线。
结论
在这项工作中,我们表明,从多个复合的,精心选择和策划的微阵列数据集的网络推断允许重建可靠的根转录相互作用网络。我们发现,这种推断出来的网络既能恢复已知的、功能特征的TF模块,又能可靠地预测这些模块的新成分,以及新模块,包括特定TF的意外角色。我们特别强调发现了一个涉及MADS-box TF的次级细胞壁合成的新模块XAL1/AGL12.我们的转录相互作用数据库进一步提供了拟南芥根系中存在的转录途径的概述,并且可能产生实验测试的多种新假设。
方法
微阵列数据
使用Affymetrix基因芯片ATH1-121501的所有微阵列实验列表已于2010年10月从EBI ArrayExpress数据库下载[8)(附加文件1).以相应的样本-数据关系文件为指导,选择所有使用根组织的实验,检索相应的CEL文件。在组织比较的实验中,例如茎与根的比较,特别注意排除非根的CEL文件。此外,为了获得高质量的同质数据集,arrayQualityMetrics Bioconductor包[95.],剔除低质量的CEL文件,以及除Columbia-0以外其他生态型样品对应的CEL文件。最后,为了避免潜在的基因调控网络可能受到干扰[96.],所有与转基因样品(突变体、过表达、启动子构建)相对应的CEL文件也被排除。这导致了使用gcRMA规范化的656个CEL文件[97.将得到的归一化数据作为ARACNe算法的输入。
对于仅使用tfs的网络,使用celutil实用程序将选定的根CEL文件转换为ASCII格式[98.]和ATH1-121501阵列名称用一个自定义名称代替。甲只TFS-CDF文件是使用XSpecies的修改版本[创建99.use_ME.pl Perl脚本One hundred., Affymetrix ATH1-121501 probe_tab文件被重命名以匹配自定义的CDF名称。这两个文件分别使用makecdfenv和AnnotationDbi包打包为R,这允许我们使用gcRMA规范化修改后的CEL文件[97.].得到的归一化数据用作ARACNe算法的输入。
转录因子名单
-e或ARACNe参数使用TF列表,以保存包含一个或多个TF的交互[21].Agris有三个rapidopsis tf数据库[1,2],Rartf [3.,4]及科技资助基金[5,6].由于DATF将Aux/IAA家族包括为TF家族,而Agris和DATF不包括,而且由于生长素在植物发育中起主要作用,我们决定通过结合三个数据库中存在的所有AGI id来创建自己的TF列表。我们进一步添加了来自TAIR10 ATH_GO_GOSLIM文件的所有AGI id,这些AGI id用Gene Ontology条目GO:0003700(“序列特异性DNA结合转录因子活性”)注释,因为数据库中缺少几个转录因子,如AGL26或AGL64。最后的TF列表包含2575个AGI id,对应于2088个探针集(附加文件8).
使用ARACNe的网络推理
使用归一化数据计算config_kernel.txt和config_threshold.txt参数需要ARACNe使用作者提供的Matlab脚本。使用Linux命令行ARACNe 32位程序在三个DPI值0.0、0.1和0.2下推断2088 TF探针集的交互,对于完整的数据集使用2088探针集列表作为-l参数,或者对于仅使用TF的数据集不使用-l参数。ARACNe程序的命令行执行形式为:ARACNe -H config_parameters -i normalized_data [-l TF-list] -e 0.0/0.1/0.2 -o output_file。
邻接文件转换
的TAIR10array_elements和别名这些表被组合起来,以获得单个表,将探针集链接到AGI id,并在可用时链接到它们对应的符号。生成的组合表用于使用自定义Perl脚本将ARACNe输出邻接文件转换为与Cytoscape兼容的、以制表符分隔的表。
根表示的tf列表
结合a) 14日龄幼苗根中实时RT-PCR检测到的转录因子列表[23], b)根样品大规模蛋白质组学筛选中鉴定的蛋白质清单,EBI PRIDE数据库实验3332,15486,15489,15517,15518,15519,15525,15526,15528 [101- - - - - -103], c) TAIR10植物本体表中标注为根或全植物表达的基因列表,即包含基因本体实验证据代码,不含微阵列证据,d) RNA-seq实验中根表达基因列表,附件SRR314814 [104]和日本DNA数据库序列读取档案的SRR331219 [105].Czechowski等人使用的引物。[23,以确定它们对预期的基因仍然是特定的。对于RNA-seq数据,使用bowtie2 (version 2.0.0-beta5;[106])——非常敏感, -N 1, -k 11和-s,0,-0.8端到端模式参数。使用自定义Perl脚本,只考虑匹配单个位点的reads来鉴定表达的基因。
BBM-2/SPT-2双突变体
BBM-2和SPT-2种子取自拟南芥生物资源中心。成人BBM-2和SPT-2植物被交叉,纯合双突变植物通过基因分型鉴定出来BBM-2变异使用BBM引物5'-ACTTTAGTGCGGCTAAATCGTAAGC-3 ', 5' -CAATAACGAACAAAATGGACCAAAG-3 '和LBb1.3引物5'- atttttgccgatttcggaac -3 ',并通过视觉鉴定植物的表现SPT-2心皮表型。单突变体和纯合双突变体的种子分别播种在0.5X Murashige和Skoog基础培养基上,0.5%蔗糖,1%植物琼脂(豪语技术实验室)板。板置于4℃,并在两天后,用一整天的光照制度转移至生长室中在22℃下(16小时光照,8小时黑暗)。照片都是6天后发芽服用。
显微镜
Col-0和xal1-2[89.植物是在标准温室条件下的土壤中种植的。采集主茎高度29-32 cm的植株,手工切下胚轴下根的横切面,在荧光显微镜下紫外光下立即观察到木质化组织的自身荧光。
缩写
- TF:
-
转录因子
- DPI:
-
数据处理不平等
- 美国国际集团ID:
-
拟南芥基因组倡议轨迹识别号码。
参考
- 1.
拟南芥转录因子数据库(AtTFDB)。http://arabidopsis.med.ohio-state.edu/AtTFDB/,
- 2.
梁晓东,李晓东,李晓东。拟南芥基因调控信息服务器的研究进展[j]。生物工程学报,2011,39(4):591 - 598。10.1093 / nar / gkq1120。
- 3.
RIKEN拟南芥转录因子数据库(RARTF)。http://rarge.psc.riken.jp/rartf/,
- 4.
Iida K,Seki M,Sakurai T,Satou M,Akiyama K,Toyoda T,Konagaya A,Shinozaki K:Rartf:全套数据库和工具拟南芥转录因子。DNA资源学报,2005,12:247-256。10.1093 / dnares / dsi011。
- 5.
拟南芥转录因子数据库(DATF)。http://datf.cbi.pku.edu.cn/,
- 6.
郭阿,他k,liu d,白s,gu x,wei l,罗j:datf:一个数据库拟南芥转录因子。生物信息学。2005,21:2568-2569。10.1093 /生物信息学/ bti334。
- 7.
蒋恩,刘伟,Zeng A, Taylor-Teeples M, Kim D, Ahnert S, Ohler U, Ware D, Walhout AJM, Benfey PN:拟南芥根中富集石柱的基因调控网络。中国生物医学工程学报,2011,27 (4):459-
- 8.
ArrayExpress。http://www.ebi.ac.uk/arrayexpress/,
- 9。
Genevestigator。https://www.genevestigator.com/gv/,
- 10.
Hruz T, Laule O, Szabo G, Wessendorp F, Bleuler S, Oertle L, Widmayer P, Gruissem W, Zimmermann P:基因调查者v3:用于转录组meta分析的参考表达数据库。生物信息学进展,2008,2008:420747-
- 11.
ATTED-II。http://atted.jp.,
- 12.
Obayashi T, Nishida K, Kasahara K, Kinoshita K: ATTED-II更新:条件特异性基因共表达,将共表达分析扩展到开花植物的广泛应用。植物营养与肥料学报。2011,32(4):429 - 434。10.1093 /卡式肺囊虫肺炎/ pcq203。
- 13.
酒吧表达式垂钓者。http://bar.utoronto.ca/ntools/cgi-bin/ntools_expression_angler.cgi,
- 14.
Winter D, Vinegar B, Nahal H, Ammar R, Wilson GV, Provart NJ:一个“电子荧光象形文字”浏览器,用于探索和分析大规模的生物数据集。公共科学图书馆。2007,2:e718-10.1371/期刊。
- 15.
AtGenExpress。http://www.weigelworld.org/resources/microarray/AtGenExpress/,
- 16.
Margolin AA,Nemenman I,Basso K,Wiggins C,Stolovitzky G,Dalla Favera R,Califano A:Aracne:一种在哺乳动物细胞背景下重建基因调控网络的算法。BMC生物信息学。2006,7(4):S7-10.1186 / 1471-2105-7-S1-S7。
- 17.
Basso K, Margolin AA, Stolovitzky G, Klein U, Dalla-Favera R, Califano A:人类B细胞调控网络的反向工程。王志强。2005,37:382-390。10.1038 / ng1532。
- 18.
Basso K, Saito M, Sumazin P, Margolin AA, Wang K, Lim W-K, Kitagawa Y, Schneider C, Alvarez MJ, Califano A, Dalla-Favera R: Integrated biochemical and computational approach identifies BCL6 direct target genes controlling multiple pathways in normal germinal center B cells. Blood. 2010, 115: 975-984. 10.1182/blood-2009-06-227017.
- 19。
Agnelli L,Forcato M,Ferrari F,Tuana G,Todoerti K,Walker Ba,Morgan GJ,Lombardi L,Bicciato S,Neri A:转录网络的重建揭示了对多发性骨髓瘤的临床结果影响的关键基因。临床癌症Res。2011,17:7402-7412。10.1158 / 1078-0432.ccr-11-0596。
- 20.
Yu X, Li L, Zola J, Aluru M, Ye H, fouree A, Guo H, Anderson S, Aluru S, Liu P, Rodermel S, Yin Y: brassinosteroid transcriptional network revealed by全基因组bsi靶基因的鉴定拟南芥.植物学报,2011,65:634-646。10.1111 / j.1365 - 313 x.2010.04449.x。
- 21.
Margolin AA, Wang K, Lim WK, Kustagi M, Nemenman I, Califano A:反向工程蜂窝网络。Nat protocol . 2006, 1: 662-671。10.1038 / nprot.2006.106。
- 22.
Zilliox MJ, Irizarry RA:微阵列数据的基因表达条形码。Nat Methods. 2007, 4: 911-913。10.1038 / nmeth1102。
- 23.
Czechowski T, Bari RP, Stitt M, Scheible W-R, Udvardi MK:超过1400的实时RT-PCR分析拟南芥转录因子:前所未有的敏感性揭示了新的根和芽特异性基因。植物学报,2004,38:366-379。10.1111 / j.1365 - 313 x.2004.02051.x。
- 24.
Sabatini S, Heidstra R, Wildwater M, Scheres B:稻草人参与了干细胞生态位的定位拟南芥根分生组织。陈志强。2003,17:354-358。10.1101 / gad.252503。
- 25.
Aida M, Beis D, Heidstra R, Willemsen V, Blilou I, Galinha C, Nussaume L, Noh Y-S, Amasino R, Scheres B: The过多基因调解图案拟南芥根干细胞生态位。细胞。2004,119:109-120。10.1016 / J.Cell.2004.09.018。
- 26.
Galinha C, Hofhuis H, Luijten M, Willemsen V, Blilou I, Heidstra R, Scheres B:作为剂量依赖的主调控因子的过剩蛋白拟南芥根发展。自然科学学报。2007,449:1053-1057。10.1038 / nature06206。
- 27.
Welch D,Hassan H,Blilou I,Immink R,Heidstra R,Scheres B:拟南芥JACKDAW和MAGPIE锌指蛋白通过限制短根作用划定不对称细胞分裂和稳定组织边界。王志强,王志强。10.1101 / gad.440307。
- 28.
阿兹培提,Benítez M, Vega I, Villarreal C, Alvarez-Buylla ER:细胞模式的单细胞和耦合GRN模型拟南芥根干细胞生态位。BMC Sys Biol. 2010, 4: 134-10.1186/1752-0509-4-134。
- 29.
Levesque MP, Vernoux T, Busch W, Cui H, Wang JY, Blilou I, Hassan H, Nakajima K, Matsumoto N, Lohmann JU, Scheres B, Benfey PN:短根发育途径的全基因组分析拟南芥.公共科学图书馆。2006,4:e143-10.1371/journal.pbio.0040143。
- 30.
Sozzani R, Cui H, Moreno-Risueno MA, Busch W, Van Norman JM, Vernoux T, Brady SM, Dewitte W, Murray JAH, Benfey PN:通过SHORTROOT连接模式和生长对细胞周期基因的时空调控。自然。2010,466:128-132。10.1038 / nature09143。
- 31.
崔华,郝颖,郭洪平,邓晓伟,黄志刚,黄志刚:基因组直接靶标分析揭示了短根通过细胞分裂素稳态在根维管模式中的作用。植物营养与肥料学报。2011,31(4):531 - 534。10.1104 / pp.111.183178。
- 32.
Ogasawara H, Kaimi R, Colasanti J, Kozaki A:转录因子JACKDAW的活性对于SHR/ scr依赖的激活至关重要稻草人和喜鹊并通过与喜鹊、稻草人和短根的相互作用进行调节。植物生态学报。2011,32(4):489- 498。10.1007 / s11103 - 011 - 9826 - 5。
- 33.
拟南芥互作组图:拟南芥互作组图网络演化的证据。中国科学:地球科学。
- 34.
Gallagher KL, Benfey PN:保守的GRAS域和核定位都是短根运动所必需的。植物学报,2009,57:785-797。10.1111 / j.1365 - 313 x.2008.03735.x。
- 35.
作者简介:王志强,男,博士,主要研究方向为细胞间运动的调节机制。《中国科学:地球科学》2004年第4期。10.1016 / j.cub.2004.09.081。
- 36.
Rademacher EH, Möller B, Lokerse AS, Llavata-Peris CI, van den Berg W, Weijers D:拟南芥的细胞表达图谱助线响应因子基因家族。植物学报,2011,68:597-606。10.1111 / j.1365 - 313 x.2011.04710.x。
- 37.
主教A, Benková E, Helariutta Y:发送混合的信息:生长素-细胞分裂素在根中的串扰。植物生态学报。2011,14:10-16。10.1016 / j.pbi.2010.08.014。
- 38.
acta botanica yunnanica(云南植物研究中心);acta botanica yunnanica(云南植物研究中心);acta botanica yunnanica(云南植物研究中心);中国科学:地球科学。10.1126 / science.1164147。
- 39.
Scacchi E,SalinaS P,Gujas B,Santuari L,Krogan N,Ragni L,Berleth T,Harttke CS:根系成绩中的跨监管事件的时空序列。pnas。2010,107:22734-22739。10.1073 / pnas.1014716108。
- 40.
Mähönen AP, Bishopp A, Higuchi M, Nieminen KM, Kinoshita K, Törmäkangas K, Ikeda Y, Oka A, Kakimoto T, Helariutta Y:细胞分裂素信号及其抑制剂AHP6在血管发育过程中调控细胞命运。中国科学:地球科学。10.1126 / science.1118875。
- 41.
b.p ishopp A, Help H, El-Showk S, Weijers D, Scheres B, Friml J, Benková E, Mähönen AP, Helariutta Y:生长素和细胞分裂素之间相互抑制的相互作用指定了根的维管模式。中国生物医学工程学报。2011,21:917-926。10.1016 / j.cub.2011.04.017。
- 42.
刘S,迪斯梅特I,Kolb的男,迈进H,尤尔根G:生长素触发基因开关。NAT细胞BIOL。2011,13:611-615。10.1038 / ncb2212。
- 43.
Ochando I, González-Reig S, Ripoll J-J, Vera A, Martínez-Laborda A:射击径向模式的变化拟南芥通过III类HD-ZIP基因的功能性等位基因INCURVATA4.国家自然科学基金青年科学基金。10.1387 / ijdb.072306io。
- 44.
钟R,泰勒JJ,叶ZH:在拟南芥突变体interfascicular纤维分化的破坏。植物细胞。1997年,9:2159年至2170年。10.1105 / tpc.9.12.2159。
- 45.
王志强,王志强,王志强,等。生长素信号对原形成层前细胞状态获取的调控拟南芥叶子。发展。2009,136:3235-3246。10.1242 / dev.037028。
- 46.
atematsu K, Nakabayashi K, Kamiya Y, Nambara E:转录因子AtTCP14在种子萌发过程中调控胚胎生长势拟南芥.植物学报,2008,53:42-52。10.1111 / j.1365 - 313 x.2007.03308.x。
- 47.
Schulze S, Schäfer BN, Parizotto EA, Voinnet O, there K:失去的分生组织基因调节拟南芥分生组织中心区的后代细胞的分化。植物杂志2010,64:668-678。10.1111 / j.1365-313X.2010.04359.x。
- 48.
Engstrom EM, Andersen CM, Gumulak-Smith J, Hu J, Orlova E, Sozzani R, Bowman J:矮牵牛花毛分生组织基因的同源性是维持茎和根不稳定性所必需的。植物营养与肥料学报。2011,32(6):741 - 748。10.1104 / pp.110.168757。
- 49.
AtGRF家族在水稻叶片和子叶生长发育中起重要作用拟南芥.植物学报,2003,36:94-104。10.1046 / j.1365 - 313 x.2003.01862.x。
- 50.
Shen Q-H, Saijo Y, Mauch S, Biskup C, Bieri S, Keller B, Seki H, Ulker B, somsich IE, schulz - lefert P: MLA免疫受体的核活性与分离特异性和基础抗病反应有关。中国科学:地球科学。10.1126 / science.1136372。
- 51.
Li H, Sun J, Xu Y, Jiang H, Wu X, Li C: bHLH-type transcription factor AtAIB正向调节ABA的反应拟南芥.植物mol biol。2007,65:655-665。10.1007 / s11103-007-9230-3。
- 52.
Wang Z, Cao G, Wang X, Miao J, Liu X, Chen Z, Qu L-J, Gu H: COI1-dependent transcription factor genes involved in ja介导的创伤应答拟南芥植物。植物学报,2008,34(5):591 - 598。
- 53.
Pandey SP, Roccaro M, Schön M, Logemann E, somsich IE: WRKY18和WRKY40调控的转录重编程促进了拟南芥白粉病的侵染。植物学报2010,64:912-923。10.1111 / j.1365 - 313 x.2010.04387.x。
- 54.
Chen Q, Sun J, Zhai Q, Zhou W, Qi L, Xu L, Wang B, Chen R, Jiang H, Qi J, Li X, Palme K, Li C: The basic helix-loop-helix transcription factor MYC2 directly suppression . (sci: cn过多茉莉酸介导的根干细胞生态位调控中的表达拟南芥.植物细胞。2011,23:3335-3352。10.1105 / TPC.111.089870。
- 55.
Wang H- y, Klatte M, Jakoby M, Bäumlein H, Weisshaar B, Bauer P:铁缺乏介导的Ib亚群应激调节BHLH基因拟南芥.植物学报。2007,26:897-908。10.1007 / s00425 - 007 - 0535 - x。
- 56.
bbhlh转录因子POPEYE调控人体对铁缺乏的反应拟南芥的根源。acta botanica sinica(云南植物研究),2010,22(4):489 - 494。10.1105 / tpc.110.074096。
- 57.
陈志强,陈志强,陈志强,等:高分辨率植物转录组对硝酸盐响应的预测网络模型。生物谷bioon . 2010, 11: R123-10.1186/gb-2010-11-12-r123。
- 58.
Dorca-Fornell C,Gregis V,Grandi V,Zonceand G,Colombo L,Kater MM:拟南芥SOC1样的基因AGL42,AGL71和AGL72促进茎尖和腋生分生组织开花。植物学报,2011,67:1006-1017。10.1111 / j.1365 - 313 x.2011.04653.x。
- 59.
大桥东K,小田Y,福田H:拟南芥在木质部分化过程中,与维管相关的NAC-DOMAIN6直接调控调控细胞程序性死亡和次生壁形成的基因。acta botanica sinica(云南植物研究),2010,22(4):441 - 446。10.1105 / tpc.110.075036。
- 60.
钟锐,李超,叶志华:二次壁NAC主开关直接目标的全局分析拟南芥.植物学报。2010,33(3):1087-1103。10.1093 / mp / ssq062。
- 61.
Yamaguchi M, Ohtani M, Mitsuda N, Kubo M, Ohme-Takagi M, Fukuda H, Demura T: VND-INTERACTING2, NAC结构域转录因子,负调控木质部导管的形成拟南芥.植物细胞。2010年,22:1249-1263。10.1105 / TPC.108.064048。
- 62.
Yamaguchi M, Mitsuda N, Ohtani M, Ohme-Takagi M, Kato K, Demura T: VASCULAR-RELATED NAC-DOMAIN7直接调控木质部导管形成的广泛基因的表达。植物学报,2011,66:579-590。10.1111 / j.1365 - 313 x.2011.04514.x。
- 63.
Cui H, Levesque MP, Vernoux T, Jung JW, Paquette AJ, Gallagher KL, Wang JY, Blilou I, Scheres B, Benfey PN:一种进化保守的机制定义了一层内皮层。中国科学:地球科学。10.1126 / science.1139531。
- 64.
克鲁兹-Ramírez的A,迪亚兹-TriviñoS,Blilou I,Grieneisen VA,Sozzani R,Zamioudis C,Miskolczi P,NieuwlandĴ,本杰明R,Dhonukshe P,Caballero的-佩雷斯Ĵ,Horvath的B,龙Y,MähönenAP,张辉,徐军,默里扎哈,Benfey PN,巴科L,MAREE AFM,Scheres B:涉及SCARECROW-视网膜母细胞瘤集成线索告知非对称干细胞分裂双稳态电路。细胞。2012年,150:1002至1015年。10.1016 / j.cell.2012.07.017。
- 65.
Kubo M, Udagawa M, Nishikubo N, Horiguchi G, Yamaguchi M, Ito J, Mimura T, Fukuda H, Demura T:原生木质部和后生木质部导管形成的转录开关。陈志强。2005,19:1855-1860。10.1101 / gad.1331305。
- 66.
Yamaguchi M, Kubo M, Fukuda H, Demura T:维管相关的NAC-DOMAIN7参与了拟南芥根和芽中所有类型的木质部导管的分化。植物学报,2008,55:652-664。10.1111 / j.1365 - 313 x.2008.03533.x。
- 67.
Benfey PN,林斯特德PJ,罗伯茨K,席费尔拜JW,豪瑟MT,Aeschbacher RA:拟南芥根的发育:四个突变体显着改变的根形态。发展。1993年,119:57-70。
- 68.
Scheres B, Di Laurenzio L, Willemsen V, Hauser MT, Janmaat K, Weisbeek P, Benfey PN:影响拟南芥根径向组织的突变在整个胚胎轴表现出特定的缺陷。发展。1995,121:53-62。
- 69.
关键词:Cytoscape 2.8,数据集成,网络可视化生物信息学。2011,27:431-432。10.1093 /生物信息学/ btq675。
- 70。
黄DW,谢尔曼BT,Lempicki RA:采用生物信息学DAVID资源大基因列表系统和综合分析。NAT PROTOC。2009年,4:44-57。
- 71。
黄德伟,谢立军,李志强:生物信息学富集工具:大基因序列综合功能分析的途径。核酸学报2009,37:1-13。10.1093 / nar / gkn923。
- 72。
Merico D,Iserlin R,Stueker O,Emili A,Bader Gd:丰富地图:基于网络的基因集的富集可视化和解释方法。Plos一个。2010年,5:e13984-10.1371 / journal.pone.0013984。
- 73。
Makkena S, Lamb RS: bHLH转录因子SPATULA通过控制根分生组织的大小调控根的生长。生物谷bioon . 2013, 13: 1-10.1186/1471-2229-13-1。
- 74.
Yamaguchi M, Goué N, Igarashi H, Ohtani M, Nakano Y, Mortimer JC, Nishikubo N, Kubo M, Katayama Y, Kakegawa K, Dupree P, Demura T:在诱导系统的控制下,VASCULAR-RELATED NAC-DOMAIN6和VASCULAR-RELATED NAC-DOMAIN7能有效诱导木质部导管分子转分化。植物营养与肥料学报,2010,29(4):531 - 534。10.1104 / pp.110.154013。
- 75.
钟R,李C,周Ĵ,麦卡锡RL,叶Z-H:参与次生细胞壁生物合成的调节的转录因子的电池拟南芥.acta botanica sinica, 2017, 36(5): 762 - 762。10.1105 / tpc.108.061325。
- 76.
Zhong Riply,Ye Z-H:MyB46转录因子是SND1的直接目标,并调节二级墙体生物合成拟南芥.植物细胞。2007,19:2776-2792。10.1105 / TPC.107.053678。
- 77.
李娥,王某,刘y,陈杰克,道格拉斯CJ:卵形家族蛋白4(OFP4)与KNAT7的相互作用调节二级细胞壁形成拟南芥.植物保护学报,2011,32(6):741 - 746。10.1111 / j.1365 - 313 x.2011.04595.x。
- 78.
阐明了纤维素合酶复合物的组装和亚基相互作用的机制拟南芥次生细胞壁。中国生物医学工程学报。2009,29(4):457 - 461。
- 79.
Berthet S, Demont-Caulet N, Pollet B, Bidzinski P, Cézard L, Le Bris P, Borrega N, Hervé J, Blondet E, Balzergue S, Lapierre C, Jouanin L:破坏LACCASE4和17导致木质素化的组织特异性改变拟南芥茎。acta botanica sinica, 2017, 36(5): 593 - 598。10.1105 / tpc.110.082792。
- 80.
Avci的U,彼佐尔德HE,伊斯梅尔IO,啤酒EP,Haigler CH:半胱氨酸蛋白酶在拟南芥根xylogenesis在完整中央液泡内XCP1和XCP2援助微自溶。植物J. 2008,56:303-315。10.1111 / j.1365-313X.2008.03592.x。
- 81.
Hossain MA, Noh - n, Kim K-I, Koh E-J, Wi S-G, Bae H- j, Lee H, Hong S-W:几丁质酶样蛋白编码的突变AtCTL2基因提高木质素积累暗生长拟南芥幼苗。植物营养与肥料学报,2010,27(6):641 - 646。10.1016 / j.jplph.2009.12.001。
- 82.
Ranocha P, Denancé N, Vanholme R, Freydier A, Martinez Y, Hoffmann L, Köhler L, Pouzet C, Renou J-P, Sundberg B, Boerjan W, Goffner D:墙很薄(WAT1),拟南芥的同源物Medicago truncatula NODULIN21,是纤维次生壁形成所需的液泡质体定位蛋白。植物学报,2010,1:469-483。
- 83.
Endo S, Pesquet E, Yamaguchi M, Tashiro G, Sato M, Toyooka K, Nishikubo N, Udagawa-Motose M, Kubo M, Fukuda H, Demura T:鉴定参与血管元件次级细胞壁形成的新成分Zinnia和拟南芥.acta botanica sinica, 2009, 31(4): 457 - 461。10.1105 / tpc.108.059154。
- 84.
Bischoff V,Nita S,Neumetzler L,Schindelasch D,Urbain A,Eshed R,Persson S,Delmer D,Scheible W-R:毛滴经的双折射及其同源物AT5G01360编码拟南芥纤维素生物合成所需的植物特异性DUF231蛋白。植物营养与肥料学报,2010,29(4):531 - 534。10.1104 / pp.110.153320。
- 85.
Persson S, Caffall KH, Freshour G, Hilley MT, Bauer S, Poindexter P, Hahn MG, Mohnen D, Somerville C:拟南芥不规则xylem8突变体缺乏glucuronoxylan和同半乳糖醛酸,这对次级细胞壁的完整性至关重要。植物学报。2007,19:237-255。10.1105 / tpc.106.047720。
- 86.
McCarthy RL, Zhong R, Ye Z-H: MYB83是SND1的直接靶点,与MYB46在拟南芥次生细胞壁生物合成调控中具有冗余作用。植物生理学杂志。2009,50:1950-1964。10.1093 /卡式肺囊虫肺炎/ pcp139。
- 87.
中subo T, Mizutani M, Suzuki S, Hattori T, Umezawa T:表征拟南芥松脂醇还原酶:一种参与木脂素生物合成的新型酶。中国生物医学工程学报。2008,29(3):453 - 457。
- 88.
Nishitani C, Demura T, Fukuda H:初级韧皮部特异性表达Zinnia线虫同源框基因。植物生理学报。2001,42(4):491 - 498。10.1093 /卡式肺囊虫肺炎/ pce156。
- 89.
Tapia-López R, García-Ponce B, Dubrovsky JG, Garay-Arroyo A, Pérez-Ruíz RV, Kim S- h, Acevedo F, Pelaz S, Alvarez-Buylla ER: An无性生殖的相关MADS-box基因,XAL1(AGL12),调控拟南芥根分生组织细胞增殖和开花过渡。植物营养与肥料学报。2008,32(4):457 - 461。10.1104 / pp.107.108647。
- 90.
Friedrichsen DM,Nemhauser J,Muramitsu T,Maloof Jn,Alonso J,Ocker Jr,Furuya M,Chory J:三种冗余芸芸胸甲类固醇早期反应基因编码预测的BHLH转录因子正常生长所需的推定的BHLH转录因子。遗传学。2002,162:1445-1456。
- 91.
Müssig C, Shin G-H, Altmann T:油菜素内酯促进拟南芥根生长。植物营养与肥料学报。2003,33(4):457 - 461。10.1104 / pp.103.028662。
- 92.
Ibañes M, Fàbregas N, Chory J, Caño-Delgado AI:油菜素内酯信号转导和生长素转运是建立油菜素内酯周期模式的必要条件拟南芥拍摄维管束。pnas。2009年,106:13630-13635。10.1073 / pnas.0906416106。
- 93.
大桥伊东,伯格曼拟南芥根维管初始种群由寂寞的公路.发展。2007,134:2959-2968。10.1242 / dev.006296。
- 94。
Sibout R,Plantegenet S,Hardtke CS:开花作为木质部膨胀的状态拟南芥下丘脑和根。Curr Biol。2008,18:458-463。10.1016 / J.Cub.2008.02.070。
- 95。
Kauffmann A, Gentleman R, Huber W: arrayqualitymetrics—用于微阵列数据质量评估的生物导体包。生物信息学。2009,25:415-416。10.1093 /生物信息学/ btn647。
- 96。
王志强,王志强,王志强,等。遗传相互作用网络的进化可塑性。王志强。2008,40:390-391。10.1038 / ng.114。
- 97。
Wu Z, Irizarry RA, Gentleman R, Martinez-Murillo F, Spencer F:基于模型的背景校正的寡核苷酸表达阵列。中国科学(d辑:地球科学),2004,19(4):593 - 597。10.1198 / 016214504000000683。
- 98.
- 99.
- One hundred.
Hammond JP, Broadley MR, Craigon DJ, Higgins J, Emmerson ZF, Townsend HJ, White PJ, May ST:使用基于基因组dna的探针选择来提高高密度寡核苷酸阵列应用于异种物种的敏感性。植物研究。2005,1:10-10.1186/1746-4811-1-10。
- 101.
蛋白质组鉴定数据库(PRIDE)。http://www.ebi.ac.uk/pride/archive,
- 102.
Baerenfaller K, Grossmann J, Grobei MA, Hull R, Hirsch-Hoffmann M, Yalovsky S, Zimmermann P, Grossniklaus U, Gruissem W, Baginsky S:基因组规模的蛋白质组学揭示拟南芥基因模型和蛋白质组动力学。中国科学:地球科学。10.1126 / science.1157956。
- 103.
关键词:拟南芥,蛋白质组,蛋白质组学,pep2pro,拟南芥中国生物医学工程学报。2011,33(3):225-237。10.1039 / c0ib00078g。
- 104.
Gan X, Stegle O,贝洱J,史蒂芬詹,Drewe P,希尔德布兰德KL, Lyngsoe R, Schultheiss SJ,奥斯本EJ, Sreedharan VT,卡尔,Bohnert R,琼G,德文特河P,凯西P Belfield EJ, Harberd NP,刻满E, Toomajian C, Kover PX,克拉克RM, Ratsch G,莫特R:多个参考基因组和转录组拟南芥.《自然》,2011,477:419-423。10.1038 / nature10414。
- 105.
DDBJ序列读取归档(DRA)。http://trace.ddbj.nig.ac.jp/dra/index_e.shtml,
- 106.
Langmead B,Trapnell C,流行女,Salzberg SL:超快和短DNA序列与人类基因组的存储器高效的对准。基因组Biol。2009年,10:R25-10.1186 / GB-2009年10月3日-R25。
致谢
我们要感谢拟南芥生物资源中心提供的种子。RACM获得了UNAM Instituto de Ciencia y Tecnología del Distrito Federal (BI09-570)和Centro de Ciencias de la Complejidad的博士后资助。SdF实验室的工作得到了墨西哥国家科学技术委员会(CONACyT) 177739号拨款的支持。ERAB实验室的工作得到如下资助:CONACyT (81542,81433, 167705, 152649, 180380,180098), PAPIIT (IN229003-3, IN204011-3, 226510-3, IB201212-2), UC-MEXUS (CN.12-623, CN.12-571)和REDES TEMÁTICAS DE INVESTIGACIÓN CONACyT: Red Complejidad, Ciencia y Sociedad。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
作者的贡献
RACM参与了研究的设计,完成了所有的生物信息学分析,进行了实验并撰写了手稿。GC参与了研究的设计,并参与了数据分析。KLGA和NMM进行了实验。SdF帮助撰写了手稿。ERAB构思了研究,参与了研究的设计,讨论了数据分析和结果,并帮助撰写了手稿。所有作者阅读并批准了最终的手稿。
电子补充材料
12870 _2013_1504_moesm4_esm.xls
附加文件4:TFsNet和FullNet中所有tf根表达的实验证据表。(XLS 586 KB)
的列表
额外的文件5:ANAC030/VND7推断在DPI 0.0, 0.1和0.2得到的FullNet中的交互作用者。(XLS 48 KB)
12870 _2013_1504_moesm6_esm.pdf
附加文件6:在DPI 0.0和1E-30的P值截止时获得的SCWS子网图。基因被表示为节点,推断的交互作用被表示为边。对应于文本中提到的输入基因的节点是彩色绿色。边缘宽度与交互的互信息(MI)值成比例,具有与较厚边缘相对应的较高的MI值。(PDF 30 KB)
表
额外的文件7:XAL1/AGL12,AT1G68810和BEE2推断在DPI 0.0, 0.1和0.2得到的FullNet中的交互作用者。仅示出了具有P值的互动器1E-50或更小。(XLS 62 KB)
作者为图像提交的原始文件
权利和权限
开放获取本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0.),允许在任何媒介上不受限制地使用、分发和复制,但须注明原作的出处。创作共用及公共领域专用豁免书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Chávez Montes, r.a., Coello, G., González-Aguilera, K.L.et al。基于aracne的推断,使用精心策划的微阵列数据拟南芥根转录调控网络。BMC植物杂志14,97(2014)。https://doi.org/10.1186/1471-2229-14-97
收到了:
接受:
发表:
关键字
- 转录调控网络
- 根
- 拟南芥
- 转录因子
- ARACNe