摘要
背景
由于本作物中发现的多态DNA标记物,培养花生的基因组研究落后于其他作物物种。有必要识别额外的DNA标志物,以进行花生的进一步遗传研究。
结果
使用SSR浓缩程序开发了栽培花生中的微卫星标记。结果表明,GA / CT重复是花生中最常分散的微卫星。设计为56种不同微卫星的引物对,其中19个在研究的基因型中显示出多态性。每个基因座的平均等位基因数为4.25,在一个基因座中发现了最多14个等位基因。这表明微卫星DNA标志物在培养的花生中的其他DNA标志物中产生更高水平的DNA多态性。
结论
期望在培养的花生中分离和表征更多的DNA标志物,用于更生产的基因组研究,例如遗传映射,标记辅助选择和基因发现。微卫星标记的发展对这些研究具有承诺。
背景
微卫星,也被称为简单序列重复或SSRs,是一个小排列的碱基(一到六个)遍布整个基因组。微卫星作为DNA标记比许多其他标记具有优势,因为它们具有高度多态性、高度丰度、共显性遗传、分析简单和易于转移[1].据报道,微卫星比RFLPs或rapd变化更大,并已被广泛应用于植物基因组研究[2-9.].微卫星相对于其他类型的遗传标记的优势将变得更加重要和明显,当它们被用于跟踪大规模育种计划中的理想性状和作为基于地图的基因克隆策略的锚点时[10.].
有许多方法可以获得微卫星标记。利用SSR探针杂交筛选基因组文库,并对杂交阳性克隆进行测序是一种传统的、费时费力的方法。在筛选富含ssr的小型插入库时,可大大减少时间和成本[11.].通过将含SSR的片段与生物素标记的探针杂交,可以通过用链霉抗生物素蛋白或固定在硝酸盐过滤器上固定的磁性珠粒捕获的生物素标记的探针来实现富集。12.].除去非杂交的DNA后的洗脱部分高度富集微卫星,富集效率在50-90%之间[13.].
栽培花生或落花生(落花生hypogaea是一种重要的油脂和蛋白质来源。虽然中国、美国和印度是最大的生产国,但它在世界各地都有种植。尽管栽培花生的各种形态、生理和农艺性状的基因型存在很大的多样性,但利用蛋白质或DNA标记检测到的DNA变异很少[15.-19.].由于DNA水平缺乏多态性,这种作物在遗传映射,标志物辅助选择,抗性基因克隆和作物进化研究中落后于遗传映射,与其他作物相比滞后。这个问题阻碍了分子技术改善栽培花生。虽然花生存在微卫星[20.那21.],微型卫星尚未完全确定、开发或利用,关于微型卫星丰富程度的详细资料仍然空白。
本研究的目的是:(1)从培养的花生基因组中鉴定微卫星;(2)检测使用这些新型微卫星的多态性,在具有多样化起源的花生戒备中的集合中。
结果
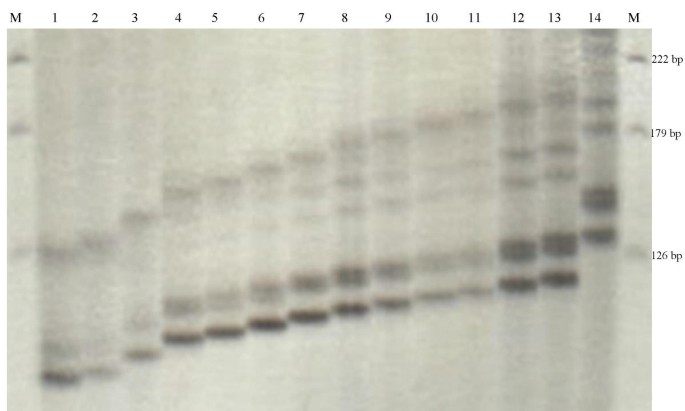
使用具有一些修饰的富集程序构建微卫星富集的文库。从初级转换板随机挑选四百个菌落并进行测序。大多数插入的长度在100 bp - 300 bp之间。发现约247(61%)的菌落含有SSR序列,其中大多数是GA / CT重复。在这些含SSR的片段中,83个是独特的(33%),而其余冗余(67%)。我们可以设计56个独特碎片中的底漆对(见附加文件1).其余序列具有太短的侧翼区域以设计引物。从56个引物对,19可以检测24种花生基因型之间的多态性(见表1).每个轨迹的平均等位基因数为4.25(见表2).由于使用引物对PM50发现的等位基因数量最多,因此还有24个基因型,包括来自野生物种的一个基因型(A. Monticola.)用来在这个特定的位点上寻找更多的等位基因。在48个基因型的调查中,PM50位点共鉴定出14个等位基因(见图)1).这是首次在栽培花生中鉴定到如此多的等位基因。利用这5个高多态性标记,可以分化24个栽培基因型(数据未显示)。我们认为,花生的微卫星标记可能像大豆等其他豆科植物一样丰富。这些初步结果表明,该系统是一种很有前途的分离和表征花生微卫星。
讨论
花生是一种独特的物种,因为rflps,同工酶和Rapds等分子标记检测到非常小的遗传变异[24.].已经鉴定了几种分子标记物以与花生中的抗性基因联系起来[25.那26.因此,通过标记辅助育种的遗传改善是有限的。虽然已经确定了相当数量的AFLP标记[27.,需要更多的DNA标记来使现有的花生连锁图谱饱和,并启动对这种植物的遗传研究。
微卫星已成为近年来遗传研究最广泛使用的分子标记之一。富集AFLP或特异性适配器扩增的DNA片段是SSR隔离的简单有效的方法,已成功应用于许多植物基因组[12.那28.-32.].通过采用来自其他植物物种的改性程序,通过本研究在花生中建立了相应的分离方案,以通过本研究开发微卫星。当基因组DNA转化为AFLP片段组件时,可以使用选择性引物扩增不同的AFLP片段,然后用SSR探针杂交。通过优化杂交和洗涤步骤,在克隆和测序后发现大多数AFLP片段涉及微卫星。为了有效地丰富含SSR的碎片,几个研究人员[12.那28.那33.组合几个SSR探针与DNA片段杂交。然而,由于杂交期间的温度不适,然后洗涤,某些类型的重复丢失了[31.].探针分别与片段杂交,以防止部分探针在本研究中被冲洗掉。然而,含有GA/CT重复序列的片段大多分别使用三种探针检测。在部分片段中发现少量GT/CA或AT/TA重复序列。结果表明,GA/CT重复序列是花生基因组中最丰富的微卫星序列。
与霍普金斯等人之前关于花生微卫星的报告相比[21.[其中,使用文库筛选方法鉴定来自26个引物对(19%)的五种多态标记,在鉴定SSR标记中,我们的富集方法更有效,其中19个引物对(34%)显示多态性。尽管基于所使用的克隆总数的SSR标记的检测百分比几乎是相同的,来自之前的报告和4.7%(19/401克隆)的4.1%(5/120克隆),从我们的结果中,浓缩程序更快,更少成本比图书馆筛查。
花生的冗余速度高,而棉花非常低[31.].它降低了微卫星在花生中发育的功效。因此,需要更大的库来识别更多的微卫星标记。可以通过用不同一组AFLP片段的SSR探针杂交来构建文库,该碎片在AFLP引物的3'末端改变不同的单碱基,这应该具有16个可能的引物组合,或者使用不同限制导致的不同的限制片段组成酶组合。
在SSR的重复次数和SSR的信息之间没有发现相关性,尽管葡萄藤和番茄报告了正相关性[34.].轨迹PM50只包含191个图案,但具有高度多态性。其他基因座,例如PM244 [(GA)74]和PM209 [(GA)53.]的重复单位更均匀,但在不同的花生基因型之间没有DNA变异。未检测到多态性,重复次数超过30次。
随着机器人、成像、计算机测序和基因分型等高通量技术和设备的发展,更可靠的多态标记将被用于构建高分辨率的遗传图谱,在栽培花生中实现标记辅助选择甚至图位克隆。
结论
本研究的结果为从栽培花生中获得多态微卫星标记提供了一条简单可靠的途径。这一结果的意义在于,开发微卫星标记不仅对花生研究人员很重要,而且对花生工业也很重要,因为栽培花生中DNA标记的缺乏,迄今为止阻碍了对这种作物的详细遗传研究。
方法
利用MasterPure plant Leaf DNA纯化试剂盒(Epicentre, Madison, WI)提取花生植株基因组DNA,并经两种酶(后III和均方误差一世)。用相应的适配器连接消化的片段,然后使用单碱选择性引物(后III-A和均方误差I-T)根据标准AFLP协议[22.].三个生物素化的SSR探针,(AT)15.,(gt)15.,(ga)15.据报道,富含其他植物物种,与扩增的AFLP分开杂交。将约200kg预扩增的AFLP片段(从100bp - 600bp的长度范围为100bp - 600bp)加入到含有4.2×SSC(柠檬酸盐,pH7.0),0.07%SDS(十二烷基硫酸钠)的单一反应混合物中,和10pmol生物素化的探针。将混合物在95℃温育5分钟,并在冰上快速冷却2分钟。然后将其保持在适当的温度(取决于每个探针的熔化温度)1小时进行退火,用于探针(GT)15.在60°C,(AT)15.在37°C,和(GA)15.在57°C。同时,通过轻轻摇动小瓶以获得均匀浆料,制备Dynabeads M-280链霉抗生物素蛋白(10μg/μl)。将约20μl珠子浆料转移至1.5ml管,然后用300μl珠洗洗涤缓冲液(1×TE + 100mM NaCl)洗涤4次。用50μL相同的缓冲液重新悬浮珠子,并加入片段探针混合物中,并在室温下持续30分钟,持续缓慢搅拌。固定后,通过施加磁场除去上清液以沉淀珠子,该珠子附着与生物素化探针杂交的SSR的含片的片段。将珠探针片段复合物在室温下用400μL非严格洗涤缓冲液(1×TE + 1M NaCl)洗涤3分钟5分钟。将复合物在室温下用400μL严格缓冲液(0.2×SSC + 0.1%SDS)进一步用400μL严格缓冲液(0.2×SSC + 0.1%SDS)洗涤三次。最后洗涤后,完全除去洗涤缓冲液,加入40μl无菌水,轻轻粘接并在95℃下温育5分钟。含有单链的洗脱溶液将SSR富集的片段克隆到PCR4-Topo载体中,并将重组转化为TOP10化学竞争力大肠杆菌遵循TOPO TA克隆试剂盒(Invitrogen公司,圣地亚哥,CA)的指导。重组菌落37℃培养14-16 h。质粒用Qiagen Qiaprep Spin Miniprep试剂盒提取纯化。使用ABI PRISM进行测序®377使用The Big Dye Terminator Kit和Beckman Coulter CEQ800使用CEQDTCS快速入门套件根据制造商的说明。
引物用Primer3软件设计http://www-genome.wi.mit.edu/cgi-bin/primer/primer3_www.cgi由sigma - gensys合成。利用SSR引物从24份不同来源的花生材料中提取基因组dna(见表)1).大多数底漆对的PCR程序如下:初始变性的94℃/ 3分钟;94°C / 30秒,65至56°C / 30秒,72°C / 1分钟,每个退火温度下两个循环;94°C / 15秒,55°C / 30秒,72°C / 1分钟20个循环;72°C / 10分钟的最终延伸[23.].对于PM36、pm42、PM45、PM50、PM53、PM60等引物对,应优化PCR条件,找到合适的退火温度,如94°C/30秒、60°C/1 min或50°C/1 min、72°C/1 min循环35次(见表)2).将PCR产物在6%变性聚丙烯酰胺凝胶中分离3小时,在250电压下并通过银染色可视化。
缩写
- 妊娠:
-
-扩增片段长度多态性
- RFLP:
-
-限制性片段长度多态性
- RAPD:
-
- 随机扩增的多晶型DNA
- 苏维埃社会主义共和国:
-
- 重复简单的序列。
参考
- 1。
Weber JL:人(DC-DA)N(DG-DT)n多态性的信息性。基因组学。1990,7:524-530。10.1016 / 0888-7543(90)90195-z。
- 2。
Akkaya MS, Bhagwat AA, Cregan PB:大豆简单序列重复DNA的长度多态性。遗传学报。1992,132:1131-1139。
- 3.
Morgante M,Olivieri AM:PCR扩增的微卫星作为植物遗传学中的标志物。工厂J.993,3(1):175-182。10.1046 / J.1365-313x.1993.t01-9-00999.x。
- 4.
Gupta M,Chyi Ys,Severson Jr,Owen JL:使用简单序列重复的单引物引物扩增来自进化的不同基因组的DNA标记。Al Appl Genet。1994年,89:998-1006。10.1007 / BF00224530。
- 5。
Sharon D,Adato A,Mhameed S,Lavi U,Hillel J,Gomolka M,Epplen C,Epplen JT:使用简单序列重复和小卫星探头的植物中的DNA指纹。Hortscience。1995,30(1):109-112。
- 6.
Hamann A,Zink D,NAGL W:在属中的微卫星指纹识别菜豆.基因组。1995,38:507-515。
- 7.
荣文J,Akkaya Ms,Bhagwat AA,Lavi U,Cregan PB:使用微卫星DNA标志物进行大豆基因型鉴定。Al Appl Genet。1995,90:43-48。10.1007 / BF00220994。
- 8.
田中科,田村,田中村,等:微卫星标记的开发与多态性水青冈属crenata和密切相关的物种,F. japonica..Al Appl Genet。1999年,99:11-15。10.1007 / S001220051203。
- 9.
Cregan PB, Mudge J, Fickus EW, Marek DD, Denny R, Shoemaker RC, Matthews BF, Jarvik T, Young ND:利用细菌人工染色体靶向分离简单序列重复标记。应用网络。1999,98:919-928。10.1007 / s001220051151。
- 10。
植物基因组简单序列重复(SSR)位点的开发与应用。发表于:植物基因组分析方法。编辑:Jauhar PP. 1996, CRC出版社。纽约,伦敦,东京,147-159。
- 11.
Billotte N,Risterucci Am,Barcelos E,Noyer JL,Amblard P,Baurens FC:油棕的开发,表征和跨征集效用(Elaeis guineensis Jacq。)微卫星标记。基因组。2001,44:413-425。10.1139 / Gen-44-3-413。
- 12.
Edwards KJ, Barker JHA, Daly A, Jones C, Karp A:植物中多个微卫星序列富集的微卫星文库。生物技术。1996,20:758-760。
- 13。
陈志强,陈志强,陈志强,等:微卫星标记的开发、遗传和跨种扩增金合欢曼尼姆.Al Appl Genet。2000,101:1282-1290。10.1007 / S001220051608。
- 14。
王志强,王志强:微卫星分离技术的研究进展。分子生态学报。2002,11:1-16。10.1046 / j.0962-1083.2001.01418.x。
- 15.
花生品种和野生品种的RFLP变异。中国科学(d辑:地球科学),1991,11:565-570。10.1007 / BF00226719。
- 16.
Halward TM, Stalker HT, Larue EA, Kochert GA:利用分子标记检测栽培花生和相关野生种质资源的遗传变异。基因组学。1991,34:1013-1020。
- 17.
HALWART TM,Stalker HT,Larue EA,Kochert GA:在花生的遗传学研究中使用单引物DNA扩增(落花生hypogaea L).植物生态学报,1998,18(4):489 - 494。10.1007 / BF00034958。
- 18.
PAIK-RO OG,SMITH RL,KNAUFT DA:限制片段长度多态性评价六种花生物种落花生部分。《中国科学:地球科学》1992,84:201-208。10.1007 / BF00224001。
- 19.
Stalker HT,Phillips Tg,Murphy JP,Jones TM:Insozyme模式的多样性落花生物种。《中国科学院院刊》1994年第1期。10.1007 / BF00222901。
- 20。
赵小平,Kochert GA:水稻微卫星(GGC)的系统发育分布和遗传定位(英文)栽培稻L).植物生态学报,1998,21(4):593 - 598。10.1007 / BF00014544。
- 21。
Hopkins Ms,Casa Am,Wang T,Mitchell Se,Dean Re,Kochert Gd,Kresovich S:在栽培花生中发现和表征多态简单序列重复(SSR)(落花生hypogaea L).作物科学,1999,39:1243-1247。
- 22。
VOS P,Bleeker M,Reijans M,Van de Lee T,Hornes M,Fritjers A,Peleman J,Kuiper M,Zabeau M:AFLP:AFLP:DNA手指印刷的新工具。核酸RES。1995年,23:4407-4414。
- 23。
基于遗传算法的微卫星长度多态性检测。生物学技术。1993年,15:582 - 584。
- 24。
跟物HT,Mozingo LG:分子标记落花生和分子标记辅助选择。花生科学。2001,28:117-123。
- 25。
GARCIA GM,Stalker HT,Shroeder E,Kochert Ga:鉴定RAPD,瘢痕和RFLP标记与线虫抗性基因紧密相关的RAPD,SCAR和RFLP标记落花生cardenasii到A. hypogaea..基因组学。1996,39:836-845。
- 26。
花生RAPD标记的鉴定落花生hypogaea)与根结线虫抗性相关联答:cardenasii.分子品种。1996年,2:307-319。10.1007 / bf00437909。
- 27。
He GH, Prakash CS:栽培花生多态DNA标记的鉴定Arachis hypogea L.).植物学报1997,97:143-149。10.1023 /: 1002949813052。
- 28.
Kijas JMH,Fowler JCS,Garbett Ca,Thomas Mr:使用与链霉抗生物素蛋白涂覆的磁性颗粒结合的生物素化的寡核苷酸序列来富集微卫星。生物学技术。1994,16:657-662。
- 29.
Fischer D,Bachmann K:大型基因组的生物体中富集富集(Allium Cepa L.).生物学技术。1998年,24:796-802。
- 30.
宋庆杰,杨志强,杨志强:小麦微卫星基因组文库的构建。在2000年国家枯萎病枯萎论坛,辛辛那提,OH。编辑:Jauhar, 2000, 50-51。
- 31。
棉花基因组研究中新的二、三核苷酸微卫星标记资源棉花学报,2001,5:103-113。
- 32。
YU JK,Mangor J,Thompson L,Edwards KJ,Slabaugh MB,Knapp SJ:精英近交锋的简单序列重复的等位基因多样性。基因组。2002,45(4):652-660。10.1139 / g02-025。
- 33。
CiFarelli Ra,Gallitelli M,Cellini F:随机扩增的杂交微卫星(RAHM):分离新类含微卫星的DNA克隆。核酸研究。1995,23(18):3802-3803。
- 34。
陈志强,陈志强,陈志强,等:不同来源番茄微卫星标记多态性与染色体定位的比较分析。应用电子学报。2002,104:229-235。10.1007 / s00122 - 001 - 0775 - 2。
致谢
作者感谢DRS。Albert Abbott和Naveen Puppala对手稿和鼓励的有用评论。这是来自乔治华盛顿雕刻农业实验站的326号贡献。该研究得到了USDA / CSREES / CBG的授权(No. 00-38814-9541)。提及本文中的商品名或商业产品仅为提供具体信息,并不意味着批准排除可能是合适的其他产品。
作者信息
隶属关系
通讯作者
额外的信息
作者的贡献
GH构思并协调项目,参与序列分析,底漆设计,并起草了手稿。RM和GG对新型引物进行了富含SSR富集的文库,克隆序列和多态性的测试。MN进行测序克隆。RP和CP参与协调并修订了稿件。所有作者阅读并认可的终稿。
作者为图像提交的原始文件
以下是与作者的原始提交的图像的链接。
权利和权限
关于这篇文章
引用这篇文章
他,G,孟,R,纽曼,M。et al。微卫星作为栽培花生的DNA标志物(落花生hypogaeaL.)。BMC植物BIOL.3,3(2003)。https://doi.org/10.1186/1471-2229-3-3
已收到:
公认:
发表:
关键字
- 微卫星标记
- 落花生Hypogaea
- 丰富程序
- 妊娠片段
- 花生基因型