摘要
背景
大多数基因拟南芥是基因家族的成员。基因家族的成员是如何产生的,基因家族的拷贝数又是如何维持的?一些基因家族可能主要通过串联复制和集群中的高出生和死亡率进化,而另一些基因家族可能通过罕见的多倍体或大规模的片段复制和随后的损失进化。
结果
我们理解基因家族进化机制的方法是构建50个大基因家族的系统发育拟南芥,识别较大的内部段重复拟南芥,将基因重复映射到节段重复上,并使用这些信息来识别每个系统发育中的哪些节点是由于节段或串联重复而产生的。举例说明六个基因家族的特征模式描述。还描述了基因家族大小的分布和基因组距离的复制模式,以便描述大基因家族的局部复制和拷贝数的模式。基因家族大小和距离复制都密切遵循幂律分布。
结论
结合有关基因组片段复制、基因家族系统发育和基因位置的信息,提供了一种评估串联复制和片段基因组复制在基因家族的生成和维持中的贡献的方法。这些差异似乎与基因家族成员在功能角色上的差异有意义对应。
背景
大多数基因拟南芥是基因家族的成员。所有预测蛋白质之间的相似性搜索显示65 - 85%拟南芥基因与基因组中至少一个其他基因同源,这取决于相似阈值([1并在本文中进行了分析)。基因家族大小范围广泛,有400多个受体激酶基因[2,3.], ~270 - 285个细胞色素P450基因[1,4,5],以及许多小家庭或独特的基因。我们在基因家族大小和分布中观察到的巨大变化可能受到许多过程的影响,包括具有高出生和死亡率的串联复制,以及由更大规模基因组事件(如多倍体或大染色体区域的复制)引起的基因复制(在本文中称为“段复制”)。我们提供了在50个大基因家族的进化中明显的基因复制模式的定量表征答:芥.
完整的排序答:芥基因组揭示了许多大规模的片段复制[1,6- - - - - -10].一些研究得出结论,至少有两轮重复可能发生在地球上答:芥基因组,由于许多损失和重排,留下了“片段复制”或“复制块”的马赛克[7,10- - - - - -14].大多数复制块似乎来自于一轮多倍体,通过各种方法估计发生在属进化之前的20 - 40 Mya芸苔属植物但在芸苔科从其他近缘芸苔科分离后[7,10- - - - - -12].存在于重复区域的基因组部分可作为评估给定基因家族中的基因是否以高于整个基因组预期的速度丢失或保留的基线。如果大多数复制块确实是在一轮多倍体中产生的,那么这种复制也可以用来提供一个内部参考点,用于比较不同基因家族成员的氨基酸取代率。
虽然多倍体是基因家族拷贝数扩展的一种机制,但串联复制或局部复制是最常见的基因家族扩展机制。串联复制通常是由于不平等的交叉造成的[15]和多次不平等的交叉可能导致基因家族的拷贝数增加或减少,或者导致基因的简单循环,而基因家族的大小没有大的变化。虽然本文没有研究,但转座因子可能也在基因复制和基因组重排中发挥了重要作用拟南芥[16].
为了确定片段和局部重复在大型基因家族进化中的相对重要性,我们开发了一种软件来识别基因家族系统发育中通过片段或局部重复产生的分支。在50个大基因家族中答:芥,我们发现这两个过程的贡献在不同基因家族之间有很大差异。我们讨论了这些差异在基因家族进化中可能的生物学意义。
结果
策略
我们的一般方法包括以下步骤。方法部分描述了详细信息、参数和软件。
1)选择初始基因家族和初始序列成员。我们从2001年开始拟南芥PIR超族,可在MIPS [17],并在随后的步骤中细化家庭成员。
2)在结构域排列的基础上缩小基因家族选择范围。我们确定了Pfam [18]每个基因家族中所有序列的结构域,评估每个家族中结构域排列的一致性,并排除结构域排列特别复杂的家族,例如几个激酶家族中的结构域。
3)迭代构建和优化基因家族比对。我们构建了T-Coffee [19],然后生成隐马尔可夫模型(hmm),将每个家族中的所有蛋白质重新排列到模型中,使用该模型研究预测的完整集拟南芥蛋白质,检索期望值小于10的序列-10,并将它们重新对齐到HMM。
4)用于系统发育分析的修剪对齐。这涉及到删除indel区域,首先删除HMM中“匹配状态”之外的残留物,然后通过视觉检查并在某些情况下删除其他对齐不良或indel区域。
5)计算系统发育。我们生成了简约和自举邻居连接树,并计算了简约拓扑的最大似然分支长度。
6)预测部分重复拟南芥基因组,使用DiagHunter [20.,21].在染色体之间氨基酸相似性“命中”的二维点图中,部分重复出现为对角线特征。形成这些特征的同源基因对将在下一步中使用。相似度为BLASTP位评分阈值500,其他参数描述在[21].
7)确定一个基因家族中的基因对,其坐标与一对复制块中的相同。任何这样的基因对都可能与该基因对的复制块同时复制。我们使用为此目的开发的OrthoParaMap软件进行了此操作和接下来的三个步骤,并在[22,23].
8)用重复区隶属度信息注释基因系统发育。推断可能是通过分段重复产生的节点,并用此信息注释系统发育。
9)使用基因位置信息来推断哪些密切相关的基因(根据基因系统发生中的位置定义)在物理上彼此“靠近”(根据基因之间的物理距离定义,如下所述)。推断可能通过串联复制产生的节点,并用该信息注释系统发育。
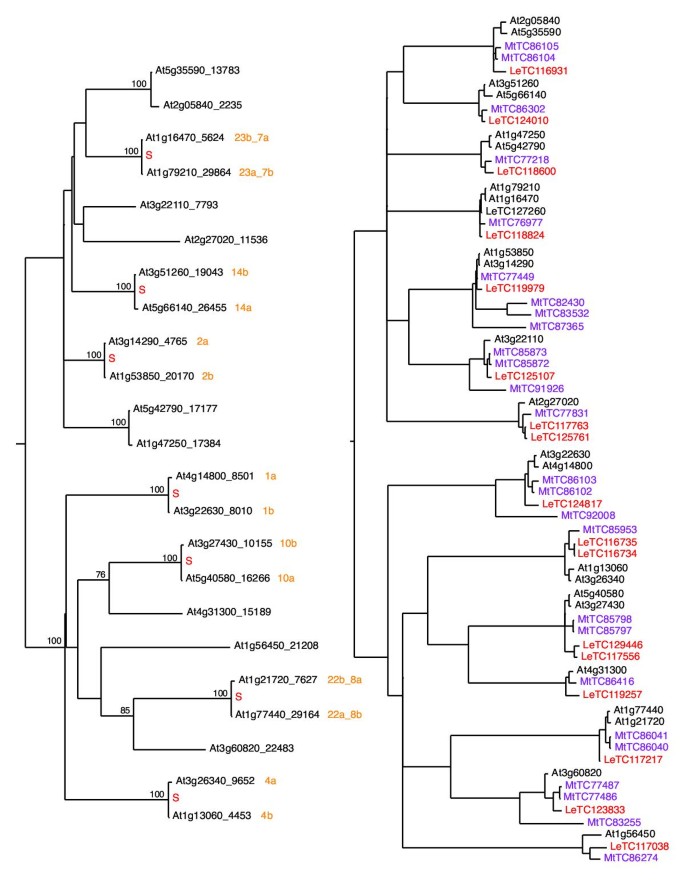
10)添加翻译自其他物种的EST共识序列,以帮助提供额外的上下文。这涉及到使用答:芥分析大豆TIGR单基因集,m . truncatula,Lotus对虾,西红柿,土豆和玉米,然后选择最长的翻译,将它们对齐到HMM,并使用与之前相同的程序重新计算系统发育答:芥(步骤5).虽然通常不是本项目的组成部分,但这些信息有助于确定一些家族的进化模式,特别是由小而高表达的蛋白质组成的家族。由于空间限制,数字5,6,7,8只包括答:芥,Medicago,和番茄序列,尽管所有序列的系统发育都包括在[24].
研究集从所有大型中选取答:芥基因家族
对许多基因家族的高通量系统发育分析在一开始就因构成基因家族的问题而变得复杂[25- - - - - -28].从概念上讲,基因家族有一个共同的祖先,由基因复制产生,并且可能具有相似的功能。基因家族中序列和功能的多样性常常给基因家族的划分带来困难。在操作上,基因家族可以根据序列相似性和结构域组成的水平来定义,但如果阈值不恰当地分割了一个发散的超家族,或不恰当地将具有共同结构域的独立基因家族分组在一起,则简单的相似性阈值可能会产生误导[25,26].
为了限制本研究的范围,并避免具有不同结构域排列的超家族所带来的一些复杂性,我们随机选择了50个至少有20个成员、共同功能域和一致的家族成员的基因家族。一致性家庭成员由HMM搜索的期望分数分布来判断(使用hmmer [29])答:芥蛋白质组。研究人员优先考虑那些HMM e值明显下降的家庭,这些家庭成员的得分最低为10分-10非会员的分数一般要低得多。除了最小的家族规模,我们选择了更深入研究的家族,尽管有些家族没有已知函数或Pfam域的成员[18].最后,我们选择了一个家族大小范围的家族,从20个成员的家族到大约225个成员的细胞色素P450超家族(尽管P450基因的总数答:芥,包括所有不同亚家族的成员,估计有275 - 285个基因[1,4,5])。本研究中使用的家庭如表所示1.
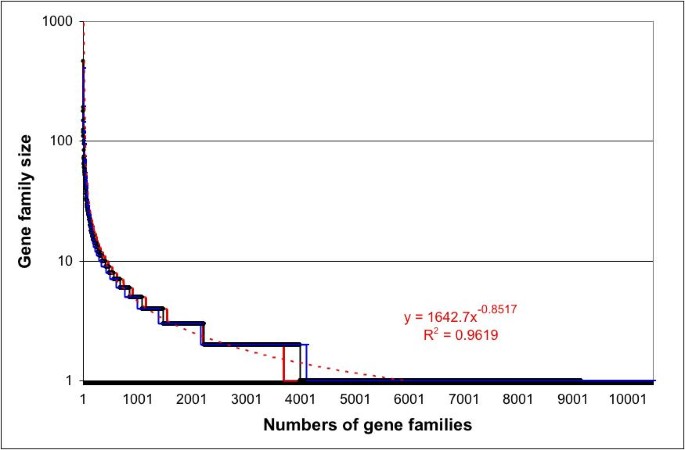
为了了解基因家族大小的分布,我们还在两个BLASTP上进行了简单的全蛋白质组同源性搜索和单连锁聚类[30.阈值。在这种情况下,单连锁聚类传递性地合并基因集,其中任何基因与该组中的其他基因足够相似。这些结果如图所示1.分布紧密地遵循幂律(图1;Y = 1642.7x-0.8517, R2= 0.96)。在这种分布中,成员多的家庭很少,成员少的家庭很多。幂律分布值得注意,部分原因是它需要一种演化和维持家庭规模的机制。任何提出的机制都需要与各个家族中个体基因复制和丢失的机制相一致。
使用BLASTP e值阈值为10-10(图1)进行单连锁聚类,得到成员不少于20人的潜在基因家族181个,成员不少于50人的潜在基因家族46个,成员不少于100人的潜在基因家族13个。至少85.7%答:芥基因在这个阈值上有一个或多个同源物。使用BLASTP阈值10-20年单连锁聚类生成140个成员不少于20人的潜在基因家族,40个成员不少于50人的潜在基因家族,10个成员不少于100人的潜在基因家族。至少80.6%答:芥基因在这个阈值有一个或多个同源物。这些BLAST和聚类结果提供了基因家族大小分布的近似描述。选择用于进一步分析的50个基因家族进一步细化,详见Methods。
串联重复量化
我们的目标是区分由染色体区域的分段复制引起的基因复制和产生附近基因副本的串联复制。这需要对基因相似性和基因组接近性进行操作定义。相似性应该在基因系统发育的背景下确定,因为不同家族的不同基因进化的速度不同。我们将串联重复的搜索限制在从末端节点到系统发育中近似中点根的平均进化距离≤75%的序列,即最大搜索深度。这在某种程度上是任意的界限,但避免了系统发育中非常早期的重复,因为这种重复的机制很难推断。为了确定两个基因在物理上是否足够接近,从而得出它们可能是通过串联复制产生的结论,我们测量了超过基因组平均水平的重复数量的平均基因组距离。
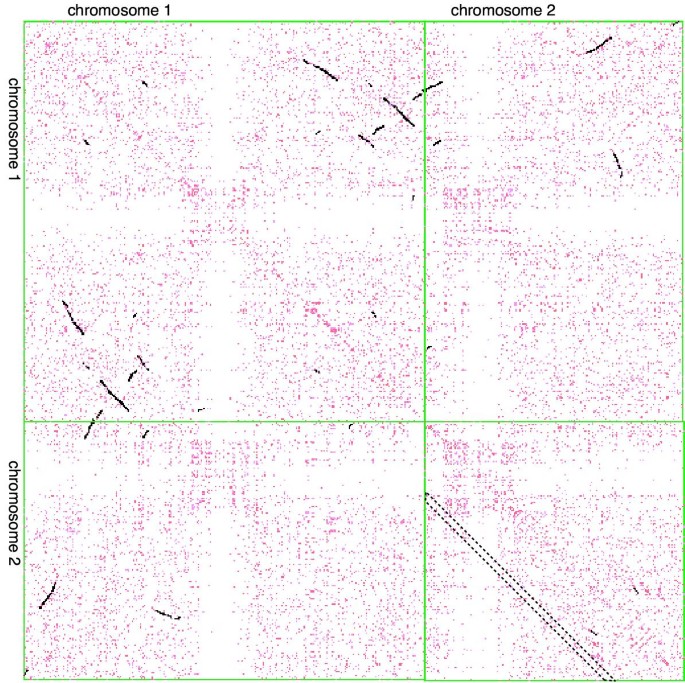
遵循Vision等人的方法。[9],我们使用点图来绘制位于不同基因组区域的两个相似序列的出现。沿着基因组区域线性序列的位置被绘制为X轴和y轴,每个点位于XY坐标上,标志着相似性“命中”(图2).染色体与自身比较的点图,例如染色体。1 by chrom。1、定位染色体内的局部串联复制和节段事件。分段事件表示为与主对角线方向相同或相反但位于主对角线以外的点的密集线性阵列。
点图中任意部分的点密度表示所比较的基因组区域之间的匹配密度。如果在一个染色体区域中发生了大量的串联复制,这将是一个密集的点状区域,位于染色体的主对角线附近。我们展示的点图不包括主对角线本身(显示每个蛋白质与其自身的相似性)。在基因之间任何给定距离上的平均密度都可以通过点阵图的对角线条计算出来。图中以其中一条为例2.
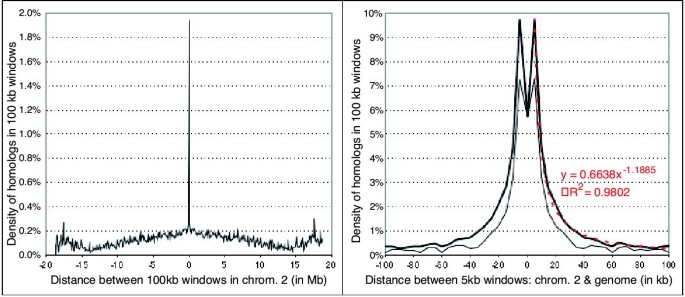
数字3.通过2号染色体点阵图显示对角线条带的平均密度。左图为整个2号染色体的比较图,右图为上游或下游100 kb以内的重复图。根据我们的分析,2号染色体没有可检测到的内部片段重复,因此具有最干净的重复图(图2)3.(左面板),峰值以0为中心表示串联副本。
通过距离观测将密度分解为更近的间隔,我们接下来在5 kb窗口中绘制密度。图中的图形3 b显示了类似的密度测量,但在5 KB窗口内,从查询的100 KB下游到染色体2的100 KB上游。得到的图与图中5条染色体非常相似3 b也显示了基因组平均。在每一种情况下,我们都发现在50 kb物理距离内有大量的局部重复。单位是每面积的同源,其中有意义的单位面积可能是(5 kb)2.使用的理由(5kb)2分母是In答:芥,平均基因密度约为每5 kb一个基因,因此如果所有基因都是同源的,则任意两个5 kb区域之间的同源基因数为1。每(5 kb)个同源物的值2在答:芥因此可以将其描述为一个密度单位(d.u.;这是本文的一个新术语)。正如预期的那样,局部重复的最高密度在5 kb处(图3 b).在任何基因从5 kb到10 kb的窗口中,明显重复的基因的密度(BLASTP阈值为10-10)在全基因组范围内为0.098 d.u.。这意味着,平均而言,在任何两个相隔5kb的5 kb窗口内,都有大约0.1个同源基因,或者10个重复基因中就有一个可能在非常近的地方有同源基因。在以任何基因为中心的100 kb窗口中,复制基因的相应密度约为0.020 - 0.035 d.u,这取决于染色体。在所有染色体中,超过50 kb未见明显的局部复制效应。因此,我们将串联复制定义为彼此之间距离在50kb以内的密切相关基因。
局部复制基因密度随距离的分布遵循指数分布(R2值为0.98,图3.).在这条曲线下积分,曲线下5 kb到100 kb区间内90%的区域都在5 kb到50 kb的较小区间内,并且表示密度高于平均背景密度0.002 d.u。再次,这支持使用50 kb作为识别局部重复的合理阈值答:芥.
串联复制和分段复制的期望值
为了比较基因家族在大小上存在显著差异时串联复制和节段复制的相对贡献,我们为每个大小类别的基因家族生成串联和节段复制事件的期望值,计算这两种机制的观察值与期望值的比值,并比较每个家族的比值。
我们模拟了在给定大小的基因组中,对于给定大小的基因家族,将偶然发生的串联复制的预期数量的分布。该模拟程序是随机放置N个基因在一个100,000 kb的基因组(大约范围的纯色DNA在答:芥),并计算彼此之间50 KB以内的基因数量。总共1000次模拟运行生成每个基因家族大小的分布。对于小基因家族,两个基因彼此靠近的概率遵循泊松分布。例如,对于一个20个成员的基因家族,观察到0.12个相邻基因的均值和方差。对于较大的基因家族,概率接近正态分布。例如,对于100个成员的基因家族,观察到相邻基因的平均值为4.0个,方差为15.4,标准偏差为3.92。模拟提供了一种解释单靠偶然性预期的串联重复的方法,我们将观测值与之进行比较(表2)2).如下图所示,对于大多数基因家族,期望值远低于观测值,因为串联复制过程并没有在整个基因组中随机分布副本。
我们计算分段重复的期望数量的目标是建立一个易于解释的归一化常数(与为标准零假设建立值的目标不同)。我们的假设是,大部分由片段重复导致的基因已经丢失,我们想要一种方法来比较家族之间的损失程度,而不仅仅是由于大量重复区域的丢失。通过我们识别分段重复块的方法[20.,21],约占全色部分的75%拟南芥基因组至少存在一个复制区。如果这些复制区域内的所有基因都被保留,那么(所有其他因素都相等)在部分复制区域内的预期基因副本的比例也将是75%。事实上,保留基因拷贝的比例远低于这一比例,但75%提供了一个基线和归一化常数,用于比较不同大小基因家族中由于节段复制而观察到的基因拷贝数(表2).
串联和分段重复的计数
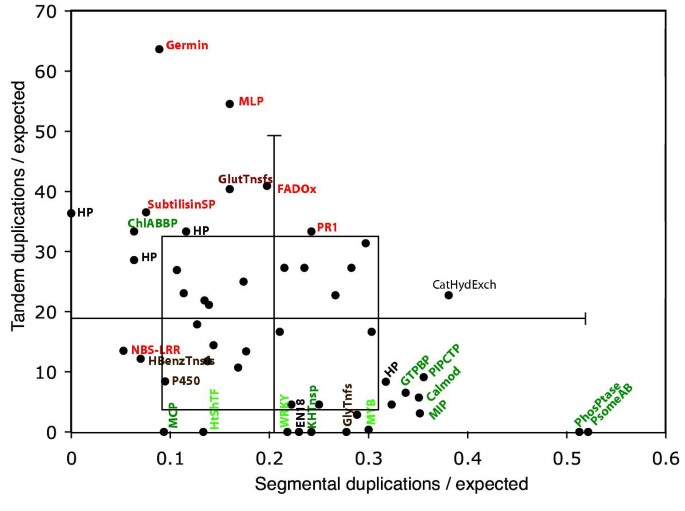
表格2显示每个家族中串联和节段重复的数量,以及这些数量与每个家族规模的串联或节段计数的预期基因组平均的比率。其他类型的事件,包括转位或部分重复的残余,没有被分类。观察到的/预期的串联重复计数之比显示了从0到63的巨大范围;一些家族显然是没有串联复制的结果,而有一个家族,Germin家族,在串联阵列中显示的基因副本是偶然预期的63倍。观测到的节段重复事件与预期的节段重复事件之比为0 ~ 0.52;有些家族丢失了模型预测的所有重复片段,而有些家族只丢失了模型预测的一半重复片段。表格2将基因家族按观察到的/预期的串联复制事件的低、中、高比率分组,然后按观察到的/预期的片段复制事件的低、中、高比率分组。为了生成这些类,将截断值设置为中值上下一个标准偏差。串联和分段类别中的几个家族也低于两个标准偏差,这些也在表中显示2.
表中所示基因家族2倾向于落入高串联/低节段复制类别,反之亦然,这在预期/观察到的分段和串联重复图中发现的中度负相关中很明显(相关系数= -0.47;R2= 0.22;p = 0.00057为方差分析f统计量;数字4).在8个低串联复制家族中,没有一个属于低段复制类别,其中两个具有段复制计数,使它们大约比段-复制中值高出两个标准差。在8个低节段复制家族中,没有一个属于低串联复制类别,有5个比观察到的/预期的串联复制事件的中位数比下降了一个以上标准偏差。与预期相比,PR1和CatHydExch等基因家族具有大量的节段或串联重复,但两者的数量都不高。无论是观察到的串联事件/预期的串联事件的比率,还是观察到的片段重复/预期的片段重复的比率,都与基因家族大小(R2数值分别为0.044和0.066,p值分别为0.14和0.08)。
在我们的50个基因家族中,低串联复制类似乎由高度保守的、管家性的或关键的调控基因家族所代表,而中、高串联复制类则由涉及病原体防御或不同酶功能的家族所代表。参与病原体防御的家族都属于中串联或高串联复制类;国家统计局- lrr [31,32],索马汀[33],葛明[34,35], pr1 [36],以及主要乳胶蛋白/PR10家族[37].低串联复制类包括三个转录因子家族中的两个(热休克和WRKY)和一些管家基因家族(线粒体载体蛋白[38,39],蛋白酶体20S亚基家族[40,41])。
多物种的基因系统发育
一些系统发育只包括答:芥序列似乎有很长的内部分支-可能表明快速进化。来自其他物种的同源序列的添加提供了一种测试这些家族中的基因是否迅速进化的手段,或者长内部分支是否表明高度保守的蛋白质序列之间存在古老的差异。这种方法如图所示5, 20S蛋白酶体亚单位家族的系统发育[41,42].右边的系统发育包括三个物种的代表:答:芥,西红柿,和m . truncatula.长内部分支末端序列的紧密聚类表明,该家族由高度保守的氨基酸序列组成,这些氨基酸序列已在这些基因组中保留了很长时间——尽管应该说,这里所代表的分类群是相当密切相关的双子叶植物,而来自基础被子植物或裸子植物的序列可能在系统发育中处于更深入的位置。类似地,来自多个物种的序列也被用于图中所示的比较7而且8.
这里采用的多物种方法通常提供了进化模式的定性而非定量措施。番茄和Medicago,我们使用易出错的翻译EST暂定共识(TC)序列。然而,对于具有高表达、相对较短的转录本的基因家族,这些信息可以估计出它们之间的最小进化距离答:芥和其他双子代基因同源物。
讨论
本文描述了串联复制和片段复制对基因家族大小和进化的相对重要性的差异答:芥基因组。串联复制显然是在基因组簇中产生新基因副本的重要引擎,在基因组簇中,不平等的交叉产生新的多样性。节段复制事件具有不同的影响,因为它们可能在整个基因组中广泛分散基因副本,而它们很少与亲本副本进行重组交换[43].为了研究这些基因组过程对多基因家族进化的共同作用,我们将基因家族分为低、中、高串联复制类和低、中、高片段复制类,并研究了每个复制类中一些研究较好的家族的属性。
基因家族大小的分布
拟南芥基因家族大小的频率分布密切服从幂律关系(图1).Huynen和van Nimwegen对其他基因组的这种分布提出了一个合理的解释[44].在他们的模型中,基因家族是由一个祖先建立的,通过复制和删除,家族规模随着时间的推移而波动,家族有可能从基因组中灭绝。该模型的要求是,一个家族中的所有成员在任何给定时间都有相同的复制或丢失概率,不同的基因家族在任何给定时间都可能有不同的概率,并且所有复制概率的平均值小于1(防止基因家族增长到无穷大)。在这些一般条件下,该模型生成存活基因家族大小的幂律分布[44- - - - - -46因此,不需要用选择来解释基因拷贝数的分布本身.
尽管如此,我们的观察表明,自从分段事件发生以来(最近的一次估计发生在20 - 40亿年),7,9,12],在不同基因家族的分段复制区域中保持了不同数量的基因拷贝。例如,在进化较慢的MYB基因家族中,由片段复制产生的基因副本在多倍体后更常被保留(表2, (24]),然而,在快速进化的大型NBS-LRR抗病家族中,局部基因组簇的重复是常见的,片段重复的保留率低得惊人。下面,我们考虑每一类基因家族的基因复制模式可能的生物学意义。
低串联,低段重复
8个基因家族被划分为低串联复制,其中大部分属于中等或高片段复制类。与全基因组平均水平相比,少数家族表现出较低的串联复制水平也表现出相对较低的片段复制水平。线粒体载体蛋白家族(MC或MCP [38,39])和热休克转录因子家族[47]每个都保留了很少的片段重复,但没有表现出明显的串联重复或聚集组织。MC蛋白作为反转运体,优先将一种溶质交换为另一种溶质[38,39,48].MC家族的结构特征成员是二聚体。可以想象,额外的基因拷贝可能会破坏这些跨膜复合物中蛋白质二聚体的化学计量学,特别是一旦复制的基因在多倍体后丢失。蛋白质复合体成员的基因复制和丢失模式是否通常不同于单体蛋白质的模式仍有待检验。
在50个基因家族中片段损失的方差中,相对较大的一部分可能是多倍体后基因组损失的随机过程的结果,因此,将出现比串联复制和损失更粗粒度的过程。在拟南芥在美国,许多兆酶复制块被保留,而其他非常大的区域已经丢失。在任何情况下,非常高或非常低的明显片段重复的更极端的情况需要在下面进一步说明;蛋白酶体20S亚基家族的高片段重复和NBS-LRR家族的低片段重复。观察到的/预期的段比值分别为0.41和0.05。
低串联,高分段重复
在低串联复制类或接近低串联复制类的家庭中,很大一部分也属于高分段复制类(表2)2).这些包括参与多种作用的蛋白质:转录因子(MYB),信号(GTP结合蛋白,钙调蛋白,磷酸蛋白磷酸酶),各种酶功能(糖基转移酶,质体色素苷),膜转运(主要固有蛋白)和细胞内务作用(蛋白酶体20S亚基)。
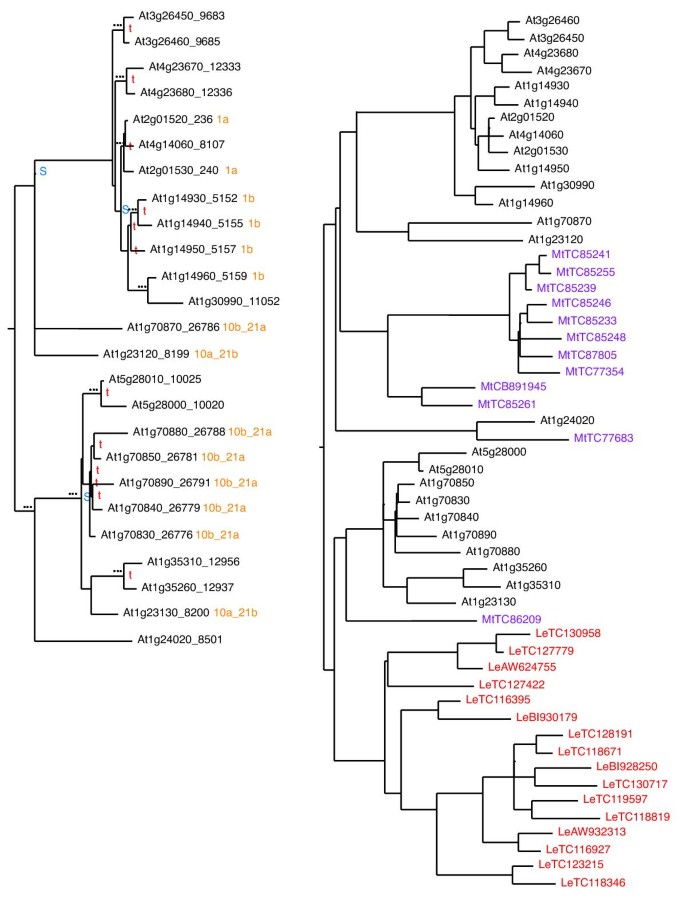
蛋白酶体20S亚基家族提供了一个有趣的研究案例,可以考虑复制过程和基因拷贝数的可能限制[22,23].在真核生物中,蛋白酶体通过降解泛素标记的蛋白质来回收蛋白质[41,42].它是一种大型蛋白质复合体,由28个亚基催化柱状结构(称为20S蛋白酶体)和一个atp依赖的19S调节粒子(由另外一组约18个亚基组成)[49].20S蛋白酶体由四个堆叠的环组成。中间的两个环各由7个20S β多肽组成,这些环夹在两个α环之间,每个α环由7个多肽组成,形成7α 7β 7β 7α结构[42].在迄今为止所描述的大多数真核生物中,7个α和7个β亚基中的每一个都彼此略有不同,需要14种蛋白酶体亚基才能组成20S蛋白酶体[50].
在答:芥20S蛋白酶体有23个基因编码20S蛋白酶体亚基[40,41,51,52——而不是14。系统发育图5暗示了额外亚基的起源。20S蛋白酶体序列有两个大的支,每个支代表α或β亚基,每个α或β支由七个支或谱系组成,代表不同的α(或β)序列[40,42)(图5).有趣的是,似乎有两组几乎完整的α和β亚基,因为我们发现23个序列由9对(总共18对)加上5个单子组成,而不是预期的14个序列。我们在这棵树上发现了7个节段复制的实例,另外两对分支的短长度表明,这些也可能代表了相同的,最近的多倍体事件的残余(图2)5).
根据生物学、系统发育学和基因组背景信息的综合分析,在一轮基因组翻倍之后,大约20-40 Mya [7,10- - - - - -12],复制的蛋白酶体亚基的大多数成员被保留下来,但有5个副本已经丢失。该基因家族中不存在串联复制,因此这两组几乎完整的20S亚基显然是通过单独的片段复制产生的。维持七个α和β谱系表明维持20S组分的化学计量学,而耐受重复亚基可能提供更大的调节或催化灵活性[49].摩尔和普鲁加南[53]描述了积极选择的优先级,驱动至少一些重复基因的固定和保存拟南芥.不管蛋白酶体亚基家族中片段重复保留率高于预期的原因是什么,这种模式与下面NBS-LRR抗性基因的例子形成了对比,后者显示了一些植物家族中基因家族成员的快速更替和主要基因谱系的丧失。
中等串联,低节段重复
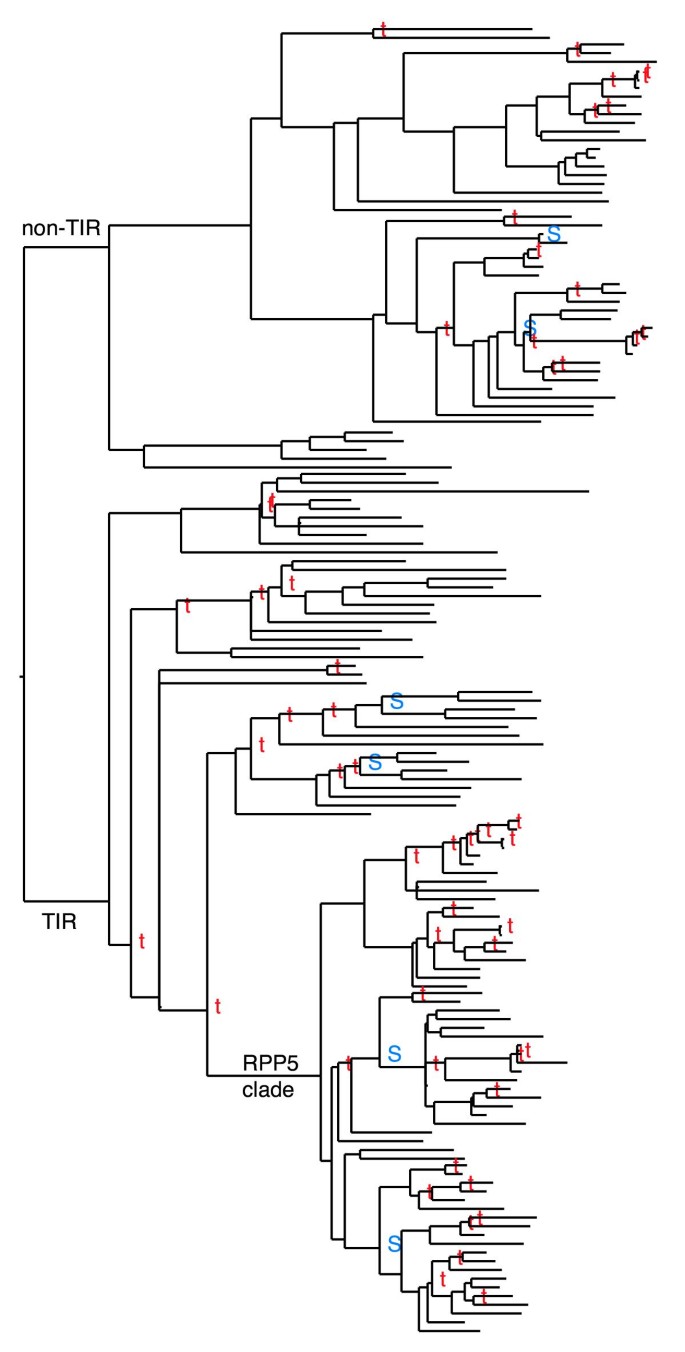
中等串联复制类中的三个家族也属于低片段复制类:蛋白质磷酸酶家族、酰基转移酶家族和NBS-LRR抗病家族。NBS-LRR抗病基因家族共有152个成员答:芥根据我们的搜索标准。家族成员已被证明对多种病原体具有抗性[31,32,54- - - - - -56并且具有巨大的经济重要性,所以我们在这里把它们作为一个例子。
我们发现了54个串联复制事件和6个片段复制事件。对于TIR亚家族中最大的分支之一(包含60个成员的RPP5分支),我们在这个单一的RPP5分支中确定了25个串联重复,其中24个发生在分段重复之后。片段复制映射到复制块,可以追溯到最近一轮多倍体,估计发生在芸苔科从其他双叶科分离之后,大约20 - 40 Mya [10,57].鉴于芸苔科物种之外没有密切的同源物[31],一个可能的情况是,一个RPP5祖先序列经历了串联复制,也许是另一个转座复制,随后通过多倍体放大,然后经历多轮串联复制。
在NBS-LRR耐药基因家族中,Baumgarten等人[43]发现片段复制在很大程度上解释了NBS-LRR同源物的全基因组分布。在这里,我们扩展了这些发现,以表明串联重复和丢失在影响中起主导作用拷贝数如RPP5同系物的扩展。事实上,多倍体后基因的净损失,加上动态扩展和损失,可能导致任何给定分支中序列数量相当不稳定。在庞大的NBS-LRR基因家族中,我们观察到几个分支,我们可以证明很少或没有串联复制[31].也许序列丢失最引人注目的例子是Poaceae中完全缺乏TIR NBS-LRR序列,尽管禾草和NBS-LRR基因家族都有丰富的序列[32,58].草类中TIR序列的缺失可以推断为草类中这种序列类型的缺失,因为在松树中发现了TIR同源物[31]和苔藓[59].就像某些分支迅速扩张一样,如RPP5分支,其他谱系,如草中的整个TIR亚家族似乎已经消失了。
高串联,低段复制
在11个高或极高串联复制家族中,5个属于低段复制类,另外4个家族的段复制水平低于中位数。高串联、低到中等节段复制家族可分为几个广泛的功能类别。串联复制水平高于基因组中位数两个标准差以上的两个家族是胚芽蛋白和主要乳胶蛋白(MLP)家族。这两个家族都参与病原体防御以及其他功能。人们发现胚芽发挥多种作用,包括萌发过程中细胞壁的形成、与应激相关的信号传递、活性氧的产生以及作为真菌毒素的草酸盐的降解[60,61].其他家族成员被证明在防御病原体中发挥作用的是枯草蛋白酶样丝氨酸蛋白酶和病原体相关的PR1家族[36].与多样性防御序列的明显优势相反,为什么叶绿素a-b结合(CAB)家族保留了大量的串联重复?先天的, CAB家族可能比高度重复的防御相关家族进化得保守得多,因为这些蛋白质形成了光系统I和II的大型多蛋白复合体[62].我们将首先描述不同寻常的CAB家族,然后是MLP家族。
高串联,低段复制,示例1:CAB
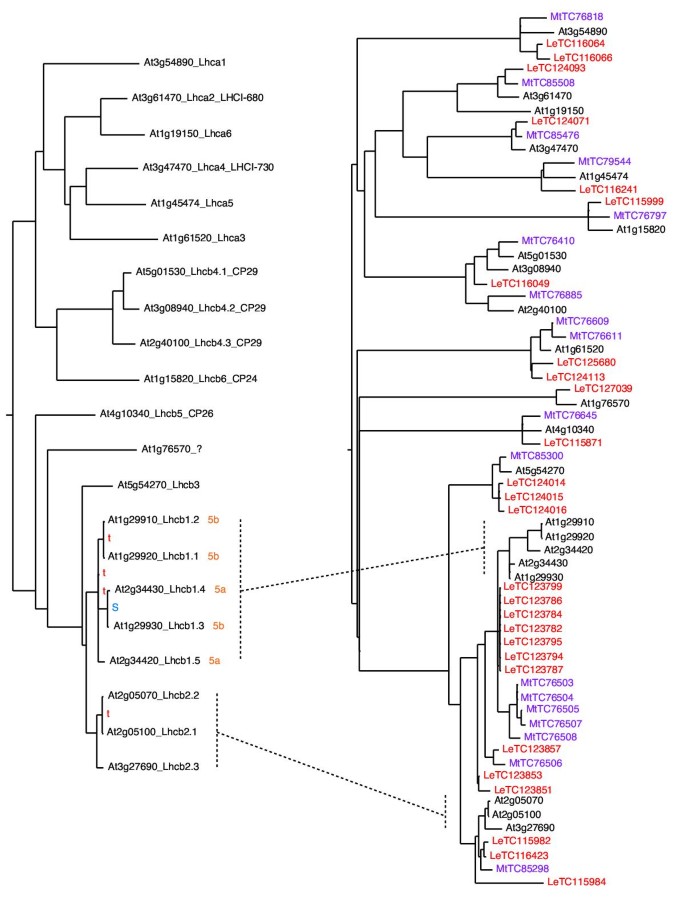
CAB蛋白是复杂多亚基光系统I和II (PS I和PS II)的组成部分[63,64].这两种光系统都由叶绿素结合核心和外围天线或光收集复合体(LHC)组成。除了光收集功能外,天线还能够通过一个称为反馈去激励的过程来耗散多余的能量[65].PS I和PS II的lhc中至少有10种不同类型的蛋白质[62,64],由核编码大型强子对撞机基因。自早期陆地植物进化以来,光系统的基本结构一直是保守的[62,64].四个大型强子对撞机标记与PS I相关的基因lhca1-4.标记与PS II相关的基因lhcb1-6,lhcb1而且lhcb2也与PS I有关[62- - - - - -64,66].尽管自早期陆地植物进化以来,光系统的基本结构一直是保守的[62,64],我们的结果显示了令人惊讶的动态拷贝数,特别是对于lhcb1,lhcb2,lhcb3.
数字7的系统发育答:芥CAB家族(左侧),作为比较,一个系统发育还包括来自番茄和m . truncatula(右侧)。在答:芥基因系统发育过程中,检测到4个串联重复和1个节段重复lhcb1,lhcb2,lhcb3.在三种基因系统发育中,存在多个lhcb1,lhcb2,lhcb3番茄和Medicago.这些平行谱系显示了每个物种谱系中最近的独立扩张,以产生彼此更相似的平行谱系,而不是来自另一个物种的同源谱系。在A. thaliana lhcb1以分枝为例,在芸苔科分化后,通过3个串联复制产生了5个平行类群,在芸苔科分化前产生了1个分段复制。在这3个物种基因树的相应分支中,至少有11个番茄序列,其中6个出现在番茄/答:芥分开,至少六个Medicago序列,其中1出现在公司/ A。芥分裂。类似的系统发育模式在玉米、大豆和马铃薯的EST数据中很明显(未显示)。相比之下,没有一个lhca基因和lhcb4-6基因表现出近期基因拷贝数的扩增。
在CAB家族的不同部分似乎有不同的进化模式在起作用。系统发育表明,在高周转率lhcb1-3基因和低周转率,另一种可能是部分重复的丢失大型强子对撞机基因。结构和功能研究表明,在PS II中,核心复合物的二聚体两侧有两个蛋白质,每个蛋白质编码lhcb4,lhcb5,lhcb6.它们的两侧依次有四个三聚体lhcb1,lhcb2,lhcb3[64,67,68].Lhcb4是功能PS II形成的必要条件,但如果表达lhcb1而且lhcb2被抑制[67,68]和抄写lhcb5被强烈上调。然而,Lhcb1而且Lhcb2蛋白质在弱光条件下确实发挥着重要作用[64]和建立适当的粒层结构,以及Lhcb5不能完全补偿这些功能[64].因此,进化的灵活性lhcb1-lhcb3基因可能提供了一种机制,在不同的光照条件下调节光收集复合物[65],而相比之下,基因组分散,进化更稳定,lhcb4,lhcb5,lhcb6基因维持PS II的光合核心。
主要乳胶蛋白(Major Latex Protein, MLP)家族编码最初从罂粟乳胶中分离出来的蛋白质[69,70]也广泛存在于各种植物和组织中[71].MLP的功能尚不清楚,但它们确实显示出与致病相关蛋白(IPR或PR10蛋白[37]),在病原体或应激挑战下表达增加[37,72- - - - - -74].这两个基因家族(MLP和IPR-PR10)的成员之间只有大约25%的同源性,但序列和结构分析表明,它们足够相似,可以被认为是单个超家族的一部分。37].有趣的是,没有答:芥与IPR-PR10亚家族同系物[37],但我们为MLP家族定位了11个串联重复和3个节段重复(图8),导致串联重复观测/预期比为54.5,段段重复观测/预期比为0.16。
在MLP中,由预测的片段重复产生的序列之间的进化距离比蛋白酶体家族中的片段重复序列之间的进化距离大。在MLP家族中,片段重复序列之间的成对距离约为15到60个PAM单位[75],但在蛋白酶体20S家族中,PAM的含量从0到约4。然而,MLP重复似乎与蛋白酶体重复来自相同的多倍体事件(Blanc et al. [6,10])。显然,在多倍体之后,MLP成员比蛋白酶体20S亚基或CAB家族的大多数成员进化得更快。如上所述,我们使用Medicago和番茄序列标记分化时间和结果支持复制由于最近的多倍体事件在拟南芥.
基因复制模式
我们观察到在基因家族中预测的串联和节段复制水平之间存在中度负相关。如果序列变异或基因拷贝数必须维持在一定范围内,那么防止串联复制的一个可能的选择来源是不平等的交叉和基因丢失将产生变异和基因拷贝的高翻转[15,76].相反,由于亚功能化,片段重复可能更常被保留,而不会增加基因重排的可能性[77,78].在片段复制和串联复制水平较高的家系中,片段复制块中保留了家族成员的基因,而部分支中的基因拷贝数因串联复制而增加。在含黄素的单加氧酶家族中发现了一个例子,该家族在树的不同节点上有6个片段重复,一个分支的成员有7个串联重复。另一个例子包括叶绿素a/b结合蛋白家族。在频谱的另一端,我们发现在我们的研究集中,很少有家庭同时具有低段重复和低串联重复。在节段和串联类别中,没有家族低于中位数的一个标准差,但有几个家族接近:线粒体载体蛋白和热休克转录因子家族分别具有9%和13%的预期节段重复。可以想象,蛋白质复制化学计量学在一些代表多亚基蛋白质复合体的家族中是至关重要的[77,78].
结论
串联复制和分段复制对产生和维持的相对贡献为50大答:芥用观察到的/预期的串联复制和观察到的/预期的片段复制的比值来表征基因家族。串联重复数与节段重复数呈负相关;没有家系表现出高水平的串联复制和节段复制。虽然基因家族大小在整个基因组中的分布可以用随机模型来解释,但通过比较大型基因家族中串联复制和片段复制的相对水平,我们可以推测基因功能可能会对拷贝数和基因组组织产生反馈,从而导致所观察到的串联复制和片段复制的模式差异很大。
方法
基因家族的选择、排列和系统发育的构建
最初的候选基因家族是在2001年确定的答:芥PIR超家族[79],以2001年MIPs为基础答:芥预测蛋白质[80].虽然有帮助,但发现有些不一致,不必要地分裂了一些家庭,产生了一些成员重叠的家庭。最初认为的家族至少包含20个基因,其中Pfam [18所有基因家族成员至少有一个共同的Pfam结构域(e值< 0.01),并且结构域的组织在整个家族中是一致的。利用预测的TIGR 2001进行基因家族成员的初步选择答:芥蛋白质。所有的比对、系统发育和分析都基于更新的基因预测(2003年TIGR答:芥4.0版本)。
使用T-Coffee对所有基因家族的预测蛋白进行比对[19]和每个家族中最多30个随机选择的蛋白质序列。这些初始的排列被用于创建hmm,然后用于重新排列完整的蛋白质组。HMM参数[29]是:“hmmbuild -archpri .7 -fast - gapmax .3”。使用hmmcaliate对hmm进行校准,然后用于搜索全预测集答:芥蛋白质序列,使用hmmer中的hmmsearch程序[29].序列得分至少10分-10一般保留为基因家族成员和得分低于这一阈值的基因被排除,尽管得分是在假定基因家族的所有得分的情况下评估的。0.1 - 10分逐渐下降的家庭-15年通常被排除在研究之外,因为很难明确地分配家庭成员。
排列准备用于系统发育重建如下。为了删除高度可变或indel区域,删除掉HMM匹配状态之外的序列位置。其余位置中匹配不到75%的基因被完全去除。对线也进行了人工检查,并删除了其他对准特别差的区域。所有基因家族的全长和修剪序列均可在[24].
计算每个基因家族的简约性和自举邻居连接树。使用Phylip套件中的protpars计算简约树[81].输入序列顺序被打乱五次,并根据每个数据顺序计算拓扑。随机选择一棵最节俭的树作为计算树枝长度的基础。使用TreePuzzle在简约拓扑上计算最大似然分支长度[82].替代模式为Adachi和Hasegawa [83],根据输入树计算氨基酸频率,允许四个Gamma速率类别的速率异质性。使用Clustalw计算相邻连接树,不进行木村距离校正,并进行1000次自举重复。所有树木可在[24].
的预测答:芥复制块和基因家族的节段和串联复制
利用DiagHunter预测内部基因组复制[20.,21].所有复制块预测(基因、基因组坐标、基因组相似性和预测重复的点图图像)均可在[24].使用OrthoParaMap套件对基因家族的片段或串联重复进行预测[22,23].
识别节段重复的方法包括识别系统发育中足够相似的基因对,这些基因对在同步性块中足够接近各自的相应区域。同构块内的基因家族成员对在系统发育中用扩展的New Hampshire (NHX)格式进行注释[84].除了13个最大的基因家族外,“足够相似”的阈值都设置为10-25年,而“足够接近”的阈值则设为50kb。因为潜在的假阳性命中同构块的数量大约与基因家族中基因数量的平方成正比,所以更严格的阈值(“相似”= 10-30年和"close" = 30 kb)用于下列家系(见表1全名):CytP450, MATE, MFS, Myb, NBS-LRR, WRKY, GSDLLipase, GTPBP, MajIntrins, Prot, oxided, Polygalns, SCDehydRed, UDPGlycTnsf。
使用OrthoParaMap套件中的ParaMap程序推断产生串联基因重复的节点[22,23].这将递归地遍历树,识别产生基因的内部节点或染色体上物理上彼此接近(<50 kb)的其他节点。
基因家族大小分布的计算
所有预测蛋白均用于BLASTP [30.]使用三种不同的e值阈值(10-10, 10-20年,和10-30年).BLAST结果使用BioPerl [85基于]的脚本。使用Perl实现的单一链接聚类方法构建了近似基因家族。2003年TIGR答:芥4.0装配和蛋白预测用于这些程序。
按距离计算基因复制密度
2003年TIGR预测的蛋白质序列答:芥根据预测的5'和3'位置中间的核苷酸位置,分配4个装配的基因组位置。每条染色体的蛋白质序列用于BLASTP [30.]针对该染色体中的所有其他序列进行搜索,以给出BLAST命中列表并查询/目标每个染色体的中点位置。对self的命中被排除。
计算预期的串联和分段重复
在假设100,000 kb基因组(大约为基因组的大小)的情况下,模拟了预期在给定大小的基因家族中偶然发生的串联复制答:芥常染色质的基因组)。基因家族的大小从20到230个基因进行了模拟,使用以10为增量的大小类。对每个基因家族大小类进行1000次模拟,计算出近似分布。
给定大小的基因家族中节段重复的归一化常数是在这样的假设下计算的,即平均基因家族中保留的节段重复的最大比例应该与基因组中“重复”存在的百分比相同(在同向块中)。预期片段重复比例的算术(在多倍体后没有局部基因丢失或重复的情况下)显示在结果部分。
额外的数据
校准,基因系统发育,注释,基因组位置的分析,与内部基因组重复的关系,以及与来自不同物种的同源est的比较http://www.tc.umn.edu/~cann0010/genefamilyevolution/
参考文献
- 1.
开花植物拟南芥基因组序列分析。自然科学,2000,30(4):344 - 344。10.1038 / 35048692。
- 2.
shu SH, Bleecker AB:拟南芥受体激酶形成了一个与动物受体激酶相关的单系基因家族。自然科学进展,2001,29(3):344 - 344。10.1073 / pnas.181141598。
- 3.
李文杰,李志刚,李志刚,李志刚。植物受体激酶信号通路研究进展。植物科学
- 4.
Feldman KA:细胞色素P450s作为作物改良的基因。植物学报,2001,4:482 - 482。
- 5.
Nelson DR:拟南芥P450统计。[http://drnelson.utmem.edu/Arabfam.html].
- 6.
Blanc G, Barakat A, Guyot R, Cooke R, Delseny M:拟南芥基因组的大量复制和重组。中国生物医学工程学报,2000,29(4):344 - 344。10.1105 / tpc.12.7.1093。
- 7.
Simillion C, Vandepoele K, Van Montagu MC, Zabeau M, Vande Peer Y:拟南芥隐藏复制历史。中国科学:自然科学,2002,29(2):366 - 366。10.1073 / pnas.212522399。
- 8.
Vandepoele K, Saeys Y, Simillion C, Raes J, Van De Peer Y:同源区域自动检测及其在拟南芥与水稻微共线性中的应用。中国生物工程学报,2002,29(4):457 - 457。10.1101 / gr.400202。
- 9.
杨晓明,李志强,李志强,等。拟南芥基因组复制的研究进展。科学通报,2000,29(3):344 - 344。10.1126 / science.290.5499.2114。
- 10.
Blanc G, Hokamp K, Wolfe KH:拟南芥基因组中最近的多倍体叠加在较老的大规模复制上。基因组学报,2003,13:137-144。10.1101 / gr.751803。
- 11.
Ermolaeva MD, Wu MM, Eisen JA, Salzberg SL:拟南芥基因组复制的年龄。中国生物医学工程学报,2003,29(3):349 - 349。10.1023 /: 1023001130337。
- 12.
杨晓明,王晓明,杨晓明,王晓明,等:基于染色体复制事件的被子植物基因组进化研究。自然科学,2003,29(4):433-438。10.1038 / nature01521。
- 13.
张玲,张志刚,张志刚,张志刚。拟南芥基因对核苷酸替代的研究进展。中国生物医学工程学报,2002,19:1464-1473。
- 14.
朱晓明,王晓明,王晓明,等:拟南芥基因组中染色体片段结构差异的研究进展。中国生物医学工程学报,2003,29(4):349 - 349。10.1093 / nar / gkg201。
- 15.
张志刚,张志刚,张志刚,张志刚。酿酒酵母染色体内重复序列的分析。中国生物医学工程学报,2000,17:1268-1275。
- 16.
Hughes AL, Friedman R, Ekollu V, Rose JR:拟南芥转座因子与重复基因组块的非随机关联。分子系统进化学报,2003,29:410-416。10.1016 / s1055 - 7903(03) 00262 - 8。
- 17.
MIPS:拟南芥PIR超家族。[http://mips.gsf.de/proj/thal/db/tables/tables_func_frame.html].
- 18.
Bateman A, Birney E, Cerruti L, Durbin R, Etwiller L, Eddy SR, Griffiths-Jones S, Howe KL, Marshall M, Sonnhammer EL: Pfam蛋白家族数据库。中国生物医学工程学报,2002,29(4):357 - 357。10.1093 / nar / 30.1.276。
- 19.
Notredame C, Holm L, Higgins DG: T-COFFEE:多序列比对的目标函数。生物信息学,1998,14:407-422。10.1093 /生物信息学/ 14.5.407。
- 20.
大炮SB: DiagHunter网站。2003年,(http://www.tc.umn.edu/~cann0010/Software.html].
- 21.
Cannon SB, Kozik A, Chan B, Michelmore R, Young ND: DiagHunter和GenoPix2D:用于基因组比较、大规模同源性发现和可视化的程序。中国生物医学工程学报,2003,4:R68-10.1186/gb-2003-4-10-r68。
- 22.
大炮SB: OrthoParaMap网站。2003年,(http://www.tc.umn.edu/~cann0010/Software.html].
- 23.
Cannon SB, Young ND: OrthoParaMap:通过整合比较基因组数据和基因系统发育来区分正同源和副同源。中国生物医学工程学报,2003,34(4):357 - 357。
- 24.
大炮SB:基因家族进化网站。2003年,(http://www.tc.umn.edu/~cann0010/genefamilyevolution/].
- 25.
高加腾,Olendzenski L:同源同源,平行同源与基因组比较。中国生物医学工程学报,1999,29(3):344 - 344。10.1016 / s0959 - 437 x(99) 00029 - 5。
- 26.
Henikoff S, Greene EA, Pietrokovski S, Bork P, Attwood TK, Hood L:基因家族:蛋白质平行嵌合体的分类。科学通报,1997,27(4):357 - 357。10.1126 / science.278.5338.609。
- 27.
多基因家族和复杂性的进化。中华药理学杂志,1997,33:34-41。
- 28.
Barker WC, Garavelli JS, Huang H, McGarvey PB, Orcutt BC, Srinivasarao GY, Xiao C, Yeh LS, Ledley RS, Janda JF, Pfeiffer F, Mewes HW, Tsugita A, Wu C:蛋白质信息资源(PIR)。中国生物医学工程学报,2000,28:41-44。10.1093 / nar / 28.1.41。
- 29.
Eddy SR: hmm:生物序列分析的剖面隐马尔可夫模型:hmm用户指南。2001,[http://hmmer.wustl.edu/].
- 30.
Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ: gapping BLAST和ppi -BLAST:新一代蛋白质数据库搜索程序。中国生物医学工程学报,1997,25:389-3402。10.1093 / nar / 25.17.3389。
- 31.
Cannon SB, Zhu H, Baumgarten AM, Spangler R, May G, Cook DR, Young ND: TIR和非TIR NBS-LRR抗性基因亚家族的多样性、分布和古代分类学关系中华生物医学杂志,2002,54:548-562。10.1007 / s00239 - 001 - 0057 - 2。
- 32.
Meyers BC, Dickerman AW, Michelmore RW, Sivaramakrishnan S, Sobral BW, Young ND:植物抗病基因编码核苷酸结合超家族中一个古老而多样的蛋白质家族成员。植物学报,1999,20:317-332。10.1046 / j.1365 - 313 x.1999.00606.x。
- 33.
Batalia MA, Monzingo AF, Ernst S, Roberts W, Robertus JD:抗真菌蛋白玉米素的晶体结构,它是索马汀样蛋白,PR-5蛋白家族的成员。生物工程学报,1996,3:19-23。
- 34.
Carter C, Graham RA, Thornburg RW:拟南芥含有一个庞大的发芽样蛋白家族:编码12个独特家族成员的cDNA和基因组序列的特征。植物化学学报,1998,38:929-943。10.1023 /: 1006038117130。
- 35.
Membre N, Bernier F, Staiger D, Berna A:拟南芥发芽样蛋白:共同特征和特异特征指向多种功能。植物学报,2000,29(3):344 - 344。10.1007 / s004250000277。
- 36.
Santamaria M, Thomson CJ, Read ND, Loake GJ:拟南芥中一个基本pr1样基因AtPRB1的启动子建立了一种器官特异性表达模式,并对乙烯和茉莉酸甲酯反应。植物化学学报,2001,47:641-652。10.1023 /: 1012410009930。
- 37.
Osmark P, Boyle B, Brisson N:细胞内致病相关蛋白与一组乳胶蛋白的序列和结构同源性。植物化学学报,1998,38(3):344 - 344。10.1023 /: 1006060224012。
- 38.
Kuan J, Saier m.h ., Jr.:转运蛋白的线粒体载体家族:结构、功能和进化关系。生物化学学报,1998,28:439 - 439。
- 39.
杨晓明,王晓明,王晓明,等:植物和哺乳动物线粒体解偶联蛋白的研究进展。科学通报2001,21:201-212。10.1023 /: 1013604526175。
- 40.
Parmentier Y, Bouchez D, Fleck J, Genschik P:拟南芥20S蛋白酶体基因家族。农学通报,1997,29(4):344 - 344。10.1016 / s0014 - 5793(97) 01228 - 3。
- 41.
Vierstra RD:泛素/26S蛋白酶体途径,是许多植物蛋白质生命的复杂的最后一章。植物科学进展,2003,8:135-142。10.1016 / s1360 - 1385(03) 00014 - 1。
- 42.
傅华,Doelling JH, Arendt CS, Hochstrasser M, Vierstra RD:拟南芥20S蛋白酶体基因家族的分子结构。中国生物工程学报,1998,29(3):357 - 357。
- 43.
杨晓明,王晓明,王晓明,王晓明,等:拟南芥抗性基因的基因组水平进化。遗传学报,2003,16(3):349 - 349。
- 44.
Huynen MA, van Nimwegen E:全基因组中基因家族大小的频率分布。中国生物医学工程学报,1998,15:583-589。
- 45.
Sornette D, Cont R:从零开始排斥的收敛乘法过程:幂律和截断幂律。中华体质杂志,1997,7:431-444。10.1051 / jp1:1997169。
- 46.
随机差分方程和随机矩阵乘积的更新理论。数学学报,2003,31(4):357 - 357。
- 47.
Kim BH, Schoffl F:拟南芥热休克转录因子1与70 kDa热休克蛋白的相互作用。应用物理学报,2002,53:371-375。10.1093 / jexbot / 53.367.371。
- 48.
Aquila H, Link TA, Klingenberg M:参与线粒体能量转移的溶质载体形成同源蛋白家族。中国农业科学,2004,27(1):1-9。10.1016 / 0014 - 5793(87) 81546 - 6。
- 49.
傅华,Doelling JH, Rubin DM, Vierstra RD:拟南芥26S蛋白酶体中6个调节粒子3a atp酶亚基的结构和功能分析。植物学报,1999,18:529-539。10.1046 / j.1365 - 313 x.1999.00479.x。
- 50.
Hochstrasser M, Johnson PR, Arendt CS, Amerik AYu, Swaminathan S, Swanson R, Li SJ, Laney J, pas - rylaarsdam R, Nowak J, Connerly PL:酿酒酵母泛素-蛋白酶体系统。中国生物医学工程学报,1999,29(3):326 - 326。10.1098 / rstb.1999.0495。
- 51.
von Arnim AG:蛋白酶体的搭便车指南。Sci抽烟可以。2001,2001: pe2。
- 52.
Gray WM, Estelle I:泛素-蛋白酶体通路在生长素反应中的作用。生物化学进展,2000,25:133-138。10.1016 / s0968 - 0004(00) 01544 - 9。
- 53.
Moore RC, Purugganan MD:复制基因进化的早期阶段。自然科学进展,2003,30(3):366 - 366。10.1073 / pnas.2535513100。
- 54.
Michelmore R, Meyers BC:植物中的抗性基因簇通过发散选择和生死过程进化而来。基因组学报,1998,8:1113-1130。
- 55.
王晓明,王晓明,王晓明,王晓明,王晓明。拟南芥nbs - lrr基因序列分析。植物学报,2003,29(3):344 - 344。10.1105 / tpc.009308。
- 56.
Jones JD:将植物抗病基因的知识运用到工作中。植物学报,2001,4:281-287。10.1016 / s1369 - 5266(00) 00174 - 6。
- 57.
勃朗G,沃尔夫K:拟南芥的拟南芥。2002年,(http://wolfe.gen.tcd.ie/athal/].
- 58.
刘松,杨军,张刚,熊勇,李震,毛林,周超,朱震,陈锐,郝波,郑伟,陈松,郭伟,陶明,朱林,袁林,杨华:水稻基因组序列草案(Oryza sativa L. ssp.)。籼稻)。科学通报,2002,29(3):344 - 344。10.1126 / science.1068037。
- 59.
Akita M, Valkonen JP:苔藓(Physcomitrella patens)中一个新的基因家族与植物抗病基因TIR-NBS类具有序列同源性和系统发育关系。中华生物医学杂志,2002,55:595-605。10.1007 / s00239 - 002 - 2355 - 8。
- 60.
陈志伟,陈志伟,陈志伟,等。萌发蛋白与萌发蛋白样蛋白的研究进展。中华实验生物学杂志,2001,39:191-200。
- 61.
施韦泽P,杜德勒R:萌发样基因家族成员在小麦表皮细胞中的瞬时表达与抗病性。植物学报,1999,20:541- 544。10.1046 / j.1365 - 313 x.1999.00624.x。
- 62.
回复:光合作用的起源和早期进化。光子学报,1997,33(3):344 - 344。
- 63.
欧丽特·阿杰,巴里·巴:串联质谱法鉴定菠菜光系统II光收集组分。光子学报,2002,29(3):344 - 344。10.1023 /: 1016132700844。
- 64.
安德森J:解剖光系统II光收集天线。(论文)。2003
- 65.
Bailey S, Walters RG, Jansson S, Horton P:拟南芥对光环境的驯化:存在独立的弱光和强光响应。植物学报,2001,29(3):344 - 344。10.1007 / s004250100556。
- 66.
Yakushevsha AE, Keegstra W, Boekema EJ, Dekker JP, Andersson J, Jansson S, Ruban AV, Horton P:拟南芥光系统II结构:CP26和CP29天线复合物的定位。生物化学,2003,42:
- 67.
Ruban AV, Wentworth M, Yakushevsha AE, Andersson J, Lee MM, Keegstra W, Dekker JP, Boekema EJ, Jansson S, Horton P:缺乏主要光收集复合物的植物保留光系统II宏观组织。《自然》2003年出版:
- 68.
Andersson J, Wentworth M, Walters RG, Howard CA, Ruban AV, Horton P, Jansson S:光合系统II缺乏主要的光收集复合体影响光合功能。植物J. 2003
- 69.
李志刚,李志刚,李志刚:罂粟乳胶蛋白基因家族的结构研究。植物化学学报,1997,20:749-752。
- 70.
奈斯勒CL:两个主要乳胶蛋白基因家族新成员的序列分析支持罂粟的三倍体杂交起源。中国生物工程学报,1994,19(3):357 - 357。10.1016 / 0378 - 1119(94) 90756 - 0。
- 71.
Stromvik MV, Sundararaman VP, Vodkin LO:一种来自大豆的新型启动子,在具有或不具有近端650个碱基对的复杂发育模式中具有活性。植物分子生物学学报,2004,29(4):357 - 357。10.1023 /: 1006312228617。
- 72.
Bufe A, Spangfort MD, Kahlert H, Schlaak M, Becker WM:桦树花粉主要过敏原Bet v1具有核酸酶活性。植物学报,1996,29(4):344 - 344。
- 73.
Flores T, alpe - giron A, Flores- diaz M, Flores HE: Ocatin。一种来自安第斯块茎作物oca的新型块茎贮藏蛋白,具有抗菌和抗真菌活性。中国生物医学工程学报,2002,29(4):344 - 344。10.1104 / pp.010541。
- 74.
Moiseyev GP, Fedoreyeva LI, Zhuravlev YN, Yasnetskaya E, Jekel PA, Beintema JJ:人参愈伤组织中两种核糖核酸酶的初级结构。细胞内致病相关植物蛋白PR-10家族的新成员。农学通报,1997,37(3):357 - 357。10.1016 / s0014 - 5793(97) 00337 - 2。
- 75.
Dayhoff MO:蛋白质序列和结构图谱。第5卷,补编3,第353-358页。美国华盛顿特区,国家生物医学研究基金会;1979.
- 76.
以杜特地动物线粒体基因组为例的基因重排的复制/随机损失模型。比较基因组学编辑:Sankoff D和Nadeau J. Dordrecht, NL, Kluwer文献出版社;2000:133 - 147。
- 77.
Lynch M, Conery JS:重复基因的进化命运和后果。科学通报,2000,29(4):531 - 531。10.1126 / science.290.5494.1151。
- 78.
林奇M, Conery JS:重复基因的进化人口统计学。中华基因工程学报,2003,3:35-44。10.1023 /: 1022696612931。
- 79.
Barker WC, Garavelli JS, Hou Z, Huang H, Ledley RS, McGarvey PB, Mewes HW, Orcutt BC, Pfeiffer F, Tsugita A, Vinayaka CR, Xiao C, Yeh LS, Wu C:蛋白质信息资源:蛋白质数据专家注释的社区资源。中国生物医学工程学报,2001,29:29-32。10.1093 / nar / 29.1.29。
- 80.
Schoof H, Zaccaria P, Gundlach H, Lemcke K, Rudd S, Kolesov G, Arnold R, Mewes HW, Mayer KF: MIPS拟南芥数据库(Arabidopsis thaliana Database, MAtDB):基于第一个完整植物基因组的综合性生物学知识资源。中国生物医学工程学报,2002,30:91-93。10.1093 / nar / 30.1.91。
- 81.
Felsenstein J: PHYLIP(系统发育推断包)版本3.6。由作者分发。华盛顿大学遗传学系,西雅图,2000
- 82.
薛晓明,王晓明,王晓明。基于并行计算的最大似然系统发育分析方法。生物信息学,2002,18:502-504。10.1093 /生物信息学/ 18.3.502。
- 83.
田立,长谷川。线粒体DNA编码蛋白质中氨基酸替代的模型。中华药理学杂志,1996,42:459-468。
- 84.
Zmasek CM, Eddy SR: ATV:注释系统发育树的显示和操作。生物信息学,2001,17:383-384。10.1093 /生物信息学/ 17.4.383。
- 85.
Stajich JE, Block D, Boulez K, Brenner SE, Chervitz SA, Dagdigian C, Fuellen G, Gilbert JG, Korf I, Lapp H, Lehvaslaiho H, Matsalla C, Mungall CJ, Osborne BI, Pocock MR, Schattner P, Senger M, Stein LD, Stupka E, Wilkinson MD, Birney E: Bioperl工具包:生命科学的Perl模块。基因组学报,2002,12:1611-1618。10.1101 / gr.361602。
确认
感谢Bridgette Barry对CAB家族的讨论,感谢Martina Stromvik对MLP家族的讨论,感谢Jeff Doyle对手稿的建议,感谢明尼苏达超级计算研究所(MSI)对计算资源的访问。这项工作部分得到了美国国家科学基金会(NSF)授予GM的DBI-9975866奖和NDY的DBI-0110206奖的支持,以及美国农业部国家需求奖学金和明尼苏达大学植物分子遗传学研究所授予SBC的奖学金。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
SBC开发了用于分析的软件,对所有基因家族进行了分析,并起草了手稿。AM对[24].SBC和AB开发了用于定量序列重复的方法拟南芥.NDY和GM在整个项目中提供建议,并帮助手稿准备和修改。所有作者都阅读并批准了最终的手稿。
权利和权限
关于本文
引用本文
加农,s.b.,米特拉,A,鲍姆加滕,A。et al。节段和串联基因复制在大基因家族进化中的作用拟南芥.BMC植物生物学4,10(2004)。https://doi.org/10.1186/1471-2229-4-10
收到了:
接受:
发表:
关键字
- 基因家族
- 串联重复
- 节段重复
- 大基因家族
- 同线性块