摘要
背景
穆萨该物种是发展中国家第四重要的作物。在此,我们报告了6252个BAC端序列的分析,以了解该基因的序列组成穆萨acuminata基因组在成本效益和高效的方式。
结果
BAC端测序得到6252个reads,共计4420944 bp,其中清洗过滤707 bp后平均读取长度为2979个克隆对。所有序列已提交到GenBank,登记号为DX451975 - DX458350。在多个数据库中检索了BAC端序列,发现其与线粒体和叶绿体(2.6%)、转座子和重复序列(36%)以及蛋白质(11%)具有显著的同源性。通过从匹配到的基因本体分配来进行蛋白质匹配的功能解释拟南芥并被证明涵盖了广泛的类别。的蛋白质匹配区域穆萨BAC端序列,测定编码区GC含量为47%。当蛋白质匹配包含起始密码子时,GC含量作为129 bp滑动窗口位置(5'到3')的函数产生“大米样”梯度。共发现352个潜在SSR标记。在4个大小类别中,最丰富的简单序列重复序列是at富集的。过滤线粒体和叶绿体匹配后,数千个BAC端序列与BLASTN显著匹配栽培稻和拟南芥基因组序列。其中,少量BAC端序列对被显示映射到dna的邻近区域栽培稻基因组代表潜在的微合成区域。
结论
数据库搜索BAC端序列和从头开始分析鉴定出这些reads可能包含转座子、重复序列、蛋白质和简单序列重复。大约600个BAC末端序列包含现有可用的未发现的蛋白质序列穆萨表达序列标签,重复或转座子数据库。此外,还可以根据与顶部蛋白命中匹配的区域估计基因统计量、GC含量和谱。少量的BAC端对序列可以映射到dna的邻近区域栽培稻代表潜在微合的区域。这些结果表明,大规模的BAC端测序策略有可能锚定一小部分的基因组穆萨acuminata到基因组栽培稻并有可能拟南芥。
背景
在能够实现极低成本基因组DNA测序的新技术开发出来之前,资助机构在选择新的植物基因组进行测序时非常挑剔。目前的技术只能以1000万到5000万美元或更多的价格产生所需数据质量的哺乳动物大小的基因组序列。许多基因组计划的最初目标通常是以低成本和有效的方式获得感兴趣的基因组的一瞥。在植物中,利用已完成的基因组通常有一些优势拟南芥和栽培稻通过比较基因组学。答:芥由于其基因组大小较小(125 Mb),因此被选为双子叶植物的模型[1]和米饭[2) (o .漂白亚麻纤维卷)是第一个被测序的谷类和单子叶植物[3.]。
穆萨香蕉和大蕉是撒哈拉以南非洲、南美洲和中美洲以及亚洲大部分地区非常重要的作物。的穆萨物种穆萨acuminata(AA基因组)和穆萨balbisiana(BB基因组),它们都有2n = 22条染色体,代表了栽培香蕉品种的两个主要祖先。的单倍体基因组穆萨物种的大小估计在560至800米之间[4- - - - - -6],是模型工厂的四倍多答:芥(125mb) [7,比……大30%以上o .漂白亚麻纤维卷(390mb) [2]。
单子叶植物的比较基因组学研究主要集中在豆科近缘单子叶植物种间的共系程度[8]。两者之间存在着广泛的微观和宏观的协同性o .漂白亚麻纤维卷、大麦、玉米和小麦[9,10染色体的保存程度在不同的染色体位置之间往往不同。远亲植物间的同源性在生物信息学上更难阐明,而且可能发生的频率较低。
为了了解序列的内容和序列的复杂性穆萨基因组,有必要对大量随机选择的克隆进行测序,这些克隆代表了整个基因组。另一种方法是从细菌人工染色体库中随机选择大量的细菌人工染色体(BACs)进行末端测序[11]。后一种方法不能提供真正的基因组随机抽样,因为用于文库构建的特定酶的限制性内切位点代表性不足的区域也将代表性不足。尽管如此,BAC端测序确实提供了基因组的准随机采样,并且具有BAC克隆似乎包含感兴趣的目标的优势,为其他分析(如荧光分析)提供了极好的材料原位对中期或粗线染色体进行杂交(FISH)或进行深度测序以发现基因。大量收集BAC端序列(BES)也是基因组测序计划的重要组成部分。在这里,我们研究了是否穆萨BES可以引导我们深入了解穆萨基因组组成使用生物信息学比较蛋白质,重复,表达序列标签(ESTs)和其他数据库。从BES,我们调查穆萨基因密度、GC含量、蛋白质和SSR含量以及推测的比较条带bac,这些条带bac代表了dna和dna之间潜在的微共胞区o .漂白亚麻纤维卷和穆萨物种。
结果与讨论
序列搜索,简单序列重复,GC分析和蛋白质发现将首先讨论,然后是基因组定位分析O.sativa和答:芥以确定比较瓷砖BACs从穆萨可能共线的库(即显示微同步)。
后端测序
野生二倍体“加尔各答4号”克隆叶片HindIII BAC文库BAC末端序列分析[j]12],生成了6252条高质量的reads,平均长度为707个核苷酸,总长度约为4.4 Mb,其中包括2,979对末端reads(表1)1)。所有序列已提交到GenBank,登记号为DX451975 - DX458350。
数据库序列搜索
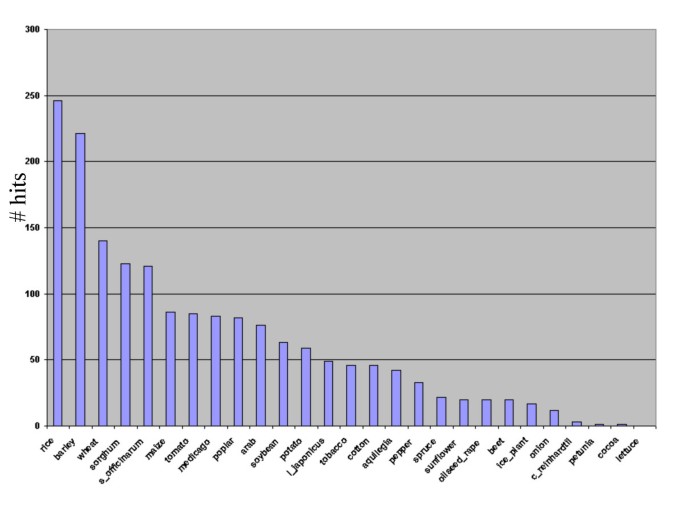
BES与TIGR非相同氨基酸数据库的比较显示,11%的序列由于匹配良好而包含“基因”区域,不包括转座子/重复(36%)。使用严格的阈值1e-5, 80%的同一性和80%的覆盖率导致2.6%的BES与叶绿体/线粒体匹配(表5)2)。在蛋白质匹配中,超过50%的情况下,BLAST匹配的最佳是O.sativa30%的人答:芥蛋白质,符合更紧密的关系之间穆萨和o .漂白亚麻纤维卷当与穆萨和答:芥。这也与TIGR植物基因指数的匹配一致,其中最高水平的同源性显示为o .漂白亚麻纤维卷其次是大麦、小麦和其他单株植物(图1)1)。在BES分析的样本中,发现36%含有与转座因子或重复序列同源的序列。大多数转座因子属于Ty1复制型(742),其次是Ty3吉普赛型(211)类型的反转录转座子(表2)2),这与先前的数据一致,即I类反转录转座子对大多数核苷酸起作用[13以及对木瓜BAC端序列的研究。
我们还发现了111个与微型倒置重复转座元件(MITEs)匹配的片段,其中最丰富的是adh-11样(46),其次是adh型d样(22)和adh型g样(12)。根据E = 1e-15处与蛋白质匹配的BES数(686)计算的基因密度预测估计每6.4 kb存在一个基因(表1)3.),这与之前的基因密度研究一致穆萨BAC研究[14]。相比之下,来自同一研究的第二个BAC给出了每10 kb中一个基因的基因密度,但是经过仔细检查,BAC的一半由转座子相关基因组成,而另一半则是非转座子相关基因。这些数据之间的差异表明,基因组织类似于禾科,基因聚集在富含基因的区域,由含有大量转座子的基因贫乏DNA隔开。与其他植物基因组相比,基因密度似乎与自动注释的报告相似o .漂白亚麻纤维卷每个基因6.2 KB [15不同于答:芥每个基因有4.5 KB [6]。
功能注释
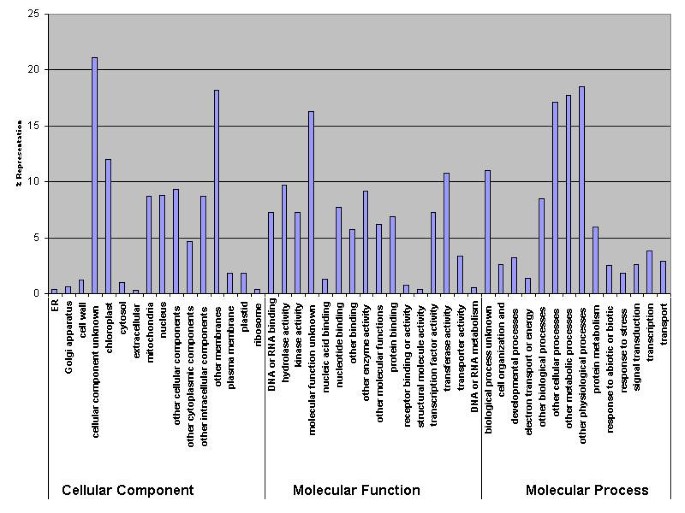
基因本体(GO)是一个功能术语的受控词汇表,允许对基因产物进行一致的注释[16]。为了将假定的功能角色分配给穆萨acuminata序列中,我们使用了答:芥蛋白质组(16]。在686个与重复序列或转座子数据库不匹配但与TIGR综合蛋白数据库匹配的BES中,664个与TIGR综合蛋白数据库匹配答:芥蛋白质,并根据最高匹配度分配GO任务。结果显示,这些基因涵盖了广泛的氧化石墨烯类别(图2)3.)。
GC概要
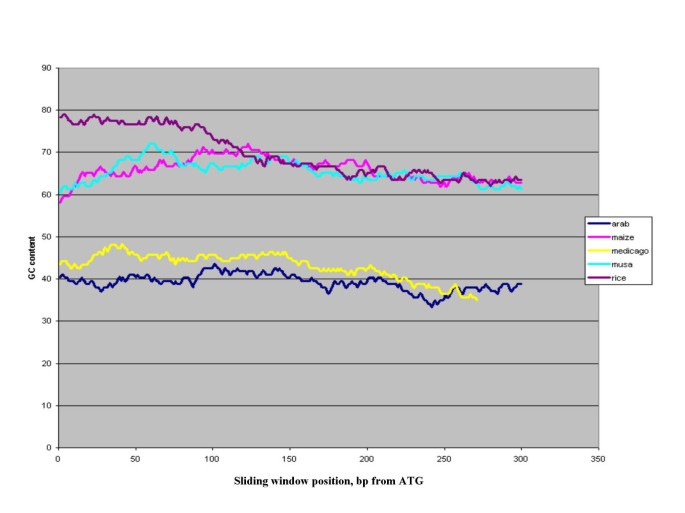
对BES与顶部蛋白命中的匹配区域进行GC分析。排除任何不包含与起始密码子匹配的BES。与此同时,也进行了类似的研究答:芥,o .漂白亚麻纤维卷、玉米和Medicago truncatula东德(BES)(图2)。答:芥和m . truncatula在整个编码序列中显示相似的GC内容。在大多数情况下穆萨,o .漂白亚麻纤维卷玉米的GC值在预测起始点前150 bp的5′端较高,在3′端逐渐降低。这一结果与前人的研究结果一致,前人的研究表明,禾本科植物具有较高的平均GC含量和不对称分布,而禾本科植物具有较低的GC含量和更对称的分布。17,18]
GC含量
生物的GC含量在基因组区、内含子区和外显子区各不相同,可低至22% (恶性疟原虫)至70%以上(玉米)。GC含量测定在BES与顶部蛋白命中的匹配区域。所有BES的平均GC含量为39%,编码序列GC含量为47%,与先前的研究一致,根据2个bac,显示总体GC含量为38%,外显子内GC含量为49% [14]。这一节和前一节已经表明,具有蛋白质匹配的BES可以在一定程度上精确地计算GC含量和GC分析。使用更大的数据集进行进一步确认,使用ESTs,- 2280穆萨est序列(19]从GenBank下载,聚类和组装得到1123个独特的序列,其中179个是contigs。这些独特的序列产生了1056个潜在的开放阅读帧,平均GC含量为51%。这些结果与以往对单子叶和双子叶中GC含量的研究一致[17]。
简单序列重复
简单序列重复序列(或微卫星)是一类分子标记,通常是多态的,广泛用于生成遗传图谱[20.]。在BAC端序列中共发现352个潜在SSR标记(表2)4)。在所有四个大小类别中,最丰富的ssr都富含at。这与之前报道的其他物种的微卫星丰度一致:聚(AT)/(TA)和富含AT的三核苷酸重复序列是同类重复序列中最丰富的答:芥在酵母中[21]。类似于蔷薇科est序列(22],二核苷酸重复序列是四类微卫星中最丰富的。本研究中发现的SSRs均未被报道过,也未发现与先前发现的Musa SSRs相匹配[23,24]。
穆萨后端平铺上o .漂白亚麻纤维卷和答:芥基因组
对于相对未表征的物种,可能与测序良好的模型物种的某些染色体区域存在同源性,高通量BAC端测序提供了将未表征物种的基因组“拼接”到已测序物种的基因组上的潜力。BES映射到o .漂白亚麻纤维卷和答:芥为了进一步表征我们的BAC文库,并测试BAC端测序方法是否对穆萨以上述方式。当Musa BESs与o .漂白亚麻纤维卷基因组序列o .漂白亚麻纤维卷程序集版本4.0 [15]), 2,646人受到重大打击o .漂白亚麻纤维卷顶级匹配的百分比从58%到98%不等。这些命中包括593对,其中55对在过滤了与线粒体和叶绿体匹配的同源性后,显示出顶突击中同一染色体。8对BES具有相似匹配o .漂白亚麻纤维卷序列,跨度为100到500 Kb(表1)5)。当Musa BESs与拟南芥基因组[7],有2177个匹配,匹配率在54% - 98%之间。在2177个命中中,403个BES对具有显著的BLAST匹配(对的两个成员)答:芥经过线粒体同源性和叶绿体同源性的筛选,共36对基因组序列显示顶胚位于同一染色体上。尽管少数BES对被证明具有相似匹配答:芥在22 ~ 500 KB的序列中,没有一个序列在正确的方向上发现,这可能代表了局部倒置。
穆萨满足顶胚击中同一染色体且与线粒体和叶绿体无同源性标准的BACs被认为是候选的假定的比较层状BACs,可能代表高度保守的基因含量和组织区域。的预测大小穆萨BACs(以及末端序列之间的距离)与序列中配对匹配的间隔进行了比较o .漂白亚麻纤维卷和答:芥基因组分别。分离穆萨超过我们任意截断的500kb的BES匹配,可能是由于两个基因组在进化过程中的重排而导致的同质区扩展。
结论
在本研究中,我们考察了两个主要观点。首先,通过基因密度、GC含量、蛋白质和SSR的发现,Musa BES可以深入了解Musa基因组;其次,这些序列可以用来识别Musa和其他物种之间潜在的微共线区域。BAC端序列与蛋白质、表达序列标签、转座子、重复序列具有同源性,可用于简单的序列重复鉴定和基因统计及GC含量的估计。在这些BES中编码的蛋白质被证明涵盖了广泛的氧化石墨烯类别。尽管Musa和O. sativa之间只有有限的微同步,但结果表明,大规模的BAC端测序策略有可能将Musa基因组的至少一小部分锚定在O. sativa序列上。大规模的BAC端测序将显示参考基因组和目标基因组之间是否存在更多的微共synsyny区域,以及由于基因特征和基因组特征的独特性,是否支持全基因组测序。BAC末端数据将与现有EST或基因组序列一起成为一个有用的指标,供资助机构在选择新的植物基因组进行测序时使用,并通过比较基因组学评估利用拟南芥和sativa完成的基因组的潜力。我们期望对其他植物或动物物种进行类似的分析,通过数据库搜索和模型物种的合成,将以一种非常经济有效的方式提供对基因组的见解。
方法
后端测序
BES由a生成穆萨利用野生二倍体“加尔各答4号”无性系叶片构建细菌人工染色体文库(穆萨acuminata无性系种群。Burmannicoides 2n = 2 × = 22),平均插入大小为100 kb [12]。
采用标准碱法制备384孔格式的DNA模板。末端测序采用Applied Biosystems (ABI) Big Dye终止化学,并在ABI 3730 xl机器上分析。使用TraceTuner进行碱基调用,使用Lucy对矢量和低质量序列进行裁剪[25]。
后端数据库搜索
将序列与TIGR植物基因索引中的所有条目进行比较[26],并使用blastx连接到TIGR非相同氨基酸数据库,该数据库包含来自GenBank、RefSeq和Uniprot等多个数据库的非相同蛋白质数据(截止值1e-5)。BAC端序列也与TIGR Repeat数据库中的重复序列进行了比较[27]和使用blastx的内部转座子数据库,截断值为1e-5。并将BAC端序列与TIGR水稻基因组序列组合进行了比较答:芥TAIR基因组序列,使用胚,截断值为1e-10。鉴定比较瓷砖BACs从穆萨与参考基因组可能共线(即显示微共音)的文库,针对参考基因组进行搜索穆萨对基因组序列进行分析,发现顶端对BES的两端与一段长度具有最高的显著性匹配o .漂白亚麻纤维卷或答:芥序列和位置上的两个区域穆萨基因组间隔在100 ~ 500 kb之间。BAC端数据集为苜蓿,拟南芥、玉米和m . truncatula用于GC分析的序列最初是从GenBank下载的,然后从estinformatics.org [28]。
EST聚类和装配
穆萨EST reads最初从GenBank下载,然后从estinformatics.org [28]和群集的和组装的[26]。
简单序列重复序列的鉴定与分析
完美二核苷酸到六核苷酸简单序列重复使用MISA进行鉴定[20.] Perl脚本,指定至少6个二核苷酸和5个四核苷酸到六核苷酸重复,最多100个核苷酸中断的复合重复和单核苷酸重复的最小长度为20个碱基。
参考文献
Meinke DW, Cherry JM, Dean C, Rounsley SD, Koornneef M:拟南芥模式植物基因组分析。科学,1998,(6):679-682。
国际水稻基因组测序计划:水稻基因组图谱序列。自然科学学报,2005,36(4):793-800。10.1038 / nature03895。
赵伟,王健,何翔,黄翔,焦艳,戴敏,魏松,傅健,陈毅,任旭,张勇,倪鹏,张健,李生,王健,王国坤,赵辉,于健,杨辉,王健:BGI-RIS,水稻基因组学集成信息资源与比较分析平台。中国生物医学工程学报,2004,32(2):377- 382。10.1093 / nar / gkh085。
李建军,李建军,李建军,等。用流式细胞术分析龙葵核DNA含量。理论与应用,1999,(8):1344-1350。10.1007 / s001220051201。
Kamate K, Brown S, Durand P, Bureau JM, De Nay D, Trinh TH:植物28个分类群的核DNA含量和碱基组成。中国生物医学工程学报,2001,44(4):622-627。10.1139 / gen - 44 - 4 - 622。
Bartos J, Alkhimova O, Dolezelova M, De Langhe E, Dolezel: Musa和Ensete (Musaceae)核糖体DNA的核基因组大小和基因组分布:分类意义。中国生物医学工程学报,2005,31(2):557 - 557。10.1159 / 000082381。
拟南芥基因组计划:开花植物拟南芥基因组序列分析。自然科学学报,2000,34(8):796-815。10.1038 / 35048692。
Singh NK, Raghuvanshi S, Srivastava SK, Gaur A, Pal AK, Dalal V, Singh A, Ghazi IA, Bhargav A, Yadav M, Dixit A, Batra K, Gaikwad K, Sharma TR, Mohanty A, Bharti AK, Kapur A, Gupta V, Kumar D, Vij S, Vydianathan R, Khurana P, Sharma S, McCombie WR, Messing J, Wing R, Sasaki T, Khurana P, Mohapatra T, Khurana JP, Tyagi AK:水稻-小麦合成的11号染色体长臂序列分析。函数集成基因组学,2004,4:102-17。10.1007 / s10142 - 004 - 0109 - y。
顾勇,孔晓东,吴晓东,等。小麦科植物基因组同源区域的比较序列分析。植物生理学报,2004,35(5):459- 467。10.1104 / pp.103.038083。
Salse J, Piegu B, Cooke R, Delseny M:对水稻(Oryza sativa L.)和玉米(Zea mays L.)之间的合成的新计算机见解强调了重组并确定了水稻基因组中的新重复。植物学报,2004,38(3):396- 396。10.1111 / j.1365 - 313 x.2004.02058.x。
赖长荣,于强,侯森,Skelton RL, Jones MR, Lewis KL, Murray J, Eustice M, Guan P, Agbayani R, Moore PH, Ming R, pressting GG:木瓜BAC末端序列的分析揭示了果树基因组的组织结构。生物医学工程学报,2006,26(1):1-12。10.1007 / s00438 - 006 - 0122 - z。
Vilarinhos AD, Piffanelli P, Lagoda P, Thibivilliers S, Sabau X, Carreel F, D'Hont A:香蕉细菌人工染色体文库的构建和鉴定。理论与应用,2003,26(6):1102- 1106。
李建军,李建军,李建军,等。玉米基因间反转录转座子的研究进展。生物医学工程学报,1998,20:43-5。10.1038/1695。
Aert R, Sagi L, Volckaert G:香蕉(Musa acuminata) BAC克隆的基因含量和密度分析。理论与应用,2004,(9):129-139。10.1007 / s00122 - 004 - 1603 - 2。
袁强,欧阳生,王安,朱伟,Maiti R, Lin H, Hamilton J, Haas B, Sultana R, Cheung F, Wortman J, Buell CR:基因组研究所Osa1水稻基因组注释数据库。植物生理学报,2005,38(1):18-26。10.1104 / pp.104.059063。
拟南芥信息资源。[http://www.arabidopsis.org]
Kuhl JC, Cheung F, Yuan Q, Martin W, Zewdie Y, McCallum J, Catanach A, Rutherford P, Sink KC, Jenderek M, Prince JP, Town CD, Havey MJ:一组独特的洋葱表达序列标记揭示了单子目芦笋和豌豆的表达序列和基因组差异。植物学报,2004,16(6):1145 - 1145。10.1105 / tpc.017202。
王国坤,王军,陶磊,谭军,张军,Passey DA,余军:禾本科基因组成梯度。中国生物医学工程学报,2002,31(2):851-856。10.1101 / gr.189102。
Santos CM, Martins NF, Horberg HM, de Almeida ER, Coelho MC, Togawa RC, da Silva FR, Caetano AR, Miller RN, Souza MT:温度胁迫下Musa acuminata ssp burmannicoides, var. Calcutta 4 (AA)叶片序列标签的表达分析。理论与应用,2005,(11):1517-1522。10.1007 / s00122 - 005 - 1989 - 5。
王晓明,王晓明,王晓明,等。大麦种质资源ssr标记的EST序列分析。理论与应用,2003,26(3):411-422。
Katti MV, Ranjekar PK, Gupta VS:真核生物基因组序列中简单序列重复序列的差异分布。中华生物医学工程学报,2001,18(1):1-7。
Jung S, Abbott A, Jesudurai C, Tomkins J, Main D:蔷薇科est中简单序列重复序列的频率、类型、分布和注释。功能集成基因组学,2005,5:136-43。10.1007 / s10142 - 005 - 0139 - 0。
Creste S, Benatti TR, Orsi MR, Risterucci AM, Figueira A:一个商业栽培品种微卫星位点的分离和鉴定穆萨acuminata。分子生态学报,2006,6(6):303-306。10.1111 / j.1471-8286.2005.01209.x。
Raboin LM, Carreel F, Noyer JL, Baurens FC, Horry JP, Bakry F, Tezenas Du Montcel H, Ganry J, Lanaud C, Lagoda PJL:香蕉三倍体出口品种的二倍体祖先:2n个恢复配子供体和n个配子供体的分子鉴定。分子育种,2005,16(6):333-341。10.1007 / s11032 - 005 - 2452 - 7。
Chou HH, Holmes MH: DNA序列质量修剪和载体去除。生物信息学,2001,17(12):1093-1104。10.1093 /生物信息学/ 17.12.1093。
王建军,李建军,李建军,等。基于基因序列的TIGR基因序列分析。中国生物医学工程学报,2009,28(2):391 - 391。10.1093 / nar / 28.1.141。
欧阳,吴建平。TIGR植物重复序列数据库:植物重复序列识别的一个集合资源。中国生物医学工程学报,2004,32(2):369 - 369。10.1093 / nar / gkh099。
estinformatics.org。[http://www.estinformatics.org]
致谢
这项工作得到了国际香蕉和大蕉改良网络(INIBAP)的支持,该网络现在是生物多样性国际的一部分,通过美国国际开发署的联动基金计划提供赠款。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
FC负责生物信息学,FC、CDT参与稿件撰写,CDT负责整体项目管理。两位作者都阅读并批准了最终的手稿。
权利和权限
本文由BioMed Central Ltd.授权发表。这是一篇基于知识共享署名许可(http://creativecommons.org/licenses/by/2.0),允许在任何媒体上不受限制地使用、分发和复制,前提是正确引用原创作品。
关于本文
引用本文
张,F,镇,c.d.a后端视图穆萨acuminata基因组。BMC Plant Biol7,29日(2007年)。https://doi.org/10.1186/1471-2229-7-29
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-7-29
关键字
- 细菌人工染色体
- 细菌人工染色体文库
- 微型倒置重复转座元件
- 穆萨Acuminata
- 蛋白质匹配