摘要
背景
广泛的刺激引起[Ca]的快速和短暂的增加2 +]cyt通过含有EF-hand基序的蛋白质传感器传递。这里,有一群栽培稻L.编码钙调蛋白(CaM)和CaM样蛋白(CML)的基因,除了钙调蛋白以外不具有其他功能域2 +分析了-binding EF-hand motif。
结果
通过对TIGR水稻数据库的功能分析和BLAST搜索,在水稻基因组中鉴定出最多243个可能具有EF-hand基序的蛋白质。利用基于氨基酸序列相似性的邻域连接树,将5个位点定义为凸轮基因和另外32种CML基因被鉴定。广泛分析基因结构、染色体位置、EF-hand基序组织、表达特征,包括RT-PCR分析和比较分析凸轮而且CML介绍了水稻和拟南芥中的基因。
结论
尽管许多蛋白质功能未知,但Ca基因家族的复杂性表明了Ca的重要性2 +-信号在调节细胞对刺激的反应,这一家族的蛋白质可能发挥关键作用的传感器。
背景
Ca2 +是所有真核细胞在响应外界刺激时触发生理变化的重要第二信使。由于Ca2 +- atp酶和Ca2 +-通道在细胞膜上,其细胞质浓度的快速和短暂的变化是可能的。在植物细胞中,广泛的刺激触发胞质[Ca2 +]不同幅度和特殊性质的增长[1,2],它们通常由蛋白质传感器传递,这些蛋白质传感器可以更好地结合Ca2 +.Ca2 +结合导致构象变化,调节它们的活性或与其他蛋白质相互作用的能力。对于Ca的大部分2 +结合蛋白,即Ca2 +-结合位点由一个称为EF手的特征螺旋-环-螺旋基序组成。每个环,包括第二侧翼螺旋的末端,提供了7个配体结合钙2 +具有五边形双金字塔几何结构。Ca2 +-结合配体位于+X*+Y*+Z*-Y*-X**-Z区域内,其中*表示中间残基。Ca的三个配体2 +配位体由残基1 (+X)、残基3 (+Y)和残基5 (+Z)中的羧酸氧提供,一个来自残基7 (-Y)中的羰基氧,两个来自残基12 (-Z)中的羧酸氧,这是一种高度保守的谷氨酸(E)。第7个配体由残基9 (-X)中的羧酸侧链提供,或由水分子提供。
在植物中,Ca2 +结合蛋白包括钙调蛋白(CaM)、钙调蛋白(Ca2 +-依赖蛋白激酶(CPK)和钙调神经磷酸酶b样蛋白(CBL) [3.- - - - - -5].最近,Reddy ASN及其同事分析了拟南芥完整的基因组序列,确定了250个编码ef -hand蛋白的基因,并将其分为6类[6].CaM,一个独一无二的Ca2 +除了Ca以外不具有其他功能域的传感器2 +-结合基序与众多cam相关蛋白属于IV类。CaM是一种小的(148个残基)多功能蛋白,可转导钙增加的信号2 +通过结合和改变多种靶蛋白的活性来浓缩。这些蛋白质的活性影响植物细胞对大量特定刺激的生理反应[7].在植物中,一个显著的特征是CaM的许多亚型可能出现在单一植物物种中。目前已从几种植物中鉴定出编码CaM及相关蛋白的大型基因家族,但除拟南芥外,编码CaM及相关蛋白的基因家族尚未在全基因组尺度上进行广泛研究。此外,已发表的关于水稻CaMs个体的研究数量非常有限[8- - - - - -10].
随着基因组DNA测序项目的完成栽培稻L.所有的序列都属于一个多基因家族,如CaM和相关蛋白可以被鉴定。初步搜寻栽培稻L.数据库揭示了大量编码cam样蛋白的基因。在拟南芥中,McCormack和Braam [11]已经从拟南芥基因组中鉴定的250个EF-hand编码基因中鉴定出IV和V组成员的特征。六个位点被定义为凸轮基因和另外50个基因是cam样的(CML)基因,编码的蛋白质主要由EF-hand Ca2 +结合主题。拟南芥中CaM及相关钙传感器蛋白的高度复杂性表明它们在Ca中的重要而多样的作用2 +信号。由于水稻基因组小,染色体与其他主要谷类作物共线性,因此被认为是单子叶和谷类植物的模式植物,了解这种蛋白质家族是如何存在于水稻基因组中的将是很有趣的。在这项研究中,我们从水稻基因组中鉴定了编码含有EF-hand基序的蛋白质的基因,这些基因与CaM有关。对所鉴定的基因和蛋白序列进行分析,包括基因结构、染色体位置、EF-hand基序组织和表达特征,并与拟南芥进行比较凸轮而且CML基因被执行。
结果与讨论
含ef -hand蛋白的鉴定及系统发育分析
为了鉴定含有ef -hand的蛋白,我们首先对栽培稻L.基因组研究所(TIGR) [12]使用5种不同的方法,包括HMMPfam, HMMSmart, BlastProDom, ProfileScan和superfamily,如“方法”部分所述。其次,利用水稻CaM1的氨基酸序列对水稻数据库进行检索。10]及CBL3 [13因为BLASTp程序中的查询和域搜索没有找到的蛋白质序列被添加到列表中。此外,我们还回顾了通过各种方法鉴定大米中含有ef -hand蛋白的文献报道。所有这些蛋白序列再次使用InterProScan分析EF hands和其他结构域[14].InterProScan是一种蛋白质结构域识别工具,结合了来自Interpro联盟成员数据库的不同蛋白质特征识别方法。15].结果,域搜索识别了245个蛋白质,但6个序列在默认设置下没有被InterProScan识别的EF手,因此它们被排除在进一步的分析中。BLAST搜索又发现了四种含有ef -hand的蛋白,文献综述没有发现额外的蛋白。总共,在大米中鉴定出最多243种假定含有ef -hand的蛋白质[见补充文件]1].这些蛋白质中有近一半不包含InterProScan预测的其他可识别的结构域。值得注意的是,有24个蛋白质包含一个EF-hand基序,仅由一个预测程序识别,可能是假阳性。
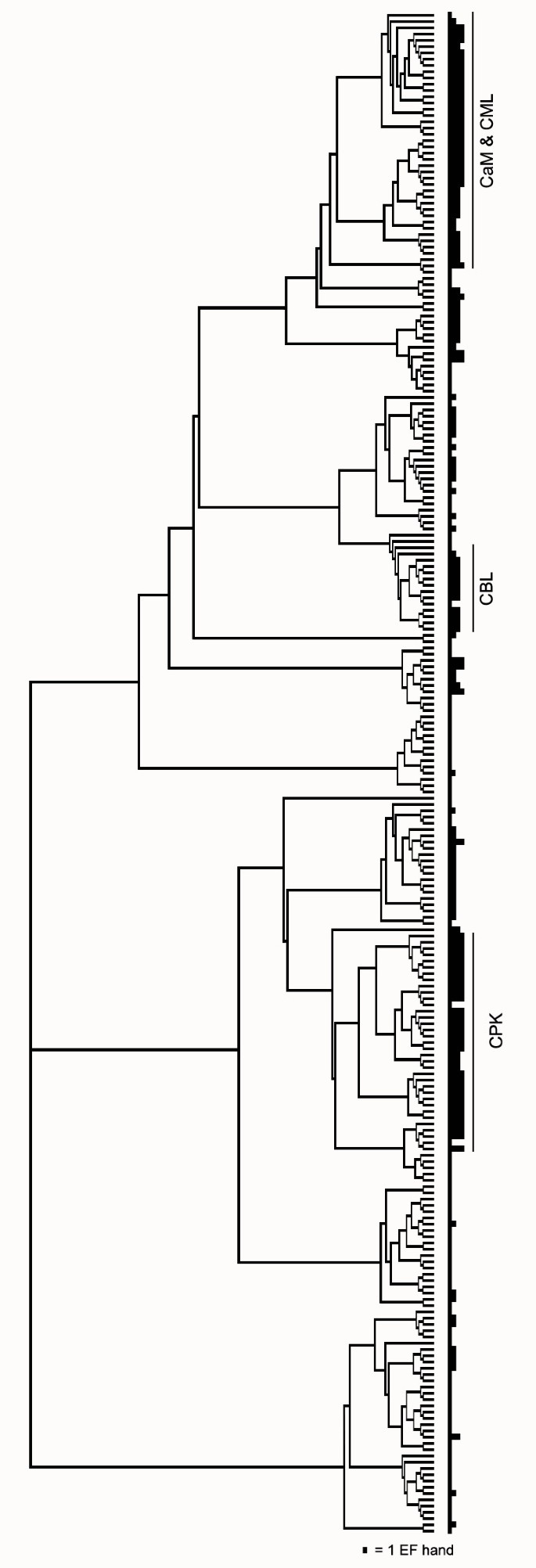
接下来,InterProScan识别出的所有含有EF-hand基序的蛋白质序列使用Clustal X [16][见附加文件2].使用邻居连接法和自举分析进行树构造。数字1显示了在没有任何基因标识符的情况下,InterProScan对每个蛋白质预测EF手的数量的树形轮廓。结果,除了Ca2 +结合EF-hand的图案分布在树上,但大多数集中在上半部分。相反,大多数位于下半部分的蛋白质含有额外的结构域,为其功能提供线索,包括转录因子、离子通道、DNA或ATP/ gtp结合蛋白、线粒体载体蛋白、蛋白磷酸酶和蛋白激酶。两组已知的含有ef -hand的蛋白质:类钙调神经磷酸酶b (CBL) [13]和Ca2 +-依赖蛋白激酶(CPK)蛋白[17]分别分组,如图所示1.我们观察到大多数含有四个EF-hand基序的蛋白质要么在CPK组中,要么位于树的顶部,围绕着典型的CaM蛋白。除两个外,所有由“CaM & CML”表示的蛋白都与OsCaM1具有至少25%的氨基酸同源性,并被选择用于进一步分析。此列表应包含与CaM相关或具有基于Ca的功能的大米蛋白2 +-类似CaM的绑定模式。这些基因的存在及其推导出的氨基酸序列是通过另一个注释数据库——水稻注释项目数据库(RAP-DB)证实的[18].
Rice CaM蛋白
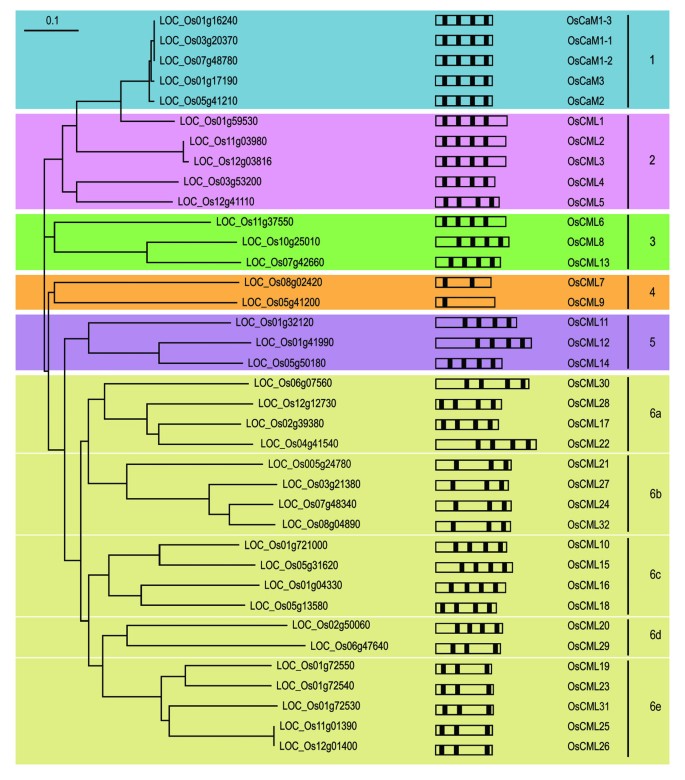
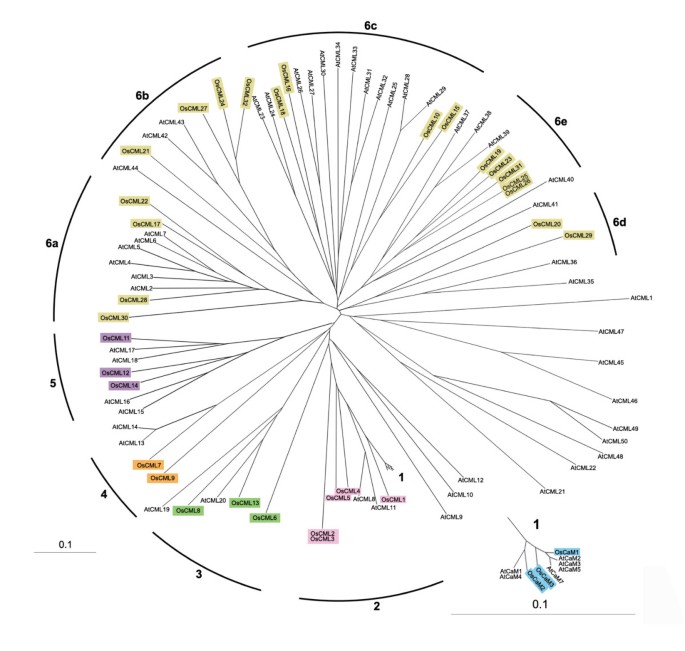
对所选蛋白质的氨基酸序列进行系统发育分析。使用邻居连接方法构造树,并使用ClustalX进行引导分析[参见附加文件]3.]生成了一个共识树,如图所示2.这一分析使我们将这些蛋白质分为6组:1-6。是什么定义了一个“真正的”CaM,并将其与扮演不同角色的CaM样蛋白区分开来在活的有机体内仍然是一个悬而未决的问题。不同的实验方法,包括生化和遗传分析,已被用来解决这个问题[19].本研究通过基于氨基酸序列相似性的系统发育分析,在第1组中鉴定出5个与其他植物已知典型CaMs氨基酸序列同一性最高(≥97%)的蛋白质。由于这些高度的氨基酸同一性,我们将它们归类为“真正的”CaMs,可能发挥典型CaMs的功能。分别命名为OsCaM1-1、OsCaM1-2、OsCaM1-3、OsCaM2和OsCaM3。它们的特点总结在表中1.
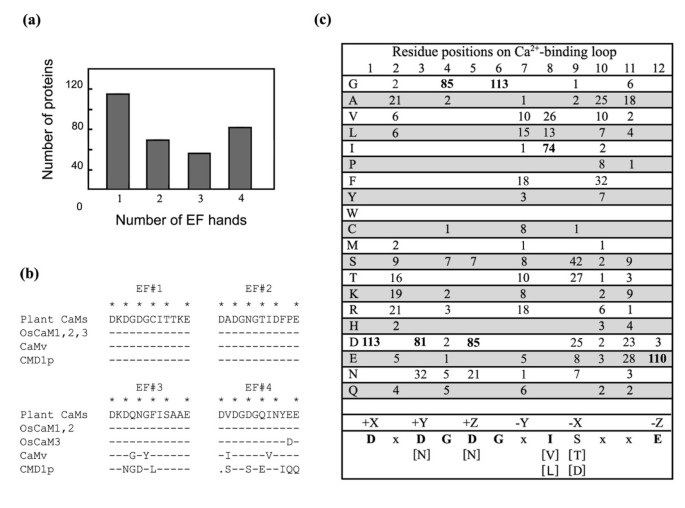
OsCam1-1;OsCam1-2而且OsCam1-3编码相同的蛋白质,然而OsCam2而且OsCam3编码一种只有两个氨基酸差异的蛋白质,它们的序列与OsCaM1蛋白有98.7%的同源性。OsCaM氨基酸序列与其他物种典型CaMs的多序列比对如图所示3.表明了它们高度的序列守恒。值得一提的是,OsCaM1氨基酸序列与大麦中典型CaMs的氨基酸序列相同(h . vulgare)和小麦(t . aestivum)反映了单子叶谷物植物之间的密切关系。平均而言,OsCaM氨基酸序列与植物、脊椎动物和酵母的氨基酸序列分别具有约99%、90%和60%的同源性。CaM-靶蛋白复合物形成机制中参与疏水相互作用的疏水残基高度保守,对CaM功能至关重要。植物CaMs中保守的8个蛋氨酸(M)残基和9个苯丙氨酸(F)残基在所有oscam中都存在。除了植物CaMs中145-146位的蛋氨酸残基与脊椎动物蛋白相比移位了一个残基外,这些残基在植物CaMs和脊椎动物CaMs之间保持着保存。由于其具有相当大的构象灵活性[20.]和弱极化的蛋氨酸残基(估计占CaM疏水斑块可达表面积的近一半)使其能够以序列无关的方式与靶蛋白相互作用[21].与植物CaMs功能相关的序列保守还包括位于116位的赖氨酸(K),假定该赖氨酸是三甲基化的。所有的OsCaM蛋白在这个位置都有一个赖氨酸残基。赖氨酸116三甲基化被认为是一种翻译后修饰,有助于调节CaM活性。EF手的母题将在后面的“EF手的数量和结构”小节中讨论。
多种CaM异构体的存在是植物CaM的一个决定性特征。尽管对基因冗余的解释仍不能排除,但越来越多的证据表明,每一种解释都是可能的凸轮基因可能具有独特而重要的功能。先前的报道表明,高度保守的CaM亚型实际上以不同的方式调节靶蛋白[22].据报道,植物组织中一些但不是全部的CaM多重异构体在特定刺激下的诱导表达[10,23因此,CaM亚型之间可能存在对靶蛋白的竞争。令人着迷的是OsCam1-1,OsCam1-2,OsCam1-3基因编码相同的蛋白质。在整个进化过程中,这些蛋白质序列是如何在自然选择压力下保持的,目前还没有明确的答案,但很可能这些基因中的每一个都具有生理意义。
水稻CaM-like (CML)蛋白
图中系统发育分析的剩余蛋白质2根据McCormack和Braam的分类,被命名为CaM-like或CML [11].像CaM一样,这些蛋白质完全由EF手组成,没有其他可识别的功能域。表中列出了它们的特点1.根据它们与OsCaM1氨基酸相同的百分比来命名,计算方法是用相同的残基数除以已对准以强调相同氨基酸的残基总数。这些蛋白质是由145 ~ 250个氨基酸残基组成的小蛋白质,与OsCaM1具有30.2% ~ 84.6%的氨基酸同源性。第2组的所有CML蛋白与OsCaM1的氨基酸序列相同超过60%。3、4、5组CML蛋白与OsCaM1的同源性分别为48.2%、46.9%、43.8%。根据这些蛋白质氨基酸序列相似性,通过自助系统进化树将第6组CML蛋白分为5个亚群:6a-6e。除了OsCML10(45.6%)外,这些蛋白质与OsCaM1的共享性不超过40.7%,平均为35.6%。6b和6e组的所有成员都包含三个EF-hand motif,尽管配置不同。
一些重要的CaM功能特征仅存在于少数CaM样蛋白中。第一个EF手残基7(-Y)的特征半胱氨酸(C)是高等植物CaM序列的标志,除了三个高度保守的CML蛋白OsCML4、OsCML5和OsCML6外,所有CaM样蛋白中都不存在。基于多序列比对,只有OsCML4、OsCML5、OsCML7、OsCML10、OsCML17、OsCML18和OsCML28是cam样蛋白中含有赖氨酸的位置与cam的Lys116相当。这些特征可能是蛋白质功能相似的指标在活的有机体内与凸轮的功能相匹配。OsCML4和OsCML5是唯一具有这两种特征的cam样蛋白。然而,在大多数OsCML蛋白中发现了CaM功能的另一个重要决定因素,即高比例的蛋氨酸(M)残基。oscml中M残留的平均百分比为4.6%,而oscam中M残留的平均百分比为6.0%。考虑到在其他蛋白质中发现的通常较低的百分比,cml中富含met的特征可能表明它们与cam有关,并且可能有类似的作用机制,即Ca构象变化引起疏水残基的暴露2 +绑定。尽管如此,一些新获得的cml特有的特征可能允许他们微调他们的Ca2 +-调节的活动到更专门的功能。
在这些蛋白质中,有三个oscmll包含一个扩展的C端基本结构域和一个CAAX (C是半胱氨酸,a是脂肪族,X是各种氨基酸)基序,一个假定的烯酰化位点(OsCML1中的CVIL和OsCML2和3中的CTIL)。OsCML1,也称为OsCaM61,被Xiao及其同事鉴定为一种新的cam样蛋白[8].据报道,CML蛋白在戊烯化时与膜相关,在未戊烯化时定位于细胞核[9].之前在矮牵牛中发现的一种名为CaM53的类似蛋白质也含有一个扩展的c端基本结构域和一个CAAX基序,这是高效的戊烯化所必需的[24].类似的亚细胞定位CaM53取决于其烯酰化状态的报道。为了找到另一种可能的修饰,所有的蛋白质都被计算机程序分析,豆蔻酰[25].因此,OsCML20被预测包含一个潜在的豆蔻酰化序列。在其余的OsCML蛋白中没有发现其他可能的豆蔻酰甘氨酸,无论是末端的还是内部的。此外,为了确定OsCML蛋白可能的定位,我们利用targetP [26].预测OsCML30含有内质网信号序列,预测OsCML21为细胞器蛋白。对于OsCaMs和其他OsCMLs,不存在靶向序列,因此,它们可能是细胞质或核蛋白
EF手的数量和结构
含有EF-手蛋白的水稻EF-手的数量在1 ~ 4个之间。图中显示了具有1、2、3或4个EF手的蛋白质的总数4.结果表明,在鉴定出的243个蛋白中,几乎所有含有4个EF手的蛋白都被纳入了我们的研究或者是CPK蛋白。所有五种OsCaM蛋白都有两对EF手,具有植物cam中常见的特征残基。Ca2 +-结合位点在植物CaMs EF手上与OsCaM1、OsCaM2、OsCaM3、脊椎动物CaM和酵母CMD1p的比较如图所示4 b.Ca2 +oscam之间的-配位残基与植物CaM一致序列的残基是不变的。其他残留在Ca2 +-binding环也是保守的,除了在OsCaM3中第4 EF手的残基11处的天冬氨酸(D)。在oscam的20个EF-hand基序中,残基1(+X)和残基3(+Y)仅被天冬氨酸(D)填充;残基5(+Z)为天冬酰胺(D)和天冬酰胺(N);残基12(-Z)是谷氨酸(E),它总是在大多数Ca的这个位置上发现2 +-绑定EF手图案。该残留物可旋转形成双齿或单齿金属离子螯合。谷氨酸提供两个配位,有利于Ca2 +在毫克2 +协调(27].残基7(-Y)通常是变化的;残基9(-X)为天冬酰胺(D)、天冬酰胺(N)、苏氨酸(T)和丝氨酸(S),这些都是植物cam中常见的氨基酸。
水稻蛋白EF手的特征.(a)含有1、2、3或4个EF-手的含EF-手蛋白质的数量。(b) oscam的EF手#1-4与典型植物CaM、脊椎动物CaM (CaMv)和酿酒酵母以植物CaMs一致性序列为标准的CaM (CMD1p);在其他序列中相同的残差用破折号(-)表示,为了比对而引入的间隙用点(.)表示。(c) Ca2 +在32个OsCML蛋白质中,-结合环显示为氨基酸(如左边所示)出现在每个位置(如上面所示)的频率。最常发现的氨基酸用粗体字母表示,并在下面显示为一致序列以及作为Ca的残基的位置2 +-以笛卡尔坐标表示的结合配体。括号内的残基是在每个位置上经常发现的替代残基,“x”是各种氨基酸。Ca类残基2 +-结合配体用星号(*)标记。
每个蛋白质序列的示意图,用封闭框表示预测EF手,如图所示2.在所有已识别的OsCaM和OsCML蛋白中,大约四分之三成对存在的EF手(59对)被24个氨基酸中断。除了两对相距小于24个氨基酸的氨基酸外,其余的氨基酸彼此之间的相对距离相似,在25-29个氨基酸之间。大多数OsCML蛋白都有两对或至少一对可识别的EF手,除了OsCML9有一个EF手和OsCML7似乎有两个独立的EF手。OsCML7和OsCML9很有趣,因为它们与OsCaM1有很高的氨基酸同源性(47.7%和46.1%),但它们只有2个和1个EF手;与其他OsCML蛋白相比,蛋氨酸(M)含量相对较低(2.8%和3.2%)。此外,具有一对可识别EF手的10个OsCML蛋白有一个额外的EF手,不与任何其他基序配对。CaM分子中EF-hand基序的配对有助于提高其对Ca的亲和力2 +,因此这些蛋白中未配对的EF手可以结合Ca2 +具有较低的亲和力,或者可能是非功能性的。
Ca配体2 +OsCML蛋白EF-hand基序的配位高度保守。公元113年2 +对-结合序列进行比对,发现氨基酸的频率如图所示4摄氏度.大多数残留在Ca2 +-结合环在OsCML蛋白中是保守的,这表明它们大多是功能性EF手。与oscam类似,残基1(+X)仅被天冬氨酸(D)填充;残基3(+Y)和残基5(+Z)通常是天冬酰胺(D)或天冬酰胺(N)。即使它们不是配位残基,6位的甘氨酸(G)是绝对保守的,疏水残基(I、V或L)总是在OsCaM和OsCML蛋白的133个EF手的8位被发现。残基12(-Z)主要是谷氨酸(E),除了OsCML7、OsCML8和OsCML13中的EF手,它们有天门冬氨酸(D)。OsCML8和OsCML13有两对EF-hand基序,OsCML7有两个EF-hand基序,在羧基端残基12处有D。凯茨及其同事[27],此前报道E12突变到D降低了EF手对Ca的亲和力2 +增加了100倍,提高了Mg的亲和力2 +10倍。这些EF手很可能结合Mg2 +而不是Ca2 +Mg的生理意义2 +-结合cam类活性尚不清楚。
凸轮而且CML基因结构和染色体分布
的结构OsCam而且OsCML通过比较它们的全长cdna和相应的基因组DNA序列来绘制基因图谱。在没有全长cDNA的情况下,使用部分cDNA和EST序列。他们的结果与TIGR数据库中的注释进行了比较和验证。37人中OsCam而且OsCML基因中,有13个基因的编码区含有内含子,而这些内含子在第5组和第6组成员中都没有发现。值得一提的是,由TIGR标注OsCam1-2而且OsCML1这些基因被证明具有一种交替拼接的mRNA,编码一种略有不同的蛋白质,但几乎没有证据支持,因此它们被从我们的列表中删除了。含内含子基因内含子-外显子结构示意图如图所示5.所有OsCam基因含有一个内含子,该内含子在编码Gly26的密码子中中断了它们的编码区域,这是所有植物的典型重排凸轮基因。
有趣的是,所有内含子OsCML基因也在相同的位置被内含子中断OsCam基因。内含子位置的保守表明它们之间的密切关系,这与这些基因编码CML蛋白1-4组成员的事实是一致的,这些成员与OsCaMs密切相关。OsCML1,OsCML2,OsCML3基因含有一个额外的内含子,该内含子位于编码传统cam的基因的最后一个残基对应的密码子上。这些蛋白具有一个扩展的c端碱基结构域和一个推测的戊烯化位点。这些内含子的位置反映了这些蛋白质中功能域的分离,并表明编码其羧基扩展的序列是在进化后期通过现有的融合而产生的凸轮附加外显子的基因。同样的,OsCML8而且OsCML13编码第3组蛋白质具有相同的基因结构,即相同的内含子数量(6)和位置。基因复制事件导致的存在OsCML8而且OsCML13OsCML8和OsCML13之间的氨基酸高度相同(60%)也支持这一结论。在这些蛋白质中,6个内含子中的一个位于编码第三个EF-hand基序的序列中,其位置与第一个EF-hand基序的Gly26相当。该内含子可能是复制事件的残余,最初在这些蛋白质中产生了两个EF-hand对。有趣的是,OsCML8和OsCML13是在残基12(-Z)处含有天冬氨酸(D)的三种oscmll中的两种。这些观察结果表明,OsCML8和OsCML13中E12向D的突变可能发生在导致它们存在的复制事件之前。
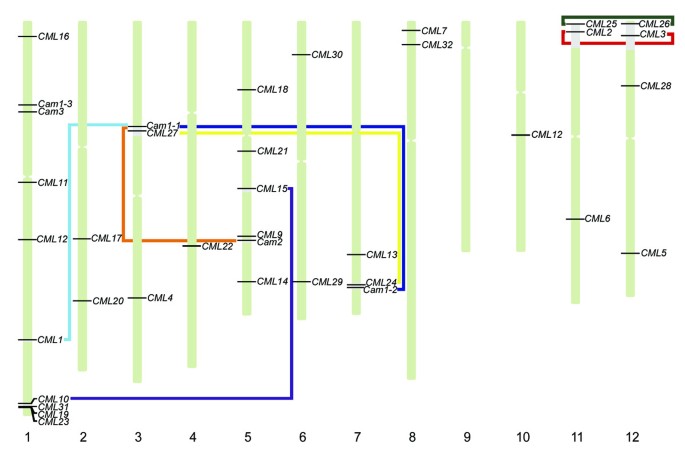
每个基因的染色体位置由TIGR数据库中的注释确定。OsCam而且OsCML基因分布在水稻的11条染色体上,如图所示61号染色体的基因数量最多(10个)。OsCam1-1在3号染色体上,OsCam1-2在7号染色体中;OsCam1-3,OsCam3在染色体1中;而且OsCam2在5号染色体上。它们的核苷酸序列具有86 - 90%的同源性,低于它们的氨基酸同源性(98-100%)。多个OsCam编码几乎相同蛋白质的基因通过自然选择得以保留,这表明每个基因的功能意义。OsCam1-1而且OsCam1-2它们编码相同的蛋白质分别被定位到染色体3和7的重复区域。OsCam1-1而且OsCam2它们也位于各自染色体的重复基因组片段中。这些观察结果表明,这些基因对来源于片段复制。此外,还有许多对/组的OsCML编码具有高度氨基酸同一性(≥60%)的蛋白质的基因。OsCML2/OsCML3(98.9%相同)和OsCML25/OsCML26(100%相同)是最密切相关的对。OsCML2而且OsCML3编码电位Ca2 +-结合蛋白在第2组中具有绝对保守的c端序列,其中包含一个烯丙基化位点(CTIL)。OsCML2而且OsCML25;而且OsCML3而且OsCML26分别被定位到11号和12号染色体最近复制的区域。因此,OsCML2/OsCML3;而且OsCML25/OsCML26可能是由片段复制事件引起的。其他密切相关的cam样基因对/组可能是由基因复制事件衍生的OsCML1/OsCam1-1;OsCML10/OsCML15;OsCML24/OsCML27;而且OsCML19/OsCML23/OsCML31.每对或组中的所有成员都有相同数量和位置的ef手图案。根据TIGR分析预测的片段复制位置以及受影响基因的染色体位置如图所示6.相反,OsCML19,OsCML23而且OsCML31在1号染色体上按串联方向排列,表明它们来源于串联复制。有趣的是,OsCML27是相邻的OsCam1-1在3号染色体和它的复制基因上,OsCML24,与OsCam1-2(OsCaM1-1和OsCaM1-2 100%相同)。因此,可能会发生局部复制和段段复制。
水稻与拟南芥的比较分析凸轮而且CML基因
对水稻CaMs和cml以及拟南芥CaMs和cml的全氨基酸序列进行系统发育分析。使用邻居连接方法构建树,并使用ClustalX进行自举分析[参见附加文件]4].在拟南芥中,基于氨基酸相似性的相邻连接树,McCormack和Braam [11]将cam和cml分为9组。我们发现一些水稻CaMs和cml与拟南芥CaMs和cml具有高度的相似性,家族成员之间的关系与之前报道的拟南芥相似,如图所示7.拟南芥和水稻的所有OsCaM蛋白高度保守(同源性为96.6%-99.3%)。有趣的是,拟南芥和水稻都有三个OsCam编码相同蛋白质的基因(ACaM2,3.,5而且OsCam1-1,1 - 2,1 - 3).水稻cml第2、3、4和5组蛋白分别与拟南芥cml第2、5、3和4组蛋白密切相关。更分散的水稻CML组6a至6e也分别分布在拟南芥CML组6、7、8、6和9的成员中。显然,两个物种的第1组都嵌在第2组中。这是由于第1组(CaMs)的任意分离造成的,即使第2组成员与第1组蛋白质具有非常高的同一性(至少50%)。因为是什么定义了一个“真正的”CaM,并将它与CaM样蛋白区分开来,后者起着不同的作用在活的有机体内尚不清楚,因此目前,只有具有极高身份度(>97%)的成员被分组在一起,以强调他们是被考虑过的,并且可能是“真正的”cam。
根据氨基酸序列比对(数据未显示),许多OsCMLs在拟南芥中有假定的同源。在第2组中,OsCML4与AtCML8和AtCML11具有高度的同一性,除了AtCML11缺少第一个内含子外,OsCML4具有相同的内含子数量(3)和位置。同样的,AtCML19而且AtCML20它们与它们的同系物(OsCML8而且OsCML13组3)具有相似的基因结构,即保留了水稻中存在的6个内含子中的5个。有趣的是,AtCML19/20和OsCML8/13蛋白在它们的一个EF手中有天冬氨酸(D)残基12(-Z),尽管不是在同一手。AtCML13和AtCML14被认为具有共同的祖先,与第4组OsCML7具有很高的同源性(74.3%和70.9%),并且在EF手的第3个EF手位置对应的EF手中均存在E12到D的突变。然而,OsCML7失去了对应于第二个位置的EF手,而在AtCML13和AtCML14中发现了第二个E12到D的突变。因此,与AtCML13和AtCML14相似,OsCML7与正则氨基酸只有一个EF通路,这可能导致结合Ca的能力受损2 +.在OsCML组5中,OsCML11与AtCML17和AtCML18非常相似,有趣的是,与其他与CaMs具有相似身份水平的CML蛋白相比,OsCML11都具有相对较低的蛋氨酸(M)百分比。OsCML11的蛋氨酸含量仅为1.4%,说明其作用于Ca2 +结合可能不同于CaM构象变化时的疏水表面暴露。
先前的报道从拟南芥基因组中鉴定出250个含有ef -hand的蛋白质[6].7个位点被定义为凸轮基因和另外50个基因CML基因(28].在这里,我们鉴定出243个含有ef -hand的蛋白质,其中5个凸轮基因与32CML基因。更少的水稻cml成员被鉴定出来,一些拟南芥cml不属于水稻蛋白质的任何一组,这可能是因为我们在这些分析中包括的水稻OsCML蛋白质与典型CaMs至少有25%的同源性,而在拟南芥中只有16%。我们注意到所有不属于水稻蛋白质组的拟南芥蛋白质与典型CaMs的同源性仅为16 - 30%。因此,这两种植物似乎都含有或多或少相似数量的ef -hand蛋白和cam样蛋白。两者也有相似的数量肌酸磷酸激酶拟南芥中有34株,水稻中有29株CBL基因(拟南芥和水稻均有10个)[13,29].然而,我们观察到的一个显著不同的特征是三个OsCML蛋白(OsCML1、OsCML2和OsCML3)具有羧基端CAAX基序进行戊烯化,但在拟南芥的cml中没有发现[11].了解这些大米蛋白具有什么功能以及烯丙化状态如何影响它们的活性将是很有趣的。
凸轮而且CML表达式
由于cDNA或EST克隆的存在表明相应基因的表达,我们对cDNA/EST水稻数据库进行了搜索。搜索显示,大多数的OsCam而且OsCML基因有相应的cDNA或EST克隆。我们已经确定了所有的EST克隆OsCam而且OsCML基因。根据EST克隆来源于哪个库,可以推断其表达特征。表中列出了在不同器官中发现的EST克隆的数量2.根据他们的EST克隆的可用性,大多数OsCam而且OsCML基因被表达。一些OsCML与其他基因相比,基因在特定器官中高度表达OsCML13而且OsCML18在花组织中。没有cDNA或EST克隆OsCML6,OsCML19,OsCML23,OsCML25.然而,这些基因的表达并不仅仅依赖于它们EST克隆的缺失。尽管如此,其余的EST克隆的可用性OsCam而且OsCML基因表明它们得到了表达,并且确实是功能基因。
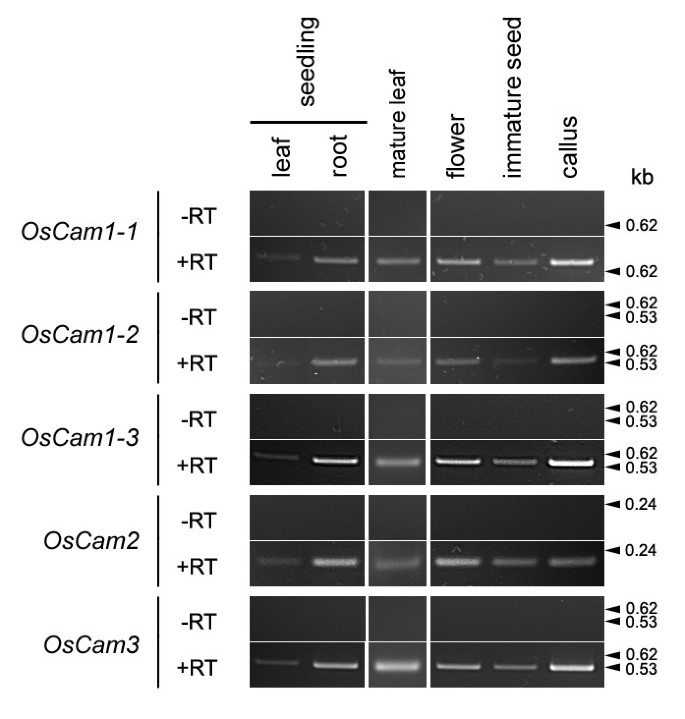
因为五OsCam基因只编码三种不同的蛋白质,它们是否具有不同的生理功能是一个有趣的问题。在这里,我们通过实验确定了每一个的表达是否OsCam基因仅限于特定的器官。从水稻叶片、根、花、未成熟种子和愈伤组织中分离总RNA,进行逆转录和PCR扩增反应。通过计算机分析这些基因对应的cDNA和EST序列选择的引物见表3..每次实验反应并行进行不添加逆转录酶的对照RT-PCR反应,以确保得到的产物可以归因于逆转录酶反应的产物。数字8显示了基于每个基因序列(698、526、551、201和520碱基对)的预期大小的带OsCam1-2,OsCam1-2,OsCam1-3,OsCam2,OsCam32周龄的幼苗、成熟的叶片、花、未成熟的种子和愈伤组织等器官或组织均检测到。对照RT-PCR反应未检测到条带。需要注意的是,本研究使用的RT-PCR条件不允许定量测定,因此无法比较不同器官或不同基因之间的表达水平。然而,可以得出结论,所有的OsCam基因在我们检查的所有器官中都有表达。
密切相关的表达凸轮基因在一个器官中并不奇怪。在其他植物物种中也有类似的报道。在烟草行业,全部13家凸轮密切相关的基因在几乎所有器官中都有表达,只有少数例外NtCam13,它只表达在词根[23].然而,NtCam13编码一种与典型植物CaMs相似度低于80%的蛋白质。同样的,ACam1-ACam5编码几乎相同蛋白质的基因都在拟南芥的叶片和角果中表达[30.,31].而凸轮表达在不同的细胞中普遍存在,蛋白质浓度在特定的细胞类型中可能有所不同。免疫定位研究表明,根冠细胞和分生区增加了CaM的积累[32].此外,水平的稳态转录凸轮据报道,基因在不同的发育阶段会被调节[33,34]以及对盐度、风、寒冷、伤害和病原体攻击等外部刺激的反应[23,35- - - - - -37].OsCam1-1在渗透胁迫下,叶片含量迅速而强烈地增加[10,38].CaM亚型的特定器官的表达调制可能在及时的方式中发挥其作用。
结论
我们在水稻基因组中鉴定出243个可能具有EF-hand基序的蛋白和37个CaMs以及相关的潜在钙传感器蛋白。由这些基因编码的大多数蛋白质的功能仍然未知。然而,CaM蛋白家族的复杂性可能反映了Ca的重要性2 +信号调节细胞对各种细胞刺激的反应,这个蛋白质家族可能起着至关重要的作用。本研究结果可为进一步研究Ca家族中的每一个成员提供依据,这对理解Ca的作用机制具有重要意义2 +调控的信号转导通路。
方法
基因结构和染色体分布的数据库检索和分析
水稻基因组研究所(TIGR)的基因组检索[39使用HMMPfam、HMMSmart、BlastProDom、ProfileScan和superfamily 5种不同的方法进行Interpro数据库匹配。含有EF-hand基序或Ca家族的蛋白质2 +每种方法分别收集到结构域PF00036、SM00054、PD000012、PS50222和蛋白家族SSP47473的结合蛋白。此外,BLAST搜索(blastp) [40]以水稻CaM1 [GenBank: NP_912914]和CBL3 [GenBank: NP_643248]的蛋白质序列作为水稻基因组查询序列。核苷酸和氨基酸序列以及有关感兴趣的每个基因的信息被获得。水稻注释项目数据库(RAP-DB)的基因注释[41]也被用来证实这些基因的存在和序列。通过比较从GenBank中获得的cDNA和基因组DNA序列,并在TIGR中搜索已识别的位点,确定基因结构和位置。当发现任何差异时,使用EST序列的信息。通过TIGR分析水稻基因组的染色体片段重复,确定基因重复。
对齐和树构造
如有必要,使用EST和cDNA序列验证编码区域的预测。通过InterProScan鉴定的含有EF手的推导出的蛋白质序列进行了系统发育分析。通过多序列比对程序ClustalX [16]使用默认设置。使用邻居连接方法进行比对并构建蛋白质树[42通过Clustal X进行引导分析(默认设置)。通过ClustalW进行多序列比对,将OsCaM蛋白与其他物种的蛋白进行比较。用于比对的序列的GenBank登录号如下:ACaM2 [GenBank: AAA32763];HvCaM [GenBank: AAA32938];T-CaM1 [GenBank: AAC49578];ZmCaM [GenBank: CAA52602];SCaM1 [GenBank: AAA34013];PCM5 [GenBank: AAA85155];MmCaM [GenBank: NP_033920];CMD1p [GenBank: AAA34504]。
蛋白质的氨基酸同一性和基序分析
推导出的氨基酸序列CaM和CaM样蛋白通过Align [43],氨基酸同一性百分比通过相同氨基酸的数量除以对齐序列的氨基酸残基总数来计算。使用InterProScan分析EF hands和其他结构域的所有蛋白质序列[44].利用InterProScan预测的信息,并将所有蛋白质的完整序列与植物EF-hand一致序列进行比较,确定EF-hand的位置。所有识别的EF手序列与ClustalX进行比对,生成一致序列。为了定位蛋白质修饰和靶向的序列,计算机程序:肉豆蔻酰[45]和targetP [46]被使用。
表达序列标签
对应的ESTs凸轮而且CML基因通过BLAST搜索确定栽培稻通过搜索GenBank中所有基因对应的UniGene条目[47].根据ESTs文库类型和文献综述确定所有基因的表达特征。
逆转录聚合酶链式反应(RT-PCR)分析
栽培稻l组织用冷冻的臼和杵在液氮中研磨。总RNA按照[48并用于逆转录。反转录由寡核苷酸(dT)启动15引物和PCR使用正向和反向寡核苷酸引物(Operon,德国)进行,见表3..确定每个基因在达到扩增平台期之前所需的周期数。Taq聚合酶在94℃2 min, 55℃1 min, 72℃2 min的程序下进行PCR扩增OsCam1-1;OsCam1-2;OsCam2;而且OsCam394°C 2分钟,58°C 1分钟,72°C 2分钟OsCam1-3.PCR产物经琼脂糖凝胶电泳分离,溴化乙锭染色和紫外荧光观察。所有用于RT-PCR的酶和化学试剂均购自Promega (Madison, WI, USA)。
参考文献
Knight H, Trewavas AJ, Knight MR:钙信号传入拟南芥对干旱和盐度的反应。植物学报,1997,12(5):1067-1078。10.1046 / j.1365 - 313 x.1997.12051067.x。
Kiegle E, Moore CA, Haseloff J, Tester MA, Knight MR:细胞类型特异性钙对干旱、盐和寒冷的响应拟南芥根。植物学报,2000,23(2):267-278。10.1046 / j.1365 - 313 x.2000.00786.x。
Zielinski RE:植物中的钙调素和钙调素结合蛋白。植物化学学报,1998,49:697-725。10.1146 / annurev.arplant.49.1.697。
Harmon AC, Gribskov M, Harper JF: cdpkks:每个Ca对应一个激酶2 +信号?。植物科学进展,2000,5:154-159。10.1016 / s1360 - 1385(00) 01577 - 6。
Luan S, Kudla J, Rodriguez-Concepcion M, Yalovsky S, Gruissem W:钙调素和钙调神经磷酸酶b样蛋白:植物特异反应偶联的钙传感器。植物学报,2002,14(增刊):S389-S400。
Day IS, Reddy VS, Shad Ali G, Reddy ASN:拟南芥含ef -hand蛋白的分析。中国生物工程学报,2002,3(10):56.1-56.24。10.1186 / gb - 2002 - 3 - 10 - research0056。
杨涛,杨文华,王晓峰,等:植物钙/钙调素介导的信号网络。植物科学进展,2003,8(10):505-512。10.1016 / j.tplants.2003.09.004。
肖超,辛华,董安,孙超,曹凯:水稻钙调素样蛋白基因的一个新发现,该基因具有一个不寻常的C端延长序列,携带一个推测的烯丙化位点。中国生物医学工程学报,1999,6(3):179-181。10.1093 / dnares / 6.3.179。
董安,辛华,于艳,孙超,曹凯,沈文华:水稻钙调蛋白OsCaM61的亚细胞定位与其烯酰化状态有关。植物分子生物学,2002,48(3):203-10。10.1023 /: 1013380814919。
植物钙调蛋白基因表达的钙信号介导和差异诱导栽培稻生物化学与分子生物学杂志,2005,38(4):432-439。
杨晓明,李志强,李志强,等。拟南芥钙调素及其钙敏感因子的研究。植物学报,2003,29(3):344 - 344。10.1046 / j.1469-8137.2003.00845.x。
袁强,欧阳山,王安,朱伟,Maiti R, Lin H, Hamilton J, Haas B, Sultana R,张F, Wortman J, Buell CR:中国科学院基因组研究所水稻基因组注释数据库。植物科学进展,2005,38(3):344 - 344。10.1104 / pp.104.059063。
Kolukinaoglu Ü, Weinl S, Blazevic D, Batistic O, Kudla J:钙传感器及其相互作用蛋白激酶:拟南芥和水稻CBL-CIPK信号网络的基因组学。中国农业科学,2004,34(4):344 - 344。10.1104 / pp.103.033068。
Quevillon E, Silventoinen V, Pillai S, Harte N, Mulder N, Apweiler R, Lopez R: InterProScan:蛋白质结构域标识符。中国生物医学工程学报,2005,33:W116-W120。10.1093 / nar / gki442。
穆德新泽西,Apweiler R, Attwood TK, Bairoch,贝特曼,宾斯D,布拉德利R,博克P,布赫P, Cerutti L,科普利R, Courcelle E, Das U,杜宾R,她W,高夫J,把手D,哈特N, Hulo N,卡恩D, Kanapin, Krestyaninova M,朗斯代尔D,洛佩兹R, Letumic我ㄧM,套装J, McDowall J,米切尔,Nikolskaya,果园,Pagni米,桥CP, Quevillon E, Selengut J, Sigrist CJA, Silventoinen城镇,Studholme DJ,沃恩R,吴CH: 2005年InterPro、进展和状态。中国生物医学工程学报,2005,33:D201-D205。10.1093 / nar / gki106。
Thompson JD, Gibson TJ, Plewniak F, Jeanmougin F, Higgins DG: CLUSTAL_X窗口界面:由质量分析工具辅助的多序列比对的灵活策略。中国生物医学工程学报,1999,25(24):476 - 482。10.1093 / nar / 25.24.4876。
Asano T, Tanaka N, Yang G, Hayashi N, Kamatsu S:水稻钙依赖蛋白激酶及其密切相关激酶基因家族的全基因组鉴定:水稻CDPKs基因家族的综合分析。植物生理学报,2005,46(2):356-366。10.1093 /卡式肺囊虫肺炎/ pci035。
Ohyanagi H, Tanaka T, Sakai H, Shigemoto Y, Yamaguchi K, Habara T, Fujii Y, Antonio BA, Nagamura Y, Imanishi T, Ikeo K, Itoh T, Gojobori T, Sasaki T:水稻注释项目数据库(rapd - db): hub for栽培稻ssp。粳稻基因组信息。中国生物医学工程学报,2006,34:D741-D744。10.1093 / nar / gkj094。
Buaboocha T, Zielinski RE:钙调素。植物生物学中的蛋白质-蛋白质相互作用。编著:McManus MT、Liang WA、Allen AC. 2002,英国谢菲尔德:谢菲尔德文献出版社,7:285-313。[Roberts JA(系列编辑:年度植物评论]
Gellman SH:蛋氨酸残基在非极性蛋白质表面序列无关识别中的作用。生物化学,1999,30:6633-6636。10.1021 / bi00241a001。
O’neil KT,降解WF:钙调蛋白如何结合其靶标:两亲性α-螺旋结构的序列独立识别。提波斯。1990, 15: 59-64。
Karita E, Yamakawa H, Mitsuhara I, Kuchitsu K, Ohashi Y:烟草钙调素在不同Ca下对植物NAD激酶的激活特征2 +浓度和ph值。植物生理学报,2004,45(10):1371-1379。10.1093 /卡式肺囊虫肺炎/ pch158。
王晓明,王晓明,王晓明,王晓明,王晓明,王晓明。烟草花叶病毒诱导13个钙调素基因的转录及转录后调控作用。中国生物医学工程学报,2001,29(3):344 - 344。10.1046 / j.1432-1327.2001.02301.x。
Rodriguez-Concepcion M, Yalovsky S, Zik M, Fromm H, Gruissem W:一种新型植物钙调素的烯丙基化状态指导蛋白的质膜或核定位。中国科学院学报,2004,18(3):366 - 366。10.1093 / emboj / 18.7.1996。
Bologna G, Yvon C, Duvaud S, Veuthey A-L: n端豆蔻酰化预测的集成神经网络。蛋白质组学。4(6):1626-1632。10.1002 / pmic.200300783。
Emanuelsson O, Nielson H, Brunak, Heijne:基于n端氨基酸序列预测蛋白质的亚细胞定位。中华分子生物学杂志,2000,30(3):344 - 344。10.1006 / jmbi.2000.3903。
王志强,王志强,王志强:钙镁结合蛋白的分子机制研究。生物物理学报,2002,32(3):344 - 344。
蔡永春,李志强,李志强,等。拟南芥钙信号通路中的CaMs和cml。植物科学进展,2005,10(8):383-389。10.1016 / j.tplants.2005.07.001。
Hrabak EM, Chan CW, Gribskov M, Harper JF, Choi JH, Halford N, Kudla J, Luan S, Nimmo HG, Sussman MR, Thomas M, Walker-Simmons K, Zhu JK, Harmon AC:拟南芥CDPK-SnRK蛋白激酶超家族。植物科学学报,2003,32(3):366 - 366。10.1104 / pp.102.011999。
王晓明,王晓明,王晓明,等:拟南芥CaM-3钙调蛋白基因的结构与表达。植物分子生物学杂志,1992,19:649-664。10.1007 / BF00026791。
Gawienowski MC, Szymanski D, Perera IY, Zielinski RE:钙调蛋白异构体拟南芥由多个不同的mrna编码。植物分子生物学,1993,22:215-225。10.1007 / BF00014930。
彭伟华,王晓明,王晓明,等。植物钙与信号转导的关系。植物生态学报,2003,26(4):349 - 349。
武泽东,刘志华,安国强,普瓦雅BW:马铃薯钙调蛋白基因家族的发育与接触诱导表达。植物分子生物学杂志,1995,27:93-703。10.1007 / BF00020223。
崔y r,曹EK,李SI, Lim有限公司加西南,曹乔丹,一个G:发育调节水稻钙调蛋白启动子的表达转基因烟草植物。生物化学学报,1996,6:541-546。
范德华,李志刚,李志刚,李志刚,李志刚:烟草钙调蛋白基因表达与钙信号通路的关系。植物科学学报,2004,29(4):457 - 457。10.1104 / pp.121.3.705。
德鲁默欧,Morère-le Paven MC, Montrichard F, Laval-Martin DL: NaCl胁迫对两种番茄钙调素转录水平和钙调素依赖性NAD激酶活性的影响。环境科学学报,2002,23:329-336。10.1046 / j.1365-3040.2000.00545.x。
Duval FD, Renard M, Jaquinod M, Biou V, Montrichard, Macherel D:萌发豌豆中三种钙调素异构体的差异表达及功能分析(Pisum一l .)种子。植物学报,2002,32:481-493。10.1046 / j.1365 - 313 x.2002.01409.x。
Kawasaki S, Borchert C, Deyholos M, Wang H, Brazille S, Kawai K, Galbraith D, Bohnert HJ:盐胁迫初期水稻基因表达谱。植物学报,2001,13:889-905。10.1105 / tpc.13.4.889。
TIGR水稻基因组注释。[http://www.tigr.org/tdb/e2k1/osa1/]
Altschul SF, Madden TL, Schäffer AA,张杰,张刚,Miller W, Lipman DJ: gapping BLAST和ppi -BLAST:新一代蛋白质数据库搜索程序。中国生物医学工程学报,1997,25:389-3402。10.1093 / nar / 25.17.3389。
水稻注释项目数据库。[http://rapdb.lab.nig.ac.jp/]
齐藤,倪明:邻居连接法:一种重建系统发育树的新方法。分子生物学杂志,1987,4(4):406-425。
浮雕成对对齐算法。[http://www.ebi.ac.uk/emboss/align/index.html]
InterProScan序列搜索。[http://www.ebi.ac.uk/InterProScan/]
豆蔻酸酯在ExPASy。[http://www.expasy.ch/tools/myristoylator/]
TargetP 1.1服务器。[http://www.cbs.dtu.dk/services/TargetP/]
国家生物技术信息中心。[http://www.ncbi.nlm.nih.gov/]
Verwoerd TC, Dekker BM, Hoekema A:一种快速分离植物rna的小规模方法。核酸研究,1989,17:2362-10.1093/nar/17.6.2362。
确认
这项工作得到了泰国国家科学技术发展局国家基因工程和生物技术中心的支持。bt - 01 - rg - 09 - 4711)。BB由朱拉隆功大学研究生院通过论文基金资助。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
BB和TB参与了数据库搜索和基因和蛋白质序列的广泛分析。BB进行了实验室工作,并编制了图表。TB进行数据分析和解读,并起草手稿。两位作者都阅读并批准了最终的手稿。
电子辅助材料
12870 _2006_145_moesm2_esm.aln
附加文件2:图中系统发育树的比对1作为ClustalX对齐文件。(aln843kb)
12870 _2006_145_moesm3_esm.aln
附加文件3:图中系统发育树的排列2作为ClustalX对齐文件。(aln16kb)
12870 _2006_145_moesm4_esm.aln
附加文件4:图中系统发育树的比对7作为ClustalX对齐文件。(aln67kb)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
杨晓东,李志强,李志强,等。水稻钙调素及其潜在钙传感器蛋白的全基因组鉴定与分析。BMC植物生物学7,4(2007)。https://doi.org/10.1186/1471-2229-7-4
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-7-4
关键字
- 逆转录聚合酶链反应
- 节段重复
- 植物凸轮
- 典型的摄像头
- 水稻注释项目数据库