摘要
背景
除了影响蛋白质加工和功能的遗传变异外,杂交种与亲本之间的差异表达基因为杂种优势或杂种活力的研究提供了重要的候选基因。基于我们对中国优质超级杂交稻(LYP9)及其亲本品种(93 - 11和PA64s在不同发育阶段的三种主要组织类型(叶、根和穗)中,我们分析了转录组并寻找与水稻杂种优势相关的候选基因。
结果
通过使用改进的标记-基因定位策略和两个最近注释的基因组组装(93-11和pa64),我们确定了10,268个额外的高质量标签,加上我们之前的结果,总共达到20,595个。我们进一步检测到8.5%和5.9%的物理定位基因在三联体中(至少在三个阶段中的一个阶段)存在差异表达P-值分别小于0.05和0.01。这些基因分布在12种主要的基因表达模式中;其中上调基因406个,下调基因469个(P< 0.05)。对鉴定基因的功能注释强调了杂交中上调的基因(其中一些是已知的酶)主要与促进叶片和根的碳同化有关。此外,我们还检测到一组与雄性不育相关的上调基因和442个与信号转导和蛋白质加工相关的下调基因,这些基因可能与水稻杂种优势有关。
结论
我们将转录序列信息与水稻基因组注释相结合,改进了标签-基因定位策略,对水稻杂种优势相关基因有了更全面的认识。不同基因型中与杂种优势相关的候选基因为探索杂种优势的分子机制提供了新的途径。
背景
杂种优势被定义为后代在数量和质量上优于其亲本,利用杂种优势原则已成为提高动植物生产力的主要做法[1]。在揭示水稻杂种优势的遗传基础方面已经投入了大量的努力(Oryza sativa, L),而这主要是由支配地位等机制来解释的[2]和上位[3.]。尽管许多研究者倾向于一种假设而不是另一种假设,但水稻杂种优势的生物学机制可能仅基于遗传方法,特别是基于经典遗传概念,无法完全表征。
近年来,有报道称杂交种与其亲本近交系之间的差异表达基因与杂种优势相关[4,5]。在小麦中,通过差异显示技术检测到多种差异表达基因,包括转录因子和参与代谢、信号转导、抗病和反转录转座子的基因,这些基因与杂种优势有关[qh]6,7]。甚至核糖体蛋白也被仔细研究过,因为它们是翻译活动和质体生物发生的指标[8]。各种技术已被应用于确定与杂种优势有关的基因,如各种基于序列和杂交的方法;一些研究产生了有趣的候选者,另一些则提出了这些候选者的表达模式[5,9]。例如,在小麦中发现了一种杂交特异性表达基因AG5(一种rna结合蛋白)[10]。另一项利用由9198个est组成的cDNA芯片研究了一个优秀水稻杂交种及其亲本在幼穗发育三个阶段的基因表达谱,结果表明存在显著的中亲本杂种优势[11]。然而,利用快速发展的基因组技术,有必要大规模地产生更多的数据。
SAGE技术是一种基于序列的方法,用于大规模研究基因表达,比基于EST(表达序列标签)的方法允许更深入的采样。它已被证明是一种非常强大的方法,用于大规模发现新的转录本,获取已表达转录本的定量信息,以及在文库之间进行定量比较[12- - - - - -14]。该技术已广泛应用于包括人类和小鼠在内的动物系统,特别是在癌症研究中,已经获得了数百个文库和近700万个SAGE标签[13,15]。在植物中,一些研究已经采用这种方法对拟南芥进行转录谱分析[16,17]和米饭[18,19]。然而,SAGE的一个瓶颈是标签到基因的定位,这是指明确确定由SAGE标签所代表的基因。其他限制包括缺乏准确的基因组序列和足够数量的SAGE数据。因此,应该鼓励产生公开数据的研究,因为杂种优势不仅仅是几个看似重要的基因的表现,而是许多基因的表现。
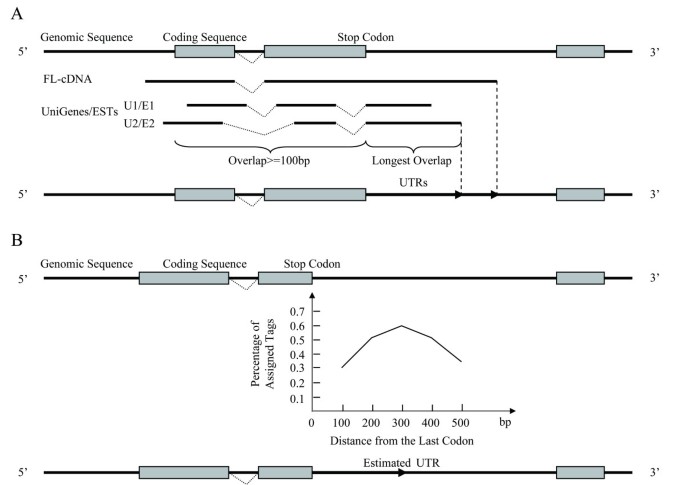
作为超级杂交水稻基因组计划(SRGP)的一部分,我们一直在研究水稻基因组,对杂种优势的分子机制特别感兴趣,重点研究一种优秀的超级杂交水稻(Liang-You-Pei-Jiu, LYP9(20.])及其亲本系,利用基因表达技术,包括EST和SAGE技术。我们当前工作的目的是从我们之前的SAGE研究中恢复更多的序列标签(基因表达信息)[21]。在我们的新分析中,SAGE标签被定位到两个新注释的基因组组合,父本栽培品种(93 - 11)和母系品种(Pei-Ai 64年代,PA64s)(华大基因未公布的数据)[22,23];当我们进行第一次分析时,后者是不可用的。SAGE标签与其自身基因组序列的完美匹配使我们能够以非常重要的方式绘制更多标签:与之前的结果相比,绘制的标签数量增加了两倍。我们还使用了三种类型的转录本,包括全长cDNA (FL-cDNA) [24],表达序列标签(est) [25,26]和UniGene数据,以及当前分析中的新策略。
结果
数据集
我们从9个SAGE文库中获得了465,164个SAGE标签,这些文库平行构建于超级杂交水稻的三个主要组织的不同生育阶段(LYP9)及其亲本(93-11和pa64)品种。这些文库是用从(1)水稻成熟乳白色期的叶片、(2)花粉成熟期的穗和(3)分蘖期的根中分离的mRNA构建的。21]。通过采用更严格的序列分析标准和质量改进方案,我们去除了与克隆连接物、载体和简单重复相匹配的污染标签,获得了68,462个唯一的经验标签;由于更严格的过滤器,这个数字比以前的数据集少21个标签。在这些独特标签中,30,595个(44.7%)标签被观察到不止一次。表中总结了不同库之间映射标签的分布情况1。我们将所有原始SAGE数据存入NCBI的基因表达综合数据库[27],这些数据可通过GEO系列登录号GSE8048访问。
评估数据集,虚拟标签和映射标签
为了获得评估数据集,我们构建了一个PCUE (P预测基因,FL-cDNA,UniGene,EST)数据库基于可用的基因组资源(见材料和方法)。我们分类了41,072个预测基因93 - 11分为三组:(1)21,676(53%)由一个或多个转录本支持,即由三个支持证据(或转录本类型)中的任何一个- FL-cDNA,UniGene,EST,(2) 19,396无支持证据,(3)10,702由所有三种类型的转录本支持。该评估数据集包含来自(3)的2480个测试标签,满足所有五个质量标准(参见材料和方法;表格2).
为了定义虚拟标签,我们需要根据预测的基因处理两类虚拟转录本:(1)由具有实际3'-UTR序列的转录本支持(图2)1)和(2)没有支持性证据,但通过添加人工3'- utr来定义(图2)1 b).从第一类开始,我们根据可变的3' UTR序列特征对13组不同的虚拟标签进行了分类(见表1)2).我们还发现,由最长的UniGene (Unimax, 97.22%)和最长的EST (ESTmax, 74.92%)构建的虚拟标签与测试标签的匹配率更高,这主要是由于它们的3'- utr更长。相比之下,由具有poly (a)信号的Uni-S和EST-S基团构建的虚拟标签的产率略差,但产率显著,分别为95.80%和71.60%。对于第二类,我们需要为要添加到预测基因中的人工utr选择一个长度范围。对于19,079个非冗余的fl - cdna(参见附加文件)1(3-UTR长度分布),其3-UTR具有明显的长度分布,平均为422 bp,中位数为295 bp,我们决定使用100 bp窗口,最佳长度范围为300 bp。利用cDNA、Unimax、ESTmax四组虚拟标签和300 bp 3'-UTR的预测基因进行进一步分析(表2)2).
我们为19,961个预测基因分配了20,595个唯一标签(表1)3.),分为三种类型:(1)16757个(81.36%)无歧义标签,(2)3316个(16.10%)物理映射到1668个基因的标签(两个或两个以上不同的标签分配给相同的预测基因),(3)698个(3.39%)物理映射到1536个基因的标签(每个标签分配给多个基因)。其中,转录本支持16430个(80%),已知证据不支持4341个(20%);后者主要是假设的转录本,要么在较低水平表达,要么特定于某些组织或发育阶段(基于我们自己的数据的微阵列和EST分析;数据未显示)。这个过程导致了更严格的标签到基因分配,与之前的结果相比,我们获得了10,268个额外的标签。此外,我们发现当前数据中缺少1610个先前映射的标签,并且通过本研究中使用的更严格的标准过滤掉了缺失的标签,与之前的数据集相比,删除了1649个fl - cdna。有45,025个未映射的标签不符合我们严格的标准(详见材料和方法)。
12种分布模式的差异表达基因
我们通过计算来定义差异表达基因P任何两个库之间的值使用先前报告的统计方法[28];该过程获得1751个(8.5%)和1216个(5.9%)显著差异表达基因P值分别< 0.05和< 0.01(表3)4).在总结总体表达谱的过程中,无论组织来源如何,我们发现了781、360和324个差异表达基因LYP9与PA64s(左vs。P),LYP9与93 - 11(左vs。N),LYP9相对于两个亲本品种(两个)在较不严格的阈值(P< 0.05)。有一个明显的偏差-具有父系样表达的基因(PLE;lvs。P)是具有母性表达(MLE;lvs。N).这种偏见表明LYP9拥有更多的差异表达基因PA64s比从93 - 11,无论它们是上调还是下调;换句话说,LYP9更类似于93 - 11比PA64s在它的整体基因表达中。
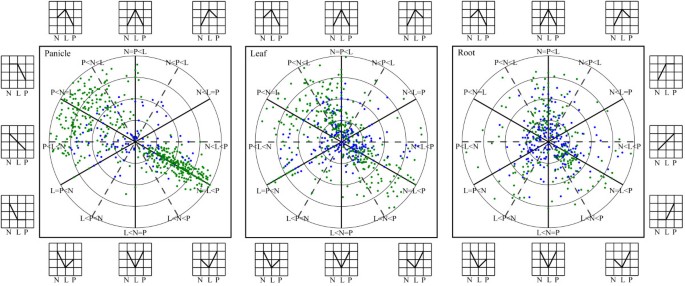
我们进一步研究了差异表达基因的谱图,将它们分类为12种不同的分布模式,根据不同的组织分别显示,并绘制了基因表达强度的折叠变化(小于16倍)P< 0.05和P< 0.01(图2).有686、568和413个基因在穗中差异表达(见附加文件)2)、叶子(参见附加文件)3.)和根(参见附加文件)4),在三和弦中P< 0.05。对应的数字分别是599、393、240P< 0.01。还列出了显示>16倍变化的基因和仅分配给pa64的基因(见附加文件)5).为了根据基因与亲本之间的关系清晰地描述基因分布,我们将12种基因分布模式划分为3种基本类型:超显性(上方4片)、低显性(下方4片)和中亲本(以水平线划分的4片)。
差异表达基因的表达模式及折叠变化。差异表达基因在穗、叶和根中93 - 11(N),PA64s(P)和它们的F1杂交体LYP9(L)所示。在每个切片中标记了12种不同的图案,并在三个面板周围显示了它们的图形指示器。绘制基因的半径表示log23个水稻品种高、低值之间的折线变化,夹角表示其之间的关系LYP9还有它的父母。差异表达基因的显著性区间为0.01 <P< 0.05和P< 0.01分别以蓝色和绿色表示。仅绘制显示<16倍变化的标签,因为超出折叠值的标签数量非常有限(在附加文件中列出)5).注:(1)各组水平线以上的5种模式所含基因在杂交中为上调基因(正杂种优势),(2)各组水平线以下的5种模式所含基因在杂交中为下调基因(负杂种优势),(3)各组水平线上有两种中亲本模式。
从总体分布上看,差异表达基因具有较高的多态性P值(P< 0.01),我们对样本进行了多次观察。首先,与其他两种组织相比,穗中的基因分布模式相当独特,也更有倾向性,因此,大多数下调的基因都非常像父系基因(或几乎与父系基因相同)93 - 11, N = L < P),且上调基因具有较强的分散性(不集中在N = L > P的实线上)。这种分散性表明,这些基因大部分大致类似于父本,但它们的表达水平接近于杂交(LYP9)或中间亲本。我们推测,这种明显的限制分布可能是由于以下一种或两种可能的偏差。一种偏见可能来自于母系品种特有的对温度敏感的雄性不育,PA64s在美国,生殖系相关基因可能通过表观遗传机制在其整体基因表达中受损。另一种可能的偏倚可能是亲本系等位基因之间的不亲和性造成的,这可能对大多数基因产生相当大的调控作用,例如种系组织中的DNA甲基化。其次,基因在叶和根中的分布有一定的相似性,尤其是在下调基因中,这些下调基因的折叠变化不像在穗中那么明显。然而,上调基因在两种组织中的分布却相当不同,其中叶片中的上调基因倾向于过显性表达,尽管有少数基因向中亲本传播。在根中,虽然与穗和叶相比,上调的基因数量要少得多(101个基因,表1)4),大多是过度支配型。最后,在总结12种模式基因分布的过程中,我们发现少数差异表达基因(25 ~ 45%)表现为加性表达(P > L > N和N > L > P;而大部分基因(380个(55%)、408个(72%)和309个(75%))在穗、叶和根中分别是非加性的。三种组织的非加性基因中,552个基因为过显性表达,394个基因为过显性表达。此外,115和32个基因的表达水平与父系相同(93 - 11)和母系(PA64s),分别;这些基因被归类为显性表达。
差异表达基因的功能分析
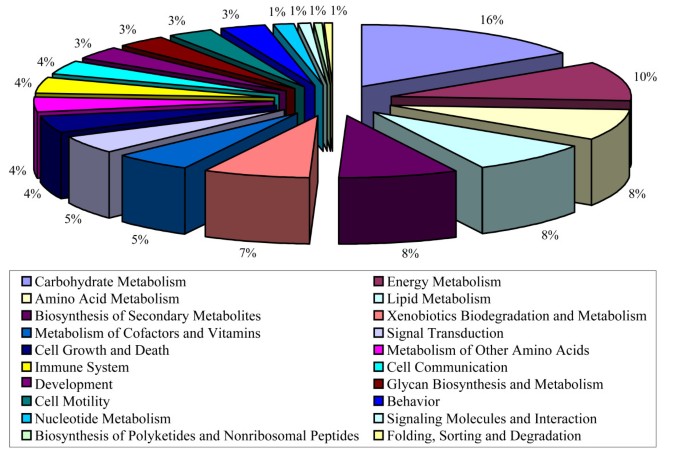
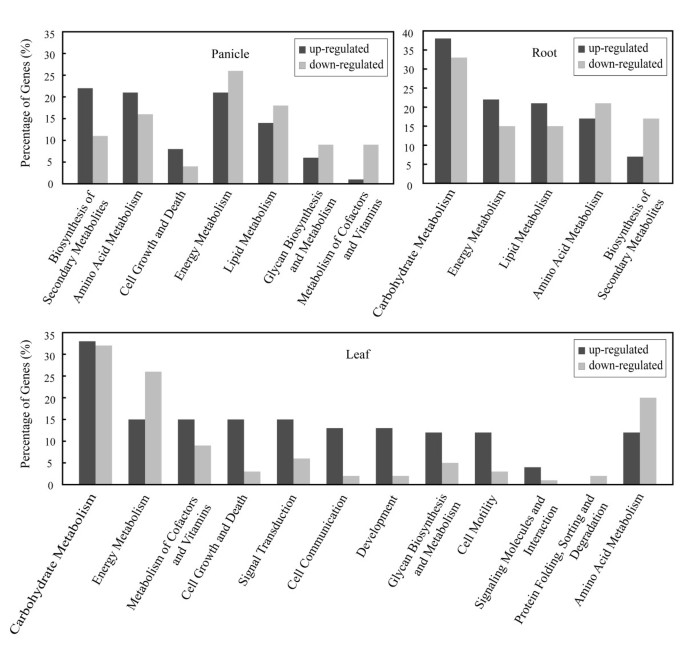
我们在两个通用数据库KEGG(京都基因与基因组百科全书)的基础上注释了217个(22.8%)和850个(89.3%)差异表达基因[29]及InterPro/Network [30.),分别。根据KEGG基因本体(KEGG Gene Ontology, KOG)分类方案,将基因进一步划分为20类3.);其中与碳水化合物代谢相关的基因最多(16%),其次是能量代谢(10%)和氨基酸代谢(8%)。例如,杂种中差异表达的基因大多与促进碳同化、能量代谢和次生代谢物的生物合成有关;这种效应不是由于整体基因分布的简单分布偏倚,因为在杂交中发现其他类别减少,例如叶片中的蛋白质分类/折叠/降解(图2)4).辅助因子和维生素在穗部的代谢也显著下调。
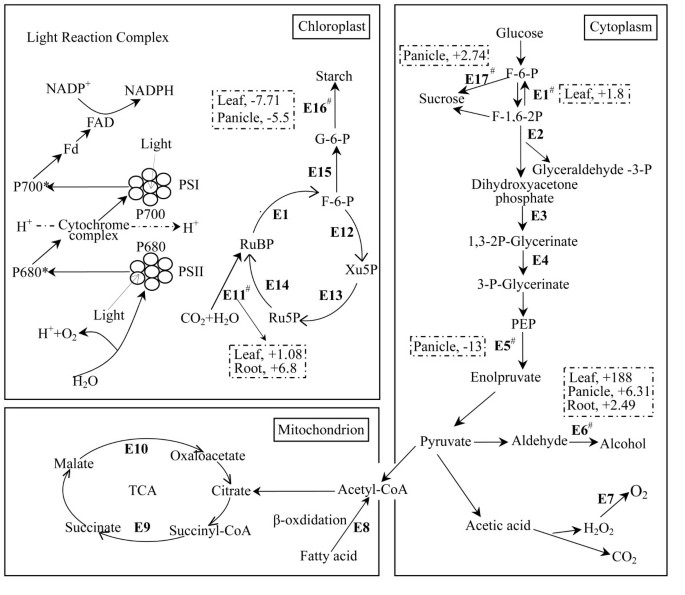
虽然与之前基于较少标签数量的结果进行总体比较得出了类似的结论,但我们认为我们目前的数据使我们能够进一步研究更多的途径和分子细节,这些在之前的分析中没有被充分利用。我们将碳代谢分为三个细胞区室:叶绿体、线粒体和细胞质(图2)5).叶绿体中参与光合作用的基因在叶片和根中均表达上调,而在穗中表达下调;这一趋势在总体分布中很容易观察到(图2)2).其中12个基因编码叶绿素a/b结合蛋白,17个是光系统I/II组分基因,以及介导CO初始反应的关键酶二磷酸核酮糖羧化酶2固定。详细的基因参与光反应被列出(见附加文件)6).我们还观察到线粒体和细胞质中涉及其他五个选定关键途径(糖酵解/糖异生,柠檬酸循环,厌氧呼吸,乙醇酸氧化和脂肪酸β氧化)的三个关键酶。第一种酶,酒精脱氢酶参与厌氧呼吸,是所有三种组织中上调最多的基因。第二种酶果糖-1,6-二磷酸酶参与糖异生,仅在叶片中上调。最后,催化磷酸烯醇丙酮酸形成丙酮酸和ATP(或碳水化合物分解)的丙酮酸激酶在叶和穗中均下调,但在根中不下调。此外,我们观察到过氧化氢酶,已知参与乙醇酸氧化途径(三种呼吸途径之一,是水稻更好地适应其水环境的独特途径),显着上调。此外,在蔗糖合成及其储存形式(淀粉)的途径上,我们发现了4个基因,分别编码β -磷酸葡萄糖糖糖化酶、1,4- α -葡聚糖分支酶、蔗糖磷酸合成酶和蔗糖合成酶,这些基因在叶片和穗中也有上调表达。这些酶被认为对超级杂交稻的高产量有贡献。
在三个主要细胞区室中参与选定的关键代谢途径的差异表达基因。基因参与光合作用、糖酵解/糖异生、柠檬酸循环(TCA循环)、厌氧呼吸、乙醇酸氧化和脂肪酸β-氧化途径。酶(#表示关键或限速酶)是:E1#的特性,6-bisphosphatase;E2,果糖二磷酸醛缩酶;E3,甘油醛3-磷酸脱氢酶;E4,磷酸甘油激酶;E5#,丙酮酸激酶;E6#,醇脱氢酶;E7,过氧化氢酶;E8,酰基辅酶a脱氢酶;E9,琥珀酰辅酶a连接酶;苹果酸脱氢酶;E11#二磷酸核酮糖羧化酶;E12汽油,转酮醇酶;E13,磷酸核酮糖- 3- epimase;E14灯头,phosphoribulokinase;E15, 1,4-葡聚糖支化酶;E16天#,蔗糖磷酸合酶;E17#,蔗糖合酶。光系统I反应中心亚基N,光系统I反应中心蛋白PsaF(亚基III), NADH:黄素氧化还原酶/NADH氧化酶和细胞色素b泛醇氧化酶。表示上(+)或下(-)调节标签的比率。轻反应配合物的详细信息在附加文件中列出6。请注意,在三个组织中,关键酶要么被上调,要么被下调;这种行为表明,混合动力车中存在积极而独特的规则。
还有许多其他功能注释基因被发现显著上调,包括快速碱化因子、蛋白酶抑制剂和MADS-box转录因子;根据数量性状位点(QTL)数据库(Gramene [QTL]),这些性状似乎都与光周期敏感型雄性不育、雄性育性恢复和花粉育性有关。31];参见附加文件7).其中,MADS-box基因(9311_Chr06_3092和9311_Chr01_4641)和快速碱化因子基因(9311_Chr12_1510)的表达量在父系中已经较高,但在杂交种中表达量高于亲本93 - 11而不是母系PA64s。这表明这些基因可能直接或间接地在花的形态发生和杂种的育性中起重要作用LYP9。
我们还发现了大量在之前的分析中不明显的下调基因,这主要是由于更多的映射标签和数据分析协议的微妙之处。这些表达抑制基因在三种组织中属于不同的功能范畴;它们大多参与穗内的能量代谢、脂质代谢、糖的生物合成和代谢,叶片内的氨基酸代谢和蛋白质加工,以及根内次生代谢物的生物合成(图2)4).在穗、叶和根中下调最多的基因分别是金属硫蛋白、肽酶M48和谷胱甘肽s转移酶。金属硫蛋白是一种富含半胱氨酸的蛋白质,可以与重金属结合,清除活性氧,保护植物免受氧化损伤。虽然在穗部表达下调最多,但在根部表达上调,对金属铁的吸收、过滤和浓缩,特别是对重金属铁的筛选起重要作用。肽酶M48是一个在蛋白质降解中起作用的蛋白家族。我们还发现了一些与蛋白质降解相关的下调基因,如泛素和泛素结合酶。谷胱甘肽s -转移酶是一种代谢外源有毒化合物的酶,利用谷胱甘肽进行解毒,用于植物的化学防御。我们推测,这两个上调和下调的基因代表了调控穗发育、快速生长、抗逆性和籽粒产量的基因的重要组成部分LYP9。显然,对这些差异表达基因的进一步验证和功能研究对于了解它们在杂种优势中的确切作用至关重要。
交叉引用SAGE数据与基于微阵列的结果
我们以有限的方式将我们的SAGE数据与基于微阵列的实验进行了比较,其中只有来自一个组织(叶片)的数据有资格进行合法比较,因为mRNA样本是从挤奶阶段的叶片中收获的,与我们用于SAGE实验的相同。微阵列数据通过使用定制设计的寡核苷酸阵列获得,该阵列包含60,727个寡核苷酸探针,代表了来自基因组组装的所有预测基因93 - 11(22]。从这一总数中,我们确定了在微阵列和SAGE数据中都发现的3355个信息性数据点,其中2312个(69%)在两种类型的实验中显示出一致的趋势(spearman系数为0.497;P< 0.0005)。我们发现,两个数据集中高表达基因之间的一致性趋势相关性相当好(spearman系数为0.743,P < 0.0005;数据未显示)。在这些基因中,根据SAGE数据,222个(39%)基因存在差异表达(P< 0.05)。我们在表中列出了23个倍数变化大于等于2的基因5。由于造成系统误差的原因是多方面的,包括采样时间、实验程序和数据归一化等,因此这些确认率与这两种实验之间的比较分析并无太大差异[13]。
讨论
标签到基因定位程序
SAGE和相关的基于测序的技术对于研究生物体内的基因表达是非常有效的,因为它们已经被应用于许多真核生物物种[17,19,32Marco Marra和他的同事最近用大量的实验数据讨论了它的优点和成功[12],尽管陷阱确实存在[13]。在我们之前的SAGE研究中,我们利用了现有的FL-cDNA序列[24]进行标签-基因定位[21],因为这些FL-cDNA序列最能代表水稻转录组,尽管数量相当有限。然而,在这个cDNA数据收集中没有发现很大比例(83%)的SAGE标签,已知这些标签没有覆盖水稻基因组的所有基因。为了克服这一限制,我们利用了一种新的标签-基因定位策略,该策略基于两个水稻基因组组合的新注释基因和其他转录序列(FL-cDNA, UniGene和ESTs)。这一过程显著提高了基因鉴定水平,增加了10268个标签,增加了68.85%的差异表达基因P值(P< 0.01),与之前的集合相比。
除了成功地将SAGE标签映射到基因组中的注释基因之外,还有几个重要的问题值得进一步讨论。首先,我们总是有标签被定位到模棱两可的位置,它们可能属于基因组序列中的多个位点(如基因家族和剪接变异体),特别是当SAGE标签的长度只有14 bp时。在我们的案例中有4014个(20%)这样的标签,我们将这些标签分配给基因组并使用它们进行功能分析。例如,尽管一个带有“AACAAGCTCA”序列的标签被分配到两个不同的位点(9311_Chr04_1718和9311_Chr05_1829),但这两个位点被两个不同的flcdna序列(AK0ah71547和AK061050)所证明,这使我们能够将它们鉴定为果糖-二磷酸醛dolase基因家族的成员。这两个基因在杂交种的根中被发现下调,它们参与糖酵解/糖异生途径。因此,绘制这些看似模糊的基因是至关重要的,特别是当它们在杂交中受到差异调节时。设计实验用位点特异性引物区分这些基因是可能的,因为大多数这些重复(或密切相关)的基因在其UTR和基因组序列上可能不相同,特别是当基因组序列很容易获得时。正如我们之前所报道的,水稻基因组有大量的重复基因[23它们中的一些实际上可能拥有杂交活力的关键信息。
第二点与部分(通常超过40%)实验标签未分配给基因的事实有关,因此我们需要找出可能的原因。将未分配的标签与基于预测的虚拟标签进行比较时国民在细胞核和细胞器(线粒体和叶绿体)基因组序列中,我们发现47,867个标签中有2,500个(5%)在基因组序列组装中缺失93 - 11342个标签(0.6%)来自线粒体(491kb)或叶绿体基因组(134kb)。这些未分配的标签很可能是由于测序错误、被内含子打断的序列、未组装的序列(包括序列间隙中的序列)和细胞器特异性序列。此外,我们在没有基于转录的证据的情况下,从技术上实现了预测基因的人工300 bp utr,并且仅从虚拟转录本中提取了3' most (canonical position)标签。这一过程当然不可能包括所有的UTR长度变异,主要是由于缺乏准确测定植物基因组中3' UTR长度的典型聚腺苷化信号[33]。为了估计这一过程的结果,我们将剩余的未分配标签总数与一个累积的虚拟标签数据集进行了比较,该数据集是通过在100-bp的间隔内(从100到500 bp)改变人工UTR长度构建的,从而进一步分配了3,119个(6.5%)额外的标签。然而,这些标签被认为是不可靠的,不包括在本分析中。然而,utr衍生的异常似乎以与序列异常相似的方式导致了受损的标签分配。其他导致未分配标签的明显因素,如实验伪影(不完全的酶消化和结扎,以及低效的克隆程序),不在这里详细讨论。
多种表达模式的差异表达基因
多年来,杂交品种与其亲本品种之间的差异基因表达被假设归因于杂种优势[5,34]。在将差异表达基因按常规划分为12种模式后,我们发现在水稻杂交种中只有25% ~ 45%或少数基因是加性表达的;这一结果与在杂交玉米中报道的类似研究结果相矛盾,在杂交玉米中发现加性表达基因是主要趋势,占77.7% [35]。造成这种差异的原因可能很复杂,因为它可能与操作授粉策略和表观遗传调控的差异有关。Meyer等人(2004)已经证明了其他授粉方法(手授粉vs.手授粉)。自花授粉)对拟南芥种子大小和幼苗早期生长速率有显著影响。与自交核相比,杂交核的基因表达模式在质量和数量上都发生了明显的变化[36],这主要是由于玉米自交系的顺式转录变异导致了F1杂交种的加性表达模式[37]。对于可能的表观遗传机制的参与,我们提到了两个物种之间转座子密度的差异,因为玉米基因组受到活跃重复序列的严重轰炸,我们推测在玉米的基因调控中可能使用了更有力的甲基化策略。在非加性表达基因中,过显性和欠显性基因都相当丰富,这在一定程度上支持了水稻杂种优势的过显性假说[34]。
在所有的差异表达基因中,我们发现了高达70%的差异表达基因(P< 0.01)表现出父本样表达(PLE)谱,尤其是在穗上,这至少部分可归因于两种合理的机制——分子印迹和母本等位基因的缺陷表达——正如在花粉成熟阶段收获的穗上经常观察到的那样,此时母系的温敏雄性不育(PA64s)可能有关[38]。例如,两个与花粉育性相关的MADS-box转录因子在杂交植物中一直被观察到上调,但它们在雄性不育植物中不表达[39,40]。快速碱化因子,一种被认为与细胞核不育和发育有关的多肽激素[41],我们通过QTL分析发现其表达上调,位于光敏性和基因性雄性不育性状中。虽然我们还不能对这些基因的精确作用描绘出合理的功能场景,但这些发现无疑为未来的分子研究提供了有用的线索。
差异表达基因的可能调控机制
植物的差异基因表达主要受转录水平的顺式和反式调控以及表观遗传和转录后调控两种机制的调控[6]。例如,CpG或CNG岛的差异甲基化[9,42]和依赖等位基因的基因调控机制[43已经在水稻和玉米的杂交和亲本之间得到了证明。然而,顺式调控元件之间的差异很难研究,而根据基因表达数据更容易识别反式调控因子。我们确实发现了超过48个转录因子,标注为差异表达基因,包括MADS-box基因,TFIIE, bZIP和Jumonji;这些基因被发现参与了陆地植物发育和分化的各个方面。一些MADS-box基因在花组织中起着花形态发生的“分子建筑师”作用。TFIIE是RNA聚合酶II转录机制的重要组成部分[44],在转录的两个不同但连续的步骤中发挥重要作用:起始前复合物形成-激活(开放复合物形成)和从起始到延伸的过渡[45]。尽管这些转录因子(多功能或多基因家族成员)对杂交活力的可能贡献可能不容易证明,但它们的存在和调控表达是深入分子和遗传学研究的初步线索。
越来越多的研究报道,重复基因的功能分化伴随着基因表达的变化,但这一过程背后的进化机制尚不清楚。有报道称,酵母中有7%的重复基因对共表达[46],我们知道基因和染色体片段复制广泛存在于水稻基因组中,包括古老的全基因组复制,最近的片段复制,以及覆盖65.7%基因组的大量正在进行的单个基因复制[23]。我们发现698个歧义标记中有7个被定位到重复的基因对上,我们怀疑高同源性的重复可能会影响基因的表达,包括在杂交后沉默或上调或下调其中一个重复基因[47]。在研究区分不同等位基因的可能的分子分析方法时,我们发现实际上可以设计等位基因特异性引物来检测重复对的表达水平。
结论
我们将转录序列信息与水稻基因组注释相结合,改进了标签-基因定位策略,获得了超过10,000个新的标签,以便更全面地了解与水稻杂种优势相关的基因。这些不同基因型间的杂种优势表达基因为探索杂种优势的分子机制提供了新的途径,包括基因表达模式的变化。
方法
PCUE数据库
我们建立了水稻(栽培稻),这些基因组资源包含(1)改进的全基因组散弹枪序列组合93 - 11[GenBank: AAAA02000000]及PA64s以及他们的注解[48[2] 19,079个非冗余fl - cdna (nr- fl - cdna);(23源自KOME [49],(3)来自NCBI的51,336条UniGene (UniGene Build #59)和1,183,931条ESTs [50]。
我们利用BLAT将收集到的转录本序列与两个基因组序列进行比对[51]获取标签注释的数据集。为对齐转录本设置的阈值参数为(1)至少90%与其基因组序列相同,(2)覆盖≥90%的转录本序列。当一个转录本对基因组序列有多个命中时,最长的共识被选择为最佳对齐(真实)位点。我们进一步选择了跨越预测基因3'端但不延伸到下一个重叠序列≥100bp的序列。结果,我们预测的基因被分成两组:由一个或多个转录本支持,没有支持数据。
评估数据集
为了评估标签-基因定位方法的准确性,我们构建了一个包含2480个FL-cDNA序列的测试数据集,该数据集满足所有五个标准:(1)ORF长度> 300 bp,(2)具有多聚(a)信号(AATAAA/ATTAAA)或多聚(a)尾部(最少5个a) [15[3]与拟南芥具有同源性(基于50%蛋白质序列相似性或100个残基),(4)具有独特的CATG标签和实验数据,(5)与独特的预测基因和相应的UniGenes或ESTs可对齐。我们进一步将该数据集分为三类:UniGene、EST和预测基因。在Unigene和EST类别中,我们有12个子集。其中有多(A)信号序列(Uni-S和EST-S)、有多(A)尾部序列(Uni-A和EST-A)、有多(A)信号和尾部序列(Uni-B和EST-B)、无多(A)信号和尾部序列(Uni-N和EST-N)的序列有8个。其他四个子集包含最长和最好的转录本,这些转录本可以通过UniGenes或ESTs (Unibest或ESTbest)进行最佳验证。为了了解3’-UTR的长度,我们使用19079个非冗余FL-cDNA来确定长度分布,发现95%的基因UTR长度短于1280 bp,平均长度为422 bp,中位数为295 bp。因此,我们添加了五种不同的长度(100-,200-,300-,400-和500-bp)来构建预测基因的虚拟utr。最后,我们通过从最后(3'-most)的直接下游序列中提取一个10-bp的标签,从上述每个子集中构建虚拟标签。国民III (CATG)站点。我们评估了与测试集匹配的虚拟标签的成功率。
虚拟标签和标签到基因的定位
由于预测的基因没有UTR,我们从两个基因组组合中提取连续的外显子,形成基因模型,并在其中添加基于已知转录本信息的UTR序列或长度为300 bp的人工UTR序列。我们获得了四组标签数据,包括基于cDNA、Unimax、ESTmax和预测基因(P-300)的标签数据。我们从我们的数据中映射了68,462个唯一的经验标签[21]筛选克隆连接子、载体、简单重复四组虚拟标签。我们从进一步的处理中排除了47,867个标签,并从我们的分析方案中总结了他们的结果(见附加文件)8).这些标签被认为是未定位标签,尽管其中45,025个标签实际上被定位到核基因组,但在外显子和UTR序列的正确位置范围出乎意料。他们中的大多数被认为是片段化的mrna,在库建设过程中被共同处理。
我们基于InterPro/Network和KEGG对所有SAGE标签进行了蛋白质家族、结构域和功能的注释。我们首先从家族类型类别中选择得分最高的主要(基于序列相似性的)注释,然后是领域类型和其他类型。如果基因没有主要注释,那么我们使用基于网络的注释[52]。P根据audi - claverie(或AC)统计数据计算图书馆之间的拷贝数之间的值[28]使用IDEG6软件[53,54]。差异表达基因的意义用P值小于0.05或0.01。分别按比值= L/[(P+N)/2](≥2)和[(P+N)/2]/L(<2)计算上调和下调基因的比值。
微阵列和QTL数据
我们使用了在我们实验室生成的乳白期叶片组织的微阵列数据。该微阵列包含60,727个寡核苷酸探针,代表了从基因组序列中预测的所有基因93 - 11(22]。我们将寡核苷酸物理映射到最新版本的基因组组装[48的阈值,即每个寡核苷酸必须与一个具有90%或更高序列一致性的独特基因匹配。我们还使用了Gramene TIGR4基因组物理位置的水稻QTL数据[31]并将差异表达基因定位到9个QTL类别。
缩写
- 比如:
-
Paternal-like表达式
- 大中型企业:
-
Maternal-like表达式
- 圣人:
-
基因表达序列分析
- QTL:
-
数量性状位点
- nr-FL-cDNAs:
-
非冗余全长cdna
参考文献
- 1.
Budak H:对杂种优势的理解。科学工程学报,2002,5(2):69-75。
- 2.
肖军,李军,袁林,Tanksley SD:分子标记QTL分析表明,显性是水稻杂种优势的主要遗传基础。遗传学报,1995,14(4):745-754。
- 3.
于世彬,李建新,徐国光,谭玉峰,高玉军,李晓华,张启峰,Maroof MAS:水稻优良杂种杂种上位性作为杂种优势遗传基础的重要性。美国国家科学促进会。[j] .中国科学:地球科学。10.1073 / pnas.94.17.9226。
- 4.
Birchler JA, Auger DL, Riddle NC:寻找杂种优势的分子基础。植物学报,2003,15(10):2236-2239。10.1105 / tpc.151030。
- 5.
孙强,吴丽,倪志,孟峰,王铮,林志:小麦双列杂交与亲本自交系叶片差异基因表达模式与杂种优势相关。植物科学(牛津)。中国生物医学工程学报,2004,31(3):651-657。
- 6.
姚毅,倪震,张勇,陈勇,丁勇,韩震,刘震,孙强:利用pcr技术鉴定小麦杂交种与亲本近交系叶片和根系差异表达基因。植物化学学报,2005,31(3):367- 378。10.1007 / s11103 - 005 - 5102 - x。
- 7.
吴丽敏,倪志峰,孟福瑞,林芝,孙启祥:小麦杂交亲本叶片差异表达cdna的克隆与鉴定。中国生物医学工程学报,2003,29(3):391 - 391。10.1007 / s00438 - 003 - 0919 - y。
- 8.
姚勇,倪忠,杜军,王鑫,吴华,孙强:小麦核糖体蛋白15个基因的分离与鉴定。植物科学(牛津)。中国生物医学工程学报,2006(3):579-586。
- 9.
熊丽珍,徐国强,张强:利用甲基化敏感扩增多态性技术检测水稻优质杂交种及其亲本胞嘧啶甲基化模式。生物医学工程学报,1999,26(3):439-446。10.1007 / s004380050986。
- 10.
倪震,孙强,刘震,吴磊,王旭:利用mRNA的差异显示技术鉴定小麦幼苗叶片中一个新的rna结合蛋白的杂交特异性表达基因。中华生物医学工程学报,2002,26(6):934-938。10.1007 / PL00008693。
- 11.
黄燕,张丽,张军,袁东,徐超,李旭,周东,王松,张强:基于9198个est基因芯片分析的水稻杂种优势与基因表达多态性。植物学报,2006,32(4):579-591。10.1007 / s11103 - 006 - 9040 - z。
- 12.
Khattra J, Delaney AD, Zhao Y, Siddiqui A, Asano J, McDonald H, Pandoh P, dhalln, Prabhu AL, Ma K, Lee S, Ally A, Tam A, Sa D, Rogers S, Charest D, Stott J, Zuyderduyn S, Varhol R, Eaves C, Jones S, Holt R, Hirst M, Hoodless PA, Marra Ma:微解剖组织,流动分类细胞和细胞系SAGE文库的大型生产。中国生物医学工程学报,2007,17(1):108-116。10.1101 / gr.5488207。
- 13.
王思敏:理解SAGE数据。趋势研究,2007,23(1):42-50。10.1016 / j.tig.2006.11.001。
- 14.
陈健,孙敏,李松,周刚,Rowley JD,王思敏:基于SAGE标签的人类基因组新转录物和新基因鉴定。美国国家科学促进会。中国生物医学工程学报,2009,35(6):1257 - 1262。10.1073 / pnas.192436499。
- 15.
Boon K, Osorio EC, Greenhut SF, Schaefer CF, Shoemaker J, Polyak K, Morin PJ, Buetow KH, Strausberg RL, De Souza SJ, Riggins GJ:正常和恶性基因表达的解剖。美国国家科学促进会。中国生物医学工程学报,2009,29(6):1128 - 1128。10.1073 / pnas.152324199。
- 16.
李建勇,李大东:利用基因表达序列分析技术揭示冷胁迫下拟南芥花粉基因表达的变化。植物生理学报,2013,32(2):517-529。10.1104 / pp.103.020511。
- 17.
Fizames C, Munos S, Cazettes C, Nacry P, Boucherez J, Gaymard F, Piquemal D, Delorme V, Commes T, Doumas P, Cooke R, Marti J, Sentenac H, Gojon A:拟南芥根系转录组基因表达序列分析。利用基因组序列进行基因鉴定。植物生理学报,2004,34(1):67-80。10.1104 / pp.103.030536。
- 18.
Matsumura H, Nirasawa S, Terauchi R:水稻(Oryza sativa L.)幼苗基因表达序列分析技术进展。植物学报,1999,20(6):719-726。10.1046 / j.1365 - 313 x.1999.00640.x。
- 19.
gibings JG, Cook BP, Oufault MR, Madden SL, Khurie S, Tumbull CJ, Dunwell JM:基于SAGE技术的水稻叶片和种子转录本分析。植物学报,2003,(1):271-285。10.1046 / j.1467-7652.2003.00026.x。
- 20.
王强,张庆东,蒋广民,卢春明,匡彦泰,吴森,李春青,焦德明:两种超高产杂交稻的光合特性。植物学报,2000,42(12):1285-1288。
- 21.
鲍健,李松,陈晨,张翔,张勇,刘森,克拉克涛,王健,曹敏,杨辉,王思敏,于杰:杂交水稻品系LYP9及其亲本基因表达序列分析。植物生理学报,2009,38(3):1216-1231。10.1104 / pp.105.060988。
- 22.
籼稻)。科学通报,2002,29(6):79-92。10.1126 / science.1068037。
- 23.
王于J, J,林W,李,李H,周J,倪P,董W,胡年代,曾C,张J,张Y,李R,徐Z,李,李X,郑H, Cong L林L,阴J,耿J,李G, J,刘J, Lv H,李J,王J,邓Y, L, X, et al:栽培稻的基因:重复的历史。中国生物医学工程学报,2005,30 (2):888 - 888 /journal.pbio.0030038。
- 24.
Kikuchi S, Satoh K, Nagata T, Kawagashira N, Doi K, Kishimoto N, Yazaki J, Ishikawa M, Yamada H, Ooka H, Hotta I, Kojima K, Namiki T, Ohneda E, Yahagi W, Suzuki K, Li CJ, Ohtsuki K, Shishiki T, Otomo Y, Murakami K, Iida Y, Sugano S, Fujimura T, Suzuki Y, Tsunoda Y, Kurosaki T, Kodama T, Masuda H, Kobayashi M,等:粳稻28000多个cDNA克隆的收集、定位和注释。科学通报,2003,31(6):376-379。10.1126 / science.1081288。
- 25.
Wu J, Maehara T, Shimokawa T, Yamamoto S, Harada C, Takazaki Y, Ono N, Mukai Y, Koike K, Yazaki J, Fujii F, Shomura A, Ando T, Kono I, Waki K, Yamamoto K, Yano M, Matsumoto T, Sasaki T:包含6591个表达序列标签位点的水稻转录本综合地图。植物学报,2002,14(3):525-535。10.1105 / tpc.010274。
- 26.
唐健,夏辉,李东,曹明,陶勇,童伟,张旭,胡顺,王健,于健,杨辉,朱丽:水稻幼穗和营养器官基因表达谱及已知基因功能鉴定穗特异性基因。中国生物医学工程学报,2009,34(5):467-476。10.1007 / s00438 - 005 - 0043 - 2。
- 27.
基因表达综合。(http://www.ncbi.nlm.nih.gov/geo/]
- 28.
李建平,李建平。基因表达谱的研究进展。中国生物医学工程学报,1997,7(10):986-995。
- 29.
京都基因与基因组百科全书。(http://www.genome.jp/kegg/]
- 30.
InterPro。(http://www.ebi.ac.uk/interpro/]
- 31.
Gramene。(http://www.gramene.org]
- 32.
张建军,张建军,张建军,张建军,等。中国生物医学工程学报,2003,31(6):393 - 393。10.1101 / gr.873003。
- 33.
张建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,等。美国国家科学促进会。中国生物医学工程学报,1994,18(4):555 - 560。10.1073 / pnas.96.24.14055。
- 34.
植物杂种优势的分子研究。植物生理学报,1999,19(4):369 - 369。10.1111 / j.1399-3054.1995.tb05324.x。
- 35.
Swanson-Wagner RA, Jia Y, DeCook R, Borsuk LA, Nettleton D, Schnable PS:在玉米F1杂交及其近交系亲本基因表达的全球比较中观察到所有可能的基因作用模式。美国国家科学促进会。中国生物医学工程学报,2009,35(6):559 - 561。10.1073 / pnas.0510430103。
- 36.
孟峰,倪志,吴玲,孙强:玉米种子发育早期异交与自交籽粒基因表达差异。植物科学,2005,32(1):23-28。10.1016 / j.plantsci.2004.07.011。
- 37.
Stupar RM, Springer NM:玉米自交系B73和Mo17的顺式转录变异导致F1杂交种的加性表达模式。遗传工程学报,2006,33(4):394 - 394。10.1534 / genetics.106.060699。
- 38.
宋丽琴,傅道德,涂建新,马春珍,杨国光:甘蓝型油菜显性雄性不育基因多等位基因遗传的分子验证[j] .华北农学报,2006,31(1):55-62。10.1007 / s00122 - 006 - 0271 - 9。
- 39.
Nagasawa N, Miyoshi M, Sano Y, Satoh H, Hirano H, Sakai H, Nagato Y: SUPERWOMAN1和下垂叶基因控制水稻花器官的特性。发展,2003,130(4):705-718。10.1242 / dev.00294。
- 40.
姜洪柱,全建新,李松,安刚:水稻B类和C类花器官鉴定基因的鉴定。植物学报,1998,38(6):1021-1029。10.1023 /: 1006051911291。
- 41.
杨树细胞培养中快速碱化因子的研究。叶片和茉莉酸甲酯处理细胞中多肽的分离、cDNA克隆及差异表达。植物生理学报,2003,31(2):814-823。10.1104 / pp.014597。
- 42.
戴勇,倪志,戴军,赵涛,孙强:小麦DNA甲基转移酶基因的分离与表达分析。生物化学与生物物理学报,2005,29(2):118-125。
- 43.
Hollick JB, Patterson GI, Asmundsson IM, Chandler VL:参数化改变玉米基因座的调控。遗传学报,2009,35(4):557 - 557。
- 44.
Maxon ME, Goodrich JA, Tjian R:转录因子IIE优先结合RNA聚合酶IIa和募集TFIIH:启动子清除模型。基因工程学报,1998,8(5):515-524。10.1101 / gad.8.5.515。
- 45.
Okamoto T, Yamamoto S, Watanabe Y, Ohta T, Hanaoka F, Roeder RG, Ohkuma Y:通过TFIIE β亚基的结构-功能研究分析TFIIE在转录调控中的作用。中国生物医学工程学报,2009,29(3):444 - 444。10.1074 / jbc.273.31.19866。
- 46.
张志,顾军,顾旭:酵母基因复制后表达差异的大小可以用调控基序进化来解释?趋势与进展,2004,20(9):403-407。10.1016 / j.tig.2004.07.006。
- 47.
李建平,李建平。植物多倍体基因表达的研究进展。趋势与进展,2005,21(10):539-543。10.1016 / j.tig.2005.07.009。
- 48.
- 49.
知识为基础的水稻分子生物学百科全书。(http://cdna01.dna.affrc.go.jp/cDNA/]
- 50.
国家生物技术信息中心。(http://www.ncbi.nih.gov/]
- 51.
BLAT -类似blast的对齐工具。中国生物医学工程学报,2002,31(4):656-664。10.1101 / gr.229202。2002年3月以前在线发表的文章。
- 52.
杨建军,杨建军,李建军,等。蛋白质相互作用网络的功能分析。生物信息学,2005,21(15):3217-3226。10.1093 /生物信息学/ bti514。
- 53.
- 54.
王晓东,王晓东,王晓东,等。基于多标签采样的差异表达基因检测。植物生理学杂志,2001,10(19):2133-2141。10.1093 /物流/ 10.19.2133。
致谢
我们非常感谢我们的微阵列团队成员提供的叶片数据。田向军和陈卫华对手稿的批判性阅读和许多建设性的讨论。本工作得到了中国科学院(KSCX1-SW-03)和科技部(2005AA235110)对JY的资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
提交人宣称,不存在相互竞争的利益。
作者的贡献
SHS和HZQ构思并进行了研究设计,进行了生物信息学分析,解释了分析结果并撰写了稿件。CC、SNH、JY参与了研究设计,并帮助修改和编辑了稿件。所有作者都阅读并批准了最终的手稿。
宋淑慧、曲红柱对这项工作也有同样的贡献。
电子补充资料
12870 _2007_190_moesm1_esm.tiff
附加文件1:基于已知的5'-UTR (A)和3'-UTR (B)的fl -cDNA的UTR大小分布。利用KOME数据库中已知的全长cDNA序列,我们绘制了UTR的大小分布,以确定人工UTR长度。(tiff 126kb)
12870 _2007_190_moesm2_esm.xls
附加文件2:穗部差异表达基因的详细信息。我们将穗部差异表达基因的表达信息、功能注释和标签定位信息整合到excel表格中。(xls 208kb)
12870 _2007_190_moesm3_esm.xls
附加文件3:叶片中差异表达基因的细节。我们将叶片差异表达基因的表达信息、功能注释和标签定位信息整合到excel表格中。(xl178kb)
12870 _2007_190_moesm4_esm.xls
附加文件4:根中差异表达基因的细节。我们在excel表格中整合了根差异表达基因的表达信息、功能注释和标签映射信息。(xl134kb)
12870 _2007_190_moesm6_esm.xls
附加文件6:参与光反应的基因。我们分类并列出了参与光反应的基因。(xl32kb)
12870 _2007_190_moesm7_esm.xls
附加文件7:穗部不育性和育性相关的差异表达基因。通过与Gramene QTL数据库的比较,列出了穗部不育性和育性相关的差异表达基因。(xl116kb)
12870 _2007_190_moesm8_esm.xls
附加文件8:本次分析中未映射的SAGE标签的潜在来源的统计结果。SAGE标签的潜在起源的一个例子是通过直接将标签映射到基因组序列中得到的。(xl62kb)
权利和权限
开放获取本文由BioMed Central Ltd.授权发表。这是一篇开放获取的文章,在知识共享署名许可(https://creativecommons.org/licenses/by/2.0),允许在任何媒体上不受限制地使用、分发和复制,前提是正确引用原创作品。
关于本文
引用本文
宋绍林,曲海,陈志强。et al。杂交水稻优良品种(Oryza sativa, L)及其亲本系基于SAGE数据。BMC Plant Biol7,49(2007)。https://doi.org/10.1186/1471-2229-7-49
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-7-49
关键字
- 重复基因对
- 数量性状位点数据
- 光周期敏感基因雄性不育
- 快速碱化因子
- 数量性状位点类别