摘要
背景
选择性剪接(Alternative splicing, AS)通过在特定情况下选择性地使用同一基因的不同外显子组合,对蛋白质多样性有显著贡献。AS的一种特殊类型是选择性第一外显子(AFEs)的使用,它的影响远远超出了蛋白质功能的微调。例如,AFEs可以改变蛋白质的n端,从而引导它们进入不同的细胞区。当备选第一外显子距离较远时,它们通常与备选启动子相关联,从而赋予了一个额外的基因表达调控水平。然而,很少有研究对AFEs的模式进行了研究,这些分析主要集中在哺乳动物的基因组上。近年来的研究表明AFEs存在于水稻基因组中,并以组织特异性的方式进行调控。我们目前对植物中的AFEs的认识仍然有限,包括它们的调控、对蛋白质多样性的贡献和进化保护等重要问题。
结果
我们系统地在水稻和水稻中鉴定了1378和645个含afe的簇拟南芥,分别。从我们的数据集中,我们根据它们的基因组组织确定了两种类型的AFEs。在I型AFEs基因中,第一外显子是相互排斥的,而大多数下游外显子是在其他转录本中共享的。相反,在II型AFEs基因中,一个基因结构的第一个外显子是另一个基因结构的内部外显子。功能分析表明,约50% ~19%的AFEs拟南芥在II型AFEs中,约5%的功能改变涉及两种基因组中蛋白质结构域的增加/缺失。表达分析表明,20~66%的水稻AFE簇是组织和/或发育特异性转录的,这与前人的观察结果一致;然而,更小的比例拟南芥这表明水稻和水稻AFEs的调控机制不同拟南芥。统计分析AFE簇的一些特征,如剪接位点强度和二级结构的形成,进一步揭示了两种植物的差异。对含afe基因对的同源检索只检测到19对保守的基因对拟南芥仅占含有afe集群的百分之几。
结论
我们对水稻和水稻中含afe基因的分析拟南芥表明AFEs具有多种功能,从调节基因表达到产生蛋白质多样性。通过对AFE簇的比较,发现两种植物的AFE簇具有不同的特征,这表明AFEs可能是在水稻(模式单子稻)分离后独立进化的拟南芥(一个模型双字子)。
背景
选择性剪接(Alternative splicing, AS)是一种重要的机制,它通过在特定情况下在不同的组织或细胞中选择性地使用同一基因的不同外显子集,对蛋白质的多样性有很大贡献[1- - - - - -3.].它已被证明存在于几乎所有的后生动物中,估计涉及30-70%的人类基因[4,5].然而,目前鉴定的AS变体偏向于包括编码序列(CDSs)在内的备选外显子[6].事实上,许多AS异构体使用可选第一外显子(AFEs)来调节其表达并产生蛋白质多样性。AFE是基因的一个剪接异构体的第一个外显子,但要么位于由同一基因产生的其他异构体的对应AFE的下游,要么完全不存在于其他异构体中。据报道,这种现象也导致了基因表达的复杂性[6,7].
迄今为止,对AFEs的研究主要集中在哺乳动物基因组上,特别是小鼠和人类。据报道,在RIKEN数据库中的全长基因中,约9%的小鼠含有AFEs [8],超过18%的人体内含有AFEs [9].AFEs可以通过替代启动子的使用产生。一些AFEs仅改变5'-非翻译区(5'-UTR),以调控翻译效率或转录本运输出细胞核的效率或目的地。在这种情况下,共享的下游外显子包含翻译起始密码子(ATGs),因此具有相同的开放阅读框(orf),并产生相同的蛋白质[6,10- - - - - -12].在其他情况下,AFEs包含替代转录起始位点(ATGs),这可能导致n端不同的蛋白质变体[2,13,14或在新的蛋白质中[15,16].
到目前为止,对植物中的AFEs进行分析的研究很少。例如,SYN1 in拟南芥已显示产生两种具有不同可选第一外显子的异构体[17].最近,一项对水稻AFEs的大规模研究发现了46个可能含有AFEs的簇,并表明它们参与了组织特异性转录[14].但是我们对植物中的AFEs的了解仍然有限。在这里,我们用系统的方法分析了它们对蛋白质多样性的贡献和它们在水稻(一种模式单子稻)和水稻之间的进化保护拟南芥(一个模型双字子)。
方法
植物基因组AFEs的系统检测
为了编制AFE数据集,我们下载了下列水稻的数据集(栽培稻lssp。粳稻),拟南芥从公共数据库:全长cdna,表达序列标签(est),参考序列(NCBI refseq)和信使rna(表1).使用GMAP程序确定每个cDNA序列的基因组位置和精确的基因结构[18].我们排除了与基因组序列相似性较低的序列(内参基因和全长cdna的一致性<95%,覆盖率<90%;<90%的一致性和<90%的ESTs覆盖),没有映射到一个独特的基因组区域,或来自细胞器(线粒体和叶绿体)。所有信息被加载到MySQL数据库中进行进一步分析。
我们首先将全长cdna和内参基因分组到基因组上的簇中,如果它们映射到相同的基因组区域,定位在相同的链上,并有重叠的序列。在每个簇中,成员根据他们的基因结构进一步分组。然后将ESTs添加到现有的集群中。根据基因组上第一个外显子的位置,EST要么作为现有基因结构的成员添加,要么作为集群中的新基因结构添加。那些不能在一个聚类中组成一个独特基因结构的ESTs被丢弃。添加ESTs后,我们计算了每个簇中每个基因结构的ESTs数量。为了得到可靠的结果,我们放弃了只有一个EST的基因结构。
由于我们的数据集中只有全长cdna才能保证转录起始位点(tss)和第一外显子的可靠性,我们在包含全长cdna且至少有两种不同基因结构的簇中寻找AFEs。我们将一个簇的第一个外显子定义为包含全长cdna的基因结构中最多的第一个外显子。然后将同一簇中的其他基因结构与该第一个外显子进行比较,以确定可能的AFEs。
在每个含有afe的基因簇中,我们通过计算其支持ESTs的数量来确定备选第一外显子的主要和次要类型。如果一个第一外显子类型比集群中的其他第一外显子具有更多的支持性ESTs,则该外显子类型被标记为“主要”类型;否则,它被标记为“次要的”。
AFEs的统计分析
根据AFEs的排列位置,我们确定了AFE簇在水稻和水稻中的染色体分布拟南芥。
为了确定调控AFEs中剪接位点选择的可能因素,如剪接位点强度、剪接结周围的共同基序和剪接位点周围的二级RNA结构形成,我们对水稻和水稻中的AFEs进行了以下统计分析拟南芥。首先,我们检查了交替剪接的第一外显子的剪接位点质量。通过使用GMAP中的外显子注释,我们以每个具有足够侧翼序列的供体(5')剪接位点为中心提取了一个500碱基对窗口,并将这些数据作为GeneSplicer [19]用于拼接位置预测。
其次,我们分析了AFEs是否倾向于在剪接位点周围形成二级结构,这可能潜在地阻碍剪接位点信号的正确识别,从而可能导致相应的外显子/内含子的跳过。我们使用维也纳RNA包的RNAfold程序[20.]来预测以每个拼接位点为中心的100基对窗口的折叠。计算每个输入序列的最小折叠能量(MFE,也称为最优折叠能量,OFE)。MFE分数越低,说明输入序列更容易形成次生结构。
第三,我们使用了模因[21,以寻找所有或部分外显子和相邻内含子序列共享的可能的公共基序。
含afe聚类的注释和功能分类
为了注释含有afe的聚类,我们将每个聚类中的内参基因或最长的全长cDNA(如果没有可用的参考序列)与Uniprot数据库中的蛋白质序列进行比较[18使用基于blast的工具。GO(基因本体)术语根据从GeneOntology Consortium网站下载的Uniprot2GO关联分配[22].GO注释是使用基于web的工具WEGO绘制的[23].利用以下方程计算超几何分布,评估含afe集群中各氧化石墨烯类富集或消耗的统计显著性:
在哪里米=生物体中按氧化石墨烯分类的基因总数,K=按特定GO类别分类的基因数量,n=按GO分类的含afe集群总数,x=根据特定GO类别分类的包含afe的集群数量,和p=一种氧化石墨烯类物质显著增加或减少的概率。
AFEs在水稻和水稻中的组织特异性表达拟南芥
为了可靠地检测某些AFE亚型的组织特异性,我们采用了Qiang Xu提出的策略等。[5,即“组织特异性评分”。为此,用组织特异性评分来衡量组织特异性TS以及两个鲁棒性值rTS和rTS~(详见参考文献。[5])。高置信度(HC)组织特异性定义为TS> 50岁rTS > 0.9和rTS~>0.9,低信度(LC)定义为TS> 0,rTS > 0.5和rTS 0.5 ~ >。
含afes的同源基因的跨基因组比较
水稻与植物之间的同源关系拟南芥通过Inparanoid [24参数设置为默认值,并启用Bootstrap选项。输出是用PERL脚本解析的。只有Bootstrap评分为100%的基因被认为是同源的。
包含afe的集群的功能
我们使用工具GetORF在EMBOSS软件包中[25为每个包含afe的集群找到假定的开放读帧。为了评估AFEs产生蛋白质多样性的潜力,我们将含有AFEs的结构分为三组:i)某个簇中的AFEs不参与ORF,且下游外显子导致所有AFEs的ORF相同;ii) AFEs含有可替代的转录起始位点(ATG),但下游外显子是相同的;iii) AFEs含有不同的转录起始位点,下游外显子不相同。
为了验证一个含有afe的结构是否会产生含有过早停止密码子(PTC)的转录本,从而被无意义介导的衰变机制(NMD)降解,我们计算了停止密码子到最后3'外显子-外显子交界处的距离。NMD候选人是根据50 nt规则定义的,如前所述[26]:如果测量距离为>50 nt,则认为含有afe的结构为NMD候选结构。
结果与讨论
植物基因组中AFEs的系统鉴定
基于大量公共数据库的序列比较,我们在水稻和水稻中分别鉴定出23,500和12,964个全长cdna包含的基因簇拟南芥,分别。这些基因簇约占42%(来自TIGR水稻基因组注释版本4的55,890个基因位点)和48.5%(来自TAIR的26,751个蛋白质编码基因)拟南芥基因组注释(Genome Annotation Release 6)水稻和水稻总表达基因拟南芥,分别。从这些数据中,我们在水稻和水稻中分别鉴定出1378和645个含有afe的簇拟南芥分别集群。在水稻中,约5.9%的表达基因表现出AFE事件。相比之下,基于5'端ESTs的最近估计约为4% [14],从cap技术为基础的cDNA文库中获得,我们的AFE比值略高。这种增加可能是由于i)我们更多的全长cdna和一般5'端ESTs集合,和/或ii)我们可能更敏感的检测方法。在拟南芥在美国,我们观察到含有AFE事件的表达基因比例相似(~5%)。
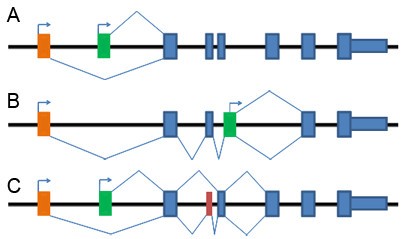
根据簇中第一外显子的基因组位置,观察到AFEs的两种模式。I型AFEs包括第一个外显子相互排斥,且同一簇内基因结构之间大多数下游外显子相同的AFEs(图1);II型AFEs包括基因结构A的第一个外显子作为基因结构B的内部外显子存在的AFEs(图1 b).需要注意的是,有时一个集群可能包含多种类型的afe。
从我们的数据集中,II型AFEs是最丰富的类型。在所有的水稻AFE事件中,II型占90%(1378例中的1241例),在645例中的546例中占83%拟南芥(表2).备选第一外显子起始位点之间的平均距离为1644 bp拟南芥水稻为1141 bp。使用Kouichi Kimura提出的>500 bp区间等。[6]作为标准,我们估计至少有257和352的II型AFE在水稻和拟南芥,分别是由于不同核心启动子的交替使用。通过将同样的标准应用于I型AFE事件,我们在水稻和水稻中确定了额外的62和22个假定的替代启动子(PAP)派生基因结构拟南芥,分别。虽然我们不能确定包含基因结构的非全长cDNA的确切转录起始位点(TSSs),但我们的数据表明,推导出的假定TSSs可能反映了真实的TSSs在活的有机体内因为每个AFE簇的基因结构由来自多个cDNA文库的多个通用5'端ESTs支持。因此,我们估计约23% ~58%的afe基因结构来源于水稻和水稻中的替代启动子拟南芥,分别。
植物基因组AFEs的统计特征
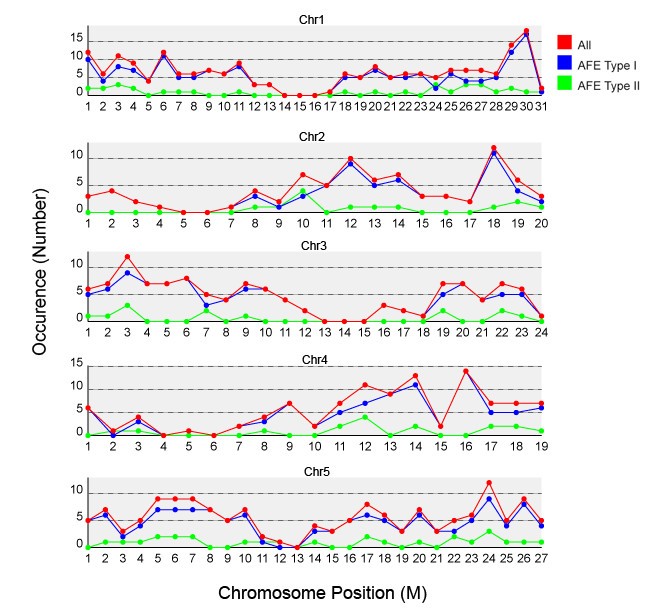
如图所示2,我们未检测到AFEs染色体分布的显著偏倚拟南芥。我们还比较了来自TAIR基因组注释的相对基因密度分布,并没有检测到任何显著的染色体区域富集或缺失。在水稻基因组中也观察到类似的趋势(见附加文件)1).
在哺乳动物基因组中,剪接位点强度在剪接位点选择和选择性剪接中起着重要作用。剪接位点周围的序列组成及其碱基与小核RNA U1配对调节了相应外显子的包合率。为了研究类似的机制是否适用于植物基因组,我们分析了AFEs的5'剪接位点(5's)强度,并将其与组成剪接外显子的强度进行了比较。如表所示3.结果表明,在水稻和水稻中,I型AFEs的5's相对于本构外显子来说相对较弱拟南芥。然而,当考虑外显子包合率时,我们发现两个基因组之间有显著差异。在拟南芥与本构外显子相比,主要表达AFE异构体的5′s强度无统计学差异(t检验与p< 0.01),而AFE亚型在剪接位点强度上与本构外显子差异显著(p= 3.2361e-012,表3.).相反,在水稻中,我们观察到主要和次要AFE亚型之间的5's强度相似。ⅱ型AFEs的分析显示,水稻和水稻之间存在类似的差异拟南芥:在主要和次要II型AFE异构体中的5's强度拟南芥与组成外显子相似,而水稻AFE主要异构体的5's强度远低于次要异构体。这些结果表明,在水稻中,调控剪接位点的选择或识别可能涉及不同的机制拟南芥。
我们进一步研究了在AFEs的5's周围形成二级结构的趋势,因为这种结构之前被认为能够调节剪接位点的识别和剪接。我们测量了AFEs和本构外显子以每5's为中心的100基窗口的最小折叠能量(MFE)。如表所示4的AFEs拟南芥与本构第一外显子相比,AFEs在第5′s形成二级结构的可能性更小,而水稻AFEs在第5′s形成二级结构的可能性更大。
为了研究可能调节第一外显子替代使用的序列基序,我们使用MEME程序搜索AFEs和周围内含子的序列。使用1E-5作为序列比对的截止点,我们没有在AFEs和周围序列的所有或子集中检测到显著富集的基序。这一结果表明,要么是一些调节序列退行性太强,无法通过模因检测到,要么是AFEs受其他机制的调节,而不是特定的序列基序。
AFEs对蛋白质多样性和功能调节的影响
为了研究第一外显子替代使用的生物学意义,我们检查了AFEs中n端编码区是否发生了改变。当假定的蛋氨酸起始密码子位于两种AFE类型的第一外显子上时,n端被认为发生了改变。
在I型AFE集群(互斥的第一外显子)中,最常见的场景涉及AFE事件,产生了具有相同orf的转录本。在这些情况下,集群中的所有基因结构共享一个包含翻译起始位点的公共下游外显子。从我们的数据集中,84和79的AFE集群在水稻和拟南芥分别属于这一类。由于蛋白质结构保持不变,组织或分期特异性之间的改变可能是这些病例的主要后果。
在II型afe基因簇中,est基因结构与全长基因结构之间的差异不仅在于第一外显子的选择性,还在于下游外显子的选择性。因此,est结构中的额外序列可能包含假定的翻译起始密码子,从而产生多个蛋白质变体。在我们的数据中,水稻和水稻中有213和298个II型AFE簇拟南芥分别是这样的案例。这些可选起始密码子大多导致蛋白质n端附加片段。然而,我们发现了一些罕见的病例(5例发生在大米中,3例发生在大米中拟南芥其中AFEs导致了多个阅读框,从而产生了新的蛋白质。
总的来说,我们在水稻中鉴定出266个可能的n端变化,在水稻中鉴定出318个拟南芥含有afe的基因簇。如表所示2在II型AFE簇中,n端蛋白的变化与假定的替代启动子的使用之间存在很强的相关性(使用Fisher精确检验进行检验)。p< 0.01)。簇中基因结构之间的距离似乎对n端蛋白质的变化有显著影响。只有一小部分I型AFE簇产生了蛋白质多样性。主要贡献者是起始密码子位置。我们观察到基因结构的5'端距离与备选起始密码子之间没有联系。
我们还研究了蛋白质n端变化对已知功能蛋白基序的影响,方法是将转录本亚型的推定ORF翻译与NCBI保守结构域数据库(CDD)进行比较[27].如表所示2I型AFE簇中约5~10%的n端变化与至少一种异构体中的已知功能蛋白结构域重叠,而II型AFE簇中约20~30%的n端变化与已知功能蛋白结构域重叠。我们发现,在II型AFE簇中,约5%的功能改变涉及整个域的添加和/或删除。这种afe引入的蛋白质调节有可能导致复杂的功能调节。
我们注意到,至少在某些情况下,可选第一外显子的使用与下游可选剪接事件相结合(图1 c),这可能导致阅读帧的转移,并使后续的异构体可能是无意义介导的mRNA衰变(NMD)的候选者。因此,我们基于TAP方法推导出了不包含全长/参考序列的基因结构的推定转录异构体[28].我们使用过早终止密码子(pcs)的定义作为帧内停止密码子驻留在最后3'外显子-外显子结上游50 bp的>,如前所述[26].筛选结果表明,在水稻和水稻中分别有284和52个AFE转录异构体拟南芥分别产生了NMD候选人。这些频率远远小于在植物AS亚型总数中观察到的频率[26].这种差异可能部分是因为afe耦合的替代剪接事件只是植物中AS事件总数的一小部分;这表明大部分AFEs包含的事件是功能性的,这与我们对AFEs与蛋白质多样性关系的分析是一致的。
包含afe事件的GO分类
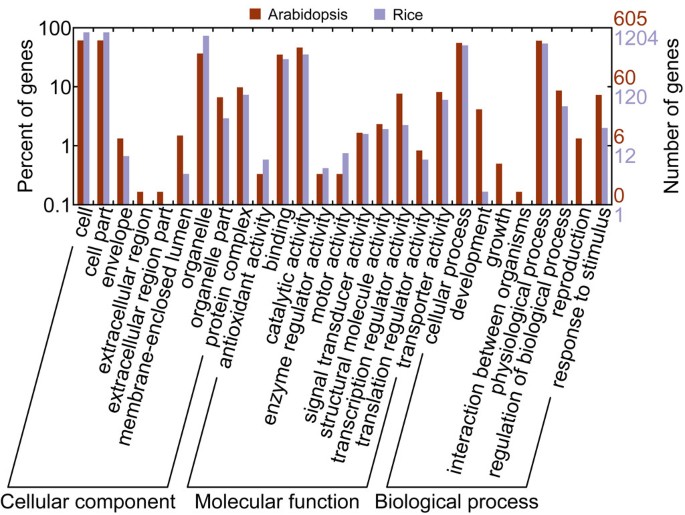
为了研究哪些类型的基因可能使用替代第一外显子以及AFEs可能带来的生物学后果,我们首先对水稻和水稻中含有AFEs的簇进行了分类拟南芥根据基因本体分类。然后我们使用了水稻和拟南芥作为参考,计算含afe聚类中某一氧化石墨烯类显著增加或减少的概率。如表所示5而且6虽然观察到不同的功能类别,但参与酶反应和细胞过程的基因在两种植物中显著富集。在水稻细胞过程调控、转运蛋白、ATP结合、细胞通讯和内源刺激反应等功能类别中也发现了afe富集簇。这些结果表明,AFEs介导的复杂转录调控可能是植物细胞适应内外环境动态变化所不可或缺的。当环境发生变化时,蛋白质的功能可以通过n端功能基序的添加或删除进行微调,或者通过改变信号多肽或转运蛋白活性来重新分配蛋白质定位。
一些GO类别显示大米和大米之间不一致拟南芥(图3.).例如,“细胞内部分”、“细胞内部分”和“细胞部分”富集于拟南芥,但在大米中减少了。需要进一步的研究来阐明这种差异。
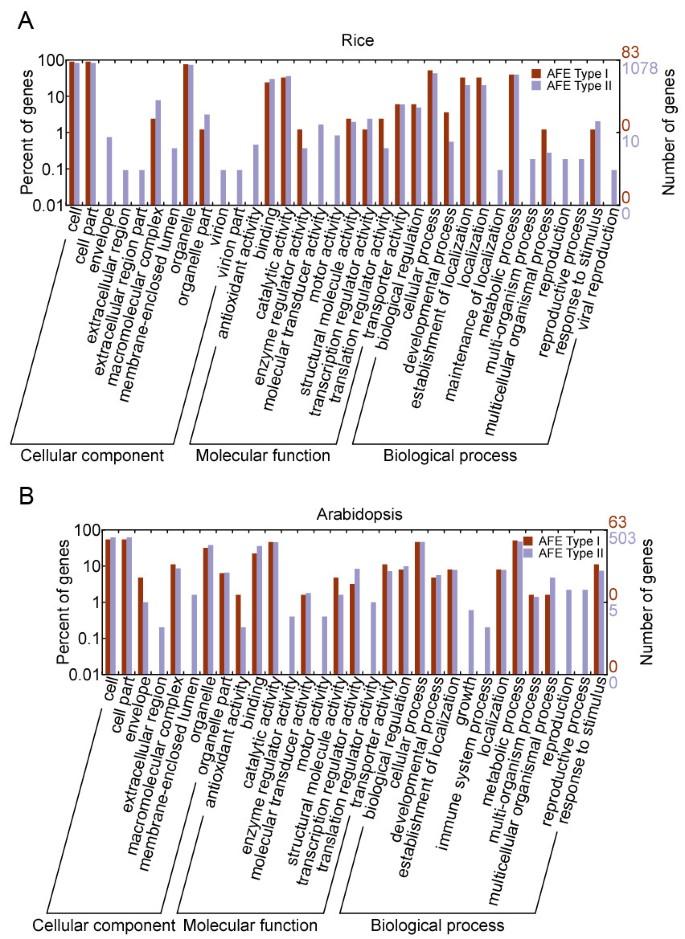
我们还比较了两种AFEs在水稻和水稻中的功能差异拟南芥。如图所示4,虽然只包含少数基因的类别(如“包膜”、“分子换能器活性”和“繁殖”)存在差异,但没有一个在统计上显著(费雪精确检验)p< 0.05)。因此,我们得出结论,在水稻和水稻中I型和II型AFE簇之间没有显著的功能偏差拟南芥。
应该注意的是,使用GO分类的至少一个缺点是,来自不同数据库的相同基因产物的GO映射有时是不同的,因此应该在一定程度上谨慎使用结果。
AFE异构体在植物基因组中的组织和发育阶段特异性表达
我们采用了徐强建议的方法等。[5来评估AFEs是否参与组织和/或发育阶段特异性表达。组织和发育阶段信息从NCBI图书馆浏览器分类下载。对于Unigene数据库中信息不明确或不完整的库,我们检查了它们的dbEST条目并进行了相应的分类。然后我们计算了每个afe基因的三个得分,即组织特异性得分TS以及两个鲁棒性值rTS和rTS ~。如表所示7通过使用高置信标准(HC,见方法),我们在水稻和水稻中鉴定了390个和31个涉及组织特异性表达的AFE聚类,以及273个和44个涉及发育阶段特异性表达的AFE聚类拟南芥,分别。用稍不严格的标准(低置信度,LC,见方法),特异性表达基因的数量增加了2到3倍。
总的来说,我们估计约20~66%的水稻AFE簇以组织特异性或发育特异性转录方式调控。我们的结果与之前的一篇报道一致,即AFEs参与了水稻的组织特异性转录[14].反之,在拟南芥,我们发现只有5~18%的含afe簇特异性表达于某些组织和/或发育阶段。
植物基因组中AFEs的进化保护
研究水稻和水稻之间AFE事件的守恒性拟南芥,我们使用每个AFE簇中最长的内参基因或全长cDNA作为代表序列。正交关系通过应用Inparanoid [24到这些序列。令我们惊讶的是,只有19个afe基因对来自水稻和拟南芥被归类为同源组,在水稻中仅占所有afe基因簇的1.4%,在拟南芥。如图所示3.,含afe基因簇的GO类别在水稻和水稻之间无差异拟南芥(费雪精确测验,p< 0.05),说明在植物基因组中进化保守存在于功能类别中,而不是单个基因。
结论
基于我们大规模的通用5'-EST和全长cDNA对水稻和拟南芥,我们估计在植物中至少有~5%的表达基因簇使用备选的第一外显子。我们进一步分析了这些交替剪接外显子的统计特征,并将其与本构剪接外显子进行了比较。结果表明,水稻和水稻的AFEs可能存在较大差异拟南芥比一般预期的要多。表达分析显示,20~66%的水稻AFE簇以组织特异性或发育特异性方式调控,这与之前的一篇报道一致[14].然而,只有5~18%的拟南芥AFE簇与组织或发育特异性表达有关。虽然含afe簇的GO分类在水稻和水稻之间没有显示出功能偏差拟南芥在两株植物间仅鉴定出19组同源afe簇。考虑到单子叶植物和双子叶植物可能使用不同的不完全兼容的拼接机器[29,30.],我们认为AFE事件可能在双子代和单子代分离后独立进化。
虽然NMD (nonsense-mediated mRNA decay, NMD)可以去除部分AFE事件,但我们发现NMD耦合的AFE事件的比例远低于植物中备选剪接事件的总比例。因此AFE事件似乎特别有可能产生生物功能转录异构体。与以前的报告不同[14,我们已经表明49%和19%的AFE事件来自拟南芥和水稻影响n端蛋白质序列,约23%的水稻和57%的拟南芥AFE事件可能源自多个启动子的替代使用。我们期待着对AFEs与体内蛋白质多样性关系的进一步研究将极大地丰富我们对基因表达调控复杂性的认识。
所有的分析工具,数据库转储和方法的详细描述可根据要求提供,通信应发送到HuSN。
参考文献
- 1.
blencwe BJ:选择性剪接:来自全球分析的新见解。中国生物医学工程学报,2006,26(1):347 - 347。10.1016 / j.cell.2006.06.023。
- 2.
Maniatis T, Tasic B:后生动物中可选的pre-mRNA剪接和蛋白质组扩展。自然学报,2002,418(6894):236-243。10.1038 / 418236。
- 3.
Lareau LF, Green RE, Bhatnagar RS, Brenner SE:替代剪接作用的演变。细胞生物学杂志,2004,14(3):273-282。10.1016 / j.sbi.2004.05.002。
- 4.
着陆器,林惇LM, Birren B, Nusbaum C, Zody MC,鲍德温J,德文郡K,杜瓦K,柯南道尔M,菲茨休W,凡克R,计D,哈里斯K, Heaford,霍德兰J,萤石L, Lehoczky J,莱文R,麦克尤恩P,麦凯南K, Meldrim J, Mesirov JP,米兰达C,莫里斯W, Naylor J,雷蒙德•C Rosetti M,桑托斯R,谢里登,Sougnez C, Stange-Thomann N, Stojanovic N,萨勃拉曼尼亚,Wyman D,罗杰斯J, Sulston J, Ainscough R,贝克,宾利D,伯顿J, Clee C,卡特N,库尔森,亡灵R, Deloukas P,邓纳姆,邓纳姆,杜宾R,法国L Grafham D,格雷戈里·S,哈伯德T, Humphray年代,狩猎,琼斯M,劳埃德·C McMurray,马修斯L,美世年代,米尔恩年代,Mullikin JC, Mungall,垂直R,罗斯M, Shownkeen R,西姆斯,沃森RH,威尔逊RK Hillier LW,麦克弗森JD,马拉马,狂欢节,富尔顿,Chinwalla, Pepin KH,吉斯WR, Chissoe SL, Wendl MC, Delehaunty KD,矿工TL, Delehaunty,克莱默JB,会做饭,富尔顿RS,约翰逊DL,风骚女子PJ,克利夫顿西南,霍金斯T, Branscomb E, Predki P,理查森P,温家宝年代,Slezak T,道根N,程摩根富林明,奥尔森,卢卡斯,艾尔C, Uberbacher E,弗雷泽M,吉布斯RA, Muzny DM,谢勒,Bouck JB, Sodergren EJ,沃雷KC,当厘米,戈雷尔JH, Metzker ML, Naylor SL, Kucherlapati RS,纳尔逊DL,魏因斯托克通用、淡比Y,富士山,服部年宏M,雅达T,丰田章男,伊藤T,马C, H,渡边Totoki Y,泰勒T, Weissenbach J, Heilig R,停下来W, Artiguenave F, Brottier P, Bruls T, Pelletier E,罗伯特·C Wincker P,史密斯博士,Doucette-Stamm L, Rubenfield M,李魏因斯托克K,嗯,杜布瓦J,罗森塔尔,是M, Nyakatura G, Taudien年代,臀部,杨H, Yu J,王J,黄G,顾J,罩L,再生草L,马丹,秦年代,戴维斯RW, Federspiel NA, Abola美联社,普氏MJ,迈尔斯RM,污物J,迪克森,Grimwood J,考克斯博士,奥尔森MV, Kaul R,雷蒙德·C,清水N,川崎K, Minoshima年代,埃文斯GA, Athanasiou M,舒尔茨R,罗伊英航,陈F,潘H,公羊J, Lehrach H,莱因哈特R, McCombie WR, de la巴斯蒂德M, Dedhia N,拦截器H, Hornischer K, Nordsiek G,《R, Aravind L,贝利是的,贝特曼,Batzoglou年代,伯尼E,博克P,布朗DG,伯吉斯CB, Cerutti L,陈HC,教会D,夹M,科普利RR, Doerks T,艾迪SR,为EE,弗瑞TS, Galagan J,吉尔伯特詹,哈蒙C,崎Y, Haussler D, Hermjakob H, Hokamp K,张成泽W,约翰逊LS,琼斯助教,Kasif年代,Kaspryzk,肯尼迪年代,肯特WJ基茨P, Koonin EV, Korf我Kulp D, D柳叶刀,劳TM, McLysaght,米凯尔森T,莫兰合资,穆德N, Pollara VJ,桥CP,舒勒克,舒尔茨J,斯莱特G,Smit AF、Stupka E、Szustakowski J、Thierry-Mieg D、Thierry-Mieg J、Wagner L、Wallis J、Wheeler R、Williams A、Wolf YI、Wolfe KH、Yang SP、Yeh RF、Collins F、Guyer MS、Peterson J、Felsenfeld A、Wetterstrand KA、Patrinos A、Morgan MJ、de Jong P、Catanese JJ、Osoegawa K、Shizuya H、Choi S、Chen YJ:人类基因组的初步测序与分析。自然学报,2001,409(6822):860-921。10.1038 / 35057062。
- 5.
Xu Q, Modrek B, Lee C:人类转录组中组织特异性剪接的全基因组检测。化学学报,2002,30(17):3754-3766。10.1093 / nar / gkf492。
- 6.
木村K,若松,铃木Y, Ota T, Nishikawa T,山下式R,山本J,关根身上M, Tsuritani K, Wakaguri H, Ishii年代,Sugiyama T,齐藤K, Isono Y,老大R, Kushida N, Yoneyama T,大冢R,神田K, T横井,近藤H, Wagatsuma M, Murakawa K,石田年代,Ishibashi T, Takahashi-Fujii,没有试图推高日圆T, Nagai K,菊池H, Nakai K, Isogai T, Sugano S:转录调制的多样化:大规模的假定的识别和描述替代人类基因的启动子。基因组研究。2006,16(1): 55-65。10.1101 / gr.4039406。
- 7.
Luzi L, Confalonieri S, Di Fiore PP, Pelicci PG:从线虫到人类Shc功能的进化。中国生物医学工程学报,2000,10(6):668-674。10.1016 / s0959 - 437 x(00) 00146 - 5。
- 8.
Zavolan M, van Nimwegen E, Gaasterland T:通过映射到小鼠基因组识别小鼠全长cdna的剪接变异。基因组研究。2002,12(9): 1377-1385。10.1101 / gr.191702。
- 9.
Landry JR, Mager DL, Wilhelm BT:复杂对照:替代启动子在哺乳动物基因组中的作用。中国农业科学,2003,19(11):640-648。10.1016 / j.tig.2003.09.014。
- 10.
Bonham K, Ritchie SA, Dehm SM, Snyder K, Boyd FM:一种替代的人类SRC启动子及其受肝核因子-1 α调控的研究。中国生物医学工程学报,2000,29(4):369 - 369。10.1074 / jbc.M004882200。
- 11.
Kelner MJ, Bagnell RD, Montoya MA, Estes LA, Forsberg L, Morgenstern R:染色体12p13.1-13.2上微粒体谷胱甘肽s转移酶基因(MGST1)的结构组织。识别正确的启动子区域和氧化应激反应的转录调控。中国生物医学工程学报,2000,27(4):369 - 369。10.1074 / jbc.275.17.13000。
- 12.
胡忠忠,庄琳,李晓燕,李晓燕,李晓燕。人催乳素受体基因结构与启动子的选择:通用启动子hPIII和新型启动子hP(N)。中华内分泌杂志1999,84(3):1153-1156。10.1210 / jc.84.3.1153。
- 13.
王旭,苏红,Bradley A:调控pcdh - γ基因表达的分子机制:多重启动子和顺式选择性剪接模型的证据。基因学报,2002,16(15):1890-1905。10.1101 / gad.1004802。
- 14.
Kitagawa N, Washio T, Kosugi S, Yamashita T, Higashi K, Yanagawa H, Higo K, Satoh K, Ohtomo Y, Sunako T, Murakami K, Matsubara K, Kawai J, Carninci P, Hayashizaki Y, Kikuchi S, Tomita M:计算分析表明,备选第一外显子参与了水稻(Oryza sativa)的组织特异性转录。生物信息学(牛津,英国)。2005,21(9): 1758-1763。10.1093 /生物信息学/ bti253。
- 15.
Quelle DE, Zindy F, Ashmun RA, Sherr CJ: INK4a肿瘤抑制基因的替代阅读框编码两种不相关的蛋白质,能够诱导细胞周期阻滞。中国生物医学工程学报,2004,24(6):356 - 356。10.1016 / 0092 - 8674(95) 90214 - 7。
- 16.
梁辉,Landweber LF:人类交替拼接基因双编码区全基因组研究。基因组研究。2006,16(2): 190-196。10.1101 / gr.4246506。
- 17.
白X, Peirson BN,董峰,薛晨,Makaroff CA:拟南芥减数分裂必需基因SYN1的分离与鉴定。植物学报,1999,11(3):417-430。10.1105 / tpc.11.3.417。
- 18.
Bairoch A, Apweiler R, Wu CH, Barker WC, Boeckmann B, Ferro S, Gasteiger E, Huang H, Lopez R, Magrane M, Martin MJ, Natale DA, O'Donovan C, Redaschi N, Yeh LSL:通用蛋白质资源(UniProt)。核酸学报,2005,33(增刊1):D154-159。
- 19.
Pertea M, Lin X, Salzberg SL: GeneSplicer:一种新的拼接位置预测方法。核酸研究。2001,29(5): 1185-1190。10.1093 / nar / 29.5.1185。
- 20.
伊沃·刘,Walter F, Peter FS, Bonhoeffer LS, Manfred T, Pet: RNA二级结构的快速折叠和比较。1993,圣菲研究所
- 21.
Bailey TL, Elkan C:通过期望最大化拟合混合模型来发现生物聚合物中的基序。中国生物医学工程学报,1994,24(2):354 - 354。
- 22.
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT, Harris MA, Hill DP, Issel-Tarver L, Kasarskis A, Lewis S, Matese JC, Richardson JE, Ringwald M, Rubin GM, Sherlock G:基因本体论:统一生物学的工具。基因本体论联盟。自然遗传学。2000,25(1): 25-29。10.1038/75556。
- 23.
叶军,方磊,郑华,张勇,陈俊,张铮,王俊,李松,李锐,Bolund L,王俊:WEGO:一种GO注释绘制web工具。核酸研究。2006, 34 (Web服务器问题):W293-7。10.1093 / nar / gkl031。
- 24.
O'Brien KP, Remm M, Sonnhammer EL: Inparanoid:一个真核生物正交学的综合数据库。核酸研究。2005, 33(数据库问题):D476-80。10.1093 / nar / gki107。
- 25.
Rice P, Longden I, Bleasby A: EMBOSS:欧洲分子生物学开放软件套件。科学通报,2000,16(6):276-277。10.1016 / s0168 - 9525(00) 02024 - 2。
- 26.
Wang BB, Brendel V:植物选择性剪接的全基因组比较分析。中国生物工程学报,2006,29(6):769 - 769。10.1073 / pnas.0602039103。
- 27.
Marchler- bauer A, Anderson JB, Cherukuri PF, DeWeese-Scott C, Geer LY, Gwadz M, He S, Hurwitz DI, Jackson JD, Ke Z, Lanczycki CJ, Liebert CA, Liu C, Lu F, Marchler GH, Mullokandov M, Shoemaker BA, Simonyan V, Song JS, Thiessen PA, Yamashita RA, Yin JJ, Zhang D, Bryant SH: CDD:蛋白质分类的保守域数据库。核酸研究。2005, 33(数据库问题):D192-6。10.1093 / nar / gki069。
- 28.
Kan Z, Rouchka EC, Gish WR, States DJ:使用基因组对齐的ESTs进行基因结构预测和可选剪接分析。中国生物医学工程学报,2004,21(5):369 - 371。10.1101 / gr.155001。
- 29.
GJ, Filipowicz W:单子叶和双子叶植物内含子核苷酸组成和二级结构对pre-mRNA剪接的不同影响。EMBO期刊。1991, 10(9): 2635-2644。
- 30.
Simpson GG, Filipowicz W:高等植物mRNA前体的剪接:剪接体机制、调控和亚核组织。植物分子生物学。1996, 32(1-2): 1-41。10.1007 / BF00039375。
确认
感谢王冰冰分享了他的Alternative Splicing分析软件,并给出了其他重要指导。中国国家自然科学基金(No. 90208029)资助了本课题的研究。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者声明没有相互竞争的利益。
作者的贡献
SNH和WHC构思了这项研究。LvCY和CQZ收集数据并进行统计分析。LvGT和WHC对数据进行了控制和分析,并起草了手稿。所有作者阅读并批准了最终稿件。
陈卫华、吕官庭对这项工作也有贡献。
电子补充材料
12870 _2007_196_moesm1_esm.png
附加文件1:水稻基因组中含afe簇的染色体分布。利用AFEs簇的排列位置确定AFEs在水稻染色体上的分布。(png 145kb)
权利与权限
开放获取本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(https://creativecommons.org/licenses/by/2.0),它允许在任何媒体上不受限制地使用、分发和复制,只要原著被恰当地引用。
关于本文
引用本文
陈,WH。,Lv, G., Lv, C.et al。植物基因组选择性第一外显子的系统分析。植物生物学7,55(2007)。https://doi.org/10.1186/1471-2229-7-55
收到了:
接受:
发表:
关键字
- 拼接的网站
- 本构外显子
- 下游的外显子
- 备用起始密码子
- 替代转录起始点