摘要
背景
核苷酸结合位点-富亮氨酸重复序列(NBS-LRR)编码基因是植物抗病基因中最大的一类。149个nbs - lrr编码基因和58个不编码lrr的相关基因约占到目前为止在拟南芥Col-0生态型中标注的所有orf的0.8%。尽管它们在基因组中的普遍存在和功能的重要性,但关于这些基因表达的信息很少。
结果
本文采用表达序列标记(EST)、大规模并行标记测序(MPSS)、微阵列分析、cDNA末端快速扩增(RACE) PCR和基因陷阱系等多种分析方法,分析了拟南芥Col-0中约170个nbs - lrr编码及其相关基因的表达模式。这些基因大多低水平表达,具有多种组织特异性。除10个基因外,至少用一种方法检测这些基因的表达。水杨酸(salicylic acid, SA)处理影响部分nbs - lrr编码基因及相关基因的表达;不同品种对SA的反应不同。对先前发表的微阵列数据的分析表明,10个nbs - lrr编码和相关基因在野生型Landsberg中表达增加erecta(左呃)在鞭毛蛋白处理后。这10个基因中有几个在SA处理后也出现表达改变,与基因的调控一致R防御反应中的基因表达及基础防御反应与水杨酸信号通路的重叠。增强子陷阱分析表明,茉莉酸和水杨酸类似物苯并噻二唑(BTH)均未诱导5个nbs - lrr编码基因和1个tir - nbs编码基因的可检测表达;然而,BTH确实诱导了其他tir - nbs编码基因的可检测表达。对许多测试基因观察到替代mRNA多聚腺苷酸位点的证据。至少在12个基因中发现了选择性剪接的证据,其中11个编码TIR-NBS-LRR蛋白。nbs - lrr编码与相关基因的表达模式、系统发育关系、基因组定位均无明显相关性。
结论
确定了多个nbs - lrr编码及相关基因的转录本。大多数存在于低水平,并表现出组织特异性表达模式。表达数据与大多数拟南芥nbs - lrr编码和相关基因在植物防御反应中的功能一致,但不排除其他生物学作用。
背景
超过40种植物抗性(R)已经从单子叶植物和双子叶植物中克隆出了对多种病原体和害虫(包括细菌、真菌、病毒、线虫和昆虫)有效的基因。这些R根据基因编码蛋白的结构,基因至少可以分为五类[1- - - - - -4].编码核苷酸结合位点-富亮氨酸重复序列(NBS- lrr)蛋白的基因是最普遍的,根据编码的n端结构域和NBS结构域的差异可分为两大类,以及几个亚类[5- - - - - -8].一个主要的组主要编码n端线圈结构域(CC-NBS-LRR或“CNL”;如。RPS2而且RPM1),而另一组的n端结构域与果蝇和人类toll样受体的细胞质结构域相似(Toll-interleukin-1受体样(TIR)结构域;TIR-NBS-LRR或“TNL”;如。16种,N而且RPP5).在拟南芥Col-0基因组中,已鉴定出149个编码nbs - lrr的基因(55个CC-NBS-LRR和94个TIR-NBS-LRR)和另外58个不编码lrr的相关基因[7,9].基于系统发育分析、蛋白基序比较和内含子位置,已在拟南芥中鉴定出4个CNL亚群、8个TNL亚群和一对不同的“NL”蛋白。7,10].这些nbs - lrr编码基因在植物基因组中以单基因、簇和超簇的形式分布[5,7,10,11].
抗病是目前证实的植物nbs - lrr编码基因的主要功能[2].NBS-LRR蛋白在抗病性中的作用仍在研究中[8].除了直接检测病原体配体外,R蛋白还可以监测('守卫')病原体毒力效应物靶标的状态,或这些蛋白作用的细胞后果[2,12- - - - - -16].LRR结构域可能参与蛋白质-蛋白质相互作用,至少部分决定耐药性特异性[17- - - - - -27].TIR区域的多态性也已被证明影响耐药性特异性[23,28].除了决定识别特异性外,LRR结构域还可能通过分子内和分子间的相互作用参与防御信号传递[27,29- - - - - -32].NBS区域包含几个保守的基序,与dna共享的NB-ARC(核苷酸结合域)同源Apaf-1,一些R基因和Ced-4)某些真核细胞死亡效应因子的作用域,如Apaf-1而且Ced-4[33].番茄I2和Mi-1这两种NBS- lrr蛋白的NBS结构域已被证明能够结合并水解ATP [34]而ATP结合形式是I2蛋白的活性结构[35],表明NBS结构域在诱导防御反应的信号转导中起着分子开关的作用。缺乏LRR结构域的CC-NBS和TIR-NBS蛋白的作用尚不清楚,但它们可能在哺乳动物系统中作为类似Myd88的适配器蛋白发挥作用[9].
已从拟南芥中分离出超过14个nbs - lrr编码基因,这些基因赋予了对细菌、卵菌、真菌或病毒病原体的抗性1).大多数nbs - lrr编码基因的亚群至少包含一种已知基因R这表明大多数nbs - lrr编码基因可能参与抗性。然而,一些更小和更分散的亚群不包含已知的抗性基因,并且有有限的证据表明NBS-LRR蛋白参与植物生物学的其他方面,如植物发育。拟南芥tir - nbs - lrr编码基因的T-DNA插入突变体改变了遮荫回避和疾病易感性[36].腺苷酸环化酶(交流例如,从玉米花粉中克隆的基因在花粉极化生长中起作用,并且与nbs - lrr编码基因具有序列相似性[37].番茄我2基因(CNL型)在侧根形成的部位表达,这表明它可能除了识别病原体外还有其他功能[38].其他四种蛋白质包括R基因产物还包含参与其他过程的蛋白质,如分别参与发育和激素接受的两种受体样激酶CLAVATA1和油菜素类固醇不敏感蛋白(BRI1),以及在植物发育中起作用的另一种受体样蛋白clavat2 [39- - - - - -42].分析拟南芥中nbs - lrr编码基因的表达模式,可能为研究其功能提供线索。
尽管越来越多的R基因被克隆,但对植物的调控知之甚少R基因的表达。RNA凝胶-印迹分析检测到大多数克隆的转录本水平较低R无挑战植物的基因[1,38,43- - - - - -48].然而,表达者寥寥无几R对基因进行了详细的研究。七TIR-NBS-LRR-encodingR基因(L6, Rpp5, N, M, RPS4, RAC1,Bs4)已被证明可编码两种或两种以上的转录本[46,49- - - - - -54];然而,选择性剪接在抗病中的作用尚不清楚。烟草的替代转录本N基因和拟南芥RPS4对由这两种介导的防御反应很重要吗R基因(50,52].对于cc - nbs - lrr编码,已经发现了罕见的替代剪接R基因。在普通菜豆中,cc - nbs - lrr编码基因被鉴定出替代转录本JA1tr而选择性剪接不受病原体感染的调控[55].病原体感染诱导耐药基因表达的报道非常少R基因,比如甜菜Hs1箴-1、大麦Mla、大米最后,Xa27[56- - - - - -59].新近克隆水稻的诱导Xa27在病原菌感染部位编码一种与其他R蛋白无同源性的蛋白的基因与耐药性相关[59].某些nbs - lrr编码的表达R基因已被证明受病原体感染以外的因素影响,如组织类型、发育阶段或环境条件[38,60- - - - - -62].
基因表达的分析方法已经从单基因方法发展到各种全球转录谱分析技术[63,64].大规模表达序列标签(EST)测序[65]和基因表达序列分析(SAGE, [66])可以定量评价基因表达,但信息量不如大规模并行签名测序(MPSS;[67,68])。MPSS以化学计量学的方式在转录本的3'端附近生成数百万个标签;因此,可以在每个样本中评估每个转录本的相对丰度,并且可以检测到罕见的转录本和以前未识别的基因。然而,它是昂贵的,很少有样品可以分析。微阵列允许一个中间数量的样品被分析,但需要先天的除非使用平铺阵列,否则表达基因的知识[69,70].目前使用微阵列分析的挑战包括应用适当的统计方法来识别表达中的显著变化,并在不同的微阵列数据集之间进行信息比较,以及将微阵列数据与来自其他方法的表达信息集成[71,72].
在本文中,我们描述了多种基因组方法来描述nbs - lrr编码及其相关基因在拟南芥中的表达。这些方法包括EST文库、MPSS、微阵列实验、基因陷阱线和RACE-PCR。超过80个基因的转录本结构被定义。我们测定了约170个nbs - lrr编码及相关基因的表达水平、组织特异性和可能的表达诱导性。大多数nbs - lrr编码及相关基因表达水平较低,且具有不同的组织特异性。如前所述为已知R基因,这些基因的表达是在植物防御反应中诱导的,只有少数的情况下被检测。本研究为进一步对单个基因进行功能分析提供了基础。
结果
表达式序列标记(EST)库中的表示
在我们的研究过程中,我们多次检查了nbs - lrr编码转录本在EST集合中出现的频率。2002年4月,从NCBI EST数据库中检索了181406条拟南芥序列,以确定其与170个nbs - lrr编码及相关基因的基因组序列的相似性。在这170个基因中,大约有一半(98个)被检测到ESTs;大多数基因(81个)每个基因有5个或更少的代表。在这种低频率下,其他表达的nbs - lrr编码和相关基因可能在这种深度EST采样中未被检测到。因此,对nbs - lrr编码及其相关基因的表达进行更高效、更灵敏的分析。当2006年7月重复同样的分析时,对622,792个拟南芥EST序列进行了搜索,以确定其与172个nbs - lrr编码及其相关基因的相似性。在分析的172个基因中,仅检测到大约三分之二(120个)的ESTs。其中大多数(94个)每个基因仍然有10个或更少的代表2;详细信息在附加文件1及网上资料库[73])。
大规模并行签名测序(MPSS)分析
利用杜邦拟南芥MPSS数据库和公共拟南芥MPSS数据库([74- - - - - -77])。平均而言,在22个杜邦库中,每个库中大约有100万个标签(表2)3.)以及Meyers等人生成的17个库中每个库中大约250万个标签4).这些标签代表给定样本中的所有转录本,每个标签的频率与每个代表基因的表达水平相关。
170个nbs - lrr编码和相关基因中的大多数在至少一个文库中被检测到,且水平较低(1至991 ppm调整百万分率(adjPPM)或百万分转录本(TPM)与内参基因相比,如EF-1α(769至4061 adjPPM)和第2幕(6-2925 adjPPM)(用于[9])。在水杨酸处理后52 h的叶片文库中,nbs - lrr编码或相关基因At3g04210表达最高,TPM水平为991 TPM (S52)。在愈伤组织文库中,At1g72900表达量第二高(683 TPM)。在未经处理的拟南芥组织中表达量最高的基因为At3g04210,该基因在叶片文库(LES)中表达于322tpm。其他基因的表达水平远低于这两个基因。大约一半的基因(73个)表达在低于32个调整ppm或TPM的极低水平。在37个MPSS文库中,只有11个基因未被检测到表达。在MPSS分析中表现出较高表达水平的基因,例如At4g33300和At3g50950,也在EST数据集中得到了很好的表达。21个预测或潜在假基因中17个在Col-0基因组中的表达[7]在Col-0组织产生的至少一个MPSS库中检测到。
nbs - lrr编码及相关基因的表达数量在MPSS文库中差异很大,从Col-0叶片文库(LEF)中检测到101个,到Col-0晚期种子文库(Ase2lm-la)中检测到15个。平均而言,每个基因只存在于39个文库中的15个(附加文件)1).四个最常见的基因At1g61190、At1g61300、At1g59124和At3g07040都编码CC-NBS-LRR蛋白,在39个研究文库中有37个被检测到。
大多数nbs - lrr编码及其相关基因在拟南芥不同组织、不同发育阶段或不同基因型中表达水平不同(附加文件)1).在拟南芥MPSS公共数据库中,有40个nbs - lrr编码及其相关基因在愈伤组织中表达量高于其他组织。一些基因优先在地上生植物部位表达(如At5g44870,叶片中58tpm,根中2tpm),而另一些基因则是根特异性的(如At5g45210,根中28tpm,叶片和其他组织中0 PPM)。部分基因主要在花中表达(如At1g63740,花中表达52 TPM,角果中表达17 TPM,根、叶和愈伤组织中表达不到5 TPM)。有些是由化学物质或激素诱导的(例如,在At1g72850中,脱落酸(ABA)诱导的Landsberg反应中,12个adjPPM vs 3个adjPPMerecta(左呃)植物和非诱导植物)。这种表达模式的范围表明nbs - lrr编码及其相关基因可能具有多种功能。
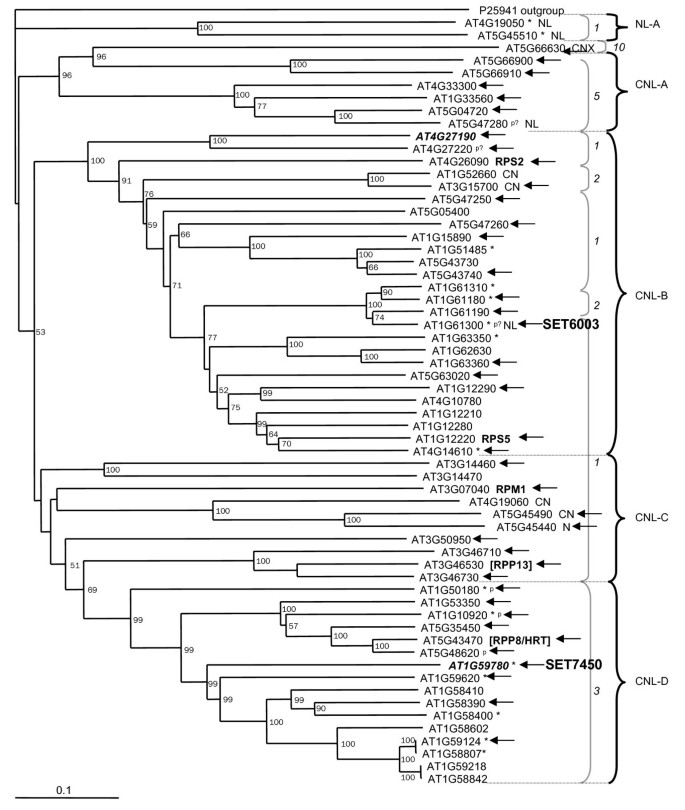
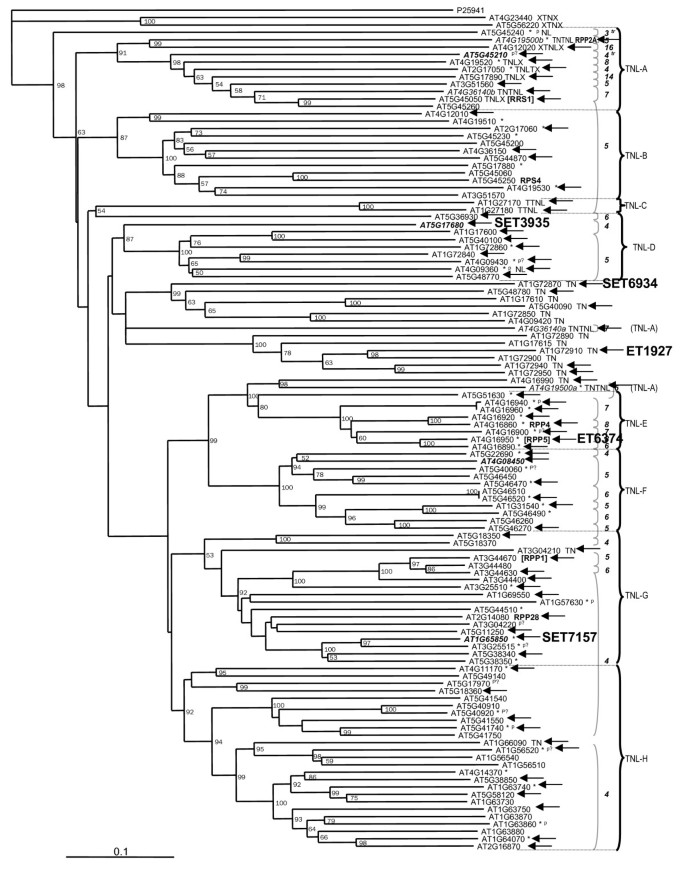
目测结果显示,所研究基因的编码蛋白结构(CC-NBS (CN)、CC-NBS- lrr (CNL)、TIR-NBS (TN)或TIR-NBS- lrr (TNL))与其表达模式之间无明显相关性。每组都含有不同表达水平和组织特异性的基因。在Meyers等人鉴定的nbs - lrr编码基因的四个CNL亚群和八个TNL亚群中[7],观察到各种各样的基因表达模式(附加文件1).结果表明,基因表达模式与在系统进化树上的位置没有相关性。染色体位置与表达模式之间也没有明显的相关性(附加文件1).
120个NBS- lrr编码或相关基因(2个发散型NLs、32个CNLs、2个CNs、1个NL (CC型)、1个NBS (CC型)、63个TNLs、5个TNLX、2个TNTNL、2个TTNL、1个NL (TIR型)、2个XTNX和7个TNs)被多个MPSS标记所代表(附加文件)1).当选择性剪接导致不同的终止密码子和多聚腺苷酸位点时,或者当多聚腺苷酸位点在a的两侧发生变化时,一个基因可以检测到多个标记分3A网站[9].共有72个基因显示可能的选择性多聚腺苷酸化。其中12个基因显示了可能的剪接,因为检测到的一些标签位于剪接位点或内含子内;这12个基因中有11个是tnl而不是cnl,这与tnl编码基因中含有多个内含子而cnl编码基因中缺乏内含子一致。RACE-PCR和随后对其中4个基因的测序证实了选择性剪接:At1g63750, At4g16860 (RPP4Col-0中的同源物),At4g16950 (RPP5Col-0中的同源物)和At5g46270。未能检测到其他8个基因的替代转录本可能是由于组织特异性或替代剪接转录本的低丰度。对于检测到多个标签的其他基因,有些是由于多聚腺苷酸位点的变化,有些是由于代表一个基因家族的几个成员的标签。
微阵列分析
为了在不同于MPSS文库的条件范围内检测表达,还使用来自Affymetrix微阵列的数据分析了nbs - lrr编码和相关基因的表达,这些数据是作为两项大型研究的组成部分生成的[78,79].这两项实验都利用了全基因组阵列(ATH1;Affymetrix),包含152个探针集,代表162个nbs - lrr编码及其相关基因,这些探针集位于Meyers等生成的系统发育树上。[7],包括13个已知的抗性基因(RPP8没有在这个数组中表示)或它们在Col-0中的同系物。
在一项实验中,研究人员分析了7份拟南芥(Col-0、Cvi-1、Est、Kin-0、Mt-0、tu -1和Van-0)在施用0.3 mM水杨酸(SA)后nbs - lrr编码及相关基因的表达变化,以鉴定表达水平多态性,如[79]及ELP网站[80].对这些数据的重新分析显示,在对照Col-0样品中,大约三分之二的nbs - lrr编码和相关基因的表达高于检测阈值(在0.02% silwet处理4小时后收集的三个重复中至少有一个重复)。At1g57630、At1g59124、At1g72900、At1g72910、At3g04210、At3g50950、At4g16950、At4g33300和At5g45510这9个基因在三个对照Col-0样本中至少有一个的表达量高于相应对照Col-0样本中当前信号的平均值。从对照样品的表达水平来看,162个nbs - lrr编码及其相关基因(152个探针组中有138个探针组)在至少一对拟南芥亲本之间的表达水平存在显著差异,说明nbs - lrr编码基因在不同亲本之间的表达存在自然差异。
与同一时间点收获的对照样品相比,SA处理未显著改变大多数nbs - lrr编码及相关基因的表达水平;然而,15个基因(3个CNLs (At3g14470、At5g04720和At5g66900)、1个CN (At5g45490)、1个CNX (At5g66630)、7个TNLs (At1g17600、At3g44630、At4g12010、At4g16860 (RPP4SA处理4小时后,Col-0植株显著诱导At5g36930、At5g41740和At5g46520)、两个TNs (At1g66090和At1g72900)和一个发散型NL (At5g45510)1而且2).此外,SA处理后28小时,4个nbs - lrr编码基因及相关基因(2个TNLs (At3g44400和At4g36150)、1个TN (At3g04210)和1个CNL (At3g50950)的表达下调。这4个基因在SA治疗后4小时也有表达增加,但没有统计学意义。
在不同拟南芥材料中,SA处理后不同时间点nbs - lrr编码及相关基因的表达受到影响(附加文件)2).在至少一株拟南芥中,共有33个探针组对SA处理表现出差异表达。一些nbs - lrr编码基因对SA处理表现出相似的反应,而另一些则没有。例如,一个cnl编码基因At1g12280的表达在SA处理4小时后的Est和SA处理28小时后的Van-0中都被下调。一个CNL基因At4g14610在SA处理后4小时在Kin-0和tu -1中表达升高。其中一个CNL基因At5g66900在SA处理4小时后在Col-0、Kin-0、Mt-0、tu -1和Van-0 5个拟南芥中表达升高,而Van-0仅在SA处理28小时后表达下调。
为了比较基础防御反应中SA对表达变化的反应,我们重新分析了Zipfel等人对鞭毛蛋白反应的数据集[78]使用与上述相同的程序。在该分析中,在两个对照野生型Landsberg中至少有一个中,只有不到一半的nbs - lrr编码和相关基因(69个)表达高于检测阈值erecta(左呃)样品。10个nbs - lrr编码及相关基因的表达(5个CNLs (At3g07040 (RPM1)、At3g50950、At4g26090 (RPS2)、At4g33300和At5g04720)、3个TNLs (At1g56510、At1g56540和At5g22690)和2个TNs (At1g72900和At1g72940)呃.有趣的是,在这10个nbs - lrr编码及相关基因中,有一个基因At1g72900在SA处理4小时后的Col-0和Van-0中也被诱导;SA处理4小时后,在Col-0和Mt-0中诱导At5g04720基因;SA处理4小时后Mt-0也诱导At4g33300和At1g56510两个基因;SA处理28 h后,一个基因At3g50950在Col-0中的表达下调,表明植物的基础防御反应与SA途径相互作用(附加文件)2).
总体而言,上述微阵列实验中检测到的nbs - lrr编码及相关基因的表达模式在阵列实验中基本一致。大多数基因被检测到低水平表达,并不是由防御信号处理诱导的。
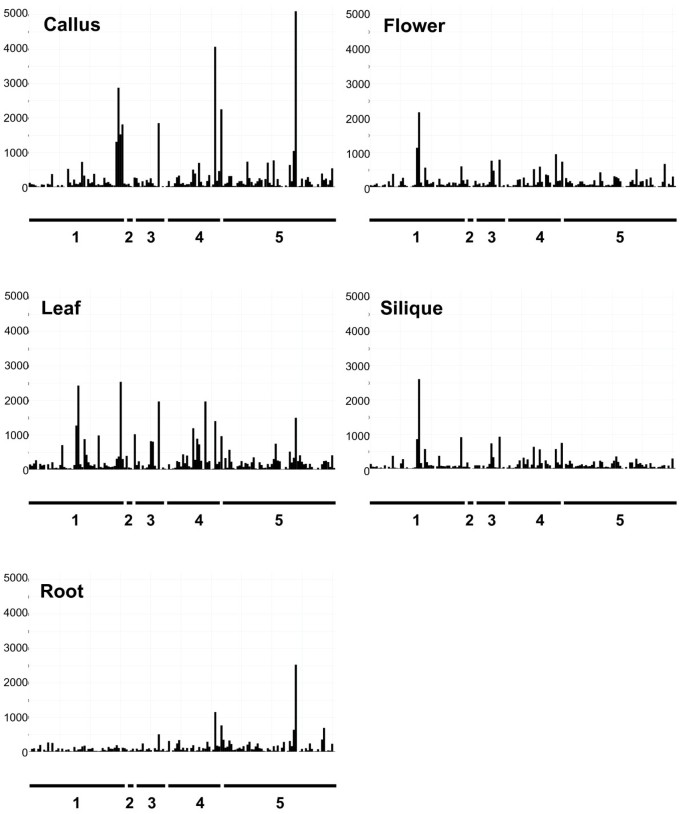
其中5个mRNA样本用于生成上述拟南芥MPSS文库(LEF、CAF、INF、ROF和SIF MPSS文库;[75- - - - - -77])也使用ATH1阵列进行分析,以交叉验证两种方法。微阵列实验中检测到的nbs - lrr编码及相关基因的表达模式与MPSS数据基本一致(表1)2;额外的文件1).这162个基因在不同组织中表达水平不同。例如,MPSS数据和Affymetrix阵列数据均表明,At4g16990在叶片中表达量最高,在花和角果中表达量较低,在愈伤组织和根中表达量最低。大多数基因通常在愈伤组织中表达最高;在花和角果中的表达水平相似(图1).在微阵列分析的叶片样品中,67个未检测到表达水平的基因中有46个在叶片MPSS文库(LEF)中有0个MPSS标记,而在微阵列分析的95个基因中有75个在MPSS叶文库中有一个以上标记。对于在ATH1阵列上由唯一MPSS标记和探针集表示的88个nbs - lrr编码及其相关基因,Spearman秩相关检验表明,MPSS数据与同一叶组织生成的Affymetrix阵列数据具有良好的相关性(相关系数0.74357,P-value < 0.001)。
报告基因陷阱
我们尝试在上述全局分析的同时使用增强子和基因陷阱系,以获得单个nbs - lrr编码或相关基因的细胞特异性表达模式的信息,并更详细地研究它们的诱导性。基因陷阱和增强子陷阱包含基因的插入β葡萄糖醛酸酶(格斯)分别在无启动子或最小启动子控制下的报告基因[81- - - - - -83].报告基因插入其内部或附近的基因的表达模式可以通过报告基因的表达来监测。基因陷阱和增强子陷阱也可用于分析染色体基因破坏导致的突变表型[81- - - - - -84].
拟南芥基因陷阱系侧翼序列冷泉港数据库[85]和新加坡IMA(分子农业生物学研究所,[86])在2002年使用BLAST在nbs - lrr编码和相关基因中进行了搜索。10个增强子诱捕系和3个基因诱捕系被插入到nbs - lrr编码或相关基因中。7个增强子陷阱系插入5个nbs - lrr编码基因和2个tir - nbs编码基因,确定了插入位点和方向(表2)5,数据2而且3.).没有基因陷阱系被证实。
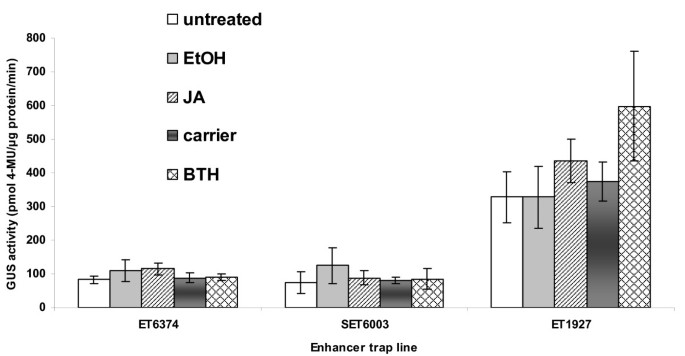
通过GUS染色和幼苗和5周龄植株的GUS定量分析,分析每个确认的增强子陷阱系中相应nbs - lrr编码及相关基因的表达模式。在5个NBS-LRR和1个TIR-NBS增强子诱捕系的整株幼苗、叶、根、花和茎中均未检测到GUS活性。这表明在微阵列和MPSS实验中检测到的这些基因的低表达水平低于GUS实验的检测阈值。我们没有发现任何证据表明任何特定的细胞类型表现出局部的、高水平的表达,因为已经观察到插入到其他一些类型的基因[81,87,88].在插入tir - nbs编码基因At1g72910的增强子陷阱系中,GUS活性染色后,整个叶片呈现非常微弱的蓝色。这种低水平的GUS活性是通过GUS定量试验确认的(图4).At1g72910是在Col-0叶片的MPSS文库和微阵列数据中检测到的表达水平较高的基因之一(表1)5).这些数据表明了组织化学检测基因陷阱系中基因活性所必需的阈值。
这7个nbs - lrr编码基因及其相关基因在上述微阵列实验中均未被水杨酸(SA)或鞭毛蛋白诱导。为了研究在微阵列实验中未检测到的时间点瞬时发生表达变化的可能性,对每一种增强子陷阱植物都施用了茉莉酸(JA)或苯并噻二唑(BTH)。GUS染色和定量GUS实验均未证明BTH或JA诱导了5个nbs - lrr编码基因和1个tir - nbs编码基因的表达(数据未显示)。另一方面,在BTH治疗5天后,At1g72910的表达被诱导了大约2倍(图4),虽然在微阵列分析中未检测到明显的SA诱导。为了检查在病原体渗透部位的有限数量的细胞中发生表达变化的可能性,七个增强子陷阱植物中的每一个都受到了挑战p .两pv。番茄应变DC3000携带avrRpt2或avrRpm1.所有品系均表现出与野生型Landsberg相似的超敏反应erecta(左呃)植物,这是增强子和基因陷阱系的遗传背景。无明显的局部诱导格斯对病原体有反应的浸润区域的基因表达。
cDNA末端快速扩增(RACE)分析
采用RACE-PCR作为敏感的定性方法检测nbs - lrr编码及其相关基因的表达,并确定转录物的5'端和3'端。转录本边界的确认是基因功能研究的先决条件,因为公共数据库中大约三分之一的nbs - lrr编码和相关基因之前注释错误,因此必须通过手动重新注释来更正[7].将我们的5'和3' RACE产物的序列与拟南芥基因组序列进行比较,以确定每个转录本的起始和终止位点(附加文件)1及网上资料库[73])。
以Col-0叶片总RNA为模板,共分析了109个nbs - lrr编码及相关基因。至少对81个基因检测到一种RACE产物(分析的35个CNL基因中的26个和50个TNL基因中的38个;表格2).在这81个基因中,有68个检测到5'和3'产物。另外6个基因只检测到5'产物,另外7个基因只检测到3'产物。其他28个基因均未检测到5'和3'产物;用RT-PCR和引物证实,这28个基因中的6个在叶片中缺乏表达,引物应该扩增了转录本的内部区域(图2而且3.、表2).对6个nbs - lrr编码基因进行RACE,这些基因在4周树龄植株的叶片中未检测到,并以7天树龄幼苗中提取的总RNA为模板。RACE再次未能检测到其中5个基因At2g17050、At3g46730、At4g08450、At4g09360和At4g27190的表达。在幼苗中仅检测到At1g65850的RACE产物。对于两个nbs - lrr编码基因At3g44400和At4g33300,除了从叶片和幼苗中生成的模板外,还以从花中提取的总RNA为模板进行RACE。RACE结果表明,这两种nbs - lrr编码基因在叶片、幼苗和花中均表现出不同的表达模式。At4g33300在幼苗中表达量最高,其次是花和叶组织,而At3g44400在叶片中表达量高于花。
上述RACE-PCR产物中,5' UTR长度为0 ~ 241 bp(平均= ~50 bp);大多数5'转录起始位点位于距离ATG起始密码子100 bp以内。转录起始位点存在一定的变异,通常每个基因可以检测到多个起始位点。3' UTR长度从6 bp到896 bp不等,大多数3' UTR长度为几百bp(平均= ~250 bp)。3'UTRs最常见的变异是由于多聚腺苷酸位点的替代。RACE与基因组序列比较发现,15个基因的5'或3' UTR区域含有内含子。7个基因在5' utr中有1个内含子,1个有2个内含子。6个基因在3' utr中有1个内含子,1个基因有3个内含子。TAIR数据库也显示5'或3' utr中存在内含子[89].根据我们的RACE结果,8个在5' UTR中含有内含子的基因中有6个在TAIR数据库中也被注释为含有内含子,同样,7个在3' UTR中含有内含子的基因中有5个也在TAIR数据库中被注释为含有内含子。
RACE-PCR产物的序列分析显示,TAIR数据库中12个nbs - lrr编码及相关基因的注释不正确,至少与我们的实验数据不一致(附加文件1).这些基因被注释了不正确的转录起始或终止位点、不同的剪接位点或额外的外显子。RACE-PCR产品验证了之前对6个基因的手工重新标注[7].其他6个基因中的错误之前没有被检测到。这六个基因的剪接事件与预测的不同。RACE-PCR数据还显示,除了MPSS数据显示的4个基因具有选择性剪接外,At1g10920的5'UTR和At1g72860的3'UTR也存在选择性剪接。RACE-PCR和测序也证实了这4个基因。
讨论
我们的综合表达分析显示,大部分nbs - lrr编码及相关基因在拟南芥中表达水平较低,而且许多基因出人意料地表现出组织特异性表达模式(附加文件)1).一些nbs - lrr编码基因和相关基因的表达受到防御信号化学物质处理的影响。大多数先前未鉴定的nbs - lrr编码及相关基因的表达模式与已知基因相似R基因,因此是一致的这些基因也在抗病功能。
不同分析方法之间的一致性
不同的分析方法在敏感性和准确性上有所不同,但从每种方法获得的表达数据基本一致。MPSS和微阵列都是最有效的全基因组转录谱分析方法,它们之间的相关性很好。与微阵列分析相比,MPSS有几个优点。MPSS更敏感和准确,因为MPSS提供了每个转录本丰度的定量评估,而不是在微阵列分析中为每个探针集生成的杂交强度。MPSS对于低水平表达的基因特别有利,因此在微阵列分析中更容易受到背景噪声的影响。例如,At5g45230和At1g17600在叶片组织微阵列分析中均以不可检测的水平表达;然而,根据MPSS数据,At1g17600在叶子库中表达明显高于At5g45230 (14 vs 0 TPM)(附加文件)1).MPSS还可以比微阵列更好地区分多基因家族成员,因此减少交叉杂交问题,这在微阵列分析中更常见。MPSS和微阵列数据之间的不一致可能是由于微阵列分析中的交叉杂交问题,或者由于一些MPSS标记代表了具有高度序列相似性的几个基因家族成员。此外,目前拟南芥基因组组装中的测序错误可能导致部分MPSS标记的分配不正确,从而导致表达测定不准确。虽然MPSS具有上述优点,但其技术可获得性和高成本限制了其广泛应用,尽管这些问题可能会通过最新的测序技术得到改善。因此,微阵列仍然是一个有用的补充,以调查MPSS数据不存在的情况,直到其他高通量测序技术可用,可以负担得起,对EST表示进行深入分析。
nbs - lrr编码基因表达水平
本研究中检测的nbs - lrr编码及相关基因大部分在无挑战植物中低水平表达,与大多数克隆植物中观察到的情况相似R基因。大多数nbs - lrr编码及相关基因的表达均有显著变化,包括已知的R在植物防御反应或两种防御信号分子SA或JA处理过程中未检测到基因。最近的其他RNA分析实验也未能检测到差异表达R基因(90,91].同样,在另一项研究抗性反应过程中基因表达变化的微阵列实验中,拟南芥nbs - lrr编码基因在胁迫介导的植物防御反应过程中没有表现出显著的表达变化RPS2,RPM1,RPS5,或RPS4(A. Bent等人,未发表)。这种在植物防御反应中缺乏基因表达的诱导类似于大多数已知的植物R基因。虽然目前还不清楚植物R蛋白在植物防御反应中的作用,但很明显,它们在防御信号通路的早期阶段起作用,要么作为主要识别分子,要么作为辅助蛋白[14- - - - - -16,92- - - - - -94].低水平的R蛋白的本构表达与识别病原体感染和诱导下游防御反应的本构能力一致。
然而,有几个迹象表明转录控制R基因的表达。至少是一个子集R在诱导基础抗性的过程中,基因被诱导到其低水平的本构表达之上;的表达式RPS2,RPM1,以及其他8个nbs - lrr编码及相关基因,通过细菌鞭毛蛋白肽flg22 [78].这可以被认为是增加植物检测潜在病原体的总体敏感性。我们的分析还揭示了野生型Col-0中15个nbs - lrr编码及相关基因,包括Col-0同源体RPP4,在SA处理4小时后诱导;在其他材料中,SA也诱导了其他几个基因。有趣的是,SA在Col-0中诱导的15个nbs - lrr编码及相关基因中,也有2个基因在flg22诱导下表达升高。这种重叠表明植物的基础防御反应和SA信号通路之间存在相互作用。MPSS数据表明,许多nbs - lrr编码基因及其相关基因的表达也受到植株发育阶段、蔗糖和植物激素ABA处理的影响。此外,我们对基因陷阱的研究数量有限R基因相关序列表明,SA同源物BTH诱导了tir - nbs编码基因At1g72910的表达。之前在拟南芥中进行的转录谱分析实验也发现,在对黄瓜花叶病毒Y株的防御反应中,nbs - lrr编码和相关基因的表达发生了改变;下调一个tir - nns - lrr编码基因At1g56510和一个tir - x编码基因At1g65400的表达,诱导另外两个tir - x编码基因At1g72940和At1g72920的表达[95].这些结果都为调控提供了依据R基因表达在植物防御反应和诱导增强水平的防御相关的监视,以应对生物挑战。
组织特异性
目前研究的一个意想不到的结果是nbs - lrr编码和相关基因表现出频繁的组织特异性表达模式。MPSS数据和微阵列数据均表明,许多nbs - lrr编码及其相关基因具有组织特异性。有些基因主要在植物的气生部分表达,而有些基因则在根中特异表达。其他的似乎是发育调节。这些差异表达模式表明,nbs - lrr编码及其相关基因在抵抗多种攻击植物不同部位的病原体方面发挥作用,或者某些nbs - lrr编码及其相关基因在不同的植物生物学过程中发挥作用。在我们的研究之前,很少有关于已知的组织特异性的数据R基因。所有已知的14种nbs - lrr编码R本研究中分析的基因或其Col-0同源物表现出组织特异性基因表达。有趣的是,番茄的nbs - lrr编码R的基因,我2,在侧根形成的部位表达,表明除了抗病性外,它还可能在侧根形成中发挥作用[38].
选择记录
对几种nbs - lrr编码基因进行了选择性剪接。根据MPSS标记的位置,12个nbs - lrr编码基因显示了选择性剪接的证据,其中4个基因,包括两个已知的Col-0同源体R的基因,RPP5而且RPP4经RACE-PCR和后续测序证实。两个基因的替代转录本(At1g63750和At4g16860)编码的截断蛋白仅包含大部分TIR结构域,同时缺乏NBS和LRR结构域。At5g46270的替代转录本编码了一个缺失大部分LRR结构域的截断蛋白,而At4g16950的替代转录本编码了只有最后几个氨基酸改变的TIR-NBS-LRR蛋白。RACE-PCR发现At1g10920和At1g72860的选择性剪接发生在5'或3'UTR上,因此不会改变氨基酸序列;然而,这种选择性剪接可能会影响转录稳定性,从而影响表达水平。已经报道了七种已知的tir - nbs - lrr编码的替代剪接R基因和一个cc - nbs - lrr编码R基因(46,49- - - - - -55].根据拟南芥基因组序列与全长cDNA和EST序列的比对,在TIGR数据库中筛选出1186个拟南芥基因[96]被注释为正在经历剪接变异,并被分为五种不同类型的剪接变异。虽然在这1186个基因中包含10个nbs - lrr编码或相关基因,但在我们的研究中,通过MPSS或RACE分析,只有一个基因显示出选择性剪接。最近一项广泛的计算分析[97]在4707个拟南芥基因(包括16个nbs - lrr编码及相关基因)中鉴定出了选择性剪接事件。这16个基因中的7个也在TIGR数据库中被鉴定出来;然而,在我们的研究中,通过MPSS或RACE分析,这16个基因中只有2个也被检测出具有选择性剪接。这种不一致可能是由于RACE和MPSS倾向于分析转录本末端的序列,或者由于缺乏对适当的组织或条件的采样。这些数据表明,至少有22个tir - nbs - lrr编码基因、1个tir - nbs - lrr编码基因、1个不同的nbs - lrr编码基因和8个cc - nbs - lrr编码基因在拟南芥中表现出可选剪接。
选择性剪接在植物中的作用R基因尚不清楚。剪接变异体可能与全长R蛋白相互作用,并在烟草抗病性中发挥调控作用N和拟南芥RPS4基因。这类似于动物toll样受体(TLRs)中选择性剪接的作用[98].烟草的替代文本N烟草花叶病毒胁迫诱导基因,两个转录本的比例似乎对抗性至关重要[50,99].拟南芥全长转录本和替代转录本的存在RPS4是必要的RPS4-介导的防御反应[52,One hundred.].然而,亚麻的替代转录16种和番茄Bs4似乎对这些基因介导的抗性并不重要[49,54].
其中一个tir - nbs编码基因At1g72910可能在植物防御反应中发挥作用,其表达由SA类似物BTH诱导。同样,6个tir - nbs编码基因At1g17610、At1g66090、At1g72890、At1g72900、At1g72950和At3g04210也可能在植物防御反应中发挥作用,因为它们的表达在研究的7个拟南芥生态型中至少有一个受到SA处理的影响。两个tir - nns编码基因At1g72900和At1g72940可能在植物的基础防御反应中起作用,它们的表达是由鞭毛蛋白诱导的。在拟南芥Col-0基因组中,有21个编码tir - nbs的基因与烟草的替代转录本具有相似的结构N、亚麻16种或拟南芥RPS4基因(9].这些功能可能类似于tir - nbs - lrr编码基因的替代转录变体。
转录后调控
R基因不需要在转录水平上被诱导来改变对病原体的抗性。可能存在多个水平的负调控,以防止在没有病原体的情况下R蛋白的不适当激活,这将对植物有害,因为防御反应的高成本和病原体独立的细胞死亡。也可能有反馈循环控制R基因表达与HR程度的关系。
我们的研究分析了稳态mRNA水平,这可能不能反映蛋白质丰度。需要进一步的工作来确定多体部分是否存在差异以及发生了什么翻译后修饰。几种表达R据报道,基因在转录后水平上受到调控。拟南芥的转录本RPM1在病原体攻击前后保持较低水平;然而,RPM1蛋白在HR过程中被降解[101].的表达式Xa21基因转录不受植物发育阶段的影响Xa21-授与性抵抗受发育调节[102].
来自动物系统的数据表明,5'UTR参与控制翻译和组织特异性表达[103,104].一些植物R包括大麦在内的5'UTR基因中含有内含子和/或上游开放阅读框(uorf)Mla6和拟南芥RPP1-WsB而且RPP1-WsC[19,105].我们的RACE分析显示,在所分析的81个nbs - lrr编码及相关基因中,有15个在其5'或3' utr中含有内含子。这些5'UTR特征可能是这些基因转录后调控的指示。
结论
大部分nbs - lrr编码基因及相关基因在未侵染植物中转录量较低。许多表现出组织特异性表达模式。大多数nbs - lrr编码基因及相关基因的转录水平在植物防御反应或植物防御信号分子处理过程中没有改变;然而,一些基因的表达发生了改变,这可能表明植物的监视水平发生了变化。我们的数据与大多数nbs - lrr编码及其相关基因的主要功能是植物抗性一致;然而,这并不排除它们参与病原体识别以外的功能。
未来关于组织特异性的意义、替代转录本的作用以及转录本与蛋白质水平之间关系的研究,以及nbs - lrr编码基因的每个主要分支下游诱导的基因谱的描述,可能会提供信息。
方法
表达序列标签(EST)分析
利用每个nbs - lrr编码或相关基因的预测cDNA序列或基因组序列(加上预测起始密码子和终止密码子的上游和下游500 bp),在NCBI EST数据库中搜索,得到每个拟南芥nbs - lrr编码或相关基因的EST表示[7]使用BLAST [106].截至7月14日th, 2006年,该数据库共包含622,792条拟南芥EST序列,包括:短的单读cDNA序列,来自差异显示实验和RACE分析的cDNA序列,以及来自RIKEN(日本理化研究所)的全长cDNA克隆的cDNA序列[107].对所有与nbs - lrr编码及相关基因匹配度大于80%的拟南芥ESTs进行了研究。EST表示是根据ESTs与相应基因的基因组或cDNA序列的比对来确定的,通常具有> 97%的序列相似性。使用TAIR BLAST工具将每个潜在的代表性EST与完整的拟南芥基因组和剪接序列进行比较[108来确认它与所代表的基因是最好的匹配。也统计了与特定nbs - lrr编码或相关基因最匹配但由于测序困难而序列同一性低于97%的ESTs。为每个代表基因计算与几个密切相关的家族成员显示相同水平的序列相似性的ESTs。早在2002年4月,除了FASTA [109]用于搜索序列相似性。NCBI EST数据库当时包含约181406个拟南芥序列。
大规模并行签名测序(MPSS)数据
MPSS通过生成短序列标签提供基因表达的全面评估,每个短序列标签长17到20 bp,从定义的位置产生(通常是第一个位置)分每个转录本内的3A限制位点5'到转录本的聚腺苷酸位点)[67,68].样本中每个基因的表达水平是通过计算代表特定基因转录的诊断序列标记的数量来确定的。
杜邦MPSS数据库包含22个拟南芥MPSS库生成的表达谱。其中一半由拟南芥Columbia (Col-0)或Landsberg构建erecta(左呃)在不同发育阶段收集的组织,一半由野生型或突变型Col-0或L构建呃用各种化学物质或激素处理的生态型植物,包括脱落酸(ABA)、地塞米松(DEX)或蔗糖(表2)3.).每个库包含大约一百万个17bp的标签。另外17个MPSS文库由拟南芥Col-0组织(野生型或开花突变体的愈伤组织、花、叶、角果和根)或水杨酸(SA)处理过的叶片组织制成,并显示在公共MPSS数据库中(表2)4, (74- - - - - -76])。每个公共图书馆都包含大约250万个17bp的标签。
为了分析杜邦MPSS数据,使用每个nbs - lrr编码或相关基因的基因组序列加上预测的起始和终止密码子的上游和下游500 bp进行搜索。在少数情况下,附加的侧翼序列与相邻基因重叠,形成较小的基因间区;然而,这些都是手动检查的,不包含可能导致分析偏差的表达标签。使用TAIR BLAST工具将每个表达的标签与完整的拟南芥基因组和剪接序列进行比较[108]以确认与指定基因的正确匹配;转录本丰度的计算仅使用与感觉链的匹配。计算每个标记的频率,然后以百万分之数(PPM)归一化,以计算样本中每个转录本的丰度。对PPM值进行了调整(adjPPM)以考虑潜在的测序错误(描述于[9])。根据标记位置,预测由可变终止密码子和可变剪接引起的可能的多聚腺苷酸化。对于Meyers等人生成的拟南芥MPSS数据[75,76],根据基因“At”号(基因标识符)对每个基因对17个MPSS文库进行基本查询。为了允许库之间的比较,签名频率被标准化为每百万转录本(TPM)。对于与多个表达特征相关的基因,所有表达特征的丰度之和,包括与拟南芥基因组有多次命中的特征,被用来表示样本中每个转录本的丰度。
为了研究基因表达与系统发育位置之间的潜在相关性,将每个nbs - lrr编码或相关基因的表达水平和组织特异性与之前描述的系统发育树进行比较[7].为了研究基因表达模式与染色体位置之间的潜在相关性,我们通过通常反映其染色体位置的基因标识符对这些编码nbs - lrr及相关基因进行排序,然后将编码nbs - lrr及相关基因的表达模式与其在排序基因列表中的位置进行可视化比较。
微阵列分析
同时使用ATH1 Affymetrix阵列进行全局表达实验,为评估nbs - lrr编码和相关基因的表达模式提供了机会。
分析水杨酸(SA)对水杨酸的响应的实验已在[79].六周的植物从七周开始生长拟南芥在0.02% Silwet L77中喷施0.3 mM SA (Col-0、Cvi-1、Est、Kin-0、Mt-0、tu -1和Van-0)。以0.02% Silwet L77处理的植株为对照。植株的空中部分在4小时、28小时或52小时后收割。每个治疗和时间点重复3次。使用Affymetrix ATH1基因芯片检测基因表达水平。ATH1阵列包含大约22,000个基因,包括152个探针集,代表162个nbs - lrr编码和基于Affymetrix注释的相关基因[110].将原始数据(CEL文件)导入GeneChip®操作软件数据库(GCOS)。使用GCOS分析Affymetrix ATH1 GeneChips阵列上所有探针集的转录本丰度。GCOS还用于评估给定转录本的存在与否(P, present;一个缺席;M,边缘)对每个探测集。为了在芯片之间进行直接比较,原始信号进行了全局缩放,使每个阵列的平均表达水平等于任意目标强度500。然后将缩放后的信号导入Excel中进行进一步分析。
另一个实验的原始阵列数据(CEL文件)[78]对鞭毛蛋白处理过的拟南芥植株进行研究[111].在这个实验中,L呃用10 μM flg22肽处理拟南芥幼苗,处理30 min后对幼苗进行分析。GCOS用于提取表达信号,并为ATH1阵列上的探针集表示的每个基因分配一个存在或不存在的调用。数据处理方式与SA诱导实验相同:原始信号被全局缩放到500的目标强度进行直接比较。
微阵列分析还对用于生成17个公共MPSS文库中的5个的相同5个总RNA样本进行了分析。基因表达水平也使用ATH1阵列进行检测。根据制造商指南(Affymetrix, Santa Clara, CA)进行互补RNA标记、杂交和信号采集。使用Affymetrix Microarray Suite version 5.0 (MAS 5.0)控制清洗、扫描和数据预处理步骤。然后将原始CEL文件导入GCOS, GCOS用于提取表达式信号并分配当前或不存在的调用。为了在芯片之间进行直接比较,原始信号在全球范围内被扩展到500的目标强度。
所有数据分析随后使用genspring GX 7.3.1软件(安捷伦科技,圣克拉拉,加州)进行。GCOS的原始输出信号未经缩放和归一化,作为genspring GX分析的输入。原始信号首先使用给定芯片上所有测量值的第50百分位进行归一化,然后将每个基因的中位数测量值调整为1。为了识别SA或鞭毛蛋白反应的差异表达基因,首先使用几个标准建立了一个显示可靠和可检测表达的探针集列表。如果探针集在至少两种比较条件中的一种的两个重复(或三个重复中的两个)中被GCOS称为存在或边缘,则它们被保留,在至少一种比较条件中,它们的变异系数(CV)小于0.3,并且它们表现出至少两倍的变化。从这个基因列表开始,在给定的治疗中表现出显著表达变化的基因被确定为通过genspring GX的参数检验(Welch t检验)的基因,不假设相等的方差并进行多次测试校正(Benjamini和Hochberg假发现率),aP-value 0.1的阈值。在进行这些参数测试时,使用对数转换的归一化信号。
利用ELP Finder工具检测7个拟南芥各nbs - lrr编码基因的表达水平多态性[112].Affymetrix ATH1 GeneChip探针id作为查询输入,并返回具有标准误差的给定接入中每个基因的平均表达值。所选材料之间的配对t检验结果也被返回。
在SAS9.1 (SAS Institute, Cary, NC)中使用Spearman秩相关检验分析了MPSS和微阵列数据之间可能的相关性。本试验分析的MPSS和微阵列数据均来自同一叶片样本,仅包括88个具有唯一代表性Affymetrix探针集和MPSS序列标记的nbs - lrr编码及其相关基因。
植物材料和生长条件
用于快速扩增cDNA末端(RACE)分析的拟南芥Col-0植株生长在土壤(Premier Pro-Mix B mix)中,环境控制室,21°C, 50%湿度,光照16小时/暗8小时,光照100 ~ 120 μEi。
基因诱捕和增强子诱捕系(Landsbergerecta(左呃)的资料来源,由冷泉港实验室或新加坡分子农业生物学研究所(IMA) [85,86].每个基因陷阱系的幼苗先在1/2 MS加50 μg/ml卡那霉素的培养基上筛选,然后将存活的幼苗移栽到土壤中。这些植物在与上述条件相同的可控环境室中生长。
基因陷阱系分析
基因诱捕系携带β-葡萄糖醛酸酶(uidA)位于序列特征位置的报告基因[81- - - - - -84].利用冷泉港实验室(Cold Spring Harbor Laboratory, CSHL)建立的基因陷阱(Gene Trap)侧边序列数据库,对207个nbs - lrr编码基因及其相关基因的基因组序列进行检索[85),Ds新加坡分子农业生物学研究所(IMA)的插入系[86]使用BLAST [106].对于每个陷阱线,组织PCR [113],通过使用基因特异性引物和a来确定插入位点和插入元素的方向uidA特定的引物。
研究茉莉酸(JA;500 μM)溶于10% v/v乙醇中,在无菌容器中喷洒于5周龄植株上。Benzothiadiazole(蓝芽;1.2 mM)用可湿性粉末载体(与BTH相同数量的载体)溶解于水中,同样地喷洒在无菌容器中5周的植株上。以10% v/v乙醇或可湿性粉末载体作为对照。在每个处理后0、24、48小时至5天的不同时间点采集整株植物,进行GUS组织化学染色和GUS活性测定。每种基因诱捕植物也都受到了挑战p .两pv。番茄应变DC3000携带avrRpt2或avrRpm1在野生型Col-0和L呃植物。分别于病原体浸润后16小时、1天和2天检测HR反应,同时进行GUS染色。
GUS活性的组织化学染色是根据[114,115].植物组织浸泡在GUS染色液(100 mM磷酸钠(pH7.0), 2 mM K3.铁(CN)6, 2 mM K4铁(CN)6, 10 mM EDTA, 0.1% (v/v) Triton X-100, 100 μg/ml氯霉素,与1 mg/ml 5-溴-4-氯-3-吲哚基-β- d -葡萄糖苷环己基铵盐(X-gluc, Gold Biotechnology Inc., St. Louis, MO, U.S.A.)。植物组织用GUS染色液在37℃下孵育24小时。
如Jefferson等人所述,使用4-甲基伞形酮葡萄糖醛酸酯(MUG, Sigma)作为底物,荧光定量测定GUS活性[114].在200 μl萃取缓冲液(50 mM NaPO4pH7, 10 mM EDTA, 0.1% Triton X-100, 0.1% Sodium Lauryl Sarcosine, 10 mM β-巯基乙醇)含有1.1 mM MUG,加入800 μl 0.2 M Na,在37℃孵育1 h后停止2有限公司3..荧光校准使用4-甲基伞形酮(MU, Sigma,圣路易斯,密苏里州,美国)。总蛋白测定以Bradford试剂为标准,BSA为标准[116].
cDNA末端快速扩增(RACE)-PCR
RACE-PCR使用Marathon™cDNA扩增试剂盒(Clontech, Mountain View, CA, USA)按照制造商说明书进行。使用TRIzol程序(Invitrogen, Carlsbad, CA)从大约四周大的野生型拟南芥Col-0植株的叶子、7天大的幼苗或花组织中提取总RNA。然后使用Dynabeads从总RNA中纯化模板mRNA®益生元(dT)25(Dynal Biotech, Lake Success, NY, USA)。RACE-PCR产物克隆到pCR中®2.1威尼斯平底渔船®vector (Invitrogen, Carlsbad, CA)并测序。序列数据使用BLAST和Sequencer 3.1 (GeneCodes, Ann Arbor, MI)进行分析。所有RACE序列存入NCBI GenBank,登录号从ES444179到ES444640,从EX654484到EX654486。
逆转录- pcr (RT-PCR)
RT-PCR使用Advantage™RT-for-PCR试剂盒(Clontech, Mountain View, CA),按照制造商说明进行。引物被设计用来扩增含有内含子的区域,以便从基因转录物中区分基因组DNA污染。
参考文献
- 1.
Hammond-Kosack KE, Jones JD:植物抗病基因。植物化学学报,1997,33(4):366 - 366。10.1146 / annurev.arplant.48.1.575。
- 2.
丹格尔,琼斯。JD:植物病原体与侵染的综合防御反应。自然科学,2001,39(1):1 - 4。10.1038 / 35081161。
- 3.
Staskawicz BJ, Mudgett MB, Dangl JL, Galan JE:植物和动物疾病的共同和对比主题。科学通报,2001,29(5):528 - 528。10.1126 / science.1062013。
- 4.
Martin GB, Bogdanove AJ, Sessa G:了解植物抗病蛋白的功能。植物生物学年度回顾。2003, 54: 23-61。10.1146 / annurev.arplant.54.031902.135035。
- 5.
Meyers BC, Dickerman AW, Michelmore RW, Sivaramakrishnan S, Sobral BW, Young ND:植物抗病基因编码核苷酸结合超家族中一个古老而多样的蛋白质家族成员。植物学报,1999,20(3):317-332。10.1046 / j.1365 - 313 - x.1999.t01 - 1 - 00606. x。
- 6.
潘强,王文杰,王晓明,等:植物NBS-LRR抗性基因在玉米和玉米基因组中的分化进化。分子进化杂志。2000, 50(3): 203-213。
- 7.
王晓明,王晓明,王晓明,王晓明,王晓明。拟南芥nbs - lrr基因序列分析。植物细胞。2003, 15(4): 809-834。10.1105 / tpc.009308。
- 8.
陈晓明,陈晓明,陈晓明,等。植物NBS-LRR蛋白的研究进展。基因组生物学。2006, 7 (4): 212-10.1186/gb-2006-7-4-212。
- 9.
Meyers BC, Morgante M, Michelmore RW: TIR-X和TIR-NBS蛋白:拟南芥和其他植物基因组中编码的TIR-NBS- lrr蛋白的两个新家族。植物学报,2002,32(1):77-92。10.1046 / j.1365 - 313 x.2002.01404.x。
- 10.
李志刚,李志刚,李志刚:拟南芥抗性基因的重组与扩增。分子生物学杂志,2002,19(1):76-84。
- 11.
杨晓明,王晓明,王晓明,等:水稻抗性基因同源物的全基因组分析。应用理论,2004,109(7):1434-1447。10.1007 / s00122 - 004 - 1758 - x。
- 12.
Van der Biezen EA, Jones JD:植物抗病蛋白和基因对基因的概念。生物化学的发展趋势。1998,23(12): 454-456。10.1016 / s0968 - 0004(98) 01311 - 5。
- 13.
Innes RW:看守货物。对植物中央报警系统的新见解。植物生理学。2004, 35(2): 366 - 366。10.1104 / pp.104.040410。
- 14.
Dodds PN, Lawrence GJ, Catanzariti AM, Teh T, Wang CI, Ayliffe MA, Kobe B, Ellis JG:亚麻抗性基因和亚麻锈病致病基因的基因特异性和共同进化的直接蛋白质相互作用。美国科学院学报,2006,103(23):8888-8893。10.1073 / pnas.0602577103。
- 15.
Jones JD, Dangl JL:植物免疫系统。自然科学学报,2006,44(6):323-329。10.1038 / nature05286。
- 16.
沈qh, Saijo Y, Mauch S, Biskup C, Bieri S, Keller B, Seki H, Ulker B, Somssich IE, Schulze-Lefert P: MLA免疫受体核活性与孤立特异性和基础抗病应答的关系。科学通报,2007,29(4):344 - 344。10.1126 / science.1136372。
- 17.
Parniske M, hamnd - kosack KE, Golstein C, Thomas CM, Jones DA, Harrison K, Wulff BB, Jones JD:番茄Cf-4/9位点连续重复基因序列交换产生新的抗病特异性。光子学报,1997,21(6):821-832。10.1016 / s0092 - 8674(00) 80470 - 5。
- 18.
Thomas CM, Jones DA, Parniske M, Harrison K, Balint-Kurti PJ, Hatzixanthis K, Jones JD:番茄Cf-4基因对支点支孢杆菌抗性的特性鉴定确定了Cf-4和Cf-9的识别特异性序列。植物细胞。1997, 9(12): 2209-2224。10.2307 / 3870580。
- 19.
Botella MA、Parker JE、Frost LN、Bittner-Eddy PD、Beynon JL、Daniels MJ、Holub EB、Jones JD:拟南芥RPP1复合抗性位点的3个基因识别了不同的Peronospora寄生致病因子。植物细胞。1998, 10(11): 1847-1860。10.2307 / 3870908。
- 20.
McDowell JM, Dhandaydham M, Long TA, Aarts MG, Goff S, Holub EB, Dangl JL:拟南芥RPP8位点抗霜霉病进化的基因内重组和多样化选择。植物细胞。1998, 10(11): 1861-1874。10.2307 / 3870909。
- 21.
Meyers BC, Shen KA, Rohani P, Gaut BS, Michelmore RW:莴苣主要抗性位点的受体样基因存在分化选择。植物细胞。1998, 10(11): 1833-1846。10.2307 / 3870907。
- 22.
王丽丽,阮德龙,宋文武,Sideris S,陈琳,Pi LY,张s,张z, Fauquet C, Gaut BS, Whalen MC, Ronald PC: Xa21D编码一种具有富含亮氨酸重复结构域的受体样分子,决定种族特异性识别,并受适应性进化的影响。植物细胞。1998, 10(5): 765-779。10.2307 / 3870663。
- 23.
Ellis JG, Lawrence GJ, Luck JE, Dodds PN:鉴定亚麻锈病抗性基因L等位基因区域,确定基因对基因特异性的差异。植物细胞。1999, 11(3): 495-506。10.2307 / 3870876。
- 24.
Noel L, Moores TL, van Der Biezen EA, Parniske M, Daniels MJ, Parker JE, Jones JD:拟南芥RPP5复合抗病性位点的种内单倍型显著分化。植物细胞。1999, 11(11): 2099-2112。10.2307 / 3871012。
- 25.
Dodds PN, Lawrence GJ, Ellis JG: 6个氨基酸的改变决定了亚麻P和P2抗锈病特异性的差异。植物细胞。2001, 13(1): 163-178。10.2307 / 3871161。
- 26.
Wulff BB, Thomas CM, smoke M, Grant M, Jones JD:域交换和基因重组确定了番茄Cf-4和Cf-9蛋白诱导avr依赖性超敏反应所需的序列。植物细胞。2001, 13(2): 255-272。10.2307 / 3871275。
- 27.
黄春华,王志强,王志强,等:番茄抗病蛋白在线虫识别和细胞死亡信号传递中的分子内相互作用。植物保护学报,2003,34(5):585-593。10.1046 / j.1365 - 313 x.2003.01749.x。
- 28.
Luck JE, Lawrence GJ, Dodds PN, Shepherd KW, Ellis JG:亚麻抗锈病蛋白富亮氨酸重复序列以外的区域在特异性测定中发挥作用。植物细胞。2000, 12(8): 1367-1377。10.2307 / 3871136。
- 29.
Warren RF, Henk A, Mowery P, Holub E, Innes RW:拟南芥抗病基因RPS5富含亮氨酸重复结构域的突变部分抑制了多种细菌和霜霉病抗性基因。植物细胞。1998, 10(9): 1439-1452。10.2307 / 3870609。
- 30.
杨晓明,王晓明,王晓明,等。植物NBS-LRR蛋白结构域在植物抗病相关细胞死亡中的作用。EMBO期刊。2002, 21(17): 4511-4519。10.1093 / emboj / cdf453。
- 31.
杨晓明,杨晓明,杨晓明,等。植物抗病蛋白信号通路:NBS-LRR蛋白及其伙伴。植物生物学的最新观点。2004, 7(4): 391-399。10.1016 / j.pbi.2004.05.009。
- 32.
Jones DA, Takemoto D:植物先天免疫-直接和间接识别一般和特定的病原体相关分子。免疫学的最新观点。2004, 16(1): 48-62。10.1016 / j.coi.2003.11.016。
- 33.
van der Biezen EA, Jones JD: NB-ARC结构域:一种由植物抗性基因产物和动物细胞死亡调节器共享的新信号基元。中国生物医学工程学报,1998,8 (7):R226-7。10.1016 / s0960 - 9822(98) 70145 - 9。
- 34.
Tameling WI, Elzinga SD, Darmin PS, Vossen JH, Takken FL, Haring MA, Cornelissen BJ:番茄R基因产物I-2和MI-1是具有ATP酶活性的功能性ATP结合蛋白。植物细胞。2002, 14(11): 2929-2939。10.1105 / tpc.005793。
- 35.
Tameling WI, Vossen JH, Albrecht M, Lengauer T, Berden JA, Haring MA, Cornelissen BJ, Takken FL: I-2 NB-ARC结构域突变损害ATP水解导致自动激活。植物生理学。2006, 40(4): 344 - 344。10.1104 / pp.105.073510。
- 36.
Faigon-Soverna, Harmon FG, Storani L, Karayekov E, Staneloni RJ, Gassmann W, Mas P, Casal JJ, Kay SA, Yanovsky MJ:拟南芥光形态发生发育中TIR-NBS-LRR蛋白的本构性避光突变。植物细胞。2006,18(11): 2919-2928。10.1105 / tpc.105.038810。
- 37.
motiinho A, Hussey PJ, Trewavas AJ, Malho R: cAMP在花花管生长和定向过程中起第二信使作用。美国科学院学报,2001,98(18):10481-10486。10.1073 / pnas.171104598。
- 38.
Mes JJ, van Doorn AA, Wijbrandi J, Simons G, Cornelissen BJ, Haring MA:镰刀菌抗性基因I-2的表达与真菌封闭部位共定位。植物学报,2000,23(2):183-193。10.1046 / j.1365 - 313 x.2000.00765.x。
- 39.
Clark SE, Williams RW, Meyerowitz EM: CLAVATA1基因编码了一种可能控制拟南芥芽和花分生组织大小的受体激酶。光子学报,1997,34(4):575-585。10.1016 / s0092 - 8674(00) 80239 - 1。
- 40.
李娟,李志军:油菜素内酯信号转导的一种富亮氨酸重复受体激酶。细胞科学学报,1997,30(5):929-938。10.1016 / s0092 - 8674(00) 80357 - 8。
- 41.
Jeong S, Trotochaud AE, Clark SE:拟南芥CLAVATA2基因编码CLAVATA1受体样激酶稳定性所需的受体样蛋白。植物细胞。1999,11(10): 1925-1934。10.2307 / 3871087。
- 42.
王志强,王志强,王志强,王志强,王志强。植物甾体激素质膜受体BRI1的研究进展。自然科学,2001,41(1):1 -3。10.1038 / 35066597。
- 43.
Grant MR, Godiard L, Straube E, Ashfield T, Lewald J, Sattler A, Innes RW, Dangl JL:拟南芥RPM1基因的双特异性抗病结构。科学通报,1995,26(3):344 - 344。10.1126 / science.7638602。
- 44.
Dixon MS, Jones DA, Keddie JS, Thomas CM, Harrison K, Jones JD:番茄Cf-2抗病基因座由两个编码富含亮氨酸重复蛋白的功能基因组成。细胞科学学报,1996,84(3):451-459。10.1016 / s0092 - 8674(00) 81290 - 8。
- 45.
Salmeron JM, Oldroyd GE, Rommens CM, Scofield SR, Kim HS, Lavelle DT, Dahlbeck D, Staskawicz BJ:番茄Prf是植物抗病基因中富含亮氨酸重复类的成员,位于Pto激酶基因簇内。中国生物医学工程学报,1996,16(1):1 - 3。10.1016 / s0092 - 8674(00) 80083 - 5。
- 46.
Parker JE, Coleman MJ, Szabo V, Frost LN, Schmidt R, van der Biezen EA, Moores T, Dean C, Daniels MJ, Jones JD:拟南芥霜霉变抗性基因RPP5与N和L6的toll和白介素-1受体相似。植物细胞。1997, 9(6): 879-894。10.1105 / tpc.9.6.879。
- 47.
Milligan SB, Bodeau J, Yaghoobi J, Kaloshian I, Zabel P, Williamson VM:番茄根结线虫抗性基因Mi是亮氨酸拉链、核苷酸结合、富含亮氨酸重复序列的植物基因家族成员。植物细胞。1998, 10(8): 1307-1319。10.2307 / 3870642。
- 48.
沈佳,Chin DB, Arroyo-Garcia R, Ochoa OE, Lavelle DO, Wroblewski T, Meyers BC, Michelmore RW: Dm3是核苷酸结合位点富含亮氨酸重复编码基因组成性表达家族的成员之一。植物与微生物相互作用,2002,15(3):251-261。10.1094 / MPMI.2002.15.3.251。
- 49.
艾利夫,马立夫,陈志强,陈志强,陈志强,陈志强:亚麻L6抗锈病基因的转录本分析。植物学报,1999,17(3):287-292。10.1046 / j.1365 - 313 x.1999.00377.x。
- 50.
刘勇,刘志刚,刘志刚。烟草花叶病毒抗性基因的克隆与鉴定。中国烟草科学,2002,23(3):379 - 379。10.1046 / j.1364-3703.2002.00110.x。
- 51.
Anderson PA, Lawrence GJ, Morrish BC, Ayliffe MA, Finnegan EJ, Ellis JG:亚麻抗锈病基因M失活与富亮氨酸重复编码区重复单元丢失相关。植物细胞。1997, 9(4): 641-651。10.2307 / 3870513。
- 52.
张晓超,Gassmann W: RPS4介导的抗病需要RPS4转录本与全长和截断的开放阅读框架的结合。植物细胞。2003, 15(10): 2333-2342。10.1105 / tpc.013474。
- 53.
Borhan MH, Holub EB, Beynon JL, Rozwadowski K, Rimmer SR:拟南芥TIR-NB-LRR基因RAC1对白锈病(Albugo candida,白锈病)具有抗性,依赖于EDS1而不是PAD4。分子植物与微生物相互作用,2004,17(7):711-719。10.1094 / MPMI.2004.17.7.711。
- 54.
Schornack S, Ballvora A, Gurlebeck D, Peart J, Baulcombe D, Ganal M, Baker B, Bonas U, Lahaye T:番茄抗性蛋白Bs4是一种预测的非核tir - rb - lrr蛋白,介导对AvrBs4严重截断衍生物和过表达AvrBs3的防御反应。植物学报,2004,37(1):46-60。10.1046 / j.1365 - 313 x.2003.01937.x。
- 55.
Ferrier-Cana E, Macadre C, Sevignac M, David P, Langin T, Geffroy V:菜豆CC-NBS-LRR基因JA1tr的转录后修饰产生了7个不同的转录本。应用理论,2005,110(5):895-905。10.1007 / s00122 - 004 - 1908 - 1。
- 56.
蔡d, Kleine M, Kifle S, Harloff HJ, Sandal NN, Marcker KA, Klein-Lankhorst RM, Salentijn EM, Lange W, Stiekema WJ, Wyss U, Grundler FM, Jung C:甜菜线虫抗性基因的位置克隆。科学通报,1997,27(4):344 - 344。10.1126 / science.275.5301.832。
- 57.
Yoshimura S, Yamanouchi U, Katayose Y, Toki S, Wang ZX, Kono I, Kurata N, Yano M, Iwata N, Sasaki T:细菌接种诱导水稻抗枯萎病基因Xa1的表达。美国国家科学院学报,1998,95(4):1663-1668。10.1073 / pnas.95.4.1663。
- 58.
Halterman DA, Wei F, Wise RP:白粉病诱导的Mla mrna是交替剪接的,包含多个上游开放阅读框。植物生理学。2003, 31(2): 353 - 353。10.1104 / pp.014407。
- 59.
顾凯,杨波,田东,吴玲,王东,Sreekala C,杨峰,褚震,王gl, White FF, Yin Z:一种iii型效应子诱导水稻抗病性的R基因表达自然科学进展,2005,35(4):344 - 344。10.1038 / nature03630。
- 60.
刘志刚,刘志刚,刘志刚,刘志刚,刘志刚。玉米Rp1-D抗锈病单倍型及其突变体的分子鉴定。植物细胞。1999, 11(7): 1365-1376。10.2307 / 3870755。
- 61.
王志霞,Yano M, Yamanouchi U, Iwamoto M, Monna L, Hayasaka H, Katayose Y, Sasaki T:水稻稻瘟病抗性Pib基因属于植物抗病基因中核苷酸结合和富亮氨酸重复序列类。植物学报,1999,19(1):55-64。10.1046 / j.1365 - 313 x.1999.00498.x。
- 62.
王志霞,山内U, Katayose Y, Sasaki T, Yano M:水稻Pib抗稻瘟病基因家族的表达在有利于侵染的环境条件和触发植物二级防御的化学信号的作用下上调。植物分子生物学。2001, 47(5): 653-661。10.1023 /: 1012457113700。
- 63.
Donson J, Fang Y, Espiritu-Santo G,邢W, Salazar A, Miyamoto S, Armendarez V, Volkmuth W:基于转录谱分析的综合基因表达分析。植物分子生物学。2002, 48(1-2): 75-97。10.1023 /: 1013722224489。
- 64.
基因表达谱:成瘾研究的方法挑战、结果和前景。化学与物理学报,2002,22(1):1- 6。10.1016 / s0009 - 3084(02) 00160 - 3。
- 65.
Adams MD, Kerlavage AR, Fleischmann RD, Fuldner RA, Bult CJ, Lee NH, Kirkness EF, Weinstock KG, Gocayne JD, White O, Sutton G, Blake JA, Brandon RC, Chiu MW, Clayton RA, Cline RT, Cotton MD, Earle-Hughes J, Fine LD, FitzGerald LM, FitzHugh WM, Fritchman JL, Geoghagen NSM, Glodek A, Gnehm CL, Hanna MC, Hedblom E, Hinkle Jr. PS, Kelley JM, Klimek KM,等:基于8300万个cDNA序列核苷酸的人类基因多样性和表达模式的初步评估。自然科学,1995,37(增刊):344 - 344。
- 66.
张玲,张志刚,张志刚,张志刚,张志刚:基因表达序列分析。科学通报,1995,27(4):344 - 344。10.1126 / science.270.5235.484。
- 67.
自然生物技术。2000, 18(6): 630-634。10.1038/76469。
- 68.
美国科学院学报,2000,97(4):1665-1670。10.1073 / pnas.97.4.1665。
- 69.
Rensink WA, Buell CR:植物基因组学的微阵列表达分析资源。植物科学发展趋势。2005,10(12): 603-609。10.1016 / j.tplants.2005.10.003。
- 70.
高等植物DNA微阵列分析。组学学报,2006,10(4):455-473。10.1089 / omi.2006.10.455。
- 71.
Armstrong NJ, van de Wiel MA:微阵列数据分析:使用基因表达数据从假设到结论。中国生物医学工程学报,2004,26(5):279-290。
- 72.
Clarke JD, Zhu T:转录组微阵列分析是理解生物系统的垫脚石:实际考虑和展望。植物学报,2006,45(4):630-650。10.1111 / j.1365 - 313 x.2006.02668.x。
- 73.
NibLrrs。[http://niblrrs.ucdavis.edu]
- 74.
拟南芥MPSS数据库。[http://mpss.udel.edu/at/]
- 75.
Meyers BC, Lee DK, Vu TH, Tej SS, Edberg SB, Matvienko M, Tindell LD:拟南芥MPSS。定量表达分析的在线资源。植物生理学。2004, 35(2): 339 - 344。10.1104 / pp.104.039495。
- 76.
Meyers BC, Tej SS, Vu TH, Haudenschild CD, Agrawal V, Edberg SB, Ghazal H, Decola S: MPSS在拟南芥全基因组转录分析中的应用。基因组研究。2004, 14(8): 1641-1653。10.1101 / gr.2275604。
- 77.
Meyers BC, Vu TH, Tej SS, Ghazal H, Matvienko M, Agrawal V, Ning J, Haudenschild CD:拟南芥转录复杂性的大规模并行序列分析。自然生物技术。2004, 22(8): 1006-1011。10.1038 / nbt992。
- 78.
李晓明,李志刚,李志刚,李志刚,李志刚,李志刚。拟南芥鞭毛蛋白对细菌抗性的影响。自然科学进展,2004,29(4):344 - 344。10.1038 / nature02485。
- 79.
Kliebenstein DJ, West MA, van Leeuwen H, Kim K, Doerge RW, Michelmore RW, St Clair DA:拟南芥基因表达多样性的基因组调查。中国生物医学工程学报,2006,32(2):339 - 339。10.1534 / genetics.105.049353。
- 80.
品行端正。[http://elp.ucdavis.edu/]
- 81.
施普林格PS, McCombie WR, Sundaresan V, Martienssen RA:拟南芥mcm2 -3-5样必需基因PROLIFERA的基因陷阱标记。科学通报,1995,26(3):357 - 357。10.1126 / science.7754372。
- 82.
孙瑞珊,王晓明,王晓明,王晓明,王晓明,王晓明,王晓明。植物发育过程中基因转移因子的研究进展。基因与发育。1995, 9(14): 1797-1810。10.1101 / gad.9.14.1797。
- 83.
Martienssen RA:功能基因组学:用转座子探测植物基因功能和表达。美国国家科学院学报,1998,95(5):2021-2026。10.1073 / pnas.95.5.2021。
- 84.
Parinov S, Sevugan M, Ye D, Yang WC, Kumaran M, Sundaresan V:拟南芥解离插入系侧翼序列分析:逆向遗传数据库。植物细胞。1999, 11(12): 2263-2270。10.2307 / 3870953。
- 85.
拟南芥基因陷阱网站在冷泉港实验室。[http://genetrap.cshl.org/]
- 86.
分子农业生物学研究所系。[http://www.arabidopsis.org/abrc/ima.jsp]
- 87.
顾Q, Ferrandiz C, Yanofsky MF, Martienssen R:拟南芥果实发育过程中的MADS-box基因介导细胞分化。发展(剑桥,英国)。1998, 25(8): 1509-1517。
- 88.
王晓明,王晓明,王晓明,等:拟南芥转座子标记及其根系发育的研究。重力空间生物通报,1998,11(2):79-87。
- 89.
TAIR。[http://www.arabidopsis.org/]
- 90.
Maleck K, Levine A, Eulgem T, Morgan A, Schmid J, Lawton KA, Dangl JL, Dietrich RA:拟南芥系统性获得性抗性转录组研究。自然遗传学。2000, 26(4): 403-410。10.1038/82521。
- 91.
陶艳,谢忠,陈伟,Glazebrook J,常海峰,韩波,朱涛,邹刚,Katagiri F:拟南芥与丁香假单胞菌亲和和不亲和互作的数量性质。植物细胞。2003, 15(2): 317-330。10.1105 / tpc.007591。
- 92.
Burch-Smith TM, Schiff M, Caplan JL, Tsao J, Czymmek K, Dinesh-Kumar SP: TIR结构域在病原衍生激发子中的新作用。公共科学图书馆,2007,5 (3):e68-10.1371/journal.pbio.0050068。
- 93.
Peart JR, Mestre P, Lu R, Malcuit I, Baulcombe DC: CC-NB-LRR蛋白NRG1与TIR-NB-LRR蛋白N共同介导烟草花叶病毒抗性。动物学报,2005,15(10):968-973。10.1016 / j.cub.2005.04.053。
- 94.
Gabriels SH, Vossen JH, Ekengren SK, van Ooijen G, Abd-El-Haliem AM, van den Berg GC, Rainey DY, Martin GB, Takken FL, de Wit PJ, Joosten MH:一种由细胞外和细胞内抗性蛋白介导的HR信号通路所需的NB-LRR蛋白。植物学报,2007,30(1):14-28。10.1111 / j.1365 - 313 x.2007.03027.x。
- 95.
马晓峰,关志,赵海峰,王晓峰,王晓峰,王晓峰,王晓峰。拟南芥抗黄瓜花叶病毒全基因组芯片研究。植物分子生物学。2004, 55(4): 501-520。10.1007 / s11103 - 004 - 0439 - 0。
- 96.
拟南芥TIGR的剪接变异。[http://www.tigr.org/tdb/e2k1/ath1/altsplicing/splicing_variations.shtml]
- 97.
王宝宝,Brendel V:植物选择性剪接的全基因组比较分析。美国国家科学院学报,2006,103(18):7175-7180。10.1073 / pnas.0602039103。
- 98.
Jordan T, Schornack S, Lahaye T: toll样植物抗性蛋白转录本的选择性剪接——与先天免疫的功能相关性?植物科学发展趋势。2002, 7(9): 392-398。10.1016 / s1360 - 1385(02) 02311 - 7。
- 99.
丁宁-库马SP,贝克。j:烟草花叶病毒抗性基因的研究进展。美国国家科学院学报,2000,97(4):1908-1913。10.1073 / pnas.020367497。
- One hundred.
Gassmann W, Hinsch ME, Staskawicz BJ:拟南芥RPS4抗菌基因是TIR-NBS-LRR抗病基因家族成员。植物学报,1999,20(3):265-277。10.1046 / j.1365 - 313 - x.1999.t01 - 1 - 00600. x。
- 101.
李志强,李志强,李志强:拟南芥RPM1抗病基因产物是一种与拟南芥超敏反应同时降解的外周质膜蛋白。美国国家科学院学报,1998,95(26):15849-15854。10.1073 / pnas.95.26.15849。
- 102.
Century KS, Lagman RA, Adkisson M, Morlan J, Tobias R, Schwartz K, Smith A, Love J, Ronald PC, Whalen MC:水稻xa21介导的抗病性的发育控制。植物学报,1999,20(2):231- 231。10.1046 / j.1365 - 313 x.1999.00589.x。
- 103.
van der Velden AW, Thomas AA: mRNA的5'非翻译区在发育过程中翻译调节中的作用。国际生物化学与细胞生物学杂志。1999, 31(1): 87-106。10.1016 / s1357 - 2725(98) 00134 - 4。
- 104.
Willis AE:生长因子和原癌基因表达的翻译控制。国际生物化学与细胞生物学杂志。1999, 31(1): 73-86。10.1016 / s1357 - 2725(98) 00133 - 2。
- 105.
Halterman D,周飞,魏飞,Wise RP, Schulze-Lefert P: MLA6 - NBS-LRR蛋白对大麦和小麦小麦青枯病(Blumeria graminis F . sp. hordei)的抗性特异性。植物学报,2001,25(3):335-348。10.1046 / j.1365 - 313 x.2001.00982.x。
- 106.
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ:基本的局部对齐搜索工具。分子生物学杂志。1990, 215(3): 403-410。
- 107.
Seki M, Narusaka M, Kamiya A, Ishida J, Satou M, Sakurai T, Nakajima M, Enju A, Akiyama K, Oono Y, Muramatsu M, Hayashizaki Y, Kawai J, Carninci P, Itoh M, Ishii Y, Arakawa T, Shibata K, Shinagawa A, Shinozaki K:拟南芥cDNA全长集的功能注释。科学通报,2002,29(6):531 - 531。10.1126 / science.1071006。
- 108.
TAIR爆炸。[http://www.arabidopsis.org/Blast/]
- 109.
Pearson WR, Lipman DJ:生物序列比较的改进工具。美国科学院学报,1988,85(8):2444-2448。10.1073 / pnas.85.8.2444。
- 110.
Affymetrix。[http://www.affymetrix.com]
- 111.
鞭毛蛋白实验原始阵列数据。[http://www.ebi.ac.uk/microarray-as/aer/dataselection?expid=295433876]
- 112.
ELP者。[http://elp.ucdavis.edu/parental_survey_ELP_finder.htm]
- 113.
Klimyuk VI, Carroll BJ, Thomas CM, Jones JD:碱处理快速制备植物材料的可靠PCR分析。植物学报,1998,3(3):493-494。10.1046 / j.1365 - 313 - x.1993.t01 - 26 - 00999. x。
- 114.
Jefferson RA, Kavanagh TA, Bevan MW: GUS融合:β -葡萄糖醛酸酶作为高等植物中敏感和多功能的基因融合标记。EMBO期刊。1987, 6(13): 3901-3907。
- 115.
Toriyama K, Thorsness MK, Nasrallah JB, Nasrallah ME:芸苔属S位点基因启动子在转基因拟南芥花药绒毡层中指导孢子体表达。发育生物学。1991,(2): 427-431。10.1016 / 0012 - 1606 (91) 90094 - j。
- 116.
Bradford MM:一种快速和灵敏的方法定量微量蛋白质利用蛋白质-染料结合的原理。分析生物化学。一九七六年,二十七:二百四八-二百四五。10.1016 / 0003 - 2697(76) 90527 - 3。
- 117.
田木,Dahlbeck D, Staskawicz B, Keen NT:丁香假单胞菌两个毒力基因的克隆及表达。glycinea。细菌学杂志。1988, 170(10): 4846-4854。
- 118.
李志强,李志强,李志强,等。拟南芥对丁香假单胞菌的抗性分析。植物学报,1998,1(3):379 - 379。
- 119.
Century KS, Holub EB, Staskawicz BJ: NDR1,拟南芥对细菌和真菌病原体的抗病所必需的基因座。美国国家科学院学报,1995,92(14):6597-6601。10.1073 / pnas.92.14.6597。
- 120.
Parker JE, Holub EB, Frost LN, Falk A, Gunn ND, Daniels MJ:拟南芥eds1突变的特征,该突变抑制了几个不同RPP基因所指定的对Peronospora寄生的抗性。植物细胞。1996, 8(11): 2033-2046。10.2307 / 3870410。
- 121.
Bent AF, Kunkel BN, Dahlbeck D, Brown KL, Schmidt R, Giraudat J, Leung J, Staskawicz BJ:拟南芥RPS2:一个富含亮氨酸的重复类植物抗病基因。科学通报,1994,26(3):344 - 344。10.1126 / science.8091210。
- 122.
Mindrinos M, Katagiri F, Yu GL, Ausubel FM:拟南芥抗病基因RPS2编码含有核苷酸结合位点和富含亮氨酸重复序列的蛋白质。细胞科学学报,1994,18(6):1089-1099。10.1016 / 0092 - 8674(94) 90282 - 8。
- 123.
Innes RW, Bent AF, Kunkel BN, Bisgrove SR, Staskawicz BJ:毒性基因avrRpt2的分子分析和所有已知丁香假单胞菌毒性基因共同调控序列的鉴定。细菌学杂志。1993, 17(3): 457 - 457。
- 124.
Whalen MC, Innes RW, Bent AF, Staskawicz BJ:拟南芥丁香假单胞菌病原菌的鉴定及其对大豆和拟南芥的致病力。植物细胞。1991, 3(1): 49-59。10.2307 / 3869199。
- 125.
Hinsch M, Staskawicz B:拟南芥抗病新位点RPs4的鉴定及其致病基因avrRps4的克隆。pisi。植物微生物相互作用。1996,9(1):55-61。
- 126.
Aarts N, Metz M, Holub E, Staskawicz BJ, Daniels MJ, Parker JE:拟南芥抗病性基因对EDS1和NDR1的不同需求确定了至少两条R基因介导的信号通路。美国国家科学院学报,1998,95(17):10306-10311。10.1073 / pnas.95.17.10306。
- 127.
杨丽娟,张志刚,张志刚,张志刚,张志刚,杨志刚。丁香假单胞菌基因间相互作用的研究。菜豆和菜豆。植物与微生物相互作用。1991,4(6):553-562。
- 128.
Simonich MT, Innes RW:拟南芥中对丁香假单胞菌avrph3基因特异的抗病基因。phaseolicola。植物与微生物相互作用,1995,8(4):637-640。
- 129.
Rehmany AP, Gordon A, Rose LE, Allen RL, Armstrong MR, Whisson SC, Kamoun S, Tyler BM, Birch PR, Beynon JL:两株拟南芥RPP1抗性基因对霜霉病致病基因的差异识别。植物细胞。2005, 17(6): 1839-1850。10.1105 / tpc.105.031807。
- 130.
van der Biezen EA, Freddie CT, Kahn K, Parker JE, Jones JD:拟南芥RPP4是TIR-NB-LRR基因RPP5多基因家族的一员,通过多种信号成分赋予霜霉变抗性。植物学报,2002,29(4):439-451。10.1046 / j.0960-7412.2001.01229.x。
- 131.
McDowell JM, Cuzick A, Can C, Beynon J, Dangl JL, Holub EB:拟南芥抗霜霉病(Peronospora parastica)基因对NDR1、EDS1、NPR1和水杨酸积累的功能需求不同。植物学报,2000,22(6):523-529。10.1046 / j.1365 - 313 x.2000.00771.x。
- 132.
Cooley MB, Pathirana S, Wu HJ, Kachroo P, Klessig DF:拟南芥HRT/RPP8抗性基因家族成员对病毒和卵菌病原体具有抗性。植物细胞。2000, 12(5): 663-676。10.2307 / 3870993。
- 133.
Takahashi H, Miller J, Nozaki Y, Takeda M, Shah J, Hase S, Ikegami M, Ehara Y, Dinesh-Kumar SP: RCY1,拟南芥RPP8/HRT家族抗性基因,对黄瓜花叶病毒的抗性需要水杨酸、乙烯和一种新的信号转导机制。植物学报,2002,32(5):655-667。10.1046 / j.1365 - 313 x.2002.01453.x。
- 134.
Bittner-Eddy PD, Crute IR, Holub EB, Beynon JL: RPP13在拟南芥中是一个简单的等位基因位点,它决定了Peronospora寄生对霜霉病的抗性。植物学报,2000,21(2):177-188。10.1046 / j.1365 - 313 x.2000.00664.x。
- 135.
Bittner-Eddy PD, Beynon JL:拟南芥霜霉病抗性基因RPP13-Nd独立于NDR1和EDS1发挥作用,不需要水杨酸的积累。植物与微生物相互作用。2001,14(3):416-421。10.1094 / MPMI.2001.14.3.416。
- 136.
Allen RL, Bittner-Eddy PD, grenille - briggs LJ, Meitz JC, Rehmany AP, Rose LE, Beynon JL:拟南芥和霜霉病的宿主-寄生虫共同进化冲突。科学通报,2004,30(3):379 - 379。10.1126 / science.1104022。
- 137.
Deslandes L, Olivier J, Theulieres F, Hirsch J, Feng DX, Bittner-Eddy P, Beynon J, Marco Y:拟南芥对番茄拉尔斯顿菌的抗性是由隐性RRS1-R基因决定的,它是一个新的抗性基因家族的成员。美国科学院学报,2002,99(4):2404-2409。10.1073 / pnas.032485099。
- 138.
Deslandes L, Olivier J, pepeters N, Feng DX, Khounlotham M, Boucher C, Somssich I, Genin S, Marco Y: RRS1-R(一种抗细菌性枯萎病的蛋白)与PopP2(一种针对植物核的III型效应子)的物理相互作用。美国科学院学报,2003,100(13):8024-8029。10.1073 / pnas.1230660100。
- 139.
Sinapidou E, Williams K, Nott L, Bahkt S, Tor M, Crute I, Bittner-Eddy P, Beynon J:拟南芥对Peronospora寄生分离物Cala2的抗性需要两个TIR:NB:LRR基因。植物学报,2004,38(6):898-909。10.1111 / j.1365 - 313 x.2004.02099.x。
- 140.
Grant JJ, Chini A, Basu D, Loake GJ:拟南芥NBS-LRR基因ADR1的靶向激活标记传递对毒性病原体的抗性。植物与微生物相互作用,2003,16(8):669-680。10.1094 / MPMI.2003.16.8.669。
- 141.
stal J, Kaliff M, Bohman S, Dixelius C:拟南芥TIR-NB-LRR基因对黑腿病(leptosperia maculans)的抗性分析。植物学报,2006,46(2):218-230。10.1111 / j.1365 - 313 x.2006.02688.x。
确认
我们感谢Dean Lavelle的技术援助、Huaqin Xu的生物信息学支持以及Belinda Martineau的编辑协助。这项工作得到了美国国家科学基金会植物基因组计划奖# 9975971的支持,r.w.m., A.F.B.等.并利用由美国国家科学基金会2010年项目奖# 0115109授予d.a.s C, R.W.M.生成的微阵列数据等,以及由美国国家科学基金会植物基因组研究奖#0110528授予B.C.M.生成的MPSS数据
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
XT进行了大部分的实验,分析了数据并起草了论文。BCM协助实验设计、数据分析和撰写。AK为拟南芥EST和基因组序列分析及数据可视化提供了生物信息学支持。MALW和DAStC参与了拟南芥种质和SA处理的微阵列实验,并协助数据解释。MM贡献了MPSS数据。AFB为实验设计和写作做出了贡献。RWM对整个实验设计、数据解释和论文写作都做出了贡献。
电子辅助材料
12870 _2007_197_moesm1_esm.xls
附加文件1:nbs - lrr编码及相关基因表达分析的详细总结。工作表1A提供了工作表1B和1C的数据结构。工作表1B提供了cc - nbs - lrr编码及相关基因的完整表达数据。工作表1C提供了tir - nbs - lrr编码及相关基因的完整表达数据。资料亦可在网上查阅[73].(xls2mb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
谭,X,迈耶斯,B.C,科兹克,A。et al。拟南芥富亮氨酸核苷酸结合位点重复编码及其相关基因的全局表达分析。BMC植物生物学7,56(2007)。https://doi.org/10.1186/1471-2229-7-56
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-7-56
关键字
- 水杨酸
- 水杨酸处理
- 大规模并行签名测序
- 增强陷阱线
- 大规模并行签名测序数据