摘要
背景
生长素/吲哚-3-乙酸(Aux/IAA)和生长素反应因子(ARF)转录因子是植物生长素反应的关键调控因子。我们确定了两个基因家族的基因组合杨树并进行了比较基因组分析拟南芥和米饭。
结果
总共35个辅助/ IAA和39东盟地区论坛基因鉴定杨树基因组。系统发育分析表明,两种双子叶植物的多个Aux/IAA和ARF亚群具有不同的扩张或收缩。激活剂东盟地区论坛研究发现,基因在大脑中被过度表达了两倍杨树基因组。PoptrIAA和PoptrARF基因家族似乎已经扩大,由于高片段和低串联重复事件。此外,表达研究表明,基因在扩大PoptrIAA3子组显示差分表达式。
结论
本研究探讨的保护和分歧的程度,在结构和演变杨树辅助/ IAA和东盟地区论坛基因家族拟南芥和米饭。本文报道的基因家族分析将有助于开展未来的功能基因组学研究,以了解这些大基因家族的分子作用如何转化为多种具有生物学意义的生长素效应。
背景
辅助/ IAAS是生长素反应基因,编码核定位蛋白[1].Aux/IAA蛋白通常有四个特征结构域;一个n端抑制结构域,一个参与蛋白质稳定性的邻近结构域,以及两个c端结构域(CTD), III和IV,通过这些结构域,Aux/IAA蛋白与Aux/IAA或ARF蛋白形成同源和异源二聚体[2].大多数ARF蛋白含有一个n端b3样DNA结合域,其中包括一个ARF家族特异性结构域,一个赋予激活或抑制活性的可变中间区域,以及在Aux/IAA中也发现的结构域III和IV [3.].无论生长素状态如何,ARF蛋白都能够结合生长素响应顺式元件(AuxRE;TGTCTC)存在于生长素应答基因编码序列的上游[4,5].Aux/IAA蛋白通过结构域III和IV与dna结合的ARF伴侣蛋白结合并抑制ARF活性。在生长素激活状态下,Aux/IAA蛋白通过与生长素修饰的SCF相互作用而泛素化TIR1并随后被26S蛋白酶体降解[6- - - - - -8].虽然一些ARFs具有典型的谷氨酰胺(Q)丰富的中间区域,从而赋予激活剂活性,但发现具有脯氨酸,丝氨酸和苏氨酸丰富的中间区域的ARFs与抑制因子活性相关[3.,9].生长素调控活性的复杂性部分是由于大尺寸的东盟地区论坛和辅助/ IAA基因家族,以及arf之间激活或抑制活性的变化,异源二聚化亲和力,表达模式以及生长素介导的转录和转录后调控。
尽管已知Aux/IAAs和ARFs影响顶端优势、维管发育、热带运动、根生长、组织和器官模式以及花和果实发育[10,但仍有许多问题悬而未决。例如,这些基因家族在森林树种中基本上未被确定,一年生和多年生木本植物之间基因家族的保护程度尚不清楚。此外,Aux/IAA和ARF相互作用和调控的机制尚不完全清楚,它们在细胞和整个生物体中的作用仍有待研究。的识别辅助/ IAA和东盟地区论坛来自不同模式植物的基因家族是制定与生理和发育过程有关的更好假设的必要步骤。最近的排序杨树基因组提供了一个额外的参考基因组,用于测试先前通过功能基因组研究获得的生长素信号转导事件的推断拟南芥.
本文总结了基于生物信息学的比较基因组研究的结果,以确定总数量辅助/ IAA和东盟地区论坛基因在杨树,以预测蛋白质结构域结构,并评估之间的保护和分歧程度杨树,拟南芥还有水稻基因家族。利用靶向RT-PCR和全基因组微阵列分析以及EST数据库调查来探索紧密同属同源基因的差异表达。此外,我们已经反映了可能的差异模式的保留,损失和扩增的重复同源基因的影响。
结果与讨论
鉴定与序列分析杨树辅助/ IAA基因
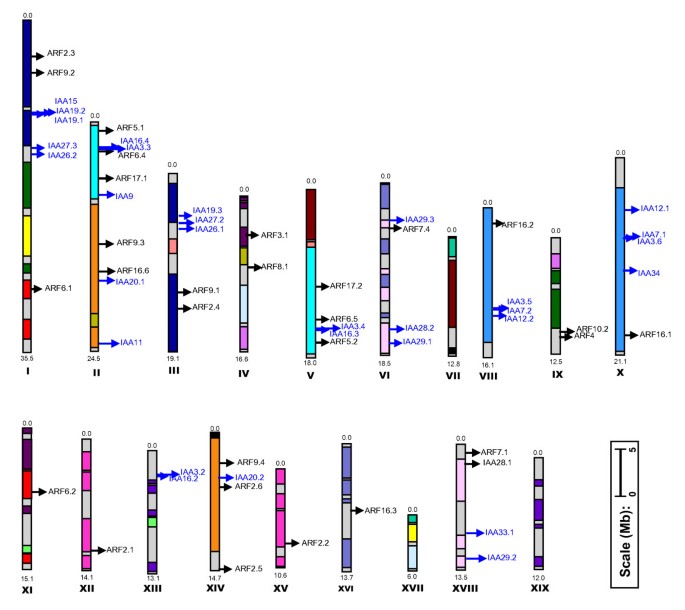
的杨树基因组有35人预测辅助/ IAA基因(以下简称基因)PoptrIAA),相比之下,人类有29个基因拟南芥基因组(以下简称基因组AtIAA) [11]和水稻基因组中的31个基因(以下简称为OsIAA) [12].在网上基因位点的定位表明PoptrIAA基因存在于19条染色体中的10条(图2)1;参见附加文件1).
的染色体位置PoptrIAA和PoptrARF基因.刻度表示5 Mb染色体距离。颜色表示大多数连锁基团的嵌合性质。共色系指同源的基因组片段,被认为产生于水螅类基因组重复65mya,并由两条染色体共享[13]。染色体数(连锁群号I-XIX)和大小(Mb)显示在每条染色体的底端。PoptrIAA和PoptrARF基因分别用蓝色和黑色字体表示。4PoptrIAA和11PoptrARF基因驻留在未组装的支架上(见附加文件)1).
任何分析杨树基因家族的进化必须考虑到属最近进化中最重要的事件;发生在大约65亿年前的全基因组复制事件,在大约92%的基因组中仍可检测到[13].基于重复基因的年龄估计和同源微同步分析(见附加文件)2), 14PoptrIAA基因对(占总基因的80%)在与最近的类水螅重复事件相关的片段重复区域内。有趣的是,在最近的全基因组或片段重复集群中,6对串联重复被表示为“古代串联基因”。这15个PoptrIAA基因(约占总数的43%)表现在7个不同的串联重复基因簇中,其中6个簇含有2个串联基因,1个簇含有3个串联基因(PoptrIAA15,PoptrIAA19.1和PoptrIAA19.2).
有可能一些表面上密切相关的基因实际上是来自未组装的单倍型的等位基因,这是这个高度杂合的基因组的猎枪组装的潜在产物。例如,PoptrIAA3.1和PoptrIAA3.2与它们的串联复制体紧密结合,PoptrIAA16.1和PoptrIAA162,PoptrIAA3.1和PoptrIAA16.1基因位于当前未组装的支架(scaffold_70)上。然而,表面上的同源物在氨基酸水平上以及在合成块中的侧翼基因顺序和身份上是不同的,这反对将支架分类为单倍型。
进一步的保守结构域分析和预测蛋白的多序列比对表明,一些杨树基因在保守域结构中包含修改(参见附加文件)3.,4和5).基于MEME-MAST蛋白基序分析,发现PoptrIAA29.1、PoptrIAA29.2和PoptrIAA29.3中缺失结构域I拟南芥直接同源,AtIAA29。同样,在PoptrIAA33.1、PoptrIAA33.2和PoptrIAA34以及AtIAA33和AtIAA34中,域I和II似乎缺失。多序列比对表明,AtIAA34 (LxLxL)中报道的“idLgLsLrt”序列为结构域I基序[14]与AtIAA14, AtIAA15, AtIAA16和AtIAA17一起保存(参见附加文件)3.),其中报告的领域I的一致序列为“TELxLxLPG”[14].
Domain II启用SCFTIR1依赖性蛋白酶体介导的Aux/IAA降解[15].Aux/IAA蛋白的快速基础降解率和生长素反应性被发现与高度保守的结构域II基序'VGWPPI/V'的改变有关[15]和域I和域II之间的“KR”基序[16].研究拟南芥表明VGWPPV基序的突变使抑制蛋白稳定[17- - - - - -23].对PoptrIAA7.1、PoptrIAA20.1、PoptrIAA 20.2、PoptrIAA 33.1、PoptrIAA 33.2、PoptrIAA 34和AtIAA20、AtIAA30、AtIAA31、AtIAA32、AtIAA33和AtIAA34的全长度氨基酸序列进行多序列比对发现,“VGWPPI/V”和“KR”基序序列均存在差异。仅在PoptrIAA27.1、PoptrIAA26.2和AtIAA28序列中观察到保守的“KR”基序存在偏差。然而,PoptrIAA7.1被发现是唯一的,因为它包含域II区域的串联复制(参见附加文件)5).中未检测到此独特功能辅助/ IAA从任何其他属报告的基因包括拟南芥,选用,葡萄属,烟草和Coffea,但在GenBank ESTs中发现来自P.trichocarpa,P.摘要,P.美洲山杨,p . tremula×美洲山杨[24],P.阿尔巴×tremula,P.trichocarpa×摘要和P.fremontii×angustifolia表明这是一个杨树特殊技能的习得。
的比较分析杨树和拟南芥辅助/ IAA基因家族
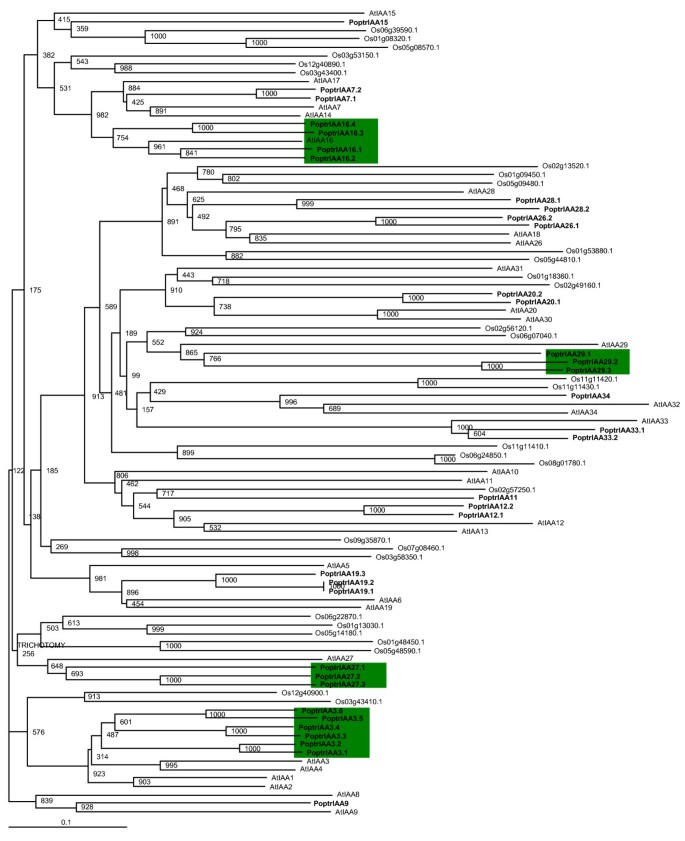
在分子水平上利用所有可用的和预测的系统发育重建杨树,拟南芥和水稻的Aux/IAA氨基酸序列显示4组PoptrIAAs (PoptrIAA3,16,27和29)已扩展为每个包含三个或更多成员(图2)2).三个AtIAAS中没有具有代表性的序列同源物杨树,表示不同的获得或延续辅助/ IAAS唯一于拟南芥以及它相对较近的祖先。
有四个同系物AtIAA16,poptriaa16.1 - 16.4的预测。杨树基因组。系统发育分组表明缺乏AtIAA16来自大米的同源物。PoptrIAA16.3和PoptrIAA16.4可能是在拟南芥中丢失的祖先基因的同同源物。这些同同源物具有完整的保守域和支持其功能的EST证据(参见附加文件)1和5).根据EST数据,PoptrIAA16.1在木材形成过程中表达(形成层和张力木材),而PoptrIAA16.3和16.4似乎在嫩叶中表达(见附加文件)6).一个PoptrIAA16.1 -如EST,以前报道过杂交杨树在木材形成和a的背景下表达PoptrIAA16.3-like EST被证明在细胞分裂和扩张中表达[24].我们的全基因组微阵列数据部分支持了这些发现,这些数据表明PoptrIAA16.1在木质部、韧皮部、皮层、根和种子中表达(见附加文件7,8和9).
然而,没有阵列证据支持的表达PoptrIAA16.3和PoptrIAA16.4但在幼嫩的叶片组织中PoptrIAA16.3在茎尖、顶端营养芽、两性新生生殖芽和老叶中表达。
已知两个Aux/IAA蛋白IAA26/PAP1(光敏色素相关蛋白1)和IAA27与光敏色素A (PHYA)相互作用[25,26]和TMV复制酶[1,27,28].杨树有三个类似iaa27的辅助/ IAA基因。EST和微阵列数据显示,该基因在茎部分生组织、花蕾以及休眠和活动形成层中表达6,7,8和9).可能与PHYA蛋白相互作用的亲和力表明PoptrIAA27在外部刺激依赖性形成层激活或生长状态中起作用。PoptrIAA26.1和PoptrIAA26.2基因与AtIAA18和AtIAA26在茎部分生组织、幼叶和衰老叶、雄絮和花蕾中表达。的RT-PCR调查PoptrIAA26.1和PoptrIAA26.2这些基因在幼叶中表达量最高。由于AtIAA26和AtIAA27蛋白显示出与PHYA的结合亲和力,很可能这些杨树序列同源物还参与介导各种树木发育过程的光调节。
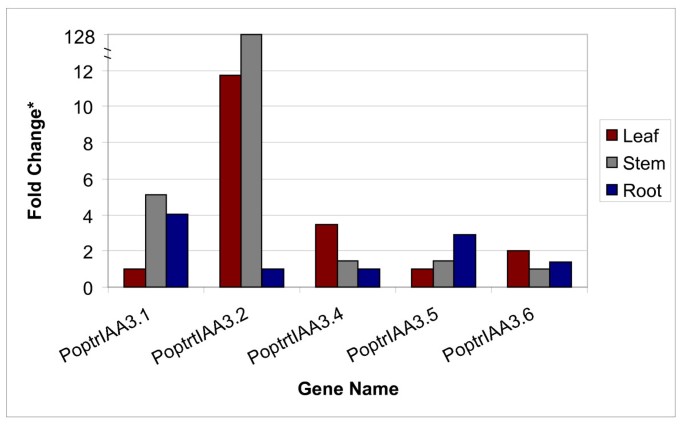
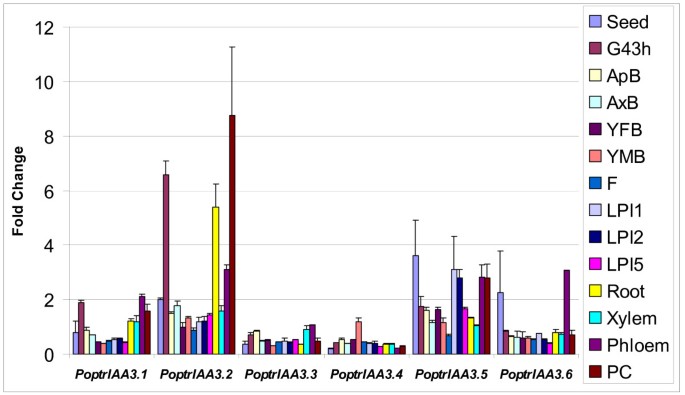
poptriaa3.1 - 3.6代表一个六人小组PoptrIAA3与…密切相关的子群AtIAA1-4.这个亚群为重复基因选择性保留后的功能分化提供了惊人的证据。而拟南芥shy2突变体,携带功能获得突变AtIAA3,叶片卷曲,向地性反应较慢,下胚轴较短,侧根较少[22的功能角色AtIAA4还有待分配。基因特异性实时RT-PCR显示PoptrIAA3亚群在叶、茎、根组织中表现出差异表达(图2)3.).PoptrIAA3.2发现其在茎中的表达量比在根中的表达量高出数倍。在对白杨的早期研究中辅助/ IAA的基因,PoptrIAA3.2样基因在木质部发育中表达量最高[24],我们的微阵列结果也支持这一观点(图2)4).EST数据表明PoptrIAA3.1和3.2似乎在形成层带和木材形成期间优先表达(见附加文件)6),微阵列数据表明,这些基因在新发芽的幼苗中也强烈表达。PoptrIAA3.6类似的est是在休眠芽和衰老叶片的文库中发现的。PoptrIAA3.4与其他的相比有不同的表达模式PoptrIAA3基因,在雄性和雌性柳絮中可检测到ESTs,并在花蕾中表达最高,通过芯片确定(图2)4).
微阵列表达式支持PoptrIAA3子群.数据以阴性对照的倍数变化表示,阴性对照包括95个th来自可能未表达的转座元件目标序列的百分位信号。误差条表示两个生物重复的标准误差。G43h,吸胀后43小时萌发;ApB,顶芽;AxB,腋芽;YFB,幼雌芽;YMB,幼雄芽;F,雌花絮,受精后;LPI1,叶片质体指数1;LPI2,叶片质体指数2; LPI5, leaf plastochron index 5; PC, phloem plus cortex. Samples are further defined in Additional file17.
鉴定与序列分析杨树ARF基因
共有39人预测东盟地区论坛基因(以下简称基因)PoptrARF)被发现杨树基因组,与23个基因相比拟南芥基因组(以下简称基因组AtARF) [2]和25个来自水稻基因组(以下简称为OsARF) [29].在网上基因位点定位显示PoptrARF基因存在于除VII、XIII、XVII和XIX以外的所有染色体上(图2)1).39个基因中有11个位于未组装的支架中。保守结构域评估显示了四个基因模型(PoptrARF3.3, 6.3, 16.5和16.6)似乎缺少一个或多个域,否则它们在最接近的序列同源中是保守的(参见附加文件)10,11和12).
十六岁PoptrARF基因对(占总数的82%)估计代表了由水螅类全基因组复制事件引起的染色体片段复制。两个基因,PoptrARF16.4和PoptrARF16.5,(占总数的5%)表示为一个串联复制对。的拟南芥基因组包含一组串联复制的7个东盟地区论坛到目前为止还没有在其他植物物种中观察到的基因,包括杨树和米饭。
的比较分析杨树和拟南芥ARF基因家族
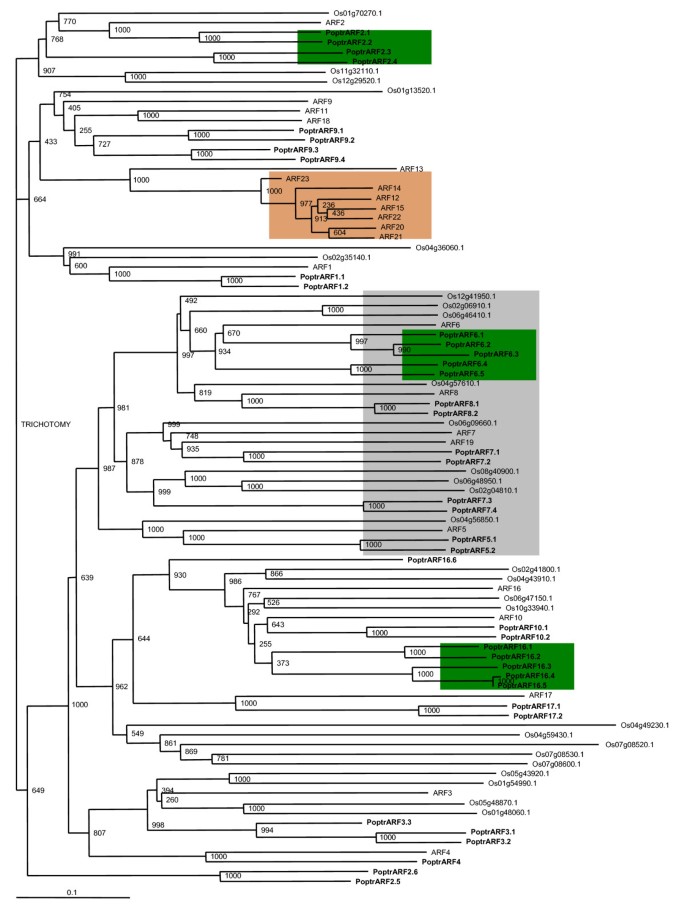
系统发育分析使用已知和预测杨树,拟南芥和水稻的ARF蛋白序列显示出不同的基因家族史,甚至在两个品种之间(图2)5).激活剂arf(由富q中间区域定义)的比值拟南芥和杨树为1:6 .6,而抑制因子和其他arf的比例为1:14 .4,表明激活因子arf在杨树进化。
一双……杨树的基因,PoptrARF7.3和PoptrARF 7.4他被发现和三个人在一起OsARF但没有明显的基因拟南芥直接同源。的功能丧失ARF7突变体表现为叶片扩张和侧根形成的改变[30.]和下胚轴向光性[31在拟南芥.PoptrARF7.3有EST支持从张力木库(见附加文件6),微阵列数据显示PoptrARF7.3在木质部组织中有更高的表达(见附加文件7,8和9).可能类似于AtARF7在气生植物形态中生长素依赖性差异生长中的作用[31],PoptrARF7.3可能参与木本茎对张力胁迫的差异生长。
至少三个不同的单例ArabidopsisARF基因(AtARF2 6和16)被发现有四个或更多的群集杨树每一个基因。PoptrARF6.4和PoptrARF6.5可能是在拟南芥中丢失的祖先基因的同同源物。AtARF 6和它的杨树同同源物有富q的中间区域。土豆ARF6,类似于AtARF6,据报道参与分生组织激活[32].据报道,芽、顶端分生组织和叶尖的含量最高ARF6转录水平。而PoptrARF6.1有形成层带和张力木组织库的EST支持;PoptrARF6.4的表达支持来自一个休眠芽库和PoptrARF6.2有EST从花蕾库的支持。此外,我们的微阵列实验表明,五名成员PoptrARF6亚组表现出不同的表达模式。PoptrARF6.1和6.4表达水平较低,在成熟叶片和韧皮部皮层样品中表达最多。相比之下,PoptrARF6.2和6.3在大多数组织类型中均有强烈表达,尤其在木质部、韧皮部、营养和生殖分生组织中表达强烈。最后,PoptrARF6.5在任何被测组织中均未显著表达(见附加文件7和8).有趣的是,AtARF6和8(双突变体)与花发育有关拟南芥[33].它们也表现出矮小,可能是由于顶端分生组织活性降低。虽然需要进一步的实验验证,但初步信息表明,该亚群在控制不同组织和发育阶段的分生组织活动中具有功能作用。
AtARF2负向调节差异生长拟南芥下胚轴(34].AtARF2据报道,突变体的花结构有缺陷[35].T-DNA插入突变体AtARF2莲座叶较大,而其他空气器官(包括花序)的数目和大小较小[33].AtARF2T-DNA插入突变体在种子和其他营养和花器官中也表现出额外的细胞分裂和扩增[35].杨树有四个假定的AtARF2直接同源(图5).PoptrARF2.5和PoptrARF2.6可能是在拟南芥中丢失的祖先基因的同同源物。基于微阵列和EST支持,PoptrARF2.12.2几乎无所不在表达,在木质部和韧皮部表达尤为强烈。PoptrARF2.3和PoptrARF2.4在营养芽和花芽以及形成层(细胞分裂)区表达。组织分布杨树ESTs和阵列结果表明,这些序列同源物可能具有相似的功能背景杨树和拟南芥.
特征描述ettin摘要)突变体显示AtARF3参与花的分生组织、雌蕊、雄蕊和花被的形成[36].杨树有三个ARF3就像基因一样PoptrARF3.1和3.2在营养芽和生殖芽中有一些EST和微阵列表达支持,但令人惊讶的是,这些基因在木质部和韧皮部中表达最强烈。PoptrARF3.3由于基因模型在初始注释中被人为截断,因此未被包含在微阵列上。
Monopteros(国会议员),即功能丧失AtARF5突变体,有严重畸形的胚轴和维管系统,有缺陷的花序,侧花完全缺乏或数量减少[37].杨树有两个假定的同源词AtARF5,PoptrARF5.1和5.2, RT-PCR和微阵列结果表明,与茎和叶相比,根中的表达略高(见附加文件)7,8和13).杨树EST和芯片数据显示PoptrARF5.2在花蕾中高度表达。这些同同源物是否可以被认为是子功能化的,还有待确定AtARF5(或共同祖先),或者它们是否在树的发育中起着额外的作用。
的演化、分化与调控辅助/ IAA和东盟地区论坛基因家族
全基因组复制随后是一系列相互串联末端融合导致了复制基因组片段的戏剧性重组杨树.随后的基因丢失和扩增事件导致了两种植物之间基因家族结构的分化,拟南芥和杨树[13,38].此外,通过串联或片段复制的扩展模式在每个基因家族的成员之间是不同的。
系统发育分析显示,IAA16和ARF4等几个亚群含有in的序列代表杨树和拟南芥但在水稻中没有,这表明这些亚群是在单子叶分化后在双子叶中获得或差异保留的。
与东盟地区论坛基因家族,辅助/ IAA观察到基因家族的扩大主要是由于片段重复拟南芥[38].片段复制也促进了这两个基因家族在水稻中的扩展[12,29].我们的研究表明PoptrIAA以及PoptrARF在水螅类生物全基因组复制和重排事件发生后,基因以高于平均水平的速率大量保留。在全基因组范围内,45,000个预测基因中约有14,000个(约32%)保留在由类水螅重复事件造成的重复对中[13].的留存率PoptrIAA和PoptrARF家庭分别占80%和82%。这与参与转录调控和信号转导的基因在复制后优先保留的预期一致[39- - - - - -41].保留串联重复的比例较低(5%)PoptrARF与基因家族相比PoptrIAA家族可能是由于与二聚体化学计量维持ARF转录因子活性相关的限制,因为串联簇确实存在于其他杨树基因家族[13].尽管43%的PoptrIAA基因以两到三个基因的串联簇表示,这些串联簇中有近86%可能在染色体水平的片段复制或全基因组复制事件后扩展并保留。这些观察结果表明PoptrIAA和PoptrARF转录因子家族主要由源自高片段(大规模)和低串联(小规模)复制事件的基因组成。与重复转录因子基因相关的亚功能化、新功能化和非功能化事件在功能分支(如差异调节生长素信号转导途径的能力)中具有更大的权重。它可以推测,主要基于我们对最近的功能角色的理解拟南芥直接同源(33- - - - - -35,42],表明存在多个ARF2同同源物杨树基因组可能反映了在开花、衰老和脱落的背景下,对细胞分裂和扩增更大的时间控制需求杨树一种多年生落叶乔木,表现出季节性的动态变化,并在数年的时间跨度内从幼树到成熟树再到繁殖阶段过渡,而不是像一年生那样在几天内拟南芥.考虑到ARF-和Aux/IAA-介导调节的复杂性,多样化的原因和影响将需要使用进化系统生物学研究进一步了解[43].
关系密切东盟地区论坛代表姐妹位点对的S已被发现具有强烈的双突变表型和重叠的表达域[33].此外,与组间成员相比,组内成员(激活因子或抑制因子ARF组)之间的相互作用亲和力更强[44].此外,单突变和双突变是由AtIAA12功能和ARF5功能丧失突变体具有相似的突变表型。有人提出ARF5可以采取积极的行动和IAA12以负调控的方式以生长素依赖的方式控制胚胎发生和根分生组织的发育[21].RT-PCR结果显示PoptrARF5和PoptrIAA12基因在根中表现出不同的表达模式(见附加文件)13).的高度表达PoptrARF5在根和低表达PoptrIAA12表明它们可能共同调控根的发育杨树以生长素依赖的方式之间异型相互作用的潜在数量杨树arf和Aux/IAAs的数量可能是这两个大基因家族成员数量的数倍。这可能有助于多年生植物获得更大发育可塑性所需的高阶生长素信号和响应机制。
生长素反应机制最近也被证明是通过小的非编码RNA物种调控的;microRNA (miRNA)和反式短干扰RNA (ta-siRNA)。AtARF 10,16和17已知受miR160的调控,miR160是一种在植物界高度保守的miRNA组[45,46),AtARF6和AtARF8受到miR167的调控[47].据预测,miR160和miR167家族在该研究中的比例都超过了两倍杨树与基因组相比拟南芥[13].miR160的靶序列已在PoptrARF10.1-10.2,poptrarf16.1 - 16.5和poptrarf17.1 - 17.2和miR167靶标poptrarf6.1 - 6.3和poptrarf8.1 - 8.2.
TAS3ta-siRNA已被证明介导转录后调控ARF3和ARF4基因表达[48,49并在幼虫向成虫相变中起关键作用拟南芥[50,51].据报道,ta-siRNA靶点序列在AtARF2,AtARF3和AtARF4以及相关的水稻和小麦序列[49].同源保守位点出现一次PoptrARF2.2和PoptrARF2.6两次PoptrARF3.1,PoptrARF3.2和PoptrARF4基因。GenBank数据库检索显示,葡萄、烟草、紫花苜蓿和番茄的arf3样序列也携带两个保守的ta-siRNA靶位点。个人PoptrARF同同源物以及miR和tasiR组中代表的不同基因为生长激素相关转录的复杂调控相互作用提供了无数的机会杨树.
结论
本研究确定了一组辅助/ IAA和东盟地区论坛基因杨树并揭示了它们的基因家族结构。比较基因组学拟南芥和rice认为,这两个基因家族有一个共同的起源,包括在所有三个基因组的ARF基因家族中保存激活子群,但经历了不同的进化史,在重要的方面存在分歧,包括缺乏大集群的串联复制AtARF基因杨树而大米,缺乏PoptrARF7.3和PoptrARF7.4直接同源的拟南芥并有一定的实质性扩张PoptrIAA和PoptrARF通过大规模分段重复的子组。的基因家族结构杨树和拟南芥与单子叶植物、水稻相比,表现出更大程度的保守性。
的杨树基因组序列和本文报道的研究结果为促进生长素反应调控基因与保守的核心植物过程以及多年生植物特征(如木材发育、长距离营养和水分运动、季节动态和抗病性)之间的联系提供了新的假设和探索机会。有针对性的反向遗传学研究,高分辨率时空表达调查以及在活的有机体内同种和异二聚化的亲和关系辅助/ IAA和东盟地区论坛为了了解这些基因的分子功能如何在整个植物水平上转化为生长素介导的多种作用,需要对分类群中的基因家族成员进行研究。对Aux/IAA和ARF蛋白基本功能背景的进一步研究将为其在农业、林业、环境和能源领域的应用提供机会。一个这样的应用包括正在进行的功能基因组学研究,以调查的作用PoptrIAA和PoptrARF碳分配和碳固存的基因。
方法
的识别辅助/ IAA和东盟地区论坛基因家族杨树
基因最初使用分配给预测的Pfam结构域id进行鉴定杨树JGI (DOE Joint Genome Institute, CA)注释管道中的基因模型[13],并使用拟南芥ARF和Aux/IAA蛋白[2,11]作为预测的BLASTP搜索中的查询杨树蛋白(杨树基因组v 1.1, 2007年1月)。杨树在此初始搜索中鉴定的蛋白质被用作预测的其他BLASTP搜索的查询序列杨树蛋白质集的穷尽鉴定分歧杨树基因家族成员。基于基因结构、保守基序的完整性、EST支持度和同源性分析对冗余和无效基因模型进行验证。我们还在研究中纳入了一个不完整的基因模型(fgenesh4_pg.C_scaffold_1006000001)PoptrARF6.3因为这个基因模型的两侧是一个序列间隙,然后是保守的ARF氨基端结构域,这可能会在即将到来的基因组版本中被纠正成一个完整的基因模型。此外,基于微阵列分析,该基因显示出非常强的表达证据。植物的序列保守与微同源性分析杨树的基因模型杨树同源(重复)基因组区域和拟南芥使用Vista Browser工具进行基因组分析,使用默认曲线计算参数;核苷酸序列“保守一致性”达70%及“最小保守宽度”达100 bp [52].本研究采用每个位点一个基因模型,其中包括一些代表提升集或参考集的JGI注释模型。还应该注意的是,在当前版本的基因组(v1.1)中,一些支架可能代表单倍型,而不是唯一的未组装的基因组区域。基因命名法是基于国际遗传工程协会制定的共识标准杨树基因组联盟(IPGC)来区分杨树trichocarpa从其他杨树物种。
染色体作图PoptrIAA和PoptrARF基因和重复基因的估计
染色体定位信息收集自杨树基因组浏览器[53].染色体位置是通过整合基因组组装和基于微卫星的遗传图谱来确定的P.trichocarpa×P.摘要[13,54].物理染色体位置通过缩放19条染色体(描绘全基因组复制区域,如Tuskan)在图形输出中表示等., 2006),然后是比例尺导向定位或定位位点。由全基因组复制事件产生的同源染色体片段的鉴定如Tuskan等人2006年所述完成。简而言之,采用双仿射Smith-Waterman算法进行全局对齐。对之间的遗传距离计算为四倍简并核苷酸位点发生翻转的比例(4DTV距离)。4DTV代表四重同义(退化)第三密码子反转。它代表了四个密码子中第三个核苷酸位置的翻转,但不会导致其编码的蛋白质中相应氨基酸特性的改变。这种对基因转录区域内的同义突变率的估计,而不是对经历选择的区域的同义突变率的估计,是在较近的进化历史中估计分化的保守方法。根据4DTV分布的离散峰,描述了与“类水杨”全基因组重复事件相对应的距离。重复片段被定义为不同连锁组或支架上含有6对或6对以上具有相似4DTV值的同源基因的区域,其中不超过25个非同源基因介入。由'salicoid'重复产生的基因对(显然是水杨科常见的)被定义为4DTV值在0.04和0.17之间。 Microsynteny in flanking regions of segmental duplicates was verified using the Genome Browser. Tandemly duplicated genes that matched the same homeolog were only counted once for this analysis.
序列与系统发育分析
保守蛋白基序由cd -search确定[55]和使用MEME-MAST程序[56,57].使用bl2seq工具确定两个基因之间的序列一致性[58].使用MUSCLE序列比对程序进行多次序列比对[59].采用保守区氨基酸序列比对和所有预测蛋白的全长序列比对两种方法构建系统发育树辅助/ IAA和东盟地区论坛基因家族杨树,拟南芥[2,9,11]和米饭[12,29].拟南芥和水稻序列分别来自TAIR和TIGR数据库。使用ClustalX 1.83程序,采用Neighbor-Joining法生成1000次bootstrap的无根phillip树。系统图在TreeView v1.6.6中可视化。保守、整理和对齐的Aux/IAA的贝叶斯系统发育分析(见附加文件)14)和ARF氨基酸序列(参见附加文件)15)使用MRBAYES(版本3.1.2)软件包[60,61].我们使用WAG替换频率矩阵[62],站点间速率变化由具有四种等概率类别的离散γ分布模拟。两次独立运行1-2百万蒙特卡洛马尔可夫链代,每代运行4条链。树木每100代采样一次,通过绘制似然分数与代数的关系来确定定相和燃烧值。根据共识计算的后验概率显示在分支上。
RNA分离及实时PCR分析
收集温室栽培植株的叶、茎、根组织材料P.trichocarpa(Nisqually-1)植物,并立即冷冻在液氮中。使用plant RNeasy试剂盒(Qiagen, CA)从植物样品中提取总RNA,然后进行DNase I处理。采用纳米滴分光光度计和凝胶电泳法测定RNA的质量和数量。根据制造商的说明,使用SuperScriptIII (Invitrogen, CA)从2微克RNA合成cDNA。利用cDNA、引物对进行实时荧光定量PCR(见附加文件)16)和iQ SYBR混合(BioRad, CA)。样品在iCycler实时PCR机(BioRad, CA)中运行一式三份,并根据18s阈值周期值进行归一化。折射率变化由公式2计算——ΔΔCt,其中ΔΔCt是特定组织中某个基因的ΔCt值与该基因在三种组织类型中任何一种的最低值ΔCt值之间的差值。ΔCt由公式估算;目的基因Ct - 18S RNA基因(对照基因)ΔCt的几何平均值。EST信息来源于GenBank和the杨树EST数据库,PopulusDB [63].
微阵列分析
我们使用了与NimbleGen Systems, Inc.共同设计的全基因组微阵列。该阵列针对来自杨树基因组测序计划的55,794个预测转录本。每个目标有三种不同的等温60聚合物探针,设计用于最大的特异性。所有探针都在阵列上复制一次,总共合成了385,000多个探针原位在玻片上[64].
用于微阵列杂交的组织来自田间和温室种植的树木,组织收集方法因组织类型而异。所有组织均取自杨树trichocarpa克隆Nisqually-1,除了花组织和种子,这些都是从俄勒冈州Corvallis附近的野生树木上收集的。组织和缩写在附加文件中列出17.12种组织类型各设2个生物重复,木质部和韧皮部各设3个生物重复。按照描述提取RNA进行RT-PCR分析。标记、杂交和扫描由NimbleGen使用他们的标准表达阵列协议进行。
数组数据(参见附加文件)18)使用NimbleGen微阵列数据处理管道(NMPP)进行归一化[65].我们使用了两步归一化程序,首先在每个组织内的重复中进行初始分位数归一化,然后进行全局归一化,将所有组织调整到相似的基线。使用所有探针和重复的平均归一化荧光值进行基因水平分析。对于阴性对照,我们使用了针对3,149个转座因子的探针,这些转座因子是最初释放的污染物杨树基因模型。这种方法是合理的,因为绝大多数这些元素在任何特定时间都是静止的,与阵列上所有其他目标组相比,来自这些探针的杂交信号明显较低,这一假设得到了支持(数据未显示)。对于每个实验,我们将表达阈值定义为95th所有阴性对照目标的荧光值的百分位数。个体基因的相对表达被评估为与阴性对照阈值的倍数变化。两倍的变化被认为是显著表达的证据。
参考文献
- 1.
王晓明,王晓明,王晓明,等。豌豆生长素诱导蛋白PS-IAA4的核靶向性研究。植物学报,1995,8(1):87-96。10.1046 / j.1365 - 313 x.1995.08010087.x。
- 2.
王晓东,王晓东,王晓东,等。生长素基因表达调控因子的研究进展。植物化学学报,2002,32(3):373-385。10.1023 /: 1015207114117。
- 3.
张建军,张建军,张建军,等。植物生长素反应因子的二聚体化与DNA结合。植物学报,1999,19(3):309-319。10.1046 / j.1365 - 313 x.1999.00538.x。
- 4.
Ulmasov T, Hagen G, Guilfoyle TJ:一种结合生长素反应元件的转录因子ARF1。科学,1997,26(5):1865-1868。10.1126 / science.276.5320.1865。
- 5.
李建军,张建军,张建军,等。生长素反应因子对基因转录的抑制作用。美国国家科学促进会。中国生物医学工程学报,2009,35(10):544 - 549。10.1073 / pnas.96.10.5844。
- 6.
Dharmasiri N, Dharmasiri S, Estelle M: F-box蛋白TIR1是一种生长素受体。自然科学,2005,35(7):441-445。10.1038 / nature03543。
- 7.
Kepinski S, Leyser O:生长素诱导的SCFTIR1- aux /IAA相互作用涉及SCFTIR1复合物的稳定修饰。美国国家科学促进会。中国生物医学工程学报,2004,31(3):381- 386。10.1073 / pnas.0402868101。
- 8.
Kepinski S, Leyser O:拟南芥F-box蛋白TIR1是一种生长素受体。自然科学,2005,35(7):446-451。10.1038 / nature03542。
- 9.
王晓明,王晓明,王晓明,等。植物生长素反应因子域在植物生长素应答转录中的作用。植物学报,2003,15(2):533-543。10.1105 / tpc.008417。
- 10.
杨晓东,杨晓东。生长素:调控、作用和相互作用。安·伯特(朗德)。中国生物医学工程学报,2009,35(5):557 - 557。10.1093 / aob / mci083。
- 11.
植物生长发育过程中Aux/IAA和ARF的遗传作用。植物化学学报,2002,32(3):387- 394。10.1023 /: 1015255030047。
- 12.
Jain M, Kaur N, Garg R, Thakur JK, Tyagi AK, Khurana JP:水稻早期生长素响应Aux/IAA基因家族的结构和表达分析。功能与整合基因组学。2006, 6(1): 47-59。10.1007 / s10142 - 005 - 0005 - 0。
- 13.
简颂年代,Tuskan GA Difazio年代,Bohlmann J,眼镜,Hellsten U,普特南N,拉尔夫,Rombauts年代,Salamov,史肯J, Sterck L, Aerts, Bhalerao RR, Bhalerao RP, Blaudez D, Boerjan W,布朗,Brunner, Busov V,坎贝尔M,卡尔森J, Chalot M,查普曼J,陈GL,库珀D, Coutinho点,女裁缝师J,隐蔽的年代,体弱的Q, R,坎宁安戴维斯J, Degroeve年代,Dejardin, Depamphilis C,德J,短剑B, Dubchak我Duplessis年代,Ehlting J,埃利斯B, Gendler K,古德斯坦D, Gribskov M, Grimwood J,挖槽机,甘特L, Hamberger B,海因策B, Helariutta Y, Henrissat B,霍利甘D,霍尔特R,黄W, Islam-Faridi N,琼斯,Jones-Rhoades M,约根森R, Joshi C, Kangasjarvi J, Karlsson J,凯莱赫C,柯克帕特里克R, Kirst M,科勒,Kalluri U,此外F, Leebens-Mack J, Leple JC, Locascio P,卢Y,卢卡斯,马丁F, Montanini B, C那不勒斯,纳尔逊博士,纳尔逊C, Nieminen K,尼尔森O, Pereda V,彼得G,菲利普·R,彼拉多G, Poliakov, Razumovskaya J,理查森P,里纳尔蒂C, Ritland K,Rouze P, Ryaboy D, Schmutz J, Schrader J, Segerman B, Shin H, Siddiqui A, Sterky F, Terry A, Tsai CJ, Uberbacher E, Unneberg P, Vahala J, Wall K, Wessler S, Yang G, Yin T, Douglas C, Marra M, Sandberg G, Van de Peer Y, Rokhsar D:黑杨木、毛卡柏(Torr.)的基因组。&灰色)。科学通报,2006,31(5):1596-1604。10.1126 / science.1128691。
- 14.
李建军,张建军,张建军,等。转录抑制区域的研究进展。植物学报,2004,16(2):533-543。10.1105 / tpc.017384。
- 15.
Ramos JA, Zenser N, Leyser O, Callis J:生长素/吲哚乙酸蛋白的快速降解需要结构域II的保护氨基酸和蛋白酶体依赖。植物细胞,2001,13(10):2349-2360。10.1105 / tpc.13.10.2349。
- 16.
Dreher KA, Brown J, Saw RE, Callis J:拟南芥Aux/IAA蛋白家族在降解和生长素响应中的多样性。植物细胞,2006,18(3):699-714。10.1105 / tpc.105.039172。
- 17.
Nagpal P, Walker LM, Young JC, Sonawala A, Timpte C, Estelle M, Reed JW: AXR2编码Aux/IAA蛋白家族成员。植物生理学报,2009,32(2):563-574。10.1104 / pp.123.2.563。
- 18.
Reed JW: Aux/IAA蛋白在拟南芥中的作用和活性。植物科学进展,2001,6(9):420-425。10.1016 / s1360 - 1385(01) 02042 - 8。
- 19.
李建军,李建军,李建军,等。植物基因IAA28的克隆及其对植物侧根发育的影响。植物学报,2001,13(3):465-480。10.1105 / tpc.13.3.465。
- 20.
Fukaki H, Tameda S, Masuda H, Tasaka M:拟南芥单根/IAA14基因功能获得突变阻断侧根形成。植物学报,2002,29(2):153-168。10.1046 / j.0960-7412.2001.01201.x。
- 21.
Hamann T, Benkova E, Baurle I, Kientz M, Jurgens G:拟南芥BODENLOS基因编码生长素反应蛋白抑制monopteros介导的胚胎模式。基因工程学报,2002,16(3):369 - 369。10.1101 / gad.229402。
- 22.
田青,Uhlir NJ, Reed JW:拟南芥SHY2/IAA3对生长素调控基因表达的抑制作用。植物学报,2002,14(2):301-319。10.1105 / tpc.010283。
- 23.
Tatematsu K, Kumagai S, Muto H, Sato A, Watahiki MK, Harper RM, Liscum E, Yamamoto KT:生长素调控蛋白Aux/IAA19与转录激活因子NPH4/ARF7共同调控拟南芥下胚轴生长和侧根形成的差异。植物学报,2004,16(2):379-393。10.1105 / tpc.018630。
- 24.
莫伊尔,施rader J, Stenberg A, Olsson O, Saxena S, Sandberg G, Bhalerao RP:环境和生长素对杂交白杨木材形成的调控。植物学报,2002,31(6):675-685。10.1046 / j.1365 - 313 x.2002.01386.x。
- 25.
崔刚,李浩,李军,权以坤,苏美娟,申斌,Luka Z, Hahn TR, Song PS:光敏色素信号通过核苷二磷酸激酶2介导。自然科学学报,1999,41(6):663 - 668。10.1038/44176。
- 26.
Soh MS, Hong SH, Kim BC, Vizir I, Park DH, Choi G, Hong MY, Chung YY, Furuya M, Nam HG:光和生长素介导的拟南芥IAA3/SHY2基因的调控。植物学报,1999,42(2):239-246。
- 27.
黄晓明,黄晓明,黄晓明,等。烟草花叶病毒复制酶蛋白与烟草花叶病毒转录因子的相互作用及其与烟草花叶病毒转录因子的关系。病毒学杂志。中国生物医学工程学报,2009,31(4):559 - 558。10.1128 / jvi.79.4.2549 - 2558.2005。
- 28.
Padmanabhan MS, Shiferaw H, Culver JN:烟草花叶病毒复制酶蛋白破坏相互作用的Aux/IAA蛋白的定位和功能。植物学报,2006,19(8):864-873。10.1094 / mpmi - 19 - 0864。
- 29.
王东,裴科,付勇,孙忠,李松,刘宏,唐凯,韩波,陶勇:水稻生长素反应因子(ARF)基因家族的全基因组分析。中国生物医学工程学报,2007,32(1):1 - 4。10.1016 / j.gene.2007.01.006。
- 30.
Wilmoth JC, Wang S, Tiwari SB, Joshi AD, Hagen G, Guilfoyle TJ, Alonso JM, Ecker JR, Reed JW: NPH4/ARF7和ARF19促进生长素诱导的叶片扩张和侧根形成。植物学报,2005,43(1):118-130。10.1111 / j.1365 - 313 x.2005.02432.x。
- 31.
Harper RM, Stowe-Evans EL, Luesse DR, Muto H, Tatematsu K, Watahiki MK, Yamamoto K, Liscum E: NPH4基因座编码生长素反应因子ARF7,这是空中拟南芥组织差异生长的条件调节因子。植物细胞,2000,12(5):757-770。10.1105 / tpc.12.5.757。
- 32.
favre - rampant O, Cardle L, Marshall D, Viola R, Taylor MA:龙葵分生组织激活过程中基因表达的变化及其对生长素反应因子基因的调控。[J] .生物医学工程学报,2004,35(3):613-622。10.1093 / jxb / erh075。
- 33.
Okushima Y, Overvoorde PJ, Arima K, Alonso JM, Chan A, Chang C, Ecker JR, Hughes B, Lui A, Nguyen D, Onodera C, Quach H, Smith A, Yu G, theology A .拟南芥生长素反应因子基因家族成员的功能基因组分析:ARF7和ARF19的独特和重复功能。植物细胞,2005,17(2):444-463。10.1105 / tpc.104.028316。
- 34.
李海,Johnson P, Stepanova A, Alonso JM, Ecker JR .:拟南芥细胞分化生长的信号通路趋同调控。开发单元,2004,7(2):193-204。10.1016 / j.devcel.2004.07.002。
- 35.
Schruff MC, Spielman M, Tiwari S, Adams S, Fenby N, Scott RJ:拟南芥生长素应答因子2基因与生长素信号、细胞分裂和种子及其他器官大小的关系。生态学报,2006,33(2):251-261。10.1242 / dev.02194。
- 36.
李建军,李建军,李建军,李建军。拟南芥花分生组织和生殖器官的研究进展。发展,1997,24(22):4481-4491。
- 37.
拟南芥基因MONOPTEROS编码一个介导胚轴形成和维管发育的转录因子。[j] .中华医学杂志,1998,17(5):1405-1411。10.1093 / emboj / 17.5.1405。
- 38.
李建军,李建军,李建军,等。水稻ARF基因的克隆与分析。植物生理学报,2009,35(3):554 - 557。10.1104 / pp.104.039669。
- 39.
Blanc G, Wolfe KH:拟南芥多倍体形成的重复基因的功能分化。植物学报,2004,16(7):1679-1691。10.1105 / tpc.021410。
- 40.
李建军,陈建军,陈建军,陈建军,陈建军,陈建军,陈建军,陈建军。美国国家科学促进会。中国生物医学工程学报,2009,31(5):559 - 559。10.1073 / pnas.0501102102。
- 41.
Seoighe C, Gehring C:基因组复制导致拟南芥蛋白质组的高度选择性扩增。科学通报,2004,20(10):461-464。10.1016 / j.tig.2004.07.008。
- 42.
王晓明,杨建军,张建军,张建军,张建军。植物生长素反应因子1和2对拟南芥衰老和花器官脱落的调控作用。发展,2005,32(20):4563-4574。10.1242 / dev.02012。
- 43.
Hughes AL:基因复制和新蛋白质的起源。美国国家科学促进会。中国生物医学工程学报,2009,32(5):591 - 592。10.1073 / pnas.0503922102。
- 44.
王晓明,王晓明,王晓明,王晓明,王晓明,王晓明,王晓明。拟南芥生长素响应因子MONOPTEROS和非光敏性下胚轴l4的克隆与表达。发展,2004,131(5):1089-1100。10.1242 / dev.00925。
- 45.
Mallory AC, Bartel DP, Bartel B: microrna定向调控拟南芥生长素反应因子17对其正常发育和早期生长素反应基因的表达至关重要。植物细胞,2005,17(5):1360-1375。10.1105 / tpc.105.031716。
- 46.
陆生植物microrna的研究进展。植物学报,2005,17(6):1658-1673。10.1105 / tpc.105.032185。
- 47.
吴文明,田青,Reed JW:拟南芥microRNA167调控ARF6和ARF8的表达模式,调控雌雄生殖。生态学报,2006,33(6):421 - 418。10.1242 / dev.02602。
- 48.
谢志强,谢志强,陈志强,陈志强,等。植物siRNA生物发生过程中microrna调控的研究进展。细胞学报,2005,21(2):207-221。10.1016 / j.cell.2005.04.004。
- 49.
Williams L, Carles CC, Osmont KS, Fletcher JC:数据库分析方法鉴定内源性反式短干扰RNA靶向拟南芥ARF2, ARF3和ARF4基因。美国国家科学促进会。中国生物医学工程学报,2009,29(3):387 - 387。10.1073 / pnas.0504029102。
- 50.
王晓明,王晓明,王晓明,王晓明,王晓明。拟南芥基因表达的研究进展。发展,2006,33(15):2973-2981。10.1242 / dev.02491。
- 51.
Fahlgren N, Montgomery TA, Howell MD, Allen E, Dvorak SK, Alexander AL, Carrington JC:生长素反应因子3通过TAS3 TA - sirna调控影响拟南芥发育时间和模式。当代生物学,2006,16(9):939-944。10.1016 / j.cub.2006.03.065。
- 52.
Frazer KA, Pachter L, Poliakov A, Rubin EM, Dubchak I: VISTA:比较基因组学的计算工具。核酸学报,2004,W273-279。10.1093 / nar / gkh458。32 Web服务器
- 53.
- 54.
Yin TM, DiFazio SP, Gunter LE, Riemenschneider D, Tuskan GA:密集遗传图谱揭示的杨树大尺度异种分离扭曲。理论应用学报,2004,29(3):451-463。10.1007 / s00122 - 004 - 1653 - 5。
- 55.
Marchler- bauer A, Anderson JB, Derbyshire MK, dewese - scott C, Gonzales NR, Gwadz M, Hao L, He S, Hurwitz DI, Jackson JD, Ke Z, Krylov D, Lanczycki CJ, Liebert CA, Liu C, Lu F, Lu S, Marchler GH, Mullokandov M, Song JS, Thanki N, Yamashita RA, Yin JJ, Zhang D, Bryant SH: CDD:一种基于交互域族分析的域域数据库。中国生物医学工程学报,2007,32(3):444 - 444。10.1093 / nar / gkl951。35数据库
- 56.
李建军,李建军。基于期望最大化的混合模型在生物聚合物中发现基序。中国生物医学工程学报,1994,22(2):359 - 361。
- 57.
贝利TL, Gribskov M:结合证据使用p值:应用于序列同源性搜索。生物信息学。1998,14(1):48-54。10.1093 /生物信息学/ 14.1.48。
- 58.
Tatusova TA, Madden TL: BLAST 2序列,一种比较蛋白质和核苷酸序列的新工具。微生物学通报,1999,32(2):557 - 557。10.1111 / j.1574-6968.1999.tb13575.x。
- 59.
Edgar RC: MUSCLE:具有高精度和高通量的多序列比对。核酸学报,2004,32(5):1792-1797。10.1093 / nar / gkh340。
- 60.
Huelsenbeck JP, Ronquist F: MRBAYES:系统发育树的贝叶斯推断。生物信息学。2001,17(8):754-755。10.1093 /生物信息学/ 17.8.754。
- 61.
Ronquist F, Huelsenbeck JP: MrBayes 3:混合模型下的贝叶斯系统发育推断。生物信息学。2003,19(12):1572-1574。10.1093 /生物信息学/ btg180。
- 62.
惠兰S,高盛N:利用最大似然方法从多个蛋白质家族衍生的蛋白质进化的一般经验模型。中华生物医学杂志,2001,18(5):691-699。
- 63.
Sterky F, Bhalerao RR, Unneberg P, Segerman B, Nilsson P, Brunner AM, charbonnell - campa L, Lindvall JJ, Tandre K, Strauss SH, Sundberg B, Gustafsson P, Uhlen M, Bhalerao RP, Nilsson O, Sandberg G, Karlsson J, Lundeberg J, Jansson S:杨树EST植物功能基因组学资源。美国国家科学促进会。中国生物医学工程学报,2004,31(3):391 - 391。10.1073 / pnas.0401641101。
- 64.
Groover AT, Mansfield SD, DiFazio SP, Dupper G, Fontana JR, Millar R, Wang Y:杨树茎尖分生组织和维管形成层的调控机制。植物学报,2006,31(6):917-932。10.1007 / s11103 - 006 - 0059 - y。
- 65.
王鑫,何宏,李磊,陈锐,邓小文,李松:NMPP:用户定制的NimbleGen微阵列数据处理管道。生物信息学。2006,22(23):2955-2957。10.1093 /生物信息学/ btl525。
致谢
作者要感谢国际杨树基因组联盟的测序,组装和注释杨树基因组和共享杨树EST数据库和Vista浏览器工具。我们也感谢S. Mane, P. Dharmawardhana和O. Crasta对微阵列数据分析的帮助,以及三位匿名评论者的有益评论。这项研究得到了美国能源部科学、生物和环境研究办公室的支持(项目名称;“基因组支持的碳封存基因的发现。杨树)及DOE/BER批准书编号:DE-FG02-06ER64185到AMB。橡树岭国家实验室由UT-Battelle, LLC为美国能源部管理,合同为DE-AC05-00OR22725。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
UCK进行了序列、系统发育和表达分析,并撰写了稿件。SPD参与序列分析,并与AMB一起生成微阵列数据并进行数据分析。GAT为这项研究的构思做出了贡献。所有作者都阅读并批准了最终的手稿。
电子补充资料
12870 _2007_200_moesm2_esm.xls
附加文件2:Vista保存摘要-微同步分析。“是”和“否”是指在Poplar v.1.0之间的100 bp滑动窗口内存在至少70%的身份,Poplar重复和拟南芥序列。(xl18kb)
12870 _2007_200_moesm3_esm.pdf
附加文件3:预测的全长氨基酸序列的多序列比对杨树,拟南芥和水稻的Aux/IAA蛋白。使用MUSCLE程序对序列进行比对。一致性序列显示在对齐的底部。(pdf 60kb)
12870 _2007_200_moesm4_esm.pdf
附加文件4:预测的保守区域的多序列比对杨树,拟南芥和水稻Aux/IAA蛋白序列。使用MUSCLE程序对序列进行比对。一致性序列显示在对齐的底部。(pdf 22kb)
12870 _2007_200_moesm5_esm.pdf
附加文件5:预测的全长氨基酸序列保守结构域的预测杨树,拟南芥和水稻的Aux/IAA蛋白。使用MEME和MAST程序预测保守结构域。Motif编号6代表保守结构域I, Motif编号3代表保守结构域II, Motif编号2代表保守结构域III, Motif编号1、4和5代表保守结构域IV。
12870 _2007_200_moesm6_esm.xls
附加文件6:EST支持杨树辅助/ IAA和东盟地区论坛基因。数字表示每个组织库中est的数量。来源:杨树EST数据库,PopulusDB。(xls28kb)
12870 _2007_200_moesm7_esm.xls
附加文件7:来自微阵列表达分析的折叠变化数据PoptrIAA和PoptrARF基因。数据呈现为目标基因相对于95的规范化表达值th转座元件目标(可能未表达)的表达值的百分位数。与阴性对照差异大于2倍的基因以粗体突出显示。示例缩写在附加文件中定义17.(xl34kb)
12870 _2007_200_moesm8_esm.xls
附加文件8:来自微阵列表达分析的规范化原始数据PoptrIAA和PoptrARF基因。数据在重复内使用分位数归一化,然后跨组织类型进行归一化。数据以相对表达式值表示。还包括95th阴性对照探针归一化表达的百分位数。示例缩写在附加文件中定义17.(xl56kb)
12870 _2007_200_moesm9_esm.pdf
附加文件9:微阵列结果的温度图表示。图是用列计算的信号强度的温度图,它是从特定组织类型的所有数据的平均值中得出的。每个图中的底线代表负控制。红色表示高表达,深蓝色表示低表达。示例缩写在附加文件中定义17.(pdf 28kb)
12870 _2007_200_moesm10_esm.pdf
附加文件10:预测的全长氨基酸序列的多序列比对杨树,拟南芥和大米ARF蛋白。使用MUSCLE程序对序列进行比对。一致性序列显示在对齐的底部。(pdf 185kb)
12870 _2007_200_moesm11_esm.pdf
附加文件11:预测的保守区域的多序列比对杨树,拟南芥和水稻ARF蛋白序列。使用MUSCLE程序对序列进行比对。一致性序列显示在对齐的底部。(pdf 45kb)
12870 _2007_200_moesm12_esm.pdf
附加文件12:预测的全长氨基酸序列保守结构域的预测杨树,拟南芥和大米ARF蛋白。使用MEME和MAST程序预测保守结构域。Motif编号1,2,3,4,7和11代表保守的B3结构域。Motif编号6,8和12代表保守的生长素响应域,Motif编号5和10代表保守的c -末端Aux/IAA域(III和IV)。
12870 _2007_200_moesm13_esm.pdf
附加文件13:的表达式分析PoptrARF5和PoptrIAA12利用实时RT-PCR检测基因。* Fold Change表示相对于该基因观察到的最低值。通过比较叶片、茎和根样品中特定基因的相对阈值来确定最低值。折射率变化由公式2计算——ΔΔCt,其中ΔΔCt是特定组织中基因的ΔCt值与该基因在三种组织类型中的最低值ΔCt值之间的差值。ΔCt由公式估算;(目的基因Ct) -(对照基因18S RNA基因ΔCt的几何平均值)。(pdf 12kb)
12870 _2007_200_moesm14_esm.pdf
附加文件14:使用MRBAYES对预测的Aux/IAA蛋白序列保守区域进行贝叶斯系统发育分析。利用MUSCLE程序对全长预测蛋白的氨基酸序列进行比对。Tree是使用保守的整理区域生成的(参见附加文件)4),如方法中所述。根据共识计算的后验概率显示在分支上。绿色方框表示子组的持续扩展杨树.(pdf 21kb)
12870 _2007_200_moesm15_esm.pdf
附加文件15:使用MRBAYES对预测ARF蛋白序列保守区域进行贝叶斯系统发育分析。利用MUSCLE程序对全长预测蛋白的氨基酸序列进行比对。Tree是使用保守的整理区域生成的(参见附加文件)11),如方法中所述。根据共识计算的后验概率显示在分支上。绿色和橙色方框表示子组的持续扩展杨树和拟南芥,分别。灰框表示富q激活器ARF子组。(pdf 39kb)
12870 _2007_200_moesm17_esm.xls
附加文件17:用于微阵列实验的组织。描述RNA的来源和阵列实验的收集方法。(xl18kb)
12870 _2007_200_moesm18_esm.xls
附加文件18:微阵列数据PoptrIAA和PoptrARF来自NimbleGen的基因家族杨树全基因组微阵列版本1.0。数据是针对指定目标的所有探针之间的平均值,按文中所述归一化。同一组织类型的不同柱是生物复制。阴性对照为3155个转座因子表达水平的第95百分位杨树基因组。(xl30kb)
权利和权限
本文由BioMed Central Ltd.授权发表。这是一篇基于知识共享署名许可(http://creativecommons.org/licenses/by/2.0),允许在任何媒体上不受限制地使用、分发和复制,前提是正确引用原创作品。
关于本文
引用本文
Kalluri, u.c., DiFazio, S.P, Brunner, A.M.et al。全基因组分析辅助/ IAA和东盟地区论坛基因家族杨树trichocarpa.BMC Plant Biol7,59(2007)。https://doi.org/10.1186/1471-2229-7-59
收到了:
接受:
发表:
关键字
- 节段重复
- 紧张的木头
- 生长素反应因子
- 形成层的区
- 生长素反应因子基因