摘要
背景
木薯是一种异源四倍体,以其对非生物胁迫的显著耐受性而闻名,是人类和动物的重要能源来源,也是许多工业过程的原材料。建立了木薯植株在正常、高温、干旱、铝和采后生理退化条件下的全长cDNA文库;利用表达序列标记(ESTs)对19968个克隆进行了序列分析。
结果
est被组装成6355个contigs和9026个singleton,这些singleton被进一步组合成10577个支架;我们发现了4621个新的木薯序列和1521个与植物蛋白数据库无显著相似性的序列。我们捕获了7796个不同基因的转录本,并能够对其中78%的基因进行功能分类,同时在拟南芥中发现了超过一半的酶注释在代谢途径中。未与其他物种转录本配对的序列注释包括许多与胁迫相关的功能类别,表明我们的文库富含胁迫诱导基因。最后,我们检测到230个假定的基因复制,其中包括活性氧信号通路中的关键酶,这些酶可能在木薯胁迫响应特征中发挥作用。
结论
在这里展示的木薯全长cDNA文库包含了与胁迫反应有关的基因转录本,以及对木薯不同研究领域重要的基因。该文库将成为基因发现、表征和克隆的重要资源;在不久的将来,它将有助于木薯基因组的注释。
背景
在生产淀粉的作物中,木薯(木薯耐大戟科的Crantz在次优条件下的碳水化合物产量高于水稻或玉米[1];全球每年生产超过1.63亿吨,其中约84%用于人类直接消费和动物饲料[2].木薯淀粉被用作多种食品和工业产品的原料,包括纸张、硬纸板、纺织品、胶合板、胶水和酒精[3.].此外,由于从木薯中生产淀粉比其他作物便宜,木薯作为燃料生产的生物质来源正受到越来越多的关注[4].一个基因组测序项目证明了人们对木薯作为一种能源作物的兴趣日益浓厚[5热带国家的生产和技术进步不断增加;例如,1973年至1990年,泰国的木薯鲜根产量从630万吨增加到2000万吨[6]而同期世界范围内的数字则以每年2.2%的速度增长[2].
由于木薯对非生物胁迫具有显著的耐受性,它生长在边缘、低肥力酸性土壤中,显示出更高的养分利用效率[7].众所周知,在干旱易发地区,它能保持健康的外表,保持光合活性,尽管速率降低[8].因为木薯非常耐旱,块茎可以在土壤中保存几年,它被认为是预防或缓解饥荒的重要储备碳水化合物来源[9].木薯有一些不寻常的特性,使它在接近最佳的环境下高产(热潮湿气候和高太阳辐射),这些特性包括提高C4磷酸烯醇式丙酮酸羧化酶,叶片寿命长,光呼吸速率低[10];然而,它通常生长在边缘高度侵蚀的土壤,降雨不确定,几乎没有农用化学品的投入。尽管木薯有一些特性使它比其他作物更能应对压力,例如气孔对环境湿度高度敏感[11],生根能力深,胁迫后恢复快[12],在这些条件下,生产力是次优和不稳定的[10].木薯产量也受到细菌和病毒疾病的威胁[13],以及节肢动物[14].此外,其淀粉含量高与蛋白质和关键微量营养素(锌、铁和维生素)的缺乏形成反差,并会产生有毒的氰化氢[15].
为了解决这些问题,传统育种方法取得了一些成功,特别是在非胁迫条件下提高鲜根产量和干物质含量[16],然而,由于该作物的杂合子遗传组成和长生长周期,这种方法的进展缓慢[17].利用生物技术改良木薯品种是一种更直接的策略,它依靠分子和细胞生物学工具来寻找理想表型的遗传决定因素[18].在木薯对生物胁迫的响应中,遗传图谱的构建和数量性状位点的鉴定取得了一定的效果。19],然而,用这种方法识别候选基因是一个耗时的过程,涉及到细菌人工染色体(BAC)文库的构建和这些克隆到遗传图谱的锚定[20.].反向遗传学方法[21]是一种更直接的解决方案,依靠对模式物种中基因功能的积累知识,可以通过调控基因的表达来评估所选基因的影响。例如,沉默P-450细胞色素已允许生产无氰转基因木薯植株[22,23].
est是一种既可协助植物表达基因的表征,又可协助分离具有已知功能的基因的核苷酸序列的工具[24].这是一种具有成本效益的基因发现方法,对基因表达的研究也很有用[25].尽管木薯序列非常重要,但大规模的木薯序列收集很少,在dbEST数据库中有36162个表达的木薯序列[26],与玉米(2961956)、水稻(1912256)、大豆(686687)、马铃薯(275813)或甘蔗(257998)的ESTs数量相比,这是一个很小的数字。随着美国能源部联合基因组研究所今年发布的木薯基因组序列草案,这种情况可能会发生改变。虽然ESTs可以帮助木薯基因组的注释,但它们大多来自由随机mRNA片段组成的文库,这使得它们不足以准确和完整地定义基因模型[27], ESTs不仅来源于部分转录本,而且还可以在组装过程中混淆交替拼接的形式[28].此外,由于ESTs的碎片性,它们在基因功能分析中的应用受到限制[29- - - - - -31].
另一方面,全长cDNA文库的构建方式是,一个插入片段代表一个转录单元,为基因的功能解剖提供完整分子的信息[28].我们从受干旱、高温和酸性条件影响的木薯叶片和根系,以及受采后生理退化(PPD)影响的根系中构建了一个完整的富集cDNA文库,PPD是木薯商业化的主要障碍[32].该文库的目的是支持木薯在非生物胁迫下的高产改良研究,提供胁迫响应基因的完整序列,扩大该物种的基因目录。文库中捕获的转录本的特征描述和非冗余克隆的选择肯定有助于基因组序列的注释[30.以及构建微阵列或功能基因组学的其他工具[33].为了描述该文库的特征,并找到捕获的转录本的数量和假定功能,从两端对近20,000个克隆进行了测序,这些ESTs虽然不太可能包括插入的全部序列,但都标记了克隆名称。由于在汇编过程中考虑到这一信息,从完整文库派生的无害环境技术原则上可以比一般的无害环境技术更准确地定义转录单位。
获得的序列注释和与木薯有密切关系的两种物种如蓖麻豆(萝藦大戟科)及杨树(杨树trichocarpa杨柳科)(34以及完整的基因集拟南芥,为通过比较基因组学的方法研究木薯基因组的进化提供了机会[35];如果有可能确定这些物种之间的基因对应关系,并在此基础上找到木薯独有的序列,那么对这些基因的更仔细的检查可能为木薯独特特征背后的机制提供线索。木薯被认为是一种异源四倍体,是由野生植物杂交产生的木薯物种(36],那么看看在木薯驯化过程中,高度杂合的基因库中有哪些基因保持了功能将是有趣的;为此,我们使用了一种检测近期复制的方法,该方法基于对其他基因组中与单个序列具有相似性的基因组的检测,希望用这种策略检测到的基因将有助于木薯研究,以改进一种已经非常出色的作物的基因。
结果
两端单通序列的测序和组装
从不同环境条件下的木薯植物叶片和根系中构建cDNA文库,从两端测序了19968个克隆(CAS01_001_A01 ~ CAS01_052_P24或52 × 384孔板);克隆体可在理研生物资源中心获得[37],序列可从日本DNA数据库(DDBJ)获取,登录号为DB920056-DB955455。
序列读取被修剪为低质量和载体污染;该过程共获得19449个克隆序列35400个。对于5'和3'序列数据显示与已知蛋白质显著序列相似的克隆,计算出的全长比为0.84,这意味着大约85%的克隆包含插入的完整编码序列(CDS)。
用CAP3将序列组装为6355个contigs和9026个singleton;然而,考虑到所有序列都带有各自的克隆id,我们能够进一步将CAP3组装的结果聚类,构建10577个代表不同转录本的支架。其中,2005(19%)在单个contig中包含各自克隆的两端,因此被认为是全长序列。
为了估计文库中不同基因的数量,必须检测其他的剪接变体;使用方法部分描述的方法,我们仅识别了2096个基因的4877个转录本。我们确定,该全长文库包含7796个不同基因的转录本,其中约26%的可选转录本。为了找到这个库中捕获的新转录本的数量,相对于GenBank中已经存在的木薯表达序列的数量,我们对我们程序集中的序列进行了BLASTN搜索,针对dbEST中截至2007年4月的36162个EST序列。任何未命中数据库的序列或e值为> 1e-100且识别率< 95%的序列都被认为是新的木薯转录本。通过这种方法,我们在我们的集合中发现了4621个新的木薯序列。此外,通过对UniProt - TrEMBL植物蛋白数据库运行BLASTX,我们发现1521个转录本与其他植物中的已知蛋白没有相似性(e值为1e-5)(见表1).
CAP3组件中的信息和测序克隆的名称被用来构建一个集群概要文件,表示每个组装的支架的克隆数量(图1);这样做是为了使用ESTstat包中实现的复合泊松过程模型提供木薯转录本总数的近似值[38,39].我们获得了50698个转录本,这是在杨树、拟南芥和水稻中估计的转录本数量的范围内2).
序列的功能注释
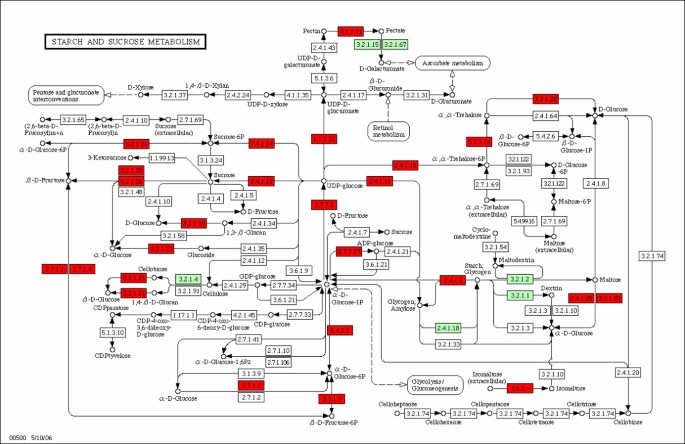
使用GoMp包对组装上定义的10577个不同转录本进行基因功能注释(参见“方法”)。因此,序列被赋予基因本体(GO)术语[40]并映射到京都基因与基因组百科全书(KEGG)的代谢途径[41基于序列相似性。在10577个序列中,8227个(78%)用这些受控词汇表的术语进行了注释,而2350个(22%)没有指定函数。KEGG Orthology (KO)系统的使用[42注释序列使我们能够以拟南芥图为模板绘制我们文库中转录本的路径图(图2).我们将木薯序列分配到114个中的101个答:芥根据电子注释,我们可能已经捕获了大约60%(1205个酶活性中的732个)的拟南芥酶活性(KO基因)(见表3.).
根据拟南芥的注释,我们捕获了一些与70%以上相关酶同源的基因全长转录本,这些几乎完整的通路包括:“糖酵解/糖异生”(100%)、“淀粉和蔗糖代谢”(76%)、“蛋白酶体”(84%)、“碳固定”(92%)、“丙酮酸代谢”(79%)、“类固醇生物合成”(70%)、“戊糖磷酸途径”(93%)和“二苯乙烯、香豆素和木质素生物合成”(73%)等。淀粉代谢的代谢途径在木薯中特别有趣;该生物聚合物的合成是一个相对简单的过程,依赖于三种主要酶的活性:ADP葡萄糖焦磷酸化酶(ADPGPase, 2.7.7.27)、淀粉合成酶(SS, 2.4.1.11)和淀粉分支酶(SBE, 2.4.1.18) [43];如图所示2,我们捕获了ADPGPase和SS的全序列,路径可视化也表明在文库中未发现SBE。在木薯中鉴定了3个ADPGPase转录本;其中包括该酶小亚基的一个序列和大亚基的两个可选剪接变体。对于SS酶,我们发现了5个序列,它们似乎是两种酶亚型的替代转录本。
分子标记是作物改良的重要工具。使用SSRFinder Perl脚本集[44]和AutoSNP包[45为捕获的10577份转录本中的1725份设计了1391个SSR和2356个SNP标记;这些标记存储在一个关系数据库中,它们链接到序列的功能注释。经过这一过程,我们获得了22个木薯转录本中的7个的SNP或SSR标记,这些转录本被鉴定为淀粉和蔗糖代谢途径中的酶,这些酶包括SS、淀粉磷酸化酶(2.4.1.1)、蔗糖磷酸合成酶(2.4.1.14)和udp -葡萄糖6脱氢酶(1.1.1.22),这些酶已知对淀粉生产有影响[46,47].在剩下的1718个与分子标记相关的基因中,563个位于85个不同通路的基因内。
为了在这种耐受性极强的作物中识别胁迫诱导基因,我们将我们的序列与RIKEN拟南芥全长(RAFL) cDNA微阵列识别的干旱和寒冷诱导基因集合进行了比较。33].表格4在我们的文库中显示了那次实验中有重大命中的基因;在拟南芥的44个胁迫诱导基因中,我们捕获了181个木薯转录本,与其中的32个具有显著的序列相似性(e-value < 1e-10)。我们发现更多木薯转录本的基因包括以下几类酶:水通道蛋白、内木葡聚糖转移酶、β -葡萄糖苷酶、硫醇蛋白酶、热休克蛋白(HSPs)、抗坏血酸过氧化物酶、硫氧还蛋白、乙烯响应元件结合(EREB)/ ap2样蛋白和过氧化氢酶。
基因对应和同源(共正交)检测
在以下各节中,“同源序列”一词将用于指定从被比较物种的最后一个共同祖先的单一祖先基因中派生出来的序列[35].这个定义允许在这些基因组中存在一个基因的单拷贝(一对一的同源关系),以及最近发生的基因复制,一个物种中的两个或多个基因与另一个物种中的一个基因是同源关系的情况。在后一种情况下,在物种形成事件后由基因复制产生的基因被称为In - parogs,它们是其他物种中相应基因的协同正交物。
我们的目标不是提供全长文库中捕获的转录本的完整分类,分为正序和反序,而是利用现有的方法来描述这个基因集合的一些有趣的特征。首先,当我们将木薯转录本与其他物种的转录本进行比较时,我们使用blast来指定互为最佳匹配(RBHs)的基因对。在这种方法中,rbh被解释为潜在的一对一正交,而协正交被忽略;这样,我们就能够在未配对的序列集(包括可能的基因复制和替代转录本)中寻找过度代表的氧化GO术语,作为识别在木薯转录本注释中特别频繁的功能类别的一种手段。其次,我们使用blast从一组已经删除了可选转录本的序列中识别假定的内谬误;这样我们就能得到一份近期潜在的基因复制列表,以供进一步分析。
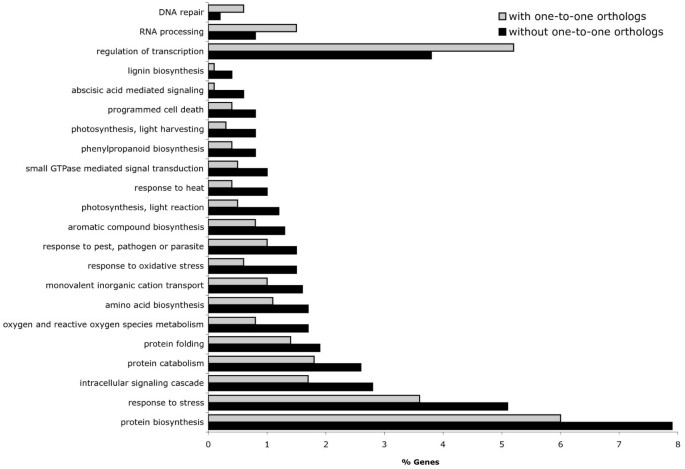
RBH标准用于定义木薯和其他三个物种之间的一对一的同源基因对:r .普通的,p . trichocarpa,答:芥.我们分别找到了3280、5392和4678个共享序列。然后,为了评估在这些条件下仅在木薯中发现的序列的功能,我们比较了被分配到一个同源对的序列的GO注释和那些没有被分配到同源对的序列的注释。结果(图4)3.),含有木薯序列(p值< 0.05,Pearson卡方检验)的GO术语没有被分配到一对一对,包括“蛋白质生物合成”、“细胞蛋白质分解代谢”、“激素介导信号传递”、“氨基酸生物合成”、“对害虫、病原体或寄生虫的反应”和“木质素生物合成”等。另一方面,富含同源对序列的氧化石墨烯术语包括:“DNA修复”、“转录调控”和“RNA处理”。
除了直接与应激反应相关的氧化石墨烯术语,如“对高光强度的反应”、“对热的反应”或“对氧化应激的反应”,没有相互最佳击打的序列经常被注释为与应激反应分子合成相关的术语,如“苯丙基生物合成”[48];此外,它们还被注释了一些术语,用来描述在压力下增强的细胞过程,如“泛素依赖性蛋白质分解代谢”[49,50和脱落酸介导的信号通路[51];或者,作为第三个例子,像“光合作用,光收集”这样的术语,我们发现主要包括叶绿素结合蛋白的同源物,这可能有助于在强光胁迫下保护光系统[52].
考虑到许多没有指定的正交序列在某种程度上与应激反应有关,我们想看看这些不匹配的序列是否对应于近期的应激相关基因的基因复制,而不是单个基因的选择性拼接形式或装配错误。为此,我们从我们的序列集合中排除了被识别为其他序列的可选剪接变体的支架。然后,我们将“同源序列”定义为彼此相似且在另一个基因组中具有相同的最佳匹配的序列(参见“方法”)。
使用这种方法和方法部分提到的附加限制,我们发现了230个可能的基因复制;这些序列的GO注释如图所示4,其中大部分是与参与初级代谢和大分子修饰的酶同源的,然而,在“对刺激的反应”类别中有几个这样的重复。仔细观察这些序列可以发现其中的酶有:单脱氢抗坏血酸还原酶(MDAR)、谷胱甘肽还原酶(GLR)、谷胱甘肽还原酶(GR)、谷氨酸半胱氨酸连接酶(GCL)、铁还蛋白NADP+还原酶(FNR)和NADPH硫氧还蛋白还原酶(NTR)似乎是重复的;如图所示5,这些酶催化活性氧(ROS)清除途径的重要步骤,此外,有丝裂原活化蛋白激酶(MAPKK)和热休克蛋白(HSP20)等酶也被复制,已知在应激反应中发挥重要作用[53].对这些基因序列区域的多重序列比对和简约树的构建支持了木薯谱系特异性扩展的观点(数据未显示)。
植物细胞中活性氧的处理。木薯中可能的基因复制用粗体和下划线表示。AOX,替代氧化酶;FNR:铁还蛋白NADPH还原酶;MAPKK,丝裂原活化蛋白激酶;MDAR monodehydroascorbate还原酶;GLR glutaredoxin;GR、谷胱甘肽还原酶;谷氨酸半胱氨酸连接酶;NADPH硫氧还蛋白还原酶; HSP20, heat shock protein 20; PSII, photosystem II; PQ, plastoquinone; Cytb6f, cytochrome b6f;电脑,质体蓝素;PSI,光系统I;Fd,;超氧化物歧化酶SOD;脱落酸ABA;亚撒,抗坏血酸盐;APX型抗坏血酸盐过氧化物酶;MDA, mohodehydroascorbate;DHA, dehydroascorbate; DHAR, DHA reductase; GSSG, oxidized glutathione; GSH, glutathione; Glu, glutamate; CAT, catalase; PrxR, peroxireductase; Trx, thioredoxin; Based on [72, 75, 104]
讨论
木薯全长cDNA文库的价值
我们建立了第一个木薯EST特征的全长cDNA文库,提供了几乎相同数量的该物种的EST数据库的序列。在这个文库中捕获的大量新序列可以被认为是木薯转录组特征很差的迹象;我们的文库没有标准化,然而,我们从木薯植物在不同环境条件下的叶和根中提取mRNA,结果是一个克隆代表了7000多个不同序列的低冗余集合(图1)1).这种低冗余度可能是不同基因表达模式对构建文库的不同条件的响应的结果,也可能是我们的ESTs集与之前专注于木薯性状(如淀粉含量和对病原体的反应)的工作之间的少量重叠[25],这可能表明我们的文库中存在许多与本研究中使用的非生物胁迫相关的基因。
全长cdna对于编码序列和非翻译区域(utr)中序列特征的详细注释非常有用[30.].虽然对前者的分析有时可以通过对氨基酸基序或蛋白质结构域的注释提供有关蛋白质结构和功能的有价值的信息[54], UTR序列可通过鉴定转录因子结合基序用于基因表达分析[55,聚腺苷酸化信号[56和其他结构特征。综上所述,我们努力的重要性不仅取决于所捕获序列的数量,还取决于文库中所代表基因的质量和相关性。我们发现库中大约85%的克隆包含完整的插入;虽然这意味着一些克隆片段是不完整的,但文库中部分cDNA的功能表征仍然允许检索序列数据,用于进一步的实验设计和分离特定基因的全长cDNA。此外,仅从EST信息,我们能够确定1949个序列的5' utr和2241个序列的3' utr,以及通过对一组已知蛋白质运行BLASTX来确定732个基因的完整编码序列,这些信息对于寻找微RNA结合位点等功能特征是有价值的[57].
我们试图通过使用策划的蛋白质功能数据库来检索GO和KO术语,以尽量减少注释错误(见“方法”)。尽管这可以防止此类错误的传播,但序列相似性并不总是保证函数关系,特别是当同一性较低时[58].在我们的数据集中,用于检索函数注释的对齐中,只有15%的对齐的识别率低于50%,超过70%的对齐的e-value小于10-30年;如Joshi和Xu所示[58],这种水平的序列相似性可以预期提供了70%到80%的可能性,两个蛋白质将具有相似的功能,即使是对于最具体的氧化石墨烯术语。威尔逊及其合作者[59]也表明,当序列恒等式大于40%时,精确函数通常是很好的保守的。我们相信,测序转录本的功能类别的总体表示应该与我们所展示的不太不同,然而,在更具体的层次上,人们应该非常谨慎地验证序列相似性的功能意义。
已知的功能被分配到78%的测序克隆中,这与之前收集的木薯EST序列形成了对比,其中多达63%的序列显示与已知蛋白质没有显著的相似性[25],我们的库中注释序列的高数量可能是由于GO中注释数量的增加。与其他物种的类似报告相比,我们为更多的序列分配了一个函数[56或小麦[29]的完整长度库,在这些情况下,没有赋值函数的序列数量分别为52和44%。我们文库中的大部分序列通过序列相似性被赋予了某种功能,这一事实有助于检测和分离已知参与相关生物过程的特定基因,或者至少是具有蛋白质基序等特征的基因,这些特征将使它们成为有趣的研究目标。
虽然大多数克隆使用氧化石墨烯连接到分子功能或生物过程,但使用KEGG途径来可视化功能分配可以更容易地评估酶活性和代谢过程,我们有转录本。我们将我们的木薯序列映射到几乎所有拟南芥的路径图上;值得注意的是,只有10577个不同的转录本,相当于从木薯的电子注释中推断出的拟南芥KEGG中所代表的途径知识的一半以上。KEGG通路由参考图组成,在参考图的上方可以绘制出物种特有的酶,因为不是所有的代谢通路都是保守的,可以构建参考图,大多数KEGG通路图是中介代谢过程,只有少数调节通路,如特定的物种答:芥是可用的(42].尽管如此,具有农艺价值的性状,如淀粉含量和品质[6,胡萝卜素的产生[60],光合作用[10]和木质素生物合成[61]是木薯改良的重要目标,很容易与一些KEGG通路映射相关(图2);鉴定参与这些过程以及其他过程的木薯基因将有助于快速选择、分离和鉴定作物改良所需的关键酶,即生物合成途径中缺失的速率限制酶或催化元素。例如,ADPGPase已被证明是淀粉生物合成中的速率限制酶,其在木薯中的过表达导致根系生物量的增加[62],对这种酶以及其他酶的分子多样性的表征可能会导致更高产量作物的发展。
尽管我们的文库面向的是应激基因,但我们捕获了淀粉生物合成等过程中重要酶的完整转录本,这一事实表明,我们在木薯研究的几个方面都拥有宝贵的资源。此外,由于我们能够为直接或间接影响淀粉生产和其他分子(如蔗糖)合成之间的平衡的基因设计分子标记[47)或葡萄糖醛酸酯;这个库提供的信息有可能为标记辅助选择提供元素,如蛋白质生物合成、胡萝卜素积累、抗病等过程。
一旦在木薯中选择了一个候选基因,就可以很容易地从相应的克隆中分离出完整的cDNA序列。该序列可用于筛选所研究位点的分子多样性,以找到适合育种计划中分子标记开发的基因变体;考虑到这一点,我们使用ESTs设计SSR和SNP分子标记;如果这些标记位于感兴趣的基因内,那么它们可能对定量性状位点(QTL)的检测有价值。此外,一旦木薯基因组被揭示,它们可以作为假分子组装的工具[63],这是对基因注释中全长序列的核心作用的补充。
我们发现,我们的文库包含7796个不同基因的转录本,与高等植物的预期基因数量相比,这是一个小数目。63,64];计算得到的50698个转录本数目为今后完成该物种基因目录的工作提供了指导。然而,这个数字很可能高估了转录本的真实数量,主要是因为我们的图书馆被认为拥有丰富的稀有转录本,这些转录本源于特定的应激条件[39].
压力相关基因的转录本
我们将正常条件下植物不同组织的RNA与PPD、干旱、高温和酸性土壤胁迫下植物的RNA汇集在一起,因此我们认为我们的文库应该含有这些条件下诱导的基因转录本。如果是这种情况,分析的目标之一应该是确定文库中应激诱导基因的类型。如前所述,我们的转录本中丰富的新木薯序列可能表明根据不同的处理捕获的基因的特异性;我们捕获了RAFL微阵列检测到的大多数应激诱导基因,这一事实支持了这一假设[33大多数在其他物种中没有指定一一对应的序列都属于直接或间接与应激反应相关的GO类别。
比较我们为检测应激诱导基因所做的有RBHs和没有RBHs的序列注释是基于以下假设:如果在一组序列中有多个拼接形式、等位基因或某一基因的拷贝,那么使用RBH准则来定义第二组中的正交序列将只将其中一个分配到第二组中的对应序列,这将留下大量未配对的序列,将丰富无正交序列组中相应的GO类别。如果我们的文库包含许多与压力相关的基因转录本,那么就更有可能发现这些序列的替代剪接变体,甚至更多的组装错误;这将导致与这些序列的功能类别相对应的GO术语注释的基因的过度表示。
对于许多没有一一对应的序列与胁迫相关基因相关的事实,一个可能的解释可能是这些序列在木薯谱系中最近的基因复制的存在。我们使用了一种非常保守的方法来检测其中的一些重复(见“方法”);用这种方法,两个或两个以上的木薯序列在至少两个其他基因组中共享它们的最佳命中,被认为是潜在的同源基因。为了创建一种方法,在处理近期和古代基因复制的同时,建立基因组之间的基因对应关系,Kellis和合作者定义了一个最佳无歧义子集作为一组基因,使集合内任何基因的所有最佳命中都包含在集合内,而集合外的任何基因的最佳命中都不包含在集合内[65];因此,我们通过找到一对潜在的同源序列来定义木薯中潜在的基因复制,其中只有一个序列是第二个基因组中的基因的最佳匹配;通过这种方式,我们避免了假阳性的报告,在这种情况下,候选人在谬误中存在正序,但两个最佳爆炸命中都只指向其中一个[66],在许多情况下是由于不同的序列长度。
如图所示4,近5%的潜在重复被标注在“对刺激的反应”GO类别中;尽管这个数字看起来很小,但我们已经看到,并不是所有与应激反应相关的序列都标注在那个类别中。例如,许多参与泛素介导的蛋白质水解的序列对应于“大分子代谢”类别中的很大一部分复制。对我们发现的230个可能的基因复制的详细检查显示,它们与几种与ROS代谢相关的酶同源;自热(67],干旱[68,酸性土壤[69]和产后抑郁[70]已被报道诱导ROS产生,我们认为这些潜在的重复是一个很好的例子,说明了完整的文库如何为木薯胁迫响应特征的潜在机制提供线索。
植物中的大多数ROS是由叶绿体的梅勒反应中电子转移产生的超氧化物向分子氧的二叉突变而产生的[71].在应激反应中,H2O2产量可以增加[72这个过程有时涉及脱落酸介导的气孔关闭和CO的减少2光合作用的水平[73].高剂量的过氧化氢可导致超敏细胞死亡,而低剂量的过氧化氢可触发对不同应激条件的保护功能[74],这种ros介导的应激反应激活依赖于一个由许多基因组成的网络,这些基因平衡了ros清除和ros产生蛋白质的结果[75].我们在木薯转录组样本中发现,H2O2清除似乎是重复的(图5);我们提出,这些重复可能至少在一定程度上解释了木薯的耐胁迫特性,这可能是由于对一些已经描述过的其他植物的ROS信号机制的更严格控制。我们的假设是建立在大量文献的基础上的,这些文献都是关于MDAR等分子的重要性的。76], FNR [77),正常(关系78], GLR [79,80),硫氧还蛋白(81和谷胱甘肽[82,83在植物对环境胁迫的敏感性方面。
据认为,Manihot属通过最近的异源多倍体化而出现,这一事件被认为是导致杂交的快速物种形成和弱种间屏障[84].我们假设,潜在的应激反应基因的复制可能起源于这一多倍体事件;因为研究表明,大多数基因在这些事件发生后会迅速沉默,除非它们的功能多样化[85],观察一些检测到的重复是否显示亚功能化的证据将是有趣的。实现这一目标的第一步将是评估不同器官的表达谱,并在各种条件下对该文库检测到的似是而非的基因复制进行评估。这可以通过构建微阵列很容易地完成,为此,全长文库将再次成为无价的资源[33].
结论
木薯研究界肯定会从这里提供的完整图书馆中受益。对测序无性系的分析已经为该作物的改良提供了诱人的研究方向。深入分析木薯的基因特征和基因家族,并对其指定的功能进行微调,可以为提高在逆境环境下的产量提供必要的元素,而且,我们从这种作物中学到的知识将引导我们在其他重要但不那么耐受力的植物物种上取得重要成就。
方法
植物材料和非生物胁迫
按照Chang报道的方法提取总RNAet al。[86]从木薯品种MTAI16在表中描述的条件下5.在不同时间点用于RNA提取和非生物胁迫处理的植物生长在温室的塑料花盆中;9个月大的植株直接从田间收获。
在高铝低pH处理中,植物被放置在含200 μM AlCl的连续曝气溶液中3.和200 μM CaCl2与pH值4.2。在热处理过程中,它们在42°C的温度下孵育。为了应对干旱,将植物从花盆中取出,然后用水清洗它们的根部,用毛巾擦干,然后置于室温下。PPD处理时,取9个月大的根,切远端和近端,用塑料覆盖块茎近端,切成2 cm的切片,然后提取RNA。
RNA的制备及全长cDNA文库的构建
保利(A)+在手册中给出的标准条件下,用μMACS mRNA Isolation Kit (Miltenyi Biotec)制备RNA。从聚(A)中构建了全长cDNA文库+利用海藻糖热激活逆转录酶生物素化CAP诱捕器法合成RNA [31].合成的双链cdna用Bam你好,Xho我,和扎成Bam你好,萨尔Lambda FLC-III向量的I个位点[87].
EST测序
用RCA方法直接从甘油砧板的384个细菌培养物中扩增每个克隆的DNA [88使用TempliPhi HT DNA扩增试剂盒(GE Healthcare,英国)。使用ABI 3700全自动毛细管DNA测序仪(Applied Biosystems)对19968个克隆进行末端测序。M13-21引物(5'-TGTAAAACGACGGCCAGT-3')和1233引物(5'-AGCGGATAACAATTTCACACAGGA-3')分别用于正向测序和反向测序。
序列数据和装配的修剪
使用Phred程序对原始序列数据进行基调用[89],丢弃每个原始序列边缘的低质量区域(Phred质量评分< 20,重复碱基数超过20)。对于矢量序列检测,我们使用sim4程序[90].省略此修剪过程后长度小于100碱基的序列数据。此外,如果一个序列中单个核苷酸的重复长度超过其总长度的10%,我们将拒绝该序列。est由CAP3 [91使用默认参数。修剪后的序列提交到登录号为DB920056-DB955455的DDBJ,同时将跟踪文件上传到跟踪存档[92根据编号1918207201至191824260。
全长cDNA文库质量
我们计算了满足以下条件的样本克隆的全长比:一个克隆同时具有5'和3'序列数据,e值在5'序列fastx34搜索[93比对NCBI-nr数据集的值小于1e-30,且对齐帧为正方向。如果以甲硫氨酸和聚(a)为起始的5'序列数据进行fastx34比对,则满足上述条件的克隆被鉴定为“全长”克隆。+Tail存在于3'序列中。
脚手架施工
为了获得非冗余的转录本集,根据克隆名称对这些转录本进行了聚集。对于这个,'。ace的文件从CAP3输出被解析来构建支架,也就是说,一组序列代表一个唯一的转录本,可以推断片段的相对位置和方向。使用克隆名称,对应于给定克隆两端的contigs或singleton通过在两个序列中间添加20 Ns连接在一起。因为20是BLAST搜索中的默认窗口大小,所以这些Ns不会干扰BLAST分析。
序列的功能注释
一旦这些支架被创建,序列就被引入到CIAT之前开发的用于DNA序列功能注释(GoMp)的管道中。简而言之,该系统使用高质量的策划注释数据库中的蛋白质序列,GO lite [94和TAIR [95],并使用BLASTX将各自的GO和KO注释转移到序列相似性显著(e-value < 1e-5)的序列上[96].在blast报告的前五次点击中,我们寻找与一个具有指定GO术语的序列和一个具有指定KO访问的序列的最佳对齐,如果这两个序列不相同,则报告两个注释,以便从两个受控词汇表分配一个功能注释。注释随后存储在关系数据库中,并可通过web cgi脚本进行导航。使用KEGG web服务API基于KO访问绘制路径图[96].
替代成绩单的识别
为了识别可选的剪接变体,我们使用了两种方法。首先,我们使用得到的CAP3组装和克隆id信息来选择由两个或两个以上的contigs组成的组,这些组由克隆的序列读取组成,其中第二个读取只组装在一个contig中。其次,我们对剩下的序列自身进行BLASTN分析,并过滤出所有超过100个碱基对的高分对的识别率都超过98%的那些对。
编码序列和utr的注释
对于编码序列和utr的注释,用于为序列分配函数的blast报告被解析,以提取已知蛋白质的开始或结束周围的对齐,然后在相同的阅读框中,这些位置被用于寻找定义编码序列和ESTs中utr的开始和结束点的开始或停止密码子。
分子标记的设计
我们使用了Perl脚本的SSRFinder集合[44],以便找到微卫星重复序列,并在其周围自动设计PCR引物。此外,e-PCR软件[97]用于去除退火到多个转录本的非特异性引物对。
AutoSNP软件[45]被用来寻找单核苷酸多态性。为此,我们创建了第二个CAP3组装,包括我们的序列和来自木薯的est MBRA685, MPER183, CM523-7, MCOL1522和SG107-35。我们使用引物3在这些snp周围设计引物对[98以及使用SBEprimer包的单碱基扩展引物[99].
基因预测r .普通的
出版的大会萝藦基因组(GenBank接入AASG01000001至AASG01036140)使用RepeatMasker屏蔽重复元素[One hundred.),拟南芥重复库。利用GenBank(2006年10月)中大戟科所有ESTs训练TIGRSCAN进行基因预测[101掩模序列。考虑orf大于200碱基对的蛋白质编码基因;这导致了一组22734个预测基因用于进一步的比较分析m .耐.虽然这是一个鸟枪基因组草图(约300 MB中的129 MB, 2倍覆盖率,最大contig长度36140,N50长度= 3683),但它仍然被认为是目前大戟科最完整的基因组序列,具有分析价值。
比较基因组学分析
的预测和/或验证转录本的序列拟南芥,杨树trichocarpa而且萝藦是从基因组序列中下载或确定的。然后使用TBLASTX进行几次互反爆炸分析,e-value < 1e-5,并进行解析,以检索其他数据库中每种木薯序列的最佳命中值和木薯中其他物种每种序列的最佳命中值。因此,我们建立了一个表,代表四个物种的最佳选择网络。
一对一的正交线图被认为是互惠的最佳爆炸命中[102],有了这个定义,我们就能把我们的序列分为在任何其他物种中具有一一对应的序列和不具有对应关系的序列。使用WEGO web服务器对两组序列的注释进行比较[103],两组中有一组表达过多(p-value < 0.05 Pearson卡方检验,每类基因数超过5个),认为转录本过多,需要进一步分析。
基于相同的blast结果网络,我们发现了in -类比,其定义如下:C1和C2是木薯序列,C1和C2是彼此的BLASTN命中值(e-value < 1e-10),并且在用于比较的数据集中,至少两个序列的最佳命中值对应于单个基因。如果数据集A中C1和C2的最佳命中是序列A1,它们在数据集B中的最佳命中是序列B1,那么在木薯中A1和B1的最佳命中必须是C1或C2。最后,对于所有满足上述条件的C1和C2对,如果其中只有一个是数据集a中序列的最佳命中,而其中只有一个是数据集B中序列的最佳命中,则认为它们是平行的。
参考文献
- 1.
木薯:热带地区的一种基本能源。科学通报,2002,27(4):555 - 562。10.1126 / science.7134971。
- 2.
农发基金、粮农组织:世界木薯经济。事实、趋势和前景。罗马,农发基金,粮农组织,2000年。
- 3.
Tonukari NJ:木薯和淀粉的未来。生物技术电子杂志。2004,7(1): 5-8。
- 4.
用流动发酵单胞菌和舒张酵母菌共固定化木薯淀粉生产乙醇。生物技术与工程学报,2001,29(6):366 - 366。10.1263 / jbb.92.560。
- 5.
Raven P, Fauquet C, Swaminathan MS, Borlaug N, Samper C:基因组测序的下一个方向是什么?科学通报,2006,31(5):563 - 566 /科学通报。
- 6.
Sriroth K, Piyachomkwan K, Wanlapatit S, Oates C:木薯淀粉技术:泰国经验。淀粉。2000,52:439-449。10.1002 / 1521 - 379 x (200012) 52:12 < 439:: AID-STAR439 > 3.0.CO;双电子。
- 7.
El-Sharkawy MA, Cadavid LF:木薯在不同生长阶段对长期水分胁迫的响应。中国农业科学,2002,38(3):333-350。10.1017 / S001447970200306X。
- 8.
木薯生物学和生理学。植物生理学报,2004,24(4):481- 481。10.1007 / s11103 - 005 - 2270 - 7。
- 9.
Raheem D, Chukwuma C:木薯食品及其与尼日利亚和其他非洲国家的关系。农业与人类价值。2001,18:383-390。10.1023 /: 1015233606665。
- 10.
El-Sharkawy MA:热带木薯光合作用、生产力、生态生理学和对环境压力响应的国际研究。植物学报,2006,44(4):481-512。10.1007 / s11099 - 006 - 0063 - 0。
- 11.
Oguntunde P:有限土壤有效水和不同蒸发需求下木薯全株水分利用和冠层导度。植物生态学报,2005,27(4):371- 371。10.1007 / s11104 - 005 - 0375 - z。
- 12.
木薯对水分胁迫的响应。植物与土壤学报,1998,21(3):366 - 366。10.1007 / BF02370950。
- 13.
Tomkins J, Fregene M, Main D, Kim H, Wing R, Tohme J:木薯(Manihot esculenta Crantz)抗虫抗病基因位置克隆的细菌人工染色体文库资源。植物生理学报,2004,27(4):555-561。10.1007 / s11103 - 004 - 5045 - 7。
- 14.
Bellotti A, Schoonhoven A:木薯的螨和害虫。昆虫学报,1998,23:39-67。10.1146 / annurev.en.23.010178.000351。
- 15.
Andersen MD, Busk PK, Svendsen I, Moller BL:从木薯(Manihot esculenta Crantz)中提取细胞色素P-450,催化生氰葡萄糖苷利纳青苷和lotaustralin生物合成的第一步。重组酶的克隆、在毕赤酵母中的功能表达及底物特异性。中国生物医学工程学报,2000,27(3):366 - 366。10.1074 / jbc.275.3.1966。
- 16.
Kawano K: 30年的木薯育种的生产力-生物和社会因素的成功。作物科学,2003,43:1325-1335。
- 17.
faquet CM, Tohme J:全球木薯遗传改良伙伴关系。植物生理学报,2004,25(4):426 - 426。10.1007 / s11103 - 004 - 0157 - 7。
- 18.
保罗KP:作物改良的植物生物技术。生物技术学报,1995,13(4):673-693。10.1016 / 0734 - 9750(95) 02010 - 1。
- 19.
Jorge V, Fregene M, Vélez C, Duque M, Tohme J, Verdier V:田间对Xanthomonas axonopidis的抗性QTL分析。manihotis木薯。中国科学(d辑)。10.1007 / s001220051683。
- 20.
Anderson JV, Delseny M, Fregene MA, Jorge V, Mba C, Lopez C, Restrepo S, Soto M, Piegu B, Verdier V, Cooke R, Tohme J, Horvath DP:木薯和大盘科其他物种的EST资源。植物生理学报,2004,27(4):533 - 536。10.1007 / s11103 - 004 - 5046 - 6。
- 21.
Waterhouse PM, Helliwell CA:通过rna诱导基因沉默探索植物基因组。科学通报,2003,4(1):29-38。10.1038 / nrg982。
- 22.
Siritunga D, Sayre RT:无氰转基因木薯的一代。植物生态学报,2003,26(3):367-373。10.1007 / s00425 - 003 - 1005 - 8。
- 23.
Jorgensen K, Bak S, Busk PK, Sorensen C, Olsen CE, Puonti-Kaerlas J, Moller BL:叶片和块茎中产氰苷含量减少的木薯植株。产氰苷的分布、合成和转运的部位以及RNA干扰技术对生物合成的阻断。植物生理学报,2004,25(1):369 - 371。10.1104 / pp.105.065904。
- 24.
罗M,见鬼P,他G,霍尔布鲁克C, Bausher M,李R:一代的表达序列标签(est)基因发现和标记发展种植花生。作物学报,2005,45:343-356。
- 25.
Lopez C, Jorge V, Piegu B, Mba C, Cortes D, Restrepo S, Soto M, Laudie M, Berger C, Cooke R, Delseny M, Tohme J, Verdier V:木薯5700个表达基因的单基因目录。植物生理学报,2004,25(4):541-554。10.1007 / s11103 - 004 - 0123 - 4。
- 26.
- 27.
Seki M, Satou M, Sakurai T, Akiyama K, Iida K, Ishida J, Nakajima M, Enju A, Narusaka M, Fujita M, ooono Y, Kamei A, Yamaguchi-Shinozaki K, Shinozaki K: RIKEN拟南芥全长(RAFL) cDNA及其在非生物胁迫条件下表达谱分析的应用中国生物医学工程学报,2004,38(4):369 - 369。10.1093 / jxb / erh007。
- 28.
Zavolan M, van Nimwegen E, Gaasterland T:通过映射到小鼠基因组识别小鼠全长cdna的剪接变异。中国生物医学工程学报,2004,12(9):1377-1385。10.1101 / gr.191702。
- 29.
Ogihara Y, Mochida K, Kawaura K, Murai K, Seki M, Kamiya A, Shinozaki K, Carninci P, Hayashizaki Y, Shin IT, Kohara Y, Yamazaki Y:六倍体小麦小穗全长cDNA文库的构建及其表达标签的大规模测序分析中国生物医学工程学报,2004,29(4):369 - 369。10.1266 / ggs.79.227。
- 30.
Alexandrov NN, Troukhan ME, Brover VV, Tatarinova T, Flavell RB, Feldmann KA:利用全长cdna发现拟南芥基因和基因组的特征。植物生物学杂志,2006,60(1):69-85。10.1007 / s11103 - 005 - 2564 - 9。
- 31.
Seki M, Narusaka M, Kamiya A, Ishida J, Satou M, Sakurai T, Nakajima M, Enju A, Akiyama K, ooono Y, Muramatsu M, Hayashizaki Y, Kawai J, Carninci P, Itoh M, Ishii Y, Arakawa T, Shibata K, Shinagawa A, Shinozaki K:拟南芥cDNA全长集合的功能注释。科学通报,2002,29(5):561 - 561。10.1126 / science.1071006。
- 32.
Cortés D, Reilly K, Okogbenin J, Beeching JR, Iglesias C, Tohme J:木薯(Manhiot esculenta Crantz)采后生理退化(PPD)的创伤反应基因定位。植物学报,2002,32(4):447 - 453。10.1023 /: 1020695719706。
- 33.
Seki M, Narusaka M, Abe H, Kasuga M, Yamaguchi-Shinozaki K, Carninci P, Hayashizaki Y, Shinozaki K:利用全长cDNA微阵列检测1300个拟南芥基因在干旱和寒冷胁迫下的表达模式植物学报,2001,13(1):61-72。10.1105 / tpc.13.1.61。
- 34.
Djerbi S, Lindskog M, Arvestad L, Sterky F, Teeri TT:黑杨木(Populus trichocarpa)基因组序列揭示了18个保守的纤维素合成酶(CesA)基因。植物学报,2005,21(5):739-746。10.1007 / s00425 - 005 - 1498 - 4。
- 35.
Koonin EV: Orthologs, parogs和进化基因组学。科学通报,2005,39:309-338。10.1146 / annurev.genet.39.073003.114725。
- 36.
Nassar NM:木薯,Manihot esculenta Crantz,遗传资源:作物起源,进化及其与野生亲缘关系。中国生物医学工程学报,2002,1(4):298-305。
- 37.
理化学生物资源中心。[http://www.brc.riken.go.jp/lab/epd/Eng/]
- 38.
王建平,Lindsay BG, Leebens-Mack J, Cui L, Wall K, Miller WC, dePamphilis CW: EST聚类误差评价与校正。生物信息学,2004,20(17):2973-2984。10.1093 /生物信息学/ bth342。
- 39.
王建平,Lindsay BG,崔玲,Wall PK, Marion J,张娟,dePamphilis CW:单文库或多文库EST测序中基因捕获预测和重叠估计。生物信息学,2005,6:300-10.1186/1471- 25-6-300。
- 40.
哈里斯马,克拉克J,爱尔兰,凯文J, ashburn M, Foulger R, Eilbeck K,刘易斯年代,马歇尔B, Mungall C,里希特J,鲁宾通用,布莱克是的,布尔特C,多兰M, Drabkin H,埃皮格JT,希尔DP,倪L, Ringwald M·R,樱桃JM,克里斯蒂KR,使用MC,德怀特党卫军,恩格尔,Fisk DG,赫希曼我,香港EL,纳什RS, Sethuraman, Theesfeld CL, Botstein D, Dolinski K, Feierbach B, Berardini T, Mundodi年代,Rhee SY, Apweiler R,巴雷尔D加们E, E的黯淡,李V, Chisholm R, Gaudet P,基布W,Kishore R, Schwarz EM, Sternberg P, Gwinn M, Hannick L, Wortman J, Berriman M, Wood V, de la Cruz N, Tonellato P, Jaiswal P, Seigfried T, White R:基因本体论(GO)数据库和信息学资源。核酸学报,2004,32(数据库问题):D258-61。
- 41.
Kanehisa M, Goto S, Hattori M, Aoki-Kinoshita KF, Itoh M, Kawashima S, Katayama T, Araki M, Hirakawa M:从基因组学到化学基因组学:KEGG的新进展。核酸学报,2006,34(数据库问题):D354-7。10.1093 / nar / gkj102。
- 42.
Kanehisa M, Goto S: KEGG:京都基因和基因组百科。核酸学报,2000,28(1):27-30。10.1093 / nar / 28.1.27。
- 43.
马丁·史密斯:淀粉生物合成。植物生态学报,2004,24(6):673 - 679。10.1105 / tpc.7.7.971。
- 44.
Sharopova N、McMullen MD、Schultz L、Schroeder S、Sanchez-Villeda H、Gardiner J、Bergstrom D、Houchins K、Melia-Hancock S、Musket T、Duru N、Polacco M、Edwards K、Ruff T、Register JC、Brouwer C、Thompson R、Velasco R、Chin E、Lee M、Woodman-Clikeman W、Long MJ、Liscum E、Cone K、Davis G、Coe EH:玉米SSR标记的开发与定位植物生理学报,2004,25(5):366 - 366。10.1023 /: 1014868625533。
- 45.
Barker G, Batley J, H OS, Edwards KJ, Edwards D:利用自snp在表达序列标签数据中基于冗余的序列多态性检测。生物信息学,2003,19(3):421-422。10.1093 /生物信息学/ btf881。
- 46.
小麦淀粉磷酸化酶与淀粉代谢的关系。植物生理学报,2004,25(4):369 - 369。10.1093 /卡式肺囊虫肺炎/ pch170。
- 47.
胡伯德,杨志强,杨志强。蔗糖磷酸合酶在香蕉成熟过程中蔗糖生物合成中的作用及其与呼吸更年期的关系。植物生理学报,1998,25(1):1- 8。
- 48.
狄克逊RA, Paiva NL:应力诱导苯丙类代谢。植物生态学报,2003,22(4):366 - 366。10.1105 / tpc.7.7.1085。
- 49.
熊林,朱建军:植物非生物胁迫信号转导的分子和遗传学研究。植物工程学报,2001,32(2):366 - 366。10.1034 / j.1399-3054.2001.1120202.x。
- 50.
泛素在植物衰老和胁迫反应中的作用。植物科学的趋势。科学通报,1996,1(10):331-335。
- 51.
Alves AAC, Setter TL:水分亏缺条件下木薯脱落酸积累与渗透调节。环境与实验植物学。2004年,第51节,第3期:259-271。10.1016 / j.envexpbot.2003.11.005。
- 52.
Li XP, Bjorkman O, Shih C, Grossman AR, Rosenquist M, Jansson S, Niyogi KK:一种调节光合光收集必需的色素结合蛋白。自然学报,2000,403(6768):391-395。10.1038 / 35000131。
- 53.
丝裂原激活蛋白激酶级联作为应激反应的调节器。中国科学(d辑:自然科学版)。10.1111 / j.1749-6632.1998.tb08987.x。
- 54.
卡瓦依J,品川,柴田K,吉野M,伊藤M, Ishii Y,荒川T, Hara, Fukunishi Y, Konno H,足立J,福田,Aizawa K, Izawa M, Nishi K, Kiyosawa H,近藤年代,山中我,齐藤T,冈崎Y, Gojobori T波诺H, Kasukawa T,齐藤R, Kadota K,松田H, ashburn M, Batalov年代,Casavant T,她W, Gaasterland T, Gissi C, B,国王Kochiwa H,屈尔P,刘易斯,松尾Y, Nikaido我Pesole G, Quackenbush J, Schriml LM,史陶比尔F,铃木R,获利,M,瓦格纳L, Washio T,酒井法子K,Okido T, Furuno M, Aono H, Baldarelli R, Barsh G,布雷克J, Boffelli D, Bojunga N, Carninci P, de Bonaldo MF Brownstein乔丹时,布尔特C,弗莱彻C, Fujita M, Gariboldi M, Gustincich年代,希尔维,霍夫曼M,休谟哒,Kamiya M,李NH,里昂P,马尔基奥尼L, Mashima J, Mazzarelli J, Mombaerts P, Nordone P, B环Ringwald M,罗德里格斯,Sakamoto N,佐佐木H,佐藤K, Schonbach C, Seya T,柴田Y,斯托奇KF,铃木H, Toyo-oka K,王KH, Weitz C,惠塔克C, Wilming L, Wynshaw-Boris,吉田K,Hasegawa Y, Kawaji H, Kohtsuki S, Hayashizaki Y:小鼠全长cDNA的功能注释。自然学报,2001,40(6):685-690。10.1038 / 35055500。
- 55.
Vandepoele K、Vlieghe K、Florquin K、Hennig L、Beemster GT、Gruissem W、Vande Peer Y、Inze D、de Veylder L:植物E2F潜在靶基因的全基因组鉴定。植物生理学报,2004,25(1):326 - 326。10.1104 / pp.105.066290。
- 56.
贾军,付军,郑军,周晓,怀军,王建军,王梅,张勇,陈晓,张军,赵军,苏铮,吕勇,王刚:胁迫诱导玉米(Zea mays L.)幼苗2073个全长cdna的注释和表达谱分析。植物学报,2006,32(5):366 - 366。10.1111 / j.1365 - 313 x.2006.02905.x。
- 57.
Rhoades MW, Reinhart BJ, Lim LP, Burge CB, Bartel B, Bartel DP:植物microRNA靶标的预测。中国生物医学工程学报,2002,38(4):533 - 533。10.1016 / s0092 - 8674(02) 00863 - 2。
- 58.
Joshi T, Xu D:序列相似度与函数相似度关系的定量评价。中国生物医学工程学报,2007,26(4):366 - 366。
- 59.
Wilson CA, Kreychman J, Gerstein M:评估基因组学注释转移:通过传统和概率评分量化蛋白质序列、结构和功能之间的关系。中华分子生物学杂志,2000,297(1):233-249。10.1006 / jmbi.2000.3550。
- 60.
Iglesias C, Mayer J, Chavez L, Calle F:木薯根中胡萝卜素含量的遗传潜力与稳定性。植物学报,1997,44(4):367-373。10.1023 /: 1002962108315。
- 61.
Demirbas A:从纤维素生物质资源中提取乙醇。绿色能源学报。2004,1(1):79-87。10.1081 / ge - 120027885。
- 62.
Ihemere U, Arias-Garzon D, Lawrence S, Sayre R:木薯基因改造提高淀粉产量。植物生态学报,2006,4(4):463 - 466。10.1111 / j.1467-7652.2006.00195.x。
- 63.
国际。水稻。基因组。测序。项目:基于地图的水稻基因组序列。自然学报,2005,436(7052):793-800。10.1038 / nature03895。
- 64.
拟南芥计划:分析开花植物拟南芥的基因组序列。自然学报,2000,408(6814):796-815。10.1038 / 35048692。
- 65.
作者:Kellis M, Patterson N, Birren B, Berger B, Lander ES:比较基因组学方法:基因组对应、基因识别和调控基序发现。中国生物医学工程学报,2004,11(2-3):319-355。10.1089 / 1066527041410319。
- 66.
沃尔DP,弗雷泽HB,赫什AE:检测假定的矫形仪。生物信息学,2003,19(13):1710-1711。10.1093 /生物信息学/ btg213。
- 67.
Dat JF, Lopez-Delgado H, Foyer CH, Scott IM:水杨酸或热驯化诱导的芥菜幼苗耐热过程中H2O2和过氧化氢酶的平行变化。植物生理学报,2004,24(4):369 - 369。10.1104 / pp.116.4.1351。
- 68.
王伟,Vinocur B, Altman A:植物对干旱、盐和极端温度的响应:基因工程的抗逆性。植物生态学报,2003,26(1):1-14。10.1007 / s00425 - 003 - 1105 - 5。
- 69.
Ezaki B, Gardner RC, Ezaki Y, Matsumoto H:转拟南芥铝诱导基因的表达可改善铝胁迫和/或氧化胁迫。植物生理学报,2004,22(3):366 - 366。10.1104 / pp.122.3.657。
- 70.
李丽丽,郭德明,郭德明,陈晓明,陈晓明。木薯采后生理退化过程中的氧化应激反应。植物生理学报,2004,25(4):366 - 366。10.1007 / s11103 - 005 - 2271 - 6。
- 71.
活性氧和氧化爆发:在植物胁迫、衰老和信号转导中的作用。中国生物医学工程学报,2005,29(5):369 - 369。
- 72.
Mittler R:氧化应激,抗氧化剂和抗应激能力。植物动态,2002,7(9):405-410。10.1016 / s1360 - 1385(02) 02312 - 9。
- 73.
蒋敏,张娟:脱落酸对玉米幼苗叶片活性氧、抗氧化防御系统及氧化损伤的影响。植物细胞物理学报,2001,42(11):1265-1273。10.1093 /卡式肺囊虫肺炎/ pce162。
- 74.
王晓燕,赵丽玲,张晓燕,等。植物抗氧化能力的研究进展。中国生物工程学报,2000,29(6):369 - 369。10.1073 / pnas.97.6.2940。
- 75.
Mittler R, Vanderauwera S, Gollery M, Van Breusegem F:植物活性氧基因网络。植物科学进展,2004,9(10):490-498。10.1016 / j.tplants.2004.08.009。
- 76.
Leterrier M, Corpas FJ, Barroso JB, Sandalio LM, del里约热内卢LA:过氧化物酶体单脱氢抗坏血酸还原酶。环境胁迫条件下基因组克隆特性及功能分析。植物生理学报,2004,25(4):369 - 369。10.1104 / pp.105.066225。
- 77.
氧化胁迫导致甲基紫罗兰处理的植物类囊体膜中铁氧还蛋白- nadp +还原酶的溶解。植物生理学报,2004,24(4):378 - 378。10.1104 / pp.115.4.1721。
- 78.
Serrato AJ, Perez-Ruiz JM, Spinola MC, Cejudo FJ:一种新的NADPH硫氧还蛋白还原酶,定位于拟南芥叶绿体,其缺乏导致对非生物胁迫的过敏。中国生物医学工程学报,2004,29(4):369 - 369。10.1074 / jbc.M404696200。
- 79.
植物glutaredoxin:仍是神秘的还原体系。中国生物医学工程学报,2004,29(4):366 - 366。10.1007 / s00018 - 004 - 3410 - y。
- 80.
王晓燕,王晓燕,王晓燕。植物glutaredoxin系统的基因组分析。中国生物医学工程学报,2006,29(4):369 - 369。10.1093 / jxb / erl001。
- 81.
高等植物硫氧还蛋白h体系的研究。植物生理与生物化学,2004,42(4):265-271。10.1016 / j.plaphy.2004.03.002。
- 82.
梅·梅,李文华,李文华,李文华。植物中谷胱甘肽的内稳态及其对环境感知和植物发育的影响。J exp机器人。中国科学:d辑,1998,34(4):369 - 369。10.1093 / jexbot / 49.321.649。
- 83.
黄晓明,王晓明,王晓明,Király Z:谷胱甘肽及其相关酶在植物对环境胁迫应答中的作用。中国地质大学学报(自然科学版),1998,38(4):561 - 561。10.1111 / j.1749-6632.1998.tb09000.x。
- 84.
木薯的细胞遗传学和进化。中国生物医学杂志,2004,23(4):1003-1014。
- 85.
Adams KL, Wendel JF:植物的多倍体和基因组进化。植物生物学杂志,2005,8(2):135-141。10.1016 / j.pbi.2005.01.001。
- 86.
张志强,张志强,张志强,等:一种简单高效的松木RNA分离方法。Plant Mol Biol Rep ISPMB。1993,11(2): 113-116。
- 87.
Carninci P, Shibata Y, Hayatsu N, Itoh M, Shiraki T, Hirozane T, Watahiki A, Shibata K, Konno H, Muramatsu M, Hayashizaki Y:平衡大小和长尺寸的全长,帽捕获cdna克隆到新型lambda-FLC家族的载体,可以提高基因发现率和功能分析。中国生物医学工程学报,2001,29(2):359 - 359。10.1006 / geno.2001.6601。
- 88.
Dean FB, Nelson JR, Giesler TL, Lasken RS:利用Phi 29 DNA聚合酶和多重引物滚圈扩增快速扩增质粒和噬菌体DNA。中国生物医学工程学报,2001,11(6):1095-1099。10.1101 / gr.180501。
- 89.
Ewing B, Hillier L, Wendl MC, Green P:使用phred的自动音序器跟踪的基调用。即精度评估。中国生物医学工程学报,2004,22(3):356 - 356。
- 90.
Florea L, Hartzell G, Zhang Z, Rubin GM, Miller W:一种将cDNA序列与基因组DNA序列进行比对的计算机程序。中国生物医学工程学报,2004,22(4):366 - 366。
- 91.
黄X,马丹A: CAP3: DNA序列组装程序。中国生物医学工程学报,1999,29(3):366 - 366。10.1101 / gr.9.9.868。
- 92.
- 93.
Pearson WR, Wood T, Zhang Z, Miller W: DNA序列与蛋白质序列的比较。基因组学,1997,46(1):24-36。10.1006 / geno.1997.4995。
- 94.
Camon E、Magrane M、Barrell D、Lee V、Dimmer E、Maslen J、Binns D、Harte N、Lopez R、Apweiler R:基因本体注释(GOA)数据库:在Uniprot中与基因本体共享知识。核酸Res. 2004, 32(数据库问题):D262-6。10.1093 / nar / gkh021。
- 95.
Rhee SY, Beavis W, Berardini TZ, Chen G, Dixon D, Doyle A, Garcia-Hernandez M, Huala E, Lander G, Montoya M, Miller N, Mueller LA, Mundodi S, Reiser L, Tacklind J, Weems DC, Wu Y, Xu I, Yoo D, Yoon J, Zhang P:拟南芥信息资源(TAIR):一个提供拟南芥生物学、研究材料和群落信息的集中、规划的模式生物数据库。核酸学报,2003,31(1):224-228。10.1093 / nar / gkg076。
- 96.
Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ: gapping BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。核酸学报,1997,25(17):3389-3402。10.1093 / nar / 25.17.3389。
- 97.
Schuler GD:电子PCR测序。基因组学报,1997,7(5):541-550。
- 98.
Rozen S, Skaletsky H: Primer3在WWW上为一般用户和生物学家程序员服务。方法分子生物学杂志,2000,32:365- 366。
- 99.
Kaderali L, Deshpande A, Nolan JP, White PS:多重基因分型的引物设计。核酸学报,2003,31(6):1796-1802。10.1093 / nar / gkg267。
- One hundred.
Smit A, Hubley R, Green P: RepeatMasker Open-3.0。1996年,(http://www.repeatmasker.org]
- 101.
Majoros WH, Pertea M, Salzberg SL: TigrScan和GlimmerHMM:两个开源的从头计算真核基因查找器。生物信息学,2004,20(16):2878-2879。10.1093 /生物信息学/ bth315。
- 102.
Overbeek R, Fonstein M, D’souza M, Pusch GD, Maltsev N:利用基因簇推断功能耦合。中国生物医学工程学报,2004,22(6):369 - 369。10.1073 / pnas.96.6.2896。
- 103.
叶军,方磊,郑华,张勇,陈俊,张铮,王俊,李松,李锐,Bolund L,王俊:WEGO:一种GO注释绘制web工具。核酸决议2006,34 (Web服务器问题):W293-7。10.1093 / nar / gkl031。
- 104.
王晓燕,王晓燕,王晓燕。氧化应激过程中的信号转导。中国生物医学工程学报,2002,32(3):366 - 366。10.1093 / jexbot / 53.372.1227。
- 105.
JGI Trichocarpa v1.1。[http://genome.jgi-psf.org/Poptr1_1/]
- 106.
拟南芥信息资源。[http://www.arabidopsis.org]
- 107.
TIGR水稻基因组注释[http://www.tigr.org/tdb/e2k1/osa1/]
确认
我们感谢理研基因组科学中心测序技术团队的所有技术人员和理研植物科学中心植物功能基因组课题组的Akiko Enju的技术支持。我们也感谢CIAT的Martin Fregene和Hernán Ceballos提供的使用基因型的信息和材料。我们也非常感谢哥伦比亚国立大学生物技术研究所和委内瑞拉国家科学计算中心将他们的机器借给我们进行计算分析。本研究得到日本理化研究所植物科学中心、日本文部科学省科学研究资助基金(青年科学家(B)18700106)和CIAT核心基金的支持。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
TS进行生物信息学分析并起草手稿,GP和FR负责计算分析的构思和执行、结果的解释并共同撰写手稿,AS进行非生物胁迫处理并准备用于文库建设的RNA, AI提供生物信息学协助处理序列数据,AT和YS对cDNA克隆进行测序,MS和KS参与了分析的协调并帮助起草了手稿,JT和MI构思了研究并对手稿进行了批判性的修改。所有作者阅读并批准了最终稿件。
樱井哲哉,Germán普拉塔,福斯托Rodríguez-Zapata对这项工作也有同样的贡献。
权利和权限
开放获取本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(https://creativecommons.org/licenses/by/2.0),它允许在任何媒体上不受限制地使用、分发和复制,只要原著被恰当地引用。
关于这篇文章
引用这篇文章
樱井,田,普拉塔,Rodríguez-Zapata, F。et al。对2万个木薯全长cDNA克隆的测序分析显示,在与应激反应相关的基因家族中存在谱系特异性扩增。BMC植物杂志7,66(2007)。https://doi.org/10.1186/1471-2229-7-66
收到了:
接受:
发表:
关键字
- 基因本体论
- 选择拼接变体
- 木薯种植
- KEGG Orthology
- 最近的基因复制