摘要
背景
植物编码大量富含亮氨酸的重复受体样激酶。豆科植物编码数个与根结形成过程相关的LRR-RLK,其配体未知。为了识别这些受体的配体,我们使用了轮廓隐马尔可夫模型和位置特异性迭代BLAST的组合,使我们能够从公开的序列数据库中检测CLV3/ESR (CLE)蛋白家族的新成员。
结果
我们从不同的植物物种中鉴定出了114个CLE蛋白家族的新成员,以及5个包含多个CLE结构域的蛋白质序列。我们能够将CLE结构域蛋白聚类为13个不同的组,基于它们在主要CLE基序中的成对相似性。此外,我们发现了与我们的序列簇一致的次要基序。基于CLE基序的分组与CLE信号肽的已知生物学功能相关,类似于基于系统发育分析和异位过表达研究的分组。我们在豆科植物中测试了两种预测的CLE信号肽的生物学功能Medicago truncatula.这些肽抑制根根尖和侧根分生组织的活性,与我们基于其他CLE信号肽聚集在同一组的功能预测一致。
结论
我们的分析提供了大量新型潜在CLE信号肽的鉴定和分类。我们发现的额外基序可能会导致未来发现处理肽酶的识别位点,以及对受体结合特异性的预测。
背景
高等植物基因组中含有大量受体样激酶(RLK) [1,2].富含亮氨酸的重复RLK (LRR-RLK)是植物RLK中最大的亚家族,并介导蛋白质-蛋白质相互作用[3.,4].一组LRR-RLK的潜在受体配体是CLV3/ESR (CLE)信号肽,由Cock和McCormick首先描述[5],并于最近审阅[6- - - - - -8].总共有65个CLE成员来自各种单子叶和双子叶植物。已知存在于非植物物种中的单一CLE信号肽是由植物寄生线虫编码的异皮线虫属甘氨酸[9],有人提出,寄生虫获得了植物信号来改变寄主的行为[10,11].除了这一例外,已有研究表明CLE信号肽是植物特异性的[5,12].
公鸡与麦考密克[5]报道了一个clv3样基因家族,他们使用位置特定迭代BLAST (PSI-BLAST)迭代搜索确定了这个家族。作者从基因组和表达序列标签(EST)数据库中检测到42个序列,得到39个相关蛋白序列。该蛋白家族被称为CLV3/ esr相关(CLE),其特征是在c端有一个跨越12个残基的保守结构域,在n端有一个疏水信号肽。该蛋白的可变区(相对于CLE基序的n端)被认为没有特定的功能,因为它可以被其他基因的核苷酸取代[13].
第一个确定的CLE成员被称为ESR基因,因为它们被证明在胚胎周围区域(ESR)特异性表达玉米胚乳(14],其mRNA在ESR区的mRNA中占主要比例[15].CLE家族中描述得最好的成员是CLAVATA 3 (CLV3),它被认为是CLV1/CLV2受体复合物的配体。该受体复合物是限制茎尖分生组织(SAM)中干细胞数量所必需的,并形成了植物LRR-RLK信号的范式。多种分析表明CLV3是CLV1/CLV2受体异源二聚体所感知的配体[16- - - - - -19].然而,配体与受体的直接结合尚未被证实。CLV3在拟南芥阻碍了第一片叶子出现后SAM器官的启动。在clv3功能丧失的突变体,干细胞积聚在芽和花分生组织的中心,形成额外的器官或未分化的组织[17].
CLE成员的功能特征表明,它们参与植物的多种发育机制,如SAM、根尖分生组织(RAM)或维管细胞分化[10,13,20.- - - - - -26].然而,单个CLE信号肽的确切功能仍然是未知的。分析在答:芥在异位表达18种不同的CLE信号肽后,显示出相似的表型,并根据CLE成员的过表达表型将其分为4组。这种分类与保守域的序列特征相关[12].然而,在活的有机体内多肽的功能可能导致更具体的表型,因为它们在植物中的表达模式可能是局部的,而与活性多肽的异位应用不相关。
在豆科植物中,根瘤的形成是由一般称为根瘤菌的固氮细菌触发的[27].根瘤菌在豆科植物根内诱导新的分生组织。这个过程涉及到至少两个已知的lrr - rlk。在感染的早期阶段,一种名为NORK(结瘤受体激酶)的LRR-RLK,紫花苜蓿) [28, DMI2(不会感染),m . truncatula) [28, SYMRK(共生受体激酶Lotus对虾) [29],或SYM19(共生),Pisum一) [30.]感知一种迄今为止未知的配体,然后激活导致结瘤的信号级联。结节分生组织的增殖受植物的限制。这个过程,所谓的结节的自动调节,是在clv1样的LRR-RLK NARK(结节自动调节受体激酶,大豆) [31, HAR1 (Hypernodulation Aberrant Root 1,l .对虾) [32], SUNN(多数性结节,m . truncatula) [33],以及SYM29 (SYMbiosis 29,p .一) [34].在所有这四种豆科植物中,这种蛋白质的功能丧失突变导致结节分生组织不受控制的增殖。结瘤的调节也与植物的氮供应有关。如果土壤中有足够的氮,根瘤就会受到抑制[35].有趣的是,CLE信号肽可能参与植物对氮的反应,作为改变CLE2的表达答:芥在氮剥夺下观察[36].
一些作者认为CLE信号肽可以作为豆科植物结瘤受体激酶自动调节的配体[21,37].因此,可以想象CLE结构域蛋白可能在结节分生组织的起始和/或维持中起着至关重要的作用。到目前为止,只有7个来自豆科植物的CLE成员被确认。他们的角色仍然未知。为了从功能上描述CLE结构域蛋白并检测其参与根结节分生组织形成的抑制,有必要从这个家族中鉴定更多的成员。由于已知的来自豆科植物的CLE结构域蛋白数量有限,我们系统地调查了大量植物序列数据库中的CLE序列。我们在已知和新的非豆科植物CLE序列的背景下分析了豆科植物的序列,以确定是否可以识别出任何豆科植物特有的CLE结构域蛋白。
由于它们的大小,许多小蛋白质,包括潜在的信号肽,通常不会被自动注释程序检测到。因此,需要更精细的生物信息学方法来识别潜在的植物信号肽,无论是在蛋白质水平还是在核苷酸水平[5,38- - - - - -42].关于CLE家族,大多数成员是使用PSI-BLAST识别的,并依赖于与已知CLE成员的序列相似性[5,43].使用motif检测和搜索工具MEME/MAST对基因序列进行搜索h·甘氨酸[9,44].一些研究也使用BLAST来鉴定有限数量的CLE信号肽[12,26,45].
结果
我们用于鉴定CLE结构域蛋白的方法类似于CLE家族第一篇报告中使用的方法[5].然而,我们的方法依赖于使用PSI-BLAST和HMMer的新组合来识别潜在的CLE家族成员[43,46,47].PSI-BLAST被用来代替BLAST来检测潜在的序列同源物,因为PSI-BLAST结合了BLAST的速度和更高的灵敏度,通过考虑以前的搜索结果,并为后续的搜索调整评分矩阵。这使得评分矩阵能够更好地反映被分析的蛋白质家族,并允许检测到简单的成对比较无法检测到的序列家族的远程成员。HMM基于多个序列比对,生成序列族的剖面隐马尔可夫模型(HMM)。考虑到使用了高质量的序列对齐,这可以提供更好的序列族表示,并允许识别更多距离较远的家族成员。缺点是对大型序列数据库的HMMer搜索相当耗时。为了充分利用这两种方法的优点,我们使用了HMMaccel [48],一个结合PSI-BLAST和HMMer的程序。PSI-BLAST用于第一步,将一个大的序列数据库减少到一个较小的序列集,显示与感兴趣的蛋白质家族最小数量的序列相似性。在这种情况下,简化后的数据库由那些生成高得分序列对的序列组成,e值最高为10,000。然后可以使用较慢但更精确的HMM方法搜索这个较小的序列集。由于对CLE结构域蛋白质认识的增加,我们可以使用先前确定的额外序列特征,n端信号序列和c端保守结构域,作为进一步的标准来分配包含motif的蛋白质序列到家族中。
CLE信号肽的鉴定
使用来自各种植物物种的序列资源生成了一个自定义数据库。我们结合了基因组计划的序列数据m . truncatula,栽培稻,杨树trichocarpa而且答:芥,以及来自TIGR基因指数的ESTs [49],以及TIGR植物转录本组合[50]来自豆科植物和各种植物。这产生了一个数据库,其中包含来自各种测序项目的数据,并包含了最大限度的序列信息,尽管是以冗余的形式。包括苔藓Physcomitrella金属盘还有绿藻衣藻reinhardtii,以推断CLE蛋白家族的进化起源。使用HMMaccel进行迭代搜索的主要输入包括项目开始时已知的45个CLE序列的多个序列比对。使用ClustalW [51,并手动细化。这种比对作为HMMaccel的输入,HMMaccel使用PSI-BLAST和HMMer组合迭代搜索上述植物数据库,以检测更多的同源物。迭代1产生169个候选,迭代2产生227个候选,迭代3产生811个候选。对第三次迭代的检查表明,许多序列被检测到,虽然显示出与已知CLE序列的一些序列相似性,但不能充分代表c端保守的12个氨基酸。这表明我们的HMM已经达到了仅基于该家族中的序列守恒就可以可靠检测到的极限。为了减少数据集中的假阳性数量,我们分析了CLANS中811个候选CLE序列[52,53].所有没有连接到包含已知CLE序列的p值阈值为1E-04的中心簇的序列都从数据集中移除。之所以选择这个阈值,是因为所有被排除的序列都不包含CLE基序的12个氨基酸,而将阈值增加到1E-05则排除了数据集中的有效代表。在将序列集重新集中到我们认为是真正积极的命中后,剩余的499个序列被用于HMMaccel搜索的第四次迭代。这种搜索的目的是检测所有真实的CLE代表,而不是生成一组只包含真实命中而没有假阳性的序列。最后的迭代还用于恢复我们可能在宗族过滤过程中无意中丢弃的任何真正序列,或者由于HMM的退化而在第三次迭代中错过的任何真正序列。迭代4返回659个序列。事实上,在迭代4中发现的序列比在迭代3中少,尽管在迭代4中使用了更多的序列来进行搜索,这表明迭代3返回了许多真阳性序列以及一些假阳性序列,随后的宗族过滤成功地排除了大多数假阳性命中,并将搜索重点重新集中在真正的CLE序列上。第四次迭代结束了我们对假定CLE信号肽序列的搜索。
作为对照,我们确定了在迭代4中是否正确识别了20个最近识别的家族成员,这些成员没有包含在初始的45个序列集中,但已经出现在数据库中。所有20个序列都可以在最终的数据集中找到。从最初的45个序列开始,我们还测试了之前迭代中的任何序列是否在后续迭代中丢失,这将表明数据集的漂移。这适用于前三次迭代,但不适用于第四次迭代,因为序列已经手动从数据集中删除了。我们无法检测到数据集的明显漂移,因为在连续迭代之间最多丢失了三个序列。45个CLE成员,作为在迭代1中执行的搜索的初始种子,在接下来的迭代中被一致地恢复。我们唯一无法检测到的已知CLE序列是CLE8 (答:芥) [5,53]及CLE15 (o .漂白亚麻纤维卷) [5],因为这些在我们的数据库中并不存在。我们所能鉴定出的CLE8最接近的同源体是在保守的CLE结构域中具有高序列同一性的其他已知CLE成员。我们无法检测到任何在整个蛋白质长度上显示出与CLE8高度相似的序列。对于CLE15,我们能够识别出两个相近的同源物(o .漂白亚麻纤维卷TIGR EST条目TC281944_+1和NP936837_+1)。多序列比对结果显示,两个EST序列均不含CLE基序,但与其余序列中的CLE15相同。这表明EST的组装发生了变化。因此,我们得出结论,最初鉴定为CLE8和CLE15的序列已从本研究使用的数据库版本中删除。所有其他已知的CLE序列在此迭代搜索过程中被识别。
接下来,我们从最终HMMaccel搜索中获得的659个序列中剔除假阳性候选序列。在主要的蛋白质序列方面,没有固定的CLE成员,在所有已知的家族成员中,CLE基序的序列都有轻微的变化。因此,Strabala等人描述的串联重复序列[12]和基于主要序列的严格标准,以可靠地将候选人分配到CLE家族。CLE家族的主要特征是保守的c端区域的氨基酸序列。第二个标准是考虑蛋白质长度(60-120个氨基酸)和motif在序列中的相对位置。通常,基序位于c端,在全长序列的最后三分之一。第三个标准是考虑等电点,因为绝大多数已知的CLE序列都有一个基本的pI。在659个序列中,我们剔除了303个不符合上述标准的序列,留下356个潜在的CLE结构域蛋白。
许多序列使用不同的标识符多次表示,因为我们的自定义数据库是通过将多个序列数据库汇集在一起生成的。为了减少我们最终集的冗余,我们使用CD-Hit[按序列相似性对356个序列进行分组。54].CD-Hit集群计算的阈值范围为70-100%。为了使数据集非冗余,序列根据其70%身份阈值进行排序,并将分配到同一聚类的所有序列分组。包含小于99%的序列的组使用MultAlin手动验证[55].这一过程最终得到179个非冗余序列,其中包括65个已知的CLE结构域蛋白和114个新的CLE结构域蛋白1,附加文件1).
这个家族的命名有混乱。我们试图保持CLE家族成员的命名客观和一致。类似于Cock和McCormick的方法[5]每个成员都被连续编号,并以“CLE”作为前缀,与物种起源无关。我们还将CLE编号分配给尚未纳入系统命名法的成员(例如CLV3、TDIF、Hg蜡烛,BnCLE19)。
我们独立地搜索了一个包含共生细菌序列的自定义数据库(Bradyrhizobium日本血吸虫,墨氏中根hizobium meliloti,中根hizobium loti)、致病菌(根农杆菌,根农杆菌)、共生真菌(球球互节,双色漆)和一系列致病真菌(例如:黑穗病,灰霉病,大豆疫霉菌),以检测是否有非植物的CLE序列。在这些物种中均未检测到CLE候选序列。
最后,我们使用从第三次迭代过滤结果中得到的HMM搜索NCBI (nr)中的非冗余蛋白数据库。该搜索返回的CLE序列仅来自植物,除先前鉴定的CLE成员来自植物外h·甘氨酸[10].此外,搜索nr数据库并没有揭示任何我们以前没有使用我们的自定义植物数据库识别的序列。
具有多个有序排列的CLE域的CLE成员
CLE家族的一般特征是成员包含一个单一的保守域。令人惊讶的是,我们从3种植物中发现了5个包含多个CLE基序的序列(CLE75, CLE76, CLE68, CLE30, CLE31)2).编码CLE75和CLE76的序列在o .漂白亚麻纤维卷基因组,起源于5号染色体上的两个不同的基因组位点。CLE68有一个条目m . truncatula基因组。CLE30和CLE31t . aestivum由Cock和McCormick鉴定,起源于t . aestivumEST数据库[5].在所有五种情况下,一个蛋白质序列中保守的CLE基序彼此非常相似,并在CLE基序中携带相同的变异。CLE68从m . truncatula是一个例外,因为第三个结构域不同于蛋白质序列中的前两个结构域。在所有情况下,CLE结构域都是有规律排列的,第一个结构域出现在50-75个氨基酸之后,这是标准CLE成员的典型特征,而进一步的结构域出现在大约30个氨基酸的间隔(图1).同样,CLE68来自m . truncatula在第一个域和第二个域之间形成一个较大间隙的异常。位于连续CLE基序之间的序列彼此相似,表明基因串联复制的融合或基因组或EST条目的错误注释。
序列分析
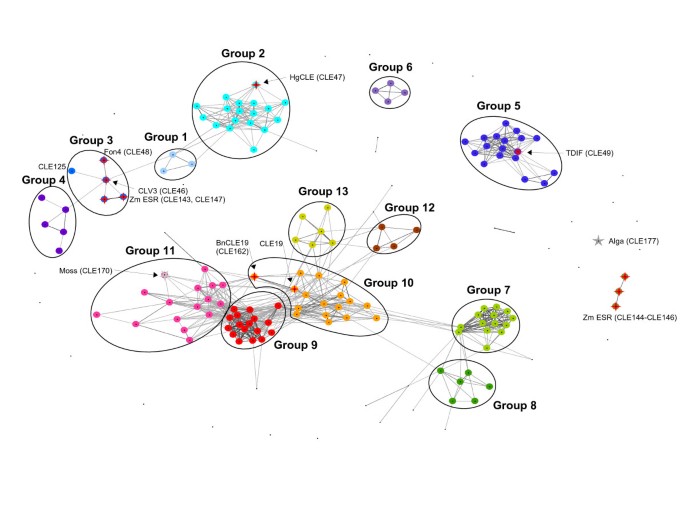
CLE成员的大部分蛋白质序列似乎是不相关的;家族内的序列相似性基本上局限于c端12-18个氨基酸的保守区域。我们进行了详细的序列分析,首先寻找CLE基序内(12-18个氨基酸)的相似性,其次测试CLE基序外是否存在序列相似性。我们使用宗族对家族的保守域进行聚类分析[52,53].宗族是一个Java工具,可视化和分析蛋白质序列的相似性基于成对相似性(BLAST),非常适合于大序列集的分析。宗族不允许得出系统发育的结论,它只允许分析蛋白质序列的相似性。对这些序列进行聚类后,将136个序列分为13个组(图2)2).我们从图中排除了携带多个CLE域的5个CLE成员,因为这些使可视化复杂化。38个序列,包括已知和新鉴定的CLE成员,不能可靠地分配到13个组中的任何一个。
CLE域序列相似性分析.基于序列相似性的174个序列在CLE域的宗族聚类。序列用圆点表示,不同的组用椭圆突出显示。同一组的序列被赋予相同的颜色。连接点的线对应的BLASTP值优于1.2E-7。CLE成员的特点HgCLE (CLE47), TDIF (CLE49)和Zm评选ESR (CLE143-CLE147),以及已知的直系物CLV3/FON4和CLE19/BnCLE19 (CLE162)被红星突出。从中找到的单个CLE成员Physcomitrella金属盘(苔藓,CLE170),聚在第11组,用灰色星星突出显示。一个假定的CLE序列衣藻reinhardtii(藻类,CLE177)也标有灰色星,但不聚集在任何基团附近。基于聚类分析建立的分组类似于之前的分类[8,12,24]。第2组含有CLE1-CLE7,之前在肽实验中被证明对RAM生长或血管细胞分化没有影响,这导致本人类矮化生长仅在发芽后21天,异位过表达。CLE9-CLE13在第7组。这些CLE成员对RAM有影响,但对肽束和血管细胞分化无影响本人在过表达研究中,可在发芽后14天和21天观察到样矮生长。CLE家族成员CLE41、CLE42、CLE44对RAM无影响,但对血管细胞分化有影响灌木样过表达表型位于第5组。
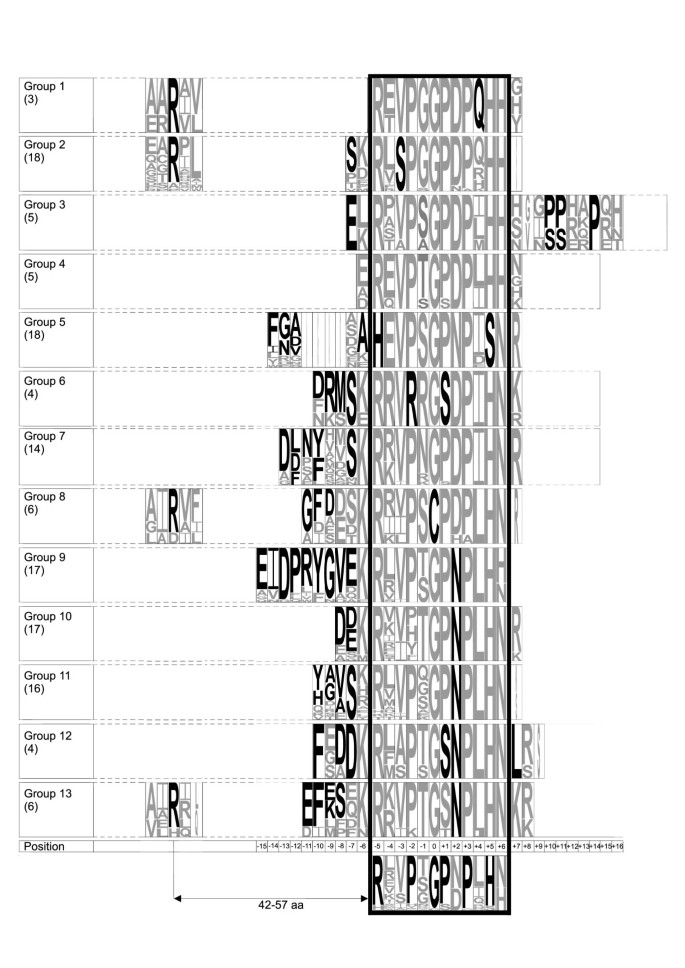
聚类完成后,我们分析了整个蛋白质序列的序列相似性,以CLE基序分组的序列在基序外是否有相似的序列区域。我们构建了序列标识,以可视化12个氨基酸CLE基序内外的保守残基。在CLE基序中,整个家族的序列共识揭示了有六个几乎不变的残基(图3.).其中包括R, P, G, P, P和H,其中前两个P残基被发现是羟基化的[24].由于G的中心保守位置,我们将G分配到位置0,并对相对于G的其他氨基酸的位置进行编号。对于N和D以及N和h,有两个位置的出现概率相同。这些保守残基可能为假定的配体的受体相互作用提供了框架。这些保守残基中一些罕见的变异发生在0位(仅在第8组中是C位而不是G位)和+1位(在第6组和第12组中是S位而不是主要的羟基化P位)。定义域中的其他位置是可变的,例如位置-4和-1。我们能够识别出组特异性残基,即根据宗族将其分离为不同的组的残基,如图所示3..
对邻近CLE基序的蛋白质序列区域的分析表明,CLE基序以外的某些区域不是随机的,而是保守的(图3.).有趣的是,这些保守的母题遵循了基于宗族的分组。这表明,在CLE蛋白编码区的其他部分,主要CLE基序的序列与进一步的序列相似区域(可能是次要序列基序)相关。
经鉴定的CLE信号肽的生物学功能Medicago truncatula
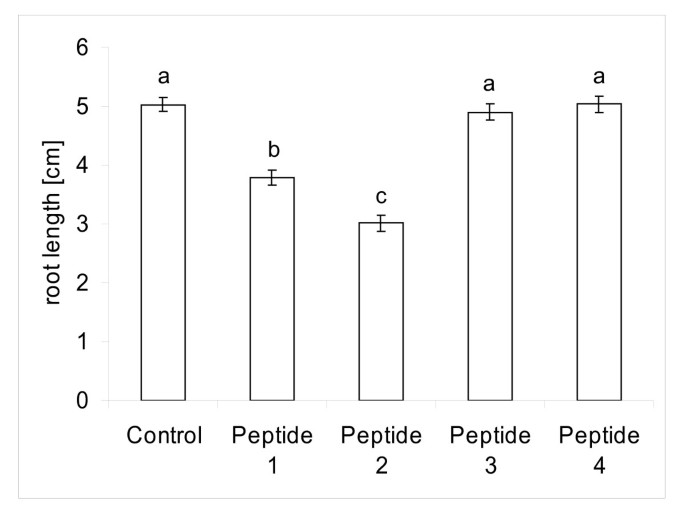
以确定其生物活性在网上为了鉴定CLE成员,我们在肽分析中测试了与保守的CLE结构域相对应的合成肽。由于大多数CLE序列被预测对RAM的生长有影响,我们使用了基于分组的预期对RAM有影响的肽(图2).我们合成了两个肽,肽1 (SKRKVPSCPDPLHN)和肽2 (SKRRVPNGPDPIHN)。选择了14个氨基酸的长度,因为这些肽在以前的报道中显示出活性[22].肽1只在CLE的一个成员CLE67中发现m . truncatula,聚集在第9组(图2,附加文件2).由于保守域的冗余,肽2在来自不同植物的8个CLE序列CLE34、CLE36、CLE64、CLE78、CLE80、CLE117、CLE118和CLE163中均有存在。由于用于聚类的CLE结构域包含多达18个氨基酸,因此后面的一些CLE序列被分为不同的组,包括第7组(CLE34, CLE78, CLE80, CLE117, CLE118, CLE163),第8组(CLE64)和一个未分组但位于第7和8组附近的序列(CLE36)。作为对照,我们分别使用氨基酸组成、分子量和等电点与肽1和肽2相同的两种随机序列的肽(肽3和肽4)。
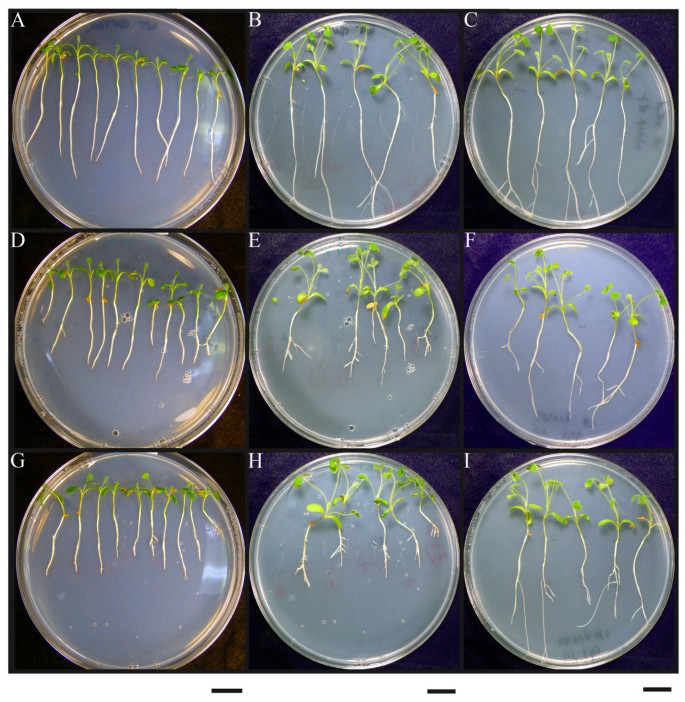
m . truncatula以该肽作为生长介质添加剂培养幼苗[22].在处理6天后,与不使用多肽和随机多肽的对照植物相比,所有经多肽1和多肽2处理的幼苗的根生长均明显终止(图2)4,图5).处理6天后,与无肽组和随机肽组相比,经肽1和肽2处理的植物根系生长显著降低(p < 0.0001,单向方差分析)。20天后,用肽1或2处理的幼苗几乎没有进一步的根生长。我们注意到在两种多肽处理中都增加了侧根的形成。与RAM类似,侧根新形成的分生组织在侧根出现后不久就终止了生长。我们通过将一半植株转移到不含多肽的新鲜培养皿中来测试多肽处理的可逆性。RAM在两周内恢复。在某些情况下,主根终止了它的生长,侧根反而拉长了。我们还观察到,从含肽的培养基中释放后,主根可以恢复生长。在这个实验中,尽管芽与琼脂没有直接接触,但琼脂中肽的存在对芽的生长没有明显的影响。
CLE多肽的生物活性Medicago truncatula.在平板实验中证实了合成的CLE肽的生物活性,这些肽对应于预测的CLE信号肽的保守域的14个氨基酸m . truncatula.以浓度为10 μM的多肽作为生长介质添加剂。上面一行(A-C)显示植物在没有肽的情况下生长,中间一行(D-F)显示肽1 (SKRKVPSCPDPLHN)的存在,下面一行(G-I)显示肽2 (SKRRVPNGPDPIHN)的存在。处理后第6天的植物生长情况(左列;A, D, G),治疗后第20天(中柱;B, E, H)和恢复的第20天,其中,幼苗处理6天,然后将其转移到无肽板上,剩余14天(右柱;C, F, I).每列底部的栏表示2厘米。
讨论
CLE成员的识别
这项研究的目的是在植物中,特别是从豆类中鉴定CLE信号肽家族的新成员。将候选人分配到家庭的总体标准是严格和有限的,这使我们能够消除许多误报。从自定义数据库中检索到的冗余序列的数量远远大于最终非冗余集中的序列的数量。这表明,在许多情况下,来自EST和基因组数据库的多个冗余序列条目被组合在一个CLE编号下。从EST和基因组数据中重复恢复了相同的CLE序列,这使得这些蛋白质极有可能实际上在植物中表达。然而,到目前为止,从具有测序基因组的植物物种中鉴定出的CLE信号肽的数量还不能被认为是完整的。这是因为我们的分析是基于从基因组中预测的蛋白质,这些蛋白质由自动开放阅读框检测进行注释。这种自动检测经常无法检测到像CLE家族成员这样的小蛋白质[38- - - - - -42].因此,我们期望在表达蛋白预测方面的改进,可能会识别更多的CLE信号肽。我们能够识别的序列集包括65个已知的和114个新的CLE序列,使识别出的潜在CLE信号肽的数量达到179个。该数据集包括28个新的豆科CLE序列。CLE家族的序列相似性分析不是基于系统进化树,而是基于成对序列比较。正如Floyd和Bowman所指出的,在CLE家族中,14个氨基酸的限制性序列守恒阻碍了系统发育分析[56].
到目前为止,我们找到了一位CLE家族的代表Physcomitrella金属盘使用EST数据库,尽管一旦这种生物的基因组公开,可能会发现更多。来自绿藻衣藻reinhardtii其中,我们使用基因组以及EST数据库和TIGR转录集,我们只能识别一个CLE序列,该序列不与任何组聚类(图2).这种假定的CLE信号肽的生物学功能衣藻将需要在未来的研究中确定。这将是有趣的发现如果CLE序列衣藻与CLE信号肽在高等植物中的作用不同,CLE信号肽显示细胞分化和分生组织活性,以及CLE信号肽是否是植物发育所需的必要遗传设备的一部分[56].
一个新的发现是CLE蛋白序列携带多个CLE基序的鉴定。我们能够检测到携带2到6个基序的多结构域CLE蛋白o .漂白亚麻纤维卷,t . aestivum而且m . truncatula,但在任何其他植物物种中都没有。这些序列来自不同的数据库和测序项目。为了降低基因组错误组装或tc条目导致含有多个cle -结构域的蛋白质出现的可能性,我们检查了蛋白质的基因组位置和EST覆盖。使用TIGRo .漂白亚麻纤维卷在基因组浏览器中,我们确定CLE75和CLE76中的基序源于单个外显子。检查来自CLE30和CLE32的tc条目t . aestivum我们在CLE30中找到了25个单独的序列reads (EST’s),在CLE31中找到了5个序列reads,涵盖了至少两个CLE motif。这为这两种多cle蛋白的来源提供了证据t . aestivum以预测的方式转录,不太可能是tc组装的产物。我们假设完整的蛋白序列在加工后释放出几个活性信号肽,这可能提供了放大效应。
CLE基序的聚类及新的次生基序的鉴定
利用宗族技术对CLE序列进行聚类分析,结果表明这些序列可分为13个组。我们观察到的基于序列相似性的分组对应于异位CLE过表达表型的分类答:芥由Strabala制作et al。[12].此外,它等价于系统发育分组,并与对根尖分生组织和组织分化影响的观察结果一致[8,24].我们在图中观察到已知功能直方线的紧密空间排列(例如FON4和CLV3,见图3组)2) [26].建立的分组允许进一步的直系同源的种间鉴定。我们假设CLE125与CLV3和FON4在同一组,是CLV3在的功能同源p . trichocarpa中的CLE143和/或CLE147z梅斯,分别。这种分组还可以缩小线虫所来自的CLE候选基因的数量h·甘氨酸可能获得了CLE信号肽。的h·甘氨酸CLE序列与第2组紧密聚集。如果发生了横向基因转移,这表明线虫已经从第2组获得了一个CLE成员,并可能使我们进一步了解第2组CLE信号肽的功能以及CLE信号肽的功能h·甘氨酸CLE信号肽。总的来说,结果表明,导致CLE成员不同群体的序列相似性与在过量肽供应(异位表达或肽添加)情况下观察到的效果之间存在联系[8,12,21- - - - - -24].然而,由于异位表达可能导致表型不反映在活的有机体内CLE信号肽的作用,未来的研究可以集中于描述每个信号肽的确切生物学功能。
在一项肽实验中,我们证实了两种在网上经鉴定的信号肽具有一定的生物学功能m . truncatula.这两种多肽都抑制了根根尖分生组织和侧根分生组织的活性,导致根生长减慢。这些肽的序列在CLE的7、8或9组成员中发现1).在这些组中聚集的其他CLE肽也被发现对根尖分生组织有负面影响,例如在研究中的CLE25和CLE26答:芥而且Zinnia线虫[8,24].此外,第9组CLE序列的成员,包括CLE9-CLE13也对RAM有影响[8,24].
剩下的一个主要问题是,为什么植物会编码如此大量的lrr - rlk,以及它们的功能和配体是什么。CLE信号肽可与CLV1/CLV2受体相关的LRR-RLKs结合,但目前对CLE肽配体与其受体的特异性尚不清楚。通过序列分析确定的组特异性和不变残基以及保守残基的变化可以确定给定信号肽靶向的受体亚群的选择性特异性。此外,我们的聚类分析显示,CLE基序以外的区域与基于主CLE基序的宗族生成的分组在序列相似性上相关。研究表明,加工过程发生在家族成员中,这意味着一种或一种酶的复合物识别部分蛋白质序列并将其裂解。在保守结构域c端添加单个精氨酸残基会导致肽活性降低[8,24].这表明,正确的切割和对保守结构域的特定识别对于信号肽的最大活性是必要的。其过程和具体机制尚不清楚。此外,目前尚不清楚是否所有肽都以与CLV3和TDIF相同的方式加工和修饰,CLV3和TDIF被发现具有12种氨基酸肽的活性。我们推测该基序的延伸可能参与了信号肽前体的特异性识别和加工。
结论
我们从多种植物中鉴定出114个新的CLE结构域蛋白,其中包括28个来自豆类的新序列,这些新序列可能是控制结瘤的LRR-RLKs的潜在配体。我们还发现了一些具有多个CLE结构域的CLE蛋白,这可能代表了一种肽信号放大机制。这些序列的聚类显示了13个不同的组,这些组被发现在CLE域外具有保守的次要基序。两种预测信号肽的生物活性得到了证实在活的有机体内.CLE信号肽可能具有改变植物发育的潜在生物技术应用,例如美国专利No. 7179963使用CLE信号肽功能z梅斯.虽然我们不能在我们的研究中测试所有识别的信号肽的生物活性,但我们希望本研究中提出的CLE结构域蛋白将允许其他研究人员测试它们在各种植物物种中的功能,以及作为LRR-RLKs的潜在配体。
方法
生物序列资源
将多个序列资源进行组合,形成一个自定义的冗余蛋白质数据库。表达式序列标签(EST)数据库答:芥(12.1版),芸苔属植物显著(版本1),c . reinhardtii(5)发布,g·马克斯(10)发布,Lotus对虾(3)发布,Lycopersicum esculentum(10.1版),m . truncatula(8)发布,烟草(版本2),o .漂白亚麻纤维卷(16)发布,茄属植物tuberosum(发行版10)和z梅斯(第16版)从TIGR基因指数下载(现在可在Dana-Farber癌症研究所基因指数项目中获得)[49].TIGR转录集(TA)从答:芥,芸苔属植物显著,c . reinhardtii,p .金属盘,g·马克斯,甘氨酸大豆,Lotus corniculatus,Lupinus白色,Lycopersicum esculentum,m .漂白亚麻纤维卷,m . truncatula,烟草,o .漂白亚麻纤维卷,菜豆coccineus,菜豆,Pisum一,茄属植物tuberosum,z梅斯已加入此集(均为2005年8月15日第1版)[50].从植物基因组中预测的蛋白质答:芥(NCBI Genbank发行5,2006年5月3日)[57),c . reinhardtii(JGI,第3版)[58),m . truncatula(基因组测序计划2006年7月17日公布)[59),o .漂白亚麻纤维卷(2005年12月30日第四版)[60),而p . trichocarpa(JGI,第1版)[61也包括在内。
序列名被截断为唯一标识符。每个序列的数据库来源信息被添加到唯一标识符(即OS-TA, OSEST, OSGEN for)中o .漂白亚麻纤维卷TA, EST或基因组序列)。在所有6个阅读帧(通用码)中将核苷酸序列翻译成蛋白质序列,并将帧信息附加到序列标识符(例如。“_ + 2”)。从基因组数据获得的翻译核苷酸序列和修改的蛋白质序列被合并到一个文件中,并使用Formatdb进行格式化(选项:-p T和-o T) [43].结果数据库包含3,631,558个序列。为了确定CLE序列是否是植物特异性的,我们在非冗余蛋白数据库(NCBI nr, version 15 June 2006.)的基础上进行了单独的搜索。
查询序列
一组45个已知CLE序列(CLE1 - CLE17, CLE19 - CLE44, HgCLE和CLV3;从Genbank和TIGR检索)合并在一个fasta文件中,使用CLUSTALW 1.83进行对齐,并手动改进[51].在多序列比对的基础上,利用HMM 2.3.2建立剖面隐马尔可夫模型(HMM) [47].原始fasta文件被重新对齐到HMM (HMMalign),并使用对齐编辑器AlnEdit进行验证[62]以检查此重新排列步骤的一致性。比对结果显示,在c端有一个高度保守的12-18个氨基酸区域,与HMM一致(对应CLV3的“HEELRTVPSGPDPLHH”)。因此,我们决定将“保守域”扩展到先前定义的12种氨基酸之外[5,24,25].这个对齐(迭代0)作为迭代1到HMMaccel的输入。此外,提取在比对中匹配的12-18个氨基酸片段,并用于构建仅由保守区域组成的HMM。HMMaccel可供下载[48].
植物数据库的Motif检索
每次迭代都从一个普通的fasta文件开始(前一次迭代的输出)。fasta文件中的所有序列都与保守域的HMM对齐。结果对齐使用AlnEdit进行验证,并转换为对齐的fasta格式(用于输入HMMaccel)。检索所有HMMaccel命中的全长序列,并将其重新对齐到保守域的HMM。结果的对齐被手动检查(AlnEdit)并转换为对齐的fasta格式(为下一次迭代输入HMMaccel)。
在整个迭代1和2中,PSI-BLAST的设置是截断e值10,000(参数-e),包含序列的e值阈值为0.005(参数-h),显示高分序列对的数量为250(参数-b),显示命中的数量为500(参数-v)。参数-b和-v在迭代3和迭代4中被更改为- b1和-v 1000。HMMaccel中hmm的参数导致返回的e值高达10,hmm需要使用5000个样本进行校准。
与之前的迭代相比,我们观察到在迭代3之后添加到数据集中的大量假阳性序列。如果不删除这些序列,数据集在迭代4中就会变得不准确。为了避免有偏差的序列删除和序列集的可重复优化,使用了序列聚类分析(宗族)[52,53].提取迭代3的811个hits的保守区域,并在CLANS中进行分析。共有312条假阳性序列从序列集中被丢弃。剩下的499个序列被提交到最终迭代中。从这499个序列派生的HMM作为附加文件可用3..迭代4后,数据集由659个蛋白质序列组成。在迭代3中返回的大量假阳性命中表明该方法已经达到了它可以解决的极限。在去除假阳性后,执行第四次迭代以减少假阴性的数量。迭代搜索的目的是在数据库中找到所有CLE肽,因此假阴性比假阳性更值得关注。
序列标识符中包含物种和数据库起源。序列的完整注释信息随后从原始fasta文件中检索。用EMBOSS 4.0.0中的PROT STATS计算等电点和分子量。包(63].从fasta文件中提取CLE结构域的蛋白长度、位置和序列。
序列分析
在CLANS中分析全长蛋白序列和保守结构域[52].在数据集中添加45个原始查询序列,以检查它们在宗族中的位置和组分配。使用全长序列,我们发现了几个具有多个结构域的序列,我们通过它们在宗族中的行为注意到了这一点。在保守区域的聚类分析中观察到CLE肽的分组。使用Kalign [64].使用WebLogo 3.0b14将主要CLE motif的对齐、扩展和附加motif可视化,以表示组的所有序列[65].
多肽合成
肽1 (SKRKVPSCPDPLHN)和肽3(随机肽1,DHKSKPPVLRPNSC)以及肽2 (SKRRVPNGPDPIHN)和肽4(随机肽2,PVHPKGNRNDISPR)由GL生物化学(中国上海)以75%纯度合成。多肽在c端带有一个游离羧基。肽1和肽2是根据CLE基序设计的,肽3和肽4不像CLE基序,因为其序列是肽1和肽2氨基酸序列的随机版本。在ExPASy上使用RandSeq工具生成随机序列[66].将多肽稀释至终浓度为10 μmol/l [22]在无菌无氮Fåhraeus培养基中[67].
肽测定
野生型m . truncatula简历。Jemalong A17种子在细砂纸上洗净,用80%技术级乙醇(5 min)、6.25%次氯酸钠溶液(5 min)和现配制的200 mg/l Augmentin杀菌®阿莫西林/克拉维酸钾;GlaxoSmithKline, Brentford UK)(5小时),用无菌milliq冲洗5次®水(Millipore, Billerica USA)。种子在没有多肽的Fåhraeus琼脂板上,在4°C (12 h)和28°C (24 h)的黑暗条件下发芽[68].在将幼苗转移到含有肽或不含肽的新鲜盘子(对照)之前,用无菌的磷酸盐缓冲盐水简单清洗幼苗。钢板用Parafilm密封®(结构探针公司,西切斯特美国)的底部和生长在一个直立的位置。用黑色纸盒盖住盘子底部的2/3,以减少根部暴露在阳光下。在恒定25°C和100 μE光照下,延长白天(白天16 h /夜晚8 h)条件下生长[68].从转移当天开始,每24小时测量一次根系生长,持续6天(t = 0d)。为了测试多肽处理的可逆性(t = 6d),将一半的植株(5株)从含有多肽的培养皿转移到新鲜培养皿(不含多肽),并生长两周(t = 20d)。在6 d和20 d的时间点拍照。使用GenStat进行统计分析®9.2 (VSN国际有限公司,Hemel Hempstead UK)。
参考文献
- 1.
Johnson KL, Ingram GC:发送正确的信号:调节受体激酶活性。植物学报,2005,8(6):648-656。10.1016 / j.pbi.2005.09.007。
- 2.
植物发育中的受体激酶信号。细胞与发育生物学杂志,2002,18:163-192。10.1146 / annurev.cellbio.18.012502.083431。
- 3.
Kobe B, Deisenhofer J:富含亮氨酸重复序列与蛋白质配体相互作用的结构基础。自然科学,1995,26(1):1 - 6。10.1038 / 374183 a0。
- 4.
松林Y,杨萍,坂上Y:高等植物肽信号及其受体。植物科学进展,2001,6(12):573-577。10.1016 / s1360 - 1385(01) 02148 - 3。
- 5.
公鸡JM,麦考密克S:一个与CLAVATA3同源的大型基因家族。植物学报,2001,26(3):344 - 344。10.1104 / pp.126.3.939。
- 6.
Fiers M, Ku KL, Liu CM: CLE肽配体及其在分生组织建立中的作用。植物学报,2007,10(1):39-43。10.1016 / j.pbi.2006.11.003。
- 7.
Germain H, Chevalier E, Matton DP:植物生物活性肽:一类扩展的信号分子。植物学报,2006,26(1):1-19。10.1139 / b05 - 162。
- 8.
孙晓明,田志明,田志明,李志明,李志明,李志明。CLV3/ esr相关(CLE)肽在植物细胞间信号分子中的作用。化学通报,2006,6(6):303-310。10.1002 / tcr.20091。
- 9.
Olsen AN, Skriver K:配体拟态?与CLAVATA3相似的植物寄生线虫多肽。植物科学进展,2003,8(2):55-57。10.1016 / s1360 - 1385(03) 00003 - 7。
- 10.
王晓华,Mitchum MG,高bl, Li CY, Diab H, Baum TJ, Hussey RS, Davis EL:一种功能类似于拟南芥CLAVATA3/ESR (CLE)的植物寄生线虫寄生基因。分子植物病理学,2005,6(2):187-191。10.1111 / j.1364-3703.2005.00270.x。
- 11.
Davis EL, Mitchum MG:线虫。豆科植物的复杂寄生虫。植物生理学杂志,2005,37(4):344 - 344。10.1104 / pp.104.054973。
- 12.
Strabala TJ, O'Donnell PJ, Smit AM, Ampomah-Dwamena C, Martin EJ, Netzler N, Nieuwenhuizen NJ, Quinn BD, Foote HCC, Hudson KR:许多CLAVATA3/ESR基因的功能获得表型,包括四个新的家族成员,与保守的CLAVATA3/ESR结构域的串联变异相关。植物生理学杂志,2006,40(4):331- 344。10.1104 / pp.105.075515。
- 13.
倪娟,陈志明,陈志明:CLAVATA3 CLE结构域的功能守恒、充分性和蛋白水解过程的证据。植物科学进展,2006,26(2):339 - 339。10.1104 / pp.105.072678。
- 14.
李文杰,李文杰,李文杰,等。玉米胚乳特异性基因ZmEsr在玉米胚乳中表达。植物学报,1997,12(1):235-246。10.1046 / j.1365 - 313 x.1997.12010235.x。
- 15.
Bonello JF, Opsahl-Ferstad HG, Perez P, Dumas C, Rogowsky PM:玉米胚乳同一区域的Esr基因表达水平不同。基因工程学报,2000,46(1-2):379 - 379。10.1016 / s0378 - 1119(00) 00088 - 3。
- 16.
Clark SE, Running MP, Meyerowitz EM: Clavata3是芽和花分生组织发育的特定调节因子,影响与Clavata1相同的过程。发展。1995,121(7):2057-2067。
- 17.
杨晓明,王晓明,王晓明,王晓明,王晓明。拟南芥干细胞命运与CLV3活性的相关性研究。科学通报,2000,29(4):344 - 344。10.1126 / science.289.5479.617。
- 18.
Rojo E, Sharma VK, Kovaleva V, Raikhel NV, Fletcher JC: CLV3定位于细胞外空间,激活拟南芥CLAVATA干细胞信号通路。植物细胞学报,2002,14(5):969-977。10.1105 / tpc.002196。
- 19.
Clark SE:芽分生组织的细胞信号。中国生物医学工程学报,2001,26(4):379 - 379。10.1038 / 35067079。
- 20.
Casamitjana-Martinez E, Hofhuis HF, Xu J, Liu CM, Heidstra R, Scheres B:拟南芥根分生组织维持与CLE19根系特异性过表达及sol1/2抑制子的关系现代生物学,2003,13(16):1435-1441。10.1016 / s0960 - 9822(03) 00533 - 5。
- 21.
Hobe M, Muller R, Grunewald M, Brand U, Simon R:在拟南芥中,一种功能相当于干细胞限制信号CLV3的蛋白质CLE40的缺失增强了根系波动。中国生物医学工程学报,2003,26(3):379 - 379。10.1007 / s00427 - 003 - 0329 - 5。
- 22.
Fiers M, Golemiec E, Xu J, van der Geest L, Heidstra R, Stiekema W, Liu CM: 14个氨基酸CLV3, CLE19和CLE40多肽通过clavata2依赖通路触发拟南芥根分生组织的消耗。植物细胞学报,2005,17(9):2542-2553。10.1105 / tpc.105.034009。
- 23.
Fiers M, Hause G, Boutilier K, Casamitjana-Martinez E, Weijers D, Offringa R, van der Geest L, Campagne MV, Liu CM:拟南芥CLV3/ esr样基因CLE19的错表达导致根分生组织消耗。基因学报,2004,327(1):37-49。10.1016 / j.gene.2003.11.014。
- 24.
Ito Y, Nakanomyo I, Motose H, Iwamoto K, Sawa S, Dohmae N, Fukuda H: Dodeca-CLE多肽作为植物干细胞分化的抑制因子。科学通报,2006,26(3):344 - 344。10.1126 / science.1128436。
- 25.
Kondo T, Sawa S, Kinoshita A, Mizuno S, Kakimoto T, Fukuda H, Sakagami Y:通过原位MALDI-TOF MS分析鉴定CLV3编码的植物肽。科学通报,2006,29(3):344 - 344。10.1126 / science.1128439。
- 26.
褚华文,钱强,梁文强,尹春春,谭海霞,姚鑫,袁忠,杨娟,黄红,罗东,马红,张德宝:水稻花器官编号4基因编码拟南芥CLAVATA3,调控水稻顶端分生组织大小。植物生理学杂志,2006,42(3):357 - 357。10.1104 / pp.106.086736。
- 27.
豆科植物结瘤的发育生物学。中国植物科学,2002,22(2):357 - 357。10.1111 / j.1469-8137.1992.tb04227.x。
- 28.
Endre G, Kereszt A, Kevei Z, Mihacea S, Kalo P, Kiss GB:调节共生结节发育的受体激酶基因。自然科学学报,2002,26(3):344 - 344。10.1038 / nature00842。
- 29.
Capoen W, Goormachtig S, De Rycke R, Schroeyers K, Holsters M: SrSymRK,共生体形成所必需的植物受体。中国科学:自然科学,2005,29(2):369- 374。10.1073 / pnas.0504250102。
- 30.
Stracke S, Kistner C, Yoshida S, Mulder L, Sato S, Kaneko T, Tabata S, Sandal N, Stougaard J, Szczyglowski K, Parniske M:细菌和真菌共生所需的植物受体样激酶。自然科学学报,2002,26(3):344 - 344。10.1038 / nature00841。
- 31.
Searle IR, Men AE, Laniya TS, Buzas DM, Iturbe-Ormaetxe I, Carroll BJ, Gresshoff PM: clavata1样受体激酶指导下的结瘤长距离信号。科学通报,2003,29(3):357 - 357。10.1126 / science.1077937。
- 32.
Nishimura R, Hayashi M, Wu GJ, Kouchi H, Imaizumi-Anraku H, Murakami Y, Kawasaki S, Akao S, Ohmori M, Nagasawa M, Harada K, Kawaguchi M: HAR1介导共生器官发育的系统性调节。自然科学学报,2002,26(4):426-429。10.1038 / nature01231。
- 33.
Schnabel E, Journet EP, de Carvalho-Niebel F, Duc G, Frugoli J:截叶苜蓿SUNN基因编码clv1样富亮氨酸重复受体激酶,调节根瘤数量和根长。植物分子生物学,2005,58(6):809-822。10.1007 / s11103 - 005 - 8102 - y。
- 34.
Krusell L, Madsen LH, Sato S, Aubert G, Genua A, Szczyglowski K, Duc G, Kaneko T, Tabata S, de Bruijn F, Pajuelo E, Sandal N, Stougaard J:芽对根发育和结瘤的控制是由受体激酶介导的。自然科学学报,2002,26(4):344 - 344。10.1038 / nature01207。
- 35.
硝酸盐对豆科植物结瘤形成和N-2固定的抑制作用。植物科学进展,1988,7(1):1-23。
- 36.
Scheible WR, Morcuende R, Czechowski T, Fritz C, Osuna D, palacioso - rojas N, Schindelasch D, Thimm O, Udvardi MK, Stitt M:拟南芥初级和次生代谢的全基因组重编程,蛋白质合成,细胞生长过程,以及氮响应的调控基础设施。植物科学进展,2004,26(1):344 - 344。10.1104 / pp.104.047019。
- 37.
植物中富含亮氨酸重复受体激酶:结构、功能和信号转导途径。中国生物医学工程学报,2004,27(3):344 - 344。
- 38.
Schopfer CR, Nasrallah ME, Nasrallah JB:芸苔属植物自我不亲和的男性决定因素。科学通报,1999,29(4):344 - 344。10.1126 / science.286.5445.1697。
- 39.
Vanoosthuyse V, Miege C, Dumas C, Cock JM:拟南芥两个大的基因家族与芸苔属基因超家族同源,该基因编码花粉外套蛋白和自交不亲和反应的雄性成分。植物分子生物学,2001,46(1):17-34。10.1023 /: 1010664704926。
- 40.
Ride JP, Davies EM, Franklin FCH, Marshall DF:拟南芥基因组序列分析揭示了植物中一个庞大的新基因家族。植物分子生物学,1999,39(5):927-932。10.1023 /: 1006178511787。
- 41.
Lease KA, Walker JC:拟南芥无注释分泌肽数据库,植物肽组学资源。植物生理学。2006, 42(3): 344 - 344。10.1104 / pp.106.086041。
- 42.
Silverstein KAT, Graham MA, Paape TD, VandenBosch KA:拟南芥300多个防御素样基因的基因组组织。植物生理学。2005, 38(2): 1 -6。10.1104 / pp.105.060079。
- 43.
Altschul SF, Madden TL, Schaffer AA, Zhang JH, Zhang Z, Miller W, Lipman DJ: gapping BLAST和ppi -BLAST:新一代蛋白质数据库搜索程序。核酸研究,1997,25(17):3389-3402。10.1093 / nar / 25.17.3389。
- 44.
Bailey TL, Baker ME, Elkan CP:在蛋白质序列中发现基序的人工智能方法:应用于类固醇脱氢酶。生物化学与分子生物学杂志,1997,32(1):29-44。10.1016 / s0960 - 0760(97) 00013 - 7。
- 45.
Sharma VK, Ramirez J, Fletcher JC:拟南芥CLV3-like (CLE)基因在不同组织中表达,并编码分泌蛋白。植物分子生物学学报,2003,51(3):415-425。10.1023 /: 1022038932376。
- 46.
Eddy SR:隐马尔科夫模型和基因组序列分析。学报学报,1998,12 (8):A1327-a1327。
- 47.
Eddy SR:剖面隐藏马尔可夫模型。生物信息学,1998,14(9):755-763。10.1093 /生物信息学/ 14.9.755。
- 48.
HMMaccel在RSBS生物信息学服务器。[http://bioinfoserver.rsbs.anu.edu.au/programs/hmmaccel/]
- 49.
Lee Y, Tsai J, Sunkara S, Karamycheva S, Pertea G, Sultana R, Antonescu V, Chan A,张F, Quackenbush J: TIGR基因指数:EST和已知基因的聚类和组装及与真核基因组的整合。中国生物医学工程学报,2005,33:D71-D74。10.1093 / nar / gki064。
- 50.
Childs KL, Hamilton JP,朱伟,Ly E,张F, Wu H, Rabinowicz PD, Town CD, Buell CR, Chan AP: TIGR植物转录本装配数据库。中国生物工程学报,2007,35:D846-D851。10.1093 / nar / gkl785。
- 51.
Thompson JD, Higgins DG, Gibson TJ: Clustal-W -通过序列加权,位置特定间隙惩罚和权重矩阵选择提高渐进式多序列对齐的灵敏度。核酸研究,1994,22(22):4673-4680。10.1093 / nar / 22.22.4673。
- 52.
Frickey T, Lupas A: CLANS:一个基于成对相似性可视化蛋白质家族的Java应用程序。生物信息学,2004,20(18):3702-3704。10.1093 /生物信息学/ bth444。
- 53.
RSBS生物信息学服务器上的宗族:RSBS生物信息学服务器上的宗族。[http://bioinfoserver.rsbs.anu.edu.au/programs/clans/index.php]
- 54.
Li WZ, Godzik A: Cd-hit:用于聚类和比较大组蛋白质或核苷酸序列的快速程序。生物信息学,2006,22(13):1658-1659。10.1093 /生物信息学/ btl158。
- 55.
论文F:层次聚类的多序列对齐。核酸研究,1988,16(22):10881-10890。10.1093 / nar / 16.22.10881。
- 56.
孙文杰,张志刚,张志刚:陆生植物的祖先发育工具包。植物学报,2007,26(1):1-35。10.1086/509079。
- 57.
拟南芥基因组计划:分析开花植物拟南芥的基因组序列。自然科学学报,2000,27(2):344 - 344。10.1038 / 35048692。
- 58.
shager J, Hauser C, Chang CW, Harris EH, Davies J, McDermott J, Tamse R, Zhang ZD, Grossman AR:莱茵衣藻基因组计划。cDNA信息的生成和使用指南。植物生理学报,2003,31(2):344 - 344。10.1104 / pp.016899。
- 59.
Bell CJ, Dixon RA, Farmer AD, Flores R, Inman J, Gonzales RA, Harrison MJ, Paiva NL, Scott AD, Weller JW, May GD: The Medicago Genome Initiative:一个模型豆类数据库。核酸研究,2001,29(1):114-117。10.1093 / nar / 29.1.114。
- 60.
松本T,吴生理改变,Kanamori H, Katayose Y,藤泽米,只N,美津浓H,山本K,安东尼奥英航,巴巴T,坂田K, Nagamura Y,青木H, Arikawa K, K, T鱼毒木,Chiden Y, Fujitsuka N, Fukunaka R,岩漠M,原田C, Hayashi, Hijishita年代,本田M,细川,川Y, Idonuma,饭岛爱,Ikeda M, Ikeno M, Ito K, Ito年代,Ito T, Ito Y, Ito Y,逐渐繁盛,Kamiya K, Karasawa W, Kurita K,片瞳年代,Kikuta,小林,小林N, Machita K,前原诚司T, Masukawa M, Mizubayashi T,向井亚纪Y,长崎H,经营着Y, Naito年代,中岛美嘉M, Nakama Y, Nakamichi Y,中村M,目黑,根岸英一,太,太,Okamoto M,小野N, Saji年代,坂口米,酒井法子K,柴田M, Shimokawa T,歌曲司法院,Takazaki Y, Terasawa K, Tsugane M,教授K,建筑师年代,Waki K,山形H,山本M,山本年代,Yamane H, Yoshiki年代,俊井R,汤川K,钟HS,矢野米,佐佐木T,元QP,蜀OT,刘J,琼斯公里,Gansberger K,莫法特K,希尔J,贝拉J, Fadrosh D, Jin SH Johri年代,金米,Overton L,里尔登,Tsitrin T, Vuong H,韦弗B, Ciecko, Tallon L杰克逊J, Pai G, Van Aken年代,T之际,Reidmuller年代,Feldblyum T,萧J, Zismann V, Iobst年代,de Vazeille AR过活CR,应K,李Y, TT,黄YC,赵问,冯Q,张L,朱镕基JJ,翁QJ、μJ, Lu YQ DL粉丝,刘黄,关JP,张YJ, SL,刘XH,张Y,香港的女朋友,汉族B, Choisne N, Demange N, Orjeda G, Samain年代,Cattolico L, Pelletier E, Couloux, Segurens B, Wincker P D 'Hont, Scarpelli称C, Weissenbach J,Salanoubat M, Quetier F,于Y,兰博T, Kim人力资源库里J, Collura K,罗MZ,杨TJ, Ammiraju js,断F C,索德伦德翼RA,帕默勒,de la巴斯蒂德M,明镜周刊L, L Nascimento, Zutavern T O ' shaughnessy,堤,Dedhia N,普雷斯顿R, Balija V, McCombie WR, Chow泰,陈HH,钟MC,陈CS,肖摩根富林明,吴惠普,萧KJ,曹国伟欧美,楚可,程CH,小时,李PF,林SJ,林YC, Liou司法院,刘SM,杏,Raghuvanshi年代,莫汉蒂,Bharti AK白肢野牛,古普塔V, Kumar D,拉维V, Vij年代,Iwama H, Gojobori T, Itoh T, Niimura Y, Fujii Y, Habara T, Sakai H, Sato Y, Wilson G, Kumar K, McCouch S, Juretic N, Hoen D, Wright S, Bruskiewich R, Bureau T, Miyao A, Hirochika H, Nishikawa T, Kadowaki K, Sugiura M, IRGS项目:基于图谱的水稻基因组序列。自然科学学报,2005,37(2):344 - 344。10.1038 / nature03895。
- 61.
简颂年代,Tuskan GA DiFazio年代,Bohlmann J,眼镜,Hellsten U,普特南N,拉尔夫,Rombauts年代,Salamov,史肯J, Sterck L, Aerts, Bhalerao RR, Bhalerao RP, Blaudez D, Boerjan W,布朗,Brunner, Busov V,坎贝尔M,卡尔森J, Chalot M,查普曼J,陈GL,库珀D, Coutinho点,女裁缝师J,隐蔽的年代,体弱的Q, R,坎宁安戴维斯J, Degroeve年代,Dejardin, Depamphilis C,德J,短剑B, Dubchak我Duplessis年代,Ehlting J,埃利斯B, Gendler K,古德斯坦D, Gribskov M, Grimwood J,挖槽机,甘特L, Hamberger B,海因策B, Helariutta Y, Henrissat B,霍利甘D,霍尔特R,黄W, Islam-Faridi N,琼斯,Jones-Rhoades M,约根森R, Joshi C, Kangasjarvi J, Karlsson J,凯莱赫C,柯克帕特里克R, Kirst M,科勒,Kalluri U,此外F, Leebens-Mack J, Leple JC, Locascio P,卢Y,卢卡斯,马丁F, Montanini B, C那不勒斯,纳尔逊博士,纳尔逊C, Nieminen K,尼尔森O, Pereda V,彼得G,菲利普·R,彼拉多G, Poliakov, Razumovskaya J,理查森P,里纳尔蒂C, Ritland K,Rouze P, Ryaboy D, Schmutz J, Schrader J, Segerman B, Shin H, Siddiqui A, Sterky F, Terry A, Tsai CJ, Uberbacher E, Unneberg P, Vahala J, Wall K, Wessler S, Yang G, Yin T, Douglas C, Marra M, Sandberg G, de Peer YV, Rokhsar D:黑杨杨毛杨(Torr. trichocarpa)的基因组。&灰色)。科学通报,2006,29(3):344 - 344。10.1126 / science.1128691。
- 62.
在RSBS生物信息学服务器的AlnEdit:在RSBS生物信息学服务器的AlnEdit。[http://bioinfoserver.rsbs.anu.edu.au/programs/alnedit/]
- 63.
Rice P, Longden I, Bleasby A: EMBOSS:欧洲分子生物学开放软件套件。遗传科学进展,2000,16(6):276-277。10.1016 / s0168 - 9525(00) 02024 - 2。
- 64.
Lassmann T, Sonnhammer ELL: Kalign -一种精确而快速的多序列对齐算法。生物信息学杂志,2005,6:
- 65.
克鲁克斯GE, Hon G, Chandonia JM, Brenner SE: WebLogo:序列标志生成器。基因组学研究,2004,14(6):1188-1190。10.1101 / gr.849004。
- 66.
Gasteiger E, Gattiker A, Hoogland C, Ivanyi I, Appel RD, Bairoch A: ExPASy:用于深入蛋白质知识和分析的蛋白质组学服务器。核酸研究,2003,31(13):3784-3788。10.1093 / nar / gkg563。
- 67.
Fahraeus G:用简单的玻璃载片技术研究了三叶草根毛结节细菌的感染。中华微生物学杂志,1997,16(2):374-381。
- 68.
Wasson AP, Pellerone FI, Mathesius U:阻断截叶苜蓿黄酮通路抑制根瘤菌对生长素运输的调控。植物细胞学报,2006,18(7):1617-1629。10.1105 / tpc.105.038232。
确认
作者感谢Helge Küster访问血管球EST数据库。此外,我们要感谢来自昆士兰大学和澳大利亚研究理事会综合豆类研究卓越中心(CILR)的澳大利亚国立大学节点的同事,他们进行了重要而富有成效的讨论。KO非常感谢来自CILR的财政支持。这项研究是由CILR (CE0348212)和澳大利亚研究委员会的研究奖学金(DP 0557692)资助的。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
KO进行了生物信息学分析和肽分析。NG设计了数据库并参与了编程。GFW和PMG参与了实验的总体设计和协调。UM进行了统计分析和部分肽分析,TF构思了motif搜索策略,并参与了编程和整体实验设计。所有作者都阅读并批准了最终的手稿。
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
Oelkers, K., Goffard, N., Weiller, G.F.et al。CLE信号肽家族的生物信息学分析。BMC植物生物学8,1(2008)。https://doi.org/10.1186/1471-2229-8-1
收到了:
接受:
发表:
关键字
- 隐藏马尔可夫模型
- 茎尖分生组织
- 根顶端分生组织
- 配置文件隐藏马尔可夫模型
- TIGR基因指数