摘要
背景
直到最近,只有少数低通量和中通量的方法被用于葡萄(葡萄藤)单核苷酸多态性(SNP)的发现和基因分型。葡萄l .)。然而,随着黑皮诺高杂合基因组序列的完成,已经有可能鉴定出数百万个电子snp (eSNPs),从而为高通量基因分型方法提供了有价值的来源。
结果
在此,我们报告了SNPlex™基因分型系统在葡萄中的首次应用,旨在锚定真核生物基因组。这种方法结合了强大的SNP检测与自动分析读出和数据分析。从黑皮诺组装基因组的非重复序列中获得813个候选eSNPs,并在90个西拉-黑皮诺杂交后代中进行了检测。获得并绘制了563个新的snp标记。采用多位移扩增(multiple displacement amplification, MDA)方法制备SNPlex基因DNA时,效率从69%提高到80%。
结论
与其他用于研究少数基因型中数千个SNP或大约1000个基因型中的几个SNP的SNP基因分型方法不同,SNPlex基因分型系统代表了一个很好的折衷方案,可以同时研究100个或更多样本中的数百个SNP。因此,SNPlex检测结合全基因组扩增(WGA)是未来在设备齐全的实验室中应用的一个很好的解决方案。
背景
近年来,单核苷酸多态性(SNPs)已成为动物和植物中最流行的遗传标记系统。在几个基因组测序项目中发现了它们的惊人数量[1],再加上最近的技术进步,使得SNP标记在标记辅助育种、EST作图以及遗传和物理图谱的整合中具有高通量应用的吸引力。
目前已有几种SNP鉴定方法,如有或无预筛选的PCR扩增子重测序、表达序列标签(EST)的电子SNP (eSNP)发现和霰弹枪基因组文库等。在后一种情况下,如果存在足够的冗余,可以通过计算筛选序列的多态性,以区分真正的多态性和测序错误[2]。
与第一代分子标记,如RFLPs(限制性片段长度多态性)和rapd(随机扩增多态性dna)不同,snp可以通过非凝胶的高通量检测来检测,节省了时间和金钱[3.]。基于不同的等位基因鉴别方法和检测平台,已经开发了几种SNP检测技术。等位基因特异性杂交、引物延伸、寡核苷酸连接和侵入性切割是四种主要的等位基因识别反应,可以与荧光、发光和质量测量等几种检测方法相结合(见[4- - - - - -6查看最近的评论)。最近,人们利用BeadArray技术在动物和植物中进行了大规模SNP表征的尝试(Illumina [7])和SNPlex™基因分型系统(应用生物系统公司,ABI [8])。选择合适的基因分型方法取决于许多因素,包括成本、多路复用和吞吐量的潜力、设备和分析开发的难度。
直到最近,只有少数低通量和中通量的方法被用于葡萄(葡萄l .) [9- - - - - -12]。黑皮诺高杂合基因组的测序[j]。13],克隆了ENTAV 115,使鉴定数百万个eSNPs成为高通量基因分型方法的潜在来源成为可能。在此,我们报告了SNPlex基因分型系统的成功应用,该系统为未来的应用研究计划提供了563个新的基于snp的葡萄基因组标记。
结果
基因组DNA (gDNA)的SNPlex及其数据分析
在949个候选eSNPs中,813个通过了SNPlex分析设计管道的设计规则,并在90°F中进行了测试1西拉与黑皮诺(entav115)杂交的两个亲本基因型后代。使用基于规则的方法,734个esnp通过了质量值,平均5个不合格SNPs,每个SNPset的通过率为98%,其余79个被丢弃,不再进行进一步分析(表5)1).在734例esnp中,171例为假阳性。其余563个esnp中(参见附加文件)1:表S1为SNP序列列表,提交给国家生物技术信息中心SNP数据库[1]), 509例遵循1:1或1:2:1基于卡方检验的孟德尔分离比,54例出现意外分离比。在后一类中,有46例亲本一方杂合,观察到3个聚类(而不是预期的2个,分离比为1:1);8例亲本双方杂合,观察到4个聚类(而不是预期的3个,分离比为1:2:1)。
全基因组扩增DNA (WGA-DNA) SNPlex及其数据分析
在同一90人的WGA-DNA上检测了3个48 plex snp集(w0607103605_0001、w0610104437_0005和w0611104858_0001)中的144个eSNPs。在这一组中,15个未通过gDNA分析质量值的eSNPs在WGA-DNA测试中被恢复,而3个通过gDNA质量值的eSNPs在WGA-DNA测试中失败(表1)2).13个eSNPs未通过gDNA和WGA-DNA测试。两种分析的基因分型数据完全一致。
重测序分析
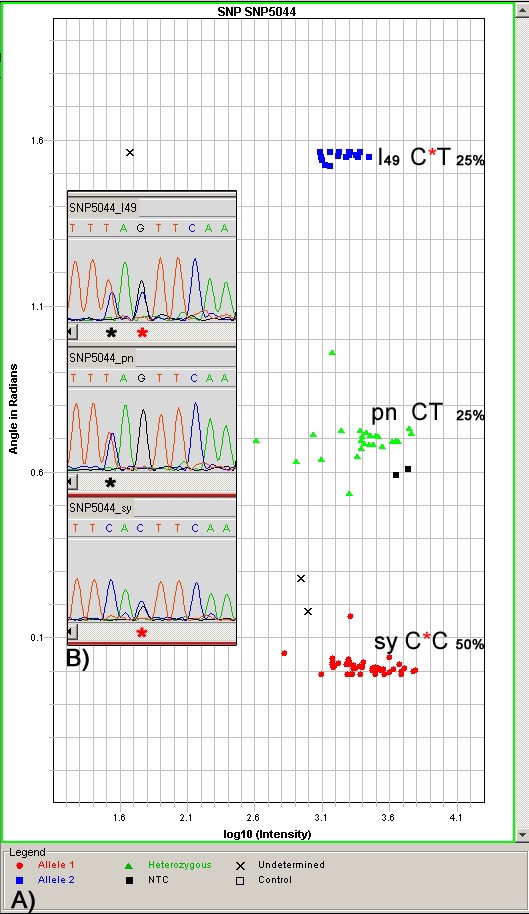
为了验证SNPlex分析获得的数据,我们对西拉、黑皮诺和6个后代中含有SNP4165、SNP4057、SNP4045、SNP0102、SNP0054和SNP5044的6个区域进行了重测序。其中4个SNP4165、SNP4057、SNP4045和SNP0102表现出孟德尔分离,相应区域的重测序证实了SNPlex分析得到的数据。对于SNP5044和SNP0054,出现了一个额外的纯合簇,在Syrah和属于额外纯合簇的后代中,在距离目标SNP 10 bp以内发现了一个意想不到的SNP(图5)1).
讨论
在这项工作中,我们报告了SNPlex基因分型系统在高等植物中的首次应用,该系统允许开发锚定葡萄藤基因组的标记。迄今为止,一些基于SSCP和微序列分析的低通量和中通量方法已被用于葡萄SNP基因分型[9,10,12]。
在葡萄基因组6.5×霰弹枪测序覆盖范围的eSNP发现的基础上,也开发了SNP标记[13]。效率为69%,平均调用率为98%,超过了最近研究中所选ESTs(38.3%)和bac端序列(35%)重测序所达到的水平[11]。虽然效率大大提高,但我们观察到在其他SNPlex基因分型研究中已经发现了10%的系统失败试验(D. Sondervan)等A.卡勒等。和S. Bevan等., SNPlex用户会议2007)和21%的假多态性。这种出乎意料的高假多态性率可能是由于SNPlex探针的设计基于组合体组装的初步阶段,其中每个单倍型在某些区域的覆盖率不够。
在WGA-DNA先导研究中,从gDNA和WGA-DNA分析中获得的基因分型数据完全一致,证实了先前基于BeadArray的SNP基因分型研究[14,15]和affmetrix技术[16]。此外,系统检测失败的数量大大减少,从10%降至2%,平均效率从69%提高到80%。这些结果是预期的,因为MDA方法[17]提供大量样品间浓度均匀的纯DNA [18],满足成功进行SNPlex检测的两个基本要求。WGA-DNA上6个SNP区域的重测序证实了SNPlex基因分型数据,并证明不存在扩增偏倚,如先前报道[16,17]。
对SNP0054和SNP5044区域进行重测序,发现在Syrah基因型中,SNP0054和SNP5044区域在与目标eSNP相邻的10 bp内出现了意想不到的额外多态性。探针退火过程中一个等位基因的优先连接可以解释人工纯合子簇的发生。
近年来,大量的SNP基因分型技术被开发出来。在为育种目的确定最合适的SNP测定时,应考虑不同方面,如准确性、可重复性和吞吐量水平。此外,还应考虑灵活性、时间和成本效益,在这方面,使用3730xl DNA分析仪(ABI)进行SNPlex分析的周转时间约为每个样品30分钟。因此,理论上每天可以产生221184个基因型(48次运行/24小时× 96根毛细血管× 48层反应)。
结论
与其他用于研究少数基因型中数千个SNP(如BeadArray和Affymetrix技术)或数千个基因型中少数SNP(如TaqMan试验)的SNP基因分型方法不同,SNPlex基因分型系统代表了一个很好的折衷方案,可以同时研究100个或更多样品中的数百个SNP。因此,在设备齐全的实验室中,使用SNPlex检测加上WGA-DNA是进行中大规模基因分型研究的一个很好的解决方案。
方法
植物材料和基因组DNA制备
90f基因组DNA1从50 ~ 100 mg幼叶中分离出西拉×黑皮诺后代和两个亲本基因型。冷冻干燥后,使用MM 300混合式磨粉机(德国哈恩的Retsch公司)研磨叶片材料,并根据制造商的协议使用dnasy 96 Plant Mini Kit (Qiagen, Valencia, California, USA)进行DNA提取。
编剧
MDA扩增10 ng的gDNA [17,19使用GenomiPhi V2 DNA扩增试剂盒(GE Healthcare, Little Chalfont, Buckinghamshire, United Kingdom),按照制造商的协议。琼脂糖凝胶电泳检测丙二醛反应的成功与否和阴性对照样品中产物的缺失。
SNP识别
黑皮诺的6.5×霰弹枪序列是发现eSNP的起点。Sanger测序从43个文库中产生了大约620万个reads,其中插入物大小不同,并组装成contigs [13]。在黑皮诺的全基因组散弹枪组装过程中,发现了大约200万个snp。其中,949个SNPs分布在19条葡萄染色体上,这些SNPs是从非重复的contigs中选择出来的。
试验设计
使用感兴趣的snp的等位基因特异性探针和优化的多路分析是由自动化多步骤流水线设计的[20.]。这些步骤包括:(1)进入含有目标snp的序列;(2)检查格式错误,如目标SNP附近的非目标多态性或序列基序与检测不相容;(3)提交通过分析设计格式检查的snp。ABI探针的设计防止了自互补和二聚化,并优化了退火效率。此外,还确定了单核苷酸多态性的最佳组合,以产生每个多重反应的最高收率。
SNPlex试验及数据分析
SNPlex在破碎的dna上进行,终浓度为45 ~ 225 ng,终体积为12.5 μl。分析了17个SNPset(14个48-plex, 3个47-plex);其中,SNPset (w0607103605_0001;w0610104437_0005和w0611104858_0001)也按照制造商的方案在片段化的GenomiPhi扩增的gDNA (WGA-DNA)上进行检测。将杂交周期中PCR产物的用量从1.5 μl改为3 μl。
样品在3730xl DNA分析仪(ABI)上运行,数据分析使用Gene Mapper v. 4.0软件(ABI)。基因型分析基于SNPlex_Rules_3730方法,遵循出厂默认规则。
重测序分析
PCR引物采用Primer3软件设计[21]根据以下标准:1)扩增片段的预期大小在200 - 600bp之间;2)引物大小在18 ~ 25个碱基之间;3)底漆熔化温度(Tm)在59 ~ 61℃之间;4)引物对自互补和互补性的比对得分和全局比对得分为8 ~ 13。
随后,在西拉、黑皮诺和6个后代中扩增出包含SNP4165、SNP4057、SNP4045、SNP0102、SNP0054和SNP5044的6个区域49,我53,我56,我57,我58和我59),使用WGA-DNA中的模板。采用以下条件进行PCR反应:1 - 20 ng DNA模板,1× PCR缓冲液(Qiagen, Valencia, California, USA),每个dNTP 0.2 mM,每个引物0.4 μM, HotStarTaq DNA聚合酶(Qiagen, Valencia, California, USA) 1 U,水至终体积12.5 μl。DNA扩增使用15分钟的初始变性/激活步骤进行,随后在94°C下30秒,57°C下30秒,72°C下2分钟进行30个循环,最后在72°C下延长10分钟。PCR产物经1.5%琼脂糖凝胶电泳鉴定,溴化乙啶染色显示。为了在扩增反应中去除未结合的dNTPs和引物,将1 μl的外切酶磷酸酶(ExoSAP-IT, GE Healthcare, Little Chalfont, Buckinghamshire, United Kingdom)加入到1 μl的PCR产物中,最终体积为6 μl,在37℃下孵育45 min,然后在72℃下孵育15 min。
PCR产物采用BigDye Terminator Cycle sequencing Ready Reaction Kit v3.1 (ABI)双向测序:PCR纯化产物6 μl, 5×测序缓冲液,引物0.32 μl, BigDye Terminator 1 μl,终体积为10 μl。测序反应采用2分钟初始变性步骤,随后在96°C 10秒,50°C 5秒和60°C 4分钟下进行40个循环。在乙醇纯化之前,PCR产物在3730xl DNA分析仪(ABI)上进行毛细管电泳。DNA序列电泳图与Pregap4/Gap4软件包(Staden package, [22]),用于调查亲本等位基因的多态位点。
参考文献
- 1.
国家生物技术信息中心SNP数据库Build 128。[http://www.ncbi.nlm.nih.gov/projects/SNP/snp_summary.cgi]
- 2.
单核苷酸多态性在作物遗传中的应用。植物生物学进展。2002,5:94-100。10.1016 / s1369 - 5266(02) 00240 - 6。
- 3.
古普塔PK, Roy JK, Prasad M:单核苷酸多态性:分子标记技术和DNA多态性检测的新范式及其在植物中的应用。科学通报,2001,31(2):524-535。
- 4.
陈晓明,苏立文PF:单核苷酸多态性基因分型:生物化学,方案,成本和通量。生物医学进展[j] . 2003,19(3): 357 - 357。10.1038 / sj.tpj.6500167。
- 5.
全基因组SNP基因分型研究进展。遗传学报,2005,37(5):559 - 561。10.1038 / ng1558。
- 6.
王晓东,王晓东,王晓东,等。基因多态性分析方法的研究进展。司法科学,2005,25(4):381 - 394。10.1016 / j.forsciint.2004.10.020。
- 7.
沈荣,范建斌,Campbell D, Chang W, Chen J, Doucet D, Yeakley J, Bibikova M, Wickham GE, McBride C, Steemers F, Garcia F, Kermani BG, Gunderson K, Oliphant A:通用头阵列的高通量SNP基因分型:单核苷酸多态性(SNPs)的检测,解释和应用。基因诱变的分子机制与基础研究。2005,37(3):70-82。10.1016 / j.mrfmmm.2004.07.022。
- 8.
Tobler A, Short S, Andersen M, Paner T, Briggs J, Lambert S, Wu P, Wang Y, Spoonde A, Koehler R, Peyret N, Chen C, Broomer A, Ridzon D, Zhou H, Hoo B, hayashbara K, Leong L, Ma C, Rosenblum B, Day J, Ziegle J, De La Vega F, Rhodes M, Hennessy K, Wenz H: SNPlex基因分型系统的可扩展平台。中国生物医学工程学报,2009,32(6):998 - 996。
- 9.
欧文斯C: SNP检测和基因分型葡萄属。植物学报,2003,36(3):139-140。
- 10.
李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军。葡萄L.),由单核苷酸多态性揭示。分子育种,2005,14:385-395。10.1007 / s11032 - 005 - 0261 - 7。
- 11.
Troggio M, Malacarne G, Coppola G, Segala C, Cartwright D, Pindo M, Stefanini M, Mank R, Moroldo M, Morgante M, Grando M, Velasco R:基于单核苷酸多态性的葡萄遗传连锁图谱(英文)葡萄l .)锚定黑皮诺细菌人工染色体组。遗传学报,2007,32(2):559 - 559。10.1534 / genetics.106.067462。
- 12.
陈建军,陈建军,李建军,等。葡萄果实中SNP检测方法的比较研究。葡萄学报,2008,47(1):21-30。
- 13.
Velasco R, Zharkikh Troggio M,卡特赖特哒,Cestaro, Pruss D, Pindo M,菲茨杰拉德L, Vezzulli年代,里德J, Malacarne G, Iliev D,科波拉G,沃代尔B,米凯莱蒂D, Macalma T, Facci M,米切尔JT, Perazzolli M·G,与P Oyzerski R, Moretto M, Gutin N, Stefanini M,陈Y, Segala C, C达文波特,Dematte L,穆拉兹,Battilana J, K Stormo,科斯塔F,道Q, Si-Ammour,哈金斯T,马屁精,Perbost C, Taillon B,斯特拉,Solovyev V,福塞特J, Sterck L, Vandepoele K, Grando M,Toppo S, Moser C, Lanchbury J, Bogden R, Skolnick M, Sgaramella V, Bhatnagar S, Fontana P, Gutin A, Van de Peer Y, Salamini F, Viola R:葡萄杂合品种基因组高质量一致序列草图。PLoS ONE。2007, 12 (2): e1326-10.1371/journal.pone.0001326。
- 14.
Pask R, Rance H, Barratt B, Nutland S, Smyth D, Sebastian M, Twells R, Smith A, Lam A, smmink L, Walker N, Todd J:组合效用研究φ29全基因组扩增和高度多重单核苷酸多态性。中国生物医学工程学报,2004,4(4):559 - 564。
- 15.
李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,等。临床化学,2005,51:1520-1523。10.1373 / clinchem.2004.047076。
- 16.
林·派斯·J, M, Beroukhim R,李JC,赵X,里希特DJ,加布里埃尔,赫尔曼·P, H,佐佐木Altshuler D,李C, Meyerson M,卖家W:基因组覆盖率和序列的忠诚φ29基于聚合酶的多链置换全基因组扩增。中国生物医学工程学报,2004,32 (2):771 - 793 /nar/gnh069。
- 17.
胡志强,王志强,吴晓明,杜勇,孙志强,吴晓明,杜军,王志强,王志强,王志强,王志强。基于全基因组法的全基因组基因扩增研究。基因工程学报,2003,13(3):954-964。10.1101 / gr.816903。
- 18.
李建军,张建军,张建军,等。全基因组扩增技术:从临床标本和珍贵标本中提取丰富的DNA。生物工程学报,2003,21:531-535。10.1016 / j.tibtech.2003.09.010。
- 19.
Dean F, Hosono S, Fang L, Wu X, Faruqi A, brayward P,孙忠,宗强,杜勇,杜军,Driscoll M,宋伟,Kingsmore SF, Egholm M, Lasken R:基于多位移扩增技术的人类基因组综合扩增。《美国国家科学院学报》上。科学通报,2009,31(2):561 - 566。10.1073 / pnas.082089499。
- 20.
应用生物系统公司。[http://www.appliedbiosystems.com]
- 21.
Rozen S, Skaletsky H:为一般用户和生物学家程序员提供的入门级WWW。分子生物学,2002,32(2):365-386。
- 22.
李建军,张建军,张建军,等:中国农业大学学报,1998。分子生物学杂志,2000,32(2):115-30。
致谢
作者要感谢Mario Libera和Diego Micheletti提供的技术支持,Alessandro Cestaro提供的生物信息学支持,以及Applied Biosystems UK支持团队负责人Charles A. Reece的有益讨论。这项工作得到了Trento自治省资助的“葡萄藤基因组”项目的支持。
本文谨以此纪念马里奥·利贝拉。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
MP进行了SNPlex试验和数据分析,重测序并起草了手稿。SV对SNPlex分析和对结果的讨论做出了贡献。GC进行基因组DNA提取和MDA样品制备。DC参与了SNPlex试验的设计。AZ进行了基因组组装和SNP发现。RV对项目进行了概念化,并参与了对结果的讨论。MT监督了基于snp的标记开发和遗传作图,并对结果的讨论做出了贡献。所有作者都阅读并批准了最终的手稿。
电子补充资料
12870 _2007_220_moesm1_esm.xls
附加文件1:表S1。563个SNP标记分别为IASMA ID号、NCBI SNP (ss) ID、SNP等位基因、5’近序列等位基因和3’近序列等位基因。(xls182kb)
作者提交的原始图片文件
下面是作者提交的原始图片文件的链接。
权利和权限
本文由BioMed Central Ltd.授权发表。这是一篇基于知识共享署名许可(http://creativecommons.org/licenses/by/2.0),允许在任何媒体上不受限制地使用、分发和复制,前提是正确引用原创作品。
关于本文
引用本文
Pindo, M., Vezzulli, S., Coppola, G.。et al。使用SNPlex™基因分型系统在葡萄藤中进行SNP高通量筛选。BMC Plant Biol8,12(2008)。https://doi.org/10.1186/1471-2229-8-12
收到了:
接受:
发表:
关键字
- 单核苷酸多态性
- 单核苷酸多态性标记
- 单核苷酸多态性基因分型
- 全基因组扩增
- 多位移放大