摘要
背景
种植花生、落花生hypogaea是最近起源的同种异体四倍体,具有AABB基因组。与许多其他多倍体一样,通过两个野生物种的杂交和自发的染色体复制,似乎在物种起源上存在严重的遗传瓶颈。因此,花生的低遗传多样性和多倍体特性阻碍了花生基因组的研究。与栽培花生相比,大多是野生的落花生物种为二倍体,遗传多样性高。二倍体研究二倍体的研究落花生因此,无论是简化遗传和物理图谱的构建,还是野生等位基因的分离和表征,基因组都具有吸引力。花生最可能的野生祖先是答:duranensis而且答:ipaensis基因型分别为AA和BB。
结果
我们在细菌人工染色体(BAC)载体中构建并鉴定了两个大插入文库,分别对应一个二倍体祖先种。库(AA和BB)分别为c。7.4和c。5.3具有低细胞器污染和平均插入大小为110和100 kb的基因组当量。这两个文库都用于分离含有遗传标记的豆科植物锚定标记(单拷贝基因)和抗性基因类似物的克隆。
结论
这些二倍体BAC文库是分离具有生物胁迫抗性的野生等位基因、比较AA和BB基因组彼此间的同源区域以及与其他豆科物种的同源区域的重要工具,并将促进物理图谱的构建。
背景
栽培花生(落花生hypogaeaL)是世界上仅次于大豆的第二大粮食豆类作物,2003/04年度产量为3300万吨[1].花生在热带和亚热带的温暖地区都有种植,但在非洲、亚洲和美国尤为重要。1].它是一个同源四倍体(AABB),最近起源于两个野生物种的杂交和自发的染色体重复[2,3.].这一多倍体事件导致了严重的遗传瓶颈[2,3.这导致了一些重要性状缺乏可变性,等位基因组合的可用性有限,从而限制了生产力。此外,栽培花生的多态性水平非常低,阻碍了遗传和基因组的表征。相比之下,花生的二倍体野生亲缘具有较高的遗传多样性,并在一系列环境和生物胁迫的进化过程中被选择,构成了丰富的等位基因多样性来源[3.].野生等位基因可以通过“再合成”途径引入到栽培花生的基因库中,基本上是人为地重新创造了类似于导致作物物种起源的事件[4].最近通过传统分类学、细胞遗传学和分子研究对野生和栽培基因组关系的认识取得了进展,为重新合成野生物种提供了新的选择[5- - - - - -8].与此同时,用于监测野生基因渗入栽培背景的新的遗传和基因组工具(见下文)为利用分子育种将野生基因有效渗入花生作物打开了前景。
栽培花生的多态性水平非常低,阻碍了遗传作图和QTL(数量性状位点)研究。因此,只发表了少量的连锁图。所有这些方法都使用了野生物种来产生足够多态的标记。限制性片段长度多态性(RFLP)图谱是Halward基于两个AA基因组物种的杂交而开发的,答:stenospermaKrapov。& WC Gregory和答:cardenasiiKrapov。& WC Gregory,以及基于TxAG-6(一种合成的双倍体)杂交的四倍体图谱答:hypogaea由Burow等人出版。[9].最近,我们建立了一个基于ssr的AA基因组图谱落花生基于答:stenosperma而且答:duranensisKrapov。& WC Gregory [10]和一幅BB基因图谱,该图谱是基于答:ipaensisKrapov。,WC Gregory & CE Simpson and答:麦格纳Krapov。,WC Gregory & CE Simpson [11].目前有54168个无害环境技术用于答:hypogaea在Genbank [12- - - - - -14],野生AA基因组为6264个答:stenosperma(15,16].
细菌人工染色体(BAC)文库是基因组研究的基础工具,在物理作图、基于基因图谱的基因克隆和基因结构与功能分析方面具有重要意义。与酵母人工染色体(YAC)载体相比,BAC载体易于处理和繁殖,相对稳定,嵌合程度低,这使得BAC载体成为克隆系统的选择[17,18].使用大插入克隆进行物理制图已经提出了许多策略:基于杂交的方法,例如使用单个cDNA或基因组克隆作为探针的交互式杂交[19],基于限制的指纹识别方法[20.]综合BAC端测序和指纹图谱分析[21]或更近的,以寡核苷酸为基础的"overgos"的使用[22].
在豆科中,BAC库可用于菜豆(23),豇豆属辐射动物(24),大豆(25),三叶草借口(26和豆科植物模型Lotus对虾(27),Medicago truncatula(28].属内落花生,已开发出一个异源多倍体栽培花生的BAC文库[29].作为这一资源的补充,我们在这里描述了两个二倍体野生物种的BAC文库的生产答:duranensis(AA基因组)和答:ipaensis(BB基因组)被确定为栽培花生最可能的祖先[8,30.].利用全基因组现场我们还进一步研究了所选二倍体基因组的亲缘性和覆盖范围答:hypogaea。
结果
现场杂交过程
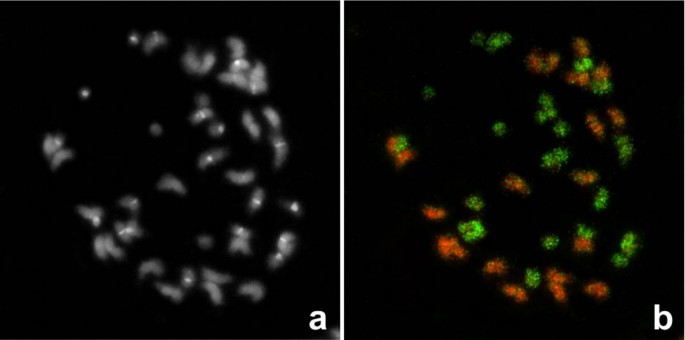
总基因组DNA答:duranensis(AA基因组)和答:ipaensis(BB基因组)当用作探针的染色体答:hypogaea在AA和BB染色体上表现出强烈而均匀的杂交模式答:hypogaea分别(无花果。1).在四倍体中,染色体亚群与相应亲本基因组之间的这种清晰的基因组区分是可能的,而不需要任何未标记的阻断DNA,这通常用于避免来自一个基因组的特定探针与来自另一个基因组的同源DNA序列之间的交叉杂交。反染色的A和B染色体显示两个基因组组成部分的总大小非常相似(图2)。1).
高分子量DNA的分离
使用标准的高分子量DNA分离方案[31,32并不能产生足够数量的高质量原子核落花生。两者都含有高含量的碳水化合物和多酚答:duranensis而且答:ipaensis叶子。这导致高粘度的叶子提取物,难以过滤,大大增加了核暴露于氧化物质作用的时间。为了克服这一问题,需要进行一些修改:在提取缓冲液中加入PVP-40,用四层粗棉布过滤叶片提取物,然后用两层miraccloth过滤,低速离心(60 × g 2分钟)和Percoll梯度(37.5%)。通过dapi染色显微镜对提取物进行分析,可以正确评估核制剂的数量和质量。
额外的纯化步骤,包括在消化前对琼脂糖塞进行PFGE 40分钟,能够清除较小的片段并消除杂质,提高克隆效率。为了在电洗脱后获得最高数量的限制性DNA,在收集管的最底部只回收50 μL,而不是标准方案中描述的300 μL [21].在双尺寸选择后,以1/4 V/I的比例进行不同比例的结扎试验,得到的转化子数量最多。总的来说,获得每个BAC文库需要5种不同的结扎方法(表2)1).
BAC文库特征
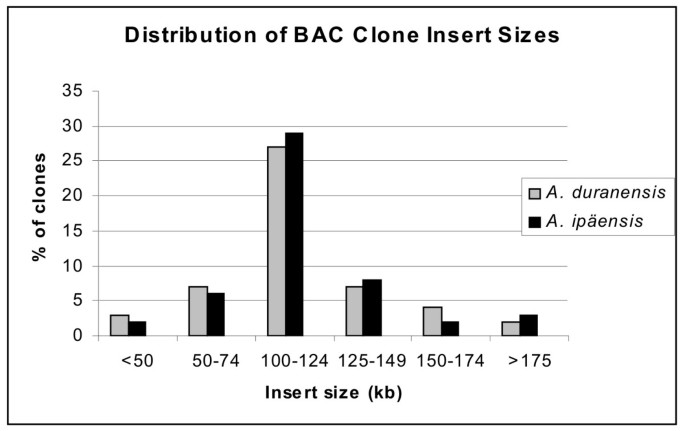
AA基因组的BAC文库(答:duranensis)包含84,096个克隆,而BB基因组文库(答:ipaensis)的人数为75,648人(表1).每个库的随机样本被分析不我消化,平均插入大小为110和100 kb答:duranensis而且答:ipaensis分别(无花果。2而且2 b;无花果。3.).
通过线粒体和叶绿体特异性探针筛选高密度过滤器来评估两个BAC文库中的细胞器污染。为答:duranensisBAC克隆的叶绿体序列污染率为0.016%,线粒体序列污染率为0.21%。为答:ipaensis0.363%的克隆被叶绿体序列污染,0.081%的克隆被线粒体DNA污染。这些值,连同显微镜下的dapi染色观察,反映了高水平的纯化落花生用改进的方法得到的核。
基于库平均插入大小和答:duranensis单倍体基因组,相当于1260 Mb [33],估计AA基因组BAC文库的覆盖率为7.4个单倍体基因组当量。然而,对于答:ipaensis在美国,dna含量的测定存在争议。辛格等人报道的单倍体基因组可能相当于2830 Mb [34是高估了2.0倍,因为测量不一致,正如已经描述的其他落花生物种(33,35因此,BB基因组BAC库为答:ipaensis可代表该物种2.7 ~ 5.3个单倍体基因组当量。考虑到A和B染色体对于四倍体的两个基因组组成部分显示出非常相似的大小(如上所述,图。1)我们认为后者是较好的估计。为了进一步测试覆盖范围,用豆科植物中单拷贝基因对应的探针作为锚定标记进行高密度筛选,并将其放置在落花生AA基因图谱(未发表数据)。在AA基因组中,每个探针平均鉴定出5.1个克隆,在BB基因组中鉴定出4.5个(表2)2).
两个BAC库的杂交落花生抗性基因类似物RGA S1_A_36在AA基因组中鉴定出两个克隆,但在BB基因组中没有2).
讨论
栽培花生是一种具有AA和BB两个核基因组成分的异体四倍体。虽然人们普遍认为这些组成基因组来自二倍体野生祖先,但涉及的确切物种一直是一些研究和讨论的问题。虽然证据还不完全明确,但分子标记、细胞遗传学、形态学和地理分布的数据分析支持这一点答:duranensis而且答:ipaensis是栽培花生的直系祖先[8,30.].
基因组原位的杂交(GISH)答:hypogaea中期染色体,总基因组DNA来自AA基因组答:duranensis和BB基因组答:ipaensis使得a和B染色体有明显的分化。首先,这一观察结果加强了人类基因组之间密切关系的证据A. duranensis, A. ipaënsis还有种植花生。其次,由于GISH在很大程度上依赖于重复序列的杂交,它也表明答:duranensis而且答:ipaensis基因组在重复序列/转座元件含量方面存在很大分歧。
相反,现有的证据表明AA和BB基因组中的基因顺序基本上是保守的[9].这种在快速进化的重复DNA中嵌入大量同位基因框架的情况似乎是植物进化中反复出现的主题[36- - - - - -39].
来自异源多倍体和两个野生祖先的BAC文库的可用性将允许对这些基因组的微同步性和重复DNA含量进行比较,特别是转座元件,并将为多倍体基因组进化的迷人领域提供见解。
此外,用于文库特征的豆科单拷贝锚定标记将允许在AA和BB之间识别和比较同源区域落花生基因组,与其他豆类基因组,特别是测序的基因组莲花而且Medicago,为豆科植物的基因组进化提供了见解。
在某些植物中构建BAC文库可能需要大量的工作,这是由于我们以前已经经历过的特殊化学结构[32].为落花生在美国,由于多酚类和碳水化合物的污染,获得高质量的HMW DNA是很困难的落花生叶子。为了获得合理数量的BAC克隆,必须在核提取的标准方案中增加若干步骤,并进行若干结扎。Yuksel和Paterson也报告了这些困难[29]用于建造一个hypogaeaBAC图书馆。
BAC文库的基因组覆盖率估计为A基因组7.4 - 5.1个单倍体基因组当量,B基因组5.3 - 4.5个单倍体基因组当量。这些差异是由于某些基因组区域内限制位点密度的变化或克隆过大或过小片段的困难[40].考虑到99%的覆盖率相当于4.7×单倍体基因组[40]答:duranensis而且一个ipaensis基因组被很好地表示出来,库将适用于许多应用。
二倍体BAC文库的可用性落花生将大大方便开发一个参考物理地图落花生。这个物理地图的构建将很快启动答:duranensisBAC图书馆。的构造所使用的映射种群的父种落花生基于ssr的地图[10].答:ipaensis也是映射总体的父类[11].这将促进遗传和物理图谱的整合。
花生的二倍体野生亲缘种具有多种抗生物和非生物胁迫的特性,因此对其进行育种开发具有很大的吸引力。用于BAC文库的两个物种对线虫和真菌疾病具有抗性[41,42].这些新的BAC资源将有助于标记和/或分离相应的抗性基因。例如,一个抗晚叶斑病的QTL是由Cercosporidium personatum被映射成十字的答:duranensis与答:stenosperma(43].与该QTL共分离的RGA S1_A_36已被用于鉴定该QTL中的两个BAC克隆答:duranensis库,而没有克隆答:ipaensis图书馆被找到了。BAC克隆的测序可以在目标区域识别微卫星,从而为分离群体的QTL跟踪提供更方便的标记。
结论
总之,在这里我们描述了AA和BB基因组BAC文库的产生落花生。这些文库将为基因的分离、物理和遗传图谱的构建和相关性、细胞遗传学分析探针的分离、两种基因组类型的进化研究以及与栽培花生同种异体四倍体基因组的比较,提供研究多倍体基因组进化的有用资源。
方法
植物材料
种子从落花生种质收集,保存在Embrapa遗传资源和生物技术- CENARGEN (Brasília-DF,巴西)。将种子放在加1%乙烯利湿的吸水纸上,可提高种子的萌发率。对于高分子量DNA的分离,答:duranensisV14167(基因组AA)和答:ipaensisKG30076 (BB基因组)在温室条件下生长。幼叶收集在液氮中,然后保存在-80°C。
探针标记和荧光原位杂交
全基因组DNA答:duranensis而且一个。ipaensis在基因组中被用作探针原位杂交(《)。探针用地高辛-11- dutp (Roche, Mannheim, Germany)或生物素-11- dutp (Sigma)标记标记。
预处理后的制剂,染色体和探针变性,条件为原位根据Moscone等人的方法进行杂交(杂交混合物中含有浓度为2.5-3.5 ng/μL的DNA探针)、杂交后清洗、阻断和氟色素偶联抗体间接检测[44].抗体由小鼠抗生物素(Dakopatts, Dako, Carpinteria, California, USA)和绵羊抗地高辛素偶联异硫氰酸荧光素(FITC) (Roche)在PBS (0.13 mol/L NaCl, 0.007 mol/L Na)中组成2HPO4, 0.003 mol/L NaH2阿宝4), 3% (w/v)牛血清白蛋白(BSA)或兔抗小鼠偶联异硫氰酸四甲基罗丹明(TRITC) (Dakopatts)和fitc偶联兔抗绵羊(Dakopatts)在PBS中,3% (w/v) BSA。用含有2 μg/mL 4',6-二氨基-2-苯基吲哚(DAPI, Sigma)的Vectashield培养基(Vector Laboratories, Burlingame, California, USA)对制剂进行反染色和裱装。
GISH后的DAPI反染导致C带样模式,主要异染色质带荧光更强烈,从而有助于染色体识别[8,44].
荧光显微镜和图像采集
染色体用Leica DMLB荧光显微镜(Leica, Heerbrugg, Switzerland)观察和拍摄,并配有计算机辅助的Leica DC 250数码相机系统。红色、绿色和蓝色图像分别使用适当的TRITC、FITC和DAPI激发滤波器以黑色和白色捕获。数字图像使用IM 1000 Leica软件进行伪彩色和组合,然后导入Adobe Photoshop, version 7.0 (Adobe, San Jose, California, USA)进行最终处理。
高分子量DNA分离
根据Meyers等,从叶片中分离出核。[31作了一些修改。将50克幼叶在液氮中研磨,细胞提取物在HB 1×提取缓冲液中,加0.2%聚乙烯吡咯烷酮(PVP 40),在4°C下孵育20分钟,释放细胞核。在缓冲液中加入PVP以减少氧化性多酚物质的产生。为了消除细胞碎片,叶片匀浆依次用四层粗棉布过滤,然后用两层miraccloth (250 μm) (Calbiochem, UK)过滤,并进行低速离心(60 × g, 2 min)。以850 × g在4℃下离心8分钟,然后用Percoll梯度(37.5%)从果胶基质中分离核[45].用不含β-巯基乙醇和Triton-100×的HB 1×萃取缓冲液20 mL洗涤核,4℃,850 × g离心8 min。最后,核球在1ml过滤后的hb1×中重悬,并嵌入1.2%低熔点琼脂糖塞(InCert琼脂糖,Cambrex-Bioscience, Rockland, Inc.)。在显微镜下使用DAPI染色对核提取的样品进行评价,以观察核的完整性和制剂的纯度。含HMW DNA的琼脂糖塞在50°C裂解缓冲液(5% Sodium Lauryl Sarcosyl, 0.625 M EDTA pH 9.0, 50 mg蛋白酶K)中孵育24小时,在4°C灭活液(0.5 M EDTA, pH 8.0, 1 mM PMSF)中洗涤1小时,然后在TE 10/10 (10 mM Tris-HCl, 10 mM EDTA, pH 8.0)中洗涤4次30分钟。使用CHEF Mapper™XA仪器(Bio-Rad,英国)进行脉冲场凝胶电泳(PFGE),在6 V/cm下进行额外的HMW DNA纯化步骤,开关时间为3秒,角度为120°,持续40分钟,目的是消除降解的DNA。琼脂糖塞最后在TE 10/1 (10 mM Tris-HCl, 1 mM EDTA, pH 8.0)中洗涤4次30分钟。4°C,然后用于限制性内切酶消化。
BAC文库建设
将含有HMW DNA的琼脂糖塞切成小块,在冰上搅拌,在1毫升中搅拌三次后III限制缓冲液(Gibco BRL,美国),每30分钟更换一次缓冲液后将III添加到每个切碎的塞中,并在冰上扩散4小时。对于部分消化,在37°C下孵育15分钟,然后通过添加总体积的十分之一的0.5 M EDTA, pH 8.0来停止反应。部分消化的HMW DNA在1% GTG SEAKEM琼脂糖凝胶中,0.5× TBE, 14°C,连续两次PFGE选择大小。第一次上胶是在6 V/cm下进行的,开关时间为1到50秒,角度为120°,持续20小时。从凝胶中切除80到150 kb和150到250 kb的两个区域,并加载到新凝胶上。然后在6 V/cm下进行第二次筛选,切换时间为3秒,角度为120°,持续20小时。从后一种凝胶中切割100至250 kb的区域,通过电洗脱(BIO-RAD/电洗脱器,英国)回收DNA。在1%琼脂糖凝胶中测定1× TAE的DNA浓度。几种结扎反应被测试包含不同比例的载体与插入物。恒定30纳克的商业载体“pIndigo BAC-5”HindIII-克隆准备”(Epicentre, USA)使用了不同数量的DNA,从50到600 ng的高分子量DNA。将一微升结扎液混合到20微升合格液中大肠杆菌电池(ElectroMAX DH10B, Invitrogen),并根据制造商的建议使用BRL Cell-Porator系统进行电转换,但充电率为355伏。在含12.5 μg/mL氯霉素、50 μg/mL 5-溴-4-氯-3-吲哚-β- d -半乳糖苷(X-Gal)和25 μg/mL异丙基硫半乳糖苷(IPTG)的2YT板(色氨酸16 g/L、酵母浸膏10 g/L、氯化钠5 g/L、琼脂16 g/L)上选择转化产物。白色菌落采用Q-Pix 2菌落采摘机器人(Genetix)采摘,转移到384孔板上,板中含80 μL的2YT, 7%甘油。微板在37℃下孵育18-20小时,-80℃保存。
BAC文库筛选及DNA分离
为估计BAC克隆插入大小,随机将单个克隆分别在100 μL的无瘤前培养,然后在3ml含氯霉素(12.5 μg/mL)的2YT液体培养基中培养。BAC DNA使用QIAGEN BIO-ROBOT 9600 (QIAGEN GmbH, Germany)进行分离。BAC DNA用不我要释放插入。消化后的克隆用PFGE在6 V下分离,切换时间从5到15秒,角度为120°,运行15小时。高密度过滤器使用Q-Pix 2机器人(Genetix)制作。每个高密度过滤器包含18432个双斑点克隆。杂交按照Clemson BAC协议进行[45].滤镜在Ferrania LifeRay xcg -film中暴露24小时。
基因探针
通过使用来自菠菜叶绿体基因、Rubisco大亚基(1.5 kb)和小麦细胞色素氧化酶线粒体基因的高密度过滤器杂交评估了两个文库中细胞器污染的估计考克斯我(1.3 kb) [32].BAC文库也与已被定义为豆科锚定标记的单拷贝基因的探针杂交[46]和落花生抗性基因类似物S1_A_36 [47] (Genbank加入AY157808)。该RGA是从AA基因组种中分离得到的答:stenosperma并且已发现它与一个抗晚叶斑病的QTL共定位Cercosporidium personatum(43].
参考文献
- 1.
粮农组织:粮农组织农业统计,2003
- 2.
杨晓明,李志强,李志强,李志强:豆科植物基因组图谱研究。植物基因组图谱。编辑:Paterson AH LRG。1996年,奥斯汀
- 3.
Halward TM, Stalker HT, Kochert G:二倍体花生RFLP连锁图谱的建立。中国生物工程学报,2003,27(3):349 - 349。10.1007 / BF01184927。
- 4.
Simpson CE, Starr JL, Church GT, Burrow MD, Paterson AH: NemaTAM花生注册。作物科学,2003,43:1561-
- 5.
Singh AK, Smartt J:花生(Arachis hypogaea L.)基因组供体的重新考察。作物遗传资源与遗传进化,1998,45:113-118。10.1023 /: 1008640631719。
- 6.
Moretzsohn MC, Hopkins MS, Mitchell SE, Kresovich S, Valls JF, Ferreira ME:基于基因组高变区分析的花生(Arachis hypogaea L.)及其野生近缘的遗传多样性中国生物医学工程学报,2004,4:11-10.1186/1471-2229-4-11。
- 7.
Valls JFM, Simpson CE:巴西、巴拉圭和玻利维亚豆科花生属新种。中华医学杂志,2005,14:35-64。
- 8.
Seijo JG, Lavia GI, Fernández A, Krapovickas A, Ducasse D, Moscone EA: 5S和18S-25S rRNA基因的FISH物理图谱证明了Arachis duranensis和A. ipaënsis是A. hypogaea(豆科)的野生二倍体祖细胞。中国植物学报,2004,29(3):344 - 344。
- 9.
杨晓明,陈晓明,陈晓明,陈晓明。人工合成双二倍体花生染色质的遗传分析。拓宽单系多倍体物种的基因库。中国生物医学工程学报,2001,39(2):344 - 344。
- 10.
Moretzsohn MC, Leoi L, Proite K, Guimaraes PM, Leal-Bertioli SC, Gimenes MA, Martins WS, Valls JF, Grattapaglia D, Bertioli DJ:基于微卫星的花生(豆科)AA基因组富基因连锁图谱。中国生物医学工程学报,2005,26(6):357 - 357。10.1007 / s00122 - 005 - 0028 - x。
- 11.
Gobbi A, Teixeira C, Moretszohn M, Guimarães PM, Leal-Bertioli S, Bertioli D, Lopes CR, Gimenes MA:花生(Arachis hypogaea-AABB) B基因组连锁图谱的建立:San Diego, Califórnia。2006,, 679-
- 12.
罗敏,梁晓强,党鹏,Holbrook CC, Bausher MG,李瑞德,郭伯忠:基于微阵列技术的花生抗曲霉菌感染和干旱胁迫差异表达基因的筛选。植物科学学报,2005,29(3):344 - 344。10.1016 / j.plantsci.2005.05.020。
- 13.
罗敏,党鹏,Bausher MG, Holbrook CC, Lee RD, Lynch RE,郭伯忠:花生对人状Cercosporidium personatum叶斑病抗性应答的转录本鉴定。植物病理学杂志,2005,30(3):381-387。10.1094 /发朵- 95 - 0381。
- 14.
罗敏,党鹏,郭伯泽,贺刚,Holbrook C, Bausher MG, Lee RD:花生基因发现与标记物开发的ESTs生成。作物科学,2005,45:346-353。
- 15.
Proite K, Leal-Bertioli SC, Bertioli DJ, Moretzsohn MC, da Silva FR, Martins NF, Guimaraes PM:来自野生花生物种的基因发现和标记开发的ESTs。中国生物医学工程学报,2007,27(3):344 - 344。
- 16.
Martins W, de Sousa D, Proite K, Guimaraes P, Moretzsohn M, Bertioli D:自动化微卫星标记开发的新软件。核酸研究,2006,34 (4):e31-10.1093/nar/gnj030。
- 17.
Dvorak J,杨志林,游FM,罗春春:小麦染色体不同重组率区域的缺失多态性。中国生物医学工程学报,2004,18(3):344 - 344。10.1534 / genetics.103.024927。
- 18.
Chalhoub B, Belcram H, Caboche M:植物基因组高效克隆到插入尺寸更大更均匀的细菌人工染色体(BAC)文库。植物学报,2004,2(3):181-188。10.1111 / j.1467-7652.2004.00065.x。
- 19.
Mozo T, Dewar K, Dunn P, Ecker JR, Fischer S, Kloska S, Lehrach H, Marra M, Martienssen R, Meier-Ewert S, Altmann T:拟南芥基因组基于bac的完整物理图谱。自然遗传学报,1999,22(3):271-275。10.1038/10334。
- 20.
Hong G:指纹与杂交相结合构建水稻contig图谱的快速准确策略。植物学报,1997,35(1):1- 3。10.1023 /: 1005741620666。
- 21.
Mayer K,舒乐问C, Wambutt R,墨菲G, Volckaert G,波尔T, Dusterhoft, Stiekema W, Entian KD, Terryn N,哈里斯B, Ansorge W,布兰德P, Grivell L, Rieger M, Weichselgartner M,德西蒙V,奥氏B,马谢R,穆勒M,克瑞斯M, Delseny M, Puigdomenech P,沃森M, Schmidtheini T, B Reichert Portatelle D, Perez-Alonso M, Boutry M,班克罗夫特,Vos P, Hoheisel J,齐默尔曼W, Wedler H,里德利P,朗廷SA McCullagh B, Bilham L,罗本J, Van der Schueren J, Grymonprez B,壮族YJ,Vandenbussche F, Braeken M, Weltjens我,沃特,Bastiaens我Aert R, Defoor E, Weitzenegger T,博特G, Ramsperger U,希尔伯特H,布劳恩M,霍尔泽E,布兰德,彼得斯,van Staveren M, Dirske W, Mooijman P,克莱因Lankhorst R,玫瑰M, Hauf J,科特P, Berneiser年代,亨佩尔年代,Feldpausch M,兰博斯年代,van den Daele H, De大尺度,Buysshaert C, Gielen J, Villarroel R, De Clercq R·蒙塔古,罗杰斯J,克罗宁,鹌鹑,Bray-Allen年代,克拉克L,道根J,大厅,凯米,林纳德N, McLay K,梅耶斯R, Pettett Rajandream马•莱恩M,贝奈斯V, Rechmann年代,Borkova D,拦截器H, Scharfe M,格林M, Lohnert TH,剂量,de Haan M, Maarse,谢弗米,Muller-Auer年代,Gabel C, Fuchs M, Fartmann B, Granderath K, Dauner D,赫茨尔,诺伊曼年代,Argiriou A, D,维托Liguori说道R, Piravandi E,马斯奈啊,奎格利F, G Clabauld, Mundlein, Felber R, Schnabl年代,希勒R,施密特W, Lecharny,奥堡年代,Chefdor F,库克R,伯杰C,蒙特福特,Casacuberta E,长臂猿T,韦伯N, Vandenbol M,Bargues M, Terol J,托雷斯,Perez-Perez, Purnelle B, E弯曲,约翰逊,顶端D,杰西T, Heijnen L,施瓦兹的年代,Scholler P,希年代,法郎P, Bielke C, Frishman D, hasse还D, Lemcke K,海鸥HW,储料器年代,扎P,贝文M,威尔逊RK, de la巴斯蒂德M, Habermann K,帕内尔L, Dedhia N, Gnoj L,舒茨的K,黄E,明镜周刊L, Sehkon M,穆雷J,表P, Cordes M, Abu-Threideh J,斯托金T, Kalicki J,坟墓T,哈蒙G,爱德华J,有P,考特尼L,云J,雅培,斯科特•K约翰逊D,风骚女子P,宾利D,富尔顿B,米勒N,希腊T,坎普K,克雷默J,富尔顿L,狂欢节E,但丁M, Pepin K,希利尔L,纳尔逊J, Spieth J,瑞安E,安德鲁斯年代,Geisel C, D门外汉,Du H,阿里•J Berghoff,琼斯K,无人机K,棉花,禅师C, B Antonoiu Zidanic M,强大的C,太阳H,拉马尔B, Yordan C,马P,钟J,普雷斯顿R,维尔D, Shekher M, Matero,沙R, Swaby本土知识,O ' shaughnessy,罗德里格斯M,霍夫曼J,直到年代,石榴,Shohdy N,长谷川,Hameed,卢迪M,约翰逊,陈E,马丽娟,马丽娟,王丽娟:拟南芥4号染色体的序列分析。自然学报,2004,29(3):339 - 344。10.1038/47134。
- 22.
Gardiner J, Schroeder S, Polacco ML, sandez - villeda H, Fang Z, Morgante M, Landewe T, Fengler K, Useche F, Hanafey M, Tingey S, Chou H, Wing R, Soderlund C, Coe EH:利用二维超杂交技术将9371个玉米表达序列标记的unigenes固定在细菌人工染色体contig图上。植物生理学报,2004,34(4):344 - 344。10.1104 / pp.103.034538。
- 23.
王志强,王志强,王志强:大豆细菌人工染色体文库的构建与鉴定。植物学报,1999,40(6):977-983。10.1023 /: 1006234823105。
- 24.
Miyagi M, Humphry M, Ma ZY, Lambrides CJ, Bateson M, Liu CJ:细菌人工染色体文库的构建及其在绿豆(Vigna radiata L. Wilczek)抗性调控的pcr标记中的应用。中国生物医学工程学报,2004,30(1):357 - 357。10.1007 / s00122 - 004 - 1821 - 7。
- 25.
Danesh D, Peñuela S, Mudge J, Denny RL, Nordstrom H, Martinez JP, Young ND:大豆细菌人工染色体文库的建立及其对包囊线虫抗性基因的克隆鉴定。中国生物工程学报,1998,26(3):357 - 357。10.1007 / s001220050727。
- 26.
Sato S, Isobe S, Asamizu E, Ohmido N, Kataoka R, Nakamura Y, Kaneko T, Sakurai N, Okumura K, Klimenko I, Sasamoto S, Wada T, Watanabe A, Kohara M, Fujishiro T, Tabata S:红三叶草(Trifolium pratense L.)基因组的综合结构分析。DNA研究,2005,12(5):301-364。10.1093 / dnares / dsi018。
- 27.
川崎S,村上Y:莲叶的基因组分析。植物科学学报,2000,19(4):379 - 379。
- 28.
Nam YW, Penmetsa RV, Endre G, Uribe P, Kim D, Cook DR:截叶苜蓿细菌人工染色体文库的构建及含乙烯反应基因的克隆鉴定。《遗传学与应用》,1999,98:3-4。
- 29.
Yuksel B, Paterson AH:花生印地二BAC文库的构建和表征。中国生物医学工程学报,2005,29(4):344 - 344。10.1007 / s00122 - 005 - 1992 - x。
- 30.
Kochert G, Stalker HT, Gimenes M, Galgaro L, Lopes CR, Moore K:同种异体四倍体驯化花生Arachis hypogaea起源和进化的RFLP和细胞遗传学证据。中国植物学报,2004,27(3):344 - 344。10.2307 / 2446112。
- 31.
张海波,赵霞,丁霞,Paterson AH, Wing RA:从植物细胞核中制备百万酶大小DNA。植物学报,1995,7:175-184。10.1046 / j.1365 - 313 x.1995.07010175.x。
- 32.
Vilarinhos AD, Piffanelli P, Lagoda P, Thibivilliers S, Sabau X, Carreel F, D'Hont A:香蕉(Musa acuminata Colla)细菌人工染色体文库的构建与鉴定。应用物理学报,2003,16(6):1102-1106。
- 33.
李志刚,李志刚,李志刚:花生基因组大小的研究。基因组学报,2001,44(5):826-830。10.1139 / gen - 44 - 5 - 826。
- 34.
陈晓峰,陈晓峰,陈晓峰:染色体DNA变异与花生进化的关系。中国生物工程学报,1996,39:890-897。
- 35.
孙文杰,李文杰,李文杰:花生和花生基因组大小变异的重新评价。基因组学报,2000,43(3):449-451。10.1139 / gen - 43 - 3 - 449。
- 36.
Gale MD, Devos KM: 10年后的植物比较遗传学。科学通报,1998,28(3):344 - 344。10.1126 / science.282.5389.656。
- 37.
McCouch SR:基因组学和Synteny。植物生理学报,2001,29(3):344 - 344。10.1104 / pp.125.1.152。
- 38.
染色体重排与转座因子。中国生物医学工程学报,2002,36:389-410。10.1146 / annurev.genet.36.040202.092802。
- 39.
Kazazian HH:移动元素:基因组进化的驱动力。科学通报,2004,34(4):344 - 344。10.1126 / science.1089670。
- 40.
张海波,吴春:BAC作为基因组测序工具。植物生理学报,2001,29(3):344 - 344。10.1016 / s0981 - 9428(00) 01236 - 5。
- 41.
花生对花生锈病抗性成分的研究。植物病理学,1983,253-256。
- 42.
孙志强,李志强,李志强:花生属植物种质资源的抗性分析。线虫学报,1989,21():654-660。
- 43.
Moretzsohn MC: Desenvolvimento e mapeamento de marcadores microssatélites e identificação de qtl ligados à productividade e à resistência à mancha preta em Arachis spp.分子生物学。Brasília, Universidade de Brasília,博士论文:2006,142
- 44.
Moscone EA, Klein F, Lambrou M, Fuchs J, Schweizer D:豆科栽培菜豆(Phaseolus) 5S和18S-25S rDNA探针的定量核型和双色FISH定位。基因组学报,1999,42(6):1224-1233。10.1139 / gen - 42 - 6 - 1224。
- 45.
Peterson DG, Tomkins JP, Frisch DA, Wing RA, Paterson AH:植物细菌人工染色体(BAC)文库的构建:图解指南。农业基因组学杂志,2000,5:
- 46.
Fredslund J, Madsen LH, Hougaard BK, Nielsen AM, Bertioli D, Sandal N, Stougaard J, Schauser L:植物比较基因组学中锚定标记开发的一般管道。BMC基因组学杂志,2006,7:207-10.1186/1471-2164-7-207。
- 47.
Bertioli DJ, Leal-Bertioli SC, Lion MB, Santos VL, Pappas G, Cannon SB, Guimaraes PM:花生属抗性基因同源物的大规模分析。分子遗传学与基因组学研究,2003,27(1):34-45。10.1007 / s00438 - 003 - 0893 - 4。
确认
这项工作是由一代挑战计划(项目#31)和主办机构资助的。Karina Proite获得了Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES)授予的博士奖学金。
作者要感谢José.F.M博士。Valls提供了宝贵的建议,并从EMBRAPA的种质库中提供了种子,同时也感谢Xavier Sabau提供的机器人技术援助。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
PMG构思了这项研究,建造了图书馆并起草了手稿。KP建造了这些库,并参与了实验计划。OG将BAC库构建协议改编为Arachis,构建了这些库,并为手稿的撰写做出了贡献。SCLB构思了这项研究,提供了资源并参与了手稿的起草。GS进行了细胞遗传学实验。CC负责温室内植物的维护。DJB构思并协调了这项研究,并起草了手稿。ADH提供了材料和资源,协调了图书馆的建设,并参与了手稿的撰写。所有作者都阅读并批准了手稿。
Patricia M Guimarães, Olivier Garsmeur, Karina Proite对这项工作做出了同样的贡献。
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
Guimarães, p.m., Garsmeur, O., Proite, K.。et al。花生异体四倍体祖先二倍体基因组BAC文库的构建。BMC植物生物学8,14(2008)。https://doi.org/10.1186/1471-2229-8-14
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-14
关键字
- 细菌人工染色体
- 落花生
- 细菌人工染色体克隆
- 细菌人工染色体库
- 晚叶斑