摘要
背景
苹果果实从开花到完全成熟需要150天。从苹果est中设计的约13000个基因(15726个45-55碱基的寡核苷酸)的阵列已用于研究果实发育过程中8个时间点的基因表达。这种基因表达的分析为苹果果实生长发育的分子理解奠定了基础。
结果
利用微阵列数据的方差分析,1955个基因的表达在这段时间内发生了显著变化。基因的表达与观察到的四种主要表达模式相协调:在花蕾中高表达;细胞分裂时高;当淀粉水平和细胞扩张速率达到峰值时高;在成熟过程中也很高。细胞周期基因与果实早期发育相关,3个核心细胞周期基因在果实发育早期显著上调。淀粉代谢基因与果实发育过程中淀粉水平的变化有关。通过与乙烯处理苹果果实的基因芯片的比较,发现了一组乙烯诱导的基因也能诱导正常的果实成熟。通过与番茄果实发育基因芯片的比较,发现了16个在苹果和番茄中表达模式相似的基因,这些基因可能在果实发育中起着重要作用。授粉后35天内发生的细胞分裂和组织规范的早期阶段与包括核心细胞周期基因在内的一组基因的上调有关。
结论
苹果果实的基因表达与特定的发育阶段相协调。该阵列的结果是可重复的,并与其他物种的实验进行了比较,以确定可能在果实发育中发挥基本作用的基因。
背景
结果作物品种是人类饮食的重要组成部分,提供营养、饮食多样性和乐趣。果实通常被认为是围绕植物发育中的种子的一种扩大的器官,或花成熟的子房以及任何相关的附属部分[1].子实体的发育和最终形式变化很大,从模式植物拟南芥的最小扩张的简单开裂(非肉质)果实,到番茄的扩张子房,再到具有几种不同扩张组织的复杂子器官,如在红果中发现的[1].所有水果都有一个共同的发育过程,即种子附近组织的扩张与种子的发育协调一致(通常,但不总是,包裹着种子)。在发育的早期阶段(受精成功前和受精成功后,有时在没有受精的情况下),果实组织经历几轮细胞分裂,随后(通常)是细胞扩张,在此期间果实以淀粉或糖的形式储存代谢物和能量(例如番茄发育[2- - - - - -4])。随后,通常在种子成熟后,果实经历一系列生化变化,将淀粉转化为更容易获得和更有吸引力的化合物,如糖,并产生挥发性次级代谢物,这些代谢物被认为是动物或昆虫的引诱剂,可以传播种子。
果实的形态学和生理学研究使人们对果实成熟和成熟过程中发生的物理和生化事件有了相当大的了解[1,3.,5],但直到最近,基因组方法才被用于研究果实的发育[4,6- - - - - -9].由于优良的遗传资源和分子和基因组方法的应用,番茄已成为研究最好的不开裂水果。番茄的驯化使果实大小从几克增加到大1000倍的品种[10].导致番茄花的卵巢壁扩张的生理事件,特别是发生在番茄成熟前后的事件已经被很好地描述(评论见Gillaspy等人。2];Giovanonni [3.])。最近,分子方法已用于研究番茄的整体基因表达[11- - - - - -13从而鉴定出大量可能与果实发育和成熟有关的基因。
在其他水果作物中,微阵列已被用于检测水果发育过程中的基因表达,特别是草莓[6],桃子[14],梨[15],以及grape [8,9].这些研究已经确定了与水果风味有关的基因和与水果发育的不同阶段相关的基因。
苹果(马吕斯×有明显Borkh。也被称为m . pumila)是蔷薇科蔷薇亚科的成员,包括梨、玫瑰和榅桲等作物品种。番茄科成员的果实由两个不同的部分组成:一个膨胀的子房对应于“核心”,与番茄果实相似;果皮:果实的皮质或可食用部分,由雄蕊、花瓣和萼片的基部融合而成[1,16],它会扩张到卵巢周围。果实从授粉到完全成熟需150天,呈简单的s型生长曲线[17,18].苹果果实发育的生理学研究主要集中在成熟度的测量上,如颜色的变化和淀粉的分解以形成可口的糖。从这些研究中可以看出,花蕾含有少量的淀粉,授粉后会迅速代谢掉。水果中的淀粉含量随着细胞的扩张而增加。授粉后约100天,淀粉含量再次开始下降,果实糖分增加,直到果实完全成熟[19].和番茄一样,苹果也经历一个乙烯依赖的成熟阶段[20.,21]而乙烯产量减少的转基因苹果不会产生皮肤颜色的变化,而且似乎也不会产生苹果特有的挥发性化合物[22].
苹果在功能上是一个二倍体,2n = 34,基因组大小适中(1C = 2.25 pg [23]大约相当于1.5 × 109使其生物学研究的基因组方法合理化。最近,EST测序方法已被用于鉴定苹果基因[24];从该测序项目中获得的Unigenes被用于设计本工作中使用的寡核苷酸。有两个研究小组发表了苹果微阵列分析[22,25].Lee等人。[25]利用3484个特征cDNA阵列,鉴定了192个苹果早期果实发育过程中表达变化的cDNA。Schaffer等人利用本文描述的相同的~13000基因(15726特征)苹果寡核苷酸阵列[22他们在水果中发现了944个对乙烯处理有反应的基因,并将基因表达的变化与水果挥发物的变化联系起来。
在本文所述的工作中,微阵列已用于研究从授粉到完全成熟的果实形成过程中发生的发育过程。在红果中,核心(子房)和皮层(托杯)组织都扩张。这项工作的最终目标是了解产生复杂苹果果实所需的事件的调节,包括来自不同花结构的细胞的分裂和扩张。利用微阵列技术,我们发现大量基因在果实发育的特定阶段协调表达。我们在苹果果实中鉴定了表达与细胞分裂周期一致的细胞分裂基因,并鉴定了可能参与水果储存和代谢淀粉的淀粉代谢酶。利用比较的方法,我们已经确定了一些在苹果和番茄果实发育中表达模式相似的基因,这些基因可能参与了果实发育的相似基本过程。
结果
苹果果实发育的芯片分析
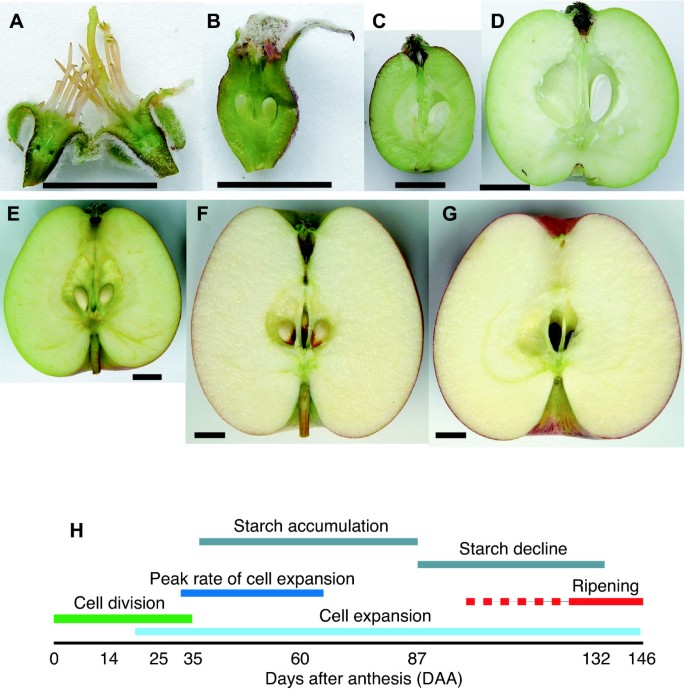
当苹果树(马吕斯有明显'Royal Gala')完全开放(超过50%的花蕾开放),对完全开放的花朵进行标记,并将树木分成两个生物重复(Rep1和Rep2)。基于苹果果实发育的生理和形态研究[17,19]选取8个时间点进行采样(图1).第一个样本是在开花后0天(DAA),同时对完全开放的花进行标记。14和25 DAA采样时间点与授粉后发生的细胞分裂时间相吻合。在35daa时,细胞分裂停止,细胞扩张速度加快,淀粉开始积累。60 DAA与细胞扩张和淀粉积累的最快速度相吻合。87 DAA时,细胞膨胀率下降,但细胞膨胀率继续降低,直到完全成熟,淀粉水平在此时间点后不久达到峰值。在采集样品的年份,收获成熟度为132 DAA,在这个阶段,淀粉水平迅速下降,果糖增加,皮肤颜色仍在变化,虽然一些风味化合物存在,但尚未形成完全的“苹果味”。到146 DAA时,果实已经“树熟”,在这一阶段,果实有强烈的颜色和充分发展的风味,几乎所有的淀粉都转化为水果糖,一些果肉软化发生。虽然在完全开花之前发生的发育事件在导致最终结果的发育过程中是重要的,但在这项工作中没有考虑完全开花之前的样本。从两个重复的样本中提取RNA,标记并杂交到由15145个unigenes设计的15726个寡核苷酸(45-55碱基长)阵列,这些寡核苷酸代表大约13000个基因。 All samples were compared (using a dye swap design) to genomic DNA (gDNA) as a common reference, making samples directly comparable, the absolute expression of all the samples is shown in Additional file1.
在果实发育过程中,协调表达的基因主要有四组
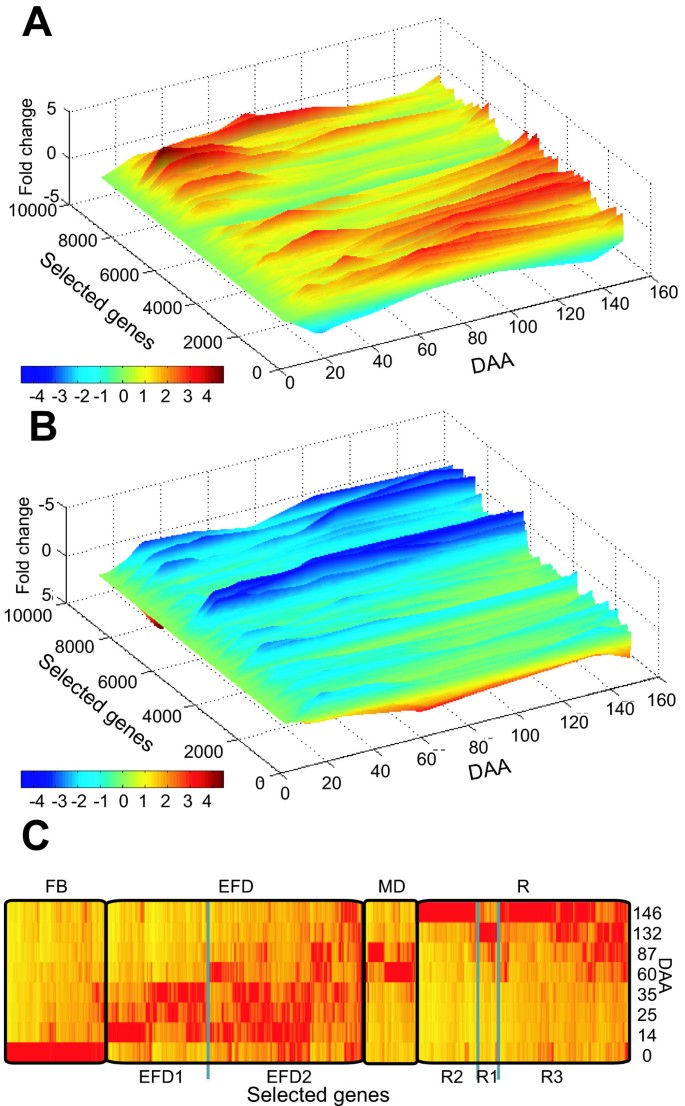
为了研究基因表达的整体变化,使用层次聚类方法对8719个在果实发育过程中表达变化的基因进行了分组(为了从表现出较小变化的基因中看到变化模式,排除了变化大于5倍的基因,这些基因的包含并不改变大多数基因的表达模式),并通过绘制三维空间的表达图进行了可视化(图2)2而且2 b).这种微阵列的全局分析显示了协调基因表达的四种主要模式。一组基因在花芽中表达,但在果实发育过程中下调,另一组基因在发育早期上调,随后下调,另外两组基因在发育中期和成熟期间上调。与番茄的结果相比[13],成熟时的整体表达模式没有明显变化,但这种差异可能反映了采样的差异。
果实发育过程中改变基因的聚类.基因表达的聚类分析。A和B,对整个阵列的表达式模式进行聚类,然后在三维空间中绘制(MATLAB, version 6.0;Mathworks)。剔除无表达变化或表达变化大于5倍的基因,剩下8719个基因。y轴为折叠变化。C,通过方差分析(FDR = 0.01)筛选出的1955个发育调控基因按其几何均值聚类。垂直线表示每个EST在0 - 146 DAA之间观察到的转录水平,最低表达量(黄色),最高表达量(红色)。主要簇有:花蕾或盛开(FB);早期果实发育(EFD); mid-development (MD); and ripening (R). The EFD and R clusters were further sub-clustered and indicated by EFD1, EFD2, R1, R2 and R3.
为了识别那些显著改变表达的基因,对整个数据集应用了单向方差分析(模型y =时间)。使用非自适应错误发现率(FDR)控制[26]的0.01,鉴定出1986个特征(对应1955个基因),在果实发育过程中基因表达发生显著变化。在果实发育过程中,层次聚类鉴定出四组表达模式相似的基因(图2摄氏度,和附加文件1,其中列出了整个数据集)。开花(FB)簇包含314个基因(315个特征),在0 DAA时高表达,其余时间低表达。早期果实发育(EFD)集群包含814个基因(819个特征),表达峰值在14 ~ 35 DAA之间。EFD簇由两个较弱的亚簇组成:EFD1,一组320个基因(326个特征),在发育早期高表达,然后在发育后期极低表达;EFD2是一组493个基因(493个特征),发育早期高表达,发育后期中表达。发育中期簇(MD)包含168个基因(169个特征),在60 DAA和87 DAA处表达高峰,其他发育阶段低表达。成熟簇(R)包含668个基因(681个特征),在果实发育后期表达低,最终达到峰值。R聚类可分为3个子聚类:R1 70个基因(70个特征)在收获成熟时表达最高(132 DAA),在其他发育阶段表达较低;R2 191基因(195个特征),在整个发育过程中表达量非常低,直到树成熟(146 DAA);r3406基因(408个特征)在树成熟时达到峰值(146 DAA),但在发育早期也有部分表达。 Both approaches to clustering identified four major groups of co-ordinately expressed genes suggesting these correspond to major phases of fruit development.
定量RT-PCR验证芯片表达
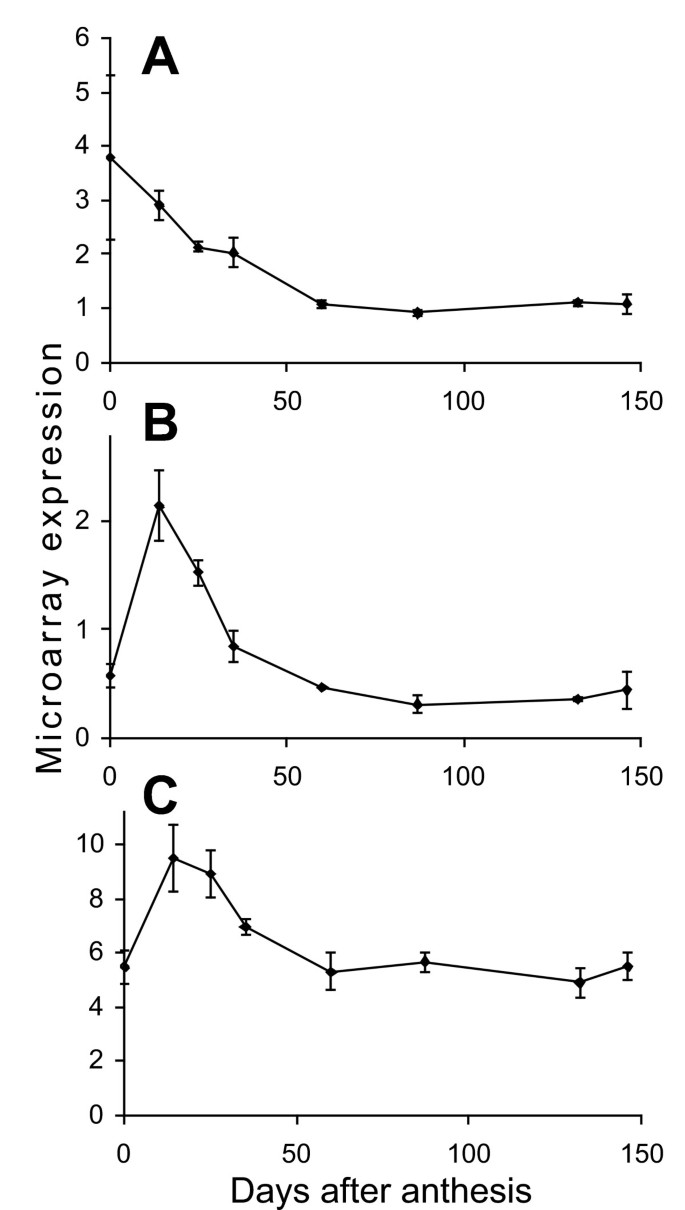
为了检验从微阵列中鉴定出的基因表达模式的可靠性,我们使用定量逆转录pcr (qRT-PCR)来检测果实发育过程中的稳态RNA水平。用于qRT-PCR的基因最初是从果实发育过程中表达发生显著变化的基因列表中选择的。调控基因的列表从最显著到最不显著依次排列,qRT-PCR基因从该列表中定期选择(大约每50个th基因)。还选择了几个基因进行qRT-PCR,以确认特定通路中的基因表达模式(见下文)。qRT-PCR结果采用3个内务基因进行归一化:肌动蛋白基因(Genbank accession CN927806);一个GAPDH基因(Genbank accession CN929227)和一个功能未知的基因(Genbank accession CN908822)。qRT-PCR表达谱与微阵列表达谱进行比较(图3.),如果他们在所有发育阶段都一致,或者如果大多数阶段一致,表达上的重大变化也一致,则被评为匹配。根据这些标准,74%(35个中的26个)的基因在微阵列实验中具有与qRT-PCR实验相同的表达模式。有趣的是,没有观察到表达模式的再现性和由方差分析确定的微阵列数据的显著性之间的关系。
在果实发育过程中,不同功能类的基因在不同时间表达
为了研究在果实发育过程中发生的基因功能变化,使用慕尼黑蛋白质序列信息中心(MIPS)定义的拟南芥蛋白质功能分类[funcat-1.3方案],确定了苹果基因的功能类。27])。对于阵列中所代表的苹果基因,根据BLAST分析选择序列相似度最高的拟南芥基因[28],阈值期望值为1 × e5,以及该拟南芥基因归属于苹果基因的MIPS功能分类。选择这个相对不严格的阈值是为了对阵列上的大多数苹果基因进行功能分类。表格1显示了苹果基因的数量,与拟南芥匹配的基因的数量,与唯一拟南芥基因匹配的数量,以及整个阵列的MIPS功能类别的数量,显示了1986年选择的特征在果实发育过程中发生变化,显示了集群和子集群。
整个数组的功能类别分布如表所示2并与1955个在果实发育过程中发生显著变化的基因的分布进行了比较。在果实发育过程中,MIPS功能类别的分布在整个阵列和所选基因之间发生变化,表明所选基因不是从整个阵列中随机选择的。例如,在果实发育基因中,与代谢相关的基因似乎有更高的代表性(20.3% vs 16.1%),这表明发育中的果实代谢更活跃。有趣的是,在所选的果实发育基因中,未分类类别略有增加(16.7% vs 15.7%),而在成熟聚类中,未分类类别的比例低于其他聚类(15.2% vs 17.4 - 17.8%),这可能反映了与果实发育早期事件相比,专注于识别和表征成熟后期基因的研究数量。
在四个主要聚类中,在果实发育中期达到峰值表达的基因与代谢相关的基因的表达减少(17.2% vs 20.1 - 21.5%),这表明这一阶段的果实发育可能代谢活性较低或使用的不同代谢基因较少。相比之下,细胞运输和运输机制功能在果实发育中期集群(2.6% vs 1.6 - 1.8%)中表现得更为突出,此时水果吸收营养和水分的速度最快。
对细胞组织功能的控制更多地体现在EFD和MD簇中(3.8%和4.6% vs FB2.7%和R2.4%),这与这一时期是果实发育阶段,果实细胞结构发生快速变化一致。在成熟簇中,“能量”类基因的比例过高(4.5%),而在发育中期的比例最低(2.1%)。此外,R2(树成熟时的峰值表达)亚簇在“代谢”类别中(25.4% vs 21.7和18.4%)的比例过高(与其他成熟亚簇R1和R3相比),与晚熟期间的能量和代谢变化相关。
值得注意的一个特征是EFD聚类中具有细胞周期分类的基因比例较高(FB 1.8%, EFD 3.4%, MD 1.4%, R 1.9%)。EFD簇包含的基因在果实发育的前30天表达达到峰值,即细胞分裂的发育阶段。17,18].这一发育阶段包括特定细胞的分裂,形成最终的苹果果实形状,由于在此期间细胞周期相关基因似乎增加,我们确定了每个簇的细胞周期分类相关基因(FB 17基因,EFD 61基因,MD 8基因,R 42基因)及其注释(表2)3.).这些列表很可能包括那些在调节水果大小和形状方面很重要的基因。例如,对这些列表的分析确定了三个核心细胞周期基因(见下文),这将是未来研究的重点。
核心细胞周期基因的表达
从形态学研究来看,苹果果实细胞在授粉后的前30天内至少经历四轮细胞分裂,细胞总数增加10倍[17,18].在30 DAA左右,构成成熟果实核心和皮层的细胞停止分裂,细胞扩张速度加快。细胞分裂和细胞扩张的控制是果实发育调控的关键部分,可能会影响最终果实的大小、质地和组织类型之间的平衡。
利用拟南芥基因组序列分析,Vanderpoele等[29]鉴定出61个核心细胞周期基因;这个列表已经扩展到88个基因,包括几个以前不认识的基因群[30.].拟南芥中的表达分析表明,许多这些核心细胞周期基因调控了稳态RNA水平[30.].为了确定这些核心细胞周期基因是否在果实发育过程中受到调控,我们鉴定了苹果的同源基因,并检测了它们的表达。由于水果样本来自多个水果,而且在一个水果中,细胞分裂不太可能同步,所以我们不期望能够检测到细胞周期中的表达变化。然而,任何发育变化的核心细胞周期基因都可能与果实形成和发育过程中细胞分裂率的控制有关。
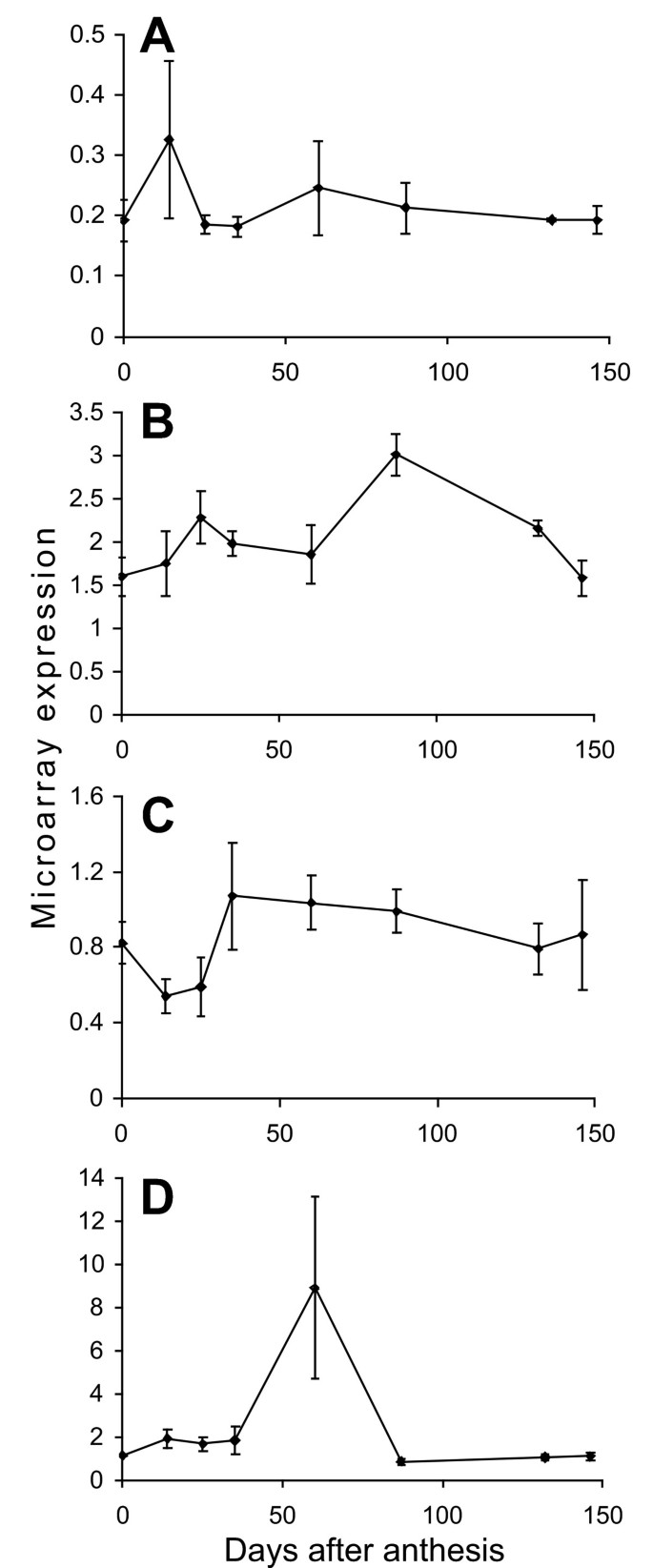
苹果阵列上的38个苹果基因与Menges等鉴定的88个拟南芥细胞周期基因具有很强的序列相似性。[30.],使用BLASTx和人工检查蛋白质序列比对(31个期望值为1 × e-40年或更好)。在这38个苹果基因中,通过方差分析选出的1955个基因中只有3个在果实发育过程中发生了显著变化(图4).ESTs 5126 (Genbank acc.;EB107042), 163128 (Genbank acc。CN943384)和173799 (Genbank acc。EB141951)均在发育早期具有较高的表达水平,但在35 DAA后下降到相对较低的水平。这3个基因与拟南芥基因At2g38620.1、At1g20930.1和At2g27960具有序列相似性(期望值为1 × e-146年, 1 × e-150年6 × e-37年分别)。At2G38620.1是CDKB1;2同源物,At1G20930.1是CDKB2;2同源物,At2g27960是CKS1同源物,这两个CDKB基因在细胞周期的进展中起作用,而CKS基因是有丝分裂特异性的支架蛋白。在这样的序列相似性水平上,不可能确定苹果基因是否代表这些基因的同源,尽管功能相似性是可能的。
淀粉代谢相关基因的表达
苹果果实的淀粉代谢是一个发育规律明确的生理过程[19].然而,在植物中调节淀粉水平的机制是复杂的,很少有人知道淀粉合成和降解酶的活性和周转是如何在水果等贮藏组织中被介导的(由Smith等人综述。[31])。为了研究苹果果实中淀粉代谢酶在转录水平上是否存在某种调控,我们检测了几种参与淀粉代谢的酶的表达模式。从京都基因和基因组百科全书(KEGG)数据库中的淀粉和蔗糖代谢途径中鉴定出参与淀粉转化的拟南芥酶[32].苹果基因与拟南芥淀粉转化基因序列相似性显著(BLAST显著性优于1 × e-100年)均纳入分析(表4).
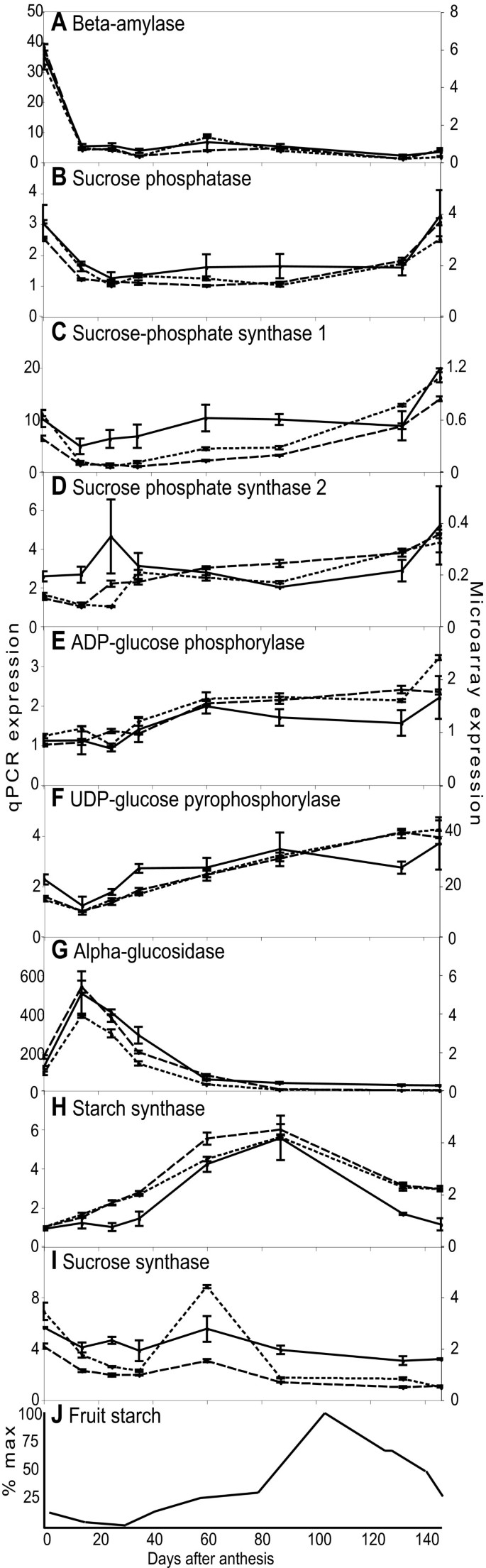
在苹果果实发育过程中具有恒定表达,因此在这一发育过程中不具有转录调控作用的基因没有进一步研究。那些低表达的也被排除在外,因为在微阵列上观察到的目标具有低信号强度的高可变性。α-淀粉酶是酶的一个例子,其检测到的转录水平低于切断值,因此没有进一步分析。总的来说,15个与淀粉代谢酶同源的苹果基因的ESTs被鉴定为在果实发育过程中变化的微阵列表达谱(表2)4),进行qRT-PCR鉴定。对于15种酶中的9种,qRT-PCR分析产生的表达谱强烈支持微阵列数据中看到的模式(图5).对于其余6种酶,qRT-PCR模式与微阵列模式不同,可能是因为RT-PCR引物扩增的等位基因或基因与微阵列寡聚物检测到的不同。
结果表明:1)β-淀粉酶基因(EB114557)在开花期转录水平较高,在果实发育的其余阶段转录水平较低,蔗糖合成酶(CN897963)表达模式相似,但表达下降速度较慢;II)蔗糖磷酸酶(EB156512)和蔗糖-磷酸合成酶基因(EB123469)的转录水平在最早和最晚的时间点达到峰值;III) adp -葡萄糖磷酸化酶(CN884033)和udp -葡萄糖焦磷酸化酶(EG631379)转录水平在果芽中最低,在果实发育过程中升高,在树熟苹果中最高;IV) α-葡萄糖苷酶(EE663791)和淀粉合成酶(EB121923)转录水平在苹果发育早期和后期均较低,在苹果发育早期和中期均达到峰值。
微阵列数据可以潜在地用于识别与协调表达途径,如淀粉代谢相关的调节基因。蔗糖磷酸酶和蔗糖-磷酸合酶图谱的相似性(图5)表示表达的协调。通过聚类分析,鉴定出Myb单结构域转录因子(EB129522),其表达模式与蔗糖磷酸酶和蔗糖-磷酸合成酶相似。瞬时表达的初步研究烟草benthamiana仅使用Myb基因,叶片没有显示两个淀粉代谢基因启动子区域的激活(数据未显示)。使用更大的启动子区域和Myb蛋白可能的结合伙伴进行进一步分析,可能会确定该基因的调节作用。
苹果果实发育候选基因的表达
虽然拟南芥不产生大的肉质果实,并且子实体的授粉后发育有限,但由于具有优良的遗传资源和基因组工具(如完整的基因组序列和全基因组微阵列),可以识别许多参与花和果实发育的重要基因。花器官的发育和受精前成熟心皮产生的相关基因已成为多项研究的主题[33].拟南芥果实授粉后的发育是有限的,虽然它可以作为开裂果实的良好模型,但尚不清楚参与拟南芥果实发育的基因是否对肉质果实的发育重要。尽管有这种保留,转录因子的重要性,如无性生殖的,富有成果的,AGL1 / AGL5,抹刀,蟹爪,ettin心皮特性和角果发育的规范表明,诸如此类的转录因子可能在肉质果实的发育中发挥重要作用[33].BLAST搜索在苹果基因微阵列上发现了寡核苷酸抹刀同系物(At4g36930, apple EST289091 Genbank acc EB132541,期望值8 × e-41年);ettin / ARF3(At2g33860,苹果EST250932, Genbank acc CN911459,期望值1 × e-163年);一个卓有成效的/ AGL8同系物(At5g60910, apple EST158712, Genbank acc EE663894,期望值7 × e-60年)和a蟹爪同源的(最同源于yabby5At2g26580,苹果EST111296, Genbank acc EB124712,期望值3 × e-42年),并绘制了这些基因的表达模式(图6).的表达式富有成果的/AGL8同系物(图6摄氏度),与AP1的相似度较高富有成果的,在苹果果实增大时增加(在细胞分裂时下降),这是有趣的,因为苹果的茎短而紧凑富有成果的突变体。
苹果和番茄果实发育的比较
阿尔巴等人最近的一项研究[13]使用了代表约8500个番茄基因的12899个EST克隆阵列来检测果实的发育和成熟,特别关注成熟前后发生的事件。虽然这项研究不包括花芽或番茄发育阶段,细胞分裂最活跃的阶段,但它是迄今为止最完整的水果发育数据集。为了识别与苹果和番茄果实发育有关的基因,我们使用了在番茄果实发育过程中发生变化的基因列表,在我们的芯片上找到了苹果的基因。
使用MegaBLAST(单词大小为12,阈值为1 × e5) Alba等人在番茄果实发育过程中改变的869个基因列表。[13]被用来鉴定在本工作中使用的阵列中存在的同源苹果基因。根据这些标准,336个番茄基因与479个苹果基因具有同源性。在这些苹果基因中,有102个被鉴定为在苹果果实发育过程中有显著的表达变化,因此在苹果和番茄中都受到转录调控。我们进一步筛选列表,只包括苹果EFD(41个基因),MD(16个基因)和R(35个基因)集群中的基因(表2)5).在FB簇中,另外10个苹果基因也被鉴定为与发育调控的番茄基因同源,但由于番茄微阵列不包括花芽样本,因此没有进一步检查。
来自苹果和番茄微阵列的表达数据被绘制为几个被识别的基因。绘制了苹果和番茄BLAST匹配质量在每个聚类中的前5个基因。还绘制了可能参与果实早期发育、中期发育和成熟过程的几个基因。由于微阵列有可能在没有先验信息的情况下识别参与过程的基因,所有没有注释的基因也被绘制出来。
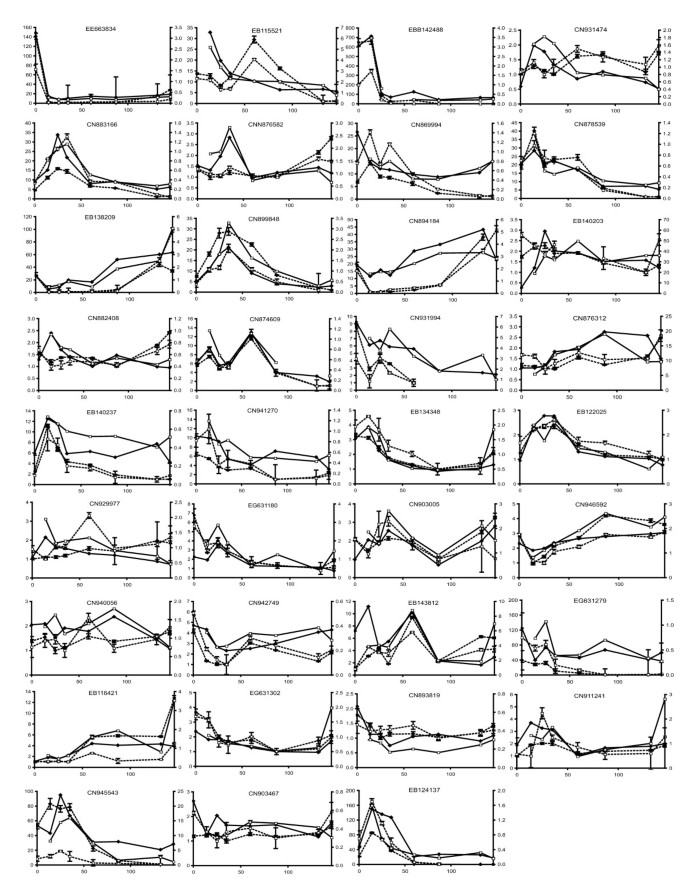
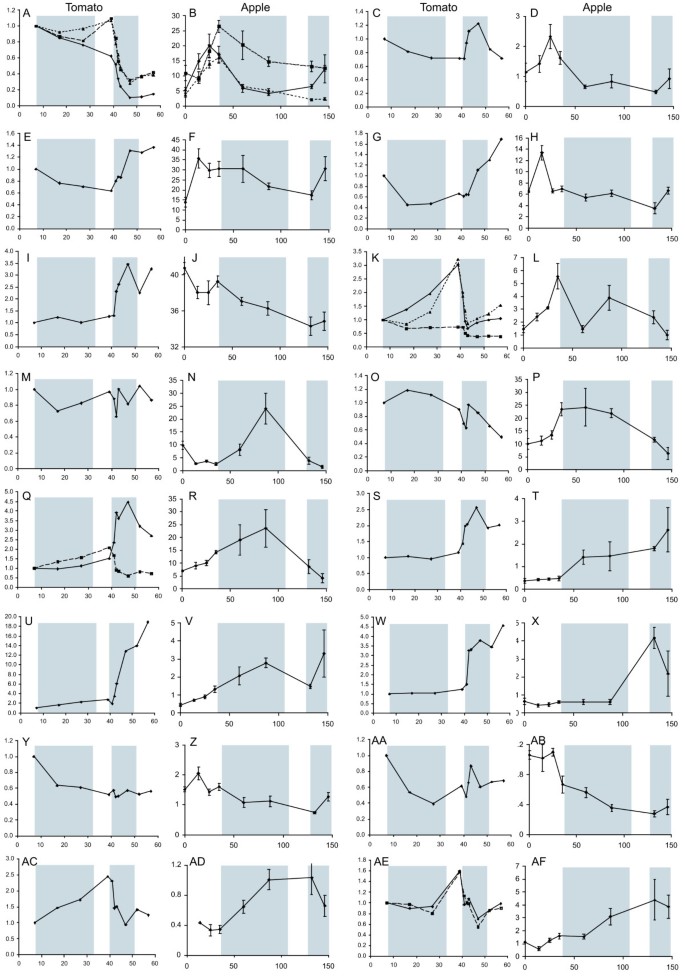
苹果和番茄果实的发育,从开花到成熟的长度不同,但我们比较了相似发育阶段的表达模式,特别是苹果和番茄细胞扩张的中期(番茄~ 8-35 DAA,苹果~ 40-110 DAA)和成熟阶段(番茄~ 40-50 DAA,苹果~ 130-150 DAA)。在47个比较表达模式的基因中,有16个在苹果和番茄中都有相似的表达模式,如图所示7,以细胞扩张和成熟阶段为重点。另外5个基因有一定的表达相似性,但26个基因表达相似性很小或没有(数据未显示)。
苹果和番茄表达的比较.通过序列比较,发现番茄和苹果基因在果实发育过程中表达变化,且相似。番茄基因相对于7 DAA的表达量,苹果为绝对表达量;x轴表示开花后的天数。每个图中的阴影区域都对应着番茄和苹果的细胞扩张和成熟的时期。A, C, E, G, I, K, M, O, Q, S, U, W, Y, AA, AC, AE番茄基因B, D, F, H, J, L, N, P, R, T, V, X, Z, AB, AD, AF苹果基因。A和B,微管蛋白同源物;C和D, IPP异构酶同源物;E和F,过氧化氢酶同系物;G和H,组蛋白2B同源物; I and J, MADS box (RIN) homologues; K and L, SAM synthase homologues; M and N, PPIase homologues; O and P, plasma membrane protein; Q, and R, α-expansin homologues; S and T, β-carotene hydroxylase homologues; U and V, Alcohol dehydrogenase homologues; W and X Phytoene synthase homologues; Y to AF Unannotated proteins. A, solid line SGN-U313081, dashed line SGN-U312305, dotted line SGN-U312306; B, solid line CN949202, dashed line EB115858, dotted line CN898685; C, SGN-U334957; D, EG631180; E, SGN-U313439; F, CN929316; G, SGN-U315396; H, CN897140; I SGN-U314473; J, EB176490; K, solid line SGN-U312527, dashed line SGN-U312579, dotted line SGN-U313529; L, EB130137; M, SGN-U313283; N, EB109090; O, SGN-U312814; P, CN943669; Q, solid line SGN-U312953, dashed line SGN-U333609; R, EB129432; S, SGN-U334905; T, EB130234; U, SGN-U314358; V, CN915191; W, SGN-U314887; X, EB144737; Y, SGN-U317999; Z, CN945062; AA, SGN-U313570; AB, CN909757; AC, SGN-U318901; AD, CN876487; AE, solid line SGN-U314449, dashed line SGN-U314453; AF, CN902741.
对于微管蛋白等基因(图7一个而且7 b), SAM合成酶(图7 k而且7 l)和一个膨胀蛋白同系物(图7问而且7 r)不止一个番茄序列与一个苹果基因同源,微管蛋白基因与三个苹果基因同源。对于微管蛋白基因,苹果和番茄的表达模式大多不同,但番茄的一个基因在细胞扩张过程中表现出稳定的表达下降,类似于苹果的基因。在三个番茄SAM合成酶基因中,只有一个(SGN-U312579)的表达模式与苹果基因相似,这表明该番茄基因可能在苹果和番茄中具有相似的功能。对于两个与苹果相似的番茄膨胀素同源基因,SGN-U312953在成熟过程中表达增加,而SGN-U333609和苹果膨胀素同源基因在细胞膨胀过程中表达增加,在成熟过程中表达下降,这表明这两个基因可能是同源基因,在细胞膨胀过程中起作用,而不是在果实软化过程中起作用。4个未标注的基因在苹果和番茄果实中具有相似的表达模式。进一步的生物信息学分析表明,CN945062可能是参与光合作用的PGR5同源蛋白,CN909757可能是F-box蛋白,在细胞扩张过程中表达的CN876487与Sec5A相似,可能参与胞吐作用。然而CN902741仍然没有注释。这些基因在果实发育中的作用仍有待确定。
苹果品种间基因表达的比较
最近的一份报告使用3484个cdna阵列检测了苹果果实发育早期基因的表达[25].这些作者在富士品种中鉴定出88种独特的苹果基因,在整个年轻果实(21 DAA)中比在整个成熟果实(175 DAA)中表达更多。在我们的EST数据库中鉴定出84个同源基因,其中42个在我们的微阵列上显示。在这42个基因中,有17个被选为在果实发育过程中发生显著变化的基因,其中13个在EFD集群中,4个在MD集群中6).
由于用于选择显著变化基因的标准相当严格,我们绘制了我们的数据与Lee等人选择的早期果实发育基因之间所有匹配的表达模式。[25]以识别任何其他具有相似表达模式的基因(数据未显示)。Lee等人鉴定出的18种基因[25]在我们的微阵列中没有得到证实,然而,另外13个基因在皇家加拉果实发育早期高表达,在成熟过程中低表达(表2)6).
乙烯反应果实发育基因的鉴定
乙烯激素在包括苹果在内的许多水果的成熟过程中起着重要作用,导致呼吸破裂和最终的果实软化[20.,21,34,35].最近的研究使用了一种转基因苹果树(表达ACC氧化酶基因的反义副本),它不会产生可检测的乙烯,以检测与苹果香气相关的基因表达变化和挥发性化合物的产生[22].这种树的果实成熟,但不会成熟或软化,除非用外源乙烯处理。Schaffer等人。[22]使用这里描述的苹果寡核苷酸阵列鉴定了944个对乙烯有反应的苹果皮层和皮肤基因。由于果实发育实验中的成熟果实样品仅由皮层组织组成,我们只鉴定了那些在皮层中变化至少2倍的基因(排除了25个表达极低的基因),从而列出了456个对果实皮层中乙烯反应强烈的基因。在这456个乙烯反应基因中,106个在正常果实发育的成熟阶段也发生了显著变化。这些乙烯响应型果实皮质成熟基因见表7并按成熟子簇进行分组。基因在三个簇之间的分布不均匀,R2簇中乙烯反应基因的比例更高(R1中70个基因中有10个(14.3%),R2中195个基因中有48个(24.6%),R3中408个基因中有48个(11.8%))。在这些基因中,有一个基因被鉴定为乙烯受体,与拟南芥中的ETR2/EIN4受体最为相似(apple EST166801, Genbank acc。EE663937)。苹果微阵列还包含四个额外假定的乙烯受体基因的寡核苷酸探针。在乙烯微阵列实验或正常果实成熟过程中,其中三个基因的表达没有显著变化(苹果EST152541, Genbank acc。CN898978,苹果EST248756, Genbank acc。CN910963,苹果EST244637, Genbank acc。CN902679)。第四个基因(苹果EST166743, Genbank acc。 EE663931, most similar to the ERS1/ETR1 receptors from Arabidopsis) was selected as induced by ethylene, and although it was not selected as significantly changing during fruit development, it does show some induction in normal fruit ripening.
讨论
通过qRT-PCR确定微阵列表达模式
在用于产生微阵列数据的每个步骤中,都可能引入可变性,导致潜在的错误。我们使用来自微阵列实验中使用的RNA样本的cDNA qRTPCR来估计我们数据的总体准确性。总体而言,我们发现qRT-PCR和微阵列结果之间有良好的相关性,通过qRT-PCR可重复75%的微阵列表达模式。然而,qRT-PCR结果中有25%的表达模式与微阵列结果不匹配。在某些情况下(~5%),这种差异似乎与基因组DNA参考样本具有非常高强度结合的基因有关。这种高水平的gDNA结合可能扭曲了所观察到的比率,或者gDNA结合可能干扰了这些基因的cDNA结合。qRT-PCR结果与微阵列结果不一致的另一个可能的解释是,微阵列上的寡核苷酸能够与样品中一个基因的多个等位基因杂交,并且qRT-PCR引物结合更具特异性。或者,该微阵列寡核苷酸可与一个基因家族的多个成员杂交。这些结果表明微阵列上的杂交条件不够严格,然而在方法的初始优化过程中,任何严格的增加都会导致信号强度的巨大损失(数据未显示)。此外,我们在阵列上有大约20个寡核苷酸被设计用于EST序列,当重新测序时,这些寡核苷酸与一致序列有一个碱基不匹配,这些寡核苷酸不绑定标记的目标,而完全匹配的寡核苷酸与相同的目标确实产生良好的信号(数据未显示),这表明杂交严格性接近最优。
在果实生长过程中,不同功能类的基因在不同时间表达
将整个序列与选定的1955个基因进行比较,发现功能类别的分布存在差异,表明从整个序列中选择的变化显著的基因是非随机选择的。考虑到器官发育、淀粉和糖的积累以及后期成熟和风味化合物的产生发生了巨大变化,与整个系列相比,“代谢”和“能量”类的增加并非不合理。其余的函数类显示整个数组与选定的1955之间只有微小的差异,这可能反映了EST序列中的一些偏差[24].由于原始EST测序中使用的大多数文库来自水果或花蕾,因此有理由期望整个序列的功能分类与果实发育中调控基因的分类相似。
当比较四个主要簇的功能分类时,可以观察到每个类别中基因比例的一些有趣变化,尽管必须谨慎地解释这些变化,因为每个簇代表一组不同的基因。代谢和能量基因功能在芽中所占比例较高,在发育过程中下降,在成熟果实中上升。这种后期增加可能反映了次级代谢产物基因表达的增加,因为在成熟过程中产生了风味化合物。当更详细地检查功能类别时,可以看到这一点。虽然FB和成熟簇的总体“代谢”分类相似(21.5% vs 20.9%),但包括萜类等已知风味成分在内的脂类、脂肪酸和类异戊二烯代谢的MIPS分类01.06从FB的2.6%转变为成熟簇的4.3%(数据未显示)。
功能分析的一个局限性是它只能提供关于某些功能已被预先确定的基因的信息。虽然基因功能分类是微阵列分析的一种有用方法,但将功能分类与其他方法(例如聚类)相结合,可以更容易地在数据中识别信息,例如识别与细胞分裂相关的基因,这些基因在发育早期表达最高。
在果实发育早期,细胞周期基因在转录水平上受到调控
苹果果实的发育包括授粉后大约30天的早期细胞分裂[17,18].细胞周期基因的调控是复杂的,但在果实发育过程中,一些核心细胞周期基因的转录调控可能参与了细胞分裂的控制。在同步拟南芥和烟草BY2细胞培养中,在转录水平上控制核心植物细胞周期基因与调节细胞周期有关[30.,36- - - - - -38].由于我们样本的性质,我们将无法检测到这种周期依赖的转录调控。然而,至少有一个核心细胞周期基因已被证明在植物发育中受到调控;CDKB1;1与拟南芥叶片发育中细胞分裂的控制有关,且CDKB1;1的表达随着拟南芥叶片的衰老而下降[39,40].CDKB1;1修饰形式的表达改变叶片中的CDKB1;1活性,改变细胞大小和核内重复。苹果果实发育芯片中两个假定的CDKB同源基因发生了显著变化,这两个苹果基因在苹果细胞分裂停止时表达量下降,这表明这些基因在调控这一过程中发挥了作用。在果实发育过程中发生显著变化的第三个核心细胞周期基因是CKS1同源基因。CKS1已被证明与CDKB蛋白相关,并被提议作为CDK活性调节的对接蛋白[41]也已被证明与参与激酶抑制剂蛋白降解的SCF复合体有关(动物中的KIPs,植物中的KRPs, [41,42])。这三种细胞周期相关基因在苹果果实细胞分裂时的表达表明它们是苹果重要的发育调节因子。改变这些基因的表达将有助于阐明它们的功能,并可能导致改变细胞数量的水果,从而改变水果的质地和大小。
G1向S的转变是植物细胞周期中的一个重要控制点,CycD3;1基因已被证明在拟南芥中限制了这一转变[43].该基因在苹果中尚未发现同源基因,但在该序列中有3个同源基因。这些同源基因在苹果果实发育过程中都没有显著变化,但其中一个(EB132575)在苹果果实发育后期下降了大约2倍。
在许多植物中,核内再复制与细胞大小的增加有关[44].拟南芥的研究表明激酶抑制剂KRP1对有丝分裂CDK复合物的抑制作用[45]及KRP2 [46和激酶Wee1 [47会导致核内重复增加。有趣的是,最近的一份报告表明,成熟的苹果果实中没有核内再复制[48].也许并不奇怪,这些基因的苹果同源基因在果实发育过程中没有发生显著变化,但是苹果Wee1同源基因在细胞分裂停止后确实表现出一些表达的增加。这些基因在苹果核内重复调控中的作用尚不清楚,但有可能通过改变这些基因的表达来诱导苹果核内重复。
果实淀粉代谢在转录水平上受到调控
虽然许多淀粉酶的生化活性已经被确定,但很难确定不同的酶通路在水果淀粉水平调节中的作用。将本研究中产生的基因表达谱与苹果发育过程中淀粉含量的已知变化相匹配是一种方法,涉及这些过程中的某些途径。虽然我们没有观察到完整通路的协调表达,但在一条通路中存在多个基因的共表达。例如,蔗糖磷酸酶(EB156512)和蔗糖-磷酸合成酶(EB123469)的表达谱反映了苹果在果实早期发育和成熟过程中淀粉含量的降低[19],表明这些酶可能是果实发育过程中淀粉降解途径的组成部分。然而,也有可能是不同的途径导致这些早期和晚期淀粉降解事件。β-淀粉酶(EB114557)和α-葡萄糖苷酶(EE663791)的高转录水平出现在发育早期,而不是在成熟过程中,这表明淀粉降解途径可能是早期发育特有的,而在发育后期不活跃。这些结果表明,不同的淀粉代谢途径在苹果果实发育中起重要作用,并在转录水平上受到调控。
在分析淀粉代谢途径的过程中观察到,对于任何给定的步骤,通常都有几个特定酶的候选基因。例如,在拟南芥数据库中有两种可塑性淀粉合成酶。两者在苹果EST数据库中都有同源基因,其中一个与两个苹果基因同源。这些候选淀粉合酶基因在苹果果实中的表达(以EB121923为代表,图5)的峰值为87 DAA,略早于果实淀粉含量的峰值(100 DAA) [19].这种表达数据与发育过程中淀粉积累模式的相关性表明,这种特殊的淀粉合酶基因参与了果实发育过程中淀粉水平的调节。这些结果表明,微阵列可以用来将转录水平与生理和生化观察相关联,以确定一个基因家族的哪个成员,甚至可能是哪个等位基因,可能参与了感兴趣的过程。
9种淀粉酶的表达谱(图5)表明,这些基因转录的发育调控与观察到的苹果发育过程中淀粉水平的变化相对应。在类似的研究中,Smith等人。[49]利用Affymetrix微阵列技术观察了拟南芥叶片中淀粉酶ε表达在一个昼夜周期内的变化。在叶片中,淀粉在光照下合成,在黑暗中降解。这些作者观察到淀粉合酶和β-淀粉酶等酶的转录水平有明显的变化。有趣的是,有证据表明,在拟南芥叶片中,淀粉的转录调节发生在24小时内,而在苹果果实中,淀粉的发育调节发生在146天内。这种源组织和库组织中淀粉的转录调节可能是协调整个植物碳水化合物分配所必需的。
果实发育微阵列实验的比较
针对同一发育过程的不同物种的微阵列实验的比较,为比较大量基因的基因表达模式提供了机会。这种比较的吸引力在于,它可以确定不同水果的共同过程,因此在所有水果的基本过程中都很重要。然而,对于一些已发表的研究,数据集的大小和/或所研究样本的差异使比较值有限[11,15,25].例如,lemire - chamley等人给出的番茄微阵列结果的特定搜索[11]在苹果和番茄中均有表达的基因[13]在果实发育早期没有发现相似的基因,可能是因为这些基因不包括在1393个番茄cdna的lemire - chamley阵列中,例如IPP异构酶同源物(SGN-U334957和EG631180),过氧化氢酶同源物(SGN-U313439和CN929316)和组蛋白2B同源物(SGN-U315396和CN897140)。苹果微阵列发现CDKB2基因在果实发育(分裂细胞)早期上调,番茄房室(扩张细胞)和番茄果皮(分裂细胞)的比较发现CDKB2;2同源物在房室组织中上调[11].
一项使用苹果(3484个cdna, 'Fuji')的微阵列实验比较了21 DAA与完全成熟的水果[25].将我们的数据与Lee等人的数据进行比较。25]可以识别那些在两个实验中没有显著变化而被排除在外的调控基因。其中一个α-膨胀蛋白同源基因EB129884,在富士微阵列的21个DAA水果中被鉴定为高表达,由于两个样品(132和146 DAA)没有检测到表达,通过方差分析被排除在Royal Gala微阵列中。在Royal Gala微阵列中,该α-扩张蛋白在14 DAA时表达最强,并保持表达至87 DAA,然后无表达检测,使其成为参与果实细胞形成和扩张的扩张蛋白的良好候选。如果没有与富士微阵列数据的比较分析,就无法确定该基因。
Alba等利用含有12899个ESTs的芯片,代表了~8500个番茄基因。13]研究了番茄果实发育过程中的基因表达,主要集中在成熟过程中。番茄果实发育芯片和苹果数据之间只有102个相同基因,这可能令人惊讶。实验设计的差异可能是造成这种小重叠的原因之一,番茄微阵列在成熟前后有更多的采样,而苹果微阵列对花芽和早期果实发育有更多的采样。这也可能是苹果和番茄果实发育差异的一个指标。
当比较相似的苹果和番茄基因的表达模式时,研究的46个基因中只有16个在苹果和番茄中都有相似的表达模式。由于大约75%的苹果微阵列表达模式在qRT-PCR中是可重复的,想必番茄微阵列也是如此,对于每对基因来说,两种模式都可重复的概率只有大约56%。因此,我们最多只能期望26对基因具有相同的表达模式。此外,由于所使用的序列相似性阈值相当低,因此也可能有一些被检测的基因对不是同源基因。然而,仅仅在苹果和番茄中鉴定出16对具有相似表达模式的基因很可能低估了水果之间的实际相似性。如果苹果和番茄的表达模式确实有相似之处,那么微阵列的表达模式很可能代表了这些基因的实际表达模式,因为这种表达模式已经在另一个物种中得到了有效的证实。当使用更完整的全基因组阵列和进行更紧密匹配的采样时,很可能会识别出更多具有相似表达的基因。随着在其他果实物种中进行进一步的微阵列实验,将标准化发育阶段的样本纳入将允许更好地比较数据集,并确定更多共同的基本过程。
在鉴定的16对番茄和苹果基因中,7对在成熟过程中表达上调,4对表达下调。这几乎肯定反映了对番茄微阵列中成熟样品的重视。在苹果和番茄的成熟过程中,β-胡萝卜素羟化酶、乙醇脱氢酶和植物素合成酶的同源物都被上调,这表明这些酶在与果实成熟相关的颜色和味道化合物的形成中起着重要作用。然而,类胡萝卜素在苹果果肉中含量并不高[50这表明,要么苹果中类胡萝卜素的产生在生物合成途径的另一个步骤被阻断,要么这些酶的产物被进一步加工成尚未在苹果中测量到的形式。IPP异构酶、过氧化氢酶、组蛋白2B和RIN MADS-box基因的同源物都在苹果和番茄的成熟过程中上调,它们在苹果芯片中也都被选为在果实发育早期上调,尽管MADS-box基因的上调可能更多地与花芽中的高表达有关。这些基因早期表达的作用尚不确定,但看看它们是否也在番茄果实发育的早期高度表达将是有趣的。一个完整的质膜蛋白同源物和一个扩展蛋白同源物在苹果和番茄中表达模式相似,并在苹果芯片的中期发育簇中选择。这一结果表明,这两个基因在果实发育过程中对细胞扩张起着重要作用。我们还鉴定了在苹果和番茄果实中具有相似表达模式的基因,但没有注释。这样的比较是有价值的,以便找到其他方法可能无法识别的特定过程的功能保守的基因。进一步的研究将使我们能够确定这些基因是否确实在果实发育中发挥重要作用。
不同苹果微阵列实验的交叉点
对两个使用相同芯片的苹果实验进行比较,有助于鉴定与果实成熟和乙烯反应有关的基因。两个数据集的组合比单独的实验提供了更多的信息。乙烯在苹果果实成熟中的重要性可以从ACC氧化酶敲除果实的不成熟中得到证明。22].当我们比较乙烯诱导和果实发育芯片的数据集时,在发育芯片的成熟集群(668个基因)中发现了106个乙烯诱导基因(在皮层)。观察到350个乙烯诱导的基因在内源性成熟过程中没有被鉴定为表达改变,这意味着这些基因在正常的果实成熟中没有作用,或者这些基因的诱导低于本工作中用于选择基因的显著性水平。这些结果表明,虽然乙烯是果实成熟中基因表达的主要调节因子,但果实成熟的很大一部分是在没有乙烯的情况下发生的。使用这种比较方法,可以确定水果成熟事件既依赖乙烯又不依赖乙烯。
结论
本文提供的数据为苹果果实发育过程中发生的分子事件提供了一幅图景,并为今后的果实发育研究提供了资源。我们已经确定了可能在一些主要过程中起重要作用的基因。将苹果的数据与其他结果植物的数据进行比较,确定了16个可能在果实发育中发挥基本作用的基因。通过对苹果实验的比较,可以区分乙烯依赖型成熟和乙烯依赖型成熟。未来的工作将确定这些基因的具体功能。通过对发育早期果实组织中CDKB和CKS表达的功能分析,可以揭示果核周围皮层组织生长的控制机制。操纵这些基因的表达可能会改变水果中的细胞大小和数量,可能会影响水果的形状、大小和质地。这些数据使我们开始了解导致发育中的种子周围组织分裂和扩张以形成果实的分子事件。
方法
树木的生长和维护及取样
苹果马吕斯×有明显Borkh。也被称为m . pumila)来自“Royal Gala”的树木生长在M9砧木上,并根据标准果园实践进行管理(除了不允许发生化学疏果)。
对于0 DAA样品,芽被剥去花瓣和叶柄,但没有进一步解剖。对于在14、25、35和60 DAA时采集的样品,只去除叶柄,对整个水果进行采样。每个样本至少收集了10个完整的水果。对于87、132和146号采集的样品,DAA只从至少10个果实中分离出皮层组织并合并。所有样品在采收时在液氮中冷冻,然后在-70°C保存。
RNA提取
在液体N下从每个组织中提取6g总RNA2使用Chang等人的改进方法。51].在加入提取缓冲液后,用1分钟的多管步骤修改该方案,第一次氯仿提取后的水相通过高压灭菌的Mira布过滤,LiCl的总浓度为2 m。分离出的RNA进行柱状纯化(使用RNAeasy Mini Kit, Qiagen, Hilden, Germany),并使用Agilent 2100 Bioanalyser (Agilent, Palo Alto, CA)检查质量和纯度。用乙醇沉淀RNA,重悬至12.5 μg/μL。
阵列设计
如Newcomb等人所述,苹果est被分为非冗余序列和unigenes。[24].对于每个EST,使用内部算法设计寡核苷酸,Tm为74°C±2°C,长度在45到55个碱基之间。具有倒置或直接重复序列和超过5个相同核苷酸的寡核苷酸被淘汰。为每个单基因从最靠近该单基因3'末端的EST中选择一个低聚子,如果一个EST中有多个可能的低聚子,则选择3'最多的低聚子。作为最终的筛选标准,用BLAST方法对苹果unigenes数据库和拟南芥蛋白质数据库进行了比较。对于与其他苹果unigenes具有高度相似性的苹果unigenes,或两个苹果unigenes与同一拟南芥蛋白具有高度相似性的苹果unigenes,只需选择一个具有代表性的苹果unigenes进行寡核苷酸设计。根据这些标准,设计了15726个苹果寡核苷酸,与15145个苹果unigenes相对应1).苹果unigenes与拟南芥和其他植物的比较表明,该阵列包含大约13000个不同的基因。低聚寡核苷酸被商业合成(Operon合成5000个,Illumina合成10726个)。低聚物在150mm NaPO4 pH 8.5中重悬,含0.00001% SDS至最终浓度为20 μM,并使用MicroGrid TAS阵列使用16个微点2500引脚共32个块打印在环氧树脂阵列载玻片(Quantifoil)上。由于寡核苷酸是随机选择和合成的,没有额外的随机化阵列是必要的。
除了样本寡核苷酸外,每个区块还包含四种类型的对照寡核苷酸(表8).第1组,苹果寡核苷酸设计自:苹果肌动蛋白单基因的3'端、中间端和5'端(MdAC1-3, EST3793, Genbank acc。CN935584),一个苹果泛素单基因(MdAC4-6, EST14223, Genbank acc。苹果伸长因子-1 α基因(Md AC7-9, EST704, Genbank acc。CN934151);苹果rubsico小亚基中的寡核苷酸(MdAC10, EST 59854, CN862467);一个苹果同源盒子unigene, 5'端(MdAC11, EST87558, Genbank acc。CN870331), 3'端(MdAC12 EST29626, Genbank acc。EB111272)和保守域(MdAC13, EST29626, Genbank acc。EB111272); an apple MADS-box gene, 3' untranslated region (MdAC14, EST58802, Genbank acc. EB175510), 3' coding region (MdAC15, EST64768, Genbank acc. EB116541) and MADS domain (MdAC16, EST15992, Genbank acc. EB114519).
第2组,与转基因植物相关的对照寡核苷酸:苏云金杆菌cry1Ac (BtAC17, Genbank acc。U89872);链霉菌属hygroscopicus磷霉素乙酰转移酶(ShAC18, Genbank acc;X17220);GFP (Av AC19, Genbank acc。AF078810);智人血红蛋白HsAC20, Genbank acc;NM_000518);GUS (EcAC21, Genbank acc;A00196);大肠杆菌潮霉素B磷酸转移酶(EcAC22, Genbank acc;K01193);荧光素酶(PpAC23);X65316);新霉素磷酸转移酶(EcAC24, Genbank acc;V00618)。
第3组,来自人类基因的控制寡核苷酸,预计不会在植物中表达:b细胞受体蛋白(HsAC25, Genbank acc。AF126021);Mysoin重链(HsAC26, Genbank acc。X13988);肌凝蛋白reg。轻链2 (HsAC27, Genbank acc。M21812);胰岛素样生长因子(HsAC28);X07868);cDNA FLJ10917fis (HsAC29, Genbank acc.; AK001779); HSPC120 (HsAC30, Genbank acc. AF161469); β2 microglobulin (HsAC31, Genbank acc. NM_004048); Phosphoglycerate kinase (HsAC32, Genbank acc. NM_000291); Tyrosine phosphatase (HsAC33, Genbank acc. L11329); G10 homolog edg-2 (HsAC34, Genbank acc. U11861).
第4组,基于拟南芥阵列数据分析,对照寡核苷酸预计在所有组织中具有相同的表达水平:泛素蛋白连接酶(Aunc1, At1g14400, Genbank acc。T21817 ESTID103C16T7);未知蛋白(Aunc2, At1g16210, Genbank acc。T04357 ESTID39A2T7);磷酸酶(aun3, At1g43900, Genbank acc。H76500 ESTID196M23T7);未知蛋白(Aunc4, At3g13060, Genbank acc。NM_112143 ESTID127j5t7);蛋白酪氨酸磷酸酶(aun5, At3g19420, Genbank acc;NM_112829 ESTID122G23T7); translation initiation factor (Aunc6, At3g19760, Genbank acc. NM_112866.3, ESTID137B19T7); helicase (Aunc7, At4g00660, Genbank acc. NM_116291.4, ESTID221A7T7).
阵列样品的标记
使用寡聚t23v引物并加入氨基烯丙基脱氧尿苷(aadU, Sigma-Aldrich, Milwaulke, WI)对总RNA样本中的mRNA进行逆转录(RT)。每个RT反应含有50 μg总RNA和10单位转录逆转录酶(Transcriptor Reverse transcrip转录酶,Roche, Indianapolis, IN), 10 μM oligodT23V, 1× first strand buffer (Roche, Indianapolis, IN), 6.6 μM DTT,以及dA、dG、dC、dT和aadU各0.5 mM、0.25 mM的核苷酸,总体积为30 μL。RNA和寡核苷酸t23v在70°C下孵育10分钟,冷却到4°C 5分钟。加入第一链缓冲液、DTT、核苷酸和酶,在42°C下孵育30分钟。加入1 μL 20 mM EDTA停止反应,加入1 μL 500 mM NaOH降解RNA,样品加热到70°C 10 min,然后用1 μL 500 mM HCl中和。
使用核子提取和纯化试剂盒(GE Healthcare)从皇家加拉叶子中分离出苹果基因组DNA。叶子(1克)按照制造商的说明加工,并加入可选的添加β- marcapto乙醇的步骤,以限制酚类化合物的氧化。DNA在500 μL TE中重悬。DNA通过26.5口径的针头切割20次。
基因组DNA第一链标记使用含有Klenow DNA I聚合酶和随机八聚体引物的Radprime DNA标记试剂盒(Invitrogen, Carlsbad, CA)的组件。每个标记反应含有2.5 μg剪切的苹果基因组DNA和40 U Klenow,以及1× Radprime缓冲液(含引物)和0.12 mM dA、dG、dC和0.06 mM dT的核苷酸,aadU,总体积为50 μL。Radprime缓冲液和苹果基因组DNA加热到95°C 10分钟,冷却到4°C 5分钟。加入核苷酸和酶,在37°C孵育1小时。
cDNA和第一链gDNA用5 μL 100 mM Na乙醇沉淀后重悬2有限公司3.(pH值9.0)。在DMSO中加入5 μL Cy3或Cy5 (Amersham Biosciences, Buckinghamshire, England),室温黑暗孵育2小时。用10 μL 4 M羟胺(Sigma, St Louis, MO)淬灭未反应染料,室温黑暗孵育10 min。标记的DNA在PCR清理柱(Qiagen, Hilden, Germany)上纯化,配对样本合并。分别测定样品在260 nm、550 nm和650 nm处的吸光度,以确定标记DNA的数量和Cy3和Cy5的标记效率。聚合酶链反应净化柱在池化后重复纯化一次以减少背景荧光。
杂交
标记样品混合到Amersham Lucidea自动切片处理器中的微阵列切片。用2 × SSC、0.3% SDS预洗玻片。样品以3 × SSC、0.2% SDS、6%液体阻断剂(RPN3601, GE Healthcare, Chalfont St Giles, United Kingdom)一起注射入滑槽。加热至45°C并混合过夜。杂交完成后,用2 × SSC、0.3% SDS洗涤1.2 min,冷却至30℃,再用2 × SSC、0.3% SDS洗涤1.2 min、2 × SSC、0.3% SDS洗涤2.4 min、0.5 × SSC、0.2% SDS洗涤2.4 min(2次),0.5 × SSC洗涤1次。通过离心干燥载玻片,并在GenePix 4000 B扫描仪(Axon Instruments)上扫描。使用GenePix4 (Axon Instruments)获取阵列载片扫描的原始数据,并通过手动检查每张载片以去除不合格的点来增强自动点对准。
归一化和分析
所有分析均按照Schaffer等人的描述进行。[22],除了使用单向方差分析模型(y =时间)。使用非自适应错误发现率(FDR)对照,以0.01阈值检测显著差异表达基因的数量[26].每个基因的表达计算为所有时间点的两个生物重复的两次技术重复(染料交换)的平均值和标准误差(0 DAA除外,其中未采集Rep2样本)。
定量rt - pcr
qRT-PCR引物的设计尽可能重叠阵列上使用的低聚物的位置,并在阵列实验中使用的相同组织样本的RNA制成的cDNA上进行qRT-PCR。提取的阵列实验总RNA用于qRT-PCR。RNA用DNAase处理(使用Turbo DNAse试剂盒,Ambion, Austin, TX)。
为每个qRT-PCR候选基因和3个对照基因设计正向和反向引物。在可能的情况下,所有引物都设计为跨越寡核苷酸阵列,最佳温度为59℃,GC含量为40-60%,扩增子长度为100 bp,引物长度为20 bp。引物序列见表9.
对每个RNA样本进行三次独立的逆转录反应。所有反应均含有2 μg RNA, 2.5 μM oligo(dT)23 V引物,0.5 mM dNTP混合物,36.5 μL H2O.样品在65°C下孵育5分钟,在冰上孵育1分钟。加入1× 1股缓冲液,5 mM DTT, 200 Units of Superscript III RT (Invitrogen, Carlsbad, CA), 50℃孵育60 min, 70℃孵育15 min。将重复反应合并并稀释至15 ng/uL。
qrt - pcr在两个生物重复样品上进行,每个反应进行四重复。每个20 μL反应含有75 ng cDNA, 200 nM正向和反向引物和2× SYBR绿色主混合物(Invitrogen, Carlsbad, CA),扩增使用ABI PRISM 7900 HT序列检测系统(Applied Biosystems, Foster City, CA)。反应经历了94°C下2分钟的变性阶段,扩增40个周期(15秒94°C, 30秒59°C, 20秒72°C)和解离阶段(15秒95°C, 30秒60°C, 15秒95°C)。表达式量化和数据分析参照Snowden等方法[52].
参考文献
- 1.
以扫K:种子植物解剖第二版。第二版。圣巴巴拉,John Wiley & Sons Inc;1977年,
- 2.
王志刚,王志刚:《水果的发展视角》。中国生物医学工程学报,2003,26(3):357 - 357。10.1105 / tpc.5.10.1439。
- 3.
果树成熟与成熟的分子生物学研究。植物化学学报,2001,29(3):344 - 344。10.1146 / annurev.arplant.52.1.725。
- 4.
Giovannoni JJ:果实发育和成熟的基因调控。中国生物医学工程学报,2004,16增刊:S170-80。10.1105 / tpc.019158。
- 5.
Seymour GB, Taylor JE, Tucker GA:水果成熟的生物化学。伦敦,查普曼&霍尔;1993年,454 -
- 6.
Aharoni A, Keizer LC, Bouwmeester HJ, Sun Z, Alvarez-Huerta M, Verhoeven HA, Blaas J, van Houwelingen AM, De Vos RC, van der Voet H, Jansen RC, Guis M, Mol J, Davis RW, Schena M, van Tunen AJ, O'Connell AP:利用DNA微阵列鉴定草莓风味生物发生中的SAAT基因。植物学报,2000,12(5):647-662。10.1105 / tpc.12.5.647。
- 7.
Trainotti L, Pavanello A, Casadoro G:不同的乙烯受体在草莓成熟过程中表达增加:这种增加是否意味着乙烯在这些非更年期水果的成熟过程中起作用?应用物理学报,2005,26(3):344 - 344。10.1093 / jxb / eri202。
- 8.
Terrier N, Glissant D, Grimplet J, Barrieu F, Abbal P, Couture C, Ageorges A, Atanassova R, Leon C, Renaudin JP, Dedaldechamp F, Romieu C, Delrot S, Hamdi S:等基因特异性寡核苷酸阵列揭示了葡萄浆果发育过程中基因表达的多方面变化。植物学报,2005,22(5):832-847。10.1007 / s00425 - 005 - 0017 - y。
- 9.
杨晓明,李志刚,李志刚,李志刚,李志刚:葡萄生长发育过程中的cDNA芯片分析。西拉子)浆果皮。中国生物医学工程学报,2005,5(1):40-58。10.1007 / s10142 - 004 - 0124 - z。
- 10.
Tanksley SD:番茄果实大小和形状变异的遗传、发育和分子基础。植物学报,2004,16增刊:S181-9。10.1105 / tpc.018119。
- 11.
Lemaire-Chamley M, Petit J, Garcia V, Just D, Baldet P, Germain V, Fagard M, Mouassite M, Cheniclet C, Rothan C:转录谱的变化与番茄早期果实组织特化有关。植物营养学报,2005,39(2):366 - 366。10.1104 / pp.105.063719。
- 12.
郑飞,唐旭,Alba RM, White JA, Ronning CM, Martin GB, Tanksley SD, Giovannoni JJ:番茄果实成熟的EST综合分析与比较基因组学。植物学报,2004,40(1):47-59。10.1111 / j.1365 - 313 x.2004.02188.x。
- 13.
Alba R, Payton P, Fei Z, McQuinn R, Debbie P, Martin GB, Tanksley SD, Giovannoni JJ:转录组和选择性代谢产物分析揭示了番茄果实发育过程中乙烯的多个控制点。植物细胞学报,2005,17(11):2954-2965。10.1105 / tpc.105.036053。
- 14.
崔诺提L, Bonghi C, Ziliotto F, Zanin D, Rasori A, Casadoro G, Ramina A, Tonutti P:利用微阵列[mu]PEACH1.0研究桃果实更年期前向更年期转变过程中的转录组变化。植物科学学报,2006,27(3):344 - 344。10.1016 / j.plantsci.2005.10.015。
- 15.
Fonseca S, Hackler JL, Zvara A, Ferreira S, Balde A, Dudits D, Pais MS, Puskas LG:利用cDNA微阵列技术监测梨果实发育、成熟和衰老过程中的基因表达。植物科学学报,2004,27(3):357 - 357。10.1016 / j.plantsci.2004.03.033。
- 16.
LH:苹果和其他柚子的形态。康奈尔农业实验站,日内瓦,纽约,美国,回忆录,1940,230:1-32。
- 17.
Denne MP:果实发育和一些影响它的树木因素。中国植物学报,2003,23(2):344 - 344。
- 18.
Denne MP:苹果小果的生长,早期疏疏对果实发育的影响。植物学报,2000,24(4):397-406。
- 19.
Brookfield P, Murphy P, Harker R, MacRae:淀粉降解和淀粉模式指数;解读和与成熟的关系。采收后生物技术,1997,11:23-30。10.1016 / s0925 - 5214(97) 01416 - 6。
- 20.
Faragher JD, Brohier RL:乙烯和苯丙氨酸解氨酶对苹果成熟过程中花青素积累的调控。园艺科学,1984,22:89-96。10.1016 / 0304 - 4238(84) 90087 - 6。
- 21.
Reid MS, Rhodes MJC, Hulme AC:苹果成熟过程中乙烯和二氧化碳的变化。中国农业科学,2007,27(4):457 - 457。10.1002 / jsfa.2740240815。
- 22.
Schaffer RJ, Friel EN, Souleyre EJF, Bolitho K, Thodey K, Ledger S, Bowen JH, Ma JH, Nain B, Cohen D, Gleave AP, Crowhurst RN, Janssen BJ, Yao JL, Newcomb RD:基因组学方法揭示苹果香气产生主要受乙烯控制在每个生物合成途径的最后一步。植物科学进展,2007,29(4):379 - 379。10.1104 / pp.106.093765。
- 23.
Marie D, Brown SC:植物DNA直方图的流式细胞术练习,70个物种的2C值。中国生物医学工程学报,2003,27(1):1-5。10.1016 / 0248 - 4900(93) 90113。
- 24.
Newcomb RD, Crowhurst RN, Gleave AP, Rikkerink EHA, Allan AC, Beuning LL, Bowen JH, Gera E, Jamieson KR, Janssen BJ, Laing WA, McArtney S, Nain B, Ross GS, Snowden KC, Souleyre EJF, Walton EF, Yauk YK: Apple表达序列标签的分析。中国生物医学工程学报,2006,26(1):457 - 457。10.1104 / pp.105.076208。
- 25.
李永平,刘海生,徐玉生,韩思,崔友,金东,莫ig,金文涛,成SK:苹果早期果实发育基因表达的微阵列分析。植物细胞代表2007
- 26.
Benjamini Y, Hochberg Y:控制错误发现率:一种实用而强大的多重测试方法。统计学报,1995,29(3):349 - 349。
- 27.
Schoof H, Zaccaria P, Gundlach H, Lemcke K, Rudd S, Kolesov G, Arnold R, Mewes HW, Mayer KF: MIPS拟南芥数据库(Arabidopsis thaliana Database, MAtDB):基于第一个完整植物基因组的综合性生物学知识资源。中国生物医学工程学报,2002,30(1):91-93。10.1093 / nar / 30.1.91。
- 28.
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ:基本的局部对齐搜索工具。中华分子生物学杂志,1999,15(3):403-410。
- 29.
范德波,李志刚,李志刚,李志刚,李志刚。拟南芥核心细胞周期基因的全基因组分析。植物细胞学报,2002,14(4):903-916。10.1105 / tpc.010445。
- 30.
Menges M, de Jager SM, Gruissem W, Murray JA:拟南芥核心细胞周期调控因子的全球分析发现了新的基因,揭示了多种高度特异性的表达谱,并为植物细胞周期控制提供了一个连贯的模型。植物学报,2005,41(4):546-566。10.1111 / j.1365 - 313 x.2004.02319.x。
- 31.
史密斯AM,塞曼SC,史密斯SM:淀粉降解。植物学报,2005,29(3):344 - 344。10.1146 / annurev.arplant.56.032604.144257。
- 32.
Kanehisa M, Goto S, Hattori M,青木木下KF, Itoh M, Kawashima S, Katayama T, Araki M, Hirakawa M:从基因组学到化学基因组学:KEGG的新进展。核酸决议2006,34(数据库问题):D354-7。10.1093 / nar / gkj102。
- 33.
Ferrandiz C, Pelaz S, Yanofsky MF:拟南芥心皮和果实发育的控制。生物化学学报,1999,29(3):344 - 344。10.1146 / annurev.biochem.68.1.321。
- 34.
Volz RK, Harker FR, Lang S:“Gala”苹果果实发育过程中硬度下降。环境科学学报,2003,26(6):377 - 382。
- 35.
李瑞仪,李志刚,李志刚,李志刚:苹果果实成熟过程中mRNA和蛋白质的变化。cv Golden Delicious)。植物营养学报,2000,27(2):379 - 379。
- 36.
Menges M, Murray JA:同步拟南芥悬浮培养的细胞周期基因活性分析。植物学报,2002,30(2):203-212。10.1046 / j.1365 - 313 x.2002.01274.x。
- 37.
孟志刚,刘志刚,王志刚,王志刚,等。拟南芥细胞悬液中基因表达的研究进展。植物分子生物学杂志,2003,53(4):423-442。10.1023 / B: PLAN.0000019059.56489.ca。
- 38.
Sorrell DA, Menges M, Healy JM, Deveaux Y, Amano C, Su Y, Nakagami H, Shinmyo A, Doonan JH, Sekine M, Murray JA:烟草品种Bright yellow 2细胞周期蛋白依赖性激酶的细胞周期调控。植物学报,2001,26(3):344 - 344。10.1104 / pp.126.3.1214。
- 39.
Boudolf V, Barroco R, Engler Jde A, Verkest A, Beeckman T, Naudts M, Inze D, De Veylder L: b1型周期蛋白依赖性激酶在拟南芥气孔复合体形成中的作用。植物细胞学报,2004,16(4):945-955。10.1105 / tpc.021774。
- 40.
Boudolf V, Vlieghe K, Beemster GT, Magyar Z, Torres Acosta JA, Maes S, Van Der Schueren E, Inze D, De Veylder L:植物特异性周期蛋白依赖激酶CDKB1;1和转录因子E2Fa-DPa控制拟南芥有丝分裂和内复制细胞的平衡。植物细胞学报,2004,16(10):2683-2692。10.1105 / tpc.104.024398。
- 41.
Dewitte W, Murray JA:植物细胞周期。植物学报,2003,29(3):344 - 344。10.1146 / annurev.arplant.54.031902.134836。
- 42.
Spruck C, Strohmaier H, Watson M, Smith AP, Ryan A, Krek TW, Reed SI:哺乳动物Cks1的CDK独立功能:SCF(Skp2)靶向CDK抑制剂p27Kip1。生物化学学报,2001,7(3):639-650。10.1016 / s1097 - 2765(01) 00210 - 6。
- 43.
Menges M, Samland AK, Planchais S, Murray JAH:拟南芥d型细胞周期蛋白CYCD3;1对g1 -S相变的限制作用。植物学报,2006,18(4):893-906。10.1105 / tpc.105.039636。
- 44.
sugiamoto - shirasu K, Roberts K:“放大”:植物的核内复制和细胞大小控制。植物学报,2003,6(6):544-553。10.1016 / j.pbi.2003.09.009。
- 45.
Weinl C, Marquardt S, Kuijt SJ, Nowack MK, Jakoby MJ, Hulskamp M, Schnittger A:植物周期蛋白依赖性激酶抑制剂ICK1/KRP1具有非细胞自主作用并抑制有丝分裂的新功能。植物细胞学报,2005,17(6):1704-1722。10.1105 / tpc.104.030486。
- 46.
Verkest A, Manes CL, Vercruysse S, Maes S, Van Der Schueren E, Beeckman T, Genschik P, Kuiper M, Inze D, De Veylder L:周期蛋白依赖性激酶抑制剂KRP2通过抑制有丝分裂CDKA;1激酶复合物控制拟南芥叶片发育过程中核内再复制周期的开始。植物细胞学报,2005,17(6):1723-1736。10.1105 / tpc.105.032383。
- 47.
Gonzalez N, Hernould M, Delmas F, Gevaudant F, Duffe P, Causse M, Mouras A, Chevalier C:番茄WEE1基因同源物的分子特征(Lycopersicon esculentum Mill.)。植物分子生物学,2004,56(6):849-861。10.1007 / s11103 - 004 - 5110 - 2。
- 48.
原田T,仓桥W,柳井M,若asa Y,佐藤T:细胞增殖和细胞增大对苹果果实大小的影响。中国园艺学报,2005,25(4):447-456。10.1016 / j.scienta.2005.02.006。
- 49.
Smith SM, Fulton DC, Chia T, Thorneycroft D, Chapple A, Dunstan H, Hylton C, Zeeman SC, Smith AM:拟南芥叶片淀粉代谢编码酶的转录组日变化为转录和转录后调节淀粉代谢提供了证据。植物科学进展,2004,26(1):379 - 379。10.1104 / pp.104.044347。
- 50.
Yano M, Kato M, Ikoma Y, Kawasaki A, Fukazawa Y, Sugiura M, Matsumoto H, Oohara Y, Nagao A, Ogawa K:日本生果和加工水果中类胡萝卜素的定量。食品科学与技术研究,2005,11(1):13-18。10.3136 / fstr.11.13。
- 51.
张春华,张志刚,张志刚,等:一种提取松材中核糖核酸的方法。植物分子生物学通报,1993,11(2):113-116。
- 52.
Snowden KC, Simkin AJ, Janssen BJ, Templeton KR, Loucas HM, Simons JL, Karunairetnam S, Gleave AP, Clark DG, Klee HJ:矮牵牛根尖显性降低基因1/矮牵牛类胡萝卜素切割基因DIOXYGENASE8影响分枝生成,并在叶片衰老、根系生长和花朵发育中发挥作用。植物细胞学报,2005,17(3):746-759。10.1105 / tpc.104.027714。
- 53.
Sol基因组网络。[http://www.sgn.cornell.edu/]
确认
我们要感谢吉姆·乔凡诺尼的支持以及对番茄阵列数据的慷慨访问。BJJ, KT, RS, LB, RB, JHB, RNC, APG, SL, SMcA, FBP和SW都得到了新西兰研究科学与技术基金会(C06X0207)和新西兰horresearch的内部资助。RA由美国国家科学基金会植物基因组资助项目05-01778资助。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
BJJ设计了实验,设计了阵列中使用的寡核苷酸,收集了样品,开发了标记和杂交方法,分析了数据并起草了手稿。KT进行标记杂交、淀粉代谢基因分析、淀粉基因qRT-PCR,并协助撰写稿件。RS协助开发混合方法,处理原始阵列数据并开发归一化算法。RA提供了番茄阵列数据的访问,并对苹果数据进行了分析,并协助完成了稿件。LB进行了标记和杂交。RB进行了标记和杂交,qRT-PCR验证,并协助完成手稿。JHB制备了RNA。RNC提供了生物信息学支持和用于选择unigenes和设计阵列中使用的寡核苷酸的脚本。APG提供测序和生物信息学支持,以确认阵列中使用的基因序列。SL负责RNA的制备和稿件的协助。 SMcA assisted in design of the experiments and managed the sample collection. FBP assisted in the design of algorithms for normalisation of the data. KCS assisted in design of the experiments and the oligos, analysed data and assisted with the manuscript. SW collected samples. All authors have read the final manuscript without any objections.
电子辅助材料
12870 _2007_224_moesm1_esm.xls
附加文件1:数组数据。包含每个数组特性的表达式数据的Excel电子表格。数据显示为除0 DAA以外的所有样本的两个生物重复和两个技术重复的表达水平的平均值、数据点数(n)和标准误差(SE),其中只有一个生物重复被采样。在某些情况下,由于技术原因,一个或多个数据点被排除在分析之外,在这种情况下,平均值和标准误差是从剩余的数据中计算出来的。原始数据存放在GEO。(xls7mb)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
杨森,b.j.,索迪,K.,谢弗,R.J.et al。苹果花蕾到成熟果实发育的基因表达分析。BMC植物生物学8日,16(2008)。https://doi.org/10.1186/1471-2229-8-16
收到了:
接受:
发表:
关键字
- 水果发展
- 苹果的水果
- 淀粉水平
- 苹果的基因
- 早期果实发育