摘要
背景
尽管最初认为替代剪接(AS)在植物中的频率低于在动物中的频率,但现在已经知道在植物中广泛存在。在这里,我们报道了豆科植物中AS的特征,豆科植物是最大和最重要的植物家族之一,基于EST对其基因组序列的比对Medicago truncatula(太),Lotus对虾(Lj).
结果
单从同源EST比对来看,可观察到的基因交替拼接的频率较低太(~10%, 1107个基因)和Lj(~3%, 92个基因)比拟南芥还有大米(都在20%左右)。然而,如果EST水平归一化,所有四种物种的AS频率是相当的。内含子保留是AS在所有四种植物中最常见的形式(约50%),豆科植物中的频率略低拟南芥和米饭。这与脊椎动物明显不同,在脊椎动物中外显子跳过最为常见。为了发现更多的AS事件,我们将其他豆科植物的est与太基因组序列。这样,额外增加了248太基因被预测是交替拼接的。我们还发现了22个AS事件在两种或两种以上的植物物种中完全保守。
结论
本研究扩大了具有高水平AS的植物类群的范围,证实了内含子保留在植物中的重要性,并证明了利用来自相关物种的ESTs来识别新的和保守的AS事件的效用。结果还表明,AS在植物中的频率与在哺乳动物中观察到的频率相当。最后,我们的结果强调了在估计备选剪接频率时规范化EST水平的重要性。
背景
选择性剪接(Alternative splicing, AS)是真核生物中一种重要的细胞过程,可将一个pre-mRNA转化为多个mRNA亚型。植物AS事件过去被认为是罕见的。然而,越来越多的计算研究现在已经证明,植物中交替拼接基因的频率比之前估计的要高[1,2].20-30%的表达基因被交替拼接拟南芥(在)和大米(Oryza sativa, Os),这是由大规模est基因组比对所揭示的[1,2].最近一项使用EST对间隙排列(EST-EST)的研究调查了11种植物,并表明不同植物的AS总体频率差异很大,有些比率与在动物中观察到的相当[3.].在哺乳动物中,外显子跳过(ExonS)是AS最常见的类型[4,5],但在在而且操作系统,内含子保留(IntronR)最丰富[1].替代受体位点(AltA)和替代供体位点(AltD)在这两种模式植物中也很常见[1,2].AS事件的一种罕见类型是替代位置(AltP),在这种情况下,替代内含子在供体和受体位点上都与其本构形式不同[1].附加文件中显示了所有五种类型的AS事件的示例1(补充图S1)。最近,一种涉及全基因组微阵列数据的新方法揭示了在~8%的基因中可以检测到IntronR在基因(6].普遍存在的IntronR事件表明内含子识别机制在其中起主导作用在而且操作系统[1].一小部分保守AS事件也被发现和证实在而且操作系统,强烈表明AS在植物中的功能重要性[1].
大多数关于哺乳动物和植物AS的计算研究使用来自同一物种的转录本序列作为它们的基因组序列。对于EST/cDNA集合相对较少的物种,来自密切相关物种的转录本序列可以成为鉴定附加AS事件的宝贵资源。即使对于具有大量EST集合的物种,包括人类和小鼠,跨物种EST比对也被用于揭示新的AS事件。通过将小鼠转录本与人类基因组比对,多达42%的人类基因显示出新的As模式[7],超过10%的人类基因座在小鼠中表现出保守的AS事件[8].另一项将跨物种策略应用于人类、小鼠和大鼠的研究发现了758个新的盒式外显子(exons)和167个新的保留内含子(IntronR)。RT-PCR验证了50~80%的检测事件,表明跨物种方法在识别新的AS事件方面具有令人印象深刻的潜力[9].在植物中,跨种转录本主要用于基因注释。例如,来自185个物种的转录本集合被映射到操作系统基因组,证实约90%的基因预测加上约500个新基因[10].类似地,大约850个新基因和1000个新AS事件被注释操作系统通过比对7种植物的est [11].跨物种转录本支持的AS事件很可能是功能性的,因为它们在物种之间是保守的。
实验研究为AS在植物中的功能提供了新的认识。许多具有不同功能的植物基因通过AS进行调控,包括(但不限于)与转录、剪接、光合作用、抗病、胁迫、开花和籽粒品质有关的基因(见[12,13])。参与剪接,特别是剪接调节的基因似乎有较高的AS频率[14].近年来的一些研究表明,富含丝氨酸/精氨酸(SR)的蛋白转录本具有广泛的AS水平,并且某些AS模式是保守的在而且操作系统[15- - - - - -18].玉米SR蛋白转录本也可交替剪接[19,20.].温度胁迫(冷和热)以及激素处理可以改变SR蛋白的as模式在,表明AS在应激反应中起着重要作用[15].一个在U2AF35同源物(atU2AF35a)也可以通过在编码区3'端去除重复边界的非规范内含子来拼接。改变U2AF35同源物的表达改变了FCA基因的剪接模式,进而导致开花时间的变化[21].U1- 70k基因编码U1小核核糖核蛋白(snRNP)中的一个核心蛋白。U1-70K的第6个内含子可以保留在在[22,一个介于两者之间的事件在而且操作系统[1].最近,IntronR事件被实验证实操作系统玉米[23].
现在已知54种植物的400多个基因是交替拼接的[24].然而,在豆科植物中只有少数的AS事件被报道(蝶形花科),是最大、最重要的植物科之一。在Lotus对虾(Lj),植物螯合肽合成酶基因(LjPCS2)可以交替拼接,其中一种亚型存在于结节(LjPCS2- 7n)中,另一种亚型存在于根(LjPCS2- 7r)中。这两种异构体编码的蛋白质只在5个氨基酸上存在差异,其中一种蛋白质(LjPCS2-7N)具有镉(Cd)耐受性,而另一种则没有,至少在酵母细胞中异位表达时没有[25].肿瘤特异性基因(LjNOD70)在肿瘤中显示IntronR事件Lj,其中剪接亚型在结节中较少[26].6个蔗糖合酶基因存在于在,操作系统而且Lj,但只有Lj同源物(LjSUS2)被交替拼接[27].在大豆(大豆,通用汽车),通过EST数据挖掘发现了一个结节特异性基因(GmPGN)。实验证实了该基因的组织特异性,并揭示了AS事件[28].在芸豆中(菜豆),单个基因(PvSBE2)可交替拼接产生两种淀粉分支酶异构体,每一种都具有不同的特征和亚细胞定位[29].一种高度丰富的新型巨逆行元素(巨魔)豌豆(Pisum一)部分拼接,可能调节全长蛋白的比例,因为保留的内含子导致截断[30.].
两种豆科植物,Medicago truncatula(太),l .对虾(Lj),正在进行大规模的基因组测序计划[31].2006年底,Medicago基因组序列联盟(MGSC)在1996个细菌人工染色体(BAC)克隆序列的基础上构建了部分基因组组合,作为构建草稿假染色体的基础。国际上共注释了42,358个基因Medicago基因组注释组[32,占总数的60%左右太基因。该数据已发布Mt1.0版本,可在[33].与此同时,Lj在GenBank中有1394条可转化人工染色体(tac)(截止2006年中期),其中488条处于第3期(已完成)。两种豆科模式植物都有相对较大的EST集合(超过150000个序列)。其他豆科植物,特别是大豆,也有大量的转录序列。这些特性使得太而且Lj对于豆科植物和其他植物的AS事件的计算比较非常理想。
在本研究中,所有来自豆科植物的转录本序列都被排列到太而且LjBAC / TAC序列。在而且操作系统转录本序列也与他们自己的基因组序列进行比对。只要作为分析基础的est的数量在不同的物种之间标准化,那么在不同的植物物种之间交替拼接基因的频率是非常相似的。在太在目前的EST覆盖范围内,约10%的表达基因被交替拼接,其中IntronR基因的数量最多。如果跨物种的ESTs与基因组对齐,就可以识别出新颖和保守的AS事件。这些结果为分析所有植物以及仅在豆科植物中发现的AS事件提供了基础。这是第一次在植物中使用est基因组比对大规模分析AS在而且操作系统这也是首次在植物中使用跨物种转录本序列进行详细比较。
结果
豆科植物外显子和内含子的特征
两个计算机程序,GeneSeqer [34]和GMAP [35],在EST序列与它们的原生基因组比对方面产生了大致相似的结果太,Lj,在,操作系统数据集。为了减少由于歧义导致的对齐工件的可能性,在进一步的分析中只使用两个程序中通常预测的对齐。此外,采用了高度严格的标准(>95%序列一致性,>80%转录本覆盖率)来限制转录本映射到未完全测序基因组中非同源的发散位置的可能性。大约有一半和三分之一的物种特异性EST集可以与电流对齐太而且Lj分别是基因组序列,其当前的序列组合大致反映了整个基因组的覆盖范围。为Lj,约15%的转录本序列映射到完成的(3期)BAC/ tac。除非另有说明,我们的分析Lj是完全基于这个子集的。如表所示1,共鉴定出11516和3298个基因/转录单位(TU,定义见METHODS)太而且Lj分别有74%和57%的人拥有多重EST支持。每基因/TU的平均ESTs数分别为10和7 in太而且Lj分别为26和30在而且操作系统.
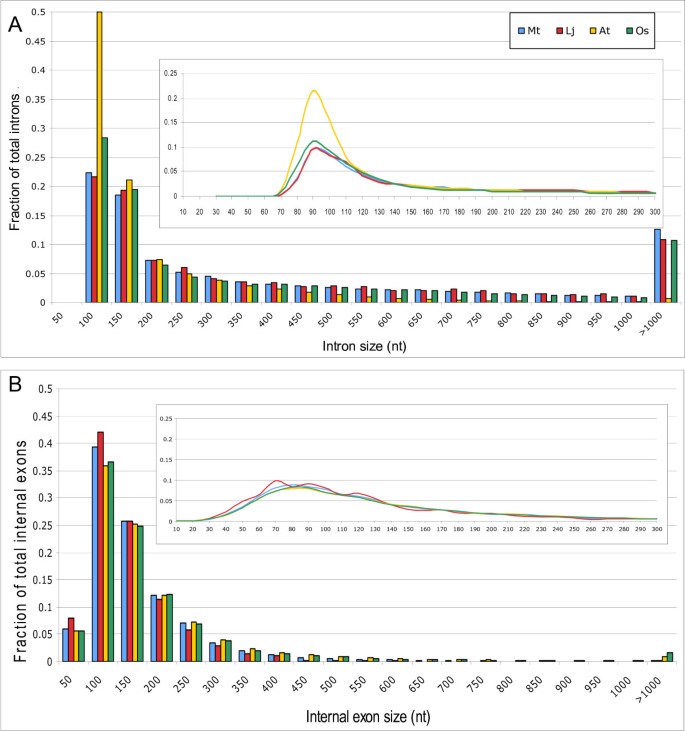
我们比较了四种植物EST比对揭示的内含子/外显子特征。内含子的大小分布也非常相似太而且Lj两个物种内含子的平均大小约为460-470 nt,中位数约为220 nt。因此,豆科植物内含子的长度明显长于in在(平均171 nt,中位数101 nt),略长于操作系统内含子(平均438 nt,中位数164 nt)。如图所示1内含子大小分布在4种物种中均在90nt附近有一个峰值。太而且Lj短于150nt的内含子比较少,而长于200nt的内含子比较多在而且操作系统.在内含子显然是四种植物中最短的。只有不到1%的内含子长度超过1000nt在,而在其他植物中这一数字超过10%。四种植物的外显子大小趋于相似,豆科植物的外显子略短在而且操作系统外显子。在太而且Lj内显子平均尺寸分别为140和127 nt,中位尺寸约为108和100 nt。在而且操作系统有内部外显子,平均值为164 nt和175 nt,中位数为113 nt和114 nt1 b显示了外显子的尺寸分布太,在而且操作系统所有显示在80 nt左右的峰值。Lj由于样本量小,数据的一致性较差。与内含子相比,小于150nt的外显子频率较高太而且Lj比在在而且操作系统在豆科植物中,长度大于200 nt的外显子频率较低。总的来说,豆科植物内含子较长,外显子略短在而且操作系统.一般来说,植物内含子比外显子长。超过40%的内含子太,Lj而且操作系统都大于300nt,而只有不到10%的外显子如此大。
如前所述[1,36]时,内含子和外显子的gc含量低约5%在比在操作系统.豆科植物内含子和外显子的gc含量与豆科植物的gc含量非常相似在,虽然太gc含量略低于两者吗在或Lj内含子区和外显子区(见附加文件)1,补充表S1和补充图S2)。g含量和a含量在所有物种中都是相似的,包括操作系统,虽然操作系统内含子含c较多,含u较少。在所有物种中,U-(T-)和A-含量的分布比G-或c -含量的分布变化更大(见附加文件)1,补充图S3)。在四种物种中,内含子和外显子之间的gc含量差异约为10%太差异最大的是11.7%和操作系统最小的为9.6%(见附加文件)1,补充表S1)。
不同的植物种类有相似水平的交替拼接基因
先前的研究表明,大约20%的表达基因是交替拼接的在而且操作系统,其中一半的AS事件是内含子保留(IntronR) [1].当我们重新检查AS频率时在而且操作系统在这项研究中,我们还发现了20%左右的频率。然而,由于EST数据集的增加,这些物种的转录本序列总数增加了80%-200%。在太而且Lj在美国,可用于分析的无害环境技术的数量要低得多。一直以来,观察到的交替剪接基因的比例要低得多,只有9.6%太2.8%Lj(表2).交替拼接基因的例子太是否显示在附加文件中1,补充图S1。所有AS的数据都存放在ASIP站点,并可在该站点查看[37].
为了比较不同物种之间的选择性剪接频率,早期的研究依赖于每个基因随机选择10个ESTs作为估计as频率的基础[4].在这里,只有一小部分(10 - 20%)的豆科基因被10个或更多的ESTs覆盖,所以这种方法是不实际的。相反,我们绘制了不同物种中EST覆盖范围相似的所有基因组的AS频率,如图所示2.太由于样本量小,剔除基因总数少于80个的类别以减少噪声Lj数据完全不包括在内,因为样本量一致太小。当以这种方式分析时,对于几乎所有的大小分类,无论什么物种,交替拼接的基因片段都是相似的。对于具有四个EST的基因(每个基因的EST中位数太)时,观察到AS频率为6-12% in太,在,操作系统一样的。对于含有9到11个EST的基因(每个基因的EST中位数操作系统而且在), 15-23%是交替拼接的。一般来说,随着转录本覆盖率的增加,交替剪接基因的比例不断增加,最终达到66%操作系统46%的人在含有数百种ESTs的基因,其水平与哺乳动物的相似[38,39].有趣的是,AS级别在操作系统是否始终高于10%在有超过40个支持ESTs的基因。
内含子r是豆科植物中最丰富的AS类型
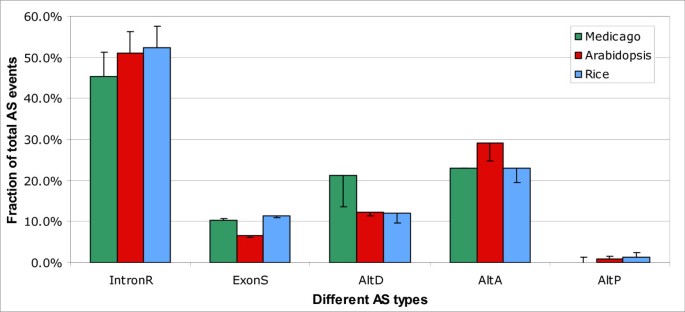
如表所示2,不同AS类型的比例相似太,在而且操作系统.(Lj数据也被列出,但不包括在分析中,因为只有大约100个as事件被识别)。植物中超过一半的AS事件是IntronR, 6-11%是ExonS,剩下的30-40%涉及不同的剪接位点(AltD/A/P)。这些数字与以前观察到的数字相当相似[1].太有较低的IntronR比例(51%)和较高的AltD比例(13%)与在而且操作系统.EST覆盖率的不同水平对AS事件的组成影响不大。如附加文件所示1(补充图S4),在所有EST水平上,不同AS类型的比率基本保持不变,特别是在在而且操作系统.IntronR在各层次中最为丰富,在各层次中所占比例相对较低太.外显子的比例始终较低在比在操作系统(和太),而AltA比值较高。
为了尽量减少EST集合中由测序错误或污染引起的虚假AS事件,我们对至少两个转录本序列支持的AS事件子集重复上述分析[40].如图所示3.在该亚群中,IntronR的比例降低了约5%。太有最低的IntronR比例(45%),比在而且操作系统.与完整数据集相比,外显子的比例保持不变。在太而且操作系统, 10-11%的AS事件是外显子,而在在.AltD比率太在子集中显著增加到21%,几乎是在而且操作系统.在在的AltA比为~30%,而在太而且操作系统.在支持每个亚型的转录本更多的子集数据中观察到类似的趋势。完整数据和子集数据都表明了这一点太IntronR的比例较低,AltD的比例较高,那么在具有较低的外显子比率但较高的AltA比率。
Medicago的跨物种EST比对揭示了数百种新的AS事件
即使是“可靠的”AS事件(如上所定义)也不一定是功能性的。因为保护通常是功能的一个很好的指标,我们部署了一个跨物种的方法,类似于以前在哺乳动物中使用的大规模方法,以确定功能性AS事件[7,9].所有可用的EST序列Lj,通用汽车和其他豆科植物都反对太•巴。跨物种方法的一个问题是潜在的高错误率[7].在这里,即使使用高达80%的身份截断,仅从GeneSeqer或GMAP校准中就可以识别出数百个as事件,其中约40%的事件在程序之间一致。我们的分析只使用了两个程序识别的常见事件,以减少由对齐错误引起的假阳性事件。如表所示3., 10-20%的非太豆科转录本序列可以映射到太BACs和聚类共7896个非冗余基因,81%的基因也具有太美国东部时间支持。从跨物种EST比对中鉴定出的内含子约70%与太EST支持内含子。基因结构来源于跨物种ESTs和太ESTs对齐基本一致,证明了跨物种ESTs在基因组注释中的价值[10].在这个分析中,总共有307个太基因(3.9%)被发现交替剪接,248个基因没有AS的证据太est序列。如果包括这些新的AS事件,估计的频率太备选剪接基因从9.6%增加到10.4%。有趣的是,从大豆ESTs中鉴定出的AS事件比从Lj尽管它们之间的进化距离相似Mt-Gm与太-Lj.在而且操作系统EST序列也被用于比较的跨物种分析,但使用相同的标准,只有1%的序列可以映射。没有推导出可靠的AS事件在而且操作系统记录序列。
从豆科植物跨种EST比对中共鉴定出367个跨种AS事件,其中IntronR占35.7%,ExonS占16.9%,AltD占16.1%,AltA占29.1%,AltP占2.2%4).与使用AS事件识别相比太单是ESTs,跨种AS事件表现出较低的IntronR比例和较高的ExonS、AltD和AltA比例。由于大多数跨物种As事件可能是保守的太与EST的原生物种相比,各AS类型在跨物种AS事件中的比例可以解释为功能性AS事件的比例。然而,在跨物种EST比对中,内含子的比例可能被低估了,因为内含子序列不像外显子序列那样保守,即使在亲缘较近的物种中也是如此。因此,一些跨物种的ESTs保留了来自原生物种的内含子,可能被80%的身份截断所过滤。在附加文件中比较了跨物种AS事件和同物种AS事件的位置和结果1(补充表S2)。
大约90%的跨物种AS事件位于开放阅读框(orf)中,远高于同一物种AS事件的比例(70-75%)。5'-UTR的跨物种和同物种AS事件似乎比3'-UTR的多(数据未显示,[1])。对于orf中的AS事件,在不改变阅读框的情况下从蛋白质中添加或删除一些氨基酸的翻译-读通事件的比例在跨物种和同物种事件中是相似的(20-24%)。AltA的翻译-读通比最高(35-40%),IntronR的翻译-读通比最低(2-10%)。有趣的是,产生无意义介导衰变(NMD)底物的AS事件的比例[41]在跨物种AS事件中高于在同一物种AS事件中。近一半的跨物种AS事件产生NMD基质,而在同物种AS事件中产生NMD基质的比例为30-40%。
从豆科植物跨种EST序列中鉴定的保守AS事件
为了确定具有多物种保存的直接证据的AS事件,采用了两种方法:(1)将所有豆科植物est与LjTACs识别由相同est之间预测的保守AS事件太而且Lj;(2)识别中保守的AS事件太与来自多个豆科植物的EST证据,均显示相同的AS模式。共保留了242个AS事件太而且Lj通过方法(1)进行识别,其中IntronR事件92例(38.0%),ExonS事件26例(10.7%),AltA事件78例(32.2%),AltD事件41例(17.0%),AltP事件5例(2.1%)。这些AS事件可以在ASIP网站上看到。方法(2)识别出22个完全保守的AS事件太(见附加文件)1,补充表S3)。22个基因中有9个也是如此在和/或操作系统共享相同AS模式的紧密同系物。例如,太假设蛋白AC156627_1具有大豆和太est支持第一个ORF内含子中的AltA事件,因此异构体利用了本构受体位点(AGCAG)上游5 nt的替代受体位点(AACAG),产生可能受NMD影响的底物。在同源物(At5g25360.1和At1g15350.1)和操作系统同源物(LOC_Os02g10720)具有完全相同的AS模式,包括替代受体位点。这种基因似乎是植物特异性的,因为无法识别非植物同源体。完全保守的AS事件的另一个例子是太AP2结构域包含AC151460_3蛋白,其中3'-UTR内含子可以保留。一个在同源和三操作系统同源物也有相同的内含子保留。在豆科植物中也有一些未被观察到的AS事件在而且操作系统.一个例子是AC124951_11,它是豆科植物中含有3'-UTR内含子交替剪接(AltD)的高表达碳酸酐酶基因。AltD事件在所有豆科植物中都是保守的(太,Lj,通用汽车等),但没有在在而且操作系统尽管存在数百个ESTs,这表明这种AS事件可能是豆类特有的。
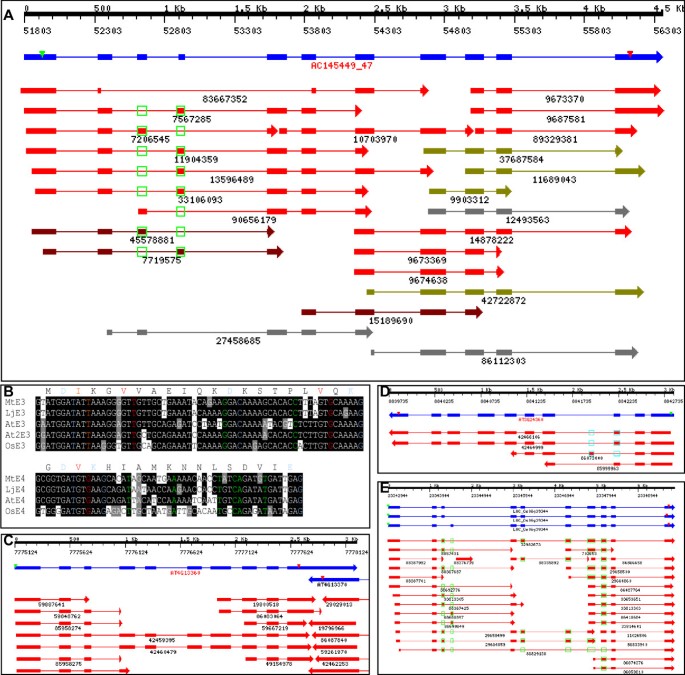
一个完全保守的外显子事件发生在烯酰辅酶a水合酶/异构酶基因中(太: AC145449_47)。如图所示4, AC145449_47的imgag注释基因结构包含11个外显子,每个外显子都有很强的EST支持。Exon3 (65nt)和Exon4 (53nt)互斥。在一个亚型中,Exon3被保留,Exon4被跳过(太: 7206545, 90656179;Lj:45578881;狼:27458685)。在另一亚型中,Exon4被保留,Exon3被跳过(太: 7567285, 11904359, 13596489, 33106093;Lj: 7719575)。因此,这两种mRNA亚型编码两种蛋白质(418 aa和414 aa),其预测的enyl - coa水合酶结构域(ECH, pfam00378)略有不同。没有异构体同时包含两个外显子,但可以跳过两者(太: 83667352)。两个基因在(At4g13360和At3g24360),在操作系统(LOC_Os06g39344)和一个inLj(LjTC_2465, AP006370.1: 88858-94512)是与AC145449_47最接近的同源物。除了At4g13360外显子(Exon3)被完整保留,在相应区域没有发现53-nt外显子的痕迹(图),其余同源基因均观察到完全相同的AS模式4汉英).序列比较发现,所有四种物种的简并密码子中都有几个核苷酸基4 b).这些碱基可能有助于外显子的识别(或跳过)。
植物烯酰基辅酶a水合酶/异构酶基因中完全保守的外显子事件.一个:同种和跨种EST对齐太基因座AC145499_47。填充框和箭头表示外显子,线表示内含子。绿色打开或填充框表示在某些est中跳过或保留外显子。顶部的黑色刻度表示BAC (AC145499)上基因位点的坐标。蓝色条表示IMGAG注释的基因模型,绿色三角形表示蛋白质翻译起始密码子,红色三角形表示停止密码子。红色条代表个体同一物种的EST序列。紫色条表示Ljest,暗黄色条代表大豆的est,灰色条代表其他豆科植物的est。B.互斥外显子的多重序列比对。E3表示外显子3,E4表示外显子4。的第二副本中的外显子是At2E3在基因(At4g13360)。编码的氨基酸太序列列在序列对齐的顶部。在所有外显子中保守的简并位(核苷酸的变化不会改变氨基酸)用颜色突出显示。C.EST对齐第二份在基因(At4g13360)。该基因中仅存在外显子E3,未检测到外显子。D、E.EST校准在而且操作系统外显子模式完全保守的基因。
讨论
不同物种AS频率的比较
在本研究中,对目前EST和基因组序列的比对显示,约10%的表达基因是交替拼接的太相比之下,只有20%在而且操作系统.这一差异主要是由于在太.我们证明,当调整具有相当EST数的基因时,三种植物的AS频率本质上是相似的。这一结论与最近一项基于EST对间隙排列的研究得出的结论不同,在不同的植物物种中观察到的变异程度更大[3.].对est数据的解释可能会被大量的基因复制事件所混淆。随着越来越多的植物基因组序列变得可用,应该很快就可以更精确地解决有关植物中AS的程度和进化的有趣问题。
另外,拼接的异构体通常丰度较低,在小的EST集合中捕获它们的机会较低,因此很难准确估计AS频率。假设一个功能事件有一定的百分比p的转录本,观察到AS事件的概率n覆盖备选剪接位点的est为1 - (1 -)p)n.例如,如果生成了一个交替拼接的异构体p= 10%的时间,n = 10个转录本序列将有65%的概率观察到该事件,而22个转录本序列将有>90%的概率观察到该事件。我们的结果表明,具有少量ESTs的基因的AS频率是相似的太,在,操作系统,表明它们都具有相似的功能AS事件级别。
在AS亚型丰度更低的情况下,显然需要更多的转录本来检测事件。尽管如此,操作系统似乎在>30 ESTs基因中AS的频率比两者都高太或在.只关注>40 ESTs基因,AS频率在操作系统是否始终(>10%)高于在.在这个分析中,我们没有包括来自操作系统亚种籼稻以消除高AS频率是由跨亚种ESTs错误引起的可能性。在任何情况下,来自EST测序或基因组污染的错误率在这三种植物中可能是相似的。因此,操作系统似乎有更高水平的低丰度AS事件在(或太).一些低丰度事件可能是在不同生长条件下植物组织构建的EST库中捕获的剪接错误,因此在植物中低丰度AS事件的较高水平操作系统可能意味着更高的错误率操作系统剪接体。
毫不奇怪,在这三种植物中,观察到的AS频率与EST数量高度相关。高表达基因(含有大量ESTs的基因)更有可能被检测到交替拼接。超过60%和40%的具有超过500个ESTs的基因被交替拼接操作系统而且在,分别。这与人类[42].如果AS异构体出现在至少1%的观察转录本中,则有一半的人类基因可交替拼接,但如果所需的丰富度水平提高到>10%,则只有20%的人类基因可交替拼接[42].这一频率与植物在相同丰度水平下的频率显著相似,表明植物调控的AS事件的频率可能并不显著低于哺乳动物。
拼接错误和功能性AS事件
AS在植物和哺乳动物中的明显区别是植物中IntronR的优势和哺乳动物中的ExonS的优势。两者都是豆科植物的模型,太而且Lj,有40-50%的AS事件是IntronR,这个级别明显低于in在而且操作系统但仍远高于哺乳动物。类似于在而且操作系统[1],短于70 nt的内含子更有可能在豆科植物中保留(数据未显示)。剪接体是一个包含数百种蛋白质的大型动态rna -蛋白质复合体。如果内含子过小,剪接体的组装和结构转化就会受到限制,可能导致剪接效率低下和IntronR [1].由于内含子的大小相当大太而且Lj在豆科植物中,由于空间位阻,内含子会被保留较少,可能导致内含子r在豆科植物中的频率较低。这些数据还表明,一些AS事件可能是拼接错误。正如我们在[1],植物中最常见的剪接错误可能是不能识别和剪接内含子,所以IntronR应该是最常见的AS类型。在哺乳动物中,内含子是通过外显子识别机制定义的,无法识别某些外显子从而跳过它们可能是最常见的错误。因此,外显子是人类最常见的AS类型。
观察到的AS事件是功能性AS事件和拼接错误的混合。其他类型的错误,如测序错误、基因组污染和校准错误,也会导致as事件的预测水平。应用了两个对准程序(GeneSeqer和GMAP),本研究中仅使用常见AS事件以最小化对准误差。通过消除ESTs,保留所有预测的内含子,可以将基因组污染最小化。然而,区分功能性AS事件和拼接错误并不是一件容易的事情。我们试图通过两种方法来实现这一目标。首先,我们选择了具有多个转录本支持的每个亚型的AS事件。由于拼接错误预计会在低频率下发生,它们在两个不同的转录本中被捕获的机会很低。在该数据集中,IntronR的频率略低,但仍然是五种AS类型中最高的,说明IntronR确实是最丰富的调控AS结果。第二种方法是通过跨种EST比较和同源基因比较寻找保守的AS事件。 A few AS events were completely conserved in太,Lj,在而且操作系统.
然而,功能性AS事件可能并不总是保守的。剪接是一个动态过程,需要数百种蛋白质和一些snrna才能准确发挥作用[14].两者都有突变反式- - -独联体-靶基因上的元素会影响剪接模式。根据突变和固定事件发生的时间,功能性AS事件可以在密切相关的物种之间共享,也可以是谱系特异性的。高表达的碳酸酐酶基因(AC124951_11) 3'-UTR的AltD事件可能是豆科植物共有的一个很好的例子。仅从EST数据很难定义特定谱系的功能性AS事件。
ASIP的集中数据场所和标准数据集
随着越来越多的植物基因组和ESTs的测序,未来将发现更多的As事件。重要的是要有一个集中的地方来存储和比较所有的AS数据。在动物系统中,一个综合数据库,ASAP [43]包括了16种动物测序的AS数据,这使得不同动物物种之间的比较非常简单。植物中也需要这样的数据库,因为对剪接信号和替代剪接的研究才刚刚开始。本研究中识别的AS数据已存入PlantGDB的ASIP数据库[37],其中存储了以前的AS数据,可以很容易地进行比较[1].此外,还建立了一个与剪接相关的基因数据库在、动物和酵母可通过PlantGDB的SRGD数据库获得[14,44].在未来,数据库将扩展到操作系统以及其他植物基因组测序,包括太,Lj和杨树。PlantGDB提供的分析程序和植物基因组浏览器应该有助于植物AS数据的深度挖掘。一个核心数据集,其中的AS事件在所有测序植物中都是保守的,这对于理解AS事件的功能,以及这一重要而有趣的现象的信号和调节非常有用。
结论
就像在在而且操作系统AS事件在两种模型豆科植物中也普遍存在太而且Lj.发现了成千上万的AS事件太通过同物种和跨物种EST序列的组合。当est的数量标准化时,在不同的植物物种中交替拼接基因的频率是相似的。与哺乳动物相比,植物被认为具有相对较低的基因交替拼接频率。我们的研究结果表明,这可能部分是由于植物物种中相对较低的EST覆盖率。在讨论的所有五种AS类型中,IntronR在不同的基因亚群中最为丰富,正如先前在在而且操作系统.我们还通过跨物种ESTs比对发现了数百个新颖和保守的AS事件。这是首次在植物中使用跨物种ESTs来探索AS的研究。对于拥有大量EST集合但缺乏基因组序列数据的物种,包括小麦和大麦,将它们的EST与密切相关的参考基因组校准,如操作系统,应该会揭示这些物种的替代剪接。
方法
数据集
的Medicago基因组序列联盟(MGSC) 1.0版本,包括本研究中分析的1826个BACs,从Medicago基因组测序计划网站[45].该组装物共包含186.2 Mb的非冗余基因组序列,估计占整个基因组的38-47%和总基因空间的55-58% [46].本研究中使用的所有其他序列数据集都是截至2006年7月17日的最新序列,这是将BACs纳入Mt1.0基因组组装的截止日期。为Lotus对虾,有1,394个BAC/ tac从NCBI下载[47使用“txid34305 [ORGN:noexp] AND HTG [KYWD]”查询的核苷酸数据库。拟南芥基因组序列和基因注释(TAIR release 6.0)从GenBank FTP网站下载[48],并从TIGR FTP网站下载了水稻基因组序列和基因注释(TIGR release 4.0) [49].
所有EST序列(包括全长cdna)均从GenBank核苷酸数据库中检索。225,920套太和150855年Lj分别使用查询(txid3880 [ORGN] AND "biomol mrna" [PROP])和(txid34305 [ORGN] AND "biomol mrna" [PROP])收集转录本序列。使用查询(txid3847 [ORGN] AND "biomol mrna" [PROP])检索大豆转录本序列(359,834),使用查询(txid3803 [ORGN:exp] NOT txid3880 [ORGN] NOT txid34305 [ORGN] NOT txid3847 [ORGN] AND "biomol mrna" [PROP])检索所有其他豆科植物的127,684个转录本序列。为在,使用查询(txid3702 [ORGN] AND "biomol mrna" [PROP] AND srcdb_ddbj/embl/genbank [PROP])检索到691,516个转录本序列。为操作系统的1,009,574个est粳稻通过查询(txid39947 [ORGN] AND "biomol mrna" [PROP] AND srcdb_ddbj/embl/genbank [PROP])检索品种组。我们有意地将转录本序列从籼稻以减少因两者的差异而可能引起的假阳性比对操作系统cultivar-groups。
转录本与基因组序列的拼接比对
将豆科转录本序列映射到太而且LjBAC集采用GeneSeqer [34]和GMAP [35].GeneSeqer的拼接站点模型设置为Medicago-特定的参数,使用程序选项"-s Medicago"。所有其他选项都使用默认参数。GMAP使用默认对齐参数。为在而且操作系统,仅在本地进行GMAP比对,从PlantGDB下载来自更大数据集的GeneSeqer比对[50].
GMAP和GeneSeqer输出对齐文件通过管道(ASpipe1.0,可通过SourceForge [51)从先前研究中使用的Perl和shell脚本开发而来[1].ASpipe从原始程序输出中提取高质量内含子/外显子/对齐的坐标和分数,并将它们存储在MySQL5.0数据库中。对于同物种EST比对,高质量比对的标准是>95%的序列一致性和>80%的覆盖率(定义为转录本序列与基因组序列对齐的部分)。高同一性(95%)截断最大限度地减少了转录本序列到不完整基因组的错误映射。对于跨物种的转录本比对,身份截断率降低到80%,从不同的转录本序列中选择了可靠的比对。冗余的EST对齐太与Mt1.0提供的非冗余基因表进行比较,去掉[33].>95%和>80%序列标识的外显子分别被认为是同种和跨种映射的可靠标识外显子。两端具有可靠外显子的内含子被认为是可靠识别的内含子。转录单元(TU)被定义为转录本序列映射和聚类的连续基因组区域。注释的基因模型可能包含多个TUs。为太,在而且操作系统,注释的基因作为分析的基础。为Lj在美国,没有基因注释,因此TUs是分析的基础。
识别可选拼接(AS)和保守的AS事件
将可靠的内含子和外显子的坐标以成对的方式进行比较,以确定AS事件的候选位点。在内含子/内含子比较中,如果两个内含子有相同的3'端,但有不同的5'端,则该事件被归类为AltD。如果两个内含子仅在3'端存在差异,则该事件被归类为AltA。AltP事件指的是内含子相互重叠,但5'端和3'端不相同。在内含子/外显子比较中,如果内含子完全被外显子覆盖,则该事件被归类为IntronR。如果一个外显子完全被内含子覆盖,这个事件就被归类为外显子。删除涉及终端外显子的ExonS事件和与ExonS事件相关的AltA/D/P事件。识别和分析AS事件的过程和算法在[1].从跨物种EST比对中鉴定出的AS事件被标记为“跨物种AS事件”。相应的,来自同物种EST对齐的事件被称为“同物种as事件”。
保守的AS事件有两种识别方法:(1)将跨物种AS事件与同物种AS事件和不同物种的跨物种AS事件进行比较;(2)确定之间的同源基因对太而且Lj并比较他们的AS事件。在第一种方法中太比较了多物种est预测的AS事件的基因组坐标。只有在交替处理的内含子/外显子的坐标相同的事件才被认为是完全保守的。在第二种方法中,通过搜索两者的ESTs来识别同源基因太而且Lj基因组。在某些情况下,矫形学在而且操作系统的注释蛋白序列通过互惠BLAST进行鉴定在,太而且操作系统.然后比较同源基因的基因结构和AS事件,以确定保守的AS事件。
缩写
- 阿尔塔:
-
替代受体位点
- AltD:
-
替代捐赠网站
- AltP:
-
可供选择的位置(供体和受体的位置是不同的)。替代拼接
- 美国东部时间:
-
表达序列标记
- 外显子:
-
外显子跳跃
- IntronR:
-
基因内区保留
- Lj:
-
Lotus对虾
- 太:
-
Medicago truncatula
- NMD:
-
nonsense-mediated衰变
- 子:
-
开放式阅读架
参考文献
- 1.
Wang BB, Brendel V:植物选择性剪接的全基因组比较分析。美国国家科学研究院。中国农业科学,2006,29(6):559 - 561。10.1073 / pnas.0602039103。
- 2.
Campbell MA, Haas BJ, Hamilton JP, Mount SM, Buell CR:水稻选择性剪接的综合分析及其与拟南芥的比较分析。中国生物医学工程学报,2006,26(3):327-10.1186/1471-2164-7-327。
- 3.
李文华,李文华,李文华。植物跨种选择性剪接的比较研究。植物生理学报,2004,24(3):366 - 366。10.1104 / pp.107.098640。
- 4.
Kim E, Magen A, Ast G:真核生物中不同水平的选择性剪接。核酸学报,2007,35(1):125-131。10.1093 / nar / gkl924。
- 5.
Gupta S, Zink D, Korn B, Vingron M, Haas SA:基于EST数据的选择性剪接全基因组识别和分类。生物信息学,2004,20:2579-2585。10.1093 /生物信息学/ bth288。
- 6.
Ner-Gaon H, Fluhr R:全基因组微阵列在拟南芥中促进了保留内含子的全局分析。DNA学报,2006,13(3):111-121。10.1093 / dnares / dsl003。
- 7.
阚志勇,王晓燕,王晓燕,王晓燕。跨物种方法检测人与小鼠的新剪接形态。中国生物工程学报,2004,9:42-53。
- 8.
Sugnet CW, Kent WJ, Ares M, Haussler D:人类和小鼠选择性剪接事件的转录组和基因组保存。生物计算。2004,66-77。
- 9.
陈方锋,陈长杰,何建勇,庄廷杰:基于跨物种est -基因组比较的人、小鼠和大鼠新型外显子和可选剪接事件的识别和进化分析。生物信息学。2006,7:136-10.1186/1471- 25-7-136。
- 10.
朱伟,Buell CR:通过比较分析改进谷物全基因组注释。基因组学报,2007,17(3):299-310。10.1101 / gr.5881807。
- 11.
陈芳芳,王士生,周明明,黄东涛,庄廷杰:植物基因及其交替剪接变异注释器。水稻基因的植物基因组注释管道和交替剪接变异鉴定与7种植物的跨种表达序列标记保存。植物生理学报,2007,29(3):366 - 366。10.1104 / pp.106.092460。
- 12.
基因组时代植物前信使rna的选择性剪接。植物学报,2007,32(4):366 - 366。10.1146 / annurev.arplant.58.032806.103754。
- 13.
Reddy ASN:植物核前mrna剪接。植物科学进展,2001,20:523- 523。10.1016 / s0735 - 2689(01) 80004 - 6。
- 14.
Wang BB, Brendel V: ASRG数据库:拟南芥pre-mRNA剪接相关基因的鉴定与研究。中国生物医学工程学报,2004,5 (12):R102-10.1186/gb-2004-5-12-r102。
- 15.
Palusa SG, Ali GS, Reddy AS:拟南芥富含丝氨酸/精氨酸蛋白的前mrna的选择性剪接:激素和应激的调节。植物学报,2007,29(6):369 - 369。
- 16.
Kalyna M, Lopato S, Voronin V, Barta A:植物SR蛋白中特定剪接事件的进化保护和调控。核酸学报,2006,34(16):4395-4405。10.1093 / nar / gkl570。
- 17.
Iida K, Go M:陆地植物SR蛋白mrna的保守选择性剪接事件的调查。分子生物学与病原学杂志,2006,23(5):1085-1094。10.1093 / molbev / msj118。
- 18.
Isshiki M, Tsumoto A, Shimamoto K:水稻中富含丝氨酸/精氨酸的蛋白质家族在pre-mRNA的组成和选择性剪接中起着重要作用。植物学报,2006,18(1):146-158。10.1105 / tpc.105.037069。
- 19.
Gupta S, Wang BB, Stryker GA, Zanetti ME, Lal SK:玉米中两种新的精氨酸/丝氨酸(SR)蛋白是差异剪接的,并利用非典型剪接位点。生物化学学报2005,1728(3):105-114。
- 20.
Gao H, Gordon-Kamm WJ, Lyznik LA: ASF/ sf2样玉米pre-mRNA剪接因子影响剪接位点的利用,其转录本交替剪接。中国生物技术学报,2004,25(3):326 - 326。10.1016 / j.gene.2004.06.047。
- 21.
王斌,Brendel V:植物中U2AF35同源物的分子特征及其系统发育。植物生理学报,2006,26(2):369 - 369。10.1104 / pp.105.073858。
- 22.
Golovkin M, Reddy AS:植物单核苷酸snRNP 70K基因的结构和表达:单核苷酸snRNP 70K pre- mrna的选择性剪接产生两种不同的转录本。植物生态学报,1998,8(8):1421-1435。10.1105 / tpc.8.8.1421。
- 23.
Gupta S, Ciungu A, Jameson N, Lal SK: U1 snRNP 70K基因的选择性剪接表达在不同植物物种间具有进化保守性。中国生物医学工程学报,2006,29(4):359 - 361。10.1080 / 10425170600856642。
- 24.
周勇,周超,叶玲,董娟,徐辉,蔡亮,张亮,魏亮:植物已知交替剪接基因的数据库及分析。中国生物医学工程学报,2003,26(6):563 - 566。10.1016 / s0888 - 7543(03) 00204 - 0。
- 25.
Ramos J, Clemente MR, Naya L, Loscos J, Perez-Rontome C, Sato S, Tabata S, Becana M:模式豆科植物japonicus的植物螯合肽合成酶。一个小的多基因家族,对镉有不同的反应和交替剪接变异。植物生理学报,2004,25(3):366 - 366。10.1104 / pp.106.090894。
- 26.
Szczyglowski K, Kapranov P, Hamburger D, de Bruijn FJ:荷花LjNOD70结节蛋白基因编码一种与转运蛋白相似的蛋白质。植物生物学杂志,1998,37(4):651-661。10.1023 /: 1006043428636。
- 27.
Horst I, Welham T, Kelly S, Kaneko T, Sato S, Tabata S, Parniske M, Wang TL:荷花TILLING突变体揭示了在无结节增强的蔗糖合酶条件下氮素的同化和固定。植物物理。2007
- 28.
郑淑贞,杨凯,朴济勇,韩克思,俞硕,黄泰,Hur CG, Kim SH, Park PB, Kim HM, Park YI, Liu JR:利用公共大豆EST数据库鉴定的两个结节特异性基因的结构、表达和定位。基因学报,2006,36(4):371 - 380。10.1016 / j.gene.2006.07.015。
- 29.
熊田芳,叶志勇,张志军,张志军,张志军,张志军,张志军,张志军:菜豆淀粉分枝酶基因的克隆及亚细胞定位研究。中国生物医学工程学报,2004,22(4):369 - 369。10.1074 / jbc.M110497200。
- 30.
Neumann P, Pozarkova D, Macas J:高度丰富的豌豆LTR逆转录转座子Ogre的组成转录和部分剪接。植物生物学杂志,2003,23(3):369 - 371。10.1023 / B: PLAN.0000006945.77043.ce。
- 31.
Young ND, Cannon SB, Sato S, Kim D, Cook DR, Town CD, Roe BA, Tabata S: Medicago truncatula和Lotus japonicus的基因空间测序。植物生理学报,2004,25(4):369 - 369。10.1104 / pp.104.057034。
- 32.
城镇CD:截断紫花苜蓿基因组注释。植物生物学杂志,2006,9(2):122-127。10.1016 / j.pbi.2006.01.004。
- 33.
Medicago基因组序列发布1.0。[http://www.medicago.org/genome/downloads/Mt1/]
- 34.
Brendel V,邢丽,朱伟:基于一致拼接比对的基因结构预测。生物信息学,2004,20(7):1157-1169。10.1093 /生物信息学/ bth058。
- 35.
Wu TD, Watanabe CK: GMAP:一种用于mRNA和EST序列的基因组定位和对齐程序。生物信息学,2005,21(9):1859-1875。10.1093 /生物信息学/ bti310。
- 36.
GJ, Filipowicz W:单子叶和双子叶植物内含子核苷酸组成和二级结构对pre-mRNA剪接的不同影响。中国地质大学学报(自然科学版),2011,30(4):369 - 369。
- 37.
植物选择性剪接(ASIP)。[http://www.plantgdb.org/ASIP/]
- 38.
Johnson JM, Castle J, Garrett-Engele P, Kan Z, Loerch PM, Armour CD, Santos R, Schadt EE, Stoughton R, Shoemaker DD:用外显子结微阵列对人类替代pre-mRNA剪接的全基因组调查。科学通报,2003,32(5):533 - 536。10.1126 / science.1090100。
- 39.
Brett D, Pospisil H, Valcarcel J, Reich J, Bork P:选择性剪接和基因组复杂性。植物学报,2002,30(1):29-30。10.1038 / ng803。
- 40.
Alexandrov NN, Troukhan ME, Brover VV, Tatarinova T, Flavell RB, Feldmann KA:利用全长cdna发现拟南芥基因和基因组的特征。植物生物学杂志,2006,60(1):69-85。10.1007 / s11103 - 005 - 2564 - 9。
- 41.
Lewis BP, Green RE, Brenner SE:选择性剪接和无意义介导的mRNA衰变在人类中广泛耦合的证据。美国国家科学研究院。2003, 100(1): 189-192。10.1073 / pnas.0136770100。
- 42.
阚卓,张志刚,王晓燕。est中功能替代剪接的选择。中国生物医学工程学报,2004,12(12):1837-1845。10.1101 / gr.764102。
- 43.
Kim N, Alekseyenko AV, Roy M, Lee C: ASAP II数据库:15种动物的选择性剪接分析和比较基因组学。核酸学报,2007,D93-98。10.1093 / nar / gkl884。35数据库
- 44.
剪接相关基因数据库。[http://www.plantgdb.org/SRGD]
- 45.
Medicago基因组测序项目。[http://www.medicago.org/genome/]
- 46.
Medicago基因组序列发布1.0白皮书。[http://www.medicago.org/genome/downloads/Mt1/Mt1.0.pdf]
- 47.
国家生物技术信息中心(NCBI)。[http://www.ncbi.nlm.nih.gov/]
- 48.
NCBI拟南芥基因组序列FTP站点。[ftp://ftp.ncbi.nih.gov/genomes/Arabidopsis_thaliana/]
- 49.
TIGR水稻基因组序列发布4.0 FTP网站。[ftp://ftp.tigr.org/pub/data/Eukaryotic_Projects/o_sativa/annotation_dbs/pseudomolecules/version_4.0/]
- 50.
董q, Lawrence CJ, Schlueter SD, Wilkerson MD, Kurtz S, Lushbough C, Brendel V: PlantGDB植物基因组资源比较。植物生理学报,2004,24(2):369 - 369。10.1104 / pp.104.059212。
- 51.
SourceForge的ASpipe项目。[https://sourceforge.net/projects/aspipe/]
确认
BBW和MOT得到了国家科学基金会拨款DBI-0321460和DBI-0606966对纽约的支持。本研究中产生的数据存放在PlantGDB的ASIP数据库中,并可通过该数据库公开获取[37],由NSF拨款DBI-0606909资助VB。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
BBW构思了这项研究,进行了研究,分析了数据并起草了手稿。交通部参与了数据分析和网页制作。VB参与了数据分析和演示,并帮助起草了稿件。NDY参与了本研究的设计,协调了数据分析,并帮助起草了稿件。所有作者阅读并批准了最终稿件。
电子补充材料
12870 _2007_225_moesm1_esm.pdf
补充文件1:补充图和表。此pdf文件包含补充的数字和表的主要稿件。(pdf 135kb)
权利与权限
开放获取本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(https://creativecommons.org/licenses/by/2.0),它允许在任何媒体上不受限制地使用、分发和复制,只要原著被恰当地引用。
关于本文
引用本文
王,BB。,O'Toole, M., Brendel, V.et al。跨种EST序列揭示了豆科植物中新颖和保守的可选剪接事件。植物生物学8日,17(2008)。https://doi.org/10.1186/1471-2229-8-17
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-17
关键字
- 选择拼接
- 细菌人工染色体
- 豆科物种
- 记录序列
- 基因内区保留