- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
水稻副同源蛋白质家族的特征gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba8gydF4y2Ba,文章号:gydF4y2Ba18gydF4y2Ba(gydF4y2Ba2008gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
植物基因组中的高基因数反映了多倍体和主要基因复制事件。gydF4y2Ba栽培稻gydF4y2Ba水稻是一种二倍体单子叶植物,基因组约390 Mb,其基因组的很大一部分经历了分段复制。这一点,再加上其他遗传事件,如串联复制,导致了大量的基因,并由此产生的蛋白质,发生在副同源家族。gydF4y2Ba
结果gydF4y2Ba
利用利用Pfam和新的蛋白质结构域的计算管道,我们对水稻的副同源科进行了表征,并将其与双子叶二倍体模型物种的副同源科进行了比较。gydF4y2Ba拟南芥gydF4y2Ba.拟南芥也经历了基因组复制,与水稻相比,其基因组和基因补体要小得多(约120 Mb)。总体而言,水稻和拟南芥非转座元件相关的蛋白质分别有53%和68%可归入副同源蛋白质家族。单基因和副同源家族基因在编码已知或假定功能的蛋白质的可能性上存在很大差异;在水稻和拟南芥中,分别有26%和66%的单例基因编码一种已知或假定的蛋白质,而73%和96%的副同源家族基因编码一种已知或假定的蛋白质。此外,还观察到特定基因功能分布的主要倾斜;水稻和拟南芥共有17个基因本体论类别在副同源家族和单例蛋白之间的差异分布具有统计学意义。与哺乳动物生物相比,我们发现水稻和拟南芥中的复制基因往往有更多可选择的剪接形式。利用大规模并行特征测序(Massively Parallel Signature Sequencing)的数据,我们发现水稻中大量重复基因表现出发散性表达,尽管在非常年轻的基因中可以看到序列发散和表达相关性之间的相关性。gydF4y2Ba
结论gydF4y2Ba
总的来说,这些数据表明,虽然在一些副同源蛋白家族成员中存在共同调节和保守功能,但进化压力导致了不同表达模式的功能分歧。gydF4y2Ba
背景gydF4y2Ba
基因复制是基因新颖性和蛋白质组复杂性的主要贡献者。复制基因的进化压力不同于单拷贝(单例)基因,已经提出了几个模型来描述复制基因的进化命运。在非/新功能化模型中,其中一个复制基因通过有害突变的积累成为假基因,尽管在极少数情况下,它可能获得新的功能[gydF4y2Ba1gydF4y2Ba].在亚功能化模型中[gydF4y2Ba2gydF4y2Ba- - - - - -gydF4y2Ba4gydF4y2Ba],复制的基因采用了祖先基因的功能子集。重复基因的功能冗余已被证明可增加生物系统的稳健性[gydF4y2Ba5gydF4y2Ba].gydF4y2Ba
基因复制在植物中经常发生,其形式有节段复制、串联复制,也有全基因组复制[gydF4y2Ba6gydF4y2Ba- - - - - -gydF4y2Ba14gydF4y2Ba].水稻的基因组复制已被报道(gydF4y2Ba栽培稻gydF4y2Ba),是重要的农业种,也是禾草科的模式种[gydF4y2Ba15gydF4y2Ba- - - - - -gydF4y2Ba19gydF4y2Ba].根据所使用的方法、参数和基因组组合,15%至62% [gydF4y2Ba15gydF4y2Ba- - - - - -gydF4y2Ba19gydF4y2Ba]的水稻基因组经历了大约7000万年前(MYA)的一轮大规模片段复制[gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba18gydF4y2Ba].在11号和12号染色体的短臂上,最近的一次重复大约发生在5 ~8 MYA [gydF4y2Ba15gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba].在串联复制方面,根据所利用的参数不同,14-29%的水稻基因是串联发生的[gydF4y2Ba21gydF4y2Ba].由串联和分段重复的基因组成的副同源家族在水稻中已进行了有限的研究,特别是在与双子叶植物物种的完整基因组进行比较的情况下,gydF4y2Ba拟南芥gydF4y2Ba[gydF4y2Ba22gydF4y2Ba- - - - - -gydF4y2Ba27gydF4y2Ba].迄今为止,在水稻中只报道了有限的副同源蛋白质家族的全基因组分析[gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba].在霍兰gydF4y2Ba等gydF4y2Ba.[gydF4y2Ba28gydF4y2Ba],拟南芥和水稻蛋白质通过基于Pfam结构域或基于blastp的相似性聚类进行共聚类,这可以将蛋白质聚类到这两个模式种之间的共同家族中,并用于鉴定种特异性蛋白质。gydF4y2Ba
在这项研究中,我们利用基于Pfam和blastp的新结构域的计算管道,将来自预测的水稻蛋白质组的蛋白质分类为副同源蛋白质家族[gydF4y2Ba30.gydF4y2Ba].虽然我们的研究重点是对水稻副同源科的分析,但为了进行比较,我们对预测的拟南芥蛋白质组进行了类似的分类,以比较和对比代表被子植物两大分支单子叶和双子叶的两个模式种的副同源科组成和特征。在水稻中,我们描述了选择性剪接、副同源家族蛋白质的功能分类、表达模式和复制年龄,并将这些数据与在单拷贝蛋白质中观察到的数据进行了比较。以拟南芥为研究对象,平行分析了副同源家族蛋白质的选择性剪接和功能域组成,并与水稻的研究结果进行了比较。为了突出我们的观察结果,我们深入研究了两个水稻蛋白家族,prolamin和Bowman-Birk抑制剂。本研究对水稻副同源基因家族进行了综合分析,并对拟南芥的副同源基因家族进行了比较分析,为研究这两种模式植物的副同源基因家族进化提供了新的视角。gydF4y2Ba
结果与讨论gydF4y2Ba
水稻和拟南芥副同源蛋白家族的分类gydF4y2Ba
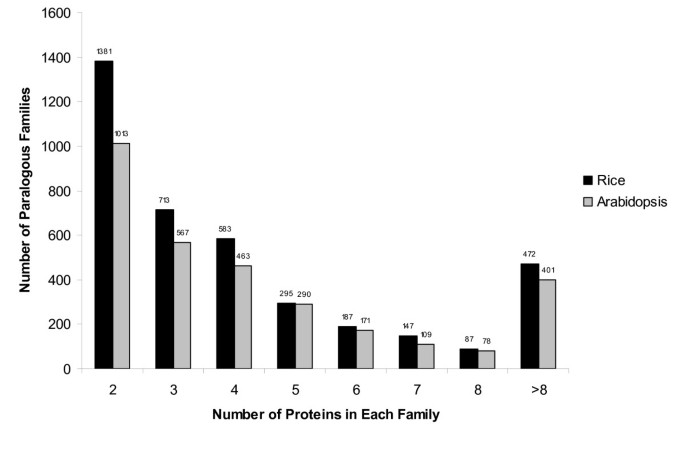
共鉴定出3,865个副同源蛋白质家族,包含21,998个蛋白质[见附加文件]gydF4y2Ba1gydF4y2Ba]从水稻基因组中预测的42,653个总非转座因子(TE)相关蛋白中,剩下20,655个假定的由单拷贝基因编码的单例蛋白。平均而言,一个水稻家族包含6个家庭成员,规模从2到214个家庭成员不等。gydF4y2Ba1gydF4y2Ba).在水稻中共鉴定出11个有超过100个成员蛋白的副同源蛋白家族,它们编码锌指蛋白、蛋白激酶、myb样蛋白和转导蛋白等蛋白质[见附加文件]gydF4y2Ba2gydF4y2Ba],与拟南芥中报道的最大蛋白质家族相似[gydF4y2Ba30.gydF4y2Ba].水稻的副同源蛋白家族基因分布在整个基因组和染色体内,其模式与单例基因相似[见附加文件]gydF4y2Ba3gydF4y2Ba].虽然副同源蛋白家族基因更频繁地位于染色质区,但这与之前的报道一致,即非te相关基因更普遍地存在于染色质区。段复制基因与副同源蛋白家族基因的比较表明,我们的分类管道是稳健的。163个片段重复区块内的2403个片段重复基因对中,1570个重复基因对(65%)被归类于同一副同源蛋白家族。对于其余的片段重复基因,175对(7%)被分类在不同的副同源蛋白家族,268对(11%)被分类在副同源蛋白家族,另一个基因被分类为单基因。我们观察到390个片段重复的基因对(16%)不包含在任何副同源蛋白家族中。请注意,在我们的计算管道中,需要四个或更多成员来定义一个基于blastp的域。因此,单对片段复制的基因不足以定义一个基于blastp的结构域。片段复制和副同源家族分类之间缺乏100%的对应关系可能是由于在一个重复的基因中获得了新的结构域或失去了现有的结构域。在我们的计算管道中,只有具有相同结构域组成的蛋白质才被归类到相同的副同源蛋白质家族。或者,这种差异可能是由于每种方法采用了不同的分类方法。 For example, LOC_Os08g37350 and LOC_Os09g28940 are segmentally duplicated genes from chromosomes 8 and 9, respectively. These two protein sequences had a 56% identity over 70% of the length of the longer sequence and were within a segmentally duplicated block of 43 collinear gene pairs. LOC_Os08g37350 has two Pfam domains (PF00443: Ubiquitin carboxyl-terminal hydrolase; PF01753: MYND finger) while LOC_Os09g28940 has only one Pfam domain (PF00443: Ubiquitin carboxyl-terminal hydrolase). As a consequence, these loci were classified in two different paralogous families (LOC_Os08g37350 is classified in Family 1545; LOC_Os09g28940 is in Family 3650). In a second example, LOC_Os11g03210 and LOC_Os12g02960 are from a segmental duplication event involving chromosomes 11 and 12 which includes 160 collinear gene pairs. LOC_Os11g03210 has a single Pfam domain (PF02798: Glutathione S-transferase, N-terminal domain) and thus is classified in Family 3362 while LOC_Os12g02960 is classified as a singleton as although it has two Pfam domains (PF02798: Glutathione S-transferase, N-terminal domain; PF00043: Glutathione S-transferase, C-terminal domain) no other protein has exactly the same domain profile. Note that in our computational pipeline, a paralogous family must have at least two members with identical domain profiles. In a third example, segmentally duplicated genes LOC_Os01g41900 and LOC_Os05g51160 are from chromosomes 1 and 5. These two genes were derived from full length cDNAs (FLcDNAs) and had a 59% identity over approximately three-quarters of the longer protein sequence. LOC_Os01g41900 has two Pfam domains (PF00249: Myb-like DNA-binding domain and PF00098: Zinc knuckle) while LOC_Os05g51160 has only one single Pfam domain (PF00249: Myb-like DNA-binding domain). As a consequence, they were classified in different families, Family 1452 and Family 3863, respectively. Manual inspection of these three sets of loci revealed that they were correctly annotated and that the lack of clustering into a single paralogous family could not be attributed to incorrect structural annotation which is another potential cause for lack of 100% correspondence between segmentally duplicated genes and paralogous families.
拟南芥平行构建了3092个副同源蛋白家族(18183个蛋白),从TAIR7释放的26819个蛋白编码基因中鉴定出8636个单拷贝基因[gydF4y2Ba31gydF4y2Ba].拟南芥蛋白质家族也有相似的大小分布,从2到182不等。gydF4y2Ba1gydF4y2Ba).在拟南芥中,最大的家族编码myb样蛋白、锌指蛋白和蛋白激酶,与先前报道的一致[gydF4y2Ba30.gydF4y2Ba].拟南芥副同源蛋白家族基因的分布与单基因相似,并且更多地位于染色质区域[见附加文件]gydF4y2Ba3 bgydF4y2Ba].gydF4y2Ba
水稻和拟南芥副同源蛋白家族的功能gydF4y2Ba
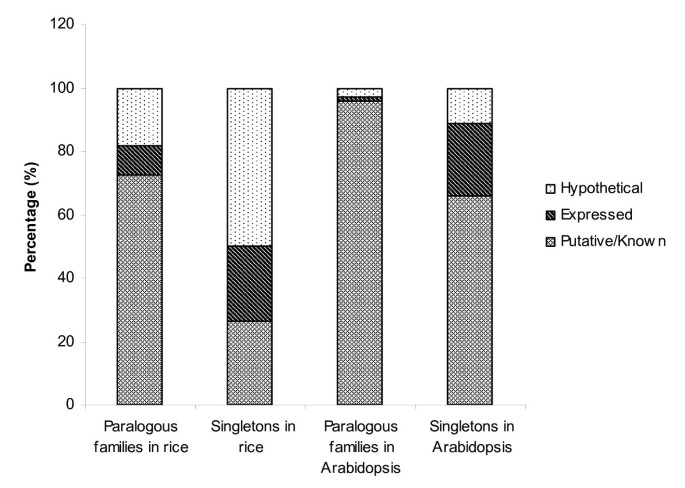
我们研究了副同源家族和单例蛋白的功能注释。在水稻和拟南芥中,共有21403个和23081个基因被注释为编码已知或假定的蛋白质,这是因为它们与功能已知的蛋白质具有很强的相似性,或者在可信边界以上存在Pfam结构域。没有已知或假定功能的基因可以由实验转录证据支持(即编码一种“表达蛋白”)或仅由基因预测gydF4y2Ba从头开始gydF4y2Ba除了其他假设蛋白质(即编码一个“假设蛋白质”)之外,基因查找器和缺乏表达支持以及与已知蛋白质的序列相似性。在水稻中,共有6913个基因编码表达蛋白,从表达序列标签(ESTs)、flcdna、大规模并行签名测序[gydF4y2Ba32gydF4y2Ba],基因表达序列分析和/或蛋白质组学数据[gydF4y2Ba33gydF4y2Ba].在拟南芥中,2270个基因编码了以ESTs和/或cDNA证据形式的实验转录本所表达的蛋白质(见方法)。其余14,337个水稻基因[gydF4y2Ba33gydF4y2Ba]和1468个拟南芥基因(见方法)编码假设的蛋白质。大多数水稻副亲缘家族基因(73%)编码一种已知或假定的蛋白质。gydF4y2Ba2gydF4y2Ba).剩余的水稻副亲缘家族基因编码表达蛋白(9%)和假设蛋白(18%)。相比之下,水稻单倍体的假设基因比例较大(50%),而已知或假定功能的基因比例较小(26%)。尽管拟南芥总体上具有比水稻更少的未知功能基因,但在拟南芥的平行分析中,可以观察到在副亲缘家族基因中具有已知或假定功能的基因的类似偏倚(图2)。gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
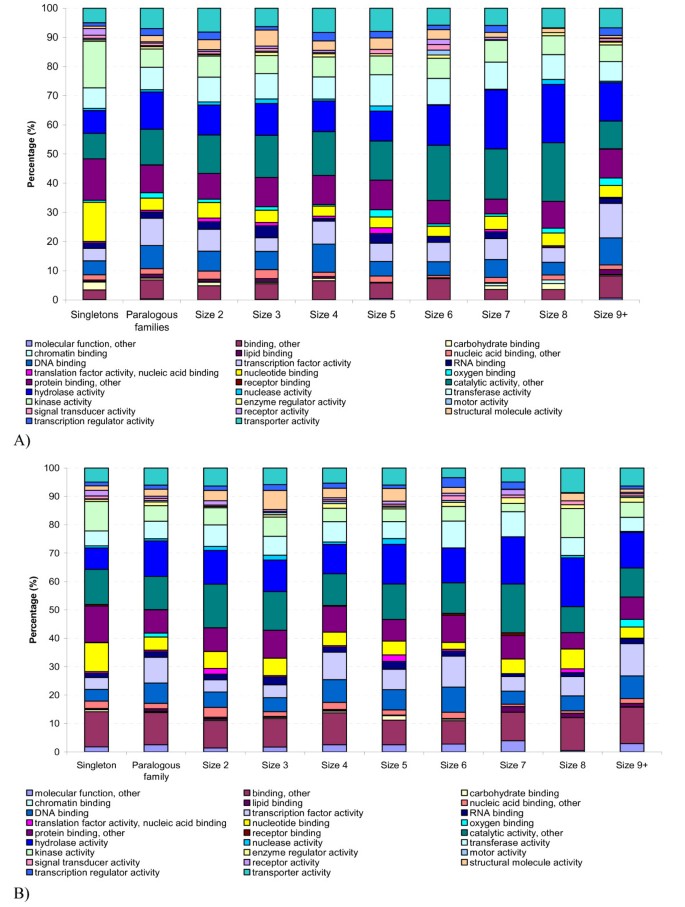
使用Plant GOSlim注释[gydF4y2Ba34gydF4y2Ba],我们比较了水稻副亲缘科的蛋白质功能与单子代的蛋白质功能。在我们的分析中确定的26个分子功能GOSlim类别中,水稻副同源蛋白家族在许多GOSlim类别中表现出与单子代不同的模式(图2)。gydF4y2Ba3gydF4y2Ba).虽然每个GOSlim类别的相对丰度随着水稻副亲缘科的大小而变化,但没有观察到明显的相关性(图2)。gydF4y2Ba3gydF4y2Ba).对每一类水稻,采用双尾双样本二项检验,比较该类水稻在副亲缘科中的丰度与单胎的丰度。多重测试使用Benjamini和Hochberg错误发现率控制在0.05水平上进行校正[gydF4y2Ba35gydF4y2Ba].统计检验显示,水稻副同源家族蛋白质中有12个类别的蛋白质显著富集,包括转录因子活性、水解酶活性、DNA结合和转运蛋白活性,而受体活性、核苷酸结合和碳水化合物结合等5个类别的蛋白质则显著降低(表2)gydF4y2Ba1gydF4y2Ba).在拟南芥的平行分析中也观察到类似的GOSlim类别的倾斜(表2)gydF4y2Ba2gydF4y2Ba和无花果。gydF4y2Ba3 bgydF4y2Ba),与之前关于拟南芥的报道一致[gydF4y2Ba36gydF4y2Ba]表明在基因复制后,具有不同功能的副同源基因会发生非随机的丢失和保留。gydF4y2Ba
副同源蛋白家族基因往往比单基因有更多的可选异构体gydF4y2Ba
选择性剪接被认为是增加遗传新颖性的一种机制。在水稻基因组中,有6253个非te相关基因有选择性剪接的证据(见方法),我们使用这组基因来检测单例蛋白家族和副同源蛋白家族基因的选择性剪接。单拷贝基因的选择性剪接比例为2094 / 20655 = 10.1%,而副同源家族基因的选择性剪接比例为4159 / 21998 = 18.9%;统计上有显著差异(gydF4y2BaχgydF4y2Ba2gydF4y2Ba检验,P < 1e-5)。为了消除由于缺乏转录证据的基因造成的任何偏差,我们将分析限制在具有EST和/或FLcDNA证据的基因上。单子中选择性剪接的比例为2094 /8,619 = 24.3%,而副同源蛋白家族基因中选择性剪接的比例为4159 /14,072 = 29.6%;统计上有显著差异(gydF4y2BaχgydF4y2Ba2gydF4y2Ba检验,P < 1e-5)。我们进一步将分析限制在结构完全由ESTs和/或flcdna支持的高置信度基因上。单子中选择性剪接的比例增加到1826 / 5964 = 30.6%,而副同源蛋白家族基因中选择性剪接的比例增加到3765 / 11235 = 33.5%;统计上有显著差异(gydF4y2BaχgydF4y2Ba2gydF4y2Ba检验,P < 1e-3)。gydF4y2Ba
为了证实我们的观察并不局限于水稻,我们对拟南芥进行了平行分析。根据TAIR7版本提供的选择性剪接数据(见方法),拟南芥单拷贝基因的选择性剪接比例为943/ 8636 = 9.8%,而副同源蛋白家族基因的选择性剪接比例为2856 / 18183 = 15.7%。这种差异在统计上也很显著(gydF4y2BaχgydF4y2Ba2gydF4y2BaP < 1e-5),与水稻相似。仅对TAIR7版本提供的EST和/或cDNA支持的拟南芥基因进行分析,结果表明,单株中选择性剪接的比例为942/ 6663 = 14.1%,而副亲缘家族中选择性剪接的比例为2852 / 15369 = 18.6%;统计上有显著差异(gydF4y2BaχgydF4y2Ba2gydF4y2Ba检验,P < 1e-5)。我们的发现与先前关于模型动物物种的报道相矛盾,在这些报道中,复制的基因往往具有较少的可选剪接异构体,从而支持了“功能共享模型”,即可选剪接和基因复制是蛋白质组功能多样性方面互补的两种机制[gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba].我们的结果表明,植物可能采用多种机制的蛋白质组复杂性,基因复制和选择性剪接。gydF4y2Ba
水稻副同源蛋白质家族的年龄gydF4y2Ba
虽然以前有关于水稻基因复制的报道[gydF4y2Ba15gydF4y2Ba- - - - - -gydF4y2Ba19gydF4y2Ba],他们利用水稻基因组的替代组装和注释数据集。为了提供本研究中确定的副亲缘家庭的年龄信息,我们根据成对分布的最大值估计了副亲缘家庭的年龄gydF4y2BadgydF4y2Ba年代gydF4y2Ba在该蛋白质家族的所有成员中计算(见方法)。我们发现大多数类人猿家族的起源可以追溯到超过1.15亿年前(MY),在这一点上,同义位点已经饱和,而年代测定就变得不可靠了(gydF4y2BadgydF4y2Ba年代gydF4y2Ba~1.5)[参见附加文件gydF4y2Ba4gydF4y2Ba].在蛋白质家族中,以两两最大gydF4y2BadgydF4y2Ba年代gydF4y2Ba值小于1.5时,分布最大gydF4y2BadgydF4y2Ba年代gydF4y2Ba相当平稳,除了最近在gydF4y2BadgydF4y2Ba年代gydF4y2Ba介于0和0.1之间[参见附加文件gydF4y2Ba4 bgydF4y2Ba].这表明,在过去的115个地球周期内,副同源家族一直在以相对稳定的速度出现,但在最近的7.5个地球周期内发生了一次重复的爆发。另外,副同源家族的出现速度与前几百万年观察到的相似,但其中约2/3在那之后很快恢复为单基因状态,解释了第一个7.5 MY之后的快速下降。较老的副类群数量相当稳定可能是由于选择性限制维持了较高的拷贝数,或者如果副类群的损失依赖于序列相似性,因此在~10%的序列分歧后,副类群的损失可以忽略不计。最后,对于每个家庭,我们确定了所有成对分布中小于1.5的最大峰值(如果有的话)gydF4y2BadgydF4y2Ba年代gydF4y2Ba值。这个峰值在所有家庭中的分布是双峰的[见附加文件gydF4y2Ba5gydF4y2Ba],证实存在大量最近重复的基因(0≤gydF4y2BadgydF4y2Ba年代gydF4y2Ba< 0.1)。此外,峰值在0.7≤gydF4y2BadgydF4y2Ba年代gydF4y2Ba≤1很可能是发生在~70 MYA的大规模片段复制事件的结果。gydF4y2Ba
水稻副同源蛋白家族的表达gydF4y2Ba
我们利用来自18个文库的MPSS数据进一步研究了副同源家族的表达模式[gydF4y2Ba32gydF4y2Ba].MPSS标签根据我们发布的4个假分子和所有注释基因模型的cDNA序列进行搜索,以确保所有MPSS标签即使跨越内含子也能被识别。我们在副同源蛋白家族中发现了11619个基因,这些基因与独特、可靠和重要的MPSS标记相关,这些基因被称为MPSS限定基因。gydF4y2Ba
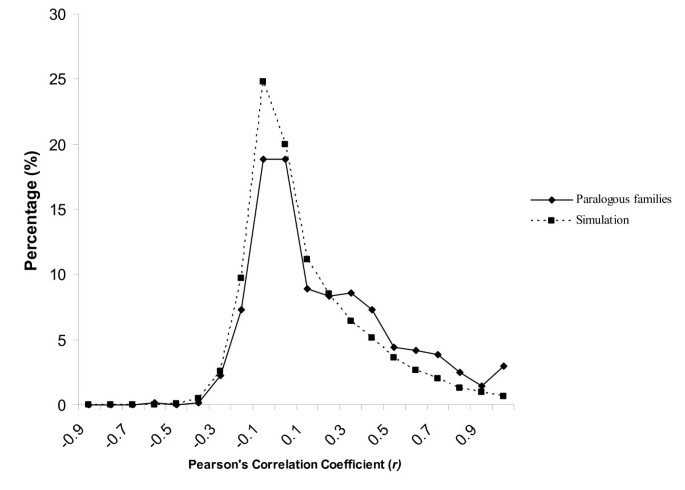
一个基因家族表达差异的相关统计可以在Gu [gydF4y2Ba39gydF4y2Ba]和GugydF4y2Ba等gydF4y2Ba.[gydF4y2Ba40gydF4y2Ba],尽管微阵列数据是这些研究的主要焦点。简而言之,我们将文库和组织中的表达相关性分析限制在具有恰好两个mpss资格基因的副同源家族(674个蛋白质家族)。为了衡量表达的相关性,皮尔逊相关系数(gydF4y2BargydF4y2Ba)对所有18个MPSS文库中674个蛋白质家族中的每一对MPSS资格基因进行表达计算。值得注意的是,我们排除了映射到多个位置的MPSS标签,因为其中大多数标签可能与密切相关的谬误线匹配,并可能使我们的分析混淆。我们采用了勃朗克和沃尔夫[gydF4y2Ba36gydF4y2Ba]以确定皮尔逊相关系数的最小截断值(gydF4y2BargydF4y2Ba)将两个重复的基因划分为表达发散的基因。基本上,通过随机洗牌单基因和皮尔逊相关系数(gydF4y2BargydF4y2Ba),每对的计算方法相似。95%的随机洗牌基因对具有相关值gydF4y2BargydF4y2Ba< 0.59。由于随机打乱的基因对应该具有不同的功能和表达模式,我们利用gydF4y2BargydF4y2Ba< 0.59为发散性表达的指标。我们的结果表明,表达式相关值(gydF4y2BargydF4y2Ba)的变异范围为-0.6 ~ 1.0,但大多数基因对的相关性较小gydF4y2BargydF4y2Ba峰值在-0.2 ~0,这与单子的观测结果相似(图2)。gydF4y2Ba4gydF4y2Ba).使用相关截止(gydF4y2BargydF4y2Ba= 0.59),共有598个(89%)具有两个符合条件的MPSS基因的副同源蛋白家族表现出不同的表达模式,与拟南芥中报道的一致[gydF4y2Ba36gydF4y2Ba]在酵母中,超过80%的旧复制基因对(gydF4y2BadsgydF4y2Ba> 1.5)表达出现分歧[gydF4y2Ba41gydF4y2Ba].gydF4y2Ba
为了更好地了解副同源蛋白家族成员在不同器官/组织中的表达模式,我们对18个MPSS文库进行了分类[gydF4y2Ba32gydF4y2Ba]按器官/组织分为四类:根、叶、生殖器官/组织和“其他组织”。在674个具有恰好两个mpss资格基因的副同源家族中,239个、168个、223个和200个副同源家族中只有一个成员分别在根、叶、生殖器官/组织和“其他组织”中表达,这表明它们的表达模式不同,可能存在组织特异性表达。为了进一步研究副同源蛋白家族成员的组织特异性或胁迫诱导的表达模式,我们计算了18个MPSS文库中来自674个副同源家族(见方法)的1348个基因的优先表达量(PEM)。PEM表示给定组织/处理中观察到的表达水平与假设所有组织/处理中表达一致的预期表达水平之比的base-10对数。PEM值为1意味着在给定组织/治疗中观察到的表达水平是预期的10倍,表明强烈的组织特异性表达。对于每个基因,将PEM严格截断值≥1的组织与复制基因对的其他成员进行比较。共有375个(375/674 = 55.6%)的副同源家族表现出很少的组织特异性表达,因为没有一个相关的PEMs值等于或大于1。299个家族表现出强烈的组织特异性表达模式;19个家族优先在同一组织或处理中表达,49个家族优先在不同组织或处理中表达,231个家族只有一个重复基因优先在组织特异性表达。gydF4y2Ba
我们进一步研究了表达发散和序列发散之间的相关性。对于每个家庭,我们计算皮尔逊相关系数(gydF4y2BargydF4y2Ba)对所有可能的mpss鉴定基因对进行检测,以测量表达差异。然后我们用gydF4y2BadsgydF4y2Ba作为每个基因对发散时间的代表。我们把分析限制在gydF4y2BadgydF4y2Ba年代gydF4y2Ba≤1.5,使同义站点不饱和。皮尔逊相关系数(gydF4y2BargydF4y2Ba)的值与gydF4y2BadgydF4y2BaS值为0.1的每个间隔,以获得更好的分辨率。也就是说,我们绘制了0 <的基因对gydF4y2BadgydF4y2Ba年代gydF4y2Ba≤0.1,0.1
副同源蛋白家族成员之间的表达模式呈正相关,表明两个成员之间保留了相似的转录调控,可能具有相似的功能。然而,我们观察到大量表达相关性很小的基因对,这可能是基因复制后亚功能化或新功能化的标志。Force等提出的重复-退化-互补(DDC)模型[gydF4y2Ba3.gydF4y2Ba]林奇和福斯[gydF4y2Ba4gydF4y2Ba]表明亚功能化是复制基因保留的主要机制,这是由调控区而不是蛋白质编码区突变积累引起的差异表达的结果。在两种不同的组织或处理中优先表达的49个家族,以及仅优先表达副同源对中的一个成员的231个家族,是亚功能化的强烈指标。由于我们的副同源蛋白家族分类要求每个家族成员具有相同的结构域轮廓,差异表达可能归因于调控区域的突变,而不是基因编码区域的突变,这与DDC模型一致。gydF4y2Ba
水稻副同源蛋白质家族的个案研究gydF4y2Ba
Prolamin蛋白家族gydF4y2Ba
Prolamin是小麦、大麦、黑麦、玉米和高粱等谷物的主要胚乳储存蛋白质之一[gydF4y2Ba43gydF4y2Ba- - - - - -gydF4y2Ba46gydF4y2Ba].由于其脯氨酸和谷氨酰胺含量高,被命名为脯氨酸。在水稻中,脯氨酸蛋白占总种子蛋白的35% [gydF4y2Ba47gydF4y2Ba].三种类型的prolamin已被鉴定gydF4y2Ba选用gydF4y2Ba按分子量计:10、13及16 kDa [gydF4y2Ba48gydF4y2Ba].水稻中主要的prolamin家族是3722家族(20名成员)和3193家族(7名成员)。两个家族的成员都有一个基于blastp的域。家族3193的成员有一个Pfam域(PF00234;蛋白酶抑制剂/种子存储/LTP家族),除了常见的基于blastp的结构域之外,因此没有聚集在3722家族中,因为在我们的计算管道中,每个家族成员都需要完全相同的结构域配置文件[参见附加文件]gydF4y2Ba7gydF4y2Ba].先前报道的prolamin基因均为单外显子基因[gydF4y2Ba49gydF4y2Ba除了四个包含单个内含子的基因,进一步检查发现基于EST比对,它们是未被正确注释的单外显子基因(数据未显示)。除4个不准确基因外,推导出的prolamin蛋白氨基酸长度在101~ 156 bp之间,有101~110和145~160 bp两个峰,与水稻prolamin蛋白的研究结果一致[gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba].gydF4y2Ba
只有5个prolamin家族成员(LOC_Os05g26720.1, LOC_Os05g26770.1, LOC_Os06g31070.1, LOC_Os12g16880.1, LOC_Os12g16890.1)与唯一、可靠和显著的MPSS标记相关,正如预期的那样,这些标记仅在3天萌发的种子中表达,丰度相对较高(分别为198、562、1042、148和670 Transcripts Per Million (TPM))[见附加文件]gydF4y2Ba8gydF4y2Ba].我们还检测了两个prolamin家族的表达,其中家族3856(123个成员)包含与prolamin家族3193相同的Pfam结构域(PF00234)[见附加文件]gydF4y2Ba7gydF4y2Ba].3856家族中共有54个基因与独特、可靠和显著的MPSS标记相关。然而,在3856家族中观察到的表达模式与prolamin家族(3722家族和3193家族)有很大的不同,因为大多数基因在多个器官/组织中表达[见附加文件]gydF4y2Ba9gydF4y2Ba].gydF4y2Ba
有趣的是,我们观察到编码prolamin蛋白家族的基因似乎紧密地定位在染色体上。共有16个prolamin蛋白家族基因位于第5号染色体上,大量te相关基因插入家族成员之间[见附加文件]gydF4y2Ba10gydF4y2Ba].其他prolamin蛋白家族基因位于6号染色体(两个基因串联),7号染色体(两个基因簇)和12号染色体(三个基因之间插入了te相关基因),这表明在整个进化过程中,prolamin蛋白家族基因的串联复制紧随转座元件的插入。这与先前关于紧致膨胀的报道是一致的gydF4y2BaαgydF4y2Ba-玉米玉米蛋白基因家族[gydF4y2Ba13gydF4y2Ba].gydF4y2Ba
Bowman-Birk抑制剂(BBI)型蛋白家族gydF4y2Ba
BBI是一种富含半胱氨酸的蛋白质,具有胰蛋白酶和凝乳蛋白酶抑制活性[gydF4y2Ba51gydF4y2Ba].它首先在大豆[gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba],后来广泛分布在单子叶和双子叶种中[gydF4y2Ba54gydF4y2Ba- - - - - -gydF4y2Ba58gydF4y2Ba].由于其在植物防御中的可能作用,它已被广泛研究[gydF4y2Ba51gydF4y2Ba,gydF4y2Ba54gydF4y2Ba,gydF4y2Ba58gydF4y2Ba]及其在癌症化学预防方面的潜在应用[gydF4y2Ba59gydF4y2Ba- - - - - -gydF4y2Ba61gydF4y2Ba].水稻中主要的BBI型蛋白质家族是Family 3328(8个成员)和Family 1493(3个成员)。虽然这两个家族都有Pfam结构域PF00228 (Bowman-Birk丝氨酸蛋白酶抑制剂家族),但家族3328也有通过BLASTP鉴定的第二个结构域[见附加文件]gydF4y2Ba11gydF4y2Ba].氨基酸组成分析显示,家族3288和家族1493的保守残基中分别有31%和47%为半胱氨酸,表明该氨基酸在BBI的蛋白酶抑制活性中具有重要作用。这些组成数据也揭示了两个BBI型蛋白家族之间的细微差异。MEGA 3.1版本生成的系统发育树[gydF4y2Ba62gydF4y2Ba家庭3328[见附加文件gydF4y2Ba12gydF4y2Ba]表明,在最初的复制事件之后,只有一个旁系基因经历了进一步的复制,这与这组BBI基因在1号染色体上的物理聚类一致[见附加文件]gydF4y2Ba13gydF4y2Ba].gydF4y2Ba
MPSS分析表明,BBI基因在广泛的组织和器官中存在差异表达,与先前报道的表达模式一致[gydF4y2Ba58gydF4y2Ba].3328家族中有7个基因具有独特、可靠和显著的MPSS标记,Pearson相关系数为-0.35 ~ 0.71。家族1493中的两个基因与独特、可靠和显著的MPSS标记相关,其表达相关性很小(gydF4y2BargydF4y2Ba= -0.12)。测定损伤后BBI基因的表达水平将是有趣的,因为家族3328中的7个蛋白质被注释为Bowman-Birk型bran胰蛋白酶抑制剂前体,据报道,这种类型在植物防御中发挥重要作用[gydF4y2Ba54gydF4y2Ba,gydF4y2Ba58gydF4y2Ba],家族1493中的两个成员被注释为创伤诱导的BBI型WIP1前体[gydF4y2Ba33gydF4y2Ba].gydF4y2Ba
结论gydF4y2Ba
我们证明,即使是相对较小的植物基因组,如水稻和拟南芥,它们的蛋白质组也有很大一部分是在副同源家族中,导致部分冗余的蛋白质组。水稻基因组中大多数副同源基因家族的起源似乎非常古老,但复制一直在以相当稳定的速度出现,复制的峰值与发生在~70 MYA的主要片段复制相一致。虽然在水稻和拟南芥同源同源家族中可以清楚地观察到蛋白质结构域的保守性,但我们确实观察到同源同源家族中蛋白质和蛋白质结构域的类型与单基因蛋白质的主要倾斜,这表明在基因组进化和基因复制过程中发生了选择的影响。副亲缘家族蛋白质的另一个潜在功能水平也可能通过选择性剪接发生,从统计上看,与水稻和拟南芥的单基因相比,副亲缘家族蛋白质中的选择性剪接更为频繁。在水稻中,虽然一些副亲本家族成员在转录上被共同调控,但表达模式的差异非常明显,从而扩大了该蛋白的功能范围。这些数据表明,植物基因组中存在多种产生蛋白质多样性的机制,这两种模式植物至少共享这些机制的一个子集。gydF4y2Ba
方法gydF4y2Ba
副同源蛋白家族的构建gydF4y2Ba
在TIGR水稻基因组注释第4版中[gydF4y2Ba33gydF4y2Ba],共注释了55,890个基因,其中与TE相关的基因有13,237个。te相关基因从所有进一步分析中被排除。由于水稻基因组中存在选择性剪接,且某些基因具有多种剪接形式,因此每当存在选择性异构体时,就使用最大的肽序列。短蛋白质序列(<50个氨基酸)被排除在分析之外。根据Haas中描述的蛋白质结构域组成,总共有42,653个水稻蛋白质序列被用于对副同源蛋白质家族进行分类gydF4y2Ba等gydF4y2Ba.[gydF4y2Ba30.gydF4y2Ba].生成蛋白质家族的基本方法包括识别结构域,然后根据结构域组织家族。两种不同类型的结构域用于生成副同源家族:Pfam/HMM结构域和基于blastp的结构域。对于Pfam/HMM结构域,预测的水稻蛋白质组在Pfam HMM结构域数据库中进行搜索[gydF4y2Ba63gydF4y2Ba]使用HMMER2 [gydF4y2Ba64gydF4y2Ba]和得分高于可信临界值的蛋白质被保留下来。对于基于BLASTP的结构域,未被Pfam HMM配置文件覆盖的肽区域,然后根据从all versus all BLASTP搜索中获得的同源性聚类[gydF4y2Ba65gydF4y2Ba].如果两种肽的e值<0.001,且>45%的同一性高于>75个氨基酸,则可以建立链接。为防止无亲缘关系的多结构域蛋白因单键而人为聚类,群落的Jaccard系数[gydF4y2Ba66gydF4y2Ba],也称为链接评分,用于聚类过程。正如哈斯所描述的gydF4y2Ba等gydF4y2Ba.[gydF4y2Ba30.gydF4y2Ba],计算链接肽序列对的链接得分gydF4y2Ba一个gydF4y2Ba而且gydF4y2BabgydF4y2Ba如下:gydF4y2Ba
选择链接得分高于临界值(0.66)的多肽生成单链接簇。然后使用CLUSTALW对聚束肽进行对齐[gydF4y2Ba67gydF4y2Ba,gydF4y2Ba68gydF4y2Ba]并用于开发基于blastp的域,如果域对齐包含四个或更多成员,则用于构建家族。然后根据结构域的类型和数量来组织蛋白质家族,包括Pfam HMM结构域和基于blastp的结构域。然后将具有相同结构域组成的蛋白质划分为假定的蛋白质家族。Paralogous拟南芥蛋白质家庭建立了类似的共有26819个蛋白质编码基因的TAIR7释放预测蛋白质组(gydF4y2Ba31gydF4y2Ba].gydF4y2Ba
片段复制基因的鉴定gydF4y2Ba
水稻基因组中的节段复制基因在第4版中定义,如前所述[gydF4y2Ba69gydF4y2Ba].简而言之,通过所有对所有BLASTP搜索(WU-BLASTP,参数“V = 5 B = 5 E = 1e-10 -filter seg”),可以识别出相似的基因对[gydF4y2Ba65gydF4y2Ba],然后通过运行DAGchainer来定义分段复制的块[gydF4y2Ba70gydF4y2Ba参数“-s -I -D 100000”。gydF4y2Ba
拟南芥蛋白质组的功能分类gydF4y2Ba
从预测蛋白质组的TAIR7版本中,总共下载了26,819个拟南芥蛋白质编码基因[gydF4y2Ba31gydF4y2Ba]并使用BLASTP搜索内部的非冗余氨基酸数据库,该数据库包含所有公开可用的蛋白质序列(例如GenBank, Swissprot等)[gydF4y2Ba65gydF4y2Ba]和Pfam HMM域数据库[gydF4y2Ba63gydF4y2Ba]使用HMMER2 [gydF4y2Ba64gydF4y2Ba].BLASTP与拟南芥序列的匹配被排除,除非它们来自Swissprot。BLASTP与保守的假设或假设蛋白的匹配也被排除。BLASTP匹配(< 1e-10和> 30%同源性超过50%覆盖率)或Pfam结构域得分高于可信截断值的拟南芥蛋白质被归类为已知或假定蛋白质。其余拟南芥基因根据TAIR7版本下载的基因集划分为表达基因或假设基因[gydF4y2Ba31gydF4y2Ba],至少有一个支持的cDNA和/或EST。gydF4y2Ba
GOSlim赋值gydF4y2Ba
指定基因本体(GO) [gydF4y2Ba71gydF4y2Ba],将预测的水稻蛋白质组与预测的拟南芥蛋白质组进行比对(TAIR6 Genome Release) [gydF4y2Ba31gydF4y2Ba]使用BLASTP。使用e值截断值1e-10,种植GOSlim注释[gydF4y2Ba34gydF4y2Ba]使用来自拟南芥的GO术语进行传递性注释。假设蛋白/表达蛋白、te相关蛋白和定义“未知”的GO术语蛋白被排除在本分析之外。拟南芥蛋白的GOSlim配位是通过TAIR7释放得到的[gydF4y2Ba31gydF4y2Ba].gydF4y2Ba
选择性剪接基因的鉴定gydF4y2Ba
在Release 4基因模型生成后,大约78万个水稻EST序列被释放[gydF4y2Ba33gydF4y2Ba].因此,我们利用PASA计划[gydF4y2Ba72gydF4y2Ba],利用最新的水稻转录数据对基因模型进行重新注释,全面识别交替剪接的基因。拟南芥的选择性剪接信息来自TAIR7释放[gydF4y2Ba31gydF4y2Ba].gydF4y2Ba
副同源蛋白家族年龄的估计gydF4y2Ba
使用CLUSTALW在默认参数设置下对每个家族进行多个蛋白质序列比对[gydF4y2Ba67gydF4y2Ba,gydF4y2Ba68gydF4y2Ba].从每个大小的蛋白质家族中gydF4y2BangydF4y2Ba,所有(gydF4y2BangydF4y2Ba2gydF4y2Ba-gydF4y2BangydF4y2Ba)/2从全局族对齐中提取成对对齐,保持所有间隙的位置和长度。每个同义站点的同义替换数量的最大似然估计(gydF4y2BadgydF4y2Ba年代gydF4y2Ba)对所有成对对齐均获得。所有计算均采用Goodman和Yang的基于密码子的替换模型[gydF4y2Ba73gydF4y2Ba]在gydF4y2BacodemlgydF4y2Ba的版本,版本3.15 [gydF4y2Ba74gydF4y2Ba],以成对模式运行(runmode = -2),密码子平衡频率由每个密码子位置的平均核苷酸频率估算(codonFreq = 2)。gydF4y2Ba
一个副同源蛋白质家族的年龄是由产生第二个成员的重复来定义的,可以用家族中亲缘关系最远的一对基因之间的差异来近似。以草类同义替换率计算,估计为~6.5 × 10gydF4y2Ba9gydF4y2Ba每年每幅地盘[gydF4y2Ba75gydF4y2Ba],表示每个站点的同义替换次数(gydF4y2BadgydF4y2Ba年代gydF4y2Ba)在同义位点不饱和的情况下(gydF4y2BadgydF4y2Ba年代gydF4y2Ba< ~ 1)。此外,在家庭内的分布高峰成对gydF4y2BadgydF4y2Ba年代gydF4y2Ba价值观显示了家庭多样化的时期。对于每个家庭,分层分布gydF4y2BadgydF4y2Ba年代gydF4y2Ba确定值,在0到1.5的范围内绘制,bin大小为0.1。的最大模态值和每个分布的模态库(通常由家谱中最古老的分裂产生)gydF4y2BadgydF4y2Ba年代gydF4y2Ba< 1.5(反映了在过去100 MY内多元化的爆发)的记录。gydF4y2Ba
大量并行签名测序数据和映射gydF4y2Ba
共有106,521个显著性(>3 TPM)和可靠(在多次测序中观察到)MPSS [gydF4y2Ba32gydF4y2Ba]标签均来自Rice MPSS项目[gydF4y2Ba32gydF4y2Ba,gydF4y2Ba76gydF4y2Ba].这些MPSS标记来自9个处理或未处理的器官/组织,包括愈伤组织、叶片、种子、冠营养分生组织、子房、柱头、花粉、穗和茎。为了降低背景噪声,采用了Haberer方法gydF4y2Ba等gydF4y2Ba.[gydF4y2Ba77gydF4y2Ba]用于删除标签,如果所有库的总最小丰度≤10 TPM,或者如果标签在至少一个库中≥5 TPM未被检测到,则总共有74,748个标签用于后续分析。最后一组MPSS标签根据TIGR水稻假分子进行搜索[gydF4y2Ba33gydF4y2Ba]使用Vmatch程序[gydF4y2Ba78gydF4y2Ba].由于标签可以跨越一个内含子(s), MPSS标签也针对注释基因的所有cDNA序列进行搜索。映射到注释基因反义序列或映射到基因组多个位置的MPSS标记被排除,这对于最小化密切相关的谬误序列之间的错误相关性很重要。如果一个基因与多个MPSS标签相关,则只使用最多的3'标签进行表达分析。分析了与独特、可靠和显著的MPSS标记相关的副同源基因。皮尔逊相关系数(gydF4y2BargydF4y2Ba)计算每个基因对的表达相关性,使用以下公式[gydF4y2Ba79gydF4y2Ba]:gydF4y2Ba
在哪里gydF4y2BangydF4y2Ba是DNA文库的数量。gydF4y2BaXgydF4y2Ba我gydF4y2Ba而且gydF4y2BaYgydF4y2Ba我gydF4y2Ba表示基因对的表达水平gydF4y2Ba我gydF4y2Bath图书馆。gydF4y2Ba
组织特异性表达分析gydF4y2Ba
为了确定某个基因是否在特定组织中优先表达,我们使用了Huminiecki设计的PEMgydF4y2Ba等gydF4y2Ba[gydF4y2Ba80gydF4y2Ba].PEM定义为loggydF4y2Ba10gydF4y2Ba(gydF4y2BaOgydF4y2Ba/gydF4y2BaEgydF4y2Ba).基本上,它将给定组织中观察到的(O)表达水平与预期的(E)表达水平进行比较,假设所有组织中表达一致。的PEM值gydF4y2Ba我gydF4y2Ba的第th基因gydF4y2BajgydF4y2Ba-th组织计算如下:gydF4y2Ba
在哪里gydF4y2Ba米gydF4y2Ba而且gydF4y2BangydF4y2Ba分别为符合mpss的基因和组织的总数。gydF4y2BaxgydF4y2Ba我gydF4y2Ba,gydF4y2BajgydF4y2Ba是第i个基因在第j个组织中的表达水平。gydF4y2Ba
缩写gydF4y2Ba
- BBI:gydF4y2Ba
-
Bowman-Birk抑制剂gydF4y2Ba
- 美国东部时间:gydF4y2Ba
-
表达序列标签gydF4y2Ba
- FLcDNA:gydF4y2Ba
-
全长cDNAgydF4y2Ba
- MPSS冲击:gydF4y2Ba
-
大规模并行签名测序gydF4y2Ba
- 我:gydF4y2Ba
-
数百万年gydF4y2Ba
- 米娅:gydF4y2Ba
-
百万年前gydF4y2Ba
- PEM:gydF4y2Ba
-
优先表达措施gydF4y2Ba
- TE:gydF4y2Ba
-
转座因子gydF4y2Ba
- TPM:gydF4y2Ba
-
每百万份成绩单gydF4y2Ba
参考文献gydF4y2Ba
- 1.gydF4y2Ba
Ohno S:基因复制的进化。Springer-Verlag,纽约;1970.gydF4y2Ba
- 2.gydF4y2Ba
Hughes AL:基因复制后功能新蛋白质的进化。中国生物医学工程学报,2004,26(4):344 - 344。10.1098 / rspb.1994.0058。gydF4y2Ba
- 3.gydF4y2Ba
Force A, Lynch M, Pickett FB, Amores A, Yan YL, Postlethwait J:利用互补退行性突变保存重复基因。中国生物医学工程学报,1999,31(4):344 - 344。gydF4y2Ba
- 4.gydF4y2Ba
Lynch M,力A:亚功能化重复基因保存的概率。中国生物医学工程学报,2000,54(1):459-473。gydF4y2Ba
- 5.gydF4y2Ba
顾z, Steinmetz LM,顾x, Scharfe C, Davis RW,李文华:重复基因在抗零突变遗传鲁棒性中的作用。自然科学学报,2003,26(3):344 - 344。10.1038 / nature01198。gydF4y2Ba
- 6.gydF4y2Ba
Grant D, Cregan P, Shoemaker RC:拟南芥基因组组织:拟南芥基因组复制与大豆与拟南芥的共时性。中国科学:自然科学,2000,29(3):349 - 349。10.1073 / pnas.070430597。gydF4y2Ba
- 7.gydF4y2Ba
沙提斯AM, Baron A, Barkan A, Martienssen RA:玉米叶绿体蛋白易位基因的复制和抑制。中国生物医学工程学报,2001,27(1):349- 356。gydF4y2Ba
- 8.gydF4y2Ba
Blanc G, Hokamp K, Wolfe KH:拟南芥基因组中最近的多倍体叠加在较老的大规模复制上。基因组学报,2003,13(2):137-144。10.1101 / gr.751803。gydF4y2Ba
- 9.gydF4y2Ba
Dias AP, Braun EL, McMullen MD, Grotewold E:最近复制的玉米R2R3 Myb基因为复制后进化分化的独特机制提供了证据。植物营养学报,2003,31(2):339 - 339。10.1104 / pp.012047。gydF4y2Ba
- 10.gydF4y2Ba
Cannon SB, Mitra A, Baumgarten A, Young ND, May G:拟南芥大基因家族进化中片段和串联基因复制的作用。中国生物医学工程学报,2004,4:10-10.1186/1471-2229-4。gydF4y2Ba
- 11.gydF4y2Ba
Leister D:植物抗病基因进化中的串联和片段基因复制与重组。植物学报,2004,20(3):116-122。10.1016 / j.tig.2004.01.007。gydF4y2Ba
- 12.gydF4y2Ba
李亚平,李亚平,姜华,毛莉:白杨MADS-box基因家族的全基因组分析。基因工程学报,2006,37(4):344 - 344。10.1016 / j.gene.2006.05.022。gydF4y2Ba
- 13.gydF4y2Ba
宋r, Llaca V, Linton E, Messing J:玉米22-kD α zein基因家族的序列、调控与进化。基因组学报,2001,11(11):1817-1825。gydF4y2Ba
- 14.gydF4y2Ba
王志刚,王志刚:玉米基因组的结构和变异。植物学报,2006,9(2):157-163。10.1016 / j.pbi.2006.01.009。gydF4y2Ba
- 15.gydF4y2Ba
王霞,石霞,郝波,葛松,罗娟:水稻基因组复制与DNA片段丢失:二倍体化的意义。植物保护学报,2005,26(3):344 - 344。10.1111 / j.1469-8137.2004.01293.x。gydF4y2Ba
- 16.gydF4y2Ba
Vandepoele K, Simillion C, Vande Peer Y:水稻和其他谷物是古代非整倍体的证据。植物学报,2003,15(9):2192-2202。10.1105 / tpc.014019。gydF4y2Ba
- 17.gydF4y2Ba
Simillion C, Vandepoele K, Saeys Y, Vande Peer Y:构建基因组图谱,揭示模糊地带的片段同源性。基因组学报,2004,14(6):1095-1106。10.1101 / gr.2179004。gydF4y2Ba
- 18.gydF4y2Ba
Paterson AH, Bowers JE, Chapman BA:在谷物分化之前的古代多倍体化,及其对比较基因组学的影响。中国科学:自然科学,2004,26(3):389 - 397。10.1073 / pnas.0307901101。gydF4y2Ba
- 19.gydF4y2Ba
Guyot R, Keller B:水稻祖先基因组复制。基因组学报,2004,47(3):610-614。10.1139 / g04 - 016。gydF4y2Ba
- 20.gydF4y2Ba
水稻11号和12号染色体测序联盟:水稻11号和12号染色体序列,富含抗病基因和最近的基因重复。中国生物医学工程学报,2005,3:20-10.1186/1741-7007-3-20。gydF4y2Ba
- 21.gydF4y2Ba
国际水稻基因组测序计划:基于图谱的水稻基因组序列。自然科学学报,2005,37(2):344 - 344。10.1038 / nature03895。gydF4y2Ba
- 22.gydF4y2Ba
Vij S, Tyagi AK:水稻胁迫相关蛋白(SAP)基因家族含A20/AN1锌指的全基因组分析及其与拟南芥的系统发育关系。Mol Genet Genomics, 2006gydF4y2Ba
- 23.gydF4y2Ba
trpathi LP, Sowdhamini R:拟南芥和水稻丝氨酸蛋白酶的交叉基因组比较中国生物医学工程学报,2006,27(3):344 - 344。gydF4y2Ba
- 24.gydF4y2Ba
马丁内斯M,亚伯拉罕Z, Carbonero P, Diaz I:拟南芥、水稻和大麦胱抑素基因家族的比较系统发育分析。中国生物医学工程学报,2005,27(5):423-432。10.1007 / s00438 - 005 - 1147 - 4。gydF4y2Ba
- 25.gydF4y2Ba
李志强,李志强,李志强,等:水稻SERK家族受体样蛋白激酶基因的表达。生物化学学报,2005,1730(3):253-258。gydF4y2Ba
- 26.gydF4y2Ba
杨晓明,李志强,李志强,等:拟南芥和水稻IQD基因家族的比较分析。中国生物医学工程学报,2005,26(3):344 - 344。gydF4y2Ba
- 27.gydF4y2Ba
袁建军,杨霞,赖娟,林红,程志明,Nonogaki H,陈峰:拟南芥、水稻和杨树的内源性- β -甘露聚糖酶基因家族。Funct integrg基因组公司,2006年gydF4y2Ba
- 28.gydF4y2Ba
李文杰,李志强,李志强,李志强。基因组聚类数据库。拟南芥和水稻序列家族分析平台。植物营养学报,2005,38(1):344 - 344。10.1104 / pp.104.059048。gydF4y2Ba
- 29.gydF4y2Ba
山崎裕田中伊藤T, T, Barrero RA, C,藤井裕久Y,希尔顿PB,安东尼奥·英航Aono H, Apweiler R, Bruskiewich R,局T,毛刺F, Costa de Oliveira,福娃G, Habara T,哈贝雷尔G,汉族B,原田E,节目,Hirochika H,霍恩D, Hokari H,细川护熙年代,杏,Ikawa H, Ikeo K, Imanishi T, Ito Y,贾斯瓦尔P,菅野米,Kawahara Y,河村建夫T,川岛H, Khurana JP,菊池,小松,森野奎KO, Kubooka H, Lieberherr D,林YC,朗斯代尔D,松本T, Matsuya, McCombie WR,扰乱J, Miyao,穆德N, Nagamura Y,南J,只N, Numa H, Nurimoto年代,马路C, Ohyanagi H, Okido T, Oota年代,大里N,帕默勒,Quetier F, Raghuvanshi年代,Saichi N,酒井法子H,酒井法子Y,坂田K,樱井T,佐藤F,佐藤Y, Schoof H,塞其M,柴田M,清水Y, Shinozaki K, Shinso Y,辛格NK, Smith-White B,武田J, Tanino M, Tatusova T, Thongjuea年代,Todokoro F, Tsugane M, Tyagi AK, Vanavichit,王一个,翅膀RA,山口K,山本M,山本N,于Y,张H,赵问,Higo K,毛刺B, Gojobori T,佐佐木T:水稻基因组的策展注释。拟南芥与粳稻的基因组比较分析。基因组学报,2007,17(2):175-183。10.1101 / gr.5509507。gydF4y2Ba
- 30.gydF4y2Ba
Haas BJ, Wortman JR, Ronning CM, Hannick LI, Smith RK JR, Maiti R, Chan AP, Yu C, Farzad M, Wu D, White O, Town CD:拟南芥基因组的完整重注释:方法、工具、协议和最终发布。中国生物医学工程学报,2005,3:7-10.1186/1741-7007-3-7。gydF4y2Ba
- 31.gydF4y2Ba
TAIR:(gydF4y2Bahttp://www.arabidopsis.orggydF4y2Ba]gydF4y2Ba
- 32.gydF4y2Ba
Rice MPSS数据库:[gydF4y2Bahttp://mpss.udel.edu/rice/gydF4y2Ba]gydF4y2Ba
- 33.gydF4y2Ba
Ouyang S, Zhu W, Hamilton J, Lin H, Campbell M, Childs K, thibard - nissen F, Malek RL, Lee Y, Zheng L, Orvis J, Haas B, Wortman J, Buell CR: TIGR水稻基因组注释资源的改进与新特征。核酸研究,2007,35(数据库issue): D883-7。10.1093 / nar / gkl976。gydF4y2Ba
- 34.gydF4y2Ba
基因本体:[gydF4y2Bahttp://www.geneontology.org/GO.slims.shtmlgydF4y2Ba]gydF4y2Ba
- 35.gydF4y2Ba
Benjamini Y, Hochberg Y:控制假阳性发现率:一种实用而强大的多重测试方法。皇家统计学会学报,1995,B辑,57:289-300。gydF4y2Ba
- 36.gydF4y2Ba
Blanc G, Wolfe KH:拟南芥进化过程中多倍体形成的复制基因的功能分化。植物细胞学报,2004,16(7):1679-1691。10.1105 / tpc.021410。gydF4y2Ba
- 37.gydF4y2Ba
Kopelman NM, Lancet D, Yanai I:选择性剪接和基因复制是逆相关的进化机制。植物学报,2005,37(6):588-589。10.1038 / ng1575。gydF4y2Ba
- 38.gydF4y2Ba
苏震,王娟,于娟,黄霞,顾旭:基因复制后选择性剪接的进化。基因组学报,2006,16(2):182-189。10.1101 / gr.4197006。gydF4y2Ba
- 39.gydF4y2Ba
顾旭:基因家族表达谱系统基因组分析的统计框架。中国生物医学工程学报,2004,37(1):344 - 344。10.1534 / genetics.167.1.531。gydF4y2Ba
- 40.gydF4y2Ba
顾晓,张卓,黄伟:酵母基因复制后表达和调控差异的快速进化。中国科学:自然科学,2005,26(3):344 - 344。10.1073 / pnas.0409186102。gydF4y2Ba
- 41.gydF4y2Ba
顾震,吕海辉,李文华:基于微阵列数据的重复基因表达快速分化。植物学报,2002,18(12):609-613。10.1016 / s0168 - 9525(02) 02837 - 8。gydF4y2Ba
- 42.gydF4y2Ba
胡晓明,张晓明,张晓明,等:拟南芥根中重复基因的表达模式。中华生物医学杂志,2005,60(2):247-256。10.1007 / s00239 - 004 - 0171 - z。gydF4y2Ba
- 43.gydF4y2Ba
王志刚,王志刚,王志刚,王志刚。大麦、黑麦和小麦种子贮藏蛋白的分子进化。中华分子生物学杂志,1998,18(3):439 - 439。10.1016 / 0022 - 2836(85) 90017 - 8。gydF4y2Ba
- 44.gydF4y2Ba
Shewry PR, Tatham AS:谷物种子脯氨酸贮存蛋白的结构与进化。生物化学学报,2000,26(1):1-12。gydF4y2Ba
- 45.gydF4y2Ba
Shewry PR, Tatham AS, Halford NG:小麦科的脯氨酸蛋白。种子蛋白质。编辑:Shewry PR, Casey R. 1999, 35-78 -gydF4y2Ba
- 46.gydF4y2Ba
Leite A, Neto GC, Vettore AL, Yunes JA, Arruda P:高粱,薏米和小米的脯胺。种子蛋白质。编辑:Shewry PR, Casey R. 1999, 141-157 -gydF4y2Ba
- 47.gydF4y2Ba
Krishnan HB, White JA:水稻种子蛋白质体的形态计量学分析(脯胺对水稻胚乳总蛋白质含量的显著贡献)。植物营养学报,2004,19(4):379 - 379。gydF4y2Ba
- 48.gydF4y2Ba
Barbier P, Ishihama A:野生水稻中prolamin基因家族核苷酸序列的变异。植物分子生物学,1990,15(1):191-195。10.1007 / BF00017744。gydF4y2Ba
- 49.gydF4y2Ba
温婷婷,Shyur LF,苏景昌,陈春春:水稻脯氨酸贮存蛋白基因RP6的核苷酸序列分析。植物营养学报,2003,26(3):344 - 344。10.1104 / pp.101.3.1115。gydF4y2Ba
- 50.gydF4y2Ba
Mullins IM, Hilu KW: 10 kDa水稻脯氨酸贮存蛋白的氨基酸变异。农业食品化学,2004,52(8):2242-2246。10.1021 / jf0305480。gydF4y2Ba
- 51.gydF4y2Ba
Ryan CA:植物中的蛋白酶抑制剂:提高对昆虫和病原体防御能力的基因。植物化学学报,2000,28(4):344 - 344。10.1146 / annurev.py.28.090190.002233。gydF4y2Ba
- 52.gydF4y2Ba
Birk Y, Gertler A, Khalef S:一种从大豆中提取的纯胰蛋白酶抑制剂。生物化学学报,2003,27(3):344 - 344。gydF4y2Ba
- 53.gydF4y2Ba
鲍曼德:大豆抗胰蛋白酶因子的分化。中华生物医学杂志,2004,27(3):344 - 344。gydF4y2Ba
- 54.gydF4y2Ba
Masumura T, Fujioka M, Matsui Y, Kumazawa Y, Tashiro M, Morita S, Tanaka K:水稻胰蛋白酶抑制基因的克隆、表达和定位模式。植物与动物基因组第十一次会议。2003gydF4y2Ba
- 55.gydF4y2Ba
Odani S, Koide T, Ono T:小麦胚芽胰蛋白酶抑制剂。Bowman-Birk型单头和双头抑制剂的分离和结构表征。J生物化学(东京)。1986, 100(4): 975-983。gydF4y2Ba
- 56.gydF4y2Ba
Tanaka AS, Sampaio MU, Marangoni S, de Oliveira B, Novello JC, Oliva ML, Fink E, Sampaio CA: Torresea cearensis种子中Bowman-Birk胰蛋白酶抑制剂的纯化及初级结构的测定。中国生物医学工程学报,1997,33(3):379 - 379。gydF4y2Ba
- 57.gydF4y2Ba
Norioka S, Ikenaka T:花生(Arachis hypogaea)中胰蛋白酶凝乳蛋白酶抑制剂(AI, AII, BI和BII)的氨基酸序列:豆科植物Bowman-Birk型抑制剂分子进化的探讨生物化学学报,2003,19(4):589- 589。gydF4y2Ba
- 58.gydF4y2Ba
曲丽娟,陈娟,刘敏,潘楠,冈本宏,林志,李超,李东,王娟,朱刚,赵霞,陈霞,顾宏,陈震:水稻新型Bowman-Birk抑制基因家族的克隆与功能分析。植物营养学报,2003,26(2):357 - 357。10.1104 / pp.103.024810。gydF4y2Ba
- 59.gydF4y2Ba
周泽,陈建平,陈建平,陈建平,陈建平:抗氧化剂和Bowman-Birk抑制剂对空间辐射的保护作用。地球科学进展,2006,26(2):327-332。10.1667 / RR3599.1。gydF4y2Ba
- 60.gydF4y2Ba
陈玉文,黄少生,林少宇,林俊杰:Bowman-Birk抑制剂通过MAP激酶磷酸酶-1的积累降低蛋白酶体功能,抑制MCF7乳腺癌细胞增殖。肿瘤发生。2005,26(7):1296-1306。10.1093 / carcin / bgi062。gydF4y2Ba
- 61.gydF4y2Ba
Dittmann KH, Mayer C, Rodemann HP: Bowman Birk蛋白酶抑制剂对正常组织放射保护改善放疗的作用。抗肿瘤药物的研究进展。2003,3(5):360-363。10.2174 / 1568011033482288。gydF4y2Ba
- 62.gydF4y2Ba
库马尔,田村K, Nei M: MEGA3:分子进化遗传学分析和序列比对的集成软件。生物工程学报,2004,5(2):150-163。10.1093 /龙头/ 5.2.150。gydF4y2Ba
- 63.gydF4y2Ba
Bateman A, Coin L, Durbin R, Finn RD, Hollich V, griffith - jones S, Khanna A, Marshall M, Moxon S, Sonnhammer EL, Studholme DJ, Yeats C, Eddy SR: Pfam蛋白家族数据库。Nucleic Acids Res. 2004, 32(数据库issue): D138-41。10.1093 / nar / gkh121。gydF4y2Ba
- 64.gydF4y2Ba
Eddy SR:剖面隐藏马尔可夫模型。生物信息学,1998,14(9):755-763。10.1093 /生物信息学/ 14.9.755。gydF4y2Ba
- 65.gydF4y2Ba
Gish W: 1996 - 2006。[gydF4y2Bahttp://blast.wustl.edugydF4y2Ba]gydF4y2Ba
- 66.gydF4y2Ba
Jaccard P:高山地区植物群的分布。新植物学家。1912,11(2):37-50。10.1111 / j.1469-8137.1912.tb05611.x。gydF4y2Ba
- 67.gydF4y2Ba
Thompson JD, Higgins DG, Gibson TJ: CLUSTAL W:通过序列加权、位置特定间隙惩罚和权重矩阵选择提高渐进式多序列对齐的灵敏度。中国生物医学工程学报,1994,22(2):473 - 480。10.1093 / nar / 22.22.4673。gydF4y2Ba
- 68.gydF4y2Ba
Chenna R, Sugawara H, Koike T, Lopez R, Gibson TJ, Higgins DG, Thompson JD:多序列对齐与Clustal系列程序。中国生物医学工程学报,2003,31(13):3497-3500。10.1093 / nar / gkg500。gydF4y2Ba
- 69.gydF4y2Ba
林海红,朱伟,陈志刚,顾晓霞,陈志刚:水稻节段复制基因中内含子的增减。中国生物医学工程学报,2006,7 (5):R41-10.1186/gb-2006-7-5-r41。gydF4y2Ba
- 70.gydF4y2Ba
Haas BJ, Delcher AL, Wortman JR, Salzberg SL: DAGchainer:一种挖掘片段基因组复制和同向性的工具。生物信息学,2004,20(18):3643-3646。10.1093 /生物信息学/ bth397。gydF4y2Ba
- 71.gydF4y2Ba
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT, Harris MA, Hill DP, Issel-Tarver L, Kasarskis A, Lewis S, Matese JC, Richardson JE, Ringwald M, Rubin GM, Sherlock G:基因本体论:生物学统一的工具。基因本体联盟。植物学报,2000,25(1):25-29。10.1038/75556。gydF4y2Ba
- 72.gydF4y2Ba
Haas BJ, Delcher AL, Mount SM, Wortman JR, Smith RK JR, Hannick LI, Maiti R, Ronning CM, Rusch DB, Town CD, Salzberg SL, White O:利用最大转录本比对程序改进拟南芥基因组注释。中国生物医学工程学报,2003,31(19):5654-5666。10.1093 / nar / gkg770。gydF4y2Ba
- 73.gydF4y2Ba
杨智,杨志勇:基于密码子的蛋白质编码DNA序列的核苷酸替代模型。分子生物学杂志,1994,11(5):725-736。gydF4y2Ba
- 74.gydF4y2Ba
杨志:PAML:最大似然系统发育分析程序包。计算应用生物科学,1997,13(5):555-556。gydF4y2Ba
- 75.gydF4y2Ba
Gaut BS, Morton BR, McCaig BC, Clegg MT:草和棕榈的替代率比较:核基因Adh的同构率差异和质体基因rbcL的平行率差异。中国科学:自然科学,2001,26(3):379 - 379。10.1073 / pnas.93.19.10274。gydF4y2Ba
- 76.gydF4y2Ba
Nakano M, Nobuta K, Vemaraju K, Tej SS, Skogen JW, Meyers BC:植物MPSS数据库:mRNA和小RNA分析的基于特征的转录资源。核酸决议2006,34(数据库问题):D731-5。10.1093 / nar / gkj077。gydF4y2Ba
- 77.gydF4y2Ba
王晓明,王晓明,王晓明,王晓明,王晓明:拟南芥复制基因中顺式元件的转录同源性和差异性分析。植物科学进展,2004,26(2):339 - 344。10.1104 / pp.104.046466。gydF4y2Ba
- 78.gydF4y2Ba
库尔兹S: Vmatch大规模序列分析软件。[gydF4y2Bahttp://www.vmatch.de/gydF4y2Ba]gydF4y2Ba
- 79.gydF4y2Ba
Rosner B:生物统计学基础第四版。克斯伯里出版社;1995.gydF4y2Ba
- 80.gydF4y2Ba
Huminiecki L, Lloyd AT, Wolfe KH:基因表达图谱,SAGEmap和TissueInfo数据库组织表达谱的一致性。生物医学工程学报,2003,4(1):31-10.1186/1471-2164-4-31。gydF4y2Ba
确认gydF4y2Ba
我们感谢张哲就统计分析提出的意见。我们感谢弗朗索瓦丝·蒂博-尼森对这篇文章的严格审查。本研究由美国国家科学基金会植物基因组研究计划资助C. R. B. (DBI-0321538)。MPSS数据由美国国家科学基金会资助B.C.M. (DBI-0321437)。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
相应的作者gydF4y2Ba
额外的信息gydF4y2Ba
作者的贡献gydF4y2Ba
HL设计了这项研究,进行了分析,并起草了手稿。SO参与了GOSlim的分析并制作了附加文件gydF4y2Ba3.gydF4y2Ba.KN和BM提供了水稻MPSS数据。AE和JS对副亲缘家族进行年龄分析。BH在水稻中鉴定了可选剪接异构体。WZ在水稻中鉴定出高置信度基因集。XG参与了alternative splicing的分析。RB设计了研究并起草了手稿。所有作者都阅读并批准了最终的手稿。gydF4y2Ba
电子辅助材料gydF4y2Ba
12870 _2007_226_moesm3_esm.tiffgydF4y2Ba
附加文件3:非转座元件相关基因在水稻和拟南芥中的分布。在图A中,12个水稻染色体中,副同源基因家族成员用蓝色表示,单拷贝基因用红色表示。节段复制块用绿色表示,着丝粒用白色方框表示。图B中,拟南芥的5条染色体中,副同源基因家族成员以蓝色表示,单拷贝基因以红色表示。(tiff 5mb)gydF4y2Ba
一)gydF4y2Ba
附加文件4:水稻副同源蛋白质家族的年龄分布。年龄分布的扩展图。gydF4y2BaB)gydF4y2Ba水稻副同源蛋白质家族的分布以扩大最大gydF4y2BadgydF4y2BaS≤1.5。(pdf 43kb)gydF4y2Ba
12870 _2007_226_moesm5_esm.pdfgydF4y2Ba
附加文件5:下的模态值分布gydF4y2BadgydF4y2Ba年代gydF4y2Ba在水稻副同源蛋白家族中≤1.5。在3865个副同源蛋白家族中,2388个蛋白家族的峰值均在1.5以下gydF4y2BadgydF4y2Ba年代gydF4y2Ba值和被绘制。(pdf 10kb)gydF4y2Ba
一)gydF4y2Ba
附加文件6:Pearson相关系数(gydF4y2BargydF4y2Ba)与gydF4y2BadsgydF4y2Ba值。gydF4y2Ba一)gydF4y2Ba0
12870 _2007_226_moesm7_esm.pdfgydF4y2Ba
附加文件7:三个相关水稻副同源蛋白家族的结构域组成示意图:3722家族、3193家族和3856家族。(pdf 11kb)gydF4y2Ba
12870 _2007_226_moesm8_esm.pdfgydF4y2Ba
附加文件8:18个文库中来自3722家族和3193家族的水稻prolamin基因的表达丰度,这些基因与独特、可靠和显著的MPSS标记相关。(pdf 7kb)gydF4y2Ba
12870 _2007_226_moesm9_esm.pdfgydF4y2Ba
附加文件9:18个文库中水稻副同源蛋白家族family 3856(含PF00234)基因的表达丰度,这些基因与独特、可靠和显著的MPSS标记相关。(pdf 15kb)gydF4y2Ba
12870 _2007_226_moesm10_esm.pdfgydF4y2Ba
附加文件10:编码水稻prolamin蛋白的基因的基因组浏览器视图,与te相关的基因插入在假定的串联重复之间。(pdf 22kb)gydF4y2Ba
12870 _2007_226_moesm11_esm.pdfgydF4y2Ba
附加文件11:具有Pfam结构域PF00228的两个水稻bbi相关副同源蛋白家族的结构域组成示意图:Family 3328和Family 1493。(pdf 10kb)gydF4y2Ba
作者提交的图片原始文件gydF4y2Ba
下面是作者提交的原始图片文件的链接。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(gydF4y2Bahttps://creativecommons.org/licenses/by/2.0gydF4y2Ba),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
林,H.,欧阳,S.,伊根,A.。gydF4y2Baet al。gydF4y2Ba水稻副同源蛋白质家族的特征。gydF4y2BaBMC植物生物学gydF4y2Ba8日,gydF4y2Ba18(2008)。https://doi.org/10.1186/1471-2229-8-18gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/1471-2229-8-18gydF4y2Ba
关键字gydF4y2Ba
- 基因对gydF4y2Ba
- 重复的基因gydF4y2Ba
- 醇溶谷蛋白gydF4y2Ba
- 域包含了gydF4y2Ba
- 重复基因对gydF4y2Ba