摘要
背景
资料超过35 000条全长栽培稻在各种处理条件下收集的cdna和相关的微阵列基因表达数据,使得识别基因启动子中保守的、可能起作用的基序变得可行独联体-在各种条件下发挥关键作用的调控要素。
结果
我们开发了一种新的工具来搜索独联体-在差异调控基因的上游、下游或编码区域的候选元素。工具首先列出顺式候选元素通过motif搜索的基础上假设是否存在顺式在给定的一组基因的调节中起重要作用的元素,它们将在统计上被过度代表,并将被保守。然后通过关联规则分析对所列候选母题的似然得分进行评估。这一策略依赖于启动子区域中过度表达的基序可能在这些基因的表达调控中发挥特定作用的想法。该工具的设计使任何生物学研究人员都可以在公开访问的互联网站点上轻松使用http://hpc.irri.cgiar.org/tool/nias/ces.我们通过使用已被充分研究的生长素诱导基因数据集来评估该工具的准确性和实用性顺式元素。测试显示了该工具在识别显著关系之间的有效性顺式候选元素和相关基因组。
结论
该工具列出了可能的顺式元件基序与感兴趣的基因相对应,这将有助于更深入地理解植物基因调控机制。
背景
随着国际水稻基因组测序计划完成水稻基因组测序[1]、北京基因研究所(华大基因)[2]和先正达[3.],许多水稻功能基因组资源已经可用,包括来自ssp的全基因组序列。粳稻Nipponbare和ssp。籼稻行93 - 11;一套水稻全长cDNA克隆及其完整和部分末端序列[4,5],基于全长cDNA序列、ESTs(表达序列标签)、MPSS(大规模并行签名测序)、SAGE(基因表达序列分析)和基因组序列中预测基因的微阵列基因表达系统;以及Tos17、Ac-Ds和t - dna的多种插入突变体[6].随着分析技术的发展,数据库不断升级,成为研究调控基因表达机制的有用资源。
顺式基因和启动子区域的元素反式-如果我们要了解调控基因表达的系统,那么作用转录因子是主要的生物学特征。候选人的识别顺式通过使用现有的序列和基因组图谱信息,结合关于基因对特定实验条件的反应的信息,与基因相对应的元素现在是可行的;这些反应已经通过使用现在公开的基因表达谱来阐明。
穷尽序列分析通过使用可用的公共数据库可以识别顺式元素候选图案进行进一步检查,但这种方法不是很有效。一个令人困惑的因素是,公共数据库是独立构建的,通常没有优化到便于将来自许多来源的信息与本地实验数据集成。对于不太熟悉生物信息学技术的实验研究人员来说,一个更令人困惑的问题是,要找到基因之间未知但在生物学上显著的关系,顺式元素,和实验条件从大量的可能组合产生的大型实验数据集。
为了解决其中的一些问题,我们开发了一种新的数据挖掘工具来识别顺式水稻基因组中的元素。它执行上面提到的复杂的生物信息学分析,然后列出顺式基因的候选元素。研究人员可以根据表达谱的相似性和其他评估标准对基因进行分组,然后该工具用相关的公共数据库信息对它们进行注释。
类似的工具以前已经开发出来了。Helden发布了RSAT,其中包括一个程序,可以检测共同调控基因上游区域的过度代表基序[7].Holt等人建立了CoReg,将共表达基因集的层次聚类与启动子元件的频率表联系起来[8].赵等人建立了TRED,它集成了一个数据库和一个预测系统独联体- - -反式-哺乳动物中的元素[9].Galuschka等人开发了AthaMAP,其中包括一个用于比较分析的程序独联体的共转录基因组中的-元素拟南芥[10].
我们的工具有以下几点特点:(i)它专注于水稻基因组,基于全长cdna,并被设计用于提取独联体-与用户指定的基因相关的候选元素。(ii)对的似然得分进行评估独联体通过比较用户选择的基因集和参考基因集中的频率计数来确定候选元素。(iii)可以评估已知信息独联体-元素序列以及由其他分析工具准备的用户指定的序列,它可以检查几个独联体元素在一起。
该工具实现了这两种功能从头开始启动子序列的Motif搜索和已知植物的搜索顺式元素,然后对识别出来的元素进行似然分析顺式在给定的一组基因的启动子中显著比例存在的基础上的元素。该评估是通过关联规则分析实现的。
在这里,我们介绍了该工具的技术细节,并使用生物学相关的样本数据集演示了其效用的实际评估。
实现
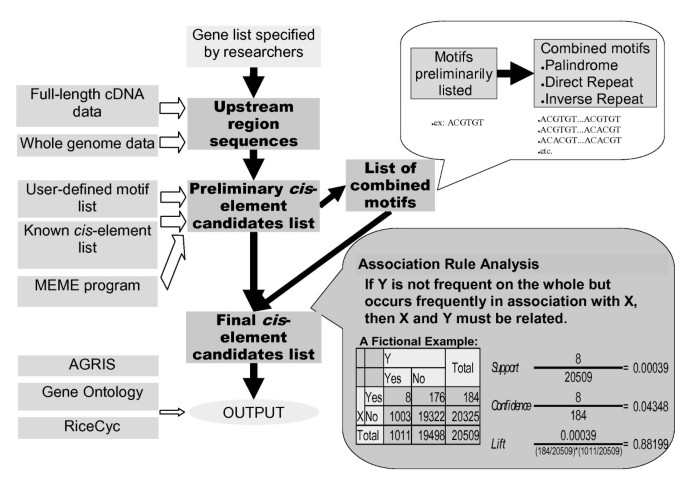
这个工具叫做Rice独联体-元素搜索器(rice),由一个顺式元素搜索管道,通过基于web的用户界面进行控制。无花果。1总结了程序。该管道首先从用户那里读取一个基因标识符列表,用于检索与所列基因对应的启动子序列。然后初步列出顺式候选元素是通过从内置的看似合理的主题列表中对齐信息来构建的,或者通过从头开始序列数据的Motif搜索。进行关联规则分析并报告,以支持结果的候选性顺式元素列表。
基因列表
RiCES假设用户已经从实验分析中识别出感兴趣的基因(例如协调调节基因簇)。标识符列表被输入到基于web的数据输入表单中。RiCES识别GenBank登录号、转录单位标识符(tu),这些标识符在TIGR假分子组合中定义[11],以及其他几个主要的基因识别系统。使用该列表,它从可用的基因组序列数据中检索指定基因两侧的相关上游、下游或编码区域序列集。
初步独联体-元素候选列表
分析的第二步是编制候选母题列表顺式元素。目前,rice支持两种方法来实现这一目标。
第一种方法取决于从头开始Motif搜索基于假设,如果有顺式在给定的一组基因的调控中起重要作用的元件,在相关的启动子序列中,它们作为保守的基序在统计上被过度代表,可以通过使用合适的基序搜索程序来识别。有几个程序实现了几个算法。我们选择使用MEME,这是一个公开的motif发现程序[12]支持期望最大化算法。在我们的分析算法中,MEME被用于识别所选基因启动子序列中看起来高度保守的6到8 bp长的基序。用户可以通过Web表单修改MEME程序的一些搜索参数。
第二种方法依赖于一个普遍的、已知的假设顺式元素在产生用户指定的基因列表的实验条件下发挥重要作用。因此,rice搜索与预先编译的已知列表相匹配的顺式元素。
植物的几个数据库顺式元素是公开可用的。的地方(13是已知的最流行的数据库之一顺式植物基因组中的元素。AtcisDB是AGRIS的一部分[14],包括有关的信息独联体-参与基因调控的元素拟南芥.
尽管这些数据库是非常有用的资源,但要将其中的信息直接交叉链接到研究人员自己的数据中并不容易。目前的数据库还不够详尽,无法区分决定功能的“核心”图案独联体-元素,来自相邻区域共存的序列。结果,很多人独联体这些数据库中的元素序列数据包括表面的核心基序,没有获得功能性的证据。这种数据的使用妨碍了有效的信息分析。
我们编制了一个新的已知的数据库顺式元素并将其纳入RiCES[参见附加文件]1].的顺式元素从实验报告中收集,如凝胶位移分析和足迹分析,按转录因子分类,并记录植物基因组中的已知活性。一些顺式考虑到它们在植物中可能(尽管未知)的作用,只在植物以外的生物体中已知的元素也被列了出来。该数据库包括四种类型的独联体-元素:(1)G-box和E-box,在许多生物中与bHLH或bZIP等常见序列结合;(2) A-box、T-box和GGTTTAG重复序列,它们与许多生物的常见序列结合,如同源结构域和Myb;(3)与植物MADS、锌指和AP2/EREBP元件结合的CArG盒和gcc盒;(4)其他顺式仅在动物中结合的元素,如HSF, PcG和HMG。
关联规则分析
分析的第三步是可能性评价顺式关联规则分析,这是一种数据挖掘方法,旨在发现数据集中观察到的特征对之间的显著关系。表现出最高可能性(特异性)的候选人被保留在决赛中顺式元素候选列表。
关联规则分析已应用于调节基因表达的机制[例如]15,16]]。我们用它来寻找确定的关系顺式元素和基因表达谱。该策略依赖于这样一种想法,即在感兴趣基因的启动子区域中过度代表的基序可以在这些基因的表达调控中发挥特定的作用。
隐含的因果关系被记录为“规则”,通过使用几个著名的可能性指数来评估,包括支持,信心,电梯[15].在样本数据集的基础上,提出了电梯指数似乎能最好地区分实验条件与顺式元素的候选人。
在规则中描述为
基因中X基序的存在意味着该基因是Y族的成员,
电梯是后验概率(如果该基因拥有基序X,则该基因属于Y组的概率)与先验概率(X拥有的概率,与Y的成员无关)的比值电梯> 1.0, X和Y的共存不是随机发生的,而是表明它们之间存在某种因果关系。如果电梯< 1.0,则认为不具有概率显著性。因此,我们将默认阈值设置为电梯到1.0,和顺式候选元素仅在满足以下条件时才包含在最终候选列表中电梯值高于此阈值。
rice还评估初步候选列表中图案的成对组合(图中右上角框)。1),考虑到多种转录元件结合可能存在蛋白-蛋白相互作用顺式元素,如实验证据所示[17,18].
输出
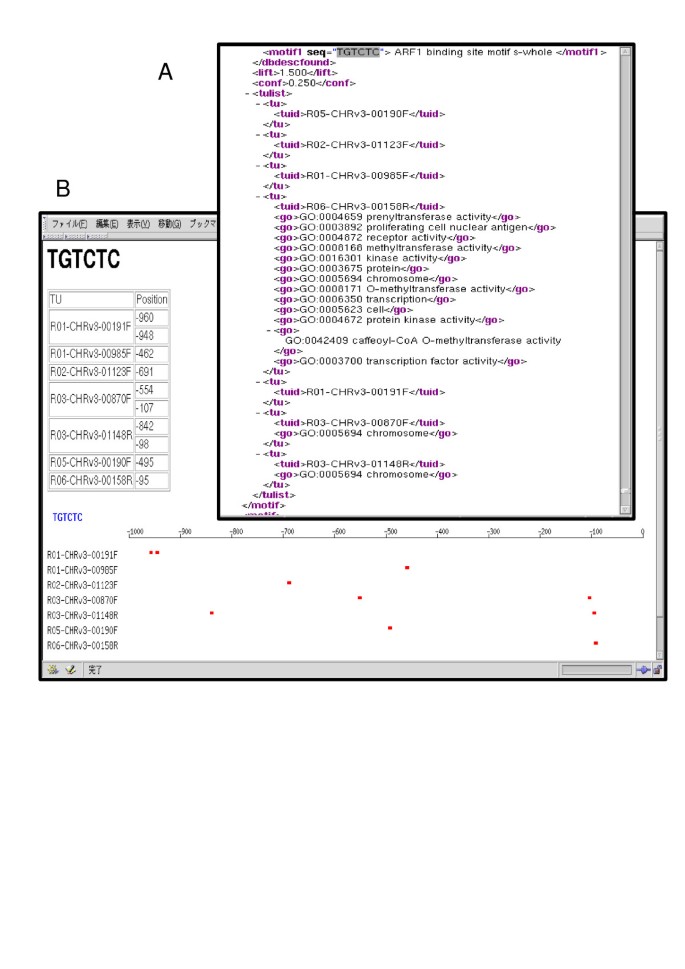
最后一个顺式元素候选列表以关联表的形式呈现,与提交的基因的标识符(当前版本使用基于TIGR基因模型注释的TU标识符)一起标注,并使用来自RiceCyc的任何可用的相应信息[19]和基因本体论[20.].RiCES还提供了候选基序的信息,包括该元素在相应的TUs启动子区域的位置、序列以及来自AtcisDB的相关信息[14].的位置顺式候选元素也以文本和图形的形式显示。
结果与讨论
许多Aux/IAA基因可被生长素诱导[23],并包含TGTCTC元素[24].该元素通常存在于生长素反应基因的上游区域。因此,通过管道检测motif的所有实例可以作为管道算法的验证。含有TGTCTC基序的生长素响应元件(AuxRE)在某些情况下需要另一个近端AuxRE来发挥生物活性[17,25].在其他情况下,AuxRE仅当其回文成分被7或8个核苷酸分开时才起作用[26].
在我们的验证测试中,MEME从上游序列的1000 bp中列出了7514个motif[见附加文件]3.],其中4128个显示高电梯value(>1.0)[参见附加文件4].在AtcisDB中对这些基序的搜索返回4,显示与“PRHA结合位点”的记录部分匹配(表4)1),该公式源自Plesch等人的报告。[27],描述生长素诱导的表达拟南芥prha同源框基因。另外4个图案包含TGTCTC元素。结果与之前的工作一致,TGTCTC被列为Aux/IAA基因单基序搜索的候选。
表格2显示预编译的验证测试结果顺式由测试元素列表生成的基因列表。分析结果为22独联体-元素候选电梯> 1.0[参见附加文件5而且6].以前的研究表明,其中一些候选者与生长素反应有某种关系。例如,RAV1的启动子区域ABP,编码生长素结合蛋白[28].的表达多叶的(LFY)受植物生长素梯度的影响拟南芥[29].摘要是另一种生长素反应因子[30.),而LFY而且摘要表达密切相关[18,31].
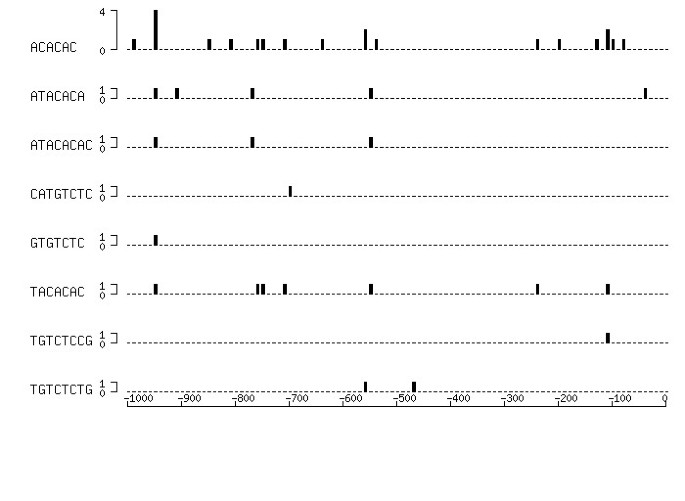
的位置顺式元素是要考虑的与功能有关的重要信息顺式元素。对于生物活动的发生,距离有些顺式来自编码区域的元素或其他协作元素受到约束。为此,rice强调了分布顺式元素的候选人。它提供了已识别的表顺式元素母题和图形母题图,帮助研究人员掌握候选元素之间的位置关系。
所列元件的位置在基因的上游区域不同,其中一些包括TGTCTC。2),并且很难检测到任何倾斜的母题分布。哥达等人。32]研究了TGTCTC基序在植物基因组中的分布答:芥,并指出25%的被研究基因在起始密码子上游1000 bp内有TGTCTC基序,14%在起始密码子上游500 bp内有TGTCTC基序。我们的结果似乎与他们的并不矛盾。
TGTCTC图案分布在许多植物物种的广泛区域(表3.).基因角色的多样性可能反映了调节基因表达和位置的机制的多样性顺式元素,即使有问题的基因可以被归类为'生长素反应基因'在更大的意义上。
一个主要的研究课题是如何接受顺式值得进一步试验的候选元素。计算和手动选择顺式候选元素应该扮演互补的角色来解决这个问题。应该强调的是顺式RiCES列出的候选元素根据关联规则分析提供的可能性进行评级。另一方面,研究人员可以通过从多个数据库中获得的相关信息来详细检查候选人的显著性。支持的数据库包括AGRIS、Gene Ontology和RiceCyc,以及上述的地图信息。
无花果。3.是TGTCTC主题输出的一个例子。输出不仅在Web浏览器中易于访问,而且还可用于进一步的统计或生物信息学分析,因为它们也以XML格式提供(图2)。3),是一种带标签的纯文本格式,可与各种电脑程序兼容。
在某些情况下,来自预编译的元素列表的分析结果将很容易与先验知识进行比较。其他仅涉及从头开始根据MEME的证据,motif搜索的结果应仔细解释,因为结果将根据所选择的选项发生相当大的变化。通过反复试验,每次都应该确定一组合适的主题搜索选项。然而,如上所述,一个主题搜索可以找到顺式候选元素的序列与已知元素的序列不完全匹配顺式元素。
虽然rice关注的是顺式元素栽培稻ssp。粳稻,该方法可以很容易地应用于其他植物物种的研究,或涉及基因表达调控的其他基因组序列基序的研究,如基因编码区域或基因序列下游的基序。通过将包含水稻全基因的参考数据集替换为其他数据集,可以使这项工作成为可能。
结论
我们在这里介绍了一个新开发的搜索工具顺式基因列表中的候选元素。实例研究表明了该工具的适用性。该工具易于使用,并且可以公开使用。我们期望它的使用将加深对植物基因表达调控机制的理解。
可用性和需求
rice在http://hpc.irri.cgiar.org/tool/nias/ces任何支持javascript的浏览器。
项目名称:世代挑战方案次级方案4
项目主页:http://www.generationcp.org/subprogramme4.php
操作系统:平台无关的
其他要求:没有一个
编程语言:Perl
许可:免费使用
非学术界人士使用的任何限制:没有
参考文献
- 1.
IRGSP项目:基于图谱的水稻基因组序列。自然科学学报,2005,37(3):344 - 344。10.1038 / nature03895。
- 2.
栽培稻l . ssp。籼稻).科学通报,2002,29(3):344 - 344。10.1126 / science.1068037。
- 3.
高夫SA,里奇D, T局域网,普雷斯特G,王R,邓恩M, Glazebrook J,会话,Oeller P, Varma H,哈德利D,和记黄埔D,马丁·C片瞳F,兰格BM, Moughamer T,夏Y, Budworth P,钟J, Miguel T Paszkowski U,张年代,科尔伯特M,太阳W,陈L,库珀B,公园,木TC、毛L,鹌鹑P, R, R,院长于Y, Zharkikh,沈R, Sahasrabudhe年代,托马斯,罐头R, Gutin, Pruss D,里德J, Tavtigian年代,米切尔J·G,肖勒T,米勒RM,博年代,阿迪N, Rubano T,图斯尼姆N, Robinson R, Feldhaus J, Macalma T, Oliphant A, Briggs S:水稻基因组草案序列(栽培稻l . ssp。粳稻).科学通报,2002,29(3):344 - 344。10.1126 / science.1068275。
- 4.
水稻全长cDNA联盟:超过28000个cDNA克隆的收集,映射和注释粳稻大米。科学通报,2003,30(3):366 - 366。10.1126 / science.1081288。
- 5.
Satoh K, Doi K,经营T,岸本N, K,铃木Otomo Y,卡瓦依J,中村M, Hirozane-Kishikawa T,神奈川年代,荒川T, Takahashi-Iida J,村田,Ninomiya N,佐佐木D,福田,Tagami M,山形H, Kurita K, Kamiya K,山本M, Kikuta,鱼毒木T, Fujitsuka N, K Ito, Kanamori H,崔我,Nagamura Y,松本T,村上K,松原K, Carninci P,崎Y,菊池:在水稻基因组织了全长cDNA映射,通过微阵列基因表达分析。公共科学学报,2007,2:e1235-10.1371/journal. peer .0001235。
- 6.
Hirochika H, Guiderdoni E, An G, Hsing Y, Eun MY, Han C, Upadhyaya N, Ramachandran S, Zhang Q, Pereira A, Sundaresan V, Leung H:水稻基因发现突变资源。中国生物医学工程学报,2004,29(4):344 - 344。10.1023 / B: PLAN.0000036368.74758.66。
- 7.
van Helden J:调控序列分析工具。中国生物医学工程学报,2003,31:339 - 339。10.1093 / nar / gkg567。
- 8.
王晓明,王晓明,王晓明,等。基于启动子序列的基因表达模块构建方法研究进展。植物科学进展,2006,29(2):366 - 366。
- 9.
赵峰,宣志,刘玲,张敏敏:TRED:转录调控元件数据库及研究平台在网上基因调控研究。中国生物医学工程学报,2005,33:D103-D107。10.1093 / nar / gki004。
- 10.
高晓明,陈晓明,陈晓明,等。一种用于共调控基因分析和鉴定的amap网络工具。中国生物医学工程学报,2007,35:D857-D862。10.1093 / nar / gkl1006。
- 11.
Ouyang S, Zhu W, Hamilton J, Lin H, Campbell M, Childs K, thibard - nissen F, Malek RL, Lee Y, Zheng L, Orvis J, Haas B, Wortman J, Buell CR: TIGR水稻基因组注释资源的改进与新特征。中国生物医学工程学报,2007,35:D883-D887。10.1093 / nar / gkl976。
- 12.
Bailey TL, Elkan C:通过期望最大化拟合混合模型来发现生物聚合物中的基序。中国生物医学工程学报,1994,2:28-36。
- 13.
Higo K, Ugawa Y, Iwamoto M, Korenaga T:植物独联体-作用调控DNA元件(PLACE)数据库。中国生物医学工程学报,1999,27(4):457 - 457。10.1093 / nar / 27.1.297。
- 14.
Davuluri RV, Sun H, Palaniswamy SK, Matthews N, Molina C, Kurtz M, Grotewold E: AGRIS:拟南芥基因调控信息服务器,拟南芥信息资源独联体-调控元件和转录因子。中国生物医学工程学报,2003,26(4):344 - 344。
- 15.
Carmona-Saez P, Chagoyen M, Rodriguez A, Trelles O, Carazo JM, Pascual-Montano A:基于关联规则发现的基因表达综合分析。中国生物医学工程学报,2006,27(4):344 - 344。
- 16.
Conklin D, Jonassen I, Aasland R, Taylor WR:核苷酸模式与基因功能类的关联:人类3'非翻译序列的应用。生物信息学,2002,18:182-189。10.1093 /生物信息学/ 18.1.182。
- 17.
刘志斌,刘志斌,刘志斌,刘志强。植物生长素响应元件的复合结构。中国生物医学工程学报,2004,27(4):344 - 344。10.1105 / tpc.7.10.1611。
- 18.
黄志刚,张志刚,张志刚,等:生长素反应因子的二聚化和DNA结合。植物学报,1999,29(4):349 - 349。10.1046 / j.1365 - 313 x.1999.00538.x。
- 19.
Gramene通路工具(RiceCyc)。[http://www.gramene.org/pathway/]
- 20.
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT, Harris MA, Hill DP, Issel-Tarver L, Kasarskis A, Lewis S, Matese JC, Richardson JE, Ringwald M, Rubin GM, Sherlock G:基因本体论:生物学统一的工具。王文杰,2000,25:25-29。10.1038/75556。
- 21.
RiceTFDB。[http://ricetfdb.bio.uni-potsdam.de/]
- 22.
阿特舒尔SF,吉什W,米勒W,迈尔斯EW,李普曼DJ:基本的局部对齐搜索工具。中华分子生物学杂志,2000,26(3):344 - 344。
- 23.
Reed JW: Aux/IAA蛋白在拟南芥中的作用和活性。植物科学进展,2001,6:420-425。10.1016 / s1360 - 1385(01) 02042 - 8。
- 24.
Tiwari SB, Wang XJ, Hagen G, Guilfoyle TJ: AUX/IAA蛋白是一种活性抑制因子,其稳定性和活性受生长素调控。植物科学学报,2001,13:2809-2822。10.1105 / tpc.13.12.2809。
- 25.
刘志斌,Hagen G, Guilfoyle TJ:大豆的G-box结合蛋白与大豆GH3启动子中的E1生长素反应元件结合,并含有富含脯氨酸的抑制结构域。植物学报,1997,15(4):397-407。10.1104 / pp.115.2.397。
- 26.
Ulmasov T, Hagen G, Guilfoyle TJ: ARF1,一种结合生长素反应元件的转录因子。科学通报,1997,27(3):344 - 344。10.1126 / science.276.5320.1865。
- 27.
李志强,李志强,李志强,等。拟南芥生长发育及生长素诱导表达的研究进展prha同源框基因。植物学报,1997,26(4):457 - 457。10.1046 / j.1365 - 313 x.1997.00635.x。
- 28.
Kagaya Y, Ohmiya K, Hattori T: RAV1是一种新型dna结合蛋白,通过两个独特的dna结合域与高等植物中特有的二部识别序列结合。中国生物医学工程学报,1999,27(4):457 - 457。10.1093 / nar / 27.2.470。
- 29.
Ezhova TA, Soldatova OP, Kalinina AIu, Medvedev SS:互动ABRUPTUS / PINOID而且多叶的花形态发生过程中的基因拟南芥(l)Heynh。遗传学报,2000,36:1682-1687。
- 30.
会议A, Nemhauser JL, McColl A, Roe JL, Feldmann KA, Zambryski PC:拟南芥花分生组织和生殖器官的ETTIN模式。《发展与发展》,1997,34(4):481- 491。
- 31.
雷明顿DL, Vision TJ, Guilfoyle TJ, Reed JW:中国市场多元化模式的对比辅助/ IAA而且东盟地区论坛基因家族。中国农业科学,2004,27(3):344 - 344。10.1104 / pp.104.039669。
- 32.
Goda H, Sawa S, Asami T, Fujioka S, Shimada Y, Yoshida S:拟南芥生长素调控和油菜素类固醇调控基因的综合比较。中国农业科学,2004,27(4):457 - 457。10.1104 / pp.103.034736。
- 33.
Nag R, Maity MK, Dasgupta M: ABA不敏感3样因子对ABA和生长素反应启动子的双DNA结合特性。中国生物医学工程学报,2005,29(3):344 - 344。10.1007 / s11103 - 005 - 1387 - z。
- 34.
杨G,中村H,市川H,北野H,小松S:OsBLE3是一种油菜素内酯增强基因,参与水稻的生长。植物化学学报,2006,27(3):344 - 344。10.1016 / j.phytochem.2006.05.026。
- 35.
徐春春,李志强,李志强,马德平:脂质转移蛋白基因的转录调控LTP3一种新的MYB蛋白植物科学学报,2005,29(3):344 - 344。10.1016 / j.plantsci.2004.07.033。
- 36.
白峰,华森,王丽娟,魏丹,王丽娟,王丽娟PsPK2,一个PINOID类似豌豆的基因(Pisum一).植物科学学报,2005,29(4):344 - 344。10.1016 / j.plantsci.2005.01.005。
- 37.
Szopa J, Lukaszewicz M, Aksamit A, Korobczak A, Kwiatkowska D:马铃薯14-3-3蛋白基因16R亚型的结构、表达和启动子分析。植物化学学报,2003,41:417-423。10.1016 / s0981 - 9428(03) 00048 - 2。
- 38.
Navarro-Avino JP, Bennett AB: Ca的角色2 +- atp酶在ABA和IAA诱导产生特异性Ca2 +信号。生物化学学报,2005,29(4):531 - 531。10.1016 / j.bbrc.2005.01.142。
- 39.
Ishiki Y, Oda A, Yaegashi Y, Orihara Y, Arai T, Hirabayashi T, Nakagawa H, Sato T: 1-氨基环丙烷-1-羧酸合成酶基因的克隆(CMe-ACS2以及ACS基因在黄化甜瓜幼苗和甜瓜果实中的表达。中国植物科学,2000,29(3):344 - 344。10.1016 / s0168 - 9452(00) 00298 - 3。
- 40.
葛林,陈辉,蒋建峰,赵颖,徐明明,徐元宇,谭克海,徐志辉,冲坤:过表达OsRAA1在转基因水稻植株中引起多效表型,包括改变叶、花、根发育和根对重力的反应。植物科学学报,2004,26(3):344 - 344。10.1104 / pp.104.041996。
- 41.
Borisov AY, Madsen LH, Tsyganov VE, Umehara Y, Voroshilova VA, Batagov AO, Sandal N, Mortensen A, Schauser L, Ellis N, Tikhonovich IA, Stougaard J: TheSym35豌豆根结发育所需的基因是一个同源基因外祖母从Lotus对虾.中国生物医学工程学报,2003,31(3):344 - 344。10.1104 / pp.102.016071。
- 42.
李艳,刘志斌,石晓霞,陈志强,陈志强,陈志强。大豆中生长素诱导因子的研究阿富汗二月启动子。植物营养学报,1994,26(3):344 - 344。10.1104 / pp.106.1.37。
- 43.
王晓明,王晓明,王晓明,等。一种具有蛋白磷酸酶活性的植物防御转录因子。中国生物工程学报,2003,26(3):366 - 366。10.1093 / emboj / cdg323。
- 44.
Esmon CA, Tinsley AG, Ljung K, Sandberg G, Hearne LB, Liscum E:生长素和生长素依赖转录的梯度在回归性生长反应之前。美国国家科学研究院。2006, 30(4): 344 - 344。10.1073 / pnas.0507127103。
- 45.
Okumoto S, Schmidt R, Tegeder M, Fischer WN, Rentsch D, Frommer WB, Koch W:拟南芥木质部薄壁组织和发育种子中特异性表达的高亲和力氨基酸转运蛋白。中国生物医学工程学报,2002,27(4):457 - 457。10.1074 / jbc.M207730200。
确认
这项工作得到了一代挑战计划SP4 2005-32项目的资助。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
KD设计了算法,完成了所有的编程,并进行了工具的可行性测试。AH帮助准备测试数据集和文献检索。TN提供了已知的内部数据库独联体-工具引用的元素。参考数据由KSa和KSu准备。RM、MJM和RB在实现和主机设置方面提出了许多技术建议。RB也更正了这篇稿件的英文。SK构思了这项研究,并参与了设计和协调。所有作者都阅读并批准了手稿。
电子辅助材料
12870 _2007_228_moesm2_esm.csv
附加文件2:可行性测试中使用的转录单位(TUs)。生长素诱导基因从RiceTFDB 2.0中提取(第一列)。对应的全长cdna由BLASTN指定(第二列),并翻译成在Pseudomolecule ver中定义的tu。4(第三列)。(csv 2kb)
12870 _2007_228_moesm3_esm.txt
附加文件3:初步清单独联体-由模因分析列出的tu候选元素,见补充表S2。详情见正文。(txt 61kb)
12870 _2007_228_moesm4_esm.csv
附加文件4:关联规则分析结果独联体- MEME列出的候选元素。第一列:检查序列。第2列:目标基因列表的28个单位内具有指定基序的单位数。第三列:KOME数据库中存储的22 943个TU中包含指定motif的TU数量。第四列:电梯价值。(csv 100kb)
12870 _2007_228_moesm5_esm.csv
附加文件5:存储在KOME数据库中的22 943 tu中对Supplementary Table S1中所示motif的序列搜索结果。第一栏:检查过的tu。第2列:在TU上游区域发现的基序。其他列:每个TU上游区域的基序位置。(CSV 467 KB)
12870 _2007_228_moesm6_esm.csv
附加文件6:序列搜索后关联规则分析结果,如补充表S6所示。第一列:检查序列。第2列:目标基因列表的28个单位内具有指定基序的单位数。第三列:KOME数据库中存储的22 943个TU中包含指定motif的TU数量。第四列:电梯价值。(CSV 1007字节)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
Doi, K,保坂,A,永田,T。et al。开发了一种新型的数据挖掘工具独联体水稻基因启动子区域的-元素。BMC植物生物学8,20(2008)。https://doi.org/10.1186/1471-2229-8-20
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-20
关键字
- 水稻基因组测序
- 主题搜索
- 候选人的主题
- 国际水稻基因组测序计划
- 提升指数