摘要
背景
葡萄科由许多不同的葡萄品种组成,生长在各种气候条件下。在过去的几年中,一些研究产生了不同的功能基因组信息葡萄属品种和栽培,包括欧洲葡萄,葡萄.我们的目标是开发一个综合的网络数据源的Vitaceae。
描述
VitisExpDB是一个在线MySQL-PHP驱动的关系数据库,它包含注释EST和基因表达数据诉酿酒用葡萄和非酿酒用葡萄葡萄品种和变种。目前,该数据库存储了来自8个物种/杂种的约32万个EST序列、它们的注释(BLAST top匹配)细节和基于基因本体的结构化词汇。每个EST在其他物种和品种中的假定同源性,以及它们的核苷酸识别率、系统发育关系和常见引物的信息都可以检索到。数据库还包含了高密度60-mer基因表达芯片的探针序列和注释特征信息,该芯片由约2万组非冗余ESTs组成。最后,该数据库包含14个经过处理的全局微阵列表达谱集。这些表达谱中的12组数据已经被映射到代谢途径上。一个用户友好的网页界面,具有多个搜索索引和广泛的超链接结果功能,允许高效的数据检索。已经添加了一些与数据库以及其他序列分析工具交互的在线生物信息学工具。此外,用户可以向数据库提交他们的ESTs。
结论
该数据库为葡萄群落提供了基因组资源,可用于收集的基因功能分析、葡萄基因组注释和基因功能鉴定。VitisExpDB数据库可通过我们的网站获得http://cropdisease.ars.usda.gov/vitis_at/main-page.htm.
背景
表达序列标签(ESTs)是一种丰富的基因组资源,截至2007年7月,基因银行存储了44,203,116个ESTs。无害环境技术是所有物种的重要基因组资源,在没有基因组序列的系统中更是如此。在这种情况下,无害环境技术被用作结构基因组注释的基础。est对于研究全局表达模式也具有根本性的重要性[1].有效生物信息学工具的使用拓宽了EST分析在基因组学、标记开发和基因组注释等领域的应用[2- - - - - -4].
的诉酿酒用葡萄欧洲葡萄种植面积超过740万公顷,是世界上最具经济价值的水果品种。葡萄是生产水果,果汁,葡萄干,葡萄酒和烈酒。目前,已经有了一个基因组草案,全面的基因组注释仍在进行中[5].最近,几个葡萄属物种基因组项目促进了公众可获得的无害环境技术的数量不断增加。有许多北美本土葡萄品种和栽培品种培育的这些品种有经济价值,也有丰富的种质资源,抵抗不同的生物和非生物胁迫(见附加文件1)。
如今,这一数字已超过32万葡萄属存入NCBI GenBank的ESTs。其中约90%属于诉酿酒用葡萄,其次为3.0%诉shuttleworthii.其余的包括无害环境技术诉arizonica,v -而且诉锐利等(表1)。这些320000年葡萄属大约200,000个ESTs诉酿酒用葡萄,v -而且诉rupestris×诉arizonica已被表征[6- - - - - -8].这些est来自于生物和非生物胁迫下不同组织和发育阶段的cDNA文库[6- - - - - -8].类似地,另外5000个est克隆自抑制消减杂交(SSH)库诉rupestris×诉arizonica混血儿的反应Xylella fastidiosa最近对感染情况进行了分析[9].据我们所知,没有PUBMED的参考文献描述了完整的可用的集合葡萄属est序列。
2004年,作为基因芯片的一部分®联盟计划,Affymetrix介绍了16K阵列基因芯片®葡萄(葡萄)基因组阵列。1.0 (Affymetrix®Inc., Santa Clara, CA),主要为诉酿酒用葡萄微阵列研究。最近,有六篇关于使用cDNA或oligo阵列测量花和浆果皮肤发育的基因表达谱的mRNA表达谱研究的报道[10- - - - - -12],以及来自葡萄枝条组织中的水分亏缺和等渗透盐胁迫[13,以及与病毒性疾病相关的表达谱[14和浆果组织的组织特异性图谱[15].GrapePLEX是一个符合miame和植物本体增强的表达数据库葡萄属微阵列研究是植物表达数据库[PLEXdb]的一部分[16].目前,来自基因芯片的表达数据集®葡萄(葡萄)基因组阵列。1.0 (Affymetrix®公司,圣克拉拉,加州)可以存入这里。
其他在线专门数据库包含葡萄属ESTs,如DFCI等葡萄基因索引数据库中存储的信息诉酿酒用葡萄ESTs(共191,616个),包括初步共识序列的信息、它们的BLAST和基因本体论细节[17].此外,还有一些函数工具,它们允许比较来自不同库的EST表达式概要文件,以及关于备选的香料表单的信息等。同样,EST序列信息及其微阵列探针信息可以从植物基因组数据库PlantGDB中获得,其中约有21万诉酿酒用葡萄ESTs被存储[18].上的信息诉酿酒用葡萄不同代谢途径的ESTs可从KEGG网站获得[19].来自不同的聚类EST序列的最新数据葡萄属具有大量EST集合的物种可从TIGR植物转录本集合数据库中获得[20.].的v -诺顿EST数据库可在密苏里州立大学山格罗夫分校获得,还包括一个名为GREED的防御基因数据库[21].
开发VitisExpDB是为了管理和允许对所有可用的葡萄EST源的方便访问,并与微阵列数据集成,特别是来自非葡萄的定制阵列的数据酿酒用葡萄品种和种,这是已知的生物和非生物抗逆性种质资源。假定的同源物已经在不同的葡萄属品种和种及与模式植物拟南芥.开发了几种搜索和检索表单以及在线生物信息学工具,以创建一个全面的数据仓库葡萄属基因组学的研究。数据库将每六个月更新一次,提供新的数据集。
结构和内容
数据库体系结构
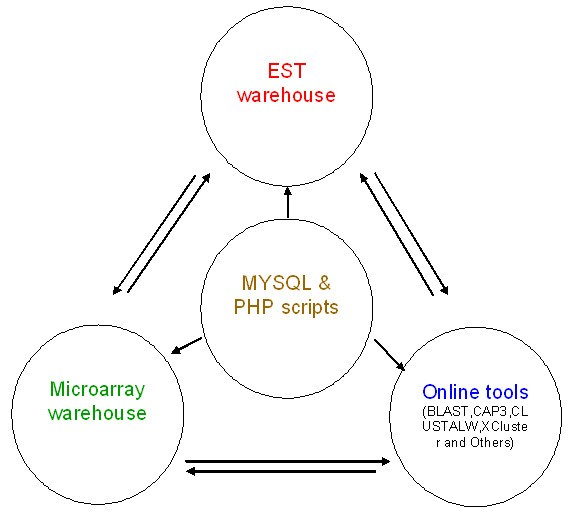
服务器使用Red Hat Enterprise Linux 4 RPM (x86)操作系统。关系数据库是使用MySQL 4.0作为后端开发的。该网站由Apache服务器提供动力。基于HTML和php的web界面已经被开发出来,可以动态地执行MYSQL查询,也可以运行不同的依赖软件1)。采用ADO数据库和BioPHP软件包与MYSQL数据库进行交互。
数据管理
est序列
EST集从NCBI数据库(UniGene for诉酿酒用葡萄其余的最好葡萄属物种,表1),并使用BLASTX算法对使用NCBI BLAST服务的整个'nr'蛋白质数据库进行搜索。通过使用Personal BLAST Navigator (PLAN)软件服务器重复相似性搜索,再次确认了这些结果[22].在这两种情况下,身份截止E值均为104是使用。基因本体论术语[23]使用高通量基因本体功能注释(Ht-Go-Fat)工具包生成[24].序列相似性搜索使用默认BLAST搜索参数,截断E值为104.生成的GO术语被超链接到最新版本“基因本体,OBO v1.2”中的定义和本体,该版本从GO网站[25].该数据库还列出了不同物种的相似基因序列葡萄属我们最近开发的nwaycompp工具[26].为此,从诉酿酒用葡萄,诉shuttleworthii,诉arizonica,v -,诉锐利使用期望得分截止为E-010的BLASTN程序进行反向BLAST搜索。潜在的应用识别这些假定同系物跨越不同葡萄属包括推断克隆est的推定功能,以及基于从保守区域设计的引物克隆其他品种的est。同样,这些假定的同源性可用于开发SSR和SNP标记,用于品种鉴定和构建标记辅助育种的遗传图谱。为诉酿酒用葡萄数据集中,从UniGene构建的#21中下载的最新表达配置文件已与PHP脚本打包,以动态生成跨越9种不同组织类型的数字EST表达配置文件。
微阵列数据集
我们设计了一个诉酿酒用葡萄而且non-viniferaest富集自定义高密度微阵列基因芯片共有20020个est(1947来自SSH文库,40来自cDNA-AFLP实验,10014来自诉酿酒用葡萄5470年,从V.shuttleworthii1219年,从v -780年,从诉rupestris×诉arizonica和588年从诉锐利)。该数据库包括分析的植物致病菌感染宿主的全局微阵列表达谱x fastidiosa它会导致葡萄的皮尔斯病。数据来自36个杂交实验,分别来自三个时间点:早期(1周)、中期(6周)和晚期(10周),分别来自抗性和易感基因型的茎和叶的感染和未感染组织的疾病发展阶段。此外,自定义微阵列上每个EST序列的DNA序列信息以及斑点探针和注释细节都是可访问的。此外,使用TAIR的路径工具组学查看器,生成的表达谱已映射到25个代谢通路[27].的归一化和折叠变化计算表达式值葡萄属而且x fastidiosa相互作用实验被映射到这些通路上。假定的同系物拟南芥基因id葡萄属为此目的使用了微阵列芯片上的ESTs。微阵列实验的细节可在网站[27].另外两个已发表的自定义微阵列数据集[11,12]也已被添加到数据库中。数据将每六个月更新一次。
效用和讨论
Web界面
在主搜索页面上,有两个侧面板,左边的面板列出了指向不同搜索页面和在线工具的超链接。这里列出了三个主要组成部分:无害环境技术、微阵列和生物信息学工具。右边的面板列出了到描述数据库内容的Web Pages的超链接。这些页面提供了实验设置、数据分析和其他相关文本等信息。为数据挖掘、分析和可视化开发了许多有用的查询接口。这包括简单的和高级的搜索表单,可以为EST和微阵列组件提供单个查询或多个查询搜索选项。
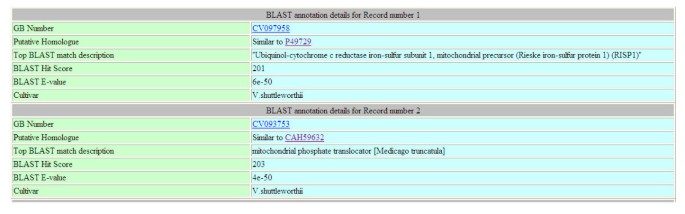
数据库的EST成分可以用GB号、GI号、基因本体ID、酶号或假设函数作为关键词进行检索。其他附加参数,如从下拉列表中选择特定的物种/品种,设置截止E值和截止BLAST评分,可以包括在内,以构建严格的查询。典型的结果页面显示与查询匹配的ESTs、单个EST序列、其描述、EC号和其基因本体分类(参见图)2A-B和参见附加文件3.)。结果页面中包含了大量超链接,将结果术语链接到数据库中的其他表格数据,以及链接到诸如NCBI和AmiGO数据库等外部数据库(参见附加文件中的图2c和D)2)。结果页面包括PHP脚本动态生成的表和图。为诉酿酒用葡萄代表所存储est的主要部分的ESTs,用户还可以检索数字表达谱以及9种不同组织类型的表达指数百分比。
EST数据库查询的示例结果页.该图显示了一个页面合成图像的上四分位数,该页面显示了查询词“线粒体”的BLAST搜索结果。完整图像请参见附加文件2.
提供了一个单独的网页来列出该专业中假定的同源基因集葡萄属使用nWayComp品种。这些信息可以从主数据库页面左侧的“Get Homologs”链接中获得。主结果页面有一个有两列的表,第一列显示比较类型,第二列显示该组合的假定同源基因集的数量(图3,并参阅附加文件4查看完整图片)。第二列中的数字被超链接到打开的HTML分析文件,该文件显示一个表,其中包含该组合的所有假定同源基因集,如图3B所示(参见附加文件)3.)。本页的第一列显示了一个排序的假定同源基因集,每个基因集一个葡萄属按其序列相似性升序排列的物种或变种。该表的第二列是假定同源基因之间序列一致性的标准差。对于每一组假定的同源基因,第3至第6列将有超链接到文件的图标,该文件将显示一个标识表,显示假定的同源基因序列标识的百分比,第3列;图3C(参见附加文件)3.),系统发育树,第4列;图3D(参见附加文件)3.), PCR克隆常用引物按扩增子大小由大到小排列,列5;图3E(参见附加文件3.), FASTA forma中的基因序列,第6列;图3F(参见附加文件3.)。
假定同系词的结果页示例.这张图显示了合成图像的上四分位数,第一列是品种组合,第二列是该组合的基因数量。完整图像请参见附加文件3..
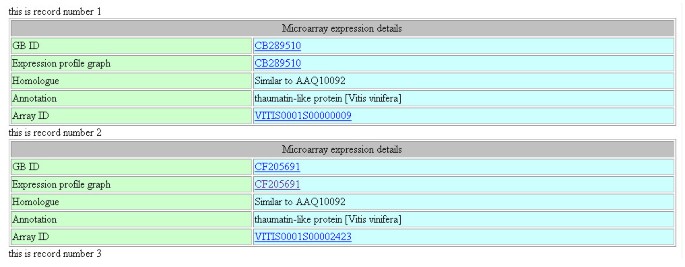
要查询微阵列数据,可以使用一个简单的表单,用户可以在其中输入一个GB数字,一个阵列ID(s)或一个假定的函数(图4 a - B和额外的文件4)。另外,还设计了一个高级的搜索表单,用户可以根据该表单构建严格的布尔搜索,如截断表达式值或选择实验的特定阶段,组织或基因型,或基于拟南芥基因,用于数据检索。典型的微阵列搜索结果将包括序列、注释和探针细节,以及动态生成的序列后记图像和注释细节,以及图形形式的表达式结果,如图4C所示(参见附加文件)4)。在高级搜索页面,提供了一个附加的表单,可用于检索微阵列探针的信息,图4D(参见附加文件)4)。
Microarray数据库查询的示例结果页.该图显示截断页面合成图像的上四分位数,显示查询词“thaumatin”的注释(BLAST顶部匹配描述)搜索结果。完整图像请参见附加文件4.
在微阵列仓库下,设计了一个独立的HTML页面,其中包含各种代谢途径的超链接图标。在研究的12个微阵列实验中,每一个都有25种不同的路径,这些路径由映射差异调控的图像生成的HTML页面创建葡萄属在微阵列数据集小节中描述的ESTs。一个单独的Web页面列出了不同的路径拟南芥基因id已与VitisExpDB中假定的同源ESTs相关联,以检索有关有趣的拟南芥基因的进一步信息。此外,一个包含所有自定义微阵列数据集的微阵列数据库正在开发中。
在线工具
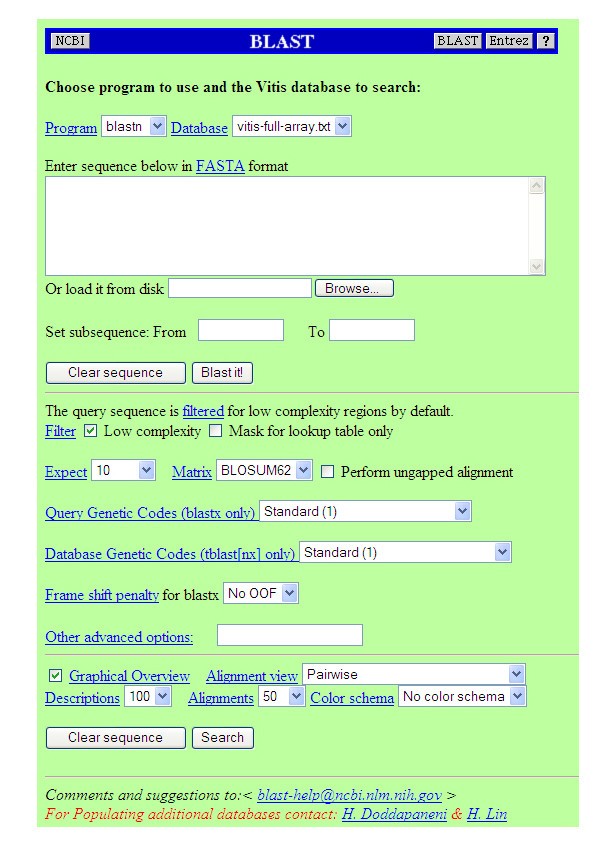
一些在线工具,如BLAST、CLUSTALW、Tandem Repeats Finder (TRF)和Cluster,已经被用于与我们的数据库进行接口。其他的,如CAP3和其他BioPHP模块(参见附加文件5),可独立访问。带注释的葡萄属BLAST数据库中增加了EST和微阵列探针序列集等数据库(和完整数组BLAST db)这将帮助葡萄科学界快速有效地识别和注释ESTs。BLAST工具接受FASTA格式的单个或多个序列文件。为了便于使用,所有NCBI的BLAST软件功能都被保留了下来5,并参阅附加文件6查看完整图片)。CLUSTALW工具可用于一次对齐最多20个序列,用户可以将感兴趣的序列的GB接入作为逗号分隔的列表输入,如图5B所示(参见附加文件)6)。每次运行的结果文件都保存为用户提供的文件名,以供后续检索,见图5C(参见附加文件)6)。已经开发了一个TRF搜索页面,用户可以在其中自定义搜索序列中的重复,并尝试不同的搜索选项,图5D(参见附加文件)6)。输出文件以用户输入选项作为前缀保存,以避免覆盖文件,如图5E所示(参见附加文件6)。这与显示静态页面有很大不同,在静态页面中,串行重复的预运行数据会像大多数EST数据库中那样显示,因此无法对单个EST序列应用不同的搜索参数。为了分析基于微阵列表达谱的功能相关基因,配置了最新的Linux版本的XCluster软件[28].可以将XCluster中的自定义结果文件下载到本地硬盘驱动器并存储起来以备将来使用。开发了一个EST序列提交表单,用户可以直接向数据库提交他们的序列。将生成一个确认电子邮件,提醒提交者提交成功。添加的序列将在进行矢量污染、适配器污染等质量控制分析后,每月进行注释,并添加到主数据库中,供后期公共检索。在主Web页面底部的左侧提供了一个到Web页面的链接,其中列出了所有主要的和有趣的内容葡萄属EST和基因组资源网页。
在线工具和后续结果页面的屏幕截图.此图显示了合成图像(BLAST web界面)的上四分位数,完整图像请参阅附加文件6.
除了拥有带有注释和基因本体管理的最新EST数据池外,VitisExpDB还具有其他独特的功能,例如关于来自不同基因的假设同源物的信息葡萄属物种,它们的信息拟南芥假定的同源性,微阵列和EST数据库的集成,转录反应对代谢途径的映射和一些数据分析工具。
结论
对于广泛的应用程序来说,VitisExpDB数据库是一个有价值的资源葡萄属遗传学与育种,基因组学,蛋白质组学和基因组注释。该数据库未来的扩展计划包括编目来自ESTs的剪接变体,基于基因组数据支持的初步共识序列识别全长ESTs,以及生成基因表达的基因组景观图。开发交叉参考工具,以便用户比较Affymetrix基因芯片阵列和其他定制阵列之间的数据,这也是未来数据库扩展的一部分。当有更多信息可用时,将频繁更新VitisExpDB数据库。
可用性和需求
资料库开放并免费提供[29].
项目名称:VitisExpDB数据库;
项目主页:http://cropdisease.ars.usda.gov/vitis_at/main-page.htm;
操作系统:平台独立;
编程语言:Perl、HTML、MySQL、PHP;
许可:GNU。
参考文献
- 1.
表达序列标签:全基因组序列的替代或补充。植物科学进展,2003,21(3):369 - 369。10.1016 / s1360 - 1385(03) 00131 - 6。
- 2.
董强,Kroiss L, Oakley FD, Wang BB, Brendel V:植物系统中EST的比较分析。方法酶学杂志,2005,32(4):418 -418。
- 3.
王晓燕,王晓燕,王晓燕。EST分析植物代谢的研究进展。植物学报,2000,3:224-228。
- 4.
Gupta PK, Rustgi S:来自高等植物基因组转录/表达区域的分子标记。功能整合基因组学,2004,4:139-162。10.1007 / s10142 - 004 - 0107 - 0。
- 5.
法意公共财团的葡萄藤基因组特征," O, Aury JM,诺尔B, Policriti, Clepet C, Casagrande, Choisne N,奥堡年代,Vitulo N, Jubin C, Vezzi, Legeai F, Hugueney P,达席尔瓦C,霍纳D,云母E, Jublot D J,也不会Bruyere C, Billault, Segurens B, Gouyvenoux M, Ugarte E, F Cattonaro, Anthouard V,维科V,德尔Fabbro C,塞德cedreic Alaux M, Di Gaspero G,杜马斯V,菲利斯N,主料,Juman我Moroldo M, Scalabrin年代,Canaguier, Le Clainche我Malacrida G,Durand E、Pesole G、Laucou V、Chatelet P、Merdinoglu D、Delledonne M、Pezzotti M、Lecharny A、Scarpelli C、Artiguenave F、Pe ME、Valle G、Morgante M、Caboche M、Adam-Blondon AF、Weissenbach J、Quetier F、Wincker P:葡萄基因组序列提示主要被子植物门的祖先六倍化。自然学报,2007,449:463-467。10.1038 / nature06148。
- 6.
Da Silva FG, Iandolino A, Al-Kayal F, Bohlmann MC, Cushman MA, Lim H, Ergul A, Figueroa R, Kabuloglu EK, Osborne C, Rowe J, Tattersall E, Leslie A, Xu J, Baek J, Cramer GR, Cushman JC, Cook DR:葡萄转录组的特征。多个表达序列标签的分析葡萄属浆果发育过程中基因表达纲目的种类和发展。植物生理学报,2004,24(3):379 - 379。10.1104 / pp.105.065748。
- 7.
Moser C、Segala C、Fontana P、Salakhudtinov I、Gatto P、Pindo M、Zyprian E、Toepfer R、Grando MS、Velasco R:不同器官表达序列标签的比较分析葡萄中国生物医学工程学报,2004,23(5):378 - 378。10.1007 / s10142 - 005 - 0143 - 4。
- 8.
Peng FY, Reid KE, Liao N, Schlosser J, Lijavetzky D, Holt R, Martinez Zapater JM, Jones S, Marra M, Bohlmann J, Lund ST: est在中国的生成葡萄属葡萄葡萄(赤霞珠)和鲜食葡萄(汉堡麝香葡萄)以及在浆果发育中具有潜在作用的新候选基因的发现。中国生物工程学报,2007,29(4):447 - 447。10.1016 / j.gene.2007.07.016。
- 9.
Lin H, Doddapaneni H, Takahashi Y, Walker MA:葡萄对Xylella fastidiosa感染。植物工程学报,2004,24(3):366 - 366。
- 10.
Terrier N, Glissant D, Grimplet J, Barrieu F, Abbal P, Couture C, Ageorges A, Atanassova R, Léon C, Renaudin JP, Dédaldéchamp F, Romieu C, Delrot S, Hamdi S:等基因特异性寡核苷酸阵列揭示葡萄浆果中基因表达的多方面变化(葡萄l .)发展。植物学报,2005,32(4):347 - 347。10.1007 / s00425 - 005 - 0017 - y。
- 11.
Waters DL, Holton TA, Ablett EM, Lee LS, Henry RJ:发育中葡萄的cDNA微阵列分析(葡萄简历。设拉子)浆果的皮肤。功能整合基因组学,2005,5:40-58。10.1007 / s10142 - 004 - 0124 - z。
- 12.
Waters DL, Holton TA, Ablett EM, Lee LS, Henry RJ:成熟葡萄酒葡萄浆果皮肤转录组。植物科学进展,2006,29(6):689 - 689。10.1016 / j.plantsci.2006.03.002。
- 13.
Cramer GR, Ergul A, Grimplet J, Tillett RL, Tattersall EA, Bohlman MC, Vincent D, sonderger J, Evans J, Osborne C, Quilici D, Schlauch KA, Schooley DA, Cushman JC:葡萄的水和盐胁迫:转录本和代谢物谱的早期和晚期变化。功能整合基因组学,2007,7:111-134。10.1007 / s10142 - 006 - 0039 - y。
- 14.
Espinoza C, Vega A, Medina C, Schlauch K, Cramer G, Arce-Johnson P:葡萄品种中相容病毒病相关基因表达。功能整合基因组学,2007,7:95-110。10.1007 / s10142 - 006 - 0031 - 6。
- 15.
Grimplet J, Deluc LG, Tillett RL, Wheatley MD, Schlauch KA, Cramer GR, Cushman JC:葡萄浆果组织中组织特异性mRNA表达谱分析。中国生物医学工程学报,2007,29(4):447 - 447。
- 16.
GrapePLEX的网址。[http://www.plexdb.org/plex.php?database=Grape]
- 17.
当时DFCI葡萄基因数据库的网址。[http://compbio.dfci.harvard.edu/tgi/cgi-bin/tgi/gimain.pl?gudb=grape]
- 18.
植物基因组数据库:PlantGDB。[http://gremlin3dev.gdcb.iastate.edu/]
- 19.
KEGG葡萄代谢途径图谱数据库的网址。[http://www.genome.jp/kegg-bin/show_organism?menu_type=pathway_maps;org=evvi]
- 20.
TIGR工厂转录本程序集的网址。[http://plantta.tigr.org/cgi-bin/plantta_release.pl]
- 21.
葡萄抗病基因ESTs和表达数据库(GREED)的网址。[http://mtngrv.missouristate.edu/CGB/NortonGeneDatabase/NortonGeneDatabase.htm]
- 22.
何静,戴鑫,赵鑫:PLAN:一个用于自动化高通量BLAST搜索和管理和挖掘结果的web平台。生物信息学,2007,8:53-10.1186/1471- 25-8-53。
- 23.
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT, Harris MA, Hill DP, Issel-Tarver L, Kasarskis A, Lewis S, Matese JC, Richardson JE, Ringwald M, Rubin GM, Sherlock G:基因本体论:统一生物学的工具。基因本体论联盟。自然遗传学,2000,25:25-29。10.1038/75556。
- 24.
高通量基因本体功能注释(Ht-Go-Fat)工具包网址[199.133.147.108/ Ht-Go-Fat .htm]
- 25.
- 26.
Yao J, Lin H, Doddapaneni H, Civerolo EL: nWayComp:一种通用的DNA和蛋白质序列比较工具。生物工程学报,2007,20-
- 27.
TAIR的路径工具组学查看器[19]。[http://www.arabidopsis.org:1555/expression.html]
- 28.
XCluster软件的Web地址。[http://genetics.stanford.edu/~sherlock/cluster.html]
- 29.
VitisExpDB的Web地址。[http://cropdisease.ars.usda.gov/vitis_at/main-page.htm]
确认
我们非常感谢加州柑橘研究委员会(CRB项目编号为5300-05F)对这项工作的部分支持,以及加州食品和农业部的皮尔斯病委员会。我们感谢PLAN服务器的作者不时地满足我们对大规模数据搜索的需求。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
HD进行了微阵列实验,设计了数据库,并开发了web界面和数据分析工具。JY开发了NWayComp软件,并参与了数据库的初步开发。HL和AW有助于相似搜索参数的开发,HL和EC有助于微阵列数据解释。EC获得了加州柑橘研究委员会的资助。HL, AW和EC协调项目。所有的作者都阅读并认可了最终的手稿。
电子辅料
12870 _2007_231_moesm1_esm.doc
附加文件1:est策划的基因背景。记录产生ESTs的葡萄藤的遗传背景细节。(DOC 28 KB)
12870 _2007_231_moesm2_esm.jpeg
附加文件2:Microarray数据库查询的示例结果页。(A)截短页面截图,显示查询词“thaumatin”的注释(BLAST top匹配描述)搜索结果。(B)显示FASTA序列和品种细节的页面视图。进一步的信息提供到NCBI的GenBank的外部链接。(C)以动态生成的图形形式显示表情轮廓的结果的页面视图,显示阶段、表情值和表情在12个数据点上的百分比。while框中的值是用于处理控件的折叠变化差异。柱状顶部给出的百分比是用该阶段的折叠变化值除以所有12个阶段的折叠变化值计算出来的。(D)显示微阵列探针信息的页面视图。(JPEG 1 MB)
12870 _2007_231_moesm3_esm.jpeg
附加文件3:EST数据库查询的示例结果页。(A)“线粒体”搜索结果的截短页面截图。BLAST匹配e值大于1e6被描述为“弱相似”小于等于“相似”(B)超链接基因本体论术语和酶的详细信息(C)相关GO术语的描述,带有AmiGO数据库中该术语的生成本体论树视图页面的超链接(D) GO术语“GO:0003954”的树视图图像。(JPEG 992 KB)
12870 _2007_231_moesm4_esm.jpeg
附加文件4:假定同系物的结果页示例。(A)第一栏显示品种组合,第二栏显示该组合的基因数量的页面视图;(B)该比较型中假定的同源基因集,第一列每行一组;(C)假定同系物集的百分比同一性;(D)使用Phylip程序构建无根邻接系统发育树,默认设置显示它们之间的关系;(E)该组常用引物删节表;和(F) FASTA格式的截短DNA序列。(JPEG 756 KB)
12870 _2007_231_moesm5_esm.doc
附加文件5:生物信息学工具的描述。文件有使用生物信息学工具的简要描述。(医生30 KB)
12870 _2007_231_moesm6_esm.jpeg
附加文件6:在线工具和后续结果页面的屏幕截图。(A) BLAST web界面(B) CLUSTALW web界面(C) CLUSTALW搜索的结果页面,带有后续数据文件的链接。用户可以将这些文件保存到本地硬盘上,以便进一步分析和解释。(D)后机基金会网页界面(E)串联重复搜索的总结结果页与结果页的链接。(JPEG 985 KB)
权利和权限
开放获取本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(https://creativecommons.org/licenses/by/2.0),它允许在任何媒体上不受限制地使用、分发和复制,只要原著被恰当地引用。
关于这篇文章
引用这篇文章
多达帕内尼,H.,林,H.,沃克,文学硕士et al。葡萄功能基因组数据库。BMC植物杂志8日,23(2008)。https://doi.org/10.1186/1471-2229-8-23
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-23
关键字
- 基因本体论
- 差减杂交
- 假定的同族体
- 葡萄属物种
- 串联重复序列仪