摘要
背景
转录和转录后水平的小rna引导基因沉默已成为动植物基因调控的一种重要模式。到目前为止,通过对水稻小RNA文库的常规测序,发现了大多数保守的miRNAs。对小RNA文库进行深度测序是发现任何生物中罕见的和谱系和/或物种特异性microRNAs (miRNAs)的有效方法。
结果
为了在水稻中识别新的miRNAs和可能受非生物胁迫调节的小RNA,从对照水稻幼苗和暴露于干旱或盐胁迫的水稻幼苗中构建了三个小RNA文库,然后进行了磷酸磷酸测序。从对照、干旱和盐胁迫文库中分别获得了58,781、43,003和80,990个独特的基因组匹配小rna。序列分析证实了大部分保守mirna在水稻中的表达。重要的是,研究发现了23个新的miRNAs,其中大部分都来自水稻基因组的一个独特位点。其中6个新的miRNAs在其他单克隆中是保守的。此外,我们鉴定了40个候选miRNAs。允许一个miRNA和它的靶mRNA之间不超过3个不匹配,我们预测了9个新的miRNA的20个靶。
结论
深度测序被证明是一种有效的策略,可以在水稻中发现23个低丰度的新miRNAs和40个候选miRNAs。
背景
在植物中,转录或转录后基因沉默是由基因组编码的21-24-nt小rna指导的。这些小rna可以分为两大类:microRNAs (miRNAs)和短干扰rna (siRNAs)。miRNAs通过引导mRNA降解或翻译抑制在转录后基因沉默中发挥作用[1- - - - - -4].根据基因调控的大小和方式,内源性sirna进一步被分为两大类。以反式作用siRNAs (tasiRNAs)和其他siRNAs为代表的21 nt大小的sinas在转录后水平指导目标mrna的切割,而24 nt大小的sinas涉及导致转录基因沉默的DNA和组蛋白修饰[5,6].
MicroRNAs最早是在秀丽隐杆线虫通过基因筛选异常发育[7,8之后在几乎所有经检查的多细胞真核生物中都发现了这种基因。成熟miRNAs是单链~ 21nt的小RNA,由单链初级转录本(pri-miRNA)形成不完善的发夹状结构,通过双链RNA识别RNase III酶(动物的Drosha和DCR1,植物的DCL1)的一系列酶活性产生。植物核定位的Dicer-like 1 (DCL1)酶将pri-miRNA加工成成熟的miRNA [9,10].在拟南芥中,另一种双链RNA结合蛋白HYPONASTIC LEAVES 1与C2H2锌指蛋白,协同协助DCL1从折叠结构中释放miRNA和miRNA* [11- - - - - -14].释放的21-nt双工通过在双工的3'端加入甲基来稳定,这一步由甲基转移酶HEN1催化。15].甲基化的miRNA-miRNA*双工被草率(出口蛋白5的植物同源物)和其他尚未确定的出口蛋白出口到细胞质[3.,16].随后,该双链被解开,只有称为miRNA的活性链被加载到rna诱导沉默复体(RISC)上。在mirna的引导下,RISC识别目标mrna上的互补位点,从而引起转录产物的切割[1,3.,17或转译停顿[2,4].
在植物中,已知约20个双子叶和单子叶之间保存良好的miRNA家族。其中,miR156/157、miR160、miR159、miR319、miR165/166、miR390和miR408 7个miRNA家族也在原始陆生植物中被发现Physcometrella而且卷柏这表明它们是高度保守的[18- - - - - -21].此外,拟南芥、大米、杨树而且Physcometrella具有许多非保守的系系或植物物种特异性miRNA家族[19,22- - - - - -26].
高通量测序方法的最新进展已经彻底改变了低丰度非保守miRNAs的识别拟南芥[22,24- - - - - -26].应用MPSS和454等测序技术,在拟南芥中发现了13个新的miRNAs [24].同样,随着测序深度的增强,在拟南芥[25].使用类似的方法结合计算方法,在其中鉴定出48个新的miRNAs拟南芥[26].这些新发现的miRNAs大多在水稻或水稻中并不保守杨树说明这些可能是特定的拟南芥.非保守miRNAs被认为是正在进化或最近进化的miRNAs,其中只有一些具有功能意义的miRNAs将被保留,其余的可能在短时间内丢失[26].
在任何生物体中鉴定一组接近完整的小rna对于理解小rna介导的基因调控和小rna的多样性具有重要意义。它为阐明复杂的小rna介导的调控网络奠定了必要的基础,这些网络控制着生物和非生物胁迫的发育、营养反应和耐受性[27- - - - - -29].大米(栽培稻L.是高通量小RNA分析的明显选择,因为它在世界范围内的农业重要性,而且是一种具有测序基因组的模式单子叶植物。已知的基因组加上高通量转录组(编码和非编码RNA)分析,将显著提高我们解开水稻小RNA引导电路的能力。到目前为止,除了一些单核细胞特异性和水稻特异性的miRNAs外,对水稻小RNA文库的计算和低深度常规测序已经成功地识别了保守的miRNAs [30.- - - - - -37].最近的两项研究报道了水稻小rna的大规模测序,其中一些序列可能是新的miRNAs [38,39],但它们作为新的mirna的注释还没有被证实。在这里,我们使用水稻小rna的深度测序来识别新的mirna和可能受胁迫调节的mirna。序列分析表明,我们在水稻中发现了23个新的miRNAs和40个候选miRNAs。其中6个新的miRNAs (osao - mir1436和5个新发现的成员属于osao - mir444家族,即osao - mir444c。1、c.2、d、e、f)在其他单性状中是保守的。Northern分析显示,这些新的mirna和候选mirna中的一些大量表达。我们预测了9个新发现的水稻miRNAs的20个新靶点。因此,本研究发现了许多新的miRNAs及其潜在的靶基因,促进了我们对水稻miRNAs的认识。
结果
水稻幼苗miRNA表达谱分析
在目前的研究中,我们对小RNA文库进行了高通量测序,以识别水稻中低丰度的候选新miRNAs和潜在的胁迫响应miRNAs。4周龄的水稻幼苗分别不处理(作为对照)或用150 mM NaCl处理24 h(盐胁迫)或脱水12 h(干旱胁迫)。从每个样本中生成一个小的RNA库,并对454种生命科学进行测序。对获得的原始序列reads(来自3个独立文库的714,202个序列)进行计算处理,去除5'和3'适配器序列,从3个文库中总共获得331,422个基因组匹配reads(分别来自未处理文库、干旱文库和盐文库的102,876个、54,016个和174,530个reads)。其余不能映射到水稻基因组的序列被丢弃,这些序列可能包含序列错误,受RNA编辑的影响,或来自未测序的基因组区域或污染物。在去除rRNA、tRNA和sn/snoRNA后,我们分别从对照文库、盐文库和干旱文库中获得了58,781、43,003和80,990个与水稻基因组完美匹配的独特小rna。
目前,miRBase列出了243个水稻miRNA (miRBase发布版本10.1),可分为62个miRNA家族。其中9个miRNA家族(miR413、miR414、miR415、miR416、miR417、miR418、miR419、miR420和miR426)仅基于水稻和拟南芥之间的序列保守进行预测[33].到目前为止,这些序列都没有在任何深度测序的拟南芥小RNA文库中找到[24- - - - - -26也不在大米中([32,34- - - - - -37],本研究)因此,截至今天,水稻中表达的miRNA家族的数量为53个。其中一些是非保守的水稻特异性miRNA家族,它们作为miRNA的特性完全基于克隆序列侧翼序列的预测折叠结构[32,34- - - - - -37].尽管如此,本研究获得的测序深度足以在水稻中已知的53个表达miRNA家族中找到46个。此前报道的7个miRNA家族没有出现在我们的序列中,可能是由于它们的丰度极低或组织或细胞特异性表达模式或其他未知原因,如偏克隆。
新microrna
在水稻中定义新的miRNAs尤其具有挑战性,因为水稻dcl1敲除突变体不可用。大米dcl1有报导说,工厂已被拆毁[35,但我们的研究没有材料。因此,本研究中的miRNA分类基于以下标准:1)预测前体的折叠结构,2)在一些情况下,小RNA序列出现在多个独立文库中,3)miR*序列出现在几个新的miRNAs的至少一个文库中,4)它们在相关的单克隆中保存(如果有的话)。大多数保守的miRNAs以基因家族的形式存在,因此在基因中有多个位点拟南芥,米饭和杨树而最近进化的miRNAs被发现在基因组中只有一个位点[24- - - - - -26].与这些最近的发现相吻合拟南芥,我们将在水稻基因组中具有单一位点的小rna命名为miRNAs。然而,基于其他考虑,如家族成员及其在相关植物物种中的保存性,本研究也将一些在水稻基因组中具有多达3个位点的离群值归类为miRNAs。例如,miR444家族的三个成员(miR444c。1, miR444c。2而且miR444e) can be mapped to more than one locus in the rice genome. Another miRNA, Osa-miR1436 with 20 hits in the rice genome has been considered as a miRNA because this appears to be conserved in山羊草属,单卧床。根据上述标准,我们将从3个独立文库中获得的23个小rna指定为水稻中的新型miRNAs(表1)1).对于所有这23个新的miRNAs,利用克隆的小rna的侧面基因组序列可以预测折叠结构1).
新的mirna的起源和频率
在植物中,大多数已知的miRNAs发现在编码基因之间,尽管少数发现在蛋白质编码基因的内含子和外显子的意义或反义方向。为了确定水稻基因组前体的位置,我们使用了TIGR GenBank数据库。在表中列出的23个新的mirna中1虽然5个新的miRNAs (osos - mir444c。1,c.2, d, e, f) belonging to Osa-miR444 family were found in the opposite strand of the protein coding genes. Sixteen of these new miRNAs can be mapped to single locus in rice genome (Table1).4个miRNAs (osos - mir444c。2,Osa-miR444e, Osa-miR1440 and Osa-miR1442) are mapped to 2 loci each, while 2 others (Osa-miR444c.1 and Osa-miR1441) are mapped to 3 loci each in the rice genome. Osa-miR1436 could be mapped to 20 loci (although hairpin structures could be predicted for the precursors from two loci only) and this has been included in the list of new miRNAs, because we found miR1436 homolog in山羊草属(图1)1).
miRNA的频率范围在控制库中的1到4948次读取之间。不出所料,得分最高的序列是保守的mirna。与保守的mirna相比,从它们的频率来看,所有的新mirna的丰度都相对较低1).4个新的miRNAs (Osa-miR1427, Osa-miR1430, Osa-miR1432和Osa-miR444c.2)在所有3个文库中都被发现,9个新的miRNAs (miR444c. 2)被发现。1,Osa-miR444e, Osa-miR444f, Osa-miR1423, Osa-miR1425, Osa-miR1431, Osa-miR1436, Osa-miR1437 and Osa-miR810b.2) appeared at least in 2 of the 3 libraries. Together, 13 of the new miRNAs appeared at least in more than one independent library. Three of the new miRNAs appeared only once (Osa-miR167*, Osa-miR444d and Osa-miR1429) in our sequences whereas the remaining 20 appeared multiple times. These findings suggest that a large number of miRNAs may be unique to the rice genome, and possibly many more low abundance miRNAs remain to be identified.
miRNA的存在*
其他大量的内源性小rna,其中一些可能来源于偶然折叠成miRNA样发夹结构的区域,使得植物中的miRNA鉴定变得复杂,因此建议在注释之前使用突变体确认生物发生的需求[28].在缺乏表达分析的情况下dcl1突变体中,miRNA*序列的检测是一个重要的标准,因为它支持从预测的折叠结构中释放miRNA双工[24- - - - - -26].在我们的文库中,至少有4个新的miRNA序列(osao - mir1435, osao - mir1430, osao - mir1432和osao - mir1431)有它们的miR*序列。
一些新发现的miRNAs是在密切相关的单克隆中保守的
为了确定这些新的候选miRNAs是否在其他植物物种中是保守的,我们搜索了核苷酸数据库中的同源物。该分析表明,2个miRNAs,即Osa-miR1436和5个Osa-miR444家族成员在其他几个单克隆细胞中是保守的(图1).利用从EST数据库中获得的miRNA周围序列,可以预测这些植物中miRNA的发夹结构(图1)2).此前,我们报道了水稻中miR436和miR444两种miRNAs的鉴定[32].在这两种情况下,只有经过处理的前体转录本才能形成发夹结构,但不使用基因组位点[32].在本研究中,我们发现了5个新成员(osos - mir444c。1, c.2, d, e, f)属于miR444家族(图1).在所有这些情况下,折叠结构的预测需要经过处理的转录本,而不需要水稻中miRNA序列周围的基因组序列(图2).Osa-miR444e和之前报道的miR444序列似乎来自于一个前体,如图所示,前体移位了5个核苷酸1 e.有趣的是,这5个新成员(osos - mir444c。1,c.2, d, e, f) are highly conserved in several monocotyledonous plants such as高粱玉米,大麦,小麦,柳枝稷,甘蔗和Brachypodium(图1 b-f).值得一提的是,尽管osos - mir444d序列在与miR444对齐时显得非常不同(图4)1 d),但两个序列均与其中一个MADS box因子(Os02g49840)具有高度的互补性。因此,我们认为osos - mir444d是miR444家族的又一新成员。总的来说,这些miRNA序列的缺失拟南芥它们在密切相关的单克隆中存在表明这6个miRNAs (osao - mir1436和osao - mir444c。1、c.2、d、e、f)是单株特异性的。
预测新的miRNAs的靶点
保守miRNAs的靶点可以以非常高的置信度预测,而在单基因组分析中,只有更广泛的成对相互作用才能以合理的置信度预测[30.].为了降低假阳性预测的比例,我们对目标预测使用了非常严格的标准,即对不超过3个不匹配的反义命中进行简单的爆炸搜索[40,尽管这种方法可能会错过一些真实的目标。此外,由于之前的研究发现植物miRNA靶点主要位于orf中,因此我们专注于在编码区寻找靶点[40].这导致了对9种新的miRNAs的20个靶点的预测2).一些有趣的预测靶标转录本是MADS-box转录因子,锌指(C3HC4)家族蛋白,ef -手家族蛋白和钙调素结合蛋白(钙信号通路中涉及的蛋白)和一些五肽重复蛋白。因此,非保守的miRNA靶基因编码了广泛的蛋白质(表2).值得注意的是,对非保守miRNAs的目标验证拟南芥并不是很成功,不像保守的miRNA靶标[26].未来的实验验证将确定这些预测靶点中有多少是水稻miRNAs真正靶向的。对于其余的新miRNAs,我们无法使用上述标准识别任何靶点。有人假设,“年轻的”miRNAs,即非保守的和最近进化的miRNAs,存在时没有实际的靶标[26].也有可能应用的严格标准可能错过了对潜在靶基因的预测。
假设新的mirna
除了表中列出的新的mirna1,我们已经确定了另外40个小rna作为候选mirna(表3.).到目前为止,已知的保守miRNA miR395已被定位到水稻miRBase的26个位点上。在水稻基因组中,这些假定的miRNAs的每个位点的数量是高度不同的(4到306个位点)。在我们列出的40个候选miRNAs中,其中一个(cpmiR188)可以映射到水稻中多达306个位点上。通过小RNA印迹分析,我们能够检测到12个候选miRNAs中的7个(spmiR63, spmiR37, dpmiR4, cpmiR7, dpmiR26, cpmiR182和cpmiR188)。其中一些对水稻基因组有大量命中的序列可能是来自重复丰富区域的sirna,而这些区域恰好预测了折叠结构。对于每一种假定的miRNAs,至少有三分之一的这些位点可以预测到折叠结构。事实上,许多位点的折叠结构可以被预测,这表明其中一些可能是miRNA的真实位点。不管这些小RNA是否来源于折叠结构或dsrna,其中一些小RNA在我们的文库中多次出现,而其他一些有最多位点的小RNA (cpmiR182有212个位点,cpmiR188有306个位点)可以通过小RNA印迹分析检测到,这表明这些小RNA被处理并积累到可检测的水平。未来使用大米的研究dcl突变体将帮助确定其中有多少是真正的mirna。我们还预测了水稻中10个候选miRNAs的22个靶点2).
水稻幼苗中保守miRNA家族的表达谱
即使在相对较低的采样深度,许多保守的miRNAs在水稻小RNA文库中被测序,表明它们的丰度更高[32].与此一致的是,在我们的序列读取中,保守的mirna构成了最丰富的前10个签名(表4).
获得的每个文库中保守miRNAs的序列计数数据表明,测序的深度足以支持对3个文库所代表的条件之间的保守miRNAs表达水平的估计。定性上,我们没有发现保守miRNAs在应力库中存在或不存在的任何显著差异,尽管在数量上存在一些显著差异。三个文库中最丰富的miRNA家族是miR1694).如果相对于miRNA的总数计算单个miRNA家族的丰度,miR169家族分别占对照、干旱和盐胁迫文库中miRNA总数的43%、37%和38%。同样,miR168和miR156两个miRNA家族也分别占这3个文库中miRNA总数的15%。
miRNA家族中的不同成员彼此之间可能略有差异,甚至可能针对不同的信使rna [41].因此,重要的是确定哪个变体在给定组织中表达更丰富,以确定其靶调控的潜力。在拟南芥中,最近谢等人。42],已经报道了73个MIRNA基因的表达,通过对几个小RNA文库的测序和对几个位点的转录起始位点的定位[42].为了检测哪一种miRNA家族成员在水稻幼苗中表达更丰富,我们根据其序列频率在未处理的水稻幼苗(对照库)中检测了已知miRNA的表达谱。我们的方法是基于序列的丰度测定,这使我们能够验证miRNA家族中的哪个miRNA位点在给定组织中表达。然而,只有当不同的成员至少在一个nt上不同,并且每个成员都由基因组中的一个位点表示时,这才有可能。水稻幼苗中表达最多的miRNA家族是miR169,由8个成员代表,对应于水稻的17个位点(表4).水稻miR169家族中4个成员的表达先前已被报道过[32].在这里,我们发现了所有9个变异在水稻中表达的证据,它们的频率高度变化(2到943)。miR169a、d、e、p和q这5个变异在水稻基因组中只有一个拷贝,而另外4个变异在水稻基因组中有多个拷贝。有趣的是5个成员(miR169a;miR169b c;miR169e;miR169f g;miR169h-m)有相似的频率(916,925,939和943),尽管在水稻基因组中只有miR169a和miR169e来源于一个位点,而其他3个成员在水稻基因组中有多个同源位点(表4).
miR156是水稻幼苗中表达量第二高的miRNA4).miR156有3个成员(miR156a-j;miR156k和miR156l),它们的序列略有不同。miR159的3个成员在水稻基因组中由12个不同的位点表达,3个成员均被发现表达。miR156a-j(同源位点产生相似的成熟序列,这使得很难判断哪个位点被表达)和miR156l位点在我们的序列中几乎相同(约565次),而第三个基因(156 k)也被表达,但与其他两个变体相比,其表达水平低50倍。
水稻miR168有两个成员(miR168a和miR168b),它们的核苷酸序列略有不同,来自两个不同的基因座。由于miRNA序列的变化,每个位点都是可区分的,因此很容易确定是否两个位点都在水稻中表达。序列分析表明,在我们的文库中只有miR168a被表达和表达了1007个reads,而miR168b没有4).这些发现与之前对水稻小RNA文库的小规模测序一致,其中观察到miR168a而不是miR168b的表达[32].这些结果表明miR168a在水稻幼苗中大量表达,miR168b的表达可能局限于本研究未检测的特定细胞或组织或发育阶段。在水稻中,miR396由3个变种和5个位点组成。此前,我们报道了水稻中仅有一个具有2个同源位点(miR396d, e)的变体的表达[32].有趣的是,miR396d, e变体似乎是单核苷酸特异性的版本。在这里,我们提供了其他两个变体表达的证据,尽管与miR396d, e相比,表达水平较低4).OsmiR396d, e在幼苗库中出现了20次,而其他两个成员miR396a, b和miR396c分别只出现了8次和5次(表2)4).同样,miR164有两个变异来自水稻的6个位点,只有miR164c与其余5个位点(miR164a, b, d, e, f)不同。miR164c有12个reads,其他成员有55个reads。miR166共有14个基因座,5个成员,均在水稻中表达,但表达频率不同。miR166i, j只出现了一次,而另一个成员出现了55次,其余成员的频率在1到55次读取之间(表4).
我们还发现了miRNA家族的所有变体以相似的频率表达的情况。例如,miR159由3个家族成员代表,在水稻中可映射到6个位点。在一个核苷酸中不同的所有3个成员都被发现以相似的丰度表达(约90 reads)。类似地,另外两个miRNAs, miR172和miR167,各有两个成员,表达量相似(表2)4).
一些保守的miRNA家族成员在水稻幼苗中低水平表达。miR162由两个同源基因座表达,表达频率极低。同样,miR319和miR397在水稻基因组中各由一个成员表达,但由两个位点表达。这两个miRNA家族被发现在低水平表达。miR394、miR395、399和408的频率最低,在这些文库中只有少量的reads (Table4).迄今为止,miR394的预测仅基于拟南芥的序列保守[30.这种基因在水稻中的表达方式是未知的。序列分析表明miR394在水稻中表达。这些发现表明,大多数保守miRNA家族的成员都得到了表达。此外,miRNA家族中不同成员/变体的表达谱在水稻幼苗中可能相似,也可能非常不同,这取决于具体的家族。在已知的非保守水稻miRNAs中,miR820, miR535, miR818和miR530出现了20次或更多,至少在3个文库中的一个(表54).因此,我们在水稻幼苗中的miRNA分析揭示了属于46个miRNA家族的79个miRNA变体的表达4).
新的和候选mirna的表达分析
新microrna
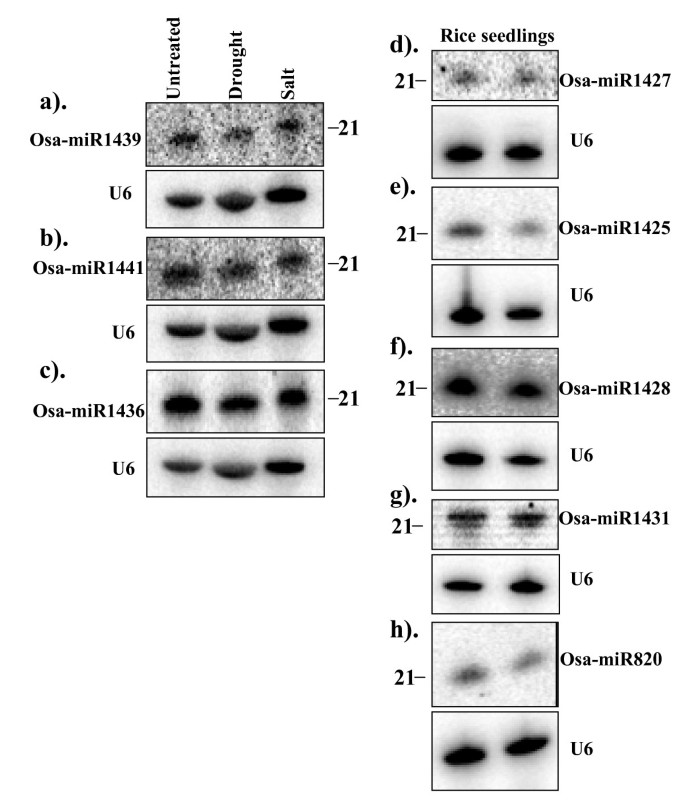
3个文库中新mirna和假定新mirna的频率变化可能表明这些小rna在胁迫下可能受到调控(表2)1和表3.).然而,观察到的这些非保守miRNAs的差异非常小,因此很难得出结论,这些miRNAs是否真的受胁迫调节。为了进一步描述一些新的和假定的新的miRNAs的表达(包括应激调节,如果有的话),我们进行了Northern blot分析。在水稻基因组中检测到的8个新miRNAs中,有5个(Osa-miR1425、Osa-miR1427、Osa-miR1428、Osa-miR1431和Osa-miR1439)是单位点的新miRNAs3.).我们还检测到另外两个具有多个位点的miRNAs,即在水稻基因组中有3个位点的Osa-miR1441和有20个位点的Osa-miR14363.).表达分析还包括水稻中已知的miRNA osos - mir820。总的来说,我们能够检测到10个新miRNAs中的7个的表达(图3.).将文库中的序列读取频率与Northern blot数据进行比较,揭示了一幅相当复杂的图景。例如,osos - mir1436在对照、干旱和盐胁迫库中分别有77、22和0个reads1),表明它可能被下调,特别是在干旱胁迫下。然而,Northern分析结果与这一评价并不一致(图3.).尽管我们的干旱文库是由最少数量的水稻基因组匹配小rna所代表的,但在干旱胁迫文库中,osos - mir1441有24个reads,在对照文库中有0个reads1)表明该miRNA可能在干旱胁迫下被轻微诱导。然而,Northern分析显示,其表达水平在对照和胁迫条件下没有差异(图3.).
候选人microrna
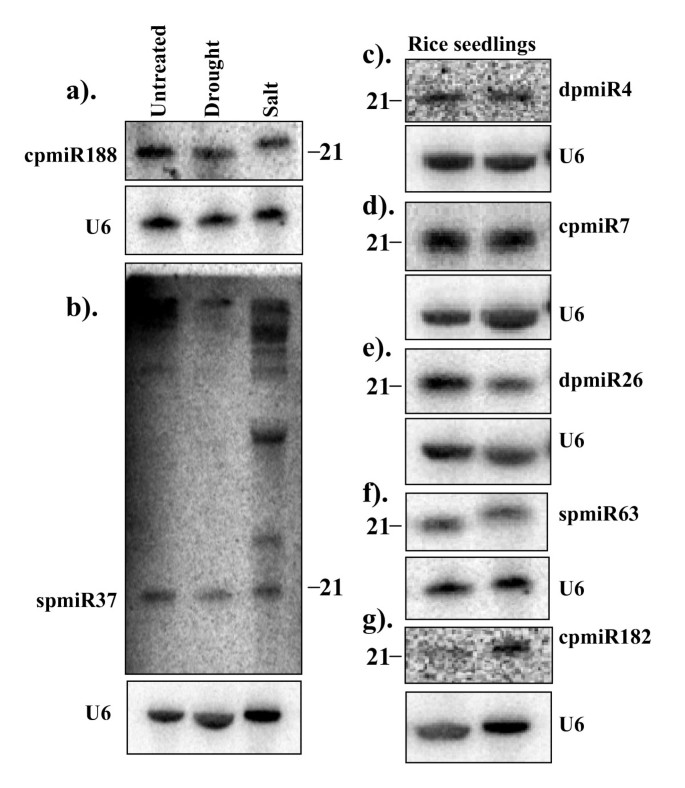
同样,我们也使用小RNA印迹分析对候选的新miRNAs进行表达分析4).在测试的13个假设的新miRNAs中,我们能够检测到其中7个的信号(spmiR63, dpmiR4, dpmiR26, cpmiR7, cpmiR182, cpmiR188,和spmiR37)(图3.).作为候选miRNA, cpmiR7在对照文库中出现了87次,在盐胁迫文库中出现了0次,这表明它可能在胁迫条件下被抑制,但在凝胶印迹分析中没有发现可检测到的差异(数据未显示)。另外三个候选miRNAs, dpmiR4, dpmiR26和spmiR63,在胁迫文库中分别有10,11和7个reads,而在对照文库中没有。北方分析表明,对照和胁迫处理的幼苗之间没有差异(数据未显示)。总的来说,序列读取数据和Northern blot结果之间似乎没有很好的相关性。这种明显的差异可能仅仅是由于序列读取数据的不可靠性造成的,因为读取数量不足。还有一种可能是,北方的分析不够敏感,无法揭示微小的差异。有人认为克隆在确定miRNA丰度方面具有更大的敏感性[26].尽管在确定mirna的相对丰度方面存在困难,但我们对一些假设的新mirna的特异性~ 21nt信号的检测表明,它们至少是真正的小rna,而不是非特异性降解产物(图4).
对于Northern分析中使用的大多数探针,我们注意到,除了预期尺寸的信号外,我们还在盐应力样本中检测到更大尺寸的信号。图中显示了一个具有显著的大信号的代表性斑点4 b.较大的信号可能对应于miRNA加工中间体,提示盐胁迫可能影响小rna的加工。
讨论
随着深度测序的应用,我们已经识别了大部分低丰度的新miRNAs [23和假定的新miRNAs [40在大米中。在水稻中发现了大量以前没有报道过的miRNAs,其中至少有6个是在单子稻中保存的,这表明可能有更多的单子稻或水稻特异性miRNAs有待鉴定。很明显,水稻完整miRNA库的鉴定需要更广泛的组织采样和更深入的测序。众所周知,在拟南芥中,花序表达更多样化的miRNAs [24- - - - - -26].当然,为了在水稻中进一步发现miRNAs,本研究中使用的幼苗必须补充额外的组织,如花序。再加上对相关单子叶植物的类似研究,将有助于确定在其他单子叶植物中有多少这些目前水稻特异性的miRNAs是被保存的。
水稻中高度保守的miRNA家族的数量基本与拟南芥相同。然而,由于单子稻和双子稻之间的功能多样化,水稻似乎进化出了谱系特异性(单子稻)miRNAs。通过对小RNA文库的深度测序,我们为来自46个miRNA家族的79个miRNA变体以及23个新miRNA和40个假定的新miRNA的表达提供了证据。非保守的植物mirna可能在较短的进化时间范围内出现和消散[25,26].建议于拟南芥[25],从与水稻密切相关的物种中提取小rna的高通量测序将有助于确定非保守miRNA基因的寿命。
每个miRNA在一个小RNA文库中被表示的次数可以作为估计其相对丰度的一个指标。在我们的对照文库中,水稻中miRNA家族的频率从1 (miR394、miR399和miR408)到4948次(miR169)不等,表明水稻幼苗中不同miRNA家族的表达差异很大。水稻幼苗中miR169的丰度非常高,这也与用小麦幼苗生成的一个小型RNA文库的数据一致[43].
40个假定的新miRNAs每个都可以映射到1到306个位点(表3.).这些序列中至少有一部分可能是潜在的miRNAs,来源于采用发夹结构的新型重复序列。在动物中,大量新出现的证据支持这一观点,即一些miRNAs来源于富含重复的区域。例如,10种哺乳动物miRNAs,包括6种人类miRNAs已被证明源自转座元件[44].在另一份报告中,确认有50种人类miRNAs位于重复(Alu)元素中[45].然而,最近的另一份报告指出,人类和老鼠体内的75个miRNAs都可能来自重复[46].有趣的是,小鼠重复聚集的miRNAs有超过2000个基因组位点分布在小鼠基因组中。此外,据估计,虽然克隆频率仅占总miRNA序列的2.6%,但重复序列衍生的miRNA占miRNA基因的23% [46].这些mirna的克隆频率很低,说明它们是低丰度的mirna。同样,一个人类miRNA家族(hsa-mir-548)被发现源自Made 1元素(螨虫)[47].有趣的是,源于hsa-mir-548的Made 1 miRNAs是由转座元件的两条链产生的,因为两条链的转录本都能够采用发夹状结构[47].Hsa-mir-548家族成员可以定位到人类基因组中的20到145个位点。我们观察到,一些假定的mirna可以映射到许多具有预测折叠结构的位点,这提高了其中一些可能是水稻中mirna的真实位点的可能性。
许多保守的miRNA家族靶向转录因子,但大多数非保守的miRNA可能靶向在广泛的生物过程中起作用的不同基因。在早期的一项研究中,我们报道了miR444及其靶基因(2个MADS box因子)在小麦、大麦、玉米、高粱和甘蔗等单株作物中是保守的,而在小麦、大麦、玉米、高粱和甘蔗中则不是拟南芥[32].5个新成员的鉴定(osos - mir444c。1,c.2, d, e, f) that are highly homologous with miR444 indicated that miR444 and their conserved MADS box factor genes as targets of this miRNA family is interesting. These observations suggest that the 2 MADS box factors may have been regulated by different members of miR444 family in monocots. Furture studies focusing on functional analysis of these regulations by disrupting the miRNA-targeted site in these MADS box factors would reveal the role of these interactions in monocots. Interestingly, miR444 target sequences were found in MADS-box transcription factor genes in grape and soybean. miR444 does not seem to be present in soybean (Subramanian et al., unpublished data), it is not known whether or not miR444 homolog is present in grapes. These observations raises two possibilities; (1) monocots may have retained miR444 which regulates MADS box factors while dicots have lost after the divergence, (2) miR444 may have evolved in monocots after the divergence. Besides the MADS box factor, we predicted C3HC4 family protein as a target for ceratin members of this miRNA family.
五肽重复基因是一个大家族,与剪接、编辑、加工和翻译等转录后过程有关,特别是在线粒体和叶绿体等细胞器中[48].PPR基因突变导致多种表型,包括细胞质雄性不育[49,50].在拟南芥中,PPR转录本被miR400、miR161.1和miR161.2靶向[51,52].最近,研究表明miRNA靶向PPRs拟南芥会产生阶段性的sirna [52].miR475和miR476杨树也针对PPR转录本[23].拟南芥和白杨中的PPR靶向miRNAs是不相关的,这导致了一个假设,即靶向PPR基因的miRNAs似乎是在拟南芥和水稻中独立进化的。类似地,我们预测osos - mir1425针对水稻中的PPR转录本(见表2)和osos - mir1425与任何靶向PPR转录本的miRNAs都不相关拟南芥而且杨树并提出了一种可能性,即Osa-miR1425是一种水稻特异性miRNA,它已经进化到抑制PPR转录本。
参与钙信号传导的4个基因(其中3个编码EF-hand蛋白,一个编码钙调素结合蛋白)是2个miRNAs (oosa - mir1432和oosa - mir444d)的潜在靶标。Ca2 +是一种无处不在的第二信使,会引发对环境刺激的生理变化[53].预测了Ca的4个组分2 +钙信号转导是2个新的miRNAs的潜在靶点,这表明miRNAs在植物的多种生理过程中涉及的主要信号机制之一——钙信号转导中的作用[53].识别这些与各种生物过程相关的预测靶点,扩大了miRNA调控下的过程和途径的广度。
方法
水稻小rna的克隆
根据制造商的说明,使用Trizol (Invitrogen, USA)从冷冻幼苗中分离总RNA。按照所述进行miRNAs的克隆[51].简单地说,NaCl和PEG沉淀富集了低分子量RNA。在变性的15%聚丙烯酰胺凝胶上分离出约100 μg的低分子量RNA。标记的RNA寡核苷酸为18和26 nt作为大小标准。取18 ~ 26 nt核苷酸,用0.4M NaCl在4℃下洗涤一夜。RNA用碱性磷酸酶(Biolabs, New England)去磷酸化,然后用乙醇沉淀回收。然后将小rna顺序连接到5' (5'-tactaatacgactcactAAA-3';大写字母,RNA;小写,DNA)和3' (5'-pUUUaaccgcatccttctcx-3';大写字母,RNA; lowercase, DNA; p, phosphate; x, inverted deoxythymidine) RNA/DNA chimeric oligonucleotide adapters. Reverse transcription was preformed after ligation with adapters, followed by PCR amplification. The resulting PCR products were sequenced by pyrosequencing method [54].
数据分析
我们分析454个小RNA库的计算方法的总体过程显示在附加文件中3..在预处理中(附加文件3.),所有的小RNA reads(以后称为reads),如果没有与两个适配器序列最近的11nt完全匹配(可能是由于测序错误),首先被删除。小于17 nt的读数随后被排除在进一步分析之外。接下来,使用EMBOSS包中的e倒置和Etandem程序去除串联重复和倒置重复[55),分别。重复元素被RepeatMasker过滤[56基于大米的Repbase [57].水稻tRNA, rRNA, snRNA, snoRNAs [58],已知的miRNAs [59和TIGR Oryza重复数据库[60]被删除。这些预处理步骤分别从对照、干旱和盐胁迫文库中获得了58,781、43,003和80,990个独特基因组匹配的小rna。
识别新mirna的方法
为了处理水稻小RNA序列的独特特征,我们开发了一种从454个序列库中识别miRNAs的新方法。我们的方法包括几个步骤,如附加文件所示3..首先,对长度小于24nt、在read library中有多个拷贝、对基因组的命中数小于24、与已知的非编码rna不匹配的reads是否为mirna进行测试,如下所示。242个已知的水稻miRNA前体(pre-miRNAs)长度从60到312 nt不等,平均值为145 nt。242个(89.7%)已知的pre-miRNAs中有217个不超过200个核苷酸。在这项工作中,我们将200 nt作为我们分析中假定的pre- mirna的长度。在待测短序列的每个基因组位点上提取两条覆盖reads的序列进行二级结构分析,其中一条覆盖reads上游160 nt和下游20 nt,另一条覆盖reads上游20 nt和下游160 nt。这两个序列的二级结构是通过rna折叠程序预测的[61].那些符合以下两个标准的序列被认为是候选的miRNA前体。首先,二级结构必须有一个发夹,在其茎区至少有18对核苷酸。第二,发夹的自由能必须小于或等于-18千卡/摩尔。还有一个中心环。在miRNA的生物生成过程中,当DCL1将pre-miRNA:miRNA*双工从pre-miRNAs中分离出来后,pre-miRNAs的其他部分会很快降解。因此,在454个库中检测到的读被认为是成熟的miRNA或miRNA*。因此,我们检查了被认为是成熟miRNA或miRNA*的reads,以满足MIRCHECK程序的要求[31].
然后,利用两类分类模型对候选前体序列进行进一步的检验。在这项工作中,我们采用了具有线性核的支持向量机(SVM) [62]作为判别模型。我们使用的SVM实现来自WEKA软件包[62使用默认参数。为了建立分类模型,miRBase version 9中242个已知的水稻pre-miRNAs [63]作为阳性样本。这些已知的pre-miRNAs的平均长度为145 nt,标准偏差为55。对于阴性样本,我们使用了水稻编码序列,该序列从TIGR水稻基因组注释数据库[64].对所选编码序列进行分段,片段长度服从正态分布,均值为145,标准差为55,与已知pre-miRNAs相同。这些片段随后用RNAfold进行折叠,并按照相同的标准进行过滤,即大于或等于18个成对核苷酸,折叠能量不大于-18 kCal/mol。还有一个中心环。随机选取242个满足上述条件的分段序列作为负样本。然后,将这484个阳性和阴性样本用于训练支持向量机模型。该模型使用前驱体内的4- 9个短序列基元作为特征。这些序列图案由WordSpy算法提取[65].为提高分类精度,一种基于包装器的特征选择方法[66进一步应用于信息性母题特征的选择。我们采用了10倍交叉验证策略。阳性样本和阴性样本均随机分为10组。将一组阳性样本和一组阴性样本组合成一个测试集。从其余9组阳性样本中提取序列基序。我们首先使用剩下的9组正样本和9组负样本训练支持向量机模型。将该模型应用于测试集,获得了得到的支持向量机模型的精度。为了寻找一组好的主题(特征),我们去掉了支持向量机模型中权重最小的5%的主题,因为它们提供的识别力最小。然后,我们利用剩余的基元重建一个新的支持向量机模型,并在测试集上测试其准确性。我们重复这个过程,建立了一系列支持向量机模型,直到我们有不到20个主题。 Finally we took as the final set of features the motifs used by the SVM model that has the highest accuracy. We iterated the whole process 10 times. At each time, one group of positive samples and one group of negative samples were left as test samples. From each of these iterations, a set of motifs were obtained for the best SVM model in that iteration. Finally we combined these sets of best motifs and used them to build a discriminative model. The final model was applied to the candidate precursor sequences to predict novel pre-miRNAs.
目标预测
已知的水稻orf从TIGR水稻基因组注释数据库下载,并用于推测miRNAs的靶预测。在我们的预测中,我们只允许mRNA靶点和假定的新miRNA之间存在3个不匹配[40].
RNA凝胶印迹分析
用Trizol试剂从未处理(对照)或暴露于盐胁迫或干旱胁迫的4周龄水稻幼苗中分离总RNA。采用PEG沉淀法从总RNA中分离出低分子量RNA。每巷装载20微克低分子量RNA,在变性的15%聚丙烯酰胺凝胶上分解,电泳转移到Hybond-N+膜上。将膜进行UV交联,在80°C下烘烤2小时。与miRNA序列互补的DNA寡核苷酸用γ-标记32P-ATP使用T4多核苷酸激酶(New England Biolabs)。膜预杂交至少1小时,并在38°C下使用完美杂交缓冲液(Sigma)杂交过夜。印迹在50℃下洗涤3次(2次用2 × SSC + 1% SDS洗涤,1次用1 × SSC + 0.5% SDS洗涤)。薄膜被短暂风干,然后暴露在磷屏上,用台风扫描薄膜获得图像。
参考文献
- 1.
唐国强,李晓燕,李晓燕。植物RNA沉默的生物化学框架。基因学报,2003,17:49-63。10.1101 / gad.1048103。
- 2.
Aukerman MJ, Sakai H: MicroRNA及其apetala2样靶基因对开花时间和花器官特性的调控。植物学报,2003,26(3):359 - 359。10.1105 / tpc.016238。
- 3.
Bartel DP: MicroRNAs:基因组学,生物发生,机制和功能。细胞科学,2004,116:281-297。10.1016 / s0092 - 8674(04) 00045 - 5。
- 4.
陈雪:拟南芥花发育过程中作为APETALA2翻译抑制因子的microRNA。科学,2004,303:2022-2025。10.1126 / science.1088060。
- 5.
Zilberman D, Cao X, Jacobsen SE: ARGONAUTE4对位点特异性siRNA积累和DNA和组蛋白甲基化的控制。科学通报,2003,29(6):716-719。10.1126 / science.1079695。
- 6.
陈士伟,齐伯曼D,谢卓,Johansen LK, Carrington JC, Jacobsen SE: RNA沉默基因控制DNA甲基化。科学通报,2004,36(3):334 - 334 / 334。
- 7.
Lee RC, Feinbaum RL, Ambros V: The秀丽隐杆线虫异质慢性基因lin-4编码与lin-14具有反义互补的小rna。Cell. 1993,75: 843-854。10.1016 / 0092 - 8674 (93) 90529 - y。
- 8.
王晓燕,王晓燕,王晓燕。异食性基因的转录后调控lin-14通过lin-4调节时间模式的形成秀丽隐杆线虫.细胞学报,1993,75:855-862。10.1016 / 0092 - 8674(93) 90530 - 4。
- 9.
Papp I, Mette MF, Aufsatz W, Daxinger L, Schauer SE, Ray A, van der Winden J, Matzke M, Matzke AJM:植物微RNA和短干扰RNA前体核加工的证据。植物生理学报,2004,24(3):366 - 366。10.1104 / pp.103.021980。
- 10.
Kurihara Y, Watanabe Y:拟南芥微rna通过Dicer-like 1蛋白功能的生物生成。美国国家科学研究院。2004, 31(4): 557 - 557。10.1073 / pnas.0403115101。
- 11.
韩mh, Goud S, Song L, Fedoroff N:拟南芥双链rna结合蛋白HYL1在microrna介导的基因调控中发挥作用。美国国家科学研究院。2004, 32(1): 1 - 4。10.1073 / pnas.0307969100。
- 12.
Vazquez F, Vaucheret H, Rajagopalan R, Lepers C, Gasciolli V, Mallory AC, Hilbert JL, Bartel DP, Crete P:内生反式-作用的sirna调节拟南芥信使rna。分子生物学杂志,2004,16:69-79。10.1016 / j.molcel.2004.09.028。
- 13.
大堂D, Rallapalli G, Schmidt DD, Martin C, Clarke J: SERRATE:植物microRNA领域的新玩家。2006, 7: 1052-1058。10.1038 / sj.embor.7400806。
- 14.
杨亮,刘铮,陆芳,董安,黄宏:SERRATE是拟南芥microRNA处理过程中一种新型核调控因子。植物学报,2006,29(4):441 - 441。10.1111 / j.1365 - 313 x.2006.02835.x。
- 15.
于斌,杨铮,李娟,Minakhina S,杨敏,Padgett RW, Steward R,陈欣:甲基化在植物microRNA生物发生中的作用。科学通报,2005,29(4):447 - 447。10.1126 / science.1107130。
- 16.
吴刚,张丽娟,张丽娟,张丽娟。拟南芥中microrna的核加工与外源分析。美国国家科学研究院。中国科学(d辑),2011,38(4):369 -369。10.1073 / pnas.0405570102。
- 17.
植物中的RNA沉默。自然学报,2004,431:356-363。10.1038 / nature02874。
- 18.
基因调控:植物中古老的microRNA靶序列。自然学报,2004,428:485-486。10.1038 / 428485。
- 19.
王晓燕,陈晓燕,陈晓燕,陈晓燕。苔藓微rna的克隆与鉴定。植物学报,2005,43:837-848。10.1111 / j.1365 - 313 x.2005.02499.x。
- 20.
Axtell MJ, Bartel DP:陆地植物中microRNAs的古老和它们的靶标。植物学报,2005,17:1658-1673。10.1105 / tpc.105.032185。
- 21.
Axtell MJ, Snyder JA, Bartel DP:陆地植物不同小rna的共同功能。植物学报,2007,19:1750-1769。10.1105 / tpc.107.051706。
- 22.
Lu C, Tej S, Luo S, Haudenschild CD, Meyers BC, Green PJ:转录组小RNA组分的鉴定。科学通报,2005,29(3):359 - 359。10.1126 / science.1114112。
- 23.
吕松,孙永华,史锐,李玲,蒋维林:拟南芥中缺失的毛杨机械应力响应小分子rna。植物科学学报,2004,25(3):366 - 366。10.1105 / tpc.105.033456。
- 24.
Lu C, Kulkarni K, Souret FF, Muthuvalliappan R, Tej SS等:拟南芥RNA依赖性RNA聚合酶-2突变体中富集的MicroRNAs和其他小RNA。基因组学报,2006,16:1276-1288。10.1101 / gr.5530106。
- 25.
Rajagopalan R, Vaucheret H, Trejo J, Bartel DP:拟南芥中多样性和进化流动的一组microRNAs。基因学报,2006,20:3407-3425。10.1101 / gad.1476406。
- 26.
Fahlgren N, Howell MD, Kasschau KD, Chapman EJ, Sullivan CM, Cumbie JS, Givan SA, Law TF, Grant SR, Dangl JL, Carrington JC:拟南芥microRNAs高通量测序:MIRNA基因频繁出生和死亡的证据。PLoS ONE。2007, 2: e219-10.1371/journal.pone.0000219。
- 27.
Mallory A, Vaucheret H:植物中microrna和相关小rna的功能。Nat Genet。2006,S31-36。10.1038 / ng1791。5
- 28.
Jones-Rhoades MJ, Bartel B, Bartel DP:植物中MicroRNAs及其调控靶点。植物学报,2006,29(3):369 - 371。10.1146 / annurev.arplant.57.032905.105218。
- 29.
孙卡,朱军,朱建军:小rna在植物非生物胁迫和营养剥夺中的作用。植物科学进展,2007,12:301-309。10.1016 / j.tplants.2007.05.001。
- 30.
Jones-Rhoades MJ, Bartel DP:植物microrna及其靶标的计算鉴定,包括胁迫诱导的miRNA。分子生物学杂志,2004,14:789 -799。10.1016 / j.molcel.2004.05.027。
- 31.
Bonnet E, Wuyts J, Rouze P, de Peer YV:在拟南芥和水稻中检测91个潜在的保守植物microRNAs,确定重要的靶基因。美国国家科学研究院。中国科学(d辑:自然科学版)10.1073 / pnas.0404025101。
- 32.
孙卡尔,贾恩PK,朱建军:水稻微rna的克隆与鉴定。植物学报,2005,17:1397-1411。10.1105 / tpc.105.031682。
- 33.
王新军,Reyes JL, Chua NH, Gaasterland T:拟南芥microrna的预测与鉴定及其mRNA靶点。中国生物学报,2004,5:R65-10.1186/gb-2004-5-9-r65。
- 34.
王建峰,周浩,陈永强,罗庆杰,曲丽华:20个来自栽培稻.核酸学报,2004,32:1688-1695。10.1093 / nar / gkh332。
- 35.
刘斌,李鹏,李旭,刘晨,曹松,褚晨,曹鑫:水稻OsDCL1功能缺失影响microRNA积累,导致发育缺陷。植物生理学报,2005,39:296-305。10.1104 / pp.105.063420。
- 36.
陈铮,张俊,孔俊,李松,付勇,李松,张宏,李勇,朱勇:水稻内源小非编码rna的多样性。遗传学报,2006,29(4):381 - 381。10.1007 / s10709 - 005 - 2486 - 0。
- 37.
罗永春,周辉,李颖,陈建勇,杨建红,陈永强,曲丽华:水稻胚性愈伤组织表达一组独特的microrna,提示microrna在植物胚后发育中的调控作用。中国科学(d辑:自然科学版)。10.1016 / j.febslet.2006.08.046。
- 38.
Johnson C, Bowman L, Adai AT, Vance V, Sundaresan V: CSRDB:谷物小RNA集成数据库和浏览器资源。核酸学报,2007,35:D829-33。10.1093 / nar / gkl991。
- 39.
Nobuta K, Venu RC, Lu C, Belo A, Vemaraju K, Kulkarni K, Wang W, Pillay M, Green PJ, Wang GL, Meyers BC:水稻mrna和小rna的表达图谱。生物技术学报,2007,25:473-477。10.1038 / nbt1291。
- 40.
Rhoades MW, Reinhart BJ, Lim LP, Burge CB, Bartel B, Bartel DP:植物microRNA靶标的预测。细胞学报,2002,110:513-520。10.1016 / s0092 - 8674(02) 00863 - 2。
- 41.
Schwab R, Palatnik JF, Riester M, Schommer C, Schmid M, Weigel D: microRNAs对植物转录组的特异性影响。开发细胞,2005,8:517-527。10.1016 / j.devcel.2005.01.018。
- 42.
Xie Z, Allen E, Fahlgren N, Calamar A, Givan SA, Carrington JC:拟南芥MIRNA基因的表达。植物生理学报,2004,24(3):366 - 366。10.1104 / pp.105.062943。
- 43.
姚勇,郭刚,倪铮,孙卡尔,杜军,朱建军,孙琼:小麦(Triticum aestivum L.) microrna的克隆与鉴定。中国生物技术学报,2007,8:R96-10.1186/gb-2007-8-6-r96。
- 44.
Smallheiser NR, Trovik VI:来自基因组重复序列的哺乳动物microRNAs。科学通报,2005,21:322-326。10.1016 / j.tig.2005.04.008。
- 45.
Borchert GM, Lanier W, Davidson BL: RNA聚合酶III转录人microRNAs。中华分子生物学杂志,2006,13:1097-1101。10.1038 / nsmb1167。
- 46.
Landgraf P, Rusu M, Sheridan R,等:基于小RNA文库测序的哺乳动物microRNA表达图谱。细胞学报,2007,29(4):347 - 347。10.1016 / j.cell.2007.04.040。
- 47.
Piriyapongsa J, Jordan IK:来自微型倒置重复转座元件的人类microRNA基因家族。科学通报,2007,2:e203-10.1371/journal.pone.0000203。
- 48.
del烦扰E, Stanley WA, Bond CS, Small ID:在细胞器转录后过程中作为序列特异性因子的五肽重复(PPR)蛋白。生物化学学报,2007,35:1643-1647。10.1042 / BST0351643。
- 49.
Bentolila S, Alfonso AA, Hanson MR:含有五肽重复的基因恢复细胞质雄性不育植物的育性。美国国家科学研究院。中国科学(d辑:自然科学版),2002,38(4):557 - 557。10.1073 / pnas.102301599。
- 50.
Akagi H, Nakamura A, Yokozeki-Misono Y, Inagaki A, Takahashi H, Mori K, Fujimura T:编码线粒体靶向PPR蛋白的bt型细胞质雄性不育修复基因Rf-1的定位克隆。中国科学(d辑),2004,38(4):449- 457。10.1007 / s00122 - 004 - 1591 - 2。
- 51.
Sunkar R,朱建科:新的和压力调节的microRNAs和其他小rna拟南芥.植物生物学杂志,2004,16:2001-2019。10.1105 / tpc.104.022830。
- 52.
Howell MD, Fahlgren N, Chapman EJ, Cumbie JS, Sullivan CM, Givan SA, Kasschau KD, Carrington JC:拟南芥中RNA依赖的RNA聚合ase6 /DICER-LIKE4通路的全基因组分析揭示了miRNA和tasirna定向靶向的依赖性。植物学报,2007,19:926-942。10.1105 / tpc.107.050062。
- 53.
Bouche N, Yellin A, sneden WA, Fromm H:植物特异性钙调素结合蛋白。植物学报,2005,36(4):356 - 366。10.1146 / annurev.arplant.56.032604.144224。
- 54.
Margulies M, Egholm M, Altman WE, Attiya S, Bader JS, Bemben LA, Berka J, Braverman MS,陈勇军,陈智,等:微组装高密度pic升反应器中的基因组测序。自然学报,2005,437:376-380。
- 55.
Rice P, Longden I, Bleasby A: EMBOSS:欧洲分子生物学开放软件套件。科学通报,2000,16:276-277。10.1016 / s0168 - 9525(00) 02024 - 2。
- 56.
- 57.
- 58.
- 59.
- 60.
- 61.
Hofacker IL:维也纳RNA二级结构服务器。核酸学报,2003,31:3429-3431。10.1093 / nar / gkg599。
- 62.
Frank E:《数据挖掘:Java实现的实用机器学习工具和技术》1999,Morgan Kaufmann,旧金山,CA
- 63.
Griffiths-Jones S, Grocock RJ, van Dongen S, Bateman A, Enright AJ: miRBase: microRNA序列、靶标和基因命名法。核酸学报,2006,34(增刊1):D140-144。10.1093 / nar / gkj112。
- 64.
- 65.
王刚,张伟:一种基于隐写分析的功能调节元件的综合识别和表征方法。中国生物医学工程学报,2006,7 (6):R49-10.1186/gb-2006-7-6-r49。
- 66.
Kohavi R, John GH:特征子集选择的包装器。人工智能。1997,97:273-324。10.1016 / s0004 - 3702 (97) 00043 - x。
确认
感谢俄克拉荷马州立大学静水分校生物化学与分子生物学系翁华女士在目标预测方面的帮助。这项研究得到了俄克拉荷马州农业实验站、OCAST植物科学研究项目和美国农业部(NRI#2007-02019)对R. Sunkar的支持,以及美国国立卫生研究院对j . k .的R01GM059138和R01GM070795的资助。朱。该研究得到了美国国家科学基金会(NSF) iss -0535257和孟山都公司(Monsanto Corporation)对w.w.z的部分资助
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
R.S.和j.k.z设计了这项研究。R.S.构建小RNA文库并进行表达分析。XZ、YZ、WZ设计了计算方法,分析了数据和结果,并撰写了计算方法和结果的章节和部分。XZ和YZ开发了软件并进行了实验。R.S.撰写了论文;j - k.z编辑了这份报纸。所有作者阅读并批准了最终稿件。
Ramanjulu Sunkar、周雪峰对这项工作也有同样的贡献。
电子补充材料
12870 _2008_233_moesm1_esm.doc
附加文件1:利用水稻中新发现的mirna前体序列预测折叠结构。利用水稻中新鉴定的mirna前体序列预测折叠结构。(doc 44kb)
12870 _2008_233_moesm2_esm.xls
附加文件2:水稻中新鉴定的miRNAs的预测靶点。水稻中新鉴定的mirna的预测靶点(XLS 16 KB)
12870 _2008_233_moesm3_esm.jpeg
附加文件3:用于鉴定水稻中新miRNAs的程序示意图。鉴定水稻中新miRNAs的过程示意图(JPEG 282 KB)
权利与权限
本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(http://creativecommons.org/licenses/by/2.0),它允许在任何媒体上不受限制地使用、分发和复制,只要原著被恰当地引用。
关于本文
引用本文
孙卡,周,X,郑,Y。et al。利用高通量测序鉴定水稻中新型和候选miRNAs。植物生物学8,25(2008)。https://doi.org/10.1186/1471-2229-8-25
收到了:
接受:
发表:
关键字
- 水稻基因组
- 水稻幼苗
- 支持向量机模型
- microrna的家庭
- 候选人microrna