摘要
背景
病毒感染的影响包括伴随的植物基因变异和细胞变化。需要一个简单的系统来评估宿主对病毒感染反应的复杂性。水稻黄斑病毒(RYMV)的基因组是一种单链RNA,结构简单。它是最著名的单子叶病毒模型。对其生物学、结构和系统地理学的研究为进一步的遗传学研究提供了适宜的背景。目前已有12条水稻染色体序列,为基因组研究,特别是物理作图和基因鉴定提供了强有力的支持。
结果
目前通过cDNA-AFLP技术获得的数据显示,两个不同水稻品种对RYMV的不同反应,即易感IR64 (水稻)和部分耐药的Azucena (金银花).这种RNA分析提供了一个新的原始数据集,使我们能够更深入地了解RYMV/水稻相互作用和宿主反应的特异性。利用SIM4子程序,我们考虑了基因的内含子/外显子结构,在12条水稻染色体上定位了281个RYMV胁迫响应(RSR)转录本,对应234个RSR基因。我们还绘制了先前发现的与部分抗性有关的失调控蛋白和基因,从而构建了第一个RYMV/水稻相互作用的全球物理图谱。水稻第4和第10染色体上的RSR转录本并非随机分布。在易感和部分抗性品种中鉴定出7个基因,这7个基因的转录本在两个品种中均有共定位。在病毒感染过程中,许多伴随的植物基因表达变化可能与感染过程、一般胁迫或防御反应引起的寄主变化有关。我们注意到一些基因(如ABC转运蛋白)在感染和区分易感和部分耐药宿主的动力学过程中受到调控。
结论
我们结合本研究的信息和之前关于RYMV感染过程中调控的蛋白质和ESTs的研究,增强了第一张RYMV/水稻互作图,从而更全面地了解与植物反应相关的基因。这一组合图谱为探索RYMV/水稻相互作用的分子机制提供了新的工具。
背景
病毒诱导转录组在不同寄主植物中的大量分析已被报道。这些研究通常对一组诱导的病毒感染变化进行了分类(综述于[1])。最一致的观察结果是,在兼容的宿主-病毒相互作用中,病毒会触发一系列应激反应,包括防御反应基因。宿主中不同信号通路的扰动是由参与病毒和宿主蛋白质之间某些特定相互作用的基因表达的变化以及不直接参与这些相互作用的基因表达的变化引起的。
的水稻黄斑驳病毒(RYMV, Sobemovirus)是水稻最具破坏性的病原体,在非洲广泛流行。它有一个简单的基因组组织,一个单链RNA编码四个开放阅读框(ORF)。它的生物学和结构是众所周知的[2,3.].这种病毒的多样化和系统地理学已得到充分记录[4,5].在栽培的亚洲和非洲水稻品种中,已经描述了对RYMV不同程度的抗性,o .漂白亚麻纤维卷而且o . glaberrima[6],在一些高地观察到部分抗性粳稻水稻品种[7].最近发现了一个位于4号染色体上的编码eIF(iso)4G蛋白的主要隐性抗性基因[8].利用双倍单倍体群体(IR64 (o.s. indica,对RYMV高度敏感)× Azucena (金银花显示了7个参与部分抗性的数量性状位点[7,9].因此,这种病毒是一个非常有吸引力的模型,因为对这种病原体的基因组研究提供了一个众所周知的背景,并为其他水稻病毒开辟了前景。
水稻基因组已在基因和物理上得到很好的绘制[10,11].日本秃(Nipponbare)品种的基因组(粳稻)及93-11 (o.s. indica)已进行整体排序[12- - - - - -20.],这12个水稻染色体可在网上购买[21].大量被预测的基因对应于功能未知的假定蛋白质,基因组的一部分对应于重复序列[22,23].然而,基于同源性搜索预测基因的假定功能似乎很有趣,从而能够识别参与特定生化途径的候选基因,如参与植物抵御病毒感染机制的病毒抗性基因。
我们已经发表了水稻细胞悬浮液感染RYMV的全球蛋白质分析,使用二维凝胶电泳。我们还在易感和部分抗性水稻品种中鉴定了应对病毒积累改变的蛋白质(防御和应激相关蛋白、翻译和蛋白质周转以及代谢蛋白)[24].此外,我们对感染RYMV的易感和部分抗性水稻品种的转录组的首次研究确定了防御、代谢和光合作用途径中的基因表达变化[25].与其他研究相比,这些结果表明在蛋白质和mRNA水平上也发生了类似的事件。在这篇文章中,我们对转录组进行了细致的研究,并报告了281个RYMV应激反应序列(通过cDNA-AFLP识别的RSR序列)的位置。这些RSR基因属于不同的功能类,特别是防御、光合作用途径和代谢,并已使用SIM4子程序将cDNA片段与基因组序列进行比对,将其映射到水稻染色体上。我们在这里介绍了第一个编译的病毒/水稻相互作用的物理图与模型RYMV/Rice。
结果
利用cDNA-AFLP发现病毒感染过程中诱导或抑制的基因
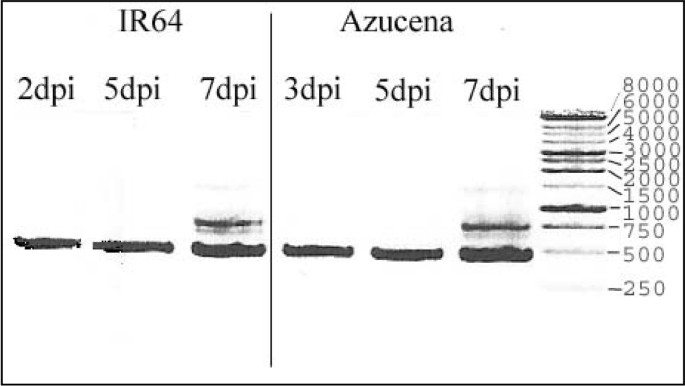
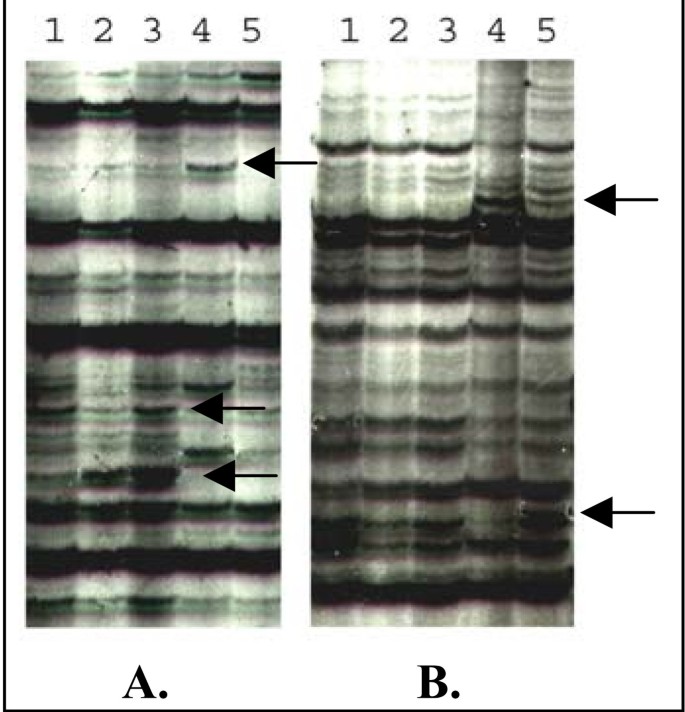
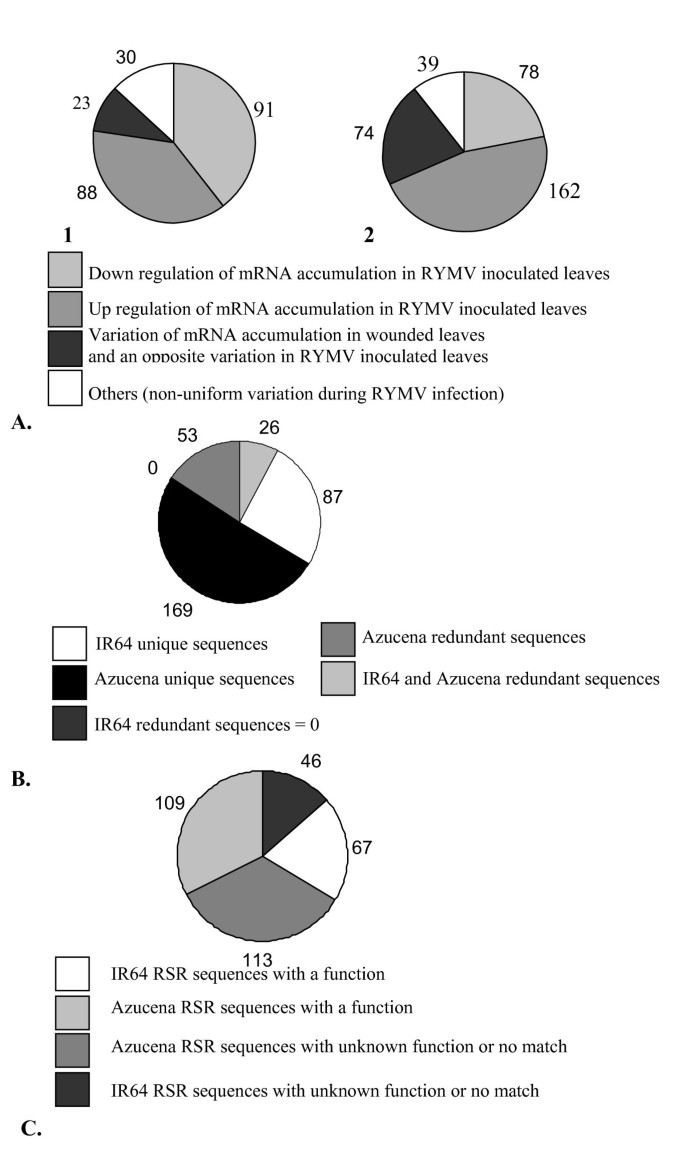
我们利用cDNA-AFLP技术分析了两个不同水稻品种的转录谱分析:IR64 (o.s. indica)对RYMV高度敏感,而Azucena (金银花)耐受RYMV。我们观察了感染后的不同时间点:IR64接种后2、5、7天,Azucena接种后3、5、7天。动力学给出了相似的模式,在感染的两个品种的叶片病毒含量(图1).两个品种在动力学的第一点以未感染的IR64和Azucena叶片作为对照,在动力学的第一点收获没有RYMV的IR64和Azucena损伤叶片作为内控,因为病毒是机械感染的。典型的AFLP凝胶切片如图所示2.利用来自IR64和Azucena品种的2万多个cDNA片段揭示了基因表达的变化模式。所得到的AFLP产物长度从40个碱基对(bp)到500个bp,凝胶上256个引物组合中的每一个都观察到60到100个条带。只考虑存在-不存在模式的带。通过cDNA- aflp筛选,鉴定出Azucena RYMV感染第一周差异积累转录本的353个cDNA片段和IR64的232个cDNA片段。其中162个(46%)AFLP-cDNA片段对应于上调转录本,78个(22%)对应于下调转录本,74个(21%)对应于损伤调控和rymv调控转录本(损伤叶片中观察到的转录本积累变化,感染叶片中观察到的转录本变化相反)。相反,88个(38%)的cDNA片段对应IR64的上调转录本,91个(39%)对应下调转录本,只有23个(10%)对应伤口调控和rymv调控转录本(图)3.).
从诱导和改变的AFLP片段编译序列
从丙烯酰胺凝胶中恢复和再扩增是棘手的步骤,因此我们无法获得所有cDNA片段的干净序列。我们分别获得了IR64和Azucena的113个(占232个片段的49%)和222个(占353个片段的63%)干净序列。这些序列被命名为RYMV应激反应(RSR)转录本。切断适配器和载体序列后,所有序列的长度均在20 ~ 300 bp之间。所有335个RSR序列(IR64为113,Azucena为222)已在GenBank中被引用(GenBank的登录号从DQ883824到DQ884159),并已相互转发。87条IR64和169条Azucena RSR序列在GenBank数据库中仅匹配一次,分别被命名为IR64或Azucena唯一序列。53个Azucena序列匹配超过一次,被命名为Azucena冗余序列。相互匹配的IR64和Azucena序列有26个,被命名为IR64和Azucena冗余序列(在这种情况下,一个IR64序列与一个或多个Azucena序列匹配,反之亦然)(图3 b).这79个RSR冗余序列实际上对应34个独特的基因。这种冗余直接归因于所使用的技术:由于cDNA- aflp是基于cDNA片段的扩增,我们期望识别单个cDNA的多个片段。
所有335条RSR序列均在NR数据库(所有非冗余GenBank CDS翻译+ RefSeq蛋白+ PDB + SwissProt + PIR + PRF)中进行blast。67条(59%)IR64序列和109条(49%)Azucena序列匹配。我们使用低强度,e值< 104,因为序列的长度较小。46条(41%)IR64和113条(51%)Azucena序列与功能未知的序列不匹配或相似1,图3 c).很大一部分AFLP序列没有分配功能,这可能是由于AFLP序列的大小较短和NR数据库的质量。在这些未分类序列中,在12个序列簇中有29个是冗余的。
所有的RSR序列被分类为功能类别(见附加文件)1).我们观察到两个品种在每个功能类的mRNA积累都有一些变化。我们的研究结果揭示了病毒感染在敏感和部分抗性品种中产生大量变化的转录本的重要变化。主要不受调控的功能类别是防御、信号传递、代谢和光合作用。我们还注意到在Azucena感染过程中编码转座子/逆转录因子的5个不同的RSR序列(DQ884156、DQ883989、DQ883835、DQ883911和DQ883916)呈负调控。
RSR序列映射到水稻染色体上
所有的cDNA-AFLP片段都被定位并与12条染色体的释放5进行比对栽培稻[21,26].该地图可在IRD平台网站上获得[见可用性部分]。共绘制了284条RSR序列,其中183条来自Azucena, 101条来自IR64。51个RSR序列还没有被绘制出来,因为分数太低,无法有足够的信心绘制它们。多个RSR序列映射在多个位置,多个RSR序列映射在同一位置,因此对应于同一基因。我们分别将46、40、54、38、25、30、35、26、20、32、26和26个RSR序列映射到12条水稻染色体上。我们从这两个品种中鉴定了20个相同位置的RSR序列,其中27个和22个位置分别具有一个以上的Azucena RSR序列和IR64 RSR序列2).这些冗余序列对应同一基因,未用于统计检验研究RSR基因在染色体上的分布。为了研究每条染色体上的RSR基因分布是否与基因的分布相对应,我们将RSR序列的分布与基因分布对应的ATG密码子的分布进行了比较。然后我们对每条染色体进行偶然性检验,alpha误差为0.5%。RSR序列在4号和10号染色体上的分布与ATG密码子的分布有显著差异。
基因表达变异的定量RT-PCR验证
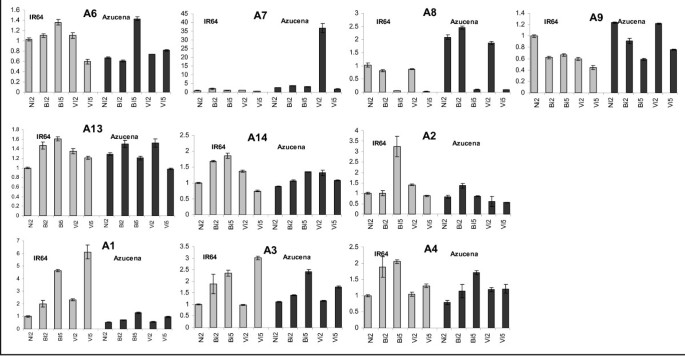
以10个基因为探针,通过定量RT-PCR确认cDNA-AFLP结果(图4).RT-PCR检测结果表明,所有相关基因均有表达变异,并不是假阳性。我们没有观察到完全相同的基因表达变化模式;然而,定量RT-PCR始终证实cDNA-AFLP鉴定的基因在RYMV感染过程中实际上是被解除调控的。定量RT-PCR也更精确地描绘了变化的时间过程。因此,我们鉴定了基因gb|DQ884155在2 dpi处短暂的上调表达,该基因编码一种假定的s受体激酶。此外,定量RT-PCR分析表明,易感品种对损伤的反应程度似乎高于耐受性品种:基因表达的变化(上调或下调)对易感品种的损伤叶片更重要。
定量RT-PCR结果。3次重复的平均值和标准误差。(NI:未接种;BI2:缓冲接种,2dpi收获;BI5:缓冲接种,5dpi收获;VI2:病毒接种,2dpi收获;VI5:病毒接种,5dpi收获-灰色图:IR64;黑色图:Azucena)。A6: IR64-gb | DQ884097 / Azucena-gb | DQ883871。A7: Azucena-gb | DQ884155。A8: IR64-gb | DQ884049。A9: IR64-gb | DQ884066。首次购物: Azucena-gb | DQ883861。阿: IR64-gb | DQ884107 / Azucena-gb | DQ883961。A2: IR64-gb | DQ884118 / Azucena-gb | DQ883962。来自IR64和Azucena共定位的RSR序列:A1: IR64-gb | DQ884103 / Azucena-gb | DQ883886。A3: IR64-gb | DQ884067 / Azucena-gb | DQ883887。A4: IR64-gb | DQ884124 / Azucena-gb | DQ884005。
讨论
病毒利用多种策略促进其在植物中的感染。大量研究已经提到了病毒感染的各种影响[27].已经有大量文献报道了宿主细胞的修饰[[1]供审阅。抑制转录后基因沉默是促进其感染的策略之一[28].在病毒感染下,伴随有许多植物基因变异和细胞变化[29].病毒感染的影响仍然复杂,因果关系仍难以确定。此外,与耐受反应相比,宿主反应的主要特征和易感反应的特异性仍然不清楚。
由于对病毒感染的反应涉及生物数量特征,且具有多因子遗传,因此对RYMV感染过程中受影响的多个基因进行全面分析,似乎是研究病毒与宿主相互作用的直接方法。利用二维电泳和ESTs分析,我们已经建立了蛋白质和基因调控的复杂模式,并确定了涉及宿主反应的细胞平衡的许多变化([25,24];数据未显示)。我们在转录和转录后水平上都发现了光合作用途径和代谢的一些变化,作为敏感反应的潜在线索。在此,我们利用cDNA-AFLP技术分析了两个水稻品种IR64 (o.s. indica)对RYMV高度敏感,而Azucena (金银花)耐受RYMV。该技术已被证明是一种强大的基于凝胶的基因组尺度转录谱分析[30.]并使我们能够在水稻感染的第一周识别出大量参与水稻- rymv相互作用的基因。
我们观察到,与易感品种相比,耐受性品种的宿主基因表达有更多的调控3).在RYMV侵染过程中,IR64的光系统II稳定性组装因子基因(RSR序列DQ884135和DQ884136)表达上调,Azucena的光系统II磷酸化蛋白基因(DQ884058)表达下调。这些结果与我们之前关于est的研究一致,即部分抗性品种光合作用通路下调,易感品种光合作用通路上调[25].然而,这两种方法(ESTs分析和cDNA-AFLP技术)差异太大,结果不可能完全重叠,我们不同的实验也没有强调过程的相同方面。ESTs方法基于大数据集分析,揭示了全局模式,而cDNA-AFLP技术允许对基因进行精细分析,即使是低表达的基因,并且可以通过RT-PCR验证表达的变化。此外,这些实验并不是同时进行的,因此可能会发生实验条件的变化。作为应激反应的一部分,病毒感染的一般特性可能会引起宿主基因产物的增加积累,特别是HSPs基因。[1].在3 dpi采收的伤叶和病毒侵染叶中,HSP70蛋白(gb|DQ883891)表达上调。许多病毒诱导热休克蛋白70基因(和其他热休克基因)的表达,在某些情况下,热休克蛋白已被证明有助于病毒感染[31].似乎大多数病毒在缺乏典型的基因对基因或抗性基因-致病基因相互作用的情况下触发了这些一般的应激反应或防御反应。RYMV感染在IR64叶片7dpi诱导了WRKY DNA结合蛋白(gb|DQ884128)的表达。WRKY6是WRKY家族的一员,Whitham已经确定它是由病毒和其他病原体感染引起的[1].RYMV既能引起宿主基因表达的特异性变化,也能引起宿主基因表达的一般性变化,这些实例表明,该病毒可诱导其他病毒诱导的常见基因表达。
本研究是首次编译RYMV/Rice相互作用图谱。我们使用Sim4物理定位了281个RSR基因[32]在水稻物理图谱上,关于内含子/外显子结构。地图是可用的(请参阅可用性和需求部分)。我们还绘制了之前确定的est和蛋白质的图谱[24,25]和参与部分抗性的7个qtl的位置[7].ESTs或cDNA-AFLP分析显示宿主基因表达的变化是感染的结果,而qtl参与了宿主耐受的直接反应。我们没有期望发现任何叠加,因为ESTs分析显示了大量表达的基因,而cDNA-AFLP技术显示了一些表达水平较低的基因,而且我们不知道部分耐药或耐药涉及的qtl是诱导还是抑制的。
对RSR序列分布的统计分析表明,RSR序列在4号染色体和10号染色体上的分布不均匀,因此可能强调了RSR序列失调控簇的假设。我们没有观察到任何具有类似功能的聚类重组RSR序列(“功能聚类”)。但我们并没有识别出所有参与对RYMV感染反应的基因,更深入的研究可以在微观水平上识别出更多的集群(特别是功能集群)。
来自IR64和Azucena的一些序列共定位(表2).部分RSR基因在RYMV感染下的基因表达模式得到了定量RT-PCR的证实,两品种之间存在一定差异。这种RSR基因可能与宿主反应的特异性有关。其中一个基因编码ABC转运蛋白(DQ883962和DQ884118 RSR序列)。该基因在敏感品种中在5 dpi处被高度抑制,而在部分抗性品种的受损叶片中过度表达(图4A2)。ABC转运体基因是cDNA-AFLP鉴定的最主要基因(Azucena有6条RSR序列,IR64有4条RSR序列),可能在植物对RYMV感染的应答中发挥重要作用。这些转运蛋白参与了大量结构和功能无关化合物的膜转运,如谷胱甘肽缀合物、脂类、无机酸、多肽、二次代谢、毒素和药物[33].研究表明,一些ABC转运蛋白,如AtPDR8,可能是在防御反应中控制细胞死亡程度的关键因素[34],并赋予重金属抗性[35].我们还观察到两个品种中鞘氨脂-1-磷酸裂解酶基因表达的变化(表2)2, DQ883945和DQ884107 RSR序列)。我们观察到,在易感品种RYMV感染的叶片中,基因表达在2 dpi时就显著下降(图4阿)。脂类影响与植物-微生物相互作用有关的发病机制和抗性机制,鞘脂类作为防御反应的激发子[36].这类在动力学过程中表达下调的基因家族可能是小rna的靶点,应进一步深入研究小rna对其表达调控的潜在影响。最后,我们注意到5个编码逆转录因子的RSR序列(DQ884156、DQ883989、DQ883835、DQ883911和DQ883916)的下调。这种负调控贯穿了紫果属侵染叶的整个动力学过程。更深层次的转录组分析现在正在进行,以确定病毒感染和这种逆转录因子下调之间的关系。
结论
本研究首次绘制了水稻/病毒相互作用的物理图谱。它为未来RYMV/水稻相互作用和其他水稻病原体的研究提供了强有力的基础,因为CMAP工具允许地图的组合。我们在易感和部分抗性品种中鉴定了宿主对RYMV感染的反应变化,并表明对RYMV的耐受现象涉及不同细胞水平的调控。在部分抗性和易感品种的反应中,一些基因似乎具有潜在的重要意义,研究它们在一个只有主要抗性qtl的近等基因系中的表达变化将是有趣的,这将避免由于两个品种遗传背景的差异而造成的偏差。此外,利用定量RT-PCR分析这些基因的表达变化可能是有趣的o . glaberrima具有不同抗性水平的物种假设测序可用。这将使进一步研究摆脱对病毒感染反应的物种间差异。
方法
植物材料
选用两个品种:IR64 (o.s. indica)和阿祖塞纳(金银花).IR64是由国际水稻研究所(IRRI)开发的高产品种,Azucena是来自菲律宾的传统旱地品种。两种品种种子分别播种,60株在28℃光照12 h, 24℃暗12 h,相对湿度70%的生长室中生长。每个品种用20株双叶期植株接种缓冲液(20 mM磷酸盐缓冲液,pH为7)或纯化的RYMV颗粒,浓度为100 μg/ml。这个实验在相同的条件下重复了两次。为了便于叶片的机械接种,在缓冲液中加入碳化硅。所有的植物都在一起种植,并同时收获,以尽量减少由于昼夜节律或收获时间与光照时间相关等因素造成的潜在问题。IR64叶片和Azucena叶片分别在接种后2、5、7 d (dpi)和3、5、7 d (dpi)收获非胁迫、损伤(缓冲接种)和RYMV接种的叶片。根据RT-PCR分析,选择这些收获日期作为病毒繁殖的适当时间,并对应于两个品种中相当的病毒积累量(图2)1).
RNA提取和ds-cDNA合成
总RNA采用Qiagen RNeasy Plant mini kit (Qiagen, Germany)和poly A提取+根据制造商说明书,使用Qiagen Oligotex mRNA纯化试剂盒(Qiagen, Germany)从200 μg总RNA中纯化RNA。总RNA的数量和质量,聚A+RNA在1%琼脂糖非变性凝胶和紫外光照下进行检测。根据制造商说明书,使用Stratagene cDNA合成试剂盒(Stratagene Inc., La Jolla, CA)从3 μg poly A+ RNA合成双链cDNA。
采用RT-PCR方法研究了RYMV在水稻中的积累。37],并根据制造商的说明(Life Technology)。439 bp片段用引物R18: 5'-GGTGTCAGCATAGTCGTAGAG-3'(3839-3819)和R17: 5'-CACACGTGCGGGGTGTGGAG-3'(3380-3400)扩增[38].
cDNA-AFLP过程
利用第一个时间点(IR64为2 dpi, Azucena为3 dpi)从未受胁迫和损伤的叶片中提取的RNA,以及IR64为2、5和7 dpi, Azucena为3、5和7 dpi的rymv感染叶片中提取的RNA进行cDNA-AFLP。每个双链cDNA样本用5u的Eco RI和Mse I (Gibco Invitrogen, Cergy Pontoise,法国)消化1微克,并将266 ng的Eco RI-和Mse I接头连接到消化的cDNA末端。适配器序列如下:Eco RI-adapter top strand, 5'- ctcgtagactgcgtac -3';生态ri -适配器底部绞线,5'-AATTGGTACGCAGTC-3';Mse i -适配器顶链,5'-GACGATGAGTCCTGAG-3';Mse i适配器底部绞线,5'-TACTCAGGACTCAT-3'。预扩增和选择性扩增引物的核心序列为:Eco RI引物,5'-GACTGCGTACCAATTC-3';Mse I引物,5'-GATGAGTCCTGAGTAA-3'。在每个引物的3'端添加选择性核苷酸,预扩增为0个核苷酸,选择性扩增为2个核苷酸(A/T/G/C和A/T/G/C)(256种不同引物组合)。利用[γ-]对Eco RI引物的5'端进行选择性扩增33P]ATP (Amersham Pharmacia Biotech, UK)和T4多核苷酸激酶(Appligene, Pleasanton, CA),根据制造商的说明。预扩增和选择性扩增在50 μl终体积中进行,终体积中含连接ds-DNA 20 ng, Eco RI和Mse I引物75 ng, dNTPs终浓度200 μl, 10 × Promega buffer (Promega, Madison, USA) 5 μl, MgCl2最终浓度为2.5 mM, 1u Taq聚合酶(Promega, Madison, USA)。PCR在PTC-200™热循环仪上进行,循环条件如下:94°C初始变性(30 s);13个“触地”循环:0.7°C下降每循环到最终退火温度56°C(30秒),72°C(1分钟);依次为33个周期:94°C (30 s);56°C(30秒);72°C(1分钟)。所得到的PCR产物在变性条件下通过聚丙烯酰胺凝胶电泳分离,然后凝胶在3mm (Whatman, Maidstone, UK)纸上干燥,并暴露于Biomax-MS x射线胶片(Kodak)。
cDNA扩增产物的分离和测序
感兴趣的条带被标记在薄膜上,从凝胶中切除并置于无菌管中。从变性凝胶中提取DNA片段,将凝胶带浸入50 μl无菌水中,在+4℃静置过夜,使DNA片段扩散。离心后,在与AFLP相同的PCR条件下,用AFLP选择性引物重新扩增。然后将DNA片段克隆到pGEM中®-T易矢量(Promega, Madison, USA)根据制造商的说明。每个AFLP条带分离并维持3个独立克隆。
测序与分析
质粒DNA采用Qiagen R.E.A.L.试剂盒制备。测序反应使用Applied Biosystems BigDye终止剂试剂盒进行,并在Applied Biosystems (Courtaboeuf France) 3100测序仪上进行分析。利用Basic Local Alignment Search Tool (BLAST)程序根据GenBank非冗余数据库和水稻est对核苷酸序列进行同源性分析[39].
定量聚合酶链反应
我们进行了两组独立的实验,包括10株健康植物,10株受伤植物和10株被RYMV感染的植物。从接种后第2天和第5天收获的14日龄叶片中提取总RNA。我们使用RNeasy kit (Qiagen)。Poly(dT) cDNA用Superscript III (Invitrogen)从3倍400 ng总RNA中制备。根据制造商的建议,在Stratagene Mx3005P仪器上使用FullVelocity SYBR Green QPCR Master Mix (Stratagene)进行定量。PCR在96孔光学反应板中进行,加热5分钟至95℃,然后在95℃下10秒循环40次,在60℃下退火-延伸30秒。目标定量使用使用Beacon Designer 4.0 (Premier Biosoft International, Palo Alto, USA)设计的特定引物对进行。表达水平归一化为ACTIN2(At3g18780)。每个实验均进行三次RT-PCR,所示值为均值正负标准差。
映射
这一步并不完全简单,因为我们将cDNA与基因组序列对齐。我们使用了12条水稻染色体的第五次释放[21].我们使用SIM4进行比对,它考虑了内含子/外显子结构,可以将表达序列标签(EST)或cDNA序列映射到基因组。因为cDNA序列来源于两个栽培稻生态型(寄主,o.s. indica(和Azucena,金银花),与基因组的生态型(Nipponbare,金银花),我们将IR64的cdna与Nipponbare的基因组序列进行比较,希望发现一些微小的多态性。一些分子分析一直显示两者之间的差异籼稻而且粳稻基因组DNA和重复序列的定量[40].然而,最近对4号染色体精细物理图谱的比较方法[41]揭示了籼稻而且粳稻物理图谱显示整体同向性,甚至包括一些种内dna序列多态性:插入/删除(indel)和单核苷酸多态性(SNP) [16].一个名为"选用“图谱比对计划”(OMAP)已开始研究进化、基因组组织、驯化、基因调控网络和作物改良[42,43].此外,每个基因内含子和外显子的平均数量、基因含量和顺序高度保守籼稻而且粳稻序列(44],因此使用Nipponbare序列作为参考来绘制和分析水稻基因序列是合理的。
第一个过滤器是整体识别率:选择所有识别率超过80%的RSR序列。所有同源性低于98%且超过50%的RSR序列均被删除。然后保留每个外显子95%以上同源性的所有序列(除了最后一个可能含有大量N的外显子)。因此,这些序列被映射到染色体上,并由第一个外显子的位置引用。
我们还绘制了之前确定参与这种相互作用的水稻/RYMV [25]使用同样的方法。使用blastx子程序定位参与相互作用的蛋白质,以比较每个肽与染色体的序列[24].地图右侧提到了这些ESTs和蛋白质。
统计分析
利用GENEPOP 3.1c版本的子程序STRUC,将每兆酶的RSR序列分布与沿染色体观察到的每兆酶ATG分布进行比较[45].本程序采用马尔可夫链法进行无偏估计P任意大小列联表的概率检验(或Fisher精确检验)的-value。对每条误差< 0.05的染色体进行偶然性检验。
可用性和需求
http://cmap.bioinfo.mpl.ird.fr/cmap/: 281条RSR序列的物理图。在每条染色体的左侧,RSR序列的起始位置为bp *100000,被鉴定为候选的ESTs和蛋白质参与了宿主反应[24,25].在左边,我们提到了参与部分抗性的7个qtl的位置[7].
参考文献
- 1.
Whitham SA, Yang C, Goodin MM:全球影响阐明植物对病毒感染的反应。MPMI。2006, 19: 1207-1215。
- 2.
Fargette D, Pinel A, Abubakar Z, Traore O, Brugidou C, Fatogoma S, Hebrard E, Choisy M, Sere Y, Fauquet C, Konate G:从基因组、系统发育和系统地理学研究推断水稻黄斑病毒的进化史。中国病毒学杂志,2004,28(3):342 - 344。10.1128 / jvi.78.7.3252 - 3261.2004。
- 3.
Qu C, Liljas L, Opalka N, Brugidou C, Yeager M, Beachy RN, Fauquet CM:三维结构域交换调节二十面体病毒群成员的稳定性。结构工程学报,2000,8(10):1095-1103。10.1016 / s0969 - 2126(00) 00508 - 6。
- 4.
Traore O, Sorho F, Pinel A, Abubakar Z, Banwo O, Maley J, Hebrard E, Winter S, Sere Y, Konate G, Fargette D:水稻黄斑病毒多样化和分散过程的大规模和高分辨率系统地理学研究。分子化学学报,2005,14:2097-2110。10.1111 / j.1365 - 294 x.2005.02578.x。
- 5.
杨晓明,张志刚,张志刚,张志刚,张志刚。水稻黄斑病毒在非洲的系统地理学研究。中华病毒学杂志,2003,34(3):344 - 344。10.1099 / vir.0.18759-0。
- 6.
水稻黄斑驳病毒的抗性评价选用物种。病毒学杂志,1993,9:65-73。
- 7.
Albar L, Lorieux M, Ahmadi N, Rimbault I, Pinel A, Sy AA, Fargette D, Ghesquière A:水稻黄斑病毒抗病qtl鉴定及其与植株形态关系的遗传学基础和定位应用理论,1998,37(3):344 - 344。10.1007 / s001220051003。
- 8.
Albar L, Bangratz-Reyser M, Hébrard E, Ndjiondjop M- n, Jones M, Ghesquière A:水稻eIF(iso)4G翻译起始因子的突变使水稻具有较高的抗性水稻黄斑驳病毒.植物学报,2006,47(3):417-426。10.1111 / j.1365 - 313 x.2006.02792.x。
- 9.
Pressoir G, Albar L, Ahmadi N, Rimbault I, Lorieux M, Fargette D, Ghesquière A:水稻黄斑病毒抗性的遗传基础和定位II:两个qtl之间互补上位性的证据应用理论与实践,1998,37(3):366 - 366。10.1007 / s001220051004。
- 10.
吴J,前原T,下川T,山本S,原田C,高崎Y,小野N,向井Y,小池K, Yazaki J,藤井F,下村A,安藤T,河野I, Waki K,山本K,矢野M,松本T,佐佐木T:包含6591个表达序列标记位点的水稻综合转录本图。植物学报,2002,14:525-535。10.1105 / tpc.010274。
- 11.
陈M,普雷斯特G, Barbazuk世行,Goicoechea杰,Blackmon B方G,金正日H,弗里希D,于Y,太阳年代,Higingbottom年代,Phimphilai J, Phimphilai D,瑟蒙德年代,Gaudette B,李P,刘J,哈特菲尔德J,主要D,法勒K,亨德森C,巴内特L,科斯塔R,威廉姆斯B,皮尔泽年代,阿特金斯M,大厅C, Budiman马服饰JP,罗M,班克罗夫特,泥火山J, Regad F, Mohapatra T,辛格NK, Tyagi AK, C,索德伦德院长RA,翼类风湿性关节炎:一个集成的物理和水稻基因组的遗传图谱。植物学报,2002,14:537- 544。10.1105 / tpc.010485。
- 12.
IRGSP:基于图谱的水稻基因组序列。自然科学学报,2005,37(3):344 - 344。10.1038 / nature03895。
- 13.
栽培稻L ssp籼稻).科学通报,2002,29(3):344 - 344。10.1126 / science.1068037。
- 14.
高夫SA,里奇D,局域网,普雷斯特G,王R,邓恩M, Glazebrook J,会话,Oeller P, Varma H,哈德利D,和记黄埔D,马丁·C片瞳F,兰格BM, Moughamer T,夏Y, Budworth P,钟J, Miguel T Paszkowski U,张年代,科尔伯特M,太阳王,陈L,库珀B,公园,木TC、毛L,鹌鹑P, R, R,院长于Y, Zharkikh,沈R, Sahasrabudhe年代,托马斯,罐头R, Gutin, Pruss D,里德J, Tavtigian年代,米切尔J·G,肖勒T,米勒RM,博年代,阿迪N, Rubano T,图斯尼姆N, Robinson R, Feldhaus J, Macalma T, Oliphant A, Briggs S:水稻基因组草案序列(栽培稻L ssp粳稻).科学通报,2002,29(3):344 - 344。10.1126 / science.1068275。
- 15.
佐佐木T,松本T,山本K,坂田K,巴巴T, Katayose Y,吴J, Niimura Y, Z、Nagamura Y,安东尼奥英航,Kanamori H,细川护熙年代,Masukawa M, Arikawa K, Chiden Y, Hayashi M, Okamoto M,安藤T,青木H, K,岩漠M,原田C, Hijishita年代,本田M,川Y, Idonuma,饭岛爱,Ikeda M, Ikeno M, Ito年代,Ito T, Ito Y, Ito Y,逐渐繁盛,Kamiya K, Karasawa W,片瞳年代,Kikuta,小林N,河野我Machita K,前原诚司T,美津浓H, Mizubayashi T,向井亚纪Y,长崎H,中岛美嘉M,中山Y、中道Y、中村M、Namiki N、根岸M、Ohta I、小野N、佐治S、酒井K、柴田M、下川T、Shomura A、宋j、高崎Y、寺泽K、辻K、Waki K、山形H、山根吉树S、吉原R、汤川K、钟H、Iwana H、远藤T、伊藤H、Hahn JH、Kim HI、Eun MY、矢野M、Jiang J、Gojobori T:水稻染色体1的基因组序列和结构。自然科学,2002,42(3):332 - 332。10.1038 / nature01184。
- 16.
顾敏,姜娟,李娟,洪光,薛勇,韩波:水稻4号染色体序列分析。自然科学学报,2002,26(3):344 - 344。10.1038 / nature01183。
- 17.
水稻3号染色体测序联盟:水稻3号染色体与不同草种间共时性的序列、注释和分析。中国生物医学工程学报,2005,30(4):344 - 344。10.1101 / gr.3869505。
- 18.
水稻染色体10测序联盟:水稻染色体10的结构、活性和进化的深入研究。科学通报,2003,30(3):344 - 344。10.1126 / science.1083523。
- 19.
水稻11号和12号染色体测序联盟:水稻11号和12号染色体序列,富含抗病基因和近期基因重复。BMC。2005, 3: 20-
- 20.
余娟,王娟,林伟,李松,李华,等:水稻基因组的复制史。公共科学图书馆,2005,3 (2):e38-10.1371/journal.pbio.0030038。
- 21.
TIGR水稻基因组注释。[http://www.tigr.org/tdb/e2k1/osa1/]
- 22.
Delseny M:为了获得水稻基因组的精确序列。中国植物学报,2003,26(6):531 - 531。10.1016 / s1369 - 5266(03) 00010 - 4。
- 23.
欧阳,李建平,李志刚:植物重复序列识别的一个集合资源——TIGR Plant Repeat数据库。中国生物医学工程学报,2004,32:D360-D363。10.1093 / nar / gkh099。
- 24.
文德龙-德博特M, Delalande F, Brizard J-P, Diemer H, Van Dorsselaer A, Brugidou C:蛋白质组分析水稻而且噢,日本苜蓿细胞悬浮液水稻黄斑驳病毒感染。蛋白质组学,2004,4:216-225。10.1002 / pmic.200300502。
- 25.
Ventelon-Debout M, Nguyen T-T-H, Wissocq A, Berger C, Laudie M, Piégu B, Cooke R, Ghesquière A, Delseny M, Brugidou C:水稻黄斑驳病毒感染的转录应答分析水稻而且粳稻品种。“万人迷”女友。2003, 27(4): 344 - 344。
- 26.
袁强,欧阳山,王安,朱伟,Maiti R, Lin H, Hamilton J, Haas B, Sultana R,张F, Wortman J, Buell CR:中国科学院基因组研究所水稻基因组注释数据库。植物生理学杂志,2005,38(4):344 - 344。10.1104 / pp.104.059063。
- 27.
赫尔R:马修斯的植物病毒学。爱思唯尔科学技术图书2001。
- 28.
Voinnet O: RNA沉默是植物对抗病毒的免疫系统。植物学报,2001,17:44-459。10.1016 / s0168 - 9525(01) 02367 - 8。
- 29.
Maule A, Leh V, Lederer C:病毒与宿主在兼容交互中的对话。植物学报,2002,5(4):279-84。10.1016 / s1369 - 5266(02) 00272 - 8。
- 30.
vylsteke M, Van Den Daele H, Vercauteren A, Zabeau M, Kuiper M: cDNA-AFLP转录调控的遗传解剖。植物学报,2006,45:439-446。10.1111 / j.1365 - 313 x.2005.02630.x。
- 31.
郭志刚,李志刚,李志刚,李志刚。腺病毒热休克反应的激活是病毒复制的关键。自然科学,2000,37(3):357 - 357。10.1038 / 35025102。
- 32.
张泽,张志军,张志军,张志军,王志军:cDNA序列与基因组DNA序列比对的计算机程序。中国生物工程学报,1998,8:967-974。
- 33.
Sánchez-Fernández R, Davies TG, Coleman JO, Rea PA: The拟南芥ABC蛋白超家族,完整目录。中国生物医学工程学报,2001,26(3):344 - 344。10.1074 / jbc.M103104200。
- 34.
Kobae Y, Sekino T, Yoshioka H, Nakagawa T, Martinoia E, Maeshima M: AtPDR8的缺失,一种质膜ABC转运蛋白拟南芥,在病原体感染时引起超敏细胞死亡。植物生理学报,2006,27(3):379 - 379。10.1093 /卡式肺囊虫肺炎/ pcj001。
- 35.
Kim DY, Bovet L, Maeshima M, Martinoia E, Lee Y: ABC输送机AtPDR8是一种镉挤压泵,具有抗重金属性能。植物学报,2007,30(2):344 - 344。10.1111 / j.1365 - 313 x.2007.03044.x。
- 36.
脂质、脂肪酶和脂质修饰酶在植物抗病中的作用。植物学报,2005,29(4):344 - 344。10.1146 / annurev.phyto.43.040204.135951。
- 37.
Brugidou C, Holt C, Yassi MN, Zhang S, Beachy R, Fauquet C:水稻黄斑驳病毒感染性全长cDNA克隆的合成及外壳蛋白的诱变。中国生物医学工程学报,2004,26(1):457 - 457。10.1016 / s0042 - 6822(95) 80025 - 5。
- 38.
Ngon A, Yassi M, Ritzenthaler C, Brugidou C, Fauquet C, Beachy RN:水稻黄斑驳病毒RNA的核苷酸序列和基因组特征。中华病毒学杂志,1994,27(4):349 - 349。
- 39.
Altschul SF, Gis W, Myers EW, Lipman DJ:基本的局部对齐搜索工具。分子生物学杂志,2000,15(3):441 - 441。
- 40.
Ohmido N, Kijima K, Akiyama Y, de Jong JH, Fukui K:总基因组DNA定量和选择的重复序列揭示了不同DNA家族同时发生的变化籼稻而且粳稻大米。中国生物医学工程学报,2000,26(3):388-394。10.1007 / s004380051182。
- 41.
基因组学报,2002,12:817-823。10.1101 / gr.48902。
- 42.
Wing RA, Ammiraju JSS, Luo M, Kim HR, Yu Y, Kudma D, Goicoechea J, Wang W, Nelson W, Rao K, Brar D, Mackill DJ, Han B, Soderlund C, Stein L, SanMiguel P, Jackson S选用地图比对项目:开启野生水稻物种遗传潜力的黄金路径。植物生态学报,2005,29(3):344 - 344。10.1007 / s11103 - 004 - 6237 - x。
- 43.
Ammiraju JSS, Luo M, Goicoechea JL, Wang W, Kudrna D, Mueller C, Talag J, Kim HR, Sisneros NB, Blackmon B, Fang E, Tomkins JB, Brar D, MacKill D, McCouch S, Kurata N, Lambert G, Galbraith DW, Arumuganathan K, Rao K, Walling JG, Gill N, Yu Y, SanMiguel P, Soderlund C, Jackson S, Wing RA选用细菌人工染色体文库资源:代表水稻属10个基因组类型的12个深覆盖大插入BAC文库的构建与分析Gen res, 2006, 16(1): 140-147。10.1101 / gr.3766306。Epub 2005年12月12日
- 44.
韩波,薛艳:水稻全基因组种内dna序列变异。植物学报,2003,6(2):134-138。10.1016 / s1369 - 5266(03) 00004 - 9。
- 45.
Raymond M, Rousset F:种群分化的精确检验。生物学报,2004,29(3):377 - 377。10.2307 / 2410454。
确认
作者感谢R. Cook、M. Laudie和C. Berger利用佩皮尼安大学的测序平台进行的所有测序。作者感谢A. Ghesquière和L. Albar的一般性评论和有益建议,以及P. Larmande的生物信息技术援助。这项工作在一定程度上得到了朗格多克-鲁西永地区和佩皮尼昂和蒙彼利埃(法国)的热纳波尔斯地区的支持。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
MVD和CB共同设计和协调了这项研究。MVD进行了分子遗传学工作和统计分析,参与了注释和作图,并起草了手稿。CTD进行了生物信息学和物理制图。TTHN参与了cDNA-AFLP检测。MB和CS进行定量RT-PCR。MD参与了研究的协调,并帮助起草了手稿。所有作者都阅读并批准了最终的手稿。
电子辅助材料
12870 _2007_234_moesm1_esm.doc
附加文件1:按功能分类的RSR序列表。所提供的表表示所有RSR序列的分类与一个假定的函数。RSR序列比对NR数据库(所有非冗余GenBank CDS翻译+ RefSeq蛋白+ PDB + SwissProt + PIR + PRF)。(doc 744 kb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
Ventelon-Debout, M., Tranchant-Dubreuil, C., Nguyen, TTH。et al。水稻黄斑病毒胁迫敏感型和耐寒型基因的研究BMC植物生物学8日,26日(2008年)。https://doi.org/10.1186/1471-2229-8-26
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-26
关键字
- 局部阻力
- 易感品种
- 水稻染色体
- 宿主基因表达
- 水稻黄斑驳病毒