摘要
背景
获取位于特定染色体上的重要表达序列标签(est)是目前比较困难的。构建单染色体EST文库是分离特定染色体上重要EST的有效策略。本研究建立了一种结合染色体显微解剖技术和杂交特异性扩增技术(HSA)快速分离黑麦1R染色体ESTs的方法。
结果
用玻璃针分离1R染色体,用蛋白酶K (PK)消化。染色体1R的DNA通过两轮PCR扩增得到一个退化寡核苷酸6-MW序列分3AI酶解位点作为引物。PCR产物用分3AI与接头HSA1连接,然后与HSA1杂交分3AI分别利用水杨酸处理和未处理的黑麦叶片的接子HSA2酶切cDNA。用HSA法提取杂交DNA片段,克隆到pMD18-T载体上。以HSA1和HSA2的部分序列为引物,通过PCR释放克隆插入片段,并对其进行测序。得到并分析的94条ESTs中,有6条已知序列位于黑麦染色体1R或小麦同源第1组染色体上;它们与小麦、大麦和/或其他植物的est高度同源稻科植物类,其中一些是由非生物或生物胁迫引起的。本研究分离到22条功能未知的ESTs,可能代表黑麦1R染色体上的一些新基因。
结论
建立了从黑麦1R染色体上快速克隆染色体特异性ESTs的新方法。本文所报道的信息对克隆和研究在1R染色体上发现的新基因有一定的参考价值。
背景
EST分析为发现所有生物体(不论其基因组大小)的基因开辟了令人兴奋的前景[1- - - - - -7]。EST独特基因的大规模定位可以为基因组和染色体的组织提供有价值的见解[8]。EST与染色体标志(短臂和长臂、常染色质、异染色质、着丝粒和端粒)和重组相关的分布对于染色体结构和进化的比较分析、基因分离以及小麦等大基因组物种的靶向基因组测序具有重要意义[qh]8- - - - - -10]。目前,大多数EST都来自cDNA文库,但由于多倍体植物基因组大且同源序列干扰严重,EST的鉴定和定位非常耗时费力。构建单染色体或染色体区域EST文库是分离特定染色体和/或特定染色体区域上重要EST的有效策略。
有几篇直接从特定染色体和/或特定染色体区域分离EST的报道,其中大多数是从人类染色体或染色体片段中分离EST,使用微解剖染色体DNA作为探针从cDNA文库中筛选ESTs [11,12]。利用微解剖介导的cDNA捕获技术成功地分离了微解剖染色体的ESTs [13,14]。
植物染色体显微解剖和微克隆研究已有十多年的历史。许多来自不同植物物种的染色体特异性DNA文库,如小麦[15,16],燕麦[17],大麦[18]和甜菜[19],都是使用这种策略构建的。我们的团队已经构建了几个植物染色体和染色体区域特异性DNA文库,并分离出与疾病相关的est基因[20.- - - - - -25]。
黑麦作为主要谷类作物物种的重要遗传资源(Secale cerealeL., 2n = 14,基因组R)对极端气候和土壤条件具有良好的适应性。众所周知,黑麦对化肥或杀虫剂等化学处理的要求最低,这使它成为特定地区(例如北欧)的生态和经济理想作物。26]。染色体1R,已被证明携带抵抗白粉病的基因[27]、茎锈病[28],叶锈病[29],黄锈[30.]和绿虫[31,尤其引起了科学家们的注意。
由于黑麦基因组大(约9000 Mb),迄今尚未建立高分辨率的连锁图谱[32],重复序列含量高,分子标记不足。基于同样的原因,构建整个黑麦基因组的EST图谱既低效又耗时。
众所周知,黑麦染色体1R中含有大量的抗性基因。SA是激活局部和全身获得性抗性(SAR)的关键信号,可以诱导一些抗性基因(R基因)的表达[33]。最近的证据表明,SA也调节细胞死亡,可能是通过一个涉及活性氧的正反馈循环[34,35]。
HSA技术是基于抑制PCR原理,选择和扩增两个复杂DNA样本的共同序列。使用寡核苷酸接头形成强钳确保了该方法的特异性,因此只有具有两种不同接头的片段才会被扩增,而具有一种接头的片段由于对接头的PCR抑制作用而不会被选择扩增[36]。本研究通过结合HSA的1R染色体显微解剖,建立了一种快速分离裸麦叶片ESTs的新方法。本研究建立的克隆特定染色体ESTs的方法尚未见报道。这将是研究特定染色体上基因的一种有用的方法。本文报道的ESTs可用于进一步克隆1R染色体上的新基因。
结果
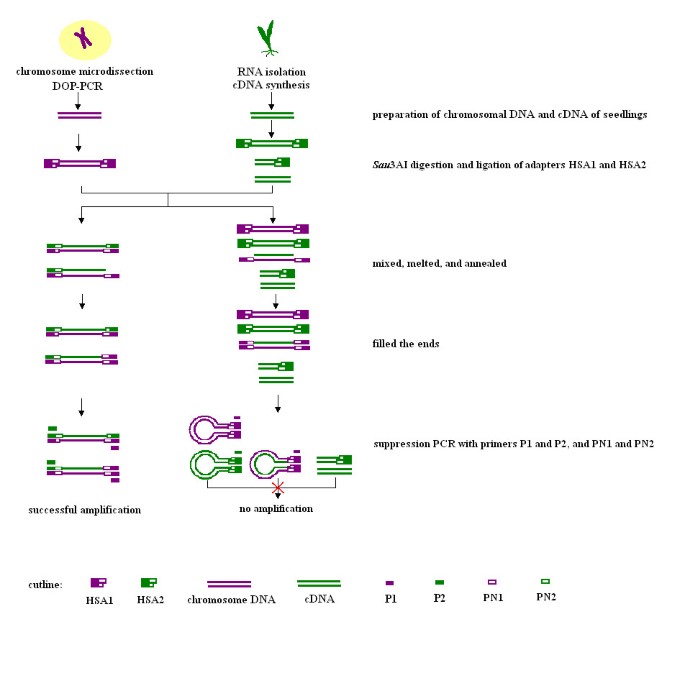
分离特定染色体表达序列的程序
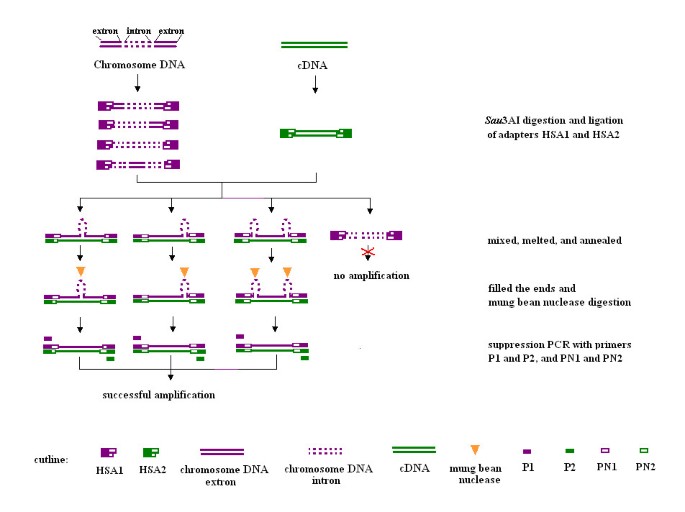
该方法的流程如图所示1.该方法的策略是染色体显微解剖法与HSA技术的结合[36得到微解剖DNA和cDNA之间的同源序列。如图所示1,对微解剖染色体进行退化寡核苷酸引物PCR (dopp -PCR)后,扩增的微解剖DNA和cDNA被分3AI和分别与两种适配器相连。上述接头和引物采用抑制减法杂交(SSH)设计[37],用钝化端改为退火端分3人工智能。将两种样品分别变性退火10 h,再混合退火10 h,得到3种杂交链:DNA-DNA杂交链、DNA-cDNA杂交链和DNA-cDNA杂交链。最后,进行两步PCR扩增以选择样品的杂交部分。只有来自不同样品、不同接头的DNA-cDNA杂交链才能呈指数扩增。由于接头的回文结构,DNA-DNA链和dna - cdna链在扩增过程中形成了长柄结构。因此,它们不能进一步扩大。
染色体1R的显微解剖、DNA扩增及鉴定
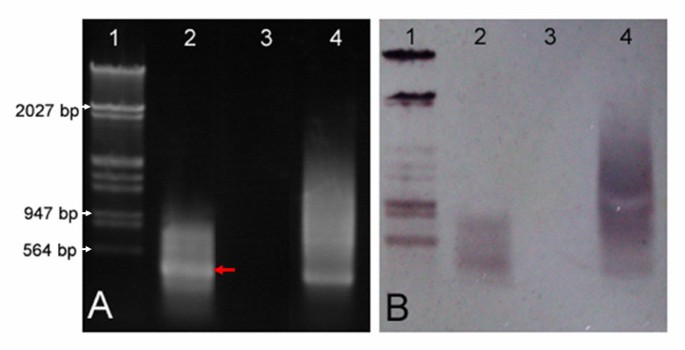
5条1R染色体成功显微解剖,并用两轮dopp - pcr扩增。第二轮PCR产物的大小在0.15 - 1.2 kb之间,主要片段在0.25-0.8 kb之间(图2)2阳性对照以10pg基因组黑麦DNA为模板,其条带更亮、更宽,大小在0.6 ~ 1.0 kb之间(图2)2阴性对照未得到产物(图2)2(第3车道),它不包含任何添加的模板DNA,因此用于监测任何可能的污染。
为了验证PCR产物的来源,将扩增产物与digi标记的黑麦基因组DNA进行杂交。杂交信号仅在阳性对照和1R染色体扩增产物中观察到(图2)2 b),表明微解剖的染色体确实来自黑麦基因组。此外,在琼脂糖凝胶中扩增的染色体1R产物的强条带(图2)2(车道2,红色箭头)被回收、纯化和测序。获得了300 bp大小的片段。与GenBank中的数据比较,该序列与的分散重复序列R173-1 (GenBank登录号X64100)一致Secale cereale,先前位于染色体1RS上[38]。这证实了扩增的DNA产物确实来自微解剖的染色体1R。
黑麦染色体1R DNA与cDNA杂交及抑制扩增
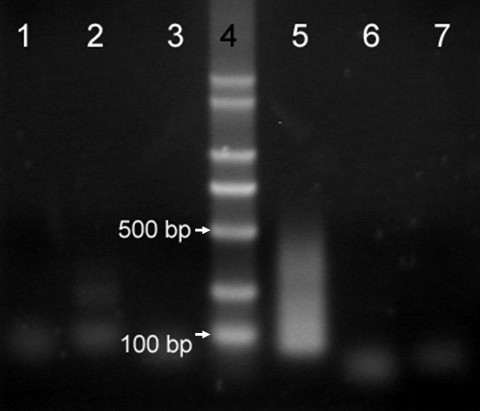
dopp - pcr得到的黑麦染色体1R DNA分别与接头HSA1连锁,并与未经SA处理的黑麦植株的hsa2连锁cDNA进行杂交。杂交后,采用单引物P1或P2、双引物P1 + P2、单引物PN1或PN2、双引物PN1 + PN2两轮抑制PCR扩增黑麦染色体1R DNA与cDNA之间的杂交片段。P1和PN1对应HSA1, P2和PN2对应HSA2。引物之间的关系如图所示1.用单引物P1、P2或双引物P1 + P2进行第一轮PCR均无明显PCR产物。在第二轮PCR中,当使用单引物PN1或PN2时,没有明显的PCR产物,而当使用双引物PN1 + PN2时,获得了大约80 - 500 bp的PCR产物(图2)3.).这些结果表明,杂交和抑制实验是成功的。
染色体1R ESTs的生成及序列分析
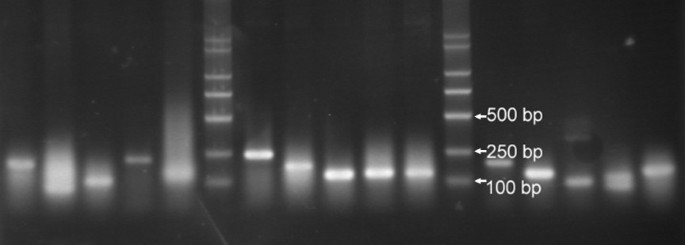
用双引物PN1和PN2扩增的PCR产物克隆到pMD18-T载体(TaKaRa, Dalian, China)上。每个培养皿中大约有100个重组克隆。从经SA诱导和未经SA诱导的黑麦叶片1r染色体EST文库中随机筛选出113个重组克隆。其中40个无性系来自sa诱导的叶片。用PN1和PN2引物PCR释放插入物并测序(图2)4).除引物序列外,113个插入片段长度为52 ~ 411bp。
113个插入片段中,污染序列(细菌DNA序列)4个,冗余序列15个,独特序列94个。在GenBank/EMBL数据库中进行Blast搜索,对这些样本进行命名、注册和分析。结果列在附加文件中1和2.染色体1R DNA具有引物PN1,而黑麦DNA具有引物PN2。成功分离出引物PN1和PN2(黑麦cDNA与1R DNA杂交产物,即1R染色体的表达序列)的序列。
在94个独特的插入中,60个在未经SA处理的情况下表达1),其中34例为sa诱导(附加文件2).这些序列分为两类:已知序列72条,与GenBank数据库中已有的EST序列同源(同源度> 80%);未知序列22条,在GenBank/EMBL数据库中没有匹配。在已知的序列中,所有序列都与est或来自黑麦、小麦和大麦的基因同源;6个已被定位到特定的染色体(表1)1) -黑麦1R染色体上的3 [38- - - - - -40],黑麦的7条染色体上都有一个[41], 2在小麦的同源族1上[42,43]。这明确地证明了我们的新方法可以分离染色体1R的ESTs。
在重组克隆中,冗余度为11%(15/113)。最常测序的插入片段是黑麦克隆F17高变DNA序列,编码大麦β -酮酰基- acp合成酶的基因[44]和大麦磷酸转运蛋白HvPT4。
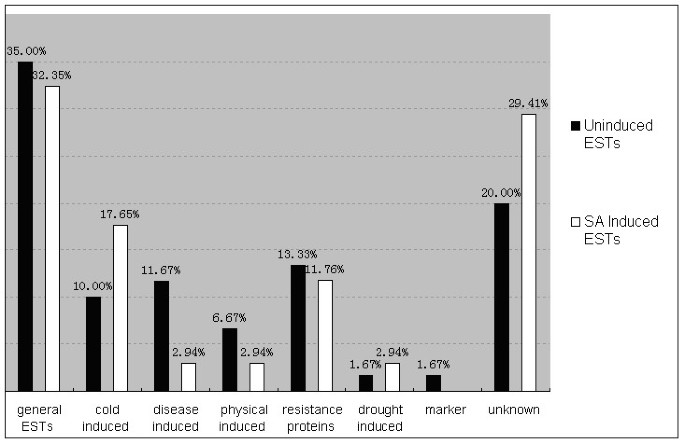
在未进行SA诱导的黑麦植株叶片中获得的60个序列中,45%是与生物和非生物胁迫相关的基因,35%与生物和非生物胁迫无关,20%是未知的基因。与未经SA诱导的黑麦叶片ESTs的功能分类相比,经SA处理的黑麦叶片中与温度诱导相关的ESTs和未知ESTs分别增加了7.65%和9.41%;与一般est相比,疾病诱导ESTs和抗性蛋白(RP)分别下降了2.65%、8.73%和1.57%(图2)5).
重组克隆的斑点杂交鉴定
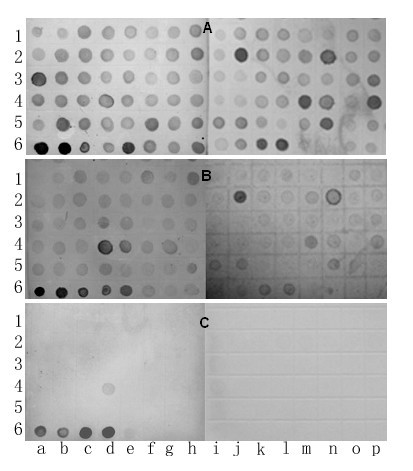
将随机选择的重组克隆的重组质粒DNA与digg标记的染色体1R二次dopp - pcr产物、SA诱导黑麦三叶期幼苗cDNA和黑麦基因组DNA进行杂交。几乎所有(99%)的重组质粒dna都能与染色体1R DOP-PCR产物杂交(图2)6),表明重组质粒DNA中的插入物与染色体1R DNA同源。重组质粒dna与黑麦幼苗的cDNA杂交时,杂交信号较弱,但部分无性系的杂交信号较强(图2)6 b).并非巧合的是,这些克隆与染色体1R的dopp - pcr产物杂交时具有更强的信号。然而,当重组克隆与黑麦基因组DNA杂交时,只有非常弱的杂交信号,甚至没有杂交信号(图2)6摄氏度).这些结果表明,所有的重组克隆都以单拷贝和低拷贝的形式存在于黑麦基因组中,这是表达基因的特征。
讨论
EST分析是克隆控制植物生物和非生物抗逆性及其他理想性状的重要新基因的有效方法[45,46]。数以百万计的植物est已在基因库数据库中登记。目前,大多数est都来自于不同发育阶段的不同组织的cDNA文库。然而,EST的鉴定和定位是费力和耗时的,特别是对于多倍体植物,因为基因组大小大,同源序列的干扰严重。通过传统遗传学和现代分子技术,许多重要的基因已经定位在特定的染色体和/或染色体的特定区域上[47,48]。因此,从特定的染色体或染色体的特定区域克隆ESTs可能是获得所需基因的有效方法。
本研究提出了一种从植物特定染色体克隆ESTs的新方法。扩增的dna有的含有PolyA尾,有的不含有PolyA尾,但都是来源于黑麦微解剖染色体1R的表达基因。我们的结果证明了这种策略是正确的和有效的。94个分离片段均含有来自两个不同接头的引物序列,其中53个片段有PolyA尾,41个片段无PolyA尾。6个序列分别位于黑麦的1R染色体和小麦的1组同源染色体上。这清楚地表明,我们的新方法可以分离染色体1R的ESTs。
以前报道过几种分离染色体表达序列(CES)或ESTs的方法。首先,在探针筛选方法中,利用来自染色体特异性文库的DNA片段作为探针筛选cDNA文库或以cDNA片段作为探针筛选染色体特异性文库,在膜上进行点杂交获得CES。利用该方法,已成功地从人和动物中分离出了CES [12,49- - - - - -52]。该方法可靠性高,但由于需要进行大量克隆操作和杂交克隆选择,耗时长。二、在杂交选择方法中[11],利用cdna对人类染色体12qHSR区微解剖产生的基因组DNA池进行杂交,并将其固定在尼龙膜上,然后对杂交片段进行PCR扩增。将染色体DNA固定在膜上,避免了多次克隆操作,提高了选择灵敏度。但由于cDNA的复杂性,会产生更多的假阳性,导致大量低丰度cDNA丢失。魏等.[51]通过使用这种方法发现,只有1/3的克隆是单/低拷贝片段。第三,在微解剖介导的cDNA捕获方法中,利用cDNA与接子杂交获得CES原位然后对杂交染色体和/或特定染色体区域进行显微解剖,随后使用接头连接的cDNA作为引物进行扩增[13,14,53]。该方法避免了染色体DNA的扩增,快速、巧妙,但假阳性率高。实际上,后两种方法都适用于CES富集。采用染色体显微解剖、DNA-cDNA液相杂交和抑制PCR相结合的方法,可以同时分离多个est或CES。与前三种方法相比,本方法更方便、经济、高效、易于控制。在我们的方法中,无需构建DNA(或cDNA)文库和点(或),即可获得ESTs或CES原位)杂交。
在以往的研究中,染色体特异性dna的扩增主要是通过连接-适配器PCR (LA-PCR)和dopp -PCR进行的。LA-PCR扩增的染色体特异性dna通常较长,但覆盖率低于dopp - pcr [17]。我们使用dopp - pcr扩增染色体1R DNA,得到长度在52 ~ 411bp之间的表达序列。这些序列中的大多数提供了足够的信息,可以与GenBank数据库中的ESTs进行比较,尽管它们比通过探针筛选方法获得的序列短[51,52]。
DNA-cDNA杂交总是存在一个常见的缺陷:由于基因内含子的存在,经常会出现不匹配的链。这一缺点导致观察到杂交序列不一定都是表达序列[36]。在这项研究中,我们使用单链特异性DNA和RNA内切酶绿豆核酸酶来切割部分不匹配的链。数字7展示了错配敏感绿豆核酸酶清除杂交产物内含子的整个过程。绿豆核酸酶对单链DNA和RNA具有高特异性;它是大多数需要单链特异性核酸酶的应用的首选酶[54]。从理论上讲,内含子不会与cdna杂交而形成环,环最终被绿豆核酸酶消化,可以避免内含子的影响。在这项研究中,没有一个分离的ESTs含有内含子。结果表明,绿豆核酸酶能有效地消化内含子。在以往的所有分离CES或ESTs的研究中,都没有使用这种内切酶来解决这个问题。
在染色体显微解剖过程中,来自细胞质和其他物种DNA的污染一直是一个严重的问题[55]。污染DNA的扩增将极大地影响染色体特异性DNA文库的质量。然而,这里的测序结果并没有显示任何来自外星DNA的严重污染。原因可能是在HSA过程中,只有DNA-cDNA杂交序列才能被检测和扩增。由于不能与黑麦叶片的1R染色体DNA或cDNA杂交,因此无法扩增。结果表明,HSA技术在我们的新方法中可以抑制污染物dna的扩增。
已知SA是几种物种获得性病原体抗性的信号分子[56,57]。它可用于诱导某些致病相关(PR)蛋白的合成,并增强对病原体的抵抗力[58,59]。简达等.[60研究发现,在正常生长条件下,向玉米幼苗的水培生长液中添加0.5 mM SA,可以防止随后的低温(2°C)胁迫。在我们的研究中,SA诱导显著增加了与温度诱导相关的ESTs和未知ESTs,而降低了一般ESTs、疾病诱导的ESTs和抗性蛋白(RP)。SA处理时间可能是与抗病性相关的ESTs数量减少的主要原因,因为不同的基因可能需要不同的SA处理时间才能达到显著表达[61]。在本研究中,我们仅使用单一处理时间(24 h)进行SA处理。推测,24小时的处理可能不适合某些抗病基因的表达。此外,SA诱导后未知ESTs增加的事实应该是有趣的。其中一些未知的est可能是与抗病相关的新基因。由于它们的功能尚不清楚,因此未被归类为抗性基因。需要对这些未知的无害环境技术进行调查。
发现新基因并检测其功能是大多数基因研究的目标。事实上,未知序列是这项研究中最有趣的部分。在本研究中,我们获得了22个未知序列(在GenBank数据库中没有匹配)和72个已知序列,它们与GenBank数据库中现有的EST序列具有高度的同源性。前者为从黑麦1R染色体上克隆新基因提供了机会。尽管我们在这里描述的方法具有插入物短的缺点,但将显微解剖和微克隆与最近开发的聚合酶克隆(Ploning)技术相结合,可能是解决这一问题的潜在方法。张等.[62]报道了一种Ploning方法,这是一种测序策略,通过使用实时等温扩增从单个细胞的DNA形成平面,从而消除了微生物的培养。他们报告说,单细胞大肠杆菌文库的基因组覆盖率为96.2%,插入长度为12 kb。通过使用phi29聚合酶和N6引物,平面导致更小的扩增偏倚和更大的插入片段[62]。
结论
总之,本研究建立了一种从植物特定染色体克隆ESTs的有效方法。这将为植物,特别是多倍体植物的基因组学和功能基因组学研究提供一种有用的方法。对黑麦1R染色体上的94个ESTs序列进行了克隆、测序和分析。这些序列可用于黑麦幼苗期染色体1R的基因表达研究,无论是否进行SA诱导。根据我们的研究结果,在未来的研究中也应该有可能克隆1R染色体上的新基因并阐明它们的功能。
方法
植物材料
本研究中使用的植物材料是Secale cerealeL. var. King II (2n = 14),由英国John Innes中心Terry Miller教授提供。
基因组DNA提取、mRNA分离及cDNA合成
黑麦种植,三叶期叶片喷施1 μg/L SA或水(对照)。使用Trizol Kit (Invitrogen, Carlsbad)从处理后24 h的植物幼叶中分离总RNA。采用poly吸引mRNA分离系统III (Promega, Madison)进行mRNA纯化。用cDNA Synthesis Kit (TaKaRa, Dalian)合成双链cDNA。采用CTAB法从黑麦嫩叶中提取基因组DNA [63]。
染色体1R的分离及其DNA扩增
染色体1R分离及其DNA扩增主要按照前面描述的程序进行[25]。简单地说,将黑麦种子浸泡在25°C的水中5-8 h,然后在潮湿的滤纸上在25°C的培养皿中黑暗发芽。种子发芽后,4℃培养24 h, 25℃暗培养,直至根长0.5-1 cm。带根种子在冰水(0°C)中处理24 h,增加中期细胞。收获根尖,在3:1乙醇:乙酸中固定5min,立即转入70%乙醇,-20℃保存。粉碎前,将根尖用2%纤维素酶(Yakukt, Tokyo)和2%果胶酶(Dingguo, Beijing)在75 mM KCl, 7.5 mM EDTA中37℃消化1 h,然后用双蒸馏水冲洗,4℃保存15-20 min。根尖在1% Carbol品红溶液中粉碎后,立即用于显微解剖。染色体1R,由它在短臂上的卫星识别[64],在倒置相衬显微镜(Olympus 1m,日本)上使用固定在LeitZ显微手术器械臂上的玻璃针对其进行显微解剖。将5条分离的1R染色体收集到0.2 ml Eppendorf管中,用10 μl PK缓冲液(0.5 mg/ml PK (Roche, Indianapolis), 1°C Taq Polymerase buffer (Promega, Madison))消化。随后,利用退化寡核苷酸(degeneredoligonucleotide, DOP)序列作为引物,采用PCR扩增染色体1R dna,该引物根据退化引物6mw [65通过改变限制位点来做一些修改Xho我到分3人工智能。设计的DOP引物序列为5′- CCGACTGATCNNNNNNATGTGG -3′。进行两轮PCR。第一轮PCR在同一试管中进行,加入4 μl 10°C Taq Polymerase buffer (Promega, Madison), 3 μl 25 mM MgCl2(Promega, Madison)、2.5 U Taq DNA聚合酶(Promega, Madison)、1 μl 10 mM dNTPs (MBI, Lithuania)、0.7 μl DOP引物(50 ng/μl)和不同量的双蒸馏水组成50 μl终体积。在最初变性(94°C, 10分钟),PCR进行5 low-stringency周期为94°C 1分钟,30°C 1.5分钟,3分钟和72°C,紧随其后的是25个high-stringency周期94°C的1分钟,1分钟55°C, 72°C 1.5分钟和最后一个扩展在72°C 10分钟。第二轮PCR是在相同的条件下完成上面所描述的,除了只有一个5μl产品从第一轮PCR作为模板,没有5 low-stringency周期。在所有程序中,除模板外,在相同的条件下进行严格的正、负对照实验。阳性对照实验以黑麦基因组DNA 10 pg为模板;阴性对照不使用DNA模板。
扩增染色体1R DNA的鉴定
为了确定扩增的微解剖DNA的来源,用黑麦基因组DNA作为探针。用CTAB法分离黑麦叶片DNA [66]。根据DIG DNA标记和检测试剂盒(Roche, Indianapolis)的方案进行探针制备、southern印迹、杂交和信号检测。用琼脂糖凝胶分离扩增的微解剖DNA中的明显条带,用DNA纯化与回收试剂盒(北京鼎果)回收纯化后测序。
从1R染色体产生ESTs
适配器
根据SSH的序列设计了适配器HSA1和HSA2 [37通过将钝端更改为不对称端(GATC)进行轻微修改,可识别为分3人工智能。
适配器HSA1
A1L 5“-GTAATACGACTCACTATAGGGCTCGAGCGGCCGCCCGGGCAGGT-3”
a1, 3 ' -CCCGTCCACTAG-5 '
适配器HSA2
A2L 5“-TGTAGCGTGAAGACGACAGAAAGGGCGTGGTGCGGAGGGCGGT-3”
a2, 3 ' -CCTCCCGCCACTAG-5 '
引物
我们根据HSA1和HSA2的序列设计了两对引物。P1和PN1对应HSA1, P2和PN2对应hsa2。
P1, 5‘-GTAATACGACTCACTATAGGGC-3’
P2, 5‘-TGTAGCGTGAAGACGACAGAA-3’
PN1 (P1的巢引物),5'-TCGAGCGGCCGCCCGGGCAGGT-3'
P2的巢引物PN2, 5'-AGGGCGTGGTGCGGAGGGCGGT-3'
黑麦染色体1R DNA与cDNA的杂交
用10u酶切黑麦1R染色体DNA (1 μg)和黑麦cDNA (1 μg)分3AI (TaKaRa, Dalian),体积为25 μl, 37℃过夜分将黑麦的1R DNA和cDNA分别与20 μl体积的HSA1和HSA2进行连锁分3AI酶切黑麦1R DNA或cDNA, 2 μl (10 μl)适配器hsa1或HSA2, 2 μl(10°C) T4连接酶缓冲液(TaKaRa,大连),1 μl (3.5 U/μl) T4连接酶(TaKaRa,大连),16°C保存过夜。结扎后,将样品在70°C下加热15分钟以使连接酶失活。将含有HSA1和HSA2的样品沉淀并重悬于2 μl杂交缓冲液(50 mM HEPES, pH 8.3;0.5 M NaCl;0.02 mM EDTA, pH 8.0;10% PEG 8000, W/V), 98℃变性10 min, 68℃孵育10 h。随后,尽快将两个样品混合在一起,68℃杂交10 h,用Taq DNA聚合酶(TaKaRa,大连,杂交DNA 1 U/μl)在72℃下填充1R DNA和黑麦cDNA的杂交DNA-cDNA双链,孵育20 min。
错配敏感核酸酶杂交DNA处理
为了消化DNA-cDNA双链中可能不匹配的片段,用0.1 U的绿豆核酸酶(TaKaRa, Dalian)在37℃下处理100 ng杂交DNA-cDNA等分15分钟。处理后的样品经苯酚/氯仿萃取纯化,并按上述方法进行沉淀[67]。
杂交DNA-cDNA的PCR扩增
经绿豆核酸酶酶切后,将杂交产物溶解于100 μl双蒸馏水中,其中1 μl作为PCR扩增模板,分别加入引物P1和P2 2 μl (5 μM,各1 μl)、10倍Taq反应缓冲液2.5 μl(北京天威)、Taq DNA聚合酶1 μl (2.5 U/μl,北京天威)和18.5 μl双蒸馏水,最终体积为25 μl。在95°C初始变性2分钟后,PCR进行30个循环,94°C 30秒,68°C 30秒,72°C 90秒,然后在72°C进行10分钟的最终延长。PCR产物用双倍蒸馏水按1:10的比例稀释。随后,在25 μl的体积中,用稀释后的PCR产物1 μl, PN1和PN2各2 μl (5 μM,各1 μl), 10倍Taq反应缓冲液2.5 μl(北京天威),Taq DNA聚合酶1 μl (2.5 U/μl,北京天威),在相同的条件下进行巢式PCR,但使用25个循环。
1R染色体ESTs序列的克隆与分析
巢式PCR产物经苯酚-氯仿提取,乙醇沉淀,溶解于40 μl双蒸馏水中。将纯化的第二轮PCR产物4 μl连接到pMD18-T载体(TakaRa, Dalian)上。共转化100 μl大肠杆菌将DH5α能态细胞转移到三个新鲜的直径为11 cm的含氨苄西林的板上。随机选取重组体的质粒,采用SDS - mini-制备法碱性裂解提取质粒[68],用PN1和PN2引物PCR释放插入物。释放的ESTs由北京Invitrogen公司测序。
为了对重组体进行表征,将质粒dna(每个0.1 ng)点缀在Hybond N上+筛选器(NEN Research, Boston, Mass.),然后与dig标记的第1R染色体二次dopp - pcr产物、SA诱导黑麦幼苗三叶期的cdna或黑麦基因组DNA杂交。根据DIG DNA标记和检测试剂盒(Roche, Indianapolis Cat.)的说明书进行点印迹杂交。没有1093657)。
数据分析
使用Blast检索程序将本研究获得的DNA序列及其推断的氨基酸序列与GenBank数据库中的DNA序列进行比较[69]。
缩写
- CES上:
-
-染色体表达序列
- 挖掘:
-
——digoxigenin
- DOP-PCR:
-
-退化寡核苷酸引物PCR
- est序列:
-
-表达序列标签
- 保险公司:
-
-杂交特异放大
- PK:
-
-蛋白酶K
- plone (:
-
-聚合酶克隆
- PN1:
-
- P1的巢引物
- PN2:
-
- P2的巢引物
- R基因:
-
-抗性基因
- 总机:
-
-抗性蛋白
- 山:
-
-水杨酸
- 特别行政区:
-
-全身获得性抵抗
- SSH:
-
-抑制减法杂交。
参考文献
- 1.
Adams MD, Kelley JM, Goceyne JD, Dubnick M, Polymeropoulos MH, Xiao H, Merril CR, Wu A, Olde B, Moreno RF, Kerlavage AR, McCombie WR, Venter JC:表达序列标记与人类基因组计划的互补DNA测序。科学通报,1991,25(5):551 - 556。10.1126 / science.2047873。
- 2.
Covitz PA, Smith LS, Long SR:根毛富集的表达序列标签Medicago truncatula互补脱氧核糖核酸库。植物生理学报,1998,17(2):559 - 561。10.1104 / pp.117.4.1325。
- 3.
Ewing RM, Kahla AB, Poirot O, Lopez F, Audic S, Claverie JM:水稻基因表达相关模式的大规模统计分析。中国生物医学工程学报,1999,9(1):959 -959。10.1101 / gr.9.10.950。
- 4.
Fernandes J, Brendel V, Gai X, Lal S, Chandler VL, Elumalai RP, Galbraith DW, Pierson EA, Walbot V:基于玉米表达序列标签频率分析和微阵列杂交的RNA表达谱比较。植物生理学报,2002,28(2):896- 896。10.1104 / pp.010681。
- 5.
Hillier L, Lennon G, Becker M, Bonaldo MF, Chiapelli B, Chisoe S, Dietrich N, DuBuque T, Favello A, Gish W: 28万个人类表达序列标签的生成与分析。中国生物医学工程学报,1996,6(6):887 - 888。10.1101 / gr.6.9.807。
- 6.
Shoemaker R, Keim P, Vodkin L, Retzel E, Clifton SW, Waterston R, Smoller D, Coryell V, Khanna A, Erpelding J, Gai X, Brendel V, Raph-Schmidt C, Shoop EG, Vielweber CJ, Schmatz M, Pape D, Bowers Y, Theising B, Martin J, Dante M, Wylie T, Granger C:大豆est的生成与分析。中国生物医学工程学报,2002,45(5):329-338。10.1139 / g01 - 150。
- 7.
Van der Hoeven R, Ronning C, Giovannoni J, Martin G, Tanksley S:基于大表达序列标签收集和选择性基因组测序的番茄基因组基因数量、组织和进化推断。植物学报,2002,14(4):441- 456。10.1105 / tpc.010478。
- 8.
气我,Echalier B,曹国伟年代,Lazo GR,巴特勒通用电气,安德森OD, Akhunov ED, Dvoøak JA Linkiewicz M, Ratnasiri, Dubcovsky J, Bermudez-Kandianis CE、格林RA, Kantety R,厘米,La值班员Munkvold JD,索雷尔科幻,索雷尔,Dilbirligi M, Sidhu D, Erayman M, Randhawa HS, Sandhu D,只有SN,吉尔KS,马哈茂德•艾哈迈迪AA,马XF, Miftahudin, Gustafson JP,康利EJ, Nduati V, Gonzalez-Hernandez杰,安德森JA,彭JH, Lapitan NLV,侯赛因公斤,Kalavacharla V, Kianian科幻,Pathan女士,张DS,阮HT,崔大伟,Fenton RD, Close TJ, McGuire PE, Qualset CO, Gill BS:多倍体小麦16000个表达序列标记位点的染色体Bin图谱及基因分布。遗传学报,2004,31(2):701-712。10.1534 / genetics.104.034868。
- 9.
侯赛因公斤,Kalavacharla V, Lazo GR, Hegstad J, Wentz MJ,西蒙斯K, Gehlhar年代,锈杰,Syamala RR, Obeori K, Suresh B Karunadharma P,曹国伟年代,安德森OD,气你,Echalier B, B,吉尔Linkiewicz, Ratnasiri, Dubcovsky J, Akhunov ED,德沃夏克J, Miftahudin,罗斯K, Gustafson JP, Sidhu D, Dilbirligi M,吉尔KS,彭JH, Lapitan NLV,格林RA, Bermudez-Kandianis CE,索雷尔,Feril O, Pathan女士,Nguyen HT Gonzalez-Hernandez杰,康利EJ,安德森JA,芬顿D, TJ, McGuire PE、Qualset CO, Kianian SF:小麦同源类群7 2148个表达序列标签位点的染色体bin图谱。遗传学报,2004,32(4):687-699。10.1534 / genetics.104.034850。
- 10.
Linkiewicz,气你,吉尔BS, Echalier B,曹国伟年代,Lazo GR,无角的DD,安德森OD, Akhunov ED,德沃夏克J, Pathan女士,阮HT,彭JH, Lapitan NLV, Miftahudin, Gustafson JP, La轮值表厘米,索雷尔,侯赛因公斤,Kalavacharla V, Kianian科幻,Sandhu D,只有SN,吉尔KS,康利EJ,安德森JA,芬顿RD, TJ, McGuire PE、Qualset有限公司Dubcovsky J: 2500 -轨迹本小麦部分同源的地图组5提供了见解与水稻基因分布和同线性。遗传学报,2004,18(2):665-676。10.1534 / genetics.104.034835。
- 11.
苏亚,Trent JM,关学祥,Meltzer PS:用染色体显微解剖直接分离均匀染色区编码的基因。美国国家科学促进会。[j] .中国科学:地球科学。10.1073 / pnas.91.19.9121。
- 12.
张敏,于丽,胡培林,毕德华,夏建辉,邓红霞,赵世义:利用显微解剖产生的探针池分离人染色体14q24.3的表达片段。生物实验学报。1997, 30(3): 241-246。
- 13.
Gracia E, Ray ME, Polymeropoulos MH, Dehejia A, Meltzer PS, Trent JM:利用微解剖介导的cDNA捕获技术分离染色体特异性ESTs。中国生物医学工程学报,1997,7(7):391 - 391。10.1101 / gr.7.2.100。
- 14.
Kim H, Kim C, Rhyu IJ, Kim JH, Lew DH, Chun YH, Park SH:新的人类胚胎大脑cdna的分离,人类染色体带,1q25和8q24.1。生物医学工程学报,2001,11(2):557 - 562。
- 15.
alalbani D, Côté MJ, Armstrong KC,陈启峰,Segal A, Robert LS:小麦微解剖染色体臂的单管PCR扩增。植物学报,1993,4(5):899-903。10.1046 / j.1365 - 313 x.1993.04050899.x。
- 16.
刘波,李建军,李建军,刘建军。小麦染色体臂基因组文库中染色体特异性DNA序列的分离与分析。植物学报,1997,11(5):959-965。10.1046 / j.1365 - 313 x.1997.11050959.x。
- 17.
陈强,Armstrong K:单个显微解剖燕麦文库的表征(燕麦属漂白亚麻纤维卷l .)染色体。中国生物医学工程学报,1995,38(3):756 - 756。
- 18.
Busch W, Martin R, Herrmann RG, Hohmann U:通过显微解剖分离的重复DNA序列。大麦芽l .)。中国生物医学工程学报,1995,38(3):1082-1090。
- 19.
Jung C, Claussen U, Horsthemke B, fish -cher F, Herrmann RG:个体DNA文库βpatellaris通过对减数分裂中期染色体的显微解剖获得的赋予线虫抗性的染色体。植物生理学报,2002,20(2):531 - 531。10.1007 / BF00040609。
- 20.
党比,胡志明,周永华,崔丽华,王丽丽,张太,李丽丽,陈志华:从植物中分离单染色体DNA文库王百合.科学通报,1998,43(2):194-199。
- 21.
姜思敏,胡军,尹文博,陈永华,王荣荣,胡志明:小麦-薄麦中间品系外源染色体上抗性基因类似物的克隆。理论与应用,2005,31(5):923-931。10.1007 / s00122 - 005 - 0022 - 3。
- 22.
姜思敏,张丽,胡军,石锐,周刚,陈永华,尹文彬,王荣荣,胡志明:大麦黄矮病毒诱导小麦Thynopyrum外源附加系差异表达基因的筛选与分析。中国生物医学工程学报,2004,17(4):414 - 421。10.1139 / g04 - 070。
- 23.
张飞,尹文博,石睿,胡玉坤,闫彦明,陈永华,周永华,胡军,王荣荣,胡志明:小麦1B染色体特异性DNA文库的构建与鉴定。植物学报,2005,35(5):369 - 369。
- 24.
周永华,党比,王辉,胡志明,王丽丽,陈志华:大豆单条染色体的显微解剖及t-微克隆文库的构建。植物学报,2001,31(2):1299 - 1295。10.1023 /: 1012043905480。
- 25.
周永华,胡志明,党比,王华,邓小东,陈志:黑麦的显微解剖与微克隆(英文)Secale cerealeL.)染色体1R。染色体工程学报,1999,28(2):559 - 559。10.1007 / s004120050375。
- 26.
Kubaláková M, Valárik M, Bartoš J, Vrána J, Cíhalíková J, Molnár-Láng M, Doleže J:黑麦(Secale cerealeL.)染色体使用流式细胞术。中国生物医学工程学报,2003,26(4):893-905。10.1139 / g03 - 054。
- 27.
van Kints TMC:小麦霉变。英国谷物致病菌毒力调查。年度报告。1986,7-12。
- 28.
马戈,王志军,李建军,李建军,李建军。黑麦茎锈病抗性基因的研究[j]。SrR)及大麦白粉病(Mla)位于染色体短臂上的共丝区。基因工程学报。2004,47(4):112-121。10.1139 / g03 - 096。
- 29.
韦惠玲,林芝A, Hackauf B, Roux SR, Ruge B, Klocke B:黑麦叶锈病抗性研究(英文)Secale cerealel .)。1.抗性基因的遗传分析与定位Pr1和Pr2.计算机工程学报,2003,19(4):432-438。10.1007 / s00122 - 003 - 1263 - 7。
- 30.
石志新,陈晓明,Line RF,梁宏,Wellings CR:抗性基因相似多态性标记的开发Yr9小麦条锈病的基因抗性。中国生物医学工程学报,2001,44(4):509-516。10.1139 / gen - 44 - 4 - 509。
- 31.
Wood EA, Sebesta EE, Starks KJ:“高乔”小黑麦对谷草裂蚜的抗性。环境昆虫学报,2004,3(3):720-721。
- 32.
Bennett MD, Gustafson JP, Smith JB:属中核DNA的变异Secale.染色体学报,1997,6(1):149-176。10.1007 / BF00327398。
- 33.
李建军,李建军,李建军,李建军,李建军,李建军,李建军,等。水杨酸对水稻获得性耐药的影响。科学,1993,61:754-756。10.1126 / science.261.5122.754。
- 34.
种子发育过程中糖的输入与代谢。植物科学,1997,2:162-165。10.1016 / s1360 - 1385(97) 01030 - 3。
- 35.
李建军,李建军,李建军,杨建军,杨建军,杨建军。水杨酸在植物免疫系统中的应用。植物学报,1997,9(6):391 - 391。10.1105 / tpc.9.2.261。
- 36.
Lecerf F, Foggia L, Mulsant P, Bonnet A, Hatey F:一种分离两种DNA样本共同部分的新方法:杂交特异性扩增(HSA)。核酸学报,2001,29 (17):e87-10.1093/nar/29.17.e87。
- 37.
Diatchenko L, Lau YF, Campbell AP, Chenchik A, Moqadam F, Huang B, Lukyanov S, Lukyanov K, Gurskaya N, Sverdlov ED, Siebert PD:抑制减杂:一种生成差异调控或组织特异性cDNA探针和文库的方法。美国国家科学促进会。[j] .中国科学:地球科学。10.1073 / pnas.93.12.6025。
- 38.
Rogowsky PM, Langridge P, Shepherd KW:基于聚合酶链反应的黑麦重复DNA序列定位。中国生物医学工程学报,2009,35(5):621-626。
- 39.
Clarke BC, Mukai Y, apels R:黑麦1R染色体短臂上的Sec-1位点(英文)Secale cerealel .)。染色体工程学报,1996,35(5):369 - 369。
- 40.
王晓明,王晓明,王晓明,等。植物硫氧还毒素亚类基因的克隆与表达。中国生物医学工程学报,2009,32(4):559 - 561。10.1046 / j.1432-1327.2000.01811.x。
- 41.
高建民,杜国祥,徐宝宝,徐志刚,Moon HP:黑麦基因组特异性RAPD产物的鉴定和染色体组织。中国生物医学工程学报,2002,45(1):157-164。10.1139 / g01 - 133。
- 42.
顾永强,Coleman-Derr D,孔晓东,Anderson OD:四种植物同源区比较序列分析揭示的基因组快速进化Triticeae基因组。植物生理学报,2004,35(1):459- 457。10.1104 / pp.103.038083。
- 43.
孔学勇,顾玉青,游峰峰,Dubcovsky J, Anderson OD .异源多倍体小麦基因组同源和旁系部分的进化动态。植物化学学报,2004,31(1):555 - 569。10.1023 / B: PLAN.0000028768.21587.dc。
- 44.
Wissenbach M:大麦的新成员内基因家族编码-酮酰基-酰基载体蛋白合成酶。植物生理学报,2004,26(4):444 - 444。10.1104 / pp.106.4.1711。
- 45.
李建军,李建军,李建军,等:植物基因组的大规模测序。植物学报,1998,1(2):136-141。10.1016 / s1369 - 5266(98) 80015 - 0。
- 46.
佐佐木T:日本的水稻基因组计划。美国国家科学促进会。中国机械工程,1998,25(5):327 - 328。10.1073 / pnas.95.5.2027。
- 47.
Peñuela S, Danesh D, Young D .大豆非tir NBS-LRR基因的靶向分离、序列分析和物理定位。理论与应用,2002,(4):261-272。10.1007 / s00122 - 001 - 0785 - 0。
- 48.
Spielmeyer W, Huang L, Bariana H, Laroche A, Gill BS, Lagudah ES: NBS-LRR序列与小麦叶片和条锈病抗性相关。理论与应用,2000,31(1):1139-1144。10.1007 / s001220051590。
- 49.
Yokoi H, Hadano S, Kogi M, Kang XL, Wakasa K, john - elkeda:利用激光显微解剖产生的微克隆探针分离人类Xq末端编码的表达序列。基因工程学报,1994,20(4):444 - 444。10.1006 / geno.1994.1194。
- 50.
高富峰,于军,童士生,齐军,Patanjali SR, Weissman SM, Patterson D:人类21号染色体表达序列的分离和精细区域定位。基因工程学报,1994,23(3):700-703。10.1006 / geno.1994.1561。
- 51.
人21号染色体20兆基GC-poor区基因鉴定及DNA序列分析。美国国家科学促进会。[j] .中国科学:地球科学。10.1073 / pnas.94.13.6862。
- 52.
魏军,王毅,冯毅,王勇,冯毅,冯毅。小鼠16号染色体DNA微解剖的cDNA直接筛选:新克隆的分离和c4 - c4区部分转录图谱的构建。中国生物医学工程学报,1996,26(8):678-687。10.1101 / gr.6.8.678。
- 53.
横山洋,大杉K, Kozaki T, Sakuragawa N:染色体区域特异性cdna的微解剖介导选择。中国生物医学工程学报,1997,26(3):391 - 391。
- 54.
李建军,李建军,李建军,等。单链特异性核酸酶的研究进展。核酸学报,2004,32(8):2632-2641。10.1093 / nar / gkh599。
- 55.
胡志明,王华,陈玉华,石荣,陈志华:植物染色体显微解剖中细胞质DNA污染及其控制研究。植物学报,2003,45(2):131-135。
- 56.
Malamy J, Carr JP, Klessig DF, Raskin I:水杨酸在烟草对病毒感染的抗性反应中的内源信号。科学,1990,25(2):100 -100。10.1126 / science.250.4983.1002。
- 57.
杨建军,杨建军,杨建军,等。烟叶中水杨酸合成途径的研究进展。植物生理学报,1993,32(3):391 - 391。
- 58.
Hooft van Huijsduijnen RAM, Alblas SW, de Rijk RH, Bol JF:水杨酸诱导不同植物对紫花苜蓿花叶病毒感染的致病相关蛋白及抗性。中国生物医学工程学报,2003,17(2):444 - 444。
- 59.
杨建军,杨建军,杨建军,杨建军,杨建军,杨建军,杨建军,杨建军,杨建军,杨建军,杨建军,杨建军。科学,1990,25(5):1004-1006。10.1126 / science.250.4983.1004。
- 60.
Janda T, Szalai G, Tari I, Páldi E:水杨酸水培处理降低玉米冷害效应(玉米l .)植物。植物学报,1999,28(4):559 - 564。10.1007 / s004250050547。
- 61.
Tan XP, Meyers BC, Kozik A, West ML, Morgante M, Clair DA, Bent AF, Michelmore RW:拟南芥富含亮氨酸核苷酸结合位点重复编码基因的表达分析。植物生态学报,2007,27(7):559 - 566。
- 62.
张凯,Martiny AC, Reppas NB, Barry KW, Malek J, Chisholm SW, Church GM:单细胞基因组的聚合酶克隆。生物工程学报,2006,24(4):668 -686。10.1038 / nbt1214。
- 63.
Murray MG, Thompson WF:高分子量植物DNA的快速分离。中国生物医学工程学报,1998,8(3):422 - 422。10.1093 / nar / 8.19.4321。
- 64.
王晓明,王晓明,王晓明,等。黑麦染色体的染色研究。遗传学报,1997,22(2):357 - 357。10.1266 / jjg.52.315。
- 65.
Telenius H, Carter NP, Bebb CE, Nordenskjöld M, Ponder BAJ, tunacliffe A:退化寡核苷酸引物PCR:用单一退化引物扩增目标DNA。基因组学,1992,13:718-725。10.1016 / 0888 - 7543 (92) 90147 - k。
- 66.
马建军,李建军,李建军,等。大麦核糖体DNA间隔长度多态性:孟德尔遗传、染色体定位和群体动态。美国国家科学促进会。[j] .中国科学:地球科学。10.1073 / pnas.81.24.8014。
- 67.
李建军,李建军,李建军,李建军,等。基于DNA杂交的DNA检测方法的研究进展。核酸学报,2004,32 (16):e130-10.1093/nar/gnh125。
- 68.
Sambrook J, Russell DW:分子克隆:实验室手册。第3版。冷泉港:冷泉港实验室出版社,2001。
- 69.
致谢
我们感谢Rob Griesbach博士和Sandra mattson小姐(美国农业部,花卉和苗圃植物研究)的英文编辑协助。本研究得到国家自然科学基金项目(30270708)、中国科学院项目(KSCX-YW-N-009)和中国科学技术部项目(2006CB101600)的资助。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
RNZ进行染色体显微解剖、杂交和统计分析,参与序列比对并撰写稿件。RS参与了本研究的设计。SMJ和YHC进行统计分析。WBY进行了部分杂交工作。HHW参与了部分染色体显微解剖。JH参与了序列比对。XQZ协助起草稿件。RRCW获得了黑麦种子并彻底修改了手稿。ZMH参与了研究的设计并起草了稿件。
电子补充资料
12870 _2007_236_moesm1_esm.doc
附文件1:未经SA诱导的黑麦植株1R染色体ESTs序列分析。该数据为未经SA诱导的黑麦植株1R染色体est序列分析提供了信息。(doc 128 kb)
12870 _2007_236_moesm2_esm.doc
附文件2:SA诱导黑麦植株1R染色体ESTs序列分析。该数据为SA诱导黑麦植株1R染色体est序列分析提供了信息。(doc 84kb)
权利和权限
开放获取本文由BioMed Central Ltd.授权发表。这是一篇开放获取的文章,在知识共享署名许可(https://creativecommons.org/licenses/by/2.0),允许在任何媒体上不受限制地使用、分发和复制,前提是正确引用原创作品。
关于本文
引用本文
周,RN。,Shi, R., Jiang, SM.et al。黑麦1R染色体EST快速分离。BMC Plant Biol8,28(2008)。https://doi.org/10.1186/1471-2229-8-28
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-28
关键字
- 水杨酸
- 特定的染色体
- 水杨酸处理
- Microdissected染色体
- 绿豆核酸酶