摘要
背景
番茄(茄属植物lycopersicon)和土豆(美国tuberosum)是两种经济上重要的作物品种,目前正在对其基因组进行测序。这项研究首次对这两个物种进行了全基因组分析,其基础是两个大的BAC端序列集合,约占番茄基因组的19%和马铃薯基因组的10%。
结果
番茄基因组比马铃薯基因组有更高的重复含量,主要是由于番茄基因组中有更多的逆转录转座子插入。另一方面,简单序列重复在马铃薯中比在番茄中更丰富。两个基因组在SSR基序的频率分布上也存在差异。根据EST和蛋白质比对,马铃薯似乎比番茄多包含6400个假定的编码区。细胞色素P450单加氧酶和丝氨酸苏氨酸蛋白激酶等主要基因家族在马铃薯中显著高于番茄。此外,P450超家族似乎在两个物种中都有惊人的扩大拟南芥这表明在大脑中有一个扩展的次生代谢途径网络茄科.番茄和马铃薯似乎都有较低水平的微共质性答:芥.观察到较高程度的同向性杨树trichocarpa特别是在15.2 - 19.4 Mb之间的区域p . trichocarpa10号染色体。
结论
这篇论文的发现首次揭示了茄科植物基因组的进化,无论是在家族内部还是相对于其他植物物种。当这些物种的全基因组序列可用时,全基因组比较和蛋白质或重复家族特异性研究可能会对这里的观察结果提供更多的解释。
背景
的茄科龙葵科是一个双子叶植物科,包括许多在农业、园艺和其他工业中使用的重要经济属。家庭成员包括有块茎的马铃薯(茄属植物tuberosum);大量结果蔬菜,如辣椒(辣椒spp)、西红柿(美国lycopersicum)、茄子(美国melongena);叶烟草(烟草);和观赏花卉从佩妮而且茄属植物属。
番茄通常被认为是一种模式作物植物物种,其具有许多高质量的遗传和基因组资源,如高密度分子图谱[1],许多特征良好的近等基因系(NILs),以及丰富的ESTs和全长cdna集合[2,3.].马铃薯是世界上最重要的农作物茄科它是继小麦、玉米和水稻之后的世界第四大粮食作物。土豆也有类似的资源,包括一张超高密度链接图[4],表现型数据的集合[5],以及一个大型的文字记录数据库[6].像大多数其他的夜生植物一样,番茄和马铃薯的基本染色体数都是12,它们的基因组之间存在全基因组共线性关系。7].
目前正在投入大量精力对这些生物的细胞核和细胞器基因组进行测序。国际番茄基因组测序计划[8正在对番茄进行测序(美国lycopersicum简历。在茄科全家族项目(SOL)的背景下研究Heinz 1706)基因组。而不是测序完整的基因组,大约950 Mb [9],只有基因丰富的染色质区域(估计为240 Mb)正在使用bac -逐层bac走行方法进行测序[10].马铃薯基因组测序联盟(PGSC) [11]的目的是为整个马铃薯(美国tuberosum,基因型RH89-039-16)基因组约840 Mb [4]使用类似的标记锚定BAC-by-BAC测序策略。
这两个测序项目都严重依赖于BAC库,其中番茄有三个BAC库(12]、MboI和EcoRI)和两个马铃薯存在(印度ii和EcoRI)。番茄文库可通过SOL基因组学网络(SGN)获得[13]和马铃薯库将很快通过PGSC [11].所有这些文库都进行了最终测序,以支持BAC-by-BAC测序和扩展,并提供全基因组调查序列的基础,以支持像这里介绍的研究。
本文对310,580个番茄BAC End Sequences (BESs)进行了详细的序列分析,分别占番茄基因组的181.1 Mb(~19%)和128,819个马铃薯BESs,分别占马铃薯基因组的87.0 Mb(~10%)(番茄和马铃薯BES数据概述见表)1).本比较基因组学研究的目的是通过重复和基因含量分析来了解番茄和马铃薯基因组之间的相似性,以及通过基因功能分析来了解番茄和马铃薯基因组之间的相似性。此外,我们还研究了这两个茄科基因组和其他几种植物基因组之间的微同步关系。来自特定文库的BESs的序列含量会因所使用的限制酶的不同而有所偏差。为了避免比较具有不同偏差的序列集,番茄-马铃薯比较只在使用相同酶的库的BESs之间进行。
结果
重复密度和分类
基于重复序列数据库的相似性搜索,在番茄BESs中有13.0%到22.9%的核苷酸被确定属于重复序列(见表2第二至第四列)。番茄文库中最常见的重复序列家族是吉普赛(5.0 - 11.6%)和Copia(4.2 - 5.3%)类的反转录转座子。另一类显著的重复序列是核糖体RNA基因(<0.1 - 8.6%)。番茄Eco (EcoRI)文库的重复密度最低,为13.0%,这可能与吉普赛反转录转座子数量较少(5.0%)有关。番茄Mbo (MboI)文库中重复序列含量最高(22.9%),其中核糖体RNA基因占比超过1 / 3(8.6%)。注意,由于重复检测是基于序列相似度,一个BES中的不同片段可以被分配到多个重复族。因此,每个重复类型的重复内容的总和可能比总重复内容略大。
与番茄BESs相比,马铃薯BESs中只有10.0% ~ 12.5%的核苷酸与已知的相似Magnoliaphytae重复(见表2,第五和第六列)。与番茄一样,大部分的重复出现在Gypsy(5.4 - 8.6%)和Copia(2.5 - 2.6%)逆转录转座子家族中,而核糖体RNA基因的比例很小(<0.1 - 0.5%)。马铃薯含有的直线和正弦元素大约是番茄的两倍(见表)2),尽管绝对比例很低。此外,在马铃薯中观察到的II类DNA转座子比例更高(1.0 - 1.2%,而在番茄中观察到的II类DNA转座子比例为0.5 - 0.7%),其中大多数无法分类。与观察到的番茄HBa (HindIII)和Eco库之间的差异一致,马铃薯PPT (EcoRI)库的重复含量总体上低于POT (HindIII)库,更具体地说,更低的Gypsy反转录转座子数量(5.4%比POT库的8.6%)。与POT文库相比,PPT文库在核糖体RNA基因中也有富集(0.5% vs小于0.1%),就像在番茄中比较Eco文库和HBa文库一样。
由于基于相似性的重复检测可能受到重复数据库的大小和多样性的限制,为了估计BESs内的冗余,我们对BESs进行了自我比较。即使严格要求给定查询序列中至少50%的核苷酸与另一个具有至少90%身份的BES匹配,番茄BESs中52.0%的核苷酸与一个或多个其他番茄BESs匹配,19.0%的核苷酸与5个或更多其他BESs匹配。马铃薯BESs的冗余度低于番茄;在马铃薯BESs中,39.0%的核苷酸对至少一个其他马铃薯BESs有影响,12.9%的核苷酸对5个或更多的马铃薯BESs有影响。这种差异不能仅仅归因于番茄的贝叶斯数量多于马铃薯的贝叶斯数量;番茄HBa库(其大小与马铃薯POT和PPT库的总和差不多)的自我比较显示,该库中50.7%的核苷酸至少匹配一个其他HBa BES, 16.8%的核苷酸匹配5个或更多其他HBa BES。在这两个物种中,与5个或更多其他BESs匹配的核苷酸的百分比仅略高于RepeatMasker分析的结果(见表)2),表明本研究使用的重复序列数据库足以检测这些物种中大部分高度丰富的重复序列。这些发现也证实了从基于相似性的重复检测中观察到的,番茄的贝叶斯比土豆的贝叶斯更重复。
简单序列重复
在番茄BESs中共检测到28,423个SSR,基序长度在1 - 5nt之间,总长度至少为15 nt,相当于每6.4 kb基因组序列中有1个SSR。术语“母题长度”在这里用来描述SSR中重复出现的母题长度;例如,ATATAT重复有两个母题长度(AT是母题)。motif长度最多的是5个核苷酸(11,177个SSRs),其次是2个核苷酸(6,588个SSRs), 4个核苷酸(4,596个SSRs), 3个核苷酸(4,135个SSRs),最后一个核苷酸(1,927个SSRs)。
在马铃薯中,共发现19,019个SSRs,其中3,964个(21%)属于I类(即包含多于10个基序重复的SSRs)。因此,马铃薯BESs基因组序列每4.6 kb有1个SSR,高于番茄(每6.4 kb有1个SSR)。与番茄一样,马铃薯SSRs中最丰富的基序长度为5个核苷酸(7922个SSRs)。然而,其次最丰富的长度是3个(3941个SSRs),其次是两个(3270个SSRs),四个(1980个SSRs)和一个(1906个SSRs)的motif长度。
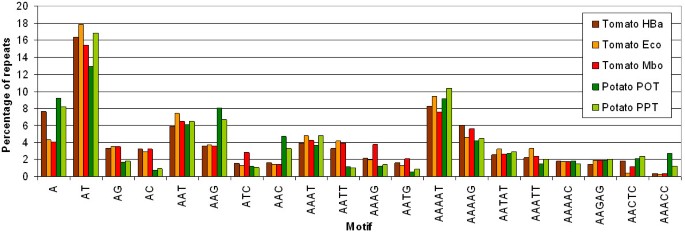
数字1显示了番茄和马铃薯BESs中SSR主基序的分布,按基序长度和相同长度基序中的相对频率排序。两个数据集中最丰富的SSR基序都是AT-rich,其中双核苷酸重复AT/TA是最丰富的(分别占番茄和马铃薯SSR的16.6%和14.7%)。AG/CT、AC/GT、AATT/AATT、AAAG/CTTT等基序在番茄中比在马铃薯中多见,而AAG/CTT、AAC/GTT、AACTC/GAGTT、AAACC/GGTTT等基序在马铃薯中多见。
如果只考虑I类SSR,番茄和马铃薯的SSR基序最多的是AT/TA(分别占所有I类SSR的50.8%和39.1%)和A/T(分别占25.8%和42.1%)。其次是番茄双核苷酸基序AC/GT(6.3%)和AG/CT(5.7%),而在马铃薯中单核苷酸基序C/G(6.0%)和三核苷酸基序AAT/ATT(4.5%)和AAG/CTT(3.7%)的出现频率分别排在第二、第三和第四。这表明,当只考虑I类SSRs时,番茄和马铃薯之间的主motif频率的差异也成立。
基因的内容
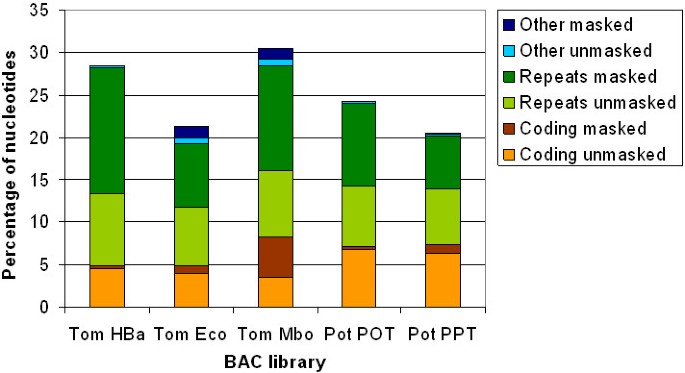
在番茄BESs中,与至少一个数据库序列匹配的核苷酸比例从Eco文库的21.3%到Mbo文库的30.5%不等。数字2根据材料和方法中描述的关键字过滤,将这些BLAST命中分为三个主要类别(“编码”、“重复”和“其他”)。然后,每个类别又被细分为“掩码的”和“不掩码的”子类别,“掩码的”表示重复序列与RepeatMasker识别的重复序列重叠,“不掩码的”表示没有这种重叠。通过这种方式,BLAST和RepeatMasker的结果被结合起来,以产生对BESs中推定的蛋白质编码核苷酸的百分比的最佳估计。“编码”类别表示匹配一个或多个数据库序列的核苷酸的百分比,并且不被关键字过滤识别为重复的。在去除与RepeatMasker识别的重复序列的重叠后,三个库中编码核苷酸的百分比从Mbo库的3.5%到HBa库的4.6%(图中“编码未屏蔽”类别)2).Mbo文库在“编码掩码”类别的三个文库中所占比例最高,这可能是该文库中大量核糖体重复序列逃脱了关键字过滤的结果。“重复”类别包含转座子和其他重复相关序列的BLAST匹配。在所有三个库中,有相当一部分的核苷酸被关键字过滤分配到“重复”类别,但与RepeatMasker识别的重复没有重叠(即“重复未掩盖”类别)。这一比例从Eco文库中的6.9%到HBa文库中的8.4%不等,可能代表RepeatMasker遗漏的重复序列和关键字过滤遗漏的真正蛋白质编码基因的组合。图中的最后一个类别2, 'other'表示通过关键字过滤识别的所有与转座子相关的重复序列(来自附加文件的除“转座子术语”以外的所有关键字术语)1).
在马铃薯POT和PPT文库中,分别有24.3和20.5%的核苷酸与蛋白库匹配。虽然这些数字略低于番茄HBa库和Eco库(分别为28.5%和21.3%),但“编码”类核苷酸的比例(6.8和6.3%)高于相应的番茄库(4.6和3.9%),这表明马铃薯可能比番茄有更大的基因库。此外,在与蛋白质数据库的比较中发现,番茄的转座子区域和其他重复相关区域的数量是马铃薯的1.5倍以上。这与重复分析中发现的转座子含量的差异是一致的。
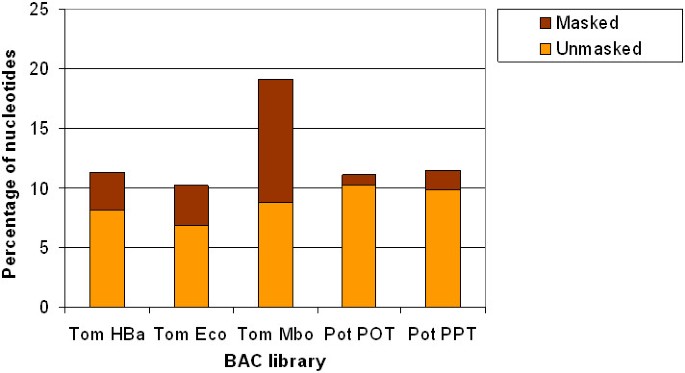
数字3.显示了BESs与特定物种EST数据库的BLASTN比较结果。比赛分为两类,“蒙面”和“不蒙面”。“被屏蔽”类别包含在EST数据库中有匹配的核苷酸,但在RepeatMasker分析中发现是重复的;“未掩饰”类别包含与重复序列不重叠的核苷酸。在番茄文库中,10.2 - 19.1%的核苷酸与一个或多个番茄EST序列匹配。Mbo库的EST覆盖率最高(19.1%),但超过一半的匹配(10.3%)被“掩盖”了。在Eco文库中,“未掩饰”类核苷酸的比例从6.8%到Mbo文库中的8.8%不等。
对于马铃薯BESs, 11.1% (POT)和11.5% (PPT)的核苷酸在马铃薯EST数据库中有匹配,这与番茄HBa和Eco与番茄数据库的比较吻合较好(分别为11.3和10.2%;参见图3.).与番茄相比,马铃薯BESs中被“掩盖”的匹配较少,这证实了BLASTX与蛋白质数据库比较的观察结果,即马铃薯BESs具有更多的蛋白质编码核苷酸和较低的重复含量。
功能注释
总共有30335个GO术语,其中585个惟一术语被分配给基于pfaam数据库中的匹配的番茄HBa BESs(参见附加文件)2,3.,4,5查看番茄和马铃薯贝叶斯中所有GO项及其对应频率的概述)。虽然Eco BESs的数量是HBa BESs的一半以上,但只有7647个GO术语(403个惟一术语)分配给它们。在potato中,分配给POT库的术语有17060个(544个惟一术语),而分配给PPT库的术语只有9312个(419个惟一术语)。比较番茄的GO注释和马铃薯的GO注释(用相同的限制性内切酶生成的库),结果发现在印地语消化的库中有18个显著过度表达的术语(在番茄HBa中有7个,在马铃薯POT中有11个;P值可在附加文件中找到3.)和9个在EcoRI消化库中显著过代表的术语(在番茄Eco中有7个,在土豆PPT中有2个;P值可在附加文件中找到2).
在这两个物种中,与EcoRI对应的词库相比,许多在indiii文库中被过多代表的词都与逆转录转座子活性有关,如DNA结合(GO:0003677)、DNA整合(GO:0015074)、rna定向DNA聚合酶活性(GO:0005634)和染色质相关术语(GO:0000785, GO:0003682, GO:0006333)。此外,与马铃薯相比,许多转座子相关术语在番茄中显著过代表(P值< 10)4;个别P值可在附加文件中找到2而且3.).这与上面讨论的RepeatMasker和BLAST分析的结果一致。令人惊讶的是,一些在两个EcoRI消化文库中过度表达的术语可能与转录因子基因有关。在番茄中,锌离子结合(GO:0008270)、dna依赖的转录调控(GO:0006355)和转录因子活性(GO:0003700)在Eco文库中都有过高的表达。对马铃薯PPT文库进行锌离子结合(GO:0008270)、核酸结合(GO:0003676)、转录因子活性(GO:0003700)富集。
对PANTHER所识别的蛋白质家族的分析揭示了相似的匹配数量趋势,无论是在番茄和土豆库内部还是在两者之间(见附加文件)6,7,8,9查看番茄和马铃薯贝叶斯中所有PANTHER项及其对应频率的概述)。在番茄中,在HBa BESs中发现了1064个不同的家庭,共28,984个命中,在Eco BESs中发现了8226个命中,代表654个家庭。对土豆POT库的分析揭示了951个不同的PANTHER家族,总共有13821个命中;然而,在PPT BESs中,716个家庭只有6926个点击量。在番茄HBa和Eco库中发现了2个和3个PANTHER家族的过度代表,而在马铃薯POT和PPT库中分别发现了11个和5个过度代表家族。
与番茄和马铃薯的HindIII文库中吉普赛反转录转座子更丰富的情况一致,GAG/POL/ENV多蛋白(PTHR10178) PANTHER家族在HindIII文库中都被发现有过多的代表,与相应的EcoRI文库相比。此外,ggag - pol相关的逆转录转座子(PTHR11439) PANTHER家族在EcoRI文库中相对更丰富,这也与hindii和EcoRI文库中Gypsy:Copia比例的差异一致(另见表)2).这两种逆转录转座子相关术语均显著(P值< 10)4;个别P值可在附加文件中找到6而且7)在番茄中所占比例高于土豆。在番茄Eco文库中,锌指CCHC结构域包含蛋白(PTHR23002)、锌指蛋白(PTHR11389)和MADS盒蛋白(PTHR11945)等转录因子相关术语显著过代表(P值为4.0*10)-13年7.8 * 107和1.5 * 106,分别验证了GO分析的结果。在马铃薯PPT文库中,没有转录因子相关的PANTHER家族显著过代表。
在番茄和马铃薯之间,马铃薯中过度表达的术语大部分对应重要的生物和生化过程。例如,含锌指CCHC结构域的蛋白(PTHR23002)和一般转录因子2相关的锌指蛋白(PTHR11697)的表达具有显著性(P值2.2*10)-16年(2)马铃薯POT的频率高于番茄HBa;后者在土豆PPT库中也有过多的代表。这也反映在GO注释中,如核酸结合(GO:0003676)和锌离子结合(GO:0008270)。这些术语相对于番茄的过度表达表明了马铃薯基因组中DNA结合蛋白的转录因子或其他基因的扩张。
另一个例子是细胞色素P450超家族(PTHR19383),它也通过铁离子结合(GO:0005506)和单加氧酶活性(GO:0004497)等术语在GO分析中发现。细胞色素P450蛋白在次生代谢物的生物合成中发挥着重要作用,这些蛋白在马铃薯中的过度表达可能表明在马铃薯中合成次生代谢物的途径网络是扩大的。
最后一个例子涉及到植物型丝氨酸-苏氨酸蛋白激酶(PTHR23258)的大家族,这种蛋白激酶已知在各种植物的抗病性中发挥重要作用(例如,番茄中的Pto基因[14])。在PANTHER数据库中,这个家族由104个不同的亚家族组成,其中71个亚家族是在番茄和马铃薯的BESs中发现的。在这71个亚科中,有15个只存在于番茄中,5个是马铃薯所特有的。在这两个物种中发现的大部分亚家族在马铃薯中都有过多的表达,如LRR受体样激酶(PTHR23258:SF462)和LRR跨膜激酶(PTHR23258:SF474)。几个亚家族在番茄中出现频率较高,包括丝氨酸/苏氨酸特异性受体样蛋白激酶(PTHR23258:SF416)和pto样激酶(PTHR23258:SF418)。因此,虽然马铃薯中丝氨酸-苏氨酸蛋白激酶的补体超过了番茄,但有几个亚家族在番茄中有所扩展。这可能反映了对不同病原体的抗性的适应,或者这些物种之间在病原抗性的主导机制上的差异。
比较基因组的映射
在135,842对番茄BESs中,他们与答:芥基因组,15283对有一个或多个匹配。这些匹配被分为五类,如表的最后五列所示3..“单端”类别表示BAC端对,其中两个序列中只有一个与答:芥基因组,并包含大部分的匹配(10,191)。配对结束匹配,即来自相同BAC的BESs与不同的染色体相匹配,被归为“非线性”类别。“有间隙”的类别包含4836个与之匹配的BAC端对答:芥配对配对之间的距离小于50kb或大于500kb的染色体。最后两类代表的BACs,其两端序列与基因组之间的距离在50到500 kb之间,要么是彼此的正确方向(“共线”),要么是彼此的重新排列(“重新排列”)。在4840对命中相同的番茄BES中答:芥染色体中,有三对属于“共线”类,有一对属于“重排”类,这表明在番茄和番茄之间存在四个假定的微共线区域答:芥.
马铃薯有55662对BESs,其中117对被映射到答:芥这对染色体的两个BESs都匹配相同的染色体。基于末端序列匹配,两个马铃薯BACs显示出假定的微共线性,其中一个是共线的,而另一个代表了可能的重排。与番茄相比,土豆很少有属于“gap”类别的BACs,尽管较小的PPT库在这一类别的序列是POT库的5倍多。有趣的是,大部分属于这一类的番茄BACs来自Eco和Mbo库(分别为1,279和3,507)。在RepeatMasker分析中,EcoRI和MboI消化文库中含有很高比例的核糖体RNA基因,事实上,这些文库中属于“缺口”类别的序列中有80%以上包含核糖体RNA基因。
重复同样的分析p . trichocarpa基因组中,只有708对番茄BES对的两端与同一染色体匹配(表中后三列之和)4).这里应该指出的是p . trichocarpa两者的染色体数量都比答:芥(19对5)和大约22000个尚未整合到染色体伪分子中的附加contig序列。仅根据这些数字,我们可以预期成对的BESs映射到同一染色体或相邻序列的数量会更少。即便如此,p . trichocarpa与番茄相比,显示出更多的微共线区域答:芥: 73对BES,在距离另一对BES 50至500 kb的距离内映射。超过三分之二的匹配(51个,表中的“共线”类别4)表明番茄与p . trichocarpa,而其余22个命中则代表各自微共线区域的重排。
与番茄的区别一致——答:芥和番茄p . trichocarpa在图谱中,有少量的马铃薯BES对(75对)可以被映射到同一染色体上p . trichocarpa,比答:芥.其中41个潜在微共线区域,其中24个共线区域。与番茄相比,马铃薯的“非线性”和较小程度上的“间隙”类别代表不足。同样,这些差异似乎源于Eco和Mbo文库中的许多BESs包含核糖体RNA基因。这些序列中的大多数都属于“非线性”的范畴p . trichocarpa比较,而不是“有差距”的类别答:芥,因为核糖体RNA基因包含在一些未组装的连续序列中,而不是在染色体假分子中。
讨论
序列的属性
基于番茄和马铃薯的文库的差异,这些部分消化的文库似乎不太可能代表基因组的无偏截面。例如,在番茄中,Mbo库比HBa库和Eco库具有更高的GC百分比。这种差异可能是由基因组消化中靶向的酶切位点的长度和GC含量引起的:indiii和EcoRI位点(分别为AAGCTT和GAATTC)的长度为6个核苷酸,GC含量为33.3%,而MboI位点(GATC)的长度为4个核苷酸,GC含量为50%。这一结果在本文提出的基因和重复含量分析的结果中很明显:结果在使用不同酶的文库中有显著差异。然而,我们认为合理的假设是,番茄和马铃薯的文库从相同的限制性内切酶的消化会有相似的序列偏差。利用这一假设,我们通过保持来自不同库的BESs的逻辑分离,只直接比较由相同限制性内切酶构建的库的BESs数据,努力将序列偏差对结果的任何影响降到最低。
番茄的贝叶斯(特别是Mbo贝叶斯)平均比土豆的贝叶斯短。番茄hindii和EcoRI文库与马铃薯文库的平均序列长度差异约为60 nt,这很可能是测序质量和设备差异的结果。然而,我们认为合理的假设是,在这个尺度上序列长度的差异不会影响本研究中所执行的基于相似性的分析的结果。
重复密度和分类
番茄和土豆库在总重复内容和重复类型之间的比例上都有所不同。例如,相对于番茄HBa库和马铃薯POT库,核糖体DNA序列分别在番茄Mbo和Eco以及马铃薯PPT库中被过度表示。这一现象在一项研究中也被观察到玉米贝丝(15],在那里它被归因于许多MboI网站的存在z梅斯核糖体DNA簇,相比之下只有一个EcoRI位点,没有hindii位点。通过类似的推理,在Eco和PPT库中吉普赛反转录转座子的表达不足可能是由于该元素中EcoRI位点的频率低于hindii和MboI位点。
由RepeatMasker识别的重复之间的差异(表2)和BLASTX(图2)表示需要番茄和马铃薯特定的重复数据库。以前已经从番茄BESs生成了一个重复数据库(L. Mueller,未发表的数据),但是使用RepeatMasker将番茄BESs与该数据库进行比较,结果大约60%的番茄BESs被注释为重复的(数据未显示)。然而,这些重复的大多数不能被分配到一个已知的重复家族。因此,尽管本文的研究结果可能低估了番茄和马铃薯BESs的实际重复含量,但RepeatMasker和BLASTX分析的结果都明确表明,番茄BESs的重复含量高于马铃薯BESs。
基因组大小和逆转录转座子含量之间的相关性先前已经在十字花科[16].在那里,人们发现逆转录转座子的含量随着基因组大小的增加而增加,从大约7%到10%答:芥(基因组大小125 Mb),到14%芸苔属植物拉伯(基因组大小530 Mb),到20%b . olacerea(基因组大小700 Mb)。与谷类作物相比,比如栽培稻(基因组大小430 Mb, 35%的逆转录转座子[17),z梅斯(基因组大小2365 Mb, 56%的逆转录转座子[15])表明,谷物中的实际逆转录转座子含量高于十字花科,与基因组大小的相关性可能在植物中普遍存在。本研究提供的数据表明,基因组扩展在茄科还与逆转录转座子扩增有关;马铃薯(基因组大小840 Mb)的逆转录转座子含量估计在8.2 (PPT)和11.4% (POT)之间,而番茄(基因组大小950 Mb)的逆转录转座子含量明显较高(Eco文库9.3%,HBa文库17.0%)。
番茄BESs中Gypsy和Copia逆转录转座子序列的比例在1:1到2:1之间,而马铃薯BESs中这一比例在2:1到3:1之间。虽然在不同的酶切库中,这个比例在每个物种中都有明显的差异,但在hindii和EcoRI酶切库中,番茄和马铃薯之间的比例差异都可以观察到(见表)2).在答:芥[18],b·拉伯[16],番木瓜[19),z梅斯[15,这个比例大约是1:1。番茄和马铃薯的基因组似乎更类似于o .漂白亚麻纤维卷吉普赛人与科比亚人的比例约为2:1 [17].番茄和马铃薯的Gypsy:Copia比例的差异表明,与番茄基因组扩增相关的逆转录转座子扩增主要是额外的Copia拷贝的结果。
简单序列重复
在番茄和马铃薯的所有大小类别中,最丰富的SSRs都是at丰富的。这与其他植物物种的发现是一致的答:芥[20.],b·拉伯[16],c .木瓜[19],大豆[21),而穆萨acuminata[22].在马铃薯和番茄中,五核苷酸重复序列是最常见的SSRs形式,而AAAAT是主要的重复基序。这与之前研究的植物物种形成鲜明对比,在植物物种中二核苷酸和五核苷酸重复通常发生频率最低[23].在许多植物物种中,如答:芥,b·拉伯[16),而o .漂白亚麻纤维卷[24,25,三核苷酸重复序列是最丰富的微卫星。然而,BES分析c .木瓜[19],g·马克斯[21),m . acuminata[22表明双核苷酸重复序列在这些植物物种中更为常见。因此,与其他研究的植物物种相比,番茄和马铃薯都显示出独特的微卫星频率分布。
与马铃薯的BESs相比,番茄的BESs具有更高的二核苷酸和四核苷酸重复的比例。这可能是因为番茄BAC端文库中有一个或多个来自番茄基因组中中心点区域的BACs被富集,因为这些区域以前被发现长时间富集I类二核苷酸和四核苷酸重复[26].然而,在所有三个番茄文库中,番茄中二核苷酸和四核苷酸重复序列的相对富集量均高于马铃薯;只有在中心区含有比番茄基因组平均水平更多的indiii、EcoRI和MboI位点时,这才与中心区富集的假设相一致。
基因的内容
经过重复屏蔽和关键字过滤,马铃薯POT和PPT BESs中与非冗余蛋白数据库匹配的核苷酸比例分别是番茄HBa和Eco BESs的1.5- 1.6倍。经过重复屏蔽和关键词过滤后,在蛋白质数据库中命中核苷酸的百分比和BESs的数量在马铃薯中都较高(POT库中为13.8%;PPT库12.9%)高于番茄(HBa库8.7%);在Eco库中为7.9%),支持了马铃薯的蛋白质编码区域比番茄更多的假设。在BESs与EST的BLASTN比较中,我们观察到土豆和番茄之间存在类似的差异,土豆的EST覆盖率比番茄高1.3- 1.4倍。此外,番茄EST与马铃薯EST的交叉比较证实了番茄EST覆盖范围的差异不是由番茄和马铃薯EST集合之间唯一转录本数量的差异引起的(数据未显示)。BLAST与蛋白质数据库和转录数据库之间的差异可能是由于在番茄转录数据中存在全长cDNA序列,而在马铃薯数据中不存在全长cDNA序列,导致编码序列内部区域在番茄BESs中过度表示。即使假设这个较保守的下限是正确的,结果仍然表明马铃薯比番茄有更多的基因库,因为番茄基因组大约只有土豆基因组的1.1倍。
在番茄和马铃薯中,与蛋白质数据库相比,与EST数据库相似的核苷酸比例更小,而与EST数据库比较的非重复编码序列的百分比(图中“未掩盖的”类别)3.)高于蛋白质数据库比较(图中“编码未掩码”类别)2).令人惊讶的是,大多数与蛋白质和转录数据库的匹配并不重叠。例如,在番茄HBa库中,分别有8.1%和4.6%的核苷酸在EST和蛋白质数据库中有匹配,而只有1.6%的核苷酸在两者中都有匹配。类似地,对于马铃薯POT库,只有2.5%的核苷酸在转录本和蛋白质序列中都有匹配,而在这些数据库中有匹配的核苷酸的单个百分比分别为10.2%和6.8%。一方面,与蛋白质数据库没有重叠的EST数据库的匹配可能代表了在非冗余蛋白质数据库中没有表示的独特的、分类单元或物种特异性的蛋白质编码基因,或者这些基因组中转录但未翻译的区域。另一方面,如果蛋白质数据库的匹配与EST数据库中的匹配不重叠,则可能表明在EST文库构建过程中采样的条件下,组织中存在未充分表达的基因,或者蛋白质数据库中存在注释错误或其他不正确的序列。
EST数据可能为这些基因组中真正的蛋白质编码区域提供了最可靠的采样,因为它是基于实验数据,其中包含了蛋白质数据库中无法提供的物种特异性序列。由于EST文库构建中通常使用的是poly-A尾的选择,因此非蛋白编码转录本的数量相对较少。从HBa和Eco库中提取与ESTs匹配且不与重复序列重叠的核苷酸作为编码序列的度量,番茄基因组(950 Mb)估计包含64.8到77.1 Mb的编码区。同样,假设基因组大小为840 Mb,马铃薯的编码区总长度将在82.5 - 85.4 Mb之间。这些数字为这些基因组的估计编码内容设置了下界,因为EST数据不太可能代表这些基因组中全长蛋白质编码序列的完整补充。
根据对27,274个UniGenes和6个BAC序列的分析,以前估计番茄的基因含量为35,000个基因[27].如果这35000个基因确实由71.0 Mb的编码序列表示(HBa和Eco文库估计的平均值),那么番茄的平均转录本长度约为2.0 kb。这比平均文本长度要长答:芥,根据TAIR7基因组注释为1.2 kb [28].假设平均转录本长度相同,马铃薯(84.0 Mb的编码序列,在两个文库中平均)将包含大约41400个基因,或比番茄多6400个基因。由于这里提供的数据仅基于短基因组序列的相似性搜索,这种差异不一定代表功能基因的差异,但也可能反映了马铃薯中更大比例的假基因或其他非功能等位基因。
功能注释
GO和PANTHER分析的结果普遍显示出类似的趋势。番茄贝叶斯比马铃薯贝叶斯有更多的GO术语和PANTHER家族。然而,土豆BESs有大量与之相关的唯一术语。这与BLASTX与非冗余蛋白数据库的比较结果一致,在该数据库中发现番茄BESs具有更高的BLAST hit的总体覆盖率,但推定蛋白编码区域的百分比较低(另见图)2).
在GO项和PANTHER族分析中,大多数项都以相对较低的频率出现。例如,在番茄HBa和马铃薯POT的比较中,分配给BESs的730个不同的GO术语中,只有131个在至少一个物种中出现了10次或更多次。这组131个GO术语包含所有18个显著性术语(P值< 104)在这一比较中的一个物种中过度存在。此外,在这131个术语中,有39个在至少一个物种中被发现至少50次,这一亚群包含了18个显著过度代表的术语中的16个。同样,在PANTHER家族分析中,在BESs中发现的1352个不同的家族中,有119个在至少一个物种中至少出现了10次,其中12个家族至少出现了50次。至少发现10次的119个科包含了13个科中的每一个,这些科在某一物种中显著过度代表;其中有10个在至少一个物种中被计数超过50次。虽然这里只显示了番茄HBa和土豆POT的比较,但其他比较显示了类似的模式,表明许多高度丰富的GO术语和PANTHER家族在番茄或土豆中显著过度代表。这些被过度代表的术语和科在马铃薯中最为丰富,代表着生物学上重要的功能和过程。在番茄中,较少的术语和科被过度代表;这些主要与基因组结构特征有关,如逆转录转座子。
转座子相关GO术语和PANTHER家族在番茄中的过代表与重复分析的结果一致,证实了番茄比马铃薯富含反转录转座子的观察结果。而在PANTHER分析中,反转录酶(PTHR19446)在马铃薯中显著过代表。乍一看,这与番茄中rna导向的DNA聚合酶活性(GO:0003964)和rna依赖的DNA复制(GO:0006278)的过度表现并不一致。然而,在番茄和马铃薯中,绝大多数的逆转录酶来源于非ltr逆转录元件(PTHR19446:SF34),这实际上与RepeatMasker分析中发现的马铃薯中非ltr逆转录转座子的较高频率一致(另见表)2).
细胞色素P450单加氧酶是植物中一个庞大的基因超家族,通常与次生代谢产物的生物合成有关。在答:芥,至少发现272个P450基因,约占该物种基因补体的1%。在o .漂白亚麻纤维卷,这个家族甚至更大,到目前为止发现了458个P450基因[29].在这些基因组中,并非所有的p450都代表真正的蛋白质编码序列;在答:芥在美国,90%的基因是真正的蛋白质编码,而在美国,这一比例为72%o .漂白亚麻纤维卷.共鉴定出66个不同的P450基因家族答:芥而且o .漂白亚麻纤维卷,其中一些基因在这两种物种中都有过高的比例。此外,有些科在一个物种中存在,而在另一个物种中完全没有[30.].在印度ii和EcoRI文库中,分别在番茄和马铃薯中发现186和209个与细胞色素P450 PANTHER家族(PTHR19383)相关的BESs。由于这些BAC端序列约占其各自基因组的14%和10%,这些数据表明P450基因在茄科.这可能是特定P450家族扩大的结果,但也可能是物种或家族特定P450进化的结果。例如,等位烯氧化物合酶目前只在茄科植物中发现,包括番茄和番茄佩妮inflata[31].与番茄相比,马铃薯中P450的过度表达可能是P450家族的另一个结果,但也可能表明这些物种之间共享的家族的扩大。
比较基因组的映射
本研究利用配对的BAC末端检测茄科植物番茄和马铃薯与模式植物有机体之间的微共微区域答:芥而且p . trichocarpa.使用类似的方法,已经观察到微同步性答:芥而且b·拉伯[16];c .木瓜而且p . trichocarpa[19];而且m . acuminata而且o .漂白亚麻纤维卷[22].
番茄和马铃薯的BACs含量较高,表现出微同步性p . trichocarpa,比答:芥.番茄/马铃薯和答:芥不可能是这些物种之间进化距离的差异。这两个答:芥而且p . trichocarpa是蔷薇科植物分支的一部分,而西红柿和马铃薯属于小行星植物分支。这可能是最近复制的结果答:芥基因组,然后是大约70%的复制基因的丢失[32].假设这种丢失是随机发生的,那么在重复之前存在的绝大多数可能的微同步区域将会由于与这种重复和随后的丢失相关的主要局部扩张和收缩而消失。这一假设被观察到只有大约1%的12000答:芥BES对以前可以在300 kb内映射到p . trichocarpa基因组,表明这些基因组的组织确实有很大的不同[19].
之前已经在番茄/马铃薯和答:芥.番茄7号染色体上含有5个基因的57 kb区域与30 kb区域共线答:芥1号染色体,尽管基因的顺序和方向暗示了两次反转事件[33].在另一项研究中,一个105 kb的BAC序列与四个不同的片段相匹配答:芥染色体2、3、4和5;然而,这四个地区中的每一个答:芥比番茄矮34].近年来,在马铃薯第5染色体上的一个耐晚疫病和根囊线虫QTL的区域之间检测到5个微共链块答:芥染色体1、3及5号[35].这些共音块跨越3到7个orf,并被非共音块打断。在这些例子中,番茄/土豆和答:芥涉及到更短的区域答:芥基因组比番茄和马铃薯的平均BAC序列长。此外,编码序列之间经常检测到(微)同步区域,而在番茄和马铃薯的BESs中编码序列的比例相对较低(< 10%),这为本文观察到的这些物种之间微同步量减少提供了一个很好的解释。
potato和答:芥也已经在全基因组水平上通过比较制图方法被识别出来。这揭示了马铃薯和马铃薯之间90个假定的共筋块答:芥覆盖了41%的马铃薯基因图谱,还有50%的答:芥物理图谱(36].这些共链块在马铃薯遗传图谱上分布不均匀,并且在基因图谱上的区域数量上是多余的答:芥在马铃薯图谱上大部分区域显示同向的基因组。番茄/马铃薯和答:芥由于BAC两端之间的距离相对较短,本研究中描述的基于be的方法所发现的不能用于确认或放弃任何假定的高阶共tenic区域。
六个配对的番茄BAC末端匹配簇在16.0 - 20.2 Mb的间隔内p . trichocarpa10号染色体。此外,7对马铃薯BESs映射到15.2 - 19.4 Mb之间的部分重叠区间,这表明在番茄/马铃薯之间存在许多不同的微同步区域,或者可能存在一个单一的大同步区域p . trichocarpa基因组。这些发现为该地区的这些物种之间的详细比较提供了一个有趣的起点,一旦更多的番茄和马铃薯基因组序列获得。
结论
本文对番茄和马铃薯的BESs进行了大规模的分析,揭示了这些亲缘关系密切的物种之间在结构和功能上的许多有趣的差异。我们已经证明,番茄基因组不仅比马铃薯基因组的重复性更强,而且这些基因组的重复组成也不同,最重要的是吉普赛(Gypsy)和科比亚(Copia)逆转录转座子的分布。与其他研究的植物基因组相比,我们发现番茄和马铃薯基因组包含大量motif长度为5的SSRs,这可能是茄科基因组的一个独特特征。
比较分析这些BESs中假定的蛋白质编码区域,发现这些区域在马铃薯基因组中富集。此外,在马铃薯中发现了几个蛋白家族的比例高于番茄,如细胞色素P450单加氧酶和丝氨酸苏氨酸蛋白激酶。相比之下,P450超家族在两个物种中都有显著的扩大答:芥这表明在大脑中有一个扩展的次生代谢途径网络茄科.
番茄和马铃薯似乎都具有较低的微共质性答:芥这可能是该物种相对较近的基因组重组的结果。观察到较高程度的同向性p . trichocarpa.进化距离的差异不太可能是微同步性增加的原因,因为两者都有答:芥而且p . trichocarpa是蔷薇科植物分支的一部分,而西红柿和马铃薯属于小行星植物分支。
综上所述,这些发现首次揭示了茄科植物基因组的进化过程,无论是在茄科植物内部还是相对于其他植物物种而言。当这些物种的完整基因组序列可用时,全基因组比较和蛋白质或重复家族特异性研究可能会对本文中有趣的观察结果提供更多的解释。
方法
BAC结束序列
从HBa (indiii), Eco (EcoRI)和Mbo (MboI)库中获得的番茄BESs从SGN FTP站点[13].对于所有的分析,使用'screened_and_trim '集(bacends_combined_screened_and_trim .v4.seq),其中低质量的区域和向量序列被修剪,小于150nt的序列被删除。此外,该文件排除了与线粒体基因组匹配的BESs拟南芥[28的叶绿体基因组n .烟草(NCBI GenBank登录NC_001879.2),基于e值截止为10的BLASTN搜索-10.马铃薯BESs经过了质量和矢量裁剪,从NCBI GenBank的GSS部分下载[37使用“RHPOKEY”查询。所有小于150nt的序列和带有BLASTN的序列(blastall 2.2.15) [38打到…答:芥线粒体或n .烟草e值小于10的叶绿体基因组-10为了与番茄的数据保持一致,都被删除了。最近,番茄和马铃薯的叶绿体基因组已经获得;然而,可以假设答:芥线粒体基因组与这些基因组足够相似,因此认为没有必要进行这种额外的过滤[39,40].
重复密度和分类
通过相似度搜索,在番茄和马铃薯的BESs中发现重复MagnoliaphytaRepBase重复数据库的部分(发布2006-10-06)[41,使用RepeatMasker 3.1.5 [42和cross_match 0.990319 [43].然后,通过对重复数据库有一次或多次命中的BESs中核苷酸的百分比计算重复密度。重复族的分类来源于RepBase数据库中的注释。使用BLASTN (blastall 2.2.15)检测BESs中的冗余,方法是将番茄和马铃薯的BES数据与自身进行比较,并删除与自身的所有匹配序列。e值截止值设置为105和BLAST命中如果没有至少覆盖50%的查询序列和90%的标识,就会被删除。
简单序列重复
微型卫星是用Sputnik软件的改良版本探测到的[44].运行参数设置为返回所有跨越至少15个核苷酸的SSRs, motif长度在1到5之间(即单核苷酸、二核苷酸、三核苷酸、四核苷酸和五核苷酸重复),最小得分为8分。用这种方法确定的微型卫星被分为两类;第一类,有10个或更多的主题重复;第II类,母题重复次数少于10次[21].
基因的内容
通过BLAST检索(BLAST search)方法估计BESs的基因含量。将BESs与NCBI GenBank非冗余蛋白数据库进行比较(发布2007-02-16)[45,并以Kazusa KTU2番茄EST数据库[46]和CAB PotatEST马铃薯EST数据库(2007年1月发布)[6使用BLASTN]。所有BLAST搜索的e值截止为105,并对最佳的5个命中值进行评估。此外,BLASTN对转录本数据库的搜索使用了90%的身份截断。
为了区分真实的、假定的蛋白质编码区域和转座子或污染相关区域,基于BLAST命中描述中的关键字匹配过滤与非冗余蛋白质数据库的BLAST匹配。在附加文件中可以找到用于过滤BLAST结果的关键字的概述1.总的来说,这些关键词描述了与细菌污染、转座子相关、叶绿体、线粒体和核糖体蛋白序列相似的序列。任何没有被任何关键字过滤的BLAST匹配都被认为代表一个非重复的蛋白质编码区域。
功能注释
通过与Pfam(版本21.0)的比较,对番茄HBa和Eco,土豆POT和PPT BESs进行了功能注释[47]和PANTHER(6.1版本)[48蛋白质家族数据库,使用InterProScan 4.3.1 [49].从InterProScan的合并输出文件中提取了Pfam注释中的GO术语,以及PANTHER注释中的PANTHER族(但不是子族)标识符。对于每个GO项和PANTHER族,统计匹配的番茄和土豆BESs的数量;如果一个GO项或PANTHER族被多次分配到同一序列,例如由于同一序列中有多个开放读帧,它只被计数一次。
随后,使用R软件套件中的双面Fisher确切检验对计数进行了两两比较。50].请注意,GO术语注释并不总是彼此独立分配(正如Fisher精确检验所要求的那样),这意味着一些术语经常或专门出现在一起,因为它们都描述了单个生物过程或功能的不同方面。然而,为了简单起见,GO项之间的这些高阶依赖性被忽略了,这可能导致对不同的过度表示项的数量的过高估计。此外,为了减少不同限制性内切酶的文库之间的偏差所造成的误差,直接的种间比较只在使用相同限制性内切酶的文库的BESs之间进行。最后,这里的零假设是,番茄和土豆的BESs之间的GO项或PANTHER族的丰度没有差异,而备择假设表明有差异。保守的P值截断值为104选择显著差异与非显著差异进行区分。
比较基因组的映射
为了确定研究的茄科植物和双子叶模式植物之间潜在的微共线区域,选择了配对的BESs并将其映射到答:芥而且杨树trichocarpa基因组序列与BLASTN对齐。135,842个番茄BACs (63,169 HBa, 33,498 Eco和39,175 Mbo)和55,662个马铃薯BACs (34,362 POT和21,300 PPT)的配对末端序列可用。全基因组序列答:芥而且p . trichocarpa已从TAIR下载[28]和JGI [51),分别。的p . thrichocarpa本研究中使用的基因组序列尚未完成,而是由19条染色体中的每条的假分子序列加上21993个contig序列中的额外177,7 Mb组成。
对于每个BES,只有与各自基因组序列e值小于10的最佳匹配5如果后续HSPs之间的距离大于2000 nt,则拒绝命中。如果BAC两端的映射距离在50 - 500 kb之间,则认为BAC与目标基因组具有微同步性。当两端相对于对方方向正确时,该区域被认为是共线的;否则,该区域被认为在两个物种之间重新排列。
缩写
- BAC:
-
=细菌人工染色体
- 贝斯:
-
= BAC结束顺序
- 环保:
-
=番茄EcoRI消化BAC库
- 美国东部时间:
-
=表达序列标签
- 走:
-
=基因本体
- HBa:
-
=番茄HindIII消化BAC库
- HSP:
-
=高分段对
- kb:
-
=个碱基
- m:
-
= Megabases
- Mbo:
-
=番茄MboI消化BAC库
- nt:
-
=核苷酸
- 锅:
-
印度ii消化BAC库
- PPT:
-
=马铃薯EcoRI消化BAC库
- 苏维埃社会主义共和国:
-
=简单序列重复。
参考文献
Tanksley SD, Ganal MW, Prince JP, de Vicente MC, Bonierbale MW, Broun P, Fulton TM, Giovannoni JJ, Grandillo S, Martin GB, Messeguer R, Miller JC, Miller L, Paterson AH, Pineda O, Röder MS, Wing RA, Wu W, Young ND:番茄和马铃薯基因组的高密度分子连锁图谱。中国生物医学工程学报,2011,29(4):381 - 381。
D'Agostino N, avversano M, Frusciante L, Chiusano ML:番茄EST数据库:利用EST数据探索番茄物种的表达模式。核酸学报2007,35(数据库问题):D901-D905。10.1093 / nar / gkl921。
矢野K,渡边M,山本N,津根T,青木K,樱井N,柴田D: MiBASE:微型番茄品种微番茄数据库。植物生物技术,2006,23:195-198。
Van Os H、Andrzejewski S、Bakker E、Barrena I、Bryan GJ、Caromel B、Ghareeb B、Isidore E、De Jong W、Van Koert P、Lefebvre V、Milbourne D、Ritter E、Rouppe Van der Voort JNAM、roussel - bourgeois F、Van Vliet J、Waugh R、Visser RGF、Bakker J、Van Eck HJ:构建马铃薯万标记超密集基因重组图谱:为加速基因分离和全基因组物理图谱提供框架。中国生物医学工程学报,2006,32(2):378 - 378。10.1534 / genetics.106.055871。
Wageningen UR植物育种CBSG马铃薯和番茄基因组数据库。[http://potatodbase.dpw.wau.nl/]
PotatEST DB。[http://biosrv.cab.unina.it/potatestdb/]
Bonierbale MW, Plaisted RL, Tanksley SD:基于公共克隆集的RFLP图谱揭示马铃薯和番茄染色体进化模式。中国生物医学工程学报,1998,25(4):359 - 359。
穆勒,Tanksley SD, Giovannoni JJ, Van Eck J,堆栈,崔D, Kim BD陈M,程Z,李C, H,雪Y,西摩G, G,主教布赖恩•G Sharma R, Khurana J, Tyagi,将D,辛格NK, Stiekema W, Lindhout P,杰西T,克莱因Lankhorst R, Bouzayen M,柴田D, Tabata年代,Granell, Botella妈,朱利亚诺克,Frusciante L,喀斯M, Zamir D:番茄测序项目,第一个项目国际茄科(SOL)的基石。比较与功能基因组学。2005,6(3):153-158。10.1002 / cfg.468。
Arumuganathan K, Earle ED:一些重要植物的核DNA含量。植物生物学杂志,1998,9(3):208-218。10.1007 / BF02672069。
Mueller LA, Solow TH, Taylor N, Skwarecki B, Buels R, Binns J, Lin C, Wright MH, Ahrens R, Wang Y, Herbst EV, Keyder ER, Menda N, Zamir D, Tanksley SD: SOL基因组网络:茄科及其他生物的比较资源。植物生理学报,2004,25(3):347 - 347。10.1104 / pp.105.060707。
马铃薯基因组测序联盟。[http://www.potatogenome.net/]
中国生物医学工程学报,2000,10(1):129-136。
索尔基因组学网络。[http://sgn.cornell.edu/]
Martin GB, Brommonschenkel SH, Chunwongse J, Frary A, Ganal MW, Spivey R, Wu T, Earle ED, Tanksley SD:基于图谱的番茄抗病蛋白激酶基因克隆。科学通报,2003,29(5):533 - 536。10.1126 / science.7902614。
Messing J, Bharti AK, Karlowski WM, Gundlach H, Kim HR, Yu Y, Wei F, Fuks G, Soderlund CA, Mayer KF, Wing RA:玉米序列组成与基因组组织。中国生物工程学报,2004,29(4):369 - 369。10.1073 / pnas.0406163101。
Hong CP, Plaha P, Koo DH, Yang TJ, Choi SR, Lee YK, Uhm T, Bang JW, Edwards D, Bancroft I, Park BS, Lee J, Lim YP: rapa基因组BAC-end序列分析及其与拟南芥的比较。分子细胞学报,2006,22(3):300-307。
国际水稻基因组测序计划:基于地图的水稻基因组序列。自然学报,2005,436(7052):793-800。10.1038 / nature03895。
拟南芥基因组计划:开花植物拟南芥基因组序列分析。自然学报,2000,408(6814):796-815。10.1038 / 35048692。
Lai CW, Yu Q, Hou S, Skelton RL, Jones MR, Lewis KL, Murray J, Eustice M, Guan P, Agbayani R, Moore PH, Ming R, Presting GG:木瓜BAC端序列分析首次揭示了果树基因组的组织。中国生物医学工程学报,2006,26(1):1-12。10.1007 / s00438 - 006 - 0122 - z。
Katti MV, Ranjekar PK, Gupta VS:简单序列重复序列在真核基因组序列中的差异分布。中华分子生物学杂志,2001,18(7):1161-1167。
Shultz JL, Kazi S, Bashir R, Afzal JA, Lightfoot DA:基于bac末端序列的大豆微卫星标记的开发及其在大豆物理和遗传图谱中的定位。中国生物医学工程学报,2007,29(6):369 - 369。10.1007 / s00122 - 007 - 0501 - 9。
张F, Town CD: Musa acuminata基因组的BAC端视图。植物生物学杂志,2007,7 (29):
Mun JH, Kim DJ, Choi HK, Gish J, Debellé F, Mudge J, Denny R, Endré G, Saurat O, Dudez AM, Kiss GB, Roe B, Young ND, Cook DR:截断紫花紫花(Medicago truncatula)基因组微卫星分布:遗传和物理图谱集成的遗传标记资源中国生物医学工程学报,2006,29(4):359 - 359。10.1534 / genetics.105.054791。
高夫SA,里奇D,局域网,普雷斯特G,王R,邓恩M, Glazebrook J,会话,Oeller P, Varma H,哈德利D,和记黄埔D,马丁·C片瞳F,兰格BM, Moughamer T,夏Y, Budworth P,钟J, Miguel T Paszkowski U,张年代,科尔伯特M,太阳王,陈L,库珀B,公园,木TC、毛L,鹌鹑P, R, R,院长于Y, Zharkikh,沈R, Sahasrabudhe年代,托马斯,罐头R, Gutin, Pruss D,里德J, Tavtigian年代,米切尔J·G,肖勒T,米勒RM,博年代,阿迪N, Rubano T,Tusneem N, Robinson R, Feldhaus J, Macalma T, Oliphant A, Briggs S:水稻(Oryza sativa L. ssp.)基因组序列草图。粳稻)。科学通报,2002,29(5):559 - 561。10.1126 / science.1068275。
籼稻)。科学通报,2002,29(5):559 - 561。10.1126 / science.1068037。
Areshchenkova T, Ganal MW:长番茄微卫星主要与中心粒区相关。中国生物技术学报,1999,42(3):536-544。10.1139 / gen - 42 - 3 - 536。
Van der Hoeven R, Ronning C, Giovannoni J, Martin G, Tanksley S:基于对大量表达的序列标签收集和选择性基因组测序的分析,推断番茄基因组中基因的数量、组织和进化。植物细胞学报,2002,14(7):1441-1456。10.1105 / tpc.010478。
TAIR。[http://www.arabidopsis.org/]
Schuler MA, Werck-Reichhart D: p450的功能基因组学。植物生态学报,2003,23(4):629-667。10.1146 / annurev.arplant.54.031902.134840。
Nelson DR, Schuler MA, Paquette SM, Werck-Reichhart D, Bak S:水稻和拟南芥的比较基因组学。单子花和双子花727个细胞色素P450基因和假基因的分析。植物生理学报,2004,25(2):366 - 366。10.1104 / pp.104.039826。
徐勇,石田华,李晓燕,陈晓燕,等。矮牵牛叶片衰老过程中液泡体定位细胞色素P450的表达。植物生物学杂志,2006,6 (8):
张文华,张文华,张文华,等:利用染色体复制事件的系统发育分析揭示被子植物的基因组进化。自然学报,2003,38(4):433-438。10.1038 / nature01521。
Rossberg M,’s K, Acarkan A, Herrero R, Schmitt T, Schumacher K, Schmitz G, Schmidt R:比较序列分析显示,番茄、拟南芥和卡普塞拉基因组的横向抑制区存在广泛的微共线性。植物细胞学报,2001,13(4):979-988。10.2307 / 3871354。
Ku HM, Vision T, Liu J, Tanksley SD:比较番茄和拟南芥基因组的序列片段:大规模复制后的选择性基因丢失创建了一个同构网络。中国生物工程学报,2000,29(6):791 - 791。10.1073 / pnas.160271297。
Ballvora A, Jöcker A, Viehöver P, Ishihara H, Paal J, Meksem K, Bruggmann R, Schoof H, Weisshaar B, Gebhardt C:马铃薯V染色体上抗病热点的茄属和拟南芥的比较序列分析揭示了保守和快速进化的基因组片段拼接。中国生物医学工程学报,2007,29 (6):773 - 773
Gebhardt C, Walkemeier B, Henselewski H, Barakat A, Delseny M, Stüber K:马铃薯(Solanum tuberosum)和拟南芥(Arabidopsis thaliana)的比较图谱揭示了马铃薯基因组中结构保守的结构域和古老的复制。植物学报,2003,34(4):529-541。10.1046 / j.1365 - 313 x.2003.01747.x。
索尔基因组学网络。[ftp://ftp.sgn.cornell.edu/]
NCBI dbGSS。[http://www.ncbi.nlm.nih.gov/dbGSS/]
Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ: gapping BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。核酸学报,1997,25:3389-3402。10.1093 / nar / 25.17.3389。
Chung HJ, Jung DJ, Park HW, Kim JH, Cha HW, Min SR, Jeong WJ, Liu JR:栽培马铃薯的叶绿体全基因组序列和与茄科植物的比较分析发现,栽培马铃薯叶绿体DNA序列存在241 bp的缺失。植物学报,2006,25(12):1369-1379。10.1007 / s00299 - 006 - 0196 - 4。
Daniell H, Lee SB, Grevich J, Saski C, Quesada-Vargas T, Guda C, Tomkins J, Jansen RK:球castanum, Solanum lycopersicum的叶绿体全基因组序列及其与茄科其他基因组的比较分析。中国生物医学工程学报,2006,29(4):359 - 359。10.1007 / s00122 - 006 - 0254 - x。
Jurka J, Kapitonov VV, Pavlicek A, Klonowski P, Kohany O, Walichiewicz J: Repbase Update,一个真核生物重复元素数据库。细胞基因学杂志,2005,10:462-467。10.1159 / 000084979。
RepeatMasker。[http://www.repeatmasker.org/]
绿色组。[http://www.phrap.org/]
小麦,大麦和水稻的EST-SSRs。[http://wheat.pw.usda.gov/ITMI/EST-SSR/LaRota/]
Micro-Tom数据库。[http://www.kazusa.or.jp/jsol/microtom/]
Finn RD, Mistry J, Schuster-Bockler B, griffith - jones S, Hollich V, Lassmann T, Moxon S, Marshall M, Khanna A, Durbin R, Eddy SR, Sonnhammer EL, Bateman A: Pfam:部落,网络工具和服务。核酸学报,2006,34(数据库问题):D247-D251。10.1093 / nar / gkj149。
Mi H, Guo N, Kejariwal A, Thomas PD: PANTHER版本6:扩展表达生物通路的蛋白质序列和功能进化数据。核酸学报2007,35(数据库问题):D247-D252。10.1093 / nar / gkl869。
穆德新泽西,Apweiler R, Attwood TK, Bairoch,贝特曼,宾斯D,博克P, V Buillard, Cerutti L,科普利R, Courcelle E, Das U,多尔蒂L, Dibley M,芬恩R,她W,高夫J,把手D, Hulo N,猎人年代,卡恩D, Kanapin, Kejariwal, Labarga, Langendijk-Genevaux PS,朗斯代尔D,洛佩兹R, Letunic我ㄧM,套装J, McAnulla C, McDowall J, Mistry J,米切尔,Nikolskaya,果园,Orengo C, Petryszak R, Selengut JD, Sigrist CJ,托马斯•PD Valentin F,威尔逊D,吴CH,叶芝C:InterPro数据库的新发展。核酸学报2007,35(数据库问题):D224-D228。10.1093 / nar / gkl841。
统计计算R项目。[http://www.r-project.org/]
联合基因组研究所。[ftp://ftp.jgi-psf.org/pub/JGI_data/Poplar/]
确认
ED得到了荷兰科学研究组织(NWO)的一笔赠款的支持。LM, RB和JG得到了国家科学基金会(NSF)的资助。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
ED构思了这项研究,进行了所有的计算分析并起草了手稿。LM, RB, JG负责番茄的BAC端测序,RV一起参与了计算分析的解释,并对手稿的最终草稿版本提供了反馈。WS和RvH参与了研究的设计和协调,并帮助起草了手稿。所有作者阅读并批准了最终稿件。
电子辅料
12870 _2007_242_moesm1_esm.doc
附加文件1:该文件描述了BLASTX对非冗余蛋白质数据库进行搜索后应用的关键字过滤,以区分真正假定的蛋白质编码区域和重复的和/或污染相关的序列。(DOC 32 KB)
12870 _2007_242_moesm2_esm.xls
附加文件2:该文件描述了在番茄和马铃薯EcoRI消化BAC末端序列的InterProScan分析中发现的基因本体术语。该表中的列描述了GO项,番茄Eco和土豆PPT库中分配了该项的BAC端序列个数,以及这两个库中该GO项相对丰度差的Fisher确切检验的P值。P值小于104表明这些库之间GO术语的丰度有显著差异。(XLS 57 KB)
12870 _2007_242_moesm3_esm.xls
附加文件3:该文件描述了在InterProScan对番茄和马铃薯HindIII消化BAC末端序列的分析中发现的基因本体术语。该表中的列描述了GO项,番茄HBa和马铃薯POT库中分配了该项的BAC端序列的数量,以及这两个库中该GO项相对丰度差的Fisher精确检验的P值。P值小于104表明这些库之间GO术语的丰度有显著差异。(XLS 72 KB)
12870 _2007_242_moesm4_esm.xls
附加文件4:该文件描述了在马铃薯hindii和EcoRI消化BAC末端序列的InterProScan分析中发现的基因本体术语。该表中的列描述了GO项、分配了该项的土豆POT和PPT库中BAC端序列的个数,以及这两个库中该GO项相对丰度差的Fisher确切检验的P值。P值小于104表明这些库之间GO术语的丰度有显著差异。(XLS 67 KB)
12870 _2007_242_moesm5_esm.xls
附加文件5:该文件描述了在番茄hindii和EcoRI消化BAC末端序列InterProScan分析中发现的基因本体术语。该表中的列描述了GO项,番茄HBa库和Eco库中分配了该项的BAC端序列的数量,以及这两个库中该GO项相对丰度差的Fisher确切检验的P值。P值小于104表明这些库之间GO术语的丰度有显著差异。(XLS 68 KB)
12870 _2007_242_moesm6_esm.xls
附加文件6:该文件描述了在番茄和马铃薯EcoRI消化BAC末端序列InterProScan分析中发现的PANTHER家族。该表中的列描述了PANTHER家族,番茄生态和土豆PPT库中分配了该术语的BAC端序列的数量,以及Fisher精确检验这两个库中该GO术语相对丰度差的P值。P值小于104表明这些库之间GO术语的丰度有显著差异。(XLS 93 KB)
12870 _2007_242_moesm7_esm.xls
附加文件7:该文件描述了在InterProScan对番茄和马铃薯HindIII消化BAC末端序列的分析中发现的PANTHER家族。该表中的列描述了PANTHER家族,番茄HBa和土豆POT库中分配了该术语的BAC端序列的数量,以及Fisher精确检验这两个库中该GO术语相对丰度差的P值。P值小于104表明这些库之间GO术语的丰度有显著差异。(XLS 124 KB)
12870 _2007_242_moesm8_esm.xls
附加文件8:该文件描述了在马铃薯hindii和EcoRI消化BAC末端序列InterProScan分析中发现的PANTHER家族。该表中的列描述了PANTHER家族,马铃薯POT和PPT库中分配了该术语的BAC端序列的数量,以及Fisher精确检验这两个库中该GO术语相对丰度差的P值。P值小于104表明这些库之间GO术语的丰度有显著差异。(XLS 110 KB)
12870 _2007_242_moesm9_esm.xls
附加文件9:该文件描述了在InterProScan对番茄hindii和EcoRI消化BAC末端序列的分析中发现的PANTHER家族。该表中的列描述了PANTHER家族,番茄HBa库和Eco库中分配了该术语的BAC端序列的数量,以及Fisher精确检验这两个库中该GO术语相对丰度差的P值。P值小于104表明这些库之间GO术语的丰度有显著差异。(XLS 112 KB)
权利和权限
开放获取本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(https://creativecommons.org/licenses/by/2.0),它允许在任何媒体上不受限制地使用、分发和复制,只要原著被恰当地引用。
关于这篇文章
引用这篇文章
E. Datema,穆勒,洛杉矶,布尔斯,R。et al。比较番茄和马铃薯的BAC端序列,发现马铃薯中特异性基因家族的过度表达。BMC植物杂志8日,34(2008)。https://doi.org/10.1186/1471-2229-8-34
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-34
关键字
- 番茄基因组
- 马铃薯基因组
- 主题长度
- 重复的数据库
- 吉普赛反转位子活动