摘要
背景
MicroRNAs (miRNAs)是最近发现的小的非编码rna,在基因表达中起着关键作用,特别是在植物和动物的转录后水平。在大量不同的植物物种中鉴定mirna对于理解mirna的进化和mirna靶向基因调控非常重要。如今,公开可用的数据库在硅内生物学中发挥着核心作用。由于在高等植物中至少有21个miRNA家族是保守的,因此使用这些数据库进行同源性搜索可以帮助识别植物中的正交同源或平行同源。
结果
我们搜索了所有公开的基因组调查序列(GSS)、高通量基因组序列(HTGS)、表达序列标签(ESTs)和非冗余(NR)核苷酸的核苷酸数据库,在155个不同的植物物种中鉴定出682个mirna。在11种植物中发现了15个以上的保守miRNA家族,在10种植物中发现了10 ~ 14个家族,在29种植物中发现了5 ~ 9个家族。在重要的豆科植物中鉴定出19个保守的miRNA家族Medicago,莲花和大豆。5个miRNA家族——miR319、miR156/157、miR169、miR165/166和miR394分别在51、45、41、40和40种不同的植物中被发现。在16个双子叶中发现了miR403同源物,而miR437和miR444同源物以及miR396家族的miR396d/e变体仅在单子叶中发现,因此为双子叶和单子叶特异性miRNAs提供了大规模的真实性。此外,我们提供了6个新发现的拟南芥miRNA同源体(miR158, miR391, miR824, miR825, miR827和miR840)和2个小rna (small-85和small-87)的保守性的计算和/或实验证据芸苔属植物spp.
结论
利用所有公开的核苷酸数据库,在155个不同的植物物种中鉴定出682个mirna。通过表达分析与计算方法相结合,我们发现迄今为止仅在拟南芥中发现的6个mirna和2个小rna也在拟南芥中保守芸苔属植物spp.这些发现将有助于通过检查小rna在人类共同祖先中的表达来追踪它们的进化拟南芥-芸苔属植物血统。
背景
细胞质对mRNA降解和翻译的控制是真核生物基因表达程序的重要策略之一。最近发现的miRNAs是转录后水平基因表达的重要调节因子。在植物中,miRNA基因由RNA聚合酶II转录,初级miRNA转录物随后被封住、剪接和聚腺苷化[1- - - - - -4].植物miRNA的加工似乎局限于细胞核,只有成熟的miRNA被输出到细胞质[2].在植物中,DCL1将主要miRNA转录物加工成miRNA-miRNA*双工,在3'端有2-nt悬垂[2].拟南芥hyponastic leaves (HYL1),一种含有双链rna结合结构域(dsRBD)的蛋白质,和SERRATE,一种C2H2锌指蛋白,协助DCL1释放miRNA双工[5- - - - - -7].然后,甲基转移酶HEN1在双工的3'端添加甲基,稳定miRNA双工[8].然后,miRNA双核苷酸通过植物的出口蛋白(草率)输出到细胞质中。9,10].只有双链的活性miRNA链,而不是客链(miRNA*)被纳入rna诱导沉默复合体(RISC)。在RISC中存在的miRNA的引导下,复合物可以识别目标转录本,并通过降解或翻译抑制来阻止蛋白质的产生[1,10- - - - - -13].
在植物中,miRNAs涉及植物生长发育的多个方面,包括叶的形态和极性、侧根的形成、激素信号传递、从幼年营养期到成体营养期、营养期到花期的过渡、花期、花器官的鉴定和繁殖[13,14].从最近的研究中,mirna在植物胁迫反应中的作用也很明显。几种mirna在不同的应激条件下受到调控,这表明mirna对其各自靶基因的转录后调控对于应对应激很重要[13,15- - - - - -20.].
由于miRNA已成为基因表达转录后调控的重要组成部分,对植物生长发育和植物胁迫反应至关重要,因此在尽可能多的植物物种中鉴定保守的miRNA同源物是非常重要的。计算方法可以成功地识别许多植物和动物中的保守miRNAs,但它们需要了解完整的基因组序列,而大多数植物物种都无法获得这方面的知识。然而,以基因组调查序列(GSSs)、高通量基因组序列(HTGSs)和非冗余核苷酸(NRs)以及表达序列标签(ESTs)为形式的大量基因组碎片数据可用于几种植物物种,并可用于鉴定保守的miRNAs。GeneBank的GSS和HTGS只代表了基因组序列的一小段,但仍然可以提供未完成基因组的更广泛的样本。NR数据库包含已完成的基因组序列和cdna。先前Zhang等。[21]仅使用ESTs识别植物中的保守miRNAs。
在这里,我们使用了可用的GSS、HTGS和NR库和ESTs来识别不同植物物种中的大量保守miRNA家族。利用BLAST搜索miRNA同源物,结合前体序列的二级结构预测,我们在155个不同的植物物种中鉴定出682个miRNA。在3种豆科植物中发现19个miRNA家族,Medicago truncatula,Lotus对虾而且大豆.此外,6个先前被认为是拟南芥特异性的mirna在芸苔属植物spp,这表明这些miRNAs是最近在拟南芥-芸苔属植物并提供了有价值的信息来追踪他们的进化。
结果
155种植物保守miRNAs的鉴定
计算鉴定miRNA的基础是保守的、成熟的miRNA序列加上可预测的miRNA周围序列的二级结构[22].我们使用NCBI BLASTN找到了至少匹配18 nt的miRNA序列(直系同源/平行同源),并为不同植物物种可能的序列变化留下3 nt。为了识别不同植物物种中的miRNA同源物,使用miRBase中的一整套拟南芥和水稻成熟miRNA序列(见URL的可用性和要求)对公开的GSS、HTGS、EST和NR数据库进行BLAST搜索。从各自的数据库中提取包含miRNA序列的miRNA前体序列,并使用mfold [23].miRNAs来源于发夹结构的5'或3'臂,这在不同的植物物种中也是保守的。为了证实这一特征,将这些发夹结构与之前报道的miRNA发夹结构进行了比较。这项研究在155种不同的植物中鉴定出了miRNAs。具体来说,我们在11种植物中发现了>15个miRNA家族,在10种植物中发现了10 ~ 14个miRNA家族,在29种植物中发现了5 ~ 9个miRNA家族。我们的调查还在一些植物物种中发现了相对保守的miRNA家族。例如,我们在玉米中发现了23个miRNA家族,在高粱中发现了19个,在小麦中发现了15个,在小麦中发现了14个柑橘sps.在一些重要的植物物种中也发现了其他值得注意的miRNA家族:葡萄中有12个,番茄中有11个,甘蔗中有10个,土豆中有7个。我们还发现裸子植物中有5个家族(miR159、miR160、miR164、miR166和miR168)保守,在裸子植物中有2个家族(miR396和miR408)保守卷柏.
有趣的是,miR319、miR156/157、miR169、miR165/166和miR394的同源物分别在51、45、41、40和40个不同的植物物种中被发现16个家族(miR159、miR160、miR167、miR170/171、miR396和miR399)在30-39种不同的植物中被发现(表1)1).同样,在20-29种不同的植物中发现了7个家族(miR164、miR168、miR172、miR393、miR395、miR398和miR408)。miR162、miR390、miR397、miR403和miR437家族在10-19种不同的植物中被发现。
在双科植物中,豆科植物是人类和动物饮食需求的重要来源,仅次于谷类植物。包括基因组学在内的分子工具正被用于快速发展Medicago truncatula,Lotus对虾而且大豆作为模型豆科植物来研究这些植物特有的一些重要的生物学问题。然而,这些重要的豆科植物中只有少数miRNA被记录在miRNA注册表中。除了miR397和miR403,我们的调查已经确定了豆科植物中剩余的19个保守miRNA家族(表397和miR403)1在双子叶和单子叶之间保守的约21个miRNA家族中,miR319同源物存在于最多的51种植物中,而miR397同源物存在于最少的14种植物中。通过搜索所有基因库来源,我们获得了更广泛的覆盖范围,无论是miRNA家族还是不同植物物种的数量。
在成熟miRNA序列相似性的基础上,这些miRNA被划分为家族,成员通常相差1至2个nt。在这里,我们在不同的植物物种中发现了16个新的miRNA,属于11个miRNA家族。这包括每个家族的一个新成员,miR158、miR159、miR160、miR172、miR390、miR395和miR408。我们还鉴定出属于miR319、miR398和miR403家族的2个新成员和属于miR169的3个新成员(表3)1).
张等。[21根据预测每个miRNA家族的植物数量,科学家将miRNA分为高度保守、中等保守和低保守,尽管不同植物物种可获得的ESTs数量差异很大。据此,miR395、miR399、miR403和miR408家族被归类为低保守[21].Zhang等人分别从9个和8个植物物种中提取了miR395和miR399同源物,这形成了作者将这些家族分类为低保守的基础[21].miR395和miR399在低营养条件下特异性上调。miR399在低磷酸盐条件下被诱导[16,18,24,25],而miR395则在低硫酸盐条件下被诱导[15].因此,原始miR395和miR399转录本在未经处理的植物产生的ESTs中表达是极不可能的。相比之下,使用GSS、HTGs、EST和NR数据库,我们分别在多达28和18个不同的植物物种中发现了miR399和miR395同源物。事实上,单独使用GSS, miR395和miR399同源物分别从9和11个不同的植物物种中检索到(表2)1).这些结果表明,这两个miRNA家族并不像之前认为的那样是低保守的miRNA。
从拟南芥和水稻中克隆miR408 [3.,26].仅通过EST数据库,就在9种植物中发现了miR408同源物。因此,Zhang等人。21]将miR408归类为低保守的mirna之一。在这项研究中,我们在23种不同的植物物种中发现了miR408同源物,包括卷柏(表1).因此,miR408是一种深度保守的mirna。miR408已被证明可以引导水稻中植物青素(plantacyanin)的靶转录物裂解[3.].同样在最近的一篇报道中,miR408被发现在卷柏以植物青素转录本为靶点[27].miR408在整个植物王国的深度保存表明植物紫素转录本水平的调控已经保存了很长时间。同样,我们在16种植物中发现了miR403同源物(表4)1);因此,miR403不是Zhang等人所分类的低保守miRNA。[21].总之,这些发现表明,仅根据现有的ESTs将mirna分类为高度、中度和低保守的mirna可能不能反映真正的保守深度。
双特异性和单特异性mirna
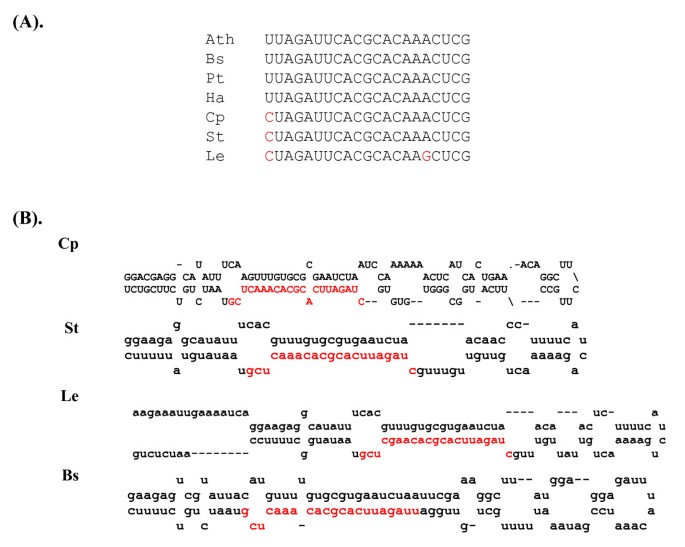
miR403最初在拟南芥中被发现,后来在杨树trichocorpa[4,26,28,29].在之前的一份报告中,miR403被认为是双柯特异性的miRNA,因为在大米中没有发现其同源物。在本研究中,我们在16种双子叶植物中发现了miR403同源体,包括杨树,木瓜,西红柿,土豆,向日葵,和芸苔属植物spp(表1和图1 a, B),但在单子叶植物中没有。在其他双子代中鉴定出miR403同源物,揭示了该miRNA家族的两个新成员。与拟南芥成熟的miR403序列相比,miR403在中5′核苷酸最多的地方存在差异木瓜和土豆,番茄中5' '最多2 nt(表1).因此,miR403家族在双子中至少由三个成员表示。在多达16个双子中鉴定出miR403,为认为它是双子特异性miRNA提供了大规模的真实性。
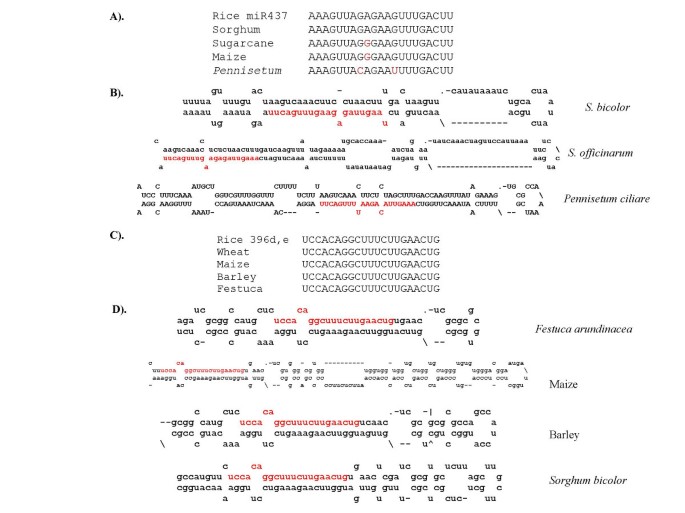
通过对水稻小RNA文库的测序,鉴定出了一些单核苷酸特异性的miRNAs [3.].水稻miR437同源物在玉米、高粱和甘蔗中发现,但在拟南芥或拟南芥中没有杨树提示miR437可能是单特异性miRNA [3.].在这项研究中,我们发现了更多的证据来支持将miR437分类为单特异性miRNA,因为从miR437中恢复了同源物狼尾草ciliare,另一单子叶(图2而且2 b).同样,miR444也被报道为单特异性miRNA [3.];在小麦、大麦、高粱、柳枝稷、甘蔗、短枝稻,野稻而且选用minuta(表1).最近,又有5个miR444家族成员被报道,这些成员都只在单子叶中保守(30)。
miR396同源物被发现高度保守[27].水稻中的miR396由两个具有5个基因座的变体(OsmiR396a,b,c和OsmiR396d,e) [3.].对应于OsmiR396a,b,c的成熟miRNA序列在双子代和单子代上是保守的。另一种变体,以OsmiR396d,e为代表,与OsmiR396a,b,c不同的是在位置8和9之间有一个额外的核苷酸“G”[3.].由于miR396d,e的确切序列尚未在拟南芥或杨树在拟南芥中未检测到其表达,被认为是miR396家族的单特异性版本[3.].与这一推测一致的是,miR396d,e同源物在另外5个单子叶中被鉴定出来高粱二色的,玉米,小麦,大麦和羊茅属arundancea-并且可以预测所有这些miRNA前体的发夹结构(图2摄氏度).因此,在几种单子叶中鉴定出miR437、miR444和miR396家族的miR396d/e变体,为认为这些mirna是单子叶特异性的观点提供了有力的支持。
拟南芥,芸苔属植物lineage-specific microrna
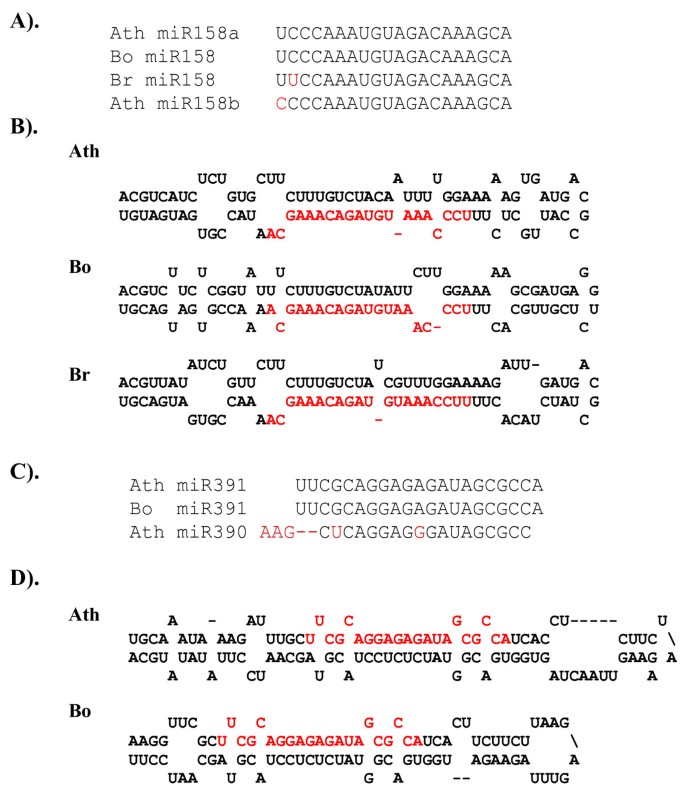
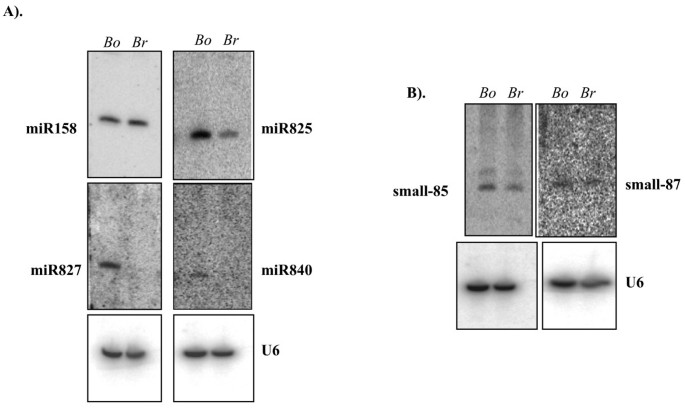
最初的实验方法导致在拟南芥中鉴定出至少四种非保守的mirna。miR158就是其中之一,在拟南芥中由miR158a和miR158b两个位点表示[31]和miR158同源物在水稻中计算/实验上也不明显[3.,15,32或用poplar [17].因此,miR158被认为是拟南芥特有的miRNA。在这里,我们发现了两个miR158同源体存在的计算证据芸苔属植物sps。(图3).此外,成熟的miR158序列和采用fold-back结构的序列具有高度保守性芸苔属植物oleracea而且芸苔属植物拉伯(图3 b).miR158在b·拉伯在5'端与拟南芥的miR158有2 nt的差异。用标记的miR158反义寡核苷酸进行Northern blot分析,发现miR158在小鼠中大量表达b . oleracea而且b·拉伯苗(图4).
miR391是最近发现的与miR390有一定序列相似性的mirna之一;因此,谢等。[4]认为它是miR390家族的一员。虽然miR390是广泛保守的miRNA之一,但miR391序列在拟南芥以外的植物中尚未被发现,这导致了miR391是一个非保守的拟南芥特异性miRNA的假设[4].我们的研究揭示了miR391同源物,并且可以预测miR391前体序列的折叠结构b . oleracea(图3 c而且3 d).
最近对拟南芥小rna的深度测序表明,拟南芥基因组编码的非保守miRNA家族比保守miRNA家族更多[19,33,34].这些新发现的拟南芥mirna被认为是非保守的,因为在水稻或水稻中还没有发现同源序列杨树基因组(19,33,34].非保守的植物mirna可能在很短的进化时间尺度内出现和消失[19,34].对与拟南芥密切相关的物种的小rna进行高通量测序将有助于确定这些瞬时miRNA基因的寿命[34].这些mirna保存的生物信息学检测芸苔属植物由于缺乏完整的基因组信息,在ESTs中对这些miRNA前体序列的搜索一直不成功,目前可能还不能提供完全的信息。由于这些新发现的miRNAs仅在高通量测序中被恢复,这表明它们的丰度极低,因此它们在ESTs中不太可能出现。研究这些新发现的miRNA同源物是否表达于芸苔属植物拟南芥是拟南芥的近亲,我们使用两种植物的RNA进行了小RNA印迹分析芸苔属植物spp.(b . oleracea而且b·拉伯).为了提高检测能力,我们使用了从4周龄苗木中分离出的低分子量RNA甘蓝和甘蓝.分析了新发现的10个mirna (miR771、miR773、miR775、miR825、miR827、miR828、miR837、miR840、miR846和miR848)的表达。我们选择这些mirna是因为它们可以在拟南芥的小rna印迹分析中检测到,并且在文库中比其他新发现的mirna相对更丰富[19,33,34].其中3个miRNAs (miR825、miR827和miR840)可在其中一个或两个细胞中检测到芸苔属植物spp,尽管它们的表达水平差异很大(图4).例如,miR825、miR827和miR840在b . oleracea比在b·拉伯(图4).令人惊讶的是,我们无法检测到miR827和miR840的信号b·拉伯(图4).计算分析显示miR824和miR828在芸苔属植物(数据未显示),尽管我们没有成功地使用探针检测miR828 in的信号芸苔属植物幼苗。miR828似乎在拟南芥的角果中特异或大量表达[34].最近,在3个基因中发现保守的miR824同源体芸苔属植物spp.[35].
计算分析显示,miR158、miR391和miR824的保守性芸苔属植物spp,我们的小RNA印迹分析证实,miR827, miR825和miR840在至少一个芸苔属植物spp(数据3.而且4).因此,在拟南芥以外未知表达的6个miRNAs (miR158、miR391、miR824、miR825、miR827和miR840)在拟南芥和拟南芥之间确实是保守的芸苔属植物.
已知拟南芥和水稻表达大量非保守的多样化小干扰rna (siRNAs) [36- - - - - -38].迄今为止唯一的例外是反式-作用sirna (Tas3a,b,c),一类高度保守的sinas [39,40].最近,Lu等人。[33]在拟南芥中发现了一些非mirna小rna。我们用小rna印迹分析来检测三种小rna (small-85, small-86和small-87)在拟南芥和拟南芥之间是否存在保守性芸苔属植物.令人惊讶的是,small-85和small-87都能被检测到芸苔属植物我们测试的物种(图4 b),这表明这两个小rna在拟南芥和芸苔属植物代表谱系特异性小rna。
植物miRNAs簇
在动物中经常发现的miRNAs簇作为多顺子一起转录[10,41- - - - - -44].虽然miRNA簇在植物中并不常见,但一些miRNA家族(miR395、miR399、miR169和miR1219)已被发现以簇的形式存在[26,45- - - - - -47].最近,在水稻和玉米中报道了两个串联miR156同源物[48,49].在这里,我们在其他几个植物物种中发现了一个miR156集群:水稻EST AK110797中两个串联miR156同源物位于相同方向370 nt内,甘蔗EST CA294779中两个miR156同源物相距190 nt, EST CL172990中两个miR156同源物相距340 nt高粱二色的在玉米EST CL985276中分离出两个相距301 nt的miR156同源体。此外,在一个基因组克隆中发现了两个非常紧密间隔的miR156同源体选用granulata(216元),选用punctata(370元)。在比较3种谷物(即水稻、高粱和玉米)的共线区时,Wang等[49]表明两个串联排列的miR156同源物在谷物中高度保守。有趣的是,我们在EST的CJ743424中发现了相隔590 nt的两个串联miR156同源物的相似排列Ipomea零是一种双子叶植物。这些发现表明,两个miR156同源物的串联排列并不局限于谷物,似乎存在于不同的远亲植物物种中。
我们还在棉花基因组克隆DX401397中发现了两个方向相同的串联miR169同源物,相隔250 nt。棉花中属于miR169家族的两个miRNAs(46)和芸苔属植物显著(49)最近被报道。由于这些同源物距离很近,有人认为它们来自两个不同的miR169原始转录本,尽管缺乏证据证明这两个miR169同源物以EST形式在一个转录本中表达。此外,miR169同源物在摘要以(DY980357),杨树tremula(CK111070)和大戟属植物esula(DV142897),但在拟南芥或水稻中没有。因此,我们展示了miR156和miR169位点在不同植物物种中的miRNA基因聚类。结果表明,在植物中至少存在4个miRNA家族(miR156、miR169、miR395和miR1219)。
讨论
最近的研究表明,mirna在高等真核生物的转录后基因表达中起着关键作用。植物miRNA保存的证据来自不同植物的基因组和EST序列数据,显示包含miRNA发夹的序列以及与已知或预测的保留miRNA互补位点的拟南芥靶标同源的序列[15,21].迄今为止,已知在双子叶和单子叶之间保守的约21个miRNA家族构成了利用公开的核苷酸数据库在不同植物物种中鉴定这些miRNA家族的基础。通过搜索这些数据库,我们在155个不同的植物物种中鉴定出了682个miRNAs。我们的分析在11种植物中得到15个保守的miRNA家族,在10种植物中得到10到14个保守的miRNA家族。我们还鉴定了相对保守的miRNA家族(即,玉米中有23个,玉米中有19个高粱小麦15英寸,小麦14英寸柑橘类葡萄中有12种,番茄中有11种,甘蔗中有10种,土豆中有7种。至少有5个家族(miR319、miR156/157、miR169、miR165/166和miR394)在40多个植物物种中被发现1).在30-39个物种中发现6个家族(miR159、miR160、miR167、miR170/171、miR396和miR399);7个(miR164、miR168、miR172、miR393、miR395、miR398和miR408)分布于20-29个种;5个(miR162, miR390, miR397, miR403和miR437)在10-19个物种中(表2)1).计算分析结合表达分析提供了6个新发现的mirna在拟南芥和拟南芥之间保守的证据芸苔属植物.此外,在拟南芥中发现的一些非mirna小rna (small-85和small-87)也在拟南芥中发现芸苔属植物(图4 b).这些发现首次大规模鉴定了谱系特异性mirna和其他小rna。
miR395和miR399分别在低硫酸盐和低磷酸盐条件下特异性诱导[15,16,18,24,25].miR399和miR395同源物分别存在于多达31和22种不同的植物物种中(表395)1).miR399在磷酸盐稳态中发挥重要作用[16,18].同样,在22种植物中发现了miR398同源物。miR398的下调与Cu/ zn -超氧化物歧化酶1 (CSD1)及2 (CSD2)对氧化应激条件的反应[13,20.].相反,miR398在Cu的作用下表达上调2 +极限条件[50].miR398诱导与的表达呈负相关CSD1而且CSD2基因,从而维持Cu2 +动态平衡和有效铜的调动2 +更重要的蛋白质,如质体青蛋白[50].miR393及其靶基因TIR1是保守的[15,26].miR393在拟南芥抗病中的作用最近已被证明[51].因此,我们发现了几种胁迫响应miRNA同源体——miR393、miR398、miR395和miR399——在不同的单子叶和双子叶中高度保守,这表明这些miRNA引导的靶基因调控被很好地保存了下来,可能是因为它们对植物的胁迫耐受性很重要[13].
最近对植物小RNA文库的深度测序清楚地表明,植物表达的非保守miRNAs比保守miRNAs更多[19,30.,34].非保守的mirna可能在较短的进化时间尺度内出现和消失[19].这种新基因的快速出现可能是由于其靶标衍生的miRNA基因的小尺寸和简单结构[52],尽管这种机制是否与大多数新出现的miRNAs相关[19,34尚不清楚。对10个新发现的miRNAs进行小rna印迹分析,证实其中3个表达于芸苔属植物幼苗。拟南芥中新发现的非保守mirna大多在花序中大量表达[33,34,36],但我们没有测试这个表达式。因此,其余7个miRNAs未检测到芸苔属植物幼苗需要进一步研究。一些新的miRNAs的表达缺失芸苔属植物可能是由于他们的损失芸苔属植物,或者它们是在分化后最近在拟南芥中进化出来的。
植物中mirna和tas3衍生的tasirna的存在是众所周知的[39,40].有趣的是,在本研究中,我们发现了两个小rna(非mirnas和非tasirnas)在拟南芥和拟南芥之间保守芸苔属植物.Small-85,最近被发现[33]并且源于一个长长的完美的折叠结构,这让人想起来自dsRNA的sirna。Small-85的积累依赖于拟南芥中的所有四个dicers [33].它只消失了四倍dcl(dcl-1,2,3,4)突变,但单独积累dcl1或者在三突变体中[33].Small-85来源于SRK基因,该基因能够采用折叠结构,其表达不依赖于RDR2 [32].
丧失自我不相容拟南芥而且芸苔属植物被认为是由于失活的自不相容(SI)系统,涉及SRK而且可控硅基因。在芸苔属植物SI系统、雌蕊(SRK)和花粉(SCR)中编码SI特异性的基因被认为是由于很少或没有重组而被保存下来的,而这种结构的破坏将导致SI的丢失。SI系统的损失答:芥Columbia-0 (Col-0)被认为是非功能性的SRK而且可控硅基因(53].卢等人。[33]假设了small-85在SRK功能丧失中的作用答:芥以其积累。在这里,我们展示了拟南芥small-85探针可以在两年内检测到预期大小范围内的强信号芸苔属植物物种,这表明small-85 RNA也在芸苔属植物幼苗。需要进一步的研究来阐明这种小RNA在自交不亲和中的作用。几种表达SRK来自营养组织中自交亲和植物物种的基因表明了这一点srk可能起到发展作用。同样,检测小85在芸苔属植物幼苗也表明其在发育中的作用芸苔属植物.
到目前为止,只有miR395同源物在拟南芥和水稻中被发现成簇存在[45].其中一些簇是共转录的,因为它们是在水稻的est中发现的[45].相似地,miR1219在Physcometrella最近报道[47].虽然miR399同源物在拟南芥和水稻中被发现间隔很近[26],其中一份文字记录中的表达是未知的。我们的分析表明,除了miR395的簇状结构外,miR156和miR169在一些植物物种中也以簇状形式存在。这些观察表明,串联复制是这种组织的原因。在某些植物中,串联重复的保留可能是由于剂量反应造成的。据估计,一般真核生物基因组中基因复制的发生率较高[54],尤其是在开花植物中[55,56].
尽管早些时候也进行了几次类似的尝试(21,28,46,57,58),但这些研究大多使用单一植物物种(例如棉花或棉花)芸苔属植物sps)或单核苷酸储存库(ESTs)。在这项研究中,我们使用了所有的核苷酸库,并考虑了所有的植物物种。此外,早期的报道(21,28,46,57,58)包括了最初被确定为mirna但最终被证明是sirna的小rna(例如,拟南芥中的miR404-miR407和水稻中的miR439、miR442和miR445)。在这里,我们使用了保守的方法,只考虑了在不同植物物种中鉴定同源物的自信注释的mirna。
与仅使用EST数据库相比,通过搜索所有可用的核苷酸数据库来鉴定保守的mirna可以更广泛、更好地覆盖不同的植物物种。我们发现了一些最近发现的拟南芥的mirna芸苔属植物拟南芥(Arabidopsis)的近亲,通过分析这些miRNAs在共同祖先中的表达,将有助于追踪这些miRNAs的进化芸苔属植物和拟南芥。拟南芥和b . oleracea是大约在1500万至2000万年前从共同祖先分化而来的密切相关物种[59].因为一些mirna在拟南芥和芸苔属植物,这些miRNAs可能存在于他们的祖先中。芸苔属植物起源的表达分析(如:番木瓜),以芸苔目(或芸苔科的姐妹科)为基础,将提供近、中、远的比较,以追踪这些mirna的进化。
结论
利用所有公开的核苷酸数据库,在155个不同的植物物种中鉴定出682个mirna。通过表达分析和计算方法相结合,我们发现迄今为止仅在拟南芥中发现的6个mirna和2个小rna也在拟南芥中保守芸苔属植物spp.这些发现将有助于通过检查小rna在人类共同祖先中的表达来追踪它们的进化拟南芥-芸苔属植物血统。
方法
对NCBI基因库进行Blast搜索
之前在拟南芥、水稻、杨树而且Physcometrella从miRBase (Release 10.0, 2007年8月)中提取非冗余的miRNA序列。我们使用这些序列在GSS, HTGS, EST和NR数据库中进行同源物的BLASTN搜索。我们采用了匹配至少18 nt的成熟miRNA序列,并为不同植物可能的序列变化留下0-3 nt。我们谨慎地使用了4-nt变异,并考虑了该4-nt变异是否在其他植物中也具有保守性。BLASTN参数与前面描述的基本相同[21,46].参数期望值为1000,描述和对齐数为1000。查询序列和数据库序列之间的默认字匹配大小为7。如果匹配的序列比查询的miRNA序列短,则手动比较对齐的部分,以确定匹配核苷酸的数量。只要有可能,就提取620 nt的前体序列(BLAST命中位点上游300 nt和下游300 nt)并用于发夹结构预测。对于GSSs,我们使用整个可用序列作为miRNA前体序列。这些前体序列被检索并用于BLASTX分析;我们去掉蛋白质编码序列,只保留非蛋白质序列。利用RNA二级结构预测软件mfold对这些潜在miRNA同源物的前体序列进行发夹状结构预测[23].我们在另一只手臂中使用了小于6个miRNA和miRNA*序列不匹配的截止值。计算研究报告miRNA前体序列具有明显高于其他非编码rna或mrna的负最小折叠自由能(mfe)和最小折叠自由能指数(MFEIs) [60].据Zhang等报道。[60],我们使用的MEFI截止值为0.85。最后,研究人员检查了发夹结构,并将其与先前报道的miRNA进行了比较,以确认成熟miRNA序列在发夹内的位置。简而言之,在将RNA序列指定为miRNA同源物时应用了以下标准:(1)折叠成适当的茎环发夹二级结构的RNA序列,(2)发夹结构的一只臂上有成熟的miRNA序列位点,(3)另一只臂上与相反的miRNA*序列错配少于6个的miRNA*序列,(4)miRNA*序列无环或断裂,(5)预测MFEIs较高和mfe为阴性的二级结构,(6)预测与之相比取代不超过3个nt的成熟miRNA答:芥、大米、杨树而且Physcometrella成熟的microrna。这些参数符合Ambros及其同事提出的标准[61].
RNA凝胶印迹分析
使用Trizol试剂从未处理(对照)或暴露于盐胁迫或干旱胁迫的四周大的水稻幼苗中分离总RNA。用PEG沉淀法从总RNA中分离出低分子量RNA。每个通道装载20 μg低分子量RNA,在变性15%的聚丙烯酰胺凝胶上分解,并通过电泳转移到Hybond-N+膜上(Amersham Biosciences, Buckinghamshire, UK)。膜经UV交联,在80°C下烘烤2小时。使用T4多核苷酸激酶(New England Biolabs)对与miRNA序列互补的DNA寡核苷酸进行末端标记γ-32P-ATP。膜预杂交至少1小时,在38°C下使用完美杂交缓冲液(Sigma)进行过夜杂交。印迹在50℃下清洗三次(2 × SSC + 1% SDS清洗两次,1 × SSC + 0.5% SDS清洗一次)。膜被简单地风干,然后暴露在磷屏上,并通过使用台风扫描薄膜获得图像。测试了两个小rna序列在芸苔属植物分别为small-85 (small-85 CAAGACAATAATCTTCTCGGCTA)和small-87 (small-87 AAGAACATCCAAGGTGTTTGT) [32].
可用性和需求
为了鉴定不同植物物种中的miRNA同源物,从miRBase中获得了一整套拟南芥和水稻成熟miRNA序列(Release 10.0, August 2007;http://microrna.sanger.ac.uk)用于BLAST搜索,并对公开的GSS、HTGS、EST和NR数据库进行搜索。
参考文献
- 1.
Aukerman MJ, Sakai H: MicroRNA及其apetala2样靶基因对开花时间和花器官特性的调控。植物学报,2003,30(4):344 - 344。10.1105 / tpc.016238。
- 2.
Kurihara Y, Watanabe Y:拟南芥Dicer-like 1蛋白功能的微rna生物发生。美国国家科学研究院。2004, 21(1): 1 - 4。10.1073 / pnas.0403115101。
- 3.
孙嘉玲,朱建科,朱建科,孙志刚:水稻microrna的克隆与鉴定。植物细胞学报。17:1397-1411。10.1105 / tpc.105.031682。
- 4.
谢喆,孙晓明,王晓明,王晓明,王晓明,王晓明,王晓明,王晓明,王晓明。拟南芥MIRNA基因表达的研究进展。中国生物医学工程学报,2005,38(4):344 - 344。10.1104 / pp.105.062943。
- 5.
韩明,Goud S, Song L, Fedoroff N:拟南芥双链rna结合蛋白HYL1在microrna介导的基因调控中发挥作用。美国国家科学研究院。2004, 30(4): 344 - 344。10.1073 / pnas.0307969100。
- 6.
robes D, Rallapalli G, Schmidt DD, Martin C, Clarke J: SERRATE:植物microRNA领域的新玩家。中国机械工程学报,2006,7:1052-1058。10.1038 / sj.embor.7400806。
- 7.
杨玲,刘震,陆峰,董安,黄华:SERRATE是拟南芥microRNA原代加工中的一种新型核调控因子。植物学报,2006,47:441 - 448。10.1111 / j.1365 - 313 x.2006.02835.x。
- 8.
于波,杨震,李娟,陈晓霞,杨敏,陈晓霞:甲基化在植物microRNA生物发生中的作用。科学通报,2005,37(3):344 - 344。10.1126 / science.1107130。
- 9.
吴国强,吴国强,吴国强,陈志伟,陈志伟,陈志伟。拟南芥microrna的核加工与输出研究。美国国家科学研究院。2005, 32(3): 366 - 366。10.1073 / pnas.0405570102。
- 10.
巴特尔DP: MicroRNAs:基因组学,生物发生,机制和功能。电子学报,2004,34(1):1- 7。10.1016 / s0092 - 8674(04) 00045 - 5。
- 11.
陈欣:拟南芥花发育中作为APETALA2翻译抑制因子的microRNA。科学通报,2004,30(3):344 - 344。10.1126 / science.1088060。
- 12.
Jones-Rhoades MJ, Bartel B, Bartel DP: MicroRNAs及其在植物中的调控靶点。植物学报,2006,29(4):344 - 344。10.1146 / annurev.arplant.57.032905.105218。
- 13.
孙佳,朱娟,朱建军:小rna在植物非生物胁迫响应和营养剥夺中的作用。植物科学进展,2007,12(7):301-309。10.1016 / j.tplants.2007.05.001。
- 14.
Mallory A, Vaucheret H:植物中microRNAs及相关小rna的功能。中国科学,2006,38(增刊):331 -36。10.1038 / ng1791。
- 15.
Jones-Rhoades MJ, Bartel DP:植物microrna及其靶标的计算鉴定,包括胁迫诱导的miRNA。生物化学学报,2004,14:787-799。10.1016 / j.molcel.2004.05.027。
- 16.
藤井红,邱廷杰,林思,昂凯,朱景江:拟南芥磷酸饥饿反应中的一个miRNA。中国生物工程学报,2005,30(4):344 - 344。10.1016 / j.cub.2005.10.016。
- 17.
吕松,孙艳华,史锐,陈志强,李玲,蒋维林:拟南芥中缺失的毛白杨机械胁迫响应microrna。中国生物医学工程学报,2005,27(3):366 - 366。10.1105 / tpc.105.033456。
- 18.
邱廷杰,昂凯,林思,吴春春,蒋顺丰,苏春林:microRNA对拟南芥磷酸盐稳态的调控。植物学报,2006,18:412-421。10.1105 / tpc.105.038943。
- 19.
Fahlgren N, Howell MD, Kasschau KD, Chapman EJ, Sullivan CM, Cumbie JS, Givan SA, Law TF, Grant SR, Dangl JL, Carrington JC:拟南芥microrna的高通量测序:MIRNA基因频繁出生和死亡的证据。PLoS ONE。2007, 2: e219-10.1371/journal.pone.0000219。
- 20.
孙卡R, Kapoor A,朱建科:拟南芥两个Cu/Zn超氧化物歧化酶基因的转录后诱导是通过下调miR398介导的,对抗氧化胁迫具有重要意义。植物科学学报,2006,30(4):344 - 344。10.1105 / tpc.106.041673。
- 21.
张波,潘X, Cannon CH, Cobb GP, Anderson TA:植物microRNA基因的保守与分化。植物学报,2006,46:243-59。10.1111 / j.1365 - 313 x.2006.02697.x。
- 22.
Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T: microRNA注释的统一系统。RNA。2003, 9: 277-9。10.1261 / rna.2183803。
- 23.
Zuker M: Mfold网络服务器,用于核酸折叠和杂交预测。核酸研究,2003,31:3406-15。10.1093 / nar / gkg595。
- 24.
昂凯,林思,吴春成,黄奕婷,苏春林,邱廷杰:pho2是由microRNA399靶基因的无义突变引起的。植物生理学报,2006,29(4):344 - 344。10.1104 / pp.106.078063。
- 25.
Bari R, Datt Pant B, Stitt M, Scheible WR: PHO2, microRNA399和PHR1定义了植物中的磷酸盐信号通路。植物生理学报,2006,29(4):388 - 388。10.1104 / pp.106.079707。
- 26.
孙卡罗,朱建科:压力调节microRNAs和其他小rna拟南芥.中国生物医学工程学报,2004,16:2001-2019。10.1105 / tpc.104.022830。
- 27.
Axtell MJ, Snyder JA, Bartel DP:陆地植物不同小rna的共同功能。植物科学学报,2007,29(3):379 - 379。10.1105 / tpc.107.051706。
- 28.
张波峰,潘小鹏,王清良,Cobb GP, Anderson TA:植物新microRNAs的EST鉴定与鉴定。中国生物医学工程学报,2005,30(4):366 - 366。10.1038 / sj.cr.7290302。
- 29.
Tuskan GA, Difazio S, Jansson S, Bohlmann J, grigoriiev I, Hellsten U, Putnam N, Ralph S, Rombauts S, Salamov A, Schein J, Sterck L, Aerts A, Bhalerao RR, Bhalerao RP, Blaudez D, Boerjan W, Brun A, Brunner A, Busov V, Campbell M, Carlson J, Chalot M, Chapman J, Chen GL, Cooper D, Coutinho PM, Couturier J, Covert S, Cronk Q, Cunningham R,等:黑杨木基因组。中国林业科学,2006,27(3):366 - 366。
- 30.
孙嘉荣,周晓霞,郑勇,张伟,朱建军:利用高通量测序鉴定水稻新mirna和候选mirna。中国生物医学工程学报,2008,8:25-10.1186/1471-2229-8-25。
- 31.
Reinhart BJ, Weinstein EG, Jones-Rhoades MW, Bartel B, Bartel DP:植物中的MicroRNAs。基因与发育。2002,16:1616-1626。10.1101 / gad.1004402。
- 32.
Bonnet E, Wuyts J, Rouze P, de Peer YV:拟南芥和水稻91个潜在保守植物microrna的检测发现了重要的靶基因。美国国家科学研究院。2004, 30(4): 457 - 457。10.1073 / pnas.0404025101。
- 33.
Lu C, Kulkarni K, Souret FF, Muthuvalliappan R, Tej SS,等:拟南芥RNA依赖RNA聚合酶-2突变体中MicroRNAs和其他小RNA富集。基因组学报,2006,16:1276-1288。10.1101 / gr.5530106。
- 34.
Rajagopalan R, Vaucheret H, Trejo J, Bartel DP:拟南芥microrna的多样性和进化流体集。基因开发,2006,20:3407-3425。10.1101 / gad.1476406。
- 35.
Kutter C, Schöb H, Stadler M, Meins F, Si-Ammour A: microrna介导的拟南芥气孔发育调控。植物细胞学报,2007,19:2417-2429。10.1105 / tpc.107.050377。
- 36.
陆超,Tej S, Luo S, Haudenschild CD, Meyers BC, Green PJ:转录组中小RNA成分的解析。科学通报,2005,29(4):457 - 457。10.1126 / science.1114112。
- 37.
孙嘉华,朱建军,孙晓燕,王晓燕:水稻内源性小干扰rna的鉴定与鉴定。中国生物医学工程学报,2005,33(4):344 - 344。10.1093 / nar / gki758。
- 38.
Nobuta K, Venu RC, Lu C, Beló A, Vemaraju K, Kulkarni K, Wang W, Pillay M, Green PJ, Wang GL, Meyers BC:水稻mrna和小rna的表达图谱。生物技术学报,2007,25:473-7。10.1038 / nbt1291。
- 39.
谢志新,谢志新,王晓明,王晓明:植物反式作用siRNA生物发生过程中microrna定向的相位调控。光子学报,2005,29(3):344 - 344。10.1016 / j.cell.2005.04.004。
- 40.
Axtell MJ, Jan C, Rajagopalan R, Bartel DP:植物siRNA生物发生的两击触发。细胞学报,2006,27(3):344 - 344。10.1016 / j.cell.2006.09.032。
- 41.
Altuvia Y, Landgraf P, Lithwick G, elant N, Pfeffer S, Aravin A, Brownstein MJ, Tuschl T, Margalit H:人类microrna的聚类和保存模式。中国生物医学工程学报,2005,34(3):344 - 344。10.1093 / nar / gki567。
- 42.
Seitz H, Royo H, Bortolin ML, Lin SP, Ferguson-Smith AC, Cavaillé J:小鼠Dlk1-Gtl2结构域的印迹microRNA基因簇。中国生物医学工程学报,2004,30(4):344 - 344。10.1101 / gr.2743304。
- 43.
Tanzer A, Stadler PF: microRNA簇的分子进化。分子生物学杂志,2004,39(3):327- 329。10.1016 / j.jmb.2004.03.065。
- 44.
Baskerville S, Bartel DP: microRNAs的微阵列分析揭示了与邻近miRNAs和宿主基因的频繁共表达。RNA。2005, 11: 241-7。10.1261 / rna.7240905。
- 45.
细胞科学进展,2005,15:631-638。10.1038 / sj.cr.7290333。
- 46.
张波,王强,王凯,潘霞,刘峰,郭涛,Cobb GP, Anderson TA:棉花microrna的鉴定及其靶标。基因学报,2007,397:26-37。10.1016 / j.gene.2007.03.020。
- 47.
杜晓峰,王晓峰,王晓峰,王晓峰,王晓峰。苔藓微rna的研究进展。植物学报,2006,44(3):344 - 344。10.1111 / j.1365 - 313 x.2006.02768.x。
- 48.
蔡国强,孙晓明,孙晓明,孙晓明,孙晓明:玉米异慢性突变体Corngrass1是由串联microRNA的过表达引起的。自然遗传学报,2007,39:544-549。10.1038 / ng2001。
- 49.
王山,朱庆林,郭晓霞,桂艳,鲍军,Helliwell C,范玲:水稻双串联microrna基因的分子进化与选择。清华大学学报(自然科学版),2007,29(4):349 - 349。10.1016 / j.febslet.2007.09.002。
- 50.
山崎浩,张志刚,张志刚,张志刚,张志刚。拟南芥微rna调控铜稳态的研究进展。生物化学学报,2007,28(3):366 - 366。10.1074 / jbc.M700138200。
- 51.
Navarro L, Dunoyer P, Jay F, Arnold B, Dharmasiri N, Estelle M, Voinnet O, Jones JD:植物miRNA通过抑制生长素信号通路促进抗菌抗性。科学通报,2006,31(4):439 -439。10.1126 / science.1126088。
- 52.
谢震,谢志刚,谢志刚,孙红艳,杨晓明,杨晓明:microRNA基因的克隆与表达拟南芥.《自然科学》,2004,36:1282-90。10.1038 / ng1478。
- 53.
Kusaba M, Dwyer K, Hendershot J, Vrebalov J, Nasrallah JB, Nasrallah ME:拟南芥异交中S位点的自交不亲和特征答:lyrata和它的自交配亲戚答:芥.植物学报,2001,13:627-43。10.1105 / tpc.13.3.627。
- 54.
Lynch M, Conery JS:重复基因的进化命运和后果。科学通报,2000,29(4):531 - 531。10.1126 / science.290.5494.1151。
- 55.
Blanc G, Wolfe KH:从复制基因的年龄分布推断模式植物中广泛的古多倍体。植物科学学报,2004,16:1667-1678。10.1105 / tpc.021345。
- 56.
Cui L, Wall PK, Leebens-Mack JH, Lindsay BG, Soltis DE, Doyle JJ, Soltis PS, Carlson JE, Arumuganathan K, Barakat A, Albert VA, Ma H, dePamphilis CW:开花植物历史上广泛的基因组复制。基因组学研究,2006,16:738-749。10.1101 / gr.4825606。
- 57.
张波,潘X, Anderson TA: 188个玉米保守microrna的鉴定及其靶标。清华大学学报(自然科学版),2006,38(4):344 - 344。10.1016 / j.febslet.2006.05.063。
- 58.
谢福林,黄淑清,郭凯,项爱民,朱友友,聂林,杨志明:新型microRNAs和靶标的计算识别芸苔属植物显著.农学通报,2007,29(4):344 - 344。10.1016 / j.febslet.2007.02.074。
- 59.
杨玉文,赖克宁,邰玉培,李文华:芸苔属被子植物线粒体DNA序列的核苷酸替代率及其分化日期。中国生物工程学报,1999,29(4):349 - 349。10.1007 / PL00006502。
- 60.
Zhang BH, Pan XP, Cox SB, Cobb, GP, Anderson TA: mirna与其他rna不同的证据。中国生物医学工程学报,2006,29(3):344 - 344。10.1007 / s00018 - 005 - 5467 - 7。
- 61.
Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T: microRNA注释的统一系统。RNA。2003, 9: 277-279。10.1261 / rna.2183803。
确认
这项工作得到了俄克拉荷马州农业实验站、OCAST植物科学研究项目和美国农业部对R. Sunkar的拨款(NRI#2007-02019)的支持。我们感谢特拉华大学的B. Meyers, P. Green和C. Lu,他们慷慨地赠送了针对small-85, small-86和small-87的LNA探针。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
RS设计了研究,RS和GJ进行了研究,RS撰写了论文。
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
桑卡尔,R.,贾格德斯瓦兰,G.。在网上在大量不同的植物物种中鉴定保守的microRNAs。BMC植物生物学8,37(2008)。https://doi.org/10.1186/1471-2229-8-37
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-37
关键字
- 小rna
- microrna的家庭
- 植物microrna
- 成熟miRNA序列
- 植物种类多样