摘要
背景
我们进一步鉴定了花器官定位基因在花序中的表达拟南芥通过比较大规模并行签名测序(MPSS)数据。从未成熟的花序组织中构建了6个RNA序列标签库,并将其与注释中的各自位点进行了匹配拟南芥基因组。这些签名库调查了野生型组织的花的转录组以及花的同种异体突变体,apetala1 apetala3,无性生殖的,一个超人/ apetala1双突变体,分化胚珠从野生型花序的雌蕊中分离。比较和对比这些MPSS花表达库,可以区分在花瓣、雄蕊、柱头型、雌蕊中富集的转录本,以及在萼片/萼片-花瓣、花瓣-雄蕊或雌蕊中富集的转录本。
结果
通过对表达文库的比较,共发现572个基因在花序中有器官富集表达。大部分器官富集的转录本多样性出现在雌蕊和雄蕊中,而很少有基因表现为萼片或花瓣的定位表达。通过与之前发表的表达数据进行比较,对计算分析进行验证,原位杂交、启动子-报告子融合和逆转录PCR。在我们的转录本过滤系统中,有许多特征良好的基因被准确地描述出来。此外,经验验证证实了MPSS预测的几个基因与先前未描述的表达模式。
结论
这种广泛的MPSS分析证实和补充了先前的微阵列花表达研究,并说明了基于序列调查的表达分析在功能基因组学中的效用。通过MPSS和类似方法积累的空间花表达数据将有助于阐明更全面的调控花发育的遗传调控网络。
背景
大部分与花发育相关的基因已经通过对表现出严重表型偏离野生型发育的突变体的表征被识别出来。这些突变体的一个有趣的子集是同源花表型组。在这些突变体中,一个螺旋花序的器官在另一个不同的螺旋中复制,而牺牲了通常存在的器官。这些突变的前提在植物发育的经典“ABC模型”中得到了解释拟南芥[1,2].根据ABC模型,MADS-box转录因子之间的相互作用包括但不限于APETALA 1(AP1),无性生殖的(AG)),APETALA 3(AP3)是萼片、花瓣、雄蕊和雌蕊发育所必需的。这些转录因子的功能缺失导致花器官的同种置换。蛋白质相互作用的“四重奏模型”解释了遗传ABC模型[3.],并提出形成五种不同的螺纹特异性四聚体复合体,能够结合DNA并激活下游负责器官发育的基因独联体-双车箱的调节[3.].在体外分析发现MADS-box转录因子之间存在异二聚体相互作用[4].此外,在活的有机体内同源矮牵牛MADS-box蛋白的相互作用也被观察到,该蛋白可能参与一个确定胚珠的第四系复合体[5].
尽管四重奏模型得到了结构上的支持,但该模型的许多监管方面还有待确定。一些花序分生组织同源基因如多叶的,不寻常的花器,LEUNIG,卷曲的叶子与编码这些四元复合体的基因上游调控有关。然而,这些复合物直接激活的下游器官特异性基因很少被发现[6].而且,下游标的如富有成果的,SPOROCYTELESS /喷嘴而且没有顶端分生组织不服从四重奏模型的单轮前提。[7- - - - - -9].为了验证其功能并了解其靶点的性质,有必要对假定的第四复合体下游的器官特异性基因表达进行表征。
基因组方法已成为一种有价值的工具,在表征器官相关基因表达和阐明全球水平的植物发育的遗传网络。通过基于杂交的方法,如cDNA和寡核苷酸微阵列,已对拟南芥花器官转录本富集进行了全基因组分析[10- - - - - -20.],代表了花转录组空间表征的有力的第一步。然而,微阵列分析和其他基于杂交的方法受到许多固有的限制,包括对RNA数量的敏感性、非特异性探针杂交和能够掩盖低表达率转录本的大量背景水平[21].此外,跨多个微阵列的定量分析需要芯片的标准化和校准,以确保等效杂交。
尽管技术进步正在解决这些微阵列问题,但签名测序(如大规模并行签名测序,MPSS)代表了微阵列的另一种选择,可以克服基于杂交的技术和其他大规模基因表达分析的传统方法固有的许多限制。MPSS反应是由现在的Illumina, Inc.(原Lynx Therapeutics, Hayward, CA)开发的,它允许对来自样本的17或20个核苷酸“签名”进行同步或并行测序,对应于不同的cDNA分子[22,23].然后将这些表达签名与基因组中相应的序列进行匹配,以确定基因表达。MPSS签名的长度通常允许与拟南芥基因组的单次匹配,并使转录的高度特异性量化成为可能[24].该技术提供的背景水平优于基于杂交的技术允许的转录本检测水平,并能够检测表达水平较低的转录[21,如许多转录因子。此外,MPSS数据采集的线性归一化性质降低了cDNA文库之间信号标准化的重要性。尽管如此,之前在MPSS测序反应中描述的“坏词”[25],以及在特定的cDNA中缺乏必要的限制位点,在不同的转录组中低表达的转录本被稀释,重复的成本高昂都是该技术的缺点。因此,在未来的元分析中集成微阵列研究和MPSS将允许改进全基因组表达表征,以及短读DNA测序技术的进步,如Illumina公司目前的“合成测序”(SBS)方法。
在本研究中,我们利用MPSS技术对花瓣、雄蕊、胚珠外的雌蕊、萼片/萼片-花瓣、花瓣-雄蕊或雄蕊-雌蕊中富集的基因进行了分析。MPSS签名与基因组注释(TAIR版本6)匹配,并集成到我们的公开web界面[26].利用该界面,交叉分析表达模式,解剖表达丰富的花器官。野生型花序被用来表征所有花器官的转录表达。突变无性生殖的(“ag), SALK_014999),用来描述雄蕊和雌蕊的完全丧失,这是由于花被轮生的生殖器官的同种置换。相反,花被螺纹的损失是通过使用apetala 1 - 10 (ap1)一种无花瓣突变体,具有增加的生殖轮生和次生无花瓣花序,以同种方式取代大多数萼片。丢失的花瓣和雄蕊表达是通过使用apetala 3 - 6(“ap3”)这种突变体的花瓣和雄蕊轮生器官已经完全被心皮和萼片所取代。虽然心皮组织减少,但双突变体Superman-2无瓣1-10(“sup ap1”)被优先选择为心皮减少,并用于表征雄蕊的富集,牺牲所有其他花器官。除了利用全花序文库来测定器官富集的基因表达外,还利用分离的分化胚珠、根和叶来测定雌蕊花表达的转录本的胚珠包容性,并在全植物范围内进行花表达的富集。
结果与讨论
花序表达谱的比较
序列调查已成为一种广泛应用的方法,以确定表达模式,并进一步阐明全基因组尺度上的发育过程。为了进一步描述花的发育特征,我们从野生型的cDNA中构建了MPSS特征库,Ap1, ap3, ag, sup Ap1在发育的前十二个阶段的花序[27以及分化的胚珠、根和叶组织。将MPSS签名与它们各自的基因注释相匹配,以相对方式描述活性表达(以每百万转录本的转录本测量;TPM)。21715个基因在检测的基因型的花花序中显著表达。为了丰富花特异性转录本,将MPSS叶片和根系表达数据与花序表达谱进行比较。只有7.9 - 13.4%的花序表达基因在叶或根中未被观察到(即0 TPM)(表4)1).因此,在花序中发现的大部分基因都不是花特异性的。尽管如此,除了ap3非花特异性空间表达基因在所有花序中的平均表达量均显著高于叶片或根组织(p < 0.001)。
在叶和根中使用0 TPM阈值选择花序特异性表达过于严格,与一些已知表达模式的基因相比(数据未显示),会导致较高的鉴别假阴性率。因此,富集花序的基因选择是通过去除在花序中表达量低于根和叶组织的基因来完成的。此外,只有那些在花序中表达大于4tpm的基因才被纳入我们的过滤标准(表1)1).使用更不严格的参数,许多花器官富集转录本被准确描述,这在我们的生物学验证中很明显;然而,提高该阈值降低了错误发现率,并增加了准确检测花表达富集的信息学能力。不同花序基因型的转录本多样性趋势不受花序富集的显著影响。在原始数据和过滤后的花序富集表达数据中,野生型的转录本多样性水平最高,ap1,ap3花序和最低的在一口ap1而且无性生殖的(表1).这可能是由于前者的生殖器官增多,而后者的花器官多样性和纯营养器官明显减少。虽然两组间转录组多样性差异显著,但两组间花序多样性差异无统计学意义(p > 0.10)。
在对花序表达进行富集后,对每个MPSS文库的表达谱进行表征,以揭示基因表达与表型之间的关系,并描述数据之间可能的偏差。基因表达的保守性是在所有文库的交集和文库的相对基础上确定的。在我们的交叉方法中,将两个花序富集筛选文库中的保守基因与两个文库中的基因数量进行比较。例如,(ap1∩ap3) / (ap1∪ap3)揭示了基于我们过滤标准中基因的存在或缺失的归一化相关性(表2).我们的文库相对方法是基于计算两个文库中花序表达水平(TPM测量)无统计学差异(p < 0.001)的基因。统计上无差异的基因数量由两个文库中表达的不同基因总数归一化,形成一个比例(表2).两种方法对具有显著不同相关性的库显示了相似的结果。
得到最相似的表达模式ap1和野生型花序。的同源转化突变体ap1而且ap3以及ap3与野生型也高度相关(表2).三种相关性之间的差异有统计学意义(p < 0.001),它们的相对秩在库交叉和基于相关性的方法中都是保守的。因此,以牺牲花被组织为代价增加雌蕊生物量对基因表达的影响不如生殖器官的损失大。此外,最大的表型差异反映在最显著的基因表达差异上。花蕊在花序内雄蕊的增加一口ap1与完全植物人相比产生了最大的差异无性生殖的尽管两个文库的转录多样性程度相似,但两种比较方法的花序均有差异。
以确定表达的其他变化ap1生成的吃晚饭突变和解决可能的表型相关性,表达谱一口ap1而且ap1比较。虽然保守基因的比例之间一口ap1而且ap1比的大一口ap1而野生型花序在库交分析中差异不显著(p > 0.05)。在文库相对分析中,基因表达的相似性一口ap1其他花序均较低2).这表明雄蕊的增加以牺牲所有其他组织为代价,比任何其他突变都更能改变花序表达谱。尽管基因表达存在显著差异,ap1揭示了与之最相似的花序表达模式一口ap1库中相对分析(p < 0.001)。
器官富集基因表达的鉴定
为了识别在花器官中高度富集和在其他器官中减少的转录本,我们使用二元系统分析了数据。单个花品系中某一特定器官或一组器官的出现被描述为“1”,如果有,则描述为“0”。基因的表达模式反映了同源突变体的器官发生概况(表3.)被认定为为有关器官的丰富成分。例如,雄蕊存在于ap1而且一口ap1;然而,他们在ap3而且无性生殖的.因此,只有那些表达大于4tpm的基因ap1而且一口ap1和缺席(0 TPM)从ap3而且ag)MPSS库被认为是“雄蕊丰富的”。为了进一步筛选全植物范围内的花器官富集,仅在花序内表达率(TPM)高于叶或根组织的转录本被纳入分析。
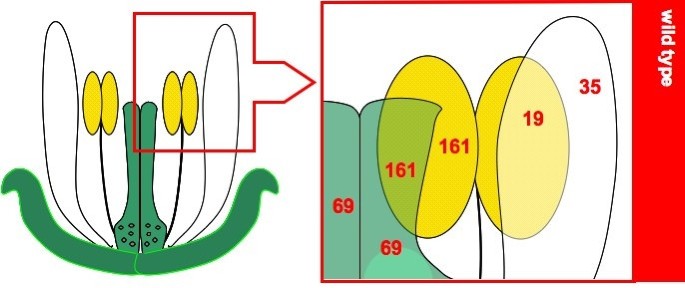
分析所有可能的花器官表达模式的能力受限于本研究中使用的突变库的多样性(表3.).一个器官或多个器官内的丰富表达只有在它们各自的器官发生谱是唯一的情况下才能被识别。例如,萼片富集的转录本不能从萼片和花瓣表达的转录本中分离出来,因为来自两个空间富集组的转录本都存在或上调无性生殖的而且apetala3突变体和内部下调ap1而且一口ap1.相似的局限性是固有的在以前的同源微阵列分析和可能导致的表征Apetala1,萼片和花瓣表达的转录本[28],在之前的数据集中以特定于separ的方式表示[10].此外,器官特异性的预测可能因同源异体转化导致的转录的附加改变而产生偏差。这方面的一个例子是萼片表达依赖于萼片和花瓣的共存。推测丰富的花瓣转录本也可能在野生型花序的萼片内表达,但在野生型花序中不表达apetala3;用于区分萼片和雌蕊缺乏表达的突变体。考虑到这些限制,在我们的MPSS分析中,转录本富集在花序器官中的存在可能只解剖了7个实例:萼片/萼片-花瓣,花瓣,雄蕊,花瓣-雄蕊,雄蕊-雌蕊,以及包含和不包含胚珠的雌蕊(见附加文件)1查看完整的成绩单列表)。
数字1揭示在分析的花器官中被鉴定为丰富的基因的数量。为了减少我们的错误发现率,每个花器官中富集的基因数量可能被低估了;然而,即使在仅基于基因存在(>0 TPM)或不存在(0 TPM)的宽松过滤参数下,我们识别的转录本多样性的相对趋势也保持不变。在生殖器官中发现了最大的转录多样性,而在营养组织或雄蕊和花瓣中活性表达较少。
在我们的分析中,共有138个基因被鉴定为具有富含雌二醇的表达。这与雄蕊内的161个基因和在两个生殖器官中表达的161个基因非常接近。雌蕊内的转录多样性水平与胚珠无关;包含胚珠的雌蕊表达和不包含胚珠的雌蕊表达共鉴定出69个基因。这表明在花发育的前12个阶段[27],胚珠不是由比柱头、花柱、胎盘或心皮壁更大的富含雌蕊的基因子集表达的。尽管在以往的显微解剖研究中发现了广泛的多样性[16]中,只有35个基因表达丰富花瓣无性生殖的但在其他突变花序中没有。尽管如此,这比在雄蕊和花瓣中都表达的19个转录本要多。因此,只在花瓣和雄蕊上富集的转录本可能比在每个器官上富集的转录本更少。这与在生殖器官中注意到的趋势形成对比,即在单个器官中发现的基因富集数量与在整个生殖组织中表达的几乎相同。不同于以前的比较,可以在单个器官或两个器官的集群中分离富集,在萼片和整个花被中富集的56个基因不能相互分离,因为本研究中使用了突变体。因此,萼片的相对转录复杂性不能与花瓣进行比较。尽管如此,我们可以得出结论,sepal富集基因的数量并不会超过在任何生殖器官中发现的基因数量。
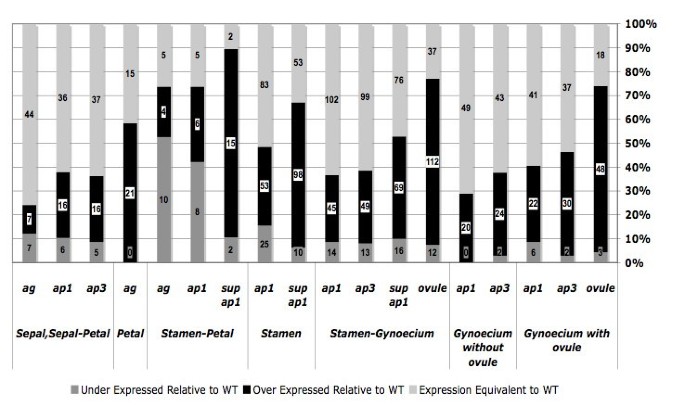
在确定MPSS测量的预测器官富集基因集的相对多样性后,比较基因表达水平。考虑到花器官发育增加的同种异型突变体应该在该器官中表达的基因富集水平显著高于野生型,分析每个器官富集的基因集,以确定相对于野生型突变体组织中显示显著过表达的基因的比例。这种方法近似于以前使用微阵列数据的方法;尽管如此,与基于表达相关性的野生型MPSS数据分析相比,之前的微阵列分析与我们基于阈值的MPSS方法之间发现了更显著的重叠。测定了器官富集基因相对于野生型差异表达的比例(图2额外的文件1).器官富集基因差异表达的平均比例(p < 0.05)为65.10%±24.12% (95% C.I.)。在差异表达基因中,大多数在预期突变体内过表达(81.20%±19.77%,95% C.I.)。因此,我们的分析并没有大幅增加检测到的基因数量,这些基因揭示了相对于野生型花序的显著增加。然而,那些表现出差异表达的花器官富集基因在积累相应器官生物量的突变体中大量过表达(图2).
为了进一步表征器官富集基因集的表达模式,我们测定了空间基因集的时间表达。尽管空间拟南芥表达的独立微阵列分析与我们的器官富集基因集之间没有发现多少共同点,但本研究积累的空间数据被叠加在genevetragator在线微阵列数据库中识别的累积时间数据上,以描述我们的器官富集基因集的时间表达[20.].多数MPSS确定花器官富集表达的基因在花发育后期10%花蕾开放后(种植后36天)被检测到。因此,我们的器官富集分析可能倾向于检测更多在发育的第12阶段附近表达的基因,在所有花器官都已发育出显著生物量之后。
器官富集基因集的功能表征
基于基因本体论,进一步对预测的器官富集表达基因进行了表征独联体-元素保存,以进一步了解支持器官发生和特定花器官维持的那些因素的性质,并确定第四系复合体在激活器官富集基因中的相对影响。使用几个公开的数据挖掘工具确定了调控和功能。将具有共同调控因子或功能的器官富集基因的丰度与全基因组平均表达水平进行比较,以确定与每个花器官相关的基因中过度代表的特征。
基因功能富集的描述是使用公开的分析工具EasyGO实现的[29].在基因集中发现了详细描述特定分子功能以及与细胞成分关联的术语的富集。在大多数器官富集的基因集中,很少有富集的词被发现,具有雄蕊富集表达的基因在脂肪酶活性(p < 0.01)和存在于膜内系统(p < 0.01)上被富集。脂肪酶的有缺陷的花药裂开已被证明可以催化茉莉酸途径中调节花开放、花粉成熟同步和花药开裂的初始步骤[30.].因此,有可能是其他的脂肪酶基因参与了类似的途径,以调节雄蕊成熟。
为了评估四元复合体中MADS-box蛋白在调节器官富集基因表达方面的作用,采用了几种算法来识别过度表达的基因独联体-每组基因中的元素。使用多伦多大学的启动子2程序以500和1000 bp的间隔扫描每个基因周围的上游和下游区域,以寻找过表达基序[31以及利兹大学和拟南芥基因调控信息服务器的已知独联体元分析仪(32].在任何一组花器官富集表达的基因中都没有显著过代表的基序。这证实了之前的微阵列功能评估,并显示在器官富集的基因集中没有显著的CArg box富集。因此,许多著名的器官丰富的表达不是由一个单一的共通物介导的独联体-调控因素,如假设的第四元络合物对CArg箱的作用[3.在花发育的后期。尽管如此,超过200个miRNAs的表达已经通过MPSS在野生型花的花序中被描述出来[33],很可能转录后调控在限制器官富集表达方面起着重要作用,其方式与miR172和AP2[27].未来的分析特征mirna在反表达模式中确定的表3.可以检测到那些负调控负责花器官维持和发育的基因表达的miRNAs。
验证实验一:MPSS与微阵列分析的对应
使用MPSS数据的阈值过滤方法生成的数据集与之前的几个微阵列研究进行比较,以揭示分析的并发性。先前的分析比较了同源突变基因表达相对于野生型发现了显著的重叠。在被MPSS鉴定为雄蕊富集的基因中,约有70%(111个基因)与之前的微阵列分析一致;然而,在之前的研究中确定的假定雄蕊富集基因的数量是我们分析中确定的7倍多。这种差异可能是由于筛选的严格程度不同,而不是由于雄蕊富集基因的实际数量不同。MPSS分析只确定最可能的器官富集候选者,并可能具有较高的假阴性率。仅基于缺乏雄蕊突变体中表达缺失的放松MPSS过滤标准显示了类似数量的雄蕊富集基因。然而,在宽松的参数下与之前的分析一致的器官富集基因的比例大大减少到约26%(267个基因)。
分离花器官的微阵列分析[16和花粉[34]揭示了雄蕊丰富的数据集中有25个基因重叠。此外,通过MPSS和解剖花器官微阵列结果的交叉识别的所有25个雄蕊富集基因[16与之前基于同种异体突变体比较的微阵列分析一致[10].这明显高于两种微阵列研究中发现的雄蕊富集基因的比例(约49%)。几种已知的雄蕊丰富的和Apetala3调节基因,包括Profilin5,Arabinogalactan6,Cyp703A2,Anther7,胼胝质Synthase5,夭折的小孢子,Taptetum1,脂质Transfer12,Anther27,在微阵列和MPSS分析中被识别。然而,在所有研究中发现的富含雄蕊的基因中,有近一半具有推定的或未知的功能,包括与一种推定的自交不亲和基因At5g26060、一种推定的磷脂酶A2(参与调节花粉成熟的茉莉酸途径)相关的基因,以及几个已知在雄蕊中具有高表达的富含甘氨酸蛋白家族的基因。
在雌蕊花、花瓣和萼片花瓣的器官富集的基因集之间注意到的共性较少(见附加文件)1).在先前的微阵列器官富集研究中,只有15%(10个基因)由MPSS确定的富含雌性激素的基因被识别出来[16].尽管如此,一些已知的富含雌性激素表达的基因被MPSS表征,这些基因在之前的微阵列分析中没有被识别,包括三个胚胎缺陷而且胚胎缺陷例如基因。通过微阵列分析,mpss识别的花瓣表达基因集仅共享两个基因[16].同样,在一组萼片/萼片-花瓣定位转录本中,只有5个基因被鉴定为微阵列分析和MPSS预测的共同基因[10,16].
MPSS与微阵列之间的相似性显著降低[10]预测的基因在雌雄异症和营养器官的富集表达表明,在确定这些器官的基因富集方面,同源突变体的比较在一种或两种分析中都不如雄蕊的比较准确。然而,尽管本研究发现生殖器官转录组多样性水平相对相似,但之前的微阵列分析发现雄蕊中表达丰富的基因明显多于雌蕊。
验证实验2:原位杂交过程
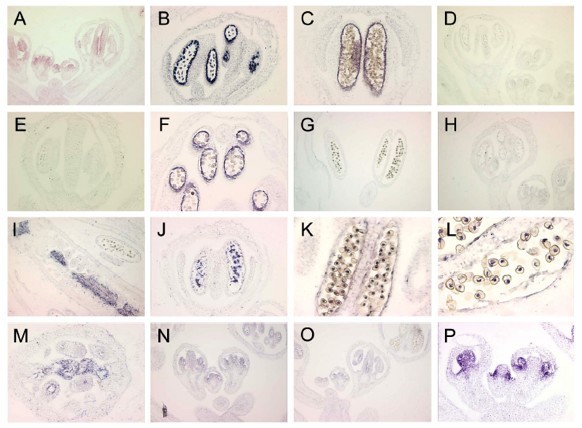
在我们严格的和更宽松的(存在与不存在)参数下确定的假定器官富集基因的先前未描述的表达被经验验证通过原位杂化。半乳糖基转移酶家族蛋白At1g33430的表达被MPSS标准确定为在雄蕊和心皮原基中表达,特别是在绒毡层和小孢子中强烈表达[图]3得了(反义)3 d, e(感觉)]。我们的MPSS数据松弛过滤系统预测At1g72290在雌蕊和雄蕊中都有表达。然而,原位杂交只显示在心皮内表达[图]3米(反义)],雄蕊内无表达。同样,At2g19070通过MPSS参数的放宽预测定位在花瓣-雄蕊内。原位杂交模式显示它只存在于绒毡层中;然而,鉴于MPSS在富含花瓣的突变体中预测的表达水平较低,严格的过滤将转录本从表达集中删除(图)3做减法(反义),3 h(感觉)]。使用宽松的参数,我们发现一个假定的雄蕊富集基因At2g42940在心皮和雄蕊原基中都有表达(图)3 n(反义),3 o(感觉)]。因此,一些为雄蕊或雌蕊富集的基因可能在两个生殖器官中表达;然而,它们也不太可能出现在营养组织中。
实证验证通过原位杂交.(两者)At1g33430(反义探针),(d e(感控探针)在雄蕊和心皮原层有信号,MPSS预测绒毡层和小孢子有较强的表达。(做减法)At2g19070(反义探针),(H)(感觉控制探头)信号在绒毡层上被识别。用MPSS预测雄蕊和花瓣的局部表达(i j)At1g54860(反义探针)信号存在于柱头乳头、中隔和发育中的小孢子上。通过MPSS分析预测其雄蕊富集。(K-L)At5g59810(反义探针)瞬态信号在四分体的一个特定小孢子上记录。MPSS预测心皮富集。(M)中隔检测到At1g72290(反义探针)信号。通过MPSS预测定位在雄蕊和心皮内。(N)At2g42940(反义探针)(O)(感控探头)雄蕊和心皮原端信号微弱。经MPSS鉴定为一份雄蕊丰富的转录本。(P)AP3(反义控制探针)。
在更严格的过滤参数用于描述器官富集基因集,一个功能未知的基因,At1g54860的表达被描述为雄蕊富集。原位杂交发现小孢子内表达强,而柱头乳头和中隔内表达较少[图]3 i j].这意味着增加过滤参数可以提高器官富集分析的准确性。然而,严格的MPSS和原位在推测的富含女性的at5g5910中记录了数据。该基因编码一种枯草酶家族蛋白,在四分体的单个小孢子的雄蕊内被严格识别原位杂交(图3 k-l].尽管如此,之前的文章已经通过一些微阵列分析、RNA凝胶印迹和标记揭示了At5g59810的柱头富集表达原位分析。
验证实验三:GUS组织化学分析
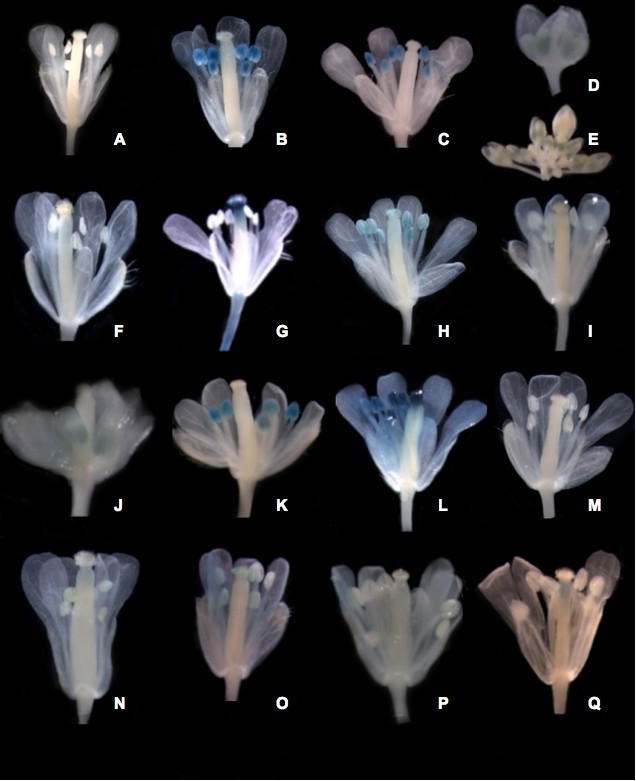
用转基因GUS报告系作为评价花器官定位分析准确性的另一手段。在几个可能富集器官的基因上游1.5 kb区域用于驱动GUS报告基因的表达。启动子:有两个或两个以上独立转化事件的GUS构建物通过组织化学分析进行表征。相同结构的转换事件之间没有空间表达差异。MPSS使用宽松的过滤参数预测的大部分器官富集表达反映在被分析启动子的gus染色活性中(图4).通过三个基因的启动子融合来评估雄蕊富集的表达。结果发现,At1g20130、At2g42940和At3g27025的启动子都能如预期的那样使雄蕊染色(图4中).然而,在At2g42940的情况下,原位分析显示心皮原基有额外的表达。另外还检测了两个可能表达丰富的雌性异位症基因。染色后,我们发现At1g07370和At1g27900的启动子:GUS融合物如预期的那样在雌蕊畸形中染色(图4做减法).MPSS预测At1g33430在两个生殖器官中均有表达。这一结果通过组织化学分析得到证实,在雄蕊和雌蕊中都发生了染色(图4 h).另外两个用MPSS预测的在雄蕊和花瓣中都有表达的基因也进行了评估。证实了我们之前的原位杂交结果显示,At2g19070只在雄蕊中表达4我).尽管存在这种差异,但研究发现,编码富含甘氨酸的油苷蛋白(GRP19)的基因At5g07550的表达可以调节花粉被膜内脂滴的大小和特征[35]在雄蕊和花瓣中都得到了证实,正如预测的那样(图4 j).该基因在花瓣中的表达在以前的文献中没有记录,可能表明额外的无特征的活性;五个独立的转换事件揭示了类似的表达模式(数据未显示)。At1g26270存在于所有花MPSS文库中,正如启动子:GUS融合所预期的那样,染色在所有花器官中普遍存在(图4 l).同样,我们对At2g35340进行了检测,结果证实了MPSS对花序组织内无表达的预测4米)尽管在花组织中已有微阵列表征[20.].
启动子的经验验证:GUS融合.(一)野生型控制。(中)At1g20130[B], At3g27025[C]At2g42940 ([D]而且[E]).MPSS-predicted stamen-enriched启动子:格斯.(做减法)At1g07370[F], At1g27900[G].MPSS-predicted carpel-enriched启动子:格斯.(H)At1g33430。MPSS-predicted雄蕊/ carpel-enriched启动子:格斯.(i j)At5g07550[我], At2g19070[J] ..MPSS-predicted雄蕊/ petal-enriched启动子:格斯.(K)At1g07930。MPSS冲击-ap3相对于其他突变体表达丰富。(左)At2g35340。mpss预测了花的表达缺失。(M)At1g26270。mpss预测了无处不在的植物表达。(N)At1g68200。MPSS冲击-ap1唯一的表情。(O)At2g43100。MPSS冲击-ap3相对于其他突变体表达丰富。(P)At3g15160。MPSS预测缺位一口ap1突变体。(问)At52900。MPSS冲击-ap3而且ag)丰富的表情。
虽然在我们的MPSS筛选参数中不存在,但相对于其他花突变体而言,在特定突变体中表达丰富的几个基因也被描述为我们筛选参数和MPSS方法的对照。与MPSS的预测相反,At1g07930基因在无雄蕊植株中表达5tpm非常弱ap3突变体,雄蕊内无表达一口ap1在雄蕊内发现了突变体(图4 k).表达在心皮和雄蕊丰富ap1At1g68200和预期的一样存在于心皮中4 n).的ap3尽管只有1个TPM在高度富集的花瓣中几乎检测不到表达水平,但发现在心皮和花瓣中表达丰富的At2g43100无性生殖的突变体(图4 o).经MPSS鉴定,At3g15160在所有花器官中均有表达一口ap1在高于10个TPM的级别;实验上,在花瓣和雌蕊中检测到表达,但在雄蕊中没有(图4 p).GUS染色显示了At3g52900的心皮特异性活性,并且在心皮富集的细胞中也被鉴定为高表达ap3通过MPSS分析,尽管在心皮缺失中也注意到显著表达(9tpm)无性生殖的突变体。这些结果表明,单个突变体中表达的缺失或存在,在富集器官中检测表达模式时并不准确;然而,对所有四种突变体的分析提高了检测能力。
除了完整的雌蕊启动子:GUS融合和基于突变体富集的比较外,在五个包含胚珠的基因中鉴定出了胚珠表达,并通过放松的MPSS参数预测雌蕊富集。在胚珠中发现了一个功能未知的基因At1g05550(图1)5).At5g24420在胚珠的珠柄和两个珠被内被鉴定,证实了基于mpss的对胚珠表达的预测(图5 b).At5g49180在珠被和珠柄中均有表达;然而,它在雌性配子体中没有表达(图5度).编码F-box蛋白的基因At3g06240的转录活性在倒生胚珠脊和背侧外胚珠的一小部分区域被发现(图5 d),但在其他完全分化的花器官中没有(数据未显示),证实了我们的MPSS分析预测的富含胚珠的表达模式。At1g27330被鉴定为表达于珠孔极和珠珠珠被以及珠心合点区(图5 e).
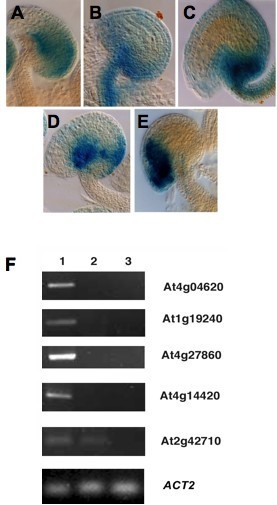
启动子赋予的表达模式:uidA融合和与胚珠表达丰富的基因对应的反转录PCR.启动子:uidA融合。除At2g47470在心皮壁和柱头中表达外(数据未显示),其他花器官(萼片、花瓣、雄蕊和雌蕊)均无GUS表达。(一)At1g05550;GUS表达于珠被和珠心,在合点区。(B)At5g24420;GUS在珠柄和珠柄中表达。(C)At5g49180;GUS在珠被和珠柄中均有表达,但在雌配子体中不表达。(D)At3g06240;GUS表达于倒伞形皮脊和背侧外被皮的一小部分区域。(E)At1g27330: GUS在珠孔极和两个珠被中表达。(F)预测在胚珠中特异表达的基因的RT-PCR表达。从单个花器官中提取总RNA,用于逆转录酶PCR分析。Lane 1:胚珠完全分化;巷2:花瓣;巷3:萼片。扩增片段尺寸:At4g04620 (231 bp);At1g19240(250个基点);At4g27860(235个基点);At4g14420(209个基点);At2g42710(223个基点)。
验证实验四:逆转录PCR验证
通过反转录PCR对5个可能表达雌雄症的基因进行MPSS器官富集过滤器的验证5 f).表达模式被发现与MPSS预测的相关。自噬微管相关蛋白ATG8b的编码基因At4g04620在胚珠中如MPSS预测的那样有表达,而在萼片或花瓣器官中没有表达。ATG8的这种亚型先前已在微阵列研究中被鉴定过[36]表明花瓣组织中花的表达水平最高,其次是雄蕊,在萼片和心皮组织中较少;然而,MPSS分析显示女性异位包括胚珠丰富表达。通过反转录PCR证实了MPSS对雌二醇富集的预测。在花瓣和萼片组织中未检测到At4g04620的表达,这表明At4g04620的独特替代转录本在胚珠(V. Pérez-España和j . ph .)中特异性表达。Vielle-Calzada,未发表的结果)。此外,逆转录PCR还发现了At4g27860、At1g19240和At4g14420的表达。在这些分析中没有发现萼片或花瓣表达的转录本。与MPSS的预测相反,反转录PCR发现了At2g42710的表达,这是胚珠和花瓣组织中大核糖体亚基的推定结构成分。这些实证分析表明,通过我们的分析,器官富集被准确地识别出来;然而,为了描述表达对单个器官的特异性,有必要采用元分析方法进一步过滤转录本,获得更健壮的数据集。
结论
我们的MPSS花转录组分析分析了额外的表达数据,以进一步证实和补充现有的基因表达空间分析使用微阵列。在我们的转录本过滤系统中,许多具有已知表达模式的特征良好的基因被准确地解剖为器官富集基因。此外,验证实验与先前未描述的几个基因的MPSS表达数据基本一致。与先前的分析一致[10],与营养组织相比,生殖结构具有最多样化和最复杂的转录组,并且在推测的器官富集基因的上游或下游没有发现明显的CArG box富集,这表明花器官的发生和维持需要大量的信号级联,而不是广泛的直接独联体-由四元复合体调控。转录本表达的空间解剖将为未来的功能研究提供有价值的参考,并为开发植物器官富集启动子驱动转基因表达提供了一个来源。此外,这些启动子可以耦合到最近描述的系统中,通过荧光激活分选分离特定的细胞类型[37],其次是更先进的基因表达分析方法,如Illumina的测序by Synthesis;比MPSS灵敏度高十倍的标记测序平台[38].这将使在未来的植物研究中基因表达的更详细的空间分析成为可能。
方法
植物材料,组织收集,核酸分离
用于MPSS和逆转录PCR分析的所有植物材料均来自拟南芥生态型Columbia-0.植株在Pro-mix土壤中生长,光照16小时,22°C,湿度60%,生长5周。这些花组织包括花序分生组织以及与前12个发育阶段相对应的花芽[27].在蛭石和珍珠岩的无菌条件下,在16小时光照下生长21天,获得相同植物的叶和根组织。一种允许胚珠分离和收获的自动微吸入器被用来收集胚珠样品(M. Arteaga-Vazquez, M. Arteaga-Sanchez M.和j . p。Vielle-Calzada unpubl。结果)。用于反转录PCR分析的萼片和花瓣组织手工解剖ag)花序。所有组织样本在天黑后2小时内采集,在-80°C冷冻后提取核酸。答:芥生态型Columbia-0用于转化启动子的植物:GUS融合质粒在相同的条件下光照16小时生长5周,然后接种花根癌土壤杆菌.
花组织利用在原位杂交验证由拟南芥的兰茨贝格erecta生态型。这主要是由于花的花序的大小比植物大Columbia-0相对的。植物在光照、温度和湿度条件与创建MPSS库时种植植物的条件相似的生长室内生长。
用于MPSS cDNA的RNA,原位使用TRIzol (Invitrogen)试剂和制造商的方案分离RT-PCR验证。用DNeasy Minispin柱提取试剂盒(Qiagen)获得用于扩增启动子序列的基因组DNA。
签名测序和基因组对应
按前面所述执行MPSS [22,23].每个花库的签名都是通过多次测序和两种不同类型的测序反应产生的[22,25];将这些测序运行和反应结合起来,计算在每个花、根和叶MPSS库中观察到的每个特征的单一规范化丰度[25].所有原始和标准化的签名数据均已在网页界面公开提供[26].这些特征与它们各自的基因座相匹配答:芥基因组序列。简单地说,潜在的MPSS签名是由所有可能的计算推导出来的DpnII基因组内限制位点(GATC)和13个相邻碱基。位于外显子、内含子、外显子-内含子剪接边界的感觉链上,或存在于注释ORF 3'端500 bp内的潜在MPSS特征,与经验推导的MPSS序列进行匹配,以确定相应基因或伪基因的表达水平。
MPSS文库过滤,花表达交叉分析,分类
本研究中实现的所有MPSS库都使用“可靠性”过滤器进行过滤,以去除潜在的错误签名并区分有效表达式级别的子集。该过滤器消除了所有当前表达式库中仅在单个序列运行中识别的所有签名。MPSS文库中使用的每个组织至少对应四个不同的测序运行。因此,在任何其他运行中未识别的签名很可能是随机MPSS测序错误的结果,据估计,这种错误的发生率为每碱基约0.25% [25].
一旦积累了可靠的MPSS表达数据,与花组织对应的文库指定为“1”(器官存在)或“0”(器官不存在)来划分同种异体花突变体中器官的出现情况(表2)3.).为了分离特定器官内表达的基因子集,通过使用我们的公共可用文库分析(LIBAN)接口,将每个基因表达对应的归一化签名数据与每个器官发生谱匹配。在缺乏该特定花器官的突变体中,花器官特异性表达的基因逻辑上具有0转录本/百万分测定(TPM)的规范化转录水平。相比之下,那些具有特定花器官的同源突变体,其转录本的规范化表达率大于4tpm,并且在花序文库中的表达率高于叶片或根MPSS文库中的表达率。
一旦确定了具有器官特异性表达的基因子集,就可以根据它们在突变体中的表达水平对其进行进一步的排序。尽管这对高表达基因的数据有偏差,但这允许在MPSS预测器官特异性表达方面建立一个相对的置信度。例如,假定的雄蕊特异性转录本首先根据其表达水平在超人/ ap1突变体,然后通过他们内部的表达水平ap1(它也会过度表达雄蕊),最后是通过野生型表达水平。
为了以相对和交叉的方式确定本研究中所测定的花组织表达的全基因组相关性,通过合并两个文库转录组,将相对于另一个文库表达未显示显著(p < 0.05)改变的基因的比例归一化。从更定性的阈值角度来看,基于我们的4个TPM阈值和花序富集的每个文库中确定的基因通过花序文库的联合进行归一化。相对于野生型的过度表达或表达不足,使用转录本每百万次二项式比例的正态近似检验(z检验)来确定[39].该分析使用Microsoft Excel和SAS JMP v. 7.0进行。
原位杂交过程
原位在切片上进行杂交兰茨贝格erecta(l)的组织。使用改进的方案进行组织固定、切片和包埋[40].反义探针和对照感探针来源于基因组的以下区域:At1g33430 (5'-CGGGGAAAGCCATAATAGTGC—CAACAGCAACTGCAATG-3');At2g19070 (5 ' -AAAAAGAAAGGGGGTTTGTGTT——ACTTCGGCAATGCTACTCTTG-3 ');At1g54860 (5 ' -TGCCTATCGCTTAATTCTGCTT——GATTGTTGTGTTTTTGTGTGAA-3 ');At5g59810 (5 ' -GACGTAAGCCCATGGTTGATGA——CGTTAGGAGTCCCATCGTCGTC-3 ');At1g72290 (5 ' -GAGAGTAAAAACGGAGGTGGTC——CTTGAGAAAACATTGATCA-3 ');At2g42940(5“-TGTTGCAGGTACAAACTACAAA——ACAGGGACCAGATGCGATTAG-3”)。这些区域被扩增并克隆到pGEM-T中;之后,通过从T7和SP6启动子位点测序确定它们的方向。使用制造商的方案(Boehringer Mannheim)进行地高辛探针合成、抗体检测和染色。 The slides were dehydrated using ethanol and xylene, and were mounted in Per-mount (Fisher Scientific). Sections were photographed through a Leitz DRB (Leica, Wetzlar, Germany) light microscope using Kodak Ektachrome 160 ASA film.
启动子:GUS融合和组织化学分析
为了构建非胚珠启动子:GUS载体,从基因组DNA中扩增每个起始密码子上游约1.5 kb的启动子片段答:芥,Col-0.采用网关克隆技术(Invitrogen)将扩增子插入pDONR221,最终插入二进制表达载体pK2GWFS7,在pK2GWFS7中使用启动子驱动报告基因β -葡糖苷酸酶的表达。用于片段扩增和质粒重组的引物为:At1g20130 (S5'-GGGGACAAGTTTGTACAAAAAAGCAGGCTTAGGATTTATTGGTGTTTCTC-3'和AS5'-GGGGACCAGTTTGTACAAGAAAGCTGGGTTGGCCACGGCTGTGGATACG-3');At3g27025 (S5'-GGGGACAAGTTTGTACAAAAAAGCAGGCTTATATCTTCGATATCATCCAA-3'和AS5'-GGGGACCAGTTTGTACAAGAAAGCTGGGTGAATGATTAGTTTATGAGAGA-3');At2g42940 (S5'- ggggacaagtttgtacaaaaaagcaggctcagcaactctgacaggccc -3'和AS5'-GGGGACCAGTTTGTACAAGAAAGCTGGGTTGTTATGAATGTTGTTATATG-3');At1g07370 (S5'- ggggacaagtttgtacaaaaaagcaggcttcaacaaagtcaaacatacagag -3'和AS5'- ggggaccagtttgtacaagaaagctgggttcgtcttagatattatag -3');At1g27900 (S5'-GGGGACAAGTTTGTACAAAAAAGCAGGCTACAAAATCAAGTGGGTTATTC-3'和AS5'-GGGGACCAGTTTGTACAAGAAAGCTGGGTATCGCAGAGAACACTCAAAGAACC-3');At1g33430 (S5'- ggggacaagtttgtacaaaaaagcaggcttgtaatcatatgttttagagc -3'和AS5'-GGGGACCAGTTTGTACAAGAAAGCTGGGTCTCCGCGCCTTAGTGC-3');At5g07550 (S5'-GGGGACAAGTTTGTACAAAAAAGCAGGCTGTATGCATGCGCACACAAGCC-3'和AS5'-GGGGACCAGTTTGTACAAGAAAGCTGGGTTGGTGGGAAGAAGTGGGG-3');At1g19070 (S5'-GGGGACAAGTTTGTACAAAAAAGCAGGCTCGTGAACAAAGGATTACG-3'和AS5'- ggggaccagtttgtacaagaaagctgggtaacacaaacccccttctt -3')。 Binary vectors were transformed into农杆菌属tumefacieriens应变gc101.先前描述的浸渍方法[41]被用来开发转基因拟南芥,在卡那霉素上选择。2 - 5个独立的T1植株分别携带每个GUS:启动子结构,进行组织化学分析以测定GUS活性。转基因和野生型花组织分别在GUS试验缓冲液(H2O, 0.1 M NaH2阿宝4, 10 mM Na2EDTA。0.5 K3.铁(CN)6, 0.1% Triton X-100和0.3% 5-溴-4-氯-3-吲哚基- β - d -葡醛酸酯(X-Gluc),在37℃孵育12 h。在22℃连续乙醇洗涤叶绿素去染色。鉴定并固定GUS染色区域。照片是用AxioVision数码相机(蔡司)拍摄的,并用GNU图像处理程序GIMP编译。
为了构建启动子:与胚珠特异性候选基因相对应的GUS载体,使用每个基因的特异性引物,通过PCR扩增启动子片段,经消化后插入pBI101质粒BamH我和后III (At1g27330和At5g24420),后三世和萨尔我(At1g05550),或萨尔我和BamHI (At5g49180和At2g47470)。用于片段扩增的引物为:At1g27330 (S5'-GCAAGCTTGGACAGGGAAGAGAGCAT-3'和AS5'-GCGGATCCACTAGTTGCGGTCCTGAT-3');At1g05550 (S5'-GCAAGCTTGATTGGGCCATCTCTTTTC-3'和AS5'-GCGTCGACGCTCCACCTCATCTTGAAGG-3');At5g49180 (S5'-GCGTCGACTGGGTTTTGTTTCCTTCAGTG-3'和AS5'-GCGGATCCGTTGAACTCTCCGAGAAGG-3');At5g24420 (S5'-GCAAGCTTGTCGTCGTCAGAGACCTTG-3'和AS5'-GCGGATCCTCTCATCGACCCAAAAGA-3');At3g06240 (S5'-CAAAGAGCGAATTTCTCGGCTAC-3'和AS AS5'-TCTCTTGGAATCTCCGGTAGTTG-3')。植物转化时,将载体转入根癌土壤杆菌ASE[65]。在野生类型上执行转换兰茨贝格erecta或Col-0通过花倾角变换[64]。从T1启动子:GUS转化子获得的幼苗在MS加50 μg/ml卡那霉素培养基中选择。摘除女性异位后进行GUS组织学检查[66]。显微观察采用DRM徕卡显微镜和Nomarsky光学。
逆转录PCR验证实验
使用5 μg总RNA和Superscript II逆转录酶(Invitrogen)合成cDNA;反转录PCR条件为:1 μg cDNA, 1 mM正向和反向引物(见下),0.6单位Taq DNA聚合酶(Invitrogen), 1 mL 10× PCR Buffer (Invitrogen)。所用引物为:At4g04620 (S5'-AAGAGTTCCCGTGATTGTGG-3'和AS5'-AACCCGTCTTCGTCTTTGTG-3');At1g19240 (S5'-CCTAATTGATCGGCCAGAAA-3'和AS5'-GACAAAGAAAACAGCGCACA-3');At4g27860 (S5'- ggtgaggaaccgagcaga -3'和S5'-AGAGAAGCAACACCGCAGAT-3');At4g14420 (S5'- ttgtctttggcagctcat3 '和AS5'-CCTGAGTTGCCTACCGTGTT-3');At2g42710 (AS5'-AAGACGCAAAAGCTGGACAT-3'和5'-GGATAACCCTTTCCCATCGT-3')。扩增包括30个周期,94°C 30秒,60°C 30秒,72°C 32秒。
参考文献
张晓燕,张晓燕,张晓燕,等:拟南芥花同源基因的遗传互作。科学通报,2001,30(1):1-20。
Coen ES, Meyerowitz EM:轮生的战争:控制花发育的基因相互作用。自然学报,1991,353(6339):31-37。10.1038 / 353031 a0。
Egea-Cortines M, Saedler H, Sommer H: MADS-box蛋白SQUAMOSA, dwarens和GLOBOSA之间的三元复合体的形成参与了反rrhinum majus花结构的控制。中国生物医学工程学报,1999,18(19):539 - 539。10.1093 / emboj / 18.19.5370。
MADS-box蛋白复合物足以将叶片转化为花器官。自然学报,2001,40(6):529 -529。10.1038 / 35054083。
Tonaco IA, Borst JW, de Vries SC, Angenent GC, Immink RG: MADS-box转录因子相互作用的体内成像。实验植物学杂志。2006, 57(1): 33-42。10.1093 / jxb / erj011。
Soltis DE, Soltis PS, Albert VA, Oppenheimer DG, dePamphilis CW, Ma H, Frohlich MW, Theissen G:缺失的环节:花的遗传结构[花的修正]和花的多样化。植物科学发展趋势。2002,7(1): 22-31。10.1016 / s1360 - 1385(01) 02098 - 2。讨论中31-24
顾强,Ferrandiz C, Yanofsky MF, Martienssen R: FRUITFULL MADS-box基因在拟南芥果实发育过程中介导细胞分化。开发(英国剑桥)。中国科学:d辑,2004,15(4):359 - 361。
Ito T, Wellmer F, Yu H, Das P, Ito N, Alves-Ferreira M, Riechmann JL, Meyerowitz EM:同源蛋白AGAMOUS通过调控无孢子细胞(SPOROCYTELESS)控制小孢子发生。自然学报,2004,43(6):356-360。10.1038 / nature02733。
Sablowski RW, Meyerowitz EM: NO顶端分生组织的同源物是花同源基因aptala3 /雌蕊分生组织的直接靶点。细胞学报,1998,92(1):93-103。10.1016 / s0092 - 8674(00) 80902 - 2。
王晓燕,李晓燕,李晓燕。拟南芥花空间基因表达的全基因组分析。植物细胞。2004, 16(5): 1314-1326。10.1105 / tpc.021741。
Yamada K, Lim J, Dale JM, Chen H, Shinn P, Palm CJ, Southwick AM, Wu HC, Kim C, Nguyen M,等:拟南芥基因组转录活性的实证分析。科学(纽约,纽约)。地球物理学报,2003,29(4):644 - 644。
Schmid M, Davison TS, Henz SR, Pape UJ, Demar M, Vingron M, Scholkopf B, Weigel D, Lohmann JU:拟南芥发育的基因表达图谱。自然遗传学。2005,37(5): 501-506。10.1038 / ng1543。
Rubinelli P,胡勇,马红:拟南芥花药特异新基因的鉴定、序列分析及表达研究。植物分子生物学。1998,37(4): 607-619。10.1023 /: 1005964431302。
Masayuki A, Tohru A, Makoto E, Katsunori H, Chikara K, Daisuke S, Kinya T, Masao W:利用拟南芥宏阵列和甘蓝的mRNA组合分析十字花科模式植物拟南芥的花药特异性基因植物有性生殖学报,2003,15(5):213-220。
爱尔兰VF: aptala3和雌蕊花同源基因作用调控的靶基因的全球鉴定。植物细胞。2003,15(1): 207-222。10.1105 / tpc.006353。
马琳,孙妮,刘旭,焦莹,赵红,邓晓伟:拟南芥发育过程中基因组的器官特异性表达。植物生理学。2005, 38(1): 1 - 8。10.1104 / pp.104.054783。
Cohen BA, Mitra RD, Hughes JD, Church GM:全基因组表达数据的计算分析揭示了基因表达的染色体域。自然遗传学。2000,26(2): 183-186。10.1038/79896。
韦默F, al维斯- ferreira M, Dubois A, Riechmann JL, Meyerowitz EM:拟南芥花发育早期基因表达的全基因组分析。公共科学图书馆遗传学。2006, 2 (7): e117-10.1371/journal.pgen.0020117。
Birnbaum K, Shasha DE, Wang JY, Jung JW, Lambert GM, Galbraith DW, Benfey PN:拟南芥根的基因表达图谱。科学(纽约,纽约)。中国农业科学,2003,29(5):561 - 561。
Zimmermann P, Hirsch-Hoffmann M, Hennig L, Gruissem W:基因研究者。拟南芥微阵列数据库和分析工具箱。植物生理学。中国科学(d辑:自然科学版),2016,38(1):1- 6。10.1104 / pp.104.046367。
拟南芥全局基因表达测量技术的比较。比较功能基因组学,2004,14(8):1641-1653。
Brenner S, Johnson M, Bridgham J, Golda G, Lloyd DH, Johnson D, Luo S, McCurdy S, Foy M, Ewan M,等:微珠阵列上大规模并行签名测序(MPSS)的基因表达分析。中国生物工程学报,2000,18(6):630-634。10.1038/76469。
美国国家科学研究院。中国科学:地球科学,2000(4):369 - 369。10.1073 / pnas.97.4.1665。
Meyers BC, Lee DK, Vu TH, Tej SS, Edberg SB, Matvienko M, Tindell LD:拟南芥MPSS。定量表达分析的在线资源。植物生理学报,2004,25(2):366 - 366。10.1104 / pp.104.039495。
Meyers BC, Tej SS, Vu TH, Haudenschild CD, Agrawal V, Edberg SB, Ghazal H, Decola S: MPSS在拟南芥全基因组转录分析中的应用。基因组研究。2004, 14(8): 1641-1653。10.1101 / gr.2275604。
Meyers BC, Lee DK, Vu TH, Tej SS, Edberg SB, Matvienko M, Tindell LD:拟南芥MPSS。定量表达分析的在线资源。植物生理学报,2004,25(2):366 - 366。10.1104 / pp.104.039495。
Smyth DR, Bowman JL, Meyerowitz EM:拟南芥早期花发育。植物生态学报,2004,25(4):366 - 366。10.1105 / tpc.2.8.755。
Mandel MA, gustaf孙- brown C, Savidge B, Yanofsky MF:拟南芥花同源基因APETALA1的分子特征分析。自然学报,1992,36(6):773 - 776。10.1038 / 360273 a0。
EasyGO:基于基因本体论的农艺物种标注和功能富集分析工具。中国生物医学工程学报,2007,29(4):366 - 366。
Ishiguro S, Kawai-Oda A, Ueda J, Nishida I, Okada K: IN ANTHER DEHISCIENCE基因缺陷编码了一种新的磷脂酶A1,它催化茉莉酸生物合成的第一步,在拟南芥中同步花粉成熟、花药开裂和花开放。植物生态学报,2004,13(10):2191-2209。10.1105 / tpc.13.10.2191。
Toufighi K, Brady SM, Austin R, Ly E, Provart NJ:植物阵列资源:E - northern,表达角度,和启动子分析。植物学报,2005,43(1):153-163。10.1111 / j.1365 - 313 x.2005.02437.x。
Manfield等人:拟南芥共表达工具(ACT):基于微阵列的基因表达分析的web服务器工具。核酸研究,2006,504-509。10.1093 / nar / gkl204。34个Web服务器
Nakano M, Nobuta K, Vemaraju K, Tej SS, Skogen JW, Meyers BC:植物MPSS数据库:用于mRNA和小RNA分析的基于签名的转录资源。核酸学报,2006,34 (supply _1): D731-735。10.1093 / nar / gkj077。
拟南芥花粉转录组的比较分析。植物生理学报,2004,24(2):366 - 366。10.1104 / pp.103.020925。
Mayfield JA、Fiebig A、Johnstone SE、Preuss D:拟南芥花粉被膜蛋白质组的基因家族。科学通报,2001,29(5):359 - 359。10.1126 / science.1060972。
Thompson AR, Doelling JH, Suttangkakul A, Vierstra RD:拟南芥ATG8和ATG12结合通路介导的营养物质自噬循环。植物生理学报,2004,24(4):378 - 378。10.1104 / pp.105.060673。
Birnbaum K, Jung JW, Wang JY, Lambert GM, Hirst JA, Galbraith DW, Benfey PN:利用荧光报告系的原生质体进行细胞类型特异性表达分析。自然的方法。科学通报,2005,2(8):615-619。10.1038 / nmeth0805 - 615。
徐婷婷,白晓霞,金德华,石松,李志勇,李志勇,朱俊:利用光裂解荧光核苷酸芯片合成四色DNA序列。美国国家科学研究院。中国农业科学,2005,29(5):359 - 359。10.1073 / pnas.0501965102。
Reinartz J, Bruyns E, ching - zhong L, Burcham T, Brenner S, Bowen B, Kramer M, Woychik R:大规模并行签名测序作为所有生物基因深度定量表达的工具。功能基因组学与蛋白质组学进展。2002,1(1):95-104。10.1093 / bfgp / 1.1.95。
Meyerowitz EM:拟南芥。遗传学年度回顾。1987年,21日:93 - 111。10.1146 / annurev.ge.21.120187.000521。
明hsan C, Ming-Kung C, shumei P:花喷雾转化能高效生成拟南芥。转基因研究进展,2000,9(6):471-486。10.1023 /: 1026522104478。
确认
本研究部分由NSF植物基因组研究奖#0110528 (B.C.M.)和NSF本科生研究经验奖(REU)资助。我们感谢Kan Nobuta博士的生物信息学援助和建议,也感谢Meyers实验室其他成员和Pam Green博士实验室成员的支持和建议。Dan Rodriguez和Cesar Alvarez-Mejía协助进行逆转录PCR和胚珠特异性报告基因构建。M.A.V.和n.s.l都获得了国家科学院Tecnología (CONACyT)的博士奖学金。JPVC实验室的研究得到了Fondos Mixtos Guanajuato、CONACyT和霍华德休斯医学研究所的支持。我们要感谢托马斯·杰克博士提供的信息ap3-6以及俄亥俄州立大学拟南芥生物资源中心的种子储备ag)种子的股票。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
生物信息学分析和Gateway Promoter构建由JP完成,早期阶段的贡献由HG完成。启动子:GUS融合验证和稿件准备由JP完成。原位用SK和HS进行杂交。用MA-V、NS-L和JPV-C对胚珠结果进行了实验和分析。HS和BCM制定了实验概念并设计了实验。BCM协助撰写手稿。
电子辅料
12870 _2007_251_moesm1_esm.xls
附加文件1:空间解剖的拟南芥材料和各自的表达水平。一份excel电子表格(.xls),包含经过筛选的花器官富集表达基因的登录号列表,以及它们在花、叶和根文库中的描述和转录水平。还包括用于确定与先前微阵列分析的对应关系的数据,以及用于确定相对于野生型组织及其相对过表达或过表达的显著表达差异的p值。(XLS 458 KB)
权利和权限
开放获取本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(https://creativecommons.org/licenses/by/2.0),它允许在任何媒体上不受限制地使用、分发和复制,只要原著被恰当地引用。
关于这篇文章
引用这篇文章
裴弗,J.A.,考希克,S.,酒井,H.。et al。拟南芥花转录组的MPSS空间解剖。BMC植物杂志8日,43(2008)。https://doi.org/10.1186/1471-2229-8-43
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-43
关键字
- 植物的器官
- 大规模并行签名测序
- 植物的组织
- 同源转化突变
- 大规模并行签名测序数据