摘要
背景
水通道蛋白,也称为主要内在蛋白(MIPs),是一种古老的通道蛋白超家族,促进水和小溶质在细胞膜上的运输。MIPs存在于几乎所有的生物体中,在植物中尤其丰富,它们形成了一组不同的蛋白质,能够运输广泛选择的底物。
结果
全基因组分析Physcomitrella金属盘结果鉴定出23个MIPs,属于7个不同的亚科,其中只有5个以前被描述过。在新发现的亚科中,有一个只在p .金属盘而另一个则被发现存在于多种双子叶植物中,并形成了一个以前未被认识的主要MIP亚家族(X Intrinsic Proteins, XIPs)。令人惊讶的是,在亚科中也有一些特定的群体拟南芥而且玉米可以在p .金属盘.
结论
我们的研究结果表明,在原始陆生植物中,MIPs的早期多样化导致了大量的亚科。在高等植物的进化过程中,这些亚科中的一些随后消失,而剩余的亚科扩展,在某些情况下多样化,导致这些亚科中形成更专门的类群。
背景
水在细胞膜上的转运对生命至关重要,为了促进水和其他小极性分子在疏水膜上的转运,生物进化出了一系列广泛的膜整体蛋白通道。这些蛋白质被称为主要内在蛋白(MIPs),形成了一个庞大的进化保守的通道蛋白超家族,存在于所有类型的生物中,包括真细菌、古生菌、真菌、动物和植物[1,2].MIPs存在于哺乳动物的许多不同组织中,可能对许多不同的疾病具有重要意义[综述于[3.]],通过直接或间接参与运输和水平衡的调节。MIPs的这种普遍的生理参与激发了人们对调控和底物特异性的分子机制的兴趣。在植物中,MIPs的功能更为复杂,其生理作用尚不清楚[综述于[4,5]]。然而,仅仅是植物中不同MIPs的数量就暗示了它们的重要性,而且很可能一些异构体通过参与水运输调节,在细胞快速伸长和干旱适应等事件中发挥关键作用[6].为了在分子水平上充分了解整个植物的水分关系和其他小极性分子的转运,有必要识别完整的MIPs及其底物特异性和表达模式。
MIPs的系统发育综合研究[7]支持两大进化类群的分类。水通道蛋白(AQPs)最初被认为是专门运输水的,甘油吸收促进剂或水甘油通道蛋白(GLPs)促进各种小中性分子的运输。虽然MIPs形成被动通道,但膜的渗透性是通过控制不同MIPs的数量来调节的,在某些情况下也通过通道的磷酸化/去磷酸化来调节。MIPs的x射线和电子晶体学结构[8- - - - - -14]显示了四聚体四元结构,其中每个单体由六个跨膜螺旋(H1到H6)组成,由五个环(a - e)连接。环B(细胞质)和环E(细胞外)形成两个半膜横跨螺旋(HB和HE),并通过两个高度保守的天冬氨酸-脯氨酸-丙氨酸(NPA)盒从相反的方向相互作用,形成一个狭窄的孔隙区域。从NPA盒子到质周侧约8 Å的收缩区域,称为芳香/精氨酸(ar/R)区域,由来自H2和H5的两个残基和来自环e的两个残基形成。该区域形成了一个主要的选择过滤器,是溶质渗透性的主要检查点[[15],以及其中的参考文献]。
植物MIPs形成了一个巨大而不同的蛋白质超家族,在植物的每个基因组中编码有30多个已识别的成员拟南芥[16,17),玉米[18),栽培稻[19].这些大量的MIPs可能反映了底物特异性、定位、转录和翻译后调控的广泛多样性。根据序列相似性将植物MIPs分为5个亚科;质膜内禀蛋白(PIPs)、液膜内禀蛋白(TIPs)、结节素-26样内禀蛋白(NIPs)、小基内禀蛋白(SIPs)和glpf样内禀蛋白(GIPs) [7,16,20.].gip到目前为止只在Physcomitrella金属盘另一种近亲苔藓[20.].每个其他亚科都可以根据序列相似性进一步划分为组[16].尽管高等植物中所有的MIPs在系统发育上都属于MIPs的AQP支系[7它们对水的特异性并不高。一些研究表明,植物MIPs对其他分子也具有渗透性,例如,据报道,TIPs有助于尿素和氨的运输[21- - - - - -23];输送甘油的NIPs [24],氨[25],乳酸[26],硼[27]和硅[28];假定pip能够促进CO2扩散(29,30.]而在SIPs中,仅报告了SIP1亚组的水运输[31].运输特异性的差异可能是由于植物MIPs的ar/R过滤器的主要差异A. thaliana, Z. mays而且o .漂白亚麻纤维卷[32,33].
p .金属盘是一种苔藓(苔藓植物),大约在4.43 - 4.9亿年前,在维管植物进化之前,从通往高等植物的谱系中分离出来[34].这使得p .金属盘在与高等植物的进化比较和发现的任何共同特征方面,一个有价值的信息来源可以预期存在于大多数陆生植物中。除了p .金属盘具有使其成为未来功能研究中有吸引力的植物模型的特性,最重要的是同源重组的可能性[关于使用的信息p .金属盘可以在David Cove的两篇出色的评论中找到[35,36]]。一个组装的基因组p .金属盘(大约在480 Mbp),基于8.1倍覆盖率,联合基因组研究所最近发布了[37,38并使基因家族进化的分析有可能扩展到基础陆生植物谱系。这种分析以前曾被描述为蛋白质的膨胀素超家族[39],我们现在对MIP超家族进行了类似的分析。与expansin研究一致,我们也假设p .金属盘是由于不需要细胞特异性表达而有一个更简单的超家族结构,这个假设被收集的数据部分证明是错误的p .金属盘.在我们的分析中,我们不仅确定了五个以前定义的亚家族(PIP, TIP, NIP, SIP和GIP),而且还发现了两个以前未分类的MIP亚家族;杂交内禀蛋白(HIPs)和未分类的X内禀蛋白(XIPs),我们发现这个亚家族也存在于许多其他植物物种中。这些数据表明,MIP亚家族在植物中很早就进化了,不同亚家族的存在反映了亚细胞定位、底物特异性、转录和/或翻译后调控的差异,这些差异在原始植物中已经很重要,而仅在高等植物中所需要的特异性(例如,维管组织和种子中的细胞特异性表达)被高等植物中出现的亚家族中后来进化的MIP群所覆盖。
在这项研究中,我们试图从进化的角度来解决植物的MIP功能,通过比较原始陆地植物(苔藓)的整套MIPsp .金属盘)与两种高等植物(答:芥而且z梅斯).通过注释整个MIP超族p .金属盘我们也为未来的植物系统功能研究奠定了基础,允许同源重组及其所有优势,如敲除/替换内源基因。
结果
的识别Physcomitrella金属盘MIPs
最近对苔藓的排序p .金属盘基因组(37,38]首次使得在一种更原始的植物中鉴定所有MIP基因成为可能,从而对MIP蛋白质超家族的分子进化做出结论。搜索小立碗虫ssp patens v1.1在JGI数据库(PpDB)中,使用完整的35个蛋白质序列答:芥MIPs (AtMIPs) [16],结果鉴定出23种不同的基因编码p .金属盘MIPs (PpMIPs)1).两个基因在核苷酸水平上是相同的,因此只有一个蛋白序列(PpPIP2;4)被纳入进一步分析,代表两个基因。PpGIP1; 1p .金属盘MIP先前由Gustavsson等人详细描述[20.]也被包括在PpMIP集合中,然后总共达到23个全长mip。4个编码部分mip样序列的基因也被鉴定出来。其中3个要么是部分的,要么包含过早的终止密码子,因此被认为是无功能的假基因(pseudopip# 1, pseudopip# 2和pseudopip# 1)。第四个序列可能代表一个有功能的MIP编码基因,但位于被识别的外显子之后的一个被大测序间隙打断的短contig中,因此不能包括在分析中(称为partialNIP#1)。人工检查了JGI基因模型,认为大多数PpMIP基因是正确的。然而,对于一些基因来说,基因组序列中编码序列的不同注释要么是cDNA序列,要么是由于亚家族特定序列和基因结构的更好保护。这些外显子的替代配位,列于表中1,在本文的所有翻译和分析中都使用了。
当这项研究开始时,23个PpMIPs中只有11个在文献中被描述[20.,40].此后,23个PpMIPs中的另一个(PpPIP2;1)已被发表[41].所有23个PpMIP序列在PpDB中都被归类为属于一个水通道蛋白真核同源群(KOG),其中大多数也有建议的分类(表2)1).基于PpMIPs和AtMIPs的系统发育z梅斯MIPs (ZmMIPs)是一种新的、更系统的PpMIPs分类,与AtMIPs和ZmMIPs命名法一致[16,18]的建议(表1).
系统发育和分类
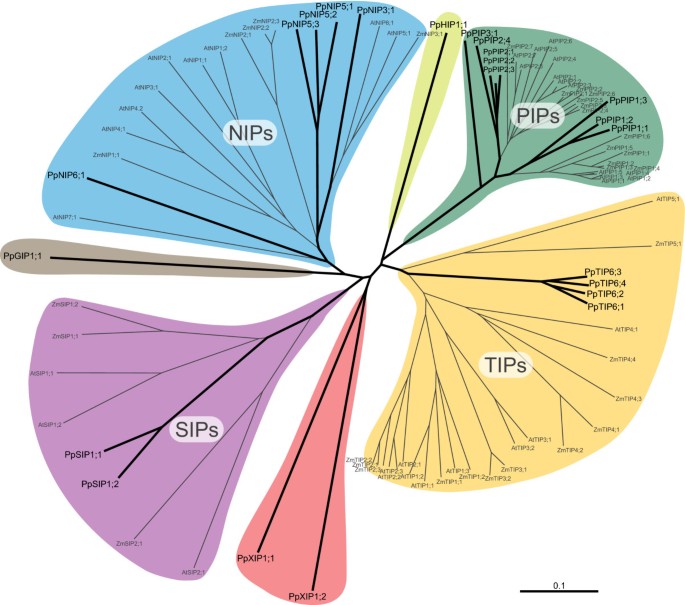
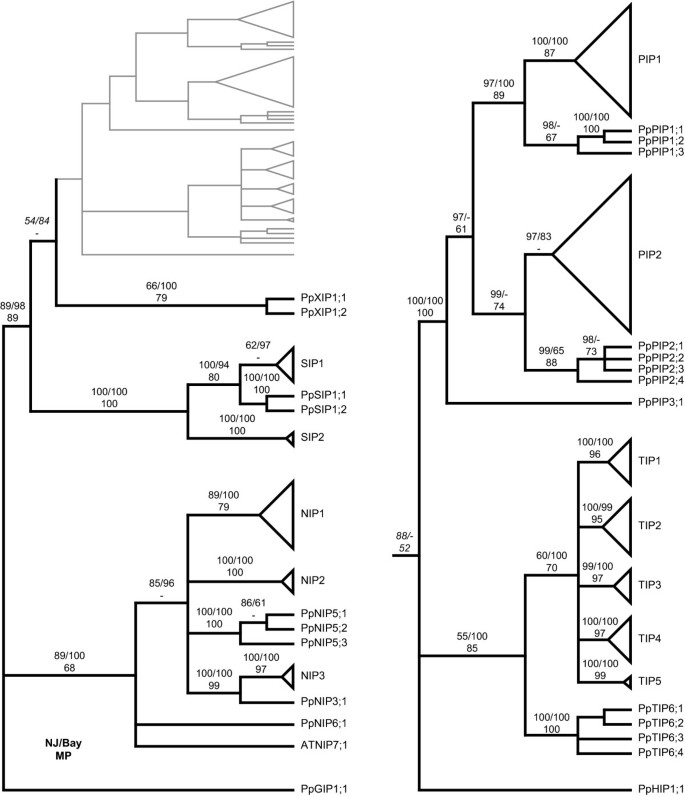
使用所有PpMIPs, AtMIPs和ZmMIPs的全长蛋白比对[见附加文件]1相邻连接(NJ)方法得到一棵树(图;1),并将其与最大简约(MP)方法和贝叶斯(Bay)方法进行比较。使用自举支持和贝叶斯后验概率构建了一个总结三种方法结果的“方法-共识”分支图,并用于对PpMIPs进行分类(图2)。2).除NIPs外,所有MIPs在亚科内亚组中的AtMIPs和ZmMIPs的分类相似。我们根据在NIPs分类中使用的命名法来命名PpNIPsz梅斯而且o .漂白亚麻纤维卷由于这四个较宽的子组允许更多的序列分歧,因此比中定义的较窄的七个子组更通用答:芥.p .金属盘未能与先前分类的亚科组分组的子组被给予连续较高的指数(例如PpPIP3, PpTIP6, PpNIP5或PpNIP6)。共分为3个pppip1、4个pppip2、1个PpPIP3、4个PpTIP6s、1个PpNIP3、3个PpNIP5s、1个PpNIP6和2个PpSIP1s。4个PpMIPs未能被归类为亚科,因为它们在中鉴定的MIPs中缺乏直系同源物答:芥而且z梅斯.其中之一是MIP异种物(水平基因转移产生的同源物)PpGIP1;1先前被鉴定为glpf样MIP,并据此命名[20.].剩下的三个是PpHIP1;1,它与TIPs和pip都有相似之处,但形成了自己的独立的亚家族,以及PpXIP1;1和PpXIP1;2,两个不同的MIPs,共享一些独特的先前未描述的基序。
为了找到三个未分类的PpMIPs (PpHIP1;1, PpXIP1;1和PpXIP1;2)的同源物,我们在NCBI和embl数据库中进行了检索。选择代表多种物种的Hits,并将相应的蛋白序列与PpPIPs、PpTIPs和PpHIP1;1或PpXIP1;1和PpXIP1;2进行比对。这些比对被用于系统发育分析,以评估新获得的序列是否有助于对三个PpMIPs进行分类。PpHIP1;1在数据库中主要被标记为TIPs或aqp4,系统发育分析得到三个聚类(PIPs, TIPs和aqp4),但PpHIP1;1仍然是所有这些亚家族的基础,因此不能被分配到这些亚家族中的任何一个(数据未显示)。对于PpXIP1;1和PpXIP1;2,命中序列多标注为Plant MIP、TIP或AQP0序列。系统发育分析产生了四个不同的亚家族,TIPs, PIPs aqp0和由未指明的植物MIPs和PpXIPs组成的第四个分支(数据未显示),见下一段的进一步分析。
XIPs是高等植物中一个未知的MIP亚家族
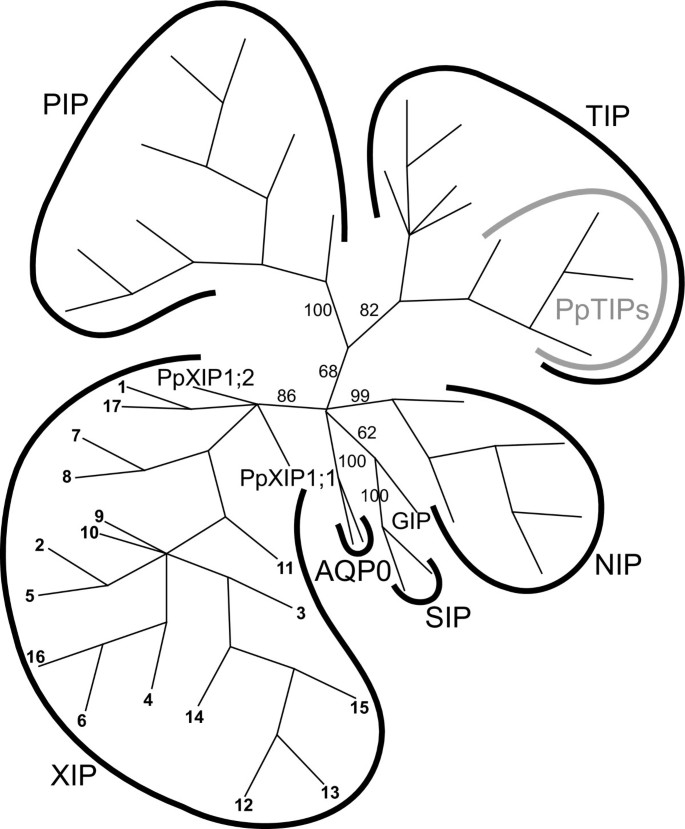
属于这第四个分支的序列总体上与MIPs的序列相似性较弱(约30%的氨基酸相同,数据未显示),既不能被分配到任何先前确定的植物MIPs类别(pip, TIPs, NIPs, SIPs和GIPs),也不能与PpHIP1;1序列相关。然而,这个新亚家族中的一些保守基序(见讨论)被鉴定出来,并基于这些代表性序列(蓖蓖子cDNA序列[GenBank:EG656577])被选择出来。该序列被用于数据库搜索,以获得更多属于该新亚家族的MIPs。他们还发现了更多具有相同保守基序的序列。这些序列之一起源于杨树trichocarpa因此p . trichocarpa检索了JGI的基因组,确定了另外4个平行谱系(表2).这些序列连同从蓖麻豆cDNA和PpXIP检索的序列以及所有PpMIP序列(PpHIP1;1除外)组合成一个用于系统发育分析的序列比对。所得到的树证实,未分类的MIPs形成了一个独特的单系支系(PpXIPs为基础类群),与分析中包括的其他MIPs不同(图2)。3.).如表所示3.在第一个NPA框和ar/R滤波器上,该支序列之间存在较大差异。我们建议,在等待进一步的描述之前,新亚家族中的MIPs应被称为X固有蛋白(XIPs),强调目前我们对这些蛋白的功能知之甚少。
基因结构
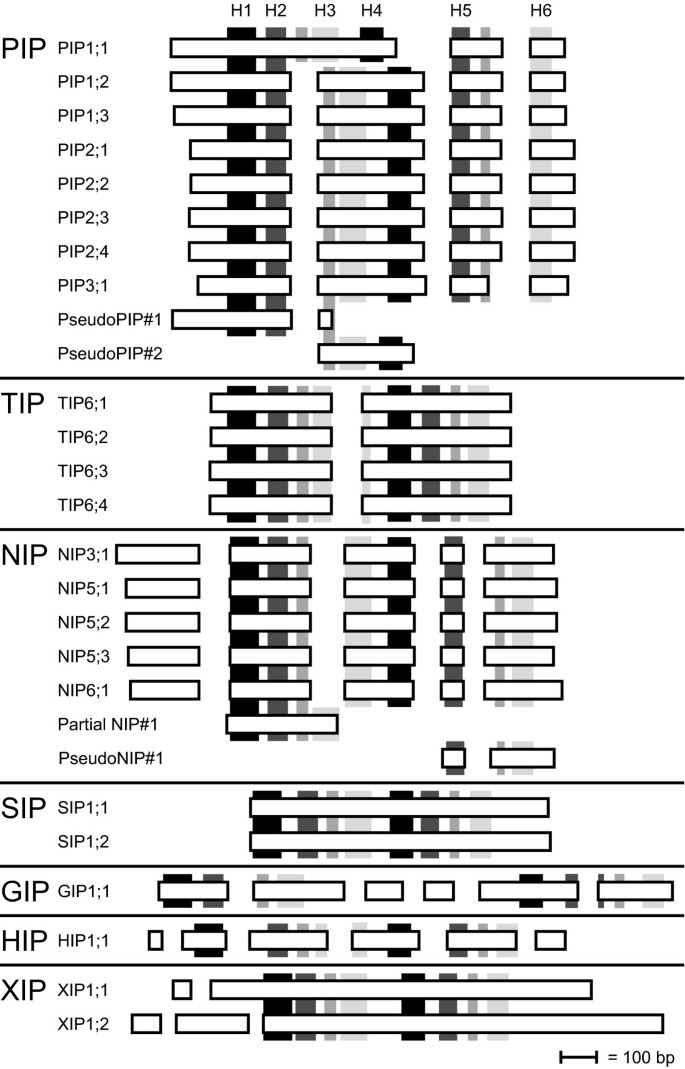
平均PpMIP发现内含子2.6个,大小为246.4 bp。这大约是内含子数量的一半,但与平均预测的大小大致相同p .金属盘全基因组分析中的基因[42].外显子/内含子模式PpMIPs在每个亚科内都高度保守,如图4.与AtMIPs显示了两者的内含子位置都是保守的pip值而且少量的酒,但不是为了提示(在p .金属盘内含子位置在5'端远35个碱基对处)和口(完全缺乏内含子p .金属盘).外显子/内含子模式也支持PpHIP和PpXIPs两者都不能归类pip值,提示,少量的酒,口也不的溢价,而是作为独立的亚家族。
鉴别五种p . trichocarpaXIP平行谱允许跨物种的基因结构比较。所有五个p . trichocarpa基因具有相同的外显子-内含子模式,在n端序列中有两个内含子(数据未显示)。ppxi1;2也是如此,但由于n端存在高度的种间变异,因此很难得出内含子位置是否完全保守的结论。
讨论
Physcomitrella金属盘主要内在蛋白质
远亲物种的蛋白质超家族的比较可以帮助我们理解蛋白质的功能,并通过注释的所有MIPsp .金属盘我们对高等植物和苔藓的MIP超家族进行了这样的比较。最初我们假设苔藓有一个相对较小的超家族,因为它们更简单(例如缺乏维管组织,因此具有不那么复杂的水运输调节)。因此,我们非常惊讶地发现p .金属盘有7个亚家族,总共包含23个不同的MIPs,这是一个意想不到的大而发散的超家族。Gustavsson等人详细分析了其中一个基因(PpGIP1;1)。[20.],因此在此讨论中省略。剩下的22个PpMIPs中有一半是由Borstlap先前描述的[40]和Lienard等人。[41],其余11个以前没有在文献中描述过。PpMIPs的基因结构支持系统发育分析,并将其划分为7个亚家族。与AtMIPs相比,PIPs和NIPs具有保守的内含子位置,而SIPs和TIPs则没有。这与两者中NIP和PIP亚家族个体组的保守性一致p .金属盘而且答:芥(下文将进一步讨论)。
PIPs -植物中最保守的MIPs
PIPs是保存良好的植物MIPs,可进一步分为pip1和pip2。pip1和pip2都是高度保守的p .金属盘这表明这些类群一定是在陆地植物进化的早期形成的,并且在植物生理学中具有根本的重要性。在高等植物中,pip1和pip2在水分关系中的生理相关性已经得到了很好的证实,最近二氧化碳也被添加到可能的底物列表中[综述于[4]]。ar/R滤波器在包括pppip在内的PIPs中是严格保守的,这表明所有的PIPs,无论在亚组中,都具有相同的底物特异性(表2)3.).PIP序列的进化也可能在许多其他方面受到限制。例如,pip存在于质膜中,为了维持质子梯度,它们对质子来说是不可渗透的。此外,磷酸化、pH和Ca可以调节pip的透水性2 +通过一个复杂的门控机制[11].从我们在这里给出的结果可以清楚地看出,在n端区域的二酸基序和d环中的组氨酸负责Ca2 +结合和pH门控分别在所有pppip1和pppip2中都是保守的。B环的磷酸化位点在所有PpPIPs中也被保守,而PIP2特异性的c端磷酸化基序仅限于pppip2。这表明,在所有表达pip的物种和组织中,pH门控机制都是通用的,例如,pH门控并不局限于高等植物根部的厌氧条件。
在p .金属盘还有一个奇PIP (PpPIP3;1),在pip1和pip2的基础上。PpPIP3;1在第二个NPA-box之后(螺旋E和螺旋6之间)有11个氨基酸缺失,这与其他pip的差异相对较大(例如缺乏Ca2 +N端结合位点和螺旋2上的保守半胱氨酸),以及ESTs的缺失,使得这一点值得怀疑米兰理工大学管理学院吉恩完全正常。
TIPs专业化发生在后来
已经有人提出p .金属盘缺乏在高等植物中观察到的TIPs的特定异构体[40]现在,有了这组完整的PpMIPs在手,这一点得到了证实。有趣的是,有人提出液泡亚型含有特定的TIP亚型[43],很容易推测高等植物的TIP基团是由于不同液泡的特殊功能需求而进化而来的。中保守蛋白的鉴定p .金属盘,参与蛋白质到不同类型液泡的分类,这表明在苔藓植物中很可能存在不止一种类型的液泡[44].这意味着TIPs不是液泡亚型的保守标记,因为在液泡中只存在一组TIPsp .金属盘表明苔藓中只有一种液泡类型具有TIPs,或者苔藓细胞中几个不同的液泡都具有相同类型的TIPs。这两种解释都与最近在高等植物中进行的实验相一致,这些实验挑战了TIPs作为液泡亚型有效标记的观点[45,46].
PpTIP6s并没有形成一个非常遥远的TIPs亚类,而是表现为在高等植物不同TIP群中发现的不同母基的保守镶嵌。例如,n端前几个氨基酸残基与TIP2s相似,而c端区域与TIP3s最相似。ar/R过滤器(HIAR)中氨基酸残基的身份与某些tip3和tip4相同,表明具有相似的特异性。事实上正是这些残基是最常见的,比较所有选择性区域的频率答:芥,z梅斯而且o .漂白亚麻纤维卷提示(H0.81我0.62一个0.72R0.75;根据[47])。这使得PpTIP6s很可能与存在于苔藓植物和维管植物最后一个共同祖先中的TIPs相似,而在这些位置发现的其他基序是后来在维管植物中不同类群TIPs进化时出现的衍生特征。高等植物TIP亚家族中特殊基团的扩展和形成可能表明,其中一些TIPs已经接管了高等植物亚家族中缺失的MIPs的功能(如HIPs和XIPs)。
NIP群体进化较早
在高等植物中,NIPs形成一个不同的亚科,种间差异较大。这也适用于NIPsp .金属盘但令人惊讶的是,这三组NIP中的一组也存在于高等植物中,这表明这一组NIPs, NIP3,已经存在于植物的共同祖先中p .金属盘和高等植物(图;2).保守的内含子位置在少量的酒在答:芥而且p .金属盘表明这种基因结构在祖先中也存在夹基因。nip与其他mip的不同之处在于,他们通常有非正统的NPA盒子。在许多高等植物的nip3中,第一和第二NPA盒分别被NPS和NPV所取代[47].PpNIP3;1中对应的基序为NPA和NPV(表1)3.),与AtNIP6相同;1(设备中两个nip3中的一个)答:芥根据单子叶分类),表明NIP3s在苔藓植物和维管植物分裂之前就具有这些基序。
两个NIP组p .金属盘(PpNIP5和PpNIP6),在ar/R过滤器中具有独特的氨基酸组合(表3.).PpNIP3;1的ar/R区与其他nip3的残基一致,支持它们是具有相同保守函数的直系物。近年来,一株NIP3已被证实在根中硼的吸收中起作用答:芥[27即使苔藓没有根,也不能排除PpNIP3;1在苔藓中硼的运输中起作用。
与大多数其他植物MIPs相比,NIPs的n端区域相对较长,并在单独的外显子上编码。由于在这一区域缺乏一般保守的基序,第一个外显子在注释中经常缺失夹基因。然而,在高等植物的NIP3s中,在n端区域发现了几个基序[48],其中一些特征在PpNIP3;1中也被保守。与高等植物相似,PpNIP3;1具有高度的脯氨酸和苏氨酸残基,并有一个序列(AKCFP),对应于高等植物中的保守基序(C [KN]C [LF] [PS])。
在高等植物中,许多NIPs在c端有一个保守的电位磷酸化基序,与磷酸化位点相对应大豆NOD26 (GmNOD26, S262)和菠菜oleraceaPIP2; 1 (SoPIP2; 1;S274) [5,49].在高等植物的NIP3s ([RK]XXR)中,该位置的丝氨酸也存在于类似的基序中年代FXR) [48但在PpNIP3;1中,丝氨酸被取代为缬氨酸。在PpNIP5;3和PpNIP6;1中存在丝氨酸,但motif中的一些基本残基并不保守。相反,在母题(KXXK年代F [HR]R)存在于PpNIP5;1和PpNIP5;2中,这表明在苔藓植物和高等植物的共同祖先中,至少有一些NIPs是由磷酸化调节的。
有趣的是,在MIP中没有NIP2类型p .金属盘,一个国家科学院的研究小组最近在水稻中发现了一种硅转运体[28].由于已知苔藓植物会积累硅[50],缺乏PpNIP2s表明这一功能是由一种不同的异构体或蛋白质类p .金属盘.
中只有sip1Physcomitrella金属盘
在答:芥SIPs有两类,SIP1s和SIP2s,它们都具有相同的基因结构,在保守位置有两个内含子[16].在p .金属盘有两个sip,但都没有内含子。令人惊讶的是,这两个PpSIPs都属于SIP1群,而高等植物的SIP2s则属于基支。这表明sip2在早期陆地植物中已经存在,但随后消失了p .金属盘其中剩余的SIP1s遭受内含子损失,或者SIP2s在分裂后迅速与SIP1s分离,导致苔藓和高等植物。内含子丢失PpSIP1s或者在一个共同的祖先中获得内含子SIP1s而且SIP2s在高等植物中同样可能发生这种情况。在大多数SIP1s中,与第一个NPA框对应的序列是NPT,有趣的是,这个不寻常的motif在PpSIP1s中也被保守,这意味着这是SIP1s在结构和功能上的重要特征。此外,ar/R滤波器与系统发育分类一致,表明SIP1s在陆生植物中具有保守功能。
HIP是一个独特的MIP,与pip和TIPs都有相似之处
有三个p .金属盘先前在植物中描述的不能被归类为任何一个亚家族的MIP序列[16,20.].其中一个,PpHIP1;1,似乎是一个相当罕见的MIP,因为我们无法识别任何orthologs。独特的基因结构表明该蛋白属于一个单独的亚家族。在系统发育分析中,PpHIP1;1倾向于与pip和TIPs聚类,尽管如图所示,这一支持度不是很强2.在看ar/R过滤器(表3.)人们也可以推测HIP与TIPs和PIPs有关,因为它在H2位(典型的TIPs)和H5位(典型的PIPs)上都有组氨酸。然而,在过滤器中有两个大的碱性氨基酸残基会对运输性能产生什么影响尚不清楚,而且由于该基因没有ESTs,它甚至可能不被表达。根据亚细胞定位预测(WoLF PSORT [51],数据未显示)PpHIP1;1比质膜更可能驻留在液泡体中。PpHIP的表达、定位和底物特异性有待进一步研究。
另外两个序列属于另一组,即XIPs,将在下一段中进一步讨论。
XIP子家族
通过对PpXIP直系同源物的搜索,发现了许多来自不同物种的XIP序列,包括来自毛锥虫(可能是相同的五种被描述为“假定的缺乏水通道蛋白拟南芥图斯坎等人。[52])。令人吃惊的是,没有一个序列来自单子叶。虽然大多数序列来自双子,但在中未发现正交序列答:芥,这可能是由于相对较近的基因组大小减少而导致的基因丢失[53].系统发育分析证实,据我们所知,这些序列来自一个以前未被认识的MIP亚家族,不同于PIPs、TIPs、NIPs、SIPs和GIPs。分析中唯一包含的非植物序列是来自变形虫的[GenBank:XM_639170]基因编码的蛋白质盘基网柄菌AX4需要指出的是,尽管该蛋白在系统发育分析中与XIPs聚类,但它被注释为一种假设蛋白,缺乏XIPs的一些特征。例如,阿米巴蛋白具有不同于所有其他XIPs的NPA盒和ar/R过滤器,以及整体高度分化的MIP序列,所有这些都使该蛋白是否具有与其他XIPs相同的功能受到质疑。还有一种石松植物,长刺苔藓的序列卷柏moellendorffii,与两个PpXIPs一起是三个最不同的序列,尽管这三个序列都可以明确地归类为XIPs。虽然大多数序列来自est,但由于XIP转录本从根、幼苗、花蕾到种子和果实等许多不同组织中分离出来,因此无法对其表达模式做出一般的结论(表2)2).根据亚细胞定位预测,XIPs可能位于质膜中(WoLF PSORT [51],数据未显示)。
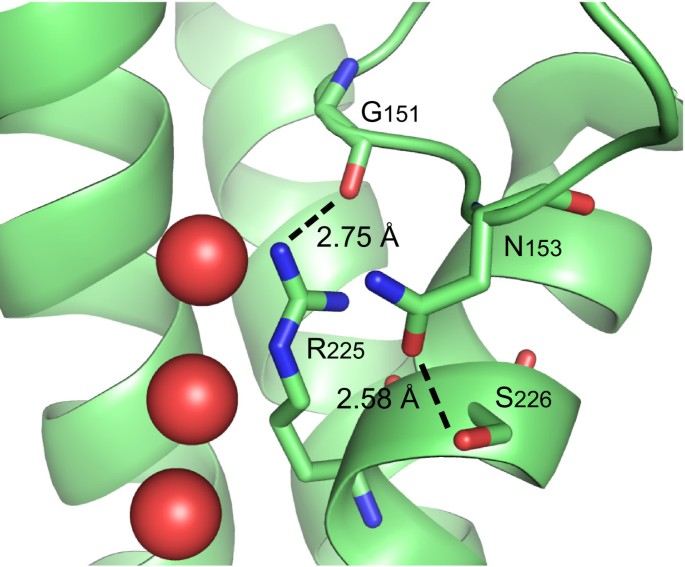
在xip的第一个NPA框中,丙氨酸被缬氨酸、亮氨酸、异亮氨酸、丝氨酸或半胱氨酸取代。除异亮氨酸外,所有这些替换都已在其他MIPs的NPA盒中观察到[47].该亚家族最保守的特征位于第2 NPA框之后,其中一个半胱氨酸氨基酸在motif NPAR中完全保守C.这种半胱氨酸只是在许多其他亚家族中发现的保守丝氨酸或苏氨酸的适度变化,如pip、TIPs、NIPs和几种哺乳动物AQPs。然而,从SoPIP2;1的解析结构可以看出,该位置的残基可以通过氢键稳定C-loop的构象([PDB:1Z98];S226 - N153,见图。5)这种相互作用似乎在结构上是保守的,在BtAQP1中也可以看到([PDB:1J4N];S198 - N129)、BtAQP0 ([PDB:1YMG];S188 - N119)和EcGlpF中供体-受体互换的BtAQP0 ([PDB:1FX8];D207 - T137)。这种稳定性可能直接影响了孔隙的渗透性,因为ar/R过滤器的精氨酸取向也通过与C-loop主干的氢键而稳定(图2)。5).有趣的是,所有的XIPs都有一个保守的半胱氨酸,从而形成LGG基序C在C-loop中,该位置可与SoPIP2中的N153对准;这表明,在XIPs中,半胱氨酸桥可能与精氨酸共价固定c环,因此胞外孔入口可能比其他MIPs更坚硬。
在第一个NPA-box前的2,7个氨基酸的螺旋末端也有一个高度保守的脯氨酸基序(PISGGHINP),也在哺乳动物aqp5中发现。在许多其他植物MIPs的螺旋5中也可以找到相应的基序,这很有趣,因为这反映了MIP蛋白质的对称性,由两个序列的直接重复组成。同样值得注意的是,除了PpXIPs,在螺旋5中缺乏高度保守的甘氨酸,允许螺旋2和5的紧密包装[54],在大多数XIPs中被亮氨酸或异亮氨酸所取代。另一种保留保守甘氨酸,但在螺旋5和第二个NPA盒子之间引入两个额外氨基酸的排列是可能的,但在本文的分析中没有使用。这种排列还将影响哪种氨基酸位于ar/R过滤器的H5位置(表2)3.).在选择的排列中,缬氨酸是H5位置上最常见的残留物,而在另一种排列中,苏氨酸将位于H5位置。在H2位置,大多数XIPs都有一个脂肪族氨基酸,这在一些NIPs和SIPs中也能找到[47].这表明XIPs主要不是水通道,尽管必须进行底物特异性实验来证实这一点。在XIPs中p .金属盘而且美国moellendorffiiar/R过滤器的H2和H5位点分别存在谷氨酰胺,高等植物的tip4和tip5位点也存在谷氨酰胺,提示这些TIPs可能在原始植物中接管了XIPs的某些功能。为了确定这个新的MIP亚家族的功能,还需要进一步的定位、特异性和表达模式的研究。
结论
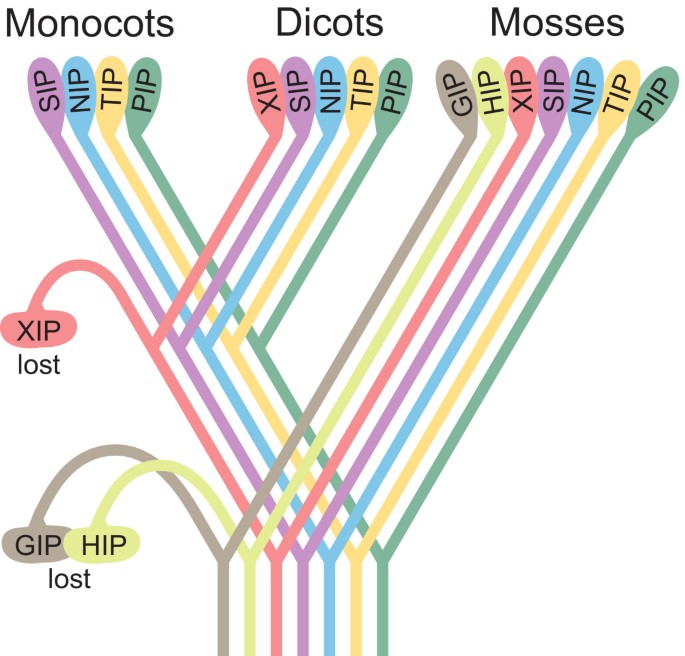
在本研究中,我们发现了数量惊人的MIP编码基因p .金属盘形成了一个由七个亚科组成的多样化的超级家族。共鉴定出23个PpMIPs;8个pip, 4个TIPs, 5个NIPs和2个SIPs, 1个GIP和3个MIPs,属于两个不同的新亚家族,HIPs和XIPs。迄今为止,在任何高等植物中都没有发现HIPs,而XIPs似乎存在于许多植物物种中,尽管在单子叶植物中没有发现。有趣的是,这些亚科中的特定群体,如pip1、pip2、NIP3s和可能的SIP1s,已经存在于高等植物和苔藓植物的共同祖先中。相比之下,TIPs的子群可能进化得更晚。这些结果表明,早期陆生植物有一个大而不同的MIP超家族,至少包括在植物中发现的七个亚科p .金属盘在高等植物的进化过程中,一些亚科消失了(图2)。6),而其余亚科进一步进化,导致亚科内的亚群多样化和形成。我们推测,一些新的亚群,或者其他一些不相关的转运蛋白,接管了高等植物中丢失的MIP亚族的功能。
方法
基因鉴定与注释
小立碗菌MIP基因是通过联合基因组研究所PpDB的TBLASTN搜索确定的[37]使用35个MIPs的完整的蛋白质序列拟南芥作为查询[16].根据亚家族序列相似性或EST支持度,手工检测与命中重叠的基因模型并保存。如果没有令人满意的模型存在,则使用基因组序列来识别新模型或修改模型的外显子(如表中所示)1).PpGIP1;1序列也被添加到序列中,因为它之前被识别为PpMIP [20.].蛋白质相应的翻译序列PpMIP基因被用于第二轮TBLASTN搜索,以识别PpDB中更不同的MIP序列,但没有发现。得到的23个PpMIPs与35个AtMIP和33个ZmMIPs一起用于翻译序列的多次比对[18].对线进行人工检查和调整,并注意保持低间隙数量,避免在功能重要特征(如npa -box和跨膜区域)中出现间隙。构成此处所述关于PpMIPs的所有系统进化分析基础的比对可在EMBL-align数据库中作为ALIGN_001168获得(该数据库可通过EMBL-EBI SRS主页访问[55]或FTP [56])。
通过EMBL核苷酸序列数据库的TFASTX3检索,寻找未分类的PpHIP, PpXIP1;1和PpXIP1;2的同源物[57]和TBLASTN检索NCBI的nr/nt、est、gss和htgs数据库[58]使用这三个的翻译序列PpMIPs.代表多种物种命中的翻译与PpHIP1;1或PpXIP1;1和PpXIP1;2以及PpPIPs和PpTIPs一起用于蛋白质比对。如上所述,对这些比对进行手动检查和调整,并用于PpHIP1;1和PpXIPs的系统发育分析,在EMBL-align数据库中分别为ALIGN_001169 ALIGN_001170。
在NCBI的nr/nt、est、gss和htgs数据库的TBLASTN搜索中,使用了发现的一个PpXIP同源物的翻译序列[GenBank:EG656577],以找到更多该类群的同源物。发现的一个正正交物来自杨树trichocarpa的TBLASTN搜索中使用了该序列的翻译p . trichocarpa在JGI的基因组中寻找谬误基因。这些平行序列与来自[GenBank:EG656577]和PpXIP搜索的同源物一起,与22个PpMIPs (PpHIP除外)一起用于翻译序列的多序列比对。采用与PpMIP-AtMIP-ZmMIP对齐相同的方式手动检查和调整对齐。这种比对形成了关于MIPs的XIP组的所有系统发育分析的基础,并在EMBL-align数据库中作为ALIGN_001171提供。
系统发育分析
通过邻居连接(NJ)、最大简约(MP)和贝叶斯推理(Bay)三种不同的系统发育方法对PpMIP序列的比对进行了分析。对于所有方法,缺口都被视为缺失数据。PAUP * 4.0 b10 [59]用于NJ和MP分析。两种方法都使用默认设置,并对每种方法进行1000次重复引导,以评估最佳树的置信度。使用MrBayes 3.0.2进行贝叶斯系统发育推理[60]在JTT下使用模糊或无信息的似然模型先验概率分布[61+I+Γ模型。两组4个平行大都会耦合蒙特卡洛马尔可夫链,其中3个加热温度增量为0.2,从随机树开始运行200万代。每100棵树取样。前25%的样本树被丢弃,并通过观察保留样本的似然分数来经验地确定固定阶段。使用贝叶斯后验概率来评估推断树的稳健性。构建了一个“方法共识”树作为概述,在这个树中,只有至少两种方法的自举或后验概率支持超过50%的分支被保留,所有其他的都被折叠。
对于PpHIP1;1, PpXIPs和XIP-group比对,PAUP*4.0b10 [59]用于NJ和MP分析(缺口被视为缺失数据)。两种方法和xip组对齐分析都使用默认设置,每种方法进行1000次重复引导,评估最佳树的置信度。所有来自PpMIP, PpHIP, PpXIPs和XIP系列分析的树都可以在nexus格式中查看[62][参见附加文件2,3.,4,5,6,7,8,9,10,11,12,13,14].
证明中增加的说明
在这项工作的发表过程中,我们成功地鉴定了穗苔中MIPs的HIP亚家族卷柏moellendorffii.PpHIP1;1和最接近的同源物美国moellendorffii高度相似(氨基酸同一性为73.7%),具有相同的npa -box和ar/R过滤动机。这证明了HIP亚族确实是MIPs的一个新的保守亚族,而不是只在Physcomitrella金属盘.
参考文献
- 1.
Agre P, Kozono D:水通道蛋白水通道:人类疾病的分子机制。中国生物医学工程学报,2003,29(1):344 - 344。10.1016 / s0014 - 5793(03) 01083 - 4。
- 2.
海曼JB, Engel A:水通道蛋白:系统发育、结构和生理。科学通报,1999,14:187-193。
- 3.
King LS, Kozono D, Agre P:从结构到疾病:水通道蛋白生物学的进化故事。自然评论。2004, 5(9): 687-698。10.1038 / nrm1469。
- 4.
植物水通道蛋白的新功能和调控特性。中国生物医学工程学报,2007,29(3):344 - 344。10.1016 / j.febslet.2007.03.021。
- 5.
Johansson I, Karlsson M, Johanson U, Larsson C, Kjellbom P:水通道蛋白在细胞和整个植物水分平衡中的作用。生物化学学报,2000,14(1):324- 324。
- 6.
Alexandersson E, Fraysse L, Sjovall-Larsen S, Gustavsson S, Fellert M, Karlsson M, Johanson U, Kjellbom P:水通道蛋白全基因家族表达与干旱胁迫调控。植物分子生物学,2005,59(3):469-484。10.1007 / s11103 - 005 - 0352 - 1。
- 7.
主要内在蛋白家族的系统发育和进化。中国生物医学工程学报,2005,26(6):397-414。10.1042 / BC20040134。
- 8.
傅东,lilibson A, Miercke LJ, Weitzman C, Nollert P, Krucinski J, Stroud RM:甘油导电通道的结构及其选择性基础。科学通报,2000,29(4):489 - 497。10.1126 / science.290.5491.481。
- 9.
Murata K, Mitsuoka K, Hirai T, Walz T, Agre P, Heymann JB, Engel A, Fujiyoshi Y:水通道蛋白-1渗透的结构决定因素。自然科学,2000,40(4):589 -605。10.1038 / 35036519。
- 10.
王志强,王志强,王志强,王志强。水通道蛋白的结构与选择性:2.5 Å水通道蛋白的x射线结构研究[j] .中国生物医学工程学报,2003,1 (3):E72-10.1371/journal.pbio.0000072。
- 11.
torroth - horfield S, Wang Y, Hedfalk K, Johanson U, Karlsson M, Tajkhorshid E, Neutze R, Kjellbom P:植物水通道蛋白门控的结构机制。自然科学进展,2006,39(4):344 - 344。10.1038 / nature04316。
- 12.
Gonen T, Cheng Y, Sliz P, Hiroaki Y, Fujiyoshi Y, Harrison SC, Walz T:双层二维AQP0晶体的脂蛋白相互作用。自然科学学报,2005,38(4):344 - 344。10.1038 / nature04321。
- 13.
Lee JK, Kozono D, Remis J, Kitagawa Y, Agre P, Stroud RM:古细菌水通道蛋白AqpM在1.68的电导结构基础Å。美国国家科学研究院。中国农业科学,2005,32(4):344 - 344。10.1073 / pnas.0509469102。
- 14.
Hiroaki Y, Tani K, Kamegawa A, Gyobu N, Nishikawa K, Suzuki H, Walz T, Sasaki S, Mitsuoka K, Kimura K,等:水通道蛋白-4结构对阵列形成和细胞粘附的影响。中华分子生物学杂志,2006,355(4):628-639。10.1016 / j.jmb.2005.10.081。
- 15.
Beitz E, Wu B, Holm LM, Schultz JE, Zeuthen T:水通道蛋白1芳香/精氨酸区域的点突变允许尿素、甘油、氨和质子通过。美国国家科学研究院。中国农业科学,2006,34(2):379 - 379。10.1073 / pnas.0507225103。
- 16.
Johanson U, Karlsson M, Johansson I, Gustavsson S, Sjovall S, Fraysse L, Weig AR, Kjellbom P:拟南芥主要内在蛋白的完整基因编码为植物主要内在蛋白的新命名提供了框架。中国植物学报,2001,26(4):344 - 344。10.1104 / pp.126.4.1358。
- 17.
李文杰,李文杰,李文杰:从基因组到功能:拟南芥水通道蛋白。中国生物医学工程学报,2002,3(1):研究0001.0001-0001.0017。
- 18.
Chaumont F, Barrieu F, Wojcik E, Chrispeels MJ, Jung R:水通道蛋白是玉米中一个庞大且高度分化的蛋白质家族。中国植物学报,2001,26(3):344 - 344。10.1104 / pp.125.3.1206。
- 19.
Sakurai J, Ishikawa F, Yamaguchi T, Uemura M, Maeshima M: 33个水稻水通道蛋白基因的鉴定及其表达与功能分析。植物生理学报,2005,26(4):344 - 344。10.1093 /卡式肺囊虫肺炎/ pci172。
- 20.
Gustavsson S, Lebrun AS, Norden K, Chaumont F, Johanson U:一种与细菌甘油通道最相似的新型植物内源性蛋白。中国生物医学工程学报,2005,39(1):379 - 379。10.1104 / pp.105.063198。
- 21.
Jahn TP, Moller AL, Zeuthen T, Holm LM, Klaerke DA, Mohsin B, Kuhlbrandt W, Schjoerring JK:植物和哺乳动物中的水通道蛋白同源物运输氨。中国生物医学工程学报,2004,27(3):344 - 344。10.1016 / j.febslet.2004.08.004。
- 22.
刘丽萍,刘丽萍,刘丽萍,冯伟仁,刘丽萍。拟南芥液泡膜内源性蛋白对尿素转运的影响。植物营养学报,2003,26(3):339 - 339。10.1104 / pp.103.027409。
- 23.
Loque D, Ludewig U, Yuan L, von Wiren N:液泡体内征蛋白AtTIP2;1和AtTIP2;3促进NH3进入液泡。植物营养学报,2005,37(2):379 - 379。10.1104 / pp.104.051268。
- 24.
Wallace IS, Roberts DM:植物水甘油酯通道内结蛋白家族两个结构亚类的不同转运选择性。生物化学,2005,44(51):16826-16834。10.1021 / bi0511888。
- 25.
张志刚,张志刚,张志刚,张志刚。丛枝菌根共生与植物水通道蛋白表达的关系。植物化学学报,2007,29(1):344 - 344。10.1016 / j.phytochem.2006.09.033。
- 26.
Choi WG, Roberts DM:拟南芥NIP2;1:缺氧胁迫诱导乳酸的主要内在蛋白转运体。生物化学杂志,2007
- 27.
Takano J, Wada M, Ludewig U, Schaaf G, von Wiren N, Fujiwara T:拟南芥主要内在蛋白NIP5;1在硼限制条件下对硼的有效吸收和植株发育至关重要。植物细胞学报,2006,18(6):1498-1509。10.1105 / tpc.106.041640。
- 28.
Ma JF, Tamai K, Yamaji N, Mitani N, Konishi S, Katsuhara M, Ishiguro M, Murata Y, Yano M:水稻中的硅转运体。自然科学学报,2006,44(6):688-691。10.1038 / nature04590。
- 29.
Flexas J, Ribas-Carbo M, Hanson DT, Bota J, Otto B, Cifre J, McDowell N, Medrano H, Kaldenhoff R:烟草水通道蛋白NtAQP1参与叶肉对CO的传导2体内。植物学报,2006,48(3):427-439。10.1111 / j.1365 - 313 x.2006.02879.x。
- 30.
Uehlein N, Lovisolo C, Siefritz F, Kaldenhoff R:烟草水通道蛋白NtAQP1是一种膜CO2毛孔具有生理功能。自然科学,2003,29(1):1 - 4。10.1038 / nature02027。
- 31.
Ishikawa F, Suga S, Uemura T, Sato MH, Maeshima M:新型水通道蛋白SIPs主要定位于ER膜,在拟南芥中表现出细胞特异性表达。中国生物医学工程学报,2005,26(3):344 - 344。
- 32.
Bansal A, Sankararamakrishnan R:水稻、玉米和拟南芥主要内在蛋白的同源性建模:跨膜螺旋关联和芳香/精氨酸选择性过滤器的比较分析。中国生物医学工程学报,2007,7:27-10.1186/1472-6807- 727。
- 33.
Wallace IS, Roberts DM:拟南芥主要内在蛋白代表性亚家族的同源性建模。基于芳香族/精氨酸选择性过滤器的分类。中国生物医学工程学报,2004,26(2):357 - 357。10.1104 / pp.103.033415。
- 34.
杜泽里EJ,斯内尔EA, Bapteste, Delsuc F,菲利普H:真核生物进化的时间:一个放松的分子钟能协调蛋白质和化石吗?美国国家科学研究院。2004, 32(4): 489 - 497。10.1073 / pnas.0403984101。
- 35.
湾D:苔藓小立碗。植物学报,2005,39:339-358。10.1146 / annurev.genet.39.073003.110214。
- 36.
Cove D, Bezanilla M, Harries P, Quatrano R:苔藓作为代谢和发育研究的模型系统。植物学报,2006,29(4):349 - 349。10.1146 / annurev.arplant.57.032905.105338。
- 37.
能源部联合基因组研究所。[http://www.jgi.doe.gov]
- 38.
Rensing SA, Lang D, Zimmer AD, Terry A, Salamov A, Shapiro H, Nishiyama T, Perroud PF, Lindquist EA, Kamisugi Y,等人:小立虫基因组揭示了植物征服土地的进化洞察力。2007年的科学。
- 39.
Carey RE, Cosgrove DJ:小立碗菌膨胀素超家族的画像:与被子植物膨胀素的比较。安·博特(伦敦)。2007, 99(6): 1131-1141。10.1093 / aob / mcm044。
- 40.
紫穗草AC:植物水通道蛋白的早期多样化。植物科学进展,2002,7(12):529-530。10.1016 / s1360 - 1385(02) 02365 - 8。
- 41.
Lienard D, Durambur G, Kiefer-Meyer MC, Nogue F, Menu-Bouaouiche L, Charlot F, Gomord V, Lassalles JP:水孔蛋白在现有植物小立碗的水分输送。植物科学进展,2008,29(3):344 - 344。10.1104 / pp.107.111351。
- 42.
李志刚,李志刚,李志刚,李志刚。小立穗苔藓中密码子编码基因的研究进展。BMC基因组学杂志,2005,6(1):43-10.1186/1471-2164-6-43。
- 43.
李志刚,李志刚,李志刚:液泡体内源性蛋白异构体对液泡功能的影响。植物学报,1999,11(10):1867-1882。10.1105 / tpc.11.10.1867。
- 44.
贝克尔B:液泡室在绿藻和陆生植物中的功能和进化。中国生物医学工程学报,2007,26(3):344 - 344。
- 45.
Hunter PR, Craddock CP, Di Benedetto S, Roberts LM, friigerio L:拟南芥液泡体和液泡腔的荧光报告蛋白识别单个液泡室。中国生物医学工程学报,2007,34(4):379 - 379。10.1104 / pp.107.103945。
- 46.
Olbrich A, Hillmer S, Hinz G, Oliviusson P, Robinson DG:大麦和豌豆根分生组织新形成的液泡具有蛋白质储存和溶解液泡的特征。中国生物医学工程学报,2007,27(4):344 - 344。10.1104 / pp.107.108985。
- 47.
傅瑞斯特。KL, Bhave M:植物主要内在蛋白(MIPs):一个对植物表型有重要影响的复杂基因家族。中国生物医学工程学报,2007,7(4):263-289。10.1007 / s10142 - 007 - 0049 - 4。
- 48.
王晓明,王晓明,王晓明,等。盐生植物盐胁迫根中2个新结节蛋白的分离及其对甘油渗透酶活性的影响。植物科学学报,2004,26(3):344 - 344。10.1016 / j.plantsci.2003.11.001。
- 49.
韦弗CD,罗伯茨DM:大豆结节蛋白26的钙依赖性蛋白激酶磷酸化位点的测定。生物化学,1992,31(37):8954-8959。10.1021 / bi00152a035。
- 50.
爱泼斯坦E:硅。植物化学学报,1999,30(3):344 - 344。10.1146 / annurev.arplant.50.1.641。
- 51.
Horton P, Park KJ, Obayashi T, Fujita N, Harada H, Adams-Collier CJ, Nakai K: WoLF PSORT:蛋白质定位预测器。核酸研究。2007年,w585 - 587。10.1093 / nar / gkm259。35 Web服务器
- 52.
Tuskan GA, Difazio S, Jansson S, Bohlmann J, Grigoriev I, Hellsten U, Putnam N, Ralph S, Rombauts S, Salamov A,等:毛杨(Torr. trichocarpa)基因组的研究。&灰色)。科学通报,2006,29(3):344 - 344。10.1126 / science.1128691。
- 53.
Johnston JS, Pepper AE, Hall AE,陈志杰,Hodnett G, Drabek J, Lopez R, Price HJ:十字花科植物基因组大小的进化。安·博特(伦敦)。2005, 35(1): 1 - 5。10.1093 / aob / mci016。
- 54.
海曼JB, Engel A:水通道蛋白序列的结构线索。中华生物医学杂志,2000,295(4):1039-1053。10.1006 / jmbi.1999.3413。
- 55.
EMBL-EBI SRS主页。[http://srs.ebi.ac.uk]
- 56.
Embl-ebi SRS ftp。[ftp://ftp.ebi.ac.uk/pub/databases/embl/align/]
- 57.
EMBL核苷酸序列数据库。[http://www.ebi.ac.uk/embl/]
- 58.
NCBI。[http://www.ncbi.nlm.nih.gov]
- 59.
Swofford D: PAUP*:使用简约(*和其他方法)的系统发育分析。2000年,马萨诸塞州桑德兰:辛纳尔联合公司,4.0b10
- 60.
Ronquist F, Huelsenbeck JP: MrBayes 3:混合模型下的贝叶斯系统发育推断。生物信息学,2003,19(12):1572-1574。10.1093 /生物信息学/ btg180。
- 61.
Jones DT, Taylor WR, Thornton JM:从蛋白质序列中快速生成突变数据矩阵。计算应用生物科学,1992,8(3):275-282。
- 62.
TreeView:在个人电脑上显示系统发育树的应用程序。计算应用生物科学,1996,12(4):357-358。
确认
我们非常感谢美国能源部联合基因组研究所对Physcomitrella金属盘并将序列公之于众。我们也要感谢协会。Nils Cronberg教授对苔藓的宝贵讨论,Virginia Balbi博士和Laura Saavedra博士对联合基因组研究所PpDB的介绍。这项工作得到了瑞典环境、农业科学和空间规划研究委员会(Formas;拨款给U.J.)。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
JÅHD进行了资料的获取、分析、解释和稿件的起草。UJ构思了这项研究,并帮助解释了数据。两位作者都参与了这项研究的设计和修改手稿,他们都阅读并批准了最终的手稿。
电子辅助材料
12870 _2007_253_moesm1_esm.pdf
附加文件1:显示PpMIPs、AtMIPs和ZmMIPs对齐的图。阴影表示氨基酸在某一位置的保存程度。实际的对齐可以从EMBL对齐数据库中以“ALIGN_001168”的形式获得。(pdf 29kb)
12870 _2007_253_moesm2_esm.tre
附加文件2:使用贝叶斯推断方法和数据集ALIGN_001168的系统发育树(nexus格式)。(tr7kb)
12870 _2007_253_moesm3_esm.tre
附加文件3:使用简约方法和数据集ALIGN_001168引导多数共识系统发育树(nexus格式)。(tre 4kb)
12870 _2007_253_moesm4_esm.tre
附加文件4:使用简约方法和数据集ALIGN_001168的系统发育树(nexus格式)。(tre 6kb)
12870 _2007_253_moesm5_esm.tre
附加文件5:使用邻居连接方法和数据集ALIGN_001168引导多数共识系统发育树(nexus格式)。(tre 3kb)
12870 _2007_253_moesm6_esm.tre
附加文件6:使用邻居连接方法和数据集ALIGN_001168的系统发育树(nexus格式)。(tre 5kb)
12870 _2007_253_moesm7_esm.tre
附加文件7:使用简约方法和数据集ALIGN_001169的系统发育树(nexus格式)。(tre 3kb)
12870 _2007_253_moesm8_esm.tre
附加文件8:使用邻居连接方法和数据集ALIGN_001169的系统发育树(nexus格式)。(tre 2kb)
12870 _2007_253_moesm9_esm.tre
附加文件9:使用简约方法和数据集ALIGN_001170的系统发育树(nexus格式)。(tre 2kb)
12870 _2007_253_moesm10_esm.tre
附加文件10:使用邻居连接方法和数据集ALIGN_001170的系统发育树(nexus格式)。(tre 2kb)
12870 _2007_253_moesm11_esm.tre
附加文件11:使用简约方法和数据集ALIGN_001171引导多数共识系统发育树(nexus格式)。(tre 3kb)
12870 _2007_253_moesm12_esm.tre
附加文件12:使用简约方法和数据集ALIGN_001171的系统发育树(nexus格式)。(tre 3kb)
12870 _2007_253_moesm13_esm.tre
附加文件13:使用邻居连接方法和数据集ALIGN_001171引导多数共识系统发育树(nexus格式)。(tre 2kb)
12870 _2007_253_moesm14_esm.tre
附加文件14:使用邻居连接方法和数据集ALIGN_001171的系统发育树(nexus格式)。(tre 3kb)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
能源部,正当,Johanson, U. Unexpected complexity of the Aquaporin gene family in the mossPhyscomitrella金属盘.BMC植物生物学8日,45(2008)。https://doi.org/10.1186/1471-2229-8-45
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-45
关键字
- 棒曲霉素
- 邻居加入
- 内在蛋白
- TBLASTN搜索
- 主要内在蛋白质