- 研究文章
- 开放获取
- 发表:
罗布斯塔咖啡基因组微卫星新标记的开发(Coffea canephoraPierre ex A. Froehner)在遗传研究中表现出广泛的跨物种转移性和实用性
BMC植物生物学体积8,文章号:51(2008)
摘要
背景
种特异性微卫星标记是遗传研究和利用mas育种进行遗传改良的理想方法。咖啡是最重要的饮料树作物之一,这种标记的可用性有限,因此需要开发更多的微卫星标记,以便有效地应用于遗传分析和咖啡改良项目。本研究旨在开发新的咖啡特异性SSR标记,并验证其在遗传多样性分析、个体化、连锁定位和可转移性方面的实用性,以用于其他相关分类群。
结果
小插入部分基因组文库Coffea canephora采用传统的Southern杂交方法,对不同SSR基序进行了研究。重复阳性克隆的特征表明,DNRs (1/15 Kb)的丰度高于TNRs (1/406 Kb)。不同dnr的相对频率为AT >> AG > AC, tnr中以AGC重复次数最多。利用SSR阳性序列设计了58对引物,其中44对引物可作为阿拉比卡和罗布斯塔基因型的单位点标记进行验证。分析显示,在测试的arabica和robusta中,平均每个标记分别有3.3和3.78个等位基因和0.49和0.62个PIC。它还揭示了所有标记的高累积PI使用兄弟姐妹为基础(106和10-12年分别为arabicas和robustas)和无偏校正估计(10-20年和10-43年分别为阿拉比卡和鲁布斯塔)。这些标记进行了Hardy-Weinberg平衡、连锁不平衡测试,并成功地用于确定所测种质(栽培和种)的一般多样性/亲缘性。在robusta连锁图谱上可映射9个标记。重要的是,这些标记在咖啡相关物种/属之间显示出92%的可转移性。
结论
利用传统的基因组文库方法开发了44个新的咖啡基因组微卫星标记,但效率较低。新标记的鉴定/验证表明,它们具有很高的信息性,有助于遗传研究,即咖啡种质资源的遗传多样性,种质保护的个体化/条形码,连锁作图,分类学研究,以及作为咖啡二级基因库的保守同源集使用。此外,不同SSR基序在咖啡基因组中的相对频率和分布表明咖啡基因组在微卫星中相对较差。
背景
咖啡树,是茜草科的一员,属Coffea其中包括100个物种。在这两个物种中,四倍体Coffea阿拉比卡L.(即阿拉比卡咖啡;2n = 4x = 44)和二倍体c . canephoraPierre ex A. Froehner(即罗布斯塔咖啡;2n = 2x = 22),用于商业种植。咖啡是最受欢迎的无酒精饮料之一,世界上40%的人口经常饮用,主要是在发达国家[1],因而在世界社会经济中占有战略地位。
全球为改善咖啡所做的努力虽然取得了成功,但由于各种因素,已被证明过于缓慢和受到严重限制。后者包括:遗传和生理组成(arabica的遗传多样性和倍性障碍低,robustas的自交不亲和/容易跨种受精),长代周期,对巨大土地资源的需求,同样缺乏易于获得和检测的遗传工具/技术进行筛选/选择。这种情况要求求助于更新、简单、实用的技术,这些技术可以为育种工作提供加速、可靠性和方向性,并允许对栽培/次生基因库进行表征,以便在遗传改良计划中适当利用现有种质资源。在这种背景下,DNA标记工具的开发和基于标记的分子连锁图的可用性对于基于mas的改良咖啡基因型的加速育种至关重要。
在不同类型的DNA标记中,基于短序列重复序列(SSR)的微卫星标记因其多等位基因性质、高多态性含量、位点特异性、可重复性、实验室间可转移性和易于自动化而有望成为最理想的标记[2].微卫星标记已被开发用于大量植物物种,并越来越多地用于确定种质多样性、连锁分析和分子育种[3.].尽管有这些优势,但迄今为止只有大约180个微卫星标记被报道用于咖啡[4- - - - - -12],表明需要扩大这些遗传信息丰富的标记库,以有效管理和改进咖啡种质资源。在这里,我们报告了一组44个新的微卫星标记,通过放射性筛选小插入部分基因组文库c . canephora(罗布斯塔咖啡)。有趣的是,所有这些标记都表现出广泛的跨物种转移性。我们还证明了它们作为遗传标记在确定种质多样性、基因型个性化、连锁定位和分类亲缘性方面的应用。
结果
本研究旨在分离新的咖啡特异性信息性ssr,作为咖啡基因组特征和连锁定位研究的遗传标记。为此,从商业栽培的罗布斯塔品种“Sln-274”构建了部分小插入基因组文库。利用放射性SSR寡核苷酸探针对文库进行筛选,分离出含有SSR的DNA片段,并对这些片段进行测序,用于从侧翼区域设计引物对,随后转化为基于pcr的SSR标记。设计的引物对被标准化用于PCR扩增,然后使用精英咖啡基因型板、用于连锁研究的映射群体和用于跨种转移的咖啡相关分类群验证其作为遗传标记的实用性。此外,利用筛选的和推测的SSR阳性克隆的序列数据,评估了罗布斯塔咖啡基因组中不同SSR基序的相对丰度。共开发了44个新的高信息性SSR标记。
Sln-274小插入部分基因组文库SSR阳性基因组序列的筛选与鉴定
从罗布斯塔品种Sln-274构建的小插入部分基因组文库包含15744个克隆。测序和印迹克隆的放射性筛选显示446个假定阳性,其中199个克隆获得了高质量的序列数据。测序克隆的平均插入尺寸为773.5 bp。考虑到后者,并且测序的克隆就大小而言是基因组文库的随机样本,克隆基因组的总大小为12.2 Mb,相当于罗布斯塔咖啡基因组的1.5% [13)(表1).利用MISA检索模块对克隆序列进行SSR检索,共检测到76个真实SSR阳性克隆(占总文库的0.48%),均含有靶向和非靶向SSR基序。总的来说,这些克隆包含92个SSRs,包括DNRs(48.3%)、TNRs(25.9%)和HO-NRs(4.8%), 24个SSRs仅包含MNRs(20.7%)(表2)1,2).在目标重复序列(筛选的ssr -寡核苷酸)中,AG重复序列含量最高(26.7%),其次是AC(12.9%)和AGC(7.8%),而CCG(0.9%)含量最低,完全没有检测到ACT(表2)2).同样,在除MNRs之外的非靶向SSR基序中,AT重复次数最多(8.6%,表2).
咖啡基因组中SSRs的频率和分布
共评估了76个靶向DNRs (DNRs和TNRs)和10个非靶向DNRs的长度、在现有文库中的分布以及它们在罗布斯塔基因组中的相对丰度(表2)2).dnr和tnr的平均长度(以重复单位计)分别为9.6和5.9。在dnr中,AT和AG具有可比性,且比AC更长,而ACG和AGC是tnr中最长的2).克隆/筛选的基因组文库的大小和已识别SSRs的观察数据与早期预测的罗布斯塔基因组大小一起被考虑[13]以获得罗布斯塔基因组中不同SSR基序的频率/分布的相对估计值。分析显示,咖啡基因组在AT型dnr (AT- dnr)中富集,估计比任何其他SSR基序(靶向和/或非靶向)多出许多倍。结果表明,罗布斯塔基因组每16 Kb (1/16 Kb)有一个AT-DNR;这几乎是第二多的DNR的20倍即.AG(约1/393 Kb)。当包括AT(占dnr总数的94%)时,dnr作为一个单一类别估计为1/15 Kb基因组,其余dnr为1/265 Kb咖啡基因组。相比之下,tnr的总频率计算为1/406 Kb,其中AGC是最主要的(约1/1300 Kb), CCG最少(约1/12200 Kb)。此外,还发现了其他一些高阶ssr(主要是AT-rich),但由于它们的数量非常低,因此没有用于估计计算。因此,本研究表明,每15 Kb的罗布斯塔咖啡基因组中有1个SSR (DNR或TNR)的丰度,其中DNR的丰度是TNRs的27倍。
微卫星标记的发展
以“SSR motif长度”(DNRs和高阶SSRs分别≥7和5重复)为主要标准,尝试设计所有鉴定出的SSR阳性序列,以设计引物对转化为microsat标记。结果,92个鉴定出的ssr中只有56个(MNRs除外)适合引物设计,引物适用性为60.9%。这些药物包括42.2%的DNRs, 40.7%的复合SSRs, 6.8%的TNRs, 5.1%的TtNRs和1.7%的HNRs。此外,还对随机选取的14个MNRs中的2个设计引物,以测试其转化为SSR标记的潜力。在不适合引物设计的ssr中,70.6%的基序长度较短,29.4%的ssr侧翼区域不适合引物建模。在设计的58对潜在引物中,52对可以成功扩增,其中44对可以进一步验证(表2)3.,4)作为有用的标记,表明引物与标记的转化率为~76%。
用于遗传研究的微卫星标记的验证
种质特性
等位基因多样性、杂合度状况及多态性程度
为了确定遗传标记的有用属性,所有新的44个微卫星标记都在16个优秀的罗布斯塔和阿拉比卡基因型上进行了测试。除了CaM54没有对阿拉比卡进行任何扩增外,所有被测基因型的标记都获得了良好的等位基因扩增。总的来说,新标记显示出低到中等程度的等位基因多样性,其中13个标记(CaM02、06、15、18、21、31、34、35、39、43、55、57、58)在所有被测阿拉比卡中均出现双等位基因。总的来说,最多有6和7个等位基因(N一个),平均获得2.7个和3.8个等位基因/标记,其中83.7%和90.9%分别为阿拉比卡和罗布斯塔基因型的多态性/信息4).阿拉比卡的7个标记(CaM08, 09, 11, 12, 22, 23, 53)和粗壮卡的4个标记(CaM11, 13, 15, 23)是单态的。各多态性标记扩增的等位基因数量分布(P米)的基因型(峰度:1.19,偏度:1.22)与健壮型(峰度:-1.08,偏度:-0.57)高度偏倚,如图所示1.
在测试的基因型中,新标记的PIC值变化很大。阿拉比卡的平均PIC值为0.49(范围为0.12 - 0.81),显著低于罗布斯塔的0.62(0.23 - 0.83)(表2)4,图1 b).此外,学生的t检验显示,在扩增等位基因的总数(N一个)和阿拉比卡和罗布斯塔基因型PIC值的估计(N一个: t = 3.18, P = 0.00, PIC: t = 3.46, P = 0.00)。
以上SSR等位基因数据用于计算杂合度估算值时,发现阿拉比卡的观测杂合度与预期杂合度之间存在极显著差异(平均Ho: 0.29,平均He= 0.50;配对t值= 3.64;P = 0.00)以及鲁棒菌(平均Ho: 0.52平均He: 0.63;配对t值= -2.54;P = 0.01)。结果表明,两个种质集均存在明显的杂合子缺陷。此外,23个P米在阿拉比卡的情况下,发现s(62.5%)处于HW平衡,而其余8个表现出明显的杂合子缺陷(表2)4)证实了杂合度数据。同样,在粗壮中,41 P的28 (65.2%)米s被发现在HW平衡和剩余的14个P米在S中,8个标记表现为显著杂合子缺失,6个标记表现为杂合子过剩。
对所有P进行了LD测试米s中,arabica和robusta的不平衡率分别为29.8%(82 / 275)和25.0% (202 / 780)(P < 0.05)。平均每个P米与3.4 (SD:±2.4,SE:±0.51)其他P米对于阿拉比卡,s为4.9 (SD:±4.0,SE:±0.63)。在阿拉比卡的标记CaM24(与其他6个标记)和粗壮树的标记CaM26(与其他8个标记)中观察到最大LD。
新型SSR标记的鉴别能力(个体化能力)
通过计算两种类型的“身份概率”(PI)估计,即兄弟姐妹为基础的和无偏的,分别考虑被测种质是相关的或不相关的,从而推断出所有新的信息性SSR标记对可能的基因型个性化的鉴别能力。PI估计值(表5),显示个体标记的基于兄弟姐妹的PI值在10左右-1对于阿拉比卡和robusta,而无偏PI估计值从10-1- 104阿拉比卡和10-1- 103罗布斯塔。相比之下,与阿拉比卡相比,测试的罗布斯塔基因池的累积pi表明新标记的鉴别能力要高得多。以兄弟姐妹为基础,计算出10、20和最具信息量的标记总数(arabica为23个,robustas为40个)的累积pi为:4.28 × 104, 8.39 × 106, 5.29 × 106阿拉比卡,5.1 × 105, 1.81 × 108, 1.22 × 10-12年罗布斯塔。同样,可比性无偏累积PI估计值为:2.14 × 10-15年, 4.59 × 10-20年, 1.09 × 10-20年阿拉比卡,2.68 × 10-20年, 4.54 × 10-32年, 2.05 × 10-43年罗布斯塔。
新型SSR标记的可映射性
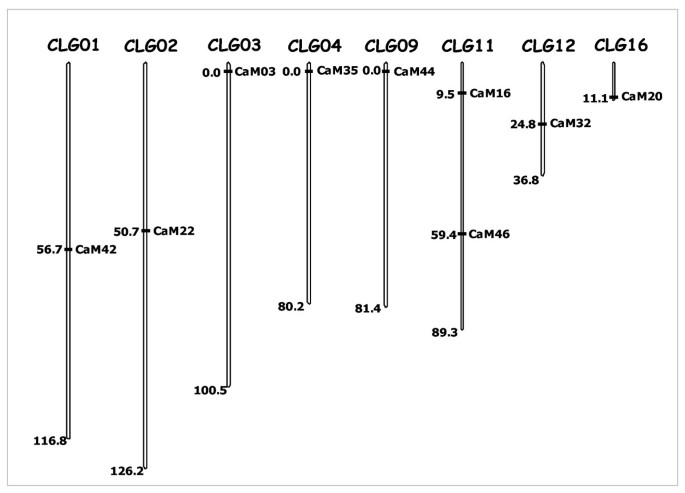
对新SSR标记在罗布斯塔连锁图谱上的拟合性进行了测试。总的来说,44个新标记中有9个(20.5%)被发现对罗布斯塔伪测试杂交定位群体的亲本具有多态性即.CXR和Kagganahalla。CaM03、16、20、22、32、35、42、44和46这9个标记可以在我们开发的罗布斯塔连锁图谱上映射[12].值得注意的是,其中7个标记(除了CaM16和CaM46)被定位在独立的LGs上,这表明新的标记随机分布在罗布斯塔基因组上(图2、表3.).
跨种/属转移性和引物保护
对13个相关的罗布斯塔衍生ssr标记进行了跨种转移性测试Coffea和两个Psilanthus物种。一般来说,这些标记在被测类群中与相同大小的等位基因产生了稳健的跨种扩增(表2)4).总的来说,观察到的平均可移植性为92%(表6,7),则为Coffea(> 93%)Psilanthusspp。(~ 82%)。此外,在不同的Coffea在分类单元中,不同植物亚段间的可移植性相当(> 91%)。因此,这些数据表明,相关咖啡品种的标记保守性非常高,在所有测试的标记中计算出约为91%。标记CaM54的保守性最低,为23%Coffea物种(Species)和27%(在所有类群中),而24个标记被发现100%保守。数据还揭示了一些私人等位基因(pa)的存在,这可能是物种特有的。总共发现了104个这样的等位基因Coffea(平均8.7 pa /种)Psilanthus种(17.5 PAs/种)均高于44个标记。这些占扩增等位基因的34%Coffea和45%的在Psilanthusspp。
栽培咖啡和野生咖啡种质之间的亲和性
通过研究野生和栽培基因库的变异和相互关系,探讨了二倍体微卫星数据在遗传多样性研究中的应用潜力。利用SSR等位基因数据计算的平均遗传距离值为0.26 (SD:±0.06;Se:±0.01),0.43 (sd:±0.06;SE:±0.01)和0.51 (SD:±0.17;SE:±0.02)。类似的估计计算不同Coffea而且Psilanthus种数:0.57 (SD:±0.12;红咖啡(二倍体+四倍体)SE:±0.04,0.54 (SD:±0.07;红咖啡(二倍体)SE:±0.05,0.58 (SD:±0.05;莫桑比克菌SE:±0.02),0.63 (SD:±0.09;Pachycoffea的SE:±0.02),Paracoffea的SE: 0.65(只有两种,因此没有SD), 0.72 (SD:±0.10;SE:±0.01)。
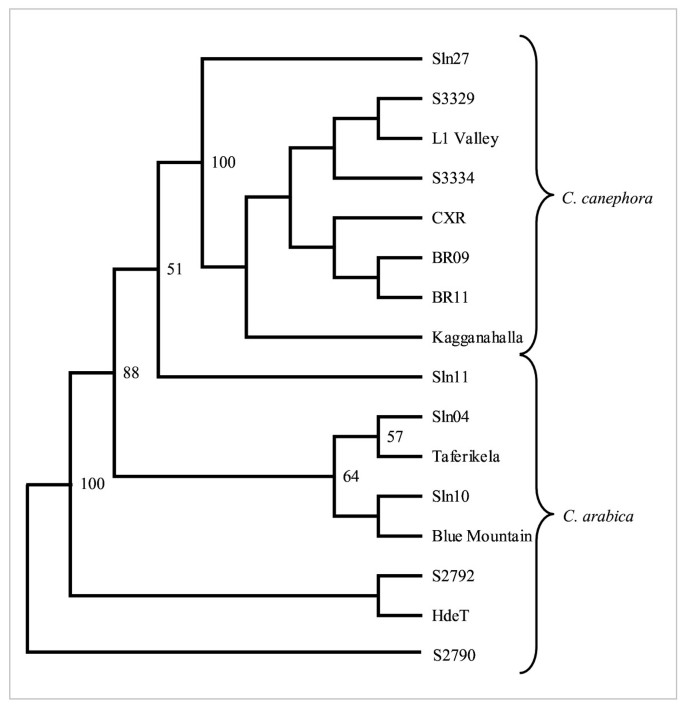
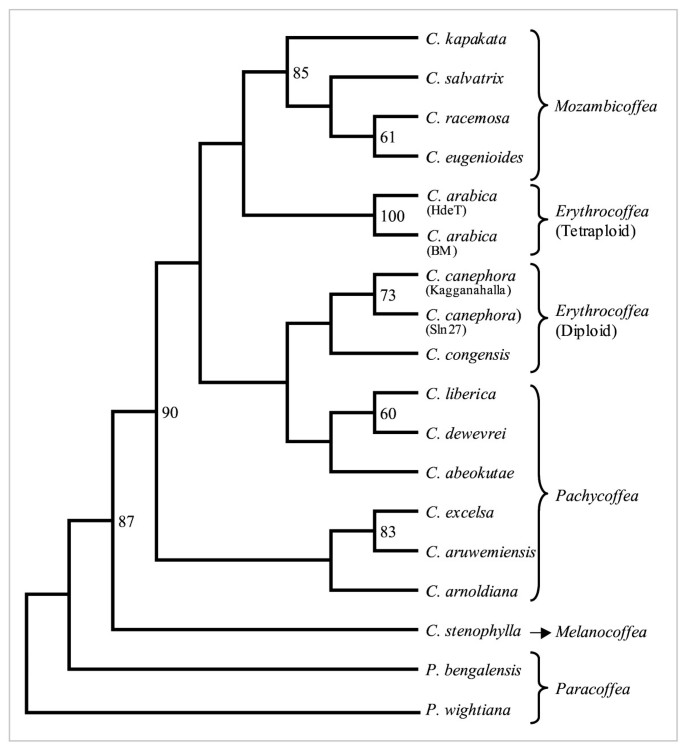
使用来自阿拉比卡和罗布斯塔的8种基因型的遗传距离估计生成的NJ表型树清楚地将测试种质分为两个不同的集群,一个代表所有的四倍体阿拉比卡,而另一个包括所有的二倍体健壮型(图)3.),并有重要的分支支援。纯阿拉比卡的选育在阿拉比卡中形成单一的聚类,而杂交种的选育则形成不同的群体。HdeT位点与S2790和S2792位点距离最近,而Sln11位点与arabica位点距离最远。类似地,对14个相关物种(12Coffea和两个Psilanthusspp。数字4)以及来自的两种基因型c·阿拉比卡而且c . canephora形成连贯的二倍体红咖啡(c . canephora,c . congensis)、四倍体红咖啡(c·阿拉比卡)、莫桑比克(c . racemosa,c . eugenioides,c . salvatrix,c . kapakata)及肿咖(c . liberica,c . dewevrei,c . abeokutae作为一个集群c . excelsa,c . arnoldiana,c . aruwemiensis作为其他集群)。Melanocoffea的单个条目,表示为c . stenophylla最具分歧的是Coffea种类,并显示与Paracoffea部分的入口接近(Psilanthusspp)。
讨论
SSR基序的分布和丰度
本研究中描述的咖啡特异性SSR标记是采用传统方法构建/筛选部分小插入基因组文库的方法开发的。任何微卫星开发工作的成功率由文库中含有SSR的克隆的比例、随后检测到的SSR数量、SSR基序的质量以及侧翼区域的质量表示。在本研究中,获得了76个优质SSR阳性无性系,共116个SSR标记1,3.).因此,结果表明,在识别潜在目标ssr阳性克隆的成功率为0.48%,在整体标记开发的成功率为0.28%。在一项评估传统文库筛选方法在16个不同植物属中微卫星标记发育成功的代表性研究中,发现ssr阳性无性系的比例在种与种之间差异显著(0.059%至5.8%,平均2.5%)[14].在本研究中观察到的SSR检测效率与之前的报道相当Acasia(0.32%, (15])及花生(0.43%,[16]),但高于大米(0.22%,[17]),马铃薯,(0.06至0.15%,[18])及小麦(0.11% [19]),而低于白云杉(0.62%,[20.])。
从这项研究中得出的估计表明,在罗布斯塔咖啡基因组中,不同SSR rs的相对分布在总体SSR丰度上相对较差(1/160 Kb为目标SSR, 1/15 Kb包括非目标SSR;表格2)与其他植物种类相比,例如拟南芥、大米、大麦(每6-8 Kb 1个)[21]和mulberry(我们未发表的数据)。然而,不同类型的基因组SSRs在咖啡基因组中的相对频率、重复长度和分布模式(表2)2)的含量与苹果[22], avacado [23],桦树[24],桃子[25),Acasia[15]和番茄[26].其中AG的检出比例高于AC(近2倍);一般来说,AG重复核被发现比任何其他SSR类型都要长。TNR的重复核总体上小于DNR,与其他DNR或TNR相比,AT(非靶向SSR)是最丰富的。相比之下,咖啡基因组中富含at的tnr的丰度相对低于大多数植物物种[16,27,28],但与一些树种,如鳄梨(ACC > AGG > AAG, [23])和桃子(含有丰富的AGG, [25])。TNR丰度的物种特异性模式也在近亲物种中得到证实,如属于同一科的水稻和小麦,但它们的基因组TNR含量有显著差异[29- - - - - -31].如上所述,在不同的研究中,包括目前关于咖啡的研究,在SSR估计(相对频率、分布和丰度)中所看到的一些变化可以归因于用于SSR搜索的标准的差异即尽管不同物种SSR的基因组组织存在先天的差异,但SSR的主要特征包括:重复核的最小长度、筛选的基因组文库大小、筛选的严格程度、用于筛选的寡核苷酸和SSR挖掘工具。
基因组SSRs的相对丰度/分布与我们早期从咖啡转录组中开发的基因-SSRs的比较[11,揭示了两个显著的差异即在两种类型的SSRs中,转录组中SSRs的丰度(1/2.16 Kb)明显较高,TNR丰度/相对分布几乎相反。重要的是,在遗传SSRs中最丰富的两个TNRs (AAG, ACT)在基因组SSRs中最不丰富或未检测到。这一观察结果将提示tnr的差异分布/组织的有趣可能性,以及用于文库构建的酶在咖啡基因组中基因丰富和基因缺乏区域的限制位点。然而,这种可能性只能通过今后进一步详细的基因组研究来解决。
新型SSR标记的开发
在咖啡中,据我们所知,迄今为止只有大约180个基因组ssr在文献中被描述[4- - - - - -11]需要继续努力开发更多的新标记,以扩大现有的曲目,以便在咖啡的遗传研究中有效部署。在这项研究中,63%的检测到的ssr被发现可用于引物设计/标记转换,与报道的苹果相比,成功率要高得多(30% [22])、木薯(37.7% [32]),大caninus(11.1% (33])、燕麦25.2% [34]及马铃薯(26.9% [18])。导致36个ssr不适合引物设计的两个主要序列属性是:较短的重复核心和不适合引物建模的低复杂性侧翼区域(AT/ gc丰富和/或易于形成二级结构的区域)。有趣的是,在目前的研究中,甚至没有一个失败是由于ssr核位于克隆序列的末尾,据报道,这是许多早期木薯、番茄、燕麦和杉木研究中的一个主要限制因素[26,32,34,35].本研究中所观察到的引物设计成功率较高,限制因素较少,这可能是由于限制性内切酶的适用性较好,以及用于基因组文库构建的基因组片段相对较长(0.5 - 1.5 kb)。用于构建基因组文库/ ssr标记开发的基因组片段大小的重要性早前也在花生中得到证实[16].
设计的引物成功生产扩增产物的比例给出了引物-标记的转化率,并表明文库建设工作的最终成功。在本研究中,设计的58对引物中,44对可以被验证为有效的ssr标记(见表)3.,4,以及以下部分的讨论),从而得到约75.8%的引物-标记转换率,与许多早期基于基因组文库的传统研究大致相当即.,葫芦[36),大[33],花生[16],西红柿,[26]和米[17].据报道,道格拉斯冷杉的引物-标记可转换性最低(4.1%),这可能是由于针叶树基因组独特的复杂性[35,37- - - - - -39].此外,对文献的调查表明,一般来说,苹果、桃子等小基因组的转换率较高,基因组大小与SSR引物扩增效率呈负相关关系,这是由于机理原因[40].
本文描述的44个新SSR标记中的两个(CaM49, 55)基于MNR重复序列。一般来说,这些标记需要更严格的评估,以确定其个体等位基因/大小,在许多情况下不容易与相似大小的混淆口吃扩增子区分(数据未显示)。因此,避免使用这种基于mnr的标记可能是谨慎的,尽管这些标记是有信息的,除非没有其他标记可用。
新ssr作为遗传标记的应用
到目前为止,有一些研究描述了咖啡特异性SSR标记的发展[4- - - - - -11];然而,其中只有少数研究为新SSRs在遗传研究中的应用提供了数据[8,11].因此,本研究的一个主要目的是测试所报道的新标记在栽培咖啡种质遗传多样性、连锁定位、构建基因型个性化参考板/条形码、跨种转移性和相关类群的分类学关系等研究中的应用潜力。
种质特性
等位基因多态性和遗传多样性水平
各种遗传参数即,等位基因多样性,PIC, HoHe计算了所有新ssr的峰度/偏度、HWE、LD,充分证明了其作为遗传标记的实用性(见表1)4).总的来说,这些标记显示出低到中等程度的等位基因/遗传多样性,这是相当的,在某些情况下比先前描述的咖啡基因组SSRs报告的更多[6,8],正如预期的那样,总是高于基因ssrs [10,11,41].不同标记扩增的等位基因总数在被测阿拉比卡和粗壮中几乎相同;然而,对于粗壮菌来说,PIC值越高,标记的信息量就越大。此外,值得注意的是,13个测试标记在所有测试的阿拉比卡中扩增了两个不同但大小相似的等位基因,这表明这是阿拉比卡基因组中固定位点重复的结果。上述观察结果可能考虑了两种咖啡的繁殖行为、基因组进化和驯化过程。由于与阿拉比卡相比,鲁巴斯塔的异交行为预计在遗传上更加多样化(导致测试标记的PIC更高),而阿拉比卡是自亲和的,也已知由于驯化过程中的遗传瓶颈而遭受狭窄的遗传基础[8,11].同样,阿拉比卡基因组的重复位点是可能的,因为它是由两个同源二倍体基因组杂交产生的异体四倍体(c . eugenioides而且c . canephora),然后是二倍体化和稳定化[42].
不同的遗传参数/测试,如HoHe、LD、HWE是有效基因库中多样性起源、演化和分布的重要指标。杂合度测定(HoHe),表明所测种质存在明显的杂合子衰变(缺陷)。峰度/偏度参数表明,新ssr的等位基因多样性不服从正态分布。同样,多态性标记的HWE和LD分析(P米S)只透露了大约2/3理查德·道金斯(63 ~ 65%)的标记在HW平衡中,约25 ~ 29%的标记在所分析的arabica和robusta中表现出显著的LD。这些结果与我们之前对基因组和遗传ssr的观察结果一致[6,10,11],并且确实反映了测试材料的遗传组成和交配行为。总的来说,这些研究表明,所测试的罗布斯塔种质资源由同种异体的、相对不相关的基因型(选择和一个杂交)组成,而自交配的阿拉比卡主要由重叠/共享谱系的杂交品种/选择组成。结果表明,这些新标记可用于确定咖啡基因库的遗传多样性。
SSR新标记的鉴别能力
植物种质资源的个性化在当今的情况下变得非常重要,因为它们的适当管理和利用以及知识产权保护可以通过使用SSRs等高多态性标记的DNA分型技术来实现。使用这种分型方法进行种质鉴定仍然是一个昂贵的主张,特别是如果目标物种如咖啡在其可用基因库中具有非常有限的多样性。为了避免这些问题并提高这些努力的效用,有人建议使用SSRs等强大的标记和通用实验指南,为咖啡种质建立参考DNA多态性数据资源/小组[12,43].这样的参考资源可以很容易地用于咖啡基因型个性化、育种/改良的种质选择以及国际合作中的种质交换[12,43].在这种情况下,确定SSR标记的PI估价值(为区分潜力提供非常有信息的指标)在种质鉴定研究中应用变得非常重要。总的来说,当单独考虑时,新标记的PI估计值范围从低到中等,但当一起测试时,对于基因型区分具有很高的信息量(累积PI)。
此外,研究发现,这些估计值反映了测试种质资源的多样性状况,因此阿拉比卡的估计值明显低于健壮树(表2)5).总的来说,分析表明需要使用3-4倍以上的标记才能在两个咖啡基因库中达到可比的鉴别水平。此外,这些数据表明,从实际的角度来看,计算基于兄弟姐妹的PI(一个更保守的歧视估计)可能是谨慎的,以决定标记的数量,可以为测试种质的个体化提供足够的可变性。这是预期的,因为基于兄弟姐妹的PI低估了由于重叠谱系/共同亲缘关系而产生的目标种质中可能的相似性/相关性。
新SSR标记的可映射性
DNA标记的主要潜在用途之一是它们作为链接基团上的健壮的基因组标记,随后可以标记到控制感兴趣的重要性状的基因上,为基于mas的育种提供了可能性。这需要生成相当密集的连接图,其中填充大量可重复访问的DNA标记,SSRs仍然是最理想的。到目前为止,很少有ssr被映射到罗布斯塔连锁图谱上[7,44]这就需要我们进行广泛的努力,以产生更多可用于连锁分析的SSR标记。在这方面,我们使用罗布斯塔咖啡的伪测试交叉映射群体测试了新标记的连锁映射的适用性。值得注意的是,有20.5%的标记在作图群体的亲本中存在多态性,这些标记均能被成功作图(图2).所映射的标记分布在不同的连锁基团上,其中一些标记映射到lg的末端,这在早期的研究中已经看到[44].因此,这些数据有力地证明了新的标记可以有效地用于咖啡的遗传连锁研究。
跨物种/通用的可转移性
新标记在栽培咖啡基因库中所表现出的中低水平的多样性,被它们的高跨种转移潜力所弥补。当测试13个基因时,所有标记都显示出强大的跨物种/-属扩增Coffea和两个Psilanthus分类单元(表7).数据显示,本文描述的标记显示出比早期发表的基因组SSR标记更好的分类群转移性[6,9,10],但相对少于我们报道的SSR基因标记[10,11].更重要的是,这些标记在相关物种之间显示出了可比性Coffea以及相关属的2种Psilanthus.这很重要,因为成功的跨物种扩增通常仅限于属内的相关物种,而在不同属的测试中则会减少[11,45].此外,有趣的是,所有被测阿拉比卡和罗布斯塔种质资源的单型/无信息的新SSRs在被测相关类群中表现出相当大的多态性。唯一的例外是标记CaM54,即使在整个Coffea因此,本文所描述的新SSR标记增强了它们作为保守同源集(COS)用于不同亲缘野生咖啡类群遗传鉴定以及咖啡分类学/共统性研究的可能性。
咖啡属植物与雪兰属植物间多样性分析及遗传亲缘关系
本研究中描述的基因组ssr,尽管多态性水平较低,但能够将属于两个栽培种质的所有16个基因型进行表型聚类,这表明了物种关系并确认了其已知的谱系(图3.).例如,分析证实了S2790和S2792的相关起源,它们是HdeT和Taferikela的双向杂交。
同样,对20个代表性样本的分析属于14Coffea和两个Psilanthus根据它们的地理分布和Chevalier的植物分类,揭示了与它们已知的分类学关系大体一致的属类亲和性[46)(图4).因此,基于新标记数据的表型树非常清楚地将所分析的相关咖啡物种按照各自的植物亚段进行分组(见结果)。重要的是,分析清楚地区分了两个副咖啡属物种(p . bengalensis而且p . wightiana)从所有其他Coffea。这些结果与较早发表的利用ssr确定物种关系的研究结果相似[8,9,11],以及其他标记方法[47- - - - - -49].密切的关系c . kapakata为莫桑比克咖啡分类单元,是唯一的黑咖啡分类单元c . stenophylla正如本文所见,早前基于EST-SSR和issr的研究也表明了这一点[11,47].这些结果表明,本研究建立的新SSR标记对探索咖啡种复合体的分类关系具有重要的参考价值。
结论
总的来说,本研究描述了44个新的微卫星标记,使用传统的方法构建/筛选部分小插入基因组文库。该方法是成功的,但困难和实验密集,成功率低~0.48%。对鉴定出的SSR阳性基因组克隆的分析提供了对咖啡基因组中不同SSR基序的相对丰度和分布模式的深入了解,与许多其他植物基因组相比,咖啡基因组的SSR丰度相对较低。总体而言,DNRs的丰度高于TNRs,在不同SSR基序中,AT的丰度最高,其次为AG、AC和ACG。TNR CCG含量最低。超过50%的鉴定ssr可以转化为可用的标记,从而获得较高的引物-标记转化率。结果表明,44个标记均具有多态性,可用于多样性分析、种质个性化、连锁定位、跨种转移和分类学研究。因此,本研究通过44个新的SSR标记丰富了现有的咖啡SSR标记库,这些SSR标记不仅适用于栽培咖啡,而且预计同样适用于相关物种的遗传研究,这些物种构成了咖啡改良的重要二级基因库。
方法
植物材料和DNA提取
本研究中16种精英基因型属于c·阿拉比卡而且c . canephora与14种相关的野生物种一起被使用,它们属于Coffea而且Psilanthus属(表7).从印度卡纳塔克邦Balehonnur中央咖啡研究所的种质库中收集每一种咖啡的叶片样本,并按照Aggarwal等人描述的方法分离DNA。[50].
基因组文库的构建及含SSR序列的分离
使用标准程序构建了部分小插入基因组文库[51]从一种优秀的罗布斯塔基因型Sln-274中分离出的总细胞DNA。大约10 μg基因组DNA被消化Rsa我和有III (NEB)限制性内切酶(NEB,美国),并在1%琼脂糖凝胶中分离。取500 ~ 1500 bp的基因组片段,用GFX色谱柱(Amersham)纯化,用T4 dna连接酶连接到pMOS钝端质粒载体(Amersham)上,最后将连接的基因组插入物克隆大肠杆菌DH10B宿主细胞电穿孔。将转化后的细胞培养一夜,分离重组白色菌落,保存在41个384孔微量滴度培养板中,并复制到Hybond-N上+尼龙膜(Amersham Biosciences, USA)获得高密度杂交过滤器用于筛选。所有15744个重组无性系均经γ- Southern杂交32p标记的两个寡核苷酸池(每个由不同的合成寡核苷酸组成,均为等色浓度),即池:(CA)15(GA)15(CAA)10(猫)10(行为)10(叫)10(AGA)10(机票)10;和Pool-II:(CTG)10(GAC)10(gg)10(GGT)10(GCC)10(GC)15.选择杂交无性系,按照标准的碱裂解法进行质粒分离[51].然后,根据制造商的详细信息,使用BigDye™化学技术,在3700 DNA分析仪上对基因组插入物使用M13通用引物进行扩增和测序(应用生物系统公司,美国)。使用Autoassembler (Applied Biosystems, USA)对序列进行对齐和编辑,最后以FASTA格式保存。
标记的发展
使用微卫星搜索模块MISA对测序克隆中的微卫星进行识别和定位(更多信息请参阅下面的可用性和要求部分),然后进行视觉评估。SSR搜索的标准对剧中重复延伸最少为:MNRs有12个重复单位,DNRs有6个重复单位,HO-NRs有5个重复单位。微卫星的分类考虑了重复基序的互补性,如AG, GA, TC和CT被视为一个单一的类别。引物对使用GENETOOL Lite 1.0版本设计,用于包含至少7个dnr序列的SSR,和/或所有其他SSRs的5个重复(更多信息请参阅下面的可用性和要求部分)。引物是商业合成的(Bioserve,印度-更多信息请参阅下面的可用性和要求部分)正向引物具有荧光标签FAM或HEX。这些新标记的细节即,位点指定、引物序列、重复基序、等位基因属性、PIC估计值和Genbank登录数汇总于表中3.,4.引物对标准化,按照前面描述的方法进行PCR [10,11].扩增产物在基于毛细管的3730 DNA分析仪(应用生物系统公司)上运行,并使用GeneMapper 3.7(应用生物系统公司)使用默认参数对产物的主要、可比较和显著峰进行精确大小测定。
统计和遗传分析
利用arabica和robusta的8个基因型的等位基因数据计算不同的统计和遗传参数。统计属性如均值,偏度,峰度,t检验等.使用Microsoft Excel函数实用程序进行计算。观察杂合度(Ho)以杂合基因型占基因型植株总数的比例计算。期望杂合度(He)按以下公式计算[52]:
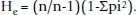
PIC值根据Botstein等计算[53]如下:
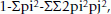
在哪里
N =微卫星标记检测到的等位基因总数,
= i的频率th等位基因,
Pj = (i+1)th所分析的基因型集合中的等位基因。
采用Fisher精确检验和Markov链算法对双等位基因多态性数据进行Hardy-Weinberg平衡(HWE)检验,预测链长为10,000,000步,去记忆步数为100,000步,采用1,000个排列进行连锁不平衡(LD)检验。对于阿拉比卡,在测试种质中显示不变的“双等位基因”存在的标记被认为是来自两个不同副本的重复位点的独立扩增,并被排除在上述等位基因属性的分析之外。HoHe使用Arlequin ver 3.1程序进行估算和HW和LD测试[52],使用Gimlet ver 1.3.2程序计算PI估计[54].私有等位基因(pa)用Convert ver软件测定。1.3.1 [55]在30个基因型中。每个微卫星位点的判别能力通过估计基于兄弟姐妹和无偏校正的PI估计来计算,累积判别能力由Waits等人描述的按递减顺序排列的连续信息标记的PI的乘积来计算。[56].跨类群可移植性(T马克)以扩增成功引物的比例计算相对于而引物保守性(C分类单元)以显示成功扩增的物种所占的比例计算相对于所有测试标记。
通过考虑基因组的随机采样,得到了所检测SSR基序之间的平均基因组距离估计值。因此,对于目标SSRs,样本基因组的大小被认为等于筛选文库的总大小,而对于非目标SSRs,使用实际测序的基因组大小来得到估计,考虑到单倍体咖啡基因组相当于809 Mb [13].最初,通过筛选的靶向SSR基因组和测序的非靶向SSR基因组,估计了罗布斯塔基因组中存在的不同DNRs和TNRs的数量即.AT-DNR。这些结果被进一步用于根据基因组中每Mb的SSR来估计不同SSR的分布,以及罗布斯塔基因组中两个这样的连续SSR重复之间的间隔。
链接映射使用JoinMap ver 3.0构建,LOD 5.0和根据软件指令的其他默认参数。根据JoinMap中指定的伪测试杂交群体共显性标记分离模型,对测试微卫星的分离等位基因数据进行评分。本研究中获得的分离数据与实验室中参考罗布斯塔种群可用的映射数据一起使用(未发表)。
遗传多样性分析
SSR数据来自P米采用基于遗传距离值的聚类分析,确定所测种质(栽培基因型/近缘种)之间的属型关系/亲缘性。最初使用双等位基因微卫星数据分析工具microsatellite Analyzer (MSA)生成100个自举距离矩阵[57]和Nei的遗传距离测量[58].根据这些距离数据,使用Phylip ver 3.6为每个矩阵分别生成邻居连接树[59通过“邻居”命令,然后生成共识树,每棵树用于栽培种质和种间关系。
可用性和需求
1微卫星:http://pgrc.ipk-gatersleben.de/misa/
2 GENETOOL精简版1.0:http://www.biotools.com/downloads/brochures/GeneTool2.pdf
3 .印度Bioserve:http://www.bioserveindia.com/
缩写
- 医嘱:
-
Di-Nucleotide重复
- C分类单元:
-
所测类群标记的保护情况
- 因为:
-
保守正交集
- 他:
-
预期的杂合性
- 何:
-
观察到的杂合性
- HO-NRs:
-
高阶核苷酸重复序列
- 曼:
-
Hexa-Nucleotide重复
- HWE:
-
哈迪温伯格平衡
- Kb:
-
个碱基
- LD:
-
连锁不平衡
- 苏维埃社会主义共和国:
-
简单序列重复
- m:
-
Megabases
- MNRs:
-
Mono-Nucleotide重复
- MSA:
-
微卫星分析
- N一个:
-
等位基因数NJ: Neighbour Joining
- 不是:
-
私有等位基因
- 图片:
-
多态信息内容
- PI:
-
同一性概率
- P米史:
-
多态标记
- tnr:
-
Tri-Nucleotide重复
- T马克:
-
标记在所有研究类群间的转移性
- TtNRs:
-
Tetra-Nucleotide重复。
参考文献
Fitter R, Kaplinsky R:随着咖啡市场变得更加差异化,谁会从产品租金中获益?价值链分析。IDS公报(特刊)。2001, 32(3): 69-82。
Powell W, Machray GC, Provan J:简单序列重复序列揭示多态性。植物科学进展,1996,1:215-222。
Gupta PK, Varshney RK:以面包小麦为重点的微卫星标记在遗传分析和植物育种中的开发和使用。《植物学报》,2000,29(3):344 - 344。
Combes MC, Andrzejewski S, Anthony F, Bertrand B, Rovelli P, Graziosi G, Lashermes P:微卫星基因座的特征Coffea阿拉比卡以及相关的咖啡品种。生物化学学报,2000,9:1178-1180。
Rovelli P, Mettulio R, Anthony F, Anzueto F, Lashermes P, Graziosi G:微卫星Coffea阿拉比卡L.咖啡生物技术和质量。编辑:Sera T, Soccol CR, Pandey A, Roussos S. Kluwer学术出版社;2000:123 - 133。
Baruah A, Naik V, Hendre PS, Rajkumar R, Rajendrakumar P, Aggarwal RK: 9个微卫星标记的分离与表征Coffea阿拉比卡L.,显示出广泛的跨种扩增。化学通报,2003,3:647-650。
Coulibaly I、Revol B、Noirot M、Poncet V、Lorieux M、Carasco-Lacombe C、Minier J、Dufour M、Hamon P: a的AFLP和SSR多态性Coffea种间回交后代[(c . heterocalyxXc . canephora) Xc . canephora].应用物理学报,2003,27(3):344 - 344。
Moncada P, McCouch S:二倍体和四倍体简单序列重复多样性Coffea物种。中国生物工程学报,2004,47:501-509。
Poncet V, Hamon P, Minier J, Carasco C, Hamon S, Noirot M:咖啡树SSR的交叉扩增和变异(Coffeaspp)。中国生物工程学报,2004,47:1071-1081。
Bhat PR, Krishnakumar V, Hendre PS, Rajendrakumar P, Varshney RK, Aggarwal RK:来自robusta咖啡品种“CXR”(一种种间杂交)的表达序列标记衍生的简单序列重复标记的鉴定和表征咖啡与咖啡同源).分子生物学杂志,2005,80-83。
Aggarwal RK, Hendre PS, Varshney RK, Bhat PR, Krishnakumar V, Singh L:咖啡及相关物种基因组分析中est衍生基因微卫星标记的鉴定、表征和利用。应用理论与实践,2007,34(3):359-372。
Hendre PS, Aggarwal RK: DNA标记:咖啡遗传改良的开发与应用。基因组辅助作物改良:基因组学在作物中的应用。主编:Varshney RK, Tuberosa R. Springer-Verlag, 2007, 2: 399-434。
Marie D, Brown SC:植物DNA直方图的流式细胞术练习,70个物种的2C值。中国生物医学工程学报,2003,27(3):344 - 344。[http://data.kew.org/cvalues/searchguide.html]
张志刚,张志刚,张志刚:微卫星分离技术研究进展。分子化学,2002,11:1-16。
张志刚,张志刚,张志刚,张志刚,张志刚。中国植物微卫星标记的开发、遗传及跨种扩增金合欢高阿.应用物理学报,2000,30(3):344 - 344。
Ferguson ME, Burow MD, Schulze SR, Bramel PJ, Paterson AH, Kresovich S, Mitchell S:花生中的微卫星识别和特征(答:hypogaeal .)。应用理论与实践,2004,30(4):344 - 344。
陈旭,徐毅,赵永g, mcouch SR:水稻全基因组微卫星框架图谱的构建(栽培稻l .)。应用理论,1997,95:553-567。
刘志刚,刘志刚,刘志刚,刘志刚。马铃薯微卫星标记及其在系统发育和指纹图谱分析中的应用。基因组学报,2001,44:50-62。
Bryan GJ, Collins AJ, Stephenson P, Orry A, Smith JB, Gale MD:六倍体面包小麦微卫星的分离与表征。应用理论,1997,94:557-563。
杨晓燕,王晓燕,王晓燕,王晓燕,王晓燕,王晓燕。云杉微卫星DNA标记的分离、鉴定、遗传及连锁分析(云杉glauca)以及它们在其他云杉物种上的用处。中国生物医学工程学报,2001,26(6):871-882。
Cardle L, Ramsay L, Milbourne D, Macaulay M, Marshall D, Waugh R:植物中物理聚类简单序列重复的计算和实验表征。中国生物工程学报,2000,29(3):344 - 344。
Guilford P, Prakash S, Zhu JM, Rikkerink E, Gardiner S, Bassett H, Forster R:微卫星马吕斯家蝇(苹果):丰度、多态性及品种鉴定。应用理论,1997,44(4):489 - 497。
Sharon D, Cregan PB, mohammeed S, Kusharska M, Hillel J, Lahav E, Lavi U:牛油果综合遗传连锁图谱。应用理论,1997,32(3):397 - 397。
Pekkinen M, Varvio S, Kulju KK, Karkkainen H, Smolander S, Vihera-Aarnio A, Koski V, Sillanpaa MJ:桦树联动图,桦木属翻车机基于微卫星和扩增片段长度多态性。中国生物工程学报,2005,48:619-625。
Sosinski B, Gannavarapu M, Hager LD, Beck LE, King GJ, Ryder CD, Rajapakse S, Baird WV, Ballard RE, Abbott AG:桃微卫星标记的表征[碧桃(l)类等)。应用理论与实践,2000,30(4):421-428。
Areshchenkova T, Ganal MW:不同来源番茄微卫星标记的多态性和染色体定位的比较分析。应用理论与实践,2002,29(3):344 - 344。
王晓明,王晓明,王晓明:植物和脊椎动物微卫星多态基序的丰度不同。中国生物医学工程学报,1993,21(4):457 - 457。
王震,Weber JL,钟刚,Tanksley SD:植物短串联DNA重复序列研究进展。应用理论,1994,18(1):1-6。
Miyao A, Zhong HS, Monna L, Yano M, Yamamoto K, Havukkala I, Minobe Y, Sasaki T:水稻基因组简单序列重复序列的特征和遗传定位。DNA决议,1996,3:233-238。
Akagi H, Yokozeki Y, Inagaki A, Fujimura T:水稻染色体的微卫星DNA标记。应用理论学报,1996,29(3):379 - 379。
宋庆杰,刘志强,李志强,李志强。小麦三核苷酸SSR基序的研究。应用理论与实践,2002,26(3):366 - 366。
Mba REC, Stephenson P, Edwards K, Melzer S, Nkumbira J, Gullberg U, Apel K, Gale M, Tohme J, Fregene M:木薯SSR标记研究(木薯耐Crantz)基因组:建立基于ssr的木薯分子遗传图谱。应用理论,2001,102:21-31。
孙GL, Salomon B, Bothmer RV:微卫星基因座的表征与分析大caninus(Tritiaceae:禾本科)。应用理论,1998,16(3):366 - 366。
Pal N, Sandhu JS, Domier LL, Kolb FL:燕麦微卫星和rflp衍生PCR标记的开发与鉴定。作物科学,2002,42:912-918。
Slavov GT, Howe GT, Yakovlev I, Edwards KJ, Krutovskii KV, Tuskan GA, Carlson JE, Strauss SH, Adams WT: Douglas-fir的高可变SSR标记:孟德尔遗传和地图位置。应用理论与实践,2004,30(3):344 - 344。
李志强,李志强,张志强,等:微卫星标记的研究进展Cucumis.应用理论,2001,32(1):61-72。
Pfeiffer A, Oliveri AM, Morgante M:挪威云杉微卫星的识别和特征(挪威云杉k)。基因组学报,1997,40:419-
Hicks M, Adams D, O'Keefe S, Hodgegetts R:黑松RAPD和微卫星标记的开发(松果体contortavar。latifolia).中国生物医学工程学报,1998,41:797-805。
杨晓燕,王晓燕,王晓燕,等。中国微卫星基因座的研究进展抗旱性中华生物医学杂志,1998,7:1260-1261。
加纳TW:基因组大小和微卫星:核大小对扩增潜力的影响。基因组学报,2002,45:212-215。
王志刚,王志刚,王志刚。植物基因微卫星标记的研究进展。生物技术,2005,23:48-55。
Lashermes P, Combes MC, Trouslot P, Anthony F, Charrier A:植物起源和遗传多样性的分子分析Coffea阿拉比卡对咖啡改良的影响。热带植物EUCARPIA会议记录。法国蒙彼利埃;1996:23-29。
Aggarwal RK, Rajkumar R, Rajendrakumar P, Hendre PS, Baruah A, Phanindranath R, Annapurna V, Prakash NS, Santaram A, Sreenivasan CS, Singh L:印度咖啡选择的指纹识别和用于创建品种鉴定分子id的参考DNA多态性板的开发。第20届国际咖啡科学会议论文集。印度班加罗尔;2004:751 - 755。
Lashermes P, Combes MC, Prakash NS, Trouslot P, Lorieux M, Charrier A:基因连锁图谱Coffea canephora:分离畸变对雌雄减数分裂的影响及重组率分析。基因组学报,2001,44:589-596。
Peakall R, Gilmore S, Keys W, Morgante M, Rafalski A:大豆跨种扩增(大豆简单序列重复序列(SSRs)在该属和其他豆科属中的意义:SSRs在植物中的可转移性。中国生物医学工程学报,1998,26(4):457 - 457。
骑士甲:全球咖啡馆。3Systematique des caféiers在Faux cafeiers。Maladieset虫易滋扰。生物百科全书28。编辑:Lechevalier P. Paris,法国;1947:356 -
Ruas PM, Ruas CF, Rampim L, Carvaljo VP, Ruas EA, Sera T:遗传关系Coffea利用ISSR (inter-simple sequence repeat)标记确定种间杂交种的种类和亲本。Genet Mol生物学,2003。
Orozco-Castillo C, Chalmers KJ, Powell W, Waugh R: RAPD和细胞器特异性PCR再次确认了属内的分类学关系Coffea.植物细胞学报,1996,15:337-341。
Lashermes P, Combes MC, Trouslot P, Charrier A:咖啡树种的系统发育关系(Coffea由核糖体DNA ITS序列推断。应用理论,1997,94:947-955。
阿加瓦尔,谢诺伊,李志强,李志强,李志强,等:印度香米和其他优良水稻基因型的荧光aflp分子鉴定。应用理论与实践,2002,30(3):366 - 366。
李文杰,李志强,李志强。分子克隆:实验室手册。纽约:冷泉港出版社;1989.
Excoffier L, Laval G, Schneider S: Arlequin ver 3.0:群体遗传数据分析的集成软件包。生物信息学学报,2005,1:47-50。
李志强,李志强,李志强,等:利用限制性片段长度多态性构建人基因连锁图谱。王志刚,王志刚,2004。
Valiere N: Gimlet:分析遗传个体识别数据的计算机程序。生物化学学报,2002,2:377-379。
Glaubitz JC: Convert:一个用户友好的程序,为常用的群体遗传软件包重新格式化二倍体基因型数据。生物化学学报,2004,4:309-310。
Waits LP, Luikart G, Taberlet P:估计自然种群中基因型的同一性概率:注意事项和指南。分子生物学学报,2001,10:249-256。
Dieringer D, Schlotterer C: MicroSatellite analyzer (MSA):大型微卫星数据集的平台独立分析工具。分子生物学杂志,2003,3:167-169。
Nei M:种群间的遗传距离。自然学家,1972,106:238-292。
Felsenstein J: PHYLIP(系统发育推断包)版本3.6。由作者分发。西雅图华盛顿大学基因组科学系;2004.
确认
作者感谢印度政府生物技术部(DBT)在“咖啡基因组学”国家网络项目下的财政支持;CCMB主任对海得拉巴的设施进行了研究;班加罗尔咖啡委员会咖啡材料研究总监;Md Ashraf Ashfaq先生在工作初期提供的帮助;T. Ramakrishna Murthy博士,科学家,CCMB校正和编辑手稿。PSH承认CSIR,印度初级和高级研究奖学金在他的博士研究期间。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
PSH构建、筛选文库、阳性克隆测序;开发并标准化了大部分的标记;验证和分析数据;起草手稿。PR、AL和AV对一些标记物进行了标准化和验证,并有助于分析。RKA负责构思、计划、监督、定稿、批准和沟通最终稿。
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
亨德利,p.s., Phanindranath, R., Annapurna, V.。et al。罗布斯塔咖啡基因组微卫星新标记的开发(Coffea canephoraPierre ex A. Froehner)在遗传研究中表现出广泛的跨物种转移性和实用性。BMC植物生物学8日,51(2008)。https://doi.org/10.1186/1471-2229-8-51
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-51
关键字
- 基因组SSRs
- 短序列重复
- 重复的核心
- 明显的杂合子缺陷
- 咖啡基因组