摘要
背景
葡萄(诉酿酒用葡萄)是用于葡萄酒生产的主要葡萄品种,全球葡萄酒产业每年价值数十亿美元。为了维持和提高葡萄产量,有必要了解葡萄品种的遗传组成。在这里,我们使用大规模并行签名测序(MPSS)进行mRNA分析,并将其与可用的表达序列标签(EST)数据相结合。这些基于标记的技术,不需要先天的基因组序列的知识,非常适合转录分析。MPSS的序列深度使我们能够捕捉和量化葡萄浆果发育过程中几乎所有特定阶段的转录本。
结果
利用大规模并行签名测序(MPSS)技术确定II期葡萄浆果转录本的数量和相对丰度。共获得2,635,293个17碱基和2,259,286个20碱基特征,分别代表至少30,737和26,878个不同序列。每个签名的平均归一化丰度为~49 TPM(转录本每百万)。MPSS签名与可用签名的比较葡萄属该物种的ESTs和单基因集表明,6430个不同的contigs和2190个单基因与至少一个MPSS签名完全匹配。在匹配的序列中,ESTs是从浆果以外的组织或不同发育阶段的浆果中鉴定出来的。与已知的葡萄ESTs不匹配的其他MPSS签名可以扩展我们对葡萄的知识诉酿酒用葡萄转录组,特别是当这些数据用于协助注释的全基因组序列葡萄.
结论
这里提供的MPSS数据不仅比之前基于EST的分析达到了更高的饱和度水平,而且在这样做的过程中,扩展了葡萄浆果在成熟开始之前的独特发育阶段的已知转录本集。MPSS数据集还揭示了反义表达的证据,以前没有在葡萄中报道过,但与其他植物物种中报道的相当。最后,我们开发了一个新的基于网络的公共资源来利用葡萄MPSS数据[1].
背景
葡萄品种(葡萄属属)是世界上种植最广泛和经济上最重要的水果作物[2].葡萄浆果的使用包括生产果汁,新鲜和干果,和蒸馏酒,虽然葡萄酒生产的栽培品种诉酿酒用葡萄拥有最高经济价值的葡萄产品。葡萄浆果是一种具有特征的双乙状形生长曲线的非更年期水果。浆果指数生长的初始阶段(阶段I)之后是滞后阶段(阶段II),在成熟或“变异”开始后恢复生长(阶段III)。浆果发育的特征是许多生物过程的变化,包括细胞分裂和增大,初级和次级代谢,以及对非生物或生物胁迫的抗性或敏感性[3.,4].这种植物物种对农业的重要性使基因组资源的开发成为高度优先事项。在这些资源中,重要葡萄组织的转录分析是一个实用的选择,可以揭示这个动态发育系统的转录复杂性和变化。
大规模并行签名测序技术[5,6是一种基于序列的测量基因表达的方法。MPSS提供的抽样深度可以识别给定样本中几乎完整的转录本目录。该方法基于一种独特的并行测序方法,首先在直径为5 μm的微珠上克隆cDNA文库;来自原始RNA样本的一个转录本被表示在一个珠子上[5].从每个珠粒中,通过连续的测序反应获得17个或更多核苷酸的“签名”序列[5- - - - - -7].这些标记来自并包括转录本中一个特定限制性内切酶位点的最多3'位点(最常见的是DpnII,生成以GATC开头的签名)[5,6].该方法的输出在概念上类似于一种可能更熟悉的方法,称为基因表达序列分析(SAGE) [8].然而,MPSS技术允许同时对给定文库中的数百万个签名进行测序[5].通过将这些标记与基因组匹配以识别特定基因,每个标记的丰度代表并测量样本组织中的基因表达水平。在该技术的几个已发表的应用中,我们之前对参考植物物种进行了全面的转录分析拟南芥还有大米[7,9].虽然MPSS、SAGE和表达序列标签(ESTs)都是用于转录分析的基于序列的技术,但由于其巨大的深度,MPSS提供了更彻底的基因表达的定性和定量描述。虽然新的测序技术,如合成测序(SBS)和454,提供了更深入的测序和更长的读取长度,但还没有一种技术在mRNA分析方面始终表现出比MPSS更好的结果[10].
在这篇报告中,我们用MPSS测量了葡萄果实发育过程中的基因表达,并与现有的表达谱进行了比较葡萄属Unigene集合[4],我们开发了一个新的基于网络的资源来利用葡萄MPSS数据。通过这一分析,我们能够注释数千个与预测基因匹配的签名,量化这些基因在发育中的浆果中的表达水平,比较从ESTs和MPSS签名频率中获得的表达谱,并扩大特定发育阶段重要葡萄器官中已知转录本的覆盖范围。由于这些数据是基于序列的,因此它们构成了一种资源,对于未来任何葡萄基因组序列的注释都是有用的。
结果
分析诉酿酒用葡萄berry MPSS数据集和签名注释
MPSS文库使用从野外生长的II阶段浆果(绿色,坚硬)中提取的RNA构建诉酿酒用葡萄简历。赤霞珠。cDNA文库克隆到珠上后,通过MPSS测序得到17个碱基和20个碱基的特征[5,6].我们注意到这些不是独立的样本,因为20个碱基的特征是通过将先前记录的17个碱基的特征扩展到3个核苷酸而获得的;由于每个额外的测序碱基的失败率较低,因此20碱基数据的原始序列计数较低。共产生2,635,293个17碱基和2,259,286个20碱基特征,分别对应30,737个和26,878个不同序列(表1)1得了)。这表示每~49个序列cDNA标签大约有一个独特序列的发现率或平均原始丰度值。
最初,为了将MPSS签名与预测的基因注释联系起来,所有可能产生MPSS签名的位点(“GATC”)都从可用的数据中识别出来葡萄属公共数据库中的Unigene数据集。这包括14,658个contigs(1,307个来自非酿酒用葡萄葡萄物种)和14,931个单胞胎(1,080个非单胞胎)酿酒用葡萄葡萄物种)。从葡萄序列的正义和反义方向提取以GATC锚定序列为起点的所有潜在特征。在该版本的contigs和singletons中,总共分别鉴定出84,834和48,490个不同的17碱基潜在签名葡萄属互补脱氧核糖核酸数据。当两个数据集结合在一起时,独特的基因组签名总数等于123,563个。总数量在网上-提取的独特的MPSS签名大约比完成报告的753,894个独特的“基因组”MPSS签名低6倍拟南芥序列(11],反映了葡萄EST数据集的不完整性以及基因间和内含子序列的缺乏。
观测到的MPSS特征根据“可靠性”和“显著性”滤波器的输出进行分类[11].这些滤波器的目的是将高质量数据(由超过指定频率阈值的签名表示)与由非常低丰度的MPSS签名生成的背景信号分开。与其他MPSS数据集一样,葡萄库由代表两个测序帧的四次测序运行生成[11].“两步”和“四步”测序帧各运行两次。可靠性过滤器询问一个签名是否出现在多个序列运行中(总共四次运行);在多次运行中观察到的特征被认为是“可靠的”。显著性过滤器仅将规范化丰度大于百万分之三(TPM)的签名标识为“显著性”。17碱基和20碱基表达签名在信度和显著性方面的分类见表1得了而且2;所有MPSS签名的96.8%对应于“可靠”和“重要”类别,与未通过过滤器的签名的极低丰度一致。该值与报告的97.5%相似拟南芥MPSS数据集[11].在与EST contigs序列完全匹配的MPSS签名中2 a - b)和单例(表2 c - d),唯一的“可靠”和“重要”签名是最大的类别(超过60%的唯一签名)。
表达的签名被映射到葡萄EST contig和单例,基于与在网上提取的“潜在签名”(见上文)。共有5,794和5,407个contigs分别被表达的可靠和显著的17碱基和20碱基MPSS签名匹配(见附加文件)1- - - - - -1 b)。平均而言,这一数字超过了已知数字的40%葡萄属sp.基因。另一方面,美国只有14%的单身人士葡萄属sp. EST集用MPSS特征进行匹配(表2摄氏度而且二维)。绝大多数的不匹配葡萄属sp序列有在网上在MPSS数据中未检测到的潜在特征。有可能对应的基因在这个样本中没有表达;或者,不匹配的contig和单例EST序列可能代表cDNA克隆的5' reads,因此不能代表大多数MPSS签名起源的3'区域。单例ESTs在未匹配集中不成比例的表现与后面的解释是一致的,因为Vitis数据集中的单例ESTs通常是5'测序反应的产物。
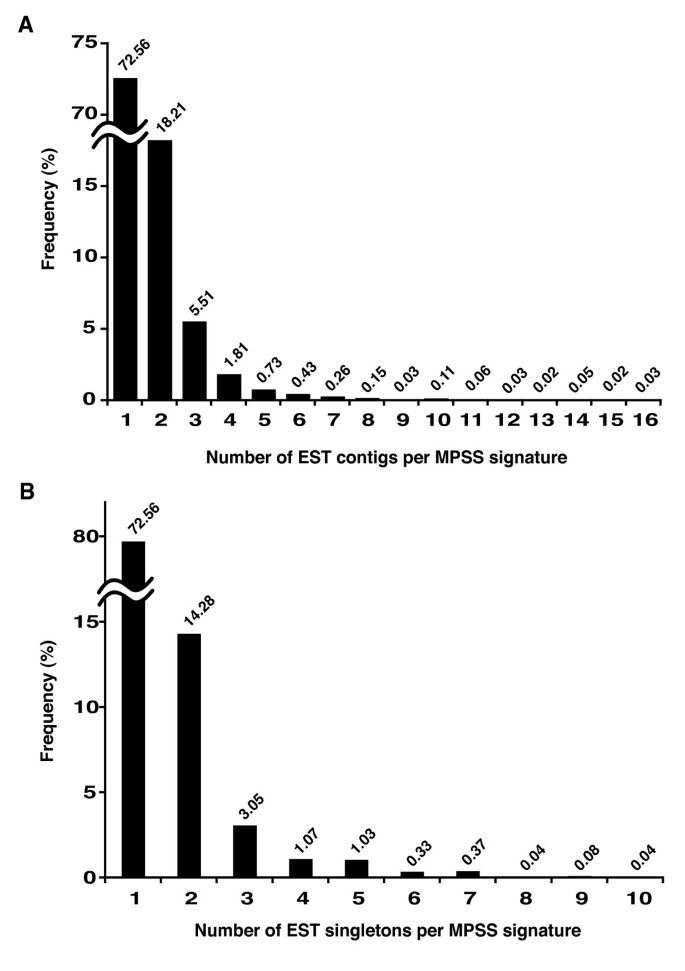
大多数签名匹配单个contig或singleton,而~40%匹配两个或更多[参见附加文件]1- - - - - -1 b].超过70%的匹配contigs和singleton显示了一个可靠且重要的MPSS签名的一对一分配(图1)[见附加文件2].其余序列对单个contig最多有16个不同签名的一对多赋值[参见附加文件]3.].17-20 bp的序列很少在不相关的基因中偶然复制[7][见附加文件4].相反,涉及基因复制或转录本处理的生物因素可能使签名与转录本的明确分配复杂化。因此,具有高序列相似性的基因家族成员很可能产生包含相同签名的不同转录本,而使用多个多聚腺苷酸位点或替代剪接位点的选择可以从同一个转录单位产生多个签名。为了估计选择性终止的频率,将5145个contigs以5'到3'的方向正确对齐。从这个子集中,鉴定出975个与至少两个MPSS签名匹配的contigs。17个核苷酸显著和可靠的MPSS特征的丰度计数被转换为相对频率值,每个特征的位置沿着3'到5'轴为975个contigs中的每个绘制2)。每个contig的签名频率从3'到5'方向呈指数下降。平均而言,约70%的签名来自3' most GATC位点,而只有~29%和~14%的签名分别来自第二和第三个3' most位点(进一步5')。因此,大多数MPSS匹配的转录本都是所有记录的3'位点最远端多聚腺苷酸化的产物。然而,与ESTs不匹配的MPSS签名(contigs或singleton)也有可能来自于更长的3'端,而其转录序列是不可用的。
义反义表达分析
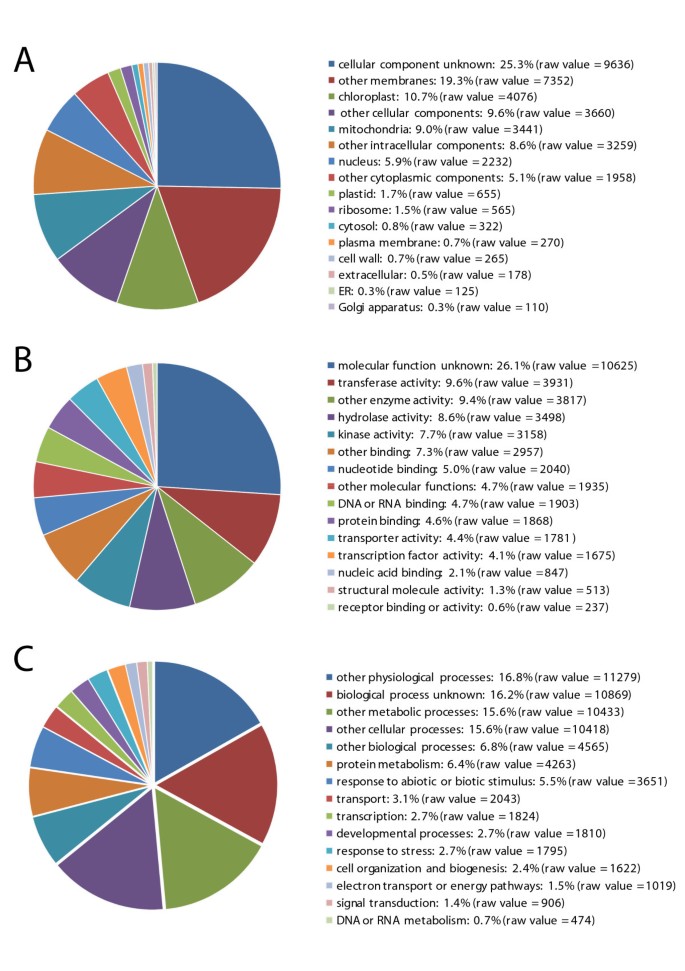
大约15%和11%的EST contigs和singleton分别在正义和反义方向上被MPSS签名匹配3 a - b)。一些序列在意义链上的MPSS签名频率要高得多,而其他序列在反义链上有更高的MPSS丰度[见附加文件]5].在已知的浆果转录组中,在两个方向上匹配的Contigs占12%(包括从EST测序和从cDNA文库中克隆的Contigs在内,总共7828个),其中与这些Contigs匹配的2891个MPSS签名占MPSS总丰度的52%。义-反义转录对可能是一个重要的转录特征,可以为转录后基因沉默提供一种机制[12]在浆果发育的动态阶段。这些contigs的功能分类显示没有特别的过度代表的类别(图3.)。此外,这些contigs在两个阅读帧方向上都没有显著的可识别的tBLASTx命中,这表明蛋白质编码只是一条链的特性。反义转录可能来自相邻基因的重叠3' utr,或者来自重叠的非编码RNA的转录。
由EST和MPSS丰度确定的表达谱
为了量化基因表达水平,我们使用了来自II期浆果文库的7,686个可靠且显著的17碱基MPSS签名的相对丰度。这些签名表示MPSS表达式数据中最健壮的子集。虽然剩余的1734个可靠但不重要的签名没有被考虑在这项分析中,但先前的分析表明,这些签名很可能代表了表达水平非常低的真实成绩单[11].这些签名所代表的转录本可能在未采样的不同特定细胞或组织层中以较高水平表达。
MPSS序列提供了给定器官或组织中转录本种群的目录,可以根据丰度进行排序。当与相关组织的EST数据相匹配时,这些数据尤其强大,因为它可以根据丰度和预测的基因功能进行排序。mpss匹配的5,791个葡萄EST contigs来自一系列cDNA文库,这些文库调查了植物发育的几个阶段,以及对生物和非生物胁迫的响应[4].其中,4753个contigs包含来自一个或多个葡萄浆果组织的ESTs,而1038个contigs包含来自其他葡萄组织而非浆果组织的ESTs(表2)4)。经MPSS标记匹配的EST contigs共有1242个,均来自于单一葡萄组织;其中,555个对应于浆果特有的EST contigs。其余的contigs均来自叶、花、叶柄、茎、芽甚至根。其余4,548个cDNA contigs和序列分别在两个或两个以上的葡萄器官中检测到4)。在7个葡萄cDNA文库中只发现了3个与mpss匹配的EST contigs。在对EST单子的类似分析中,绝大多数与以前只在浆果cDNA文库中观察到的转录本相对应,但只有207个是II期浆果(表2)4 b)。在先前与浆果文库无关的contigs和单倍体中,有来自花和叶cDNA文库的。MPSS签名为确认转录本的存在和相对转录水平提供了有价值的信息。这些转录本中的许多可能在以前仅基于EST数据被错误地识别为组织特异性,因为EST测序不够深入,无法在不同组织中检测到这些低丰度转录本。MPSS数据表明,给定组织中的基因库存是复杂的,在不同组织中可能有比以前描述的多得多的重叠,这只能通过非常深入的est测序来识别。
基于标记的转录分析技术(如ESTs、SAGE和MPSS)的一个优点是在分析之前不需要预先选择目标。虽然使用基于ests的方法的新转录本的发现率受到测序工作的程度和给定cDNA文库中的冗余的限制,但未匹配或低丰度的MPSS签名可以用作基于PCR的方法的引物,以扩展当前已知的基因集葡萄属[13].共有18,631个17碱基MPSS特征与已知的葡萄EST序列不匹配,其中5,900个特征显著且可靠;这些最有可能代表以前没有被确定为转录或转录变异的新基因。我们使用葡萄基因组的可用序列来验证这一假设,该序列由57,662个contigs组成,包含487,125,096个碱基对[14].总共有20,661个17-mer和17,867个20-mer不同的MPSS特征与基因组contig序列匹配。其中,有9,125个17-mer和7,771个不同的17-mer MPSS签名只与基因组contigs匹配,而不与ESTs匹配。以17人签名为基准,MPSS数据显示的转录本多样性比现有公共EST资源记录的多44%。
在网上由EST得到的表达式配置文件5)和MPSS特征频率(表6)显示了排名最高的基因的相对丰度的差异和共同点。例如,两个数据集的一个共同特征是几种几丁质酶、金属硫蛋白样蛋白和存储蛋白的丰度相对较高,以及一个假定的转录因子和一个延伸因子1-α。另一方面,两个六聚多泛素和一个质膜水通道蛋白是基于MPSS标记而不是基于EST计数排名靠前的基因,而对于非特异性脂质转移蛋白a,情况恰恰相反(存在于顶级ESTs中,而不是在MPSS标记中)7)。在这些单例ESTs中,有与细胞壁修饰相关的转录本(木葡聚糖特异性真菌内切葡聚糖酶抑制剂蛋白和一种伸展素样蛋白)、非生物/生物应激因子(过氧化氢酶和过氧化氢氧化酶)、真核翻译起始因子和几种注释较差的转录本。
从EST或MPSS特征计数中观察到contigs的相对丰度有显著差异。从两个浆果SII文库中获得的est序列中,195个contigs约占50%,只有10个contigs与过滤后的MPSS签名匹配。基于MPSS频率排名前20位的contigs占410,925个(占所有匹配EST contigs序列的56.7%),这表明MPSS数据的曲线更陡峭,可能多样性水平更低。相比之下,基于EST计数的20个最频繁的contig仅占这两个库EST总数的29.4%。
正如预期的那样,MPSS签名从诉酿酒用葡萄浆果阶段II也匹配几个非酿酒用葡萄类中的EST单例和contig葡萄属Unigene集。虽然转录组的非酿酒用葡萄物种的特征很少,基于MPSS签名频率的排名最高的转录本的比较(表8而且9)显示出不同物种之间的显著相似性。
一个访问葡萄MPSS数据的网站
为方便公众查阅及使用MPSS数据,我们开发了一个资料库及网页界面[15].数据库和界面是先前描述的网站的自定义版本[16].与拟南芥或水稻MPSS位点利用这些物种的完整基因组序列不同,我们的葡萄数据库侧重于EST contigs。这就需要开发专门的工具和方法。例如,est的不完整性质需要BLAST工具,允许用户识别与其感兴趣的基因最接近的葡萄序列。可以通过输入葡萄contig标识符或EST代码、MPSS签名序列、感兴趣的葡萄序列或一组contig标识符来访问MPSS数据。本网站提供的转录活性数据可作为葡萄个体基因或基因家族分析的起点。
讨论
我们通过比较和结合两种基于标记的方法:ESTs和MPSS,探索了葡萄浆果发育特定阶段的表达模式。这两种方法描述了相似的转录本丰度模式,尽管有一些明显的差异可能与方法本身有关。原则上,由于测序的深度,MPSS数据在表达和相对丰度方面应比ESTs数据提供更彻底和定量的绝对转录本种群表达[7,11].当从任何给定文库中测序的cDNA克隆数量较低或在样本组织中仅表达低水平的基因时,这尤其正确。为了使EST频率代表绝对转录本频率,测序工作必须很大,取样必须无偏倚。如果公共领域数据库的资源足够大,可以通过结合这些数据库中现有的图书馆信息来克服由特定组织构建的图书馆达到饱和的目标。然而,文库建设和EST测序所使用的不同方案,缺乏对生长条件、基因型的完全控制,甚至缺乏描述特定发育阶段的标准化指南,使得难以实现无偏抽样。另一方面,MPSS分析也存在偏差。例如,一些高度转录的基因(基于EST频率分析)与任何MPSS签名都不匹配,可能是由于序列中缺乏GATC位点或技术因素。缺乏合适的DpnII .部分地点拟南芥与其他高通量技术相比,转录本是MPSS转录谱阴性结果的一个来源[17].此外,MPSS大大低估了包含IIS型限制性内切酶识别位点的签名的表达BbvI(用于MPSS测序),或在测序帧中包含某些四核苷酸单词的签名[11].以前基于标记的方法的高成本限制了生物复制作为实验方法的一部分;这类数据对于确定这些技术所产生的生物变异和技术噪声的程度是非常必要的[7].随着成本的降低,下一代技术可能更容易实现这一目标。多种转录分析方法的联合应用可能提供最可靠的转录水平测定。
在葡萄MPSS数据集中,当多个特征匹配到一个contig时,这些特征通常在丰度上变化显著。然而,这些数据与从est转录本的主要形式中获得的最丰富的MPSS签名是一致的[1].基于MPSS在不同组织和治疗中的替代转录本聚腺苷酸化的评估可以通过识别差异终止转录本来深入了解这种基因调控机制。与多个contigs匹配的标记的注释和分析是一项更困难的任务,但这些数据的验证可以通过使用微阵列和专门设计的探针来确定所有匹配基因的相对表达,或者通过使用不同的“锚定酶”重复MPSS实验,例如国民III (CATG)代替Dpn二世(比如)。
全基因组复制的发生可能推动植物王国的基因组多样化和物种形成[18].据报道,陆地棉中全基因组多倍体化导致同源基因的基因和器官特异性沉默和不平等表达水平[19- - - - - -21]而类似的现象也可能是黄籽商品大豆品种的原因[22].与重复相关的基因表达变化可能在葡萄表型中发挥作用的程度在很大程度上是未知的。由于葡萄基因组的祖先多倍体特性[23- - - - - -25],可能发生了导致同源基因相互作用或沉默的复制事件。通过比较ESTs和MPSS转录谱数据,确定了大量反义表达的证据。哺乳动物系统中最初的整个转录组分析表明,高达20%的转录本形成义-反义(S/AS)对[26- - - - - -31].来自大规模小鼠cDNA测序项目的最新分析[32]和人类染色体的高分辨率转录图谱[33结果显示,在所有小鼠和人类转录本中,S/AS对分别存在高达72%和50%。在浆果转录组中观察到的S/AS频率与文献中报道的相似拟南芥,其中大约22%的已知基因具有组织特异性的自然反义转录对[7].考虑到基因组中不同基因和区域对S/AS对形成的贡献不相等[32],整个转录组分析肯定会比本研究中用有限的转录组覆盖所确定的结果更准确地描述葡萄中这种现象的程度。
反义表达有两个不同的来源:独联体- - -反式-编码反义[27- - - - - -29].前者对应于来自与感RNA相同遗传位点的相反链的转录本。独联体-编码的反义转录本往往与意义链完全重叠,形成长而完全匹配的RNA双链[28].人类中大约50%的义-反义对类别属于这一类别[29].反式-编码的反义转录本来源于替代位点,并倾向于与原始位点的意义链有部分重叠[27,28].葡萄中dsRNA或小rna的内源性群体的功能仍有待更详细的实验来阐明,这最好使用短读测序方法来实现[34].
基于标记的转录分析方法为发现新的表达序列提供了独特的优势。来自浆果发育特定阶段的MPSS签名揭示了葡萄中潜在的6345个新转录本。这些转录本可以更充分地确定,以扩大已知的和实验验证的集合葡萄属以pcr为基础的方法[13],或者最终将签名与葡萄基因组序列进行比对。在缺乏全基因组序列信息的情况下,基于pcr的方法对于由于低拷贝数或rna依赖cDNA合成的技术限制而难以通过基于est的方法识别的转录本可能变得尤为重要。全基因组测序诉酿酒用葡萄基因组,结合数据丰富的基于标记(ESTs和MPSS签名频率)和基于微阵列的转录数据,将极大地有助于我们理解基因组组织、转录活性和表型之间的复杂关系。由于自动化基因组注释系统既容易出错,又随着实验数据的结合而大大改进,因此EST和MPSS数据在未来几年将被证明对基因发现和基因组序列注释非常宝贵。
结论
我们进行了完整的转录分析诉酿酒用葡萄利用MPSS结合EST数据对果实过渡到成熟阶段进行分析。从MPSS数据中鉴定出大约30,000个不同的签名,每个签名代表一个不同的转录本,并将这些签名映射到EST序列上。与一个EST匹配的MPSS签名数量从1到16不等,这表明在该地区存在大量的替代转录本诉酿酒用葡萄.此外,还发现了大量与反义取向ESTs相匹配的MPSS特征。虽然反义转录本的存在在许多植物物种中都有报道,但这是第一次发现反义转录本存在于植物中诉酿酒用葡萄.除了与EST匹配的签名外,还识别出大量与EST不匹配的MPSS签名。虽然一小部分可能是由于测序错误,但我们认为大多数主要是由于当前EST数据集的序列覆盖深度较低;支持这种解释的事实是,签名匹配的比例诉酿酒用葡萄通过合并全基因组序列数据,序列几乎翻了一番。高容量、短读测序技术,特别是下一代千兆酶方法,有可能为人类基因组序列的持续注释做出重要贡献诉酿酒用葡萄.葡萄MPSS数据可从特拉华大学MPSS网站[1], EST数据集可通过加州大学戴维斯分校农业与环境科学学院基因组学设施(CGF)网站[35].
方法
植物材料和取样程序
用于MPSS测序的cDNA是从野外生长的II期浆果(绿硬)样本中构建的诉酿酒用葡萄简历。赤霞珠(Cabernet Sauvignon),克隆8葡萄,位于加州大学戴维斯分校的Tyree Teaching Vineyard。为了确保样本的代表性,浆果从多个集群和单个集群的不同位置取样。在此阶段的浆果子样本用于生成cDNA文库和表达序列标签(ESTs),如先前报道[4].有关样品处理和存储的更多细节,请参见Goes da Silva等人,2005年。
MPSS数据生成与分析
所有MPSS的执行基本上如上所述[5,6],该文库在Illumina, Inc.(原Solexa, Inc.;海沃德,CA)。原始和标准化的MPSS数据可在特拉华大学MPSS网站[1].我们将MPSS签名与诉酿酒用葡萄加州大学戴维斯分校环境科学研究中心网页[35]并为每个被识别出完全匹配的序列分配签名。一个签名与EST数据集的匹配数量被记录为每个签名的“命中数”。我们合并了测序运行,并计算了先前报道的单个标准化丰度[11].利用BLASTX批量搜索,并分析每个Contig的第一个EST和最后一个EST的主题指标,进行5'到3'方向的Contig定向。数据分析在MS Excel (Microsoft, Seattle, WA)和SAS V.8统计包(SAS Institute, Cary, NC)中进行,或在定制的MySQL数据库中进行[16]和SigmaPlot 8.0版本中的数字(Systat软件公司,圣何塞,加利福尼亚州)。
参考文献
- 1.
葡萄MPSS数据库。[http://mpss.udel.edu/grape/]
- 2.
Vivier MA, Pretorius IS:为葡萄酒行业量身定制的基因葡萄。生物技术,2002,20 (11):472-10.1016/S0167-7799(02)02058-9。
- 3.
欧拉特N, Diakou-Verdin P, Carde JP, Barrieu F, Gaudillére JP, Moing A:葡萄浆果发展综述。中国农业科学,2002,26(3):457 - 457。
- 4.
Goes da Silva F, Iandolino A, Al-Kayal F, Bohlmann MC, Cushman MA, Lim H, Ergul A, Figueroa R, Kabuloglu EK, Osborne C, Rowe J, Tattersall E, Leslie A, Xu J, Baek J, Cramer GR, Cushman JC, Cook DR:葡萄转录组的特征。多个葡萄品种表达序列标签的分析及浆果发育过程基因表达纲要的建立。植物营养学报,2005,39(2):344 - 344。10.1104 / pp.105.065748。
- 5.
生物技术学报,2000,18(6):630-634。10.1038/76469。
- 6.
美国国家科学研究院。2000, 97(4): 1665-1670。10.1073 / pnas.97.4.1665。
- 7.
Meyers BC, Vu TH, Tej SS, Ghazal H, Matvienko M, Agrawal V, Ning J, Haudenschild CD:拟南芥转录复杂性的大规模并行序列分析。生物技术学报,2004,22(8):1006-1011。10.1038 / nbt992。
- 8.
张玲,张志刚,张志刚,张志刚,张志刚:基因表达序列分析。科学通报,1995,27(4):344 - 344。10.1126 / science.270.5235.484。
- 9.
Nobuta K, Venu RC, Lu C, Belo A, Vemaraju K, Kulkarni K, Wang W, Pillay M, Green PJ, Wang G, Meyers BC:水稻mrna和小rna的表达图谱。中国生物工程学报,2007,25 (4):473-10.1038/nbt1291。
- 10.
埃霍尔姆格里斯M, M,奥特曼,我们提亚,巴德JS, Bemben洛杉矶,贝尔卡J,布雷弗曼女士,陈YJ陈Z,杜尔某人,Du L, Fierro JM,戈麦斯十五,公元前古德温,他W, Helgesen年代,何鸿燊CH, Irzyk GP, Jando SC, Alenquer多层互连,Jarvie TP, Jirage KB,金正日JB,小骑士,Lanza JR Leamon JH,莱夫科维茨SM,雷米,李J,洛曼KL,卢H, akhijani用VB,麦克达德克。麦凯纳MP,迈尔斯电子战,Nickerson E,高贵的小,植物R,举办的英国石油公司,罗南MT,罗斯GT, Sarkis GJ,西蒙斯摩根富林明,辛普森JW, Srinivasan M, Tartaro KR,托马斯,Vogt KA, Volkmer GA, Wang SH, Wang Y, Weiner MP, Yu P, Begley RF, Rothberg JM:微制造高密度皮升反应器的基因组测序。自然科学学报,2005,37 (6):337 -
- 11.
Meyers BC, Tej SS, Vu TH, Haudenschild CD, Agrawal V, Edberg SB, Ghazal H, Delcola S: MPSS在拟南芥全基因组转录分析中的应用。基因组学报,2004,14:1641-1653。10.1101 / gr.2275604。
- 12.
Borsani O,朱娟,Verslues PE, Sunkar R,朱景江:拟南芥耐盐性的内源性sirna调控。光子学报,2005,29(3):344 - 344。10.1016 / j.cell.2005.11.035。
- 13.
陈娟,孙敏,李松,周刚,罗利JD,王思敏:利用SAGE标记识别人类基因组中的新转录本和新基因。美国国家科学研究院。2002, 99(19): 12257-12262。10.1073 / pnas.192436499。
- 14.
Genoscope。[http://www.cns.fr/externe/English/Projets/Projet_ML/organisme_ML.html]
- 15.
Nakano M, Nobuta K, Vemaraju K, Tej SS, Skogen JW, Meyers BC:植物MPSS数据库:mRNA和小RNA分析的基于特征的转录资源。核酸决议2006,34(数据库问题):D731-5。10.1093 / nar / gkj077。
- 16.
Meyers BC, Lee DK, Vu TH, Tej SS, Edberg SB, Matvienko M, Tindell LD:拟南芥MPSS。定量表达分析的在线资源。中国农业科学,2004,26(2):344 - 344。10.1104 / pp.104.039495。
- 17.
张志刚,张志刚,张志刚,等。拟南芥基因表达量测定技术的研究进展。比较与功能基因组学。2004,5(3):245-252。10.1002 / cfg.397。
- 18.
Cui L, Wall PK, Leebens-Mack JH, Lindsay BG, Soltis DE, Doyle JJ, Soltis PS, Carlson JE, Arumuganathan K, Barakat A, Albert VA, Ma H, dePamphilis CW:开花植物历史上广泛的基因组复制。基因组学报,2006,16(6):738-749。10.1101 / gr.4825606。
- 19.
Adams KL, Cronn R, Percifield R, Wendel JF:多倍体复制的基因对转录组和器官特异性相互沉默的贡献不平等。美国国家科学研究院。2003, 100(8): 4649-4654。10.1073 / pnas.0630618100。
- 20.
杨晓明,王晓明,王晓明,等:棉花种间二倍体不同器官乙醇脱氢酶基因的双向沉默。中国生物医学工程学报,2005,31(4):339 - 339。10.1534 / genetics.105.047357。
- 21.
胡晓燕,王晓燕,王晓燕,王晓燕,等。多倍体复制基因表达水平的研究进展。遗传学报,2006,173(3):1823-1827。10.1534 / genetics.106.058271。
- 22.
张志刚,张志刚,张志刚,张志刚:查尔酮合成酶基因簇介导的组织特异性基因沉默。植物细胞学报,2004,16(4):819-835。10.1105 / tpc.021352。
- 23.
法国-意大利葡萄基因组鉴定公共联盟:葡萄基因组序列表明主要被子植物门的祖先六倍化。大自然。《自然》第449卷第7161期自然出版集团,2007:463-10。1038 / nature06148。
- 24.
Olmo HP:葡萄。作物植物的进化。编辑:Simmon NW伦敦,朗曼;1976:294 - 298。
- 25.
黄志刚,王晓明,王志刚,王志刚,等:被子植物基因组大小的研究进展。学报学报,2003,29(4):344 - 344。10.3732 / ajb.90.11.1596。
- 26.
Fahey ME, Moore TF, Higgins DG:人类基因组中的重叠反义转录。比较与功能基因组学。2002,3(3):244-253。10.1002 / cfg.173。
- 27.
陈娟,孙敏,Kent WJ,黄旭,谢辉,王伟,周刚,石荣智,Rowley JD:人类转录本中超过20%的转录本可能形成义反义对。中国生物医学工程学报,2004,32(16):4812-4820。10.1093 / nar / gkh818。
- 28.
Shendure J, Church G:人类和小鼠基因组中意义-反义转录的计算发现。中国生物医学工程学报,2002,3 (9):research0044-10.1186/gb-2002-3-9-research0044。
- 29.
王志强,王志强,王志强,等:哺乳动物细胞正义反义转录物的系统鉴定。中国生物工程学报,2004,22 (1):104-10.1038/nbt925。
- 30.
Yelin R, Dahary D, Sorek R, Levanon EY, Goldstein O, Shoshan A, Diber A, Biton S, Tamir Y, Khosravi R, Nemzer S, Pinner E, Walach S, Bernstein J, Savitsky K, Rotman G:反义转录在人类基因组中广泛存在。生物技术学报,2003,21(4):379-386。10.1038 / nbt808。
- 31.
Kiyosawa H, Yamanaka I, Osato N, Kondo S, Hayashizaki Y:具有FANTOM2克隆集的反义转录本及其基因调控的意义。基因组学报,2003,13 (6b): 1324-1334。10.1101 / gr.982903。
- 32.
京基因组勘探研究小组和基因组科学组和幽灵财团,片年代,Tomaru Y, Kasukawa T, Waki K,录像,中村M, Nishida H, Yap CC,铃木,卡瓦依J,铃木H, Carninci P,崎Y,井C,弗里斯M, Ravasi T,彭日成KC, Hallinan J, Mattick J,休谟哒,Lipovich L, Batalov年代,PG,美津浓Y, Faghihi MA Sandelin,粉笔,Mottagui-Tabar年代,梁Z, Lenhard B, Wahlestedt C:在哺乳动物的转录组反义转录。科学通报,2005,29(4):344 - 344。10.1126 / science.1112009。
- 33.
Cheng J, Kapranov P, Drenkow J, Dike S, Brubaker S, Patel S, Long J, Stern D, Tammana H, Helt G, Sementchenko V, Piccolboni A, Bekiranov S, Bailey DK, Ganesh M, Ghosh S, Bell I, Gerhard DS, Gingeras TR: 5-Nucleotide分辨率下10条人类染色体的转录图谱。科学通报,2005,38(4):344 - 344。10.1126 / science.1108625。
- 34.
Meyers BC, Souret FF, Lu C, Green PJ:植物中microRNA的发现。生物技术学报,2006,17 (2):139-
- 35.
加州大学戴维斯分校农业与环境科学学院基因组研究所。[http://cgf.ucdavis.edu]
确认
我们感谢Wu Huizhuan和Mayumi Nakano在葡萄MPSS web界面上的工作,以及Richi Gupta, Anna Leslie和Brian Chan的生物信息学帮助。这项工作得到了来自美国国家科学基金会植物基因组研究计划(奖励#0110528和#0321437给B.C.M.),美国农业部(SCA 58-5302-2-788给D.R.C)和加州食品和农业部(合同02-0150给D.R.C)的研究资助。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
人工智能进行研究并分析数据;KN进行了计算研究;FGdS分析数据;DRC和BCM设计了实验。所有的作者都参与了手稿的写作。
电子辅助材料
12870 _2007_261_moesm1_esm.doc
附加文件1:匹配grape EST contigs的过滤MPSS签名。表A: 17个mer签名。表B: 20个mer签名(DOC)
12870 _2007_261_moesm2_esm.xls
附加文件2:与contigs和singleton匹配的MPSS签名数。所有的唯一签名(包括17和20)被分为以下8类:可靠(R)、不可靠(nR)、显著(S)、不显著(nS)、可靠而显著(RS)、可靠而不显著(RnS)、不可靠而显著(nRS)和不可靠而不显著(nRnS)。在正义和反义两种方向上对每个类别的签名数和频率进行了识别。面板A:与EST contigs匹配的17个mer MPSS签名。面板B:与EST contigs匹配的20 mer MPSS签名。面板C:与EST单例匹配的17 mer MPSS签名。面板D:与EST单例匹配的20 mer MPSS签名。(XLS)
12870 _2007_261_moesm3_esm.pdf
附加文件3:Iandolino。葡萄ESTs与MPSS特征匹配的频率分布。该文件中的表格显示了与ESTs匹配的MPSS签名的频率。EST contigs(面板A和B)的频率范围为1到16,EST singleton(面板C和D)的频率范围为1到10。每个表中的数据基于我们用于排序MPSS签名的过滤器进行分类:RS,可靠和显著性;RnS,可靠但不显著;nRS,不可靠但显著;nRnS,不可靠,不显著。面板A:与EST contigs匹配的17个mer MPSS签名。面板B:与EST contigs匹配的20 mer MPSS签名。面板C:与EST单例匹配的17 mer MPSS签名。 Panel D: 20-mer MPSS signatures matched to EST singletons. (PDF 72 KB)
12870 _2007_261_moesm4_esm.pdf
附加文件4:由多个MPSS签名匹配的葡萄EST contigs示例。显示了一个具有3个MPSS签名匹配的EST contigs。该contig (CTG1027770)与“假定的转录因子btf3样mRNA”相似。面板A显示了在该contig中识别的所有MPSS签名,以及丰度水平及其在该contig上的坐标。面板B显示这个contig的序列,面板A的所有意义MPSS签名用蓝色表示。大写字母表示预测的ORF,小写字母表示预测的utr。最丰富的签名(#2)的位置与最多的3'一致。DpnII站点,MPSS测量的位置。其他的标记可能来自于MPSS文库构建过程中其他转录本、选择性聚腺苷酸化或不完全消化产生的标记。(pdf 51kb)
12870 _2007_261_moesm5_esm.pdf
附加文件5:在相关和不相关的contigs中出现相同的MPSS签名。给出了对EST连续段进行多次命中的MPSS签名的示例。在这个特定的示例中,MPSS签名“GATCAAGACTGATGAAA”(显示为红色)在三个EST contig中被标识,其中两个具有相同的注释,第三个不同。在每个编码序列的开头,列出了与拟南芥同源物最密切相关的同源物及其BLAST期望值。(PDF)
12870 _2007_261_moesm6_esm.xls
附加文件6:带有表达意义和反义MPSS签名的EST contigs。显示所有MPSS签名同时匹配正义方向和反义方向的EST contig。每个contig对拟南芥注释版本5 (TIGR5)进行BLAST,并在“blastdef”下列出这些contig的潜在功能以及基因ID(“ginumber”)和BLAST期望值(“evalue”)。这些contigs起源于两种不同的EST修正(Stage II berry GH和GS),这两种修正源于不同的植物葡萄属物种。GH和GS的EST ID编号以及物种名称分别列在“Berry SII-GH”、“Berry SII-GS”和“species”下。(XLS)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
Iandolino, A., Nobuta, K., da Silva, F.G.et al。葡萄的比较表达谱分析(葡萄)基于est和MPSS特征的频率分析。BMC植物生物学8,53(2008)。https://doi.org/10.1186/1471-2229-8-53
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-53
关键字
- 反义转录
- 葡萄浆果
- 大规模并行签名测序
- 大规模并行签名测序数据
- 大规模并行签名序列签名