摘要
背景
栽培花生或落花生(落花生hypogaeaL.)是世界上第四大油籽作物,主要生长在热带、亚热带和暖温带气候。由于它的起源是通过单一的和最近的多倍体事件,随后在育种努力中进行了连续的选择,栽培花生的遗传背景有限。在这些物种中,微卫星或简单序列重复(SSR)标记在育种应用中非常有用。栽培花生种质资源的多态性水平较低,需要大量的多态微卫星标记。
结果
利用TMV2基因型构建了微卫星富集文库。从3072个假定的ssr阳性克隆中测序了720个,提供了490个SSRs。其中完美型为71.2%,不完美型为13.1%,复合型为15.7%。其中GT/CA重复基序最常见(37.6%),其次是GA/CT重复基序(25.9%)。引物对共可设计170个ssr,并在两个基因型上进行了初步优化。104对(61.2%)引物在32个栽培花生基因型中存在多态性,46对(44.2%)引物在32个栽培花生基因型中存在多态性。多态性SSR标记检测到2 ~ 5个等位基因,平均每个位点2.44个。这些标记的多态性信息含量(PIC)值在0.12 ~ 0.75之间,平均为0.46。基于46个标记获得的112个等位基因,构建了32个基因型之间的表型图。大多数基因型代表亚种hypogaea被归为一类,而基因型属于亚种fastigiata主要分为两类。
结论
新开发的104个SSR标记扩展了栽培花生的SSR标记库。这些标记在栽培花生种质资源中具有较高的PIC值,对栽培花生种质资源分析、连锁定位、多样性研究和系统发育关系等具有重要意义落花生物种。
背景
种植的花生或落花生,落花生hypogaeaL., (2n = 4x= 40)是世界上大多数热带和亚热带地区的主要作物,全世界种植的落花生68%产于亚洲(2300万吨),24%产于非洲(800万吨),其余8%(350万吨)产于北美、加勒比、欧洲和大洋洲[1].种子可直接供人类食用,也可作为油和蛋白质的来源[2].此外,在世界许多地区,植物残余物作为牛饲料极为重要[3.].在树木作物成熟之前,这种作物作为树木种植园的收入来源正变得越来越重要。在非洲和亚洲,花生被间作于玉米、高粱和大豆之间,在少数地区,间作于成熟的椰子树之间。4].
与在栽培花生中观察到的丰富的表型多样性相反,迄今为止在栽培基因库中观察到的遗传多样性要低得多。栽培花生中这种低水平的遗传变异归因于它起源于在进化时间尺度上相对较近的单一多倍体事件[5].然而,迄今为止观察到的低水平分子多态性的其他促成因素可能是所使用的标记技术和所测试样本的多样性数量[6].
一般来说,分子标记,特别是微卫星或简单序列重复序列(SSRs)已被证明对许多物种的作物改良非常有用[7].然而,在落花生中,由于该物种的遗传变异水平较低,分子标记在育种中的应用受到了限制。然而,近年来,人们在花生SSR标记的开发上付出了巨大的努力[8- - - - - -10].SSR标记的开发传统上需要克隆和测序,因此与随机扩增多态性dna (rapd)、扩增片段长度多态性(AFLP)等PCR任意引物技术相比,成本更高,人力更密集[7].然而,一旦SSR标记被开发出来,它们在育种活动中的应用,特别是使用高通量方法的应用变得非常经济有效。为了从基因组DNA文库中分离SSR,已经开发了几种创建SSR富集基因组文库的方法。11- - - - - -13]或在基因组文库建设后[14].
通过不同的方法,已在花生中开发出> 500 ssr [15].利用这些SSR标记,在花生AA基因组和bb基因组的遗传图谱绘制和多样性研究方面取得了良好进展[8,9,16- - - - - -22].而在栽培种质中,这些SSR标记的多态性水平很低[8,19- - - - - -22].这也是为什么尽管花生的SSR标记数量适中,但迄今为止还没有一份基于栽培种质资源的遗传图谱发表。为了克服花生种质资源多态性水平低的问题,一种简单的解决方法是在花生种质资源中开发临界数量的SSR标记,从而获得约200-300个多态性SSR标记的资源库。
本研究采用微卫星富集基因组文库方法,旨在从花生中分离和鉴定新的微卫星标记。本研究旨在完善栽培落花生种质资源的多态性SSR标记库,为栽培落花生种质资源的遗传定位和性状定位提供依据。
结果与讨论
SSR-enriched图书馆
从TMV2基因型出发,采用Fischer和Bachmann改良方法构建SSR富集文库[23].该文库对CA和CT SSR重复序列进行了富集。从这个文库中,从32个96孔板中挑选了3072个克隆。用地高辛标记的SSR探针(CA和CT)杂交得到720个(23.4%)推测阳性克隆。测序结果显示,插入体大小为50 ~ 792 bp,平均长度为309 bp。大多数(43.9%)克隆含有中等大小(200 bp-400 bp)的插入体,34.6%的克隆含有小插入体(50 bp-200 bp), 21.5%的克隆含有> - 500 bp的插入体。
用串联重复查找器(Tandem Repeat Finder, TRF)分析上述序列数据,有490个(68%)克隆包含一个或多个SSRs。与其他花生SSR分离研究相比,所构建文库的富集效率较高。和目前的研究一样,在He和同事的研究中,61%的克隆被发现含有SSRs [20.],在Gimenes及其同事的研究中,56%的克隆具有SSRs [18Wang和同事报道43.7%的克隆含有SSRs [24].然而,在其他一些富集SSRs的文库中获得了非常低的富集效率(10%至31%)[8,9,21].事实上,这种富集效率取决于许多因素,包括用于文库构建的限制性内切酶的选择,用于富集的SSR探针等。本研究采用的方法似乎是花生SSR分离最有效的富集策略。
通过多序列比对分析,在ssr富集的基因组文库中观察到26%的冗余水平ClustalW计划;共观察到5份1个克隆,4份5个克隆,3份10个克隆,2份65个克隆。含有ssr的冗余克隆的比例(26%)与其他利用微卫星富集的其他植物基因组文库的研究相当,例如橄榄树(齐墩果欧洲公司L., 16.6%) [25],洋葱(洋葱L. 24.3%) [23].与花生SSR分离研究相比,在花生中观察到高达67%的冗余[8,20.,21],本研究中采用的策略似乎相当有效,可以分离出更高比例的新颖和独特的ssr。本研究中观察到的冗余水平可能是由于花生基因组中存在多个SSRs副本,这些SSRs可能同时存在于栽培的A和B基因组中答:hypogaea.观察到的某些SSRs在多个克隆中重复的偏倚也可以解释为在富集过程中(适配器结扎,单链富集DNA的扩增,镀前的细菌生长),可以任意选择一些片段而不是其他片段。
尽管在富集过程中添加了过量的适配器,7.2%的克隆被鉴定为连接产物,在最初的限制/连接步骤中,由于内部的存在Rsa我和反光镜锁定我限制网站。在富集克隆过程的PCR步骤中可形成另一种类型的连接[27].这种嵌合体通常仍未被检测到,并可能导致某些引物对在引物评估中无法扩增基因组DNA模板[28].
SSRs的发生和特征
对720个克隆进行序列分析,490个(68%)克隆存在一个或多个SSRs。根据韦伯的定义[29], 71.2%的ssr是完美的,13.1%的ssr是不完美的(当ssr被少数碱基对打断时),15.7%的ssr是复合的(当多个ssr被少数碱基对隔开时),其中9.8%的ssr是完美的,5.9%的ssr是复合的不完美的。花生的不同SSR分离研究中,不同SSR类的分布相似[8,9,18].
在重复基序方面,GT/CA重复基序最常见,占所有重复类型的37.6%,其次是GA/CT重复,占25.9%。以往对植物基因组中微卫星丰度的研究表明,AT是出现频率最高的二核苷酸重复基序,其次是AG/CT和GT/CA [30.- - - - - -34].AT重复序列是自我互补的,很难通过群体杂交进行筛选,因此文库中没有对AT进行富集。本研究中花生ssr分离物中GA/CT、GT/CA、AT和ATT重复基序的丰度与前人对花生ssr分离物的报道一致[8,9,20.,24].与在同一杂交中使用不同重复基序的混合相比,使用单独的GA和GT滤波器可以提高检测完美GA/CT和GT/CA重复基序或重复频率的能力。然而,一些研究表明,当使用不同SSR寡核苷酸的混合物富集文库时,化合物SSR的检索比例更高(高达75%)[18,35,36].
GT/CA和GA/CT双核苷酸重复基序的最大重复单位数分别为48和50个单位;整体重复图案数量从7到50不等。事实上,在一些研究中,为较长的重复基序开发的标记被发现更能用于栽培花生种质的多态性检测[9].除了包含SSRs的GT和GA重复序列外,一些包含重复基序的SSRs - (AAG)n(CAA)n(TAA)n(测试)n(GTT)n(TTC)n(有条件现金转移支付)n(AAAG)n(TTTC)n(TTCTC)n(CTTTT)n(CTCTTT)n和(GTGTTT)n也分离出2-11个重复数。有趣的是,大多数含有这些重复序列的克隆都有额外的GT/CA或GA/CT重复序列。Gimenes和他的同事[18]还观察到37%具有不同重复序列的SSRs,就像本研究中一样,这些SSRs与所使用的寡核苷酸探针不完全互补。然而,重复基序ATT在大豆等几种豆科植物中含量丰富,信息丰富。37],鹰嘴豆[38]和pea [39]在本研究中并没有大量观察到。就花生而言,关于SSRs的分离和分布有许多报告,但只有两项研究[8,9]表明AAT重复基序的丰度。在本研究中,从3072个克隆中随机选取了720个克隆进行测序,可能对更多的克隆进行测序可以显示AAT重复基序的丰度。
标记的发展
利用Primer3程序对含有SSR的序列进行引物设计。遵循标准标准:底漆长度- 18-27 bp;Tm - 57 - 63°C;GC含量- 40-60%,正向引物和反向引物最大Tm差- 1.5°C,为170个含SSR克隆设计引物对[见附加文件]1].其中47.1%的引物对设计为完美重复,18.2%设计为不完美重复,34.7%设计为复合重复。对于其余的序列,引物设计是不可能的,因为在某些情况下,序列质量较差,而在某些情况下,ssr太靠近插入的开始或结束。设计引物的百分比,相对于测序的克隆数量(23.6%)高于Moretzsohn及其同事对花生的一些研究[9](10.5%)、何及同事[20.](14.0%)及Ferguson及其同事[8](21.3%),但低于Moretzsohn和同事等其他一些报告[21](41.4%)和王志强及其同事[24)(43.7%)。这可能与插入物的大小范围、用于基因组DNA文库构建的限制性内切酶以及用于SSR富集的方法等有关。[7].
利用新设计的SSR标记对TMV2和ICG 99001两个基因型进行扩增。在这组引物中,只有104对(61.2%)引物在检测的基因型中产生了可评分的扩增子(表2)1).引物对的功能相对低于Ferguson等人的研究[8]和莫瑞兹松及其同事[9他们分别在84.9%和81.6%的病例中观察到扩增。值得注意的是,上述研究均采用了多种PCR谱图和PCR优化策略,而本研究为节省成本和时间,未重复优化非扩增标记物的PCR条件。在104种工作引物中,有89对(85.6%)引物对在65°C -60°C触地型上进行了优化,14对(13.5%)引物对在60°C -55°C触地型上进行了优化,只有1对(0.9%)引物对在55°C -45°C特定轮廓上进行了优化。通过使用不同的PCR条件和图谱,很有可能提高新开发的标记的功能率。
同样值得注意的是,小于20个重复单位的标记在约73%的情况下产生扩增子,而包含较高重复单位数量的标记(> 20)在16.7% - 54.5%的情况下仅产生扩增子。这是可能的,较高的重复单位数量使Taq聚合酶不稳定,使其无法与模板DNA一起延伸[7].
SSR多态性
为了评价新开发的SSR标记在4×花生基因型多态性检测中的潜力,对104对引物进行了32个基因型的PCR检测2).结果表明,只有46个(44.2%)标记在种质资源中表现出多态性(表2)3.).46条引物中,30条引物用于完美ssr, 16条引物用于不完美ssr。本研究所观察到的标记多态性高于或相当于早期花生栽培种质SSR多样性的研究。例如,在两项不同的研究中,He和同事观察到29.2% [40]和33.9%的标记[20.]而Moretzsohn等研究中35.8%的标记存在多态性[9].在所有这些研究中,与本研究一样,SSR标记都是从基因组DNA文库中分离出来的。可能是本研究开发的SSR标记信息更丰富,也可能是所调查的种质资源更加多样化。SSR标记的多态性程度随着重复总长度的增加而增加,这一普遍理论支持了新开发的标记较高的信息性[7,9,29].这里开发的大多数标记(> 80%)包含对应SSRs的超过10个重复单元1).然而,Ferguson和他的同事[8]与本研究相比,他们观察到更高的标记多态性(48.7%),这可以归因于他们研究中检测的种质资源的多样性。
近年来,由于越来越重视功能分子标记的开发,SSR标记从表达序列标记(ESTs)发展而来[10].Luo和同事们[22]有20%的标记显示出多态性;Moretzsohn和他的同事[9]在栽培种质中检测到7.5%的多态性标记。基于est的SSR标记的多态性水平较低,可归因于它们起源于基因组的高度保守比例[41].我们认为,花生等遗传背景较窄的作物需要更多来自基因组DNA文库的SSR标记,而不是来自cDNA文库或ESTs。
46个多态标记所检测到的等位基因数在2 ~ 5之间,平均每个位点2.44个等位基因3.).这些多态性标记的PIC值变化范围为0.12 (IPAHM 92) - 0.75 (IPAHM 123),平均为0.46(表2)3.).在SSR类别和基序方面,三核苷酸SSR表现出较高的等位基因数量(平均每个位点2.5个)和PIC值(平均每个标记0.53个),其次是二核苷酸(平均每个位点2.45个等位基因;PIC值-平均0.45个标记)和复合SSRs(平均等位基因-每个位点2.35个;PIC值-每个标记平均0.44)。在二核苷酸ssr中,GA/CT重复序列比GT/CA重复序列(平均等位基因-每个位点2.6个,PIC值-每个标记平均0.50个)显示出更多的信息性(平均等位基因-每个位点2.0个,PIC值-每个标记平均0.33个)。Ferguson和同事[8]以及Moretzsohn及其同事[9]观察到GA/CT重复基序具有更高的信息量。因此,为了在栽培花生中开发更多的多态性标记,我们建议分离和开发基于GA/CT重复序列的SSR标记。
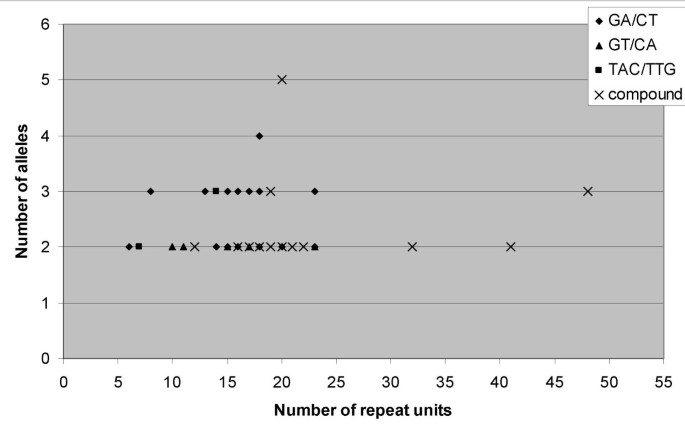
为了了解SSR标记的多态性与相应SSR的重复单位长度之间可能存在的关系,我们将重复单位长度与检测到的等位基因数量做了两个散点图(图2)。1)和计算出的PIC值(图;2).等位基因数与重复单位长度之间的散点图显示,在13 ~ 20个重复之间等位基因数变化最大,在低重复数或极高重复数中等位基因数较少。然而,它并没有像Ferguson及其同事指出的那样,在等位基因的数量和重复单位长度之间提供任何结论性的关系[8]和莫瑞兹松及其同事[9重复序列较长的基因座更有可能是可变的。事实上,在多态SSR标记中,含有最多重复单位的IPAHM 147标记(41)只提供了2个等位基因,而含有20个重复单位的IPAHM 103标记则显示了最多的等位基因(5)。这可能是因为在本研究中大多数多态SSR标记检测到2个和3个等位基因。
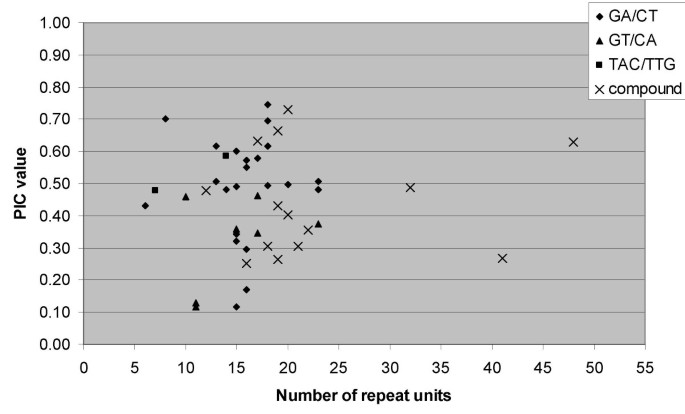
PIC值与重复单位长度的散点图显示,较高的PIC值(> 0.50)出现在13 ~ 20个重复之间,而较低的PIC值出现在低重复数(< 13)或极高重复数(> 20)之间。值得注意的是,这种关系似乎是SSR类特有的,因为它对化合物SSR更一致。据我们所知,目前还没有关于花生中PIC值与重复单位长度之间关系的报道,因此无法对观测结果进行直接比较。
通过对重复单位长度与等位基因数和PIC值的关系的研究,发现重复单位数与SSR多态性之间的关系不一致。早前已有报道,多态程度随重复长度的增加而增加[9,29,38,39],包括花生在内的其他一些研究表明SSR多态性与重复单位长度没有关系或弱相关[8,20.,42,43].
多样性分析和遗传关系
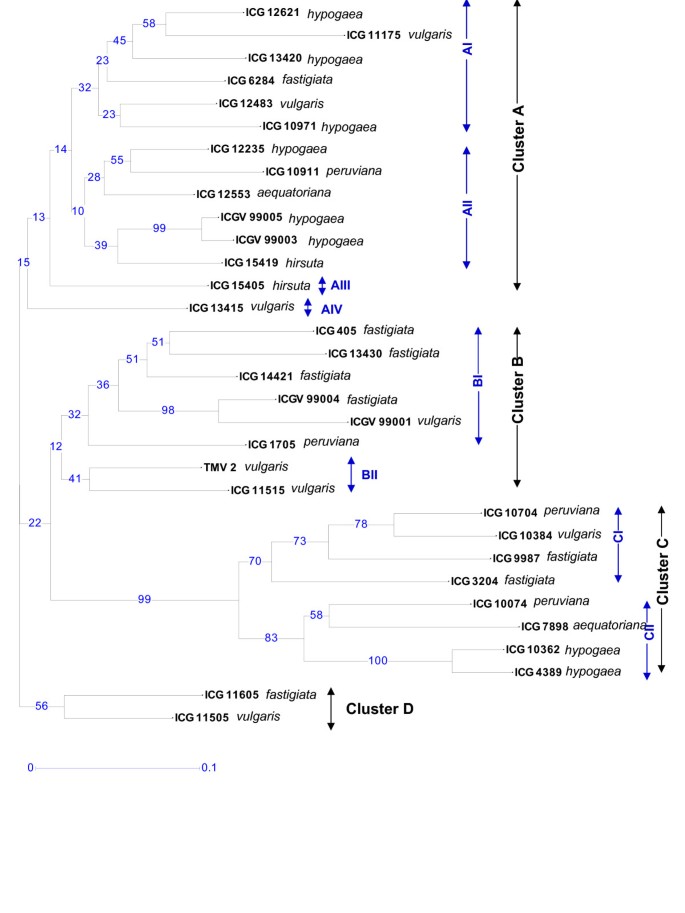
利用多态性标记获得的栽培落花生各种质资源的独特DNA指纹图谱,构建了一个表型图,以了解所调查的栽培落花生种质资源之间的关系。利用DARwin程序构建的基于DICE相似系数的树状图将种质资源划分为A、B、C和D 4个主要聚类(图2)。3.).聚类A包含14个基因型,聚类B包含8个基因型,聚类C和聚类D分别包含8个和2个基因型。在这些主要聚类下,基因型进一步分组为亚聚类。例如,集群A包含四个子集群(AI, AII, AIII和AIV),集群B (BI, BII)和集群C (CI和CII)各包含两个子集群。
大多数基因型(10个中的8个)代表亚种hypogaea(6hypogaea和2物种基因型)在主簇a中被发现fastigiata不同类群中两个亚种的基因型划分与花生植物品种的分类一致[44].然而,在早期的研究中[45代表两个亚种的基因型仅被分为两组。
聚类B包含4个(10个)属于变种的基因型fastigiata三种基因型属于变种寻常的.聚类C也包含来自于fastigiata包括2个基因型变种的亚种fastigiata2、品种peruviana各有一个品种寻常的而且aequatoriana.D簇仅包含2个属于亚种的基因型fastigiata.主集群下的大多数节点都由高引导值支持。
重要的是要注意植物品种的定位aequatoriana而且peruviana亚种fastigiata或hypogaea在文献中一直存在争议。例如,在过去,根据形态和生理特征,两个植物品种被划分为亚种fastigiata(包括其他品种fastigiata而且寻常的),而AFLP标记提示与aequatoriana而且peruviana来hypogaea而不是亚种fastigiata[45].目前的研究只包括两项aequatoriana和三个peruviana基因型,他们被归为所有三个主要的聚类(A, B和C)。然而,正如He和Prakash [45],五种基因型中有四种代表aequatoriana而且peruviana亚种表现出更密切的关系hypogaea基因型(AII和CII聚类),只有一个基因型peruviana亚种(ICG 1705)与亚种有一定的相似性fastigiata.植物品种peruviana根据RAPD和ISSR标记资料,除4个品种外,还将其划分为一个操作分类单元。fastigiata,寻常的(两者都属于亚种fastigiata),跑步者而且群(两者都属于亚种hypogaea) [46].
值得注意的是,两个抗叶锈病品系(ICGV 99003和ICGV 99005)被归为一类(AII),另外两个抗晚叶斑病品系(ICGV 99001和ICGV 99004)同样被归为一类(BI)。抗叶锈病基因型与抗晚叶斑病基因型具有同源性或同源性。其中,ICG 405、ICG 1705和ICG 6284 3个抗早叶斑病品种分布在树状图中。在聚类b下出现了易感叶锈病、晚叶斑病和早叶斑病3种病害的亲本TMV2。树状图显示了具有较高遗传多样性的潜在亲本基因型,可用于构建叶锈病、晚叶斑病和早叶斑病的定位群体。甚至需要在育种计划中选择和利用不同品种,以增强育种群体的多样性,以在未来获得选择收益[47].
结论
本研究结果为从栽培花生中获取微卫星标记提供了一种可靠、高效的方法。在栽培花生中分离和鉴定更多的DNA标记,以便进行更有效的基因组研究,如遗传作图、标记辅助选择和基因发现。SSR富集文库的构建和测序共获得400个SSR,但可设计引物对的只有170个,其中104个引物对具有功能。本研究为栽培花生提供了一套104个SSR标记。为了评价新开发的标记在种质资源分析中的潜力,对这些标记在32个基因型上进行了筛选,结果显示多态性水平合理。新开发的标记平均每个位点检测到2.44个等位基因,平均PIC值为0.46。本研究还揭示了SSR标记多态性的性质、重复类的类型或长度对相关SSR标记多态性的影响。最后,本研究建立的SSR标记对种质资源分析、群体遗传结构和系统发育关系具有重要意义。
方法
植物材料
为了构建富含ssr的基因组文库,使用了属于西班牙植物品种的花生种质系TMV2。利用TMV2和ICG 99001两个基因型对新开发的SSR标记进行PCR分析优化,利用32个基因型对栽培花生种质资源的潜在多态性标记进行鉴定(表2)2).在这32个基因型中,有10个基因型代表亚种hypogaea (2 .多样化物种8 .多样化hypogaea)其余22个基因型属于亚种fastigiata(10到多样化fastigiata7 .多样化寻常的3 .多样化peruviana2 .多样化aequatoriana).
DNA提取
采用改良的ctab方法从展开的叶片中分离总基因组DNA [48].按照制造商(岛津株式会社,日本)的建议,使用紫外- 160A分光光度计在1%琼脂糖凝胶和DNA浓度上检查DNA质量。
ssr富集库的构建
Fischer和Bachmann的修正方案[23]构建SSR富集文库。6微克的基因组DNA被钝端生成限制性内切酶消化Rsa一、琼脂糖凝胶电泳确认消化后反光镜锁定I适配器,由21-mer (5'-CTCTTGCTTACGCGTGGACTA-3')和磷酸化的25-mer (5'-pTAGTCCA)CGCGTAAGCAAGAGCACA-3')使用50 ng适配器/μg基因组DNA连接到限制性内切片段的钝端。在37°C下结扎2小时,以允许限制性内切酶消化继续进行,从而防止DNA片段相互重新结扎。然后用1% TAE琼脂糖凝胶分离结扎产物,从凝胶中切割100-900 bp和900-1500 bp的片段,并用GFX gel Band Purification Kit (Amersham Biosciences, USA)进行纯化。然后将这些结构物热变性并杂交成生物素化的微卫星寡核苷酸。用75 μl 6×SSC和150 nM生物素化低聚物(GT)进行杂交。15和(GA)15在T点过夜hyb= T米5°C。杂交种随后与链霉亲和素涂层磁珠结合(Dynabeads M -280链霉亲和素- Dynal,挪威)。为了捕获目标序列,对100-900 bp片段在室温下孵育15分钟,对900-1500 bp片段在43℃下孵育1小时。随后通过一系列洗涤去除非杂交基因组DNA;2× SSC 2次;0.1% SDS(每个5分钟,25℃),在1×SSC中两次(每个5分钟,25℃),最后在1×SSC中两次,在Thyb分别为2分钟和5分钟。结合的DNA以单链片段的形式在TE预热至95°C中洗脱。
将杂交后的DNA片段作为模板,以21基团寡核苷酸为引物(56℃退火温度,30个循环)进行PCR。聚合酶链反应后,用GFX柱纯化试剂盒(Amersham Biosciences, USA)对相似样品进行组合和纯化。纯化的PCR产物用反光镜锁定I通过在37°C孵育过夜获得矢量兼容的粘性末端片段。在连接到修饰的pUC19载体(pJV1)之前,使用MicroSpin™色谱柱(Amersham Biosciences, USA)对限制性内切片段进行纯化(Edwards等人,1996年[11])的线性化BssHII和去磷酸化。将结扎后的载体片段转化为活性载体大肠杆菌DH5α细胞(Invitrogen, USA),镀于含氨苄西林(100 μg/ml)的LB琼脂上。为了允许蓝白选择,在培养皿上涂上5-溴-4-氯-3-吲哚基-β- d -半乳糖吡喃甙(X-gal;80 μg/ml)加β- d -硫半乳糖yranoside (IPTG;80μg / ml)。白色菌落在含有氨苄西林的LB上,在用尼龙膜进行菌落提升之前,按照制造商(Amersham Biosciences, USA)的建议,取下并镀在网格中。杂交在42°C下进行过夜,使用地高辛标记的探针,其中包含所搜索的SSR基元(Roche,德国)。
ssr阳性克隆的测序
杂交鉴定的SSR阳性克隆在含100 μg/ ml氨苄青霉素的3 ml LB肉汤中培养过夜。质粒DNA使用GFX™Micro Plasmid Prep Kit (Amersham Biosciences, USA)提取。随后,在ABI 3700测序仪上使用M13 Forward 24-mer测序引物,采用双脱氧核苷酸链终止法对质粒DNA进行测序。基地呼叫使用Phred [49].序列数据使用50 bp的滑动窗口进行质量修剪,最低平均Phred评分为20。
SSR鉴定与引物设计
测序数据分析使用ClustalW程序以确定库中的冗余率。使用Tandem Repeat Finder软件分析非冗余序列[50].随后利用Primer3程序将含有SSR的序列用于引物设计。引物从重复基序的侧翼区域内设计;二核苷酸重复基序长度大于14 bp,三核苷酸长度大于15 bp,四核苷酸长度大于16 bp。
微卫星位点的扩增和可视化
所有引物均在5 μl反应体积内进行PCR反应,分别在65 ~ 55℃(89条标记)、60 ~ 55℃(14条标记)和55 ~ 45℃(1条标记)3个触点条件下进行。对5 ng基因组DNA进行PCR,引物数量不同,Mg2 +, dNTPs和TaqDNA聚合酶。关于产生PCR扩增子的每个引物对(标记)的这些反应成分的详细信息在附加文件中给出1.触地扩增程序包括94°C 2分钟,94°C 45秒30个循环,退火温度(65-55°C/60 - 55°/ 55-45°C) 60秒,72°C 60秒,72°C最终延伸10分钟,72°C。
PCR产物在未变性的6%聚丙烯酰胺凝胶上,在250 V下,在1× TBE缓冲液中分离2.5 ~ 3小时,用银染色观察,银染色由Kolodny [51].在检查的基因型中扩增子的存在或不存在分别被评分为1或0。
统计分析
每个微卫星位点的多态性信息含量(PIC)由Weir [52]:
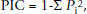
在哪里P我是频率的我基因型中的Th等位基因。
利用DARwin v 5.0.153程序,以0-1方式获得微卫星位点上所有等位基因的等位基因数据,计算简单匹配系数后的个体间遗传不相似性[53].利用生成的不相似矩阵,利用DARwin程序生成邻域连接(NJ)后UPGMA (Unweighted Pair Group Method with Arithmetic mean)树状图。
参考文献
Dwivedi SL, Crouch JH:亚洲开发银行支持的高粱、花生和鹰嘴豆分子育种项目研讨会论丛。ICRISAT。2003年,28-43。
Weiss EA:油籽作物。英国伦敦:朗曼;1983:660 -
Savage GP, Keenan JI:花生仁的成分及营养价值。花生作物:改良的科学依据。编辑:Smart J.伦敦,英国:查普曼和霍尔;1994:173 - 213。
林志强,刘志强,刘志强。植物形态形成、细胞遗传学及应用落花生物种。王志强。1987,41:1-39。
杨德明,李志强,李志强,李志强:豆科植物基因组图谱研究(Fam.)蝶形花科).植物基因组图谱。编辑:Paterson AH。美国奥斯汀:兰德斯公司;1996:211 - 227。
王晓明,王晓明,王晓明,王晓明:花生遗传多样性的分子多态性分析,落花生hypogaea中国农业科学,1998,30(4):349 - 349。
Gupta PK, Varshney RK:以面包小麦为重点的微卫星标记在遗传分析和植物育种中的开发和使用。《植物学报》,2000,29(3):344 - 344。
Ferguson ME, Burow MD, Schultz SR, Bramel PJ, Paterson AH, Kresovich S, Mitchell S:花生中的微卫星识别和特征(答:hypogaeal .)。应用理论与实践,2004,30(4):344 - 344。
Moretzsohn MC, Leoi L, Proite K, Guimaraes PM, Leal-Bertioli SCM, Gimanes MA, Martin WS, Valls JFM, Grattapaglia D, Bertioli D:一种基于微卫星的AA基因组富基因连锁图落花生(蝶形花科)。应用物理学报,2005,39(2):366 - 366。
梅斯ES,瓦尔什尼RK,马哈拉克什米V, Seetha K, Gafoor A, Leeladevi Y, Crouch JH:在网上豆科aeschynomenoid/dalbergoid和genistoid演化支中简单序列重复标记的开发及其可转移性落花生hypogaea、花生。植物科学学报,2007,44(3):344 - 344。
Edwards KJ, Barker JHA, Daly A, Jones C, Karp A:植物微卫星文库中多种微卫星序列的富集。生物学技术。1996, 20: 759-760。
Karagyozov L, Kalcheva ID, Chapman VM:简单序列重复序列高富集随机小插入基因组文库的构建。核化学学报,1993,21:3911-3912。
Kijas JMH, Fowler JCS, Garbett CA, Thomas MR:利用与链霉亲和素包被磁性颗粒结合的生物素化寡核苷酸序列富集柑橘基因组中的微卫星。生物学技术。1994, 16: 656-662。
伊藤涛,王志刚,王志刚:基于三倍亲和捕获的DNA序列特异性纯化。美国国家科学研究院。1992, 29(4): 489 -498。
Varshney RK, Hoisington DA, Upadhyaya HD, Gaur PM, Nigam SN, Saxena K, Vadez V, Sethy NK, Bhatia S, Aruna R, Gowda MVC, Singh NK:半干旱热带地区谷物豆类作物的分子遗传学和育种。基因组学在作物改良中的应用。编著:Varshney RK, Tuberosa R. Dordrecht,荷兰:施普林格. 2007,II: 207-242。
王涛,王涛,王涛,王涛,王涛,王涛,王涛:多态简单序列重复序列(SSRs)的发现与表征。作物科学,1999,39:1243-1247。
Palmieri DA, Hoshino AA, Bravo JP, Lopes CR, Gimenes MA:牧草品种微卫星位点的分离与表征落花生pintoi(属落花生).分子生物学杂志,2002,2:551-553。
Gimenes MA, Hosino AA, Barbosa AVG, Palmieri DA, Lopes CR:栽培花生微卫星标记的表征和转移性(落花生hypogaea).中国生物医学工程学报,2007,7:9-
何国强,陈晓明,陈晓明,等:花生多态DNA标记的鉴定(落花生hypogaea。l)。《自然科学》,1997,30(3):344 - 344。
何g,孟R, Newman M,高GM, Pittman RN, Prakash CS:花生微硒石的DNA标记(落花生hypogaeal .)。植物学报,2003,30 (3):329 -
Moretzsohn MC, Hopkins MS, Mitchell SE, Kresovich S, Valls JFM, Ferreira ME:花生的遗传多样性(落花生hypogaeaL.)及其野生近缘种基于基因组的高变区分析。植物学报,2004,4:11-
罗M,见鬼P,郭,他G,霍尔布鲁克CC, Bausher MG,李采访:一代表达序列标签(est)基因发现和标记发展种植花生。作物科学,2005,45:346-353。
Fischer D, Bachmann K:微卫星富集生物大基因组(洋葱l .)。生物技术,1998,24(5):796- 800,802。
王婷婷,杨晓东,陈晓东,余少林,刘广志,唐云云,徐建忠:花生中简单序列重复序列的分离。选择Jour生物技术,2007,10:473-480。
王晓燕,王晓燕,王晓燕,等。橄榄树简单序列重复序列(SSRs)的研究进展(齐墩果欧洲公司l .)。应用物理学报,2000,30(3):344 - 344。
Fisher PJ, Richardson TE, Gardner RC:单副本和多副本微卫星的特征松果体放射虫纲.应用理论,1998,26(3):369 - 379。
kobilizkova A, Dolezel J, Macas J:用3'修饰的寡核苷酸减法消除微卫星富集DNA文库中的扩增伪影。生物学技术。1998, 25: 32-38。
王晓明,王晓明,王晓明,王晓明,王晓明,王晓明,王晓明,王晓明,王晓明,王晓明,王晓明,王晓明天竺葵属植物.应用物理学报,2000,30(3):344 - 344。
韦伯JL:人的信息量(cC-dA)n(dG-dT)n多态性。基因组学杂志,1990,7:524-530。
热带树木基因组中双碱基重复序列的丰度和序列分析。基因组学报,1991,34(1):66-71。
张晓东,张晓东,张晓东:植物和脊椎动物微卫星基序的多态丰度存在差异。中国生物医学工程学报,1993,21(4):457 - 457。
Morgante M, Olivieri AM: pcr扩增微卫星作为植物遗传学标记。植物学报,1998,3(1):1 - 5。
王震,Weber JL,钟刚,Tanksley SD:植物短串联重复序列研究综述。应用理论,1994,18(1):1-6。
Powell W, Machray GC, Provan J:简单序列重复序列揭示多态性。植物科学进展,1996,1:215-222。
李文杰,李文杰,李文杰,李文杰,李文杰,李文杰等:生菜微卫星检索(摘要以l .)。基因组学报,1998,42:139-150。
张志刚,张志刚,张志刚,张志刚,张志刚。中国植物微卫星标记的开发、遗传及跨种扩增金合欢高阿.应用物理学报,2000,30(3):344 - 344。
Akkaya MS, Bhagwat AA, Cregan PB:大豆简单序列重复DNA的长度多态性。遗传学杂志,1992,132:1131-1139。
Hüttel B, Winter P, Weising K, Choumane W, Weigand F, Kahl G:鹰嘴豆微卫星标记的序列标记(中投arietinuml .)。中国生物工程学报,1999,42:210-217。
Burstin J, Deniot G, Potier J, Weinachter C, Aubert G, Baranger A:微卫星多态性Pisum一.植物学报,2001,30(4):344 - 344。
何g,孟R,高华,郭斌,Newman M, Pittman RN, Prakash CS:花生植物品种的简单序列重复标记(落花生hypogaea。l)。《自然科学》,2005,29(3):344 - 344。
王志刚,王志刚,王志刚。植物基因微卫星标记的研究进展。生物技术,2005,23(1):48-55。
Love J, Knight A, Mc Aleer M, Todd J:利用PCR分析的微卫星构建高分辨率小鼠基因组图谱。中国生物医学工程学报,1990,18:4123-4130。
俞凯,朴秀娟,李志强,李志强等:大豆微卫星DNA序列的丰度与变异(Phaseolus和Vigna).基因组学报,1999,42:27-34。
Krapovickas A, Gregory WC:分类分类落花生(豆科)。《科学》,1994,8:1-186。
何国强,Prakash C:栽培花生植物品种间亲缘关系评价(落花生hypogaeal)使用AFLP标记。中国农业科学,2001,30(4):347-353。
Raina SN, Rani V, Kojima T, Ogihara Y, Singh KP, Devarumath RM: RAPD和ISSR指纹在花生遗传多样性、品种鉴定和系统发育关系分析中的应用(落花生hypogaea)栽培品种和野生种。基因组学报,2001,44:763-772。
Krishna GK, Zhang J, Burow M, Pittman RN, Delikostadinov SG, Lu Y, Puppala N: Valencia花生遗传多样性分析(落花生hypogaeal)使用微卫星标记。细胞生物学杂志,2004,9:685-697。
马志刚,刘志刚,刘志刚:热带分子育种的高通量DNA提取方法。植物化学学报,2003,23(4):457 - 457。
Ewing B, Hillier L, Wendl MC, Green P:使用phred的自动序列跟踪的基调用。一、准确性评估。中国生物工程学报,1998,18(4):379 - 379。
串联重复序列查找器:一个分析DNA序列的程序。中国生物医学工程学报,1999,27(4):457 - 457。[http://tandem.bu.edu/trf/trf.html]
Kolodny GM:一种提高聚丙烯酰胺凝胶中核酸带银染色分辨率和灵敏度的改进方法。中国生物医学工程学报,2004,27(1):457 - 457。
Weir BS:基因数据分析。离散遗传数据的方法。马萨诸塞州桑德兰:Sinauer Associates;1990:125 -
Perrier X, jacquond - collet JP: DARwin软件。2006年,(http://darwin.cirad.fr/darwin/]
确认
作者感谢A. Gafoor先生的技术援助和H. D. Upadhyaya博士提供本研究中分析的种质资源。感谢亚洲开发银行和印度农业研究委员会(印度国家基金)分别资助了“亚洲半干旱热带地区贫困农民的快速作物改良”和“栽培花生生物和非生物胁迫耐受力的基于基因的遗传图谱和分子标记”的研究项目,使这项研究成为可能。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
LMC和ESM完成了大部分研究工作,VDQ、TDL和RKV分析了数据。JHC、ESM和RKV参与了工作的设计和规划,并解释了结果。RKV在LMC的帮助下对手稿进行了批判性的起草和编辑。所有作者都阅读并批准了最终的手稿。
电子辅助材料
12870 _2008_263_moesm1_esm.doc
附加文件1:新开发的SSR标记的特征及多态性现状所提供的数据代表了新SSR标记的详细信息,如标记名称、Genbank登录id、引物序列、PCR条件和扩增状态。(doc 751 kb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
卡克,l.m.,梅斯,e.s.,克劳奇,J.H.et al。新型微卫星标记的分离、鉴定及其在栽培花生多样性评价中的应用(落花生hypogaea).BMC植物生物学8,55(2008)。https://doi.org/10.1186/1471-2229-8-55
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-55
关键字
- 简单序列重复标记
- 叶锈病
- 重复的主题
- 多态信息内容
- 植物品种