摘要
背景
叶绿体基因组序列是非减数分裂的,通常是单代遗传,因此对物种间的相互关系非常有价值。我们的研究对象,Fagopyrum esculentum是石竹科蓼科的一种。石龙类和小行星的亲和性仍不确定,这可能是由于分类群的采样不足造成的。在这样的背景下,能够获得完整的叶绿体基因组序列Fagopyrum变得非常中肯。
结果
我们报道了栽培荞麦野生祖先的叶绿体全基因组序列。Fagopyrum esculentumssp。ancestrale.利用先前描述的基于pcr的方法和通用引物快速确定了该序列,该引物设计在叶绿体基因组多序列比对的支架上。荞麦叶绿体基因组的基因含量和顺序与之相似菠菜oleracea.然而,存在一些独特的结构差异:内含子的存在rpl2基因,移码突变rpl23基因和延伸的倒置重复区包括ycf1基因。系统发育分析了44个完整质体基因组中的61个蛋白质编码基因序列,为石竹科(包括蓼科)与菊科的姐妹关系提供了有力的支持。进一步,我们的分析也为Amborella是所有被子植物的姐妹,但有趣的是,在贝叶斯系统发育推论中基于前两个密码子位置Amborella与尼法尔斯联合。
结论
比较基因组学分析显示Fagopyrum叶绿体基因组具有其他几个叶绿体基因组所描述的特征基因含量和组织。然而,它有一些独特的结构特征,不同于以前报道的完整的叶绿体基因组序列。系统发育分析数据集,包括来自非核心石岩类的新序列,支持石岩类和小行星之间的姐妹关系。
背景
叶绿体被假设是从古代的内共生蓝藻进化而来的。它们是半自主的,拥有自己的基因组,编码一组蛋白质,协调光合作用过程和其他家务功能。叶绿体基因的非减数分裂和主要的单亲本遗传使它们成为植物系统发育研究中最具信息的实体。技术的进步和随之而来的序列捕获时间的缩短,使得最近几个叶绿体基因组的测序成为可能。植物系统发育学是这些技术进步的最大受益者。从全基因组序列分析中得到的系统发育树是完全或接近完全分解的,具有高度支持的节点。此外,对叶绿体基因进化速率的分析可以提供关于节点支持的信息,最近在Saxifragales的研究中证明,来自叶绿体倒置重复区域的缓慢进化基因为深层差异提供了支持[1].尽管有这些数据集,但基于完整叶绿体基因组序列的系统发育很容易因不完整的分类单元采样而产生伪影[2- - - - - -4].因此,从其他类群获得完整的叶绿体基因组序列对于系统发育研究是非常必要的。
本研究报告的完整的叶绿体基因组序列Fagopyrum esculentumssp。ancestrale荞麦的野生祖先[5].本种属于蓼科。根据APGII [6蓼科是石竹科的一种;然而,这个科在其中代表了一个独立的类群,即所谓的非核心石竹科[7]有时它被当作一个单独的目,Polygonales [8].目前尚缺乏对该群体的系统发育和基因组学研究。此外,石龙目作为一个整体的亲缘关系也仍有争议。以叶绿体基因组序列为基础的系统发育已获得菠菜(石龙目唯一具有已知叶绿体基因组序列的代表)是小行星的姐妹(例如参见[9,10])。其他一些研究纳入了较少数量的基因,但更广泛的分类单元采样,将它们置于包括小行星和罗西亚目在内的进化支的底部[6,11].为了验证石蕊亚目和小行星的姐妹关系是否由于石蕊亚目分类单元欠采样所致,还需要更多的序列信息。因此,将荞麦叶绿体基因组序列纳入综合系统发育分析有望有助于解决石楠类的亲和问题。
的序列Fagopyrum叶绿体基因组除了具有系统发育意义外,还可以为更实际的方面提供有用的信息。普通荞麦(f . esculentum)是一种广泛种植的多用途作物[12].获得叶绿体基因组序列可以突出荞麦的其他重要生理性状。此外,叶绿体基因组序列可用于开发物种特异性转化载体(有关综述见[13,14])。因此,对荞麦叶绿体基因组核苷酸序列的了解,为质体基因工程在荞麦中的应用开辟了一条途径。
方法
植物材料
的种子Fagopyrum本研究中所用物种均来自全俄豆科和大豆科作物研究所。植物材料红脚鹬,感冒,Reynoutria而且Coccoloba采自莫斯科国立大学植物园。
DNA提取、扩增和测序
按照制造商的说明,使用NucleoSpin植物DNA试剂盒(machery - nagel)从新鲜叶片组织中分离总细胞DNA。采用Encyclo PCR试剂盒(Evrogen JSC, Moscow, Russia)在PTC-220 DNA Engine Dyad (MJ Research)上进行PCR扩增。扩增时,参考[15],即将退火温度从55℃降低到50℃,进行35次触地PCR。对于一些引物对,通过改变退火温度的梯度PCR优化进行PCR。对于Long PCR,根据预期的扩增子大小,在68℃下扩增5 - 7分钟。PCR产物使用Gel Extraction & PCR Cleanup Kit (Cytokine Ltd, S.Petersburg, Russia)进行纯化。在ABI 3100测序仪上使用Big Dye Terminator v.3.1测序试剂盒(ABI, USA)进行自动测序。
测序策略和引物设计
荞麦叶绿体基因组序列是使用基于PCR的方法获得的,类似于之前在[15].的反向重复区域Fagopyrum利用ASAP引物扩增叶绿体基因组并进行测序;使用从已知被子植物叶绿体基因组序列多次比对开发的PCR引物扩增大、小单拷贝区域(引物在附加文件中列出)1).这些通用引物能够扩增整个叶绿体基因组FagopyrumPCR片段重叠,大小从0.5 kb到9 kb不等。由于在对齐的叶绿体基因组中缺乏序列守恒和很少的结构变化(例如,在IR-LSC和IR-SSC连接中),必须产生长片段。使用引物行走法对较大片段进行扩增和测序(完整的引物列表可在附加文件中获得)2).
Contig组装和注释
引物对生成的序列首先使用Blast 2序列(bl2seq)工具进行比对[16],可在NCBI网站上获得,以开发contigs,然后使用BioEdit软件进行组装[17].基因组注释草稿使用细胞器注释包DOGMA [18].利用BLAST相似性搜索对预测的标注进行进一步验证[19].
系统发育分析
为了进行系统发育分析,收集了一组来自44个叶绿体基因组的61个蛋白质编码基因1).所分析的物种代表了迄今已报道的被子植物叶绿体基因组序列的所有主要世系。
基因序列被解析以检测移码突变,并在必要时进行编辑。将序列翻译成衍生氨基酸序列,用MUSCLE ver进一步比对。3.6 [20.]然后进行人工校正。核苷酸序列比对覆盖在氨基酸序列比对上。
采用PAUP* ver进行最大简约(MP)方法进行系统发育分析。4.0 b8 [21].利用MrBayes程序进行了系统发育的贝叶斯推理。3.1.2 (22].使用Tree-Puzzle程序进行可选拓扑测试[23].
MP分析涉及启发式搜索,使用树等分和重连接(TBR)分支交换和100个随机添加重复。非参数自举分析[24],共100个重复,TBR分支交换。核苷酸和氨基酸序列都包括在分析中。
使用氨基酸和核苷酸序列数据集也进行了贝叶斯分析。核苷酸序列分析采用不同的划分策略:每个基因作为单独的分区(61个分区),基因按功能组合(4个分区:光合代谢、光合装置、基因表达等)和按密码子位置划分(3个分区)。对于61个氨基酸分区中的每一个,通过Modelgenerator ver中的BIC(贝叶斯信息准则)确定最合适的替代模型。0.43 [25].同样,对于每个核苷酸分区,最合适的核苷酸替代模型由Modeltest ver中的AIC(赤池信息准则)确定。3.7 [26].
贝叶斯分析在两次运行中分别使用三个链进行。每条链从随机树开始,生成2500000个氨基酸数据重复和5000000个核苷酸数据重复。这样得到的树每100代取样一次。不变位点的比例和伽玛分布率的形状在不同分区之间是不相关的。使用收敛诊断确定丢弃树的数量。
结果与讨论
荞麦叶绿体基因组的总体结构和基因含量
本研究中报道的核苷酸序列的GenBank登录号为EU254477。完整的叶绿体基因组Fagopyrum esculentumssp。ancestrale由159599个核苷酸碱基组成。这超过了开花植物叶绿体基因组的平均大小~155 Kb,特别是,几乎比其最近亲缘植物的叶绿体基因组大9 Kb菠菜oleracea(150725个基点)。观察到的尺寸增加是由于反向重复(IR)区域的扩张。IR区大小为30684,大单拷贝区(LSC)和小单拷贝区(SSC)分别为84888和13343 bp。整个质体的总AT含量为62%,LSC和SSC分别为63%和68% AT,倒置重复中富含59% AT。这与其他陆地植物的叶绿体基因组(菠菜oleracea- 63%,烟草的抗旱性- 62%,Lotus对虾- 64%,玉米- 62%)。IR区AT%较低反映核糖体RNA基因AT含量较低。
荞麦叶绿体基因组的基因顺序和含量与小麦相同菠菜(无花果。1).这种相似性不仅在功能基因中可见,而且在假基因中也可见。在荞麦和菠菜叶绿体基因组,所代表的序列rpl23而且ycf15基因被内部的停止密码子中断,表明它们的假基因状态。后一种情况在被子植物中很常见:对该地区进化模式的详细研究表明ycf15基因不是蛋白质编码基因[27].相反,rpl23基因已知存在于大多数开花植物中并发挥作用[28].因此,该基因的伪基因化可能代表了石竹类所特有的特征。到目前为止,四个序列的rpl23地区已报道石炭类;甜菜(甜菜属)、菠菜、荞麦和硅宾latifolia(石竹科)。对4个序列进行比较分析,发现均存在突变;但它们的确切结构是不同的(附加文件3.).在荞麦中有一个4 bp的插入,它会影响阅读框,导致停止密码子的产生。β而且菠菜共享一个公共的14 bp删除,这也改变了阅读帧。有趣的是在硅宾这个地区似乎受影响较小。它没有任何移码突变,但该基因在5'端附近有一个终止密码子。这一观察结果不能作为该基因无功能的唯一证据,因为在叶绿体中常见的RNA编辑现象可以将停止密码子编辑为意义密码子。测序神器可能是停止密码子存在的另一种合理解释。来评估是否伪化rpl23基因是石蕊质体的一种常见结构特征,其他物种的基因序列信息还需补充。转录rpl23基因已在菠菜中实验证实,但本研究未检测到蛋白质产物[29].因此,它似乎是一个表达的假基因。类似的情况也可能存在于荞麦的情况中。
之间共享的另一个特征Fagopyrum而且菠菜质体是存在两个基因的-psbL而且ndhD-使用ACG启动密码子。C到U RNA编辑介导的从原始ACG密码子生成AUG起始密码子是在叶绿体基因组衍生转录本中观察到的一种常见现象。RNA编辑已被实验证明psbL[30.),ndhD烟草中的基因。编辑ndhD基因在菠菜和金鱼中也有报道[31].自psbL而且ndhD基因编码对叶绿体功能至关重要的蛋白质,因此可以有把握地假设它们的转录本被编辑进去了Fagopyrum.这可以在未来的研究中借助cDNA测序或其他实验证据来证实。有趣的是,有两个结构特征是独一无二的Fagopyrum质体系。首先是IR-SSC边界相对于其他质体基因组的位置。在菠菜IRa/SSC边界位于的3′区ycf1基因。剩下的ycf1基因存在于SSC;的3'区域的副本ycf1位于IRb的基因产生ycf1IRb/SSC边界的假基因。荞麦具有扩大的IR区域和两个全长拷贝ycf1基因就这样产生了。
其次,荞麦质体基因组由18个含内含子的基因组成,而菠菜则减少到17个。这种差异是由于损失rpl2菠菜中的内含子[32].内含子的丢失rpl2基因在开花植物的几个谱系中独立存在[33],它被认为是在石蕊纲核心成员中观察到的一个特征。于是,出现了rpl2在荞麦(蓼科)中,内含子突出了其与石梨科核心植物的区别。的rpl2内含子也在Rumex(蓼科的另一代表),以及与蓼科很接近,常与石竹科(Caryophyllales)有关的Plumbaginaceae [33].
荞麦叶绿体基因组中反向重复区域的扩增
叶绿体基因组倒重复区(IR)的大小和边界在不同的植物物种中是不同的[34].以往对红外边界的研究主要集中在红外和LSC结[34,35],揭示了红外区域的多个膨胀和收缩实例,范围从几个碱基对到超过15 Kb。
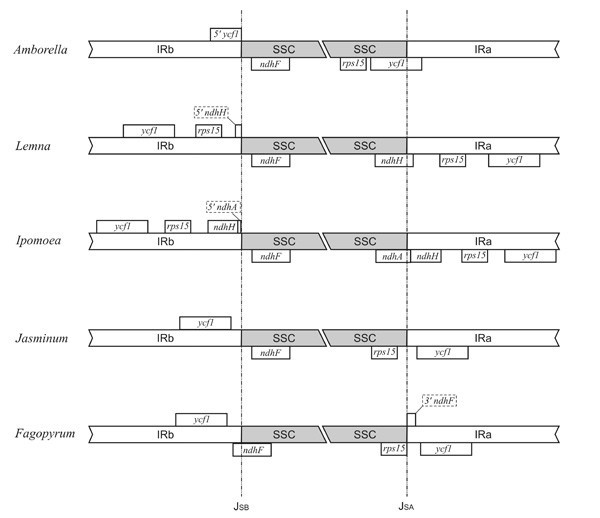
与其他开花植物相比,荞麦红外区域的扩大平均增加了5 Kb。所观察到的扩展和其他增强红外区域的顺序Fagopyrum以前曾报道过物种[36,37].该区域被证明与其他双子叶叶绿体基因组中IRa邻近的小单拷贝(SSC)区域高度相似,这使我们能够得出结论,这种扩展是由于SSC序列的包含。
在本研究中产生的序列信息证实了IR的扩展Fagopyrum esculentumssp。ancestrale.与大多数被子植物不同的是,扩张包含了ycf1基因是在所有已知双子叶和一些单子叶叶绿体基因组中发现的保守的叶绿体ORF。它的确切功能尚不清楚,但与另一个保守的叶绿体ORF一起ycf2,它已被证明对烟草叶绿体的功能至关重要[38].在大多数情况下,红外区域只包含的一部分ycf1另一部分位于质体的SSC区。的长度ycf1被复制的基因在156 bp之间睡莲属到1583 bpAmborella[27].但是,包含的ycf1在单子叶中也报道了IR区的基因浮萍属小[39],用星号表示迎春花[40),番薯紫竹[41].考虑到这些类群属于不同的开花植物谱系,扩展ycf有一个基因很可能是从一个祖先中独立出现在这些群体中的每一个Amborella像叶绿体基因组。再者,准确的模式ycf1基因扩增在不同物种中存在差异(图;2).例如,在素馨属IR/SSC边界位于ycf1-ndhF分隔符区域位于一个边界上,而另一个边界位于rps15-ycf1垫片。在浮萍属复制包括rps15基因和5'区ndhH基因。在番薯这个扩展甚至进一步扩展,包括了completeycf1的基因,rps15的基因,ndhH基因,和一个短的区域ndhA基因的第一个外显子。对于荞麦,扩展区域包括ycf1-ndhF间隔器和70bp的3'端ndhF基因。
有趣的是,扩展区域的序列Fagopyrum esculentumssp。ancestrale与Aii等人报道的序列不同。[37].作者在该区域报道了3711 bp的反演。这种反转影响了转录的连续性ycf1基因,导致读取框中断。在本研究中报道的序列中观察到的差异可以通过使用不同的荞麦基因型来合理地解释。然而,报道的反转可以潜在地用于追踪各种荞麦品种的起源。重要的是,在这项工作中报道的一组荞麦特异性引物有望使这一和其他潜在有趣的结构特征的未来研究成为可能。
我们进一步研究了与之相关的其他物种IR的扩展Fagopyrum esculentumspp。ancestrale使用基于PCR的方法和两组引物(详情见附加文件)4).一套底漆退火到ndhF而且rps15SSC内的基因。另一种底漆对固化和退火都是通用的ycf1基因。它在叶绿体基因组的SSC中只有一个不具有IR膨胀的退火位点(例如,菠菜),但在IR中有两个退火位点(在正方向和反方向)ycf1由于红外膨胀(如Fagopyrum).
这些研究表明,在研究中采样的所有蓼科成员中都存在观察到的IR膨胀,其中包括另外两个荞麦物种f . esculentum(f . tataricum而且f . homotropicum).相似的红外膨胀观察到红脚鹬,感冒,Reinoutria而且Coccoloba物种(图;3.).对于荞麦品种,IRb/SSC和SSC/IRa边界的测序进一步证实了IR的扩展(登录号EU272335 - EU272336)F.tataricum和EU272337 - EU272338f . homotropicum).因此,从这些研究可以清楚地看出,膨胀不仅仅是一个特征Fagopyrum,也适用于其他相关属,这可能代表了蓼科的一个共同特征。从荞麦品种中获得的不同序列的比较分析显示,IR/SSC边界的精细结构存在微小变化。在f . esculentum而且f . homotropicumSSC/IR边界相同,位于ndhF基因(IRb/SSC)和上游2 bprps15基因(SSC / IRb)。总的来说,rps15-ycf1在上述物种中,间隔区包含在IR中F. tataricum rps15基因和21 bprps15-ycf1基因间间隔区位于SSC中。基于核和叶绿体位点的系统发育分析f . esculentum而且f . homotropicum彼此密切相关[42,43].这些物种的IR/SSC边界的精细结构与这些数据一致,进一步的研究可以用于说明其他的系统发育关系Fagopyrum.
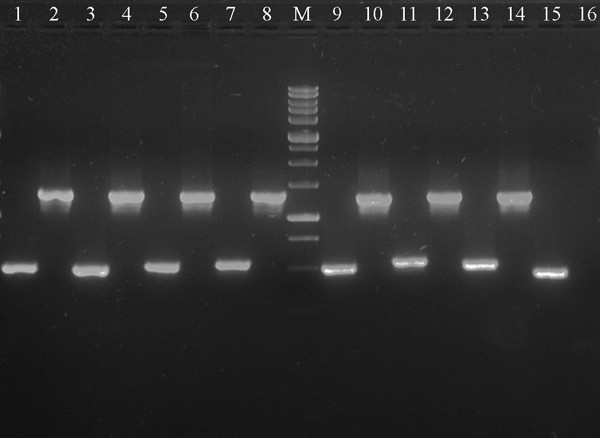
蓼科植物IR区扩展.溴化乙锭染色1.5%琼脂糖凝胶显示PCR扩增rps15-ycf1而且ycf1-ndhF所选蓼科类群的间隔菠菜oleracea.1,2 -Fagopyrum esculentumssp。ancestrale rps15-ycf1而且ycf1-ndhF片段分别为3,4 -f . homotropicumSSCrps15-ycf1而且ycf1-ndhF, 5,6 -F. tataricum rps15-ycf1而且ycf1-ndhF, 7,8 -大白菜-ycf1而且ycf1-ndhF, 9,10 -唐古提黄rps15-ycf1而且ycf1-ndhF, 11, 12 -粳稻rps15-ycf1而且ycf1-ndhF, 13,14 -葡萄球菌rps15-ycf1而且ycf1-ndhF, 15, 16 -菠菜15-ycf1而且ycf1-ndhF(ycf1-ndhF-没有放大)。M是0.25 - 10 Kb DNA阶梯(SibEnzyme Ltd, Moscow, Russia),最下方可见通道对应0.5 Kb。
系统发育分析
以确定的相对位置Fagopyrum在被子植物中进行了有效质体基因组序列的比较系统发育分析。该数据集包括来自44个不同类群的61个连接的蛋白质编码基因序列,其中包括两个裸子植物物种作为外群。核苷酸和氨基酸数据集在排除了模糊对齐位置后分别包含42504和14168个字符。
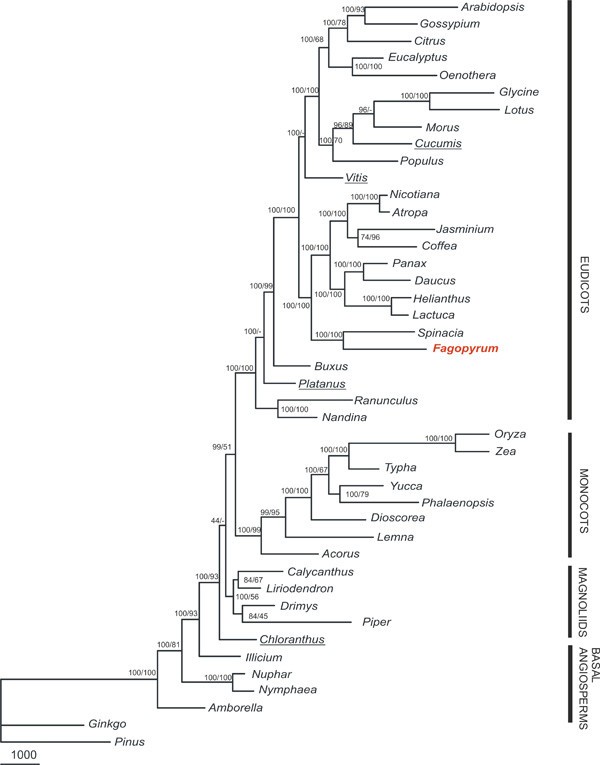
所有对齐的核苷酸位置的最大简约(MP)分析结果是一个完全解析的树,其中大多数节点在自举分析中获得了高支持(图2)。4),除了放置珠兰.MP分析氨基酸数据也产生了单一的树,但其拓扑结构因位置不同而不同葡萄属(它成为rosids和asterids的姐妹,boothstrap支持100%),Cucumis(它与桑属自举支持64%),法国梧桐(Ranunculales的姐妹,低引导支持)和珠兰(低引导支持的magnoliids的姐妹)。
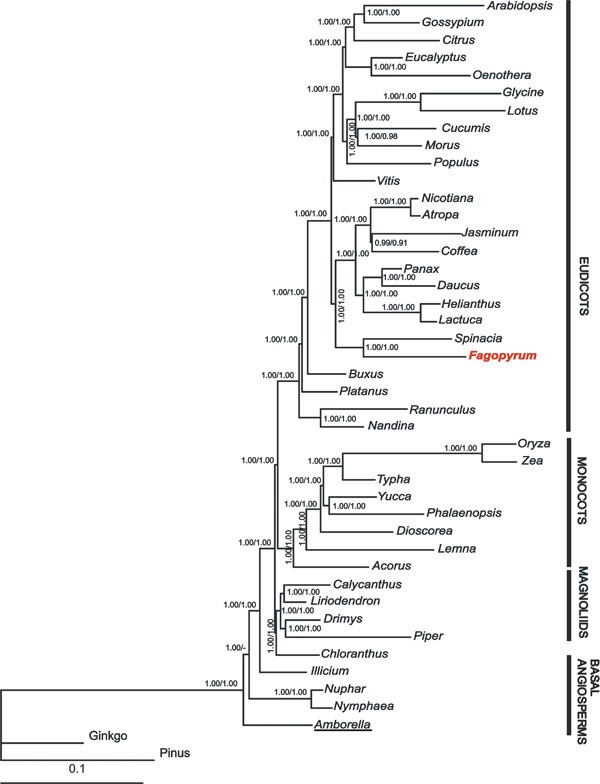
从分割的核苷酸矩阵和氨基酸序列分析中得到的贝叶斯树的拓扑结构是相同的(图2)。5),而不考虑所选择的分区方案。从贝叶斯分析中排除第三个密码子位置导致了一个相似的树,除了Amborella与尼法尔斯联合。
之前已经证明,复杂数据集的划分极大地提高了贝叶斯推理的性能[44- - - - - -46].因此,我们在分析中使用了不同的分区方案(61个分区、4个分区和3个分区),但是,对结果树的比较显示结果树拓扑没有差异。同时,如果比较边际似然的调和均值(分别为61,4和3个分区的-455638.67,-455391.40,-449073.19),则将数据集划分为3个分区似乎更符合逻辑。
总的来说,我们推断的叶绿体系统发育与最近发表的分子树非常相似,在分子树中,木兰、单子叶和真子叶的相互关系和单系性得到了强有力的支持[9,10,47,48].在所有的衍生系统进化树中,Fagopyrum而且菠菜(两者都代表石楠科理智lato)与小行星形成姐妹支系。为了确定是否可以区分石蕊作为rosids姐妹群(AT1)或rosids和asterids姐妹群(AT2)的替代拓扑,Shimodaira-Hasegawa测试[49],并进行预期似然权重[50]采用RELL优化计算[51].根据这些测试的结果,石蕊的替代位置明显差于最优拓扑,支持石蕊和小行星的密切关系(p <0.001, c<0.001对于AT1和AT2)。
蔷薇属的一种枝系,基部为葡萄属,是小行星属和石竹属的姐妹。特别是在我们的数据集中包含桑属序列导致稳定Cucumis在我们所有的分析中,珠壳虫I是单系的。在最近发表的基于叶绿体序列的系统发育树中,对Cucumis是不稳定的。它要么与桃金娘科结合(桉树、月见草)或嵌套在MP树的eurosids I中[9,47,52].密切的关系桑属而且Cucumis已在之前演示过[53]在我们的贝叶斯树中,这些物种形成一个具有高后验概率值的聚类。基于我们的MP分析和之前报道的64个质体基因组的分析,这种分组是有问题的[10把他们作为一个等级。然而,很明显,珠虫I可能被认为是单系的。
毛茛属植物而且南天竹属似乎构成了最基部的分枝。进行了是所有衍生树中最接近核心的同盟者,而法国梧桐在基底胚珠之间不能确定。在氨基酸序列自举重采样的MP分析中,不支持它的中间位置。
Amborella在被子植物系统发育中,若虫科、金毛科和木兰科仍然是问题最多的类群。它们的系统发育关系仍然不明确,取决于分析过程中使用的方法和数据。没有关于Amborella(睡莲属或所有其他被子植物的姐妹)和珠兰根据叶绿体基因组序列的系统发育分析,(仅为木兰科植物或木兰科植物、单子叶植物和真木兰科植物的姐妹植物)可被拒绝[9,10].显然,有限的分类单元采样是现阶段叶绿体系统发育分析的第一个薄弱环节,因此,只有从基础被子植物和裸子植物中引入更多的代表,才能得到可靠的关系。这同样适用于单子叶和双子叶,只要许多重要的世系缺失。人们不应该被高的自举或后经验概率值所迷惑,因为在系统发育分析中,当使用大量的特征(如整个叶绿体基因组序列)而使用少量的分类单元时,往往会获得对可疑分支的高支持。例如,Calycanthus在一些树上,它被视为eudicots的姐妹,具有较高或中等的支持度[53- - - - - -57]直到分析中纳入了更多的玉兰花[47].为了克服分类单元采样的问题,有必要确定最有效的基因进行系统发育分析,然后在更多的分类单元中分析这些基因。最近RPO编码质体rna -聚合酶亚基的基因已被证明可以产生与整个质体基因组系统发育相似的系统发育树的拓扑结构[58].另一个例子是使用叶绿体倒重复序列区编码的缓慢进化基因,这有助于解决Saxifragales内部的系统发育关系[1].
ACG起始密码子在被子植物rpl2、psbL和ndhD基因中的系统发育分布
被子植物质体中有几个叶绿体基因具有非典型起始密码子ACG。对于某些物种(烟草、金鱼、菠菜、玉米),有强有力的实验证据表明该密码子被编辑为标准AUG密码子[30.,31,59这表明类似的机制可能存在于其他物种中。由于RNA编辑模式被认为是广泛平行进化的主体,因此它们并不一定具有系统发育方面的信息[60].然而,对于三个质体基因-rpl2,psbL而且ndhD- RNA编辑与系统发育之间的相关性已被报道[61].本研究基于7种被子植物质体的序列;结论是RNA编辑在psbL基因出现在被子植物的共同祖先中,然后在单子叶植物中丢失。编辑rpl2基因只在单子叶中出现ndhD基因仅在双子代中观察到[61].
从被子植物中获得的大量完整的叶绿体基因组序列以及对开花植物系统发育的认识的提高使这一结论得以重新评估。我们对基因起始密码子中潜在的RNA编辑位点进行了研究rpl2,psbL而且ndhD迄今为止已测序的44种种子植物叶绿体基因组(包括Fagopyrum),并以本文报道的系统发育树为框架,研究了其在不同进化谱系中的分布(附加文件5).首先,我们的分析表明,在种子植物中,ACG起始密码子在种子植物中rpl2基因不仅是单子叶植物的特征,也是一些早期分化被子植物的特征(Amborella,珠兰和玉兰属)和下花萼(毛茛属和法国梧桐).然而,它是缺席的八角若虫属也是被子植物基系的代表。因此,很难断定这个位点的RNA编辑是在被子植物的共同祖先中出现,然后在被子植物中丢失的八角和睡莲目。或者,它可能是后来演化的,在ANITA级别的成员分化和它在Amborella可能是由于平行进化。编辑ndhD基因似乎是在被子植物的共同祖先中进化而来,然后消失在睡莲属在一些单子叶中。在迄今为止研究过的所有草中ndhD基因具有标准的ATG起始密码子[62- - - - - -65这表明这个家族的所有成员都有可能是共同的。ACG起始密码子的分布模式psbL基因更复杂。它的存在是大多数早期分化被子植物的特征(除了珠兰而且Calycanthus),但在所有采样的单子叶植物和几个不同的双子叶植物谱系中均不存在。我们假设该基因中ACG起始密码子的存在(以及RNA编辑)是被子植物的一种祖先特征状态,而它的缺失是由于多次并行的损失。然而,这一观察结果需要进一步使用更广泛的分类单元抽样进行验证。
结论
荞麦的完整序列(Fagopyrum esculentumssp。ancestrale)叶绿体DNA已使用基于PCR的方法生成,验证了该方法的实用性,特别是对于未重排的被子植物叶绿体基因组。这是蓼科和非核心石竹科植物的第一个质体基因组测序结果。荞麦质体的基因含量和顺序与小麦相似菠菜oleracea,其相对于核心石竹科。然而,揭示了两种结构上的差异。首先是内含子的存在rpl2二是反向重复区域的扩展。扩展区域的确切结构与以前报告的数据不同,并且包含一个完整的ORFycf1基因。
44个类群61个蛋白质编码基因的系统发育分析,包括新获得的叶绿体基因组序列(Fagopyrum)为Caryophyllales sensu lato(包括Polygonaceae)与asterids的姐妹关系提供了有力的支持。以前叶绿体基因组系统发育研究的大多数其他结论也得到了证实,特别是叶绿体基因组的位置Amborella(或Amborella+若虫属)为基性被子植物,姐妹关系珠兰和木兰属及毛茛属的位置(毛茛属植物+南天竹属),作为最早的分支血统的eudiicots。
植物起始密码子中潜在RNA编辑位点分布的研究rpl2,psbL而且ndhD在被子植物中发现的基因与系统发育有一定的相关性,但也证实了RNA编辑的进化是一个广泛的平行变化的主题。
缩写
- APG:
-
被子植物系统发育类群
- 尽快:
-
质体扩增、测序、注释
- 英国石油公司:
-
碱基对
- cp:
-
叶绿体
- 信条:
-
双细胞器基因组注释器
- 红外光谱:
-
反向重复
- Kb:
-
千碱基
- LSC:
-
大单份
- 议员:
-
最大的吝啬
- 子:
-
开式阅读架
- 聚合酶链反应:
-
聚合酶链反应
- SSC:
-
小单份
- 为:
-
树bisection-reconnection。
参考文献
- 1.
Jian S, Soltis PS, Gitzendanner MA, Moore MJ, Li R, Hendry TA, Qiu Y-L, Dhingra A, Bell CD, Soltis DE:求解Saxifragales中古老的快速辐射。系统生物学,2008,57(1):38-57。10.1080 / 10635150801888871。
- 2.
Soltis DE, Albert VA, Savolainen V, Hilu K, Qiu YL, Chase MW, Farris JS, Stefanoviæ S, Rice DW, Palmer JD, Soltis PS:基因组尺度数据,被子植物关系,和“结束不一致”:系统发育的警示故事。植物科学进展,2004,9:477-483。10.1016 / j.tplants.2004.08.008。
- 3.
Stefanoviæ S, Rice DW, Palmer JD:长枝吸引,分类单元采样,和最早的被子植物:Amborella或单子叶?中国生物医学工程学报,2004,4(1):35-10.1186/1471-2148-4-35。
- 4.
Degtjareva GV, Samigullin TH, Sokoloff DD, Valiejo-Roman CM:基因抽样与分类单元抽样:是Amborella(amborelaceae)是所有现存被子植物的姐妹类群。朱文斌,2004,39(1):1 - 7。
- 5.
Ohnishi O:发现普通荞麦的野生祖先。金荞麦,1991,11:5-10。
- 6.
被子植物系统发育组(APG II):开花植物目和科被子植物系统发育组分类的更新:APG II。环境科学学报,2003,29(3):349 - 349。10.1046 / j.1095 8339.2003.t01 - 1 - 00158. x。
- 7.
Cuènoud P, Savolainen V, Chatrou LW, Powell M, Grayer RJ, Chase M:基于核18S rDNA和质粒rbcL、atpB和matK DNA序列的石龙叶植物分子系统发育。中国生物医学工程学报,2002,29(3):344 - 344。10.3732 / ajb.89.1.132。
- 8.
Judd WS, Campbell CS, Kellogg EA, Stevens PF:植物系统学:系统发育方法。Sinauer Associates, Inc.,桑德兰,马萨诸塞州;2002.
- 9.
Hansen DR, Dastidar SG, Cai Z, Penaflor C, Kuehl JV, Boore JL, Jansen RK: 4种早期分化被子植物全叶绿体基因组序列的系统发育和进化意义:进行了(黄杨科),珠兰(金粟兰科),薯蓣属(薯蓣科)八角(Schisandraceae)。分子系统发育与进化,2007,45(2):547-563。10.1016 / j.ympev.2007.06.004。
- 10.
Jansen RK, Cai Z, Raubeson LA, Daniell H, dePamphilis CW, leeben - mack J, Mueller KF, Guisinger-Bellian M, Haberle RC, Hansen AK, Chumley TW, Lee S-B, Peery R, McNeal JR, Kuehl JV, Boore JL:分析来自64个质体基因组的81个基因,解析被子植物的关系并识别基因组尺度的进化模式。美国国家科学研究院。2007, 44(3): 369- 374。10.1073 / pnas.0709121104。
- 11.
Soltis D, Soltis P, Chase M, Mort M, Albach D, Zanis M, Savolainen V, Hahn W, Hoot S, Fay M, Axtell M, Swensen S, Nixon K, Farris J:从18S rDNA, rbcL和atpB序列组合数据集推断被子植物系统发育。中国生物工程学报,2004,26(3):366 - 366。
- 12.
Campbell GC:荞麦Fagopyrum esculentumMoench。促进保护和利用未充分利用和被忽视的作物。19 Inst Plant Genet Crop Plant Res. Gatersleben/IPGRI,罗马;1997.
- 13.
葛伟奇,丹尼尔。H:叶绿体基因工程:最新进展和未来展望。植物科学进展,2005,24(2):83-107。10.1080 / 07352680590935387。
- 14.
Dhingra, James VA, Koop HU, Mok MC, Paepe RD, Gallo M, Folta KM:烟草。转基因:生物技术植物百科全书。编辑:Kole C, Hall TC布莱克威尔出版;2008.
- 15.
Dhingra, Folta K: ASAP:质体的扩增、测序和注释。中国生物医学工程学报,2005,6:176-10.1186/1471-2164-6-176。
- 16.
Tatusova TA, Madden TL: BLAST 2 SEQUENCES,一种比较蛋白质和核苷酸序列的新工具。微生物学通报,1999,27(3):357 - 357。10.1111 / j.1574-6968.1999.tb13575.x。[http://www.ncbi.nlm.nih.gov/blast/bl2seq/wblast2.cgi]
- 17.
大厅TA: BioEdit:一个用户友好的生物序列比对编辑器和分析程序,适用于Windows 95/98/NT。化学学报,1999,41:95-98。
- 18.
王志刚,王志刚,杨森等:基于DOGMA的细胞器基因组自动注释。生物信息学,2004,20:352 - 355。10.1093 /生物信息学/ bth352。[http://dogma.ccbb.utexas.edu/]
- 19.
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ:基本的局部对齐搜索工具。中华分子生物学杂志,2000,26(3):344 - 344。
- 20.
Edgar RC: MUSCLE:多序列比对,高精度,高通量。自然科学进展,2004,32(5):1792-1797。10.1093 / nar / gkh340。
- 21.
Swofford DL: PAUP*:使用简约(*和其他方法)的系统发育分析,ver。4.0.2003年,桑德兰MA: Sinauer Associates
- 22.
朗奎斯特F, Huelsenbeck JP: MRBAYES 3:混合模型下的贝叶斯系统发育推断。生物信息学,2003,19:1572-1574。10.1093 /生物信息学/ btg180。
- 23.
薛晓明,王晓明,王晓明。基于并行计算的最大似然系统发育分析方法。生物信息学,2002,18:502-504。10.1093 /生物信息学/ 18.3.502。
- 24.
Felsenstein J:系统发生的置信限制:一种使用自举法的方法。《科学进展》,1985,39:783-791。10.2307 / 2408678。
- 25.
Keane TM, Creevey CJ, Pentony MM, Naughton TJ, McInerney JO:对氨基酸基质选择方法及其在经验数据上的应用的评估表明,对基质选择的临时假设是不合理的。中国生物医学工程学报,2006,6:29-10.1186/1471-2148-6-29。
- 26.
Posada D, Crandall KA: MODELTEST:测试DNA替代模型。生物信息学,1998,14:817-818。10.1093 /生物信息学/ 14.9.817。
- 27.
Raubeson LA, Peery R, Chumley TW, Dziubek C, Fourcade MH, Boore JL, Jansen RK:比较叶绿体基因组学:包括被子植物新序列的分析黄睡莲advena而且毛茛属植物macranthus.中国生物医学工程学报,2007,30(4):344 - 344。
- 28.
Sugiura M:叶绿体基因组。植物分子生物学杂志,1992,19(4):349 - 349。10.1007 / BF00015612。
- 29.
托马斯F,马斯内O,多恩AM,布里亚特JF,马彻R:表达的rpl23日,rpl2,石头剪刀菠菜叶绿体中的19个基因。自然科学进展,1998,16(6):461- 472。10.1093 / nar / 16.6.2461。
- 30.
Kudla J, Igloi GL, Metzlaff M, Hagemann R, Kössel H:烟草叶绿体中RNA编辑导致可翻译的psbL mRNA在起始密码子内通过C到U的取代形成。中国农业科学,2002,23(4):497 - 497。
- 31.
Neckermann K, Zeltz P, Igloi GL, Kossel H, Maier RM: RNA编辑在叶绿体基因组起始密码子守恒中的作用。中国生物工程学报,2004,29(3):357 - 357。10.1016 / 0378 - 1119(94) 90290 - 9。
- 32.
Schmitz-Linneweber C, Maier RM, Alcaraz JP, Cottet A, Herrmann RG, Mache R:菠菜质体染色体(菠菜oleracea):完整的核苷酸序列和基因组织。植物分子生物学,2001,45(3):307-315。10.1023 /: 1006478403810。
- 33.
Downie SR, Olmstead RG, Zurawski G, Soltis DE, Soltis PS, Watson JC, Palmer JD:六个独立的叶绿体DNA丢失rpl双子叶植物中的内含子:分子和系统发育意义。自然科学进展。1994,45:1245-1259。10.2307 / 2409731。
- 34.
Goulding SE, Olmstead RG, Morden CW, Wolfe KH:叶绿体反转重复的涨落。中国生物医学工程学报,1996,26(3):344 - 344。10.1007 / BF02173220。
- 35.
普朗科特GM, Downie SR: Apioideae亚科Apioideae叶绿体倒置重复的扩张和收缩。系统工程学报,2000,25:648-667。10.2307 / 2666726。
- 36.
Kishima Y, Ogura K, Mizukami K, Mikami T, Adachi T:荞麦品种叶绿体DNA分析:系统发育关系,生殖系统起源和延长的倒置重复序列。中国植物科学,1995,18(3):344 - 344。10.1016 / 0168 - 9452 (95) 04130 - m。
- 37.
Aii J, Kishima Y, Mikami T, Adachi T:荞麦叶绿体基因组IR的扩增是由于SSC序列的加入,该序列可以通过反转介导。现代遗传学,1997,31(3):276-279。10.1007 / s002940050206。
- 38.
Drescher A, Ruf S, Calsa T, Carrer H, Bock R:高等植物叶绿体基因组编码的两个最大的开放阅读框是必需基因。植物学报,2000,22(2):97-104。10.1046 / j.1365 - 313 x.2000.00722.x。
- 39.
马尔达诺夫AV,拉文NV,库兹涅佐夫BB,萨米古林TH,安东诺夫AS,科尔加诺娃TV,斯克里亚宾KG:浮萍完整序列(浮萍属小)叶绿体基因组:与其他被子植物的结构组织和系统发育关系。J Mol Evol。
- 40.
李海林,李志强,李志强,金桂娟:植物叶绿体基因组的基因重定位素馨属而且Menodora(夹竹桃科)是由于多重,重叠倒置。Mol生物学报,2007,24(5):1161-1180。10.1093 / molbev / msm036。
- 41.
Mc Neal JR, Kuehl JV, Boore JL, de Pamphilis CW:完整的质体基因组序列表明寄生植物属中光合基因的保留有很强的选择作用五角.中国生物医学工程学报,2007,27(3):377 - 388。
- 42.
Yasui Y, Ohnishi O:种间关系Fagopyrum(蓼科的核苷酸序列所揭示的:而且accD基因及其基因间区。学报学报,1998,35(4):344 - 344。10.2307 / 2446346。
- 43.
西本Y, Ohnishi O, Hasegawa M:核和叶绿体DNA树的拓扑不一致提示蓼科蓼属尾叶群的杂交。基因与遗传系统。2003,38(3):357 - 357。10.1266 / ggs.78.139。
- 44.
Nylander JAA, Ronquist F, Huelsenbeck JP, Nieves-Aldrey JL:组合数据的贝叶斯系统发育分析。中国生物医学工程学报,2004,53:47-67。10.1080 / 10635150490264699。
- 45.
Brandley MC, Schmitz A, Reeder TW:分区贝叶斯分析,分区选择和科学蜥蜴的系统发育关系。中国生物医学工程学报,2005,54(3):373-390。10.1080 / 10635150590946808。
- 46.
Brown JM, Lemmon AR:贝叶斯系统发育中数据划分的重要性和贝叶斯因子的效用。中国生物医学工程学报,2007,56(4):643-655。10.1080 / 10635150701546249。
- 47.
蔡z, Penaflor C, Kuehl JV, Leebens-Mack J, Carlson JE, dePamphilis CW, Boore JL, Jansen RK:植物全质体基因组序列Drimys,鹅掌楸而且风笛手:对木兰类的系统发育关系的影响。中国生物医学工程学报,2006,26(6):457 - 457。
- 48.
Saarela FM, Rai HS, Doyle JA, Endress PK, Mathews S, Marchant AD, Briggs BG, Graham SW:被子植物系统发育树的一个新分支——Hydatellaceae。自然科学学报,2007,29(3):344 - 344。10.1038 / nature05612。
- 49.
Shimodaira H, Hasegawa M:对数可能性的多重比较及其在系统发育推断中的应用。中国生物医学工程学报,1999,26(3):344 - 344。
- 50.
Strimmer K, Rambaut A:推断可能错误指定的基因树的置信集。中国生物医学工程学报,2002,26(4):457 - 457。10.1098 / rspb.2001.1862。
- 51.
Kishino H, Hasegawa M:从DNA序列数据估计进化树拓扑的最大似然估计的评估,以及人科的分支顺序。中华药理学杂志,1989,29:170-179。10.1007 / BF02100115。
- 52.
吴春生,王云云,刘明生,周明生:植物叶绿体基因组(cpDNA)铁树taitungensis和56个cp蛋白编码基因Gnetum parvifolium:对现存种子植物cpDNA进化和系统发育的深入了解。分子生物学杂志,2007,24(6):1366-1379。10.1093 / molbev / msm059。
- 53.
Ravi V, Khurana PJ, Tyagi AK, Khurana P:桑树叶绿体基因组:全核苷酸序列、基因组织及比较分析。树遗传与基因组,2006,3(1):49-59。10.1007 / s11295 - 006 - 0051 - 3。
- 54.
Leebens-Mack J, Raubeson LA, Cui L, Kuehl J, Fourcade M, Chumley T, Boore JL, Jansen RK, dePamphilis CW:基础被子植物叶绿体基因组系统发育的识别:基于Felsenstein区。中国生物医学工程学报,2005,26(3):344 - 344。10.1093 / molbev / msi191。
- 55.
Jansen RK, Kaittanis C, Saski C, Lee SB, Tomkins J, Alverson AJ, Daniell H:植物系统发育分析葡萄属基于完整叶绿体基因组序列的蔷薇科植物:分类单元取样和系统发育方法对解决蔷薇科植物间关系的影响。中国生物医学工程学报,2006,6:32-10.1186/1471-2148-6-32。
- 56.
鲍usher MG, Nameirakpam DS, Lee S, Jansen RK, daniel H:植物叶绿体全基因组序列素类(l)山脊菠萝:与其他被子植物的组织和系统发育关系。中国生物医学工程学报,2006,6:21-10.1186/1471-2229-6-21。
- 57.
Ruhlman T, Lee SB, Jansen RK, Hostetler JB, Tallon LJ, Town CD, Daniell H:全质体基因组序列胡萝卜胡萝卜:被子植物的生物技术和系统发育意义。中国生物医学工程学报,2006,27(3):344 - 344。
- 58.
Logacheva MD, Penin AA, Samigullin TH, Vallejo-Roman CM, Antonov AS:用叶绿体基因组序列研究开花植物的系统发育:寻找“幸运基因”。生物化学(Mosc)。2007, 12: 1324-1330。10.1134 / S0006297907120061。
- 59.
Hoch B, Maier RM, Appel K, Igloi GL, Kossel H:通过创建起始密码子编辑叶绿体mRNA。自然学报,1997,33(3):388 - 388。10.1038 / 353178 a0。
- 60.
Freyer R, Kiefer-Meyer M-C, Kossel H:陆地植物主要世系中质体RNA编辑的发生。美国国家科学研究院。1997, 44(3): 489 - 497。10.1073 / pnas.94.12.6285。
- 61.
Tsudzuki T, Wakasugi T, Sugiura M:高等植物叶绿体RNA编辑位点的比较分析。中华生物医学杂志,2001,53(4):327-332。10.1007 / s002390010222。
- 62.
Asano T, tsuzuki T, Takahashi S, Shimada H, Kadowaki K:甘蔗全核苷酸序列(蔗糖officinarum)叶绿体基因组:四种单子叶叶绿体基因组的比较分析。中国生物医学工程学报,2004,11(2):93-99。10.1093 / dnares / 11.2.93。
- 63.
Hiratsuka J, Shimada H, Whittier R, Ishibashi T, Sakamoto M, Mori M,近藤C, Honji Y, Sun C,孟B, Li Y, Kanno A, Nishizawa Y, Hirai A, Shinozaki K, Sugiura M:水稻的完整序列(栽培稻)叶绿体基因组:不同tRNA基因之间的分子间重组是谷物进化过程中质体DNA反转的主要原因。中国生物医学工程学报,1998,26(3):344 - 344。10.1007 / BF02464880。
- 64.
Saski C, Lee SB, Fjellheim S, Guda C, Jansen R, Luo H, Tomkins J, Rognli O, Daniell H, Clarke J:植物叶绿体全基因组序列大麦芽,高粱二色的而且Agrostis多茎目,并与其他草的基因组进行比较分析。中国生物医学工程学报,2007,29(4):379 - 379。10.1007 / s00122 - 007 - 0567 - 4。
- 65.
Maier RM, Neckermann K, Igloi GL, Kossel H:玉米叶绿体基因组全序列:基因含量、分化热点及转录编辑对遗传信息的微调。中华分子生物学杂志,1995,26(3):344 - 344。10.1006 / jmbi.1995.0460。
- 66.
若树T, tsuzuki J, Ito S, Nakashima K, tsuzuki T, Sugiura M:全部损失ndh通过对黑松的整个叶绿体基因组进行测序确定的基因松果体thunbergii.美国国家科学研究院。1994, 91: 9794-9798。10.1073 / pnas.91.21.9794。
- 67.
Goremykin VV, Hirsch-Ernst KI, Wolfl S, Hellwig FH:通过对木伞伞属植物叶绿体基因组序列的分析,认为木伞伞属植物不是基性被子植物。中国生物医学工程学报,2003,20:1499-1505。10.1093 / molbev / msg159。
- 68.
Goremykin VV, Hirsch-Ernst KI, Wolfl S, Hellwig FH:植物叶绿体基因组睡莲属阿尔巴:全基因组分析和最基础被子植物的鉴定问题。中国生物医学工程学报,2004,21:1445-1454。10.1093 / molbev / msh147。
- 69.
Goremykin VV, Hirsch-Ernst KI, Wolfl S, Hellwig FH:基底被子植物叶绿体基因组Calycanthus floridus-结构和系统发育分析。植物科学学报,2003,26(3):357 - 357。10.1007 / s00606 - 003 - 0056 - 4。
- 70.
Goremykin VV, Holland B, Hirsch-Ernst KI, Hellwig FH:分析菖蒲属菖蒲叶绿体基因组及其系统发育意义。中华生物医学杂志,2005,22:1813-1822。10.1093 / molbev / msi173。
- 71.
常春林,林昌昌,林毅普,陈海辉,陈文华,程春春,林春春,刘明生,常春昌,周明生:植物叶绿体基因组蝴蝶兰属阿佛洛狄忒(兰科):进化速率与草的比较分析及其系统发育意义。中国生物医学工程学报,2006,23:279-291。10.1093 / molbev / msj029。
- 72.
Sato S, Nakamura Y, Kaneko T, Asamizu E, Tabata S:植物叶绿体基因组的完整结构拟南芥.DNA学报,1999,6:283-290。10.1093 / dnares / 6.5.283。
- 73.
Schmitz-Linneweber C, Regel R, Du TG, Hupfer H, Herrmann RG, Maier RM:块状染色体Atropa颠茄并与之比较烟草: RNA编辑在植物物种形成过程中产生分化的作用。中国生物医学工程学报,2002,19:1602-1612。
- 74.
Nalapalli S, Bausher MG, Lee SB, Jansen RK, Daniell H:咖啡的完整核苷酸序列(Coffea阿拉比卡L.)叶绿体基因组:被子植物之间的生物技术和系统发育关系的组织和意义。中国生物工程学报,2007,5:339-953。10.1111 / j.1467-7652.2007.00245.x。
- 75.
Kim JS, Jung JD, Lee JA, Park HW, Oh KH, Jeong WJ, Choi DW, Liu JR, Cho KY:黄瓜的完整序列和组织(Cucumis巨大成功l .简历。Baekmibaekdadagi)叶绿体基因组。植物细胞学报,2006,25(4):334-340。10.1007 / s00299 - 005 - 0097 - y。
- 76.
Steane DA:塔斯马尼亚蓝胶叶绿体基因组的完整核苷酸序列,Eucaliptus桉(桃金娘科)。中国生物医学工程学报,2005,30(4):344 - 344。10.1093 / dnares / dsi006。
- 77.
李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚:植物叶绿体全基因组序列分析大豆并与其他豆科植物基因组进行了比较分析。中国生物医学工程学报,2005,29(3):349 - 349。10.1007 / s11103 - 005 - 8882 - 0。
- 78.
Lee SB, Kaittanis C, Jansen RK, Hostetler JB, Tallon LJ, Town CD, Daniell H:植物叶绿体全基因组序列陆地棉:与其他被子植物的组织和系统发育关系。BMC基因组学杂志,2006,7:61-10.1186/1471-2164-7-61。
- 79.
李文杰,张志刚,张志刚,等。菊科植物菊苣属和菊苣属植物质体基因组结构的比较分析。机械工程学报,2007,44(3):344 - 344。10.3732 / ajb.94.3.302。
- 80.
Kato T, Kaneko T, Sato S, Nakamura Y, Tabata S:豆科植物叶绿体基因组的完整结构,Lotus对虾.DNA res 2000, 7: 323-330。10.1093 / dnares / 7.6.323。
- 81.
Moore MJ, Dhingra A, Soltis PS, Shaw R, Farmerie WG, Folta KM, Soltis DE:快速准确的血管植物质体基因组焦磷酸测序。中国生物医学工程学报,2006,6:17-10.1186/1471-2229-6-17。
- 82.
Shinozaki K, Ohme M, Tanaka M, Wakasugi T, Hayashida N, Matsubayashi T, Zaita N, Chunwongse J, Obokata J, Yamaguchi-Shinozaki K, Ohto C, Torazawa K,孟BY, Sugita M, Deno H, Kamogashira T, Yamada K, Kusuda J, Takaiwa F, Kato A, Tohdoh N, Shimada H, Sugiura M:烟草叶绿体基因组全核苷酸序列:基因组织和表达。中国科学院学报(自然科学版),2011,30(4):344 - 344。
- 83.
Hupfer H, Swaitek M, Hornung S, Herrmann RG, Maier RM, Chiu WL, Sears B:全核苷酸序列月见草补给线质体染色体,代表质体I中的5个可区分的月见草属质体系。中国生物医学工程学报,2006,26(3):366 - 366。
- 84.
金桂杰,李海林:高丽参叶绿体全基因组序列(人参schinsengNees)和17种维管植物序列进化的比较分析。DNA学报,2004,11:247-261。10.1093 / dnares / 11.4.247。
- 85.
奥村S, Sawada M, Park YW, Hayashi T, Shimamura M, Takase H, Tomizawa K:杨树的转化(杨树阿尔巴)质体和外源蛋白在叶绿体中的表达。转基因植物学报,2006,15(5):637-646。10.1007 / s11248 - 006 - 9009 - 3。
确认
本研究由俄罗斯基础研究基金会(项目No06-04-96315 MDL和AAP),支持领先科学学校的计划(项目No140.2008.4 AAP和1275.2008.4 MDL和MK-920.2007.4 AAP。作者非常感谢A.N. Fesenko(全俄罗斯豆类和大植物科学研究所,奥廖尔,俄罗斯)提供的种子Fagopyrum品种,S. V. Kuptsov(莫斯科国立大学植物园,莫斯科,俄罗斯)提供其他蓼科植物材料,N. V. Ravin(中心≪生物工程≫RAS,莫斯科,俄罗斯)共享数据浮萍属小并向A. B. Shipunov(爱达荷州立大学,莫斯科,爱达荷州,美国)征求对初稿的有益意见。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
MDL参与了本研究的设计,设计了通用引物和分类学特异性引物进行扩增和测序,完成了所有PCR反应,参与了contig组装并进行了注释和初稿的编写。THS进行了系统发育分析,并撰写了系统发育部分的手稿。AD提供了倒置重复区扩增的ASAP引物,编辑并参与了稿件的撰写。AAP参与了研究的设计,组装,并开发了图形。所有作者都阅读并批准了最终的手稿。
电子辅助材料
12870 _2008_267_moesm1_esm.doc
附加文件1:开发用于荞麦叶绿体基因组扩增和测序的保守引物。表格(doc 200kb)
12870 _2008_267_moesm2_esm.doc
附加文件2:分类单元特异性引物。包含荞麦专用引物的列表。引物根据其在荞麦叶绿体基因组中的位置命名。例如,对于引物4080F和4621R, 4080和4621分别是正向链和反向链上引物序列的起始点。在IR区退火的引物根据其在IRa和IRb上的位置有双重名称。(doc 172kb)
12870 _2008_267_moesm3_esm.txt
附加文件3:对齐rpl23被子植物和裸子植物的同源物,说明在rpl23石竹目。β而且菠菜有14bp缺失(对齐位置131-145),硅宾在17对位有换人。这种替换(G代替T或C)创建了一个停止密码子TAG。Fagopyrum具有和插入4bp(对准位置49-53)。(txt 18kb)
12870 _2008_267_moesm4_esm.doc
附加文件4:IR扩增PCR检测的细节,包括引物位置和预期扩增子长度。表格(doc 27kb)
12870 _2008_267_moesm5_esm.jpeg
附加文件5:潜在RNA编辑位点的分布rpl2,psbL而且ndhD在被子植物。填充方形表示存在ACG起始密码子,薄方形表示存在典型的ATG起始密码子。蓝色代表rpl2,红色为psbL黑色代表ndhD.问号表示不明确的字符状态(由于基因丢失或缺乏序列数据)。通过对核苷酸数据集的最大简约性分析,推导出系统发育树。(jpeg 844 kb)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
洛加切娃,医学博士,萨米古林,t.h.,丁格拉,A。et al。比较叶绿体基因组学和系统发育Fagopyrum esculentumssp。ancestrale-栽培荞麦的野生祖先。BMC植物生物学8,59(2008)。https://doi.org/10.1186/1471-2229-8-59
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-59
关键字
- 叶绿体基因组
- 质体基因组
- Rps15基因
- 大单份
- 反向重复区