摘要
背景
蔷薇科包括许多不同属的大量具有重要经济价值的二倍体和多倍体果实和观赏种。这些属的基本染色体数目为x= 7、8和9,它们都有紧凑且相对相似的基因组大小。远亲属之间的比较映射已执行到有限的范围内,包括蔷薇科之间的比较马吕斯(马齿苋亚科)和李属(亚Prunoideae);然而,迄今为止还没有发表比较数据马吕斯或李属成为蔷薇亚科的一员在本文中,我们比较了草莓属是蔷薇科的一员李属是普鲁尼亚科的一员。
结果
的二倍体基因组李属(2 n=2 x= 16)和草莓属(2 n=2 x= 14),通过对71个锚定标记(40个限制性片段长度多态性(RFLPs), 29个来自表达序列标记(ESTs)的索引或单核苷酸多态性(SNPs)和两个简单序列重复(SSRs)在这两个属的参考图上的映射进行比较。这些标记提供了良好的覆盖李属(78%)和草莓属(78%)基因组,最大缺口为22 cM,平均密度为7.3 cM/标记李属32厘米和8.0厘米/标记草莓属.
结论
我们的结果表明了一个清晰的同向模式,其中一个物种的每条染色体的大多数标记都映射到另一个物种的一条或两条染色体上。大量的重排(36),其中大部分由反转产生(27),其余(9)由易位或裂变/聚变事件产生。我们为比较这两个具有经济价值且亲缘关系较远的蔷薇科属的基因或DNA序列的位置提供了第一个框架。
背景
根据不同植物类群之间同源标记的图谱位置进行基因组比较,已经确定了植物王国科内物种的基因组,如茄科[1,2], Poaceae [3.],豆科[4]和芸苔科[5]在有限数量的染色体重排上有所不同,这意味着不同物种之间的大量染色体区域,甚至整个染色体都是共线的[6].然而,当在不同科的成员之间进行比较时,共时性的保守性大大降低,这可以通过将几种作物物种的基因组与的基因组进行比较来证明拟南芥[7- - - - - -9].
蔷薇科包括大量二倍体和多倍体物种(3,000种)[10包括重要的作物,例如属于该属的作物李属(杏仁和所有核果:桃、杏、樱桃和李子),马吕斯(苹果),Pyrus(梨),罗莎(玫瑰)悬钩子属植物(覆盆子)和草莓属(草莓)。它们的基本染色体数目是x= 7、8和9,它们都具有紧凑且相对相似的基因组大小,二倍体物种的基因组大小为~170 Mbp in草莓属(2n = 2x = 14) [11~300 Mbp in李属(2n= 2x= 16) [12].大多数多倍体物种的基因组大小与组成它们的二倍体基因组大小大致成正比,即双二倍体苹果(2)n= 2x= 34)的基因组大小约为750 Mbp [12]和栽培的草莓,一种异基因八倍体(2n= 8x= 56),基因组大小约为800 Mbp [11].
在有限的范围内对蔷薇科进行了比较测绘[13].属李属,来自Prunoideae亚科,是第一个被研究的,因为存在高密度参考图[14,15],基于高度多态性的种间(杏仁×桃)F2用可转移到属内其他物种(主要是rflp和SSRs)的标记构建。对16张已出版的地图进行了分析李属物种,每个至少有28个标记的共同参考地图,建立了李属基因组基本上是共线的,迄今为止研究的所有二倍体物种(桃子、杏仁、杏、樱桃、青梅、p . davidiana,p . ferganenesis) [13].苹果和梨属于Maloideae亚科,它们的基因组比较显示出高度的同向性,从这些属的共同标记的研究中无法推断出重大的染色体重排[16,17].目前还没有关于蔷薇亚科(包括草莓、玫瑰和覆盆子)成员间同系性研究的数据发表。然而,属于不同亚科的属的基因组之间有部分的比较:李属(Prunoideae)和马吕斯(Maloideae)。这项工作是用34个标记(23个RFLPs和11个同工酶)完成的,在可能的比较中,检测到高度的同步性,以及至少一次大规模的染色体重排[14].
在这篇论文中,我们比较了李属而且草莓属.为此,我们使用了前面提到的李属参考地图及草莓属参考地图[18],基于种间二倍体f . vesca×f . nubicolaF2人口。的草莓属利用蔷薇科不同种的表达序列标签(EST)序列中存在的生长信息,以ssr和标记为主,通过蔷薇科基因组数据库[19].在这里,我们建立了一个包含两个基因组的共同标记的框架,并发现尽管保存了较大的染色体片段,但正如预期的那样,许多染色体重排分离了二倍体草莓属而且李属基因组,支持从DNA序列数据推断出的它们在家族中的遥远位置[20.].
结果
草莓属而且李属地图
共有71个锚定标记可用于植物二倍体基因组的比较李属而且草莓属.这些是:a)在已映射的65个单拷贝探针中,从相同数量的探针中发现了40个多态rflp李属(表12 .答案b草莓属从135个高度同源的基因中选择得到的ESTs李属的515个ESTs中的李属转录谱图(表2);这些标记是根据它们在地图上的位置选择的,因为它们位于没有被rflp覆盖的区域或被判断为基因组比较感兴趣的区域,c) 8李属从转录本图谱中与已知序列不同源的13个ESTs中选取草莓属est序列(表2), d) 8草莓属从已知功能的基因中获得的STSs草莓属[21)(表2)和e)两个SSRs(表2).
两种映射策略被用于在T×E和FV×FN链接映射上放置新标记(表2)3.).55个标记:全部40个rflp, 8个草莓属sts,五个草莓属显示内含子长度多态性和两个ssr的ESTs通过基因分型对FV×FN群体的所有个体进行定位。其余16个标记使用bin映射方法进行映射,其中一组6个植物(bin集)允许使用二倍体草莓映射群体进行wselective'或'bin'映射[22].在T×E种群中,只有10个新的标记被绘制出来:8个草莓属ESTs被bin映射[15],并用整个种群绘制了这两个ssr。
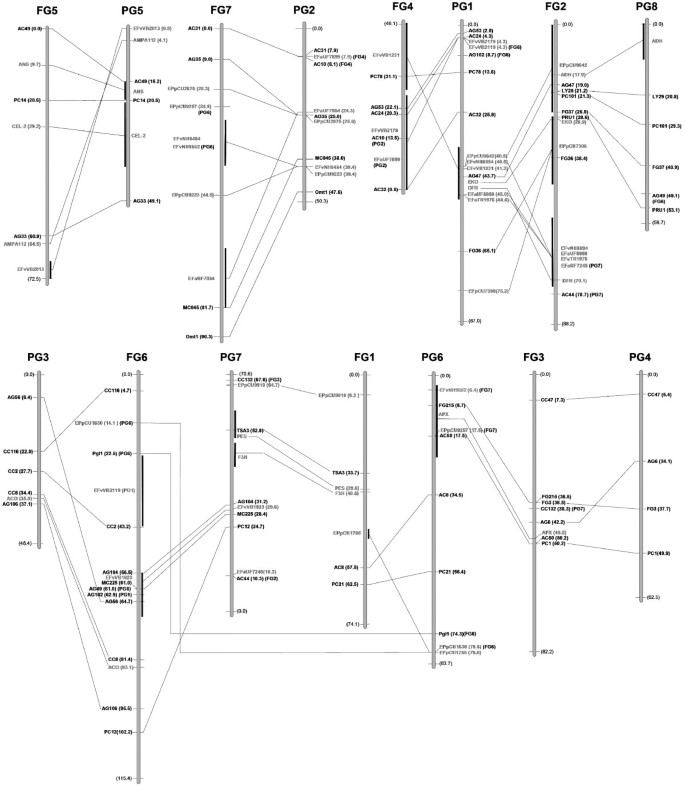
的草莓属使用MapMaker按照材料和方法构建的地图,包含228个标记,其中172个是以前的地图[18],并在整个人群中研究了55个新的标记。萨金特等人在地图上绘制的三个标记。[18], CFVCT028, CFVCT05和UFFxa03B05,无法使用本工作中为映射设置的条件进行定位。检测到7个期望连锁群(FG1-FG7), FV×FN图谱覆盖的总遗传距离为568.8 cM,平均密度为2.5 cM/标记,比值为0.30 Mb/cM。与Sargent等人获得的原始地图相比,得到的地图显示了微小的重排。[18大部分发生在FG2的上部。此地图在附加文件中提供1: 'FV×FN参考地图',包括作为补充信息。的李属地图上共有564个标记,即[14]加上两个SSRs映射在这里。地图覆盖距离为511.3 cM,平均密度为0.91 cM/标记,比值为0.59 Mb/cM。
在草莓基因组的7个连锁群中,71个锚定标记的分布相对均匀1),在FG1和FG5上有7个标记,在FG2上有16个标记,平均标记密度在FG1上为10.6 cM/标记,在FG2上为5.5 cM/标记。这些标记所覆盖的地图距离为441.3 cM,占所有标记所构建地图的78%。>只有3个30 cM的大间隙,一个在FG3末端(32.0 cM),一个在FG2中间(31.7 cM),最长的在FG7下部(37.2 cM)。这71个标记的分布李属图谱相似,PG1上最多有17个标记,PG4上最少有4个标记,每个连锁群的标记密度为15.6 (PG4) ~ 5.0 (PG2) cM/标记。锚点标记的地图覆盖率为399.5 cM,占完整地图的78%。最长的标记之间的间隙较小李属超过20cm的只有4个:PG7为21.7个;PG6为21.9,PG8为20.8,PG5为28.6。所有的缝隙都在草莓属而且李属除了PG6和FG3上的极端标记外,其他地图上至少有一个标记在缺口的极端标记之间,这表明这是每张地图上最长的缺口。
地图对比李属而且草莓属
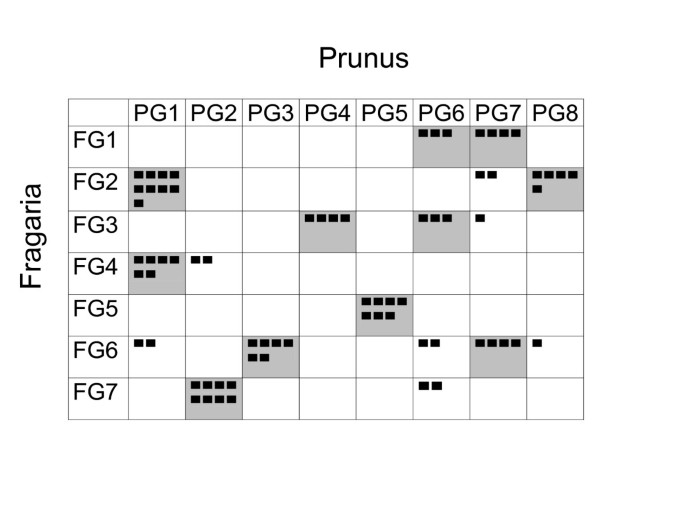
图中总结了共时性的总体模式2,可以看出,两个物种的大多数连锁群在另一个物种的一个或两个连锁群中标记最多。最简单的例子是两个物种的G5,它们的所有标记都是相同的,而最复杂的例子是FG6,它包含了五个不同的标记李属虽然该连锁群上的大多数标记属于PG3和PG7。只考虑李属链接组,其中5个只包含其中一个组的大部分或全部标记草莓属(PG2-FG7, pg2 -FG6, PG4-FG3, PG5-FG5和PG8-FG2),以及两组中的其他三组(PG1-FG2和FG4, PG6-FG1和FG3, PG7-FG1和FG6)。
如果一个物种的两个或多个连锁群在另一个物种的连锁群中存在标记,则表明自两个物种从共同祖先分化以来,它们之间发生了裂变/融合或易位事件。这些重新安排中有9个发生在草莓属和李属基因组,基于我们的研究结果。
的共线区域内标记的共线性李属而且草莓属只是部分的。最共线的两个组FG5和PG5只需要一个反转事件就可以将所有标记以相同的顺序放置,但在其他链接组中需要更多。如材料和方法部分所示,FG1上的标记需要两次反转才能与PG7和PG6上的标记顺序相同李属FG3和FG4为三次逆转录,FG7为四次逆转录,FG6为六次逆转录,FG2为八次逆转录。总的来说,至少需要27个反转来解释两个基因组的标记顺序差异。控件分隔的断点总数的估计下界草莓属而且李属因此基因组是36个(9个易位和27个倒置)。
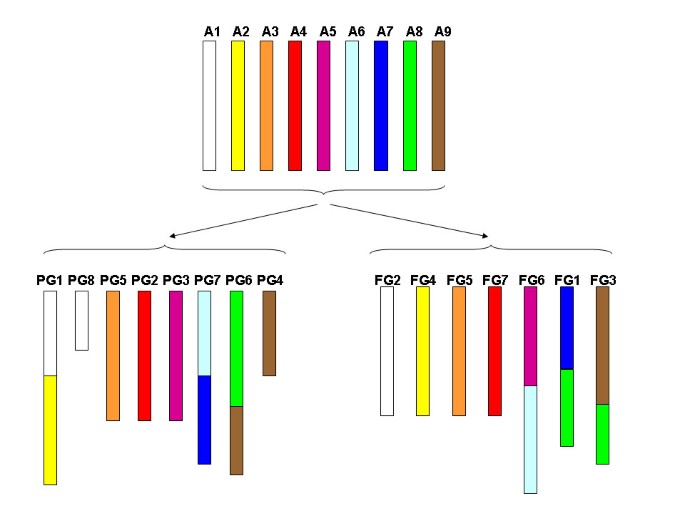
一种可能的演化方案草莓属而且李属从一个假设的祖先基因组x= 9 [23]如图所示3..在这个方案中,我们只考虑了主要的染色体重排,即融合/裂变和相互易位,涉及用于比较的两个以上的标记。在这种情况下,祖先基因组经历了两次融合(分别对应于FG6和FG1的A5/A6和A7/A8),以及FG1和A9中部分A8染色体的相互易位,从而产生FG3,成为FG1x= 7个草莓基因组。为李属x= 8个基因组,祖先基因组进行了3次融合,A1/A2形成PG1, A6/A7形成PG7, A8/A9形成PG6, 2次分裂,A1的部分在A1/A2染色体上形成PG8, A9的部分在A8/A9染色体上形成PG4。
关于某些染色体进化的一些推论可以基于两种图谱之间的比较来制定(图1),这两个基因组都是从一个祖先的基因组进化而来的(图3.),以及蔷薇科的细胞遗传学和图谱比较资料有限。最简单的比较是FG5和PG5,它们显示出显著的结构守恒水平,两者之间的标记顺序李属而且草莓属只差一个反转事件。FG7和PG2的大部分标记是相同的,表明它们起源于单一的祖先染色体(A4),但FG7包含PG6的短片段,PG2不完全包含在FG7中,有一小部分位于FG4上。PG1是一个较长的连锁基团,比其他基团包含更多的标记,很可能与染色体1重合李属[14],明显比该属的其他染色体长[24].根据核型观察,这样长的染色体不存在于马吕斯[25]或in草莓属[26].地图对比马吕斯而且李属[14)提供了额外的证据,证明长李属在双二倍体的至少一个组成基因组中,染色体可以分裂成两个马吕斯染色体。我们的数据表明,PG1源于两条祖先染色体A1和A2的融合,它们分别对应于草莓中的FG4和FG2。PG1中发生裂变/聚变的区域李属-苹果比较(距离PG1顶部38-44 cM)也与李属-草莓属(估计在距离PG1顶部25-45厘米的区域)。有趣的是,大多数的PG8,其中之一李属具有最少数量标记和最小遗传距离的染色体似乎整合到FG2中,在PG8和FG2中,研究的6个锚定标记中的5个顺序相同。这种情况可能是A1在裂变的结果李属但不是在草莓属.
讨论
的地图之间的比较李属和二倍体草莓属已经用71种常见标记进行了检查。大部分都已经在地图上了李属地图和被添加到草莓地图这里。这些标记对两个基因组都有很好的覆盖:两者参考图总距离的78%李属[14]和草莓[18].平均锚标记密度为7.3 cM/标记,最大间隙为22 cM (PG6)李属和8.0 cM/标记,最大间隙为32 cM (FG3)草莓属.的总地图距离李属(519厘米)和草莓属(569 cM)的地图使用所有可用标记构建时相似[14,18].考虑到基因组的大小李属大约是草莓的两倍,这些结果表明,每物理单位距离的整体重组率为草莓属FV×FN杂交种高于杏仁×桃F1个人,产生了T×E F2人口。
为了比较两个基因组,我们选择了65个RFLP探针,全部来自李属或马吕斯种,杂交效果好,均为单拷贝李属基因组。以草莓为样品,在相同的严格条件下对探针进行了研究李属16个(24%)产生的杂交效果差或没有杂交,这表明它们不存在于草莓基因组中,或者它们的序列与草莓基因组有很大差异李属.一些没有杂交的探针位于同一区域李属其中覆盖较短遗传距离(每个区域<10 cM)的几个探针没有杂交的基因组,如PG1的中心区域和PG6的上端(表2)1).未在PG1区杂交的3个探针分别来自桃cDNA文库(PC85和PC15)和桃基因组文库(PC85和PC15)p . ferganensis(FG16)。其中2个(PC15和FG16)进行了测序1),并与已知蛋白质具有同源性。PG6区域有4个标记(AG13、AG54、AG40和PLG59),均来自李属基因组库。只有AG40被测序,其序列与蛋白序列无同源性。这些结果表明,草莓基因组中相应的区域,或者至少是测试的特定序列可能被删除。鉴于其中一些探针与通常以基因家族形式存在的蛋白质(如聚半乳糖醛酸酶(FG16)和防御素蛋白1 (PC15))同源,另一种解释是它们可能对应于这些与草莓中存在高度序列差异的基因的副本。
在40个杂交良好且多态的RFLP探针中,有14个(35%)在草莓中检测到2个位点李属均为单位点(表1).Tanksley等人[27他们在辣椒中绘制的42个番茄cDNA探针中,有5个(12%)在每个物种中都有不同的拷贝数。这些拷贝数的差异可能是由于存在于祖先基因组中的复制DNA片段的差异缺失造成的李属而且草莓属产生。这些重复的存在使基因组比较复杂化,因为如果两者只有一个位点可以被绘制,就像在这里绘制的RFLPs草莓属在一半的病例中,标记的位置可以解释为存在虚假的遗传重排。
用蔷薇科探针检测到的单拷贝RFLPs (T×E)数量在草莓群体中分离,沿种群分布良好李属地图,是不足以很好的覆盖的李属基因组。这部分是由于大约一半的探针用于RFLP映射李属拥有多于一份副本[28,29]并且T×E中使用的大量探针(30%)来自于蔷薇科以外的科[14].为了解决这个问题,我们使用了可用的EST和物理地图信息李属在大多数未被发现的区域找到ESTs。然后我们用一种高效而昂贵的方法来映射这些est,重新排序,使用一种廉价的映射策略,bin映射。这使我们能够覆盖大部分的缺口李属并将该物种无锚定标记的最大间隔缩短至22 cM。鉴于信息的快速增长速度李属转录本图,这一策略很可能让我们在不久的将来更详细地分析同音。
数字2说明了水平之间的同步性李属而且草莓属是高的,大多数标记映射到一个物种的同一个连锁群映射到另一个物种的一个或两个连锁群。然而,共线性只是部分的,估计有9个染色体重排涉及两条染色体(易位或融合/裂变事件)和27个倒置。易位和聚变/裂变事件的数量是高的,如果我们把它与其他同类比较,其中这个数字已经估计。只有辣椒和番茄的遥远的茄科基因组,估计有10个这样的重排[30.],得出了类似的结果。然而,倒位的优势超过其他重排,我们发现在李属-草莓属基因组比较在植物王国中很常见,例如西红柿和土豆之间的比较[31]、番茄和茄子[2]、番茄及胡椒[30.),芸苔属植物黑质而且拟南芥[32或a。芥而且答:lyrata[33].
着丝粒或着丝粒周围的异色区域通常是断点发生的地方,导致染色体重排。茄科植物的大部分倒置和易位[30.,34]及Poaceae [35]在着丝粒或着丝粒附近有断点。这一信息可能有助于推断某些基因的着丝粒的位置李属或草莓属染色体。例如,根据我们假设祖先染色体A1和A2之间发生融合事件形成PG1的位置,我们可以推断出着丝粒位于该染色体的中心部分(距顶部30-45 cM)。这与桃1号染色体的稳心特性一致。舒伯特(36]提出了一个染色体融合的模型,在一个端心染色体或端心染色体与另一个染色体之间的相互易位可能产生一个更大的融合染色体和一个最终丢失的小染色体。按照这个模型,FG2、FG4或两者中的一个,或它们的祖先染色体(A1和A2),可能是末端中心的。其他稳心染色体可能是由其他物种的两条染色体的部分(可能是整个易位臂)组成的;这可能是PG7、PG6、FG1、FG6和FG3的情况(见图)3.).在PG6的情况下,先前在桃中报道过与PG8相互易位[37].该断点估计位于PG6顶部18-39 cM处,与FG1和FG3片段的连接区域(18-35 cM)重合,可能对应于这三条染色体的着丝点区域。另一方面,PG8的易位断点[37]位于其远端区域,这表明它是一个端心染色体。
估计每条染色体每百万年(My)的结构突变率为0.14(±0.06)个[6]从植物中各种大共时性比较的分析中得出。考虑到这个速率,我们估计至少有36个染色体重排之间的数目李属而且草莓属,假设共同祖先的初始染色体数量为x= 9,使我们可以估计这两个物种的差异大约从29亿年开始。这将这两个属作为蔷薇科中遥远的类群,类似于Poaceae中的玉米和高粱(~24 Mya),但比茄科中的番茄和辣椒(~40 Mya),玉米和水稻或小麦和水稻更接近,两者的分化时间都估计为~66 Mya [6].
我们的结果表明,在基因组之间有足够的同步性草莓属而且李属将其中一个物种的标记、基因或数量性状位点(QTL)位置信息用于另一个物种。例如,决定草莓产生传代体(营养繁殖体)能力的基因R/r)位于FG2上,与PG1上Evergrowing基因(Evg/evg),决定叶片的连续生产[38]和一个决定桃花期的QTL位于[39].草莓季节性开花与永久开花的基因(年代/年代)映射到FG6的一个区域(靠近SSR EMFn017),在我们的比较中,该区域与决定桃花期的一个主要QTL所在的PG7片段大致吻合[39].然而,这些比较是初步的,需要更详细的研究,但这是对其他可能的比较的第一次洞察,可能促进对蔷薇科关键性状的遗传知识的发展。
方法
植物材料
以两个作图群体的亲本和子代为比较对象李属而且草莓属基因组,即F2种间李属参考映射总体(N = 82)由F1杏仁之间的交叉(p . dulcis)品种“德克萨斯”和桃子(p . persica)品种“Earlygold”(缩写为T×E)和F2种间二倍体草莓属参考映射总体(N = 76)由F1交叉的f . vesca815×f . nubicola601(缩写为FV×FN)。T×E图谱由562个标记组成(185个SSRs, 361个rflp, 11个同工酶和5个STSs) [14],而FV×FN图谱目前由182个标记组成(175个SSRs, 6个基因特异性标记和1个SCAR) [18].使用的大多数标记都是从T×E中选择的,随后映射到FV×FN中。
DNA提取
为草莓属,每个个体收集1克年轻的膨胀叶,在-80℃保存,然后进行DNA分离。采用Doyle和Doyle的CTAB方法从叶片样品中分离基因组DNA [42],其次是DNeasy miniprep试剂盒纯化(Qiagen)。使用Gene Quant II分光光度计(Pharmacia Biotech)测量DNA浓度。为李属, DNA按[28].
RFLP标记的
共65个探头来自各种李属RFLP分析(表1).这些探针是从用于建造的李属参考地图[14]是单拷贝的,覆盖整个基因组的距离大约为10-25厘米。采用Viruel等方法进行RFLP分析[28].DNA探针首先在FV×FN种群的亲本中进行杂交,随后在所有F2个人。
基于草莓属或李属est序列
另外一组标记是由李属转录本图谱,即与参考连锁图谱的已映射标记(通常为RFLPs)位于桃物理图谱相同BAC或相同BAC contig上的EST unigenes集合[43].我们分析了515个T×E ESTs目前锚定在李属转录谱图(在蔷薇科基因组数据库中找到)中找到同源EST序列草莓属使用TBLASTX程序[44].所选序列(仅为e值<1.00E-15的序列)2),然后用TBLASTN进行分析,选择同源性最强的区域进行引物设计。引物的设计包括假定的草莓属基于拟南芥基因组序列的内含子[45],并给出预期扩增子大小>800 bp,使用程序Primer3 [46].
对于SNP检测,草莓属在父母中pcr扩增est草莓属在温度梯度PCR中映射群体,以获得每个EST引物对的最佳退火温度。PCR反应按[47],扩增产物用ABI PRISM 3700 DNA分析仪进行测序,并用STADEN包进行序列比对,检测两个亲本系之间的snp [48]并通过肉眼检查DNA色谱图来验证。同样的程序被用于李属我们没有找到同源的转录本图谱的ESTs草莓属est序列。
用于这些标记开发的引物序列见表4.EST衍生标记的术语为:E (EST),一个两个字母的代码,表示获得EST的物种(Fv, Fa或Pp表示f . vesca,F. x安纳萨而且p . persica),另外两个字母表示获取EST的地点(即NH代表新罕布什尔大学,VB代表弗吉尼亚生物信息学研究所,UF代表佛罗里达大学,TR代表Trisaia研究中心,CU代表克莱姆森大学),以及与EST EMBL登录号的后四位数字对应的四个数字。
草莓属gene-specific标记
另外8个先前在FV×FN种群中绘制的基因特异性位点[21]在T×E人群中进行了定位。用于扩增其中四种基因(CEL-2、PES、ANS和APX)的引物对先前已由Sargent等人描述。[21]而其余四种(EKO、F3H、DFR和ADH)的引物对则按照Sargent等人的程序设计。[21]来自EMBL数据库的基因序列,列于表中4.引物对用于扩增该基因亲本的产物李属按照Sargent等人的程序绘制人口。[47]和snp的检测结果如上所述。
微卫星标记
在5' UTR中发现了1个SSR (ACO)李属1-氨基环丙烷-1-羧酸盐氧化酶基因;AF129073)用于定位该基因,该基因已经被定位草莓属[21],浏览T×E地图。所用引物列于表中4.另一种杏SSR, AMPA112,先前由Hagen等人描述。[49],在两个参考种群中都是多态的,也被包括在地图比较中。PCR反应由[50].
地图构建草莓属bin映射李属而且草莓属
Dirlewanger等人的T×E地图[14],使用MapMaker/EXP v. 3.0构建[51],被用作地图比较的标准。FV×FN地图的数据,最初使用Joinmap 3.0构建[52]萨金特等人[18],用于新位点的定位草莓属.但是,为了使两张地图具有可比性,使用了FV×FN地图的相同数据加上这里获得的数据,但地图是用MapMaker重新构建的。利用Kosambi映射函数将重组单元转换为遗传距离。映射过程遵循在中构造的先前映射的指导原则李属[29].的八个连杆群的常用符号李属是G1到G8,是7人草莓属组是I-VII。为了便于地图比较,我们在这里使用了术语PG1-PG8李属和FG1-FG7草莓属.
T×E映射种群的六个植物的子集,具有大量的重组断点和在李属基因组由Howad等人选择。[15].这组植物的基因型(bin set)鉴定了64个片段(bin)李属地图平均大小为7.8厘米。遵循类似的方法,Sargent等人从FV×FN映射种群中选择了六种植物的bin集。[22].的草莓属bin组检测到46个平均长度为12.6 cM的基因组片段。使用这两个物种的bin集,我们可以用更低的成本和努力来确定一些标记的位置,而不是用整个种群来绘制。
映射策略
表格3.总结了本研究中所有新标记所采用的定位策略。所有使用的rflp都已映射李属使用整个T×E后代,并被映射到草莓属使用整个FV×FN种群。的草莓属而且李属ESTs在亲本中具有多态性草莓属测图人群或测序草莓属bin集和bin-mapped,或在整个FV×FN种群中进行映射,当检测到明显的内含子长度多态性时。八草莓属以前在FV×FN中映射的ESTs被bin映射到李属.SSRs被映射进去李属而且草莓属使用这些种群中的所有个体。每个指标的数据都由两名研究人员独立评分。相互矛盾的结果被重新审查,如果有分歧,采取了最保守的选择。
估计染色体重排的数量
来估计染色体重排发生在李属而且草莓属由于他们来自一个共同的祖先,我们详细阐述了每个标记的列表草莓属连杆组与其在上的相关位置李属连锁图。以FG1为例,该链接群有7个锚点标记,顺序分别为7.2 (PG7,位置2)、7.3、7.5、7.4、6.10、6.6、6.7。然后,我们推断出将这些标记的位置按相同顺序排列的最小突变数李属.在FG1的例子中,我们计算了一个易位(PG7和PG6之间)和两个反转,一个涉及7.4 - 7.5片段,另一个涉及6.6 - 6.7片段,从而给出最终的标记顺序:7.2、7.3、7.4、7.5、6.10、6.7和6.6。另外一个突变(可能是由易位引起的)必须发生才能解释6.10和6.7之间的差距。在分析接受易位片段的染色体时,考虑了这种突变。我们为每个易位计算一个断点,为每个反转计算一个断点。这是一个较低的边界,因为如果涉及染色体的远端部分,则倒置可能需要一个断点,如果对应于染色体的内部片段,则需要两个断点。最后,我们仅将包含两个或两个以上标记的区域视为易位,因为单位点易位更有可能是假的。
参考文献
Tanksley SD, Ganal MW, Prince JP, De Vicente MC, Bonierbale MW,等:番茄和马铃薯基因组的高密度分子连锁图谱。遗传学杂志,1992,132:1141-1160。
Doganlar S, Frary A, Daunay MC, Lester RN, Tanksley SD:茄子遗传连锁图谱比较(茄属植物melongena)及其对茄科基因组进化的意义。中国生物医学工程学报,2002,29(3):344 - 344。
Devos KM, Gale MD:基因组关系:当前研究中的草模型。中国生物医学工程学报,2000,29(4):344 - 344。10.1105 / tpc.12.5.637。
崔洪红,文建辉,金金杰,朱海燕,白建明,Mudge J, Roe B, Ellis N, Doyle J, Kiss GB, Young ND, Cook DR:作物和模式豆科植物基因组保护的估算。美国国家科学研究院。2004, 30(4): 457 - 457。10.1073 / pnas.0402251101。
Schranz ME, Song BH, Windsor AJ, Mitchell-Olds T:十字花科的比较基因组学:全科视角。植物学报,2007,10:168-175。10.1016 / j.pbi.2007.01.014。
张春华,林玉玲,张志刚,等:超越单双子系差异的高等植物遗传图谱的构建。自然科学学报,1996,14:366 - 366。10.1038 / ng1296 - 380。
Salse J, Piégu B, Cooke R, Delseny M: Synteny between拟南芥基因组水平上的水稻:在正在进行的水稻基因组测序项目中,这是一种识别保护的工具。中国生物医学工程学报,2002,30:2316-2328。10.1093 / nar / 30.11.2316。
Dominguez I, Graziano E, Gebhardt C, Barakat A, Berry S, Arús P, Delseny M, Barnes S:植物基因组考古:双子叶植物物种中保存的祖先染色体片段的证据。中国生物工程学报,2003,1:91-99。10.1046 / j.1467-7652.2003.00009.x。
荣杰,鲍ers JE, Schulze SR, Waghmare VN, Rogers CJ,等:比较基因组学Gossypium而且拟南芥:揭示古代和现代多倍体的后果。中国生物工程学报,2005,30(4):344 - 344。10.1101 / gr.3907305。
Judd WS, Campbell CS, Kellogg EA, Stevens PF:植物系统学,系统发育方法。桑德兰M.A. Sinauer Associates;1999.
Akiyama Y, Yamamoto Y, Ohmido N, Oshima M, Fukui K:草莓核DNA含量的估算(草莓属采用双系统流式细胞术与拟南芥进行比较。中国生物医学工程学报,2001,26(3):344 - 344。
Arumuganathan K, Earle ED:一些重要植物物种的核DNA含量。植物分子生物学学报,1999,9:888 - 888。10.1007 / BF02672069。
Arús P, Yamamoto T, Dirlewanger E, Abbott AG:蔷薇科的Synteny。《植物育种评论》。编辑:Janick J. 2005,伦敦,John Wiley and Sons Inc, 175-211。
Dirlewanger E, Graziano E, Joobeur T, Garriga-Caldere F, Cosson P, Howad W, Arus P:蔷薇科水果作物的比较定位和标记辅助选择。美国国家科学研究院。2004, 30(4): 344 - 344。10.1073 / pnas.0307937101。
Howad W, Yamamoto T, Dirlewanger E, Testolin R, Cosson P, Cipriani G, Monforte AJ, Georgi L, Abbott AG, Arus P:利用少数植物制图:微卫星饱和度的选择性制图李属参考地图。中国生物医学工程学报,2005,27(3):357 - 357。10.1534 / genetics.105.043661。
Yamamoto T, Kimura T, Saito T, Kotobuki K, Matsuta N, Liebhard R, Gessler C, Weg van de WE, Hayashi T:日本和欧洲梨的遗传连锁图谱与苹果共识图谱一致。植物学报,2004,663:51-56。
Pierantoni L, Cho KH, Shin IS, Chidoni R, Tartarini S, Dondini L, Kang SJ, Sansavini S:苹果ssr对两种欧洲梨F的表征和转移性1人群。应用理论与实践,2004,29(4):419 - 424。10.1007 / s00122 - 004 - 1775 - 9。
Sargent DJ, Clarke J, Simpson DW, Tobutt KR, Arus P, Monfort A, Vilanova S, denoes - rothan B, Rousseau M, Folta KM, Bassil NV, Battey NH:二倍体的增强微卫星图草莓属.应用理论与实践,2006,29(3):349- 359。10.1007 / s00122 - 006 - 0237 - y。
Jung S, Jesudurai C, Staton M, Du ZD, Ficklin S, Cho IH, Abbott A, Tomkins J, Main D: GDR(蔷薇科基因组数据库):蔷薇科基因组学和遗传学研究的综合网络资源。中国生物医学工程学报,2004,30(4):344 - 344。[http://www.bioinfo.wsu.edu/gdr/]
Potter D, Eriksson T, Evans RC, Oh S, Smedmark JEE, Morgan DR, Kerr M, Robertson KR, Arsenault M, Dickinson TA, Campbell CS:蔷薇科植物的系统发育与分类。植物学报,2007,26(3):344 - 344。10.1007 / s00606 - 007 - 0539 - 9。
Sargent DJ, Rijs A, Nier S, Simpson DW, Tobutt KR:功能标记的开发和定位草莓属以及它们在其他属中的转移性和映射潜力。应用理论与实践,2007,29(3):344 - 344。10.1007 / s00122 - 006 - 0441 - 9。
Sargent DJ, Cipriani G, Vilanova S, Gil-Ariza D, Arús P, Simpson DW, Tobutt KR, Monfort A:二倍体103个标记的bin映射群体的建立和选择性映射草莓属参考映射人口FV×FN。中国生物工程学报,2008,51:120-127。10.1139 / g07 - 107。
Morgan DR, Soltis DE, Robertson KR:研究的系统性和进化意义加拿大皇家银行蔷薇科的L序列变异。中国生物医学工程学报,2004,29(4):489 - 497。10.2307 / 2445770。
萨莱斯G,莫拉斯A:试探性的原生质体与染色体的结合李属.植物学报,1997,27:363-368。
杨晓明,李志刚,李志刚:苹果单倍体核型分析(马吕斯有明显).中国园艺学报,2000,21(4):329 - 329。
Iwatsubo Y, Naruhashi N:三种植物的核型草莓属(蔷薇科)。中国生物医学工程学报,2004,29(4):489 -497。
Tanksley SD, Bernatzky R, Lapitan NL, Prince JP:辣椒和番茄基因序列的保守性和序性。美国国家科学研究院。1988, 85: 6419-6423。10.1073 / pnas.85.17.6419。
马丽娟,王晓明,王晓明,等:杏仁基因RFLP和同工酶标记的多态性分析。应用理论,1995,19(3):366 - 366。10.1007 / BF00223907。
Joobeur T, Viruel MA, de Vicente MC, Jauregui B, Ballester J, Dettori MT, Verde I, Truco MJ, Messeguer R, Batlle I, Quarta R, Dirlewanger E, Arus P:饱和连杆图的构造李属使用杏仁×桃子F2后代。应用理论,1998,37(4):434 - 441。10.1007 / s001220050988。
Livingstone K, Lackney VK, Blauth JR, Wijk RV, Jahn MK:基因组图谱辣椒以及茄科植物基因组结构的演化。遗传学杂志,1999,152:1183-1202。
Bonierbale MW, Plaisted RL, Tanksley SD:基于一组普通克隆的RFLP图谱揭示了马铃薯和番茄的染色体进化模式。遗传学报,1988,30(4):344 - 344。
拉格克兰茨U:比较映射之间拟南芥而且芸苔属植物黑质表明芸苔属植物基因组的进化是通过广泛的基因组复制伴随着染色体融合和频繁的重排。遗传学报,1998,15(3):344 - 344。
Yogeeswaran K, Frary A, York TL, Amenta A, Lesser AH, Nasrallah JB, Tanksley SD, Nasrallah ME:比较基因组分析拟南芥在进化历史中推断染色体重排事件答:芥.基因组学报,2006,15:505-515。10.1101 / gr.3436305。
Paterson AH, Bowers JE, Burow MD, Draye X, Elsik CG, Jiang CX, Katsar CS, Lan TH, Lin YR, Ming RG, Wright RJ:植物染色体比较基因组学。中国生物医学工程学报,2000,29(3):344 - 344。10.1105 / tpc.12.9.1523。
杨晓明,杨晓明,杨晓明,杨晓明。着丝粒位点与谷粒染色体进化的关系。中国生物医学工程学报,1997,27(3):344 - 344。
舒伯特I:染色体进化。中国植物学报,2007,30(3):357 - 357。10.1016 / j.pbi.2007.01.001。
Jáuregui B, de Vicente MC, Messeguer R, Felipe A, Bonnet A, Salesses G, Arus P: 'Garfi'杏仁和'Nemared'桃子之间的相互易位。应用理论,2001,32(3):379 - 379。10.1007 / s001220000511。
Bielenberg DG, Wang Y, Fan S, Reighard GL, Scorza R, Abbott AG:在长生桃突变体中存在影响几个候选基因的缺失。王志强,2004,30(3):344 - 344。10.1093 / jhered / esh057。
Silva C, Garcia-Mas J, Sanchez AM, Arús P, Oliveira MM:杏树花期研究(李属dulcis(Mill) D.A. Webb):候选基因方法。应用物理学报,2005,30(3):357 - 357。10.1007 / s00122 - 004 - 1918 - z。
莫奈R, Guye A, Roy M, Dachary N:桃孟德尔遗传学:一个简短的回顾和新结果。农学通报,1996,16:321-329。10.1051 /农业:19960505。
Folta KM, Davis TM:草莓基因和基因组学。植物科学进展,2006,26(3):349 - 349。10.1080 / 07352680600824831。
Doyle JJ, Doyle JL:一种用于少量新鲜叶片组织的快速DNA分离程序。植物化学通报,1987,19:11-15。
Horn R, Lecouls AC, Callahan A, Dandekar A, Garay L, McCord P, Howad W, Chan H, Verde I, Main D, Jung S, Georgi L, Forrest S, Mook J, Zhebentyayeva T, Yu YS, Kim HR, Jesudurai C, Sosinski B, Arus P, Baird V, Parfitt D, Reighard G, Scorza R, Tomkins J, Wing R, Abbott AG:果树模式种桃的候选基因数据库和转录谱图。应用理论与实践,2005,30(1):1 - 4。10.1007 / s00122 - 005 - 1968 - x。
Atschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ: gapping BLAST和ppi -BLAST:新一代蛋白质数据库搜索程序。中国生物医学工程学报,1997,25:389-3402。10.1093 / nar / 25.17.3389。[http://www.ncbi.nlm.nih.gov/BLAST/Blast.cgi]
拟南芥基因组序列。[http://arabidopsis.org]
Primer3。[http://frodo.wi.mit.edu/cgi-bin/primer3/primer3_www.cgi]
Sargent DJ, Hadonou AM, Simpson DW:多态微卫星标记的发展和特征草莓属冬青这是一种野生二倍体草莓。生物化学学报,2003,3:555 -552。10.1046 / j.1471-8286.2003.00507.x。
Staden R: Staden序列分析包。中国生物工程学报,1996,15(3):344 - 344。10.1007 / BF02900361。
张志刚,张志刚,张志刚,张志刚,张志刚,陈志刚,陈志刚,陈志刚,陈志刚,陈志刚:植物cDNA微卫星的研究进展(李属armeniacal .)。生物化学学报,2004,4:742-745。10.1111 / j.1471-8286.2004.00802.x。
P:一套来自野生草莓的多态SSR标记(草莓属vesca)可转移到其他二倍体草莓属物种和Fragaria x ananassa.化学通报,2006,6:197-200。10.1111 / j.1471-8286.2005.01191.x。
李丽娟,李丽娟,李丽娟,等:MAPMAKER:一种用于构建实验群体和自然群体遗传连锁图谱的交互式计算机包。基因组学杂志,1987,1:174-181。10.1016 / 0888 - 7543(87) 90010 - 3。
Van Ooijen JW, Voorrips RE: Joinmap 3.0:遗传连锁图计算软件。荷兰瓦赫宁根国际植物研究中心;2001.
确认
这项研究得到了西班牙教育部项目INIA (RTA2007-00063-00-00)的部分资金支持。东马林的蔷薇基因组学由Defra赞助。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
AM和PA构想并设计了实验。SV用RFLPs进行了实验草莓属在绘制种群的过程中,dj们进行了绘制基因特异性标记的实验工作草莓属而且李属bin绘制种群,AM开展了EST的实验工作草莓属人口。PA协调所有作者投稿的手稿准备工作,并阅读并批准最终手稿。
电子辅助材料
12870 _2007_275_moesm1_esm.pdf
附加文件1:FV×FN参考地图.的二倍体草莓属使用MapMaker根据Sargent et al.(2006)的数据和本文添加的新标记(黑体和红色)构建参考地图。(PDF)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
维拉诺瓦,S,萨金特,d.j., Arús, P。et al。两个远亲蔷薇科基因组的同步性守恒:李属(核果)和草莓属(草莓)。BMC植物生物学8,67(2008)。https://doi.org/10.1186/1471-2229-8-67
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-67
关键字
- 连锁群
- 祖先基因组
- 锚标记
- 祖先染色体
- 内含子长度多态性