摘要
背景
新近发展的多果型黑莓(悬钩子属植物L.)栽培品种,结合形态标记辅助黑莓育种家对无刺进行选择的悠久历史,引起了人们对使用分子标记促进黑莓育种的兴趣。然而,没有专门针对栽培黑莓的基因图谱、分子标记,甚至是序列。本研究的目的是通过生成和注释第一个黑莓表达序列标签(EST)库,从ESTs中设计引物来扩增包含简单序列重复序列(SSR)的区域,并在两个黑莓品种中测试EST- ssrs子集的有效性,从而开始这些工具的开发。
结果
从许多无刺商业品种的祖先Merton thorless的扩张叶组织中获得了18,432个克隆的cDNA文库。在注释的3000个基因中,表达最多的是那些与能量、细胞结构和防御有关的基因。从含有SSRs的单个序列中,设计了673对引物。在随机选取的33对引物中,10对引物平均检测到1.9个多态PCR产物。
结论
这一比例预测该文库可能产生多达940个SSR引物对,检测1786个多态性。这可能足以生成一个遗传图谱,可用于将分子标记与表型性状相关联,使分子标记辅助育种成为可能,以补充现有的形态标记辅助育种。
背景
最近发布的两款黑莓(悬钩子属植物L.)具有新性状的品种,primocane结果[1),有可能大幅扩张黑莓行业。所有其他的黑莓品种都在夏天在一种叫做floricanes的藤条上结果,这种藤条在前一年生长。初果类果实栽培品种在花藤上生产果实,然后在夏末和初秋在春季出现的只有几个月大的藤上生产较小的第二次作物,从而延长了种植者、营销人员和消费者的潜在果实生产周期。或者,甘蔗可以在晚秋割到地上,全年的作物都可以在第二年春天出现的甘蔗上生产。由于藤条能够避免冬季的伤害,这种类型的黑莓生产可以扩展到以前被认为太冷不适合种植黑莓的地区。在季节和地理上扩大黑莓生产对行业的潜在影响,导致人们希望开发新的品种,将primocane结果与其他重要特性(如无刺)结合起来。
使primocane结果子的等位基因是隐性的[2],因此一个四倍体四体黑莓品种需要四个拷贝才能在原果上产生果实。该性状的表现受环境和植物活力的影响,因此异花授粉的后代在第一个甚至第二个结果年都不能从视觉上识别出来。如果每年对数千个后代进行评估,将有助于尽早消除不受欢迎的基因型。因此,primocane结果将是一个很好的标记辅助选择的候选者。利用基因座周围的多个标记,黑莓育种家可以识别出应该是primocane结果子或在杂合状态下携带该性状的后代。
迄今为止,人们对栽培黑莓分子标记连锁图谱的开发一直缺乏积极的兴趣,也没有发表过黑莓连锁图谱。然而,尽管其高度杂合的四倍体基因组,黑莓是发展标记辅助选择的一个很好的候选者。事实上,黑莓育种者已经在使用类似的方法来挑选对该行业来说另一个非常重要的特征——无刺的幼苗。像primocane结果子一样,无刺的遗传是通过单个位点上的隐性等位基因[3.].无刺选择可在苗期根据子叶边缘毛的缺失进行。这两个特征被认为是紧密相连的,而不是等位基因在单个位点上的上位效应,因为极少数没有子叶毛的幼苗后来会产生刺(J.R. Ballington,个人交流)。因此,一种形态标记辅助选择已经被黑莓育种家使用了很多年,这表明分子标记辅助选择在像primocane结果这样的性状上有被采用的潜力。
利用连锁作图策略,如单剂量限制性片段作图方法,可以建立一个分子标记与primocane结果或任何其他性状的连锁[4]或使用专为四体体物种设计的软件,如Tetraploid Map [5].由于双重还原,来自多个种群的饱和黑莓图谱将是染色体臂的图谱,而不是整个染色体,但与重要性状的有用标记联系仍然是很有可能的。
来自黑莓序列的分子连锁图和分子标记都不存在或不可用。低比例的SSR引物是从草莓(草莓属L.)放大了黑莓的产品,在接受测试的产品中,只有不到三分之一[6].84种ssr中r . idaeus无性系种群。idaeusL.(红覆盆子),品种Glen Moy [7],不超过26%的人扩增了测试的两种黑莓基因型中的任何一种,而在该研究中测试的142个蔷薇科SSR引物对中,只有18%的人检测到两种黑莓基因型之间的多态性[8].
Lewers等人,[6]从存储在GenBank中的草莓EST序列中生成SSR标记,并用它们来扩增草莓DNA。草莓、黑莓和覆盆子同属一个亚科,草莓亚科。GenBank中大约14%的草莓ESTs含有至少5次重复的ssr序列。在由6个或6个以上重复序列设计的引物对中,68%的引物检测到被测材料之间的多态性,而在由少于6个重复序列设计的引物对中,43%的引物检测到多态性。因此,我们有理由期望从黑莓的est中可以开发出有用的SSR标记,而且如果产生了足够多的est,可以期望有足够多的SSR标记用于遗传作图。然而,目前GenBank中还没有可能用于分子标记开发的黑莓序列。因此,这项工作的目标是开发一个黑莓EST库,并使用得到的序列开发SSR标记。
结果与讨论
RNA和文库的定量
从3.5 g黑莓叶中提取的总RNA为1.2 mg,从聚合的总RNA中分离出1.8 μg的mRNA。1.8 μg mRNA反转录得到的cDNA产量为145 ng。将整个145 ng与100 ng pDONR222载体结扎,转化产量为7.6 × 106菌落形成单位。一项对96个无性系插入物大小的调查显示,通过限制性内切酶消化,插入物的平均大小为1.7 kbp,长度从大约200个碱基到超过2 kb不等。
序列分析,contig组装和同源性
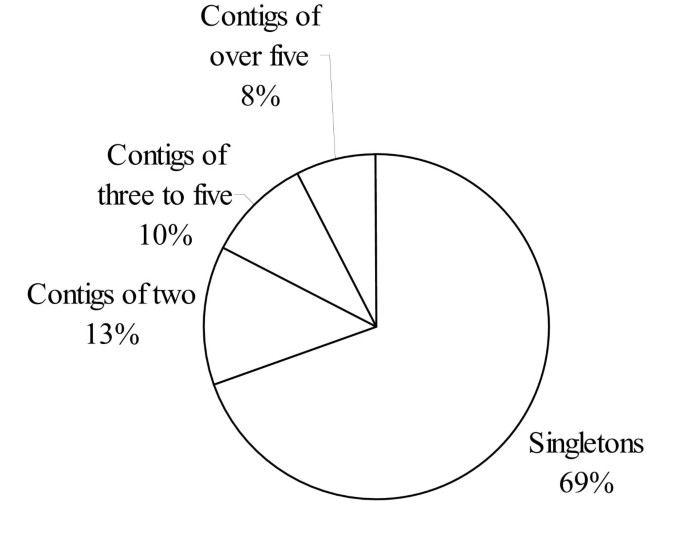
修剪后,从3884个克隆中获得3000条高质量序列(77%)。GenBank中长度超过100 bp的最高质量序列为FF682838 ~ FF685515,共2678条。平均读取长度为889 bp,平均phred值为28。测序成功率较低很可能是由于与pDONR222载体相关的二级结构的固有形成。在这3000条序列中,2614条(87%)与之前存入GenBank的序列相似(期望值< 1e-6),而386条(13%)不相似。在GenBank中与其他序列相似的2614个序列中,有580个(2614中的22%)与功能未知的假定蛋白质相似。当这3000个序列被组装成contigs时,919个(31%)被组装成301个contigs,而2081个(69%)单体仍然是分开的(图2)。1).在301个contigs中,288个(96%)与GenBank中的序列相似,13个(4%)与GenBank中的序列不相似。在GenBank中与其他序列相似的288个contigs中,有44个(占288个contigs的15%)包含100个克隆,被注释为具有未知功能。
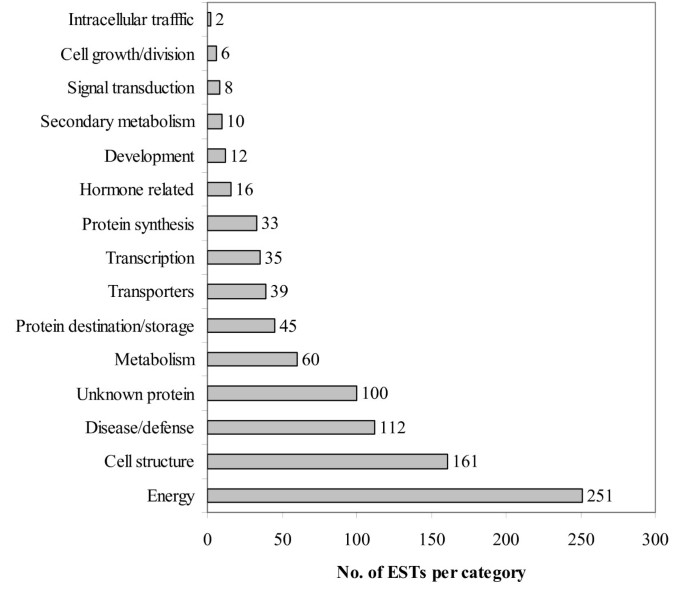
与先前报道的基因相似的contig序列是按照相对丰度排序的,由contig中的ESTs数量决定(附加文件)1),然后按其假定的功能类别进行分组(图。2) [9,10].在克隆、测序和组装成contigs的最丰富的mrna中,涉及能量(特别是光合作用和电子传递)的mrna最多。5个contigs由49个克隆组成(5%的克隆组装成contigs),与核酮糖1,5-二磷酸羧化酶(RUBISCO)小亚基序列相似,RUBISCO是光合作用卡尔文循环的关键。第二大丰富的功能类别是细胞结构,特别是与叶绿体有关的结构。17个contigs,包括114个克隆(占contigs中克隆的12%),与叶绿素a/b结合蛋白序列相似。考虑到cDNA文库是由叶组织的RNA制成的,这些结果并不令人惊讶。
第三大功能类别是疾病和防御相关的,有112个克隆组合成39个contigs。在这一功能类别中,大多数contigs(78个克隆)与应激反应相关的基因相似,69个克隆与热休克蛋白相似。同样,考虑到用于图书馆建设的叶片组织是在7月的下午从植物中收集的,这也许并不令人惊讶。其他与疾病和防御相关基因相似的contigs包括4个(14个克隆)与重金属相关基因相似,4个(11个克隆)与解毒基因相似,4个(9个克隆)与抗病基因相似。大量与疾病和防御相关的克隆表明,这个文库也将有助于研究与耐受非生物和生物胁迫潜在相关的基因。
SSR鉴定与检测
共检测到1026个GC含量为40-60%且motif两侧至少有20个碱基对序列的ssr,用于引物设计。一些序列及其引物是由序列中相同扩增区域的重复或同一序列中两个重复区域的重复引起的。剔除重复区域相同、引物相同的重复,保留扩增同一序列不同区域的引物对和扩增同一区域的不同引物对。保留这些引物对被认为是有价值的,以确保每个区域有一个工作对被识别,并允许基因家族成员的扩增。该类型引物有94对,其中579对引物由独特序列设计,共673条含SSR的引物序列,约占3000条高质量序列的22%。含有ssr序列的百分比略高于草莓(14%-15%)[6,11]和苹果(马吕斯有明显(17%) [12],以及其他两个蔷薇科物种,这些估计可能会因用于搜索ssr的ESTs方法而有所不同[13].
假设SSR区域的存在不会影响池中单个mRNA的相对丰度,那么这673条含有SSR的序列可以预期以与获得的3000条高质量序列相同的比例和方式组装成contigs。因此,大约31%(209个序列)预计将是68个contigs的成员,而69%(464个序列)预计将保持独立。假设组装成contigs的序列确实是同一基因和位点的一部分,我们可以期望68 + 464,总共532个引物对,放大来自独特基因组区域的产物。
为了测试引物对的功效,从673个引物对的较大子集中随机选择了33个引物对。其中16个序列是在考虑全部3000个序列时形成的9个contigs的一部分。仅考虑33个序列的子集,11个序列组成了3个contigs(附加文件2),符合基于整个库的期望。在33对用“APF-12”和“Arapaho”DNA检测的SSR引物中,21对(64%)扩增了一个产物,比例略低[[13].在这21个引物中,9个检测到至少一种大小多态性,一对引物扩增了一种基因型的产物,而不是另一种基因型,总共有10对引物检测到多态性,约占随机选择的33个引物的30%。
对于三个Contig中的一个,Contig 219,其序列与叶绿素a/b结合前体相似,只有一半的引物对扩增了产物,并且没有检测到多态性。另外两个contigs 112和131分别包含两个序列,每个contig扩增产物中都有两个序列的引物对,但每个contig只检测到一个引物对的多态性。因此,在检测到多态性的10对引物中,没有2对来自同一contig的序列。这些发现支持了测试可能扩增同一区域的引物对的决定,以试图确保至少有一对引物对在作图中有用。
10个引物对每个基因型最多扩增6个产物。四联体基因组中每个位点的等位基因数预计为4个,因此在比较两个四倍体基因型与SSR引物对时,最多可能有8个多态产物。甚至更多的可能是涉及基因在串联重复序列或松散集群中复制的位点[14].观察到的扩增产物数量可能低于预期数量,这是由于几个因素造成的,包括一个或多个位点的产物大小相同,以及在合成四倍体植物自育后代中观察到的一些位点缺失[15].
检测多态性的引物对数量(33个引物中有10个或约30%)低于80%至90%的综述报告范围[16].大多数研究都是在同一物种的几个不同种质上测试新的引物对,预计会有更高水平的多态性,但两个个体之间的多态性率对我们确定这个文库在遗传作图中的有用性的目标更有价值。此外,许多在遗传图谱中有用的多态性在四体黑莓的亲代筛选中是看不到的。检测到多态性的10对引物每对扩增出最多3个多态性产物(存在于一种基因型中,而不存在于另一种基因型中),平均每对引物有1.9个多态性。当这些引物对与映射种群一起使用时,数量可能会增加。如果单型PCR产物扩增的位点(例如A)在双亲中均以单剂量(simplex = Aaaa)或双剂量(duplex = Aaaa)存在,则产生的一些分离子将缺乏该位点(nuliplex = Aaaa),其中一些位点可以用TetraploidMap等软件程序进行映射[5].如果同源染色体对之间没有优先配对,父母双方都有单一剂量的位点上的分离子将以大约3:1的比例分离。即使引物对扩增了来自父母双方的产物,因此似乎没有检测到多态性,但在亲本双方都有双重剂量的位点上的分离子将以35:1的比例分离,而在亲本一方有单一剂量而另一方有双重剂量的位点上的分离子将以11:1的比例分离。如果扩增产物被映射为优势标记,由于从重组事件中获取信息的困难,建议从映射数据集中删除11:1或35:1分离比例的产物,但仍应包括3:1分离比例的产物(双亲均扩增产物)[5].
对进一步测序结果的期望
事实上,从这个库中获得的3000个序列中有69%是单例序列(冗余率,或新序列已经在数据集中被表示的几率为31%),这表明许多新的黑莓序列可以从这个18432个克隆库的继续分析中获得,尽管从5'端测序的库往往有更高的重叠不足率来形成contigs,这在一定程度上增加了单例序列的数量[17].与任何植物中预期的独特基因数量相比,初始样本量非常小(甚至局限于单个组织或阶段)。具有相对确定基因估计的植物,例如拟南芥(l)呵呵,有更多的基因,一个非标准化文库可以预期与许多基因有序列相似性,而不是在这个原始的小样本中发现的基因。很可能,随着从库中获得的序列越多,将有更大比例的序列组装成contigs [18,19].其他蔷薇科作物的单株百分比的比较与这一假设是一致的。在草莓EST库的1800个序列中观察到的单子百分比为65% [11],有点类似于在黑莓EST库中观察到的情况。然而,9984个桃子中的35% (碧桃(l)类等)。ESTs是单胞胎[20.],而在151687份苹果est中,只有17%是单胞胎[12].
组装成contigs似乎有助于发现与其他基因的序列相似性,因为3000个高质量序列中有近13%与GenBank中的其他基因不相似,而由3000个序列形成的301个contigs中只有约4%与GenBank中的其他序列没有显著相似性。由多个ESTs组成的Contigs具有更长的一致性序列和更少的错误。这种长度和质量的增加增加了识别统计上相似的蛋白质序列的概率。此外,高表达的基因倾向于在小样本中形成contigs,高表达使它们更有可能先前被识别并出现在数据库中。
结论
综上所述,本文首次发表了栽培黑莓的ESTs分析。序列和注释可通过GenBank和Rosaceae基因组数据库获得[21].所得到的序列被用于开发SSR标记,最终用于遗传作图。33个SSR引物对的子集预测,共鉴定的673个SSR引物对可检测到30%的多态性,导致202个SSR可检测到多态性,平均每个引物对可检测到1.9个多态性,或约384个多态性(表2)1).虽然这个数字可能足以识别至少一个与primocane结果相关的SSR标记,但不太可能足以生成一个足够的四倍体黑莓图谱。如果我们测序的3884个克隆的子集代表了整个18432个克隆,那么我们可以期望从这个文库中检测到940个SSR引物对(22%的序列),以检测到1786个多态性。由于冗余,检测多态性的引物对的实际数量将会减少,而随着更多的文库被测序,冗余预计将会增加。另一方面,由于某些SSR引物对在双亲中检测单剂量或双剂量存在的位点,实际可被映射的位点数量将大于估计。结果应该是有足够数量的分离位点来生成一个相当饱和的图谱,可以被其他人用来更有效地识别与其他群体中其他重要园艺性状相关的SSR标记。
方法
基因型选择和mRNA分离
四倍体黑莓品种Merton thorless (PI 553276)被选为EST开发,因为它是无刺性状的来源[3.在许多商业种植的无刺品种中。2004年7月,从贝尔茨维尔农业研究中心北部农场的植物中收获了“默顿无刺”的年轻叶子。叶子用铝箔包裹,放在液氮中,运输到实验室进行提取。使用RNeasy Plant Mini Kit (Qiagen, Inc., Valencia, california)对木本植物组织进行修饰,从3.5 g叶片组织中提取总RNA [9].将7种提取的RNA聚合在一起,以提高总产量。没有进行浓缩。Poly(A)Purist™Kit (Ambion, Austin, Tex.)用于从总RNA中分离mRNA。
文库制作和测序
cDNA文库在Gateway系统(Invitrogen Corp., Carlsbad, california)中使用pDONR222载体和ElectroMAX™DH10B™T1噬菌体抵抗细胞(Invitrogen Corp.)按照制造商说明克隆。卡那霉素用于筛选含有载体的菌落。共选取18432个克隆,排列到96孔板中进行测序。M13F底漆和ABI PRISM®BigDye™终结者v3.1反应混合物(应用生物系统公司,福斯特城,加利福尼亚州)被用于从3884个克隆的5'端测序。循环排序如下:96°C for 5 min, 35个循环(96°C for 45 s, 50°C for 5 s, 60°C for 4 min)。3884个克隆的序列数据由ABI 3730xl遗传分析仪(Applied Biosystems)积累。
跟踪文件处理
使用phred[将序列跟踪文件转换为FASTA文件和质量评分文件。22基础调用程序。使用序列比较程序Cross_Match识别和掩盖载体和宿主污染(如物种特异性线粒体RNA, rRNA, tRNA和snoRNA) [23].很少有属特异性和种特异性的叶绿体序列。向量修剪去除最长的非掩码序列,进一步修剪去除读取两端的低质量碱基(小于phred得分20)。如果序列缺乏polyA尾(以消除叶绿体基因组编码序列),具有大于5%的模糊碱基,或具有小于100个高质量碱基(最低phred评分为20),则序列被丢弃。PolyA尾巴通过在序列的前350个碱基之后找到至少9个A的第一次运行来搜索。
组装高质量的序列和注释
过滤后的库文件使用contig汇编程序CAP3 [24].更严格的参数(-p 90。-d 60)用于防止过度组装和帮助识别潜在的谬误。结合contig数据集和singleton数据集,得到unigene数据集。unigene数据集的注释包括使用fastx3.4算法将过滤后的库和contig共识库文件与GenBank nr蛋白数据库进行成对比较[25].只有当期望截断值小于1e-6时,序列才被认为相似。该序列还与来自TAIR的拟南芥蛋白进行了比较[26],以及Swiss-Prot蛋白质数据库[27].具有假定身份的contig被分为14个功能组,然后使用前面描述的蓝莓[9].分类基于蛋白质的已知功能,参考BioCyc-MetaCyc:代谢途径百科全书网站[28],参考基因本体(Go)数据库[29],并在PubMed中搜索相关摘要[30.].
底漆设计及测试
使用基于SSRIT软件的cusisrs .pl脚本在单基因数据集中识别简单序列重复序列(SSRs) [31,32]并进一步筛选以获得最佳引物开发(40-60% GC含量和motif两侧至少20个碱基对序列)。引物使用引物3从周围序列设计[33].引物序列是从单个序列而不是contigs设计的,以避免不正确的序列组装可能导致的非扩增问题。在无刺'Arapaho'上测试了33个引物对的子集[34),以及产生原始果的‘APF-12’(Prime-Jim®) [1].DNA提取、聚合酶链式反应(PCR)和PCR产物的大小按照Stafne等人的描述进行。[8].
参考文献
Clark JR, Moore JN, Lopez-Medina J:阿肯色大学发布的原始果实黑莓品种。HortScience。2004, 39: 662。
杨晓明,王晓明,王晓明,等:四倍体直立黑莓原生果实遗传模型的建立。环境科学学报,2004,25(2):366 - 366。
斯科特DH,达罗GM,墨克DP:默顿无刺黑莓的亲本育种。地球科学进展,1997,33(4):366 - 366。
吴kk, Burnquist W, Sorrells ME, Tew TL, Moore PH, Tankesley SD:利用单剂量限制性片段检测和估计多倍体连锁。《应用理论》,1998,33(3):344 - 344。10.1007 / BF00224274。
罗志伟,刘志强,王志强,王志强,王志强。基于分子标记的四倍体遗传连锁图谱的构建。中国生物医学工程学报,2001,27(3):344 - 344。
Lewers KS, Bassil NV, Styan SMN, Hokanson SC:草莓genbank衍生和基因组简单序列重复(SSR)标记及其在草莓,黑莓和红黑覆盆子上的应用。生态环境学报,2005,29(3):344 - 344。
Graham J, Smith K, MacKenzie K, Jorgenson L, Hackett C, Powell W:红覆盆子遗传连锁图谱的构建(悬钩子属植物idaeus无性系种群。idaeus)基于aflp、基因组- ssr和EST-SSR标记。应用理论与实践,2004,29(4):344 - 344。10.1007 / s00122 - 004 - 1687 - 8。
Stafne ET, Clark JR, Weber CA, Graham J, Lewers KS:覆盆子和黑莓遗传定位的SSR标记。环境科学学报,2005,29(3):344 - 344。
王志刚,王志刚,王志刚,等。蓝莓花蕾冷驯化相关基因表达分析。植物科学学报,2004,26(3):344 - 344。10.1016 / j.plantsci.2003.11.013。
Ablett E, Seaton G, Scott K, Shelton D, Graham MW, Baverstock P, Slade Lee L, Henry R:葡萄叶和浆果中ESTs基因的整体表达模式分析。中国植物科学,2000,39(3):344 - 344。10.1016 / s0168 - 9452(00) 00335 - 6。
Folta KM, Staton M, Stewart PJ, Jung S, Bies DH, Jesdurai C, Main D:八倍体草莓的ESTs和SSR标记草莓属×ananassa).中国生物医学工程学报,2005,5:12-22。10.1186 / 1471-2229-5-12。
Newcomb RD, Crowhurst RN, Gleave AP, Rikkerink EHA, Allan AC, Beuning LL, Bowen JH, Gera E, Jamieson KR, Janssen BJ, Laing WA, McArtney S, Nain B, Ross GS, Snowden KC, Souleyre EJF, Walton EF, Yauk Y-K: Apple表达序列标签的分析。植物生理学报,2006,29(4):357 - 357。10.1104 / pp.105.076208。
王志刚,王志刚,王志刚。植物基因微卫星标记的研究进展。生物技术,2007,23:48-55。10.1016 / j.tibtech.2004.11.005。
Lewers K, Heinz R, Beard H, Marek L, Matthews B:大豆A2连锁群黑色种皮附近基因密集区域的物理图谱Rhg4位点。应用理论与实践,2002,29(4):344 - 344。10.1007 / s00122 - 001 - 0780 - 5。
Gaeta RT, Pires JC, inigez - luy F, Leon E, Osborn TC:再合成的基因组变化芸苔属植物显著以及它们对基因表达和表型的影响。植物学报。2007,doi: 10.1105/tpc.107.054346
Ellis JR, Burke JM: EST-SSRs作为群体遗传分析的资源。遗传与遗传学报,2007,29(3):344 - 344。10.1038 / sj.hdy.6801001。
Wang J- pz, Lindsay BG, Leebens-Mack J, Cui L, Wall PK, Miller WC, dePamphilis CW: EST聚类误差评估与校正。生物信息学,2004,20:2973-2984。10.1093 /生物信息学/ bth342。
王金鹏,林德伟,崔玲,王金鹏,王金鹏,张娟,deamphilis CW: EST测序中单文库或多文库基因捕获预测和重叠估计。生物信息学,2005,6:300-310。10.1186 / 1471-2105-6-300。
李丽娟,李志刚,李志刚,李志刚。EST分析中贝叶斯非参数预测方法的研究。生物信息学,2007,8:339-359。10.1186 / 1471-2105-8-339。
Horn R, Lecouls A-C, Callahan A, Dandekar A, Garay L, McCord P, Howad W, Chan H, Verde I, Main D, Jung S, Georgi L, Forrest S, Mook J, Zhebentyayeva T, Yu Y, Kim HR, Jesudurai C, Sosinski B, Arús P, Baird V, Parfitt D, Reighard G, Scorza R, Tomkins J, Wing R, Abbott AG:桃的候选基因数据库和转录谱图。应用理论与实践,2005,30(1):1 - 4。10.1007 / s00122 - 005 - 1968 - x。
Jung S, Staton M, Lee T, Blenda A, Svancara R, Abbott A, Main D: GDR(蔷薇科基因组数据库):蔷薇科基因组学和遗传学数据的综合网络数据库。核酸决议2007
刘志刚,刘志刚,刘志刚。基于phred的自动序列跟踪的基本调用方法。一、准确性评估。基因组学研究,1998,8:175-185。
Gordon D, Abanjian C, Green P:序列整理的图形工具。基因组学研究,1998,8:95-202。
欢X,马丹A:一个DNA序列组装程序。基因组学研究,1999,9:868-877。10.1101 / gr.9.9.868。
Pearson JD, Lipman DJ:生物序列比较的改进工具。自然科学进展,1998,29(2):344 - 344。10.1073 / pnas.85.8.2444。
Rhee SY, Beavis W, Berardini TZ, Chen G, Dixon D, Doyle A, Garcia-Hernandez M, Huala E, Lander G, Montoya M, Miller N, Mueller LA, Mundodi S, Reiser L, Tacklind J, Weems DC, Wu Y, Xu I, Yoo D, Yoon J, Zhang P:拟南芥信息资源(TAIR):一个模式生物数据库,为拟南芥生物学、研究材料和群落提供集中的、有管理的门户。中国生物医学工程学报,2003,31(4):344 - 344。10.1093 / nar / gkg076。
Bairoch A, Boeckmann B, Ferro S, Gasteiger E: Swiss-Prot:进化与稳定之间的杂耍。生物工程学报,2004,5:39-55。10.1093 /龙头/ 5.1.39。
代谢途径百科全书。[http://metacyc.org/]
基因本体(Go)数据库。[http://www.geneontology.org/]
PubMed。[http://www.ncbi.nlm.nih.gov/PubMed/]
Jung S, Abbott A, Jesudurai C, Tomkins J, Main D: rosaceaceae ESTs中简单序列重复序列的频率、类型、分布和注释。中国生物医学工程学报,2005,30(5):366 - 366。10.1007 / s10142 - 005 - 0139 - 0。
Temnykh S, DeClerck G, Lukashova A, Lipovich L, Cartinhour S, McCouch S:水稻微卫星的计算和实验分析:频率、长度变异、转座子关联和遗传标记潜力。基因组学报,2001,11:1441-1452。10.1101 / gr.184001。
Rozen S, Skaletsky HJ:面向普通用户和生物学家程序员的WWW上的Primer3。生物信息学方法与协议。编辑:Krawetz S, Misener S. 2000,托托瓦,新泽西州:Humana出版社,365-386。(系列:分子生物学方法第132卷。
Moore JN, Clark JR:“阿拉帕霍”直立无刺黑莓。HortScience。1993, 28: 861-862。
确认
作者希望感谢北美荆棘种植者协会对本研究的部分资助;Drs。John Clark和Eric Stafne的“阿拉帕霍”和“APF-12”叶片组织;Kate Rappaport女士、Ernalyn Peralta女士和Allie Bradley女士获得SSR扩增数据;以及处理反应的蒂娜·斯彭女士。感谢Tad Sonstagard博士的管理,并感谢Beltsville地区研究中心对基因分型设备的支持。还要感谢博士们。Deb Fravel, Angela Baldo, David Hyten, Ann Callahan以及匿名评论者的有益评论。在本出版物中提及商品名称或商业产品仅为提供特定信息的目的,并不意味着美国农业部或克莱姆森大学的推荐或认可。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
KSL种植了这些植物并计划了这项研究。KSL和ALD收获叶片组织。ALD在LJR的指导下提取RNA。BJC首先克隆、挑选并检查库的质量。DCH和CAS在JPT指导下对ESTs进行测序。MES在DSM和JPT的指导下进行了跟踪文件处理、序列注释、contig组装、SSR搜索和EST-SSR引物设计。LJR将注释基因按功能分类。KSL在PCR中监督DNA的提取和EST-SSR引物对的检测。KSL起草了手稿,所有作者阅读并批准了最终的手稿。
电子辅助材料
12870 _2008_277_moesm1_esm.xls
附加文件1:黑莓(悬钩子属植物l .)包含5个以上序列的EST contigs。黑莓序列最接近匹配的Entrez蛋白数据库序列(匹配序列)的描述和登录号,以及匹配序列的功能类别。(xls 22 kb)
12870 _2008_277_moesm2_esm.xls
附加文件2:来自黑莓的SSR引物对的检测(悬钩子属植物l .)含有EST序列的SSR。包括序列组装的contigs,序列相似度匹配,序列中的重复motif, motif的重复次数,在开放阅读框中的放置(ORF,“Yes”表示ORF中存在SSR,“No”表示ORF中不存在SSR),扩增成功,以及检测到的多态性类型(“No”表示无多态性,“Size”表示所测两种基因型之间存在大小多态性,或“+/-”表示引物对扩增了两种基因型中至少一种产物,但不扩增另一种)。(xls 22 kb)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
刘易斯,k.s.,萨斯基,c.a.,卡斯伯森,B.J.et al。黑莓(悬钩子属植物L.)表达序列标记库,用于开发简单序列重复标记。BMC植物生物学8日,69(2008)。https://doi.org/10.1186/1471-2229-8-69
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-69
关键字
- 主要对
- 简单序列重复标记
- 高质量序列
- 简单序列重复素数对
- 开发简单序列重复标记