摘要
背景
近年来,各种作物物种序列资源的快速积累确保了遗传学方法的改进,包括数量性状位点分析以及整体群体分析和自然变异的关联作图。由于麦族包括小麦和大麦等重要谷物,整合这些作物的遗传标记信息应该有效地加速基于地图的麦族物种遗传研究,并导致发现与植物生产力有关的关键位点,从而有助于可持续粮食生产。因此,整合小麦和大麦遗传标记信息并进一步开发遗传标记需要信息学应用和语义知识库,以推进小麦科传统标记辅助遗传分析和群体基因组学研究。
描述
麦类已映射表达序列标签(EST)数据库(TriMEDB)提供了与大麦及其小麦同源物相关的已映射cDNA标记的信息以及各种注释。当前版本的TriMEDB提供了大麦和小麦ESTs的地图定位数据,这些数据是从3个已发表的大麦连锁图(苏格兰作物研究所的大麦单核苷酸多态性数据库,莱布尼茨植物遗传学和作物植物研究所的大麦转录本图,以及HarvEST大麦。1.63)和1个二倍体小麦图谱。这些数据被导入到CMap中,以便可视化这些ESTs的图谱位置以及这些ESTs与公共基因模型和代表性cDNA序列的相互关系。检索到的每个EST标记对应的cDNA序列被分配到水稻基因组中,以预测外显子-内含子结构。此外,为了在公共领域的小麦科植物中生成一组独特的EST标记,将3472个标记组合在一起,形成2737个独特的标记群作为contigs。利用这些contigs对从不同EST图谱资源获得的连锁图谱进行两两比较。
结论
TriMEDB提供了转录遗传标记的信息,并作为一个语义知识库,为加速麦类QTL分析和群体遗传学研究提供了信息学工具。
背景
现有遗传标记的积累和饱和直接促进了标记辅助遗传研究的发展,并具有广泛的应用前景。设计用于广泛覆盖基因组的遗传标记不仅允许通过数量性状位点(QTL)分析检测和识别与复杂性状相关的单个基因,而且还可以探索遗传多样性和自然变异的群体结构[1- - - - - -3.].近年来各种作物物种序列资源的迅速积累,确保了遗传学方法与比较基因组学相结合的改进[4].作物基因组资源的日益丰富,极大地促进了作物进化的阐明;这一阐明涉及到关键位点的发现,从而有助于基于基因的驯化和改进[5].
麦族包括重要的作物,如小麦(小麦L.)和大麦(大麦芽l .)。有必要积累和饱和可用标记,并设计一个核心标记集用于高效的基于图谱的研究,以发现与这些作物不同品种中发生的表型变化相关的关键位点,这些位点与生产力有关。这些努力将通过分子育种的应用促进可持续粮食生产。为了建立全面的基因发现序列资源和可靠的基因表达数据库,大规模采集了普通小麦和大麦的表达序列标签(ESTs) [6,7].近年来,这些作物的ESTs和非冗余序列的数量急剧增加。2008年3月1日,NCBI的UniGene数据库分别包含41227条和23078条具有代表性的小麦和大麦聚类est序列。这些全面的EST集合在全基因组遗传标记的开发中具有潜在的应用价值。迄今为止,不同的大麦图谱已经使用est衍生标记构建[8- - - - - -10];因此,麦类基因组整体遗传标记的潜在可用性增加了。从ESTs中设计的遗传标记数量超过3000个;这个数字与这些ESTs在大麦基因组中的染色体位置一起公布。最近,二倍体小麦的遗传图谱(小麦属植物monococcumL.)是利用大麦est衍生的可转移标记构建的[11].这种方法清楚地表明,从大麦EST数据中获得的EST标记可以直接用作小麦基因组的遗传标记。因此,由于二倍体小麦和大麦之间的同源连锁群具有显著的保守性,因此,将大麦EST标记与小麦EST标记进行相互关联和整合是增强和积累大麦和小麦潜在EST标记的有效和可靠的权宜之。11,12].
最近,一种使用多个定位群体进行qtl跨群体比较和整合的遗传方法已被报道;这种方法对于广泛研究全基因组复杂性状的遗传结构是非常有效的。例如,利用由7个重组自交系(RI)和1个双单倍体(DH)群体组成的多个定位群体检测到大麦种子休眠qtl,以整合和扩展先前已知的qtl和表型的使用,以评估不同的种质资源。因此,群体中的保守qtl和一致性qtl已通过共识标记区间确定[13].近年来,嵌套关联映射(nested association mapping, NAM)策略作为一种新的复杂性状解剖策略,同时利用了连锁分析和关联映射的优势,已在玉米中得到应用;该策略涉及对由不同亲本和一个共同亲本系产生的RI系群体的共同亲本特异性(CPS)标记进行基因分型[14].对于全基因组性质的遗传方法,统一和整合现有的cDNA标记以生成跨种群的共同标记集将促进多个种群和/或自然变异的高通量基因分型。
因此,将现有的cDNA标记整合到一个语义知识库中,可以提供各种注释,并生成一组统一的相同标记,这对于改进基于全基因组图的方法肯定是有用的。在此,我们报告了麦类EST数据库(TriMEDB),该数据库提供了大麦中已映射的cDNA标记的信息,以及各种注释及其在小麦中的同源物。当前版本的TriMEDB包含2737个唯一的cdna映射到链接映射上,查询数据的结果显示在web界面上。
结构与内容
EST标记源
当前版本的TriMEDB提供了大麦和小麦的ESTs信息及其从3个已发表的大麦连锁图和1个二倍体小麦图中检索到的地图位置数据。此信息可导入CMap [15],并用于将ESTs的地图位置可视化。来自莱布尼茨植物遗传学和作物植物研究所(IPK)的1052个基于EST的大麦标记的EST序列、聚合酶链反应(PCR)引物和图谱位置,通过使用国家生物技术信息中心(NCBI) GenBank中可用的登录号检索。苏格兰作物研究所(SCRI) 332个标记的大麦SNP数据库和CMap的数据来自SCRI网站。使用HarvEST barley ver也检索了1848标记的contigs序列和地图位置。1.63.从Hori的文献中获得240个二倍体小麦的PCR标记等.(2007)。这些PCR标记用于dbEST (GenBank EST release #163.0)中小麦和大麦EST集的相应序列检测。通过研究每个引物对对应的cDNA序列,进一步分析每个引物对所找到的最长的ESTs。
EST标记在公共序列资源基因模型中的分配
从每个cDNA数据库中获得了EST标记的核苷酸序列,该数据库提供了聚集或组装cDNA的代表性序列。为此,我们搜索了NCBI UniGene的PCR引物序列和扩增标记序列[16], TIGR基因指数[17],收获[10],以及植物基因组数据库(PlantGDB) [18]通过匹配PCR引物的序列身份或使用BLASTN以<1e × 10的阈值进行相似性搜索,对大麦进行检测-200年.利用BLASTN相似性搜索技术对各序列资源进行同源标记序列的识别,阈值e <1e × 10-130年.
EST标记的统一
为了为Triticeae部落生成一个独特的EST标记集,使用CAP3对来自4个地图的3472个EST进行了组装[19]使用默认参数设置;这些ESTs被分为2737个独特的标记组作为contigs。这些contigs被用作虚拟标记来比较同源染色体的连锁图谱。
水稻基因组标记的分配
为了鉴定每个大麦EST标记的水稻基因同源物,并根据水稻同源序列预测外显子-内含子结构,使用BLASTN进行同源性搜索,阈值e值<1e × 10-20年与国际水稻基因组测序计划(IRGSP)的水稻基因组序列进行比较。4.0摘自水稻注释项目数据库(RAP-DB) [20.].利用该数据库对同源区域进行近似定位和提取,并将标记物的cDNA序列与BLASTN搜索得到的覆盖两侧同源区域5kb序列的水稻基因组片段进行SIM4比对[21]使用默认参数设置。预测的外显子-内含子结构数据与通过Generic genome Browser从RAP-DB中获得的水稻基因组注释数据一起应用于内部数据库[22].
数据库和web界面
数据库采用MySQL和Perl CGI脚本实现,web界面运行在Apache web服务器上。实现了CMap来可视化链接映射。实现了通用基因组浏览器(Generic Genome Browser),以显示每个标记cdna的外显子-内含子结构,并将其与从RAP-DB中获得的水稻同源序列和注释水稻基因组进行比较。
实用程序
标记搜索
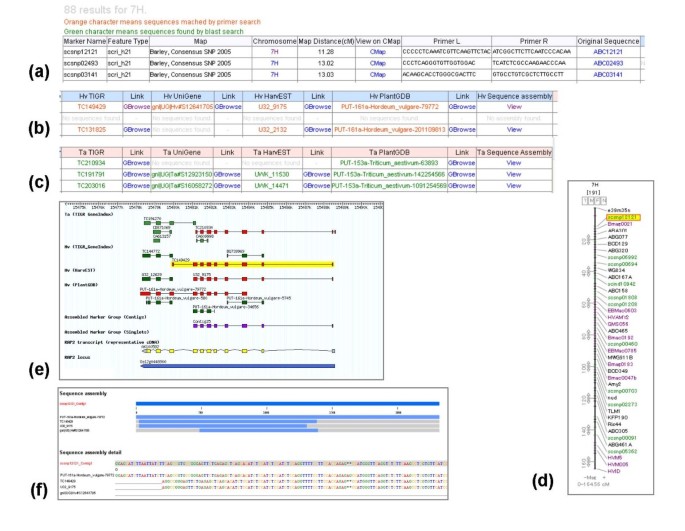
用户可以使用两个不同的查询来搜索大麦和二倍体小麦的EST标记:标记名称和染色体名称(图2)。1).可以根据标记类型(如SNP和cleaved amplification polymorphic sequence (CAPS))以及链接映射的名称对这两个查询中的每个查询进行过滤。通配符'*'在搜索文本字段中是可以接受的,即为检索位于某个染色体上的所有标记提供的字段。搜索标记的结果以电子表格的形式显示,由3个主要部分组成,如图所示。2、2,2摄氏度.左侧分区由9列组成,显示一般信息,如标记名称、标记类型、原始映射群体、染色体位置和映射位置,以及从每个标记资源中检索到的PCR引物和/或原始序列(图2)。2).其余2个区分别展示了从TIGR Gene Index、NCBI UniGene、HarvEST和PlantGDB 4个单独序列资源中获得的cdna的代表性序列。这些代表性序列对应于引物序列搜索和/或序列相似性搜索所识别的每个标记。在其他两个部门中,一个提供大麦相关信息,另一个提供小麦相关信息。这2个部门中的每一个都有3种类型的超链接(图。2 b 2 c).染色体名称的超链接链接到可以在CMap上可视化的连锁图(图2)。二维).序列标识符的超链接链接到一个页面,该页面显示每个序列资源的单个fasta格式序列,以及使用原始标记序列作为查询获得的BLASTN的同源搜索结果。“Gbrowse”的超链接链接到一个本地通用基因组浏览器,该浏览器提供了从RAP-DB中释放的注释水稻基因组,并显示了基于水稻基因组同源序列衍生的标记cdna的外显子-内含子结构(图2)。2 e).序列组装列中的“View”超链接链接到显示contig组装的页面,其中包含来自序列资源的相应序列,并为目前可用的最长cDNA序列提供contig组装(图2)。2 f).
组合标记搜索
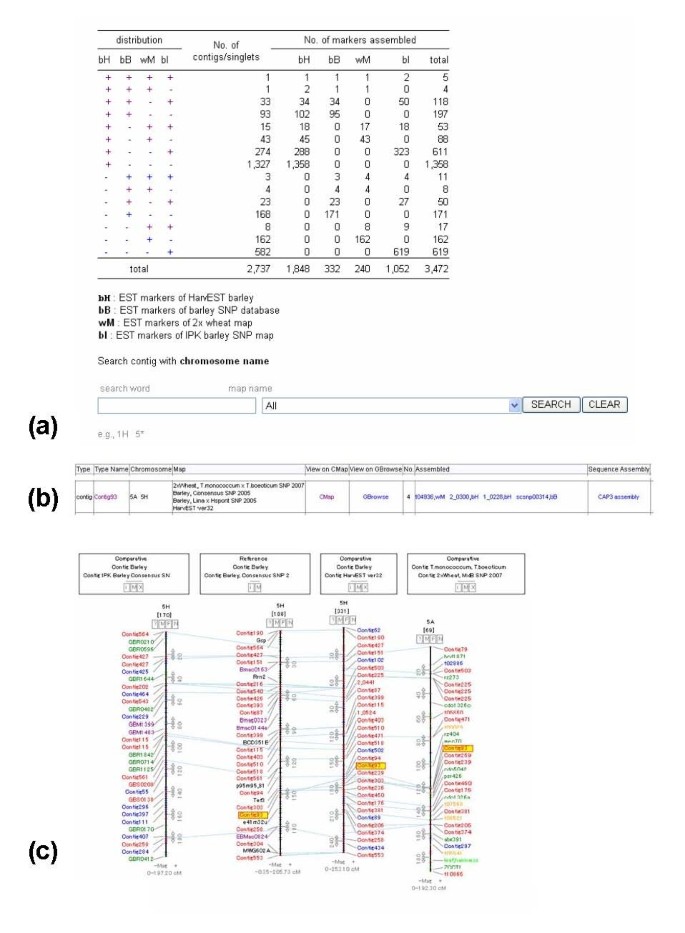
组装的标记搜索页面提供了一个接口,用于检索从CAP3程序集派生的2737个惟一的标记组,总共有3472个标记。如图所示。3,该网页的网页界面描述了标记的数量和4个单个EST标记资源的分布情况。每个显示单个标记分布的超链接链接到表示contig列表的页面,以及contig识别标签和到CMap的超链接,组装标记的列表,以及到contig组装的链接(图2)。3 b).用户可以通过使用染色体名称和链接映射名称来搜索组装的标记,以便检索位于目标染色体上的统一标记集(图2)。3).链接到本页的链接映射提供了多个链接映射的比较说明,这些链接映射是使用这些虚拟标记统一到相同的contigs中获得的;该图谱不仅可用于识别不同连锁图谱中常见的标记,还可用于识别可能从其他连锁图谱转移的候选标记(图2)。3 c).
表示统一标记的Web界面。从大麦和二倍体小麦的4个个体标记资源中获得的相同标记的分布状况。用户可以通过单击分布模式中的每个链接,并输入染色体名称和链接映射名称(A)来访问统一标记列表。统一标记及其成员以contigs形式列出,并超链接到CMap,在CMap中可以比较来自不同资源的链接映射。例如,2个大麦图谱和一个二倍体小麦图谱共用的Contig93被超链接到CMap、Gbrowse、组装的标记信息和CAP3组装(b)。相同标记的Contig组装用于虚拟标记,以比较图谱资源之间的链接图谱。描述了3个不同大麦图谱中同源群5的连锁图谱的比较,以及使用它们共享的组合标记群进行的二倍体小麦图谱的比较(c)。
讨论与结论
积累与大麦和小麦遗传标记相关的信息,并将与这些标记相关的各种注释整合到语义知识库中,已经有效地促进了基于地图的麦类基因组学方法。我们结合了目前大麦和小麦的EST标记,已经被用于将2700多个cdna映射到大麦和小麦的同源染色体上。这些独特的标记将被认为是多态性的初始候选,可用于通过使用新的映射群体生成连锁图,而这些映射群体又可用于QTL分析。这些独特的标记将被大麦和小麦共享。由于预测的外显子-内含子结构和水稻基因组的信息也应该有助于设计PCR引物来扩增合适的内含子以发现多态性,它们也应该有利于小麦科的有效通量基因组定位[23].
遗传标记由多个数据库资源提供,这些数据库资源有一个web界面,允许用户浏览遗传连锁图。GrainGenes是麦类基因组学的热门网站;它还提供了小麦、大麦、黑麦和燕麦的遗传标记和连锁图谱数据[24].Gramene是一个植物比较基因组学数据库;它提供了各种植物的基因图谱[25].TriMEDB专注于映射est;与这些先前发布的数据库相比,TriMEDB由于其3个特定特征,可以更大程度地利用小麦科的遗传ESTs图谱。(1)大麦和二倍体小麦的EST基因标记已被分配到聚类EST序列数据库,即小麦和大麦的公共数据库UniGene、HarvEST、TIGR Gene Index和Plant GDB。有了这个数据库,用户可以在两个物种中找到同源的标记序列,如图所示。2 b而且2摄氏度.(2)在序列相似性的基础上,将聚类est序列映射到水稻基因组上,以预测小麦科和水稻之间的外显子-内含子结构和相似性。(3)在该数据库中,EST标记被聚类并组装成contig组,以统一来自各种资源的EST标记。因此,用户可以直接比较大麦之间以及大麦和二倍体小麦之间的遗传图谱。我们相信,TriMEDB的这些特征将有助于改善谷物和草的基因组学。
补充图。附加文件中的S11举例说明使用TriMEDB作为小麦类EST标记的语义知识库,以解决基于地图的草基因组学研究中遇到的查询问题。TriMEDB可以用于查询,例如涉及搜索标记以进行全基因组基因分型,以构建链接图或链接不平衡(LD)映射。统一的contig标记更适用于多定位群体和小麦科跨种间的QTL比较。如图所示。3.,将来自不同遗传图谱的大麦EST标记组装成相同的标记组,导入CMap进行比较。因此,TriMEDB可以作为跨麦类基因组学的信息学门户[9].此外,它还可用于大麦各品种间遗传知识的整合分析以及大麦与小麦同源染色体上保守qtl的比较。
此外,所获得的标记群可作为一组核心标记,用于小麦科植物群体结构的高通量基因分型。驯化和适应导致小麦和大麦种植面积的扩大[26].TriMEDB肯定会对小麦基因组学做出贡献,因为它将有助于发现与适应各种环境相关的关键位点,并可能有助于发现驯化品种的表型变异。TriMEDB也可以应用于EST标记积累到被检测qtl的靶染色体区域,通过搜索更多的标记,将标记分配在常用定位标记的区间上。基于小麦科/水稻基因组共线性寻找候选基因可能有助于浏览Gbrowse上显示的水稻基因组注释。TriMEDB可以将任何映射EST数据合并为语义知识,只要该数据包含标记名称和以下任何序列标识符:公共数据库的登录ID、PCR引物序列或标记序列[参见附加文件中的补充图S2]1].因此,可以将映射的EST信息更新并进一步积累到TriMEDB上。TriMEDB作为一个新的功能遗传标记库的这些方面将使我们能够使用多个分离群体、群体分析和全基因组关联映射以及在麦类物种中使用这些联合方法来促进强大的QTL分析。该数据库可作为标记饱和、QTL区域缩小和克隆以及比较草基因组学等项目的平台。
因此,TriMEDB对于传统的基于地图的分析,如QTL分析和关联映射,以及将促进麦类植物分子育种的进化群体基因组学研究特别有用。
可用性和需求
项目名称:TriMEDB: Triticeae映射EST数据库
项目首页:http://trimedb.psc.riken.jp/
操作系统:独立于平台
编程语言:Perl
其他要求:无
许可证:不需要
对非院士使用的任何限制:无
参考文献
- 1.
Varshney RK, Graner A, Sorrells ME:基因组学辅助育种的作物改良。植物科学进展,2005,10:621-630。10.1016 / j.tplants.2005.10.004。
- 2.
费剑锋,万杰,王志刚,王志刚,王志刚,王志刚。基于水稻亚种遗传育种的SNP资源籼稻而且粳稻基因组比对。基因组学报,2004,14:1812-1819。10.1101 / gr.2479404。
- 3.
Caicedo AL, Williamson SH, Hernandez RD, Boyko A, Fledel-Alon A, York TL, Polato NR, Olsen KM, Nielsen R, McCouch SR, Bustamante CD, Purugganan MD:驯化水稻核苷酸多态性的全基因组模式。公共科学图书馆,2007,3:1745-1756。10.1371 / journal.pgen.0030163。
- 4.
胡培生,王晓明,王晓明:谷物知识:谷物模型的基因组学。中国生物工程学报,2005,30(4):344 - 344。10.1101 / gr.3725905。
- 5.
伯克JM,伯格JC,查普曼MA:作物进化:从遗传学到基因组学。中国生物医学工程学报,2007,6:525-532。
- 6.
Zhang H, Sreenivasulu N, Weschke W, Stein N, Rudd S, Radchuk V, Potokina E, Scholz U, Schweizer P, Zierold U, Langridge P, Varshney RK, Wobus U, Graner A:基于表达序列标签的大麦转录组大规模分析植物学报,2004,30(4):379 - 379。10.1111 / j.1365 - 313 x.2004.02209.x。
- 7.
Mochida K, Kawaura K, Shimosaka E, Kawakami N, Shin-I T, Kohara Y, Yamazaki Y, Ogihara Y:大量表达序列标签的组织表达图谱及其在普通小麦胁迫响应基因硅质筛选中的应用。中国生物医学工程学报,2006,27(3):344 - 344。10.1007 / s00438 - 006 - 0120 - 1。
- 8.
罗斯托克斯N, Borevitz JO, Hedley PE, Russell J, Mudie S, Morris J, Cardle L, Marshall DF, Waugh R:大麦转录组单特征多态性的发现。中国生物工程学报,2005,6:R54-10.1186/gb-2005-6-6-r54。
- 9.
Stein N, Prasad M, Scholz U, Thiel T, Zhang H, Wolf M, Kota R, Varshney RK, Perovic D, Grosse I, Graner A:大麦基因组1000位点转录本图谱:禾草整合基因组学的新定位点。应用理论与实践,2007,29(1):1 -8。10.1007 / s00122 - 006 - 0480 - 2。
- 10.
- 11.
Hori K, Takehara S, Nankaku N, Sato K, Sasakuma T, Takeda K:大麦EST标记增强了二倍体小麦的图谱饱和度和QTL定位。育种科学,2007,57:39-45。10.1270 / jsbbs.57.39。
- 12.
Dubcovsky J, Luo MC, Zhong GY, Bransteitter R, Desai A, Kilian A, Kleinhofs A, Dvorák J:二倍体小麦遗传图谱,小麦属植物monococcumL.,并与图进行比较大麦芽遗传学杂志。143:983-999。
- 13.
Hori K, Sato K, Takeda K:大麦新种质多定位群体种子休眠QTL检测。应用物理学报,2007,29(3):369 - 366。10.1007 / s00122 - 007 - 0620 - 3。
- 14.
于俊,王晓明,王晓明,王晓明。玉米巢式关联定位的遗传设计与统计分析。遗传学报,2008,38(4):539-551。10.1534 / genetics.107.074245。
- 15.
- 16.
Wheeler DL, Barrett T, Benson DA, Bryant SH, Canese K, Chetvernin V, Church DM, Dicuccio M, Edgar R, Federhen S, Feolo M, Geer LY, Helmberg W, Kapustin Y, Khovayko O, Landsman D, Lipman DJ, Madden TL, Maglott DR, Miller V, Ostell J, Pruitt KD, Schuler GD, Shumway M, Sequeira E, Sherry ST, Sirotkin K, Souvorov A, Starchenko G, Tatusov RL, Tatusova TA, Wagner L, Yaschenko E:国家生物技术信息中心数据库资源。核酸决议2008,D13-D21。36个数据库
- 17.
Childs KL, Hamilton JP, Zhu W, Ly E,张F, Wu H, Rabinowicz PD, Town CD, Buell CR, Chan AP: TIGR植物转录组件数据库。核酸研究,2007,D846-851。10.1093 / nar / gkl785。35数据库
- 18.
Duvick J, Fu A, Muppirala U, Sabharwal M, Wilkerson MD, Lawrence CJ, Lushbough C, Brendel V:比较植物基因组学资源。核酸决议2008,D959-D965。36个数据库
- 19.
黄X, Madan A: CAP3: DNA序列组装程序。基因组学报,1999,9:868-877。10.1101 / gr.9.9.868。
- 20.
Ohyanagi H, Tanaka T, Sakai H, Shigemoto Y, Yamaguchi K, Habara T, Fujii Y, Antonio BA, Nagamura Y, Imanishi T, Ikeo K, Itoh T, Gojobori T, Sasaki T:水稻注释项目数据库(rapd - db): hub for栽培稻ssp。粳稻基因组信息。核酸决议2006,D741-D744。10.1093 / nar / gkj094。34个数据库
- 21.
张泽,张志军,张志军,张志军,王志军:cDNA序列与基因组DNA序列比对的计算机程序。中国生物工程学报,1998,8:967-974。
- 22.
Stein LD, Mungall C, Shu S, Caudy M, Mangone M, Day A, Nickerson E, Stajich JE, Harris TW, Arva A, Lewis S:通用基因组浏览器:模型生物系统数据库的构建模块。中国生物工程学报,2002,30(4):344 - 344。10.1101 / gr.403602。
- 23.
Ishikawa G, Yonemaru J, Saito M, Nakamura T:基于pcr的标志性独特基因(PLUG)标记能有效地将小麦同源基因分配到A、B和D基因组上。中国生物医学工程学报,2007,30(4):344 - 344。
- 24.
Carollo V, Matthews DE, Lazo GR, Blake TK, Hummel DD, Lui N, Hane DL, Anderson OD: GrainGenes 2.0。对小谷物群落来说是一种改良的资源。植物科学学报,2005,29(3):344 - 344。10.1104 / pp.105.064485。
- 25.
Liang C, Jaiswal P, Hebbard C, Avraham S, Buckler ES, Casstevens T, Hurwitz B, McCouch S, Ni J, Pujar A, Ravenscroft D, Ren L, Spooner W, Tecle I, Thomason J, Tung CW, Wei X, Yap I, Youens-Clark K, Ware D, Stein L: Gramene:一种生长植物比较基因组资源。核酸项目,2008,D947-D953。36个数据库
- 26.
杜晓峰,李志刚,李志刚:基因组可塑性是多倍体小麦驯化成功的关键因素。科学通报,2007,37(3):344 - 344。10.1126 / science.1143986。
确认
作者感谢IPK的A. Graner博士和N. Stein博士,冈山大学的K. Sato博士,SCRI的R. Waugh博士和加州大学的T. Close博士允许将发布的数据整合到TriMEDB中。作者还感谢NIAS的K. Hori博士的有益讨论。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
KM设计项目,进行数据分析,协助设计数据库,并起草文稿。DS还设计了该项目并分析了已发表的基因图谱。TY进行数据分析,并协助设计网页界面和数据库。TS执行数据库系统管理。KS担任该项目的主要研究员。所有作者都对手稿的写作做出了贡献,并阅读并批准了最终提交的版本。
电子辅助材料
12870 _2008_280_moesm1_esm.pdf
附加文件1:补充图S1是基于地图的分析和TriMEDB使用的示意图。补充图S2是TriMEDB数据库结构的示意图。(pdf 56kb)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
望田K,斋正D,吉田T。et al。TriMEDB:一个数据库,整合转录标记和促进tribe Triticeae的遗传研究。BMC植物生物学8日,72(2008)。https://doi.org/10.1186/1471-2229-8-72
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-72
关键字
- 数量性状位点
- 水稻基因组
- 数量性状位点分析
- 聚合酶链反应引物
- 天生的重组