摘要
背景
阐明多细胞生物的代谢网络结构和功能是功能基因组学的一个新兴目标。我们描述了遗传模式植物中三个核心代谢过程的共表达网络拟南芥:脂肪酸生物合成、淀粉代谢和氨基酸(亮氨酸)分解代谢。
结果
这些共表达网络形成了由编码酶的基因组成的模块,这些酶代表了通常被认为是定义每种途径的反应。然而,这些模块也包含了一组更广泛的基因,编码转运蛋白、辅因子生物合成酶、前体产生酶和调节分子。我们通过实验验证了一个假设,即与淀粉代谢模块紧密共表达的基因之一,一个假定的激酶AtPERK10,将在这个过程中发挥作用。事实上,AtPERK10基因敲除系的淀粉积累发生了改变。此外,共表达数据定义了一种与分解代谢相关的新的分层转录水平结构,其中执行更小、更具体任务的基因似乎被招募到具有更广泛分解代谢功能的高阶模块中。
结论
这些核心代谢途径中的每一个都被构建为一个共表达转录本模块,这些转录本在广泛的环境和遗传扰动和发育阶段共同积累,并代表了与支持每个代谢途径功能的共同任务相关的一组扩展的大分子。实验证明,共表达分析可以为理解基因功能提供丰富的方法。
背景
生物系统的特点是它们在面对环境和发育信号时保持稳态的同时实现净代谢相互转换的能力。这种能力与生物体的遗传蓝图紧密相连,并表现为生物体基因组对不同信号和提示作出反应时遗传潜力的受控表达。控制有机体遗传潜能表达的机制包括那些调节基因转录、RNA加工、稳定性和翻译以及多肽加工及其组装成复合物的机制。这些复合物中的一些是酶催化剂,而另一些是结构或调节的。一个代谢网络从最广泛的意义上讲,它可以定义为包括催化、结构和调节基因的集合,这些基因表达为mrna、蛋白质和代谢物,协同工作以实现净代谢转换。
在过去十年中,功能基因组学领域取得的进展提供了在rna、蛋白质和代谢物水平上对全球基因组表达进行分析的能力。这些数据分别定义了转录组、蛋白质组和代谢组。从概念上讲,从实验数据中识别代谢网络是可能的,这些实验数据揭示了分子亚群(mrna、蛋白质/蛋白质复合物或代谢物)丰度的相关性。鉴于生物体的行为可以受到影响转录组、蛋白质组和代谢组的多种机制的调控,因此研究转录组可以在多大程度上揭示代谢网络是很重要的。在单细胞真核生物中酿酒酵母,其优势是细胞群同质,基因组相对简单,只有6,608个基因(SGD计划"Saccharomyces genome Database", 2008年1月13日[1]),代谢网络的运作,包括糖酵解和嘌呤代谢,仅从转录组数据集就已揭示[2- - - - - -4].但是,这种代谢网络能否在具有更大、更复杂基因组的生物中被检测到,或者在代谢网络的证据可能被与细胞分化相关的噪声淹没的多细胞生物中被检测到呢?
从表达数据中了解到的代谢过程的组织和已知代谢途径之间的对应关系的途径引导方法是选择途径的基因集,并在每个途径中寻找显著的共表达。该方法已用于揭示复杂生物中不同组织中属于同一代谢途径的基因的转录共表达,例如青蛙中的克雷布斯循环酶[5]或乳糖生物合成酶[6].另一种模块引导的方法是根据一组基因的表达模式对其进行分类,并研究这些集群与可识别的生物过程的对应关系。该方法用于揭示44个模块,对应于几种代谢途径秀丽隐杆线虫,包括脂质和氨基酸生物合成[7].
近年来,出现了丰富的表情数据,并创建了几个微阵列数据在线存储库,包括NASCArrays [8], genevarcheator [9],丛数据库[10], MetaOmGraph [11, ArrayExpress [12], Vanted [13]、虚拟植物[14], atted-ii [15],拟南芥共表达数据挖掘工具[16], MapMan [17],及PageMan [18].在不同条件下查询转录组的数据的可用性允许创建对许多扰动的生物反应的纲要,这可能指导微阵列实验的解释和新基因的注释[19- - - - - -22].由于在技术上已经可以分析一个生物体的整个转录组(确定蛋白质组和代谢组仍然是一个技术挑战),我们测试了仅使用转录组数据来揭示代谢网络在拟南芥中的运作的可行性。Gachon等的研究表明,拟南芥中吲哚、苯丙素和类黄酮生物合成酶的基因是共表达的[23].类似的共表达已被证明用于合成纤维素和其他细胞壁组分的酶[24- - - - - -26]和类异戊二烯生物合成途径[27].对AraCyc代谢途径中1330个拟南芥基因的转录协调性进行分析,发现同一途径基因之间的共表达高于不同途径基因之间的共表达[28].在最近的拟南芥基因网络分析中,根据表达谱纲要,一些代谢过程也可以被确定为相关的子网络[29].
在这里,我们重点研究了拟南芥的三个核心代谢途径:脂肪酸生物合成、淀粉代谢和亮氨酸分解代谢,并分析了它们在Affymetrix ATH1芯片上表达的22,746个基因的表达。我们的方法结合了路径引导和模块引导的方法,我们将预先选择的已知和假定的基因从路径中分组到模块中,然后将功能分配给模块中假定的候选基因。我们利用了这样一个假设,即网络中的一个节点可以被假设为具有与其某个半径附近的大多数节点相似的属性(例如,功能或位置,如果节点代表基因)。这一假设最近越来越受欢迎,因为它是为未知基因分配功能的一种方式[28,30.,31].从这些途径的基因中创建的共表达网络形成了模块,包括每个途径中反应的酶,表明了途径中催化步骤的扩展,也允许识别与模块相关的转运蛋白、辅因子和调控分子。事实上,在淀粉模块中鉴定出的一个基因已被独立地显示出来。32参与淀粉代谢的调节。我们使用敲除突变体来实验评估淀粉模块中第二个假定的调节基因。
结果与讨论
脂肪酸生物合成、淀粉代谢和亮氨酸分解代谢形成综合模块
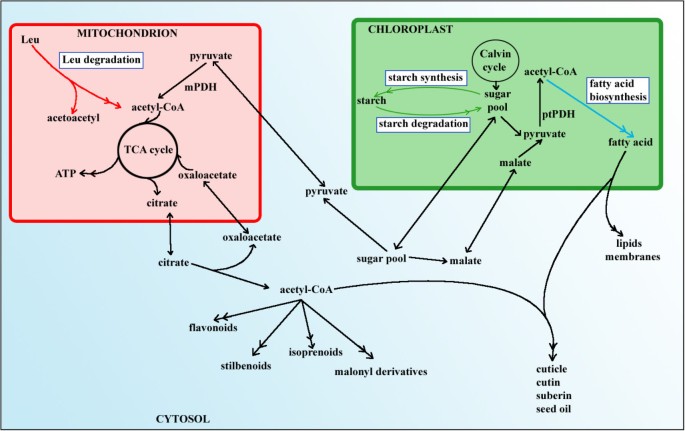
为了确定共表达网络如何在代谢过程中延伸,我们选择了三个核心代谢途径进行分析:脂肪酸生物合成、淀粉代谢和线粒体亮氨酸分解代谢(图2)。1).选择这些途径的部分原因是作者对其结构和生物学意义的广泛了解(例如,[33- - - - - -39])。脂肪酸生物合成是利用乙酰辅酶a生成酰基部分的过程,酰基部分用作细胞膜、信号分子和储存能量的分子(如种子油)的组成部分[40].淀粉是葡萄糖的主要聚合物储存形式,可在叶片中短暂储存,也可在种子中长期储存[41].亮氨酸分解代谢提供了乙酰辅酶a的替代来源,在没有光合作用的情况下维持呼吸和代谢过程[42].我们收集了一份在这三种代谢途径中具有证实或假定功能的基因列表;这些基因不仅编码“教科书途径”的酶,而且编码对合成辅助因子和转运蛋白很重要的酶,这些辅助因子和转运蛋白被认为参与了这些过程。具体来说,一组126个与这些途径相关的基因被确定基于以下三个标准之一:1)被实验证据证明属于这些途径之一的基因;2)基于序列相似性和亚细胞定位,在这些途径之一中赋予“假定”功能的基因;或3)编码转运蛋白和酶的基因,合成这些途径所需的辅因子[见附加文件]1].
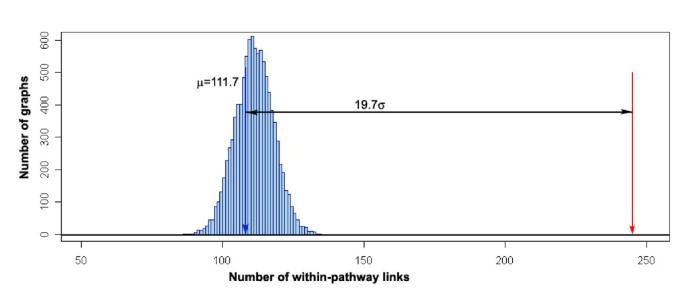
公开可用的Affymetrix ATH1芯片转录组数据集包括956个生物样本,用于推断该芯片上表达的22,746个拟南芥基因的转录本共积累模式(详情请参阅方法部分)。这些数据来自纳斯卡射线的72个实验[43]和PLEXdb [44],这代表了拟南芥的广泛发育、环境和遗传扰动[见附加文件2].丢弃再现性差的数据,将数据归一化到相同的均值和范围(可在MetaOmGraph [11])。我们计算了这些数据中126个选定基因的Pearson成对相关值。结果被可视化为一个图,这样基因形成节点,如果它们之间的相关性值高于选定的阈值,则由一条边连接。使用0.5、0.6和0.7的Pearson相关阈值可得到三个共表达网络,共表达网络由107、77和62个基因组成,分别由733、444和204条边连接。2).这些网络中节点(基因)的定位是由图形布局软件产生的,该软件将高度互联的节点紧密地放置在一起[45].因此,该节点在图上的绝对位置没有意义,只有它与其他节点的相对位置:最密集的节点表示共表达最高的基因。在这些共表达网络中,来自相同代谢途径的基因之间的相关性数量明显大于具有相似链接结构的随机生成网络(具体而言,在阈值为0.6的网络中,通路内边的比例为245/444,而在随机重新连接的网络中,其均值为112/444;这相当于20个标准差的差异;无花果。3.).随着相关阈值从0.5增加到0.7,出现了三个紧密相连的不同簇,其成员基因与本研究的三个代谢途径密切对应。我们指的是图中可识别的三个紧密连接的簇。2 b为模块。
三种核心代谢途径内基因的共表达.相关性高于阈值的实体用边连接;比较三个Pearson相关阈值下的网络:一个0.5;B0.6;而且C0.7点。随着相关阈值的增大,通路内连接由噪声较大的通路间连接逐渐变为通路内连接。节点颜色表示分配给每个基因的代谢功能(蓝色:脂肪酸合成,绿色:淀粉代谢,红色:亮氨酸分解代谢,黄色:转运或辅因子合成,白色:乙酰辅酶a生成)[见附加文件]1基因名称]。网络布局由GraphExplore软件生成。最密集的节点表示共表达量最高的基因。在这三个共表达网络中,来自相同代谢途径的基因之间的链接数量明显大于具有相似链接结构的随机生成网络。未显示孤立节点。
淀粉是一种复杂分子,其代谢由许多已知催化功能但生理功能不明确的基因组成的网络[46,47].事实上,有些基因可能同时在淀粉的分解代谢和生物合成中起作用(例如,普鲁兰酶[48])。在0.6相关水平下,淀粉代谢模块包含28个基因,编码所有已知的淀粉代谢酶。该模块包含编码蛋白质的基因,到目前为止,还没有实验证据表明这些蛋白质参与淀粉代谢,例如β -淀粉酶At2g32290 (BAM2)和三种磷酸葡萄糖酶样蛋白(At4g11570, PGM-like2;At1g70730, PGM-like3和At1g70820, PGM-like4)。另一方面,α -葡萄糖苷酶,一种历史上被认为参与淀粉降解的酶,尽管最近马铃薯的实验数据表明情况并非如此[49],与拟南芥淀粉代谢模块不相关(图;2 b).尽管在拟南芥叶片中,淀粉的合成和降解过程在昼夜周期中暂时分离(综述于[50];[51]),以及许多淀粉代谢基因的转录([52,53];Foster和Wurtele,未发表的数据),这没有反映在拟南芥的共表达网络中;相反,这两个过程集中在一个模块中。有两个因素可能特别有助于淀粉合成和降解转录物处于一个共同的模块。首先,拟南芥没有块茎等主要的淀粉储存器官,因此,具有高度淀粉合成能力的拟南芥器官也可能能够迅速转向淀粉降解,所有相关转录本的总体水平可能较高。事实上,抄本的日波动就是这种情况。其次,由于几种淀粉代谢酶似乎在淀粉的降解和合成中都起作用[48(事实上,这种说法的全部程度还不清楚),这些基因的表达可能与淀粉降解和淀粉合成有关。
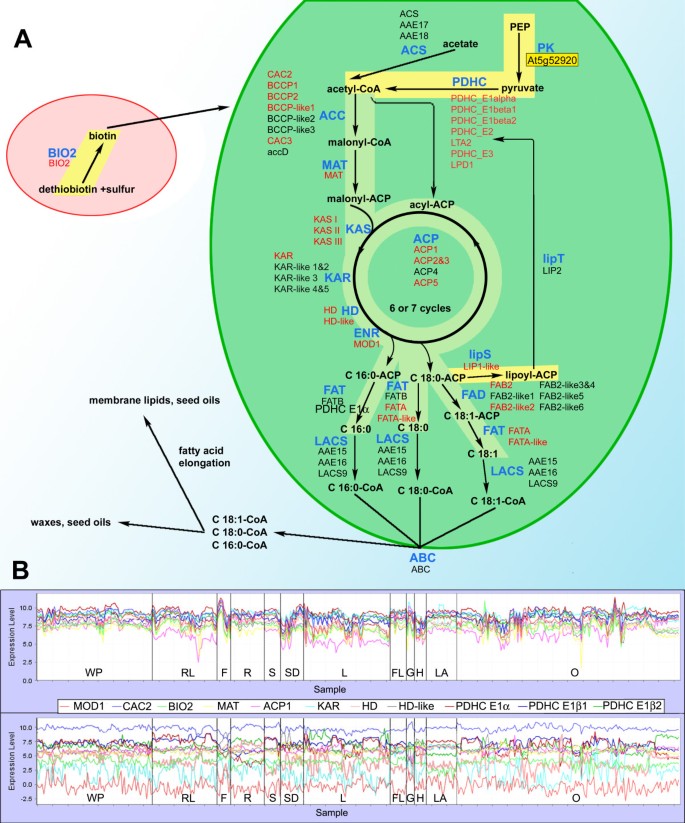
脂肪酸合成模块包含编码从乙酰辅酶a生物合成18碳脂肪酸所需的全套酶的基因:乙酰辅酶a羧化酶(CAC2、CAC3、BCCP1、BCCP2和BCCP1样)、丙二酰辅酶a:ACP转酰基酶(MAT)、酰基载体蛋白(ACP1、ACP2&3和ACP5)、β -酮酰-[ACP]合成酶(KAS I、KAS II和KAS III)、β -酮酰-[ACP]脱水酶(KAR)、β -羟基酰基-[ACP]脱水酶(HD和HD样)、烯酰-ACP还原酶(MOD1)、硬脂酰-[ACP]去饱和酶(FAB2和FAB2样)和酰基-[ACP]硫酯酶(FATA和FATA样)(图1)4).该模块还包含了从丙酮酸生成乙酰辅酶a(脂肪酸生物合成前体)的可塑性丙酮酸脱氢酶的四个亚基的所有基因。丙酮酸脱氢酶基因与脂肪酸生物合成的明显共表达可能表明乙酰辅酶a的主要代谢通量的方向,即使有其他可塑性乙酰辅酶a的命运(例如,硫代葡萄糖苷和氨基酸,如亮氨酸和半胱氨酸的生物合成)。有趣的是,该模块包含生物素生物合成中最终反应的基因[54- - - - - -56]和硫辛酸生物合成,分别为乙酰辅酶a羧化酶和丙酮酸脱氢酶的专性辅因子。相比之下,乙酰辅酶a合成酶(ACS)基因没有表现出任何表达相关性,这与该酶不需要为脂肪酸生物合成提供乙酰辅酶a的实验证据相一致[33,57,58].
脂肪酸生物合成基因的共表达.一个-脂肪酸在叶绿体中的生物合成。酶功能的候选基因(蓝色的酶名)用黑色或红色字体显示;红色位点ID的基因是共表达的。这些基因在图2B中形成连接网络,编码从乙酰辅酶a生物合成18碳脂肪酸所需的所有酶(浅绿色背景上的反应序列),以及产生乙酰辅酶a底物的基因,生物素和硫辛酸的生物合成基因,为乙酰辅酶a羧化酶和丙酮酸脱氢酶(浅黄色背景上的反应序列)专门的辅因子。丙酮酸激酶基因(黄色背景,通过搜索与MOD1共表达的基因识别),丙酮酸生产所需的基因,也与脂肪酸生物合成途径的基因共表达[见附加文件]1基因名和位点id之间的对应关系]。B-来自脂肪酸生物合成模块的11个最紧密共表达基因在956个微阵列芯片上的表达谱(上面板)和11个随机选择基因的表达谱(下面板)进行比较。组织和器官样本实验表示:WP,整株植物;RL:莲座叶;F、水果;R,根;年代,拍摄;SD、种子;L,叶;FL,花;G,雄配子体; H, hypocotyls; LA, leaf apex; O, other.美国广播公司,酰基转运体;ACC,异构体乙酰辅酶a羧化酶;ACS,乙酰辅酶a合成酶;生物圈二号,生物素合成酶;位,烯酰-[ACP]还原酶;时尚,脂肪酰基-[ACP]去饱和酶;脂肪,脂肪酰基-[ACP]硫酯酶;高清, 3-羟基酰基-[ACP]脱水酶;冰斗, 3-酮酰- [ACP]还原酶;内, 3-酮酰-[ACP]合酶;虫胶,质体长链酰基辅酶a合成酶;嘴唇,脂酸合成酶;lipT,脂酸转移酶;垫,丙二酰辅酶a:ACP转酰基酶;PDHC,丙酮酸脱氢酶配合物;PK,丙酮酸激酶。
亮氨酸降解模块包含线粒体亮氨酸分解代谢的四种已知酶的基因。5):支链氨基酸转氨酶(BCAT)、支链α -酮酸脱氢酶(BCDH)、异缬草酰辅酶a脱氢酶(IVD)和甲基巴豆酰辅酶a羧化酶(MCC)的四个亚基中的三个。模块中只包含一个BCAT (BCAT2, At1g10070)。这个基因不是Schuster和Binder所假设的[59]参与亮氨酸的降解。与这些结果一致的是,我们模块中的8个基因(IVD除外)中有7个基于偏相关在拟南芥基因网络中形成了一个相干的子网络[29].
在所有基因转录本的空间中可以识别模块
为了测试这些途径特异性模块是否可以在整个基因组的表达中被检测到,并确定编码途径催化功能之外的基因是否被共同调控,我们计算了单个途径特异性枢纽基因与Affymetrix芯片上表示的22,746个基因之间的Pearson相关性。用作每个代谢途径枢纽的基因有:At2g05990,编码enyyl - acp还原酶,这是在新创脂肪酸生物合成[60];歧化酶编码淀粉代谢中枢基因At2g40840 [61];以及亮氨酸分解代谢中心基因At4g34030,该基因编码甲基巴豆酰辅酶a羧化酶β亚基[39,42)(表1).
尽管大约32000个基因的基因组相关的复杂性(TAIR7基因组发布,2007年4月23日[62]),并且与用于微阵列分析的rna来自混合了代谢不同的细胞和组织区室的植物样本相关的挑战,仍然有可能找到共同代谢途径的基因之间的表达相关性。具体来说,大多数与脂肪酸生物合成中心基因、淀粉代谢中心基因和亮氨酸分解代谢中心基因表现出最高相关性的基因是为这些代谢途径中所需的酶编码的结构基因(表2)1).类似地,对于模块中的每个基因,我们在ATH1阵列的22,746个基因中找到了与之相关的基因(高于设置的阈值),并检查了这些相关基因中有多少也在同一个模块中(“in”链接),有多少不在模块中(“out”链接)。我们发现,在0.7阈值时,脂肪酸生物合成模块22个基因中有19个基因的“内”链数高于“外”链数,在0.8阈值时,8个亮氨酸分解代谢基因中有7个,16个淀粉代谢基因中有9个。因此,这些基因之间优先相互连接,与其他基因的连接较少,这将它们定义为拟南芥基因组中的一个模块。
对共表达的分析也提供了查询基因组复制结果的方法。与其他植物基因组一样,拟南芥经历了全基因组复制(多倍体化),随后是激进基因沉默、基因翻转和基因家族的扩张,最近的一次被认为发生在大约2500万年前[63,64].在我们的分析中,14个拟南芥丙酮酸激酶编码基因之一的At5g52920与脂肪酸生物合成中心基因高度相关(表5g52920)1).丙酮酸激酶催化磷酸烯醇丙酮酸转化为丙酮酸,丙酮酸是用于脂肪酸生物合成的乙酰辅酶a的直接前体[65].因此,这些数据有助于限制与多倍体化相关的复杂性,并表明在基因组中出现的14个丙酮酸激酶基因中,At5g52920最有可能主要用于脂肪酸生物合成。
此外,丙酮酸脱氢酶和丙酮酸激酶与脂肪酸生物合成基因的共表达意味着通常被认为是“脂肪酸合成”的功能途径(乙酰辅酶a到脂肪酸)可以扩展到丙酮酸激酶的底物磷酸烯醇丙酮酸(PEP)开始(图2)。4).
与代谢途径共表达的潜在调控基因
一组潜在的调控基因,其表达模式与每个通路特异性中枢基因高度相关,在该分析中被确定(基因标记为星号,表1).虽然这些相关性不能推断出因果关系,但候选调节基因的鉴定可以描述假说,并为鉴定这些基因的调节功能提供实验方法。
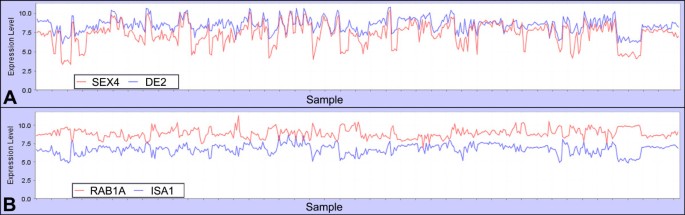
这样一个假定的调节基因编码蛋白酪氨酸磷酸酶/激酶基因SEX4 (At3g52180)(表1和无花果。6).它在这个模块中的存在将导致我们假设参与淀粉代谢的调节。事实上,这个基因最近被实验证明在淀粉代谢中有调节作用;At3g52180基因敲除等位基因具有淀粉过剩表型[32].类似地,一个基因编码一个假定的蛋白激酶(At1g26150, AtPERK10 [66])与淀粉代谢模块中的基因相关。我们直接检查了该基因是否可能在调节淀粉合成中发挥作用。
我们获得了三个独立的等位基因,分别命名为Atperk10 -1,Atperk10-2,Atperk10-3,对应于基因AtPERK10位点的T-DNA插入。用Lugol溶液为淀粉染色的纯合突变系显示淀粉积累增加(数据未显示)。数字7对野生型和野生型淀粉含量进行了定量比较Atperk10-3等位基因的纯合子。
有趣的是,淀粉中心基因与两个rablike gtpase编码基因(At5g47200;RAB1A和At4g17530;RAB1C;无花果。6 b).在真核生物中,来自这个小gtp酶家族的蛋白质在不同的过程中起调控作用并充当信号分子[67].拟南芥中与rablike GTPase最接近的同源蛋白是RAB1B (At1g02130),定位于内质网,与哺乳动物系统中一样,它对高尔基体的发育很重要[68].RAB1A和RAB1C被预测具有分泌功能,可能具有与RAB1B类似的功能,或者具有某种新的植物特异性调节功能,其似乎与淀粉代谢负相关。
脂肪酸合成模块包含含有pentatricopeptide repeat的蛋白质(由At5g50390编码)。因为这类蛋白质被认为参与调控细胞器转录本的稳定性[69],这种相关性可能表明该基因在协调核-塑性相互作用以调节脂肪酸生物合成方面的作用,例如,通过控制乙酰辅酶a羧化酶的质体编码亚单位基因(accDAtCg00500)。这是最直接参与脂肪酸生物合成的质体编码生化功能。
亮氨酸分解代谢模块包含一个假定的转录调节因子At5g49450,它可能编码bZIP家族转录因子。
分解代谢的层次模块化
这些转录组水平的分析为拟南芥代谢的层次模块化提供了新的见解。分层组织可以被认为是一个紧密连接的模块嵌套在一个更大的模块中,仍然与周围的网络不同且可分离。亮氨酸分解代谢的层次结构尤其明显,它形成了一个功能一致且紧密连接的模块,嵌套在一个超模块中[见图]。8和附加文件3.].
已经观察到代谢和转录网络模块的分层组织大肠杆菌[70- - - - - -72]和42种其他生物的代谢网络[73,74].层次模块化组织被假定为解释生物网络观察到的无标度特征、高聚类系数和短路径长度等特征的拓扑[74,75].这种结构可使代谢通量更好地适应不断变化的条件[74].
对于拟南芥,在拓扑和功能水平上观察到亮氨酸分解代谢纳入整体分解代谢模块。从拓扑的角度来看,参与亮氨酸分解代谢的基因形成了一个完全连接的子网络,同时它们也表现出与共同的较大基因集的较弱连接。因此,一个较小的、连接较强的模块嵌套在较大的、连接较弱的模块中。从功能的角度来看,一个功能(亮氨酸分解代谢)形成了一个更大的、更广泛的生物过程的可分离部分。超级模块包含的基因似乎具有更广泛但常见的生物任务,即通过分解代谢维持细胞能量平衡。具体来说,26个基因与亮氨酸分解代谢模块中的8个基因高度相关(定义在图中)。2 b)包括可能参与蛋白质转换(At1g76410)、氨基酸分解代谢(At1g06570、At1g08630、At2g14170、At3g47340、At5g63620,以及亮氨酸分解途径的8个基因)、碳水化合物分解代谢(At1g18270、At5g20250、At5g49360)、脂质分解(At3g51840、At5g41080)和细胞壁降解(At4g30270)的基因。5个基因被注释为与衰老、衰老或对光或蔗糖刺激的反应相关(At2g43400, At3g47340, At4g30270, At4g35770, At5g20250)。由于植物是光自养生物,当光合作用不能维持细胞能量平衡时,例如在衰老、种子萌发、碳剥夺或自噬过程中,这种分解代谢过程可能会变得至关重要[37,39,42,76].与这一概念一致,我们发现这个超级模块中的基因在实验条件下被共同诱导到最高水平的表达,这些条件预计会限制光合作用碳固定,并诱导分解代谢过程以维持能量平衡(例如,氧化,冷,热和干旱胁迫-来自NASCArrays的实验:nascarray -28, nascarray -79, nascarray -70;质体-核体通信中断,nascarray -89;影响光敏色素反应的突变和光照条件,nascarray -89, nascarray -124;蔗糖饥饿,Contento等人。[76]和nascarray -14;和己糖信号,nascarray -40 [8])。
该分解代谢超级模块还包含具有明确或部分定义的分子功能,但生理功能不明确的基因(如At1g76410,锌指蛋白;At5g21170,蛋白激酶;At5g16340, amp结合蛋白)。这些基因在这个分解代谢超级模块中的包含可能表明它们在分解代谢过程中具有更广泛的生理功能;这一假设可以通过新的遗传和生化分析进行实验验证。
结论
这项研究揭示了转录组学数据可以揭示复杂真核生物代谢网络迄今为止未被怀疑的方面。这些被分析的核心代谢途径中的每一个都被构建为一个共表达模块,其转录本在广泛的环境和遗传扰动和发育阶段共同积累。这些分析进一步表明,模块可以被招募到分层组织(超级模块)中,这暗示了超级模块是由高度交互网络中协调净代谢变化的共同信号调节的可能性。从转录组数据中检测到这种分层组织的事实表明,至少有一部分高阶代谢网络可能在转录水平上受到调控。
方法
转录组数据
实验数据和元数据来自在线微阵列存储库:NASCArrays [43]和PLEXdb [44].从NASCArrays获得的数据,来自72个实验,包括956个Affymetrix ATH1微阵列幻灯片,已经用Affymetrix MAS 5.0算法归一化到100的共同平均值。我们缩放了来自PLEXdb的数据,将平均值设置为100。
通过目视检查在散点图上对实验的再现性进行定性评估,丢弃生物学重复较差的芯片。剩余的生物重复平均得到424个样本。基于载玻片名称和实验信息,人工确定生物重复(实验中相同处理的重复)。大多数幻灯片都有描述性的名称,总结治疗和复制状态。因此,作为同一处理的生物复制的载玻片具有相同的载玻片名称,仅在“Repx后缀,例如,
Shirras_1-3_Non-Calcicole_Rep1
Shirras_1-4_Non-Calcicole_Rep2
随后,通过Yang等人描述的基于中值绝对偏差(MAD)的尺度归一化方法,将数据归一化到相同的范围,[77].表达式的值xij微阵列芯片j乘以这个因子,在那里疯了定义为
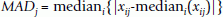
常数C是的算术平均值疯了
标准化数据可在线获取[11].ATH1探针set- locus ID mapping于3月31日从TAIR获得圣2007年(78,79].实验信息包含在附加文件中2.
通道数据
各途径所涉及基因的选择由:拟南芥脂肪酸合成基因脂质基因数据库[80,81];拟南芥淀粉代谢网络项目[82]促进淀粉代谢;亮氨酸分解代谢的原始参考文献[42,83,84].我们在原始参考文献的基础上进一步考虑每个候选基因。
共表达基因网络
用于初始网络构建的126个基因(图;2),包括脂肪酸合成、淀粉代谢和亮氨酸分解代谢的候选功能,在附加文件中显示1.使用皮尔逊积矩系数计算这些基因的相关矩阵,定义为
Pearson相关性值高于0.3具有统计学高度显著性(多次试验Bonferroni校正后p值< 0.00001 [85])。我们还通过对相应数据向量进行10,000次排列来评估高于0.6的相关性的有效性。具有基于排列的p值< 0.0001的相关性(在我们的情况下,检测的所有基因对)被考虑进行进一步分析[见附加文件]4].图中的网络。2通过在高于阈值的相关基因对之间放置一条边来构建。通过网络可视化软件生成的分组来定义网络中的模块。
共表达网络中通路内边的富集
为了测试来自同一代谢途径(即脂肪酸生物合成、淀粉代谢或亮氨酸分解代谢类别)的基因之间是否存在比偶然预期更多的相关性,我们比较了来自实验数据的共表达网络(图2)。2 b)到10,000个计算生成的随机网络。在这些随机网络中,每个节点的边的总数和邻居的数量被设置为与原始图中相同。在r语言中实现了一个生成随机图的函数,通过随机填充邻接矩阵来构造图,其中节点的度是有约束的,不允许自循环。事实上,在我们的共表达网络中,通路内边的比例明显高于随机图中的比例(245/444对平均值112/444,这等于约20个标准差的差异,图。3.).
与Hub基因的相关性
计算三个中心基因(At2g40840, At4g34030, At2g05990)与芯片上所有其他基因之间的Pearson相关性(表21)使用我们的公开软件MetaOmGraph [11].MetaOmGraph能够有效利用内存处理大型数据集,并与MetNet Exchange套装中其他公开可用的生物信息学工具集成[86,87].
属于分解代谢超模块的基因[见图]。8和附加文件3.]通过与来自亮氨酸分解代谢模块的8个基因中的每一个在0.5阈值以上相关的所有基因的交叉列表来识别。
植物材料
三个独立的等位基因命名Atperk10-1,Atperk10-2,Atperk10-3在INRA-Versailles产生的突变体集合中发现了与AtPERK10基因位点上的T-DNA插入相对应的[88,89], gabi-kat [90,91]和索尔克[92,93].T-DNA分别插入5'-UTR、外显子1和内含子6。
淀粉量化
两株植物(100 - 400mg鲜重)在50ml 80% (v/v)乙醇中煮沸。然后将脱色的植物用2 ml Konte组织研磨机在80%乙醇中研磨,在室温下3000 g离心10分钟。用80%乙醇清洗颗粒两次。离心后,将不溶性物质悬浮在1毫升蒸馏水中,煮沸30分钟。使用商业Glc检测试剂盒(目录编号:;E0207748;R-Biopharm, Darmstadt, Germany),根据试剂盒方案中的说明。该方法利用淀粉葡萄糖苷酶将淀粉样品完全消化成Glc,并通过Glc氧化酶试剂的应用来测量Glc。计算了6个生物重复的平均值,每个重复使用2个单株。
软件
计算(归一化、相关矩阵、基于排列的p值、随机图生成、超几何分布)在R软件中进行[94].RNA谱图在MetaOmGraph中绘制。图中代谢基因的网络。2使用GraphExplore进行可视化[95].路径数据来自MetNetDB数据库。R代码可根据要求提供。
参考文献
酵母基因组数据库。[http://www.yeastgenome.org/]
马格温尼,金J:利用一阶条件独立性估计基因组共表达网络。中国生物医学工程学报,2004,5:R100-
Segal E, Shapira M, Regev A, Pe er D, Botstein D, Koller D, Friedman N:模块网络:从基因表达数据中识别调控模块及其条件特异性调控因子。王志强,2003,30(4):366 - 366。
Segal E, Yelensky R, Koller D:从DNA序列和基因表达中转录模块的全基因组发现。生物信息学,2003,19:i273-282。
Baldessari D, Shin Y, Krebs O, Konig R, Koide T, Vinayagam A, Fenger U, Mochii M, Terasaka C, Kitayama A, Peiffer D, Ueno N, Eils R, Cho KW, Niehrs C:基因表达谱和聚类分析非洲爪蟾蜍光滑的.机械开发,2005,122:441-475。
Zhang W, Morris QD, Chang R, Shai O, Bakowski MA, Mitsakakis N, Mohammad N, Robinson MD, Zirngibl R, Somogyi E, Laurin N, Eftekharpour E, Sat E, Grigull J, Pan Q, Peng WT, Krogan N, Greenblatt J, Fehlings M, van KD, Aubin J, Bruneau BG, rosant J, Blencowe BJ, Frey BJ, Hughes TR:小鼠基因表达的功能景观。中国生物医学杂志,2004,3:21-
Kim SK, Lund J, Kiraly M, Duke K, Jiang M, Stuart JM, Eizinger A, Wylie BN, Davidson GS:基因表达图谱秀丽隐杆线虫.科学通报,2001,29(3):357 - 357。
NASCArrays。[http://affymetrix.arabidopsis.info/narrays/experimentbrowse.pl]
Genevestigator。[https://www.genevestigator.ethz.ch/]
PLEXdb。[http://plexdb.org/]
MetaOmGraph。[http://metnet.vrac.iastate.edu/MetNet_MetaOmGraph.htm]
ArrayExpress。[http://www.ebi.ac.uk/arrayexpress/]
Vanted。[http://vanted.ipk-gatersleben.de/]
虚拟工厂。[http://virtualplant-prod.bio.nyu.edu/cgi-bin/virtualplant.cgi]
ATTED-II。[http://www.atted.bio.titech.ac.jp/]
拟南芥共表达数据挖掘工具。[http://www.arabidopsis.leeds.ac.uk/act/]
MapMan。[http://gabi.rzpd.de/projects/MapMan/]
PageMan。[http://mapman.mpimp-golm.mpg.de/pageman/]
Hughes TR, Marton MJ, Jones AR, Roberts CJ, Stoughton R, Armour CD, Bennett HA, Coffey E, Dai H, He YD, Kidd MJ, King AM, Meyer MR, Slade D, Lum PY, Stepaniants SB, Shoemaker DD, Gachotte D, Chakraburtty K, Simon J, Bard M, Friend SH:通过表达简介的功能发现。细胞,2000,102:109-126。
Lamb J, Crawford ED, Peck D, Modell JW, Blat IC, Wrobel MJ, Lerner J, Brunet JP, Subramanian A, Ross KN, Reich M, Hieronymus H, Wei G, Armstrong SA, haggonymus SJ, Clemons PA, Wei R, Carr SA, Lander ES, Golub TR:连通性图:使用基因表达签名连接小分子、基因和疾病。科学通报,2006,27(3):329 - 329。
Nielsen HB, Mundy J, Willenbrock H:响应重叠(FARO)的功能关联,一种匹配基因表达表型的功能基因组学方法。PLoS ONE。2007, 2: e676-
Weston DJ, Gunter LE, Rogers A, Wullschleger SD:植物环境胁迫表型的基因、共表达模块和分子特征的连接。中国生物医学工程学报。2008,2:16-
Gachon CM, Langlois-Meurinne M, Henry Y, Saindrenan P:拟南芥次生代谢酶的转录协同调控:功能和进化意义。植物化学学报,2005,28(3):329 - 329。
Brown DM, Zeef LA, Ellis J, Goodacre R, Turner SR:利用表达谱和逆向遗传学鉴定拟南芥次级细胞壁形成的新基因。中国生物医学工程学报,2005,27(3):344 - 344。
王晓明,王晓明,王晓明,王晓明。基于微阵列数据集的纤维素合成基因筛选。自然科学进展,2005,32(3):344 - 344。
Usadel B, Kuschinsky AM, Steinhauser D, Pauly M:转录共反应分析作为识别壁生物合成机制新组分的工具。中国生物工程学报,2005,29(3):344 - 344。
Wille A, Zimmermann P, Vranová E, Fürholz A, Laule O, Bleuler S, Hennig L, Prelic A, von Rohr P, Thiele L, Zitzler E, Gruissem W, Bühlmann P:中异戊二烯类基因网络的稀疏图形高斯建模拟南芥.中国生物医学工程学报,2004,5:R92-
陈琳,陈丽娟,陈丽萍,陈丽萍,陈丽萍,陈丽萍,陈丽萍拟南芥.植物科学学报,2006,29(4):344 - 344。
马珊珊,龚强,洪杰:基于图形高斯模型的拟南芥基因网络。基因组学报,2007,17:1614-1625。
许维科斯基B, Uetz P, Fields S:酵母蛋白相互作用网络。中国生物工程学报,2000,18:1257-1261。
Schlitt T, Palin K, Rung J, Dietmann S, Lappe M, Ukkonen E, Brazma A:从基因网络到基因功能。基因组学报,2003,13:2568-2576。
Niittyla T, comparoto - moss S, Lue WL, Messerli G, Trevisan M, Seymour MD, Gatehouse JA, Villadsen D, Smith SM, Chen J, Zeeman SC, Smith AM:相似蛋白磷酸酶控制植物淀粉代谢和哺乳动物糖原代谢。中国生物医学工程学报,2006,29(2):344 - 344。
柯杰,黄志刚,李志刚,李志刚,李志刚:丙酮酸脱氢酶和乙酰辅酶A合成酶在拟南芥种子脂肪酸合成中的作用。中国生物医学工程学报,2000,29(3):349 - 349。
柯建军,温廷敏,吴淑娟,王晓明,王晓明。异聚体乙酰辅酶a羧化酶核和叶绿体编码基因时空表达的坐标调控。植物科学学报,2002,22(3):357 - 357。
吴淑娟,王志强,王志强,等:植物3-甲基巴豆酰辅酶a羧化酶的鉴定。方法酶学杂志,2000,324:280-292。
陈建平,张志刚,张志刚,张志刚:生物素对拟南芥3-甲基巴豆酰辅酶a羧化酶表达的调控作用。植物科学进展,2003,31(4):349 - 349。
张志刚,张志刚,张志刚,等:拟南芥3-甲基巴豆酰辅酶A羧化酶表达的代谢和环境调控。植物科学学报,2002,29(3):366 - 366。
王志强,王志强,王志强,等:植物生物素羧化酶。生物化学学报,2003,29(3):344 - 344。
王志强,王志强,王志强,王志强,王志强,王志强,王志强。3-甲基巴豆酰辅酶a羧化酶亚基的分子特性研究。中国生物医学工程学报,2000,27(3):357 - 357。
脂质生物合成。中国生物医学工程学报,2004,27(3):357 - 357。
Gibon Y, Blasing OE, palacioss - rojas N, Pankovic D, Hendriks JH, Fisahn J, Hohne M, Gunther M, Stitt M:调节日间淀粉周转:夜间糖的消耗导致碳水化合物利用的暂时抑制,糖的积累和adp -葡萄糖焦磷酸化酶的翻译后激活在接下来的光照期。植物学报,2004,39:847-862。
杨晓明,陈晓明,王晓明,王晓明,王晓明,等:3-甲基巴豆酰辅酶A羧化酶是植物线粒体亮氨酸分解代谢途径的组成部分。植物科学学报,1998,18(3):344 - 344。
克根·DJ,詹姆斯·N,奥克耶尔,希金斯,乔瑟姆·J,梅:NASCArrays:由NASC转录组服务生成的微阵列数据存储库。中国生物医学工程学报,2004,32:D575-577。
沈玲,龚娟,陈志强,李志强,李志强,李志强,陈志强。植物基因组学表达分析数据库的研究进展。中国生物医学工程学报,2005,33:D614-D618。
Fruchterman T, Reingold E:力指向放置的图形绘制。软件技术与应用。2001,29(4):379 - 379。
Myers AM, Morell MK, James MG, Ball SG:理解支链淀粉晶体生物合成的最新进展。植物科学学报,2002,22(3):489 - 497。
史密斯AM,塞曼SC,史密斯SM:淀粉降解。植物学报,2005,29(3):344 - 344。
丁格斯,柯洛尼·C,詹姆斯·MG,迈尔斯·AM:玉米普鲁兰酶型去分支酶的突变分析表明其在淀粉代谢中具有多种功能。中国生物医学工程学报,2003,29(3):366 - 366。
Taylor MA, Ross HA, McRae D, Stewart D, Roberts I, Duncan G, Wright F, Millam S, Davies HV:马铃薯α -葡萄糖苷酶基因编码糖蛋白处理α -葡萄糖苷酶ii类活性。转基因植物中酶活性及下调效应的研究。植物学报,2000,24(3):344 - 344。
王晓明,王晓明,王晓明:植物淀粉的生物合成与降解。植物化学与分子生物学学报,2004,19(4):379 - 379。
王晓明,王志强,王志强,王志强,王志强,王志强:拟南芥淀粉合成的研究进展。颗粒的合成、组成和结构植物学报,2002,29(4):516-529。
Smith SM, Fulton DC, Chia T, Thorneycroft D, Chapple A, Dunstan H, Hylton C, Zeeman SC, Smith AM:拟南芥叶片淀粉代谢编码酶的转录组日变化为转录和转录后调节淀粉代谢提供了证据。植物科学进展,2004,26(1):379 - 379。
Blasing OE, Gibon Y, Gunther M, Hohne M, Morcuende R, Osuna D, Thimm O, Usadel B, Scheible WR, Stitt M:糖和昼夜节律调节对拟南芥昼夜基因表达的全局调控有重要贡献。植物学报,2005,17:3257-3281。
王文杰,王文杰,王文杰,王文杰。植物生物素合成酶基因编码的研究进展拟南芥.植物科学学报,1996,15(3):344 - 344。
巴顿DA,约翰逊M,沃德ER:生物素合成酶来自拟南芥.cDNA的分离与表达特性。植物营养学报,2004,27(1):366 - 366。
Baldet P, Ruffet ML:高等植物生物素合成:cDNA编码的分离拟南芥生物素营养不良突变体bioB105的功能互补等效的生物b基因产物大肠杆菌K12的。学院科学III。1996, 319(2): 99-106。
鲍X,福克M,波拉德M, Ohlrogge J:理解在活的有机体内叶组织中脂肪酸合成的碳前体供应。植物学报,2000,22:39-50。
黄志刚,林敏,李志刚,李志刚:乙酰辅酶A合成酶在植物叶片中的作用拟南芥.生物化学学报,2002,29(4):357 - 357。
Schuster J, Binder S:线粒体支链转氨酶(AtBCAT-1)能够启动人体几乎所有组织中亮氨酸、异亮氨酸和缬氨酸的降解拟南芥.植物化学学报,2005,29(4):344 - 344。
牟志,何颖,戴艳,刘霞,李娟:脂肪酸合成酶缺乏导致植物细胞过早死亡和形态的显著改变。中国生物医学工程学报,2000,29(4):344 - 344。
Lu Y, Sharkey TD:淀粉酶在光合细胞细胞液中麦芽糖代谢中的作用。植物学报,2004,29(3):366 - 366。
TAIR。[http://www.arabidopsis.org/]
Blanc G, Wolfe KH:从复制基因的年龄分布推断模式植物中广泛的古多倍体。植物科学学报,2004,16:1667-1678。
李文杰,李文杰:植物多倍体与基因组进化。中国植物学报,2005,30(4):531 - 531。
彭志强,李志强,李志强:白质丙酮酸激酶的分子调控特性芸苔属植物显著(油菜籽)悬浮细胞。生物化学学报,2002,29(4):344 - 344。
赵珍,赵志刚,赵志刚,赵志刚,赵志刚,赵志刚。拟南芥富脯氨酸伸展素样受体激酶基因家族的生物信息学和实验分析。植物生理学报,2004,30(4):344 - 344。
西蒙斯M,高井Y: Ras GTPases:唱歌调。Sci抽烟可以。2001, 2001: pe1 -
杨志刚,杨志刚,杨志刚:花粉管中Rho家族GTPase通过两种拮抗下游途径控制肌动蛋白动态和顶端生长。中国生物医学工程学报,2005,29(3):344 - 344。
Nakamura T, Schuster G, Sugiura M, Sugita M:叶绿体rna结合和五磷酸肽重复蛋白。生物化学学报,2004,32:571-574。
马华文,布尔杰,曾爱平:网络科学中的层次结构与模块大肠杆菌一种新的自上而下的方法揭示了转录调节网络。中国生物医学工程学报,2004,27 (3):344 - 344
李志刚,李志刚,李志刚,李志刚。大肠埃希氏菌转录网络进化的层次结构和反馈机制。中国科学院学报(自然科学版),2007,27(1):1 - 7。
Sales-Pardo M, Guimerà R, Moreira AA, Amaral LA:提取复杂系统的层次组织。中国科学:自然科学,2007,29(1):1 - 7。
奥尔泰·ZN, Barabási AL:生命的复杂性金字塔。科学通报,2002,29(4):344 - 344。
王晓峰,王晓峰,王晓峰,王晓峰,Barabási AL:代谢网络模块化的分层组织。科学通报,2002,29(3):457 - 457。
Clauset A, Moore C, Newman ME:层次结构和网络中缺失环节的预测。自然科学,2008,453:98-101。
王志刚,王志刚,王志刚,等:拟南芥悬浮培养细胞对Suc饥饿应答的转录组分析。植物科学学报,2004,26(3):329 - 329。
杨永华,刘平,林德明,彭伟,倪娟,陈晓娟,陈晓明:cDNA微阵列数据归一化的研究进展。中国生物医学工程学报,2002,30:e15-
Rhee SY, Beavis W, Berardini TZ, Chen G, Dixon D, Doyle A, Garcia-Hernandez M, Huala E, Lander G, Montoya M, Miller N, Mueller LA, Mundodi S, Reiser L, Tacklind J, Weems DC, Wu Y, Xu I, Yoo D, Yoon J, Zhang P:拟南芥信息资源(TAIR):一个模式生物数据库,为拟南芥生物学、研究材料和群落提供集中的、有管理的门户。中国生物医学工程学报,2003,31(4):344 - 344。
Beisson F, Koo AJ, Ruuska S, Schwender J, Pollard M, Thelen JJ, Paddock T, Salas JJ, Savage L, Milcamps A, Mhaske VB, Cho Y, Ohlrogge JB:拟南芥酰基脂代谢相关基因。2003年对候选基因的普查,器官中表达序列标签分布的研究,以及基于网络的数据库。植物科学学报,2003,32(3):366 - 366。
拟南芥脂质基因数据库。[http://lipids.plantbiology.msu.edu/]
拟南芥淀粉代谢网络。[http://www.starchmetnet.org/]
杜丽娟,李志强,李志强,李志强,等:拟南芥中支链氨基酸转氨酶基因家族的研究进展。植物学报,2002,29(4):349 - 349。
Taylor NL, Heazlewood JL, Day DA, Millar AH:硫辛酸依赖的α -酮酸在线粒体中的氧化分解代谢为拟南芥支链氨基酸分解代谢提供了证据。中国农业科学,2004,27(4):344 - 344。
Bonferroni CE: Teoria statistica delle classe calcolo delle probability。《佛罗伦萨高等经济商业科学研究所》,1936,8:3-62。
MetNet交换。[http://metnet.vrac.iastate.edu/]
吴特莱,李娟,张慧,张华,福斯特CM, Fatland B, Dickerson J, Brown A, Cox Z, Cook D, Lee EK, Hofmann H:拟南芥生物遗传晶格的构建与建模。中国生物医学工程学报,2003,23(4):349 - 349。
INRA-Versailles。[http://dbsgap.versailles.inra.fr/publiclines/informations.php]
Samson F, Brunaud V, Balzergue S, Dubreucq B, Lepiniec L, Pelletier G, Caboche M, Lecharny A: FLAGdb/FST:的侧翼插入点(FSTs)映射数据库拟南芥本文分别转化株。中国生物医学工程学报,2002,30:94-97。
GABI-KAT。[http://www.gabi-kat.de]
罗索MG,李Y, Strizhov N, Reiss B, Dekker K, Weisshaar B: a拟南芥T-DNA诱变种群(GABI-Kat)用于侧翼序列标记的逆向遗传。中国生物医学工程学报,2003,23(3):357 - 357。
Leisse TJ,阿隆索JM,斯金CJ,陈H,希恩P,史蒂文森DK,齐默尔曼J,巴拉哈斯P, Cheuk R, Gadrinab C,海勒C, Jeske, Koesema E,迈耶斯CC,帕克H, Prednis L,安萨里Y,白菜N,迪恩H, Geralt M, Hazari N, Hom E,圆锥形石垒,穆赫兰C, Ndubaku R,施密特,古兹曼P, Aguilar-Henonin L,施密德M,魏盖尔D,卡特德玛珊德T, Risseeuw E, Brogden D,泽柯,克罗斯比西城,贝瑞CC,艾克尔JR:全基因组的插入突变拟南芥.科学通报,2003,30(3):344 - 344。
R开发核心团队:R统计计算基金会,2004年,奥地利维也纳
GraphExplore。[http://graphexplore.cgt.duke.edu]
确认
作者想对拟南芥科学界通过NASC和PLEXdb数据库提供他们的微阵列表达数据表示感谢。我们感谢D. Cook和D. Nettleton对微阵列数据规范化的帮助,感谢MetNet小组的建议和促进讨论,感谢Susan Blauth对微阵列数据进行了有益的讨论和初步描述Atperk10-3纯合突变体。我们特别感谢A. Myers, L. Li, C. Foster和M. James在淀粉代谢方面非常有用的专业知识。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
WIM设计了这项研究,进行了生物信息学分析并起草了手稿。JP进行突变体筛选和淀粉染色定量。NR实现了MetaOmGraph软件进行分析。BJN对数据的解释以及对手稿的批判性修改和写作做出了贡献。ESW协调了这项研究,并为其设计、解释结果和撰写手稿做出了贡献。所有作者都阅读并批准了最终的手稿。
电子辅助材料
12870 _2008_284_moesm1_esm.xls
附加文件1:研究中使用的基因.表中列出了在研究中使用的基因的名称、Affy id、位点id和参考文献。在0.6相关阈值处形成共表达模块的基因(图;2 b)显示在颜色背景上(蓝色:脂肪酸生物合成;红色:亮氨酸分解代谢;绿色:淀粉代谢)。(xl113kb)
12870 _2008_284_moesm3_esm.doc
附加文件3:含有亮氨酸分解代谢途径的超模块中的基因.表中列出了超模块中含有亮氨酸分解代谢途径的基因(如图所示)。8),被识别为与亮氨酸分解代谢模块中的8个基因中的每一个相关超过0.5阈值的所有基因列表的交集。(doc 48 kb)
12870 _2008_284_moesm4_esm.doc
附加文件4:共表达网络中基于排列的相关性支持.图显示了图中126个基因共表达网络中基于排列的相关性支持。2 b.对于Pearson相关性高于0.6的每一对基因,该真实相关值(红星)将与相应表达数据载体的10,000个排列中获得的相关值分布进行比较(绿星,最大值;黑星,意思;蓝星,最小值;橙色条,上下铰链之间的值)。(doc 1mb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
门岑,W.I,彭,J,兰森,N。et al。拟南芥三大核心代谢过程:脂肪酸生物合成、亮氨酸分解代谢和淀粉代谢。BMC植物生物学8日,76(2008)。https://doi.org/10.1186/1471-2229-8-76
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-76
关键字
- 丙酮酸激酶
- 脂肪酸生物合成
- 淀粉合成
- 绝对偏差中位数
- 淀粉代谢