摘要
背景
在植物细胞器中,特定的信使rna (mRNAs)受到转换编辑,这一过程通常转换密码子的第一个或第二个核苷酸,从而转换编码的氨基酸。在mrna上没有发现转换位点的系统模式,并且转换位点很少编码位于蛋白质活性位点的残基。RNA编辑在植物细胞器中的作用和起源尚不清楚。
结果
在这里,我们研究了由编辑密码子编码的氨基酸残基与蛋白质中这些残基的结构特征之间的关系,例如蛋白质-蛋白质界面、二级结构元素或蛋白质结构核心。我们发现由编辑密码子编码的残基明显偏向于参与螺旋和蛋白质结构核。RNA编辑可以将亲水氨基酸的密码子转化为疏水氨基酸。因此,只有经过编辑的mRNA形式才能被翻译成在适当位置具有螺旋偏好和核形成残基的多肽,这通常是蛋白质形成功能三维(3D)结构所必需的。
结论
我们对植物细胞器中受RNA编辑影响的蛋白质残基的位置进行了一项新的分析。这项研究证明,RNA编辑位点通常存在于对3D结构形成很重要的位置。如果没有RNA编辑,蛋白质折叠将无法正常发生,从而影响基因表达。我们认为,RNA编辑可能具有进化优势,它可以作为一种机制,通过允许DNA中GC含量的增加,同时保持蛋白质折叠和活性所需的编码残基所必需的RNA密码子,从而降低DNA损伤的易感性。
背景
RNA编辑是转录后在RNA中插入、删除和转换核苷酸的过程,与RNA剪接和3'加工不同[1,2].RNA编辑的插入/缺失类型首次在原生动物着丝体mrna中被发现[3.],而这种RNA编辑的转换类型首次在编码载脂蛋白B的哺乳动物mRNA (飞机观测) [4,5],随后在植物线粒体mRNA中发现coxII[6]和叶绿体mRNArpl2[7].从那时起,转换编辑主要被发现在陆地植物线粒体和叶绿体基因组转录的mrna中[8- - - - - -12].在线粒体中拟南芥时,mRNAs中至少有441个核苷酸受到RNA编辑[13].在黑松的叶绿体中,至少有26个位点被编辑[14];在角苔的叶绿体中Anthoceros formosae,已鉴定出942个RNA编辑位点[11].植物细胞器中的大多数转换编辑事件发生在mrna的蛋白质编码区域内,包括胞苷到尿苷(C-U)的转换,有时也包括尿苷到胞苷(U-C)的转换[2].早期分析[15]的研究没有发现任何规则来解释为什么蛋白质序列中的特定残基会受到密码子变化的影响,而其他残基则不受影响。
RNA编辑通常会增加编码氨基酸序列与同源序列的一致性百分比,这意味着RNA编辑在编码蛋白质功能中起着重要作用。在玉米叶绿体中rpl2时,AUG起始密码子由ACG [16].在细胞色素c氧化酶亚基II,由线粒体DNA编码玉米,铜配体残基的密码子由Thr的密码子转化为Met的密码子;在这个位点上需要Met来结合铜离子,而铜离子又是电子转移的先决条件,这是细胞色素的生物学功能c氧化酶(17].在小麦线粒体中ORF240,相当于细胞色素c生物合成蛋白,RNA编辑将其中一个血红素结合残基的密码子转换为编码适合血红素相互作用的氨基酸[18].未经审查的psbF菠菜叶绿体的mRNA导致光系统ii缺陷表型[19];未经审查的petB烟草叶绿体的mRNA导致血红素与细胞色素的附着缺陷b6[20.];未编辑的乙酰辅酶a羧化酶羧转移酶β没有功能[21].这些例子都是证明RNA编辑功能重要性的例外情况;然而,对于细胞器中的大多数RNA编辑事件,其功能的重要性尚未明确阐明。
基因组测序和结构基因组学项目已经产生了大量的数据,包括RNA编辑位点、细胞器基因组序列和蛋白质3D结构。在本报告中,我们结合这些数据并计算研究了RNA编辑功能角色的影响。我们通过蛋白质3D结构定义蛋白质功能,并发现RNA编辑转换的残基对结构重要位点有显著的偏向。
结果
DNA序列数据库中的转换型RNA编辑
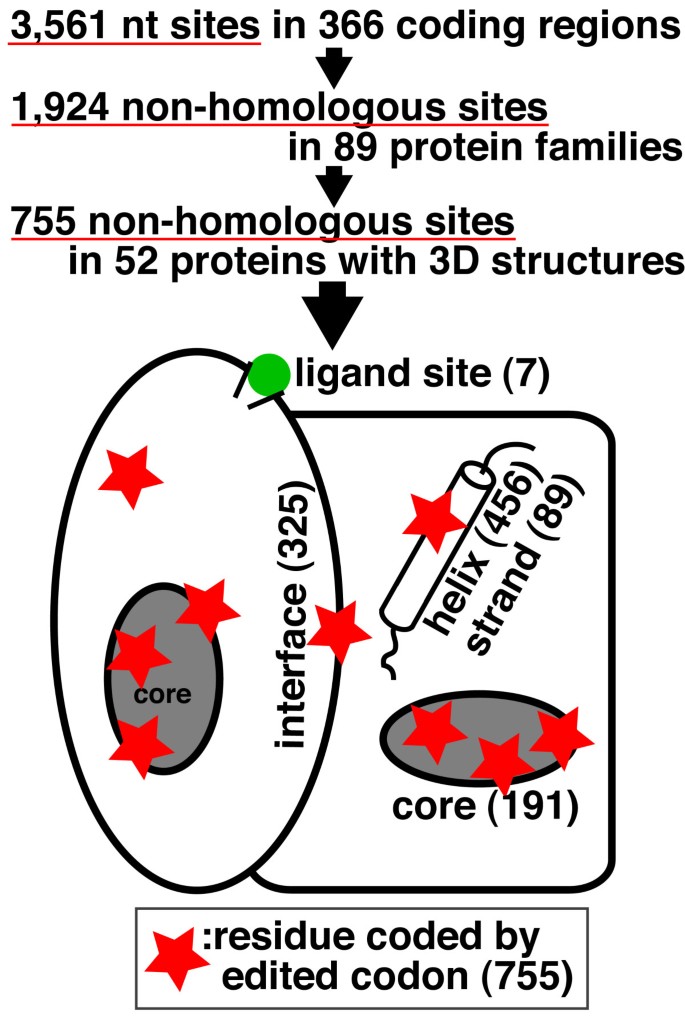
在Genbank发布的158中,植物细胞器中有365个基因在其蛋白编码区域内的3560个核苷酸上进行转换编辑(表2)1).其中,1219个RNA编辑事件发生在密码子的第一个核苷酸上,1983个事件发生在第二个核苷酸上,358个事件发生在第三个核苷酸上。密码子中被编辑的核苷酸的数量不限于一个。在3560个事件中,有129对RNA编辑事件针对同一个基因中的密码子。密码子的第1、2个核苷酸被编辑的有86个,第2、3个核苷酸被编辑的有33个,第1、3个核苷酸被编辑的有10个。
通过序列识别对365个基因产物进行分类,结果得到88个蛋白质家族(附加文件)1而且2).有1923个独特的RNA编辑事件;其中大多数是C-U转换(表2).在表2,有13个事件涉及其他类型的转换;所有这些事件都在编码线粒体细胞色素的mRNA中观察到b6从Pfiesterra piscicida[22].
这些数据表明,约90%(=(1219 + 1983)/3560)的RNA编辑事件发生在密码子的第一个或第二个核苷酸上;这一观察表明,RNA编辑事件经常改变编码氨基酸的身份。表格3.显示了1923个RNA编辑事件中氨基酸残基的转换模式。前五种模式是Ser-Leu转换(333),其次是Pro-Leu (325), Ser-Phe (248), Pro-Ser(101)和Arg-Trp(83)。这些转换主要恢复进化上保守的氨基酸残基,在同源蛋白的多序列比对中发现(图。1).转换模式见表2而且3.与以往研究中报道的结果相似[16,23,24].
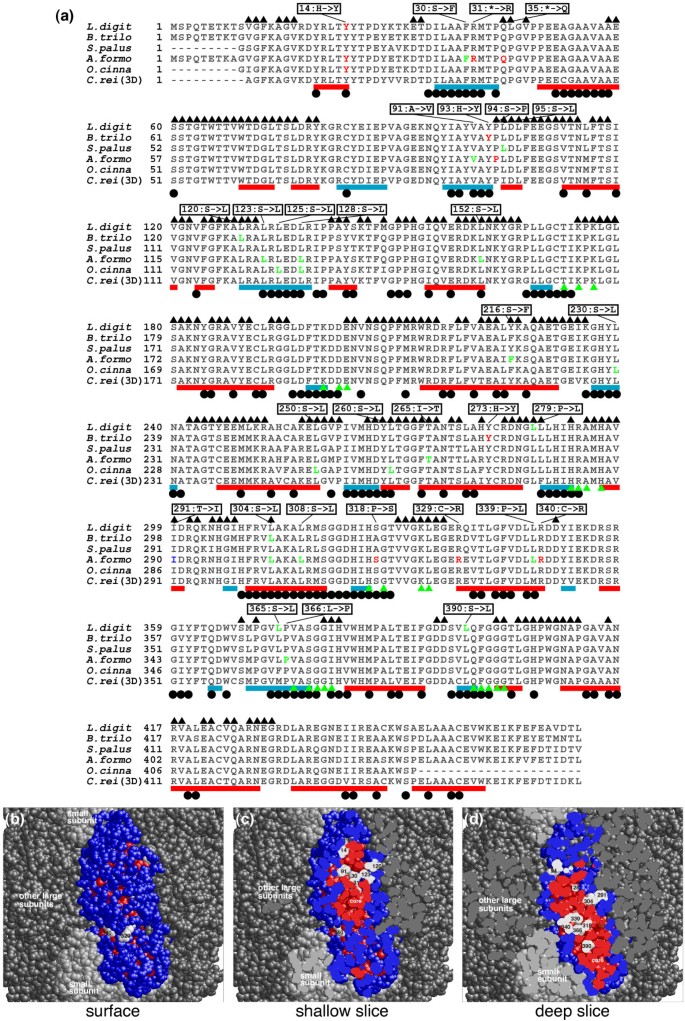
RuBisCO大亚基中RNA编辑转化的氨基酸残基。(一)来自叶绿体基因RuBisCO大亚基的多序列比对石松属植物digitatum(l .数字),Bazzania trilobata(b . trilo),泥炭藓palustre(美国杭),Anthoceros formosae(答:formo),Osmunda菜var。fokiensis(o . cinna),衣藻reinhardtii(c·丽(3 d))。关于RNA编辑位点的数据收集自[10].氨基酸序列c . reinhardtiiRuBisCO与其他序列进行比对,确定蛋白质数据库(ID: 1UW9[26])。经RNA编辑转换的氨基酸残基的颜色如下:红色表示密码子的第一个核苷酸被编辑,绿色表示第二个,青色表示第二个和第三个。每个对齐行下面的红框表示螺旋结构中的残留物;蓝色方框表示链结构中的残留物。一行下方的黑点表示结构核心中的残留物;行上的三角形表示小的子单元或其他大的子单元在界面上的残留。绿色三角形表示中间类似物(2-羧arabin醇-1,5-二磷酸)的结合位点。方框中描述了RNA编辑对氨基酸残基的转化模式。(B)RuBisCO大亚基超分子形式的三维结构。中间有颜色的分子为焦点大亚基,浅灰色分子为RuBisCO小亚基,深灰色分子为RuBisCO大亚基。在中间的分子上,红色的残基形成了结构核心,白色的残基是经过RNA编辑转换的残基。白渣上的数字与(A)中的数字对应。(C)(B)的横截面来描绘构造核心。切片平面与图页平行。(D)(B)的横切面,切片平面平行于图页,深于(C)。
蛋白质的3D结构编辑转录本
在88个mrna进行转换编辑的蛋白质家族中,52个家族成员的3D结构数据已存入蛋白质数据库[25];因此,它们的三维结构可以被建模(表1).在上面描述的1923个RNA编辑位点中,755个位点编码了这些3D结构中的残基。
功能残基与RNA编辑位点的相关性
我们根据这52个蛋白质的三维结构分配了功能残基,并整理了功能残基和编辑密码子编码的残基列表。图中总结了编辑后的站点和功能站点之间的对应关系2.
配体结合残留物
我们比较了RuBisCO中配体结合残基和RNA编辑转化残基,其中配体类似物的结合位点是已知的[26],并发现配体结合位点上的残基没有被RNA编辑转化(图2)。1).在755个RNA编辑位点中,只有7个位点位于配体结合位点(附加文件)1).首先,通过编辑密码子编码乙酰辅酶a羧化酶羧转移酶β中的锌配体残基。在乙酰辅酶a羧化酶中,由叶绿体编码的羧转移酶βaccD基因的铁线蕨capillus-veneris,将CGC密码子编码Arg73转换为UGC编码Cys。Cys是锌指基序的一部分,质体中基序的缺失accD丧失的酶活性[27].第二,细胞色素中的cu配体残基c氧化酶I和II由经过RNA编辑的密码子编码。在线粒体中coxI基因的冷杉云杉,落叶松sp。Megacerossp.和苏铁科植物sp., CAU密码子编码His,转换为UAU编码Tyr;这种Tyr残基连接了从血红素到Cu的电子转移途径。将Tyr替换为Phe重新定位了Cu,这表明Tyr在将Cu定位到适当的电子路径中发挥了作用[28,29].在许多线粒体中coxII基因中,ACG编码Thr被编辑为AUG编码Met;Met残基是Cu的配体。铜离子coxII也在电子转移路径上。当这种残基突变为苏氨酸时,CoxII几乎完全失去了与铜结合的能力[30.].第三,通过RNA编辑靶向NADH脱氢酶亚基I中的Fe-S配体残基。CGC密码子编码Arg70转换为UGC编码Cys inndhI叶绿体的信使rna答:capillus-veneris.与Cys残基结合的Fe-S团簇是电子转移途径的一部分[31].最后,F1 atp酶α中的Mg-ATP结合残基被编辑的密码子编码。在线粒体基因中编码Pro的CCU烟草转换为UCU编码Ser。而残基在三维结构确定的序列中对Pro进行编码[32],因此编辑似乎对ATP的结合没有影响。
二级结构残留物
许多密码子被RNA编辑转换的残基被发现是蛋白质二级结构的一部分,特别是螺旋结构(附加文件)1).在755个RNA编辑位点中,456个(约60%)位点被发现位于α-或3位点10-螺旋结构和89位于β-片结构。RNA编辑位点在螺旋上的偏斜分布具有统计学意义(p< 8.38 × 109).在目前的数据中,有131例经RNA编辑转换的残基呈螺旋状,但最初在DNA中编码为Pro。在这些情况下,一个未编辑的氨基酸序列会有一个扭曲或截断的螺旋,这将导致蛋白质3D结构的缺陷(因为Pro是一个螺旋破坏者[33]),可能会产生较不稳定的蛋白质。因此,编辑通过将残基恢复到适合二级结构形成的残基来修复蛋白质的似是而非的缺陷。
亚单位界面残馀
在当前的研究中,我们指定的3D结构的所有蛋白质都是多亚基复合体;稳定的亚基相互作用是这些蛋白质发挥作用的重要因素。RuBisCO由一个具有整体桶状结构的异源二聚体(大亚基和小亚基)的八聚体组成。每个大的子单元都与两个小的子单元和另外两个大的子单元有接口。界面残留物如图所示。1通过黑色三角形。在RNA编辑转化的密码子编码的30个唯一残基中,有13个残基位于界面上,其中大多数残基由亲水性残基转化为疏水性残基(图2)。1).在52个蛋白质家族中的755个RNA编辑位点中,325个位点位于亚基的界面(附加文件)1).在亚单位界面中,Ser转化为亮氨酸、Ser转化为苯、Arg转化为色氨酸的次数分别为64次、57次和23次。这些转化都将残基的物理化学性质从亲水转变为疏水。这种类型的转换增加了界面残基的疏水性,这是蛋白质-蛋白质界面的重要性质之一[34].
蛋白质结构核心
构建蛋白质三维结构的关键步骤是结构核心的形成。RuBisCO大亚基在结构核中有139个(~30%)残基(图2)。1),通过RNA编辑转换的残基偏向于形成核心的残基。在无花果。1罪犯,经RNA编辑转化的残基以白色显示,这些白色残基聚集在红色的核形成残基内。在RuBisCO中30个独特的RNA编辑位点中,有18个残基(60%)位于结构核中,考虑到只有30%的残基位于结构核中,这些残基在蛋白质结构核中被过度代表。的残余Anthoceros formosaeRuBisCO对应的残基为339衣藻reinhardtiiRuBisCO (3D已知)最初编码为Pro,密码子的RNA编辑将编码的残基转化为Leu(图。1).残留物是结构核心的一部分,位于α螺旋上。如果残基没有被RNA编辑转化,那么Pro会破坏α螺旋和结构核心,改变蛋白质的局部体积,即使所得到的肽能够折叠(图2)。1 d).
在52种蛋白质的755个RNA编辑位点中,有648个RNA编辑位点导致了残基转化。其他107个(= 755-648)RNA编辑位点要么参与停止密码子,要么参与不转化氨基酸残基的RNA编辑。在残基转化的648个RNA编辑位点中,191个(~30%)靶向于蛋白质结构核心中的残基。在52个具有3D结构的蛋白质中,有12370个残基,其中2331个残基(约19%)包含在蛋白质结构核中。因此,得到观测分布的概率小于4.54 × 10-10.然而,由于蛋白质结构核心中的残基往往是疏水的,RNA编辑经常将残基转化为疏水的残基(表2)3.),这可能是RNA编辑位点倾向于位于蛋白质结构核心的自然结果。因此我们只对所有序列上52个蛋白质的Leu和Phe残基进行显著性检验,发现概率为7.81 × 105,仍然足以支持RNA编辑位点选择性地位于蛋白质结构核内残基的密码子的假设。
讨论
我们从核苷酸序列数据库中收集了蛋白质编码区域内已知的RNA编辑位点,并将RNA编辑位点的位置与这些蛋白质中功能和结构上的重要位点进行了比较。在以往的研究中,通过RNA编辑转化的残基在氨基酸序列中被认为是无规则分布的[15],只有少数RNA编辑被发现靶向蛋白质的活性位点[16].在这项研究中,我们证明了由编辑密码子编码的残基偏向于螺旋、蛋白质-蛋白质界面和蛋白质结构核心。螺旋和蛋白质结构核的倾斜在统计上特别显著。这些分布表明,RNA编辑位点由于某种生物学原因位于螺旋和蛋白核心。
Mulligan等人最近的研究[35]用一种复杂的统计方法证明,被编辑的密码子经常在核苷酸序列中分组,这些组被不包含编辑位点的长间隙隔开。我们在这里的分析可能会给他们的非随机分布的编辑网站的结构背景。沿着氨基酸序列的编辑密码子组可能对应于蛋白质结构核心内的一簇残基,编辑密码子组之间的长间隙可能对应于不形成蛋白质结构核心的区域,即表面。
蛋白质3D结构对RNA编辑功能的影响
众所周知,螺旋的突变对蛋白质的稳定性有重大影响。在T4溶菌酶中,将Pro引入螺旋结构导致显著的扭结结构,稳定性降低约2.5 kcal/mol [36].考虑到蛋白质的整体热力学稳定性是由~ 5-15 kcal/mol的自由能差来实现的[37],这种缩氨酸折叠态和未折叠态自由能差的减小很容易对蛋白质的稳定性产生影响。在细胞色素螺旋的RNA编辑位点人工反向突变Leu到Prob6进行;该研究的作者发现,突变的蛋白质不能形成蛋白质复合物,显然是因为细胞色素b6本身没有适当折叠[20.].因此,原始DNA序列的直接翻译似乎通过蛋白质稳定性和/或第四纪结构的形成对生物功能产生影响;RNA编辑恢复残基,使蛋白质稳定和功能。
蛋白质结构核心的突变也会对蛋白质稳定性产生重大影响。RNA编辑修复的典型转化之一,核心中Leu突变为Ser,相当于在残基侧链上损失一个甲基,引入一个羟基;因此,蛋白质结构核心中可能会形成亲水空腔。实验测量了蛋白质中空腔形成的影响;降低稳定性~3.3 kcal/mol [38].核糖核酸酶T1核心的Val转变为Ser使蛋白质的稳定性降低了4.7 kcal/mol [39].因此,在未编辑的多肽中,蛋白质结构核心中的两到三个空腔可能对蛋白质稳定性有显著影响。Sakaki等。[21]在残基267处表达了分别含有Leu和Ser的豌豆编辑过的和未编辑过的乙酰辅酶a羧转移酶β,并测定了该蛋白在α亚基复合物中的功能。除了在未编辑的配合物中检测到无活性外,作者还发现与编辑的配合物相比,未编辑的配合物的溶解度较低。洗脱液的冻结和解冻会影响未编辑的复合物,导致不可溶的复合物,而扰动不影响编辑的复合物。根据我们的三维结构分析,残基267位于一个蛋白质结构核中;因此,该实验直接表明了结构核中的RNA编辑控制着蛋白质的稳定性。在线粒体中z梅斯时,编码核糖体蛋白S12的未经编辑的mRNA被翻译成一种不能并入核糖体的多肽[40].来自未编辑mRNA的产物具有亲水残基,而不是蛋白质核心形成所需的疏水残基,因此不会形成足够稳定的结构来参与超分子的形成。Islas-Osuna等人。[41]表明细胞色素上的几个RNA编辑位点b从葡萄藤中分离出来的蛋白要么位于蛋白结构核心,要么位于与其他亚基的界面上。他们对一种特定蛋白质的分析与我们的全部数据分析一致。
通过RNA编辑,螺旋和结构核心内氨基酸残基的转换恢复了有助于形成稳定3D结构的残基。未经编辑的产物通常不稳定或不能折叠,并可能扰乱蛋白质所涉及的细胞器中的蛋白质网络。一种转录后切换蛋白质核心内氨基酸的机制可以用来调节蛋白质的功能,而不调节蛋白质基因的转录。此外,细胞中经过编辑和未经编辑的产物的混合物可以产生多种具有不同稳定性的蛋白质,这些蛋白质都来自单一基因。如果可以通过调控核基因组中编码的RNA编辑酶来修改细胞中被编辑产物的比例[42,43],那么涉及编辑产物的生物功能的效率可以在转录后控制。
RNA编辑进化的启示
在线粒体和叶绿体中观察到的转换编辑之间的相似性表明,这两种机制起源于共同的来源[16].结合下面总结的先前研究中的讨论,我们提出了一种不同于先前提出的植物细胞器中RNA编辑起源的进化方案[44,45].
有三组观察结果可能与细胞器中RNA编辑机制的进化讨论有关。首先,人们早就注意到,RNA编辑的目标C后面经常跟着一个嘧啶核苷酸,通常是U [16].我们统计了在我们的数据集中DNA上编辑C之前和之后的每个核苷酸的数量,发现在前面的位置,T远远超过了其他类型的核苷酸(表4).T的这种偏倚表明,RNA编辑恢复了DNA上表达不足的TT二核苷酸序列。第二,已知植物线粒体基因组序列富含GC,线粒体基因组中RNA编辑位点的数量与其G+C含量呈正相关[46].RNA编辑被认为是一种补偿从T到C遗传漂变的机制[46],即抵消GC压力。气相色谱压力的原因可能不同,但建议的物理原因之一是避免嘧啶二聚体的形成[47].第三,细胞器中的RNA编辑几乎只在陆地植物中发现。Yoshinaga等人提出,已经获得了叶绿体中的RNA编辑,以影响植物的陆地适应[11].当植物开始向陆地迁移时(大约4亿年前),臭氧层还没有保护土地免受紫外线的照射[48,49].水生植物因水而免受紫外线的有害影响(即在DNA上形成嘧啶二聚体)[50,陆地植物需要一种新的方法来保护自己。
上述研究推测,在植物细胞器中引入RNA编辑是对抗嘧啶二聚体形成的积极选择对策。在这种情况下,RNA编辑应该被引入几乎所有的TT位点。由于臭氧层的形成,紫外线减少后,蛋白质核心的RNA编辑位点仍然存在,而对蛋白质结构形成不是必需的RNA编辑位点,即位于表面的RNA编辑位点可以以中性的方式消失。如果直接翻译编码残基对蛋白质3D结构稳定性的影响很小,那么在该位点上的RNA编辑可能会消失。通过RNA编辑转换的残基与其在蛋白质结构中的位置之间的相关性可能是这一过程的结果。
可以考虑RNA编辑技术出现的另一种情况。线粒体和叶绿体分别是氧气燃烧和氧气生产的主要部位。分子氧可以改变DNA碱基的化学结构。为了减轻氧分子对细胞器dna的影响,可以通过基因转移到细胞核来减少细胞器基因组中dna的数量,并通过RNA编辑来补偿细胞器dna中的突变。在动物线粒体中,已经观察到基因转移到核基因组。在植物细胞器中,已经观察到dna上的基因转移和T向C的改变。DNA上的T - C转换会减少AT碱基对的数量,而AT比GC碱基对更容易受到氧分子的影响[51].
结论
我们发现由编辑密码子编码的残基与负责二级结构和蛋白质结构核心形成的残基之间存在统计学上的显著相关性。这种相关性表明,RNA编辑通过调节蛋白质稳定性间接影响蛋白质功能,有时也对蛋白质酶活性至关重要。RNA编辑机制可以通过修复mRNA的核苷酸序列来编码稳定的蛋白质,从而调控植物细胞器中蛋白质功能的表达。
方法
收集用于转换RNA编辑的数据
Genbank/EMBL/DDBJ中RNA编辑位点描述[52- - - - - -54]并不是标准化的,并且有许多努力(包括这里的一个)来启动一个RNA编辑站点的数据库[55,56].为了识别RNA编辑位点,我们进行了全文搜索,在Genbank数据库release 158的“misc_feature”行“/note”中找到了一串同时匹配“RNA”和“editing”的字符。我们编写了一个计算机程序来提取蛋白质编码区域的RNA编辑条目,并将编辑和未编辑的mrna翻译成氨基酸序列。在此过程中,我们在Genbank注释中遇到了大量的错误,并根据文献或与储户的交流对这些注释进行了更正。大多数错误表现为misc_feature线中描述的核苷酸位置数与沉积的核苷酸序列中的RNA编辑位点之间的差异。对AB254134、AJ006146、AY820131、AY521591、BA000029、DQ645537、DQ984517、X69720、X92735、Y17812进行修复。我们无法纠正所有的错误,因为我们无法联系到所有的存款人。有明显错误的表项被丢弃。用于RNA编辑:转录本中,我们将RNA编辑位点描述从参考文献[10];D14882、D43696、L11055、L11056、L13485。当我们遇到一对具有相同氨基酸序列和mrna中相同RNA编辑模式的蛋白质序列时,我们从数据集中删除了其中一个条目。
RNA编辑转录物的蛋白质3D结构
我们对编辑mrna预测的氨基酸序列与PDB中已知3D结构的蛋白质氨基酸序列进行了同源性搜索[25]使用BLAST [57].当序列身份为25%或更多时,我们使用PDB中的3D结构来分配编辑和未编辑mrna编码的产物的结构属性。
蛋白质上的功能残基
基于文献和蛋白质三维结构,我们收集了四种类型的蛋白质功能。(1)配体结合残基:大多数蛋白质的三维结构是通过配体和辅因子确定的。结合这些配体的残基是根据测量残基与配体的溶剂可及性来确定的。当有配体和没有配体计算的残基的溶剂可及性之间的差异不消失时,则该残基被指定为配体结合残基。溶剂可达性计算采用Sharke和Rupley的改进方法[58],水半径为1.4Å。(2)蛋白-蛋白界面:经编辑的mrna蛋白产物大多为超分子组分,其三维结构以蛋白复合物形式确定。与其他亚基相互作用的残基通过测量残基与其他亚基的溶剂可及性来确定。(3)二级结构:我们使用DSSP分配二级结构[59].(4)蛋白质结构核:结构核按以下步骤确定;i)计算溶剂可达性,找到可达性为零的残基,并计算这些残基之间的所有碳原子距离;如果距离不超过4.0Å,则这对残基是蛋白质结构核心的组成部分。ii)计算溶剂可达性,找出可达性大于0但不大于0.05的残基,计算残基中一个碳原子与i中一个碳原子的距离;如果距离为4.0Å或更短,则该残基是蛋白质结构核心的一部分。
参考文献
- 1.
基冈LP,加洛A,奥康奈尔MA: RNA编辑器的许多角色。植物学报,2001,2(11):869-878。
- 2.
Gott JM, Emeson RB: RNA编辑的功能和机制。科学通报,2000,34:499-531。
- 3.
Benne R, Burg van den J, Brakenhoff JP, Sloof P, van Boom JH, Tromp MC:锥虫线粒体中框架转移coxII基因的主要转录本包含4个不在DNA中编码的核苷酸。细胞学报,1986,46:819-826。
- 4.
Powell LM, Wallis SC, Pease RJ, Edwards YH, Knott TJ, Scott J:一种新型的组织特异性RNA加工在肠道中产生载脂蛋白b48。细胞,1987,50:831-840。
- 5.
陈胜,Habib G,杨春,顾志文,Lee BR,翁萨,Silberman SR,蔡世杰,Deslypere JP, Rosseneu M, Gotto AM,李文华,Chan L:载脂蛋白B-48是一种具有器官特异性框架内终止密码子的信使RNA的产物。科学通报,1987,38(3):363-366。
- 6.
Covello PS, Gray MW:植物线粒体RNA编辑。自然科学学报,1998,29(3):366 - 366。
- 7.
Hoch B, Maier RM, Appel K, Igloi GL, Kössel H:通过创建启动密码子编辑叶绿体mRNA。自然学报,1997,33(3):388 - 388。
- 8.
Hiesel R, Combettes B, Brennicke A:除苔藓植物外,所有主要陆地植物组的线粒体RNA编辑的证据。美国国家科学研究院。1994, 29(4): 529 - 529。
- 9.
林s,张华,Spencer DF, Norman JE, Gray MW:鞭毛藻线粒体mrna的广泛编辑。中国生物医学工程学报,2002,23(3):727-739。
- 10.
Freyer R, Kiefer-Meyer M-C, Kössel H:陆地植物各主要谱系中质体RNA编辑的发生。美国国家科学研究院。1997, 44(3): 489 - 497。
- 11.
Yoshinaga K, Iinuma H, Masuzawa T, Ueda K:角草叶绿体rbcL转录本中除了C到U的替换外,U到C的大量RNA编辑和绿色植物RNA编辑的起源。中国生物医学工程学报,1996,29(4):344 - 344。
- 12.
Kugita M, Yamamoto Y, Fujikawa T, Matsumoto T, Yoshinaga K:角苔叶绿体中的RNA编辑使一半以上的基因具有功能。中国生物医学工程学报,2003,29(4):344 - 344。
- 13.
Giege P, Brennicke A:拟南芥线粒体RNA编辑对orf 441 C到U变化的影响。美国国家科学研究院。1999, 26(3): 344 - 344。
- 14.
Wakasugi T, Hirose T, Horihata M, Tsudzuki T, Kössel H, Sugiura M:在黑松叶绿体RNA水平上创建一个新的蛋白质编码区域:裸子植物叶绿体的RNA编辑模式与被子植物不同。美国国家科学研究院。1996, 93: 8766-8770。
- 15.
葛雷MW, Covello PS:植物线粒体和叶绿体的RNA编辑。王志强,1997,7:64-71。
- 16.
Bock R:无意义的意义:叶绿体的遗传信息如何被RNA编辑改变。生物化学学报,2000,32(3):549-557。
- 17.
Covello PS, Gray MW:细胞色素c氧化酶中cua结合位点的RNA序列和性质。2月。1990, 26(2): 1 -7。
- 18.
Bonnard G, Grienenberger JM:一个被提出编码ABC转运体跨膜结构域的基因在小麦线粒体中表达。中国生物医学工程学报,2004,26(3):344 - 344。
- 19.
Bock R, Kössel H, Maliga P:烟草质体基因组引入异源编辑位点:缺乏RNA编辑导致突变表型。中国科学院学报,2004,26(4):344 - 344。
- 20.
杨晓明,杨晓明,杨晓明,Kössel杨晓明:细胞色素突变研究进展b6在衣藻reinhardtii揭示了玉米和烟草中petB编辑对脯氨酸向亮氨酸转化的功能意义。植物分子生物学,1997,33:79-86。
- 21.
Sasaki Y, Kozaki A, Ohmori A, Iguchi H, Nagano Y:植物中功能性乙酰辅酶A羧化酶所需的叶绿体RNA编辑。中国生物医学工程学报,2001,26(3):349 - 349。
- 22.
张华,林生:线粒体细胞色素b鞭毛藻mRNA编辑:可能的生态和进化关联。中国生物医学工程学报,2005,26(6):538-545。
- 23.
Maier RM, Zeltz P, Kössel H, Bonnard G, Gualberto JM, Grienenberger JM:植物线粒体和叶绿体的RNA编辑。植物分子生物学杂志,1996,32:343-365。
- 24.
Brennicke A, Marchfelder A, Binder S: RNA编辑。中国生物医学工程学报,1999,26(3):329 - 329。
- 25.
Berman HM, Henrick K, Nakamura H:宣布全球蛋白质数据库。中国生物工程学报,2003,10 (12):980-
- 26.
Karkehabadi S, Taylor TC, Spreitzer RJ, Andersson I:催化受损核酮糖-1,5-二磷酸羧化酶/加氧酶晶体结构亚基间相互作用的改变。生物化学,2005,44:113-120。
- 27.
Kozaki A, Mayumi K, Sasaki Y:豌豆乙酰辅酶A羧化酶核编码和叶绿体编码亚基之间的硫醇-二硫化物交换。中国生物医学工程学报,2001,27(3):397 - 397。
- 28.
Tsukihara T,青山H, Yamashita E, Tomizaki T, Yamaguchi H, Shinzawa-Itoh K, Nakashima R, Yaono R, Yoshikawa S:牛心脏细胞色素c氧化酶氧化金属位点的结构2.8Å。科学通报,1995,26(3):349 - 349。
- 29.
Trumpower BL, Gennis RB:线粒体和细菌呼吸中细胞色素复合物的能量转导:耦合电子转移反应到跨膜质子易位的酶学。生物化学学报,1994,29(3):344 - 344。
- 30.
Kelly M, Lappalainen P, Talbo G, Haltia T, Oost van der J, Saraste M:两个半胱氨酸,两个组氨酸和一个蛋氨酸是一个双核紫铜中心的配体。中国生物医学工程学报,2003,26(4):457 - 457。
- 31.
Sazanov LA, Burrows PA, Nixon PJ:质体ndh基因编码nadh特异性脱氢酶:从豌豆类囊体膜中分离复杂I类似物。美国国家科学研究院。1998, 95: 1319-1324。
- 32.
鲍勒MW,蒙哥马利MG,莱斯利AGW,沃克JE:叠氮化物如何抑制ATP水解f -ATP酶。美国国家科学研究院。2006, 30(4): 344 - 344。
- 33.
Chou PY, Fasman GD:从氨基酸序列预测蛋白质的二级结构。中国生物医学工程学报,1997,47:445 - 448。
- 34.
王志强,王志强。基于神经网络的大分子识别。中国生物医学工程学报,2002,29(4):344 - 344。
- 35.
Mulligan RM, Chang KLC, Chou CC:植物线粒体基因组RNA编辑位点的计算分析揭示了相似的信息含量和零星分布的编辑位点。中华生物医学杂志,2007,24:1971-1981。
- 36.
Blaber M,张晓军,Matthews BW:氨基酸α螺旋倾向的结构基础。科学通报,1993,26(3):344 - 344。
- 37.
Creighton TE:蛋白质折叠。中国生物工程学报,2004,27(1):1-16。
- 38.
Loladze VV, Ermolenko DN, Makhatadze GI:蛋白质内部极性和非极性氨基酸残基埋藏的热力学结果。中华分子生物学杂志,2002,32(3):343-357。
- 39.
Vos SD, Backmann J, Prevost M, Steyaert J, Loris R:核糖核酸酶T1的疏水核操作。生物化学,2001,40:10140-10149。
- 40.
Phreaner CG, WIlliams MA, Mulligan RM: rps12转录本的不完全编辑导致植物线粒体中多态多肽的合成。植物科学学报,1996,8:397 - 397。
- 41.
Islas-Osuna MA, Silva-Moreno B, Caceres-Carrizosa N, Garcia-Robles JM, Sotelo-Mundo RR, yepizi - plascencia GM:葡萄线粒体细胞色素的编辑b蛋白质的mRNA和分子模型。生物化学学报,2006,29(4):531 - 531。
- 42.
Zeltz P, Hess WR, Neckermann K, Borner T, Kossel H:大麦和玉米叶绿体rpoB转录本的编辑与叶绿体翻译无关,并表现出不同的模式。中国农业科学,2004,27(3):489 - 497。
- 43.
Halter CP, Peeters NM, Hanson MR:玉米无核糖体质体的RNA编辑。王志强,2004,30(4):331-337。
- 44.
李志刚,李志刚,李志刚,李志刚。叶绿体RNA编辑的研究进展。中国生物医学工程学报,2006,23:1212 - 1221。
- 45.
Covello PS, Gray MW: RNA编辑的进化。遗传学报,1993,9:265-268。
- 46.
Malek O, Lättig K, Hiesel R, Brennicke A, Knoop V:苔藓植物RNA编辑与陆生植物分子系统发育。中国农业科学,1996,16(4):344 - 344。
- 47.
辛格CE,艾姆斯BN:阳光紫外线和细菌DNA碱基比。科学通报,1997,17(3):344 - 344。
- 48.
高桥A,大石T:利用国际空间站的暴露设施研究太阳紫外线辐射的生物效应的意义。生物科学学报,2004,18:255-260。
- 49.
Berkner LV, Marshall LC:主要大气成分的历史。美国国家科学研究院。1965, 53: 1215-1226。[http://www.pnas.org/content/53/6/1215.full.pdf+html]
- 50.
Häder DP, Sinha RP:太阳紫外线辐射诱导水生生物DNA损伤:潜在的环境影响。生物工程学报,2005,29(1):1- 3。
- 51.
Lindahl T: DNA一级结构的不稳定性和衰变。自然科学学报,1993,29(3):339 - 344。
- 52.
Benson DA, Karsch-Mizrachi I, Lipman DJ, Ostell J, Wheeler DL: GenBank。核科学通报,2007,35:D21-D25。
- 53.
Kulikova T, Akhtar R, Aldebert P, Althorpe N, Andersson M, Baldwin A, Bates K, Bhattacharyya S, Bower L, Browne P, Castro M, Cochrane G, Duggan K, Eberhardt R, Faruque N, Hoad G, Kanz C, Lee C, Leinonen R, Lin Q, Lombard V, Lopez R, Lorenc D, McWilliam H, Mukherjee G, Nardone F, Pastor MPG, Plaister S, Sobhany S, Stoehr P, Vaughan R, Wu D, Zhu W, Apweiler R: 2006 EMBL核苷酸序列数据库。核科学通报,2007,35:D16-D20。
- 54.
Sugawara H, Abe T, Gojobori T, Tateno Y: DDBJ致力于INSDC中细菌基因的评估和分类。核科学通报,2007,35:D13-D15。
- 55.
何涛,杜鹏,李勇:基于网络的RNA编辑位点注释数据库。核工业学报,2007,35:D141-144。
- 56.
Picardi E, Regina TMR, Brennicke A, Quagliariello C: REDIdb: RNA编辑数据库。中国环境科学,2007,35:D173-D177。
- 57.
Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ: gapping BLAST和ppi -BLAST:新一代蛋白质数据库搜索程序。核化学学报,1997,25:3389-3402。
- 58.
摇动A, Rupley JA:环境与蛋白质原子的溶剂暴露。溶菌酶和胰岛素。中华生物医学杂志,2003,19(3):351-371。
- 59.
Kabsch W, Sander C:蛋白质二级结构词典:氢键的模式识别和几何特征。生物聚合物。1983,22:2577-2637。
确认
KY感谢Kazuko Kaji女士和Atsuko Doi女士从文献中收集RNA编辑位点的描述,并感谢Kazuhiro Kobayashi先生和Masataka Oikawa先生协助数据分析。KY和MG由日本科学促进协会(JSPS) KAKENHI(18370061)的科学研究资助(B)资助。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
KY建立数据库,计算相关性,并起草稿件。MG发起并监督了这项研究。所有作者都阅读并批准了最终的手稿。
电子辅助材料
12870 _2008_287_moesm1_esm.xls
附加文件1:补充表1。已知3D结构的52个蛋白质中RNA编辑转换的残基列表。该表还包含二级结构、溶剂可及性和基因来源的物种名称。(xls 174kb)
12870 _2008_287_moesm2_esm.xls
附加文件2:补充表2。在37个没有已知3D结构的蛋白质中,RNA编辑转换的残基列表。(xl194kb)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
李志强,李志强,李志强。植物细胞器蛋白质三维结构中氨基酸残基与功能残基的相关性研究。BMC植物生物学8,79(2008)。https://doi.org/10.1186/1471-2229-8-79
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-8-79
关键字
- 溶剂可及性
- 编码氨基酸
- 功能性残留
- 电子传递途径
- 植物细胞器